Method Article
Turnusol Testi kullanarak Bakterilerin Kolorimetrik Tespiti
Bu Makalede
Özet
We describe a protocol for colorimetric detection of E. coli using a modified litmus test that takes advantage of an RNA-cleaving DNAzyme, urease, and magnetic beads.
Özet
There are increasing demands for simple but still effective methods that can be used to detect specific pathogens for point-of-care or field applications. Such methods need to be user-friendly and produce reliable results that can be easily interpreted by both specialists and non-professionals. The litmus test for pH is simple, quick, and effective as it reports the pH of a test sample via a simple color change. We have developed an approach to take advantage of the litmus test for bacterial detection. The method exploits a bacterium-specific RNA-cleaving DNAzyme to achieve two functions: recognizing a bacterium of interest and providing a mechanism to control the activity of urease. Through the use of magnetic beads immobilized with a DNAzyme-urease conjugate, the presence of bacteria in a test sample is relayed to the release of urease from beads to solution. The released urease is transferred to a test solution to hydrolyze urea into ammonia, resulting in an increase of pH that can be visualized using the classic litmus test.
Giriş
Bacterial pathogens are one of the major causes of global morbidity and mortality. Outbreaks from hospital-acquired infections, food-borne pathogens, and bacterial contaminants in the environment pose serious and on-going threats to public health and safety. To prevent these outbreaks, effective tools are needed that permit pathogen detection in a timely fashion under a variety of settings. Simple but still effective tests that are portable and cost-effective are greatly coveted, especially in regions that are susceptible to outbreaks but cannot afford expensive testing facilities.1-3 Although there exists a multitude of methods to detect bacteria, many of them are not suitable as screening or on-site testing tools because they require long test times, expensive instruments and complicated testing procedures.
Colorimetric tests are particularly attractive for point-of-care or field applications as color changes can be easily detected by the naked eye. The litmus test for pH is simple, quick, and effective. Although it is a very old technology, it is still widely used today because of its simplicity and effectiveness. Surprisingly, this simple test had never been modified to achieve the detection of other analytes before we recently developed an approach of modifying this test for E. coli testing.4
The expanded litmus test for E. coli employs three additional components: an E. coli activated RNA-cleaving DNAzyme (EC1), 5 urease, and magnetic beads. DNAzymes refer to synthetic single-stranded DNA molecules with catalytic activity.6 They can be isolated from random-sequence DNA pools using in vitro selection.7,8 They are highly stable and can be produced cost-effectively using high-efficiency automated DNA synthesis.9 For these reasons, DNAzymes, particularly RNA-cleaving DNAzymes, have been widely examined for biosensing applications.6,10,11 RNA-cleaving DNAzyme sensors have been developed to detect metal ions,12-16 small molecules,17,18 bacterial pathogens5,19-21 and cancer cells.22 Given the great availability of target-induced RNA-cleaving DNAzymes, any assay that utilizes a DNAzyme can be potentially expanded to detect a diverse range of analytes.
Urease is chosen for its ability to hydrolyze urea into ammonia,23,24 resulting in a pH increase. Urease is also highly efficient, stable and amenable for conjugation to other biomolecules. Therefore, we postulated that a conjugate of an RNA-cleavage DNAzyme with urease would allow the use of litmus test for the detection of other targets.5
The action of the RNA-cleaving DNAzyme is relayed to urease-mediated increase of pH through the use of magnetic beads that are immobilized with the DNAzyme-urease conjugate. Because the activity of the DNAzyme under investigation is strictly dependent on E. coli, the presence of this bacterium in the test solution will result in the release of urease from the magnetic beads to the solution, which is then taken and used to hydrolyze urea in a reporter solution that contains a pH-sensitive dye. The final outcome of this procedure is a color change that can be conveniently reported by the dye or pH paper.
Protokol
Reaktifler ve Tamponlar hazırlanması 1.
- 0.5 M etilendiamin tetraasetik asit (EDTA),
- 2 L'lik bir beher içinde, deiyonize damıtılmış 800 ml su ile 186.1 gr EDTA ekleyin (GKD 2 O). NaOH peletleri ile 8.0 çözeltinin pH'ının ayarlanması. 1.0 L'lik son bir hacme O GKD 2 ekleyin ve 4 ° C'de otoklav ve mağaza bir otoklav cam şişe çözüm aktarın.
- 10 x Tris-borat EDTA (10x TBE)
- 4 L'lik bir plastik kabı içinde, tüm bileşenler kadar bir karıştırma çubuğu kullanılarak 4 L Karışım bir son hacme 432 g Tris-bazı, 200 g borik asit, 0.5 M EDTA (pH 8.0) ve GKD 2 O 80 ml ilave çözündürülür. 4 ° C'de otoklav ve mağaza 1 L cam şişe çözüm aktarın.
- % 10 denatüre edici poliakrilamid Hazır
- F kadar çözeltisi ve GKD 2 A: (1 29), 4 L'lik bir plastik kap olarak, 1681,7 g üre 400 ml 10x TBE, 40% akrilamid / bisakrilamid 1 L eklemeNominal hacmi üre çözünene kadar bir karıştırma çubuğu kullanılarak 4 L bir karışımıdır. 4 ° C 'de 1 L'lik amber cam şişe ve saklamak için bir çözüm aktarın.
- Yükleme Tamponu 2x Jel (2x GLB)
- 200 ml'lik bir cam kabın içinde, 44 g üre, 8 g sakroz, 10 mg bromofenol mavi, 10 mg iksilen siyanol FF,% 10 sodyum dodesil sülfat 400 ul ve 10 x TBE ve 4 ml. 40 ml'lik bir son hacme O GKD 2 ekleyin ve bir manyetik karıştırma çubuğu ile karıştırılarak 50 ° C'de, hafif ısıtma ile bileşenleri çözülür. 4 ° C'de Aktarım 1 mi, 1.5 ml mikrofüj tüplerine kısım saklayın.
Not: Ön kullanımı herhangi bir katı yeniden çözünmesi için 90 ° C'de kısa bir ısıtma gerektirir.
- 200 ml'lik bir cam kabın içinde, 44 g üre, 8 g sakroz, 10 mg bromofenol mavi, 10 mg iksilen siyanol FF,% 10 sodyum dodesil sülfat 400 ul ve 10 x TBE ve 4 ml. 40 ml'lik bir son hacme O GKD 2 ekleyin ve bir manyetik karıştırma çubuğu ile karıştırılarak 50 ° C'de, hafif ısıtma ile bileşenleri çözülür. 4 ° C'de Aktarım 1 mi, 1.5 ml mikrofüj tüplerine kısım saklayın.
- 1 M Tris-hidroklorür (Tris-HCl) (pH 7.5)
- Bir cam şişe içerisinde, Tris-bazın 12.1 g ve GKD 2 O 70 ml ilave edilir ve katı madde çözülene kadar karıştırın. 1 M hidroklorik asit (HCI) ile pH'ı 7.5'e ayarlayın. 100 mi, otoklav ve taştan bir son hacme GKD 2 O eklemere 4 ° C'de
- 5 M sodyum klorür (NaCI)
- Bir cam şişe içerisinde, GKD 2 O 150 ml NaCI 58.4 g çözülür GKD 2 O. ile 200 ml ses seviyesini ayarlamak 4 ° C'de saklayın.
- DNA, Seyreltme Tampon maddesi
- bir cam şişe içinde, 1 M Tris-HCl (pH 7.5), 8.0 5 M NaCl mi ve 0.5 M EDTA, 0.4 mi (pH 8.0) içinde 2.0 ml karıştırılır. GKD 2 O, 4 ° C'de otoklav ve mağaza ile 200 ml ses seviyesini ayarlayın.
- 1 M 4- (2-hidroksietil) -1-piperazinetansülfonik asit (HEPES) (pH 7,4)
- Bir cam şişe içerisinde, GKD 2 O 80 ml HEPES, 2.38 g çözülür 5 N NaOH kullanılarak 7.4 pH ayarlamak ve 100 ml lik bir son hacme GKD 2 O ekleyin.
- 1 M magnezyum klorür (MgCI2)
- Bir cam şişe içerisinde, MgCl2 -6H 2 O 2.03 g ekleme ve GKD 2 O ile 100 ml'ye ses getirmek
- Reaksiyon Tamponu (RB)
- 50 ml konik bir tüp içinde, 1 M HEPES (pH 7.4), 5 M NaCI, 1.5 mL, 1 M MgCl2, 0.75 mi, ve Tween-20 5 ul 50 ul ekle. 50 ml'lik bir son hacme O GKD 2 ekleyin. çözelti karıştırılır ve kullanılana kadar 4 ° C de, bir şırınga veren filtre (0.22 um) ve deposunu kullanan bir konik tüp içine filtre.
- Bağlama Tamponu (BB)
- 50 ml konik bir tüp içinde, bir son hacim için O GKD 2; 1 M Tris-HCI (pH 7.5), 500 ul, NaCI 8.8 g, MgCl2, 1 M, 50 ul, Tween 20 5 ul 50 ml. çözelti karıştırılır ve kullanılana kadar 4 ° C de, bir şırınga veren filtre (0.22 um) ve deposunu kullanan bir konik tüp içine filtre.
- Substrat solüsyonu (SS)
- 50 ml'lik konik bir tüp içinde, NaCI 5.8 g, 1 M 3 mL MgCl2, 1.5 g üre ve GKD 2 O 40 ml ilave 10 mM HCI kullanılarak 5,0 çözeltinin pH'ının ayarlanması. Çözelti tamponlu olduğundan, HCI kullanılarak pH ayarlamakdikkatle küçük HCI alikotları eklenmesi ile. 50 ml'lik bir son hacme O GKD 2 ekleyin.
- çözelti karıştırılır ve kullanılana kadar 4 ° C de, bir şırınga veren filtre (0.22 um) ve deposunu kullanan bir konik tüp içine filtre.
- Luria Bertani (LB) Broth
- Beher, GKD 2 O 1 L 20.0 g LB tozu çözmek 4 ° C'de bir cam şişe, otoklav ve saklamak için aktarın.
- % 1.5 LB agar
- 250 ml'lik bir şişe içinde, 1.5 g agar ve LB suyu 100 ml. 4 ° C'de otoklav ve saklayın.
- agar Tabaklar
- Bir mikrodalga LB agar tekrar çözündürülmesi ve çözeltiye ~ 50 ° C soğutulur. Petri kaplarına solüsyonu dökün ~ 5 plakaları yapmak ve onları katılaşmaya sağlar.
2. sentezi ve E. saflaştırma coli- duyarlı DNAzim EC1
- Şablon aracılığı Enzimatik Ligasyonu ile EC1 sentezi
- comm arındırınticari olarak uygundur, sentezlenmiş oligonükleotidler BS1, DE1 ve T1,% 10, standart protokollere göre poliakrilamid jel elektroforezi (dPAGE) denatüre edici ile (Tablo 1 'de verilen sekansları).
- BS1, DE1 ve T1 100 uM stok hazırlayın. kullanılıncaya kadar -20 ° C'de saklayın.
- 1.5 ml mikrofüj tüpüne, enzim ile ilgili olarak (500 mM Tris-HCI (pH 7.6), 100 mM MgCl2 sağladığı GKD 2 O 38.5 ul, DE1 5 ul ve 10 x T4 polinükleotid kinaz reaksiyon tamponu 5 ul 50 mM ditiotreitol (DTT), 1.0 mM spermidin).
- adenosin trifosfat (ATP) (100 mM) 1 ul ekle. T4 polinükleotid kinaz 5 birim (10 U / ul) eklenir. reaksiyon karışımına dikkatli bir pipetleme karıştırın.
- 30 dakika için 37 ° C 'de reaksiyon inkübe edin.
- 5 dakika boyunca 90 ° C'ye kadar ısıtılarak, reaksiyonun sönmesi.
- GKD 2 O 118 ul, BS1 5 ul ve T1 5 ul ekleyin. 2 mi, 90 ° C tepkime ısısın, ve 10 dakika boyunca oda sıcaklığına kadar soğutulur.
- Enzim, tedarikçi tarafından sağlanan 10 x T4 DNA ligaz reaksiyon tamponu 20 ul ekle (400 mM Tris-HCI (pH 7.8), 100 mM MgCl2, 100 mM DTT, 5 mM ATP). T4 DNA ligazı, 10 birim (5 U / ul) ilave edilir ve dikkatli bir şekilde pipetle karıştırılır.
- 2 saat oda sıcaklığında inkübe edin.
- 3 M sodyum asetat (NaOAc) (pH 5.2) 20 ul soğuk% 100 etanol 500 ul ekle. Vorteks ile çözüm karıştırın ve 30 dakika için -20 ° C dondurucu içinde bir tüp yerleştirmek.
- 4 ° C'de 20 dakika boyunca 20,000 x g'de mikrofüj santrifüj. Dikkatle pipetleme süpernatant kaldırmak.
- 4 ° C'de 10 dakika boyunca 20,000 x g hızında tekrar soğuk% 70 etanol ve santrifüj ile pelet yıkayın. Bir kez daha süpernatant kaldırmak ve 10 dakika süre ile, vakum altında pelet kurutulur.
- GKD 2 O 15 ul DNA tekrar ve daha sonra 2x GLB 15 ul ekleyin.
- 90 ° C'ye kadar kuvvetli bir şekilde karıştırın ve ıslak sertleştirilmesi için2 dak. Aşağıda tarif edildiği gibi,% 10 dPAGE tam uzunlukta DNA saflaştırılır.
- % 10 dPAGE Kurma
- Temiz iki cam levha (bir tam plaka ve bir çontuklu), iki 0.75 mm kalınlığında aralama ve 12 kuyu tarak. İki her iki tarafta spacers ve üstüne çentikli plaka ile düz bir yüzeye bir cam plaka yatıyordu. Birlikte tedarikçi tarafından sağlanan dört klipler kullanarak iki tabak Clip.
- 150 ml'lik bir plastik kabı içinde,% 10 dPAGE 40 ml, tetrametiletilendiamin 40 ul (TEMED) ve% 10 amonyum persülfat (APS), 400 ul dökün. bileşenleri karıştırın ve yavaş yavaş plakalar arasına çözüm dökün.
- Tarak yerleştirin ve sonra jel üzerinde 10 dakika polimerize izin verir. Jel polimerize edildikten sonra, yavaş yavaş tarak kaldırmak ve GKD 2 O. ile kuyu yıkayın
- jel elektroforezi aparat üzerine plakaları monte edin. aşırı ısınmayı önlemek için üretilen ısıyı dağıtmak için bir metal plaka kullanın.
- Üst ve alt bölmelere üzerine 1x TBE dökünve kuyular iyi tampon batık emin olun. Bir pipet veya şırınga kullanarak 1x TBE ile kuyu yıkayın.
- yükleme örnekleri önce 15 dakika için 35 mA'da için cihaz ve önceden çalıştırılmış ayarlayın.
- % 10 dPAGE den Bağlanmış EC1 elüsyon
- Adım 2.2.6 ardından, alt boya (Bromophenol mavi) kadar 35 mA jel çalıştırmak cam plakanın alt ulaşır. Bu, yaklaşık 1.5 saat sürer. Gücü kapatın ve cihazdan plakaları çıkarın.
- kağıt havlu birkaç yaprak tabak Lay ve dikkatle cam plakalar gelen aralayıcılar kaldırın. Dikkatli bir şekilde üst cam plaka kaldırmak ve plastik wrap ile jel sarın.
- ikinci cam plakayı kaldırın ve tekrar plastik wrap ile karşılamak için jel çevirin. plastik wrap kırışıklıkları önlemek için özen gösterin.
- 4 farklı DNA bantları (Tam bağlanan EC1, DE1, BS1 ve T1) üretecek UV gölgeleme (260 nm) kullanarak bağlandı ürünü gözünüzde canlandırın.
- Tüketim 1.5 ml mikrofuge'de tüp steril bir jilet ve transferi ile üst bant (EC1). ince bir hamur haline dönene kadar steril bir pipet (200 ul uç boyutu) ile jel Crush. DNA elüsyon tamponu 550 ul ekle gömülü fluorofor korumak için alüminyum folyo ile tüp kapağı ve 15 dakika boyunca çalkalayın.
- 5 dakika boyunca 20.000 xg'de jel çözüm Santrifüj ve dikkatli bir 1.5 ml mikrofuge'de tüp süpernatant 400 ul aktarın. pipetleme sırasında jel parçaları çekilmesi kaçının.
- transfer süpernatan ile mikrofüj tüpe 40 3 M NaOAc (pH 5.2) içinde ul soğuk% 100 etanol içinde 1.0 ml. Vorteks ile çözüm karıştırın ve 30 dakika için -20 ° C dondurucu içinde bir tüp yerleştirmek.
- 4 ° C'de 20 dakika boyunca 20,000 x g'de mikrofüj santrifüj. Dikkatle pipetleme süpernatant kaldırmak.
- 4 ° C'de 10 dakika boyunca 20,000 x g hızında tekrar soğuk% 70 etanol ve santrifüj ile pelet yıkayın. Bir kez daha, kaldırmakSüpernatan 10 dakika için vakum altında pelet kurutulur.
- 260 nm'de UV absorbansı ölçerek EC1 konsantrasyonunun belirlenmesi. 10 uM stok yapmak ve kullanılana kadar -20 ° C'de örnek saklayın.
DNA üreazın 3. Konjugasyon
- Sukinimidil 4- (N-maleimidometil) sikloheksan-1-karboksilat (SMCC), hisse senedi hazırlanması
- DMSO 676 ul 1 mg SMCC çözülür. kullanılana kadar buz üzerinde girdap ve yer.
- Üreaz Stokta hazırlanması
- 1 x PBS (Mg + 2 veya Ca + 2), 1 ml 1 mg üreaz çözülür. Buz üzerinde kullanıma kadar yerleştirin.
Not: Kristalize üreaz çözünmesi yavaştır ve yumuşak karıştırma denatürasyon önlemek için gereklidir.
- 1 x PBS (Mg + 2 veya Ca + 2), 1 ml 1 mg üreaz çözülür. Buz üzerinde kullanıma kadar yerleştirin.
- Üreaz-DNA sentezi (UrDNA)
- 2.5 mi mikrofüj tüpüne 100 uM LD1 10 ul ekle. GKD 2 O 140 ul 10x PBS 40 ul ve vorteks ekleyin.
- SMCC s 80.5 ul ekleyintak, bir tezgah üstü santrifüj kullanılarak DMSO, girdap ve kısaca santrifüj 159.5 ul.
- 60 dakika boyunca 37 ° C'de inkübe edin. mikrofuge'ye kapağın altında yoğuşmayı önlemek.
- 1x PBS 200 ul 3 M NaOAc (pH 5.2), 60 ul soğuk% 100 etanol içinde 1.5 ml. Vorteks ile çözüm karıştırın ve 30 dakika boyunca -20 ° C'de inkübe edin.
- 4 ° C'de 20 dakika boyunca 20,000 x g'de santrifüjleyin çözeltisi. Süpernatantı ve vakum altında pelet kurulayın. aşırı kurumayı önlemek.
- Kurutulmuş pelet, üreaz stokunun 400 ul, 5 saat oda sıcaklığında inkübe edin.
- Önceden yıkanmış 100K MWCO santrifüj filtre kolonuna ham konjügat 200 ul aktarın. 5 dakika boyunca 14.000 x g'de sütun santrifüjleyin.
- 5 dakika boyunca 14.000 x g'de kolon ve santrifüj ham konjügat kalan 200 ul aktarın. sütunu kaldırmak ve yeni bir 2.0 ml mikrofuge'de tüp ( "toplama tüpüne") olarak baş aşağı yerleştirin.
- Santrifüjuge 2 dakika süreyle 1000 xg'de (ters sütun ile) toplama tüpü. toplama tüpünü çıkarın ve konjugatlarının ek kurtarma için membran yıkamak için santrifüj sütuna 1x PBS 30 ul ekleyin.
- Bir kez daha sütunu ters ve geri toplama tüpünün içine koyun. 2 dakika süreyle 1000 xg'de (ters sütun ile) toplama tüpü santrifüj. Çıkarın ve sütun atmayın.
- kullanılana kadar 4 ° C 'de UrDNA saklayın.
Manyetik boncuklar üzerine EC1 ve UrDNA 4. Montaj
- gibi manyetik boncuk (MB) stok karıştırın ve 1.5 ml mikrofüj tüpe MB süspansiyon 100 ul aktarın. MB izole bir manyetik raf tutucu mikrofuge'ye tüpü yerleştirin.
- pipetle ile süpernatantı ve tüp tampon (BB) bağlayıcı 150 ul ekleyin. tutucudan tüpü çıkarın ve dikkatlice homojen bir çözüm MB tekrar süspansiyon tüp dokunun. t süspansiyon sıçramasını önlemektüp ya da kap op. Böyle bir durumda, kısaca geri süspansiyona kalıntı dönmeye bir tezgah üstü santrifüj kullanın.
- Adımı yineleyin 4.2 iki kez daha.
- Bu süspansiyona 10 uM EC1 10 ul ekle. Dikkatle tüp dokunarak karıştırın.
- Hafif 30 dakika boyunca çalkalayarak çözelti inkübe edin. MB çökmesini önlemek için tüpü her 2-3 dakikada bir dokunun.
- MB izole ve pipetleme süpernatant kaldırmak için geri manyetik raf mikrofuge'ye tüpü yerleştirin. (Adım 4.2 de tarif edildiği gibi) BB 150 ul MB üç kez yıkayın.
- Yıkama işlemi tamamlandıktan sonra, BB 150 ul bir toplam MB askıya. Bu çözeltiye, UrDNA 15 ul ilave edin ve 2 dakika boyunca 45 ° C'ye kadar çözelti ısıtılır. Solüsyon oda sıcaklığına kadar soğutulur ve 2 saat süreyle inkübe edin.
- MB izole ve pipetleme süpernatant kaldırmak için geri manyetik raf mikrofuge'ye tüpü yerleştirin.
- Reaksiyon tampon maddesi (RB), 100 ul ekle. t kaldırO manyetik raf tüp mikrofuge'ye ve dikkatle MB tekrar süspansiyon.
- adım 4.9 tekrarlayarak MB üç kez yıkayın.
NOT: yıkanmış süpernatant hızla Melezleşmemiş UrDNA hala mevcut olup olmadığını bir yanlış pozitif sinyal neden olabilir, hangi belirlemek için test edilebilir. Test yıkama çözeltisine kırmızı 50 mM üre 10 ul ve% 0.04 fenol 10 ul ekleyerek yapılabilir. yıkama çözeltisi bir renk değişimine neden olmaz kadar MB yıkamak devam edin. 100 ul süspansiyon kullanilana kadar 4 ° C'de saklanır.
Bakteriyel hücreler 20 5. hazırlanması
- Kültürleme E. Stoklar gelen coli
- Plaka E. E. coli K12, bir alev altında veya biyolojik bir güvenlik kabini, bir gliserol stokundan LB agar kaplan üzerine (MG1655).
- Steril bir pipet ucu kullanarak, yavaşça LB agar delinmesiyle önlemek için gliserol stok ve hafifçe çizgi bir agar plaka yüzeyi dokunun.
- Kesikli invertPlaka ve 14 saat boyunca 37 ° C'de inkübe edin.
- 3 haftalık bir süre için 4 ° C sıcaklıkta Parafilm ile plaka ve mağaza kapatın.
- Kültürleme E. Hücre Sayımı için coli
- steril 14 ml kültür tüpünde, LB suyu 2 ml dağıtın.
- Steril bir pipet ucu kullanarak, adım 5.1 hazırlanan çizgili agar plakasından tek bir koloni almak ve kültür tüpüne aktarın.
- 37 ° C 'de kültür inkübe ve 14 saat boyunca 230 rpm'de çalkalanır.
- Seri olarak 10-misli aralıklarla bakteri kültürü seyreltin.
- Her bir seyreltilmiş numune için eşit, ayrı LB agar plakaları üzerinde beş 100 ul alikotları plaka. plakalar ters çevirin ve 14 saat boyunca 37 ° C'de inkübe edin.
- Her bir seyreltme ortalama hücre konsantrasyonu elde etmek üzere her numune hücreleri sayın.
NOT: 10 7 hücreleri genellikle E. için bir referans turnusol testi kurmak için kullanılan E. Bu seviyeye kadar coli E. coli hızlı bir renk chan tetikleyebilirge. Sonuçlar bölümünde tartışıldığı gibi Ancak, düzgün idam turnusol testi gibi düşük 500 hücreleri tespit edebilir.
- E. hazırlama Test coli hücreleri
- arzu edilen bir hücre süspansiyonu için, transfer 1.5 ml mikrofüj tüpüne kültürlendi, 1 mL stok.
- 4 ° C'de 10 dakika boyunca 6000 x g'de santrifüjleyin hücreleri. Dikkatle hücre pelletini bozmadan süpernatant kaldırmak.
- Hücre topağı, reaksiyon tampon maddesi 10 ul ilave edin ve tekrar süspansiyon hücreleri. 5 dakika boyunca bir hücre süspansiyonu sonikasyon. 5 dakika için bir buz kutusu hücre süspansiyonu aktarın.
- 5 dakika daha hücre süspansiyonu sonikasyon.
- 4 ° C'de 10 dakika boyunca 13,000 x g'de hücre süspansiyonu santrifüj. Test için supernatant (10 ul) kullanır.
6. Litmus Testi
- 1.5 ml mikrofüj tüpüne, mikrofüj tüpüne reaksiyon tampon maddesi (RB) 100 ul ilave edildi ve vorteks yoluyla tüp ön yıkama vetampon atarak.
- Aktarım yıkanmış mikrofüj tüpe monte EC1 (protokol 4) 15 ul.
- Bir manyetik rafa mikrofuge'ye tüp yerleştirerek manyetik boncuk yıkayın. pipetleme süpernatantı. , Raftan mikrofuge'ye tüpünü çıkarın RB 100 ul ekleyin ve dikkatlice manyetik boncuklar tekrar süspansiyon.
- adım 6.3 yineleyerek MB iki kez yıkayın.
- , Manyetik rafa geri mikrofuge'ye tüpü yerleştirin süpernatant kaldırmak ve 10 ul E. ekleyin E. coli örnek adım 5.3 hazırlanan.
- yavaşça mikrofuge'de tüp dokunarak dikkatlice örnek ve manyetik boncuk karıştırın.
- 1 saat boyunca oda sıcaklığında reaksiyon inkübe edin.
- Reaksiyona, GKD 2 O 90 ul ekleyin ve bir manyetik raf üzerine mikrofuge'ye tüp yerleştirin.
- Sonra manyetik ayırma yaklaşık 3 dakika, dikkatle 0.5 ml mikrofuge'de tüp süpernatant 85 ul aktarın. yavaş yavaş süpernatant geri çekinherhangi bir manyetik boncuk toplama kaçının.
- Yukarıdaki mikrofüj tüpe% 0,04 fenol kırmızısı 15 ul ve alt-tabaka çözeltisi 100 ul ilave edin.
- renk değişikliği kaydetmek için belirli zaman aralıklarında bir fotoğraf çekmek.
Not: pH değişikliği bir mikroelektrot içeren bir pH metre kullanılarak izlenebilir. Başlangıç pH yaklaşık 5,2-5,5 olmalıdır (çözüm sarı renkte). Aksi takdirde, çözelti, 1 mM asetat tampon pH 5.0) ilavesiyle ayarlanabilir.
Sonuçlar
Bakteriyel test edeceği prensibi Şekil 1 'de açıklanmıştır Test üç temel malzeme kullanır. Belirli bir bakteri, üreaz ve manyetik boncuklar ile aktive olan bir RNA klivaj DNAzim. DNAzim ilgi konusu bir bakterinin yüksek düzeyde spesifik tespit elde etmek için moleküler tanıma elemanı olarak kullanılır. Üreaz ve manyetik boncuk DNAzıme RNA klivaj aktivitesinin sinyal transdüksiyonunu elde etmek için kullanılır. Bu üreaz-DNAzim konjugatlarının ihtiva manyetik boncuk oluşturulmasını içerir. Hedef bakterinin varlığı, DNAzim parçalayarak kendi RNA bağlantı. Bu işlem, manyetik boncuklardan üreaz ayrışmasına neden olur. serbest üreaz kolay manyetik boncuk ayrılır ve üre ve pH'a duyarlı bir boya içeren bir raportör çözeltisi içinde bir renk değişikliği üretmek için de kullanılabilir. Üreaz c tetikleyen pH artışı eşliğinde amonyak içine üre hidrolizBoyanın enk değişimi.
Şekil 2 nerede EC1, E. bakteriyel bir turnusol testi sunuyor E. coli tepki gösteren RNA klivaj DNAzım, DNAzıme olarak kullanılmıştır, ve fenol kırmızısı, pH raporlama boya olarak kullanılmıştır. EC1 daha önce in vitro seçimi tekniği kullanılarak rasgele dizi DNA havuzu grubumuz ile izole edilmiştir. 5 Daha önceki çalışmalar EC1 E için son derece spesifik olduğu gösterilmiştir E. coli ve 5,19. Diğer bakteri karşı çok az aktivite sergiler EC1 E. bir protein molekülü ile aktive edilir olduğu bulunmuştur Bu protein belirteç kimliği deşifre edilmemiş olmakla birlikte coli., yüksek tanıma özgünlüğü bu proteinin E. özgü olduğunu göstermektedir E. coli. bildirici çözeltisi 5.5 bir başlangıç pH değerine sahip ayarlanır. Bu pH'da fenol kırmızı sarı bir renk sergiler. üreaz olarak, amonyak içine muhabiri solüsyonunun bazlık üre hidrolizKatkı artar. Bu san bir pembe rengin kademeli değişikliği ile yansıtılır. Şekil 2'de gösterildiği gibi renk değişikliği derinliği, aşağıdaki iki parametrelere bağlıdır: E. sayısı DNAzim aktivasyon aşaması, üre hidroliz adımı için izin verilen süre kullanılan coli hücreleri. Daha E. coli hücreleri ilerleyici sarı-to-pembe renk geçişi E. gözlem yansıyan güçlü renk değişikliklere neden coli hücreleri seri artan 5 ile 10 x 5-7 (10-kat, her zaman artış). Diğer taraftan, üre hidrolizi için daha uzun bir zaman E. az sayıda tespiti için izin E. coli hücreleri (1 saat tepkimede 5000 hücreleri ve 2 saat tepkimede 500 hücre).
Bakteriyel test edeceği pH değişikliği, aynı zamanda, bir el pH metre kullanılarak izlenebilir ve Örnek sonuçlar Şekil 3'te gösterilmektedir. Was bulduğu 10 7 E. varlığı coli hücreleri 10 dakika içinde 3 adet pH kademeli artış ile sonuçlanmıştır. Bunun aksine, E. yokluğu coli hücreleri aynı ayarı altında saptanabilir pH değişiklikleri neden olmadı.
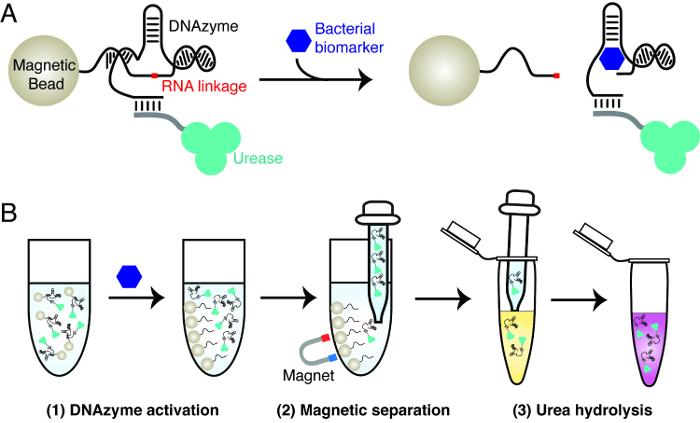
Şekil 1: Bakteriyel turnusol test tasarım ilkesi, ilgi konusu bir bakteriden, belirli bir belirteç ile bir RNA klivaj DNAzim (A) aktivasyonu.. belirteç mevcudiyetinde, RNA-yaran DNAzim çözeltisine manyetik boncuklardan etiketli üreaz salınmasına yol açar manyetik boncuk yarılır ilgili RNA bağı hareket. (B) üç adımlı deney prosedürü. Adım 1: DNAzim aktivasyonu, panel A, aşama 2'de tarif edildiği gibi: Manyetik Ayırma - serbest üreaz manyetik boncuk ayrılır. Aşama 3: Üre hidroliz R12; serbest üreaz, bir üre ihtiva eden raportör çözeltisi içine ilave edilir. Üreaz bir pH-duyarlı boya ile rapor edilebilir pH değişikliği ile sonuçlanan, amonyak içine üre hidrolize. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.

Şekil 2: E. ile Turnusol testi E. coli E. kullanarak coli tepki gösteren DNAzim EC1. E. değişen numaraları ile Temsilcisi renk değiştiren sonuçlar Her bir test tüpüne, yukarıda verilen coli hücreleri. Fenol kırmızı pH'a duyarlı bir boya olarak kullanılmıştır. E. olmadan bir test E. coli, bir negatif kontrol olarak kullanılmıştır. Daha E. coli hücreleri daha fazla üreaz moleküllerinin salgılanmasına neden bekleniyor, b eşliky güçlü rengi değişir. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
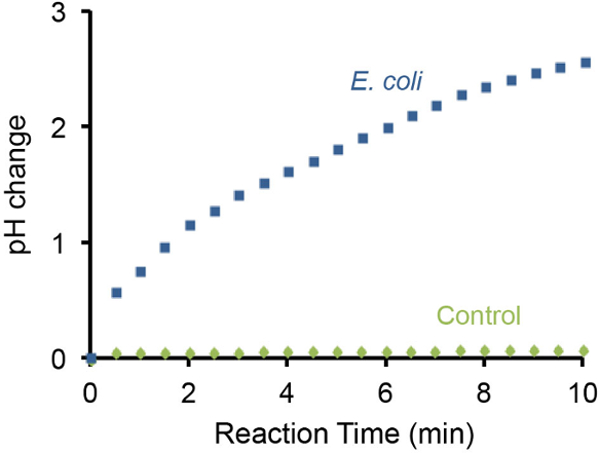
Şekil 3: pH metre kullanılarak pH artışı İzleme 10 7 E. neden pH değişikliği. coli hücreleri taşınabilir pH metre kullanılarak izlenmiştir. E. olmadan bir test E. coli, bir negatif kontrol olarak kullanılmıştır. 10 7 E. varlığı Test çözeltisi içinde coli hücreleri 10 dakika içinde ~ 3 pH birimi tarafından bazlığını artırabilir. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
| isim | Sesekans (5'-3 ') | Not |
| BS1 | BTTTT TTTTT TTTAC TCTTC CTAGC FRQGG TTCGA TCAAG bir | B: 5'-Biotin; R: adenin ribonükleotid; F: floresein-dT; S: DABCYL-dT |
| DE1 | GATGT GCGTT GTCGA GACCT GCGAC CGGAA CACTA CACTG TGTGG GGATG GATTT CTTTA CAGTT GTGTG TTGAA CGCTG TGTCA AAAAA AAAA | |
| T1 | GACAA CGCAC ATCTC TTGAT CGAAC Cı | |
| LD1 | XTTTT TTTTT TTTTT TTGAC ACAGC GTTCA bir | X: 5'-NH2 |
Tablo 1: sentetik oligonükleotit dizileri.
Tartışmalar
Şekil 1 'de gösterildiği gibi bir turnusol test etmek için bir bakteri duyarlı DNAzıme RNA bölünme etkinliğinin etki yaptı, üreaz ve manyetik ayırma kullanımı ile mümkün olmaktadır. Bakteriyel tespiti için modifiye test edeceği gösteri rağmen E ile yapılan E. coli bağımlı RNA-yaran DNAzim, 5,19,20 tasarım genellikle herhangi RNA-yaran DNAzıme için uzatılabilir. Yeni hedefler için rasgele dizi havuzlarından yeni RNA-yarma DNAzımler izole etmek farklı analitler ve çeşitli metodolojileri için RNA-yaran DNAzimler büyük kullanılabilirliği göz önüne alındığında, biz modifiye turnusol testi platformu ilgi çeşitli hedeflerin tespiti uzatılabilir bekliyoruz .
E. için turnusol testi Raporlama reaksiyon süresi sırasıyla 1 ve 2 saat olmak üzere ayarlandığı zaman coli algılama 5,000 ve 500 hücreleri tespit edebilir. Popüler polimeraz zincir reaksiyonu (PCR) vesandviç immün testi enzim-bağlı (ELISA) yöntemleri yaklaşık 10 4 -10 5 E. algılama sınırlarını elde edebilirsiniz Benzer test zamanlarında coli hücreleri. 25,26 Böylece, bakteriyel bir turnusol testi karşılaştırılabilir algılama hassasiyetini sunar.
Bakteriyel turnusol testi yürütmek kolay ve canlı renk değişiklikleri üretebilir rağmen, çeşitli faktörler önemli ölçüde test sonuçlarını etkileyebilir. İlk olarak, üreaz kalitesi çok önemlidir. Farklı kaynaklardan gelen üreaz kullanılan ve test sonuçları önemli ölçüde değişebilir bulduk. Biz Malzemeler bölümünde belirtilen kaynaktan üreaz kullanılması tavsiye edilir.
DNAzim / üreaz / manyetik boncuk montaj özel dikkat gerektirir. melezleşmemiş UrDNA kaldırmak için manyetik boncuk Kapsamlı yıkama yanlış pozitif sonuçların önlenmesi için gereklidir. Bakım ayrıca l iç yüzeyinde kalan manyetik boncuk birikmesini önlemek için alınması gerekengörmek zor olabilir mikrofuge'de tüp, kimliği. Bir kez orada, manyetik boncuk artık manyetik ayırma tabi tutulur ve böylece muhabir reaksiyonunda yalancı pozitif sinyaller yol açabilir bazı melezleşmemiş UrDNA taşıyabiliyor. Manyetik ayırma adımı sırasında daha uzun bir süre, 10 dakika boyunca manyetik rafa mikrofüj tüpü terk bilmek de önemlidir. boncuk toplamak veya yıkama verimliliği azaltır ve partiden partiye tutarsızlığı tanıtmak olabilir mikrofuge'ye tüp, yapışabilir. Yıkama çözeltisi içinde% 0.01 Tween-20 dahil edilmesi partiden partiye tutarlılık artırabilir ve uygulanmalıdır.
Manyetik kurecikler manyetik boncuklar üzerine DNAzim-üreaz konjugatlarının monte çapa olarak kullanılan streptavidin ile kaplanmıştır. streptavidin ve üreaz Hem depolama sırasında denatüre edilebilir protein molekülleridir. Normal olarak 4 haftaya kadar 4 ° C de bir araya DNAzim-üreaz manyetik boncuk depolamakve daha tutarlı sonuçlar elde etmek için düzenli olarak taze toplu yapmak.
Bakım ayrıca DNAzim aktivasyonunu takiben manyetik ayırma adımı (adım 6.9) manyetik boncuk alarak kaza önlemek için alınması gereken. Bizim deneyim, hücresel enkaz ve çözüm diğer partiküller manyetik ayırma verimliliği azaltabilir ve bu nedenle, bazı manyetik boncuk istemeden pipetleme sırasında alınabilir. Bu yanlış pozitif sonuçlara neden olur. uzun bir ayrılık süresi (örneğin 5-10 dk), pipet üzerinde bir basınç yavaş salınım süpernatant nazik çekilmesi izin verir ve manyetik ayırma ek bir tura yüzer tabi: Biz problemi hafifletmek için aşağıdaki önlemleri tavsiye .
Son olarak, çok sayıda numune test edilir bir deney sırasında üreazın tarafından bildirici stok çözeltisi kaza kirlenmesini önlemek için önemlidir. u yüksek reaktif göz önüne alındığındaSerbest Bırakma, bu doğanın kirlenmesi yanlış pozitif sonuçlara yol açabilir.
Açıklamalar
The authors have nothing to disclose.
Teşekkürler
The funding for this research project was provided by the Natural Sciences and Engineering Research Council of Canada (NSERC) via a Discovery Grant to YL.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Ethylenediaminetetraacetic acid (EDTA) | VWR AMRESCO | 0105 | |
| Sodium Hydroxide (NaOH) pellets | BIO BASIC CANADA INC. | SB6789 | |
| Tris-base | VWR AMRESCO | 0497 | |
| Boric acid | AMRESCO | 0588 | |
| Urea | VWR AMRESCO | M123 | |
| 40% acrylamide/bisacrylamide (29:1) solution | BIO BASIC CANADA INC. | A0007 | |
| Sucrose | Bioshop Canada inc. | SUC507 | |
| Bromophenol blue | Bioshop Canada inc. | BRO777 | |
| Xylenecyanol FF | SIGMA-ALDRICH | X-4126 | |
| 10% sodium dodecyl sulfate | Bioshop Canada inc. | SDS001 | |
| Hydrochloric Acid (HCl) | CALEDON LABORATORIES LTD | 6026 | |
| Sodium Chloride (NaCl) | Bioshop Canada inc. | SOD001 | |
| 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid (HEPES) | Bioshop Canada inc. | HEP001 | |
| Magnesium Chloride (II) hexahydrate | VWR AMRESCO | 0288 | |
| Tween 20 | Bioshop Canada inc. | TW508 | |
| Adenosine Triphospahte (ATP) | AMRESCO | 0220 | |
| Sodium Acetate trihydrate (NaOAc) | SIGMA-ALDRICH | S8625 | |
| Ethanol | Commercial Alcohols | P016EAAN | |
| Tetramethyleneethylenediamine (TEMED) | AMRESCO | 0761 | |
| 10% Ammonium persulfate (APS) | BIO BASIC CANADA INC. | AB0072 | |
| Succinimidyl 4-(N-maleimidomethyl) cyclohexane-1-carboxylate (SMCC) | ThermoFisher SCIENTIFIC | 22360 | |
| Dimethyl sulfoxide (DMSO) | CALEDON LABORATORIES | 803540 | |
| Urease | SIGMA-ALDRICH | U0251 | |
| 1x Phosphate Buffered Saline (PBS) | ThermoFisher SCIENTIFIC | 70011-069 | |
| 0.04% Phenol red | SIGMA-ALDRICH | P3532 | |
| 10x T4 polynucleotide kinase reaction buffer | Lucigen | 30061-1 | |
| 10x T4 DNA ligase reaction buffer | Bio Basics Canada | B1122-B | |
| T4 DNA ligase (5 U/μl) | Thermo Fischer Scientific | B1122 | |
| Luria Bertani (LB) Broth | AMRESCO | J106 | |
| Agar | AMRESCO | J637 | |
| T4 polynucleotide kinase (10 U/μl) | Lucigen | 30061-1 | |
| E. coli K12 (MG1655) | ATCC | ATCC700926 | |
| Centrifuge | Beckman Coulter, Inc. | 392187 | |
| Glass plates | CBS scientific | ngp-250nr | |
| 0.75 mm thick spacers | CBS scientific | VGS-0725r | |
| 12-well comb | CBS scientific | VGC-7512 | |
| UV Lamp | UVP | 95-0017-09 | |
| Spectrophotometer (NanoVue) | GE Healthcare | N/A | |
| Metal plate | CBS scientific | CPA165-250 | |
| Vortex | VWR International | 58816-123 | |
| Gel electrophoresis apparatus | CBS scientific | ASG-250 | |
| Petri dishes | VWR International | 25384-342 | |
| 100 kDa MWCO centrifugal filters | EMD Millipore | UFC510024 | |
| Magnetic Bead (BioMag) | Bangs Laboratories Inc | BM568 | |
| Magnetic Seperation Rack | New England BioLabs | S1506S | |
| Microfuge tubes | Sarstedt | 72.69 | |
| Syringe filter (0.22 μm) | VWR International | 28145-501 | |
| 14 ml culture tube | VWR International | 60818-725 | |
| Cell culture incubator | Eppendorf Scientific | M13520000 | |
| Branson Ultrasonic cleaner | Branson | N/A | |
| Camera (Canon Powershot G11) | Canon | N/A | |
| 50 ml conical tube | VWR International | 89004-364 |
Referanslar
- Daar, A. S., et al. Top ten biotechnologies for improving health in developing countries. Nat. Genet. 32, 229-232 (2002).
- Newman, J. D., Turner, A. P. Home blood glucose biosensors: a commercial perspective. Biosens. Bioelectron. 20, 2435-2453 (2005).
- Turner, A. P. Biosensors: sense and sensibility. Chem. Soc. Rev. 42, 3184-3196 (2013).
- Tram, K., Kanda, P., Salena, B. J., Huan, S. Y., Li, Y. F. Translating Bacterial Detection by DNAzymes into a Litmus Test. Angew. Chem. Int. Ed. 53, 12799-12802 (2014).
- Ali, M. M., Aguirre, S. D., Lazim, H., Li, Y. Fluorogenic DNAzyme probes as bacterial indicators. Angew. Chem. Int. Ed. 50, 3751-3754 (2011).
- Schlosser, K., Li, Y. Biologically inspired synthetic enzymes made from DNA. Chem. Biol. 16, 311-322 (2009).
- Tuerk, C., Gold, L. Systematic evolution of ligand by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Breaker, R. R. Making catalytic DNAs. Science. 290, 2095-2096 (2000).
- Liu, J., Cao, Z., Lu, Y. Functional nucleic acid sensors. Chem. Rev. 109, 1948-1998 (2009).
- Navani, N. K., Li, Y. Nucleic acid aptamers and enzymes as sensors. Curr. Opin. Chem. Biol. 10, 272-281 (2006).
- Li, J., Lu, Y. A highly sensitive and selective catalytic DNA biosensor for lead ions. J. Am. Chem. Soc. 122, 10466-10467 (2000).
- Liu, J., Lu, Y. A colorimetric lead biosensor using DNAzyme-directed assembly of gold nanoparticles. J. Am. Chem. Soc. 125, 6642-6643 (2003).
- Liu, Z., Mei, S. H. J., Brennan, J. D., Li, Y. Assemblage of signaling DNA enzymes with intriguing metal specificity and pH dependence. J. Am. Chem. Soc. 125, 7539-7545 (2003).
- Liu, J., et al. A catalytic beacon sensor for uranium with parts-per-trillion sensitivity and millionfold selectivity. Proc. Natl. Acad. Sci. USA. 104, 2056-2061 (2007).
- Huang, P. J., Vazin, M., Liu, J. In vitro selection of a new lanthanide-dependent DNAzyme for ratiometric sensing lanthanides. Anal. Chem. 86, 9993-9999 (2014).
- Chiuman, W., Li, Y. Simple fluorescent sensors engineered with catalytic DNA 'MgZ' based on a non-classic allosteric design. PLoS One. 2, e1224(2007).
- Xiang, Y., Lu, Y. Using personal glucose meters and functional DNA sensors to quantify a variety of analytical targets. Nat. Chem. 3, 697-703 (2011).
- Aguirre, S. D., Ali, M. M., Salena, B. J., Li, Y. A sensitive DNA enzyme-based fluorescent assay for bacterial detection. Biomolecules. 3, 563-577 (2013).
- Aguirre, S. D., Ali, M. M., Kanda, P., Li, Y. F. Detection of Bacteria Using Fluorogenic DNAzymes. J. Vis. Exp. (63), e3961(2012).
- Shen, Z., et al. A catalytic DNA activated by a specific strain of bacterial pathogen. Angew. Chem. Int. Ed. 54, (2015).
- He, S., et al. Highly specific recognition of breast tumors by an RNA-cleaving fluorogenic DNAzyme probe. Anal. Chem. 87, 569-577 (2015).
- Sumner, J. B., Hand, D. B. Isoelectric point of crystalline urease. J. Am. Chem. Soc. 51, 1255-1260 (1929).
- Karplus, P. A., Pearson, M., Hausinger, R. P. 70 Years of crystalline urease: What have we learned. Acc. Chem. Res. 30, 330-337 (1997).
- Omiccioli, E., Amagliani, G., Brandi, G., Magnani, M. A new platform for Real-Time PCR detection of Salmonella spp., Listeria monocytogenes and Escherichia coli O157 in milk. Food Microbiol. 26, 615-622 (2009).
- Cui, S., Schroeder, C. M., Zhang, D. Y., Meng, J. Rapid sample preparation method for PCR-based detection of Escherichia coli O157:H7 in ground beef. J. Appl. Microbiol. 95, 129-134 (2003).
- Ibekwe, A. M., Watt, P. M., Grieve, C. M., Sharma, V. K., Lyons, S. R. Multiplex fluorogenic real-time PCR for detection and quantification of Escherichia coli O157:H7 in dairy wastewater wetlands. Appl. Environ. Microbiol. 68, 4853-4862 (2002).
- Strachan, N. J., Ogden, I. D. A sensitive microsphere coagulation ELISA for Escherichia coli O157:H7 using Russell's viper venom. FEMS Microbiol Lett. 186, 79-84 (2000).
- de Boer, E., Beumer, R. R. Methodology for detection and typing of foodborne microorganisms. Int. J. Food Microbiol. 50, 119-130 (1999).
- Gracias, K. S., McKillip, J. L. A review of conventional detection and enumeration methods for pathogenic bacteria in food. Can. J. Microbiol. 50, 883-890 (2004).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır