Method Article
كشف اللونية من البكتيريا عن طريق محك الاختبار
In This Article
Summary
We describe a protocol for colorimetric detection of E. coli using a modified litmus test that takes advantage of an RNA-cleaving DNAzyme, urease, and magnetic beads.
Abstract
There are increasing demands for simple but still effective methods that can be used to detect specific pathogens for point-of-care or field applications. Such methods need to be user-friendly and produce reliable results that can be easily interpreted by both specialists and non-professionals. The litmus test for pH is simple, quick, and effective as it reports the pH of a test sample via a simple color change. We have developed an approach to take advantage of the litmus test for bacterial detection. The method exploits a bacterium-specific RNA-cleaving DNAzyme to achieve two functions: recognizing a bacterium of interest and providing a mechanism to control the activity of urease. Through the use of magnetic beads immobilized with a DNAzyme-urease conjugate, the presence of bacteria in a test sample is relayed to the release of urease from beads to solution. The released urease is transferred to a test solution to hydrolyze urea into ammonia, resulting in an increase of pH that can be visualized using the classic litmus test.
Introduction
Bacterial pathogens are one of the major causes of global morbidity and mortality. Outbreaks from hospital-acquired infections, food-borne pathogens, and bacterial contaminants in the environment pose serious and on-going threats to public health and safety. To prevent these outbreaks, effective tools are needed that permit pathogen detection in a timely fashion under a variety of settings. Simple but still effective tests that are portable and cost-effective are greatly coveted, especially in regions that are susceptible to outbreaks but cannot afford expensive testing facilities.1-3 Although there exists a multitude of methods to detect bacteria, many of them are not suitable as screening or on-site testing tools because they require long test times, expensive instruments and complicated testing procedures.
Colorimetric tests are particularly attractive for point-of-care or field applications as color changes can be easily detected by the naked eye. The litmus test for pH is simple, quick, and effective. Although it is a very old technology, it is still widely used today because of its simplicity and effectiveness. Surprisingly, this simple test had never been modified to achieve the detection of other analytes before we recently developed an approach of modifying this test for E. coli testing.4
The expanded litmus test for E. coli employs three additional components: an E. coli activated RNA-cleaving DNAzyme (EC1), 5 urease, and magnetic beads. DNAzymes refer to synthetic single-stranded DNA molecules with catalytic activity.6 They can be isolated from random-sequence DNA pools using in vitro selection.7,8 They are highly stable and can be produced cost-effectively using high-efficiency automated DNA synthesis.9 For these reasons, DNAzymes, particularly RNA-cleaving DNAzymes, have been widely examined for biosensing applications.6,10,11 RNA-cleaving DNAzyme sensors have been developed to detect metal ions,12-16 small molecules,17,18 bacterial pathogens5,19-21 and cancer cells.22 Given the great availability of target-induced RNA-cleaving DNAzymes, any assay that utilizes a DNAzyme can be potentially expanded to detect a diverse range of analytes.
Urease is chosen for its ability to hydrolyze urea into ammonia,23,24 resulting in a pH increase. Urease is also highly efficient, stable and amenable for conjugation to other biomolecules. Therefore, we postulated that a conjugate of an RNA-cleavage DNAzyme with urease would allow the use of litmus test for the detection of other targets.5
The action of the RNA-cleaving DNAzyme is relayed to urease-mediated increase of pH through the use of magnetic beads that are immobilized with the DNAzyme-urease conjugate. Because the activity of the DNAzyme under investigation is strictly dependent on E. coli, the presence of this bacterium in the test solution will result in the release of urease from the magnetic beads to the solution, which is then taken and used to hydrolyze urea in a reporter solution that contains a pH-sensitive dye. The final outcome of this procedure is a color change that can be conveniently reported by the dye or pH paper.
Protocol
1. إعداد الكواشف والمخازن
- 0.5 M ثنائي أمين الإيثيلين رباعي حمض الخل (EDTA)
- في دورق 2 لتر، إضافة 186.1 ز EDTA إلى 800 مل من الماء منزوع الأيونات المقطر ([ده 2 O). ضبط درجة الحموضة من الحل إلى 8.0 باستخدام كريات هيدروكسيد الصوديوم. إضافة ده 2 O إلى الحجم النهائي من 1.0 لتر ونقل حل لزجاجة autoclavable عن التعقيم وتخزينها في 4 درجات مئوية.
- EDTA 10 × تريس، بورات (10X TBE)
- في 4 لتر كوب من البلاستيك، إضافة 432 غرام تريس، قاعدة، و 200 غرام حامض البوريك، 80 مل من 0.5 M EDTA (8.0 درجة الحموضة) وده 2 O إلى الحجم النهائي من 4 ل. مزيج باستخدام بقضيب حتى كل المكونات تذاب. نقل حل لقوارير الزجاج 1 لتر من التعقيم وتخزينها في 4 درجات مئوية.
- 10٪ بولي أكريلاميد تغيير طبيعة الأوراق المالية
- في 4 لتر كوب من البلاستيك، إضافة 1681.7 ز اليوريا، 400 مل 10X TBE، 1 لتر من 40٪ مادة الأكريلاميد / bisacrylamide (29: 1) حل وده 2 O حتى وحجم إينال هو 4 L. ميكس باستخدام بقضيب حتى يذوب اليوريا. نقل الحل إلى 1 لتر الزجاجات العنبر الزجاج وتخزينها في 4 درجات مئوية.
- 2X جل تحميل العازلة (2X GLB)
- في كوب زجاج 200 مل، إضافة 44 ز اليوريا، 8 ز السكروز، 10 ملغ برموفينول الأزرق، و 10 ملغ سيانول زايلين FF، 400 ميكرولتر من 10٪ كبريتات الصوديوم دوديسيل، و 4 مل من 10 × TBE. إضافة ده 2 O إلى الحجم النهائي من 40 مل وتذوب المكونات مع التدفئة خفيفة عند 50 درجة مئوية في حين خلط مع شريط مغناطيسي. نقل 1 مل قسامة إلى 1.5 مل أنابيب microfuge وتخزينها في 4 درجات مئوية.
ملاحظة: استخدام المسبقة يتطلب تسخين وجيزة في 90 درجة مئوية إلى تنحل أي مواد صلبة.
- في كوب زجاج 200 مل، إضافة 44 ز اليوريا، 8 ز السكروز، 10 ملغ برموفينول الأزرق، و 10 ملغ سيانول زايلين FF، 400 ميكرولتر من 10٪ كبريتات الصوديوم دوديسيل، و 4 مل من 10 × TBE. إضافة ده 2 O إلى الحجم النهائي من 40 مل وتذوب المكونات مع التدفئة خفيفة عند 50 درجة مئوية في حين خلط مع شريط مغناطيسي. نقل 1 مل قسامة إلى 1.5 مل أنابيب microfuge وتخزينها في 4 درجات مئوية.
- 1 M تريس، هيدروكلوريد (تريس، حمض الهيدروكلوريك) (7.5 درجة الحموضة)
- في زجاجة، إضافة 12.1 غرام من تريس القاعدة و 70 مل من ده 2 O وتخلط حتى يذوب الصلبة. ضبط درجة الحموضة إلى 7.5 باستخدام 1 M حمض الهيدروكلوريك (حمض الهيدروكلوريك). إضافة ده 2 O إلى الحجم النهائي من 100 مل، الأوتوكلاف وستوإعادة في 4 درجات مئوية
- 5 M كلوريد الصوديوم (كلوريد الصوديوم)
- في زجاجة، ويحل 58.4 غرام من كلوريد الصوديوم في 150 مل من ده 2 O. ضبط مستوى الصوت إلى 200 مل مع DDH 2 O. تخزينها في 4 درجات مئوية.
- الحمض النووي الاحتياطي شطف
- في زجاجة، وخلط 2.0 مل من 1 M تريس، حمض الهيدروكلوريك (7.5 درجة الحموضة)، 8.0 مل من 5 M كلوريد الصوديوم و 0.4 مل من 0.5 M EDTA (8.0 درجة الحموضة). ضبط مستوى الصوت إلى 200 مل مع DDH 2 O، الأوتوكلاف وتخزينها في 4 درجات مئوية.
- 1 M 4- (2-هيدروكسي) حامض -1-piperazineethanesulfonic (HEPES) (7.4 درجة الحموضة)
- في زجاجة، ويحل 2.38 غرام من HEPES في 80 مل من ده 2 O. ضبط درجة الحموضة إلى 7.4 باستخدام 5 N هيدروكسيد الصوديوم وإضافة ده 2 O إلى الحجم النهائي من 100 مل.
- 1 M كلوريد المغنيسيوم (MgCl 2)
- في زجاجة، إضافة 2،03 غرام من MgCl 2 -6H 2 O وتبرزي حجم 100 مل مع DDH 2 O.
- رد فعل العازلة (RB)
- في 50 مل أنبوب مخروطي الشكل، إضافة 50 ميكرولتر من 1 M HEPES (7.4 درجة الحموضة)، و 1.5 مل من 5 M كلوريد الصوديوم، 0.75 مل من 1 M MgCl 2، و 5 ميكرولتر من توين-20. إضافة ده 2 O إلى الحجم النهائي من 50 مل. مزيج من حل وتصفية في أنبوب مخروطي آخر باستخدام فلتر يحركها حقنة (0.22 ميكرون)، وتخزينها في 4 درجات مئوية حتى الاستخدام.
- الاحتياطي ملزم (BB)
- في 50 مل أنبوب مخروطي الشكل، إضافة 500 ميكرولتر من 1 M تريس، حمض الهيدروكلوريك (7.5 درجة الحموضة)، 8.8 غرام من كلوريد الصوديوم، و 50 ميكرولتر من 1 M من MgCl 2، و 5 ميكرولتر من توين 20. اضافة ده 2 O إلى الحجم النهائي 50 مل. مزيج من حل وتصفية في أنبوب مخروطي آخر باستخدام فلتر يحركها حقنة (0.22 ميكرون)، وتخزينها في 4 درجات مئوية حتى الاستخدام.
- الركيزة الحل (SS)
- في 50 مل أنبوب مخروطي الشكل، إضافة 5.8 غرام من كلوريد الصوديوم، 3 مل من 1 M MgCl 2، 1.5 غرام اليوريا، و 40 مل من ده 2 O. ضبط درجة الحموضة من الحل إلى 5.0 باستخدام 10 ملم حمض الهيدروكلوريك. لأنه لم يتم تخزينها مؤقتا الحل، وضبط درجة الحموضة باستخدام حمض الهيدروكلوريكبعناية من خلال إضافة قسامات حمض الهيدروكلوريك الصغيرة. إضافة ده 2 O إلى الحجم النهائي من 50 مل.
- مزيج من حل وتصفية في أنبوب مخروطي آخر باستخدام فلتر يحركها حقنة (0.22 ميكرون)، وتخزينها في 4 درجات مئوية حتى الاستخدام.
- لوريا Bertani (LB) مرق
- في كوب، ويحل 20.0 غرام من مسحوق LB في 1 لتر من ده 2 O. نقل إلى زجاجة، الأوتوكلاف، وتخزينها في 4 درجات مئوية.
- 1.5٪ LB أجار
- في قارورة 250 مل، إضافة 1.5 غرام أجار و 100 مل من مرق LB. الأوتوكلاف وتخزينها في 4 درجات مئوية.
- لوحات أجار
- تنحل أجار LB في الميكروويف وتبريد حل ل~ 50 درجة مئوية. صب الحل في أطباق بتري، مما يجعل ~ 5 لوحات، والسماح لهم لترسيخ.
2. تحضير وتنقية E. coli- استجابة DNAzyme EC1
- توليف EC1 بواسطة قالب وساطة الأنزيمية من ربط
- تنقية بالاتصالاتأليغنوكليوتيد] توليفها ercially BS1، DE1، وT1 (تسلسل المنصوص عليها في الجدول رقم 1) بنسبة 10٪ تبديل طبيعة الكهربائي للهلام بولي أكريلاميد (dPAGE) وفقا لبروتوكولات موحدة.
- إعداد الأوراق المالية 100 ميكرومتر من BS1، DE1، وT1. تخزين في درجة حرارة -20 درجة مئوية حتى الاستخدام.
- في أنبوب 1.5 مل microfuge، إضافة 38.5 ميكرولتر من ده 2 O و 5 ميكرولتر من DE1، و 5 ميكرولتر من 10X T4 عازلة رد فعل عديد النوكليوتيد كيناز التي يقدمها المورد انزيم (500 ملي تريس، حمض الهيدروكلوريك (الرقم الهيدروجيني 7.6)، و 100 ملي MgCl 2، 50 ملي dithiothreitol (DTT)، 1.0 ملي إنتاج الأسبرميدين).
- إضافة 1 ميكرولتر من ثلاثي فوسفات الأدينوزين (ATP) (100 ملم). إضافة 5 وحدات من كيناز T4 عديد النوكليوتيد (10 U / ميكرولتر). مزيج من قبل pipetting بعناية خليط التفاعل.
- احتضان رد الفعل عند 37 درجة مئوية لمدة 30 دقيقة.
- إخماد رد فعل عن طريق التسخين إلى 90 درجة مئوية لمدة 5 دقائق.
- إضافة 118 ميكرولتر من ده 2 O و 5 ميكرولتر من BS1، و 5 ميكرولتر من T1. حرارة التفاعل إلى 90 درجة مئوية لمدة 2 ميلن ثم بارد لدرجة حرارة الغرفة أكثر من 10 دقيقة.
- إضافة 20 ميكرولتر من 10 × T4 DNA العازلة رد فعل يغاز التي يقدمها المورد انزيم (400 ملي تريس، حمض الهيدروكلوريك (الرقم الهيدروجيني 7.8)، و 100 ملي MgCl 2، 100 ملي DTT و 5 ملي ATP). إضافة 10 وحدة من يغاز T4 الحمض النووي (5 U / ميكرولتر) وتخلط بعناية من قبل pipetting.
- يحضن في درجة حرارة الغرفة لمدة 2 ساعة.
- إضافة 20 ميكرولتر من 3 خلات الصوديوم M (NaOAc) (الرقم الهيدروجيني 5.2)، و 500 ميكرولتر من البرد الإيثانول بنسبة 100٪. مزيج الحل من قبل vortexing ووضع الأنبوب في الثلاجة -20 درجة مئوية لمدة 30 دقيقة.
- الطرد المركزي microfuge في 20000 x ج لمدة 20 دقيقة على 4 درجات مئوية. إزالة بعناية طاف قبل pipetting.
- غسل بيليه مع الباردة الايثانول 70٪ وأجهزة الطرد المركزي مرة أخرى في 20000 x ج لمدة 10 دقيقة على 4 درجات مئوية. مرة أخرى إزالة طاف وتجفيف بيليه في ظل فراغ لمدة 10 دقيقة.
- Resuspend والحمض النووي مع 15 ميكرولتر من ده 2 O ثم إضافة 15 ميكرولتر من 2X GLB.
- دوامة بقوة والحرارة إلى 90 درجة مئوية لمدة2 دقيقة. تنقية الحمض النووي بالطول بنسبة 10٪ dPAGE كما هو موضح أدناه.
- إعداد 10٪ dPAGE
- لوحات نظيفة اثنين من الزجاج (لوحة واحدة كاملة وحقق واحدة)، واثنين من 0.75 ملم الفواصل سميكة، ومشط 12-جيدا. وضع لوحة كوب واحد على سطح مستو مع اثنين من الفواصل على كل جانب ولوحة حقق على القمة. مقطع لوحات اثنين معا باستخدام مقاطع أربعة المقدمة من المورد.
- في 150 مل دورق البلاستيك، صب 40 مل من 10٪ dPAGE، 40 ميكرولتر من tetramethylethylenediamine (TEMED)، و 400 ميكرولتر من فوق كبريتات الأمونيوم 10٪ (APS). خلط المكونات وتصب الحل بين لوحات ببطء.
- إدراج مشط ومن ثم السماح للهلام لتتبلمر أكثر من 10 دقيقة. مرة واحدة يتم بلمرة هلام، وإزالة ببطء المشط وتدفق الآبار مع DDH 2 O.
- تركيب لوحات على الجهاز الكهربائي للهلام. استخدام لوحة معدنية لتبديد الحرارة المتولدة لمنع ارتفاع درجة الحرارة.
- صب TBE 1X على غرف العلوية والسفليةوالتأكد من أن الآبار المغمورة جيدا في المخزن المؤقت. تدفق الآبار مع 1X TBE باستخدام ماصة أو حقنة.
- تعيين جهاز لتشغيل عند 35 أمبير وما قبل التشغيل لمدة 15 دقيقة قبل عينات التحميل.
- شطف من Ligated EC1 من 10٪ dPAGE
- الخطوة التالية 2.2.6، تشغيل هلام في 35 مللي أمبير حتى القاع صبغ (برموفينول الأزرق) تصل إلى الجزء السفلي من لوحة من الزجاج. هذا ينبغي أن تأخذ حوالي 1.5 ساعة. إيقاف الطاقة وإزالة لوحات من الجهاز.
- وضع لوحات على بضع ورقات من مناشف ورقية وإزالة بعناية الفواصل من لوحات الزجاج. بعناية إزالة لوحة أعلى الزجاج والتفاف هلام مع غلاف بلاستيكي.
- الوجه هلام فوق لإزالة لوحة من الزجاج الثانية، وتغطي مع غلاف بلاستيكي مرة أخرى. الحرص على تجنب التجاعيد فى الاغطية البلاستيكية.
- تصور المنتج ligated باستخدام الأشعة فوق البنفسجية التظليل (260 نانومتر)، والتي سوف تنتج 4 نطاقات الحمض النووي مميزة (EC1 ligated بالكامل، DE1، BS1، وT1).
- المكوس على الشريط العلوي (EC1) بشفرة حلاقة معقمة ونقل إلى أنبوب 1.5 مل microfuge. سحق هلام مع طرف ماصة معقمة (200 ميكرولتر حجم غيض) حتى يتحول إلى عجينة غرامة. إضافة 550 ميكرولتر من العازلة شطف الحمض النووي، وتغطية الأنبوب بورق الألمنيوم لحماية fluorophore جزءا لا يتجزأ، ويهز لمدة 15 دقيقة.
- أجهزة الطرد المركزي في حل هلام في 20000 x ج لمدة 5 دقائق ونقل بعناية 400 ميكرولتر من طاف لأنبوب 1.5 مل microfuge آخر. تجنب سحب القطع هلام خلال pipetting ل.
- إلى microfuge أنبوب مع طاف نقل إضافة 40 ميكرولتر من 3 M NaOAc (الرقم الهيدروجيني 5.2)، و 1.0 مل من البرد الإيثانول بنسبة 100٪. مزيج الحل من قبل vortexing ووضع الأنبوب في الثلاجة -20 درجة مئوية لمدة 30 دقيقة.
- الطرد المركزي microfuge في 20000 x ج لمدة 20 دقيقة على 4 درجات مئوية. إزالة بعناية طاف قبل pipetting.
- غسل بيليه مع الباردة الايثانول 70٪ وأجهزة الطرد المركزي مرة أخرى في 20000 x ج لمدة 10 دقيقة على 4 درجات مئوية. مرة أخرى، وإزالةطاف وتجفيف بيليه في ظل فراغ لمدة 10 دقيقة.
- تحديد تركيز EC1 من خلال قياس امتصاص الأشعة فوق البنفسجية في 260 نانومتر. جعل المخزون 10 ميكرومتر وتخزين العينة عند درجة حرارة -20 درجة مئوية حتى الاستخدام.
3. الإقتران من اليورياز على الحمض النووي
- إعداد Succinimidyl 4- (N-maleimidomethyl) الهكسان الحلقي-1-الكربوكسيل (SMCC) سهم
- حل 1 SMCC ملغ في 676 ميكرولتر من DMSO. دوامة ومكان على الجليد حتى الاستخدام.
- إعداد اليورياز المالية
- حل 1 ملغ اليورياز في 1 مل من 1 × برنامج تلفزيوني (لا المغنيسيوم 2+ أو الكالسيوم 2+). ضع على الجليد حتى الاستخدام.
ملاحظة: تبلور اليورياز بطيء إلى حل، وهناك حاجة خلط لطيف لتجنب تمسخ.
- حل 1 ملغ اليورياز في 1 مل من 1 × برنامج تلفزيوني (لا المغنيسيوم 2+ أو الكالسيوم 2+). ضع على الجليد حتى الاستخدام.
- توليف اليورياز الحمض النووي (UrDNA)
- إضافة 10 ميكرولتر من 100 ميكرومتر LD1 إلى أنبوب 2.5 مل microfuge. إضافة 140 ميكرولتر من ده 2 O، 40 ميكرولتر من 10X برنامج تلفزيوني، ودوامة.
- إضافة 80.5 ميكرولتر من SMCC الصورةتوك، 159.5 ميكرولتر من DMSO، الدوامة، وأجهزة الطرد المركزي لفترة وجيزة باستخدام جهاز للطرد المركزي الفوق.
- احتضان عند 37 درجة مئوية لمدة 60 دقيقة. تجنب التكثيف تحت سقف microfuge.
- إضافة 200 ميكرولتر من برنامج تلفزيوني 1X، 60 ميكرولتر من 3 M NaOAc (الرقم الهيدروجيني 5.2)، و 1.5 مل من البرد الإيثانول بنسبة 100٪. مزيج الحل من قبل vortexing واحتضان عند -20 درجة مئوية لمدة 30 دقيقة.
- أجهزة الطرد المركزي الحل في 20000 x ج لمدة 20 دقيقة على 4 درجات مئوية. إزالة طاف وتجفيف بيليه في ظل فراغ. تجنب الإفراط في تجفيف.
- لبيليه المجففة إضافة 400 ميكرولتر من الأسهم اليورياز ويحضن في درجة حرارة الغرفة لمدة 5 ساعة.
- نقل 200 ميكرولتر من المكورات الخام إلى 100K MWCO عمود الطرد المركزي فلتر غسلها مسبقا. أجهزة الطرد المركزي لعمود في 14000 x ج لمدة 5 دقائق.
- نقل المتبقية 200 ميكرولتر من المكورات الخام إلى العمود والطرد المركزي في 14000 x ج لمدة 5 دقائق. إزالة العمود ووضعه رأسا على عقب في 2.0 مل أنبوب microfuge جديدة ( "أنبوب جمع").
- Centrifأويغه أنبوب جمع (مع العمود المقلوب) في 1000 x ج لمدة 2 دقيقة. إزالة أنبوب جمع وإضافة 30 ميكرولتر من برنامج تلفزيوني 1X إلى العمود الطرد المركزي لغسل غشاء للانتعاش إضافي من تقارن.
- مرة أخرى عكس العمود والمكان مرة أخرى في أنبوب جمع. أجهزة الطرد المركزي في أنبوب جمع (مع العمود المقلوب) في 1000 x ج لمدة 2 دقيقة. إزالة والتخلص من العمود.
- تخزين UrDNA في 4 درجات مئوية حتى الاستخدام.
4. جمعية EC1 وUrDNA على الخرز المغناطيسي
- خلط الأوراق المالية حبة المغناطيسي (MB) بشكل جيد ونقل 100 ميكرولتر من MB تعليق لأنبوب 1.5 مل microfuge. وضع microfuge أنبوب على حامل حامل المغناطيسي لعزل جماعة الإخوان المسلمين.
- إزالة طاف قبل pipetting وإضافة 150 ميكرولتر من العازلة ملزمة (BB) إلى أنبوب. إزالة أنبوب من صاحب والاستفادة بعناية أنبوب ل resuspend جماعة الإخوان المسلمين إلى حل متجانس. تجنب الرش تعليق لرالمرجع من الأنبوب أو غطاء. إذا حدث هذا، استخدم جهاز للطرد المركزي الفوق تدور لفترة وجيزة على بقايا إلى تعليق.
- كرر الخطوة 4.2 مرتين أخريين.
- لهذا تعليق إضافة 10 ميكرولتر من EC1 10 ميكرومتر. مزيج بعناية من قبل التنصت على أنبوب.
- احتضان الحل مع خفيفة تهتز لمدة 30 دقيقة. الاستفادة من أنبوب كل 2-3 دقيقة لتجنب الأمطار من جماعة الإخوان المسلمين.
- وضع microfuge أنبوب مرة أخرى على الرف المغناطيسي لعزل جماعة الإخوان المسلمين وإزالة طاف قبل pipetting. غسل MB ثلاث مرات مع 150 ميكرولتر من BB (كما هو موضح في الخطوة 4.2).
- وبمجرد أن غسل كاملة، تعليق MB في ما مجموعه 150 ميكرولتر من BB. إلى هذا الحل، إضافة 15 ميكرولتر من UrDNA والحرارة الحل إلى 45 درجة مئوية لمدة 2 دقيقة. تبريد حل لدرجة حرارة الغرفة، واحتضان لمدة 2 ساعة.
- وضع microfuge أنبوب مرة أخرى على الرف المغناطيسي لعزل جماعة الإخوان المسلمين وإزالة طاف قبل pipetting.
- إضافة 100 ميكرولتر من رد فعل العازلة (RB). إزالة رانه microfuge أنبوب من رف المغناطيسي و resuspend بعناية جماعة الإخوان المسلمين.
- غسل MB ثلاث مرات بتكرار الخطوة 4.9.
ملاحظة: طاف غسلها ويمكن اختبار بسرعة لتحديد ما إذا كان الغير مهجنة UrDNA لا يزال قائما، مما قد يتسبب في إشارة إيجابية كاذبة. ويمكن أن يتم الاختبار عن طريق إضافة 10 ميكرولتر من 50 ملي اليوريا و 10 ميكرولتر من 0.04٪ الفينول الأحمر إلى محلول الغسيل. الاستمرار في غسل MB حتى لا تتسبب في محلول الغسيل تغيير اللون. يتم تخزين تعليق 100 ميكرولتر في 4 درجات مئوية حتى الاستخدام.
5. إعداد الخلايا البكتيرية 20
- زراعة E. القولونية من الأسهم
- لوحة E. القولونية K12 (MG1655) على لوحات أجار LB من المخزون الجلسرين تحت لهب أو في خزانة السلامة البيولوجية.
- باستخدام طرف ماصة معقمة، المس بلطف الأسهم الجلسرين وخفيفة متتالية على سطح لوحة أجار لتجنب ثقب آغار LB.
- عكس مجزعلوحة واحتضان عند 37 درجة مئوية لمدة 14 ساعة.
- ختم لوحة مع بارافيلم وتخزينها في 4 درجة مئوية لمدة أقصاها 3 أسابيع.
- زراعة E. القولونية عن عد الخلايا
- في أنبوب ثقافة 14 مل العقيمة، الاستغناء 2 مل من مرق LB.
- باستخدام طرف ماصة معقمة، واختيار مستعمرة واحدة من لوحة أجار يشوبه أعدت في الخطوة 5.1 وتحويلها إلى أنبوب الثقافة.
- احتضان الثقافة في 37 درجة مئوية ويهز في 230 دورة في الدقيقة لمدة 14 ساعة.
- متسلسل تمييع ثقافة البكتيرية في فترات 10 أضعاف.
- لكل عينة الواحد، بالتساوي لوحة خمسة 100 مكل على لوحات أجار LB منفصلة. عكس لوحات واحتضان عند 37 درجة مئوية لمدة 14 ساعة.
- عد الخلايا من كل عينة للحصول على متوسط تركيز خلية من كل تخفيف.
ملاحظة: 10 7 خلايا غالبا ما تستخدم لإعداد اختبار إشارة الحقيقي لE. القولونية حيث أن هذا المستوى من E. القولونية يمكن أن تؤدي إلى اللون سريع تشانجنرال الكتريك. ومع ذلك، يمكن اختبارا تنفيذها بشكل صحيح كشف ما يصل الى 500 خلية، كما نوقش في قسم النتائج.
- إعداد E. خلايا القولونية للاختبار
- للتعليق الخلية المطلوبة، نقل 1 مل من الأسهم مثقف لأنبوب 1.5 مل microfuge.
- الطرد المركزي الخلايا في 6000 x ج لمدة 10 دقيقة على 4 درجات مئوية. إزالة بعناية طاف دون الإخلال بيليه الخلية.
- إضافة 10 ميكرولتر من العازلة رد فعل على بيليه الخلية وresuspend الخلايا. يصوتن تعليق خلية لمدة 5 دقائق. نقل تعليق الخلية إلى مربع الجليد لمدة 5 دقائق.
- يصوتن تعليق خلية لمدة 5 دقائق أخرى.
- الطرد المركزي تعليق خلية في 13000 x ج لمدة 10 دقيقة على 4 درجات مئوية. استخدام طاف للاختبار (10 ميكرولتر).
6. محك الاختبار
- في أنبوب 1.5 مل microfuge، prewash أنبوب عن طريق إضافة وvortexing ل100 ميكرولتر من رد فعل العازلة (RB) في microfuge أنبوب والتخلص من العازلة.
- نقل 15 ميكرولتر من EC1 تجميعها (بروتوكول 4) إلى microfuge أنبوب غسلها.
- تغسل حبات المغناطيسي عن طريق وضع microfuge أنبوب على الرف المغناطيسي. إزالة طاف قبل pipetting. إزالة microfuge أنبوب من الرف، إضافة 100 ميكرولتر من RB، و resuspend بعناية حبات مغناطيسية.
- غسل MB اثنين من أكثر الأوقات بتكرار الخطوة 6.3.
- وضع microfuge أنبوب مرة أخرى على الرف المغناطيسي، وإزالة طاف وإضافة 10 ميكرولتر E. أعدت القولونية عينة من الخطوة 5.3.
- خلط العينة والمغناطيسية الخرز بعناية من خلال استغلال بلطف على microfuge أنبوب.
- احتضان رد الفعل في درجة حرارة الغرفة لمدة 1 ساعة.
- إلى رد فعل، إضافة 90 ميكرولتر من ده 2 O ووضع microfuge أنبوب على الرف المغناطيسي.
- بعد ما يقرب من 3 دقائق من الفصل المغناطيسي، نقل بعناية 85 ميكرولتر من طاف لأنبوب 0.5 مل microfuge. سحب طاف ببطء لتجنب جمع أي حبات مغناطيسية.
- إلى microfuge أنبوب أعلاه إضافة 15 ميكرولتر من 0.04٪ الفينول الأحمر، و 100 ميكرولتر من حل الركيزة.
- التقاط صورة على فترات زمنية محددة لتسجيل تغيير اللون.
ملاحظة: تغيير في درجة الحموضة يمكن أيضا رصد باستخدام مقياس درجة الحموضة مع مسرى مكروي. يجب أن يكون الرقم الهيدروجيني بدءا حوالي 5،2-5،5 (الحل هو الأصفر). إن لم يكن، والحل يمكن تعديلها بإضافة 1 ملي خلات العازلة درجة الحموضة 5.0).
النتائج
ويفسر مبدأ اختبارا الجرثومي في الشكل 1 يستخدم الاختبار ثلاث مواد رئيسية هي: وDNAzyme الشق الحمض النووي الريبي التي يتم تفعيلها من بكتيريا معينة، اليورياز وحبات مغناطيسية. يتم استخدام DNAzyme باعتبارها العنصر الاعتراف الجزيئي لتحقيق الكشف محددة للغاية للبكتيريا من الفائدة. تستخدم اليورياز وحبات مغناطيسية لتحقيق نقل الإشارة من النشاط الحمض النووي الريبي انشقاق من DNAzyme. وهذا ينطوي على إنشاء حبات مغناطيسية التي تحتوي على تقارن اليورياز-DNAzyme. في وجود البكتيريا المستهدفة، وDNAzyme يشق لها الحمض النووي الريبي الربط. هذا الإجراء يؤدي إلى تفكك اليورياز من حبات مغناطيسية. واليورياز صدر يمكن فصلها بسهولة من حبات مغناطيسية وتستخدم لتوليد تغيير اللون في حل المراسل، الذي يحتوي على اليوريا وصبغة حساسة درجة الحموضة. اليورياز hydrolyzes اليوريا إلى الأمونيا، يرافقه زيادة درجة الحموضة الذي يقوم بتشغيل جتغيير OLOR للصباغة.
ويبين الشكل 2 اختبارا البكتيرية حيث EC1، وهو E. القولونية -responsive DNAzyme الشق RNA، كان يستخدم في DNAzyme، وكان يستخدم أحمر الفينول مثل صبغ الإبلاغ عن الرقم الهيدروجيني. وقد EC1 عزل من قبل مجموعتنا من مجموعة الحمض النووي العشوائي تسلسل باستخدام تقنية في المختبر الاختيار. 5 أظهرت دراساتنا السابقة أن EC1 غير محددة للغاية لE. القولونية ويسلك الحد الأدنى من النشاط تجاه البكتيريا الأخرى. 5،19 وقد وجد أن EC1 يتم تفعيلها من خلال جزيء البروتين من E. القولونية. على الرغم من أن هوية هذه العلامات البيولوجية البروتين لم يتم فك شفرتها، تقترح عالية التحديد الاعتراف بأن هذا البروتين فريد من نوعه لE. القولونية. تم تعيين حل مراسل حتى يكون لها درجة الحموضة الأولية من 5.5. وفي هذا الرقم الهيدروجيني، أحمر الفينول يسلك اللون الأصفر. كما اليورياز hydrolyzes اليوريا إلى الأمونيا، وقاعدية سول مراسلزيادات ution. ويتجلى ذلك من خلال التغيير التدريجي من اللون من الأصفر إلى الوردي. عمق تغيير لون يعتمد على المعلمتين التالية، كما يتضح من الشكل 2: عدد E. خلايا القولونية المستخدمة في تفعيل خطوة DNAzyme والوقت المسموح به للخطوة اليوريا التحلل. المزيد E. أسفرت خلايا القولونية في يتغير لون أقوى، التي تعكسها والمراقبة من E. الأصفر إلى الوردي تحول لون عندما التدريجي وزادت خلايا القولونية متسلسل 5-5 × 10 7 (10 أضعاف زيادة في كل مرة). وفي الوقت نفسه، وقتا أطول لالتحلل اليوريا يسمح للكشف عن أعداد أقل من E. خلايا القولونية (5000 الخلايا في رد فعل 1 ساعة و 500 الخلايا في رد فعل 2 ساعة).
تغيير الرقم الهيدروجيني للاختبارا البكتيرية يمكن أيضا رصد باستخدام متر المحمولة الرقم الهيدروجيني وأوضحت نتائج ممثلة في الشكل (3). وواالصورة وجدت أن وجود 10 7 E. أسفرت خلايا القولونية في زيادة تدريجية في درجة الحموضة بمقدار 3 وحدات في غضون 10 دقيقة. في المقابل، فإن غياب E. لم خلايا القولونية لا تسبب تغيرات درجة الحموضة للكشف تحت نفس الإعداد.
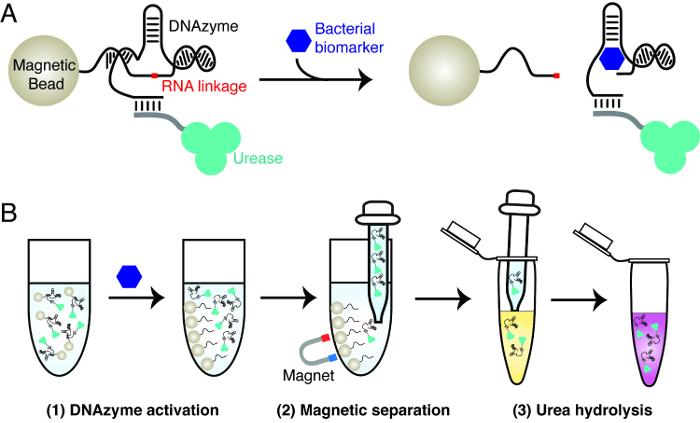
الشكل 1: مبدأ تصميم اختبارا البكتيري (A) تفعيل لDNAzyme الشق الحمض النووي الريبي العلامات البيولوجية محددة من بكتيريا من الفائدة. في وجود العلامات البيولوجية، وDNAzyme الشق RNA ثبتوا على يشق حبات مغناطيسية الربط RNA، مما أدى إلى الإفراج عن اليورياز الموسومة من حبات مغناطيسية إلى حل. (ب) ثلاث خطوات إجراء الفحص. الخطوة 1: تفعيل DNAzyme، كما هو موضح في لوحة A. الخطوة 2: الفصل المغناطيسي - يتم فصل اليورياز صدر من حبات مغناطيسية. الخطوة 3: اليوريا التحلل R12؛ وأضاف اليورياز صدر في حل مراسل التي تحتوي على اليوريا. اليورياز hydrolyzes اليوريا إلى الأمونيا، مما أدى إلى تغير في درجة الحموضة الذي يمكن الكشف عنه بواسطة صبغة حساسة درجة الحموضة. الرجاء انقر هنا لعرض نسخة أكبر من هذا الرقم.

الشكل 2: اختبار عباد الشمس مع E. القولونية E. باستخدام القولونية -responsive DNAzyme EC1. التمثيلية تغيير لون النتائج مع أعداد متفاوتة من E. خلايا القولونية الواردة أعلاه كل أنبوب اختبار. تم استخدام أحمر الفينول مثل صبغة حساسة درجة الحموضة. اختبار دون E. وقد استخدم القولونية كعنصر تحكم السلبية. المزيد E. ومن المتوقع أن تتسبب في إطلاق المزيد من الجزيئات اليورياز خلايا القولونية، يرافقه بذ يتغير لون أقوى. الرجاء انقر هنا لعرض نسخة أكبر من هذا الرقم.
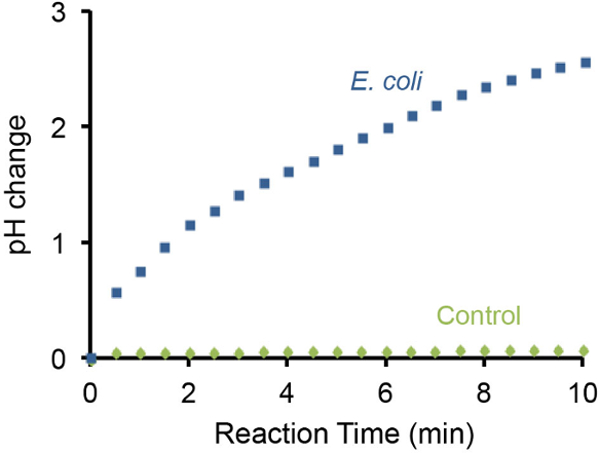
الشكل (3): رصد زيادة درجة الحموضة باستخدام مقياس درجة الحموضة وتغيير درجة الحموضة الناجمة عن 10 7 هاء. تم رصد خلايا القولونية باستخدام مقياس درجة الحموضة المحمولة. اختبار دون E. وقد استخدم القولونية كعنصر تحكم السلبية. وجود 10 7 E. خلايا القولونية في حل الاختبار يمكن أن تزيد من قاعدية من قبل وحدات ~ 3 درجة الحموضة في 10 دقيقة. الرجاء انقر هنا لعرض نسخة أكبر من هذا الرقم.
| اسم | سيquence (5'-3 ') | ملاحظة |
| BS1 | BTTTT TTTTT TTTAC TCTTC CTAGC FRQGG TTCGA TCAAG و | باء: 5'-البيوتين. R: ريبونوكليوتيد الأدينين. F: فلوريسئين من DT. س: dabcyl من DT |
| DE1 | GATGT GCGTT GTCGA GACCT GCGAC CGGAA CACTA CACTG TGTGG GGATG GATTT CTTTA CAGTT GTGTG TTGAA CGCTG TGTCA AAAAA AAAA | |
| T1 | GACAA CGCAC ATCTC TTGAT CGAAC C | |
| LD1 | XTTTT TTTTT TTTTT TTGAC ACAGC GTTCA و | X: 5'-NH 2 |
الجدول 1: متواليات من أليغنوكليوتيد الاصطناعية.
Discussion
يتم ترجمة العمل من النشاط النووي الريبي انشقاق من DNAzyme-البكتيريا تستجيب للاختبارا ممكن من خلال استخدام اليورياز والفصل المغناطيسي، كما يتضح من الشكل 1. على الرغم من أن مظاهرة من اختبارا تعديل للكشف عن البكتيريا هي القيام به مع E. القولونية معتمد على DNAzyme الشق RNA، 5،19،20 تصميم ويمكن تمديد عموما لأي DNAzyme الشق الحمض النووي الريبي. ونظرا لتوافر كبير من DNAzymes الشق RNA للالتحاليل المختلفة ومنهجيات مختلفة لعزل DNAzymes جديدة الشق الحمض النووي الريبي من حمامات العشوائي تسلسل للأهداف جديدة، ونحن نتوقع أن محك منصة الاختبار تعديل يمكن أن تمتد إلى الكشف عن أهداف متنوعة من الفائدة .
الاختبار الحقيقي لE. كشف القولونية يمكن الكشف عن 5000 و 500 خلايا عندما يتم تعيين وقت رد الفعل التقارير إلى أن يكون 1 و 2 ساعة، على التوالي. رد الفعل الشعبية البلمرة المتسلسل (PCR) وشطيرة انزيم مرتبط المناعي فحص (ELISA) أساليب يمكن أن تحقق حدود الكشف عن ما يقرب من 10 4 -10 5 هاء خلايا القولونية في الأوقات العصيبة مماثلة. 25،26 وهكذا، فإن الاختبار الحقيقي البكتيرية يقدم كشف حساسية قابلة للمقارنة.
على الرغم من أن الاختبار الحقيقي للبكتيريا من السهل القيام بها ويمكن أن تنتج تغييرات اللون نابضة بالحياة، يمكن أن العديد من العوامل تؤثر تأثيرا كبيرا على نتائج الاختبار. أولا، نوعية اليورياز مهم جدا. وقد استخدمنا اليورياز من مصادر مختلفة، وجدت نتائج الاختبار يمكن أن تختلف بشكل كبير. نحن نوصي باستخدام اليورياز من المصدر المحدد في قسم المواد.
يحتاج تجميع DNAzyme / اليورياز / حبات مغناطيسية اهتماما خاصا. الغسل الكامل من الخرز المغناطيسي لإزالة UrDNA الغير مهجنة ضروري لمنع نتائج إيجابية كاذبة. يحتاج إلى رعاية أيضا التي يجب اتخاذها لتجنب تراكم حبات مغناطيسية المتبقية على السطح الداخلي لللمعرف من microfuge أنبوب، والذي قد يكون من الصعب أن نرى. مرة واحدة هناك، لم تعد تخضع حبات مغناطيسية إلى الفصل المغناطيسي، وبالتالي، يمكن أن تحمل بعض UrDNA الغير مهجنة التي يمكن أن تؤدي إلى إشارات إيجابية كاذبة في رد فعل المراسل. ومن المهم أيضا لتجنب ترك microfuge أنبوب على الرف المغناطيسي لمدة أطول من 10 دقيقة خلال خطوة فصل المغناطيسية. حبات قد تجميع أو التمسك microfuge أنبوب، الأمر الذي قد يقلل من كفاءة الغسيل وإدخال التناقض دفعة إلى دفعة. إدراج 0.01٪ توين 20 في الحل غسل يمكن تحسين الاتساق دفعة إلى دفعة، وينبغي تنفيذه.
والمغلفة حبات مغناطيسية مع streptavidin، الذي كان يستخدم كمركز تجمع لتجميع تقارن DNAzyme-اليورياز على حبات مغناطيسية. كلا streptavidin واليورياز هي عبارة عن جزيئات البروتين التي يمكن التشويه والتحريف أثناء التخزين. ونحن عادة تخزين حبات DNAzyme-اليورياز المغناطيسي تجميعها في 4 درجة مئوية لمدة تصل إلى 4 أسابيعوجعل دفعات جديدة بشكل منتظم لتحقيق نتائج أكثر اتساقا.
يحتاج إلى عناية أيضا التي يجب اتخاذها لتجنب الخطأ أخذ حبات مغناطيسية في خطوة الفصل المغناطيسي (الخطوة 6.9) بعد تفعيل DNAzyme. من تجربتنا، والحطام الخلوي والجسيمات الأخرى في الحل يمكن أن تقلل من كفاءة الفصل المغناطيسي، وبالتالي، بعض حبات مغناطيسية يمكن أن تؤخذ عن غير قصد من خلال pipetting ل. وهذا يؤدي إلى نتائج إيجابية كاذبة. ونحن نوصي التدابير التالية للتخفيف من حدة المشكلة: وقتا أطول فصل (مثل 5-10 دقيقة)، بيان أبطأ من الضغط على ماصة للسماح للانسحاب لطيف من طاف، وإخضاع طاف لجولة إضافية من الفصل المغناطيسي .
وأخيرا، من المهم تجنب التلوث الحادث من محلول المخزون مراسل بواسطة اليورياز خلال التجربة حيث يتم اختبار عينات متعددة. ونظرا لتفاعل عالية من شrease، تلوث من هذا النوع يمكن أن يؤدي إلى نتائج إيجابية كاذبة.
Disclosures
The authors have nothing to disclose.
Acknowledgements
The funding for this research project was provided by the Natural Sciences and Engineering Research Council of Canada (NSERC) via a Discovery Grant to YL.
Materials
| Name | Company | Catalog Number | Comments |
| Ethylenediaminetetraacetic acid (EDTA) | VWR AMRESCO | 0105 | |
| Sodium Hydroxide (NaOH) pellets | BIO BASIC CANADA INC. | SB6789 | |
| Tris-base | VWR AMRESCO | 0497 | |
| Boric acid | AMRESCO | 0588 | |
| Urea | VWR AMRESCO | M123 | |
| 40% acrylamide/bisacrylamide (29:1) solution | BIO BASIC CANADA INC. | A0007 | |
| Sucrose | Bioshop Canada inc. | SUC507 | |
| Bromophenol blue | Bioshop Canada inc. | BRO777 | |
| Xylenecyanol FF | SIGMA-ALDRICH | X-4126 | |
| 10% sodium dodecyl sulfate | Bioshop Canada inc. | SDS001 | |
| Hydrochloric Acid (HCl) | CALEDON LABORATORIES LTD | 6026 | |
| Sodium Chloride (NaCl) | Bioshop Canada inc. | SOD001 | |
| 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid (HEPES) | Bioshop Canada inc. | HEP001 | |
| Magnesium Chloride (II) hexahydrate | VWR AMRESCO | 0288 | |
| Tween 20 | Bioshop Canada inc. | TW508 | |
| Adenosine Triphospahte (ATP) | AMRESCO | 0220 | |
| Sodium Acetate trihydrate (NaOAc) | SIGMA-ALDRICH | S8625 | |
| Ethanol | Commercial Alcohols | P016EAAN | |
| Tetramethyleneethylenediamine (TEMED) | AMRESCO | 0761 | |
| 10% Ammonium persulfate (APS) | BIO BASIC CANADA INC. | AB0072 | |
| Succinimidyl 4-(N-maleimidomethyl) cyclohexane-1-carboxylate (SMCC) | ThermoFisher SCIENTIFIC | 22360 | |
| Dimethyl sulfoxide (DMSO) | CALEDON LABORATORIES | 803540 | |
| Urease | SIGMA-ALDRICH | U0251 | |
| 1x Phosphate Buffered Saline (PBS) | ThermoFisher SCIENTIFIC | 70011-069 | |
| 0.04% Phenol red | SIGMA-ALDRICH | P3532 | |
| 10x T4 polynucleotide kinase reaction buffer | Lucigen | 30061-1 | |
| 10x T4 DNA ligase reaction buffer | Bio Basics Canada | B1122-B | |
| T4 DNA ligase (5 U/μl) | Thermo Fischer Scientific | B1122 | |
| Luria Bertani (LB) Broth | AMRESCO | J106 | |
| Agar | AMRESCO | J637 | |
| T4 polynucleotide kinase (10 U/μl) | Lucigen | 30061-1 | |
| E. coli K12 (MG1655) | ATCC | ATCC700926 | |
| Centrifuge | Beckman Coulter, Inc. | 392187 | |
| Glass plates | CBS scientific | ngp-250nr | |
| 0.75 mm thick spacers | CBS scientific | VGS-0725r | |
| 12-well comb | CBS scientific | VGC-7512 | |
| UV Lamp | UVP | 95-0017-09 | |
| Spectrophotometer (NanoVue) | GE Healthcare | N/A | |
| Metal plate | CBS scientific | CPA165-250 | |
| Vortex | VWR International | 58816-123 | |
| Gel electrophoresis apparatus | CBS scientific | ASG-250 | |
| Petri dishes | VWR International | 25384-342 | |
| 100 kDa MWCO centrifugal filters | EMD Millipore | UFC510024 | |
| Magnetic Bead (BioMag) | Bangs Laboratories Inc | BM568 | |
| Magnetic Seperation Rack | New England BioLabs | S1506S | |
| Microfuge tubes | Sarstedt | 72.69 | |
| Syringe filter (0.22 μm) | VWR International | 28145-501 | |
| 14 ml culture tube | VWR International | 60818-725 | |
| Cell culture incubator | Eppendorf Scientific | M13520000 | |
| Branson Ultrasonic cleaner | Branson | N/A | |
| Camera (Canon Powershot G11) | Canon | N/A | |
| 50 ml conical tube | VWR International | 89004-364 |
References
- Daar, A. S., et al. Top ten biotechnologies for improving health in developing countries. Nat. Genet. 32, 229-232 (2002).
- Newman, J. D., Turner, A. P. Home blood glucose biosensors: a commercial perspective. Biosens. Bioelectron. 20, 2435-2453 (2005).
- Turner, A. P. Biosensors: sense and sensibility. Chem. Soc. Rev. 42, 3184-3196 (2013).
- Tram, K., Kanda, P., Salena, B. J., Huan, S. Y., Li, Y. F. Translating Bacterial Detection by DNAzymes into a Litmus Test. Angew. Chem. Int. Ed. 53, 12799-12802 (2014).
- Ali, M. M., Aguirre, S. D., Lazim, H., Li, Y. Fluorogenic DNAzyme probes as bacterial indicators. Angew. Chem. Int. Ed. 50, 3751-3754 (2011).
- Schlosser, K., Li, Y. Biologically inspired synthetic enzymes made from DNA. Chem. Biol. 16, 311-322 (2009).
- Tuerk, C., Gold, L. Systematic evolution of ligand by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Breaker, R. R. Making catalytic DNAs. Science. 290, 2095-2096 (2000).
- Liu, J., Cao, Z., Lu, Y. Functional nucleic acid sensors. Chem. Rev. 109, 1948-1998 (2009).
- Navani, N. K., Li, Y. Nucleic acid aptamers and enzymes as sensors. Curr. Opin. Chem. Biol. 10, 272-281 (2006).
- Li, J., Lu, Y. A highly sensitive and selective catalytic DNA biosensor for lead ions. J. Am. Chem. Soc. 122, 10466-10467 (2000).
- Liu, J., Lu, Y. A colorimetric lead biosensor using DNAzyme-directed assembly of gold nanoparticles. J. Am. Chem. Soc. 125, 6642-6643 (2003).
- Liu, Z., Mei, S. H. J., Brennan, J. D., Li, Y. Assemblage of signaling DNA enzymes with intriguing metal specificity and pH dependence. J. Am. Chem. Soc. 125, 7539-7545 (2003).
- Liu, J., et al. A catalytic beacon sensor for uranium with parts-per-trillion sensitivity and millionfold selectivity. Proc. Natl. Acad. Sci. USA. 104, 2056-2061 (2007).
- Huang, P. J., Vazin, M., Liu, J. In vitro selection of a new lanthanide-dependent DNAzyme for ratiometric sensing lanthanides. Anal. Chem. 86, 9993-9999 (2014).
- Chiuman, W., Li, Y. Simple fluorescent sensors engineered with catalytic DNA 'MgZ' based on a non-classic allosteric design. PLoS One. 2, e1224(2007).
- Xiang, Y., Lu, Y. Using personal glucose meters and functional DNA sensors to quantify a variety of analytical targets. Nat. Chem. 3, 697-703 (2011).
- Aguirre, S. D., Ali, M. M., Salena, B. J., Li, Y. A sensitive DNA enzyme-based fluorescent assay for bacterial detection. Biomolecules. 3, 563-577 (2013).
- Aguirre, S. D., Ali, M. M., Kanda, P., Li, Y. F. Detection of Bacteria Using Fluorogenic DNAzymes. J. Vis. Exp. (63), e3961(2012).
- Shen, Z., et al. A catalytic DNA activated by a specific strain of bacterial pathogen. Angew. Chem. Int. Ed. 54, (2015).
- He, S., et al. Highly specific recognition of breast tumors by an RNA-cleaving fluorogenic DNAzyme probe. Anal. Chem. 87, 569-577 (2015).
- Sumner, J. B., Hand, D. B. Isoelectric point of crystalline urease. J. Am. Chem. Soc. 51, 1255-1260 (1929).
- Karplus, P. A., Pearson, M., Hausinger, R. P. 70 Years of crystalline urease: What have we learned. Acc. Chem. Res. 30, 330-337 (1997).
- Omiccioli, E., Amagliani, G., Brandi, G., Magnani, M. A new platform for Real-Time PCR detection of Salmonella spp., Listeria monocytogenes and Escherichia coli O157 in milk. Food Microbiol. 26, 615-622 (2009).
- Cui, S., Schroeder, C. M., Zhang, D. Y., Meng, J. Rapid sample preparation method for PCR-based detection of Escherichia coli O157:H7 in ground beef. J. Appl. Microbiol. 95, 129-134 (2003).
- Ibekwe, A. M., Watt, P. M., Grieve, C. M., Sharma, V. K., Lyons, S. R. Multiplex fluorogenic real-time PCR for detection and quantification of Escherichia coli O157:H7 in dairy wastewater wetlands. Appl. Environ. Microbiol. 68, 4853-4862 (2002).
- Strachan, N. J., Ogden, I. D. A sensitive microsphere coagulation ELISA for Escherichia coli O157:H7 using Russell's viper venom. FEMS Microbiol Lett. 186, 79-84 (2000).
- de Boer, E., Beumer, R. R. Methodology for detection and typing of foodborne microorganisms. Int. J. Food Microbiol. 50, 119-130 (1999).
- Gracias, K. S., McKillip, J. L. A review of conventional detection and enumeration methods for pathogenic bacteria in food. Can. J. Microbiol. 50, 883-890 (2004).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved