Method Article
リトマス試験を用いて細菌の比色検出
要約
We describe a protocol for colorimetric detection of E. coli using a modified litmus test that takes advantage of an RNA-cleaving DNAzyme, urease, and magnetic beads.
要約
There are increasing demands for simple but still effective methods that can be used to detect specific pathogens for point-of-care or field applications. Such methods need to be user-friendly and produce reliable results that can be easily interpreted by both specialists and non-professionals. The litmus test for pH is simple, quick, and effective as it reports the pH of a test sample via a simple color change. We have developed an approach to take advantage of the litmus test for bacterial detection. The method exploits a bacterium-specific RNA-cleaving DNAzyme to achieve two functions: recognizing a bacterium of interest and providing a mechanism to control the activity of urease. Through the use of magnetic beads immobilized with a DNAzyme-urease conjugate, the presence of bacteria in a test sample is relayed to the release of urease from beads to solution. The released urease is transferred to a test solution to hydrolyze urea into ammonia, resulting in an increase of pH that can be visualized using the classic litmus test.
概要
Bacterial pathogens are one of the major causes of global morbidity and mortality. Outbreaks from hospital-acquired infections, food-borne pathogens, and bacterial contaminants in the environment pose serious and on-going threats to public health and safety. To prevent these outbreaks, effective tools are needed that permit pathogen detection in a timely fashion under a variety of settings. Simple but still effective tests that are portable and cost-effective are greatly coveted, especially in regions that are susceptible to outbreaks but cannot afford expensive testing facilities.1-3 Although there exists a multitude of methods to detect bacteria, many of them are not suitable as screening or on-site testing tools because they require long test times, expensive instruments and complicated testing procedures.
Colorimetric tests are particularly attractive for point-of-care or field applications as color changes can be easily detected by the naked eye. The litmus test for pH is simple, quick, and effective. Although it is a very old technology, it is still widely used today because of its simplicity and effectiveness. Surprisingly, this simple test had never been modified to achieve the detection of other analytes before we recently developed an approach of modifying this test for E. coli testing.4
The expanded litmus test for E. coli employs three additional components: an E. coli activated RNA-cleaving DNAzyme (EC1), 5 urease, and magnetic beads. DNAzymes refer to synthetic single-stranded DNA molecules with catalytic activity.6 They can be isolated from random-sequence DNA pools using in vitro selection.7,8 They are highly stable and can be produced cost-effectively using high-efficiency automated DNA synthesis.9 For these reasons, DNAzymes, particularly RNA-cleaving DNAzymes, have been widely examined for biosensing applications.6,10,11 RNA-cleaving DNAzyme sensors have been developed to detect metal ions,12-16 small molecules,17,18 bacterial pathogens5,19-21 and cancer cells.22 Given the great availability of target-induced RNA-cleaving DNAzymes, any assay that utilizes a DNAzyme can be potentially expanded to detect a diverse range of analytes.
Urease is chosen for its ability to hydrolyze urea into ammonia,23,24 resulting in a pH increase. Urease is also highly efficient, stable and amenable for conjugation to other biomolecules. Therefore, we postulated that a conjugate of an RNA-cleavage DNAzyme with urease would allow the use of litmus test for the detection of other targets.5
The action of the RNA-cleaving DNAzyme is relayed to urease-mediated increase of pH through the use of magnetic beads that are immobilized with the DNAzyme-urease conjugate. Because the activity of the DNAzyme under investigation is strictly dependent on E. coli, the presence of this bacterium in the test solution will result in the release of urease from the magnetic beads to the solution, which is then taken and used to hydrolyze urea in a reporter solution that contains a pH-sensitive dye. The final outcome of this procedure is a color change that can be conveniently reported by the dye or pH paper.
プロトコル
試薬および緩衝液の調製
- 0.5 Mエチレンジアミン四酢酸(EDTA)
- 2リットルのビーカーで、脱イオン蒸留水800ミリリットル(のddH 2 O)に186.1グラムのEDTAを追加します。 NaOHペレットを用いて、8.0への溶液のpHを調整します。 1.0 Lの最終容量までのddH 2 Oを追加し、4℃でオートクレーブ処理し、ストアのオートクレーブ可能なガラス瓶にソリューションを転送します。
- 10×トリス - ホウ酸EDTA(10×TBE)
- 4 Lのプラスチックビーカーでは、すべてのコンポーネントまで攪拌棒を用いて、4 L.ミックスの最終容量432グラムトリスベース、200グラムのホウ酸、0.5 M EDTA(pHは8.0)とのddH 2 Oの80ミリリットルを追加します。溶解されます。 4℃でオートクレーブ処理し、ストアの1 Lのガラス瓶にソリューションを転送します。
- 10%変性ポリアクリルアミド証券
- 4 Lのプラスチックビーカー内で、1681.7グラムの尿素を追加し、400ミリリットル10倍TBE、40%アクリルアミド/ビスアクリルアミドの1 L(29:1)からfまでのソリューションとのddH 2 Oinalボリュームは、尿素が溶解するまで攪拌棒を用いて、4 L.ミックスです。 4℃で1 L琥珀色のガラスびんとストアにソリューションを転送します。
- 2倍ゲルローディングバッファー(2×GLB)
- 200ミリリットルのガラス製ビーカーに、44グラムの尿素、8グラムのスクロース、10mgのブロモフェノールブルー、10 mgのキシレンシアノールFF、10%ドデシル硫酸ナトリウム400μlの、および10×TBEの4ミリリットルを追加します。 40 mlの最終体積までのddH 2 Oを加え、磁気攪拌棒で混合しながら、50℃で穏やかに加熱して成分を溶解します。 4℃で1.5ミリリットルマイクロチューブや店舗への転送1ミリリットルのアリコート。
注:使用前には、任意の固形物を再溶解するために90℃で短時間加熱を必要とします。
- 200ミリリットルのガラス製ビーカーに、44グラムの尿素、8グラムのスクロース、10mgのブロモフェノールブルー、10 mgのキシレンシアノールFF、10%ドデシル硫酸ナトリウム400μlの、および10×TBEの4ミリリットルを追加します。 40 mlの最終体積までのddH 2 Oを加え、磁気攪拌棒で混合しながら、50℃で穏やかに加熱して成分を溶解します。 4℃で1.5ミリリットルマイクロチューブや店舗への転送1ミリリットルのアリコート。
- 1 Mトリス - 塩酸(トリス-HCl)(pH7.5)を
- ガラス瓶に、トリスベースの12.1グラムとのddH 2 Oの70ミリリットルを追加し、固体が溶解するまで混合します。 1 M塩酸(HCl)を使用してpHを7.5に調整します。オートクレーブSTO 100 mlの最終容量までのddH 2 Oを加えます4℃で再
- 5 M塩化ナトリウム(NaCl)で
- ガラス瓶に、のddH 2 Oの150ミリリットル中のNaClの58.4グラムを溶解ddH 2 Oで200mlにボリュームを調整します4℃で保存。
- DNA溶出バッファー
- ガラス瓶に、1 Mトリス塩酸(pH7.5)、8.0 5 MのNaCl mlおよび0.5 M EDTA 0.4mlの液(pH 8.0)2.0 mlと混合します。 ddH 2 O、4℃でオートクレーブや店舗で200mlにボリュームを調整します。
- 1 M 4-(2-ヒドロキシエチル)-1-ピペラジンエタンスルホン酸(HEPES)(pH7.4)で
- ガラス瓶に、のddH 2 O 80ml中HEPES 2.38グラム溶解5 N NaOHを用いてpHを7.4に調整し、100ミリリットルの最終容量までのddH 2 Oを追加します。
- 1 M塩化マグネシウム(MgCl 2を)
- ガラス瓶に、のMgCl 2・6H 2 Oの2.03グラムを追加し、のddH 2 Oで100mlにボリュームをもたらします
- 反応バッファー(RB)
- 50ミリリットルの円錐管で、1 M HEPES(pH7.4)で、5 M NaClを1.5ミリリットル、1 MのMgCl 2の0.75ミリリットル、およびTween-20の5μlに50μlを添加します。 50ミリリットルの最終体積のddH 2 Oを追加します。溶液を混合し、使用するまで4℃でシリンジ駆動フィルター(0.22μm)で、店舗を使用して別のコニカルチューブにフィルタリングします。
- 結合緩衝液(BB)
- 50ミリリットルコニカルチューブでは、1Mトリス塩酸(pH7.5)を500μl、NaClを8.8グラムを追加し、のMgCl 2の1 M50μlの、およびTween 20の5μlのは、最終容量までのddH 2 Oを追加します。 50ミリリットルの。溶液を混合し、使用するまで4℃でシリンジ駆動フィルター(0.22μm)で、店舗を使用して別のコニカルチューブにフィルタリングします。
- 基質溶液(SS)
- 50ミリリットルの円錐管では、NaClを5.8グラム、1 Mの3ミリリットルのMgCl 2、1.5グラムの尿素、およびのddH 2 Oの40ミリリットルを追加します。 10mMのHClを用いて5.0に溶液のpHを調整します。溶液が緩衝されていないため、塩酸を用いてpHを調整します慎重に小さな塩酸アリコートの添加によって。 50ミリリットルの最終体積のddH 2 Oを追加します。
- 溶液を混合し、使用するまで4℃でシリンジ駆動フィルター(0.22μm)で、店舗を使用して別のコニカルチューブにフィルタリングします。
- ルリア・ベルターニ(LB)ブロス
- ビーカーに、のddH 2 Oを1Lの20.0 gでLB粉末を溶解します4℃でガラスびん、オートクレーブ、およびストアに転送します。
- 1.5%LB寒天
- 250ミリリットルフラスコに、1.5グラム寒天とLBブロスの100ミリリットルを追加します。 4℃でオートクレーブや店舗。
- 寒天プレート
- 電子レンジでLB寒天を再溶解し、溶液を〜50℃に冷却します。 〜5のプレートを作り、ペトリ皿にソリューションを注ぎ、それらを固化することができます。
2.合成とEの精製coli-応答ザイムEC1
- テンプレート媒介酵素連結によりEC1の合成
- COMMを精製ercially合成されたオリゴヌクレオチドBS1、DE1、及びT1 10%が、標準的なプロトコルに従って、ポリアクリルアミドゲル電気泳動(dPAGE)を変性させることによって( 表1に提供された配列)。
- BS1、DE1、およびT1の100μMのストックを準備します。使用するまで-20℃で保管してください。
- 1.5mlマイクロチューブでは、酵素のサプライヤー(500 mMトリス塩酸(pHは7.6)、100 mMのMgCl 2をにより提供のddH 2 Oの38.5μlを、DE1を5μl、および10×T4ポリヌクレオチドキナーゼ反応緩衝液5μlを追加50mMのジチオスレイトール(DTT)、1.0 mMのスペルミジン)。
- アデノシン三リン酸(ATP)(100 mM)の1μLを加えます。 T4ポリヌクレオチドキナーゼ(10 U /μl)を5単位を追加します。慎重に反応混合物をピペットで混ぜます。
- 37℃で30分間反応をインキュベートします。
- 5分間90℃に加熱して反応をクエンチしました。
- ddH 2 Oの118μlを、BS1の5μlを、およびT1の5μlを添加します。 2マイルのために90°Cまで反応を加熱nは、その後、10分間かけて室温まで冷却。
- 酵素供給業者によって提供さ10×T4 DNAリガーゼ反応緩衝液20μl(400 mMトリス塩酸(pHは7.8)、100 mMのMgCl 2を、100mMのDTT、5mMのATP)を追加します。 T4 DNAリガーゼ10単位(5 U /μl)を加え、注意深くピペッティングにより混合します。
- 2時間室温でインキュベートします。
- 3 M酢酸ナトリウム(NaOAcを)(pHは5.2)を20μl、冷たい100%エタノール500μlのを追加します。ボルテックスにより溶液を混合し、30分間-20℃の冷凍庫にチューブを配置します。
- 遠心分離機、4℃で20分間20,000×gでマイクロフュージ。ピペットで注意深く上清を除去します。
- 4℃で10分間20,000×gで再び冷70%エタノール及び遠心分離機でペレットを洗浄。再度上清を除去し、10分間真空下でペレットを乾燥させます。
- ddH 2 Oの15μlのDNAを再懸濁した後、2×GLBの15μlを添加します。
- 90°Cに勢いよくボルテックスし、熱のために2分。後述のように10%dPAGEにより全長DNAを精製します。
- 10%dPAGEの設定
- きれいな2枚のガラス板(1フルプレートと1ノッチ付き)、2 0.75ミリメートルの厚さのスペーサー、および12ウェルコーム。 2それぞれの側にスペーサ及び上部に切り欠き板が平らな表面上の1つのガラス板を置きます。サプライヤーによって設けられた4つのクリップを使用して、2つのプレートをクリップ。
- 150 mlのプラスチックビーカーに、10%dPAGE 40mlを、テトラメチルエチレンジアミンの40μL(TEMED)、および10%の過硫酸アンモニウム(APS)の400μLを注ぎます。成分を混合し、ゆっくりとプレート間のソリューションを注ぎます。
- コームを挿入した後、ゲルを10分間かけて重合することができます。ゲルが重合された後、ゆっくりとコームを削除してのddH 2 Oでウェルをフラッシュ
- ゲル電気泳動装置にプレートをマウントします。過熱を防ぐために、発生した熱を放散するための金属板を使用します。
- 上部と下部のチャンバーに1×TBEを注ぎますウェルはよくバッファーに沈めていることを確認してください。ピペットまたはシリンジを使用して、1×TBEでウェルを洗浄します。
- サンプルをロードする前に15分間、35ミリアンペアと実行前に実行されるように装置を設定します。
- 10%dPAGEから連結したEC1の溶出
- 底色素(ブロモフェノールブルー)は、ガラス板の底部に到達するまで、ステップ2.2.6に続いて、35ミリアンペアでゲルを実行します。これは、約1.5時間を取る必要があります。電源をオフにして、装置からプレートを取り外します。
- 紙タオルの数枚のプレートを置き、慎重にガラス板からスペーサーを削除します。慎重トップガラス板を取り外し、ラップでゲルを包みます。
- 第二のガラスプレートを取り外して、再度ラップでカバーするためにゲルを裏返し。ラップのしわを避けるように注意してください。
- 4つの異なるDNAバンド(完全に連結したEC1、DE1、BS1、およびT1)を生成しますUVシャドーイング(260 nm)を用いて連結産物を可視化します。
- 消費税1.5mlマイクロチューブに滅菌カミソリの刃と転送にトップバンド(EC1)。それは細かいペースト状になるまで滅菌ピペットチップ(200μlの先端サイズ)を有するゲルをつぶします。 、DNAの溶出緩衝液550μLを加える埋め込み蛍光団を保護するためにアルミホイルでチューブをカバーし、そして15分間振とうします。
- 5分間20,000×gでゲル溶液を遠心し、慎重に別の1.5mlマイクロチューブに上清400μlのを転送します。ピペット操作中にゲル片を回収することは避けてください。
- 転送上清を持つマイクロチューブに冷たい100%エタノールの3 MのNaOAc(pHは5.2)の40μlを添加し、1.0ミリリットル。ボルテックスにより溶液を混合し、30分間-20℃の冷凍庫にチューブを配置します。
- 遠心分離機、4℃で20分間20,000×gでマイクロフュージ。ピペットで注意深く上清を除去します。
- 4℃で10分間20,000×gで再び冷70%エタノール及び遠心分離機でペレットを洗浄。もう一度、削除上清を、10分間真空下でペレットを乾燥させます。
- 260 nmのUV吸光度を測定することにより、EC1の濃度を決定します。 10μMのストックを作成し、使用するまで-20℃でサンプルを保存します。
DNAへのウレアーゼの3コンジュゲート
- スクシンイミジル4-(N-マレイミドメチル)シクロヘキサン-1-カルボキシレート(SMCC)ストックの調製
- DMSOの676μlの中1mgのSMCCを溶解させます。使用するまで氷上に渦と場所。
- ウレアーゼストックの調製
- 1×PBS(なしのMg 2+またはCa 2+)1mlに1mgのウレアーゼを溶解させます。使用するまで氷上に置いてください。
注:結晶化ウレアーゼを溶解するのに遅く、穏やかな混合は、変性を回避するために必要とされます。
- 1×PBS(なしのMg 2+またはCa 2+)1mlに1mgのウレアーゼを溶解させます。使用するまで氷上に置いてください。
- ウレアーゼ-DNAの合成(UrDNA)
- 2.5ミリリットルマイクロチューブに100μMLD1の10μLを加えます。 ddH 2 Oの140μlを、10×PBSを40μl、およびボルテックスを追加します。
- SMCC秒の80.5μlを添加しますタック、卓上遠心機を用いて、DMSO、渦、および短時間遠心機の159.5μlの。
- 60分間37℃でインキュベートします。マイクロフュージキャップの下に結露を避けてください。
- 1×PBS200μlの、3 MのNaOAc(pHは5.2)60μlの、冷100%エタノールの1.5ミリリットルを追加します。ボルテックスで溶液を混合し、30分間-20℃でインキュベートします。
- 遠心分離機、4℃で20分間20,000×gでソリューションを提供します。上清を除去し、真空下でペレットを乾燥させます。過乾燥は避けてください。
- 乾燥したペレットにウレアーゼ株式の400μlを添加し、5時間室温でインキュベートします。
- 予め洗浄100K MWCO遠心フィルターカラムに粗製の結合体の200μlのを転送します。 5分間、14,000×gでカラムを遠心。
- 5分間、14,000×gで、カラムと遠心への粗結合体の残りの200μlのを転送します。列を削除し、新しい2.0ミリリットルマイクロチューブ(「コレクションチューブ」)に逆さまに置きます。
- Centrif2分1,000×gで(反転列を持つ)コレクションチューブUGE。コレクションチューブを外し、コンジュゲートの追加の回復のために、膜を洗浄し、遠心カラムに1×PBSの30μlを添加します。
- もう一度列を反転し、コレクションチューブに戻す置きます。 2分1,000×gで遠心分離(逆列を持つ)コレクションチューブ。列を削除し、処分します。
- 使用するまで4℃でUrDNAを保管してください。
磁気ビーズ上にEC1とUrDNAの4アセンブリ
- よく磁気ビーズ(MB)の株式を混合し、1.5mlマイクロチューブにMB懸濁液100μlを移します。 MBを単離するための磁気ラックホルダーにマイクロチューブを置きます。
- ピペットで上清を除去し、チューブに結合バッファー(BB)の150μlを添加します。ホルダーからチューブを外し、慎重に均質な溶液にMBを再懸濁するためにチューブをタップします。トンにサスペンションをはね避けてくださいチューブまたはキャップのオペアンプ。この問題が発生した場合、簡単に戻って懸濁液に残留物をスピンする卓上型遠心分離機を使用しています。
- 繰り返しステップ4.2をさらに2回。
- この懸濁液に、10μMのEC1の10μlを添加します。慎重にチューブをタップして混ぜます。
- 30分間の穏やかな振とうで溶液をインキュベートします。 MBの沈殿を回避するために、チューブごとに2〜3分をタップします。
- MBを分離し、ピペッティングにより上清を除去するために戻って磁気ラック上のマイクロチューブを置きます。 (ステップ4.2で説明したように)BB150μlでMB 3回洗浄します。
- 洗濯が完了すると、BB150μlの合計でMBを一時停止します。この溶液に、UrDNAの15μlを添加し、2分間45℃にソリューションを加熱します。溶液を室温に冷却し、2時間インキュベートします。
- MBを分離し、ピペッティングにより上清を除去するために戻って磁気ラック上のマイクロチューブを置きます。
- 反応バッファー(RB)の100μLを加えます。トンを削除します彼は、磁気ラックからチューブを微量遠心し、慎重にMBを再懸濁します。
- ステップ4.9を繰り返して、MB 3回以上洗浄します。
注:洗浄上清をすぐに偽陽性シグナルをもたらし得る、ハイブリダイズしないUrDNAがまだ存在するかどうかを決定するために試験することができます。試験は、洗浄溶液に50mMの尿素及び0.04%フェノールレッドを10μlを10μl添加することによって行うことができます。洗浄液は、色の変化を起こさないまでMBを洗浄し続けます。 100μlの懸濁液を使用するまで4℃で保存されています。
細菌細胞20の5準備
- 培養E.株式から大腸菌
- プレートE.大腸菌 K12(MG1655)炎の下で、または生物学的安全キャビネット内のグリセロールストックからLB寒天プレート上に。
- LB寒天を穿刺を避けるために、寒天プレートの表面を滅菌したピペットチップを使用して、静かにグリセロールストックを触れ、軽く連勝。
- ストリークを反転プレートとは、14時間、37℃でインキュベートします。
- パラフィルムでプレートをシールし、3週間の最大のために4℃で保存します。
- 培養E.細胞の計数のための大腸菌
- 滅菌した14 mlの培養チューブに、LB培地を2mlを分注します。
- 滅菌したピペットチップを使用して、ステップ5.1で調製した画線寒天プレートから単一コロニーをピックアップし、培養チューブに移します。
- 37℃で培養物をインキュベートし、14時間、230 rpmで振とうします。
- 逐次10倍の間隔で細菌培養物を希釈します。
- 各希釈サンプルについて、均等に独立したLB寒天プレート上で5100μlアリコートをプレート。プレートを反転し、14時間、37℃でインキュベートします。
- 各希釈液の平均細胞濃度を得るために、各サンプルの細胞を数えます。
注:10 7細胞は、しばしばE.のための基準リトマス試験を設定するために使用されていますE.このレベルとして大腸菌 大腸菌は、迅速なカラーちゃんをトリガすることができますGE。結果セクションで説明したようにしかし、適切に実行リトマス試験は、と低い500個の細胞を検出することができます。
- E.の準備テストのための大腸菌細胞
- 目的の細胞懸濁液を、1.5mlマイクロチューブに培養された株式の譲渡1ミリリットルのため。
- 4℃で10分間、6000×gで細胞を遠心。慎重に細胞ペレットを乱すことなく、上清を除去します。
- 細胞ペレットに反応緩衝液10μlを加え、細胞を再懸濁。 5分間細胞懸濁液を超音波処理します。 5分間のアイスボックスに細胞懸濁液を転送します。
- さらに5分間、細胞懸濁液を超音波処理します。
- 4℃で10分間13,000×gで細胞懸濁液を遠心。テストのための上清(10μl)を使用してください。
6.リトマス試験
- 1.5mlマイクロチューブでは、マイクロチューブ中の反応バッファー(RB)の100μLを添加し、ボルテックスすることにより、チューブを予備洗浄し、バッファを破棄。
- 転送洗浄マイクロチューブに組み立てEC1(プロトコル4)15μlの。
- 磁気ラック上にマイクロチューブを配置することによって磁気ビーズを洗浄します。ピペットで上清を取り除きます。ラックからマイクロチューブを外し、RBの100μlを添加し、慎重に磁気ビーズを再懸濁します。
- ステップ6.3を繰り返して、MBさらに2回洗浄します。
- 磁気ラックにバックマイクロチューブを置き、上清を除去し、10μlのE.を追加大腸菌サンプルは、ステップ5.3から調製しました。
- 静かにマイクロチューブをタップして、慎重に試料及び磁性ビーズを混ぜます。
- 1時間室温で反応をインキュベートします。
- 反応に、のddH 2 Oの90μlを添加し、磁気ラック上にマイクロチューブを配置します。
- 後の磁気分離の約3分、慎重に0.5ミリリットルマイクロチューブに上清の85μLを移します。にゆっくりと上清を撤回任意の磁気ビーズを収集することは避けてください。
- 上記のマイクロチューブに0.04%のフェノールレッド、および100μlの基質溶液の15μlを添加します。
- 色の変化を記録するために特定の時間間隔で写真を撮ります。
注:pHの変化はまた、微小電極でpH計を用いてモニターすることができます。最初のpHは約5.2〜5.5でなければならない(溶液は黄色です)。そうでない場合、溶液は、1mMの酢酸緩衝液のpH 5.0)を添加することによって調整することができます。
結果
細菌のリトマス試験の原理は、図1で説明されたテストは、3つの重要な材料使用しています。特定の細菌、ウレアーゼおよび磁気ビーズによって活性化されるRNA切断DNAザイムを。 DNAザイムは、目的の細菌の高度に特異的な検出を達成するために、分子認識要素として使用されます。ウレアーゼおよび磁気ビーズをDNAザイムのRNA切断活性のシグナル伝達を達成するために使用されます。これは、ウレアーゼDNAザイムコンジュゲートを含む磁気ビーズを作成することを含みます。標的細菌の存在下で、DNAザイム切断のRNA結合。このアクションは、磁気ビーズからのウレアーゼの解離を生じさせます。放出ウレアーゼが簡単磁気ビーズから分離し、尿素およびpH感受性色素を含有するレポーター溶液の色変化を生成するために使用することができます。ウレアーゼは、cをトリガのpHの増加を伴う、アンモニアに尿素を加水分解する色素のolor変化。
図2は、細菌のリトマス試験EC1、Eを提示します大腸菌は、応答性RNA切断DNAザイムを、DNAザイムとして使用し、フェノールレッドは、pHレポーティング色素として使用しました。 EC1は、以前のin vitro選択技術を用いて、ランダム配列DNAプールから我々のグループによって単離した。5我々の以前の研究は、EC1はE.に対して高度に特異的であることを示しましたcoliおよび他の細菌に向かって最小の活性を示す。EC1はE.からタンパク質分子によって活性化されることが見出された5,19このタンパク質バイオマーカーの同定が解読されていないが大腸菌、高い認識の特異性は、このタンパク質がE.に対して一意であることを示唆しています大腸菌 。レポーター溶液は5.5の初期pHを持つように設定されています。このpHでは、フェノールレッドは、黄色を呈します。ウレアーゼとしては、アンモニアにレポーターゾルの塩基度を尿素を加水分解utionが増加します。これは、黄色からピンクに色の緩やかな変化によって反射されます。 図2に示すように、色の変化の深さは、次の二つのパラメータに依存します:E.数DNAザイムの活性化工程及び尿素加水分解工程のために許可された時間内に使用される大腸菌細胞 。詳細E. coli細胞は、プログレッシブ黄色〜ピンク色遷移Eの観察によって反射、強力な色の変化をもたらしました大腸菌細胞は、直列(10倍毎に増加)5×10 7、5から増加しました。一方、尿素加水分解のために長い時間がEのより少ない数の検出を許可します大腸菌細胞(2時間の反応で1時間の反応および500細胞中の5000細胞)。
細菌のリトマス試験のpH変化はまた、ハンドヘルドpH計を用いて監視することができ、代表的な結果を図3に示されている。これWAsが10 7 Eの存在することを発見しましたcoli細胞を、10分以内に3単位てpHの漸増をもたらしました。対照的に、Eの欠如大腸菌細胞は、同じ設定で検出可能なpH変化を引き起こしませんでした。
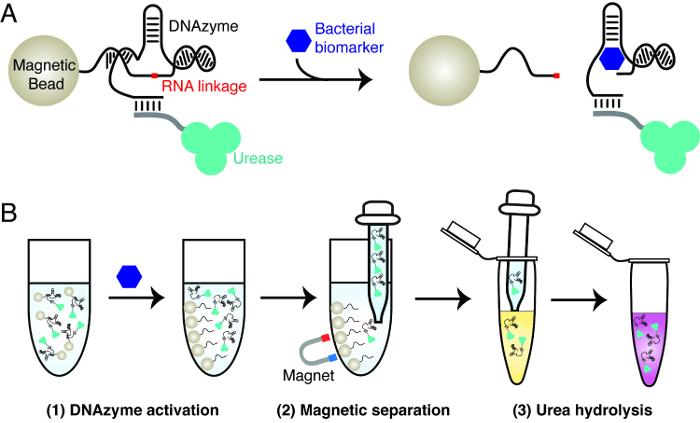
図1: 細菌のリトマス試験の設計原理関心の細菌からの特定のバイオマーカーによるRNA切断DNAザイムの(A)の活性化。バイオマーカーの存在下では、RNA切断DNAザイムは、溶液への磁気ビーズからタグ付けされたウレアーゼのリリースで、その結果、磁気ビーズを切断のRNA結合を固定化しました。 (B)三段階アッセイ手順。ステップ1:DNAザイム活性、パネルAの工程2に記載されているように:磁気分離 - 放出ウレアーゼを磁気ビーズから分離されています。ステップ3:尿素加水分解のR12;放出ウレアーゼは、尿素を含むレポーター溶液に添加します。ウレアーゼは、pH感受性色素によって報告することができpHの変化が生じ、アンモニアに尿素を加水分解する。 この図の拡大版をご覧になるにはこちらをクリックしてください。

図2:E. とリトマス試験 Eを 使用して 大腸菌 大腸菌 応答性DNAザイムEC1。E.の様々な数と代表色変化の結果各試験管の上方に設けられた大腸菌細胞 。フェノールレッドは、pH感受性色素として使用しました。 E.なしテスト大腸菌は、陰性対照として使用しました。詳細E.大腸菌細胞は、Bを伴って、より多くのウレアーゼ分子の放出を引き起こすことが予想されますyの強い色が変化します。 この図の拡大版をご覧になるにはこちらをクリックしてください。
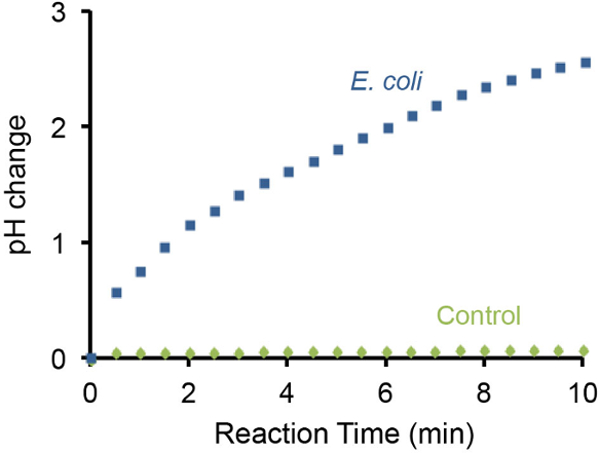
図3:pH メーターを用いてpHの上昇を監視 10 7 大腸菌によって引き起こされるpHの変化。 coli細胞は、携帯型pHメーターを用いてモニターしました。 E.なしテスト大腸菌は、陰性対照として使用しました。 10 7 Eの存在試験溶液中の大腸菌細胞は10分間で〜3のpH単位によって塩基性を向上させることができます。 この図の拡大版をご覧になるにはこちらをクリックしてください。
| 名 | セquence(5'-3 ') | 注意 |
| BS1 | BTTTT TTTTT TTTAC TCTTC CTAGC FRQGG TTCGA TCAAG A | B:5'-ビオチン; R:アデニンリボヌクレオチド; F:フルオレセイン-DT; Q:ダブシル-DT |
| DE1 | GATGT GCGTT GTCGA GACCT GCGAC CGGAA CACTA CACTG TGTGG GGATG GATTT CTTTA CAGTT GTGTG TTGAA CGCTG TGTCA AAAAA AAAA | |
| T1 | GACAA CGCAC ATCTC TTGAT CGAAC C | |
| LD1 | XTTTT TTTTT TTTTT TTGAC ACAGC GTTCA A | X:5'-NH 2 |
表1: 合成オリゴヌクレオチドの配列。
ディスカッション
図1に示すように、リトマス試験に細菌応答性DNAザイムのRNA切断活性の作用の翻訳は、ウレアーゼと磁気分離を使用することによって可能となる。細菌検出のための修正されたリトマス試験のデモンストレーションではあるがE.で行わ大腸菌依存性RNA切断DNAザイム、5,19,20デザインは、一般的に任意のRNA切断DNAザイムのために拡張することができます。異なる検体と新たな目標のためにランダム配列プールからの新しいRNA切断DNAザイムを単離するための様々な方法論のためのRNA切断DNAザイムの偉大な可用性を考えると、我々は修正されたリトマス試験プラットフォームは関心の多様な標的の検出に拡張することができることを期待します。
E.のためのリトマス試験報告反応時間は、それぞれ、1及び2時間に設定されている場合大腸菌検出は5,000と500の細胞を検出することができます。人気のポリメラーゼ連鎖反応(PCR)とサンドイッチ酵素結合免疫吸着アッセイ(ELISA)法は、約10 4〜10 5 Eの検出限界を達成することができます同様の試験時間の大腸菌細胞。25,26このように、細菌のリトマス試験は、同等の検出感度を提供しています。
細菌のリトマス試験を行うことが容易であり、鮮やかな色の変化を生成することができますが、いくつかの要因が大きく、試験結果に影響を与えることができます。まず、ウレアーゼの品質は非常に重要です。我々は、異なるソースからウレアーゼを使用し、試験結果が著しく変化することができる発見しました。我々は、材料のセクションで指定した送信元からのウレアーゼの使用をお勧めします。
DNAザイム/ウレアーゼ/磁気ビーズのアセンブリは特別な注意が必要です。ハイブリダイズしていないUrDNAを除去するために磁気ビーズを徹底的に洗浄は偽陽性の結果を防止する必要があります。注意はまた、Lの内側表面上の残留磁気ビーズの蓄積を避けるように注意する必要があります見えにくいかもしれマイクロチューブ、のID。そこから、磁気ビーズはもはや磁気分離に供されていないため、レポーター反応で偽陽性シグナルにつながることができますいくつかのハイブリダイズしていないUrDNAを運ぶことができます。磁気分離工程の間よりも長い10分間磁気ラック上のマイクロチューブを残さないようにすることも重要です。ビーズは凝集または洗浄効率を低下させ、バッチ間の矛盾を導入することができるマイクロチューブに付着することができます。洗浄溶液中の0.01%のTween-20を含めることは、バッチ間の一貫性を向上させることができ、実施されるべきです。
磁気ビーズは、磁気ビーズ上にDNAザイム、ウレアーゼ結合体を組み立てるためのアンカーとして使用されたストレプトアビジンでコーティングされています。ストレプトアビジンおよびウレアーゼの両方が貯蔵中に変性することができるタンパク質分子です。当社は通常4週間まで4℃で組み立てられたDNAザイム、ウレアーゼ磁気ビーズを保存しますそしてより一貫性のある結果を達成するために、定期的に新鮮なバッチを作ります。
ケアはまた、誤ってDNAザイムの活性化に続く磁気分離工程(ステップ6.9)に磁気ビーズを取って避けるように注意する必要があります。我々の経験から、溶液中の細胞破片および他の粒子は、磁気分離効率を低下させることができ、したがって、いくつかの磁気ビーズが意図せずにピペット中に取り出すことができます。これは、偽陽性の結果になります。我々は問題を軽減するために、以下の対策をお勧めします(例えば5〜10分など)より長い分離時間、ピペットの圧力のより遅い放出上清の穏やかな撤退を可能にするために、磁気分離の追加のラウンドに上清を施します。
最後に、多数のサンプルが試験される実験の経過中にウレアーゼによるレポーター原液の事故汚染を回避することが重要です。 Uの高い反応性を考えますrease、この種の汚染は偽陽性の結果につながることができます。
開示事項
The authors have nothing to disclose.
謝辞
The funding for this research project was provided by the Natural Sciences and Engineering Research Council of Canada (NSERC) via a Discovery Grant to YL.
資料
| Name | Company | Catalog Number | Comments |
| Ethylenediaminetetraacetic acid (EDTA) | VWR AMRESCO | 0105 | |
| Sodium Hydroxide (NaOH) pellets | BIO BASIC CANADA INC. | SB6789 | |
| Tris-base | VWR AMRESCO | 0497 | |
| Boric acid | AMRESCO | 0588 | |
| Urea | VWR AMRESCO | M123 | |
| 40% acrylamide/bisacrylamide (29:1) solution | BIO BASIC CANADA INC. | A0007 | |
| Sucrose | Bioshop Canada inc. | SUC507 | |
| Bromophenol blue | Bioshop Canada inc. | BRO777 | |
| Xylenecyanol FF | SIGMA-ALDRICH | X-4126 | |
| 10% sodium dodecyl sulfate | Bioshop Canada inc. | SDS001 | |
| Hydrochloric Acid (HCl) | CALEDON LABORATORIES LTD | 6026 | |
| Sodium Chloride (NaCl) | Bioshop Canada inc. | SOD001 | |
| 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid (HEPES) | Bioshop Canada inc. | HEP001 | |
| Magnesium Chloride (II) hexahydrate | VWR AMRESCO | 0288 | |
| Tween 20 | Bioshop Canada inc. | TW508 | |
| Adenosine Triphospahte (ATP) | AMRESCO | 0220 | |
| Sodium Acetate trihydrate (NaOAc) | SIGMA-ALDRICH | S8625 | |
| Ethanol | Commercial Alcohols | P016EAAN | |
| Tetramethyleneethylenediamine (TEMED) | AMRESCO | 0761 | |
| 10% Ammonium persulfate (APS) | BIO BASIC CANADA INC. | AB0072 | |
| Succinimidyl 4-(N-maleimidomethyl) cyclohexane-1-carboxylate (SMCC) | ThermoFisher SCIENTIFIC | 22360 | |
| Dimethyl sulfoxide (DMSO) | CALEDON LABORATORIES | 803540 | |
| Urease | SIGMA-ALDRICH | U0251 | |
| 1x Phosphate Buffered Saline (PBS) | ThermoFisher SCIENTIFIC | 70011-069 | |
| 0.04% Phenol red | SIGMA-ALDRICH | P3532 | |
| 10x T4 polynucleotide kinase reaction buffer | Lucigen | 30061-1 | |
| 10x T4 DNA ligase reaction buffer | Bio Basics Canada | B1122-B | |
| T4 DNA ligase (5 U/μl) | Thermo Fischer Scientific | B1122 | |
| Luria Bertani (LB) Broth | AMRESCO | J106 | |
| Agar | AMRESCO | J637 | |
| T4 polynucleotide kinase (10 U/μl) | Lucigen | 30061-1 | |
| E. coli K12 (MG1655) | ATCC | ATCC700926 | |
| Centrifuge | Beckman Coulter, Inc. | 392187 | |
| Glass plates | CBS scientific | ngp-250nr | |
| 0.75 mm thick spacers | CBS scientific | VGS-0725r | |
| 12-well comb | CBS scientific | VGC-7512 | |
| UV Lamp | UVP | 95-0017-09 | |
| Spectrophotometer (NanoVue) | GE Healthcare | N/A | |
| Metal plate | CBS scientific | CPA165-250 | |
| Vortex | VWR International | 58816-123 | |
| Gel electrophoresis apparatus | CBS scientific | ASG-250 | |
| Petri dishes | VWR International | 25384-342 | |
| 100 kDa MWCO centrifugal filters | EMD Millipore | UFC510024 | |
| Magnetic Bead (BioMag) | Bangs Laboratories Inc | BM568 | |
| Magnetic Seperation Rack | New England BioLabs | S1506S | |
| Microfuge tubes | Sarstedt | 72.69 | |
| Syringe filter (0.22 μm) | VWR International | 28145-501 | |
| 14 ml culture tube | VWR International | 60818-725 | |
| Cell culture incubator | Eppendorf Scientific | M13520000 | |
| Branson Ultrasonic cleaner | Branson | N/A | |
| Camera (Canon Powershot G11) | Canon | N/A | |
| 50 ml conical tube | VWR International | 89004-364 |
参考文献
- Daar, A. S., et al. Top ten biotechnologies for improving health in developing countries. Nat. Genet. 32, 229-232 (2002).
- Newman, J. D., Turner, A. P. Home blood glucose biosensors: a commercial perspective. Biosens. Bioelectron. 20, 2435-2453 (2005).
- Turner, A. P. Biosensors: sense and sensibility. Chem. Soc. Rev. 42, 3184-3196 (2013).
- Tram, K., Kanda, P., Salena, B. J., Huan, S. Y., Li, Y. F. Translating Bacterial Detection by DNAzymes into a Litmus Test. Angew. Chem. Int. Ed. 53, 12799-12802 (2014).
- Ali, M. M., Aguirre, S. D., Lazim, H., Li, Y. Fluorogenic DNAzyme probes as bacterial indicators. Angew. Chem. Int. Ed. 50, 3751-3754 (2011).
- Schlosser, K., Li, Y. Biologically inspired synthetic enzymes made from DNA. Chem. Biol. 16, 311-322 (2009).
- Tuerk, C., Gold, L. Systematic evolution of ligand by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Breaker, R. R. Making catalytic DNAs. Science. 290, 2095-2096 (2000).
- Liu, J., Cao, Z., Lu, Y. Functional nucleic acid sensors. Chem. Rev. 109, 1948-1998 (2009).
- Navani, N. K., Li, Y. Nucleic acid aptamers and enzymes as sensors. Curr. Opin. Chem. Biol. 10, 272-281 (2006).
- Li, J., Lu, Y. A highly sensitive and selective catalytic DNA biosensor for lead ions. J. Am. Chem. Soc. 122, 10466-10467 (2000).
- Liu, J., Lu, Y. A colorimetric lead biosensor using DNAzyme-directed assembly of gold nanoparticles. J. Am. Chem. Soc. 125, 6642-6643 (2003).
- Liu, Z., Mei, S. H. J., Brennan, J. D., Li, Y. Assemblage of signaling DNA enzymes with intriguing metal specificity and pH dependence. J. Am. Chem. Soc. 125, 7539-7545 (2003).
- Liu, J., et al. A catalytic beacon sensor for uranium with parts-per-trillion sensitivity and millionfold selectivity. Proc. Natl. Acad. Sci. USA. 104, 2056-2061 (2007).
- Huang, P. J., Vazin, M., Liu, J. In vitro selection of a new lanthanide-dependent DNAzyme for ratiometric sensing lanthanides. Anal. Chem. 86, 9993-9999 (2014).
- Chiuman, W., Li, Y. Simple fluorescent sensors engineered with catalytic DNA 'MgZ' based on a non-classic allosteric design. PLoS One. 2, e1224(2007).
- Xiang, Y., Lu, Y. Using personal glucose meters and functional DNA sensors to quantify a variety of analytical targets. Nat. Chem. 3, 697-703 (2011).
- Aguirre, S. D., Ali, M. M., Salena, B. J., Li, Y. A sensitive DNA enzyme-based fluorescent assay for bacterial detection. Biomolecules. 3, 563-577 (2013).
- Aguirre, S. D., Ali, M. M., Kanda, P., Li, Y. F. Detection of Bacteria Using Fluorogenic DNAzymes. J. Vis. Exp. (63), e3961(2012).
- Shen, Z., et al. A catalytic DNA activated by a specific strain of bacterial pathogen. Angew. Chem. Int. Ed. 54, (2015).
- He, S., et al. Highly specific recognition of breast tumors by an RNA-cleaving fluorogenic DNAzyme probe. Anal. Chem. 87, 569-577 (2015).
- Sumner, J. B., Hand, D. B. Isoelectric point of crystalline urease. J. Am. Chem. Soc. 51, 1255-1260 (1929).
- Karplus, P. A., Pearson, M., Hausinger, R. P. 70 Years of crystalline urease: What have we learned. Acc. Chem. Res. 30, 330-337 (1997).
- Omiccioli, E., Amagliani, G., Brandi, G., Magnani, M. A new platform for Real-Time PCR detection of Salmonella spp., Listeria monocytogenes and Escherichia coli O157 in milk. Food Microbiol. 26, 615-622 (2009).
- Cui, S., Schroeder, C. M., Zhang, D. Y., Meng, J. Rapid sample preparation method for PCR-based detection of Escherichia coli O157:H7 in ground beef. J. Appl. Microbiol. 95, 129-134 (2003).
- Ibekwe, A. M., Watt, P. M., Grieve, C. M., Sharma, V. K., Lyons, S. R. Multiplex fluorogenic real-time PCR for detection and quantification of Escherichia coli O157:H7 in dairy wastewater wetlands. Appl. Environ. Microbiol. 68, 4853-4862 (2002).
- Strachan, N. J., Ogden, I. D. A sensitive microsphere coagulation ELISA for Escherichia coli O157:H7 using Russell's viper venom. FEMS Microbiol Lett. 186, 79-84 (2000).
- de Boer, E., Beumer, R. R. Methodology for detection and typing of foodborne microorganisms. Int. J. Food Microbiol. 50, 119-130 (1999).
- Gracias, K. S., McKillip, J. L. A review of conventional detection and enumeration methods for pathogenic bacteria in food. Can. J. Microbiol. 50, 883-890 (2004).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved