Method Article
Colorimetrico rilevamento di batteri Uso tornasole Test
In questo articolo
Riepilogo
We describe a protocol for colorimetric detection of E. coli using a modified litmus test that takes advantage of an RNA-cleaving DNAzyme, urease, and magnetic beads.
Abstract
There are increasing demands for simple but still effective methods that can be used to detect specific pathogens for point-of-care or field applications. Such methods need to be user-friendly and produce reliable results that can be easily interpreted by both specialists and non-professionals. The litmus test for pH is simple, quick, and effective as it reports the pH of a test sample via a simple color change. We have developed an approach to take advantage of the litmus test for bacterial detection. The method exploits a bacterium-specific RNA-cleaving DNAzyme to achieve two functions: recognizing a bacterium of interest and providing a mechanism to control the activity of urease. Through the use of magnetic beads immobilized with a DNAzyme-urease conjugate, the presence of bacteria in a test sample is relayed to the release of urease from beads to solution. The released urease is transferred to a test solution to hydrolyze urea into ammonia, resulting in an increase of pH that can be visualized using the classic litmus test.
Introduzione
Bacterial pathogens are one of the major causes of global morbidity and mortality. Outbreaks from hospital-acquired infections, food-borne pathogens, and bacterial contaminants in the environment pose serious and on-going threats to public health and safety. To prevent these outbreaks, effective tools are needed that permit pathogen detection in a timely fashion under a variety of settings. Simple but still effective tests that are portable and cost-effective are greatly coveted, especially in regions that are susceptible to outbreaks but cannot afford expensive testing facilities.1-3 Although there exists a multitude of methods to detect bacteria, many of them are not suitable as screening or on-site testing tools because they require long test times, expensive instruments and complicated testing procedures.
Colorimetric tests are particularly attractive for point-of-care or field applications as color changes can be easily detected by the naked eye. The litmus test for pH is simple, quick, and effective. Although it is a very old technology, it is still widely used today because of its simplicity and effectiveness. Surprisingly, this simple test had never been modified to achieve the detection of other analytes before we recently developed an approach of modifying this test for E. coli testing.4
The expanded litmus test for E. coli employs three additional components: an E. coli activated RNA-cleaving DNAzyme (EC1), 5 urease, and magnetic beads. DNAzymes refer to synthetic single-stranded DNA molecules with catalytic activity.6 They can be isolated from random-sequence DNA pools using in vitro selection.7,8 They are highly stable and can be produced cost-effectively using high-efficiency automated DNA synthesis.9 For these reasons, DNAzymes, particularly RNA-cleaving DNAzymes, have been widely examined for biosensing applications.6,10,11 RNA-cleaving DNAzyme sensors have been developed to detect metal ions,12-16 small molecules,17,18 bacterial pathogens5,19-21 and cancer cells.22 Given the great availability of target-induced RNA-cleaving DNAzymes, any assay that utilizes a DNAzyme can be potentially expanded to detect a diverse range of analytes.
Urease is chosen for its ability to hydrolyze urea into ammonia,23,24 resulting in a pH increase. Urease is also highly efficient, stable and amenable for conjugation to other biomolecules. Therefore, we postulated that a conjugate of an RNA-cleavage DNAzyme with urease would allow the use of litmus test for the detection of other targets.5
The action of the RNA-cleaving DNAzyme is relayed to urease-mediated increase of pH through the use of magnetic beads that are immobilized with the DNAzyme-urease conjugate. Because the activity of the DNAzyme under investigation is strictly dependent on E. coli, the presence of this bacterium in the test solution will result in the release of urease from the magnetic beads to the solution, which is then taken and used to hydrolyze urea in a reporter solution that contains a pH-sensitive dye. The final outcome of this procedure is a color change that can be conveniently reported by the dye or pH paper.
Protocollo
1. Preparazione dei reagenti e tamponi
- 0,5 M Acido etilendiamminotetraacetico (EDTA)
- In un becher 2 L, aggiungere 186,1 g di EDTA a 800 ml di acqua deionizzata distillata (DDH 2 O). Regolare il pH della soluzione a 8,0 con NaOH pellets. Aggiungere DDH 2 O ad un volume finale di 1,0 L e trasferire la soluzione in una bottiglia di vetro autoclavabile per autoclave e conservare a 4 ° C.
- EDTA 10 × Tris-borato (10x TBE)
- In un becher di plastica 4 L, aggiungere 432 g di Tris-base, acido borico 200 g, 80 ml di 0,5 M EDTA (pH 8,0) e DDH 2 O per un volume finale di 4 L. Mescolare con ancoretta finché tutti i componenti sono disciolti. Trasferire la soluzione in bottiglie di vetro da 1 litro per la sterilizzazione in autoclave e conservare a 4 ° C.
- 10% di poliacrilammide denaturante della
- In un becher di plastica 4 L, aggiungere 1681,7 g di urea, 400 ml 10x TBE, 1 L di 40% di acrilammide / bisacrilammide (29: 1) e soluzione DDH 2 O fino alla fil volume inale è 4 L. Mescolare con un ancoretta fino a quando l'urea viene sciolta. Trasferire la soluzione in bottiglie di vetro ambrato 1 L e conservare a 4 ° C.
- 2x Gel Caricamento Buffer (2x GLB)
- In un bicchiere di vetro da 200 ml, aggiungere 44 g di urea, 8 g di saccarosio, 10 mg blu di bromofenolo, 10 mg xilene cianolo FF, 400 ml di 10% sodio dodecil solfato e 4 ml di 10 × TBE. Aggiungere DDH 2 O per un volume finale di 40 ml e sciogliere i componenti con blando riscaldamento a 50 ° C sotto agitazione con ancoretta magnetica. Trasferire 1 ml aliquota provette da 1,5 ml microcentrifuga e conservare a 4 ° C.
NOTA: l'uso Prima richiede breve riscaldamento a 90 ° C per riportare in soluzione una qualsiasi solido.
- In un bicchiere di vetro da 200 ml, aggiungere 44 g di urea, 8 g di saccarosio, 10 mg blu di bromofenolo, 10 mg xilene cianolo FF, 400 ml di 10% sodio dodecil solfato e 4 ml di 10 × TBE. Aggiungere DDH 2 O per un volume finale di 40 ml e sciogliere i componenti con blando riscaldamento a 50 ° C sotto agitazione con ancoretta magnetica. Trasferire 1 ml aliquota provette da 1,5 ml microcentrifuga e conservare a 4 ° C.
- 1 M Tris-cloridrato (Tris-HCl) (pH 7.5)
- In una bottiglia di vetro, aggiungere 12,1 g di Tris-base e 70 ml di DDH 2 O e miscelare fino a che il solido è sciolto. Aggiustare il pH a 7,5 con 1 M acido cloridrico (HCl). Aggiungere DDH 2 O per un volume finale di 100 ml, autoclave e store a 4 ° C
- 5 M cloruro di sodio (NaCl)
- In una bottiglia di vetro, sciogliere 58,4 g di NaCl in 150 ml di DDH 2 O. Regolare il volume a 200 ml con DDH 2 O. Conservare a 4 ° C.
- DNA Elution Buffer
- In una bottiglia di vetro, mescolare 2,0 ml di 1 M Tris-HCl (pH 7,5), 8,0 ml di 5 M NaCl e 0,4 ml di 0,5 M EDTA (pH 8,0). Regolare il volume a 200 ml con DDH 2 O, autoclave e conservare a 4 ° C.
- 1 M 4- (2-idrossietil) -1-acido piperazineethanesulfonic (HEPES) (pH 7,4)
- In una bottiglia di vetro, sciogliere 2,38 g di HEPES in 80 ml di DDH 2 O. Portare il pH a 7,4 con NaOH 5 N e aggiungere DDH 2 O per un volume finale di 100 ml.
- 1 M cloruro di magnesio (MgCl 2)
- In una bottiglia di vetro, aggiungere 2,03 g di MgCl 2 -6h 2 O e portare il volume a 100 ml con DDH 2 O.
- Reaction Buffer (RB)
- In una provetta conica da 50 ml, aggiungere 50 ml di 1 M HEPES (pH 7,4), 1,5 ml di 5 M NaCl, 0,75 ml di 1 M MgCl 2, e 5 ml di Tween-20. Aggiungere DDH 2 O per un volume finale di 50 ml. Mescolare la soluzione e filtrare attraverso un altro tubo conico usando un filtro a siringa condotto (0,22 micron) e conservare a 4 ° C fino all'utilizzo.
- Binding Buffer (BB)
- In una provetta conica da 50 ml, aggiungere 500 ml di 1 M Tris-HCl (pH 7,5), 8,8 g di NaCl, 50 ml di 1 M di MgCl 2, e 5 ml di Tween 20. Aggiungere DDH 2 O ad un volume finale di 50 ml. Mescolare la soluzione e filtrare attraverso un altro tubo conico usando un filtro a siringa condotto (0,22 micron) e conservare a 4 ° C fino all'utilizzo.
- Soluzione di substrato (SS)
- In una provetta conica da 50 ml, aggiungere 5,8 g di NaCl, 3 ml di 1 M MgCl 2, 1,5 g di urea, e 40 ml di DDH 2 O. Regolare il pH della soluzione a 5,0 con HCl 10 mM. Poiché la soluzione non viene tamponata, regolare il pH utilizzando HClattenzione attraverso l'aggiunta di piccole aliquote HCl. Aggiungere DDH 2 O per un volume finale di 50 ml.
- Mescolare la soluzione e filtrare attraverso un altro tubo conico usando un filtro a siringa condotto (0,22 micron) e conservare a 4 ° C fino all'utilizzo.
- Luria Bertani (LB) Broth
- In un bicchiere, sciogliere 20,0 g LB polvere in 1 l di DDH 2 O. Trasferimento in una bottiglia di vetro, autoclave, e conservare a 4 ° C.
- 1,5% LB agar
- In un pallone da 250 ml, aggiungere 1,5 g agar e 100 ml di brodo LB. Autoclave e conservare a 4 ° C.
- piastre di agar
- Riprendere il agar LB in un forno a microonde e raffreddare la soluzione a ~ 50 ° C. Versare la soluzione in capsule di Petri, rendendo ~ 5 piatti e permettere loro di solidificare.
2. Sintesi e purificazione di E. coli- reattivo DNAzyme EC1
- Sintesi di EC1 by Template Mediated enzimatica legatura
- purificare commoligonucleotidi sintetizzati ercially BS1, DE1 e T1 (sequenze fornite nella Tabella 1) del 10% denaturazione elettroforesi su gel di poliacrilammide (dPAGE) secondo protocolli standard.
- Preparare una 100 micron magazzino di BS1, DE1, e T1. Conservare a -20 ° C fino al momento dell'uso.
- In una provetta da 1,5 ml microcentrifuga, aggiungere 38,5 ml di DDH 2 O, 5 ml di DE1, e 5 ml di 10x T4 tampone di reazione polinucleotide chinasi forniti dal fornitore enzima (500 mm Tris-HCl (pH 7,6), 100 mM MgCl 2, 50 mM ditiotreitolo (DTT), 1,0 mm spermidina).
- Aggiungere 1 ml di adenosina trifosfato (ATP) (100 mM). Aggiungere 5 unità di T4 polinucleotide chinasi (10 U / mL). Mescolare pipettando accuratamente la miscela di reazione.
- Incubare la reazione a 37 ° C per 30 min.
- Quench la reazione mediante riscaldamento a 90 ° C per 5 min.
- Aggiungere 118 ml di DDH 2 O, 5 ml di BS1 e 5 ml di T1. Riscaldare la reazione a 90 ° C per 2 min e poi raffreddare a temperatura ambiente superiore a 10 min.
- Aggiungere 20 ml di 10 × T4 DNA ligasi tampone di reazione fornito dal fornitore enzima (400 mM Tris-HCl (pH 7,8), 100 mM MgCl 2, 100 mM DTT, 5 mM ATP). Aggiungere 10 unità di T4 DNA ligasi (5 U / mL) e con attenzione mescolare pipettando.
- Incubare a temperatura ambiente per 2 ore.
- Aggiungere 20 ml di acetato di 3 M di sodio (NaOAc) (pH 5,2), 500 ml di etanolo al 100% freddo. Mescolare la soluzione con vortex e posizionare il tubo in un congelatore C -20 ° per 30 min.
- Centrifugare la microcentrifuga a 20.000 xg per 20 minuti a 4 ° C. rimuovere con attenzione il surnatante pipettando.
- Lavare il pellet con freddo al 70% di etanolo e centrifugare nuovamente a 20.000 xg per 10 min a 4 ° C. Una volta rimuovere il supernatante e asciugare il pellet sotto vuoto per 10 min.
- Risospendere il DNA con 15 ml di DDH 2 O e quindi aggiungere 15 ml di 2x GLB.
- Vortex vigorosamente e si riscalda a 90 ° C per2 min. Purificare il DNA integrale del 10% dPAGE come descritto di seguito.
- Impostazione 10% dPAGE
- piastre pulite due di vetro (un piatto pieno e uno dentellati), due da 0,75 mm di spessore, distanziali e un pettine 12-pozzetti. Lay uno lastra di vetro su una superficie piana con due distanziali su ogni lato e la piastra dentata sopra. Agganciare le due piastre insieme con le quattro clip forniti dal fornitore.
- In un becher di plastica 150 ml, versare 40 ml di 10% dPAGE, 40 ml di tetrametiletilendiammina (TEMED), e 400 ml di persolfato di ammonio 10% (APS). Miscelare i componenti e versare la soluzione tra le piastre lentamente.
- Inserire il pettine e quindi consentire il gel di polimerizzare su 10 min. Una volta che il gel è polimerizzato, rimuovere lentamente il pettine e lavare i pozzetti con DDH 2 O.
- Montare le piastre su dell'apparato elettroforesi su gel. Utilizzare una piastra metallica per dissipare il calore generato per evitare il surriscaldamento.
- Versare TBE 1x sulle camere superiore e inferioree garantire che i pozzi sono ben immersi nel buffer. Lavare i pozzetti con 1x TBE con una pipetta o una siringa.
- Impostare l'apparecchiatura per l'esecuzione a 35 mA e pre-corsa per 15 minuti prima caricato i campioni.
- Eluizione di legatura EC1 dal 10% dPAGE
- Seguendo passo 2.2.6, eseguire il gel a 35 mA fino colorante inferiore (blu di bromofenolo) raggiunge il fondo della piastra di vetro. Questo dovrebbe prendere circa 1,5 ore. Spegnere l'alimentazione e rimuovere le piastre dall'apparecchio.
- Posare le lastre su alcuni fogli di carta assorbente e rimuovere con attenzione i distanziatori dalle lastre di vetro. Rimuovere con cura la lastra di vetro superiore e avvolgere il gel con pellicola trasparente.
- Capovolgere il gel sopra per rimuovere la seconda lastra di vetro e coprire con pellicola trasparente di nuovo. Fare attenzione a evitare le rughe della pellicola trasparente.
- Visualizza il prodotto legatura utilizzando UV shadowing (260 nm), che produrrà 4 bande di DNA distinte (EC1 completamente legatura, DE1, BS1 e T1).
- Excise la fascia superiore (EC1) con una lama di rasoio sterile e trasferimento in una provetta da 1,5 ml microcentrifuga. Schiacciare il gel con una punta di pipetta sterile (200 dimensioni punta ml) fino a quando non si trasforma in una pasta fine. Aggiungere 550 pl di tampone di eluizione DNA, coprire il tubo con un foglio di alluminio per proteggere il fluoroforo incorporato e agitare per 15 min.
- Centrifugare la soluzione di gel a 20.000 xg per 5 minuti e trasferire con cautela 400 ml di surnatante in un'altra provetta da 1,5 ml microcentrifuga. Evitare il ritiro pezzi di gel durante il pipettaggio.
- Per la provetta per microcentrifuga con il surnatante trasferito aggiungere 40 ml di 3 M NaOAc (pH 5.2) e 1,0 ml di freddo 100% di etanolo. Mescolare la soluzione con vortex e posizionare il tubo in un congelatore C -20 ° per 30 min.
- Centrifugare la microcentrifuga a 20.000 xg per 20 minuti a 4 ° C. rimuovere con attenzione il surnatante pipettando.
- Lavare il pellet con freddo al 70% di etanolo e centrifugare nuovamente a 20.000 xg per 10 min a 4 ° C. Ancora una volta, rimuovere lasupernatante e asciugare il pellet sotto vuoto per 10 min.
- Determinare la concentrazione di EC1 misurando l'assorbanza UV a 260 nm. Fare un 10 mM magazzino e conservare il campione a -20 ° C fino al momento dell'uso.
3. Coniugazione del Ureasi al DNA
- Preparazione di succinimidyl 4- (N-maleimidomethyl) cicloesano-1-carbossilato (SMCC) Stock
- Sciogliere 1 mg SMCC in 676 ml di DMSO. Vortex e posto sul ghiaccio fino al momento dell'uso.
- Preparazione di Ureasi della
- Sciogliere 1 mg ureasi in 1 ml di 1 × PBS (senza Mg 2+ o Ca 2+). Mettere in ghiaccio fino al momento dell'uso.
NOTA: cristallizzato ureasi è lenta a dissolversi ed è necessario delicata miscelazione per evitare la denaturazione.
- Sciogliere 1 mg ureasi in 1 ml di 1 × PBS (senza Mg 2+ o Ca 2+). Mettere in ghiaccio fino al momento dell'uso.
- Sintesi di Ureasi-DNA (UrDNA)
- Aggiungere 10 ml di 100 mM LD1 in una provetta da 2,5 ml microcentrifuga. Aggiungere 140 ml di DDH 2 O, 40 ml di PBS 10x, e vortice.
- Aggiungere 80,5 ml di SMCC stac, 159,5 ml di DMSO, vortex e centrifugare brevemente con una centrifuga da banco.
- Incubare a 37 ° C per 60 min. Evitare la formazione di condensa sotto il tappo microcentrifuga.
- Aggiungere 200 ml di PBS 1x, 60 ml di 3 M NaOAc (pH 5.2) e 1,5 ml di freddo 100% di etanolo. Mescolare la soluzione nel vortex e incubare a -20 ° C per 30 min.
- Centrifugare la soluzione a 20.000 xg per 20 min a 4 ° C. Rimuovere il surnatante e asciugare il pellet sotto vuoto. Evitare di essiccazione.
- Al pellet essiccati aggiungere 400 ml di ureasi magazzino e incubare a temperatura ambiente per 5 ore.
- Trasferire 200 ml di coniugato greggio ad un 100k MWCO colonna di filtro centrifugo pre-lavati. Centrifugare la colonna a 14000 xg per 5 min.
- Trasferire i restanti 200 ml di coniugato greggio alla colonna e centrifugare a 14000 g per 5 min. Rimuovere la colonna e metterlo a testa in giù in un nuovo 2,0 ml provetta per microcentrifuga ( "tubo di raccolta").
- centrifuge il tubo di raccolta (con la colonna invertita) a 1.000 xg per 2 min. Rimuovere il tubo di raccolta e aggiungere 30 ml di PBS 1x alla colonna di centrifuga per lavare la membrana per ulteriore recupero di coniugati.
- Ancora una volta invertire la colonna e inserire di nuovo nel tubo di raccolta. Centrifugare il tubo di raccolta (con la colonna invertita) a 1.000 xg per 2 min. Rimuovere e smaltire la colonna.
- Conservare il UrDNA a 4 ° C fino all'utilizzo.
4. Montaggio di EC1 e UrDNA su biglie magnetiche
- Mescolare il magazzino tallone magnetica (MB) bene e trasferire 100 ml di sospensione MB in una provetta da 1,5 ml microcentrifuga. Posizionare il tubo microcentrifuga su un supporto magnetico cremagliera per isolare la MB.
- Rimuovere il surnatante pipettando e aggiungere 150 ml di Binding Buffer (BB) al tubo. Rimuovere il tubo dal supporto e toccare con attenzione il tubo per sospendere di nuovo la MB a una soluzione omogenea. Evitare di spruzzare la sospensione al top del tubo o tappo. In questo caso, utilizzare una centrifuga da banco a girare brevemente il residuo torna alla sospensione.
- Ripetere il punto 4.2 altre due volte.
- A questa sospensione aggiungere 10 ml di 10 mM EC1. Mescolare accuratamente toccando il tubo.
- Incubare la soluzione con lieve agitazione per 30 min. Premere il tubo ogni 2-3 min per evitare la precipitazione del MB.
- Posizionare la provetta per microcentrifuga indietro sulla griglia magnetica per isolare il MB e rimuovere il surnatante pipettando. Lavare il MB per tre volte con 150 ml di BB (come descritto al punto 4.2).
- Dopo il lavaggio è completo, sospendere la MB in un totale di 150 ml di BB. A questa soluzione, aggiungere 15 ml di UrDNA e riscaldare la soluzione a 45 ° C per 2 min. Raffreddare la soluzione a temperatura ambiente e incubare per 2 ore.
- Posizionare la provetta per microcentrifuga indietro sulla griglia magnetica per isolare il MB e rimuovere il surnatante pipettando.
- Aggiungere 100 ml di tampone di reazione (RB). rimuovere tegli microcentrifuga tubo dal rack magnetica e con attenzione risospendere il MB.
- Lavare i MB altre tre volte ripetendo il passaggio 4.9.
NOTA: Il supernatante lavato può essere rapidamente testato per determinare se non ibridizzate UrDNA è ancora presente, che può risultare in un falso segnale positivo. Il test può essere fatto aggiungendo 10 ml di 50 mM di urea e 10 ml di 0,04% di fenolo rossi per la soluzione di lavaggio. Continuare a lavare il MB fino a quando la soluzione di lavaggio non causa un cambiamento di colore. La sospensione 100 microlitri è conservato a 4 ° C fino all'utilizzo.
5. Preparazione di cellule batteriche 20
- Coltura E. coli dalle scorte
- Piastra E. coli K12 (MG1655) su piastre di agar LB da uno stock di glicerolo sotto una fiamma o in un armadio di sicurezza biologica.
- Utilizzando una punta pipetta sterile, toccare delicatamente lo stock di glicerolo e leggermente striscia la superficie di una piastra di agar per evitare la puntura del agar LB.
- Invertire la striatipiastra e incubare a 37 ° C per 14 ore.
- Sigillare la piastra con parafilm e conservare a 4 ° C per un massimo di 3 settimane.
- Coltura E. coli per Cell conteggio
- In una provetta 14 ml sterile, dispensare 2 ml di brodo LB.
- Utilizzando una punta pipetta sterile, scegliere una singola colonia dalla piastra di agar striato preparato al punto 5.1 e trasferirlo al tubo della cultura.
- Incubare la cultura a 37 ° C e agitare a 230 giri al minuto per 14 ore.
- In serie diluire la coltura batterica in intervalli di 10-fold.
- Per ciascun campione diluito, uniformemente piatto cinque aliquote di 100 microlitri su piatti separati LB agar. Capovolgere le piastre e incubare a 37 ° C per 14 ore.
- Contare le cellule di ogni campione per ottenere la concentrazione media di cellule di ciascuna diluizione.
NOTA: 10 7 cellule vengono spesso utilizzati per impostare una cartina di tornasole di riferimento per E. coli come questo livello di E. coli può innescare un rapido colore change. Tuttavia, una cartina di tornasole eseguito correttamente in grado di rilevare partire da 500 celle, come discusso nella sezione Risultati.
- Preparazione E. Le cellule coli per test
- Per una sospensione cellulare desiderato, il trasferimento di 1 ml di coltura in una provetta da 1,5 ml microcentrifuga.
- Centrifugare le cellule a 6000 xg per 10 min a 4 ° C. rimuovere con attenzione il surnatante senza disturbare il pellet.
- Aggiungere 10 ml di tampone di reazione al pellet e risospendere le cellule. Sonicare la sospensione cellulare per 5 min. Trasferire la sospensione cellulare ad una scatola di ghiaccio per 5 min.
- Sonicare la sospensione cellulare per altri 5 minuti.
- Centrifugare la sospensione cellulare a 13.000 xg per 10 min a 4 ° C. Utilizzare il surnatante per la prova (10 ml).
6. cartina di tornasole
- In una provetta da 1,5 ml microcentrifuga, prelavaggio il tubo con l'aggiunta e vortex 100 ml di tampone di reazione (RB) nella provetta per microcentrifuga escartando il buffer.
- Trasferimento 15 ml di EC1 assemblato (protocollo 4) alla provetta per microcentrifuga lavato.
- Lavare le sfere magnetiche ponendo la provetta da microcentrifuga su un rack magnetica. Rimuovere il surnatante pipettando. Rimuovere la provetta per microcentrifuga dal rack, aggiungere 100 ml di RB, e risospendere attentamente le sfere magnetiche.
- Lavare i MB altre due volte ripetendo il passaggio 6.3.
- Posizionare la provetta per microcentrifuga indietro sulla griglia magnetica, rimuovere il surnatante e aggiungere il 10 microlitri E. campione coli preparato dal punto 5.3.
- Mescolare le perline campione e magnetici accuratamente picchiettando delicatamente sul tubo microcentrifuga.
- Incubare la reazione a temperatura ambiente per 1 ora.
- Per la reazione, aggiungere 90 ml di DDH 2 O e posizionare il tubo microcentrifuga su una griglia magnetica.
- Dopo circa 3 min di separazione magnetica, trasferire accuratamente 85 ml di surnatante in una provetta da 0,5 ml microcentrifuga. Prelevare il surnatante lentamenteevitare di raccogliere eventuali sfere magnetiche.
- Per la provetta per microcentrifuga sopra aggiungere 15 ml di 0,04% rosso fenolo e 100 ml di soluzione di substrato.
- Scattare una fotografia a intervalli di tempo specifici per registrare il cambiamento di colore.
NOTA: Il cambiamento di pH può anche essere monitorato con un pH-metro con un microelettrodo. Il pH di partenza deve essere di circa 5,2-5,5 (la soluzione è gialla). In caso contrario, la soluzione può essere regolato mediante aggiunta di 1 mM tampone acetato pH 5.0).
Risultati
Il principio della prova del nove batterica è spiegato in Figura 1 Il test utilizza tre materiali principali:. Un DNAzyme RNA-scissione che viene attivato da un batterio specifico, ureasi e perline magnetiche. Il DNAzyme viene utilizzato come elemento di riconoscimento molecolare per ottenere un rilevamento altamente specifica di un batterio di interesse. Ureasi e sfere magnetiche sono utilizzati per ottenere trasduzione del segnale delle attività RNA-scissione del DNAzyme. Questo comporta la creazione di sfere magnetiche che contengono coniugati ureasi-DNAzyme. In presenza del batterio bersaglio, la scinde DNAzyme suo RNA linkage. Questa azione provoca la dissociazione di ureasi da perline magnetiche. L'ureasi rilasciato può essere facilmente separato dal biglie magnetiche e utilizzata per generare un cambiamento di colore in una soluzione giornalista, contenente urea e un colorante sensibile al pH. Ureasi idrolizza l'urea in ammoniaca, accompagnato dalla crescita di pH che fa scattare il ccambiamento olor del colorante.
La figura 2 presenta una cartina di tornasole batterica dove EC1, un E. coli -responsive RNA-scissione DNAzyme, è stato utilizzato come DNAzyme e rosso fenolo è stato utilizzato come colorante pH segnalazione. EC1 stato precedentemente isolato dal nostro gruppo da un pool DNA casuale sequenza con la tecnica di selezione in vitro. 5 nostri studi precedenti hanno dimostrato che EC1 è altamente specifico per E. coli e mostra un'attività minima verso altri batteri. 5,19 Si è trovato che EC1 viene attivato da una molecola di proteina da E. coli. Anche se l'identità di questo biomarker proteina non è stato decifrato, la specificità alto riconoscimento suggerisce che questa proteina è unico per E. coli. La soluzione giornalista è impostato per avere un pH iniziale di 5,5. A questo pH, rosso fenolo presenta un colore giallo. Come ureasi idrolizza l'urea in ammoniaca, la basicità del sol giornalistaaumenta ution. Ciò si riflette dal graduale cambiamento di colore dal giallo al rosa. La profondità del cambiamento di colore dipende dai due parametri seguenti, come illustrato dalla Figura 2: il numero di E. coli utilizzati nella fase di attivazione DNAzyme e il tempo concesso per la fase di urea idrolisi. Più E. coli portato a cambiamenti di colore forti, riflessa dalla osservazione di un progressivo colore giallo-rosa transizione di colore quando E. coli sono state serialmente passato da 5 a 5 x 10 7 (10-fold aumenta ogni volta). Nel frattempo, un tempo più lungo per idrolisi dell'urea consentito per la rilevazione di un numero minore di E. coli (5.000 cellule nella reazione di 1 ora e 500 cellule nella reazione di 2 ore).
Il cambiamento di pH la cartina tornasole batterica può essere monitorata utilizzando un misuratore portatile pH e risultati rappresentativi sono illustrati nella figura 3. Si was trovato che la presenza di 10 7 E. coli determinato graduale aumento del pH da 3 unità entro 10 min. Al contrario, l'assenza di E. coli non ha causato variazioni di pH rilevabili sotto la stessa impostazione.
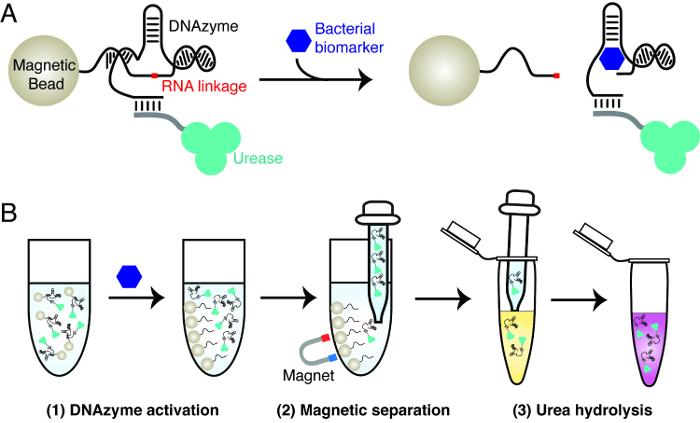
Figura 1: Il principio costruttivo di cartina di tornasole batterica (A) Attivazione di un DNAzyme RNA-fende da un biomarker specifico da un batterio di interesse.. Alla presenza del biomarker, la DNAzyme RNA-scissione immobilizzato su perline scinde il legame magnetico RNA, con conseguente rilascio del ureasi contrassegnati da sfere magnetiche a soluzione. (B) Procedura di test in tre fasi. Fase 1: attivazione DNAzyme, come descritto nel pannello A. Fase 2: Separazione magnetica - dell'ureasi rilasciato è separato da perline magnetiche. Fase 3: Urea idrolisi R12; l'ureasi rilasciato viene aggiunto in una soluzione di urea giornalista contenente. Ureasi idrolizza l'urea in ammoniaca, con un conseguente cambiamento di pH che può essere segnalato da un colorante sensibile al pH. Si prega di cliccare qui per vedere una versione più grande di questa figura.

Figura 2: prova del nove con E. coli utilizzando E. coli -responsive DNAzyme CE1. I risultati di variazione cromatica rappresentativi con un numero variabile di E. coli fornite sopra di ciascuna provetta. Rosso fenolo è stato utilizzato come il colorante sensibile al pH. Una prova senza E. coli è stato usato come controllo negativo. Più E. coli sono tenuti a causare il rilascio di più molecole ureasi, accompagnato by cambiamenti di colore più forti. Cliccate qui per vedere una versione più grande di questa figura.
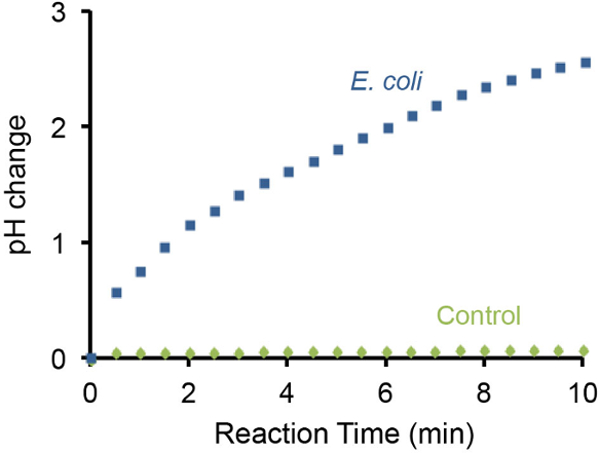
Figura 3: monitoraggio del pH utilizzando un pHmetro La variazione di pH provocati dalle 10 7 E.. coli è stato monitorato mediante un pH-metro portatile. Una prova senza E. coli è stato usato come controllo negativo. La presenza di 10 7 E. coli nella soluzione di prova può aumentare la basicità da unità di pH ~ 3 in 10 min. Cliccate qui per vedere una versione più grande di questa figura.
| Nome | Sesequenza (5'-3 ') | Nota |
| BS1 | BTTTT TTTTT TTTAC TCTTC CTAGC FRQGG TTCGA TCAAG A | B: 5'-biotina; R: ribonucleotide adenina; F: fluoresceina-dT; Q: DABCYL-dT |
| DE1 | GATGT GCGTT GTCGA GACCT GCGAC CGGAA CACTA CACTG TGTGG GGATG GATTT CTTTA CAGTT GTGTG TTGAA CGCTG TGTCA AAAAA AAAA | |
| T1 | GACAA CGCAC ATCTC TTGAT CGAAC C | |
| LD1 | XTTTT TTTTT TTTTT TTGAC ACAGC GTTCA A | X: 5'-NH 2 |
Tabella 1: Sequenze di oligonucleotidi sintetici.
Discussione
La definizione dell'azione dell'attività RNA clivaggio di un DNAzyme batterio sensibile ad un banco di prova è reso possibile attraverso l'uso di ureasi e separazione magnetica, come illustrato nella figura 1. Sebbene la dimostrazione della cartina tornasole modificati per rivelare batterica è fatto con una E. coli-dipendente RNA-scissione DNAzyme, 5,19,20 il design può essere generalmente esteso per qualsiasi DNAzyme RNA-scissione. Data la grande disponibilità di DNAzimi RNA-scissione per i diversi analiti e varie metodologie di isolare nuovi DNAzymes RNA-scissione da piscine casuale sequenza di nuovi obiettivi, ci aspettiamo che la piattaforma cartina di tornasole modificato può essere esteso al rilevamento di diversi obiettivi di interesse .
La prova del nove per E. rilevamento coli in grado di rilevare 5.000 e 500 cellule quando il tempo di reazione segnalazione è impostato per essere 1 e 2 ore, rispettivamente. La reazione popolare a catena della polimerasi (PCR) epanino enzyme-linked immunosorbent assay (ELISA) metodi possono raggiungere limiti di rilevabilità pari a circa 10 4 -10 5 E. coli in tempi di test simili. 25,26 Così, la prova del nove batterica offre sensibilità di rilevamento comparabile.
Sebbene la cartina tornasole batterica è facile da realizzare e può produrre vibranti cambiamenti di colore, diversi fattori possono influenzare significativamente i risultati dei test. In primo luogo, la qualità di ureasi è molto importante. Abbiamo usato ureasi da fonti diverse e trovato i risultati del test possono variare in modo significativo. Si consiglia l'uso di ureasi dall'origine specificata nella sezione Materiali.
Il montaggio delle sfere magnetiche / ureasi / DNAzyme ha bisogno di particolare attenzione. lavaggio accurato di sfere magnetiche per rimuovere UrDNA unhybridized è necessario per evitare risultati falsi-positivi. Cura deve anche essere adottate per evitare l'accumulo di perline magnetiche residue sulla superficie interna della lid della provetta per microcentrifuga, che può essere difficile da vedere. Una volta lì, le sfere magnetiche non sono più sottoposti a separazione magnetica e, quindi, potrebbe portare un po 'di UrDNA unhybridized che può portare a segnali falsi-positivi nella reazione giornalista. E 'anche importante evitare di lasciare il tubo per microcentrifuga sul sostegno magnetico per più di 10 minuti durante la fase di separazione magnetica. Le perle possono aggregare o aderire alla provetta per microcentrifuga, che possono ridurre l'efficienza di lavaggio e di introdurre incoerenza da lotto a lotto. L'inclusione di 0,01% Tween-20 nella soluzione di lavaggio può migliorare la coerenza da lotto a lotto e dovrebbe essere attuata.
Le sfere magnetiche sono rivestiti con streptavidina, che è stato utilizzato come ancoraggio per assemblare coniugati DNAzyme-ureasi sulle sfere magnetiche. Sia streptavidina e ureasi sono molecole proteiche che possono essere denaturato durante la conservazione. Noi di solito memorizzare le perle DNAzyme-ureasi-magnetici assemblati a 4 ° C per un massimo di 4 settimanee rendere lotti freschi regolarmente per ottenere risultati più coerenti.
La cura deve anche essere presa per evitare accidentalmente prendere sfere magnetiche in fase di separazione magnetica (passo 6,9) dopo l'attivazione DNAzyme. Dalla nostra esperienza, detriti cellulari e altri particolati nella soluzione può ridurre l'efficienza di separazione magnetica, pertanto alcune sfere magnetiche possono essere involontariamente prese durante pipettamento. Questo si tradurrà in risultati falsi-positivi. Si raccomanda le seguenti misure per alleviare il problema: un tempo di separazione più lungo (come ad esempio 5-10 minuti), un rilascio più lento della pressione sulla pipetta per consentire dolce ritiro del surnatante, e sottoponendo il surnatante in una serie supplementare di separazione magnetica .
Infine, è importante evitare la contaminazione incidente della soluzione giornalista stock di ureasi nel corso di un esperimento in cui vengono testati campioni multipli. Data l'elevata reattività di uRease, la contaminazione di questo tipo può portare a risultati falsi-positivi.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
The funding for this research project was provided by the Natural Sciences and Engineering Research Council of Canada (NSERC) via a Discovery Grant to YL.
Materiali
| Name | Company | Catalog Number | Comments |
| Ethylenediaminetetraacetic acid (EDTA) | VWR AMRESCO | 0105 | |
| Sodium Hydroxide (NaOH) pellets | BIO BASIC CANADA INC. | SB6789 | |
| Tris-base | VWR AMRESCO | 0497 | |
| Boric acid | AMRESCO | 0588 | |
| Urea | VWR AMRESCO | M123 | |
| 40% acrylamide/bisacrylamide (29:1) solution | BIO BASIC CANADA INC. | A0007 | |
| Sucrose | Bioshop Canada inc. | SUC507 | |
| Bromophenol blue | Bioshop Canada inc. | BRO777 | |
| Xylenecyanol FF | SIGMA-ALDRICH | X-4126 | |
| 10% sodium dodecyl sulfate | Bioshop Canada inc. | SDS001 | |
| Hydrochloric Acid (HCl) | CALEDON LABORATORIES LTD | 6026 | |
| Sodium Chloride (NaCl) | Bioshop Canada inc. | SOD001 | |
| 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid (HEPES) | Bioshop Canada inc. | HEP001 | |
| Magnesium Chloride (II) hexahydrate | VWR AMRESCO | 0288 | |
| Tween 20 | Bioshop Canada inc. | TW508 | |
| Adenosine Triphospahte (ATP) | AMRESCO | 0220 | |
| Sodium Acetate trihydrate (NaOAc) | SIGMA-ALDRICH | S8625 | |
| Ethanol | Commercial Alcohols | P016EAAN | |
| Tetramethyleneethylenediamine (TEMED) | AMRESCO | 0761 | |
| 10% Ammonium persulfate (APS) | BIO BASIC CANADA INC. | AB0072 | |
| Succinimidyl 4-(N-maleimidomethyl) cyclohexane-1-carboxylate (SMCC) | ThermoFisher SCIENTIFIC | 22360 | |
| Dimethyl sulfoxide (DMSO) | CALEDON LABORATORIES | 803540 | |
| Urease | SIGMA-ALDRICH | U0251 | |
| 1x Phosphate Buffered Saline (PBS) | ThermoFisher SCIENTIFIC | 70011-069 | |
| 0.04% Phenol red | SIGMA-ALDRICH | P3532 | |
| 10x T4 polynucleotide kinase reaction buffer | Lucigen | 30061-1 | |
| 10x T4 DNA ligase reaction buffer | Bio Basics Canada | B1122-B | |
| T4 DNA ligase (5 U/μl) | Thermo Fischer Scientific | B1122 | |
| Luria Bertani (LB) Broth | AMRESCO | J106 | |
| Agar | AMRESCO | J637 | |
| T4 polynucleotide kinase (10 U/μl) | Lucigen | 30061-1 | |
| E. coli K12 (MG1655) | ATCC | ATCC700926 | |
| Centrifuge | Beckman Coulter, Inc. | 392187 | |
| Glass plates | CBS scientific | ngp-250nr | |
| 0.75 mm thick spacers | CBS scientific | VGS-0725r | |
| 12-well comb | CBS scientific | VGC-7512 | |
| UV Lamp | UVP | 95-0017-09 | |
| Spectrophotometer (NanoVue) | GE Healthcare | N/A | |
| Metal plate | CBS scientific | CPA165-250 | |
| Vortex | VWR International | 58816-123 | |
| Gel electrophoresis apparatus | CBS scientific | ASG-250 | |
| Petri dishes | VWR International | 25384-342 | |
| 100 kDa MWCO centrifugal filters | EMD Millipore | UFC510024 | |
| Magnetic Bead (BioMag) | Bangs Laboratories Inc | BM568 | |
| Magnetic Seperation Rack | New England BioLabs | S1506S | |
| Microfuge tubes | Sarstedt | 72.69 | |
| Syringe filter (0.22 μm) | VWR International | 28145-501 | |
| 14 ml culture tube | VWR International | 60818-725 | |
| Cell culture incubator | Eppendorf Scientific | M13520000 | |
| Branson Ultrasonic cleaner | Branson | N/A | |
| Camera (Canon Powershot G11) | Canon | N/A | |
| 50 ml conical tube | VWR International | 89004-364 |
Riferimenti
- Daar, A. S., et al. Top ten biotechnologies for improving health in developing countries. Nat. Genet. 32, 229-232 (2002).
- Newman, J. D., Turner, A. P. Home blood glucose biosensors: a commercial perspective. Biosens. Bioelectron. 20, 2435-2453 (2005).
- Turner, A. P. Biosensors: sense and sensibility. Chem. Soc. Rev. 42, 3184-3196 (2013).
- Tram, K., Kanda, P., Salena, B. J., Huan, S. Y., Li, Y. F. Translating Bacterial Detection by DNAzymes into a Litmus Test. Angew. Chem. Int. Ed. 53, 12799-12802 (2014).
- Ali, M. M., Aguirre, S. D., Lazim, H., Li, Y. Fluorogenic DNAzyme probes as bacterial indicators. Angew. Chem. Int. Ed. 50, 3751-3754 (2011).
- Schlosser, K., Li, Y. Biologically inspired synthetic enzymes made from DNA. Chem. Biol. 16, 311-322 (2009).
- Tuerk, C., Gold, L. Systematic evolution of ligand by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Breaker, R. R. Making catalytic DNAs. Science. 290, 2095-2096 (2000).
- Liu, J., Cao, Z., Lu, Y. Functional nucleic acid sensors. Chem. Rev. 109, 1948-1998 (2009).
- Navani, N. K., Li, Y. Nucleic acid aptamers and enzymes as sensors. Curr. Opin. Chem. Biol. 10, 272-281 (2006).
- Li, J., Lu, Y. A highly sensitive and selective catalytic DNA biosensor for lead ions. J. Am. Chem. Soc. 122, 10466-10467 (2000).
- Liu, J., Lu, Y. A colorimetric lead biosensor using DNAzyme-directed assembly of gold nanoparticles. J. Am. Chem. Soc. 125, 6642-6643 (2003).
- Liu, Z., Mei, S. H. J., Brennan, J. D., Li, Y. Assemblage of signaling DNA enzymes with intriguing metal specificity and pH dependence. J. Am. Chem. Soc. 125, 7539-7545 (2003).
- Liu, J., et al. A catalytic beacon sensor for uranium with parts-per-trillion sensitivity and millionfold selectivity. Proc. Natl. Acad. Sci. USA. 104, 2056-2061 (2007).
- Huang, P. J., Vazin, M., Liu, J. In vitro selection of a new lanthanide-dependent DNAzyme for ratiometric sensing lanthanides. Anal. Chem. 86, 9993-9999 (2014).
- Chiuman, W., Li, Y. Simple fluorescent sensors engineered with catalytic DNA 'MgZ' based on a non-classic allosteric design. PLoS One. 2, e1224(2007).
- Xiang, Y., Lu, Y. Using personal glucose meters and functional DNA sensors to quantify a variety of analytical targets. Nat. Chem. 3, 697-703 (2011).
- Aguirre, S. D., Ali, M. M., Salena, B. J., Li, Y. A sensitive DNA enzyme-based fluorescent assay for bacterial detection. Biomolecules. 3, 563-577 (2013).
- Aguirre, S. D., Ali, M. M., Kanda, P., Li, Y. F. Detection of Bacteria Using Fluorogenic DNAzymes. J. Vis. Exp. (63), e3961(2012).
- Shen, Z., et al. A catalytic DNA activated by a specific strain of bacterial pathogen. Angew. Chem. Int. Ed. 54, (2015).
- He, S., et al. Highly specific recognition of breast tumors by an RNA-cleaving fluorogenic DNAzyme probe. Anal. Chem. 87, 569-577 (2015).
- Sumner, J. B., Hand, D. B. Isoelectric point of crystalline urease. J. Am. Chem. Soc. 51, 1255-1260 (1929).
- Karplus, P. A., Pearson, M., Hausinger, R. P. 70 Years of crystalline urease: What have we learned. Acc. Chem. Res. 30, 330-337 (1997).
- Omiccioli, E., Amagliani, G., Brandi, G., Magnani, M. A new platform for Real-Time PCR detection of Salmonella spp., Listeria monocytogenes and Escherichia coli O157 in milk. Food Microbiol. 26, 615-622 (2009).
- Cui, S., Schroeder, C. M., Zhang, D. Y., Meng, J. Rapid sample preparation method for PCR-based detection of Escherichia coli O157:H7 in ground beef. J. Appl. Microbiol. 95, 129-134 (2003).
- Ibekwe, A. M., Watt, P. M., Grieve, C. M., Sharma, V. K., Lyons, S. R. Multiplex fluorogenic real-time PCR for detection and quantification of Escherichia coli O157:H7 in dairy wastewater wetlands. Appl. Environ. Microbiol. 68, 4853-4862 (2002).
- Strachan, N. J., Ogden, I. D. A sensitive microsphere coagulation ELISA for Escherichia coli O157:H7 using Russell's viper venom. FEMS Microbiol Lett. 186, 79-84 (2000).
- de Boer, E., Beumer, R. R. Methodology for detection and typing of foodborne microorganisms. Int. J. Food Microbiol. 50, 119-130 (1999).
- Gracias, K. S., McKillip, J. L. A review of conventional detection and enumeration methods for pathogenic bacteria in food. Can. J. Microbiol. 50, 883-890 (2004).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon