Method Article
La detección colorimétrica de bacterias mediante prueba de fuego
En este artículo
Resumen
We describe a protocol for colorimetric detection of E. coli using a modified litmus test that takes advantage of an RNA-cleaving DNAzyme, urease, and magnetic beads.
Resumen
There are increasing demands for simple but still effective methods that can be used to detect specific pathogens for point-of-care or field applications. Such methods need to be user-friendly and produce reliable results that can be easily interpreted by both specialists and non-professionals. The litmus test for pH is simple, quick, and effective as it reports the pH of a test sample via a simple color change. We have developed an approach to take advantage of the litmus test for bacterial detection. The method exploits a bacterium-specific RNA-cleaving DNAzyme to achieve two functions: recognizing a bacterium of interest and providing a mechanism to control the activity of urease. Through the use of magnetic beads immobilized with a DNAzyme-urease conjugate, the presence of bacteria in a test sample is relayed to the release of urease from beads to solution. The released urease is transferred to a test solution to hydrolyze urea into ammonia, resulting in an increase of pH that can be visualized using the classic litmus test.
Introducción
Bacterial pathogens are one of the major causes of global morbidity and mortality. Outbreaks from hospital-acquired infections, food-borne pathogens, and bacterial contaminants in the environment pose serious and on-going threats to public health and safety. To prevent these outbreaks, effective tools are needed that permit pathogen detection in a timely fashion under a variety of settings. Simple but still effective tests that are portable and cost-effective are greatly coveted, especially in regions that are susceptible to outbreaks but cannot afford expensive testing facilities.1-3 Although there exists a multitude of methods to detect bacteria, many of them are not suitable as screening or on-site testing tools because they require long test times, expensive instruments and complicated testing procedures.
Colorimetric tests are particularly attractive for point-of-care or field applications as color changes can be easily detected by the naked eye. The litmus test for pH is simple, quick, and effective. Although it is a very old technology, it is still widely used today because of its simplicity and effectiveness. Surprisingly, this simple test had never been modified to achieve the detection of other analytes before we recently developed an approach of modifying this test for E. coli testing.4
The expanded litmus test for E. coli employs three additional components: an E. coli activated RNA-cleaving DNAzyme (EC1), 5 urease, and magnetic beads. DNAzymes refer to synthetic single-stranded DNA molecules with catalytic activity.6 They can be isolated from random-sequence DNA pools using in vitro selection.7,8 They are highly stable and can be produced cost-effectively using high-efficiency automated DNA synthesis.9 For these reasons, DNAzymes, particularly RNA-cleaving DNAzymes, have been widely examined for biosensing applications.6,10,11 RNA-cleaving DNAzyme sensors have been developed to detect metal ions,12-16 small molecules,17,18 bacterial pathogens5,19-21 and cancer cells.22 Given the great availability of target-induced RNA-cleaving DNAzymes, any assay that utilizes a DNAzyme can be potentially expanded to detect a diverse range of analytes.
Urease is chosen for its ability to hydrolyze urea into ammonia,23,24 resulting in a pH increase. Urease is also highly efficient, stable and amenable for conjugation to other biomolecules. Therefore, we postulated that a conjugate of an RNA-cleavage DNAzyme with urease would allow the use of litmus test for the detection of other targets.5
The action of the RNA-cleaving DNAzyme is relayed to urease-mediated increase of pH through the use of magnetic beads that are immobilized with the DNAzyme-urease conjugate. Because the activity of the DNAzyme under investigation is strictly dependent on E. coli, the presence of this bacterium in the test solution will result in the release of urease from the magnetic beads to the solution, which is then taken and used to hydrolyze urea in a reporter solution that contains a pH-sensitive dye. The final outcome of this procedure is a color change that can be conveniently reported by the dye or pH paper.
Protocolo
1. Preparación de los reactivos y búferes
- M de ácido 0,5 etilendiaminotetraacético (EDTA)
- En un vaso de precipitados de 2 L, agregar 186,1 g EDTA a 800 ml de agua destilada-desionizada (ddH 2 O). Ajustar el pH de la solución a 8,0 usando gránulos de NaOH. Añadir ddH2O a un volumen final de 1,0 L y transferir la solución a una botella de vidrio en autoclave para la esterilización en autoclave y se almacena a 4 ° C.
- EDTA 10 × Tris-borato (10x TBE)
- En un vaso de precipitados de plástico 4 L, añadir 432 g de Tris-base, 200 g de ácido bórico, 80 ml de 0,5 M EDTA (pH 8,0) y ddH 2 O hasta un volumen final de 4 L. Mix usando una barra de agitación hasta que todos los componentes se disuelven. Transferir la solución a botellas de vidrio de 1 L de autoclave y se almacena a 4 ° C.
- 10% de poliacrilamida desnaturalizante Stock
- En un vaso de precipitados de plástico 4 L, añadir 1.681,7 g de urea, 400 ml de 10x TBE, 1 L de 40% de acrilamida / bisacrilamida (29: 1) solución y ddH 2 O hasta que la final volumen es de 4 L. Mezclar con una barra de agitación hasta que se disuelve la urea. Transferir la solución a botellas de vidrio ámbar de 1 L y se almacena a 4 ° C.
- 2x tampón de carga de gel (2x GLB)
- En un vaso de precipitados de vidrio de 200 ml, añadir 44 g de urea, 8 g de sacarosa, 10 mg de azul de bromofenol, 10 mg de xileno cianol FF, 400 l de 10% de dodecil sulfato de sodio, y 4 ml de 10 x TBE. Añadir ddH 2 O hasta un volumen final de 40 ml y disolver los componentes con calentamiento suave a 50 ° C mientras se mezcla con una barra de agitación magnética. Transferir 1 ml de alícuota de 1,5 ml tubos de microcentrífuga y se almacena a 4 ° C.
NOTA: El uso anterior requiere breve calentamiento a 90 ° C para volver a disolver cualquier sólido.
- En un vaso de precipitados de vidrio de 200 ml, añadir 44 g de urea, 8 g de sacarosa, 10 mg de azul de bromofenol, 10 mg de xileno cianol FF, 400 l de 10% de dodecil sulfato de sodio, y 4 ml de 10 x TBE. Añadir ddH 2 O hasta un volumen final de 40 ml y disolver los componentes con calentamiento suave a 50 ° C mientras se mezcla con una barra de agitación magnética. Transferir 1 ml de alícuota de 1,5 ml tubos de microcentrífuga y se almacena a 4 ° C.
- 1 M Tris-hidrocloruro (Tris-HCl) (pH 7,5)
- En una botella de vidrio, añadir 12,1 g de Tris-base y 70 ml de ddH2O y mezclar hasta que se disuelva el sólido. Ajustar el pH a 7,5 utilizando ácido clorhídrico 1 M (HCl). Añadir ddH 2 O hasta un volumen final de 100 ml, autoclave y store a 4 ° C
- 5 M de cloruro de sodio (NaCl)
- En una botella de vidrio, se disuelven 58,4 g de NaCl en 150 ml de ddH2O Ajuste el volumen a 200 ml con ddH 2 O. Almacenar a 4 ° C.
- DNA tampón de elución
- En una botella de vidrio, mezclar 2,0 ml de 1 M Tris-HCl (pH 7,5), 8,0 ml de 5 M de NaCl y 0,4 ml de 0,5 M EDTA (pH 8,0). Ajuste el volumen a 200 ml con ddH2O, autoclave y se almacena a 4 ° C.
- 1 M 4- (2-hidroxietil) -1-piperazinetanosulfónico (HEPES) (pH 7,4)
- En una botella de vidrio, se disuelven 2,38 g de HEPES en 80 ml de ddH2O Ajustar el pH a 7,4 usando NaOH 5 N y añadir ddH 2 O hasta un volumen final de 100 ml.
- 1 M de cloruro de magnesio (MgCl 2)
- En una botella de vidrio, añadir 2,03 g de MgCl 2 6H 2 O y llevar el volumen a 100 ml con ddH 2 O.
- El tampón de reacción (RB)
- En un tubo cónico de 50 ml, añadir 50 l de 1 M HEPES (pH 7,4), 1,5 ml de 5 M NaCl, 0,75 ml de 1 M MgCl 2 y 5 l de Tween-20. Añadir ddH 2 O a un volumen final de 50 ml. Mezclar la solución y filtrar en otro tubo cónico utilizando un filtro de jeringa impulsada (0,22 m) y se almacena a 4 ° C hasta su uso.
- Tampón de unión (BB)
- En un tubo cónico de 50 ml, añadir 500 l de 1 M Tris-HCl (pH 7,5), 8,8 g de NaCl, 50 l de 1 M de MgCl 2 y 5 l de Tween 20. Añadir ddH 2 O hasta un volumen final de 50 ml. Mezclar la solución y filtrar en otro tubo cónico utilizando un filtro de jeringa impulsada (0,22 m) y se almacena a 4 ° C hasta su uso.
- Solución de sustrato (SS)
- En un tubo cónico de 50 ml, añadir 5,8 g de NaCl, 3 ml de 1 M MgCl 2, 1,5 g de urea, y 40 ml de ddH 2 O. Ajustar el pH de la solución a 5,0 usando HCl 10 mM. Debido a que la solución no es amortiguada, ajustar el pH utilizando HClcuidado a través de la adición de pequeñas alícuotas de HCl. Añadir ddH 2 O a un volumen final de 50 ml.
- Mezclar la solución y filtrar en otro tubo cónico utilizando un filtro de jeringa impulsada (0,22 m) y se almacena a 4 ° C hasta su uso.
- Luria Bertani (LB) Broth
- En un vaso de precipitados, se disuelven 20,0 g de polvo LB en 1 l de ddH 2 O. Transferencia a una botella de vidrio, autoclave, y se almacena a 4 ° C.
- 1,5% de agar LB
- En un matraz de 250 ml, añadir 1,5 g de agar y 100 ml de caldo LB. Autoclave y se almacena a 4 ° C.
- Las placas de agar
- Se vuelve a disolver el agar LB en un horno de microondas y enfriar la solución a ~ 50 ° C. Verter la solución en placas de Petri, haciendo ~ 5 placas y dejar que se solidifique.
2. Síntesis y purificación de E. colitis sensibles ADNzima EC1
- Síntesis de CE1 por la plantilla mediada por ligación enzimática
- purificar commoligonucleótidos sintetizados ercially BS1, DE1, y T1 (secuencias proporcionadas en la Tabla 1) por 10% de desnaturalización de electroforesis en gel de poliacrilamida (dPAGE) de acuerdo con protocolos estándar.
- Preparar una solución madre 100 mM de BS1, DE1, y T1. Almacenar a -20 ° C hasta su uso.
- En un tubo de microcentrífuga de 1,5 ml, añadir 38,5 l de ddH 2 O, 5 l de DE1, y 5 l de tampón de reacción de polinucleótido quinasa 10x T4 proporcionados por el proveedor de enzima (500 mM Tris-HCl (pH 7,6), 100 mM MgCl 2, ditiotreitol 50 mM (DTT), espermidina 1,0 mM).
- Añadir 1 l de trifosfato de adenosina (ATP) (100 mM). Añadir 5 unidades de polinucleótido quinasa de T4 (10 U / l). Mezclar pipeteando cuidadosamente la mezcla de reacción.
- Incubar la reacción a 37 ° C durante 30 min.
- Se detiene la reacción por calentamiento a 90 ° C durante 5 min.
- Añadir 118 l de ddH2O, 5 l de BS1, y 5 l de T1. La reacción se calienta a 90 ° C durante 2 millasn y después se enfría a temperatura ambiente durante 10 min.
- Añadir 20 l de 10 × T4 ADN ligasa tampón de reacción proporcionado por el proveedor de la enzima (400 mM Tris-HCl (pH 7,8), 100 mM MgCl 2 mM, DTT 100 mM, ATP 5). Añadir 10 unidades de ADN ligasa de T4 (5 U / l) y mezclar cuidadosamente mediante pipeteo.
- Incubar a temperatura ambiente durante 2 hr.
- Añadir 20 l de acetato de sodio 3 M (NaOAc) (pH 5,2), 500 l de etanol frío al 100%. Mezclar la solución mediante agitación y se coloca el tubo en un congelador C -20 ° durante 30 min.
- Centrifugar la microcentrífuga a 20.000 xg durante 20 min a 4 ° C. Retirar con cuidado el sobrenadante con la pipeta.
- Lavar el precipitado con etanol frío al 70% y se centrifuga de nuevo a 20.000 xg durante 10 min a 4 ° C. Una vez más, eliminar el sobrenadante y secar el pellet al vacío durante 10 min.
- Resuspender el ADN con 15 l de ddH2O y luego añadir 15 l de 2x GLB.
- Vortex vigorosamente y se calienta a 90 ° C durante2 minutos. Se purifica el ADN de longitud completa en un 10% dPAGE como se describe a continuación.
- Configuración de un 10% dPAGE
- Limpiar las placas de vidrio de dos (un plato completo y uno con muescas), dos de 0,75 mm de espesor, separadores y un peine de 12 pocillos. Coloque una placa de vidrio sobre una superficie plana con dos espaciadores en cada lado y la placa dentada en la parte superior. Cortar las dos placas juntas utilizando los cuatro clips proporcionados por el proveedor.
- En un vaso de precipitados de plástico de 150 ml, verter 40 ml de 10% dPAGE, 40 l de tetrametiletilendiamina (TEMED) y 400 l de persulfato de amonio al 10% (APS). Mezclar los componentes y se vierte la solución lentamente entre las placas.
- Inserte el peine y luego permitir que el gel se polimeriza durante 10 minutos. Una vez que se polimeriza el gel, retire lentamente el peine y lavar los pocillos con ddH2O
- Montar las placas en el aparato de electroforesis en gel. Utilice una placa de metal para disipar el calor generado para evitar el sobrecalentamiento.
- Verter TBE 1x a las cámaras superior e inferiory asegurar que los pocillos son bien sumergidos en el tampón. Lavar los pocillos con TBE 1x utilizando una pipeta o una jeringa.
- Ajuste el aparato para funcionar a 35 mA y pre-una duración de 15 minutos antes de cargar las muestras.
- La elución de ligado EC1 de 10% dPAGE
- Después de la etapa 2.2.6, ejecute el gel a 35 mA hasta que el colorante de fondo (azul de bromofenol) alcanza la parte inferior de la placa de vidrio. Esto debe tomar aproximadamente 1,5 horas. Desconecte la alimentación y retire las placas del aparato.
- Colocar las placas en unas cuantas hojas de papel toalla y retirar con cuidado los separadores de las placas de vidrio. Retirar con cuidado la placa de vidrio superior y envolver el gel con una envoltura de plástico.
- Da la vuelta al gel sobre quitar la segunda placa de vidrio y cubrir con plástico nuevo. Tenga cuidado para evitar las arrugas de la envoltura de plástico.
- Visualizar el producto ligado mediante el uso de UV sombreado (260 nm), que producirá 4 bandas de ADN distintos (EC1 totalmente ligado, DE1, BS1, y T1).
- Impuestos Especiales de la banda superior (EC1) con una hoja de afeitar estéril y transferir a un tubo de microcentrífuga de 1,5 ml. Aplastar el gel con una punta de pipeta estéril (tamaño de la punta 200 l) hasta que se convierte en una pasta fina. Añadir 550 l de tampón de elución de ADN, cubrir el tubo con papel de aluminio para proteger el fluoróforo incrustado, y agitar durante 15 min.
- Centrifugar la solución de gel a 20.000 xg durante 5 min y transferir cuidadosamente 400 l del sobrenadante a otro tubo de microcentrífuga de 1,5 ml. Evitar la retirada de piezas de gel durante el pipeteado.
- Para el tubo de microcentrífuga con el sobrenadante se transfirió añadir 40 l de 3 M NaOAc (pH 5,2), y 1,0 ml de etanol frío al 100%. Mezclar la solución mediante agitación y se coloca el tubo en un congelador C -20 ° durante 30 min.
- Centrifugar la microcentrífuga a 20.000 xg durante 20 min a 4 ° C. Retirar con cuidado el sobrenadante con la pipeta.
- Lavar el precipitado con etanol frío al 70% y se centrifuga de nuevo a 20.000 xg durante 10 min a 4 ° C. Una vez más, retire elsobrenadante y secar el pellet al vacío durante 10 min.
- Determinar la concentración de EC1 midiendo la absorbancia UV a 260 nm. Hacer a 10 mM de valores y almacenar la muestra a -20 ° C hasta su uso.
3. Conjugación de ureasa para DNA
- Preparación de succinimidil 4- (N-maleimidometil) ciclohexano-1-carboxilato de etilo (SMCC) Stock
- Disolver 1 mg de SMCC en 676 l de DMSO. Vortex y el lugar en hielo hasta su uso.
- Preparación de la ureasa de la
- Disolver 1 mg de ureasa en 1 ml de 1 × PBS (sin Mg2 + o Ca2 +). Colocar en hielo hasta su uso.
NOTA: Se cristalizó ureasa es lento para disolver y se necesita un mezclado suave para evitar la desnaturalización.
- Disolver 1 mg de ureasa en 1 ml de 1 × PBS (sin Mg2 + o Ca2 +). Colocar en hielo hasta su uso.
- Síntesis de la ureasa-DNA (UrDNA)
- Añadir 10 l de 100 mM LD1 a un tubo de microcentrífuga de 2,5 ml. Añadir 140 l de ddH2O, 40 l de 10x PBS, y remolino.
- Añadir 80,5 l de SMCC stac, 159,5 l de DMSO, vortex y centrifugar brevemente utilizando una centrífuga de mesa.
- Incubar a 37 ° C durante 60 min. Evitar la condensación debajo de la tapa de microcentrífuga.
- Añadir 200 l de 1x PBS, 60 l de 3 M NaOAc (pH 5,2), y 1,5 ml de etanol frío al 100%. Mezclar la solución por agitación e incubar a -20 ° C durante 30 min.
- Centrifugar la solución a 20.000 xg durante 20 min a 4 ° C. Eliminar el sobrenadante y secar el precipitado al vacío. Evitar el exceso de secado.
- Para el sedimento se secó añadir 400 l de ureasa de stock y se incuba a temperatura ambiente durante 5 hr.
- Transferir 200 l de conjugado en bruto a una columna de filtro centrífugo 100k MWCO previamente lavado. Centrifugar la columna a 14.000 xg durante 5 min.
- Transferir los 200 l restantes de conjugado crudo a la columna y se centrifuga a 14.000 xg durante 5 min. Retire la columna y colocarlo boca abajo en un nuevo tubo de microcentrífuga de 2,0 ml ( "tubo de extracción").
- CentrifUGE el tubo de recogida (con la columna invertida) a 1.000 xg durante 2 min. Retire el tubo de recogida y añadir 30 l de PBS 1x a la columna centrífuga para lavar la membrana para la recuperación adicional de los conjugados.
- Una vez más invertir la columna y colocar de nuevo en el tubo de recogida. Centrifugar el tubo de recogida (con la columna invertida) a 1.000 xg durante 2 min. Retirar y desechar la columna.
- Almacenar el UrDNA a 4 ° C hasta su uso.
4. Montaje de EC1 y UrDNA sobre perlas magnéticas
- Mezclar la población de perlas magnéticas (MB) y así transferir 100 l de suspensión MB a un tubo de microcentrífuga de 1,5 ml. Se coloca el tubo de microcentrífuga en un soporte de estante magnético para aislar el MB.
- Eliminar el sobrenadante por pipeteo y añadir 150 l de tampón (BB) de unión al tubo. Retire el tubo del soporte y golpee suavemente el tubo para resuspender el MB a una solución homogénea. Evitar salpicaduras de la suspensión de la top del tubo o tapa. Si esto sucede, utilice una centrífuga de mesa a girar brevemente el residuo de nuevo a la suspensión.
- Repita el paso 4.2 dos veces más.
- A esta suspensión se añaden 10 l de 10 mM EC1. Mezclar con cuidado con un toque en el tubo.
- Incubar la solución con agitación suave durante 30 min. Toque en el tubo de 2-3 min para evitar la precipitación de la MB.
- Se coloca el tubo de microcentrífuga de nuevo en el estante magnético para aislar el MB y eliminar el sobrenadante con la pipeta. Lavar el MB tres veces con 150 ml de BB (como se describe en el paso 4.2).
- Una vez que el lavado sea totalmente efectivo, suspender la MB en un total de 150 l de BB. A esta solución, añadir 15 l de UrDNA y se calienta la solución a 45 ° C durante 2 min. Enfriar la solución a temperatura ambiente y se incuba durante 2 hr.
- Se coloca el tubo de microcentrífuga de nuevo en el estante magnético para aislar el MB y eliminar el sobrenadante con la pipeta.
- Añadir 100 l de tampón de reacción (RB). Retire tque microfuga tubo del bastidor magnético y resuspender cuidadosamente el MB.
- Lavar los tres MB más veces repitiendo el paso 4.9.
NOTA: El sobrenadante se lavó rápidamente puede ser probado para determinar si sin hibridar UrDNA aún está presente, lo que puede dar lugar a una falsa señal positiva. La prueba se puede realizar mediante la adición de 10 l de urea 50 mM y 10 l de 0,04% de rojo de fenol a la solución de lavado. Continuar para lavar el MB hasta que la solución de lavado no causa un cambio de color. La suspensión 100 l se almacena a 4 ° C hasta su uso.
5. Preparación de células bacterianas 20
- El cultivo E. coli y Cotizaciones
- Placa E. coli K12 (MG1655) en placas de agar LB a partir de un stock de glicerol en virtud de una llama o en una cabina de seguridad biológica.
- Con una punta de pipeta estéril, toque suavemente la población de glicerol y la ligera racha de la superficie de una placa de agar para evitar perforar el agar LB.
- Invertir el veteadola placa e incubar a 37 ° C durante 14 horas.
- Sellar la placa con Parafilm y se almacena a 4 ° C durante un máximo de 3 semanas.
- El cultivo E. coli para Recuento celular
- En un tubo de cultivo de 14 ml estéril, prescindir de 2 ml de caldo LB.
- Con una punta de pipeta estéril, se toma una única colonia de la placa de agar con rayas preparado en la etapa 5.1 y transferirla al tubo de cultivo.
- Incubar el cultivo a 37 ° C y agitar a 230 rpm durante 14 horas.
- En serie diluir el cultivo bacteriano en intervalos de 10 veces.
- Para cada muestra diluida, de manera uniforme la placa cinco 100 ml de alícuotas sobre placas de agar LB separadas. Invertir las placas e incubar a 37 ° C durante 14 horas.
- Contar las células de cada muestra para obtener la concentración celular media de cada dilución.
NOTA: 10 7 células a menudo se utilizan para configurar una prueba de fuego de referencia para E. coli ya que este nivel de E. coli puede desencadenar un color rápida Change. Sin embargo, una prueba de fuego debidamente ejecutada puede detectar tan bajo como 500 células, como se discutió en la sección Resultados.
- Preparación E. Las células de E. coli para la prueba
- Para una suspensión celular deseada, la transferencia de 1 ml de Stock cultivaron a un tubo de microcentrífuga de 1,5 ml.
- Centrifugar las células a 6000 xg durante 10 min a 4 ° C. Retirar con cuidado el sobrenadante sin alterar el sedimento celular.
- Añadir 10 l de tampón de reacción al sedimento celular y resuspender las células. Sonicar la suspensión celular durante 5 min. Transferir la suspensión celular a una caja de hielo durante 5 min.
- Sonicar la suspensión de células durante otros 5 min.
- Centrifugar la suspensión celular a 13.000 xg durante 10 min a 4 ° C. Utilice el sobrenadante para el ensayo (10 l).
6. prueba de fuego
- En un tubo de microcentrífuga de 1,5 ml, prelavado del tubo y añadiendo vórtex 100 l de tampón de reacción (RB) en el tubo de microcentrífuga ydesechar el tampón.
- Transferencia de 15 l de EC1 ensamblado (protocolo 4) en el tubo de microcentrífuga de lavado.
- Lavar las perlas magnéticas colocando el tubo de microcentrífuga sobre una rejilla magnética. Eliminar el sobrenadante con la pipeta. Retire el tubo de microcentrífuga del bastidor, añadir 100 l de RB, y volver a suspender cuidadosamente las perlas magnéticas.
- Se lavan las dos MB más veces repitiendo el paso 6.3.
- Se coloca el tubo de microcentrífuga de nuevo en el estante magnético, eliminar el sobrenadante y añadir el 10 l E. muestra coli preparó a partir de la etapa 5.3.
- Mezclar las perlas magnéticas de muestra y cuidadosamente golpeando suavemente en el tubo de microcentrífuga.
- Incubar la reacción a temperatura ambiente durante 1 hr.
- Para la reacción, añadir 90 l de ddH2O y colocar el tubo de microcentrífuga sobre una rejilla magnética.
- Después de aproximadamente 3 min de separación magnética, transferir cuidadosamente 85 l del sobrenadante a un tubo de 0,5 ml de microcentrífuga. Retirar el sobrenadante lentamente aevitar la recogida de cualquier perlas magnéticas.
- Para el tubo de microcentrífuga por encima de añadir 15 l de 0,04% de rojo de fenol, y 100 l de solución de sustrato.
- Tome una fotografía a intervalos de tiempo específicos para registrar el cambio de color.
NOTA: El cambio en el pH también se puede monitorizar utilizando un medidor de pH con un microelectrodo. El pH de partida debe ser de aproximadamente 5.2 a 5.5 (la solución es de color amarillo). Si no, la solución se puede ajustar mediante la adición de 1 mM tampón de acetato pH 5,0).
Resultados
El principio de la prueba de fuego bacteriano se explica en la Figura 1 La prueba utiliza tres materiales principales:. Una ADNzima RNA-cleaving que se activa por una bacteria específica, ureasa y perlas magnéticas. La ADNzima se utiliza como el elemento de reconocimiento molecular para lograr una detección altamente específica de una bacteria de interés. La ureasa y perlas magnéticas se utilizan para conseguir la transducción de señales de la actividad de ARN-escisión de la ADNzima. Esto implica la creación de cuentas magnéticas que contienen conjugados de ureasa-ADNzima. En presencia de la bacteria diana, la ADNzima escinde su vinculación ARN. Esta acción da lugar a la disociación de la ureasa a partir de perlas magnéticas. La ureasa liberado puede ser fácilmente separada de perlas magnéticas y se utiliza para generar un cambio de color en una solución de reportero, que contiene urea y un colorante sensible al pH. La ureasa hidroliza la urea en amoníaco, acompañado por el aumento del pH que activa la ccambio olor del tinte.
La figura 2 presenta una prueba de fuego bacteriano en EC1, una E. coli -responsive RNA-cleaving ADNzima, se utilizó como la ADNzima, y se usó rojo de fenol como el colorante pH de reporte. EC1 fue previamente aislado por nuestro grupo a partir de un conjunto de ADN de secuencia aleatoria usando la técnica de selección in vitro. 5 Nuestros estudios previos han demostrado que EC1 es altamente específico para E. coli y exhibe actividad mínima hacia otras bacterias. 5,19 Se ha encontrado que EC1 es activado por una molécula de proteína de E. coli. Aunque el identidad de este biomarcador de proteína no ha sido descifrado, la alta especificidad de reconocimiento sugiere que esta proteína es única para E. coli. La solución reportero está preparado para tener un pH inicial de 5,5. A este pH, rojo fenol exhibe un color amarillo. Como ureasa hidroliza la urea en amoníaco, la basicidad del sol reporteroution aumenta. Esto se refleja en el cambio gradual del color del amarillo al rosa. La profundidad del cambio de color depende de los dos parámetros siguientes, como se ilustra en la Figura 2: el número de E. coli células utilizadas en la etapa de activación ADNzima y el tiempo permitido para la etapa de hidrólisis de la urea. Más E. células de E. coli dieron como resultado cambios de color más fuertes, que se refleja por la observación de un E.-amarillo a rosa transición de color cuando progresiva células de E. coli se incrementaron en serie 5-5 x 10 7 (10 veces aumentar cada vez). Mientras tanto, un tiempo más largo para la hidrólisis de urea permitido para la detección de un número menor de E. coli células (5000 células en la reacción de 1 hora y 500 células en la reacción de 2 horas).
El cambio de pH de la prueba de fuego bacteriano también se puede controlar utilizando un medidor de pH de mano y resultados representativos se ilustran en la Figura 3. Was encontró que la presencia de 10 7 E. células de E. coli resultó en incremento gradual de pH por 3 unidades dentro de 10 min. En contraste, la ausencia de E. células de E. coli no causó cambios en el pH detectables bajo el mismo entorno.
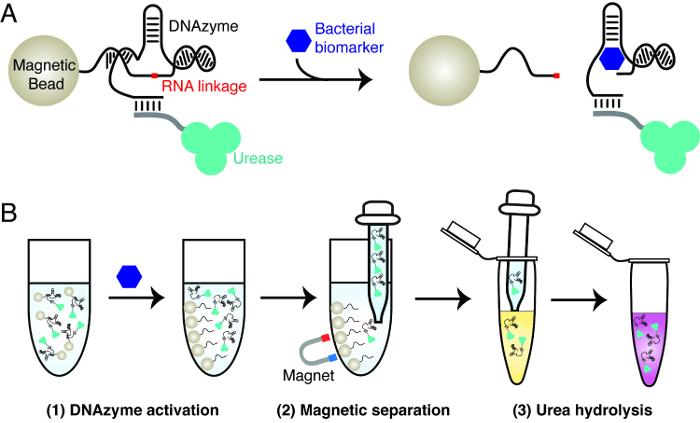
Figura 1: El principio de diseño de prueba de fuego bacteriano (A) Activación de una ADNzima RNA-cleaving por un biomarcador específico de una bacteria de interés.. En presencia del biomarcador, la ADNzima RNA-cleaving inmovilizados sobre perlas magnéticas escinde la unión de ARN, lo que resulta en la liberación de la ureasa etiquetado de perlas magnéticas a la solución. (B) Procedimiento de ensayo de tres pasos. Paso 1: activación ADNzima, como se describe en el panel A. Paso 2: La separación magnética - la ureasa liberado se separó de las perlas magnéticas. Paso 3: La urea hidrólisis R12; se añade la ureasa se libera en una solución reportero que contiene urea. La ureasa hidroliza la urea en amoníaco, lo que resulta en un cambio en el pH que puede ser informado por un colorante sensible al pH. Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Prueba de fuego con E. coli utilizando E. coli -responsive ADNzima EC1. Los resultados que cambian de color representativos con un número variable de E. coli células proporcionadas por encima de cada tubo de ensayo. Rojo fenol fue utilizado como el colorante sensible al pH. Una prueba sin E. coli se utilizó como control negativo. Más E. Se espera que las células de E. coli para producir la liberación de más moléculas de ureasa, acompañado bY más fuertes cambios de color. Haga clic aquí para ver una versión más grande de esta figura.
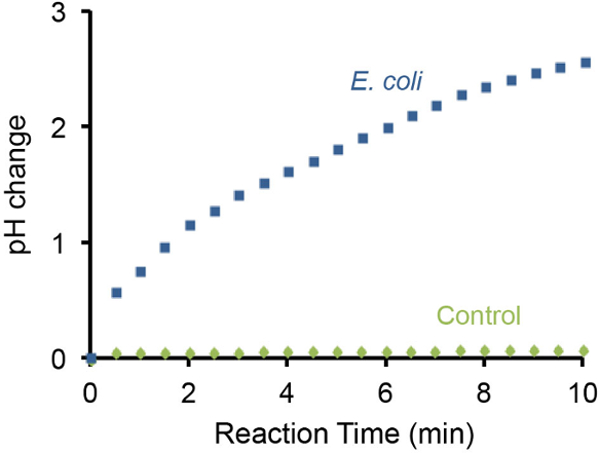
Figura 3: Supervisión de aumento del pH con un medidor de pH El cambio de pH causado por 10 7 E.. células de E. coli se controló utilizando un medidor de pH portátil. Una prueba sin E. coli se utilizó como control negativo. La presencia de 10 7 E. células de E. coli en la solución de ensayo pueden aumentar la basicidad por unidades de pH ~ 3 en 10 minutos. Por favor, haga clic aquí para ver una versión más grande de esta figura.
| Nombre | Sesecuencia (5'-3 ') | Nota |
| BS1 | BTTTT TTTTT TTTAC TCTTC CTAGC FRQGG TTCGA TCAAG Un | B: 5'-biotina; R: ribonucleótidos de adenina; F: fluoresceína-dT; Q: dabcilo-dT |
| DE1 | GATGT GCGTT GTCGA GACCT GCGAC CGGAA CACTA CACTG TGTGG GGATG GATTT CTTTA CAGTT GTGTG TTGAA CGCTG TGTCA AAAAA AAAA | |
| T1 | GACAA CGCAC ATCTC TTGAT CGAAC C | |
| LD1 | XTTTT TTTTT TTTTT TTGAC ACAGC GTTCA Un | X: 5'-NH 2 |
Tabla 1: Secuencias de oligonucleótidos sintéticos.
Discusión
La traducción de la acción de la actividad RNA escisión de una ADNzima bacteria sensible a una prueba de fuego se hace posible mediante el uso de la ureasa y la separación magnética, como se ilustra en la Figura 1. Aunque la demostración de la prueba de fuego modificado para la detección bacteriana es hecho con una E. coli dependiente de ARN de escisión ADNzima, 5,19,20 el diseño en general se puede ampliarse para cualquier ADNzima ARN de escisión. Dada la gran disponibilidad de DNAzymes RNA-cleaving para diferentes analitos y diversas metodologías para aislar nuevos DNAzymes RNA-cleaving de piscinas de secuencia aleatoria de nuevos objetivos, esperamos que la plataforma de prueba de fuego modificado se puede extender a la detección de diversas dianas de interés .
La prueba de fuego para E. detección coli puede detectar 5.000 y 500 células cuando el tiempo de reacción de notificación se establece para que sea 1 y 2 h, respectivamente. La reacción popular cadena de la polimerasa (PCR) ysándwich unido a enzima ensayo inmunoenzimático (ELISA) métodos pueden alcanzar límites de detección de aproximadamente 10 4 -10 5 E. células de E. coli en tiempos de prueba similares 25,26. Por lo tanto, la prueba de fuego bacteriano ofrece una sensibilidad de detección comparables.
Aunque la prueba de fuego bacteriano es fácil de realizar y puede producir cambios de colores vibrantes, varios factores pueden afectar significativamente los resultados de las pruebas. En primer lugar, la calidad de la ureasa es muy importante. Hemos utilizado la ureasa de diferentes fuentes y ha encontrado los resultados de la prueba pueden variar significativamente. Se recomienda el uso de la ureasa de la fuente especificada en la sección de Materiales.
El conjunto de perlas magnéticas / ureasa / ADNzima necesita una atención especial. lavado a fondo de las perlas magnéticas para eliminar UrDNA sin hibridar es necesaria para evitar resultados falsos positivos. Care también tiene que tener cuidado para evitar la acumulación de perlas magnéticas residuales en la superficie interior de la lIdentificación del tubo de microfuga, que puede ser difícil de ver. Una vez allí, las perlas magnéticas ya no están sometidos a la separación magnética y, por tanto, podrían llevar a algunos UrDNA no hibridada que puede conducir a falsos positivos señales en la reacción indicadora. También es importante para evitar dejar el tubo de microcentrífuga en el estante magnético durante más de 10 min durante la etapa de separación magnética. Las perlas pueden agregar o pegar al tubo de microfuga, que puede reducir la eficacia del lavado e introducir inconsistencia de lote a lote. La inclusión de un 0,01% de Tween-20 en la solución de lavado puede mejorar la consistencia de lote a lote y debe ser implementado.
Las perlas magnéticas se revisten con estreptavidina, que se utilizó como el ancla de montar conjugados ADNzima-ureasa sobre las perlas magnéticas. Tanto estreptavidina y ureasa son moléculas de proteínas que pueden ser desnaturalizadas durante el almacenamiento. Nos suelen almacenar los granos ADNzima-ureasa magnéticos montados a 4 ° C durante un máximo de 4 semanasy hacer lotes frescos regularmente para conseguir resultados más consistentes.
Cuidado también tiene que tener cuidado para evitar accidentalmente tomar perlas magnéticas en la etapa de separación magnética (paso 6.9) tras la activación ADNzima. En nuestra experiencia, los desechos celulares y otras partículas en la solución puede reducir la eficiencia de la separación magnética, y por lo tanto, algunas perlas magnéticas pueden ser sacado involuntariamente durante el pipeteado. Esto dará lugar a resultados falsos positivos. Recomendamos las siguientes medidas para aliviar el problema: un tiempo de separación más larga (por ejemplo, 5 a 10 min), una liberación más lenta de la presión sobre la pipeta para permitir la retirada suave del sobrenadante, y someter el sobrenadante a una ronda adicional de separación magnética .
Por último, es importante evitar la contaminación de la solución de accidente reportero de stock de ureasa durante el curso de un experimento en el que se ponen a prueba múltiples muestras. Dada la alta reactividad de uRease, la contaminación de esta naturaleza puede dar lugar a resultados falsos positivos.
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
The funding for this research project was provided by the Natural Sciences and Engineering Research Council of Canada (NSERC) via a Discovery Grant to YL.
Materiales
| Name | Company | Catalog Number | Comments |
| Ethylenediaminetetraacetic acid (EDTA) | VWR AMRESCO | 0105 | |
| Sodium Hydroxide (NaOH) pellets | BIO BASIC CANADA INC. | SB6789 | |
| Tris-base | VWR AMRESCO | 0497 | |
| Boric acid | AMRESCO | 0588 | |
| Urea | VWR AMRESCO | M123 | |
| 40% acrylamide/bisacrylamide (29:1) solution | BIO BASIC CANADA INC. | A0007 | |
| Sucrose | Bioshop Canada inc. | SUC507 | |
| Bromophenol blue | Bioshop Canada inc. | BRO777 | |
| Xylenecyanol FF | SIGMA-ALDRICH | X-4126 | |
| 10% sodium dodecyl sulfate | Bioshop Canada inc. | SDS001 | |
| Hydrochloric Acid (HCl) | CALEDON LABORATORIES LTD | 6026 | |
| Sodium Chloride (NaCl) | Bioshop Canada inc. | SOD001 | |
| 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid (HEPES) | Bioshop Canada inc. | HEP001 | |
| Magnesium Chloride (II) hexahydrate | VWR AMRESCO | 0288 | |
| Tween 20 | Bioshop Canada inc. | TW508 | |
| Adenosine Triphospahte (ATP) | AMRESCO | 0220 | |
| Sodium Acetate trihydrate (NaOAc) | SIGMA-ALDRICH | S8625 | |
| Ethanol | Commercial Alcohols | P016EAAN | |
| Tetramethyleneethylenediamine (TEMED) | AMRESCO | 0761 | |
| 10% Ammonium persulfate (APS) | BIO BASIC CANADA INC. | AB0072 | |
| Succinimidyl 4-(N-maleimidomethyl) cyclohexane-1-carboxylate (SMCC) | ThermoFisher SCIENTIFIC | 22360 | |
| Dimethyl sulfoxide (DMSO) | CALEDON LABORATORIES | 803540 | |
| Urease | SIGMA-ALDRICH | U0251 | |
| 1x Phosphate Buffered Saline (PBS) | ThermoFisher SCIENTIFIC | 70011-069 | |
| 0.04% Phenol red | SIGMA-ALDRICH | P3532 | |
| 10x T4 polynucleotide kinase reaction buffer | Lucigen | 30061-1 | |
| 10x T4 DNA ligase reaction buffer | Bio Basics Canada | B1122-B | |
| T4 DNA ligase (5 U/μl) | Thermo Fischer Scientific | B1122 | |
| Luria Bertani (LB) Broth | AMRESCO | J106 | |
| Agar | AMRESCO | J637 | |
| T4 polynucleotide kinase (10 U/μl) | Lucigen | 30061-1 | |
| E. coli K12 (MG1655) | ATCC | ATCC700926 | |
| Centrifuge | Beckman Coulter, Inc. | 392187 | |
| Glass plates | CBS scientific | ngp-250nr | |
| 0.75 mm thick spacers | CBS scientific | VGS-0725r | |
| 12-well comb | CBS scientific | VGC-7512 | |
| UV Lamp | UVP | 95-0017-09 | |
| Spectrophotometer (NanoVue) | GE Healthcare | N/A | |
| Metal plate | CBS scientific | CPA165-250 | |
| Vortex | VWR International | 58816-123 | |
| Gel electrophoresis apparatus | CBS scientific | ASG-250 | |
| Petri dishes | VWR International | 25384-342 | |
| 100 kDa MWCO centrifugal filters | EMD Millipore | UFC510024 | |
| Magnetic Bead (BioMag) | Bangs Laboratories Inc | BM568 | |
| Magnetic Seperation Rack | New England BioLabs | S1506S | |
| Microfuge tubes | Sarstedt | 72.69 | |
| Syringe filter (0.22 μm) | VWR International | 28145-501 | |
| 14 ml culture tube | VWR International | 60818-725 | |
| Cell culture incubator | Eppendorf Scientific | M13520000 | |
| Branson Ultrasonic cleaner | Branson | N/A | |
| Camera (Canon Powershot G11) | Canon | N/A | |
| 50 ml conical tube | VWR International | 89004-364 |
Referencias
- Daar, A. S., et al. Top ten biotechnologies for improving health in developing countries. Nat. Genet. 32, 229-232 (2002).
- Newman, J. D., Turner, A. P. Home blood glucose biosensors: a commercial perspective. Biosens. Bioelectron. 20, 2435-2453 (2005).
- Turner, A. P. Biosensors: sense and sensibility. Chem. Soc. Rev. 42, 3184-3196 (2013).
- Tram, K., Kanda, P., Salena, B. J., Huan, S. Y., Li, Y. F. Translating Bacterial Detection by DNAzymes into a Litmus Test. Angew. Chem. Int. Ed. 53, 12799-12802 (2014).
- Ali, M. M., Aguirre, S. D., Lazim, H., Li, Y. Fluorogenic DNAzyme probes as bacterial indicators. Angew. Chem. Int. Ed. 50, 3751-3754 (2011).
- Schlosser, K., Li, Y. Biologically inspired synthetic enzymes made from DNA. Chem. Biol. 16, 311-322 (2009).
- Tuerk, C., Gold, L. Systematic evolution of ligand by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Breaker, R. R. Making catalytic DNAs. Science. 290, 2095-2096 (2000).
- Liu, J., Cao, Z., Lu, Y. Functional nucleic acid sensors. Chem. Rev. 109, 1948-1998 (2009).
- Navani, N. K., Li, Y. Nucleic acid aptamers and enzymes as sensors. Curr. Opin. Chem. Biol. 10, 272-281 (2006).
- Li, J., Lu, Y. A highly sensitive and selective catalytic DNA biosensor for lead ions. J. Am. Chem. Soc. 122, 10466-10467 (2000).
- Liu, J., Lu, Y. A colorimetric lead biosensor using DNAzyme-directed assembly of gold nanoparticles. J. Am. Chem. Soc. 125, 6642-6643 (2003).
- Liu, Z., Mei, S. H. J., Brennan, J. D., Li, Y. Assemblage of signaling DNA enzymes with intriguing metal specificity and pH dependence. J. Am. Chem. Soc. 125, 7539-7545 (2003).
- Liu, J., et al. A catalytic beacon sensor for uranium with parts-per-trillion sensitivity and millionfold selectivity. Proc. Natl. Acad. Sci. USA. 104, 2056-2061 (2007).
- Huang, P. J., Vazin, M., Liu, J. In vitro selection of a new lanthanide-dependent DNAzyme for ratiometric sensing lanthanides. Anal. Chem. 86, 9993-9999 (2014).
- Chiuman, W., Li, Y. Simple fluorescent sensors engineered with catalytic DNA 'MgZ' based on a non-classic allosteric design. PLoS One. 2, e1224(2007).
- Xiang, Y., Lu, Y. Using personal glucose meters and functional DNA sensors to quantify a variety of analytical targets. Nat. Chem. 3, 697-703 (2011).
- Aguirre, S. D., Ali, M. M., Salena, B. J., Li, Y. A sensitive DNA enzyme-based fluorescent assay for bacterial detection. Biomolecules. 3, 563-577 (2013).
- Aguirre, S. D., Ali, M. M., Kanda, P., Li, Y. F. Detection of Bacteria Using Fluorogenic DNAzymes. J. Vis. Exp. (63), e3961(2012).
- Shen, Z., et al. A catalytic DNA activated by a specific strain of bacterial pathogen. Angew. Chem. Int. Ed. 54, (2015).
- He, S., et al. Highly specific recognition of breast tumors by an RNA-cleaving fluorogenic DNAzyme probe. Anal. Chem. 87, 569-577 (2015).
- Sumner, J. B., Hand, D. B. Isoelectric point of crystalline urease. J. Am. Chem. Soc. 51, 1255-1260 (1929).
- Karplus, P. A., Pearson, M., Hausinger, R. P. 70 Years of crystalline urease: What have we learned. Acc. Chem. Res. 30, 330-337 (1997).
- Omiccioli, E., Amagliani, G., Brandi, G., Magnani, M. A new platform for Real-Time PCR detection of Salmonella spp., Listeria monocytogenes and Escherichia coli O157 in milk. Food Microbiol. 26, 615-622 (2009).
- Cui, S., Schroeder, C. M., Zhang, D. Y., Meng, J. Rapid sample preparation method for PCR-based detection of Escherichia coli O157:H7 in ground beef. J. Appl. Microbiol. 95, 129-134 (2003).
- Ibekwe, A. M., Watt, P. M., Grieve, C. M., Sharma, V. K., Lyons, S. R. Multiplex fluorogenic real-time PCR for detection and quantification of Escherichia coli O157:H7 in dairy wastewater wetlands. Appl. Environ. Microbiol. 68, 4853-4862 (2002).
- Strachan, N. J., Ogden, I. D. A sensitive microsphere coagulation ELISA for Escherichia coli O157:H7 using Russell's viper venom. FEMS Microbiol Lett. 186, 79-84 (2000).
- de Boer, E., Beumer, R. R. Methodology for detection and typing of foodborne microorganisms. Int. J. Food Microbiol. 50, 119-130 (1999).
- Gracias, K. S., McKillip, J. L. A review of conventional detection and enumeration methods for pathogenic bacteria in food. Can. J. Microbiol. 50, 883-890 (2004).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados