Method Article
Kolorimetrischen Nachweis von Bakterien mit Lackmus-Test
In diesem Artikel
Zusammenfassung
We describe a protocol for colorimetric detection of E. coli using a modified litmus test that takes advantage of an RNA-cleaving DNAzyme, urease, and magnetic beads.
Zusammenfassung
There are increasing demands for simple but still effective methods that can be used to detect specific pathogens for point-of-care or field applications. Such methods need to be user-friendly and produce reliable results that can be easily interpreted by both specialists and non-professionals. The litmus test for pH is simple, quick, and effective as it reports the pH of a test sample via a simple color change. We have developed an approach to take advantage of the litmus test for bacterial detection. The method exploits a bacterium-specific RNA-cleaving DNAzyme to achieve two functions: recognizing a bacterium of interest and providing a mechanism to control the activity of urease. Through the use of magnetic beads immobilized with a DNAzyme-urease conjugate, the presence of bacteria in a test sample is relayed to the release of urease from beads to solution. The released urease is transferred to a test solution to hydrolyze urea into ammonia, resulting in an increase of pH that can be visualized using the classic litmus test.
Einleitung
Bacterial pathogens are one of the major causes of global morbidity and mortality. Outbreaks from hospital-acquired infections, food-borne pathogens, and bacterial contaminants in the environment pose serious and on-going threats to public health and safety. To prevent these outbreaks, effective tools are needed that permit pathogen detection in a timely fashion under a variety of settings. Simple but still effective tests that are portable and cost-effective are greatly coveted, especially in regions that are susceptible to outbreaks but cannot afford expensive testing facilities.1-3 Although there exists a multitude of methods to detect bacteria, many of them are not suitable as screening or on-site testing tools because they require long test times, expensive instruments and complicated testing procedures.
Colorimetric tests are particularly attractive for point-of-care or field applications as color changes can be easily detected by the naked eye. The litmus test for pH is simple, quick, and effective. Although it is a very old technology, it is still widely used today because of its simplicity and effectiveness. Surprisingly, this simple test had never been modified to achieve the detection of other analytes before we recently developed an approach of modifying this test for E. coli testing.4
The expanded litmus test for E. coli employs three additional components: an E. coli activated RNA-cleaving DNAzyme (EC1), 5 urease, and magnetic beads. DNAzymes refer to synthetic single-stranded DNA molecules with catalytic activity.6 They can be isolated from random-sequence DNA pools using in vitro selection.7,8 They are highly stable and can be produced cost-effectively using high-efficiency automated DNA synthesis.9 For these reasons, DNAzymes, particularly RNA-cleaving DNAzymes, have been widely examined for biosensing applications.6,10,11 RNA-cleaving DNAzyme sensors have been developed to detect metal ions,12-16 small molecules,17,18 bacterial pathogens5,19-21 and cancer cells.22 Given the great availability of target-induced RNA-cleaving DNAzymes, any assay that utilizes a DNAzyme can be potentially expanded to detect a diverse range of analytes.
Urease is chosen for its ability to hydrolyze urea into ammonia,23,24 resulting in a pH increase. Urease is also highly efficient, stable and amenable for conjugation to other biomolecules. Therefore, we postulated that a conjugate of an RNA-cleavage DNAzyme with urease would allow the use of litmus test for the detection of other targets.5
The action of the RNA-cleaving DNAzyme is relayed to urease-mediated increase of pH through the use of magnetic beads that are immobilized with the DNAzyme-urease conjugate. Because the activity of the DNAzyme under investigation is strictly dependent on E. coli, the presence of this bacterium in the test solution will result in the release of urease from the magnetic beads to the solution, which is then taken and used to hydrolyze urea in a reporter solution that contains a pH-sensitive dye. The final outcome of this procedure is a color change that can be conveniently reported by the dye or pH paper.
Protokoll
1. Vorbereitung der Reagenzien und Puffer
- 0,5 M Ethylendiamintetraessigsäure (EDTA)
- In einem 2 - Liter - Becher, fügen 186,1 g EDTA zu 800 ml entionisiertem destilliertem Wasser (ddH 2 O). Der pH-Wert der Lösung auf 8,0 unter Verwendung von NaOH-Pellets. In ddH 2 O auf ein Endvolumen von 1,0 l und die Lösung in einem autoklavierbaren Glasflasche für Autoklaven und lagern bei 4 ° C.
- 10 × Tris-Borat-EDTA (10x TBE)
- In einem 4 L - Plastikbecher, fügen Sie 432 g Tris-Base, 200 g Borsäure, 80 ml 0,5 M EDTA (pH 8,0) und ddH 2 O auf ein Endvolumen von 4 L. Mix mit einem Rührstab , bis alle Komponenten gelöst. Die Lösung wird auf 1 Liter Glasflaschen für Autoklaven und lagern bei 4 ° C.
- 10% denaturierende Polyacrylamid-Auf
- In einem 4 L - Plastikbecher, fügen 1681,7 g Harnstoff, 400 ml 10x TBE, 1 l 40% Acrylamid / Bisacrylamid (29: 1) Lösung und ddH 2 O bis final Volumen 4 L. Mix einen Rührstab mit bis sich der Harnstoff gelöst ist. Die Lösung wird auf 1 Liter Braunglasflaschen und lagern bei 4 ° C.
- 2x Gelladepuffer (2x GLB)
- In einem 200 ml Becherglas, fügen 44 g Harnstoff, 8 g Saccharose, 10 mg Bromphenolblau, 10 mg Xylolcyanol FF, 400 ul 10% Natriumdodecylsulfat, und 4 ml 10 x TBE. Hinzufügen ddH 2 O auf ein Endvolumen von 40 ml und lösen sich die Komponenten unter gelindem Erwärmen auf 50 ° C unter Rühren mit einem magnetischen Rührstab gemischt wird . 1 ml aliquoten bis 1,5 ml Mikrozentrifugenröhrchen und lagern bei 4 ° C.
HINWEIS: Vor Gebrauch erfordert kurzes Erhitzen bei 90 ° C keine Feststoffe wieder zu lösen.
- In einem 200 ml Becherglas, fügen 44 g Harnstoff, 8 g Saccharose, 10 mg Bromphenolblau, 10 mg Xylolcyanol FF, 400 ul 10% Natriumdodecylsulfat, und 4 ml 10 x TBE. Hinzufügen ddH 2 O auf ein Endvolumen von 40 ml und lösen sich die Komponenten unter gelindem Erwärmen auf 50 ° C unter Rühren mit einem magnetischen Rührstab gemischt wird . 1 ml aliquoten bis 1,5 ml Mikrozentrifugenröhrchen und lagern bei 4 ° C.
- 1 M Tris-Hydrochlorid (Tris-HCl) (pH 7,5)
- In einer Glasflasche, fügen 12,1 g Tris-Base und 70 ml ddH 2 O und mischen , bis der Feststoff gelöst ist. Einstellung des pH auf 7,5 mit 1 M Salzsäure (HCl). Hinzufügen ddH 2 O auf ein Endvolumen von 100 ml, Autoklaven und store bei 4 ° C
- 5 M Natriumchlorid (NaCl)
- In eine Glasflasche, lösen sich 58,4 g NaCl in 150 ml ddH 2 O. Stellen Sie die Lautstärke auf 200 ml mit ddH 2 O. Lagerung bei 4 ° C.
- DNA Elution Buffer
- In eine Glasflasche wurden 2,0 ml von 1 M Tris-HCl (pH 7,5) mischen, 8,0 ml 5 M NaCl und 0,4 ml 0,5 M EDTA (pH 8,0). Stellen Sie die Lautstärke auf 200 ml mit ddH 2 O, Autoklaven und lagern bei 4 ° C.
- 1 M 4- (2-Hydroxyethyl) -1-piperazinethansulfonsäure (HEPES) (pH 7,4)
- In einer Glasflasche, lösen sich 2,38 g HEPES in 80 ml ddH 2 O. Den pH - Wert auf 7,4 unter Verwendung von 5 N NaOH und fügen ddH 2 O auf ein Endvolumen von 100 ml.
- 1 M Magnesiumchlorid (MgCl 2)
- In einer Glasflasche, fügen 2,03 g MgCl 2 · 6H 2 O und bringen das Volumen auf 100 ml mit ddH 2 O.
- Reaktionspuffer (RB)
- In einem 50 ml konischen Röhrchen wurden 50 ul 1 M HEPES (pH 7,4), 1,5 ml 5 M NaCl, 0,75 ml 1 M MgCl 2 und 5 & mgr; l Tween-20 hinzu. Hinzufügen ddH 2 O auf ein Endvolumen von 50 ml. Mischen Sie die Lösung und den Filter in eine andere konische Röhrchen mit einer Spritze gesteuerten Filter (0,22 & mgr; m) und bei 4 ° C bis zur Verwendung.
- Binding Buffer (BB)
- In einem 50 ml konischen Röhrchen, 500 ul 1 M Tris-HCl (pH 7,5), 8,8 g NaCl, 50 ul 1 M MgCl 2 und 5 & mgr; l Tween 20. In ddH 2 O auf ein Endvolumen von 50 ml. Mischen Sie die Lösung und den Filter in eine andere konische Röhrchen mit einer Spritze gesteuerten Filter (0,22 & mgr; m) und bei 4 ° C bis zur Verwendung.
- Substratlösung (SS)
- In einem 50 ml konischen Röhrchen, fügen Sie 5,8 g NaCl, 3 ml 1 M MgCl 2, 1,5 g Harnstoff und 40 ml ddH 2 O. Der pH-Wert der Lösung auf 5,0 unter Verwendung von 10 mM HCl. Da die Lösung nicht gepuffert ist, pH-Einstellung mit HClvorsichtig durch die Zugabe von kleinen Aliquots HCl. Hinzufügen ddH 2 O auf ein Endvolumen von 50 ml.
- Mischen Sie die Lösung und den Filter in eine andere konische Röhrchen mit einer Spritze gesteuerten Filter (0,22 & mgr; m) und bei 4 ° C bis zur Verwendung.
- Luria Bertani (LB) Broth
- In einem Becherglas löst man 20,0 g LB - Pulver in 1 l ddH 2 O. Übertragung auf eine Glasflasche, Autoklaven und lagern bei 4 ° C.
- 1,5% LB-Agar
- In einem 250 ml Kolben, fügen 1,5 g Agar und 100 ml LB-Brühe. Autoklav und lagern bei 4 ° C.
- Agar-Platten
- Abgeblasen, und der LB-Agar in einer Mikrowelle und die Lösung auf ~ 50 ° C. Füllen Sie die Lösung in Petrischalen, so dass ~ 5 Platten und es ihnen ermöglichen, sich zu verfestigen.
2. Synthese und Reinigung von E. Colireaktions DNAzyms EC1
- Synthese von EC1 durch Vorlage Mediated enzymatische Ligation
- Reinige commercially synthetisierte Oligonukleotide BS1, DE1 und T1 (Sequenzen in Tabelle 1) , um 10% denaturierende Polyacrylamidgelelektrophorese (DPAGE) nach Standardprotokollen.
- Bereiten Sie eine 100 & mgr; M Lager von BS1, DE1 und T1. Lagerung bei -20 ° C bis zur Verwendung.
- In einem 1,5 ml Mikrozentrifugenröhrchen, fügen 38,5 & mgr; l ddH 2 O, 5 ul DE1 und 5 ul 10fach T4 Polynucleotidkinase - Reaktionspuffer durch Enzym - Lieferanten zur Verfügung gestellt (500 mM Tris-HCl (pH 7,6), 100 mM MgCl 2, 50 mM Dithiothreitol (DTT), 1,0 mM Spermidin).
- 1 & mgr; l von Adenosintriphosphat (ATP) (100 mM). Werden 5 Einheiten T4-Polynukleotidkinase (10 U / ul). Mischen Sie durch sorgfältig die Reaktionsmischung Pipettieren.
- Inkubieren der Reaktion bei 37 ° C für 30 min.
- Quenche die Reaktion durch Erhitzen auf 90 ° C für 5 min.
- Hinzufügen , 118 & mgr; l ddH 2 O, 5 ul BS1, und 5 ul T1. Die Reaktion wird auf 90 ° C für 2 min und dann auf Raumtemperatur abkühlen über 10 min.
- Geben Sie 20 & mgr; l von 10 × T4 - DNA - Ligase - Reaktionspuffer durch das Enzym Lieferanten zur Verfügung gestellt (400 mM Tris-HCl (pH 7,8), 100 mM MgCl 2, 100 mM DTT, 5 mM ATP). In 10 Einheiten T4-DNA-Ligase (5 U / ul) und vorsichtig durch Pipettieren mischen.
- Inkubieren bei Raumtemperatur für 2 Std.
- Geben Sie 20 & mgr; l 3 M Natriumacetat (NaOAc) (pH 5,2), 500 ul kaltem 100% Ethanol. Mischen Sie die Lösung durch Verwirbelung und legen Sie den Schlauch in einem -20 ° C Gefrierschrank für 30 min.
- Zentrifugieren Sie die Mikrofuge bei 20.000 × g für 20 min bei 4 ° C. Entfernen Sie vorsichtig den Überstand durch Pipettieren.
- Waschen des Pellets mit kaltem 70% Ethanol und zentrifugiere wieder bei 20.000 xg für 10 min bei 4 ° C. Wieder den Überstand zu entfernen, und das Pellet unter Vakuum für 10 min trocknen.
- Resuspendieren der DNA mit 15 ul ddH 2 O und dann 15 ul 2x GLB hinzufügen.
- Kräftig vortexen und Hitze auf 90 ° C für2 Minuten. Reinige den Volllängen-DNA um 10% DPAGE wie unten beschrieben.
- Einrichten 10% DPAGE
- Reinigen Sie zwei Glasplatten (eine volle Platte und einem gekerbten), zwei 0,75 mm dicken Abstandshalter und eine 12-Well-Kamm. Legen Sie eine Glasplatte auf einer ebenen Fläche mit zwei Abstandshalter auf jeder Seite und der gekerbten Platte an der Spitze. Clip die beiden Platten zusammen mit den vier Clips vom Lieferanten zur Verfügung gestellt.
- In einem 150 ml Kunststoffbecher, gieße 40 ml 10% DPAGE, 40 ul Tetramethylethylendiamin (TEMED) und 400 ul 10% Ammoniumpersulfat (APS). Die Komponenten mischen und gießen Sie die Lösung zwischen den Platten langsam.
- Setzen Sie den Kamm und dann lassen Sie das Gel über 10 Minuten zu polymerisieren. Sobald das Gel polymerisiert wird, langsam den Kamm zu entfernen und die Vertiefungen mit ddH 2 O spülen
- Montieren Sie die Platten auf das Gel Elektrophorese-Vorrichtung. Verwenden Sie eine Metallplatte zu leiten Wärme erzeugt eine Überhitzung zu vermeiden.
- Gießen 1x TBE auf den oberen und unteren Kammernund sicherzustellen, dass die Vertiefungen werden auch in den Puffer eingetaucht. Spülen Sie die Brunnen mit 1x TBE mit einer Pipette oder Spritze.
- Stellen Sie das Gerät bei 35 mA und Vorlauf für 15 Minuten vor dem Laden der Proben zu laufen.
- Die Elution von ligierte EC1 von 10% DPAGE
- Nach dem Schritt 2.2.6, führen Sie das Gel bei 35 mA, bis der Boden Farbstoff (Bromphenolblau) erreicht den Boden der Glasplatte. Dies sollte etwa 1,5 Stunden dauern. Schalten Sie das Gerät aus und entfernen Sie die Platten aus dem Gerät.
- Legen Sie die Platten auf ein paar Blatt Papier Handtücher und sorgfältig die Abstandshalter von den Glasplatten zu entfernen. Entfernen Sie vorsichtig die obere Glasplatte und wickeln Sie das Gel mit Plastikfolie.
- Drehen Sie das Gel auf die zweite Glasplatte entfernen und die Abdeckung mit Plastikfolie wieder. Achten Sie darauf, Falten der Plastikfolie zu vermeiden.
- Visualisieren Sie die ligierte Produkt durch UV-Shadowing (260 nm) verwendet, die vier verschiedene DNA-Banden produzieren wird (Fully ligierten EC1, DE1, BS1, und T1).
- Verbrauch das obere Band (EC1) mit einer sterilen Rasierklinge und in ein 1,5 ml Mikrozentrifugenröhrchen. Crush das Gel mit einer sterilen Pipettenspitze (200 ul Spitze Größe), bis er in einer feinen Paste verwandelt. In 550 & mgr; l DNA-Elutionspuffer, decken Sie das Rohr mit Aluminiumfolie des eingebetteten Fluorophors zu schützen, und schütteln für 15 min.
- Zentrifugieren Sie die Gel-Lösung bei 20.000 xg für 5 min und transferieren es vorsichtig 400 ul des Überstandes auf einen anderen 1,5 ml Mikrozentrifugenröhrchen. Vermeiden Sie Gelstücke beim Pipettieren zurückzog.
- An den Mikrozentrifugenröhrchen mit dem übertragenen Überstand werden 40 ul 3 M NaOAc (pH 5,2) und 1,0 ml kaltem 100% Ethanol. Mischen Sie die Lösung durch Verwirbelung und legen Sie den Schlauch in einem -20 ° C Gefrierschrank für 30 min.
- Zentrifugieren Sie die Mikrofuge bei 20.000 × g für 20 min bei 4 ° C. Entfernen Sie vorsichtig den Überstand durch Pipettieren.
- Waschen des Pellets mit kaltem 70% Ethanol und zentrifugiere wieder bei 20.000 xg für 10 min bei 4 ° C. Wieder einmal, entfernen Sie dieÜberstand und das Pellet unter Vakuum für 10 min trocknen.
- Bestimmung der Konzentration der EC1 durch UV-Absorption bei 260 nm gemessen wird. Machen Sie eine 10 uM Lager und lagern Sie die Probe bei -20 ° C bis zur Verwendung.
3. Konjugation von Urease zu DNA
- Herstellung von Succinimidyl-4- (N-maleimidomethyl) cyclohexan-1-carboxylat (SMCC) Stock
- Man löst 1 mg SMCC in 676 ul DMSO. Vortex und auf Eis bis zum Gebrauch.
- Herstellung von Urease Auf
- Man löst 1 mg Urease in 1 ml 1 × PBS (ohne Mg 2+ oder Ca 2+). Auf Eis bis zum Gebrauch.
HINWEIS: Crystallized Urease ist langsam aufzulösen und schonendes Mischen ist erforderlich, eine Denaturierung zu vermeiden.
- Man löst 1 mg Urease in 1 ml 1 × PBS (ohne Mg 2+ oder Ca 2+). Auf Eis bis zum Gebrauch.
- Synthese von Urease-DNA (UrDNA)
- Zugabe von 10 ul 100 uM LD1 zu einem 2,5 ml Mikrozentrifugenröhrchen. In 140 ul ddH 2 O, 40 ul 10x PBS und Wirbel.
- In 80,5 ul SMCC stock, 159,5 ul DMSO, Wirbel, und kurz Zentrifuge ein Benchtop-Zentrifuge.
- für 60 min bei 37 ° C inkubieren. Vermeiden Sie Kondensation unter der Mikrofuge Kappe.
- Mit 200 & mgr; l 1x PBS, 60 ul 3 M NaOAc (pH 5,2) und 1,5 ml kaltem 100% Ethanol. Mischen Sie die Lösung durch Vortexen und Inkubation bei -20 ° C für 30 min.
- Zentrifugieren Sie die Lösung bei 20.000 × g für 20 min bei 4 ° C. Entfernen Sie den Überstand und trocknen Sie das Pellet unter Vakuum. Vermeiden Sie Übertrocknung.
- Zu dem getrockneten Pellet 400 ul Urease Lager hinzufügen und bei Raumtemperatur für 5 Stunden inkubiert.
- Übertragung von 200 ul rohem Konjugat zu einem vorgewaschenen 100k MWCO Zentrifugalfilter Säule. Zentrifugieren Sie die Säule bei 14.000 xg für 5 min.
- Übertragen, um die verbleibenden 200 ul Rohkonjugat auf die Säule und Zentrifugieren bei 14.000 xg für 5 min. Entfernen Sie die Spalte und legen Sie es auf den Kopf in einem neuen 2,0 ml Mikrozentrifugenröhrchen ( "Sammelröhrchen").
- Zentri- fugalkruge das Sammelrohr (mit der invertierten Spalte) bei 1000 × g für 2 min. Entfernen Sie die Sammelröhrchen und fügen Sie 30 ul 1x PBS auf die zentrifugalen Spalte die Membran für zusätzlichen Gewinnung von Konjugate zu waschen.
- Wieder einmal die Spalte umkehren und wieder in das Sammelrohr platzieren. Zentrifugieren Sie das Sammelrohr (mit der invertierten Spalte) bei 1000 × g für 2 min. Entfernen Sie die Spalte zu entsorgen.
- Lagern Sie die UrDNA bei 4 ° C bis zur Verwendung.
4. Montage von EC1 und UrDNA auf Magnetic Beads
- Mischen Sie die magnetischen Kügelchen (MB) Lager gut und Transfer 100 ul MB Suspension in ein 1,5 ml Mikrozentrifugenröhrchen. Setzen Sie den Mikrozentrifugenröhrchen auf einem Magnethalterahmen für den MB zu isolieren.
- Entfernen Sie den Überstand durch Pipettieren und fügen 150 ul Bindungspuffer (BB) an das Rohr. Entfernen Sie den Schlauch aus dem Halter und vorsichtig den Schlauch tippen Sie auf die MB zu einer homogenen Lösung zu suspendieren. Vermeiden Sie die Suspension auf die t spritztop des Rohres oder Kappen. Wenn dies der Fall ist, verwenden Sie einen Tischzentrifuge kurz um den Rückstand zu drehen zurück in die Suspension.
- Wiederholen Sie Schritt 4.2 zwei weitere Male.
- Zu dieser Suspension 10 ul von 10 uM EC1 hinzuzufügen. sorgfältig mischen, indem Sie auf das Rohr klopfen.
- Inkubieren Sie die Lösung mit milden Schütteln für 30 min. Tippen Sie das Röhrchen alle 2-3 Minuten, um die Fällung des MB zu vermeiden.
- Setzen Sie den Mikrozentrifugenröhrchen wieder auf dem Magnet Rack mit dem MB zu isolieren und den Überstand mit einer Pipette entfernen. Waschen Sie die MB dreimal mit 150 ul BB (wie in Schritt 4.2 beschrieben).
- Sobald das Waschen abgeschlossen ist, setzt sie die MB in insgesamt 150 ul BB. Zu dieser Lösung werden 15 ul UrDNA und die Lösung auf 45 ° C für 2 min erwärmt. Die Lösung wird auf Raumtemperatur und Inkubation für 2 Stunden.
- Setzen Sie den Mikrozentrifugenröhrchen wieder auf dem Magnet Rack mit dem MB zu isolieren und den Überstand mit einer Pipette entfernen.
- 100 l Reaktionspuffer (RB). entfernen ter Mikrozentrifugenröhrchen aus dem magnetischen Rack und sorgfältig die MB resuspendieren.
- Waschen Sie die MB noch drei Mal für Schritt 4.9 wiederholen.
HINWEIS: Der gewaschene Überstand schnell getestet werden können, um festzustellen, ob unhybridisiert UrDNA noch vorhanden ist, was zu einem falsch-positiven Signal führen kann. Der Test kann durch Zugabe von 10 ul 50 mM Harnstoff und 10 ul 0,04% Phenolrot in die Waschlösung durchgeführt werden. Fortsetzung der MB zu waschen, bis die Waschlösung nicht um eine Farbänderung verursacht. Die 100 & mgr; l Suspension wird bei 4 ° C bis zur Verwendung gelagert.
5. Herstellung von Bakterienzellen 20
- Die Anzucht E. coli aus Aktien
- Platte E. coli K12 (MG1655) auf LB - Agar - Platten aus einem Glycerin - Stamm unter einer Flamme oder in einem biologischen Sicherheitsschrank.
- Mit einer sterilen Pipettenspitze, sanft berühren die Glycerolstammlösung und leicht Streifen die Oberfläche einer Agarplatte Punktierung des LB-Agar zu vermeiden.
- Kehren Sie die strichenenPlatte und für 14 Stunden bei 37 ° C inkubiert.
- Verschließen Sie die Platte mit Parafilm und lagern bei 4 ° C für maximal 3 Wochen.
- Die Anzucht E. coli für Zellzählung
- In einem sterilen Kulturröhrchen 14 ml, verzichtet werden 2 ml LB-Brühe.
- Mit einer sterilen Pipettenspitze, wählen Sie eine einzelne Kolonie von der Agarplatte ausgestrichen, hergestellt in Schritt 5.1 und überträgt es auf dem Kulturröhrchen.
- Inkubieren Sie die Kultur bei 37 ° C und Schütteln bei 230 Umdrehungen pro Minute für 14 Stunden.
- Abwechselnd verdünnen, um die Bakterienkultur in 10-fach-Intervallen.
- Für jede verdünnte Probe, die Platte gleichmäßig fünf 100 ul Aliquots auf separaten LB-Agarplatten. Kehren Sie die Platten und Inkubation bei 37 ° C für 14 Stunden.
- Zählen Sie die Zellen jeder Probe die durchschnittliche Zellkonzentration von jeder Verdünnung zu erhalten.
HINWEIS: 10 7 Zellen werden häufig eine Referenz Lackmustest einzurichten für E. verwendet coli als dieser Ebene von E. coli kann eine schnelle Farb chan auslösenge. Allerdings kann ein ordnungsgemäß ausgeführt Lackmus-Test so günstig wie 500 Zellen erkennen, wie im Abschnitt Ergebnisse diskutiert.
- Vorbereiten E. coli - Zellen für die Prüfung
- Für eine gewünschte Zellsuspension, 1 ml der gezüchteten Lager in ein 1,5 ml Mikrozentrifugenröhrchen.
- Zentrifugieren der Zellen bei 6000 × g für 10 min bei 4 ° C. Entfernen Sie vorsichtig den Überstand ohne das Zellpellet zu stören.
- Zugabe von 10 & mgr; l Reaktionspuffer zu dem Zellpellet und Resuspendieren der Zellen. Beschallen die Zellsuspension für 5 min. Übertragen Sie die Zellsuspension für 5 min auf einen Eisschrank.
- Beschallen die Zellsuspension für weitere 5 min.
- Zentrifugieren Sie die Zellsuspension bei 13.000 × g für 10 min bei 4 ° C. Verwenden Sie den Überstand für die Prüfung (10 ul).
6. Lackmus-Test
- In einem 1,5 ml Mikrozentrifugenröhrchen, Vorwäsche das Röhrchen durch Zugabe und Vortexen 100 & mgr; l Reaktionspuffer (RB) in dem Mikrozentrifugenröhrchen undVerwerfen des Puffers.
- Transfer 15 ul montiert EC1 (Protokoll 4) mit dem Mikrozentrifugenröhrchen gewaschen.
- Waschen Sie die magnetischen Kügelchen durch das Mikrozentrifugenröhrchen auf einem magnetischen Rack platzieren. Entfernen Sie den Überstand durch Pipettieren. Entfernen Sie die Mikrozentrifugenröhrchen aus dem Rack, 100 & mgr; l von RB und resuspendieren sorgfältig die magnetischen Kügelchen.
- Waschen Sie die MB noch zwei Mal für Schritt 6.3 wiederholen.
- Setzen Sie den Mikrozentrifugenröhrchen wieder auf dem Magnetträger, entfernen Sie den Überstand und fügen Sie die 10 & mgr; l E. coli Probe aus Schritt 5.3 vorbereitet.
- Mischen Sie die Probe und magnetische Kügelchen vorsichtig durch leichtes Klopfen auf den Mikrozentrifugenröhrchen.
- Inkubieren der Reaktion bei Raumtemperatur für 1 Std.
- Zu der Reaktion werden 90 & mgr; l ddH 2 O und legen Sie das Mikrozentrifugenröhrchen auf einem Magnethalter.
- Nach ca. 3 min magnetischer Trennung, übertragen vorsichtig 85 ul des Überstandes auf eine 0,5 ml Mikrozentrifugenröhrchen. Ziehen Sie den Überstand langsam aufvermeiden alle magnetischen Kügelchen zu sammeln.
- Zu der obigen Mikrozentrifugenröhrchen 15 ul 0,04% Phenolrot hinzuzufügen, und 100 ul Substratlösung.
- Machen Sie ein Foto in bestimmten Zeitintervallen Farbwechsel aufzeichnen.
HINWEIS: Der pH-Änderung kann auch mit einer Mikroelektrode mit einem pH-Meter überwacht werden. Der Start pH-Wert sollte etwa 5,2-5,5 sein (die Lösung ist gelb). Wenn nicht, kann die Lösung durch Zugabe von 1 mM Acetatpuffer pH 5,0) eingestellt werden.
Ergebnisse
Das Prinzip des bakteriellen Lackmus - Test ist in Abbildung 1 erläutert Der Test verwendet drei Schlüsselmaterialien. Ein RNA - spaltender DNAzyme die von einem bestimmten Bakterium, Urease und magnetische Kügelchen aktiviert. Die DNAzyme wird als molekulares Erkennungselement verwendet hochspezifischen Nachweis eines Bakteriums von Interesse zu erreichen. Urease und magnetische Kügelchen verwendet werden Signaltransduktion der RNA-Spaltungsaktivität der DNAzyme zu erreichen. Dazu gehört die Schaffung von magnetischen Kügelchen, die Urease-DNAzym-Konjugate enthalten. In Gegenwart des Zielbakteriums, das DNAzyme spaltet seine RNA-Bindung. Diese Aktion führt zur Dissoziation von Urease aus magnetischen Kügelchen. Das freigesetzte Urease kann leicht aus magnetischen Kügelchen getrennt werden und verwendet, um eine Farbänderung in einer Reporterlösung zu erzeugen, die Harnstoff und einen pH-empfindlichen Farbstoff enthält. Urease hydrolysiert Harnstoff in Ammoniak, durch die Erhöhung des pH begleitet, die die c-Triggerolor Veränderung des Farbstoffs.
Abbildung 2 stellt eine bakterielle Lackmus - Test , wo EC1, ein E. coli -responsive RNA-Spaltung DNAzyme, wurde als die DNAzyme verwendet und Phenolrot wurde als der pH-Berichterstattungs Farbstoff verwendet. EC1 wurde zuvor von unserer Gruppe aus einem random-Sequenz DNA - Pool unter Verwendung der Technik der in vitro - Selektion isoliert. 5 Unsere frühere Studien haben gezeigt , dass EC1 ist hoch spezifisch für E. coli und eine minimale Aktivität gegenüber anderen Bakterien. 5,19 Es wurde gefunden , daß EC1 durch ein Proteinmolekül aus E. aktiviert wird , coli. Obwohl die Identität dieses Proteins biomarker nicht entschlüsselt worden ist , schlägt die hohe Erkennungsspezifität , dass dieses Protein an E. einzigartig coli. Die Reporter-Lösung eingerichtet wird einen anfänglichen pH-Wert von 5,5 haben. Bei diesem pH zeigt Phenolrot eine gelbe Farbe. Als Urease hydrolysiert Harnstoff zu Ammoniak, um die Basizität des Reporter solution erhöht. Dies wird durch die allmähliche Änderung der Farbe von gelb nach rosa reflektiert. Die Tiefe der Farbänderung wird auf den folgenden zwei Parametern abhängig, wie Abbildung 2 dargestellt: die Zahl der E. coli - Zellen in der DNAzyme Aktivierungsschritt und die Zeit für die Harnstoff Hydrolyseschritt erlaubt verwendet. Mehr E. coli - Zellen führte zu einer stärkeren Farbveränderungen, die sich durch die Beobachtung eines progressiven Gelb-to-rosa Farbübergang , wenn E. coli - Zellen wurden seriell von 5 erhöhte sich auf 5 x 10 7 (10-fache jedes Mal erhöhen). Inzwischen für die Detektion von kleineren Anzahlen von E. eine längere Zeit für die Harnstoffhydrolyse erlaubt coli - Zellen (5000 Zellen in 1 h Reaktions und 500 Zellen in 2-Stunden - Reaktion).
Die pH - Änderung des bakteriellen Lackmus - Test kann auch ein Handheld - pH - Meter und repräsentative Ergebnisse sind in Abbildung 3 dargestellt überwacht werden. Es was festgestellt , dass die Anwesenheit von 10 & sup7 ; E. coli - Zellen führte zu einer allmählichen Erhöhung des pH - Wertes von 3 Einheiten innerhalb von 10 min. Im Gegensatz dazu ist die Abwesenheit von E. coli - Zellen nicht die Ursache nachweisbar pH - Veränderungen unter der gleichen Einstellung.
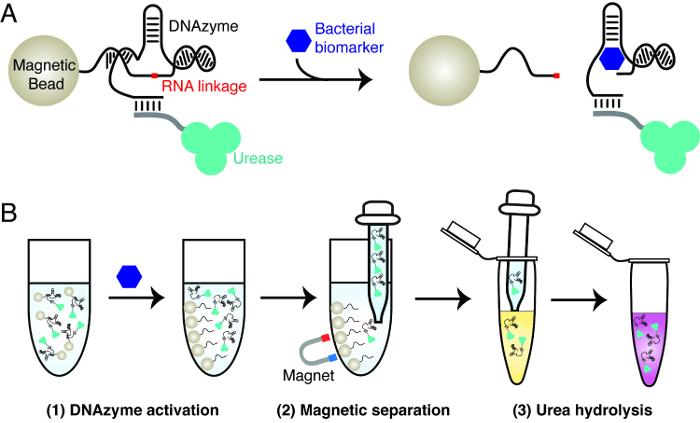
Abbildung 1: Das Konstruktionsprinzip der bakteriellen Lackmus - Test (A) Aktivierung eines RNA - spaltendes DNAzym durch einen spezifischen Biomarker aus einem Bakterium von Interesse.. In Gegenwart des Biomarkers, die RNA-Spaltung DNAzyme auf magnetischen Kügelchen spaltet die RNA-Bindung immobilisiert ist, in der Freisetzung des markierten Urease aus magnetischen Kügelchen zu Lösung führt. (B) Drei-Stufen - Testverfahren. Schritt 1: DNAzyms Aktivierung, wie in Feld A. Schritt 2 beschrieben: Magnetische Trennung - die frei Urease von magnetischen Kügelchen getrennt ist. Schritt 3: Harnstoffhydrolyseprozess R12; die frei Urease wird in eine harnstoffhaltige Reporterlösung gegeben. Urease hydrolisiert Harnstoff in Ammoniak, was zu einer Änderung des pH - Werts, der von einem pH-sensitiven Farbstoff gemeldet werden können. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.

Abbildung 2: Lackmus - Test mit E. coli unter Verwendung von E. coli -responsive DNAzyms EC1. Repräsentative Farbwechsel - Ergebnisse mit einer unterschiedlichen Anzahl von E. coli - Zellen über jedem Teströhrchen vorgesehen. Phenolrot wurde als der pH-sensitiven Farbstoff verwendet. Ein Test ohne E. coli wurde als Negativkontrolle verwendet. Mehr E. coli - Zellen wird erwartet , dass die Freisetzung von mehr Urease - Molekülen zu verursachen, begleitet by kräftigere Farbe ändert. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
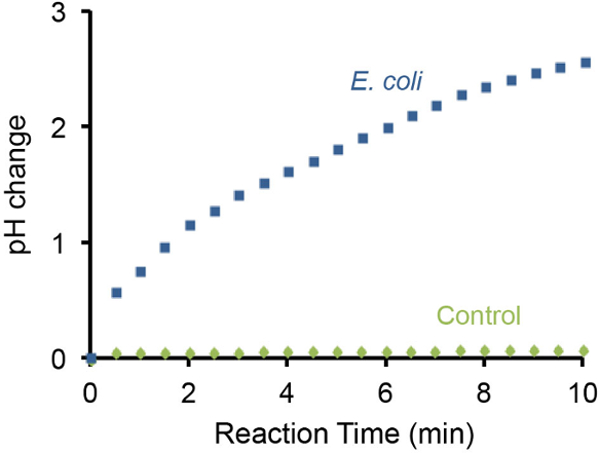
Abbildung 3: Überwachung pH - Erhöhung eines pH - Meters Die Änderung des pH - Wertes durch 10 7 E.. coli - Zellen wurde unter Verwendung eines tragbaren pH - Meter überwacht. Ein Test ohne E. coli wurde als Negativkontrolle verwendet. Die Gegenwart von 10 & sup7 ; E. coli - Zellen in der Testlösung die Basizität von ~ 3 pH - Einheiten in 10 min zu erhöhen. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
| Name | Sequenz (5'-3 ') | Hinweis |
| BS1 | BTTTT TTTTT TTTAC TCTTC CTAGC FRQGG TTCGA TCAAG A | B: 5'-Biotin; R: Adenin Ribonukleotid; F: Fluorescein-dT; F: Dabcyl-dT |
| DE1 | GATGT GCGTT GTCGA GACCT GCGAC CGGAA CACTA CACTG TGTGG GGATG GATTT CTTTA CAGTT GTGTG TTGAA CGCTG TGTCA AAAAA AAAA | |
| T1 | GACAA CGCAC ATCTC TTGAT CGAAC C | |
| LD1 | XTTTT TTTTT TTTTT TTGAC ACAGC GTTCA A | X: 5'-NH 2 |
Tabelle 1: Sequenzen von synthetischen Oligonukleotiden.
Diskussion
Die Übersetzung von der Wirkung der RNA - Spaltungsaktivität eines Bakteriums-responsive DNAzyme zu einer Lackmus - Test wird möglich gemacht durch die Verwendung von Urease und magnetische Trennung, wie 1 durch veranschaulicht ist . Obwohl der Nachweis des modifizierten Lackmus - Test für Bakteriendetektion mit einem E. getan coli -abhängigen RNA-Spaltung DNAzyme, 5,19,20 das Design im Allgemeinen für jede RNA-Spaltung DNAzyme erweitert werden kann. Angesichts der großen Verfügbarkeit von RNA-Spaltung DNAzyme für verschiedene Analyten und verschiedene Methoden zur Isolierung neuer RNA-Spaltung DNAzyme von zufälliger Sequenz Pools für neue Ziele, erwarten wir, dass die modifizierte Lackmus-Test-Plattform zur Erkennung von unterschiedlichen Ziele von Interesse erweitert werden kann .
Der Lackmus - Test für E. coli Nachweis kann 5000 und 500 Zellen erkennen , wenn die Berichtsreaktionszeit eingestellt ist 1 und 2 h eingestellt sind. Die beliebte Polymerase-Kettenreaktion (PCR) undSandwich - Enzym-linked immunosorbent assay (ELISA) Verfahren können Nachweisgrenzen von 10 etwa 4 -10 5 E. erreichen coli - Zellen in ähnlichen Testzeiten. 25,26 So ist die bakterielle Lackmus - Test bietet vergleichbare Nachweisempfindlichkeit.
Obwohl die bakterielle Lackmus-Test ist einfach durchzuführen und lebendige Farbveränderungen erzeugen können, können mehrere Faktoren erheblich Testergebnisse beeinflussen. Erstens ist die Qualität der Urease sehr wichtig. Wir haben Urease aus verschiedenen Quellen verwendet, und fand die Testergebnisse erheblich variieren kann. Wir empfehlen die Verwendung von Urease aus der Quelle in dem Abschnitt Materialien angegeben.
Die Montage von DNAzym / Urease / magnetische Kügelchen erfordert besondere Aufmerksamkeit. Gründliches Waschen von magnetischen Kügelchen nicht hybridisierten UrDNA zu entfernen ist notwendig, falsch-positive Ergebnisse zu verhindern. Sorgfalt muss auch die Ansammlung von Restmagnetkügelchen auf der Innenfläche des l zu vermeiden werdenid des Mikrozentrifugenröhrchen, das nur schwer zu sehen. Dort angekommen, werden die magnetischen Kügelchen nicht mehr einer magnetischen Trennung unterworfen und so konnten einige nicht hybridisierten UrDNA tragen, die zu falsch-positiven Signalen in der Reporter Reaktion führen kann. Es ist auch das Mikrozentrifugenröhrchen für länger als 10 min bei der magnetischen Trennschritt auf das Magnetzahnstange zu vermeiden, wichtig, zu verlassen. Die Perlen auf der Mikrozentrifugenröhrchen aggregieren oder hängen bleiben, was die Wascheffizienz reduzieren kann und von Charge zu Charge Inkonsistenz einzuführen. Die Aufnahme von 0,01% Tween-20 in der Waschlösung kann von Charge zu Charge Konsistenz verbessern und umgesetzt werden sollten.
Die magnetischen Perlen werden mit Streptavidin beschichtet, die als Anker verwendet wurde DNAzyme-Urease-Konjugate an die magnetischen Kügelchen zu montieren. Sowohl Streptavidin und Urease sind Proteinmoleküle, die während der Lagerung denaturiert werden kann. Wir speichern typischerweise die versammelten DNAzym-Urease-magnetische Kügelchen bei 4 ° C für bis zu 4 Wochenund machen frische Chargen regelmäßig konsistentere Ergebnisse zu erzielen.
Pflege muss auch in der magnetischen Trennschritt magnetischen Kügelchen zu vermeiden getroffen werden (Schritt 6.9) folgende DNAzyms Aktivierung versehentlichen Einnahme. Aus unserer Erfahrung, Zelltrümmer und andere Partikel in der Lösung kann die magnetische Trenneffizienz, zu reduzieren und daher einige magnetische Kügelchen werden können unbeabsichtigt beim Pipettieren entnommen. Dies wird in falsch-positiven Ergebnissen führen. Wir empfehlen folgende Maßnahmen zur Linderung des Problems: eine längere Trennungszeit (wie etwa 5-10 min), eine langsamere Freisetzung von Druck auf die Pipette sanft Rückzug des Überstandes zu ermöglichen, und Aussetzen der Überstand auf eine zusätzliche Runde der magnetischen Trennung .
Schließlich ist es wichtig, Unfall Kontamination der Reporter-Vorratslösung im Verlauf eines Experiments durch Urease zu vermeiden, wenn mehrere Proben getestet. Die hohe Reaktivität von u gegebenenrease kann zu falsch-positiven Ergebnissen Kontamination dieser Art führen.
Offenlegungen
The authors have nothing to disclose.
Danksagungen
The funding for this research project was provided by the Natural Sciences and Engineering Research Council of Canada (NSERC) via a Discovery Grant to YL.
Materialien
| Name | Company | Catalog Number | Comments |
| Ethylenediaminetetraacetic acid (EDTA) | VWR AMRESCO | 0105 | |
| Sodium Hydroxide (NaOH) pellets | BIO BASIC CANADA INC. | SB6789 | |
| Tris-base | VWR AMRESCO | 0497 | |
| Boric acid | AMRESCO | 0588 | |
| Urea | VWR AMRESCO | M123 | |
| 40% acrylamide/bisacrylamide (29:1) solution | BIO BASIC CANADA INC. | A0007 | |
| Sucrose | Bioshop Canada inc. | SUC507 | |
| Bromophenol blue | Bioshop Canada inc. | BRO777 | |
| Xylenecyanol FF | SIGMA-ALDRICH | X-4126 | |
| 10% sodium dodecyl sulfate | Bioshop Canada inc. | SDS001 | |
| Hydrochloric Acid (HCl) | CALEDON LABORATORIES LTD | 6026 | |
| Sodium Chloride (NaCl) | Bioshop Canada inc. | SOD001 | |
| 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid (HEPES) | Bioshop Canada inc. | HEP001 | |
| Magnesium Chloride (II) hexahydrate | VWR AMRESCO | 0288 | |
| Tween 20 | Bioshop Canada inc. | TW508 | |
| Adenosine Triphospahte (ATP) | AMRESCO | 0220 | |
| Sodium Acetate trihydrate (NaOAc) | SIGMA-ALDRICH | S8625 | |
| Ethanol | Commercial Alcohols | P016EAAN | |
| Tetramethyleneethylenediamine (TEMED) | AMRESCO | 0761 | |
| 10% Ammonium persulfate (APS) | BIO BASIC CANADA INC. | AB0072 | |
| Succinimidyl 4-(N-maleimidomethyl) cyclohexane-1-carboxylate (SMCC) | ThermoFisher SCIENTIFIC | 22360 | |
| Dimethyl sulfoxide (DMSO) | CALEDON LABORATORIES | 803540 | |
| Urease | SIGMA-ALDRICH | U0251 | |
| 1x Phosphate Buffered Saline (PBS) | ThermoFisher SCIENTIFIC | 70011-069 | |
| 0.04% Phenol red | SIGMA-ALDRICH | P3532 | |
| 10x T4 polynucleotide kinase reaction buffer | Lucigen | 30061-1 | |
| 10x T4 DNA ligase reaction buffer | Bio Basics Canada | B1122-B | |
| T4 DNA ligase (5 U/μl) | Thermo Fischer Scientific | B1122 | |
| Luria Bertani (LB) Broth | AMRESCO | J106 | |
| Agar | AMRESCO | J637 | |
| T4 polynucleotide kinase (10 U/μl) | Lucigen | 30061-1 | |
| E. coli K12 (MG1655) | ATCC | ATCC700926 | |
| Centrifuge | Beckman Coulter, Inc. | 392187 | |
| Glass plates | CBS scientific | ngp-250nr | |
| 0.75 mm thick spacers | CBS scientific | VGS-0725r | |
| 12-well comb | CBS scientific | VGC-7512 | |
| UV Lamp | UVP | 95-0017-09 | |
| Spectrophotometer (NanoVue) | GE Healthcare | N/A | |
| Metal plate | CBS scientific | CPA165-250 | |
| Vortex | VWR International | 58816-123 | |
| Gel electrophoresis apparatus | CBS scientific | ASG-250 | |
| Petri dishes | VWR International | 25384-342 | |
| 100 kDa MWCO centrifugal filters | EMD Millipore | UFC510024 | |
| Magnetic Bead (BioMag) | Bangs Laboratories Inc | BM568 | |
| Magnetic Seperation Rack | New England BioLabs | S1506S | |
| Microfuge tubes | Sarstedt | 72.69 | |
| Syringe filter (0.22 μm) | VWR International | 28145-501 | |
| 14 ml culture tube | VWR International | 60818-725 | |
| Cell culture incubator | Eppendorf Scientific | M13520000 | |
| Branson Ultrasonic cleaner | Branson | N/A | |
| Camera (Canon Powershot G11) | Canon | N/A | |
| 50 ml conical tube | VWR International | 89004-364 |
Referenzen
- Daar, A. S., et al. Top ten biotechnologies for improving health in developing countries. Nat. Genet. 32, 229-232 (2002).
- Newman, J. D., Turner, A. P. Home blood glucose biosensors: a commercial perspective. Biosens. Bioelectron. 20, 2435-2453 (2005).
- Turner, A. P. Biosensors: sense and sensibility. Chem. Soc. Rev. 42, 3184-3196 (2013).
- Tram, K., Kanda, P., Salena, B. J., Huan, S. Y., Li, Y. F. Translating Bacterial Detection by DNAzymes into a Litmus Test. Angew. Chem. Int. Ed. 53, 12799-12802 (2014).
- Ali, M. M., Aguirre, S. D., Lazim, H., Li, Y. Fluorogenic DNAzyme probes as bacterial indicators. Angew. Chem. Int. Ed. 50, 3751-3754 (2011).
- Schlosser, K., Li, Y. Biologically inspired synthetic enzymes made from DNA. Chem. Biol. 16, 311-322 (2009).
- Tuerk, C., Gold, L. Systematic evolution of ligand by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Breaker, R. R. Making catalytic DNAs. Science. 290, 2095-2096 (2000).
- Liu, J., Cao, Z., Lu, Y. Functional nucleic acid sensors. Chem. Rev. 109, 1948-1998 (2009).
- Navani, N. K., Li, Y. Nucleic acid aptamers and enzymes as sensors. Curr. Opin. Chem. Biol. 10, 272-281 (2006).
- Li, J., Lu, Y. A highly sensitive and selective catalytic DNA biosensor for lead ions. J. Am. Chem. Soc. 122, 10466-10467 (2000).
- Liu, J., Lu, Y. A colorimetric lead biosensor using DNAzyme-directed assembly of gold nanoparticles. J. Am. Chem. Soc. 125, 6642-6643 (2003).
- Liu, Z., Mei, S. H. J., Brennan, J. D., Li, Y. Assemblage of signaling DNA enzymes with intriguing metal specificity and pH dependence. J. Am. Chem. Soc. 125, 7539-7545 (2003).
- Liu, J., et al. A catalytic beacon sensor for uranium with parts-per-trillion sensitivity and millionfold selectivity. Proc. Natl. Acad. Sci. USA. 104, 2056-2061 (2007).
- Huang, P. J., Vazin, M., Liu, J. In vitro selection of a new lanthanide-dependent DNAzyme for ratiometric sensing lanthanides. Anal. Chem. 86, 9993-9999 (2014).
- Chiuman, W., Li, Y. Simple fluorescent sensors engineered with catalytic DNA 'MgZ' based on a non-classic allosteric design. PLoS One. 2, e1224(2007).
- Xiang, Y., Lu, Y. Using personal glucose meters and functional DNA sensors to quantify a variety of analytical targets. Nat. Chem. 3, 697-703 (2011).
- Aguirre, S. D., Ali, M. M., Salena, B. J., Li, Y. A sensitive DNA enzyme-based fluorescent assay for bacterial detection. Biomolecules. 3, 563-577 (2013).
- Aguirre, S. D., Ali, M. M., Kanda, P., Li, Y. F. Detection of Bacteria Using Fluorogenic DNAzymes. J. Vis. Exp. (63), e3961(2012).
- Shen, Z., et al. A catalytic DNA activated by a specific strain of bacterial pathogen. Angew. Chem. Int. Ed. 54, (2015).
- He, S., et al. Highly specific recognition of breast tumors by an RNA-cleaving fluorogenic DNAzyme probe. Anal. Chem. 87, 569-577 (2015).
- Sumner, J. B., Hand, D. B. Isoelectric point of crystalline urease. J. Am. Chem. Soc. 51, 1255-1260 (1929).
- Karplus, P. A., Pearson, M., Hausinger, R. P. 70 Years of crystalline urease: What have we learned. Acc. Chem. Res. 30, 330-337 (1997).
- Omiccioli, E., Amagliani, G., Brandi, G., Magnani, M. A new platform for Real-Time PCR detection of Salmonella spp., Listeria monocytogenes and Escherichia coli O157 in milk. Food Microbiol. 26, 615-622 (2009).
- Cui, S., Schroeder, C. M., Zhang, D. Y., Meng, J. Rapid sample preparation method for PCR-based detection of Escherichia coli O157:H7 in ground beef. J. Appl. Microbiol. 95, 129-134 (2003).
- Ibekwe, A. M., Watt, P. M., Grieve, C. M., Sharma, V. K., Lyons, S. R. Multiplex fluorogenic real-time PCR for detection and quantification of Escherichia coli O157:H7 in dairy wastewater wetlands. Appl. Environ. Microbiol. 68, 4853-4862 (2002).
- Strachan, N. J., Ogden, I. D. A sensitive microsphere coagulation ELISA for Escherichia coli O157:H7 using Russell's viper venom. FEMS Microbiol Lett. 186, 79-84 (2000).
- de Boer, E., Beumer, R. R. Methodology for detection and typing of foodborne microorganisms. Int. J. Food Microbiol. 50, 119-130 (1999).
- Gracias, K. S., McKillip, J. L. A review of conventional detection and enumeration methods for pathogenic bacteria in food. Can. J. Microbiol. 50, 883-890 (2004).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten