Method Article
Detecção Colorimétrico de bactérias usando teste decisivo
Neste Artigo
Resumo
We describe a protocol for colorimetric detection of E. coli using a modified litmus test that takes advantage of an RNA-cleaving DNAzyme, urease, and magnetic beads.
Resumo
There are increasing demands for simple but still effective methods that can be used to detect specific pathogens for point-of-care or field applications. Such methods need to be user-friendly and produce reliable results that can be easily interpreted by both specialists and non-professionals. The litmus test for pH is simple, quick, and effective as it reports the pH of a test sample via a simple color change. We have developed an approach to take advantage of the litmus test for bacterial detection. The method exploits a bacterium-specific RNA-cleaving DNAzyme to achieve two functions: recognizing a bacterium of interest and providing a mechanism to control the activity of urease. Through the use of magnetic beads immobilized with a DNAzyme-urease conjugate, the presence of bacteria in a test sample is relayed to the release of urease from beads to solution. The released urease is transferred to a test solution to hydrolyze urea into ammonia, resulting in an increase of pH that can be visualized using the classic litmus test.
Introdução
Bacterial pathogens are one of the major causes of global morbidity and mortality. Outbreaks from hospital-acquired infections, food-borne pathogens, and bacterial contaminants in the environment pose serious and on-going threats to public health and safety. To prevent these outbreaks, effective tools are needed that permit pathogen detection in a timely fashion under a variety of settings. Simple but still effective tests that are portable and cost-effective are greatly coveted, especially in regions that are susceptible to outbreaks but cannot afford expensive testing facilities.1-3 Although there exists a multitude of methods to detect bacteria, many of them are not suitable as screening or on-site testing tools because they require long test times, expensive instruments and complicated testing procedures.
Colorimetric tests are particularly attractive for point-of-care or field applications as color changes can be easily detected by the naked eye. The litmus test for pH is simple, quick, and effective. Although it is a very old technology, it is still widely used today because of its simplicity and effectiveness. Surprisingly, this simple test had never been modified to achieve the detection of other analytes before we recently developed an approach of modifying this test for E. coli testing.4
The expanded litmus test for E. coli employs three additional components: an E. coli activated RNA-cleaving DNAzyme (EC1), 5 urease, and magnetic beads. DNAzymes refer to synthetic single-stranded DNA molecules with catalytic activity.6 They can be isolated from random-sequence DNA pools using in vitro selection.7,8 They are highly stable and can be produced cost-effectively using high-efficiency automated DNA synthesis.9 For these reasons, DNAzymes, particularly RNA-cleaving DNAzymes, have been widely examined for biosensing applications.6,10,11 RNA-cleaving DNAzyme sensors have been developed to detect metal ions,12-16 small molecules,17,18 bacterial pathogens5,19-21 and cancer cells.22 Given the great availability of target-induced RNA-cleaving DNAzymes, any assay that utilizes a DNAzyme can be potentially expanded to detect a diverse range of analytes.
Urease is chosen for its ability to hydrolyze urea into ammonia,23,24 resulting in a pH increase. Urease is also highly efficient, stable and amenable for conjugation to other biomolecules. Therefore, we postulated that a conjugate of an RNA-cleavage DNAzyme with urease would allow the use of litmus test for the detection of other targets.5
The action of the RNA-cleaving DNAzyme is relayed to urease-mediated increase of pH through the use of magnetic beads that are immobilized with the DNAzyme-urease conjugate. Because the activity of the DNAzyme under investigation is strictly dependent on E. coli, the presence of this bacterium in the test solution will result in the release of urease from the magnetic beads to the solution, which is then taken and used to hydrolyze urea in a reporter solution that contains a pH-sensitive dye. The final outcome of this procedure is a color change that can be conveniently reported by the dye or pH paper.
Protocolo
1. Preparação de Reagentes e Buffers
- Ácido m-0,5 etilenodiaminotetracético (EDTA)
- Numa proveta de 2 litros, adicionar 186,1 g de EDTA em 800 ml de água desionizada-destilada (ddH2O). Ajustar o pH da solução a 8,0 usando peletes de NaOH. Adicionar ddH2O para um volume final de 1,0 L e transferir a solução para um frasco de vidro autoclavável para esterilização em autoclave e armazenar a 4 ° C.
- EDTA 10 × tampão Tris-borato (10x TBE)
- Em um copo de plástico de 4 L, adicionar 432 g de base Tris, 200 g de ácido bórico, 80 ml de 0,5 M de EDTA (pH 8,0) e ddH2O para um volume final de 4 L. mistura utilizando uma barra de agitação até que todos os componentes são dissolvidos. Transferir a solução para garrafas de vidro de 1 L de autoclavagem e armazenar a 4 ° C.
- 10% de poliacrilamida desnaturante da
- Em um copo de plástico de 4 L, adicione 1681,7 g de ureia, 400 ml de 10x TBE, 1 L de 40% de acrilamida / bisacrilamida (29: 1) e solução de ddH2O até ao fvolume de inal é de 4 L. Mix usando uma barra de agitação até que a ureia é dissolvido. Transferir a solução para 1 L frascos de vidro âmbar e armazenar a 4 ° C.
- 2x Gel carregando Buffer (2x GLB)
- Em um copo de vidro de 200 ml, adicionar 44 g de ureia, 8 g de sacarose, 10 mg de azul de bromofenol, 10 mg de cianol de xileno FF, 400 ul de sulfato de dodecilo de sódio a 10%, e 4 ml de 10 x TBE. Adicionar ddH2O para um volume final de 40 ml e dissolve-se os componentes com aquecimento suave a 50 ° C, enquanto se mistura com uma barra de agitação magnética. Transferência alíquota de 1 ml para tubos de microcentrífuga de 1,5 ml e armazenar a 4 ° C.
NOTA: O uso anterior requer breve aquecimento a 90 ° C para redissolver qualquer sólido.
- Em um copo de vidro de 200 ml, adicionar 44 g de ureia, 8 g de sacarose, 10 mg de azul de bromofenol, 10 mg de cianol de xileno FF, 400 ul de sulfato de dodecilo de sódio a 10%, e 4 ml de 10 x TBE. Adicionar ddH2O para um volume final de 40 ml e dissolve-se os componentes com aquecimento suave a 50 ° C, enquanto se mistura com uma barra de agitação magnética. Transferência alíquota de 1 ml para tubos de microcentrífuga de 1,5 ml e armazenar a 4 ° C.
- 1 M de Tris-cloridrato (Tris-HCl) (pH 7,5)
- Em um frasco de vidro, adicionar 12,1 g de Tris-base e 70 ml de ddH2O e misturar até o sólido estar dissolvido. Ajustar o pH para 7,5 utilizando ácido clorídrico 1 M (HCl). Adicionar ddH2O para um volume final de 100 ml, autoclave e STOre a 4 ° C
- 5 M de cloreto de sódio (NaCl)
- Em um frasco de vidro, dissolver 58,4 g de NaCl em 150 ml de ddH 2 O. Ajustar o volume para 200 ml com DDH 2 O. Armazenar a 4 ° C.
- Tampão de Eluição de DNA
- Em um frasco de vidro, misturar 2,0 ml de Tris-HCl 1 M (pH 7,5), 8,0 ml de NaCl 5 M e 0,4 ml de EDTA 0,5 M (pH 8,0). Ajustar o volume a 200 ml com ddH2O, autoclave e armazenar a 4 ° C.
- 1 H 4- (2-hidroxietil) -1-piperazinoetanossulfónico (HEPES) (pH 7,4)
- Em um frasco de vidro, dissolver 2,38 g de HEPES em 80 ml de ddH 2 O. Ajustar o pH para 7,4 utilizando NaOH 5 N e adicionar ddH2O para um volume final de 100 ml.
- 1 M de cloreto de magnésio (MgCl2)
- Em um frasco de vidro, adicionar 2,03 g de MgCl 2 -6H 2 O e levar o volume até 100 ml com ddH 2 O.
- Tampão de Reacção (RB)
- Num tubo de 50 ml, adicionar 50 ul de HEPES 1 M (pH 7,4), 1,5 ml de NaCl 5 M, 0,75 mL de 1 M de MgCl2, e 5 ul de Tween-20. Adicionar ddH2O para um volume final de 50 ml. Misturar a solução e filtra-se para outro tubo cónico utilizando um filtro de seringa-driven (0,22 um) e armazenar a 4 ° C até à sua utilização.
- Tampão de Ligação (BB)
- Num tubo de 50 ml, adicionar 500 ul de Tris-HCl 1 M (pH 7,5), 8,8 g de NaCl, 50 ul de 1 M de MgCl2, e 5 ul de Tween 20. Adicionar ddH2O para um volume final de 50 ml. Misturar a solução e filtra-se para outro tubo cónico utilizando um filtro de seringa-driven (0,22 um) e armazenar a 4 ° C até à sua utilização.
- Solução substrato (SS)
- Num tubo de 50 ml, adicionar 5,8 g de NaCl, 3 ml de 1 M de MgCl2, 1,5 g de ureia, e 40 ml de ddH 2 O. Ajuste o pH da solução para 5,0 utilizando HCl a 10 mM. Uma vez que a solução não é tamponada, ajustar o pH usando HCIcuidadosamente através da adição de pequenas alíquotas de HCl. Adicionar ddH2O para um volume final de 50 ml.
- Misturar a solução e filtra-se para outro tubo cónico utilizando um filtro de seringa-driven (0,22 um) e armazenar a 4 ° C até à sua utilização.
- Luria Bertani (LB) Caldo
- Numa proveta, dissolver 20,0 g LB pó em 1 L de ddH 2 O. Transfira para uma garrafa de vidro, autoclave, e armazenar a 4 ° C.
- 1,5% de agar LB
- Em um balão de 250 ml, adicionar 1,5 g de agar e 100 ml de caldo LB. Autoclave e armazenar a 4 ° C.
- As placas de agar
- Redissolve-se o agar LB num forno de microondas e arrefecer a solução até ~ 50 ° C. Verter a solução em placas de Petri, tornando ~ 5 placas e permitir-lhes a solidificar.
2. Síntese e purificação de E. coli- responsiva ADNzima EC1
- Síntese de EC1 by Template Mediated enzimática Ligadura
- purificar commercially oligonucleótidos sintetizados BS1, DE1, e T1 (sequências proporcionadas na Tabela 1) em 10% de electroforese em gel de poliacrilamida desnaturante (dPAGE) de acordo com protocolos padrão.
- Prepare um estoque de 100 M de BS1, DE1 e T1. Armazenar a -20 ° C até à sua utilização.
- Num tubo de microcentrífuga de 1,5 ml, adicionar 38,5 mL de ddH2O, 5 ul de DE1, e 5 ul de 10 x T4 tampão de reacção de cinase de polinucleótidos fornecidos pelo fornecedor da enzima (Tris-HCl 500 (pH 7,6), MgCl2 100, ditiotreitol 50 mM (DTT), 1,0 mM de espermidina).
- Adicionar 1 ml de adenosina trifosfato (ATP) (100 mM). Adicionam-se 5 unidades de cinase de polinucleótido de T4 (10 U / ul). Misturar por pipetagem cuidadosamente a mistura de reacção.
- Incubar a reacção a 37 ° C durante 30 min.
- Extingue-se a reacção por aquecimento a 90 ° C durante 5 min.
- Adicionar 118 uL de ddH2O, 5 ul de BS1, e 5 ul de T1. Aquece-se a reacção a 90 ° C durante 2 min e, em seguida, arrefecer até à temperatura ambiente ao longo de 10 min.
- Adicionar 20 ul de 10 x T4 ADN ligase de tampão de reacção fornecido pelo fornecedor da enzima (Tris-HCl 400 (pH 7,8), MgCl2 100 mM, DTT a 100 mM, ATP 5 mM). Adicionam-se 10 unidades de ligase do DNA de T4 (5 U / uL) e mistura-se cuidadosamente por pipetagem.
- Incubar à temperatura ambiente durante 2 h.
- Adicionar 20 ul de 3 M de acetato de sódio (NaOAc) (pH 5,2), 500 ul de etanol frio a 100%. Misture a solução por agitação em vórtice e colocar o tubo num congelador a -20 ° C durante 30 min.
- Centrifuga-se o microcentrifuga a 20000 xg durante 20 min a 4 ° C. Remova cuidadosamente o sobrenadante por pipetagem.
- Lava-se a pastilha com etanol frio a 70% e centrifuga-se novamente a 20000 xg durante 10 min a 4 ° C. Mais uma vez, remove-se o sobrenadante e secar o sedimento sob vácuo durante 10 min.
- Ressuspender o ADN com 15 uL de ddH2O e, em seguida, adicionar 15 ul de 2x GLB.
- Vortex vigorosamente e aquecer a 90 ° C durante2 min. Purifica-se o ADN de comprimento completo por 10% dPAGE como descrito abaixo.
- Configurando 10% dPAGE
- placas limpas dois de vidro (um prato completo e um entalhadas), duas de 0,75 mm de espessura espaçadores, e um pente de 12 poços. Lay uma placa de vidro em uma superfície plana, com dois espaçadores de cada lado e a placa entalhada no topo. Corte as duas placas em conjunto, utilizando os quatro grampos fornecidos pelo fornecedor.
- Em um copo de plástico de 150 ml, verter 40 mL de 10% dPAGE, 40 ul de tetrametiletilenodiamina (TEMED) e 400 ul de persulfato de amónio a 10% (APS). Misturar os componentes e despeje a solução entre as placas lentamente.
- Insira o pente e, em seguida, permitir que o gel para polimerizar durante 10 minutos. Uma vez que o gel é polimerizado, retire lentamente o pente e lavar os poços com DDH 2 O.
- Montar as placas num aparelho de electroforese em gel. Use uma placa de metal para dissipar o calor gerado para evitar o superaquecimento.
- Despeje TBE 1x para as câmaras superiores e inferiorese assegurar que as cavidades são bem submerso no tampão. Lavar os poços com TBE 1x, utilizando uma pipeta ou uma seringa.
- Definir o aparelho para executar a 35 mA e pré-run durante 15 minutos antes de colocar as amostras.
- Eluição de ligado EC1 de 10% dPAGE
- Seguindo o passo 2.2.6, correr o gel a 35 mA até o corante de fundo (azul de bromofenol) atinge o fundo da placa de vidro. Isso deve demorar cerca de 1,5 horas. Desligue a alimentação e retire as placas do aparelho.
- Coloque as placas em algumas folhas de toalhas de papel e retire cuidadosamente os espaçadores das placas de vidro. Remova cuidadosamente a placa de vidro superior e enrole o gel com filme plástico.
- Virar o gel sobre para remover a segunda placa de vidro e cubra com filme plástico novamente. Tome cuidado para evitar rugas do filme plástico.
- Visualizar o produto ligado utilizando sombreamento de UV (260 nm), o que irá produzir 4 bandas de DNA distintas (EC1 totalmente ligado, DE1, BS1, e T1).
- Excise a banda superior (EC1) com uma lâmina de barbear estéril e transferir para um tubo de 1,5 ml de microcentrífuga. Esmagar o gel com uma ponta de pipeta estéril (200 tamanho da ponta ul) até que ele se transforma em uma pasta fina. Adicionar 550 ul de tampão de eluição de ADN, cobrir o tubo com folha de alumínio para proteger o fluoróforo incorporado, e agitar durante 15 min.
- Centrifugar a solução de gel a 20000 xg durante 5 min e transferir cuidadosamente 400 ul do sobrenadante para outro tubo de microcentrífuga de 1,5 ml. Evitar retirar pedaços de gel durante a pipetagem.
- Para o tubo de microcentrífuga com o sobrenadante transferido adicionar 40 uL de NaOAc 3 M (pH 5,2), e 1,0 ml de etanol frio a 100%. Misture a solução por agitação em vórtice e colocar o tubo num congelador a -20 ° C durante 30 min.
- Centrifuga-se o microcentrifuga a 20000 xg durante 20 min a 4 ° C. Remova cuidadosamente o sobrenadante por pipetagem.
- Lava-se a pastilha com etanol frio a 70% e centrifuga-se novamente a 20000 xg durante 10 min a 4 ° C. Mais uma vez, remover osobrenadante e secar o sedimento sob vácuo durante 10 min.
- Determinar a concentração de EC1, medindo a absorvância de UV a 260 nm. Adicione um estoque de 10? M e armazenar a amostra a -20 ° C até à sua utilização.
3. Conjugação de urease para ADN
- Preparação de 4- (N-maleimidometil) ciclo-hexano-1-carboxilato (SMCC) da
- Dissolve-se 1 mg de SMCC em 676 ul de DMSO. Vortex e colocar no gelo até o uso.
- Preparação de urease da
- Dissolve-se 1 mg de urease em 1 ml de 1 x PBS (sem Mg2 + ou Ca2 +). Colocar em gelo até à sua utilização.
NOTA: cristalizado urease é lento para dissolver e mistura suave é necessária para evitar a desnaturação.
- Dissolve-se 1 mg de urease em 1 ml de 1 x PBS (sem Mg2 + ou Ca2 +). Colocar em gelo até à sua utilização.
- Síntese de ADN em urease (UrDNA)
- Adicionar 10 uL de 100 uM LD1 para um tubo de microcentrífuga de 2,5 ml. Adicionar 140 uL de ddH2O, 40 ul de PBS 10x, e vortex.
- Adicionar 80,5 ul de SMCC stac, 159,5 ul de DMSO, vórtice, e centrifuga-se brevemente usando uma centrífuga de bancada.
- Incubar a 37 ° C durante 60 min. Evitar a condensação sob a tampa de microcentrífuga.
- Adicionar 200 ul de 1x PBS, 60 ul de NaOAc 3 M (pH 5,2), e 1,5 ml de etanol frio a 100%. Misture a solução por vórtice e incubar à temperatura de -20 ° C durante 30 min.
- Centrifugar a solução a 20000 xg durante 20 min a 4 ° C. Remover o sobrenadante e o sedimento secar sob vácuo. Evitar o excesso de secagem.
- Para o sedimento seco adicionar 400 ul de estoque de urease e incubar à temperatura ambiente durante 5 h.
- Transferir 200 ul de conjugado em bruto a uma coluna de centrífuga de filtro pré-lavada 100K MWCO. Centrifugar a coluna a 14000 xg durante 5 min.
- Transferir os restantes 200 ul de conjugado em bruto para a coluna e centrifugar a 14000 xg durante 5 min. Remova a coluna e colocá-lo de cabeça para baixo em um novo tubo de 2.0 ml de microcentrífuga ( "tubo de coleta").
- CentrifUGE o tubo de recolha (com a coluna invertida) a 1000 xg durante 2 min. Remover o tubo de recolha e adicionar 30 ul de 1x PBS para a coluna centrífuga de lavar a membrana para recuperação adicional de conjugados.
- Mais uma vez inverter a coluna e colocar de volta para o tubo de recolha. Centrifuga-se o tubo de recolha (com a coluna invertida) a 1000 xg durante 2 min. Retirar e descartar a coluna.
- Armazenar o UrDNA a 4 ° C até à sua utilização.
4. Montagem de EC1 e UrDNA sobre esferas magnéticas
- Misturar o estoque esférulas magnéticas (MB) bem e transferir 100 ul de suspensão MB para um tubo de microcentrífuga de 1,5 ml. Colocar o tubo de microcentrífuga num suporte magnético cremalheira para isolar o MB.
- Remover o sobrenadante por pipetagem e adicionar 150 ul de tampão (BB) de ligação ao tubo. Remover o tubo do suporte e toque cuidadosamente o tubo para ressuspender o MB a uma solução homogénea. Evite espirrar a suspensão à top do tubo ou tampa. Se isso acontecer, use uma centrífuga de bancada para girar rapidamente o resíduo de volta para a suspensão.
- Repita o passo 4.2 mais duas vezes.
- A esta suspensão adiciona 10 ul de EC1 10? M. Misturar cuidadosamente tocando no tubo.
- Incubar a solução com agitação suave durante 30 min. Toque no tubo a cada 2-3 minutos, para evitar a precipitação do MB.
- Colocar o tubo de microcentrífuga para trás sobre a grelha magnética para isolar o MB e remover o sobrenadante por pipetagem. Lava-se a MB três vezes com 150 ul de BB (tal como descrito no passo 4.2).
- Uma vez que a lavagem estar completa, a suspensão MB em um total de 150 ul de BB. A esta solução, adicionar 15 ul de UrDNA e aquecer a solução a 45 ° C durante 2 min. Arrefece-se a solução até à temperatura ambiente e incubar durante 2 horas.
- Colocar o tubo de microcentrífuga para trás sobre a grelha magnética para isolar o MB e remover o sobrenadante por pipetagem.
- Adicionar 100 ul de tampão de reacção (RB). Retirar tele microcentrífuga tubo do rack magnético e ressuspender cuidadosamente o MB.
- Lave os MB mais três vezes repetindo o passo 4.9.
NOTA: O sobrenadante lavada pode ser rapidamente testado para determinar se não hibridada UrDNA ainda está presente, que pode resultar em um sinal falso positivo. O teste pode ser feito por adição de 10 ul de ureia 50 mM e 10 uL de 0,04% de vermelho de fenol na solução de lavagem. Continuar a lavagem do MB até que a solução de lavagem não causar uma mudança de cor. A suspensão de 100 ul é armazenado a 4 ° C até à sua utilização.
5. Preparação de células bacterianas 20
- A cultura E. coli de Stocks
- Placa E. coli K12 (MG1655) em LB placas de agar de um estoque de glicerol sob uma chama ou em uma cabine de segurança biológica.
- Usando uma ponta de pipeta estéril, gentilmente tocar o estoque de glicerol e levemente raia a superfície de uma placa de agar para evitar a perfuração do ágar LB.
- Inverta os listradosplaca e incubar a 37 ° C durante 14 h.
- Selar a placa com parafilme e armazenar a 4 ° C durante um máximo de 3 semanas.
- A cultura E. coli para contagem celular
- Num tubo de cultura de 14 mL estéril, preparar 2 ml de caldo LB.
- Utilizando uma ponta de pipeta estéril, escolher uma única colónia da placa de agar estrias preparado no passo 5.1 e transferi-lo para o tubo de cultura.
- Incubar a cultura a 37 ° C e agitar a 230 rpm durante 14 horas.
- Serialmente dilua a cultura bacteriana, em intervalos de 10 vezes.
- Para cada amostra diluída, placa uniformemente cinco alíquotas de 100 ul em placas de agar LB separadas. Inverter as placas e incuba-se a 37 ° C durante 14 h.
- Contar as células de cada amostra para obter a concentração de células médio de cada diluição.
NOTA: 10 7 células são muitas vezes utilizados para configurar um teste de referência para E. coli como este nível de E. coli pode desencadear uma cor rápida change. No entanto, um teste executado adequadamente pode detectar tão baixo quanto 500 células, como discutido na secção Resultados.
- Preparando E. As células de E. coli for Testing
- Para obter uma suspensão de células desejada, transferir 1 ml da cultura estoque para um tubo de microcentrífuga de 1,5 ml.
- Centrifugar as células a 6000 xg durante 10 min a 4 ° C. Cuidadosamente remover o sobrenadante sem perturbar o sedimento celular.
- Adicionar 10 ul de tampão de reacção para a pelete de células e ressuspender as células. Sonicar a suspensão de células durante 5 min. Transferir a suspensão de células para uma caixa de gelo durante 5 min.
- Sonicar a suspensão de células durante mais 5 min.
- Centrifugar a suspensão de células a 13.000 xg durante 10 min a 4 ° C. Utilizar o sobrenadante para o ensaio (10 ul).
6. Teste Litmus
- Num tubo de microcentrífuga de 1,5 ml, pré-lavagem do tubo por adição de vortex e 100 ul de tampão de reacção (RB) no tubo de microcentrifuga edescartando o tampão.
- Transferência de 15 ul de EC1 montados (protocolo 4) para o tubo de microcentrífuga lavada.
- Lavam-se as esferas magnéticas, colocando o tubo de microcentrífuga em uma cremalheira magnética. Remover o sobrenadante por pipetagem. Retire o tubo de microcentrífuga do rack, adicione 100 ml de RB, e ressuspender cuidadosamente as esferas magnéticas.
- Lave os MB mais duas vezes repetindo o passo 6.3.
- Colocar o tubo de microcentrífuga para trás sobre a grelha magnética, remover o sobrenadante e adicionar 10 uL da E. coli amostra preparada a partir do passo 5.3.
- Misturar a amostra e magnéticos pérolas cuidadosamente batendo suavemente no tubo de microcentrífuga.
- Incubar a reacção a temperatura ambiente durante 1 h.
- Para a reacção, adicionar 90 uL de ddH2O e coloque o tubo de microcentrífuga para um rack magnético.
- Após cerca de 3 minutos de separação magnética, transferir cuidadosamente a 85 ul de sobrenadante para um tubo de microcentrífuga de 0,5 ml. Retirar o sobrenadante lentamente aevitar a coleta de quaisquer esferas magnéticas.
- Para o tubo de microcentrífuga de cima adicionar 15 ul de 0,04% de vermelho de fenol e 100 ul de solução de substrato.
- Tirar uma fotografia em intervalos de tempo específicos para registrar a mudança de cor.
NOTA: A mudança no pH também pode ser monitorado usando um medidor de pH com um microeletrodo. O pH de partida deverá ser de aproximadamente 5,2-5,5 (a solução é amarelo). Se não, a solução pode ser ajustado pela adição de Tampão Acetato de 1 mM, pH 5,0).
Resultados
O princípio do teste decisivo bacteriana é explicado na Figura 1 O ensaio utiliza três materiais principais:. Uma ADNzima de clivagem de ARN que é activado por uma bactéria específica, urease e esferas magnéticas. A ADNzima é utilizado como o elemento de reconhecimento molecular para alcançar a detecção altamente específica de uma bactéria de interesse. A urease e esferas magnéticas são usados para alcançar transdução de sinal da actividade ARN-clivagem da ADNzima. Isto envolve a criação de esferas magnéticas que contêm conjugados de urease-ADNzima. Na presença da bactéria alvo, a ADNzima cliva a sua ligação de ARN. Esta acção provoca a dissociação da urease de esferas magnéticas. A urease pode ser libertado facilmente separados dos grânulos magnéticos e utilizado para gerar uma alteração da cor numa solução repórter, que contém ureia e um corante sensível ao pH. A urease hidrolisa a ureia em amónia, acompanhada pelo aumento de pH que desencadeia a Cmudança olor do corante.
A Figura 2 apresenta uma prova de fogo bacteriano onde EC1, uma E. coli -responsive-ARN clivagem ADNzima, foi usado como o ADNzima, e vermelho de fenol foi usado como o corante de relatório de pH. CE1 foi previamente isolada pelo nosso grupo a partir de um conjunto de ADN de sequência aleatória, utilizando a técnica de selecção in vitro. 5 Os nossos estudos anteriores mostraram que EC1 é altamente específico para a E. coli e exibe uma actividade mínima no sentido de outras bactérias. 5,19 Verificou-se que EC1 é activado por uma molécula de proteína a partir de E. coli. Embora a identidade deste biomarcador proteína não tenha sido decifrada, a especificidade de reconhecimento de alta sugere que esta proteína é única para E. coli. A solução repórter é configurado para ter um pH inicial de 5,5. A este pH, de vermelho de fenol apresenta uma cor amarela. Como urease hidrolisa a ureia em amoníaco, a basicidade do Sol repórterution aumenta. Isto reflecte-se pela mudança progressiva da cor de amarelo para cor de rosa. A profundidade da alteração da cor é dependente dos dois parâmetros que se seguem, tal como ilustrado pela Figura 2: o número de E. células de E. coli utilizadas na etapa de ativação ADNzima eo tempo permitido para a etapa de hidrólise de ureia. Mais E. células de E. coli resultaram em mudanças de cor mais forte, que se reflecte pela observação de uma E. amarelo-para-rosa quando a transição de cor progressiva células de E. coli foram aumentados em série 5-5 x 10 7 (10 vezes aumentar cada vez). Enquanto isso, um tempo mais longo para a hidrólise de ureia permitiu a detecção de números pequenos de E. células de E. coli (5000 células na reacção de 1 h e a 500 células na reacção 2 horas).
A mudança de a prova de fogo bacteriano de pH também pode ser monitorizado utilizando um medidor portátil de pH e os resultados representativos são ilustrados na Figura 3. Was descobriram que a presença de 10 7 E. coli resultou em aumento gradual de pH em 3 unidades dentro de 10 min. Em contraste, a ausência de E. coli não provocou alterações de pH detectáveis com a mesma configuração.
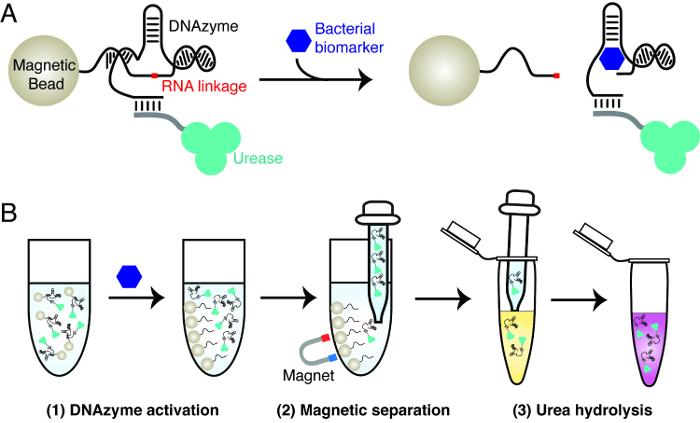
Figura 1: O princípio de construção de prova de fogo bacteriano (A) Activação de uma ADNzima de clivagem de ARN por um biomarcador específico a partir de uma bactéria de interesse.. Na presença do biomarcador, o ADNzima de clivagem de ARN imobilizado sobre esferas magnéticas cliva a ligação de RNA, resultando na libertação da urease com etiquetas de esferas magnéticas a solução. (B) Procedimento de ensaio de três passos. Passo 1: ADNzima activação, tal como descrito no painel A. Passo 2: Separação magnética - a urease libertado é separado do esferas magnéticas. Passo 3: Urea hidrólise R12; a urease libertado é adicionada a uma solução repórter contendo ureia. Urease hidrolisa uréia em amônia, resultando em uma mudança no pH que pode ser relatado por um corante sensível ao pH. Por favor clique aqui para ver uma versão maior desta figura.

Figura 2: teste de Litmus com E. coli utilizando E. coli -responsive ADNzima EC1. resultados de mudança de cor representativos com números variados de E. coli fornecidos acima de cada tubo de ensaio. Vermelho de fenol foi usado como o corante sensível ao pH. Um teste sem E. coli foi utilizado como controlo negativo. Mais E. células de E. coli são esperados para provocar a libertação de mais moléculas de urease, acompanhada by mudanças de cores mais fortes. Por favor clique aqui para ver uma versão maior desta figura.
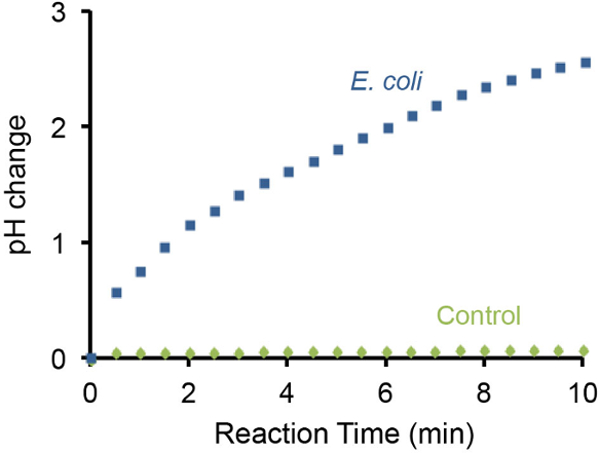
Figura 3: Monitorando o aumento do pH utilizando um medidor de pH A mudança de pH causada por 10 7 E.. células de E. coli foi monitorizado utilizando um medidor de pH portátil. Um teste sem E. coli foi utilizado como controlo negativo. A presença de 10 7 E. coli na solução de teste pode aumentar a basicidade por unidades ~ 3 pH em 10 min. Por favor clique aqui para ver uma versão maior desta figura.
| Nome | Sequência (5'-3 ') | Nota |
| BS1 | BTTTT TTTTT TTTAC TCTTC CTAGC FRQGG TTCGA TCAAG A | B: 5'-biotina; R: ribonucle�ido adenina; F: fluoresceína-dT; Q: dabcil-dT |
| DE1 | GATGT GCGTT GTCGA GACCT GCGAC CGGAA CACTA CACTG TGTGG GGATG GATTT CTTTA CAGTT GTGTG TTGAA CGCTG TGTCA AAAAA AAAA | |
| T1 | GACAA CGCAC ATCTC TTGAT CGAAC C | |
| LD1 | XTTTT TTTTT TTTTT TTGAC ACAGC GTTCA A | X: 5'-NH2 |
Tabela 1: Sequências de oligonucleótidos sintéticos.
Discussão
A tradução da acção da actividade de clivagem de ARN de uma ADNzima bactéria responde a um teste decisivo é tornada possível através da utilização de urease e separação magnética, como ilustrado pela Figura 1. Embora a manifestação do teste decisivo modificado para detecção de bactérias é feito com uma E. coli dependente de RNA-clivagem ADNzima, 5,19,20 o projeto pode ser estendido geralmente para qualquer ADNzima de clivagem de RNA. Dada a grande disponibilidade de DNAzymes de clivagem de ARN para diferentes analitos e várias metodologias para isolar novos DNAzymes de clivagem de ARN a partir de pools de seqüência aleatória de novas metas, esperamos que a plataforma de teste decisivo modificado pode ser estendido para a detecção de diversos alvos de interesse .
O teste decisivo para E. coli detecção pode detectar 5.000 e 500 células, quando o tempo de reacção de informação é definido como 1 e 2 horas, respectivamente. A reacção populares cadeia da polimerase (PCR) esandwich enzyme-linked immunosorbent assay (ELISA) métodos podem atingir os limites de detecção de aproximadamente 10 -10 4 5 E. coli em testes vezes semelhantes 25,26. Assim, o teste decisivo bacteriana oferece a sensibilidade de detecção comparáveis.
Embora o teste decisivo bacteriana é fácil de realizar e pode produzir mudanças de cores vibrantes, vários fatores podem afetar significativamente os resultados dos testes. Em primeiro lugar, a qualidade de urease é muito importante. Temos usado urease de diferentes fontes e encontrou os resultados do teste podem variar significativamente. Recomendamos o uso de urease da origem especificada na seção Materiais.
A montagem de ADNzima / urease / esferas magnéticas precisa de atenção especial. Lavagem minuciosa de contas magnéticas para remover UrDNA não hibridada é necessário para evitar resultados falso-positivos. Cuidado também deve ser tomado para evitar a acumulação de esferas magnéticas residuais na superfície interior do LID do tubo de microcentrífuga, o que pode ser difícil de ver. Uma vez lá, as esferas magnéticas não estão mais sujeitos a separação magnética e assim, pode levar algum UrDNA não hibridada que podem levar a sinais falsos positivos na reacção repórter. É também importante para evitar deixar o tubo de microcentrifugação na cremalheira magnética durante mais de 10 min durante o passo de separação magnética. Os grânulos podem agregar ou manter o tubo de microcentrífuga, o que pode reduzir a eficiência de lavagem e introduzem inconsistência de lote para lote. A inclusão de 0,01% de Tween-20 na solução de lavagem pode melhorar a consistência de lote para lote e deve ser implementado.
As esferas magnéticas revestidas com estreptavidina são, o qual foi utilizado como âncora para montar conjugados ADNzima-urease sobre as pérolas magnéticas. Tanto a estreptavidina e de urease são moléculas de proteína que podem ser desnaturadas durante o armazenamento. Nós normalmente armazenam as contas ADNzima-urease-magnéticos montados a 4 ° C por até 4 semanase fazer lotes frescos regularmente para obter resultados mais consistentes.
Cuidados também devem ser tomados para evitar acidentalmente tomar esferas magnéticas na etapa de separação magnética (passo 6.9) após a ativação ADNzima. A partir da nossa experiência, os detritos celulares e outras partículas na solução pode reduzir a eficiência da separação magnética, e por conseguinte, algumas esferas magnéticas pode ser inadvertidamente retirada durante a pipetagem. Isto irá resultar em falsos-positivos. Recomendamos as seguintes medidas para aliviar o problema: um maior tempo de separação (por exemplo, 5-10 min), uma versão mais lenta de pressão sobre a pipeta para permitir a retirada suave do sobrenadante, e submetendo o sobrenadante para uma rodada adicional de separação magnética .
Finalmente, é importante para evitar a contaminação do acidente solução estoque repórter pela urease durante o curso de uma experiência em que múltiplas amostras são testadas. Dada a elevada reactividade de uRease, a contaminação desta natureza pode levar a resultados falso-positivos.
Divulgações
The authors have nothing to disclose.
Agradecimentos
The funding for this research project was provided by the Natural Sciences and Engineering Research Council of Canada (NSERC) via a Discovery Grant to YL.
Materiais
| Name | Company | Catalog Number | Comments |
| Ethylenediaminetetraacetic acid (EDTA) | VWR AMRESCO | 0105 | |
| Sodium Hydroxide (NaOH) pellets | BIO BASIC CANADA INC. | SB6789 | |
| Tris-base | VWR AMRESCO | 0497 | |
| Boric acid | AMRESCO | 0588 | |
| Urea | VWR AMRESCO | M123 | |
| 40% acrylamide/bisacrylamide (29:1) solution | BIO BASIC CANADA INC. | A0007 | |
| Sucrose | Bioshop Canada inc. | SUC507 | |
| Bromophenol blue | Bioshop Canada inc. | BRO777 | |
| Xylenecyanol FF | SIGMA-ALDRICH | X-4126 | |
| 10% sodium dodecyl sulfate | Bioshop Canada inc. | SDS001 | |
| Hydrochloric Acid (HCl) | CALEDON LABORATORIES LTD | 6026 | |
| Sodium Chloride (NaCl) | Bioshop Canada inc. | SOD001 | |
| 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid (HEPES) | Bioshop Canada inc. | HEP001 | |
| Magnesium Chloride (II) hexahydrate | VWR AMRESCO | 0288 | |
| Tween 20 | Bioshop Canada inc. | TW508 | |
| Adenosine Triphospahte (ATP) | AMRESCO | 0220 | |
| Sodium Acetate trihydrate (NaOAc) | SIGMA-ALDRICH | S8625 | |
| Ethanol | Commercial Alcohols | P016EAAN | |
| Tetramethyleneethylenediamine (TEMED) | AMRESCO | 0761 | |
| 10% Ammonium persulfate (APS) | BIO BASIC CANADA INC. | AB0072 | |
| Succinimidyl 4-(N-maleimidomethyl) cyclohexane-1-carboxylate (SMCC) | ThermoFisher SCIENTIFIC | 22360 | |
| Dimethyl sulfoxide (DMSO) | CALEDON LABORATORIES | 803540 | |
| Urease | SIGMA-ALDRICH | U0251 | |
| 1x Phosphate Buffered Saline (PBS) | ThermoFisher SCIENTIFIC | 70011-069 | |
| 0.04% Phenol red | SIGMA-ALDRICH | P3532 | |
| 10x T4 polynucleotide kinase reaction buffer | Lucigen | 30061-1 | |
| 10x T4 DNA ligase reaction buffer | Bio Basics Canada | B1122-B | |
| T4 DNA ligase (5 U/μl) | Thermo Fischer Scientific | B1122 | |
| Luria Bertani (LB) Broth | AMRESCO | J106 | |
| Agar | AMRESCO | J637 | |
| T4 polynucleotide kinase (10 U/μl) | Lucigen | 30061-1 | |
| E. coli K12 (MG1655) | ATCC | ATCC700926 | |
| Centrifuge | Beckman Coulter, Inc. | 392187 | |
| Glass plates | CBS scientific | ngp-250nr | |
| 0.75 mm thick spacers | CBS scientific | VGS-0725r | |
| 12-well comb | CBS scientific | VGC-7512 | |
| UV Lamp | UVP | 95-0017-09 | |
| Spectrophotometer (NanoVue) | GE Healthcare | N/A | |
| Metal plate | CBS scientific | CPA165-250 | |
| Vortex | VWR International | 58816-123 | |
| Gel electrophoresis apparatus | CBS scientific | ASG-250 | |
| Petri dishes | VWR International | 25384-342 | |
| 100 kDa MWCO centrifugal filters | EMD Millipore | UFC510024 | |
| Magnetic Bead (BioMag) | Bangs Laboratories Inc | BM568 | |
| Magnetic Seperation Rack | New England BioLabs | S1506S | |
| Microfuge tubes | Sarstedt | 72.69 | |
| Syringe filter (0.22 μm) | VWR International | 28145-501 | |
| 14 ml culture tube | VWR International | 60818-725 | |
| Cell culture incubator | Eppendorf Scientific | M13520000 | |
| Branson Ultrasonic cleaner | Branson | N/A | |
| Camera (Canon Powershot G11) | Canon | N/A | |
| 50 ml conical tube | VWR International | 89004-364 |
Referências
- Daar, A. S., et al. Top ten biotechnologies for improving health in developing countries. Nat. Genet. 32, 229-232 (2002).
- Newman, J. D., Turner, A. P. Home blood glucose biosensors: a commercial perspective. Biosens. Bioelectron. 20, 2435-2453 (2005).
- Turner, A. P. Biosensors: sense and sensibility. Chem. Soc. Rev. 42, 3184-3196 (2013).
- Tram, K., Kanda, P., Salena, B. J., Huan, S. Y., Li, Y. F. Translating Bacterial Detection by DNAzymes into a Litmus Test. Angew. Chem. Int. Ed. 53, 12799-12802 (2014).
- Ali, M. M., Aguirre, S. D., Lazim, H., Li, Y. Fluorogenic DNAzyme probes as bacterial indicators. Angew. Chem. Int. Ed. 50, 3751-3754 (2011).
- Schlosser, K., Li, Y. Biologically inspired synthetic enzymes made from DNA. Chem. Biol. 16, 311-322 (2009).
- Tuerk, C., Gold, L. Systematic evolution of ligand by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Breaker, R. R. Making catalytic DNAs. Science. 290, 2095-2096 (2000).
- Liu, J., Cao, Z., Lu, Y. Functional nucleic acid sensors. Chem. Rev. 109, 1948-1998 (2009).
- Navani, N. K., Li, Y. Nucleic acid aptamers and enzymes as sensors. Curr. Opin. Chem. Biol. 10, 272-281 (2006).
- Li, J., Lu, Y. A highly sensitive and selective catalytic DNA biosensor for lead ions. J. Am. Chem. Soc. 122, 10466-10467 (2000).
- Liu, J., Lu, Y. A colorimetric lead biosensor using DNAzyme-directed assembly of gold nanoparticles. J. Am. Chem. Soc. 125, 6642-6643 (2003).
- Liu, Z., Mei, S. H. J., Brennan, J. D., Li, Y. Assemblage of signaling DNA enzymes with intriguing metal specificity and pH dependence. J. Am. Chem. Soc. 125, 7539-7545 (2003).
- Liu, J., et al. A catalytic beacon sensor for uranium with parts-per-trillion sensitivity and millionfold selectivity. Proc. Natl. Acad. Sci. USA. 104, 2056-2061 (2007).
- Huang, P. J., Vazin, M., Liu, J. In vitro selection of a new lanthanide-dependent DNAzyme for ratiometric sensing lanthanides. Anal. Chem. 86, 9993-9999 (2014).
- Chiuman, W., Li, Y. Simple fluorescent sensors engineered with catalytic DNA 'MgZ' based on a non-classic allosteric design. PLoS One. 2, e1224 (2007).
- Xiang, Y., Lu, Y. Using personal glucose meters and functional DNA sensors to quantify a variety of analytical targets. Nat. Chem. 3, 697-703 (2011).
- Aguirre, S. D., Ali, M. M., Salena, B. J., Li, Y. A sensitive DNA enzyme-based fluorescent assay for bacterial detection. Biomolecules. 3, 563-577 (2013).
- Aguirre, S. D., Ali, M. M., Kanda, P., Li, Y. F. Detection of Bacteria Using Fluorogenic DNAzymes. J. Vis. Exp. (63), e3961 (2012).
- Shen, Z., et al. A catalytic DNA activated by a specific strain of bacterial pathogen. Angew. Chem. Int. Ed. 54, (2015).
- He, S., et al. Highly specific recognition of breast tumors by an RNA-cleaving fluorogenic DNAzyme probe. Anal. Chem. 87, 569-577 (2015).
- Sumner, J. B., Hand, D. B. Isoelectric point of crystalline urease. J. Am. Chem. Soc. 51, 1255-1260 (1929).
- Karplus, P. A., Pearson, M., Hausinger, R. P. 70 Years of crystalline urease: What have we learned. Acc. Chem. Res. 30, 330-337 (1997).
- Omiccioli, E., Amagliani, G., Brandi, G., Magnani, M. A new platform for Real-Time PCR detection of Salmonella spp., Listeria monocytogenes and Escherichia coli O157 in milk. Food Microbiol. 26, 615-622 (2009).
- Cui, S., Schroeder, C. M., Zhang, D. Y., Meng, J. Rapid sample preparation method for PCR-based detection of Escherichia coli O157:H7 in ground beef. J. Appl. Microbiol. 95, 129-134 (2003).
- Ibekwe, A. M., Watt, P. M., Grieve, C. M., Sharma, V. K., Lyons, S. R. Multiplex fluorogenic real-time PCR for detection and quantification of Escherichia coli O157:H7 in dairy wastewater wetlands. Appl. Environ. Microbiol. 68, 4853-4862 (2002).
- Strachan, N. J., Ogden, I. D. A sensitive microsphere coagulation ELISA for Escherichia coli O157:H7 using Russell's viper venom. FEMS Microbiol Lett. 186, 79-84 (2000).
- de Boer, E., Beumer, R. R. Methodology for detection and typing of foodborne microorganisms. Int. J. Food Microbiol. 50, 119-130 (1999).
- Gracias, K. S., McKillip, J. L. A review of conventional detection and enumeration methods for pathogenic bacteria in food. Can. J. Microbiol. 50, 883-890 (2004).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados