Method Article
Колометрической Обнаружение бактерий с помощью лакмусовой тест
В этой статье
Резюме
We describe a protocol for colorimetric detection of E. coli using a modified litmus test that takes advantage of an RNA-cleaving DNAzyme, urease, and magnetic beads.
Аннотация
There are increasing demands for simple but still effective methods that can be used to detect specific pathogens for point-of-care or field applications. Such methods need to be user-friendly and produce reliable results that can be easily interpreted by both specialists and non-professionals. The litmus test for pH is simple, quick, and effective as it reports the pH of a test sample via a simple color change. We have developed an approach to take advantage of the litmus test for bacterial detection. The method exploits a bacterium-specific RNA-cleaving DNAzyme to achieve two functions: recognizing a bacterium of interest and providing a mechanism to control the activity of urease. Through the use of magnetic beads immobilized with a DNAzyme-urease conjugate, the presence of bacteria in a test sample is relayed to the release of urease from beads to solution. The released urease is transferred to a test solution to hydrolyze urea into ammonia, resulting in an increase of pH that can be visualized using the classic litmus test.
Введение
Bacterial pathogens are one of the major causes of global morbidity and mortality. Outbreaks from hospital-acquired infections, food-borne pathogens, and bacterial contaminants in the environment pose serious and on-going threats to public health and safety. To prevent these outbreaks, effective tools are needed that permit pathogen detection in a timely fashion under a variety of settings. Simple but still effective tests that are portable and cost-effective are greatly coveted, especially in regions that are susceptible to outbreaks but cannot afford expensive testing facilities.1-3 Although there exists a multitude of methods to detect bacteria, many of them are not suitable as screening or on-site testing tools because they require long test times, expensive instruments and complicated testing procedures.
Colorimetric tests are particularly attractive for point-of-care or field applications as color changes can be easily detected by the naked eye. The litmus test for pH is simple, quick, and effective. Although it is a very old technology, it is still widely used today because of its simplicity and effectiveness. Surprisingly, this simple test had never been modified to achieve the detection of other analytes before we recently developed an approach of modifying this test for E. coli testing.4
The expanded litmus test for E. coli employs three additional components: an E. coli activated RNA-cleaving DNAzyme (EC1), 5 urease, and magnetic beads. DNAzymes refer to synthetic single-stranded DNA molecules with catalytic activity.6 They can be isolated from random-sequence DNA pools using in vitro selection.7,8 They are highly stable and can be produced cost-effectively using high-efficiency automated DNA synthesis.9 For these reasons, DNAzymes, particularly RNA-cleaving DNAzymes, have been widely examined for biosensing applications.6,10,11 RNA-cleaving DNAzyme sensors have been developed to detect metal ions,12-16 small molecules,17,18 bacterial pathogens5,19-21 and cancer cells.22 Given the great availability of target-induced RNA-cleaving DNAzymes, any assay that utilizes a DNAzyme can be potentially expanded to detect a diverse range of analytes.
Urease is chosen for its ability to hydrolyze urea into ammonia,23,24 resulting in a pH increase. Urease is also highly efficient, stable and amenable for conjugation to other biomolecules. Therefore, we postulated that a conjugate of an RNA-cleavage DNAzyme with urease would allow the use of litmus test for the detection of other targets.5
The action of the RNA-cleaving DNAzyme is relayed to urease-mediated increase of pH through the use of magnetic beads that are immobilized with the DNAzyme-urease conjugate. Because the activity of the DNAzyme under investigation is strictly dependent on E. coli, the presence of this bacterium in the test solution will result in the release of urease from the magnetic beads to the solution, which is then taken and used to hydrolyze urea in a reporter solution that contains a pH-sensitive dye. The final outcome of this procedure is a color change that can be conveniently reported by the dye or pH paper.
протокол
1. Приготовление реагентов и буферные
- 0,5 М этилендиаминтетрауксусной кислоты (ЭДТА)
- В 2 л лабораторный стакан, добавляют 186,1 г ЭДТА в 800 мл деионизованной дистиллированной воды (DDH 2 O). Доводят рН раствора до 8,0 с помощью NaOH гранул. Добавить DDH 2 O до конечного объема 1,0 л и передача раствора на автоклавируемом стеклянная бутылка для автоклавирования и хранят при температуре 4 ° С.
- 10 × Трис-борат ЭДТА (10x КЭ)
- В пластиковом стакане емкостью 4 л, добавляют 432 г Трис-основание, 200 г борной кислоты, 80 мл 0,5 М ЭДТА (рН 8,0) и DDH 2 O до конечного объема 4 L. Mix не используя мешалку , пока все компоненты растворены. Переносят раствор до 1 л стеклянные бутылки для автоклавирования и хранить при температуре 4 ° С.
- 10% денатурирующих полиакриламидном Stock
- В пластиковый стакан 4 л, добавить 1681.7 г мочевины, 400 мл 10x TBE 1 л 40% акриламид / бис - акриламид (29: 1) раствор и DDH 2 O до FИнал объем 4 L. Mix, используя мешалку, пока мочевины не растворится. Переносят раствор до 1 л бутылках из желтого стекла и хранят при температуре 4 ° С.
- 2x Гель буфера для загрузки (2x GLB)
- В стекл нный стакан емкостью 200 мл, добавляют 44 г мочевины, 8 г сахарозы, 10 мг бромфеноловый синий, 10 мг ксилолцианола FF, 400 мкл 10% додецилсульфата натрия и 4 мл 10 × КЭ. Добавить DDH 2 O до конечного объема 40 мл и растворения компонентов с мягким нагреванием при 50 ° С при перемешивании с помощью магнитной мешалки. Передача 1 мл аликвоты по 1,5 мл микроцентрифужных труб и хранить при температуре 4 ° С.
Примечание: Перед использованием этой команды требует кратковременного нагревания при 90 ° С для повторного растворения любых твердых веществ.
- В стекл нный стакан емкостью 200 мл, добавляют 44 г мочевины, 8 г сахарозы, 10 мг бромфеноловый синий, 10 мг ксилолцианола FF, 400 мкл 10% додецилсульфата натрия и 4 мл 10 × КЭ. Добавить DDH 2 O до конечного объема 40 мл и растворения компонентов с мягким нагреванием при 50 ° С при перемешивании с помощью магнитной мешалки. Передача 1 мл аликвоты по 1,5 мл микроцентрифужных труб и хранить при температуре 4 ° С.
- 1 М Трис-гидрохлорида (трис-HCl) (рН 7,5)
- В стеклянную колбу, добавляют 12,1 г трис-основания и 70 мл DDH 2 O и перемешивают до тех пор , пока твердое вещество не растворится. Доводят рН до 7,5 с использованием 1 М соляной кислоты (HCl). Добавить DDH 2 O до конечного объема 100 мл, автоклав и STOповторно при температуре 4 ° C
- 5 М хлорида натрия (NaCl)
- В стеклянную колбу, растворяют 58,4 г NaCl в 150 мл DDH 2 O. Отрегулируйте громкость до 200 мл с DDH 2 O. Хранить при температуре 4 ° С.
- ДНК-буфера для элюции
- В стеклянную колбу, смешивают 2,0 мл 1 М Трис-HCl (рН 7,5), 8,0 мл 5 М NaCl и 0,4 мл 0,5 М ЭДТА (рН 8,0). Отрегулируйте громкость до 200 мл с DDH 2 O, автоклав и хранят при температуре 4 ° C.
- 1 М 4- (2-гидроксиэтил) -1-пиперазинэтансульфонова кислота (HEPES) (рН 7,4)
- В стеклянной бутылке, растворяют 2,38 г HEPES в 80 мл DDH 2 O. Доводят рН до 7,4 с использованием 5 н NaOH и добавляют DDH 2 O до конечного объема 100 мл.
- 1 М хлорид магния (MgCl 2)
- В стеклянной бутылке, добавьте 2,03 г MgCl 2 -6Н 2 O и доводят объем до 100 мл с DDH 2 O.
- Реакция буфера (РБ)
- В 50 мл коническую пробирку, добавляют 50 мкл 1 М HEPES (рН 7,4), 1,5 мл 5 М NaCl, 0,75 мл 1 М MgCl 2, и 5 мкл Tween-20. Добавить DDH 2 O до конечного объема 50 мл. Приготовьте раствор и фильтруют в другую коническую трубку с помощью шприца управляемый фильтр (0,22 мкм) и хранят при температуре 4 ° С до использования.
- Связывающем буфере (ВВ)
- В 50 мл коническую пробирку, добавляют 500 мкл 1 М Трис-HCl (рН 7,5), 8,8 г NaCl, 50 мкл 1 М MgCl 2 и 5 мкл Tween 20. Добавить DDH 2 O до конечного объема 50 мл. Приготовьте раствор и фильтруют в другую коническую трубку с помощью шприца управляемый фильтр (0,22 мкм) и хранят при температуре 4 ° С до использования.
- Раствор субстрата (SS)
- В 50 мл коническую пробирку, добавляют 5,8 г NaCl, 3 мл 1 М MgCl 2, 1,5 г мочевины и 40 мл DDH 2 O. Доводят рН раствора до 5,0 с помощью 10 мМ HCl. Поскольку решение не буферизуется, регулирования рН с помощью HClтщательно путем добавления небольших аликвот HCl. Добавить DDH 2 O до конечного объема 50 мл.
- Приготовьте раствор и фильтруют в другую коническую трубку с помощью шприца управляемый фильтр (0,22 мкм) и хранят при температуре 4 ° С до использования.
- Лурия Бертани (LB) Бульон
- В химический стакан, растворяют 20,0 г LB порошка в 1 л DDH 2 O. Переложить в стеклянную колбу, автоклав, и хранят при температуре 4 ° С.
- 1,5% LB-агар
- В 250 мл колбу, добавляют 1,5 г агара и 100 мл LB бульона. Автоклавы и хранить при температуре 4 ° С.
- Чашки с агаром
- Растворяться агар LB в микроволновой печи и охлаждения раствора до ~ 50 ° C. Налейте раствор в чашки Петри, что делает ~ 5 пластин и позволить им затвердеть.
2. Синтез и очистка Е. coli- отзывчивым ДНКзим EC1
- Синтез EC1 Шаблона опосредованного ферментативного лигирования
- очищают комлинкercially синтезированные олигонуклеотиды БС1, DE1, и Т1 (последовательности , представленные в Таблице 1) 10% денатурирующих электрофореза в полиакриламидном геле (dPAGE) в соответствии со стандартными протоколами.
- Подготовьте 100 мкМ запас BS1, DE1 и T1. Хранить при -20 ° С до использования.
- В 1,5 мл микроцентрифужную пробирку, добавляют 38,5 мкл DDH 2 O, 5 мкл DE1 и 5 мкл 10х Т4 реакционного буфера полинуклеотид - киназы , предоставляемые поставщиком фермента (500 мМ Трис-HCl (рН 7,6), 100 мМ MgCl 2, 50 мМ дитиотреитола (ДТТ), 1,0 мМ спермидин).
- Добавить 1 мкл аденозинтрифосфата (АТФ) (100 мм). Добавьте 5 единиц Т4 полинуклеотидной киназы (10 ед / мкл). Смешать осторожно пипеткой реакционную смесь.
- Инкубируют реакционную смесь при температуре 37 ° С в течение 30 мин.
- Гасят реакции при нагревании до 90 ° С в течение 5 мин.
- Добавьте 118 мкл DDH 2 O 5 мкл BS1 и 5 мкл T1. Реакционную смесь нагревают до 90 ° С в течение 2 милиN, а затем охлаждают до комнатной температуры в течение 10 мин.
- Добавьте 20 мкл 10 × Т4 ДНК - лигазы реакции предоставленный буфер поставщиком фермента (400 мМ Трис-HCl (рН 7,8), 100 мМ MgCl 2, 100 мМ ДТТ, 5 мМ АТФ). Добавьте 10 единиц ДНК-лигазы Т4 (5 ед / мкл) и тщательно перемешать с помощью пипетки.
- Инкубируют при комнатной температуре в течение 2 часов.
- Добавьте 20 мкл 3 М ацетата натрия (NaOAc) (рН 5,2), 500 мкл холодного 100% этанола. Приготовьте раствор путем встряхивания и поместить трубку в C морозильной камере -20 ° С в течение 30 мин.
- Центрифуга микроцентрифуге при 20000 х г в течение 20 мин при температуре 4 ° С. Осторожно удалите супернатант с помощью пипетки.
- Промывают осадок с холодным 70% этанолом и снова центрифуге при 20000 х г в течение 10 мин при 4 ° C. Снова удалить супернатант и высушить осадок под вакуумом в течение 10 мин.
- Ресуспендируют ДНК с 15 мкл DDH 2 O , а затем добавить 15 мкл 2х GLB.
- Вортексе и нагревают до 90 ° С в течение2 мин. Очищают ДНК полной длины, с помощью 10% -ного dPAGE, как описано ниже.
- Настройка 10% dPAGE
- Гладко две стеклянные пластины (одна полная и одна тарелка с надрезом), два 0,75 мм толщиной прокладки, и расчесывать 12-луночного. Положите одну стеклянную пластину на плоскую поверхность с двумя прокладками на каждой стороне и рифленые пластины на вершине. Клип две пластины вместе с помощью четырех клипов, предоставленных поставщиком.
- В пластиковый стакан емкостью 150 мл, заливают 40 мл 10% -ного dPAGE 40 мкл тетраметилэтилендиамина (TEMED) и 400 мкл 10% персульфата аммония (APS). Смешайте компоненты и вылить раствор между пластинами медленно.
- Вставьте гребень, а затем позволить гель полимеризуется в течение 10 мин. После того , как гель полимеризуется, медленно удалить расческу и промыть лунки с DDH 2 O.
- Установите пластины на гель-электрофореза устройства. Используйте металлическую пластину для рассеивания тепла для предотвращения перегрева.
- Налейте 1x КЭ на верхней и нижней палати гарантировать, что скважины хорошо погружают в буфер. Промыть лунки с 1x КЭ с помощью пипетки или шприца.
- Установите устройство для запуска при 35 мА и предварительный прогон в течение 15 мин до загрузки образцов.
- Элюирование с наложенной лигатурой, EC1 с 10% dPAGE
- После этапа 2.2.6, запустить гель при 35 мА пока нижний краситель (бромофенолового синий) достигает нижней части стеклянной пластины. Это должно занять примерно 1,5 часа. Выключите питание и удалите пластины из аппарата.
- Положите тарелки на несколько листов бумажных полотенец и аккуратно удалите распорки из стеклянных пластин. Осторожно снимите верхнюю стеклянную пластину и обернуть гель с полиэтиленовой пленкой.
- Флип гель над удалить вторую стеклянную пластину и снова накрыть полиэтиленовой пленкой. Будьте осторожны, чтобы избежать морщин полиэтиленовой пленкой.
- Визуализируйте лигирован продукт с помощью УФ-затенения (260 нм), что будет производить 4 отдельные полосы ДНК (полностью сшита ec1, dE1, BS1, и T1).
- Акцизный верхняя полоса (EC1) со стерильным лезвием бритвы и передачи в 1,5 мл пробирку микроцентрифуге. Раздавить гель с стерильным наконечником пипетки (200 мкл размера наконечника), пока она не превратится в тонкую пасту. Добавить 550 мкл ДНК-буфера для элюции, накройте трубу с алюминиевой фольгой для защиты встроенного флуорофор, и встряхивают в течение 15 мин.
- Центрифуга раствора геля при 20000 х г в течение 5 мин и осторожно передавать 400 мкл супернатанта в другую 1,5 мл микроцентрифужную пробирку. Избегайте снятия геля частей во время пипеткой.
- К пробирке с передаваемым супернатанта добавляют 40 мкл 3 М NaOAc (рН 5,2) и 1,0 мл холодного 100% этанола. Приготовьте раствор путем встряхивания и поместить трубку в C морозильной камере -20 ° С в течение 30 мин.
- Центрифуга микроцентрифуге при 20000 х г в течение 20 мин при температуре 4 ° С. Осторожно удалите супернатант с помощью пипетки.
- Промывают осадок с холодным 70% этанолом и снова центрифуге при 20000 х г в течение 10 мин при 4 ° C. Еще раз, удалитесупернатант и высушить осадок под вакуумом в течение 10 мин.
- Определить концентрацию EC1 путем измерения УФ-поглощению при 260 нм. Делают 10 мкМ запас и хранить пробу при -20 ° С до использования.
3. Сопряжение уреазы к ДНК
- Приготовление сукцинимидилового 4- (N-малеимидометил) циклогексан-1-карбоксилат (SMCC) Фото
- Растворите 1 мг SMCC в 676 мкл ДМСО. Vortex и место на льду до использования.
- Приготовление уреазы запаса
- Растворите 1 мг уреазы в 1 мл 1 × PBS (без Mg 2+ или Ca 2+). Поместите на льду до использования.
Примечание: Кристаллизированный уреазы медленно растворяться и нежное перемешивание необходимо, чтобы избежать денатурации.
- Растворите 1 мг уреазы в 1 мл 1 × PBS (без Mg 2+ или Ca 2+). Поместите на льду до использования.
- Синтез уреазы-ДНК (UrDNA)
- Добавляют 10 мкл 100 мкМ LD1 в 2,5 мл пробирке с. Добавьте 140 мкл DDH 2 O, 40 мкл 10х PBS, и вихрь.
- Добавить 80,5 мкл ККАП sTock, 159,5 мкл ДМСО, вихря и кратко центрифуге с использованием лабораторной центрифуге.
- Инкубируют при 37 ° С в течение 60 мин. Избегайте конденсации влаги под микроцентрифуге крышкой.
- Добавить 200 мкл 1x PBS, 60 мкл 3 М NaOAc (рН 5,2) и 1,5 мл холодного 100% этанола. Приготовьте раствор путем встряхивания и инкубировать при температуре -20 ° С в течение 30 мин.
- Центрифуга решение при 20000 х г в течение 20 мин при температуре 4 ° С. Удалить супернатант и высушить осадок под вакуумом. Избегайте пересушивания.
- К высушенному осадку добавляют 400 мкл уреазы запаса и инкубировать при комнатной температуре в течение 5 часов.
- Перенести 200 мкл неочищенного конъюгата в предварительно промытую 100k MWCO центробежной колонке фильтра. Центрифуга колонку при 14000 х г в течение 5 мин.
- Перенесите оставшиеся 200 мкл сырого конъюгата в колонку и центрифуге при 14000 х г в течение 5 мин. Удалить столбец и поместите его вниз головой в новый 2,0 мл пробирке ( "коллекторной трубки").
- ЦентробежнаяУГЭ отборная труба (с перевернутой колонке) при 1000 мкг в течение 2 мин. Извлеките пробирку для сбора и добавить 30 мкл 1x PBS центробежной колонку для промывки мембраны для дополнительного извлечения конъюгатов.
- Еще раз перевернуть колонку и поместить обратно в пробирку. Центрифуга отборная труба (с инвертированным Column) при 1000 х г в течение 2 мин. Снимите и выбросьте колонку.
- Хранить UrDNA при температуре 4 ° С до использования.
4. Монтаж EC1 и UrDNA на магнитные шарики
- Смешайте магнитный шарик (MB) наличие хорошо и передавать 100 мкл суспензии МБ до 1,5 мл в пробирке. Поместите трубку на микроцентрифуге магнитный держатель стойки для изоляции МБ.
- Удалить супернатант с помощью пипетки и добавляют 150 мкл буфера для связывания (BB) к трубе. Снимите трубку из держателя и осторожно нажмите на трубку для ресуспендирования МБ до однородного раствора. Избегайте разбрызгивания суспензии в тОп трубки или колпачка. Если это произойдет, использовать центрифугу для стендовых испытаний на короткое время раскручивать остаток обратно в суспензии.
- Повторите шаг 4,2 еще два раза.
- К этой суспензии добавляют 10 мкл 10 мкМ EC1. Тщательно перемешать, нажав на трубе.
- Выдержите раствор с мягким встряхивания в течение 30 мин. Нажмите пробирку каждые 2-3 мин, чтобы избежать осаждения МБ.
- Поместите трубку обратно микроцентрифуге на магнитной стойке, чтобы изолировать МБ и удалить супернатант с помощью пипетки. Вымойте MB три раза с 150 мкл BB (как описано в пункте 4.2).
- После промывки завершена, приостановить МБ в общей сложности на 150 мкл ВВ. К этому раствору добавляют 15 мкл UrDNA и нагревают раствор до 45 ° С в течение 2 мин. Охлаждают раствор до комнатной температуры и инкубировать в течение 2 ч.
- Поместите трубку обратно микроцентрифуге на магнитной стойке, чтобы изолировать МБ и удалить супернатант с помощью пипетки.
- Добавляют 100 мкл реакционного буфера (RB). Удалить тон трубку от микроцентрифуге магнитной стойки и осторожно ресуспендируют MB.
- Вымойте MB еще три раза, повторяя шаг 4.9.
Примечание: промытую надосадочную жидкость можно быстро испытания, чтобы определить, является ли негибридизированным UrDNA по-прежнему присутствует, что может привести к ложному положительному сигналу. Испытание может быть сделано путем добавления 10 мкл 50 мМ мочевины и 10 мкл 0,04% фенола красного в моющем растворе. Продолжайте промывать МБ до тех пор, пока промывной раствор не вызывает изменения цвета. 100 мкл суспензии хранят при температуре 4 ° С до использования.
5. Приготовление бактериальных клеток 20
- Культивирование Е. палочки из запасов
- Пластина E. палочки K12 (1655) на LB чашки с агаром из глицерина запаса под пламенем или в кабинете биологической безопасности.
- С помощью стерильной пипетки, осторожно прикоснитесь к глицерине и слегка полоса, поверхность агаровой пластины, чтобы избежать прокалывания агар LB.
- Инвертировать прожилкамипластины и инкубировать при 37 ° С в течение 14 ч.
- Уплотнение плита с парафильмом и хранить при температуре 4 ° С в течение не более 3-х недель.
- Культивирование Е. палочки для сотового Counting
- В стерильной культуральную пробирку объемом 14 мл, дозировать 2 мл LB бульона.
- С помощью стерильной пипетки, выбрать одну колонию с прожилками агаром, приготовленным на стадии 5.1, и передать ее в культуральную пробирку.
- Инкубируйте культуру при 37 ° С и встряхивают при 230 оборотах в минуту в течение 14 часов.
- Серийно разбавить бактериальной культуры в интервалах в 10 раз.
- Для каждого разведенного образца, равномерно пластины пять 100 мкл аликвоты на отдельных чашках с агаром LB. Инверсия пластины и инкубировать при 37 ° С в течение 14 ч.
- Подсчитайте клетки каждого образца, чтобы получить среднюю концентрацию клеток каждого разведения.
Примечание: 10 7 клеток часто используются , чтобы создать эталонные бумажка для E. палочка , как этот уровень E. палочка может вызвать быстрый цвет чанGE. Тем не менее, тест выполнен правильно лакмусовой может обнаружить столь же низко как 500 клеток, как описано в разделе Результаты.
- Подготовка Е. Клетки палочки для тестирования
- Для получения требуемой суспензии клеток, перенос 1 мл культуральной запаса до 1,5 мл в пробирке.
- Центрифуга клеток при 6000 х г в течение 10 мин при температуре 4 ° С. Осторожно удалите супернатант, не нарушая клеточный осадок.
- Добавьте 10 мкл реакционного буфера к осадку клеток и клетки вновь суспендируют. Разрушать ультразвуком суспензии клеток в течение 5 мин. Передачи клеточной суспензии в коробку со льдом в течение 5 мин.
- Разрушать ультразвуком суспензии клеток в течение еще 5 мин.
- Центрифуга клеточной суспензии при 13000 х г в течение 10 мин при температуре 4 ° С. Используйте супернатант для тестирования (10 мкл).
6. лакмусовая бумажка
- В 1,5 мл микроцентрифуге пробирку, предварительно промыть трубку путем добавления и встряхиванием 100 мкл реакционного буфера (RB) в пробирке иотбрасывания буфера.
- Передача 15 мкл собранной EC1 (протокол 4) к промытым пробирке.
- Вымойте магнитные шарики, поместив трубку на микроцентрифуге магнитной стойке. Удалить супернатант с помощью пипетки. Снимите трубку из микроцентрифуге стойки, добавьте 100 мкл РБ и осторожно ресуспендируют магнитные шарики.
- Вымойте MB еще два раза, повторяя шаг 6.3.
- Поместите трубку обратно микроцентрифуге на магнитной стойке, удалить супернатант и добавить 10 мкл E. Образец палочки получают из стадии 5.3.
- Смешайте образец и магнитные гранулы тщательно, осторожно постукивая по пробирке.
- Инкубируют реакционную смесь при комнатной температуре в течение 1 часа.
- К реакции добавляют 90 мкл DDH 2 O и поместите трубку на микроцентрифуге магнитную стойку.
- Примерно через 3 мин магнитной сепарации, тщательно передачи 85 мкл супернатанта в 0,5 мл микроцентрифужную пробирку. Вывод супернатант медленноВо избежание взимания каких-либо магнитных шариков.
- К указанному выше микроцентрифужную пробирку добавляют по 15 мкл 0,04% фенола красного, и 100 мкл раствора субстрата.
- Сфотографировать через определенные промежутки времени, чтобы записать изменения цвета.
Примечание: Изменение рН также можно контролировать с помощью рН-метра с микроэлектродом. Исходное значение рН должно быть приблизительно 5,2-5,5 (раствор желтого цвета). Если нет, то решение может быть отрегулирована путем добавления 1 мМ ацетатного буфера, рН 5,0).
Результаты
Принцип теста с бактериальной лакмусовой объясняется на рисунке 1 Тест использует три основных материалов:. РНК-расщепляющие ДНКзима , который активируется с помощью специфической бактерией, уреазы и магнитных шариков. ДНКзим используется в качестве элемента молекулярного распознавания для достижения высокой специфической детекции бактерии, представляющей интерес. Уреазы и магнитные гранулы используются для достижения трансдукцию сигнала РНК-расщепления активности ДНКзима. Это предполагает создание магнитных шариков, которые содержат уреазы-ДНКазима конъюгатов. В присутствии мишени бактерии, то ДНКзим расщепляет ее РНК-связь. Это действие приводит к диссоциации уреазы из магнитных шариков. Освобожденный уреазы могут быть легко отделены от магнитных гранул и используется для генерации изменение цвета в растворе репортер, который содержит мочевину и рН-чувствительный краситель. Уреазный гидролизует мочевину в аммиак, сопровождающееся повышением рН, запускающего грOlor изменение красителя.
Рисунок 2 представляет собой тест с бактериальной лакмусовой где EC1, Е. палочка -responsive РНК-расщепляющие ДНКазима, был использован в качестве ДНКзима, и фенол красный был использован в качестве рН-отчетности красителя. EC1 был ранее выделен нашей группой из пула ДНК случайной последовательности с использованием методики отбора в пробирке. 5 Наши предыдущие исследования показали , что EC1 является весьма специфичным для E. палочка и проявляет минимальную активность в отношении других бактерий. 5,19 Было обнаружено , что ЕС1 активируется молекулы белка из E. палочка. Хотя личность этого белка биомаркера не была расшифрована, высокая специфичность распознавания позволяет предположить , что этот белок является уникальным для E. палочки. Решение репортер установлен, чтобы иметь исходное значение рН 5,5. При этом рН фенол красный проявляет желтый цвет. Поскольку уреаза гидролизует мочевину в аммиак, основность репортер золесоциологическое загрязнение увеличивается. Это отражается постепенным изменением цвета от желтого до розового цвета. Глубина изменения цвета зависит от следующих двух параметров, как показано на рисунке 2: количество E. Клетки палочки , используемые в стадии активации ДНКзима и времени , отведенного на стадии гидролиза мочевины. Больше E. Клетки палочки привели к сильным изменениям цвета, отражается наблюдения прогрессивного желтого до розового цвета перехода , когда Е. Клетки палочки были последовательно увеличено с 5 до 5 × 10 7 (10-кратное увеличение каждый раз). В то же время, более длительное время для гидролиза мочевины позволило для обнаружения меньшего числа E. Клетки coli (5000 клеток в реакции 1 ч и 500 клеток в 2-часовой реакции).
Изменение рН теста с бактериальной лакмусовой также можно контролировать с помощью измерителя карманным рН и репрезентативные результаты показаны на рисунке 3. Это Was обнаружили , что присутствие 10 7 E. клетки палочки приводит к постепенному увеличению рН на 3 единицы в течение 10 мин. В противоположность этому , отсутствие E. Клетки палочка не вызывает обнаруживаемые изменения рН в соответствии с той же установкой.
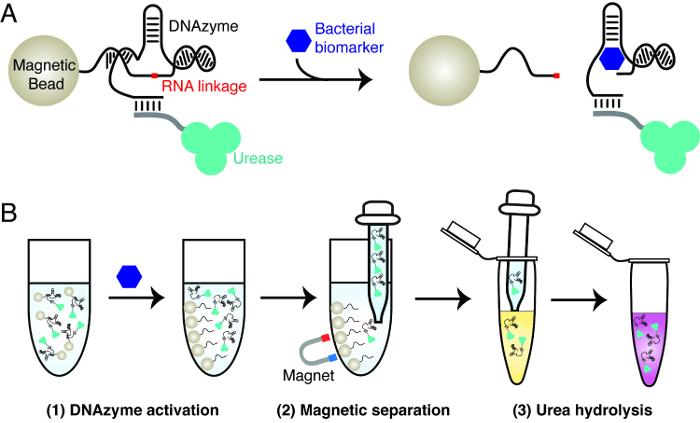
Рисунок 1: Принцип конструкции теста с бактериальной лакмусовой (А) Активация РНК-расщепляющие ДНКзима специфическим биомаркером из интересующей бактерии.. При наличии биомаркера, РНК-расщепляющие ДНКзим иммобилизованного на магнитных гранул расщепляет РНК-связь, что приводит к освобождению меченого уреазы из магнитных гранул в растворе. (B) Трехступенчатая процедура анализа. Шаг 1: активация ДНКзим, как описано в панели A. Шаг 2: магнитная сепарация - освобожденный уреазы отделен от магнитных шариков. Шаг 3: Мочевина гидролиз R12; освобожденный уреазы добавляется в мочевины раствор, содержащий репортером. Уреазный гидролизует мочевину в аммиак, что приводит к изменению рН , которые могут регистрироваться с помощью рН-чувствительного красителя. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.

Рисунок 2: лакмусовая бумажка с E. палочки с помощью E. палочки -responsive ДНКзим EC1. Типичные результаты изменения цвета с различным числом E. Клетки палочки приведенные выше каждой пробирки. Фенол красный был использован в качестве рН-чувствительный краситель. Испытание без E. палочка, использовали в качестве отрицательного контроля. Больше E. Клетки палочки , как ожидается, вызвать высвобождение более молекул уреазы, сопровождающееся бу более сильные изменения цвета. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
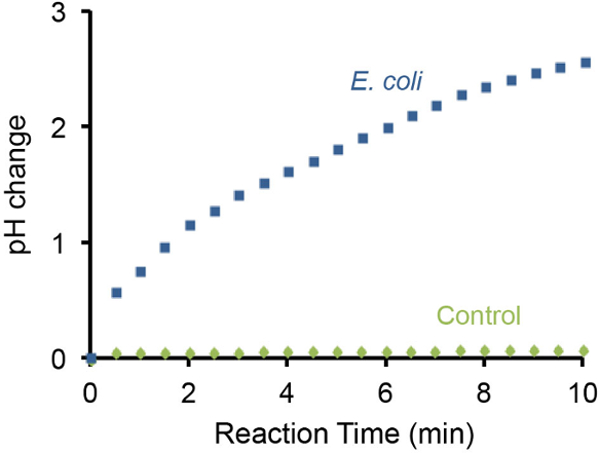
Рисунок 3: Мониторинг увеличение рН с помощью рН - метра Изменение рН вызвано 10 7 E.. Клетки палочки контролировали с помощью портативного рН - метра. Испытание без E. палочка, использовали в качестве отрицательного контроля. Присутствие 10 7 E. палочка клеток в исследуемом растворе может увеличить щелочность единицами ~ 3 рН в течение 10 мин. Пожалуйста , нажмите здесь , чтобы посмотреть увеличенную версию этой фигуры.
| имя | Seствием (5'-3 ') | Заметка |
| BS1 | BTTTT TTTTT TTTAC TCTTC CTAGC FRQGG TTCGA TCAAG | В: 5'-биотин; R: аденин рибонуклеотид; F: флуоресцеин-дТ; Q: dabcyl-дТ |
| DE1 | GATGT GCGTT GTCGA GACCT GCGAC CGGAA CACTA CACTG TGTGG GGATG GATTT CTTTA CAGTT GTGTG TTGAA CGCTG TGTCA AAAAA AAAA | |
| T1 | GACAA CGCAC ATCTC TTGAT CGAAC C | |
| LD1 | XTTTT TTTTT TTTTT TTGAC ACAGC GTTCA | Х: 5'-NH 2 |
Таблица 1: Последовательности синтетических олигонуклеотидов.
Обсуждение
Перевод действия расщепления РНК - активности бактерии реагирующих ДНКзима к лакмусовой бумажкой стало возможным благодаря использованию уреазы и магнитной сепарации, как показано на рисунке 1. Хотя демонстрация модифицированного лакмусовой бумажкой для бактериального обнаружения является сделано с E. -зависимая палочка РНК-расщепляющие ДНКзима, 5,19,20 конструкция может быть расширена , как правило для любой РНК-расщепляющие ДНКзима. Учитывая большое наличие РНК-расщепляющие DNAzymes для различных аналитов и различных методик для выделения новых РНК-расщепляющие DNAzymes из случайных последовательностей пулов для новых целей, мы ожидаем, что модифицированная тестовая платформа бумажка может быть расширена до обнаружения различных целей, представляющих интерес ,
Лакмусовой бумажкой для E. обнаружение палочка может обнаружить 5000 и 500 клеток , когда время реакции отчетности устанавливается равным 1 и 2 ч соответственно. Популярная полимеразной цепной реакции (ПЦР) исэндвич - иммуноферментный анализ (ELISA) методы могут достигнуть пределов обнаружения приблизительно 10 4 -10 5 E. палочки клетки в подобных времена испытаний. 25,26 Таким образом, тест бактериальной лакмусовой предлагает сопоставимую чувствительность обнаружения.
Хотя тест бактериальной лакмусовой легко носить с собой, и может производить яркие изменения цвета, некоторые факторы могут существенно повлиять на результаты испытаний. Во-первых, качество уреазы является очень важным. Мы использовали уреазы из различных источников и нашли результаты испытаний могут значительно различаться. Мы рекомендуем использовать уреазу из источника, указанного в разделе Материалы.
Сборка ДНКзима / уреазы / магнитных шариков нуждается в особом внимании. Тщательное мытье магнитных шариков для удаления негибридизированным UrDNA необходимо для предотвращения ложных положительных результатов. Уход также необходимо принимать, чтобы избежать накопления остаточных магнитных гранул на внутренней поверхности лИдентификатор трубки микроцентрифужную, что может быть трудно увидеть. После того, как там, магнитные шарики, больше не подвергается магнитной сепарации и, таким образом, может выполнять некоторые негибридизированным UrDNA, что может привести к ложноположительных сигналов в реакции репортера. Важно также, чтобы избежать выхода из трубки микроцентрифужную на магнитной стойке в течение более чем 10 минут в течение стадии магнитной сепарации. Гранулы могут агрегировать или прилипает к пробирке, что может уменьшить эффективность стирки и ввести партии к партии противоречивость. Включение 0,01% твин-20 в моющем растворе может улучшить консистенцию от партии к партии и должны быть реализованы.
Магнитные шарики покрывают стрептавидином, который был использован в качестве якоря для сборки ДНКзим-уреазы конъюгатов на магнитных шариков. Оба стрептавидин и уреазы являются белковые молекулы, которые могут быть денатурированные в процессе хранения. Мы обычно хранят собранные ДНКзим-уреазы-магнитных шариков при 4 ° С в течение до 4-х недельи сделать свежие порции регулярно, чтобы достичь более стабильных результатов.
Уход также необходимо соблюдать осторожность во избежание случайного принятия магнитных шариков на стадии магнитной сепарации (шаг 6.9) после активации ДНКзима. По нашему опыту, продукты распада клеток и других частиц в растворе может уменьшить магнитную эффективность разделения, и, следовательно, некоторые магнитные гранулы могут быть непреднамеренно вынимают во время пипеткой. Это приведет к ложно-положительных результатов. Мы рекомендуем следующие меры для смягчения проблемы: большее время разделения (например, 5-10 мин), более медленное высвобождение давления на пипетку, чтобы позволить нежный вывод надосадочной жидкости и подвергают супернатант к дополнительному раунду магнитной сепарации ,
И, наконец, важно, чтобы избежать несчастных случаев загрязнения репортер маточного раствора путем уреазы в ходе эксперимента, в котором тестируются множество образцов. С учетом высокой реакционной способности Urease, загрязнение такого рода может привести к ложно-положительных результатов.
Раскрытие информации
The authors have nothing to disclose.
Благодарности
The funding for this research project was provided by the Natural Sciences and Engineering Research Council of Canada (NSERC) via a Discovery Grant to YL.
Материалы
| Name | Company | Catalog Number | Comments |
| Ethylenediaminetetraacetic acid (EDTA) | VWR AMRESCO | 0105 | |
| Sodium Hydroxide (NaOH) pellets | BIO BASIC CANADA INC. | SB6789 | |
| Tris-base | VWR AMRESCO | 0497 | |
| Boric acid | AMRESCO | 0588 | |
| Urea | VWR AMRESCO | M123 | |
| 40% acrylamide/bisacrylamide (29:1) solution | BIO BASIC CANADA INC. | A0007 | |
| Sucrose | Bioshop Canada inc. | SUC507 | |
| Bromophenol blue | Bioshop Canada inc. | BRO777 | |
| Xylenecyanol FF | SIGMA-ALDRICH | X-4126 | |
| 10% sodium dodecyl sulfate | Bioshop Canada inc. | SDS001 | |
| Hydrochloric Acid (HCl) | CALEDON LABORATORIES LTD | 6026 | |
| Sodium Chloride (NaCl) | Bioshop Canada inc. | SOD001 | |
| 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid (HEPES) | Bioshop Canada inc. | HEP001 | |
| Magnesium Chloride (II) hexahydrate | VWR AMRESCO | 0288 | |
| Tween 20 | Bioshop Canada inc. | TW508 | |
| Adenosine Triphospahte (ATP) | AMRESCO | 0220 | |
| Sodium Acetate trihydrate (NaOAc) | SIGMA-ALDRICH | S8625 | |
| Ethanol | Commercial Alcohols | P016EAAN | |
| Tetramethyleneethylenediamine (TEMED) | AMRESCO | 0761 | |
| 10% Ammonium persulfate (APS) | BIO BASIC CANADA INC. | AB0072 | |
| Succinimidyl 4-(N-maleimidomethyl) cyclohexane-1-carboxylate (SMCC) | ThermoFisher SCIENTIFIC | 22360 | |
| Dimethyl sulfoxide (DMSO) | CALEDON LABORATORIES | 803540 | |
| Urease | SIGMA-ALDRICH | U0251 | |
| 1x Phosphate Buffered Saline (PBS) | ThermoFisher SCIENTIFIC | 70011-069 | |
| 0.04% Phenol red | SIGMA-ALDRICH | P3532 | |
| 10x T4 polynucleotide kinase reaction buffer | Lucigen | 30061-1 | |
| 10x T4 DNA ligase reaction buffer | Bio Basics Canada | B1122-B | |
| T4 DNA ligase (5 U/μl) | Thermo Fischer Scientific | B1122 | |
| Luria Bertani (LB) Broth | AMRESCO | J106 | |
| Agar | AMRESCO | J637 | |
| T4 polynucleotide kinase (10 U/μl) | Lucigen | 30061-1 | |
| E. coli K12 (MG1655) | ATCC | ATCC700926 | |
| Centrifuge | Beckman Coulter, Inc. | 392187 | |
| Glass plates | CBS scientific | ngp-250nr | |
| 0.75 mm thick spacers | CBS scientific | VGS-0725r | |
| 12-well comb | CBS scientific | VGC-7512 | |
| UV Lamp | UVP | 95-0017-09 | |
| Spectrophotometer (NanoVue) | GE Healthcare | N/A | |
| Metal plate | CBS scientific | CPA165-250 | |
| Vortex | VWR International | 58816-123 | |
| Gel electrophoresis apparatus | CBS scientific | ASG-250 | |
| Petri dishes | VWR International | 25384-342 | |
| 100 kDa MWCO centrifugal filters | EMD Millipore | UFC510024 | |
| Magnetic Bead (BioMag) | Bangs Laboratories Inc | BM568 | |
| Magnetic Seperation Rack | New England BioLabs | S1506S | |
| Microfuge tubes | Sarstedt | 72.69 | |
| Syringe filter (0.22 μm) | VWR International | 28145-501 | |
| 14 ml culture tube | VWR International | 60818-725 | |
| Cell culture incubator | Eppendorf Scientific | M13520000 | |
| Branson Ultrasonic cleaner | Branson | N/A | |
| Camera (Canon Powershot G11) | Canon | N/A | |
| 50 ml conical tube | VWR International | 89004-364 |
Ссылки
- Daar, A. S., et al. Top ten biotechnologies for improving health in developing countries. Nat. Genet. 32, 229-232 (2002).
- Newman, J. D., Turner, A. P. Home blood glucose biosensors: a commercial perspective. Biosens. Bioelectron. 20, 2435-2453 (2005).
- Turner, A. P. Biosensors: sense and sensibility. Chem. Soc. Rev. 42, 3184-3196 (2013).
- Tram, K., Kanda, P., Salena, B. J., Huan, S. Y., Li, Y. F. Translating Bacterial Detection by DNAzymes into a Litmus Test. Angew. Chem. Int. Ed. 53, 12799-12802 (2014).
- Ali, M. M., Aguirre, S. D., Lazim, H., Li, Y. Fluorogenic DNAzyme probes as bacterial indicators. Angew. Chem. Int. Ed. 50, 3751-3754 (2011).
- Schlosser, K., Li, Y. Biologically inspired synthetic enzymes made from DNA. Chem. Biol. 16, 311-322 (2009).
- Tuerk, C., Gold, L. Systematic evolution of ligand by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Breaker, R. R. Making catalytic DNAs. Science. 290, 2095-2096 (2000).
- Liu, J., Cao, Z., Lu, Y. Functional nucleic acid sensors. Chem. Rev. 109, 1948-1998 (2009).
- Navani, N. K., Li, Y. Nucleic acid aptamers and enzymes as sensors. Curr. Opin. Chem. Biol. 10, 272-281 (2006).
- Li, J., Lu, Y. A highly sensitive and selective catalytic DNA biosensor for lead ions. J. Am. Chem. Soc. 122, 10466-10467 (2000).
- Liu, J., Lu, Y. A colorimetric lead biosensor using DNAzyme-directed assembly of gold nanoparticles. J. Am. Chem. Soc. 125, 6642-6643 (2003).
- Liu, Z., Mei, S. H. J., Brennan, J. D., Li, Y. Assemblage of signaling DNA enzymes with intriguing metal specificity and pH dependence. J. Am. Chem. Soc. 125, 7539-7545 (2003).
- Liu, J., et al. A catalytic beacon sensor for uranium with parts-per-trillion sensitivity and millionfold selectivity. Proc. Natl. Acad. Sci. USA. 104, 2056-2061 (2007).
- Huang, P. J., Vazin, M., Liu, J. In vitro selection of a new lanthanide-dependent DNAzyme for ratiometric sensing lanthanides. Anal. Chem. 86, 9993-9999 (2014).
- Chiuman, W., Li, Y. Simple fluorescent sensors engineered with catalytic DNA 'MgZ' based on a non-classic allosteric design. PLoS One. 2, e1224 (2007).
- Xiang, Y., Lu, Y. Using personal glucose meters and functional DNA sensors to quantify a variety of analytical targets. Nat. Chem. 3, 697-703 (2011).
- Aguirre, S. D., Ali, M. M., Salena, B. J., Li, Y. A sensitive DNA enzyme-based fluorescent assay for bacterial detection. Biomolecules. 3, 563-577 (2013).
- Aguirre, S. D., Ali, M. M., Kanda, P., Li, Y. F. Detection of Bacteria Using Fluorogenic DNAzymes. J. Vis. Exp. (63), e3961 (2012).
- Shen, Z., et al. A catalytic DNA activated by a specific strain of bacterial pathogen. Angew. Chem. Int. Ed. 54, (2015).
- He, S., et al. Highly specific recognition of breast tumors by an RNA-cleaving fluorogenic DNAzyme probe. Anal. Chem. 87, 569-577 (2015).
- Sumner, J. B., Hand, D. B. Isoelectric point of crystalline urease. J. Am. Chem. Soc. 51, 1255-1260 (1929).
- Karplus, P. A., Pearson, M., Hausinger, R. P. 70 Years of crystalline urease: What have we learned. Acc. Chem. Res. 30, 330-337 (1997).
- Omiccioli, E., Amagliani, G., Brandi, G., Magnani, M. A new platform for Real-Time PCR detection of Salmonella spp., Listeria monocytogenes and Escherichia coli O157 in milk. Food Microbiol. 26, 615-622 (2009).
- Cui, S., Schroeder, C. M., Zhang, D. Y., Meng, J. Rapid sample preparation method for PCR-based detection of Escherichia coli O157:H7 in ground beef. J. Appl. Microbiol. 95, 129-134 (2003).
- Ibekwe, A. M., Watt, P. M., Grieve, C. M., Sharma, V. K., Lyons, S. R. Multiplex fluorogenic real-time PCR for detection and quantification of Escherichia coli O157:H7 in dairy wastewater wetlands. Appl. Environ. Microbiol. 68, 4853-4862 (2002).
- Strachan, N. J., Ogden, I. D. A sensitive microsphere coagulation ELISA for Escherichia coli O157:H7 using Russell's viper venom. FEMS Microbiol Lett. 186, 79-84 (2000).
- de Boer, E., Beumer, R. R. Methodology for detection and typing of foodborne microorganisms. Int. J. Food Microbiol. 50, 119-130 (1999).
- Gracias, K. S., McKillip, J. L. A review of conventional detection and enumeration methods for pathogenic bacteria in food. Can. J. Microbiol. 50, 883-890 (2004).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены