Method Article
איתור מדד-צבע של חיידקים באמצעות מבחן לקמוס
In This Article
Summary
We describe a protocol for colorimetric detection of E. coli using a modified litmus test that takes advantage of an RNA-cleaving DNAzyme, urease, and magnetic beads.
Abstract
There are increasing demands for simple but still effective methods that can be used to detect specific pathogens for point-of-care or field applications. Such methods need to be user-friendly and produce reliable results that can be easily interpreted by both specialists and non-professionals. The litmus test for pH is simple, quick, and effective as it reports the pH of a test sample via a simple color change. We have developed an approach to take advantage of the litmus test for bacterial detection. The method exploits a bacterium-specific RNA-cleaving DNAzyme to achieve two functions: recognizing a bacterium of interest and providing a mechanism to control the activity of urease. Through the use of magnetic beads immobilized with a DNAzyme-urease conjugate, the presence of bacteria in a test sample is relayed to the release of urease from beads to solution. The released urease is transferred to a test solution to hydrolyze urea into ammonia, resulting in an increase of pH that can be visualized using the classic litmus test.
Introduction
Bacterial pathogens are one of the major causes of global morbidity and mortality. Outbreaks from hospital-acquired infections, food-borne pathogens, and bacterial contaminants in the environment pose serious and on-going threats to public health and safety. To prevent these outbreaks, effective tools are needed that permit pathogen detection in a timely fashion under a variety of settings. Simple but still effective tests that are portable and cost-effective are greatly coveted, especially in regions that are susceptible to outbreaks but cannot afford expensive testing facilities.1-3 Although there exists a multitude of methods to detect bacteria, many of them are not suitable as screening or on-site testing tools because they require long test times, expensive instruments and complicated testing procedures.
Colorimetric tests are particularly attractive for point-of-care or field applications as color changes can be easily detected by the naked eye. The litmus test for pH is simple, quick, and effective. Although it is a very old technology, it is still widely used today because of its simplicity and effectiveness. Surprisingly, this simple test had never been modified to achieve the detection of other analytes before we recently developed an approach of modifying this test for E. coli testing.4
The expanded litmus test for E. coli employs three additional components: an E. coli activated RNA-cleaving DNAzyme (EC1), 5 urease, and magnetic beads. DNAzymes refer to synthetic single-stranded DNA molecules with catalytic activity.6 They can be isolated from random-sequence DNA pools using in vitro selection.7,8 They are highly stable and can be produced cost-effectively using high-efficiency automated DNA synthesis.9 For these reasons, DNAzymes, particularly RNA-cleaving DNAzymes, have been widely examined for biosensing applications.6,10,11 RNA-cleaving DNAzyme sensors have been developed to detect metal ions,12-16 small molecules,17,18 bacterial pathogens5,19-21 and cancer cells.22 Given the great availability of target-induced RNA-cleaving DNAzymes, any assay that utilizes a DNAzyme can be potentially expanded to detect a diverse range of analytes.
Urease is chosen for its ability to hydrolyze urea into ammonia,23,24 resulting in a pH increase. Urease is also highly efficient, stable and amenable for conjugation to other biomolecules. Therefore, we postulated that a conjugate of an RNA-cleavage DNAzyme with urease would allow the use of litmus test for the detection of other targets.5
The action of the RNA-cleaving DNAzyme is relayed to urease-mediated increase of pH through the use of magnetic beads that are immobilized with the DNAzyme-urease conjugate. Because the activity of the DNAzyme under investigation is strictly dependent on E. coli, the presence of this bacterium in the test solution will result in the release of urease from the magnetic beads to the solution, which is then taken and used to hydrolyze urea in a reporter solution that contains a pH-sensitive dye. The final outcome of this procedure is a color change that can be conveniently reported by the dye or pH paper.
Protocol
1. הכנת ריאגנטים חוצצים
- 0.5 M Ethylenediaminetetraacetic חומצה (EDTA)
- בכוס 2 L, להוסיף EDTA 186.1 גרם ל -800 מ"ל מים מזוקקים deionized (DDH 2 O). התאם את ה- pH של התמיסה ל 8.0 באמצעות כדורי NaOH. הוסף DDH 2 O לנפח סופי של 1.0 L ולהעביר הפתרון בקבוק זכוכית autoclavable עבור מעוקר ולאחסן ב 4 מעלות צלזיוס.
- 10 × טריס- borate EDTA (10x TBE)
- בכוס פלסטיק 4 L, להוסיף 432 גרם טריס בסיס, 200 גרם חומצת בור, 80 מ"ל של 0.5 EDTA M (pH 8.0) ו DDH 2 O לנפח סופי של 4 מיקס ל באמצעות בר ומערבבים עד שכל הרכיבים הם מומסים. מעבירים את הפתרון בקבוקי זכוכית 1 ליטר עבור מעוקר ולאחסן ב 4 מעלות צלזיוס.
- 10% denaturing polyacrylamide במלאי
- בכוס פלסטיק 4 L, להוסיף 1681.7 אוריאה גרם, 400 מ"ל 10x TBE, 1 ליטר של 40% acrylamide / bisacrylamide (29: 1) פתרון DDH 2 O עד fנפח Inal הוא 4 ל מערבב בעזרת בר ומערבב עד האוריאה נמסה. מעבירים את הפתרון עד 1 L בקבוקי זכוכית ענבר ולאחסן ב 4 ° C..
- ג'ל 2x טעינת חוצץ (2x GLB)
- בתוך כוס זכוכית 200 מ"ל, להוסיף 44 גרם אוריאה, 8 סוכרוז גרם, 10 מ"ג bromophenol כחול, 10 מ"ג FF קסילן cyanol, 400 μl של סולפט dodecyl נתרן 10%, ו -4 מ"ל של 10 × TBE. הוסף DDH 2 O לנפח סופי של 40 מיליליטר ו לפזר את הרכיבים עם חימום קל על 50 מעלות צלזיוס תוך ערבוב עם בר ומערבבים מגנטי. העברת 1 מ"ל aliquot 1.5 מ"ל microfuge צינורות ולאחסן ב 4 ° C..
הערה: שימוש לפני דורשת חימום קצר על 90 מעלות צלזיוס כדי redissolve כל מוצקים.
- בתוך כוס זכוכית 200 מ"ל, להוסיף 44 גרם אוריאה, 8 סוכרוז גרם, 10 מ"ג bromophenol כחול, 10 מ"ג FF קסילן cyanol, 400 μl של סולפט dodecyl נתרן 10%, ו -4 מ"ל של 10 × TBE. הוסף DDH 2 O לנפח סופי של 40 מיליליטר ו לפזר את הרכיבים עם חימום קל על 50 מעלות צלזיוס תוך ערבוב עם בר ומערבבים מגנטי. העברת 1 מ"ל aliquot 1.5 מ"ל microfuge צינורות ולאחסן ב 4 ° C..
- 1 M טריס-הידרוכלוריד (Tris-HCl) (7.5 pH)
- בתוך בקבוק זכוכית, להוסיף 12.1 גרם של טריס-בסיס ו -70 מ"ל של DDH 2 O ומערבבים עד להמסה מוצק. התאם את ה- pH ל -7.5 באמצעות 1 חומצה הידרוכלורית M (HCl). הוסף DDH 2 O לנפח סופי של 100 מ"ל, החיטוי STOמחדש ב 4 ° C
- 5 כלוריד נתרן M (NaCl)
- בתוך בקבוק זכוכית, לפזר 58.4 גרם של NaCl ב -150 מ"ל של DDH 2 O. כוון את עוצמת הקול כדי 200 מ"ל עם DDH 2 O. חנות ב 4 מעלות צלזיוס.
- DNA הצפת elution
- בתוך בקבוק זכוכית, לערבב 2.0 מ"ל של 1 M טריס- HCl (pH 7.5), 8.0 מ"ל של 5 M NaCl ו 0.4 מ"ל של 0.5 מ 'EDTA (pH 8.0). כוון את עוצמת הקול כדי 200 מ"ל עם DDH 2 O, החיטוי ולאחסן ב 4 מעלות צלזיוס.
- 1 M 4- (2-hydroxyethyl) חומצה -1-piperazineethanesulfonic (HEPES) (pH 7.4)
- בתוך בקבוק זכוכית, לפזר 2.38 גרם של HEPES ב -80 מ"ל של DDH 2 O. התאם את ה- pH ל -7.4 באמצעות 5 N NaOH ולהוסיף DDH 2 O לנפח סופי של 100 מ"ל.
- 1 מגנזיום כלוריד M (MgCl 2)
- בתוך בקבוק זכוכית, להוסיף 2.03 גר 'MgCl 2 -6H 2 O ולהביא את נפח 100 מ"ל עם DDH 2 O.
- מאגר תגובה (RB)
- בתוך צינור 50 מ"ל חרוטי, להוסיף 50 μl של 1 M HEPES (pH 7.4), 1.5 מ"ל של 5 M NaCl, 0.75 מ"ל של 1 M MgCl 2, ו -5 μl של Tween-20. הוסף DDH 2 O לנפח סופי של 50 מ"ל. מערבבים את הפתרון ולסנן לתוך צינור אחר חרוטי באמצעות מסנן מונחה מזרק (0.22 מיקרומטר) ולאחסן ב 4 ° C עד השימוש.
- חיץ מחייב (BB)
- בתוך צינור 50 מ"ל חרוטי, להוסיף 500 μl של 1 M טריס- HCl (pH 7.5), 8.8 גרם של NaCl, 50 μl של 1 M של MgCl 2, ו -5 μl של Tween 20. להוסיף DDH 2 O לנפח סופי של 50 מ"ל. מערבבים את הפתרון ולסנן לתוך צינור אחר חרוטי באמצעות מסנן מונחה מזרק (0.22 מיקרומטר) ולאחסן ב 4 ° C עד השימוש.
- פתרון המצע (SS)
- בתוך צינור 50 מ"ל חרוטי, להוסיף 5.8 גרם של NaCl, 3 מ"ל של 1 M 2 MgCl, 1.5 אוריאה ז, ו -40 מ"ל של DDH 2 O. התאם את ה- pH של התמיסה עד 5.0 באמצעות 10 מ"מ HCl. בגלל הפתרון אינו שנאגר, להתאים את ה- pH באמצעות HClבזהירות באמצעות התוספת של aliquots HCl הקטן. הוסף DDH 2 O לנפח סופי של 50 מ"ל.
- מערבבים את הפתרון ולסנן לתוך צינור אחר חרוטי באמצעות מסנן מונחה מזרק (0.22 מיקרומטר) ולאחסן ב 4 ° C עד השימוש.
- לוריא Bertani (LB) מרק
- בכוס, לפזר אבקת 20.0 גרם LB ב 1 ליטר של DDH 2 O. מעביר בקבוק זכוכית, חיטוי, ולאחסן ב 4 ° C..
- אגר 1.5% LB
- בבקבוק 250 מ"ל, להוסיף אגר 1.5 גרם ו 100 מ"ל של מרק LB. חיטוי ולאחסן ב 4 מעלות צלזיוס.
- צלחות אגר
- Redissolve אגר LB במיקרוגל ומצננים הפתרון ~ 50 מעלות צלזיוס. יוצקים את הפתרון אל צלחות פטרי, מה שהופך ~ 5 צלחות ולאפשר להם לחזק.
סינתזה 2. וטיהור של E. coli- תגובה DNAzyme EC1
- סינתזה של EC1 ידי תבנית מתווכת אנזימתי קשירה
- לטהר commercially מסונתז oligonucleotides BS1, DE1, ו- T1 (רצפים מופיע בלוח 1) על ידי 10% denaturing ג'ל אלקטרופורזה polyacrylamide (dPAGE) על פי פרוטוקולים סטנדרטיים.
- הכינו מלאי 100 מיקרומטר של BS1, DE1, ו- T1. חנות ב -20 ° C עד השימוש.
- בתוך צינור 1.5 מ"ל microfuge, להוסיף 38.5 μl של DDH 2 O, 5 μl של DE1, ו -5 μl של חיץ התגובה קינאז polynucleotide 10x T4 שמספק הספק אנזים (500 מ"מ טריס-HCl (pH 7.6), 100 מ"מ MgCl 2, 50 מ"מ dithiothreitol (DTT), 1.0 spermidine מ"מ).
- הוסף 1 μl של אדנוזין אדנוזין (ATP) (100 מ"מ). הוסף 5 יחידות של קינאז polynucleotide T4 (10 U / μl). מערבבים על ידי pipetting את תערובת התגובה בזהירות.
- דגירת התגובה על 37 מעלות צלזיוס למשך 30 דקות.
- להרוות את התגובה על ידי חימום עד 90 מעלות צלזיוס למשך 5 דקות.
- להוסיף 118 μl של DDH 2 O, 5 μl של BS1, ו -5 μl של T1. מחממים את התגובה עד 90 מעלות צלזיוס למשך 2 ק"מn ולאחר מכן להתקרר לטמפרטורת החדר במשך 10 דקות.
- הוסף 20 μl של 10 × חיץ התגובה האנזים T4 DNA שמספק לו הספק אנזים (400 מ"מ טריס-HCl (pH 7.8), 100 מ"מ MgCl 2, 100 מ"מ DTT, 5 מ"מ ATP). הוספת 10 יחידות של ה- DNA האנזים T4 (5 U / μl) ומערבבים בזהירות על ידי pipetting.
- דגירה בטמפרטורת החדר למשך 2 שעות.
- הוסף 20 μl של 3 M נתרן אצטט (NaOAc) (5.2 pH), 500 μl של אתנול 100% קר. מערבבים את הפתרון על ידי vortexing ומניחים את הצינורית במקפיא -20 מעלות צלזיוס למשך 30 דקות.
- צנטריפוגה microfuge ב -20,000 XG במשך 20 דקות ב 4 °. מוציאים בזהירות את supernatant ידי pipetting.
- שטוף את הכדור עם אתנול ו צנטריפוגות 70% קרים שוב ב -20,000 XG במשך 10 דקות ב 4 ° C. שוב להסיר את supernatant ומייבשים גלולה תחת ואקום במשך 10 דקות.
- Resuspend את ה- DNA עם 15 μl של DDH 2 O ולאחר מכן להוסיף 15 μl של 2x GLB.
- וורטקס במרץ וחום עד 90 מעלות צלזיוס במשך2 דק '. לטהר את הדנ"א באורך מלא על ידי 10% dPAGE כמתואר להלן.
- הגדרת 10% dPAGE
- נקיה שתי צלחות זכוכית (צלחת מלאה אחת מחורצים אחד), שני מפרידים עבים 0.75 מ"מ, מסרק 12 גם. נח צלחת זכוכית אחד על משטח שטוח עם שני מפרידים בכל צד ואת הצלחת מחורצים על העליונה. קליפ שני הלוחות יחד באמצעות ארבעה קליפים שמספק לו הספק.
- בכוס פלסטיק 150 מ"ל, יוצקים 40 מ"ל של 10% dPAGE, 40 μl של tetramethylethylenediamine (TEMED), ו -400 μl של persulfate אמוניום 10% (APS). מערבבים את המרכיבים ויוצקים הפתרון בין הלוחות לאט.
- הכנס את המסרק ולאחר מכן לאפשר את הג'ל לפלמר במשך 10 דקות. לאחר ג'ל הוא polymerized, לאט להסיר את המסרק ולשטוף את הבארות עם DDH 2 O.
- הר את הצלחות על מנגנון ג'ל אלקטרופורזה. השתמש בצלחת מתכת כדי לפזר חום שנוצר כדי למנוע התחממות יתר.
- יוצקים TBE 1x על תאי העליון והתחתוןולהבטיח כי הבארות שקועות היטב למאגר. לשטוף את הבארות עם TBE 1x בעזרת פיפטה או מזרק.
- הגדר את המנגנון לרוץ ב 35 mA וברח מראש במשך 15 דקות לפני דגימות טעינה.
- Elution של ligated EC1 מ -10% dPAGE
- בעקבות צעד 2.2.6, הפעל את הג'ל על 35 מילי-אמפר עד (כחול bromophenol) צבע התחתון מגיע לתחתית צלחת זכוכית. זה אמור לקחת בערך 1.5 שעות. כבה את הכוח להסיר את הצלחות מן המנגנון.
- הנח את הצלחות על כמה גיליונות של מגבות נייר ולהסיר את מפרידי בקפידה מתוך לוחות הזכוכית. מוציא בזהירות את צלחת הזכוכית העליונה ועוטף את הג'ל בניילון הנצמד.
- תהפכו את הג'ל על להסיר את צלחת הכוס השנייה ומכסים בניילון נצמד שוב. תשמרי על עצמך כדי למנוע קמטים של בניילון נצמד.
- דמיינו את המוצר ligated באמצעות UV הצללה (260 ננומטר), אשר תייצר 4 להקות ה- DNA נפרדות (EC1 ligated מלא, DE1, BS1, ו- T1).
- בלו הלהקה העליונה (EC1) עם סכין גילוח סטרילית והעביר צינור 1.5 מיליליטר microfuge. למחוץ את הג'ל עם קצה פיפטה סטרילית (200 גודל עצה μl) עד שהוא הופך למשחה עדינה. להוסיף 550 μl של חיץ elution DNA, לכסות את הצינור עם רדיד אלומיניום על מנת להגן על fluorophore המוטבע, ולנער במשך 15 דקות.
- צנטריפוגה הפתרון ג'ל ב -20,000 XG במשך 5 דקות ובזהירות להעביר 400 μl של supernatant כדי צינור 1.5 מ"ל microfuge אחר. הימנע משיכת חתיכות ג'ל במהלך pipetting.
- אל הצינור microfuge עם supernatant הועבר להוסיף 40 μl של 3 M NaOAc (pH 5.2), ו 1.0 מ"ל של אתנול 100% קר. מערבבים את הפתרון על ידי vortexing ומניחים את הצינורית במקפיא -20 מעלות צלזיוס למשך 30 דקות.
- צנטריפוגה microfuge ב -20,000 XG במשך 20 דקות ב 4 °. מוציאים בזהירות את supernatant ידי pipetting.
- שטוף את הכדור עם אתנול ו צנטריפוגות 70% קרים שוב ב -20,000 XG במשך 10 דקות ב 4 ° C. שוב, להסיר אתsupernatant ומייבשים גלולה תחת ואקום במשך 10 דקות.
- קבע את הריכוז של EC1 על ידי מדידת ספיגת UV ב 260 ננומטר. הפוך מניה 10 מיקרומטר ולאחסן את המדגם ב -20 ° C עד השימוש.
הצמיד 3. באוראז DNA
- הכנת succinimidyl 4- (N-maleimidomethyl) cyclohexane-1-carboxylate (SMCC) במלאי
- ממיסים 1 SMCC מ"ג ב 676 μl של DMSO. וורטקס ומניחים על הקרח עד לשימוש.
- הכנת מאגר באוראז
- ממיסים באוראז 1 מ"ג 1 מ"ל של 1 × PBS (ללא Mg 2 + או Ca 2 +). מניחים על קרח עד לשימוש.
הערה: התגבש באוראז איטית להתמוסס ערבוב עדין נדרש על מנת להימנע denaturation.
- ממיסים באוראז 1 מ"ג 1 מ"ל של 1 × PBS (ללא Mg 2 + או Ca 2 +). מניחים על קרח עד לשימוש.
- סינתזה של באוראז-דנ"א (UrDNA)
- הוסף 10 μl של 100 מיקרומטר LD1 לצינור 2.5 מ"ל microfuge. להוסיף 140 μl של DDH 2 O, 40 μl של 10x PBS, ו מערבולת.
- להוסיף 80.5 μl של SMCC sטוק, 159.5 μl של DMSO, מערבולת, ו צנטריפוגות בקצרה באמצעות צנטריפוגות benchtop.
- לדגור על 37 מעלות צלזיוס למשך 60 דקות. למנוע התעבות תחת כובע microfuge.
- הוסף 200 μl של 1x PBS, 60 μl של 3 M NaOAc (pH 5.2), ו 1.5 מ"ל של אתנול 100% קר. מערבבים את הפתרון על ידי vortexing ו דגירה ב -20 מעלות צלזיוס במשך 30 דקות.
- צנטריפוגה הפתרון ב 20,000 XG במשך 20 דקות ב 4 °. הסר את supernatant ומייבשים גלולה תחת ואקום. הימנעו על-ייבוש.
- כדי גלולה יבשים להוסיף 400 μl של המניות באוראז דגירה בטמפרטורת החדר במשך 5 שעות.
- העבר 200 μl של המצומד גולמי טור מסנן צנטריפוגלי מראש שטף 100k MWCO. צנטריפוגה העמודה 14,000 XG במשך 5 דקות.
- מעבירים את המצומד 200 μl של גולמי הנותרים לעמודה צנטריפוגות ב 14,000 XG במשך 5 דקות. הסר את העמודה ולמקם אותו במהופך בתוך שפופרת 2.0 מ"ל microfuge חדש ( "צינור איסוף").
- CentrifUge צינור האיסוף (עם הטור ההפוך) XG ב 1000 למשך 2 דקות. הסר את צינור איסוף ולהוסיף 30 μl של 1x PBS לעמודה צנטריפוגלי לשטוף את הממברנה להתאוששות נוספת של conjugates.
- שוב להפוך את העמודה ולמקם בחזרה לתוך צינור האיסוף. צנטריפוגה צינור האיסוף (עם הטור ההפוך) XG ב 1000 למשך 2 דקות. סר ולהיפטר הטור.
- אחסן את UrDNA ב 4 ° C עד השימוש.
4. האסיפה של EC1 ו UrDNA על חרוזים מגנטיים
- מערבבים את חרוז מגנטי (MB) המניה היטב ולהעביר 100 μl של השעיה MB לצינור 1.5 מ"ל microfuge. מניחים את הצינור microfuge על בעל מתלה מגנטי לבידוד MB.
- הסר את supernatant ידי pipetting ולהוסיף 150 μl של חיץ מחייב (BB) אל הצינור. הסר את הצינור מבעל ובזהירות לחץ על הצינור כדי resuspend MB לפתרון הומוגנית. הימנע מתיז ההשעיה אל tאופ של הצינור או כובע. אם זה יקר, השתמש בצנטריפוגה benchtop לסובב את השאריות בקצרה חזרה ההשעיה.
- חזור על שלב 4.2 פעמים נוספות.
- כדי השעיה זו להוסיף 10 μl של 10 מיקרומטר EC1. בזהירות ומערבבים ידי הקשה על הצינור.
- דגירה הפתרון עם רעד עדין למשך 30 דקות. לחץ על הצינור כל 2-3 דקות כדי למנוע את המשקעים של MB.
- מניחים את הצינור microfuge חזרה על השבכה מגנטי לבודד את MB ולהסיר את supernatant ידי pipetting. שטפו את MB שלוש פעמים עם 150 μl של BB (כמתואר בשלב 4.2).
- לאחר הכביסה תושלם, להשעות את MB ב סך של 150 μl של BB. לפתרון זה, להוסיף 15 μl של UrDNA ומחמם פתרון 45 מעלות צלזיוס למשך 2 דקות. מצננים את הפתרון בטמפרטורת החדר דגירה במשך שעה 2.
- מניחים את הצינור microfuge חזרה על השבכה מגנטי לבודד את MB ולהסיר את supernatant ידי pipetting.
- הוספת 100 μl של חיץ התגובה (RB). הסר tהוא microfuge שפופרת מהמדף מגנטי ובזהירות resuspend את MB.
- לשטוף שלוש פעמים MB יותר על ידי חזרה על שלב 4.9.
הערה: supernatant השטף ניתן לבדוק במהירות כדי לקבוע אם unhybridized UrDNA עדיין קיים, דבר אשר עלול לגרום איתות חיובית כוזבת. הבדיקה יכולה להיעשות על ידי הוספת 10 μl של 50 אוריאה מ"מ ו 10 μl של 0.04% פנול אדום לפתרון לשטוף. המשך לשטוף את MB עד הפתרון לשטוף אינו גורם שינוי צבע. ההשעיה 100 μl מאוחסן על 4 מעלות צלזיוס עד לשימוש.
5. הכנת תאים חיידקיים 20
- Culturing E. coli מ מניות
- פלייט E. coli K12 (MG1655) על צלחות אגר LB ממלאי גליצרול תחת להבה או בתוך ארון בטיחות ביולוגית.
- בעזרת קצה פיפטה סטרילית, לגעת בעדינות את המניות גליצרול בקלילות פס פני השטח של צלחת אגר כדי למנוע ניקוב אגר LB.
- הפוך את המפוספסותצלחת לדגור על 37 מעלות צלזיוס למשך 14 שעות.
- חותם את הצלחת עם Parafilm ולאחסן ב 4 מעלות צלזיוס למשך תקופה מקסימלית של 3 שבועות.
- Culturing E. coli עבור ספירת תאים
- בתוך צינור תרבות סטרילי 14 מ"ל, לוותר 2 מ"ל של מרק LB.
- בעזרת קצה פיפטה סטרילית, לקחת קולוניה אחת מהצלחת אגרה מפוספס המוכן בשלב 5.1 ולהעביר אותו אל צינור התרבות.
- דגירת התרבות ב 37 מעלות צלזיוס ולנער ב 230 סל"ד במשך 14 שעות.
- סדרתי לדלל את תרבית החיידקים במרווחים של פי 10.
- עבור כל דגימה בדילול מלא, צלחת שווה חמישה 100 aliquots μl על צלחות אגר נפרד LB. הפוך את הצלחות לדגור על 37 מעלות צלזיוס למשך 14 שעות.
- ספירת התאים של כל דגימה כדי לקבל את ריכוז התאים הממוצע של כל דילול.
הערה: 10 7 תאים משמשים לעתים קרובות כדי להגדיר מבחן לקמוס התייחסות E. coli כמו זה ברמה של E. coli יכול לעורר צבע מהיר צ'אןGE. עם זאת, מבחן לקמוס מבוצע כהלכה יכול לזהות נמוך כמו 500 תאים, כפי שפורט בסעיף התוצאות.
- הכנת E. תאי coli לבחינות
- במשך השעית תא רצויה, להעביר 1 מיליליטר של מנייה תרבותית לצינור 1.5 מיליליטר microfuge.
- צנטריפוגה התאים ב 6000 XG במשך 10 דקות ב 4 °. מוציאים בזהירות את supernatant מבלי להפריע גלולה התא.
- הוסף 10 μl של חיץ התגובה התא גלולה ו resuspend התאים. Sonicate השעית התא למשך 5 דקות. מעביר את השעית התא אל תיבת קרח למשך 5 דקות.
- Sonicate השעית התא למשך 5 דקות אחרות.
- צנטריפוגה ההשעיה תא 13,000 XG במשך 10 דקות ב 4 °. השתמש supernatant לבדיקה (10 μl).
6. מבחן לקמוס
- בתוך צינור 1.5 מ"ל microfuge, כביסה מוקדמת הצינור על ידי הוספה vortexing 100 μl של חיץ התגובה (RB) בצינור microfuge והשלכת למאגר.
- העברת 15 μl של EC1 התאספו (פרוטוקול 4) אל הצינור microfuge שטף.
- שטפו את החרוזים מגנטי על ידי הנחת צינור microfuge על מדף מגנטי. הסר את supernatant ידי pipetting. הסר את צינור microfuge מהמדף, להוסיף 100 μl של RB, ובזהירות resuspend החרוזים המגנטיים.
- שטוף את פעמי MB עוד שני על ידי חזרה על שלב 6.3.
- מניח את צינור microfuge חזרה על השבכה המגנטית, להסיר את supernatant ולהוסיף 10 μl E. מדגם coli מוכן משלב 5.3.
- מערבבים את החרוזים מדגם ומגנטיות בקפידה על ידי הקשה בעדינות על הצינור microfuge.
- דגירה התגובה בטמפרטורת החדר למשך שעה 1.
- כדי התגובה, להוסיף 90 μl של DDH 2 O ובמקום צינור microfuge על מתלה מגנטית.
- לאחר כ -3 דקות של הפרדה מגנטית, להעביר בזהירות 85 μl של supernatant לצינור 0.5 מ"ל microfuge. למשוך את supernatant לאטלמנוע איסוף כל חרוזים מגנטיים.
- אל הצינור microfuge מעל להוסיף 15 μl של פנול אדום 0.04%, ו -100 μl של פתרון המצע.
- לצלם בזמנים קצובים להקליט שינוי צבע.
הערה: השינוי ב- pH יכול גם להיות במעקב באמצעות מד pH עם microelectrode. ה- pH מתחיל צריך להיות כ 5.2-5.5 (הפתרון הוא צהוב). אם לא, הפתרון יכול להיות מותאם על ידי תוספת של 1 מ"מ Acetate הצפת pH 5.0).
תוצאות
העיקרון של הנייר לקמוס חיידקי מוסבר באיור 1 המבחן משתמש בשלושה חומרי מפתח:. DNAzyme ביקוע-RNA כי הוא מופעל על ידי חיידק ספציפי, באוראז וחרוזים מגנטיים. DNAzyme משמש כאלמנט הכרה המולקולרי להשיג זיהוי ספציפי מאוד של חיידק עניין. באוראז וחרוזי מגנטים משמשים להשגה הולכים אותות של פעילות RNA-המחשוף של DNAzyme. זה כרוך ביצירת חרוזים מגנטיים המכילים conjugates באוראז-DNAzyme. בנוכחות של חיידק היעד, הצמדת RNA שלו ודבק DNAzyme. פעולה זו מעלה את הניתוק של באוראז מן החרוזים מגנטיים. באוראז שוחרר ניתן להפריד בקלות מן חרוזים מגנטיים רגילים לחולל שינוי צבע בתמיסה הכתב, המכיל אוריאה לצבוע pH רגיש. באוראז hydrolyzes אוריאה לתוך אמוניה, בליווי עלייה של pH שמפעיל את גשינוי olor של הצבע.
איור 2 מציג מבחן לקמוס חיידקי שם EC1, א' coli -responsive RNA-ביקוע DNAzyme, שימש DNAzyme, ו פנול אדום שימש לצבוע דיווח-pH. EC1 היה מבודד בעבר על ידי הקבוצה שלנו מתוך מאגר DNA אקראי רצף בטכניקה של בחירה במבחנה. 5 המחקרים הקודמים שלנו הראו כי EC1 הוא מאוד ספציפי עבור E. coli ותערוכות פעילות מינימלית כלפי חיידקים אחרים. 5,19 כבר נמצא כי EC1 מופעל על ידי מולקולת חלבון מן E. coli. למרות הזהות של סמן ביולוגי חלבון זה לא פוענחה, את הספציפיות הכרה הגבוהות עולה כי חלבון זה הוא ייחודי E. coli. פתרון הכתב מוגדר להיות בעל pH ראשונית של 5.5. ב- pH זה, פנול אדום מפגין צבע צהוב. כפי באוראז hydrolyzes אוריאה לתוך אמוניה, הבסיסית של סול הכתבעליות ution. הדבר בא לידי ביטוי על ידי השינוי ההדרגתי של צבע מצהוב ורוד. עומק השינוי בצבע תלוי בשני הפרמטרים הבאים, כפי שמודגם על ידי איור 2: מספר E. קולי תאים המשמשים שלב ההפעלה DNAzyme ואת הזמן המוקצב לשלב הידרוליזה אוריאה. עוד E. קולי תאי ביאה לשינויי צבע חזקים, המשתקפים התצפית של מעבר צבע צהוב-אל-ורוד מתקדם כאשר E. קולי תאים הוגדלו באופן סדרתי מן 5 5 x 10 7 (פי 10 להגדיל בכל פעם). בינתיים, זמן רב יותר עבור הידרוליזה אוריאה מותר לצורך זיהוי של מספרים קטנים של E. קולי תאים (5,000 תאים התגובה 1 שעה ו 500 התאים התגובה 2 שעות).
השינוי pH של נייר לקמוס חיידקיים יכולים גם להיות במעקב באמצעות מד pH כף יד ותוצאות נציג הם באיור 3. WA זההים נמצא כי הנוכחות של 10 7 א קולי תאים הביאו לעלייה הדרגתית של pH על ידי 3 יחידות בתוך 10 דקות. לעומת זאת, בהיעדר E. קולי תאים לא לגרום לשינויים pH לזיהוי תחת אותה הגדרה.
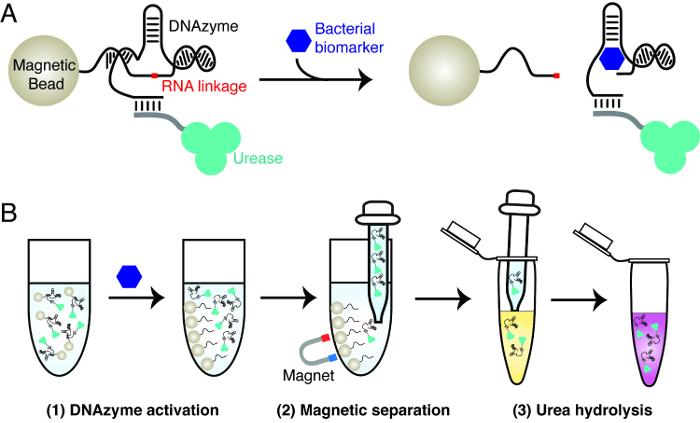
איור 1: עיקרון העיצוב של נייר לקמוס בקטריאלי (א) הפעלה של DNAzyme ביקוע-RNA על ידי סמן ביולוגי ספציפי מחיידק של עניין.. בנוכחות של הסמן הביולוגי, את DNAzyme ביקוע-RNA משותק על מסלקת חרוזים מגנטית הצמדת RNA, וגורם לשחרור של באוראז מתויג מן החרוזים מגנטיים לפתרון. (ב) הליך assay שלושה שלבים. שלב 1: הפעלת DNAzyme, כמתואר בלוח א שלב 2: הפרדה מגנטי - באוראז שוחרר מופרד חרוזים מגנטיים. שלב 3: R הידרוליזה אוריאה12; באוראז שוחרר מתווסף לתוך פתרון כתב המכיל אוריאה. באוראז hydrolyzes אוריאה לתוך אמוניה, וכתוצאה מכך שינוי pH שניתן יהיה לדווח על ידי לצבוע pH רגיש. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.

איור 2: נייר לקמוס עם E. coli באמצעות E. EC1 DNAzyme -responsive coli. נציג תוצאות צבע משתנה עם מספרים שונים של E. קולי תאים בתנאי מעל כל מבחנה. פנול אדום שימש לצבוע pH רגיש. מבחן ללא E. coli שימש כביקורת שלילית. עוד E. קולי תאים צפויים לגרום לשחרור מולקולות urease יותר, מלווה בy שינויי צבע חזקים. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
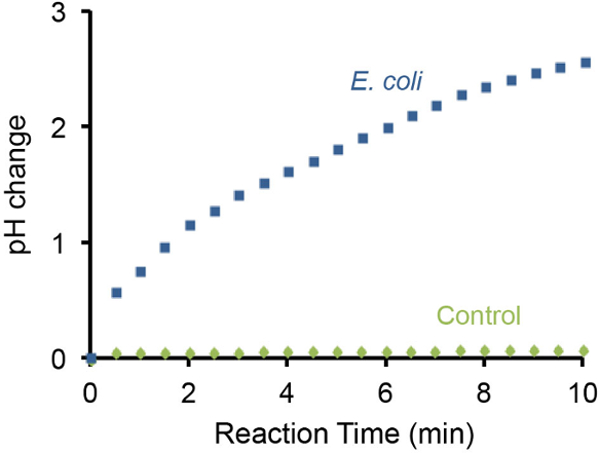
איור 3: ניטור גידול pH באמצעות מד pH השינוי של pH נגרם על ידי 10 7 א. קולי תאים היה פיקוח באמצעות מד pH נייד. מבחן ללא E. coli שימש כביקורת שלילית. הנוכחות של 10 7 א coli תאים בתמיסת הבדיקה יכולים להגדיל את בסיסיות על ידי ~ 3 יחידות pH ב 10 דקות. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
| שֵׁם | Sequence (5'-3 ") | הערה |
| BS1 | BTTTT TTTTT TTTAC TCTTC CTAGC FRQGG TTCGA TCAAG | B: 5'-ביוטין; R: ribonucleotide אדנין; F: והעמסה-DT; ש: dabcyl-DT |
| DE1 | GATGT GCGTT GTCGA GACCT GCGAC CGGAA CACTA CACTG TGTGG GGATG GATTT CTTTA CAGTT GTGTG TTGAA CGCTG TGTCA AAAAA AAAA | |
| T1 | GACAA CGCAC ATCTC TTGAT CGAAC C | |
| LD1 | XTTTT TTTTT TTTTT TTGAC ACAGC GTTCA | X: 5'-NH 2 |
טבלה 1: רצפים של oligonucleotides הסינטטי.
Discussion
התרגום של הפעולה של פעילות מחשוף RNA של DNAzyme חיידק-מגיב מבחן לקמוס מתאפשר באמצעות השימוש באוראז ופרדה מגנטית, כפי שמודגם על ידי איור 1. למרות ההפגנה של נייר לקמוס השונה לגילוי חיידקים היא נעשה עם E. coli תלוי RNA-ביקוע DNAzyme, 5,19,20 העיצוב יכול להתארך באופן כללי לכל DNAzyme ביקוע-RNA. בהינתן הזמינות הרבה של DNAzymes ביקוע-RNA עבור analytes שונים ומתודולוגיות שונות לבודד DNAzymes חדש ביקוע-RNA מבריכות אקראית רצף עבור מטרות חדשות, אנו מצפים כי פלטפורמת מבחן לקמוס שונה יכול להתארך עד זיהוי של מטרות מגוונות של עניין .
מבחן לקמוס עבור E. זיהוי קולי יכול לזהות תאים 5,000 ל 500 כאשר זמן התגובה הדיווח מוגדר להיות hr 1 ו -2, בהתאמה. תגובת השרשרת של פולימראז פופולרי (PCR)כריך אנזים צמוד assay immunosorbent (ELISA) שיטות יכולות להשיג לתחום גילוי של כ 10 4 -10 5 א קולי תאים בזמני בדיקות דומים. 25,26 לפיכך, המבחן לקמוס חיידקים מציע רגישויות גילוי דומות.
למרות הבדיקה לקמוס חיידקים קלה לבצע והוא יכול לייצר שינויי צבעי חיים, מספר גורמים יכולים להשפיע על תוצאות בדיקה באופן משמעותי. ראשית, האיכות באוראז חשובה מאוד. השתמשנו באוראז ממקורות שונים ומצאתי את תוצאות הבדיקה יכולה להשתנות באופן משמעותי. אנו ממליצים על השימוש באוראז מהמקור מפורט בסעיף החומרים.
ההרכבה של DNAzyme / באוראז / חרוזים מגנטיים צריכה תשומת לב מיוחדת. שטיפה יסודית של חרוזים מגנטיים להסיר UrDNA unhybridized נחוצה כדי למנוע תוצאות חיוביות שגויות. טיפול גם צריך לקחת כדי למנוע הצטברות של חרוזים מגנטיים שיורית על פני השטח הפנימי של lid של הצינור microfuge, אשר עשוי להיות קשה לראות. ברגע שיש, החרוזים המגנטיים לא חשופים יותר זמן הפרדה מגנטית וכך, יכולים לשאת כמה UrDNA unhybridized שיכולים להוביל אותות חיוביים שגויים תגובת הכתב. כמו כן, חשוב להימנע מיציאת צינור microfuge על המדף המגנטי למשך זמן ארוך יותר מ -10 דקות במהלך שלב הפרדה המגנטי. החרוזים יכולים לצבור או מקל על צינור microfuge, אשר עשוי להפחית את יעילות הכביסה להציג תצווה ל-תצווה עקביות. הכללה של 0.01% Tween-20 בפתרון הכביסה יכולה לשפר את האחידות תצווה ל-תצווה והיא צריכה להיות מיושמת.
חרוזים מגנטיים הם מצופים streptavidin, אשר שימש כעוגן להרכיב conjugates DNAzyme-באוראז על חרוזים מגנטיים. שניהם streptavidin ו באוראז הם מולקולות חלבון שניתן מפוגל במהלך האחסון. אנחנו בדרך כלל לאחסן את החרוזים DNAzyme-באוראז-מגנטי התאספו בבית 4 ° C עד 4 שבועותולעשות קבוצות טריות באופן קבוע כדי להשיג תוצאות עקביות יותר.
טיפול גם צריך לנקוט כדי למנוע לקיחת חרוזים מגנטיים בטעות בשלב הפרדה מגנטי (שלב 6.9) לאחר הפעלת DNAzyme. מניסיוננו, פסולת הסלולר וחלקיקים אחרים הפתרון יכולים להפחית את יעילות הפרדה המגנטית, ולכן, כמה חרוזים מגנטיים ניתן בכוונה להוציא במהלך pipetting. זה יצמצם את תוצאות חיוביות שגויות. אנו ממליצים על הצעדים הבאים כדי להקל על הבעיה: זמן הפרדה יותר (כגון 5-10 דקות), שחרור איטי של לחץ על פיפטה לאפשר נסיגה עדינה של supernatant, והכפיף את supernatant סבב נוסף של פרדה מגנטית .
לבסוף, חשוב כדי למנוע זיהום תאונה של פתרון כתב המניות על ידי באוראז במהלך ניסוי שבו דגימות מרובות נבדקות. בהתחשב תגובתיות גבוהה של urease, זיהום מסוג זה יכול להוביל לתוצאות חיוביות שגויות.
Disclosures
The authors have nothing to disclose.
Acknowledgements
The funding for this research project was provided by the Natural Sciences and Engineering Research Council of Canada (NSERC) via a Discovery Grant to YL.
Materials
| Name | Company | Catalog Number | Comments |
| Ethylenediaminetetraacetic acid (EDTA) | VWR AMRESCO | 0105 | |
| Sodium Hydroxide (NaOH) pellets | BIO BASIC CANADA INC. | SB6789 | |
| Tris-base | VWR AMRESCO | 0497 | |
| Boric acid | AMRESCO | 0588 | |
| Urea | VWR AMRESCO | M123 | |
| 40% acrylamide/bisacrylamide (29:1) solution | BIO BASIC CANADA INC. | A0007 | |
| Sucrose | Bioshop Canada inc. | SUC507 | |
| Bromophenol blue | Bioshop Canada inc. | BRO777 | |
| Xylenecyanol FF | SIGMA-ALDRICH | X-4126 | |
| 10% sodium dodecyl sulfate | Bioshop Canada inc. | SDS001 | |
| Hydrochloric Acid (HCl) | CALEDON LABORATORIES LTD | 6026 | |
| Sodium Chloride (NaCl) | Bioshop Canada inc. | SOD001 | |
| 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid (HEPES) | Bioshop Canada inc. | HEP001 | |
| Magnesium Chloride (II) hexahydrate | VWR AMRESCO | 0288 | |
| Tween 20 | Bioshop Canada inc. | TW508 | |
| Adenosine Triphospahte (ATP) | AMRESCO | 0220 | |
| Sodium Acetate trihydrate (NaOAc) | SIGMA-ALDRICH | S8625 | |
| Ethanol | Commercial Alcohols | P016EAAN | |
| Tetramethyleneethylenediamine (TEMED) | AMRESCO | 0761 | |
| 10% Ammonium persulfate (APS) | BIO BASIC CANADA INC. | AB0072 | |
| Succinimidyl 4-(N-maleimidomethyl) cyclohexane-1-carboxylate (SMCC) | ThermoFisher SCIENTIFIC | 22360 | |
| Dimethyl sulfoxide (DMSO) | CALEDON LABORATORIES | 803540 | |
| Urease | SIGMA-ALDRICH | U0251 | |
| 1x Phosphate Buffered Saline (PBS) | ThermoFisher SCIENTIFIC | 70011-069 | |
| 0.04% Phenol red | SIGMA-ALDRICH | P3532 | |
| 10x T4 polynucleotide kinase reaction buffer | Lucigen | 30061-1 | |
| 10x T4 DNA ligase reaction buffer | Bio Basics Canada | B1122-B | |
| T4 DNA ligase (5 U/μl) | Thermo Fischer Scientific | B1122 | |
| Luria Bertani (LB) Broth | AMRESCO | J106 | |
| Agar | AMRESCO | J637 | |
| T4 polynucleotide kinase (10 U/μl) | Lucigen | 30061-1 | |
| E. coli K12 (MG1655) | ATCC | ATCC700926 | |
| Centrifuge | Beckman Coulter, Inc. | 392187 | |
| Glass plates | CBS scientific | ngp-250nr | |
| 0.75 mm thick spacers | CBS scientific | VGS-0725r | |
| 12-well comb | CBS scientific | VGC-7512 | |
| UV Lamp | UVP | 95-0017-09 | |
| Spectrophotometer (NanoVue) | GE Healthcare | N/A | |
| Metal plate | CBS scientific | CPA165-250 | |
| Vortex | VWR International | 58816-123 | |
| Gel electrophoresis apparatus | CBS scientific | ASG-250 | |
| Petri dishes | VWR International | 25384-342 | |
| 100 kDa MWCO centrifugal filters | EMD Millipore | UFC510024 | |
| Magnetic Bead (BioMag) | Bangs Laboratories Inc | BM568 | |
| Magnetic Seperation Rack | New England BioLabs | S1506S | |
| Microfuge tubes | Sarstedt | 72.69 | |
| Syringe filter (0.22 μm) | VWR International | 28145-501 | |
| 14 ml culture tube | VWR International | 60818-725 | |
| Cell culture incubator | Eppendorf Scientific | M13520000 | |
| Branson Ultrasonic cleaner | Branson | N/A | |
| Camera (Canon Powershot G11) | Canon | N/A | |
| 50 ml conical tube | VWR International | 89004-364 |
References
- Daar, A. S., et al. Top ten biotechnologies for improving health in developing countries. Nat. Genet. 32, 229-232 (2002).
- Newman, J. D., Turner, A. P. Home blood glucose biosensors: a commercial perspective. Biosens. Bioelectron. 20, 2435-2453 (2005).
- Turner, A. P. Biosensors: sense and sensibility. Chem. Soc. Rev. 42, 3184-3196 (2013).
- Tram, K., Kanda, P., Salena, B. J., Huan, S. Y., Li, Y. F. Translating Bacterial Detection by DNAzymes into a Litmus Test. Angew. Chem. Int. Ed. 53, 12799-12802 (2014).
- Ali, M. M., Aguirre, S. D., Lazim, H., Li, Y. Fluorogenic DNAzyme probes as bacterial indicators. Angew. Chem. Int. Ed. 50, 3751-3754 (2011).
- Schlosser, K., Li, Y. Biologically inspired synthetic enzymes made from DNA. Chem. Biol. 16, 311-322 (2009).
- Tuerk, C., Gold, L. Systematic evolution of ligand by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Breaker, R. R. Making catalytic DNAs. Science. 290, 2095-2096 (2000).
- Liu, J., Cao, Z., Lu, Y. Functional nucleic acid sensors. Chem. Rev. 109, 1948-1998 (2009).
- Navani, N. K., Li, Y. Nucleic acid aptamers and enzymes as sensors. Curr. Opin. Chem. Biol. 10, 272-281 (2006).
- Li, J., Lu, Y. A highly sensitive and selective catalytic DNA biosensor for lead ions. J. Am. Chem. Soc. 122, 10466-10467 (2000).
- Liu, J., Lu, Y. A colorimetric lead biosensor using DNAzyme-directed assembly of gold nanoparticles. J. Am. Chem. Soc. 125, 6642-6643 (2003).
- Liu, Z., Mei, S. H. J., Brennan, J. D., Li, Y. Assemblage of signaling DNA enzymes with intriguing metal specificity and pH dependence. J. Am. Chem. Soc. 125, 7539-7545 (2003).
- Liu, J., et al. A catalytic beacon sensor for uranium with parts-per-trillion sensitivity and millionfold selectivity. Proc. Natl. Acad. Sci. USA. 104, 2056-2061 (2007).
- Huang, P. J., Vazin, M., Liu, J. In vitro selection of a new lanthanide-dependent DNAzyme for ratiometric sensing lanthanides. Anal. Chem. 86, 9993-9999 (2014).
- Chiuman, W., Li, Y. Simple fluorescent sensors engineered with catalytic DNA 'MgZ' based on a non-classic allosteric design. PLoS One. 2, e1224 (2007).
- Xiang, Y., Lu, Y. Using personal glucose meters and functional DNA sensors to quantify a variety of analytical targets. Nat. Chem. 3, 697-703 (2011).
- Aguirre, S. D., Ali, M. M., Salena, B. J., Li, Y. A sensitive DNA enzyme-based fluorescent assay for bacterial detection. Biomolecules. 3, 563-577 (2013).
- Aguirre, S. D., Ali, M. M., Kanda, P., Li, Y. F. Detection of Bacteria Using Fluorogenic DNAzymes. J. Vis. Exp. (63), e3961 (2012).
- Shen, Z., et al. A catalytic DNA activated by a specific strain of bacterial pathogen. Angew. Chem. Int. Ed. 54, (2015).
- He, S., et al. Highly specific recognition of breast tumors by an RNA-cleaving fluorogenic DNAzyme probe. Anal. Chem. 87, 569-577 (2015).
- Sumner, J. B., Hand, D. B. Isoelectric point of crystalline urease. J. Am. Chem. Soc. 51, 1255-1260 (1929).
- Karplus, P. A., Pearson, M., Hausinger, R. P. 70 Years of crystalline urease: What have we learned. Acc. Chem. Res. 30, 330-337 (1997).
- Omiccioli, E., Amagliani, G., Brandi, G., Magnani, M. A new platform for Real-Time PCR detection of Salmonella spp., Listeria monocytogenes and Escherichia coli O157 in milk. Food Microbiol. 26, 615-622 (2009).
- Cui, S., Schroeder, C. M., Zhang, D. Y., Meng, J. Rapid sample preparation method for PCR-based detection of Escherichia coli O157:H7 in ground beef. J. Appl. Microbiol. 95, 129-134 (2003).
- Ibekwe, A. M., Watt, P. M., Grieve, C. M., Sharma, V. K., Lyons, S. R. Multiplex fluorogenic real-time PCR for detection and quantification of Escherichia coli O157:H7 in dairy wastewater wetlands. Appl. Environ. Microbiol. 68, 4853-4862 (2002).
- Strachan, N. J., Ogden, I. D. A sensitive microsphere coagulation ELISA for Escherichia coli O157:H7 using Russell's viper venom. FEMS Microbiol Lett. 186, 79-84 (2000).
- de Boer, E., Beumer, R. R. Methodology for detection and typing of foodborne microorganisms. Int. J. Food Microbiol. 50, 119-130 (1999).
- Gracias, K. S., McKillip, J. L. A review of conventional detection and enumeration methods for pathogenic bacteria in food. Can. J. Microbiol. 50, 883-890 (2004).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved