Method Article
Détection colorimétrique de bactéries à l'aide de test Litmus
Dans cet article
Résumé
We describe a protocol for colorimetric detection of E. coli using a modified litmus test that takes advantage of an RNA-cleaving DNAzyme, urease, and magnetic beads.
Résumé
There are increasing demands for simple but still effective methods that can be used to detect specific pathogens for point-of-care or field applications. Such methods need to be user-friendly and produce reliable results that can be easily interpreted by both specialists and non-professionals. The litmus test for pH is simple, quick, and effective as it reports the pH of a test sample via a simple color change. We have developed an approach to take advantage of the litmus test for bacterial detection. The method exploits a bacterium-specific RNA-cleaving DNAzyme to achieve two functions: recognizing a bacterium of interest and providing a mechanism to control the activity of urease. Through the use of magnetic beads immobilized with a DNAzyme-urease conjugate, the presence of bacteria in a test sample is relayed to the release of urease from beads to solution. The released urease is transferred to a test solution to hydrolyze urea into ammonia, resulting in an increase of pH that can be visualized using the classic litmus test.
Introduction
Bacterial pathogens are one of the major causes of global morbidity and mortality. Outbreaks from hospital-acquired infections, food-borne pathogens, and bacterial contaminants in the environment pose serious and on-going threats to public health and safety. To prevent these outbreaks, effective tools are needed that permit pathogen detection in a timely fashion under a variety of settings. Simple but still effective tests that are portable and cost-effective are greatly coveted, especially in regions that are susceptible to outbreaks but cannot afford expensive testing facilities.1-3 Although there exists a multitude of methods to detect bacteria, many of them are not suitable as screening or on-site testing tools because they require long test times, expensive instruments and complicated testing procedures.
Colorimetric tests are particularly attractive for point-of-care or field applications as color changes can be easily detected by the naked eye. The litmus test for pH is simple, quick, and effective. Although it is a very old technology, it is still widely used today because of its simplicity and effectiveness. Surprisingly, this simple test had never been modified to achieve the detection of other analytes before we recently developed an approach of modifying this test for E. coli testing.4
The expanded litmus test for E. coli employs three additional components: an E. coli activated RNA-cleaving DNAzyme (EC1), 5 urease, and magnetic beads. DNAzymes refer to synthetic single-stranded DNA molecules with catalytic activity.6 They can be isolated from random-sequence DNA pools using in vitro selection.7,8 They are highly stable and can be produced cost-effectively using high-efficiency automated DNA synthesis.9 For these reasons, DNAzymes, particularly RNA-cleaving DNAzymes, have been widely examined for biosensing applications.6,10,11 RNA-cleaving DNAzyme sensors have been developed to detect metal ions,12-16 small molecules,17,18 bacterial pathogens5,19-21 and cancer cells.22 Given the great availability of target-induced RNA-cleaving DNAzymes, any assay that utilizes a DNAzyme can be potentially expanded to detect a diverse range of analytes.
Urease is chosen for its ability to hydrolyze urea into ammonia,23,24 resulting in a pH increase. Urease is also highly efficient, stable and amenable for conjugation to other biomolecules. Therefore, we postulated that a conjugate of an RNA-cleavage DNAzyme with urease would allow the use of litmus test for the detection of other targets.5
The action of the RNA-cleaving DNAzyme is relayed to urease-mediated increase of pH through the use of magnetic beads that are immobilized with the DNAzyme-urease conjugate. Because the activity of the DNAzyme under investigation is strictly dependent on E. coli, the presence of this bacterium in the test solution will result in the release of urease from the magnetic beads to the solution, which is then taken and used to hydrolyze urea in a reporter solution that contains a pH-sensitive dye. The final outcome of this procedure is a color change that can be conveniently reported by the dye or pH paper.
Protocole
1. Préparation des réactifs et tampons
- 0,5 M Ethylenediaminetetraacetic acide (EDTA)
- Dans un bécher de 2 litres, ajouter 186,1 g d' EDTA à 800 ml d'eau déminéralisée distillée (ddH 2 O). Ajuster le pH de la solution à 8,0 en utilisant des pastilles de NaOH. Ajouter ddH 2 O à un volume final de 1,0 L et transférer la solution dans une bouteille en verre autoclavable pour autoclavage et conserver à 4 ° C.
- EDTA 10 x Tris-borate (TBE 10x)
- Dans un 4 L bêcher en plastique, ajouter 432 g de Tris-base, 200 g d' acide borique, 80 ml d'EDTA 0,5 M (pH 8,0) et ddH 2 O à un volume final de 4 L. Mélanger à l' aide d' une barre d'agitation jusqu'à ce que tous les composants sont dissous. Transférer la solution dans des bouteilles en verre 1 L pour autoclavage et conserver à 4 ° C.
- 10% polyacrylamide dénaturant Stock
- Dans un 4 L bêcher en plastique, ajouter 1681,7 g d' urée, 400 ml 10x TBE, 1 L de 40% d' acrylamide / bisacrylamide (29: 1) solution et ddH 2 O jusqu'à ce que le fle volume inal est de 4 L. Mélanger à l'aide d'une barre d'agitation jusqu'à ce que l'urée est dissoute. Transférer la solution à 1 L bouteilles de verre ambré et conserver à 4 ° C.
- 2x Gel Loading Buffer (2x GLB)
- Dans un bécher de 200 ml en verre, ajouter 44 g d'urée, 8 g de saccharose, 10 mg de bleu de bromophénol, 10 mg de xylène-cyanol FF, 400 ul de 10% de dodécylsulfate de sodium et 4 ml de 10 x TBE. Ajouter ddH 2 O à un volume final de 40 ml et dissoudre les composants avec un léger chauffage à 50 ° C tout en mélangeant avec un barreau d'agitation magnétique. Transfert aliquote de 1 ml à 1,5 ml microtubes et conserver à 4 ° C.
NOTE: L'usage antérieur nécessite un bref chauffage à 90 ° C pour redissoudre tout solide.
- Dans un bécher de 200 ml en verre, ajouter 44 g d'urée, 8 g de saccharose, 10 mg de bleu de bromophénol, 10 mg de xylène-cyanol FF, 400 ul de 10% de dodécylsulfate de sodium et 4 ml de 10 x TBE. Ajouter ddH 2 O à un volume final de 40 ml et dissoudre les composants avec un léger chauffage à 50 ° C tout en mélangeant avec un barreau d'agitation magnétique. Transfert aliquote de 1 ml à 1,5 ml microtubes et conserver à 4 ° C.
- 1 M de Tris-chlorhydrate (Tris-HCl) (pH 7,5)
- Dans une bouteille en verre, ajouter 12,1 g de Tris base , et 70 ml de ddH 2 O et mélanger jusqu'à ce que le solide soit dissous. Ajuster le pH à 7,5 en utilisant 1 M d'acide chlorhydrique (HCl). Ajouter ddH 2 O jusqu'à un volume final de 100 ml, autoclave et store à 4 ° C
- 5 M de chlorure de sodium (NaCl)
- Dans une bouteille en verre, dissoudre 58,4 g de NaCl dans 150 ml de ddH 2 O. Réglez le volume à 200 ml avec ddH 2 O. Conserver à 4 ° C.
- Tampon d'élution de l'ADN
- Dans un flacon en verre, mélanger 2,0 ml de 1 M de Tris-HCl (pH 7,5), 8,0 ml de NaCl 5 M et 0,4 ml d'EDTA 0,5 M (pH 8,0). Réglez le volume à 200 ml avec ddH 2 O, autoclave et conserver à 4 ° C.
- 1 M de 4- (2-hydroxyéthyl) -1-piperazineethanesulfonic (HEPES) (pH 7,4)
- Dans une bouteille en verre, dissoudre 2,38 g d'HEPES dans 80 ml de ddH 2 O. Ajuster le pH à 7,4 en utilisant du NaOH 5 N et O 2 ddH ajouter à un volume final de 100 ml.
- 1 M de chlorure de magnésium (MgCl 2)
- Dans une bouteille en verre, ajouter 2,03 g de MgCl 2 6H 2 O et amener le volume à 100 ml avec ddH 2 O.
- Reaction Buffer (RB)
- Dans un tube conique de 50 ml, ajouter 50 pl de 1 M de HEPES (pH 7,4), 1,5 ml de NaCl 5 M, 0,75 ml de 1 M de MgCl2, et 5 pi de Tween-20. Ajouter ddH 2 O à un volume final de 50 ml. Mélanger la solution et filtrer dans un autre tube conique en utilisant un filtre à seringue (0,22 um) et conserver à 4 ° C jusqu'à utilisation.
- Un tampon de liaison (BB)
- Dans un tube conique de 50 ml, ajouter 500 pi de 1 M de Tris-HCl (pH 7,5), 8,8 g de NaCl, 50 pi de 1 M de MgCl2, et 5 pi de Tween 20. Ajouter ddH 2 O à un volume final de 50 ml. Mélanger la solution et filtrer dans un autre tube conique en utilisant un filtre à seringue (0,22 um) et conserver à 4 ° C jusqu'à utilisation.
- Solution de substrat (SS)
- Dans un tube conique de 50 ml, ajouter 5,8 g de NaCl, 3 ml de 1 M de MgCl2, 1,5 g d' urée et 40 ml de ddH 2 O. Ajuster le pH de la solution à 5,0 en utilisant du HCl 10 mM. Parce que la solution ne soit pas tamponné, ajuster le pH en utilisant HClattentivement l'addition de petites portions de HCl. Ajouter ddH 2 O à un volume final de 50 ml.
- Mélanger la solution et filtrer dans un autre tube conique en utilisant un filtre à seringue (0,22 um) et conserver à 4 ° C jusqu'à utilisation.
- Luria Bertani (LB) Broth
- Dans un bécher, dissoudre 20,0 g LB poudre dans 1 L de ddH 2 O. Transférer dans un flacon en verre, autoclave, et conserver à 4 ° C.
- 1,5% de gélose LB
- Dans un ballon de 250 ml, on ajoute 1,5 g d'agar et 100 ml de bouillon LB. Autoclave et conserver à 4 ° C.
- Les plaques d'agar-agar
- Dissoudre la gélose LB dans un micro-ondes et refroidir la solution à ~ 50 ° C. Versez la solution dans des boîtes de Pétri, en faisant ~ 5 plaques et leur permettre de se solidifier.
2. Synthèse et Purification de E. coliformes sensibles DNAzyme EC1
- Synthèse de EC1 par modèle Mediated Enzymatic Ligation
- Purifier commdes oligonucléotides synthétisés ercially BS1, DE1 et T1 (séquences fournies dans le tableau 1) de 10% électrophorèse sur gel dénaturant de polyacrylamide (DPage) selon des protocoles standards.
- Préparer un 100 uM stock d'BS1, DE1 et T1. Conserver à -20 ° C jusqu'à utilisation.
- Dans un tube de 1,5 ml, ajouter 38,5 pi de ddH 2 O, 5 ul de DE1, et 5 ul de tampon de réaction polynucleotide kinase 10x T4 fournies par le fournisseur d'enzyme (500 mM de Tris-HCl (pH 7,6), 100 mM de MgCl2, dithiothréitol 50 mM (DTT), 1,0 mM de spermidine).
- Ajouter 1 pi de l'adénosine triphosphate (ATP) (100 mM). Ajouter 5 unités de polynucleotide kinase T4 (10 U / ul). Mélanger par pipetage avec précaution le mélange réactionnel.
- Laisser incuber la réaction à 37 ° C pendant 30 min.
- Stopper la réaction par chauffage à 90 ° C pendant 5 min.
- Ajouter 118 ul de ddH 2 O, 5 pi de BS1, et 5 pi de T1. Chauffer le mélange réactionnel à 90 ° C pendant 2 min et puis refroidir à la température ambiante pendant 10 min.
- Ajouter 20 ul de 10 x tampon de T4 ADN ligase réaction fourni par le fournisseur d'enzyme (400 mM de Tris-HCl (pH 7,8), 100 mM de MgCl2, 100 mM DTT, 5 mM d' ATP). Ajouter 10 unités de T4 ADN ligase (5 U / pl) et mélanger délicatement par pipetage.
- Incuber à température ambiante pendant 2 h.
- Ajouter 20 ul de 3 M d'acétate de sodium (NaOAc) (pH 5,2), 500 ul d'éthanol à 100% froid. Mélanger la solution par tourbillonnement et placer le tube dans un congélateur à -20 ° C pendant 30 min.
- Centrifuger le microcentrifugeuse à 20.000 xg pendant 20 min à 4 ° C. Retirez délicatement le surnageant par pipetage.
- Laver le culot avec froid à 70% d'éthanol et centrifuger à nouveau à 20.000 xg pendant 10 min à 4 ° C. Une fois que enlever à nouveau le surnageant et le culot sécher sous vide pendant 10 min.
- Resuspendre l'ADN avec 15 pi de ddH 2 O, puis ajouter 15 pl de 2x GLB.
- Vortexer vigoureusement et chauffer à 90 ° C pendant2 minutes. Purifier l'ADN pleine longueur de 10% DPage comme décrit ci-dessous.
- Configuration 10% DPage
- plaques Clean deux en verre (une plaque complète et une crantées), deux de 0,75 mm d'épaisseur, espaceurs et un peigne à 12 puits. Poser une plaque de verre sur une surface plane avec deux entretoises de chaque côté et la plaque crantée sur le dessus. Coupez les deux plaques ensemble en utilisant les quatre clips fournis par le fournisseur.
- Dans un bêcher en plastique de 150 ml, verser 40 ml de 10% DPage, 40 ul de tétraméthyléthylènediamine (TEMED), et 400 pi de 10% de persulfate d'ammonium (APS). Mélanger les composants et verser la solution entre les plaques lentement.
- Insérez le peigne, puis laisser le gel se polymériser pendant 10 min. Une fois que le gel est polymérisé, retirer lentement le peigne et rincer les puits avec ddH 2 O.
- Monter les plaques sur l'appareil d'électrophorèse sur gel. Utilisez une plaque métallique pour dissiper la chaleur générée pour éviter la surchauffe.
- Verser TBE 1x sur les chambres supérieure et inférieureet faire en sorte que les puits soient bien immergées dans le tampon. Rincer les puits avec 1x TBE à l'aide d'une pipette ou d'une seringue.
- Réglez l'appareil pour fonctionner à 35 mA et de pré-course pour 15 min avant le chargement des échantillons.
- Elution de ligaturé EC1 de 10% DPage
- Suite à l'étape 2.2.6, exécutez le gel à 35 mA jusqu'à ce que le colorant de fond (bleu de bromophénol) atteint le fond de la plaque de verre. Cela devrait prendre environ 1,5 heures. Coupez l'alimentation et retirer les plaques de l'appareil.
- Poser les plaques sur quelques feuilles de serviettes en papier et retirez soigneusement les espaceurs des plaques de verre. Retirez délicatement la plaque de verre supérieure et envelopper le gel avec une pellicule de plastique.
- Retournez le gel sur pour enlever la deuxième plaque de verre et couvrir d'une pellicule de plastique à nouveau. Prenez soin d'éviter les rides de la pellicule de plastique.
- Visualiser le produit ligaturé en utilisant l'observation UV (260 nm), qui produira 4 bandes d'ADN distinctes (EC1 entièrement ligaturé, DE1, BS1 et T1).
- Excise la bande supérieure (EC1) avec une lame de rasoir stérile et transférer dans un tube de 1,5 ml. Écraser le gel avec une pointe de pipette stérile (200 pi taille de la pointe) jusqu'à ce qu'il se transforme en une pâte fine. Ajouter 550 pi de tampon d'élution d'ADN, couvrir le tube de papier d'aluminium pour protéger le fluorophore incorporé, et agiter pendant 15 min.
- Centrifuger la solution de gel à 20 000 xg pendant 5 min et transférer soigneusement 400 ul du surnageant dans un autre tube de 1,5 ml. Évitez de retirer des morceaux de gel pendant le pipetage.
- Au microtube avec le surnageant a été transféré ajouter 40 ul de NaOAc 3 M (pH 5,2) et 1,0 ml d'éthanol à 100% froid. Mélanger la solution par tourbillonnement et placer le tube dans un congélateur à -20 ° C pendant 30 min.
- Centrifuger le microcentrifugeuse à 20.000 xg pendant 20 min à 4 ° C. Retirez délicatement le surnageant par pipetage.
- Laver le culot avec froid à 70% d'éthanol et centrifuger à nouveau à 20.000 xg pendant 10 min à 4 ° C. Encore une fois, retirez lasécher le surnageant et le culot sous vide pendant 10 min.
- Déterminer la concentration de EC1 en mesurant l'absorbance UV à 260 nm. Faire 10 uM stock et stocker l'échantillon à -20 ° C jusqu'à utilisation.
3. Conjugaison de uréase à l'ADN
- Préparation du succinimidyl 4- (N-maléimidométhyl) cyclohexane-1-carboxylate (SMCC) Stock
- Dissoudre 1 mg de SMCC dans 676 ul de DMSO. Vortex et placer sur la glace jusqu'à utilisation.
- Préparation de uréase Stock
- Dissoudre 1 mg uréase dans 1 ml de 1 x PBS (sans Mg2 + ou Ca2 +). Placer sur la glace jusqu'à utilisation.
NOTE: Confit uréase est lente à se dissoudre et mélange doux est nécessaire pour éviter la dénaturation.
- Dissoudre 1 mg uréase dans 1 ml de 1 x PBS (sans Mg2 + ou Ca2 +). Placer sur la glace jusqu'à utilisation.
- Synthèse de uréase-ADN (UrDNA)
- Ajouter 10 pi de 100 uM LD1 à un tube de 2,5 ml de microcentrifugation. Ajouter 140 ul de ddH 2 O, 40 pl de PBS 10x, et vortex.
- Ajouter 80,5 ul de SMCC stock, 159,5 pi de DMSO, vortex et centrifuger brièvement à l'aide d'une centrifugeuse de paillasse.
- Incuber à 37 ° C pendant 60 min. Eviter la condensation sous le chapeau de microcentrifugation.
- Ajouter 200 ul de 1 x PBS, 60 ul de NaOAc 3 M (pH 5,2) et 1,5 ml d'éthanol à 100% froid. Mélanger la solution par tourbillonnement et on incube à -20 ° C pendant 30 min.
- Centrifuger la solution à 20.000 xg pendant 20 min à 4 ° C. Retirer le surnageant et sécher le culot sous vide. Évitez de trop sécher.
- Pour le culot séché ajouter 400 pi de uréase stock et incuber à température ambiante pendant 5 heures.
- Transférer 200 ul de conjugué brut à une colonne pré-lavés filtre centrifuge 100k MWCO. Centrifuger la colonne à 14 000 xg pendant 5 min.
- Transférer les 200 ul restants du conjugué brut à la colonne et centrifuger à 14 000 g pendant 5 min. Retirer la colonne et placez-le à l'envers dans un nouveau tube de 2,0 ml de microcentrifugation ( "tube de collecte»).
- centrifuge le tube de collecte (à la colonne inversée) à 1000 g pendant 2 min. Retirer le tube de collecte et d'ajouter 30 pl de PBS 1x à la colonne centrifuge pour laver la membrane pour la récupération supplémentaire de conjugués.
- Encore une fois inverser la colonne et placer dans le tube de collecte. Centrifuger le tube de collecte (à la colonne inversée) à 1000 g pendant 2 min. Retirer et jeter la colonne.
- Stocker le UrDNA à 4 ° C jusqu'à utilisation.
4. Assemblée des EC1 et UrDNA sur Magnetic Beads
- Mélanger le stock bille magnétique (MB) bien et transférer 100 pl de suspension MB à un tube de 1,5 ml. Placer le tube de microcentrifugation sur un support en rack magnétique pour isoler le MB.
- Enlever le surnageant par pipetage et ajouter 150 ul de tampon (BB) de liaison au tube. Retirer le tube du support et frapper avec précaution le tube pour remettre le MB à une solution homogène. Évitez les éclaboussures de la suspension à la top du tube ou une casquette. Si cela se produit, utilisez une centrifugeuse de paillasse pour faire tourner brièvement le résidu de retour à la suspension.
- Répétez l'étape 4.2 deux fois plus.
- A cette suspension, ajouter 10 pi de 10 uM EC1. Mélanger soigneusement en tapant sur le tube.
- Incuber la solution sous agitation douce pendant 30 minutes. Appuyez sur le tube chaque 2-3 min pour éviter la précipitation de la MB.
- Placer le tube de microcentrifugation de retour sur le support magnétique pour isoler le MB et retirer le surnageant par pipetage. Laver le MB trois fois avec 150 pi de BB (comme décrit dans l'étape 4.2).
- Une fois que le lavage est terminé, suspendre le MB dans un total de 150 pi de BB. A cette solution, ajouter 15 ul de UrDNA et chauffer la solution à 45 ° C pendant 2 min. Refroidir la solution à température ambiante et laisser incuber pendant 2 h.
- Placer le tube de microcentrifugation de retour sur le support magnétique pour isoler le MB et retirer le surnageant par pipetage.
- Ajouter 100 ul de tampon de réaction (RB). Retirer til microcentrifugeuse tube du support magnétique et mélanger soigneusement le MB.
- Laver les MB trois fois par étape 4.9 répéter.
NOTE: Le surnageant lavé peut être testé rapidement pour déterminer si non hybridée UrDNA est toujours présente, ce qui peut entraîner un signal faux positif. Le test peut être effectué en ajoutant 10 pi de 50 mM d'urée et de 10 pi de 0,04% de rouge de phénol à la solution de lavage. Continuer à laver le MB jusqu'à ce que la solution de lavage ne provoque pas un changement de couleur. La suspension de 100 ul est stockée à 4 ° C jusqu'à utilisation.
5. Préparation des cellules bactériennes 20
- La culture E. coli provenant de stocks
- Plate E. coli K12 (MG1655) sur LB agar plaques à partir d' un stock de glycérol sous une flamme ou dans une enceinte de sécurité biologique.
- En utilisant une pointe de pipette stérile, appuyez doucement sur le stock de glycérol et légèrement strie la surface d'une plaque de gélose pour éviter la perforation de la gélose LB.
- Inversez le striéplaque et incuber à 37 ° C pendant 14 heures.
- Sceller la plaque avec du Parafilm et conserver à 4 ° C pendant un maximum de 3 semaines.
- La culture E. coli pour Comptage des cellules
- Dans un 14 ml tube de culture stérile, passer 2 ml de bouillon LB.
- En utilisant une pointe de pipette stérile, prélever une seule colonie de la plaque de gélose striée préparé à l'étape 5.1 et le transférer dans le tube de culture.
- Incuber la culture à 37 ° C et agiter à 230 tours par minute pendant 14 heures.
- En série diluer la culture bactérienne dans des intervalles de 10 fois.
- Pour chaque échantillon dilué, plaque uniformément cinq 100 aliquotes sur des plaques d'agar LB séparées. Intervertir les plaques et incuber à 37 ° C pendant 14 heures.
- Compter les cellules de chaque échantillon afin d'obtenir la concentration cellulaire moyenne de chaque dilution.
NOTE: 10 7 cellules sont souvent utilisées pour mettre en place un test décisif de référence pour E. coli comme ce niveau de E. coli peut déclencher une couleur rapide change. Cependant, un test décisif correctement exécuté peut détecter aussi peu que 500 cellules, tel que discuté dans la section Résultats.
- Préparation E. Cellules coli pour les essais
- Pour une suspension cellulaire désirée, le transfert de 1 ml de bouillon de culture à un tube de 1,5 ml.
- Centrifuger les cellules à 6000 g pendant 10 min à 4 ° C. Retirez délicatement le surnageant sans perturber le culot cellulaire.
- Ajouter 10 ul de tampon de réaction au culot cellulaire et remettre en suspension les cellules. Sonication de la suspension cellulaire pendant 5 min. Transférer la suspension cellulaire à une boîte de glace pendant 5 min.
- Sonication de la suspension cellulaire pendant encore 5 min.
- Centrifuger la suspension cellulaire à 13000 xg pendant 10 min à 4 ° C. Utiliser le surnageant pour l'essai (10 ul).
6. Test de Litmus
- Dans un tube de 1,5 ml, un prélavage du tube en ajoutant et en tourbillonnement 100 ul de tampon de réaction (RB) dans le tube de microcentrifugeuse etjeter le tampon.
- Transfert 15 pi de EC1 assemblé (protocole 4) sur le tube de microcentrifugation lavé.
- Laver les billes magnétiques en plaçant le tube de microcentrifugeuse sur un support magnétique. Retirer le surnageant par pipetage. Retirez le tube de microcentrifugation du rack, ajouter 100 pi de RB, et resuspendre soigneusement les billes magnétiques.
- Laver les MB deux fois par étape 6.3 répéter.
- Placer le tube de microcentrifugation de retour sur le support magnétique, éliminer le surnageant et ajouter 10 ul E. échantillon coli préparé à l' étape 5.3.
- Mélanger les échantillons et magnétiques perles soigneusement en tapotant doucement sur le tube de microcentrifugeuse.
- Laisser incuber la réaction à température ambiante pendant 1 h.
- Pour la réaction, ajouter 90 ul de ddH 2 O et placez le tube microfuge sur une grille magnétique.
- Au bout d'environ 3 min de séparation magnétique, transvaser soigneusement 85 ul du surnageant dans un tube microfuge de 0,5 ml. Retirer le surnageant lentementéviter de recueillir toutes les billes magnétiques.
- Au microtube ci-dessus ajouter 15 pl de 0,04% de rouge de phénol, et 100 ul de solution de substrat.
- Prenez une photo à des intervalles de temps spécifiques pour enregistrer le changement de couleur.
REMARQUE: le changement du pH peut également être contrôlé à l'aide d'un pH-mètre avec une microélectrode. Le pH de départ devrait être d'environ 5,2-5,5 (la solution est jaune). Sinon, la solution peut être ajustée par addition de 1 mM de tampon acétate pH 5,0).
Résultats
Le principe de l'épreuve décisive bactérienne est expliqué dans la Figure 1 Le test utilise trois matériaux clés:. Un DNAzyme ARN-clivant qui est activé par une bactérie spécifique, uréase et des billes magnétiques. La DNAzyme est utilisé comme élément de reconnaissance moléculaire pour obtenir une détection hautement spécifique d'une bactérie d'intérêt. Uréase et des billes magnétiques sont utilisées pour réaliser la transduction du signal de l'activité ARN-clivage de la DNAzyme. Cela implique la création de billes magnétiques qui contiennent des conjugués uréase-DNAzyme. En présence de la bactérie cible, clive la liaison de l'ARN DNAzyme. Cette action entraîne la dissociation de l'uréase à partir de billes magnétiques. L'uréase libéré peut être facilement séparé des billes magnétiques et servent à produire un changement de couleur dans une solution de rapporteur qui contient de l'urée et un colorant sensible au pH. Uréase hydrolyse l'urée en ammoniac, accompagnée de l'augmentation du pH qui déclenche le color changement du colorant.
La figure 2 présente un test décisif bactérienne où EC1, un E. coli -responsive ARN clivant DNAzyme, a été utilisé comme DNAzyme et le rouge de phénol a été utilisé comme colorant du pH de rapports. EC1 a déjà été isolé par notre groupe à partir d' un pool d'ADN aléatoire séquence en utilisant la technique de sélection in vitro. 5 Nos études antérieures ont montré que EC1 est hautement spécifique de E. coli et présente une activité minimale vers d' autres bactéries. 5,19 On a trouvé que EC1 est activé par une molécule de protéine à partir de E. coli. Bien que l'identité de cette protéine marqueur biologique n'a pas été décrypté, la haute spécificité de reconnaissance suggère que cette protéine est unique à E. coli. La solution de journaliste est mis en place pour avoir un pH initial de 5,5. A ce pH, le rouge de phénol présente une couleur jaune. Comme uréase hydrolyse l'urée en ammoniac, la basicité du sol reporteurution augmente. Cela se traduit par le changement progressif de la couleur du jaune au rose. La profondeur du changement de couleur dépend des deux paramètres suivants, comme illustré par la figure 2: le nombre de E. cellules de E. coli utilisées dans l'étape d'activation DNAzyme et le temps alloué à l'étape d'hydrolyse d' urée. Plus E. les cellules coli ont entraîné des changements de couleurs plus fortes, reflétée par l'observation d'un jaune-à-rose transition de couleur lorsque E. progressive cellules coli ont été en série ont augmenté de 5 à 5 x 10 7 (10 fois plus à chaque fois). Pendant ce temps, un temps plus long pour l' hydrolyse de l' urée a permis la détection de petits nombres de E. cellules de E. coli (5000 cellules dans la réaction de 1 heure et à 500 cellules dans la réaction 2 heures).
Le changement de pH de l'épreuve décisive bactérienne peut également être contrôlée à l' aide d' un pH - mètre de poche et des résultats représentatifs sont illustrés à la figure 3. Il was a constaté que la présence de 10 7 E. cellules de E. coli ont donné lieu à augmentation progressive du pH par des unités 3 à 10 min. En revanche, l'absence de E. cellules coli ne provoquent des changements de pH détectables dans le cadre du même réglage.
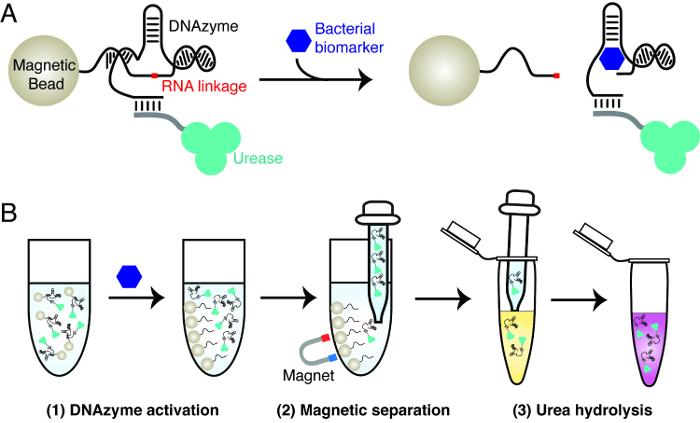
Figure 1: Le principe de conception de test décisif bactérienne (A) Activation d'un DNAzyme d'ARN-cliver par un biomarqueur spécifique d'une bactérie d'intérêt.. En présence du biomarqueur, la DNAzyme ARN clivant immobilisé sur des billes magnétiques clive la liaison de l'ARN, ce qui entraîne la libération de l'uréase étiqueté à partir de billes magnétiques à la solution. (B) en trois étapes procédure de dosage. Etape 1: activation de DNAzyme, tel que décrit dans le panneau A. Étape 2: La séparation magnétique - l'uréase libéré est séparé des billes magnétiques. Étape 3: hydrolyse de l'urée R12; l'uréase libéré est ajouté dans une solution contenant de l'urée rapporteur. Uréase hydrolyse l' urée en ammoniac, ce qui entraîne un changement de pH qui peut être rapporté par un colorant sensible au pH. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.

Figure 2: Test de Litmus avec E. E. coli en utilisant DNAzyme EC1 de coli. Résultats de changement de couleur représentatifs avec un nombre variable de E. cellules coli fournies ci - dessus chaque tube à essai. Le rouge de phénol a été utilisé comme colorant sensible au pH. Un essai sans E. coli a été utilisé comme témoin négatif. Plus E. coli cellules devraient provoquer la libération de plusieurs molécules d'uréase, accompagné by changements de couleurs fortes. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
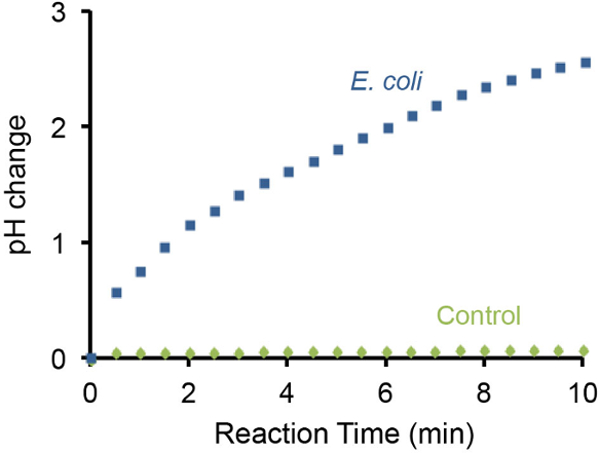
Figure 3: Contrôle de l' augmentation du pH à l' aide d' un pH mètre Le changement de pH provoqué par E. 10 7. cellules de E. coli a été contrôlée en utilisant un pH - mètre portatif. Un essai sans E. coli a été utilisé comme témoin négatif. La présence de 10 7 E. cellules coli dans la solution d'essai peut augmenter la basicité par des unités ~ 3 de pH dans 10 min. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
| prénom | Seséquence (5'-3 ') | Remarque |
| BS1 | BTTTT TTTTT TTTAC TCTTC CTAGC FRQGG TTCGA TCAAG A | B: 5'-biotine; R: ribonucléotide adénine; F: fluorescéine dT; Q: dabcyl-dT |
| DE1 | GATGT GCGTT GTCGA GACCT GCGAC CGGAA CACTA CACTG TGTGG GGATG GATTT CTTTA CAGTT GTGTG TTGAA CGCTG TGTCA AAAAA AAAA | |
| T1 | GACAA CGCAC ATCTC TTGAT C CGA-Canada | |
| LD1 | XTTTT TTTTT TTTTT TTGAC ACAGC GTTCA A | X: 5'-NH 2 |
Tableau 1: Séquences d'oligonucléotides synthétiques.
Discussion
La traduction de l'action de l'activité ARN de clivage d'une DNAzyme bactérie sensible à un test décisif est rendu possible grâce à l'utilisation de l' uréase et la séparation magnétique, comme illustré par la figure 1. Bien que la démonstration de la mise à l' épreuve modifiée pour la détection bactérienne est fait avec un E. coli dépendante de l' ARN-clivant DNAzyme, 5,19,20 la conception peut généralement être prolongé pour tout ARN clivant DNAzyme. Compte tenu de la grande disponibilité de DNAzymes d'ARN-clivant pour différents analytes et diverses méthodes pour isoler les nouveaux DNAzymes d'ARN-clivant de pools séquence aléatoire pour de nouvelles cibles, nous nous attendons à ce que la plate-forme de test décisif modifié peut être étendue à la détection de diverses cibles d'intérêt .
Le test décisif pour E. coli détection peut détecter 5000 et 500 cellules lorsque le temps de réaction de référence est réglé pour être 1 et 2 h, respectivement. La réaction en chaîne par polymérase populaire (PCR) eten sandwich enzyme linked immunosorbent assay (ELISA) peuvent atteindre des limites de détection d'environ 10 4 -10 5 E. cellules coli dans les temps de tests similaires. 25,26 Ainsi, le test décisif bactérienne offre une sensibilité de détection comparable.
Bien que le test décisif bactérien est facile à réaliser et peut produire des changements de couleurs vives, plusieurs facteurs peuvent affecter de manière significative les résultats des tests. Tout d'abord, la qualité de l'uréase est très important. Nous avons utilisé l'uréase provenant de sources différentes et a trouvé les résultats des tests peuvent varier de façon significative. Nous recommandons l'utilisation d'uréase à partir de la source spécifiée dans la section des matériaux.
L'assemblage de DNAzyme / uréase / billes magnétiques nécessite une attention particulière. Un lavage soigneux des billes magnétiques pour enlever UrDNA non hybridée est nécessaire pour éviter des résultats faussement positifs. Des précautions doivent également être prises pour éviter l'accumulation de billes magnétiques résiduels sur la surface intérieure de la lid du tube microfuge, qui peut être difficile à voir. Une fois là, les billes magnétiques ne sont plus soumis à une séparation magnétique et donc, pouvaient transporter certains UrDNA non hybridée qui peut conduire à des signaux faux positifs dans la réaction de reporter. Il est également important d'éviter de laisser le tube de microcentrifugeuse sur le support magnétique pendant plus de 10 minutes pendant l'étape de séparation magnétique. Les billes peuvent agréger ou coller au tube microfuge, ce qui peut réduire l'efficacité de lavage et d'introduire l'incohérence de lot à lot. Inclusion de 0,01% de Tween-20 dans la solution de lavage peut améliorer la cohérence de lot à lot et devrait être mis en œuvre.
Les billes magnétiques sont revêtues de streptavidine, qui a été utilisé en tant que point d'ancrage pour assembler des conjugués DNAzyme-uréase sur des billes magnétiques. À la fois la streptavidine et uréase sont des molécules de protéines qui peuvent être dénaturées lors du stockage. Nous conservons généralement les billes DNAzyme-uréase-magnétiques assemblés à 4 ° C pendant jusqu'à 4 semaineset de faire des lots frais régulièrement pour obtenir des résultats plus cohérents.
Des précautions doivent également être prises pour éviter accidentellement prendre des billes magnétiques dans l'étape de séparation magnétique (étape 6.9) après l'activation DNAzyme. D'après notre expérience, les débris cellulaires et d'autres particules dans la solution peuvent réduire l'efficacité de la séparation magnétique, et donc, des billes magnétiques peuvent être involontairement pris au cours de pipetage. Cela se traduira par des résultats faussement positifs. Nous recommandons les mesures suivantes pour atténuer le problème: un temps plus long de séparation (comme 5-10 min), une libération plus lente de la pression sur la pipette pour permettre le retrait en douceur du surnageant, et on soumet le surnageant à une série supplémentaire de séparation magnétique .
Enfin, il est important d'éviter la contamination accident de la solution reporter du stock par uréase au cours d'une expérience où plusieurs échantillons sont testés. Compte tenu de la forte réactivité de urease, la contamination de cette nature peut conduire à des résultats faussement positifs.
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
The funding for this research project was provided by the Natural Sciences and Engineering Research Council of Canada (NSERC) via a Discovery Grant to YL.
matériels
| Name | Company | Catalog Number | Comments |
| Ethylenediaminetetraacetic acid (EDTA) | VWR AMRESCO | 0105 | |
| Sodium Hydroxide (NaOH) pellets | BIO BASIC CANADA INC. | SB6789 | |
| Tris-base | VWR AMRESCO | 0497 | |
| Boric acid | AMRESCO | 0588 | |
| Urea | VWR AMRESCO | M123 | |
| 40% acrylamide/bisacrylamide (29:1) solution | BIO BASIC CANADA INC. | A0007 | |
| Sucrose | Bioshop Canada inc. | SUC507 | |
| Bromophenol blue | Bioshop Canada inc. | BRO777 | |
| Xylenecyanol FF | SIGMA-ALDRICH | X-4126 | |
| 10% sodium dodecyl sulfate | Bioshop Canada inc. | SDS001 | |
| Hydrochloric Acid (HCl) | CALEDON LABORATORIES LTD | 6026 | |
| Sodium Chloride (NaCl) | Bioshop Canada inc. | SOD001 | |
| 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid (HEPES) | Bioshop Canada inc. | HEP001 | |
| Magnesium Chloride (II) hexahydrate | VWR AMRESCO | 0288 | |
| Tween 20 | Bioshop Canada inc. | TW508 | |
| Adenosine Triphospahte (ATP) | AMRESCO | 0220 | |
| Sodium Acetate trihydrate (NaOAc) | SIGMA-ALDRICH | S8625 | |
| Ethanol | Commercial Alcohols | P016EAAN | |
| Tetramethyleneethylenediamine (TEMED) | AMRESCO | 0761 | |
| 10% Ammonium persulfate (APS) | BIO BASIC CANADA INC. | AB0072 | |
| Succinimidyl 4-(N-maleimidomethyl) cyclohexane-1-carboxylate (SMCC) | ThermoFisher SCIENTIFIC | 22360 | |
| Dimethyl sulfoxide (DMSO) | CALEDON LABORATORIES | 803540 | |
| Urease | SIGMA-ALDRICH | U0251 | |
| 1x Phosphate Buffered Saline (PBS) | ThermoFisher SCIENTIFIC | 70011-069 | |
| 0.04% Phenol red | SIGMA-ALDRICH | P3532 | |
| 10x T4 polynucleotide kinase reaction buffer | Lucigen | 30061-1 | |
| 10x T4 DNA ligase reaction buffer | Bio Basics Canada | B1122-B | |
| T4 DNA ligase (5 U/μl) | Thermo Fischer Scientific | B1122 | |
| Luria Bertani (LB) Broth | AMRESCO | J106 | |
| Agar | AMRESCO | J637 | |
| T4 polynucleotide kinase (10 U/μl) | Lucigen | 30061-1 | |
| E. coli K12 (MG1655) | ATCC | ATCC700926 | |
| Centrifuge | Beckman Coulter, Inc. | 392187 | |
| Glass plates | CBS scientific | ngp-250nr | |
| 0.75 mm thick spacers | CBS scientific | VGS-0725r | |
| 12-well comb | CBS scientific | VGC-7512 | |
| UV Lamp | UVP | 95-0017-09 | |
| Spectrophotometer (NanoVue) | GE Healthcare | N/A | |
| Metal plate | CBS scientific | CPA165-250 | |
| Vortex | VWR International | 58816-123 | |
| Gel electrophoresis apparatus | CBS scientific | ASG-250 | |
| Petri dishes | VWR International | 25384-342 | |
| 100 kDa MWCO centrifugal filters | EMD Millipore | UFC510024 | |
| Magnetic Bead (BioMag) | Bangs Laboratories Inc | BM568 | |
| Magnetic Seperation Rack | New England BioLabs | S1506S | |
| Microfuge tubes | Sarstedt | 72.69 | |
| Syringe filter (0.22 μm) | VWR International | 28145-501 | |
| 14 ml culture tube | VWR International | 60818-725 | |
| Cell culture incubator | Eppendorf Scientific | M13520000 | |
| Branson Ultrasonic cleaner | Branson | N/A | |
| Camera (Canon Powershot G11) | Canon | N/A | |
| 50 ml conical tube | VWR International | 89004-364 |
Références
- Daar, A. S., et al. Top ten biotechnologies for improving health in developing countries. Nat. Genet. 32, 229-232 (2002).
- Newman, J. D., Turner, A. P. Home blood glucose biosensors: a commercial perspective. Biosens. Bioelectron. 20, 2435-2453 (2005).
- Turner, A. P. Biosensors: sense and sensibility. Chem. Soc. Rev. 42, 3184-3196 (2013).
- Tram, K., Kanda, P., Salena, B. J., Huan, S. Y., Li, Y. F. Translating Bacterial Detection by DNAzymes into a Litmus Test. Angew. Chem. Int. Ed. 53, 12799-12802 (2014).
- Ali, M. M., Aguirre, S. D., Lazim, H., Li, Y. Fluorogenic DNAzyme probes as bacterial indicators. Angew. Chem. Int. Ed. 50, 3751-3754 (2011).
- Schlosser, K., Li, Y. Biologically inspired synthetic enzymes made from DNA. Chem. Biol. 16, 311-322 (2009).
- Tuerk, C., Gold, L. Systematic evolution of ligand by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science. 249, 505-510 (1990).
- Ellington, A. D., Szostak, J. W. In vitro selection of RNA molecules that bind specific ligands. Nature. 346, 818-822 (1990).
- Breaker, R. R. Making catalytic DNAs. Science. 290, 2095-2096 (2000).
- Liu, J., Cao, Z., Lu, Y. Functional nucleic acid sensors. Chem. Rev. 109, 1948-1998 (2009).
- Navani, N. K., Li, Y. Nucleic acid aptamers and enzymes as sensors. Curr. Opin. Chem. Biol. 10, 272-281 (2006).
- Li, J., Lu, Y. A highly sensitive and selective catalytic DNA biosensor for lead ions. J. Am. Chem. Soc. 122, 10466-10467 (2000).
- Liu, J., Lu, Y. A colorimetric lead biosensor using DNAzyme-directed assembly of gold nanoparticles. J. Am. Chem. Soc. 125, 6642-6643 (2003).
- Liu, Z., Mei, S. H. J., Brennan, J. D., Li, Y. Assemblage of signaling DNA enzymes with intriguing metal specificity and pH dependence. J. Am. Chem. Soc. 125, 7539-7545 (2003).
- Liu, J., et al. A catalytic beacon sensor for uranium with parts-per-trillion sensitivity and millionfold selectivity. Proc. Natl. Acad. Sci. USA. 104, 2056-2061 (2007).
- Huang, P. J., Vazin, M., Liu, J. In vitro selection of a new lanthanide-dependent DNAzyme for ratiometric sensing lanthanides. Anal. Chem. 86, 9993-9999 (2014).
- Chiuman, W., Li, Y. Simple fluorescent sensors engineered with catalytic DNA 'MgZ' based on a non-classic allosteric design. PLoS One. 2, e1224 (2007).
- Xiang, Y., Lu, Y. Using personal glucose meters and functional DNA sensors to quantify a variety of analytical targets. Nat. Chem. 3, 697-703 (2011).
- Aguirre, S. D., Ali, M. M., Salena, B. J., Li, Y. A sensitive DNA enzyme-based fluorescent assay for bacterial detection. Biomolecules. 3, 563-577 (2013).
- Aguirre, S. D., Ali, M. M., Kanda, P., Li, Y. F. Detection of Bacteria Using Fluorogenic DNAzymes. J. Vis. Exp. (63), e3961 (2012).
- Shen, Z., et al. A catalytic DNA activated by a specific strain of bacterial pathogen. Angew. Chem. Int. Ed. 54, (2015).
- He, S., et al. Highly specific recognition of breast tumors by an RNA-cleaving fluorogenic DNAzyme probe. Anal. Chem. 87, 569-577 (2015).
- Sumner, J. B., Hand, D. B. Isoelectric point of crystalline urease. J. Am. Chem. Soc. 51, 1255-1260 (1929).
- Karplus, P. A., Pearson, M., Hausinger, R. P. 70 Years of crystalline urease: What have we learned. Acc. Chem. Res. 30, 330-337 (1997).
- Omiccioli, E., Amagliani, G., Brandi, G., Magnani, M. A new platform for Real-Time PCR detection of Salmonella spp., Listeria monocytogenes and Escherichia coli O157 in milk. Food Microbiol. 26, 615-622 (2009).
- Cui, S., Schroeder, C. M., Zhang, D. Y., Meng, J. Rapid sample preparation method for PCR-based detection of Escherichia coli O157:H7 in ground beef. J. Appl. Microbiol. 95, 129-134 (2003).
- Ibekwe, A. M., Watt, P. M., Grieve, C. M., Sharma, V. K., Lyons, S. R. Multiplex fluorogenic real-time PCR for detection and quantification of Escherichia coli O157:H7 in dairy wastewater wetlands. Appl. Environ. Microbiol. 68, 4853-4862 (2002).
- Strachan, N. J., Ogden, I. D. A sensitive microsphere coagulation ELISA for Escherichia coli O157:H7 using Russell's viper venom. FEMS Microbiol Lett. 186, 79-84 (2000).
- de Boer, E., Beumer, R. R. Methodology for detection and typing of foodborne microorganisms. Int. J. Food Microbiol. 50, 119-130 (1999).
- Gracias, K. S., McKillip, J. L. A review of conventional detection and enumeration methods for pathogenic bacteria in food. Can. J. Microbiol. 50, 883-890 (2004).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon