Method Article
Identificação de inibidores de PD-1/PD-L1 com tecnologia de ressonância plasmônica de superfície
Neste Artigo
Resumo
Este protocolo descreve um ensaio de bloqueio para inibidores de PD-1/PD-L1 usando tecnologia de ressonância plasmônica de superfície. Ele emprega uma estratégia de imobilização em duas etapas e um sistema tampão personalizado para medir com precisão as unidades de resposta, facilitando a avaliação das taxas de bloqueio de compostos ou produtos biológicos. Além disso, suporta a identificação de alto rendimento de inibidores de PD-1/PD-L1.
Resumo
A interrupção da interação PD-1/PD-L1 é uma estratégia promissora para a imunoterapia contra o câncer. Plataformas de triagem confiáveis são essenciais para avaliar a eficácia dos inibidores de PD-1/PD-L1. Um ensaio de bloqueio de PD-1/PD-L1 humano previamente estabelecido utilizando a tecnologia de Ressonância Plasmônica de Superfície (SPR) (plataforma de triagem SPR inibidora de PD-1/PD-L1 de primeira geração) demonstrou resultados comparáveis aos obtidos por meio de Fluorescência Homogênea Resolvida no Tempo (HTRF) e ensaios baseados em células, com potencial para triagem em larga escala. Aqui, uma versão otimizada deste ensaio (plataforma de triagem SPR inibidora de PD-1 / PD-L1 de segunda geração) é apresentada, apresentando um processo de acoplamento de duas etapas que combina acoplamento de amina e bioestreptavidina para melhorar o controle de orientação de PD-1 no chip e reduzir o consumo de proteína PD-1. A plataforma atualizada foi validada com sucesso usando o inibidor de PD-1/PD-L1 BMS-1166, mostrando efeitos de bloqueio comparáveis ao método anterior baseado em SPR e outras técnicas estabelecidas, como ELISA. Esses resultados confirmam a confiabilidade da abordagem. Esta plataforma de triagem SPR otimizada oferece uma ferramenta confiável e de alto rendimento para identificar novos inibidores de PD-1/PD-L1, avançar na pesquisa de imunoterapia contra o câncer e destacar o potencial da SPR na triagem de inibidores de checkpoint imunológico.
Introdução
As terapias de bloqueio de checkpoint imunológico, particularmente aquelas direcionadas à Morte Celular Programada-1 (PD-1) e ao Ligante de Morte Celular Programada 1 (PD-L1), estão na vanguarda das estratégias de imunoterapia contra o câncer. As terapias anti-PD-1/PD-L1 receberam aprovação para uso em vários tipos de câncer, como câncer hematológico, cutâneo, pulmonar, hepático, de bexiga urinária e renal1. PD-1 é uma glicoproteína transmembrana pertencente à superfamília das imunoglobulinas, caracterizada por um único domínio semelhante a uma variável de imunoglobulina (IgV) no N-terminal, um pedúnculo de aproximadamente 20 aminoácidos separando o domínio IgV da membrana plasmática, um domínio transmembrana e uma cauda citoplasmática contendo motivos de sinalização baseados em tirosina2. A PD-L1, identificada como um dos ligantes para a PD-1, é uma proteína transmembrana do tipo I com região transmembrana, dois domínios extracelulares - constante de imunoglobulina (IgC) e IgV - e um domínio citoplasmático relativamente curto que desencadeia vias de sinalização intracelular3. A via inibitória PD-1 / PD-L1 serve como um ponto de verificação imunológico crítico que regula a ativação e a autoimunidade das células T4. O PD-1 é expresso em células T, onde interage com o PD-L1, inibe a sinalização do receptor de células T e bloqueia a estimulação de moléculas CD28 e CD80 em células apresentadoras de antígenos e células T5. Os tecidos cancerígenos exploram esse mecanismo fisiológico superexpressando PD-L1 durante a fase de escape, criando assim um ambiente imunossupressor que promove o crescimento e a progressão do tumor6. Os inibidores de PD-1 e PD-L1 interrompem essa interação, permitindo que o sistema imunológico evite a supressão induzida pelo tumor e reinicie o processo de morte tumor-celular mediado por células T7.
Com base na base estabelecida pelo papel proeminente das terapias de bloqueio de checkpoint imunológico, o desenvolvimento de inibidores de PD-1 / PD-L1 marcou um avanço significativo na imunoterapia contra o câncer. A Food and Drug Administration (FDA) dos EUA endossou nove inibidores de checkpoint imunológico que visam especificamente a via PD-1 / PD-L1. Estes incluem seis inibidores de PD-1 - pembrolizumabe, dostarlimabe, nivolumabe, cemiplimabe, oripalimabe e tislelizumabe - e três inibidores de PD-L1 - atezolizumabe, avelumabe e durvalumabe 8,9. Essas terapias têm sido efetivamente utilizadas para tratar uma variedade de cânceres, como melanoma, câncer de pulmão, câncer urotelial, câncer cervical, câncer gástrico ou gastroesofágico e outros tumores sólidos10. Apesar de sua eficácia, as terapias baseadas em anticorpos monoclonais enfrentam limitações significativas, incluindo baixas taxas de resposta, altos custos, meias-vidas prolongadas, eventos adversos graves relacionados ao sistema imunológico e restrições ao parto intravenoso ou subcutâneo 11,12,13. Consequentemente, a pesquisa está cada vez mais focada no desenvolvimento de inibidores de moléculas pequenas direcionados ao eixo PD-1 / PD-L1. Essas pequenas moléculas oferecem vantagens distintas, como melhor penetração celular, modulação de diversos alvos biológicos, biodisponibilidade oral aprimorada e custos reduzidos, com o objetivo de alcançar resultados terapêuticos comparáveis com menos efeitos adversos14. No entanto, o desenvolvimento de inibidores de pequenas moléculas direcionados à interação PD-1/PD-L1 está em seus estágios iniciais, principalmente devido à falta de uma plataforma confiável de triagem de alto rendimento. Essas plataformas são essenciais para avaliar rapidamente vastas bibliotecas de pequenas moléculas e identificar compostos principais para validação e otimização adicionais. Superar esse desafio é fundamental para o avanço da imunoterapia contra o câncer.
A tecnologia de Ressonância Plasmônica de Superfície (SPR) é amplamente empregada na detecção de várias biomoléculas, incluindo antígenos de anticorpos, enzimas, ácidos nucléicos e medicamentos, e é particularmente eficaz na triagem de medicamentos de moléculas pequenas15,16. Ao contrário de outras técnicas biofísicas, a SPR oferece detecção sem marcadores, dados cinéticos em tempo real e uma ampla faixa de detecção. Em contraste, a calorimetria de titulação isotérmica carece de insights cinéticos em tempo real e requer volumes de amostra maiores, limitando o rendimento. A termoforese em microescala é propensa a interferência de buffer e não pode fornecer dados cinéticos, enquanto a interferometria de biocamada tem limitações específicas da aplicação com base no tamanho e nas propriedades moleculares. A fluorescência homogênea resolvida no tempo requer rotulagem e é suscetível à interferência fluorescente. Reconhecemos que o HTRF é outra tecnologia adequada para explorar os inibidores de PD-1 / PD-L1. Uma limitação inerente do HTRF, em comparação com o SPR, é a extinção da fluorescência causada por interações externas com o processo de excitação intramolecular (por exemplo, transferência de elétrons, FRET e branqueamento), a sensibilidade é muito baixa no processo de triagem de drogas devido ao pequeno intervalo de janela e interferência de compostos de biblioteca fluorescente ou proteínas biológicas17. Esses recursos posicionam a SPR como uma ferramenta superior para a descoberta de medicamentos. Nossos estudos anteriores demonstraram que a SPR é capaz de determinar o efeito de bloqueio de pequenas moléculas contra PD-1/PD-L1, o que é vantajoso em relação a outras técnicas que exigem altos requisitos de tecnologia de marcação, múltiplas etapas, baixa especificidade e alto custo no processo de descoberta de medicamentos18.
Este estudo apresenta uma plataforma otimizada baseada em SPR, integrando um processo de acoplamento de duas etapas que utiliza acoplamento de amina e bioestreptavidina para melhorar a orientação do PD-1 no chip e minimizar o uso de proteínas. Essa abordagem atualizada foi validada com sucesso usando o inibidor de PD-1 / PD-L1 BMS-1166 como um aglutinante de controle positivo, demonstrando efeitos de bloqueio comparáveis ao nosso método SPR anterior e a outras técnicas estabelecidas, como ELISA19,20. Isso não apenas confirma a confiabilidade e reprodutibilidade de nosso protocolo, mas também ilustra a eficácia de nossa plataforma modificada em facilitar a triagem de alto rendimento de inibidores de PD-1 / PD-L1. A incorporação da etapa de captura de bioestreptavidina fornece orientação proteica direcionada ao local em vez de aleatória, permitindo concentração reduzida de PD-1 (40 μg/mL vs. 10 μg/mL) e economia de custos, permitindo que o usuário final imobilize estreptavidina (SA) em um chip CM5, uma alternativa mais barata aos chips SA pré-imobilizados comercializados. Isso o torna vantajoso para triagens em larga escala e econômicas de bibliotecas de compostos/peptídeos. Embora métodos adicionais de caracterização, incluindo ensaios in silico, in vitro e in vivo, sejam essenciais para avaliar o potencial clínico dos inibidores de PD-1/PD-L1 contra o câncer, nossa plataforma de triagem aprimorada baseada em SPR se destaca como uma ferramenta eficiente para triagem em larga escala de inibidores de PD-1/PD-L1.
Protocolo
Os reagentes e equipamentos estão listados na Tabela de Materiais.
1. Imobilização da proteína estreptavidina (SA) no chip CM5
- Configure o método de imobilização no instrumento SPR : Abra/novo modelo de assistente , escolha a imobilização, defina o tipo de chip para CM5 e as células de fluxo por ciclo para 1. Verifique a imobilização da célula de fluxo 1 e da célula de fluxo 2.
- Defina Amine como método de imobilização. Defina o objetivo para o nível de imobilizado, concentração de ligante: 40 μg / mL de estreptavidina, nível alvo: 2000 RU, solução de lavagem: 50 mM NaOH. Em seguida, verifique o prime antes de executar.
- Preparar os seguintes tubos: R2 B1 e R2 C1 - 40 μg/mL de estreptavidina; R2 B2 e R2 C2: 50 mM de NaOH; R2 B3 e R2 C3: EDC; R2 B4 e R2 C4: TAN; R2 B5 e R2 C5: Vazio; R2 B6 e R2 C6: etanolamina.
NOTA: EDC e NHS ativam os grupos carboxila no chip CM5, permitindo o acoplamento covalente com aminas no ligante. A etanolamina é usada como um tampão de bloqueio para evitar a ligação inespecífica durante a imobilização. - Dilua 20 mL de tampão HBS-EP+ 10× em 180 mL de água deionizada (DI) para preparar 200 mL de solução tampão de corrida 1× HBS-EP+.
- Adicione 1 mL de água livre de DNase a 1 mg de estreptavidina e incube em temperatura ambiente por 30 min. Em seguida, dilua a solução de estreptavidina a 40 μg/ml em tampão acetato (pH 4,5). Prepare os reagentes do kit de reagentes do acoplamento de amina de acordo com as instruções do fabricante e coloque todos os layouts do reagente relevantes conforme indicado na etapa 1.2.
- Substitua o chip do sensor de manutenção pelo chip CM5, coloque o tubo A no buffer HBS-EP+ de 1× preparado, reabra o método de imobilização e insira os tubos de acordo com o layout da etapa 1.2. Execute o método e salve o arquivo de resultados.
- Manutenção pós-execução: substitua o chip CM5 pelo chip do sensor de manutenção, coloque o Tubo A em uma garrafa cheia de água deionizada e execute a escorva. Depois de retirar o chip CM5, lave o chip com algumas gotas de água DI e seque ao ar. Coloque o chip em um tubo de 50 mL a 4 °C.
NOTA: O SA imobilizado permite que o chip CM5 funcione como um chip SA.
2. Imobilização da proteína PD-1 no chip SA
- Defina o método de imobilização no instrumento SPR : Abrir/novo modelo de assistente , escolha imobilização, defina o tipo de chip para SA e células de fluxo por ciclo para 1. Verifique imobilizar célula de fluxo 1 e célula 2.
- Defina a captura de SA-biotina como o método. Para a imobilização em branco do conjunto da célula 1, para a célula 2, defina o objetivo do nível imobilizado, 10 μg/mL PD-1 como ligante, nível alvo: 4000 RU e, em seguida, verifique o primer antes de executar.
NOTA: Prepare os seguintes tubos - R2 B1 e R2 C1: 1 M de NaCl, 50 mM de NaOH; R2 B2 e R2 C2: 50% de isopropanol/50 mM de NaOH/1 m de NaCl; R2 C3: 10 μg/mL PD-1.
- Defina a captura de SA-biotina como o método. Para a imobilização em branco do conjunto da célula 1, para a célula 2, defina o objetivo do nível imobilizado, 10 μg/mL PD-1 como ligante, nível alvo: 4000 RU e, em seguida, verifique o primer antes de executar.
- Dissolva 58,44 mg de NaCl em 1 mL de NaOH 50 mM para preparar a solução de NaCl 1 M e 50 mM de NaOH. Dissolva 58,44 mg de NaCl e 4,0 mg de NaOH em 500 μL de água e, em seguida, adicione 500 μL de isopropanol para preparar a solução de 50% de isopropanol / 50 mM de NaOH / 1 M de NaCl.
- Prepare a solução de PD-1: Adicione 200 μL de água livre de DNase a 100 μg de PD-1 humano biotinilado (Fc e Avitagged) e estabilize à temperatura ambiente por 30 min, depois dilua para 10 μg / mL em tampão acetato (pH 5,0).
- Coloque todos os layouts de reagente conforme descrito na etapa 2.1. Coloque o tubo A na solução tampão de execução HBS-EP+ de 1×, ejete o chip do sensor de manutenção e insira o chip SA (chip CM5 revestido pela proteína estreptavidina da etapa 1). Reabra o método de imobilização, insira o rack de reagentes 2 e verifique as posições e, em seguida, execute o método para o tempo de execução estimado.
- Repita a etapa 1.6.
3. Reconhecimento de regeneração para PD-1 e PD-L1
- Configure o método de reconhecimento de regeneração: Abra/novo modelo de assistente , escolha Regeneration Scouting, defina o caminho do fluxo: 2-1, 4-3, tipo de chip para SA, verifique o ciclo de condicionamento de execução, registre a solução como HBS-EP+, tempo de contato como 30 s, número de injeções como 3, solução como PD-L1, tempo de contato: 30 s, taxa de fluxo: 30 μL/min.
- Para parâmetros de regeneração, a vazão é de 30 μL/min e o período de estabilização é de 300 s. No planejamento experimental, defina o número de condições como 4 e o número de ciclos para cada condição como 2. Defina as condições como 4, solução de regeneração: Glicina 1.5, 2.0, 2.5, 3.0, tempos de contato: 30 s, verifique a escorva antes de executar.
- Defina cada concentração de PD-L1 como uma posição de poço de amostra separada em um layout de microplaca de 96 poços: R1 A1 a R1 A9: 1 μM PD-L1; R1 A10 a R1 A12: amortecedor HBS-EP+; R1 B1: Glicina 1,5; R1 B2: Glicina 2; R1 B3: glicina 2,5; R1 B4: Glicina 3.
NOTA: O tampão glicina com pH diferente é usado como tampão de regeneração. - Prepare a solução PD-L1: Adicione 200 μL de água livre de DNase a 100 μg de proteína PD-L1 Fc Tag humana (9,42 μM) e, em seguida, dilua a 1 μM em tampão HBS-EP+.
- Colocar todos os reagentes relevantes no esquema conforme descrito no passo 3.2. Coloque o tubo A no buffer HBS-EP+ de 1× e, em seguida, substitua o chip do sensor de manutenção pelo chip SA. Reabra o método Regeneration Scouting , siga a posição do tubo da etapa 3.2, insira o rack de reagentes e execute o método para o tempo de execução estimado.
- Repita a etapa 1.6.
4. Validação da interação PD-1/PD-L1
NOTA: Para validação, um relatório publicado anteriormente18 foi seguido com pequenos ajustes.
- Use os mesmos parâmetros do relatório publicado em Configurações gerais, Etapas do ensaio e Tipos de ciclo, defina o tempo de contato para 60 s, o tempo de dissociação para 60 s e a taxa de fluxo para 30 μL/min. Em variáveis de método, variáveis de avaliação e comandos, use a mesma configuração, exceto para usar Glicina 2.0 como Regeneração, taxa de fluxo de configuração para 30 μL/min, passa pelo caminho de fluxo de 1, 2, 3, 4.
- Em seguida, configure run e escolha o caminho do fluxo: 2-1, 4-3. Insira PD-L1 com concentrações de 0 μM, 0,037 μM, 0,111 μM, 0,333 μM e um peso molecular de 51.300 Da. Verifique todas as etapas do ensaio para verificação e selecione o primer antes da execução.
- Em seguida, defina cada concentração de PD-L1 como uma posição de poço de amostra separada em um layout de rack de reagentes 2: R2 B1: PD-L1 0 μM; R2 B2: PD-L1 0,037 μM; R2 B3: PD-L1 0,111 μM; R2 B4: PD-L1 0,333 μM; R2 A1: Glicina 2.0 como tampão de regeneração; R2 A2: HBS-EP+ como buffer de inicialização.
- Prepare 200 mL de solução tampão de corrida 1× HBS-EP+. Prepare as concentrações de PD-L1: dilua a proteína PD-L1 a 9,42 μM a 0,333 μM, 0,111 μM e 0,037 μM em HBS-EP+. Em seguida, colocar todos os reagentes relevantes no esquema descrito no passo 4.1.2.
- Insira o tubo A na solução de buffer de execução HBS-EP+ de 1×, ejete o chip do sensor de manutenção e insira o chip SA. Reabra o método de reconhecimento de regeneração , siga o posicionamento do tubo da etapa 4.1, insira o rack de reagentes 2 e execute o método pela duração estimada.
- Repita a etapa 1.6.
5. Ensaio de bloqueio PD-1 / PD-L1 com inibidor de molécula pequena: BMS-1166
NOTA: Para o ensaio de bloqueio, um relatório publicado anteriormente18 foi seguido com pequenos ajustes.
- Use os mesmos parâmetros do relatório publicado em Configurações gerais, Etapas do ensaio e Tipos de ciclo, defina o tempo de contato para 60 s, o tempo de dissociação para 60 s e a taxa de fluxo para 30 μL/min. Em variáveis de método, variáveis de avaliação e comandos também usam a mesma configuração, exceto para usar Glicina 2.0 como Regeneração, taxa de fluxo de configuração para 30 μL/min, passa pelo caminho de fluxo de 1, 2, 3, 4.
- Em seguida, configure run e escolha o caminho do fluxo: 2-1, 4-3. Insira a solução da amostra: PD-L1 (0,111 μM, peso molecular de 51.300 Da) com BMS-1166 a 0 μM, 0,125 μM, 0,625 μM, 3,125 μM. Verifique todas as etapas do ensaio para verificação e selecione o primer antes da execução.
- Em seguida, defina cada concentração de PD-L1 como uma posição de poço de amostra separada em um layout de rack de reagentes 2: R2 B1: PD-L1 (0,111 μM) + BMS-1166 0 μM R2 B2: PD-L1 (0,111 μM) + BMS-1166 0,125 μM; R2 B3: PD-L1 (0,111 μM) + BMS-1166 0,625 μM; R2 B4: PD-L1 (0,111 μM) + BMS-1166 3,125 μM; R2 A1: Glicina 2.0 para regeneração.
- Prepare 200 mL de solução tampão de corrida 1× HBS-EP+. Prepare a mistura BMS-1166 / PD-L1: Dissolva 5 mg de BMS-1166 em 77,99 μL de dimetilsulfóxido (DMSO) para preparar uma solução estoque de 100 mM. Dilua a solução estoque com proteína PD-L1 (0,11 μM) até as concentrações alvo de BMS-1166 a 0 μM, 0,125 μM, 0,625 μM e 3,125 μM em HBS-EP+. Colocar todos os reagentes relevantes dispostos conforme descrito no passo 5.1.2.
- Coloque o tubo A na solução tampão de execução HBS-EP+ de 1×, ejete o chip do sensor de manutenção e insira o chip SA. Reabra o método de reconhecimento de regeneração , siga a posição do tubo de 5.1 e, insira o rack de reagentes 2 e execute o método para o tempo de execução estimado.
- Repita a etapa 1.6.
Resultados
Imobilização de SA no chip CM5
Os dados foram analisados por meio da saída do instrumento SPR e do software de análise associado, indicando o alcance bem-sucedido da RU alvo (2000 RU) da proteína SA na célula de fluxo 1 e na célula de fluxo 2. As células de fluxo 1 e 2 foram imobilizadas com SA (40 μg/mL) na superfície do chip CM5 com uma resposta final de 1902,3 RU na célula de fluxo 1 (Figura 1A) e 1900,7 RU na célula de fluxo 2 (Figura 1B).
Imobilização de PD-1 no chip SA
A análise de dados com base na saída do instrumento SPR e software associado indicou uma unidade de resposta baixa (RU) para a célula em branco na célula de fluxo 1 e a obtenção bem-sucedida da RU alvo (4000 RU) para o ligante PD-1 na célula de fluxo 2. A célula de fluxo 1 foi imobilizada como um branco, produzindo uma resposta final de -161,0 RU ( Figura 2A ), enquanto a célula de fluxo 2 foi imobilizada com a proteína PD-1 biotinilada (10 μg / mL) revestida no chip SA, resultando em uma resposta final de 3698,5 RU ( Figura 2B ).
Reconhecimento de regeneração para PD-1 e PD-L1.
A respecção de regeneração foi conduzida com PD-1 imobilizado na célula de fluxo 2 e PD-L1 em solução (0,1 μM) em vários níveis de pH de glicina (1,5, 2, 2,5 e 3) para determinar a solução de regeneração, resultando em uma linha de base estável e resposta da amostra. Os dados foram analisados por meio da saída do instrumento SPR e do software de análise, resultando na glicina (pH 2,0) como a condição ideal de regeneração devido a mudanças mínimas na resposta para a linha de base e a resposta da amostra, indicando perda mínima da proteína PD-1 e remoção bem-sucedida da proteína PD-L1 da superfície do chip. No pH da glicina 2,0, a resposta basal permaneceu relativamente constante e a resposta do analito foi estável e próxima da resposta no início do experimento, indicando o tampão de regeneração mais ideal entre as quatro condições de pH da glicina testadas. Um pH mais alto é insuficiente e um pH mais baixo é muito severo; O pH 2,0 é identificado como a condição de regeneração mais adequada (Figura 3).
Validação da interação PD-1/PD-L1
Os dados foram analisados por meio do software de avaliação correspondente. Na seção Cinética/Afinidade, selecione "limite de superfície", escolha a curva 2-1, defina o modelo cinético para ligação 1:1 e ajuste o parâmetro RI para um ajuste constante para determinar a taxa de associação (ka), a taxa de dissociação (kd) e a constante de dissociação de equilíbrio (KD). A interação de ligação de PD-L1 em concentrações variadas com PD-1 foi quantificada, produzindo uma resposta mensurável (Figura 4). Os parâmetros de ligação analisados incluíram uma taxa de associação (ka) de 3,611 × 104 (1/Ms), uma taxa de dissociação (kd) de 0,0236 (1/s) e uma constante de dissociação de equilíbrio (KD) de 6,536 × 10-7 M.
Ensaio de bloqueio PD-1/PD-L1 com inibidor de molécula pequena estabelecido: BMS-1166
Os dados foram analisados por meio do correspondente Em Cinética/Afinidade, selecione limite de superfície, escolha a curva 2-1 e obtenha a curva visualizada do resultado e da unidade de resposta. O efeito de bloqueio da interação de ligação PD-1/PD-L1 foi observado com BMS-1166 (0-3,125 μM) com 0,11 μM de proteína PD-L1 em tampão HBS EP+ (Figura 5). A unidade de resposta mais alta é identificada apenas pelo PD-L1, enquanto com o aumento da concentração de BMS-1166, a unidade de resposta de ligação diminui proporcionalmente.

Figura 1: Imobilização de SA no chip CM5. As curvas de imobilização da proteína estreptavidina na célula de fluxo 1 (A) e na célula de fluxo 2 (B) do chip CM5 são mostradas. Primeiro, cinco pré-concentrações da proteína SA fluíram sobre as células de fluxo 1 e 2, seguidas por uma lavagem de NaOH e estabelecimento de uma linha de base estável. Em seguida, EDC e NHS foram adicionados e, em seguida, uma lavagem com cloridrato de etanolamina foi realizada. Em seguida, cinco pulsos de PD-1 foram realizados para atingir o nível alvo (2000 RU), seguidos por uma lavagem com cloridrato de etanolamina para remover ligantes ligados eletrostaticamente e desativar os ésteres NHS que permanecem não reativos. Clique aqui para ver uma versão maior desta figura.
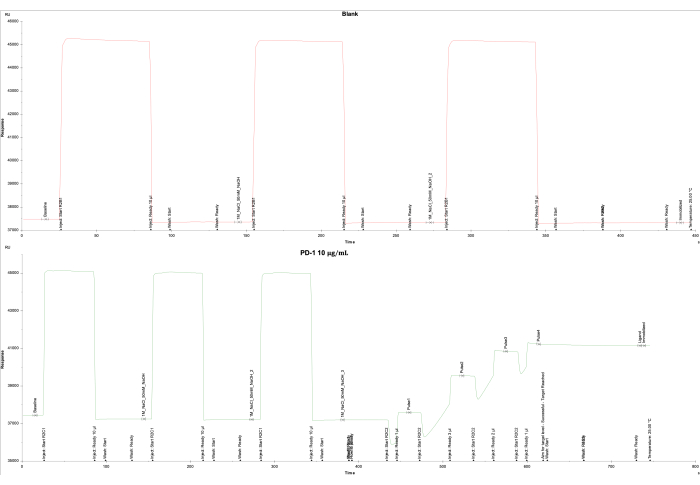
Figura 2: Imobilização de PD-1 no chip SA. As curvas de imobilização de (A) a célula de fluxo em branco 1 revestida apenas com SA e (B) a proteína PD-1 no chip SA na célula de fluxo 2 são mostradas. Para o branco (célula de fluxo 1), 1 M de NaCl e 50 mM de NaOH foram injetados três vezes, seguidos de uma lavagem com 50% de isopropanol/1 M de NaCl/50 mM de NaOH. Para a proteína PD-1 (célula de fluxo 2), 1 M de NaCl e 50 mM de NaOH foram injetados três vezes, seguidos por cinco pulsos de proteína PD-1 para atingir o nível alvo de 4000 RU e uma lavagem final de 50% de isopropanol/1 M de NaCl/50 mM de NaOH. Clique aqui para ver uma versão maior desta figura.

Figura 3: Reconhecimento de regeneração para PD-1 e PD-L1. A linha de base e a resposta da amostra foram obtidas para quatro condições de regeneração com glicina em pH 1,5, 2, 2,5 e 3 para determinar a solução de regeneração ideal antes do teste da amostra. Clique aqui para ver uma versão maior desta figura.

Figura 4: Validação da interação PD-1/PD-L1. Cinética de ligação de diferentes concentrações de PD-L1 (0,037 μM, 0,111 μM e 0,333 μM) a PD-1 na superfície do chip revestida com estreptavidina. PD-L1 demonstra associação distinta (0-60 s) e fases de dissociação (61-120 s) com PD-1. Clique aqui para ver uma versão maior desta figura.
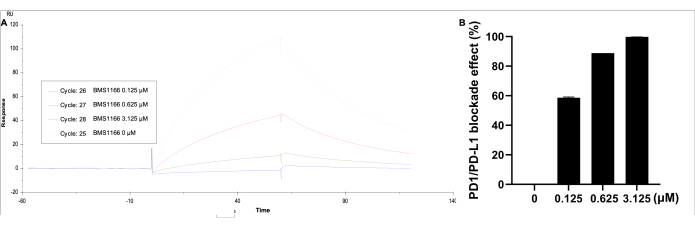
Figura 5: Análise SPR de PD-1 revestido em um chip CM5 imobilizado SA, com PD-L1 (0,11 μM) e BMS-1166 (0-3,125 μM) em solução. (A) Uma resposta SPR representativa em tempo real das reações de ligação entre PD-1 / PD-L1 com BMS-1166. (B) O efeito de bloqueio percentual BMS-1166 na cinética de ligação da interação PD-1 / PD-L1. Clique aqui para ver uma versão maior desta figura.

Figura 6: Comparação das estratégias de imobilização da proteína PD-1. Acoplamento de amina (A) vs. Acoplamento estreptavidina-biotina de duas etapas (B). O acoplamento de amina apresenta desafios, incluindo disponibilidade limitada de locais de ligação acessíveis, impedimento estérico à ligação e modificação potencial dos locais de ligação PD-1 durante a imobilização. Em contraste, a abordagem de estreptavidina-biotina de duas etapas aumenta a disponibilidade de locais de ligação livres no PD-1 imobilizado, facilitando a interação aprimorada com o PD-L1 em solução. Clique aqui para ver uma versão maior desta figura.
Discussão
Nas últimas décadas, várias abordagens de imunoterapia - incluindo vacinas contra o câncer, inibidores de checkpoint imunológico e terapias com células T CAR - avançaram significativamente no tratamento do câncer21. Os pontos de verificação imunológicos desempenham um papel crucial na prevenção de danos colaterais mediados por células imunes durante as respostas patogênicas e na supressão da autoimunidade. Um exemplo importante é a interação entre PD-L1 e PD-1, que forma um importante ponto de verificação imunológica, permitindo que as células cancerígenas evitem a vigilância imunológica. Direcionar a via PD-1 / PD-L1 com anticorpos monoclonais alcançou um sucesso notável em oncologia clínica. No entanto, devido às limitações associadas às terapias com anticorpos monoclonais e ao aumento da incidência de eventos adversos relacionados ao sistema imunológico, há um interesse crescente no desenvolvimento de inibidores de moléculas pequenas direcionados a PD-1 / PD-L1 9,22.
As estratégias atuais de triagem para inibidores de PD-1/PD-L1 de moléculas pequenas concentram-se principalmente em técnicas de bioensaio, como ELISA, ensaios repórter baseados em células e ensaios de células T. Técnicas biofísicas, incluindo SPR e interferometria de biocamada (BLI) são amplamente utilizadas para a caracterização de afinidades de ligação, mas seu potencial para serem usadas como ferramentas de triagem é subestimado23. Este estudo desenvolveu um ensaio de bloqueio PD-1 / PD-L1 otimizado baseado em SPR, que oferece uma plataforma de alto rendimento adequada para a descoberta de inibidores de PD-1 / PD-L1 de moléculas pequenas. O SA foi imobilizado a ~ 2000 RU, seguido por PD-1 biotinilado (~ 4000 RU) por meio da interação biotina-estreptavidina. Essa ligação robusta garantiu um revestimento PD-1 seguro com orientação ideal, minimizando a ligação inespecífica e reduzindo o uso de proteínas (10 μg/mL). A imobilização dirigida ao local melhorou a eficiência em comparação com os métodos convencionais. O tampão glicina (pH 2,0) foi usado para regenerar a superfície do sensor entre os ciclos, mantendo a integridade experimental e evitando ligações inespecíficas.
Em comparação com os métodos imunológicos padrão e ELISA, essa abordagem SPR ofereceu detecção em tempo real, sem marcadores, com alta sensibilidade e especificidade. Além disso, permite a triagem de alto rendimento com um tempo de execução de 120 s/amostra e complementa os bioensaios na avaliação da eficiência do bloqueio de compostos e produtos biológicos direcionados a PD-1/PD-L1. O nível de imobilização alcançado foi comparável ao da plataforma anterior (3698,5 RU vs. 3688,5 RU), com uma afinidade de interação PD-1/PD-L1 semelhante (KD = 6,536 × 10-7 M vs. 1,295 × 10-7 M). O inibidor de BMS-1166 demonstrou uma taxa de dissociação mais alta em uma concentração mais baixa, com efeitos de bloqueio comparáveis à plataforma anterior (99,8% vs. 94,2% a 3,125 μM). O BMS-1166 demonstrou taxas de bloqueio de PD-1 / PD-L1 suportadas por valores de IC50 de 1,4 nM e 96 nM em HTRF e ensaios baseados em células, respectivamente24. Além disso, outros estudos relataram valores de IC50 de 3,9 nM e 1574 nM usando HTRF e métodos de ensaio de co-cultura de bloqueio de checkpoint imunológico25. Nossos resultados anteriores mostraram que os valores de IC50 do BMS-1166 foram de 85,4 nM, o que é consistente com esses achados anteriores18. Outra vantagem desta plataforma de peneiramento é sua robustez e alto rendimento. Este método foi amplamente aplicado na triagem de alto rendimento usando um formato de placa de 384 poços, com BMS-1166 e BMS-202 incluídos como controles positivos para cada 10 amostras. As faixas de taxa de bloqueio foram de 29,8% a 38,1% para BMS-1166 e 6,0% a 10,4% para BMS-202 a 10 nM (n = 11 por placa).
É provável que o DMSO tenha efeitos variados em membranas biológicas heterogêneas, dependendo de sua composição e estrutura locais, potencialmente influenciando as funções biológicas associadas à membrana. Em concentrações relativamente baixas, o DMSO pode alterar as propriedades da proteína na solução, levando à desnaturação, agregação ou degradação. Além disso, o DMSO pode modificar as propriedades aparentes de ligação das proteínas26,27. Além disso, para abordar as preocupações anteriores sobre a interferência do DMSO, reduzimos a concentração de DMSO para 0,003% (0,01% de DMSO foi usado na plataforma anterior) neste protocolo.
Em um estudo anterior, as proteínas PD-1 foram imobilizadas em chips sensores usando acoplamento de amina, que emprega a química EDC / NHS para ativar grupos carboximetil no chip, formando ligações covalentes com grupos amina em PD-1. Devido à presença de vários grupos amina nos 288 aminoácidos do PD-1, esse método resulta inerentemente em orientações aleatórias. Para obter uma orientação mais específica, empregamos uma estratégia de acoplamento de captura com PD-1 humano biotinilado (Fc, Avitag) neste estudo. O resíduo de lisina única no Avitag é enzimaticamente biotinilado na região Fc de PD-1, permitindo uma imobilização precisa por meio da interação de alta afinidade entre biotina e estreptavidina (Figura 6). Teoricamente, essa abordagem garante que o domínio variável do PD-1 permaneça exposto, facilitando a interação ideal com o PD-L1 no buffer. No entanto, essa suposição requer validação adicional usando técnicas como crio-EM ou cristalografia para confirmar a orientação da proteína.
A glicina 2.0 foi usada como um tampão de regeneração leve, removendo efetivamente o PD-L1 sem reduzir o PD-1 imobilizado. No entanto, para inibidores fortemente ligados ou agregados de proteínas, tampões de regeneração mais fortes, como 0,5% de SDS ou 50-100 mM de NaOH, podem ser necessários para evitar interferência com amostras subsequentes.
Este estudo tem várias limitações. Primeiro, o alto custo da instrumentação SPR pode limitar a acessibilidade, embora o protocolo possa ser adaptado para outros sistemas SPR com recursos semelhantes. Parâmetros como concentração de proteínas e tempos de associação/dissociação podem ser ajustados para se adequar a plataformas SPR específicas. Outra limitação é sua dependência de proteínas recombinantes, que podem não replicar completamente as interações de proteínas nativas. Esses fatores devem ser considerados ao interpretar os dados de SPR e comparar os resultados com técnicas complementares, como ensaios baseados em células ou modelos in vivo .
Apesar dessas limitações, o método SPR otimizado oferece uma abordagem rápida, em tempo real, de alto rendimento e sem rótulos para a triagem de inibidores de moléculas pequenas da interação PD-1 / PD-L1. Ele aprimora significativamente as técnicas biofísicas para caracterizar pequenas moléculas e produtos biológicos direcionados à interação PD-1 / PD-L1. Com sua alta sensibilidade, a plataforma é particularmente valiosa para estudar interações de checkpoint imunológico e interações proteína-proteína (PPI) mais amplas, tornando-a uma ferramenta poderosa para o avanço da descoberta de medicamentos IBP.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Os autores reconhecem a instalação central do RI-INBRE na Universidade de Rhode Island, apoiada por Grant P20GM103430 do Centro Nacional de Recursos de Pesquisa (NCRR), um componente dos Institutos Nacionais de Saúde (NIH). Esta pesquisa foi apoiada por um Prêmio Piloto da Faculdade de Farmácia da Universidade de Rhode Island, um Prêmio Pequeno Prêmio do Rhode Island Life Science Hub (RILSH) e uma bolsa da Fundação Rhode Island, todos concedidos a Chang Liu, Ph.D.
Materiais
| Name | Company | Catalog Number | Comments |
| 50 mM NaOH | Cytiva Life Sciences | 100358 | |
| 50 mM NaOH | Fisher Scientific | 905376 | |
| 96-Well Polystyrene Microplates | Cytiva Life Sciences | BR100503 | |
| Amine Coupling Kit | Cytiva Life Sciences | 35120 | |
| Biacore T200 SPR System and Evaluation Software 3.2 | Cytiva Life Sciences | 28975001 | |
| Biotinylated Human PD-1 Fc, Avitag Protein | Acro Biosystems | PD1-H82F1 | |
| BMS1166 | MedChemExpress | HY-102011 | |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | 276855 | |
| DNase Free Water | Fisher Scientific | 188506 | |
| Glycine 1.5 | Cytiva Life Sciences | BR100354 | |
| Glycine 2.0 | Cytiva Life Sciences | BR100355 | |
| Glycine 2.5 | Cytiva Life Sciences | BR100356 | |
| Glycine 3.0 | Cytiva Life Sciences | BR100357 | |
| HBS-EP+ Buffer | Cytiva Life Sciences | BR100669 | |
| Human PD-L1 Fc Tag Protein | Acro Biosystems | PD-1-H5258 | |
| Isopropanol | Fisher Scientific | BP2618-1 | |
| Microplate Foil, 96-Well | Cytiva Life Sciences | 28975816 | |
| NaCl | Sigma-Aldrich | 746398 | |
| Plastic Vials 7 mm | Cytiva Life Sciences | BR100212 | |
| Rubber Caps, Type 3 | Cytiva Life Sciences | BR100502 | |
| Series S Sensor Chip CM5 | Cytiva Life Sciences | 29149603 | |
| Sodium Acetate 4.5 | Cytiva Life Sciences | 100350 | |
| Sodium Acetate 5.0 | Cytiva Life Sciences | 100351 | |
| Streptavidin | Sigma-Aldrich | S4762 |
Referências
- Ribas, A., Wolchok, J. D. Cancer immunotherapy using checkpoint blockade. Science. 359 (6382), 1350-1355 (2018).
- Zhang, X., et al. Structural and functional analysis of the costimulatory receptor programmed death-1. Immunity. 20 (3), 337-347 (2004).
- Freeman, G. J., et al. Engagement of the PD-1 immunoinhibitory receptor by a novel B7 family member leads to negative regulation of lymphocyte activation. J Exp Med. 192 (7), 1027-1034 (2000).
- Han, Y., Liu, D., Li, L. PD-1/PD-L1 pathway: Current researches in cancer. Am J Cancer Res. 10 (3), 727-742 (2020).
- Lyu, N., et al. Recognition of PDL1/L2 by different induced-fit mechanisms of PD1: A comparative study of molecular dynamics simulations. Phys Chem Chem Phys. 22 (3), 1276-1287 (2020).
- Taube, J. M., et al. Colocalization of inflammatory response with B7-H1 expression in human melanocytic lesions supports an adaptive resistance mechanism of immune escape. Sci Trans Med. 4 (127), 127ra37 (2012).
- Sun, X., et al. Immune-related adverse events associated with programmed cell death protein-1 and programmed cell death ligand 1 inhibitors for non-small cell lung cancer: A PRISMA systematic review and meta-analysis. BMC Cancer. 19 (1), 558 (2019).
- Alkholifi, F. K., Alsaffar, R. M. Dostarlimab an inhibitor of PD-1/PD-L1: A new paradigm for the treatment of cancer. Medicina. 58 (11), 1572 (2022).
- Uzar, W., et al. An updated patent review on PD-1/PD-L1 antagonists (2022-present). Expert Opin Ther Pat. 34 (8), 627-650 (2024).
- Ai, L., et al. Research status and outlook of PD-1/PD-L1 inhibitors for cancer therapy. Drug Des Devel Ther. 14, 3625-3649 (2020).
- Conroy, M., Naidoo, J. Immune-related adverse events and the balancing act of immunotherapy. Nat Commun. 13 (1), 392 (2022).
- Guzik, K., et al. Small-molecule inhibitors of the Programmed Cell Death-1/Programmed Death-Ligand 1 (PD-1/PD-L1) interaction via transiently induced protein states and dimerization of PD-L1. J Med Chem. 60 (13), 5857-5867 (2017).
- Sifniotis, V., Cruz, E., Eroglu, B., Kayser, V. Current advancements in addressing key challenges of therapeutic antibody design, manufacture, and formulation. Antibodies. 8 (2), 36 (2019).
- Beck, H., Härter, M., Haß, B., Schmeck, C., Baerfacker, L. Small molecules and their impact in drug discovery: A perspective on the occasion of the 125th anniversary of the Bayer Chemical Research Laboratory. Drug Discov Today. 27 (6), 1560-1574 (2022).
- Nguyen, H. H., Park, J., Kang, S., Kim, M. Surface plasmon resonance: A Versatile technique for biosensor applications. Sensors (Basel, Switzerland). 15 (5), 10481-10510 (2015).
- Liu, C., Seeram, N. P., Ma, H. Small molecule inhibitors against PD-1/PD-L1 immune checkpoints and current methodologies for their development: A review. Cancer Cell Int. 21 (1), 239 (2021).
- Hu, K., Li, X. -. J., Asmamaw, M. D., Shi, X. -. J., Liu, H. -. M. Establishment of high-throughput screening HTRF assay for identification small molecule inhibitors of Skp2-Cks1. Sci Rep. 11 (1), 21105 (2021).
- Puopolo, T., et al. Establishment of human PD-1/PD-L1 blockade assay based on surface plasmon resonance (SPR) biosensor. Bio-protoc. 13 (15), e4765 (2023).
- Ding, M., Chen, Y., Lang, Y., Cui, L. The role of cellular prion protein in cancer biology: A potential therapeutic target. Front Oncol. 11, 742949 (2021).
- Li, H., Seeram, N. P., Liu, C., Ma, H. Further investigation of blockade effects and binding affinities of selected natural compounds to immune checkpoint PD-1/PD-L1. Front Oncol. 12, 995461 (2022).
- Kamrani, A., et al. New immunotherapeutic approaches for cancer treatment. Pathol Res Pract. 248, 154632 (2023).
- Yan, Y., Zhang, L., Zuo, Y., Qian, H., Liu, C. Immune checkpoint blockade in cancer immunotherapy: mechanisms, clinical outcomes, and safety profiles of PD-1/PD-L1 inhibitors. Arch Immunol Ther Exp. 68 (6), 36 (2020).
- Chandrasekharan, G., Unnikrishnan, M. High throughput methods to study protein-protein interactions during host-pathogen interactions. Eur J Cell Biol. 103 (2), 151393 (2024).
- Zhang, Y., et al. BMS-202, a PD-1/PD-L1 inhibitor, decelerates the profibrotic effects of fibroblasts derived from scar tissues via ERK and TGFβ1/Smad signaling pathways. Immun Inflamm Dis. 10 (1), e591 (2022).
- Surmiak, E., et al. PD-L1 inhibitors: Different classes, activities, and mechanisms of action. Int J Mol Sci. 22 (21), 11797 (2021).
- Feoli, A., Sarno, G., Castellano, S., Sbardella, G. DMSO-Related effects on ligand-binding properties of lysine methyltransferases G9a and SETD8. ChemBioChem. 25 (4), e202300809 (2024).
- Tjernberg, A., Markova, N., Griffiths, W. J., HalléN, D. DMSO-Related effects in protein characterization. SLAS Discov. 11 (2), 131-137 (2006).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados