Method Article
표면 플라즈몬 공명 기술을 사용한 PD-1/PD-L1 억제제 식별
요약
이 프로토콜은 표면 플라즈몬 공명 기술을 사용하여 PD-1/PD-L1 억제제에 대한 차단 분석을 설명합니다. 2단계 고정 전략과 맞춤형 완충 시스템을 사용하여 반응 단위를 정확하게 측정하고 화합물 또는 생물학적 제제에 대한 차단율 평가를 용이하게 합니다. 또한 PD-1/PD-L1 억제제의 고처리량 식별을 지원합니다.
초록
PD-1/PD-L1 상호작용의 중단은 암 면역요법에 대한 유망한 전략입니다. 신뢰할 수 있는 스크리닝 플랫폼은 PD-1/PD-L1 억제제의 효능을 평가하는 데 필수적입니다. SPR(Surface Plasmon Resonance) 기술(1세대 PD-1/PD-L1 억제제 SPR 스크리닝 플랫폼)을 활용하여 이전에 확립된 인간 PD-1/PD-L1 차단 분석은 HTRF(Homogeneous Time-Resolved Fluorescence) 및 세포 기반 분석을 통해 얻은 것과 유사한 결과를 보여주었으며 대규모 스크리닝 가능성이 있습니다. 본 연구에서는 이 분석법의 최적화된 버전(2세대 PD-1/PD-L1 억제제 SPR 스크리닝 플랫폼)을 제시하며, 아민과 바이오 스트렙타비딘 결합을 결합하여 칩의 PD-1 방향 제어를 강화하고 PD-1 단백질 소비를 줄이는 이중 단계 결합 프로세스를 특징으로 합니다. 업데이트된 플랫폼은 PD-1/PD-L1 억제제 BMS-1166을 사용하여 성공적으로 검증되었으며, 이전의 SPR 기반 방법 및 ELISA와 같은 기타 확립된 기술과 유사한 차단 효과를 보여주었습니다. 이러한 결과는 접근 방식의 신뢰성을 확인합니다. 이 최적화된 SPR 스크리닝 플랫폼은 새로운 PD-1/PD-L1 억제제를 식별하고, 암 면역요법 연구를 진전시키며, 면역관문억제제 스크리닝에서 SPR의 잠재력을 강조하기 위한 고처리량의 신뢰할 수 있는 도구를 제공합니다.
서문
면역 관문 차단 요법, 특히 PD-1(Programmed Cell Death-1) 및 PD-L1(Programmed Cell Death-Ligand 1)을 표적으로 하는 요법은 암 면역 요법의 최전선에 서 있습니다. 항PD-1/PD-L1 치료제는 혈액암, 피부암, 폐암, 간암, 방광암, 신장암 등 다양한 유형의 암에 사용할 수 있도록 승인을 받았다1. PD-1은 면역글로불린 슈퍼패밀리에 속하는 막관통 당단백질로, N-말단에서 단일 면역글로불린 변수(IgV)와 유사한 도메인, 원형질막에서 IgV 도메인을 분리하는 약 20개의 아미노산 줄기, 막관통 도메인, 티로신 기반 신호 모티프를 포함하는 세포질 꼬리를 특징으로 합니다2. PD-1의 리간드 중 하나로 확인된 PD-L1은 막관통 영역, 두 개의 세포외 도메인(면역글로불린 상수(IgC) 및 IgV)과 세포 내 신호 전달 경로3을 유발하는 상대적으로 짧은 세포질 도메인을 특징으로 하는 I형 막관통 단백질입니다. PD-1/PD-L1 억제 경로는 T 세포 활성화와 자가면역을 조절하는 중요한 면역 관문 역할을 합니다4. PD-1은 T세포에서 발현되어 PD-L1과 상호작용하고, T세포 수용체 신호를 억제하며, 항원제시세포와 T세포에서 CD28 및 CD80 분자의 자극을 차단합니다5. 암 조직은 탈출 단계에서 PD-L1을 과발현하여 이러한 생리학적 메커니즘을 이용하여 종양의 성장과 진행을 촉진하는 면역억제 환경을 조성합니다6. PD-1 및 PD-L1 억제제는 이러한 상호작용을 방해하여 면역체계가 종양 유발 억제를 회피하고 T세포 매개 종양-세포 사멸 과정을 다시 시작할 수 있도록 합니다7.
면역관문차단 요법의 탁월한 역할을 바탕으로 PD-1/PD-L1 억제제의 개발은 암 면역 요법의 중요한 발전을 이루었습니다. 미국 식품의약국(FDA)은 PD-1/PD-L1 경로를 특이적으로 표적으로 하는 9가지 면역관문억제제를 승인했습니다. 여기에는 6가지 PD-1 억제제(펨브롤리주맙, 도스타리맙, 니볼루맙, 켐플리맙, 오리팔리맙, 티스렐리주맙)와 3가지 PD-L1 억제제(아테졸리주맙, 아벨루맙, 더발루맙 8,9)가 포함됩니다. 이러한 치료법은 흑색종, 폐암, 요로상피암, 자궁경부암, 위암 또는 위식도암 및 기타 고형 종양과 같은 다양한 암을 치료하는 데 효과적으로 활용되어 왔다10. 단클론 항체 기반 치료법은 그 효능에도 불구하고 낮은 반응률, 높은 비용, 반감기 연장, 심각한 면역 관련 부작용, 정맥 주사 또는 피하 전달 제한 등 상당한 한계에 직면해 있습니다 11,12,13. 이에 따라 PD-1/PD-L1 축을 표적으로 하는 저분자 억제제 개발에 대한 연구가 점점 더 집중되고 있습니다. 이러한 소분자는 세포 침투 개선, 다양한 생물학적 표적의 조절, 경구 생체이용률 향상, 비용 절감 등의 뚜렷한 이점을 제공하며, 부작용이 적으면서 유사한 치료 결과를 달성하는 것을 목표로 합니다14. 그러나 PD-1/PD-L1 상호작용을 표적으로 하는 저분자 억제제의 개발은 초기 단계에 있으며, 이는 주로 신뢰할 수 있는 고처리량 스크리닝 플랫폼이 없기 때문입니다. 이러한 플랫폼은 방대한 소분자 라이브러리를 신속하게 평가하고 추가 검증 및 최적화를 위해 선도 화합물을 식별하는 데 필수적입니다. 이러한 문제를 극복하는 것은 암 면역 요법을 발전시키는 데 매우 중요합니다.
SPR (Surface Plasmon Resonance) 기술은 항체 항원, 효소, 핵산 및 약물을 포함한 다양한 생체 분자를 검출하는 데 광범위하게 사용되며 특히 저분자 약물 스크리닝에 효과적입니다15 , 16 . 다른 생물물리학 기법과 달리 SPR은 무표지 검출, 실시간 동역학 데이터 및 광범위한 검출 범위를 제공합니다. 대조적으로, 등온 적정 열량측정법은 실시간 역학 통찰력이 부족하고 더 많은 시료 부피가 필요하여 처리량을 제한합니다. Microscale Thermophoresis는 완충 간섭이 발생하기 쉽고 역학 데이터를 제공할 수 없는 반면, Biolayer Interferometry는 분자 크기 및 특성에 따라 응용 분야별 제한이 있습니다. Homogeneous Time-Resolved Fluorescence는 라벨링이 필요하며 형광 간섭에 취약합니다. 우리는 HTRF가 PD-1/PD-L1 억제제를 탐구하는 데 적합한 또 다른 기술이라는 것을 인정합니다. SPR과 비교했을 때 HTRF의 내재적 한계 중 하나는 분자 내 여기 과정(예: 전자 전달, FRET 및 표백)과의 외부 상호 작용으로 인한 형광 소멸, 작은 창 범위로 인해 약물 스크리닝 과정에서 감도가 너무 낮다는 점, 형광 라이브러리 화합물 또는 생물학적 단백질의 간섭입니다17. 이러한 특징은 SPR을 신약 개발을 위한 우수한 도구로 자리매김하고 있습니다. 이전 연구에서는 SPR이 PD-1/PD-L1에 대한 소분자의 차단 효과를 측정할 수 있음을 입증했으며, 이는 신약 개발 과정에서 높은 라벨링 기술 요구 사항, 여러 단계, 낮은 특이성 및 높은 비용이 필요한 다른 기술에 비해 유리합니다18.
이 연구는 칩의 PD-1 배향을 강화하고 단백질 사용을 최소화하기 위해 아민과 바이오 스트렙타비딘 결합을 모두 활용하는 이중 단계 결합 프로세스를 통합하는 최적화된 SPR 기반 플랫폼을 소개합니다. 이 업데이트된 접근법은 PD-1/PD-L1 억제제 BMS-1166을 양성 대조군 바인더로 사용하여 성공적으로 검증되었으며, 이전 SPR 방법 및 ELISA19,20과 같은 다른 확립된 기술 모두에 필적하는 차단 효과를 입증했습니다. 이는 당사 프로토콜의 신뢰성과 재현성을 확인할 뿐만 아니라 PD-1/PD-L1 억제제의 고처리량 스크리닝을 촉진하는 데 있어 수정된 플랫폼의 효과를 보여줍니다. 바이오 스트렙타비딘 포획 단계를 통합하면 무작위 단백질 배향이 아닌 현장 지향을 제공하여 PD-1 농도를 낮추고(40μg/mL 대 10μg/mL) 최종 사용자가 streptavidin(SA)을 상용화된 사전 고정 SA 칩에 대한 저렴한 대안인 CM5 칩에 고정화할 수 있도록 하여 비용을 절감할 수 있습니다. 따라서 화합물/펩타이드 라이브러리의 대규모, 비용 효율적인 스크리닝에 유리합니다. 암에 대한 PD-1/PD-L1 억제제의 임상적 잠재력을 평가하기 위해서는 인실리코(in silico), 체외(in vitro) 및 생체 내 분석(in vivo assay)을 포함한 추가적인 특성 분석 방법이 필수적이지만, 당사의 향상된 SPR 기반 스크리닝 플랫폼은 PD-1/PD-L1 억제제의 대규모 스크리닝을 위한 효율적인 도구로 두드러집니다.
프로토콜
시약 및 장비는 재료 표에 나열되어 있습니다.
1. CM5 칩에서 스트렙타비딘(SA) 단백질의 고정화
- SPR 기기에서 부동화 방법을 설정합니다: Open/new wizard 템플릿, 부동화를 선택하고, 칩 유형을 CM5로 설정하고, 사이클당 플로우 셀을 1로 설정합니다. immobilize flow cell 1 및 flow cell 2를 확인합니다.
- 아민을 고정 방법으로 설정합니다. 고정 수준, 리간드 농도: 40μg/mL 스트렙타비딘, 목표 수준: 2000RU, 세척 용액: 50mM NaOH에 대한 목표 설정. 다음으로, 실행하기 전에 프라임을 확인하십시오.
- 다음 튜브를 준비하십시오 : R2 B1 및 R2 C1 - 40 μg / mL 스트렙타비딘; R2 B2 및 R2 C2 : 50 mM의 NaOH; R2 B3 및 R2 C3: EDC; R2 B4 및 R2 C4: NHS; R2 B5 및 R2 C5: 비어 있음; R2 B6 및 R2 C6: 에탄올아민.
참고: EDC와 NHS는 CM5 칩의 카르복실기를 활성화하여 리간드의 아민과 공유 결합을 가능하게 합니다. 에탄올아민은 고정화 중 비특이적 결합을 방지하기 위해 차단 완충액으로 사용됩니다. - HBS-EP+ 완충액 20mL를 탈이온수(DI) 180mL에 10× 희석하여 1× HBS-EP+ 러닝 버퍼 용액 200mL를 준비합니다.
- 스트렙타비딘 1mg에 DNase가 없는 물 1mL를 넣고 실온에서 30분 동안 배양합니다. 다음으로, 스트렙타비딘 용액을 아세테이트 완충액(pH 4.5)에서 40μg/mL로 희석합니다. 제조업체의 지침에 따라 아민 커플링 시약 키트 시약을 준비하고 1.2단계에 표시된 대로 모든 관련 시약의 레이아웃을 배치합니다.
- 유지보수 센서 칩을 CM5 칩으로 교체한 다음 튜브 A를 준비된 1× HBS-EP+ 버퍼에 넣고 고정 방법을 다시 연 다음 1.2단계 레이아웃에 따라 튜브를 삽입합니다. 메서드를 실행하고 결과 파일을 저장합니다.
- 실행 후 유지 관리: CM5 칩을 유지 보수 센서 칩으로 교체하고 튜브 A를 탈이온수가 채워진 병에 넣고 프라임을 실행합니다. CM5 칩을 꺼낸 후 DI 워터 몇 방울로 칩을 세척하고 자연 건조시킵니다. 칩을 4°C의 50mL 튜브에 넣습니다.
참고: 고정된 SA를 사용하면 CM5 칩이 SA 칩으로 작동할 수 있습니다.
2. SA 칩에서 PD-1 단백질의 고정화
- SPR 기기에서 고정 방법을 설정합니다: Open/new wizard 템플릿, 고정을 선택하고, 칩 유형을 SA로 설정하고, cycle 당 flow cells를 1로 설정합니다. immobilize flow cell 1 및 cell 2를 확인합니다.
- SA-비오틴 캡처를 Method로 설정합니다. 셀 1의 경우 빈 고정을 설정하고, 셀 2의 경우 고정 수준을 목표로 하고, 리간드로 10μg/mL PD-1을 설정하고, 목표 수준: 4000RU를 설정한 다음 실행하기 전에 프라임을 확인합니다.
참고 : 다음 튜브를 준비하십시오 - R2 B1 및 R2 C1 : NaCl 1M, NaOH 50mM; R2 B2 및 R2 C2 : 50 % 이소프로판올 / NaOH 50 mM / NaCl 1 m; R2 C3: 10μg/mL PD-1.
- SA-비오틴 캡처를 Method로 설정합니다. 셀 1의 경우 빈 고정을 설정하고, 셀 2의 경우 고정 수준을 목표로 하고, 리간드로 10μg/mL PD-1을 설정하고, 목표 수준: 4000RU를 설정한 다음 실행하기 전에 프라임을 확인합니다.
- 58.44mg의 NaCl을 1mL의 50mM NaOH에 용해시켜 1M의 NaCl과 50mM의 NaOH 용액을 준비합니다. 500μL 물에 NaCl 58.44mg과 NaOH 4.0mg을 용해한 다음 500μL의 이소프로판올을 첨가하여 50% 이소프로판올/50mM NaOH/1M NaCl 용액을 준비합니다.
- PD-1 용액 준비: 100μg의 Biotinylated Human PD-1(Fc 및 Avitagged)에 DNase가 없는 물 200μL를 추가하고 실온에서 30분 동안 안정화한 다음 아세테이트 완충액(pH 5.0)에서 10μg/mL로 희석합니다.
- 2.1단계에서 설명한 대로 모든 시약 레이아웃을 배치합니다. 튜브 A를 1× HBS-EP+ 러닝 버퍼 용액에 넣은 다음 유지보수 센서 칩을 꺼내고 SA 칩(1단계에서 스트렙타비딘 단백질로 코팅된 CM5 칩)을 삽입합니다. 고정 방법을 다시 열고 시약 랙 2를 삽입하고 위치를 확인한 다음 예상 실행 시간에 대해 방법을 실행합니다.
- 1.6단계를 반복합니다.
3. PD-1 및 PD-L1 재생 정찰
- 재생 스카우팅 방법 설정: Open/new wizard 템플릿, Regeneration Scout 선택, 흐름 경로 설정: 2-1, 4-3, 칩 유형을 SA로 설정, 실행 컨디셔닝 주기 확인, 용액을 HBS-EP+로 기록, 접촉 시간 30초, 주입 횟수 3회, 용액을 PD-L1 단위, 접촉 시간: 30초, 유속: 30μL/min.
- 재생 매개변수의 경우 유속은 30μL/min이고 안정화 기간은 300초입니다. 실험 설계에서 조건 수를 4로 설정하고 각 조건에 대한 주기 수를 2로 설정합니다. 조건을 4, 재생 용액 : 글리신 1.5, 2.0, 2.5, 3.0, 접촉 시간 : 30 초로 설정 한 다음 실행하기 전에 프라임을 확인하십시오.
- 96웰 마이크로플레이트 레이아웃에서 각 PD-L1 농도를 별도의 샘플 웰 위치로 설정: R1 A1 - R1 A9: 1μM PD-L1; R1 A10 - R1 A12: HBS-EP+ 버퍼; R1 B1 : 글리신 1.5; R1 B2 : 글리신 2; R1 B3 : 글리신 2.5; R1 B4: 글리신 3.
참고: pH가 다른 글리신 완충액이 재생 완충액으로 사용됩니다. - PD-L1 용액 준비: 100μg의 Human PD-L1 Fc Tag 단백질(9.42μM)에 DNase가 없는 물 200μL를 첨가한 다음 HBS-EP+ 완충액에서 1μM로 희석합니다.
- 3.2단계에서 설명한 대로 레이아웃에 모든 관련 시약을 배치합니다. 튜브 A를 1× HBS-EP+ 버퍼에 넣은 다음 유지보수 센서 칩을 SA 칩으로 교체합니다. Regeneration Scouting Method를 다시 열고, 3.2단계의 튜브 위치를 따르고, 시약 랙을 삽입한 다음 예상 실행 시간 동안 Method를 실행합니다 .
- 1.6단계를 반복합니다.
4. PD-1/PD-L1 상호 작용의 검증
참고: 검증을 위해 이전에 발표된 보고서18 다음에 약간의 조정이 이루어졌습니다.
- General Settings(일반 설정), Assay Steps(분석 단계) 및 Cycle Types(주기 유형)에서 게시된 보고서와 동일한 파라미터를 사용하고, 접촉 시간을 60초로, 해리 시간을 60초로, 유속을 30μL/min으로 설정합니다. Method 변수, 평가 변수 및 명령에서 Glycine 2.0을 재생으로 사용하는 것을 제외하고는 동일한 설정을 사용하고, 유속을 30μL/min으로 설정하고, 1, 2, 3, 4의 유동 경로를 통과합니다.
- 그런 다음 실행을 설정하고 흐름 경로( 2-1, 4-3)를 선택합니다. 0 μM, 0.037 μM, 0.111 μM, 0.333 μM 농도 및 51,300 Da의 분자량을 가진 PD-L1을 입력합니다. 검증을 위해 모든 분석 단계를 확인하고 실행 전에 prime을 선택합니다.
- 그런 다음 각 PD-L1 농도를 시약 랙 2 레이아웃에서 별도의 샘플 웰 위치로 설정합니다: R2 B1: PD-L1 0μM; R2 B2 : PD-L1 0.037 μM; R2 B3 : PD-L1 0.111 μM; R2 B4 : PD-L1 0.333 μM; R2 A1 : 재생 완충액으로서의 글리신 2.0; R2 A2: HBS-EP+를 시작 버퍼로 사용합니다.
- 200mL의 1× HBS-EP+ 러닝 버퍼 용액을 준비합니다. PD-L1 농도 준비: PD-L1 단백질을 9.42μM에서 0.333μM, 0.111μM 및 0.037μM로 HBS-EP+에 희석합니다. 그런 다음 4.1.2단계에서 설명한 대로 레이아웃에 모든 관련 시약을 배치합니다.
- 튜브 A를 1× HBS-EP+ 러닝 버퍼 용액에 삽입하고 유지보수 센서 칩을 배출한 다음 SA 칩을 삽입합니다. 재생 스카우팅 방법을 다시 열고, 4.1단계의 튜브 위치를 따르고, 시약 랙 2를 삽입하고, 예상 기간 동안 방법을 실행합니다 .
- 1.6단계를 반복합니다.
5. 저분자 억제제를 사용한 PD-1/PD-L1 봉쇄 분석: BMS-1166
참고: 봉쇄 분석의 경우, 이전에 발표된 보고서18 에 약간의 조정이 가해졌습니다.
- General Settings(일반 설정), Assay Steps(분석 단계) 및 Cycle Types(주기 유형)에서 게시된 보고서와 동일한 파라미터를 사용하고, 접촉 시간을 60초로, 해리 시간을 60초로, 유속을 30μL/min으로 설정합니다. 분석법 변수에서 평가 변수 및 명령도 동일한 설정을 사용하지만, Glycine 2.0을 재생으로 사용하는 것을 제외하고는 유속을 30μL/min으로 설정하고 1, 2, 3, 4의 유동 경로를 통과합니다.
- 그런 다음 실행을 설정하고 흐름 경로( 2-1, 4-3)를 선택합니다. 샘플 용액 입력: PD-L1(0.111 μM, 분자량 51,300 Da)과 BMS-1166을 0 μM, 0.125 μM, 0.625 μM, 3.125 μM에서 입력합니다. 검증을 위해 모든 분석 단계를 확인하고 실행 전에 prime을 선택합니다.
- 그런 다음 각 PD-L1 농도를 시약 랙 2 레이아웃에서 별도의 샘플 웰 위치로 설정합니다: R2 B1: PD-L1(0.111μM) + BMS-1166 0μM R2 B2: PD-L1(0.111μM) + BMS-1166 0.125μM; R2 B3 : PD-L1 (0.111 μM) + BMS-1166 0.625 μM; R2 B4 : PD-L1 (0.111 μM) + BMS-1166 3.125 μM; R2 A1: 재생을 위한 글리신 2.0.
- 200mL의 1× HBS-EP+ 러닝 버퍼 용액을 준비합니다. BMS-1166/PD-L1 혼합물 준비: 77.99μL 디메틸 설폭사이드(DMSO)에 5mg의 BMS-1166을 용해시켜 100mM 원액을 준비합니다. PD-L1 단백질(0.11μM)이 함유된 원액을 HBS-EP+에서 0μM, 0.125μM, 0.625μM 및 3.125μM에서 BMS-1166의 목표 농도로 희석합니다. 5.1.2단계에 설명된 대로 모든 관련 시약 레이아웃을 배치합니다.
- 튜브 A를 1× HBS-EP+ 러닝 버퍼 용액에 넣은 다음 유지보수 센서 칩을 꺼내고 SA 칩을 삽입합니다. 재생 스카우팅 방법을 다시 열고 5.1부터 튜브 위치를 따르고 시약 랙 2를 삽입한 다음 예상 실행 시간 동안 방법을 실행합니다 .
- 1.6단계를 반복합니다.
결과
CM5 칩에서 SA의 고정화
SPR 기기 및 관련 분석 소프트웨어의 출력을 통해 데이터를 분석했으며, 이는 플로우 셀 1 및 플로우 셀 2에서 SA 단백질의 목표 RU(2000RU)가 성공적으로 달성되었음을 나타냅니다. 플로우 셀 1과 2는 플로우 셀 1에서 1902.3RU(그림 1A), 플로우 셀 2에서 1900.7RU의 최종 반응으로 CM5 칩 표면에서 SA(40μg/mL)로 고정되었습니다(그림 1B).
SA 칩에서 PD-1의 고정화
SPR 기기 및 관련 소프트웨어의 출력을 기반으로 한 데이터 분석은 플로우 셀 1의 blank cell에 대해 낮은 반응 단위(RU)와 flow cell 2에서 PD-1 리간드에 대한 목표 RU(4000RU)의 성공적인 달성을 나타냈습니다. 플로우 셀 1은 블랭크로 고정되어 -161.0RU의 최종 반응을 보였으며(그림 2A), 플로우 셀 2는 SA 칩에 코팅된 비오틴화된 PD-1 단백질(10μg/mL)로 고정되어 최종 반응은 3698.5RU였습니다(그림 2B).
PD-1 및 PD-L1에 대한 재생 정찰.
재생 스카우팅은 PD-1을 플로우 셀 2에 고정하고 PD-L1을 다양한 글리신 pH 수준(1.5, 2, 2.5 및 3)에서 용액(0.1μM)에 담아 안정적인 베이스라인 및 샘플 반응을 도출하는 재생 용액을 측정하기 위해 수행했습니다. SPR 기기 및 분석 소프트웨어의 출력을 통해 데이터를 분석한 결과, 베이스라인 및 샘플 반응 모두에 대한 반응의 변화가 최소화되어 최적의 재생 조건으로 글리신(pH 2.0)이 생성되었으며, 이는 PD-1 단백질 손실이 최소화되고 칩 표면에서 PD-L1 단백질이 성공적으로 제거되었음을 나타냅니다. 글리신 pH 2.0에서 기준선 반응은 비교적 일정하게 유지되었고 분석물 반응은 안정적이었으며 실험 시작 시 반응에 가까웠으며, 이는 테스트된 4가지 글리신 pH 조건 중 가장 최적의 재생 완충액을 나타냅니다. 높은 pH는 충분하지 않고 낮은 pH는 너무 가혹합니다. pH 2.0이 가장 적합한 재생 조건으로 확인되었습니다(그림 3).
PD-1/PD-L1 상호 작용 검증
데이터는 해당 평가 소프트웨어를 사용하여 분석되었습니다. Kinetics/Affinity 섹션에서 "surface-bound"를 선택하고, 곡선 2-1을 선택하고, kinetics 모델을 1:1 바인딩으로 설정하고, RI 매개변수를 상수 적합으로 조정하여 association rate(ka), dissociation rate(kd) 및 equilibrium dissociation constant(KD)를 결정합니다. 다양한 농도에서 PD-L1과 PD-1의 결합 상호작용을 정량화하여 측정 가능한 반응을 생성했습니다(그림 4). 분석된 결합 매개변수에는 3.611 × 104(1/Ms)의 연관률(ka), 0.0236(1/s)의 해리율(kd) 및 6.536 × 10-7M의 평형 해리 상수(KD)가 포함되었습니다.
확립된 소분자 억제제를 사용한 PD-1/PD-L1 봉쇄 분석: BMS-1166
데이터는 해당 자료를 사용하여 분석하였다. Kinetics/Affinity(역학/친화도)에서 surface-bound(표면 경계)를 선택하고 곡선 2-1을 선택한 다음 결과 및 응답 단위의 시각화된 곡선을 얻습니다. PD-1/PD-L1 결합 상호작용의 차단 효과는 HBS EP+ 완충액에서 0.11μM PD-L1 단백질과 함께 BMS-1166(0-3.125μM)에서 관찰되었습니다(그림 5). 가장 높은 반응 단위는 PD-L1 단독으로 식별되는 반면, BMS-1166 농도가 증가하면 결합 반응 단위가 비례적으로 감소합니다.

그림 1: CM5 칩에서 SA의 고정화. CM5 칩 플로우 셀 1(A) 및 플로우 셀 2(B)에서 스트렙타비딘 단백질의 고정 곡선이 표시되어 있습니다. 먼저, SA 단백질의 5개의 사전 농도가 플로우 셀 1과 2를 통해 흐른 다음 NaOH 세척 및 안정적인 기준선 설정이 수행되었습니다. 다음으로, EDC와 NHS를 첨가 한 다음 에탄올 아민 염산염 세척을 수행했습니다. 다음으로, 목표 수준(2000RU)에 도달하기 위해 PD-1의 5개 펄스를 수행한 다음, 에탄올아민 염산염 세척을 수행하여 정전기로 결합된 리간드를 제거하고 반응성이 없는 NHS-에스테르를 비활성화했습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
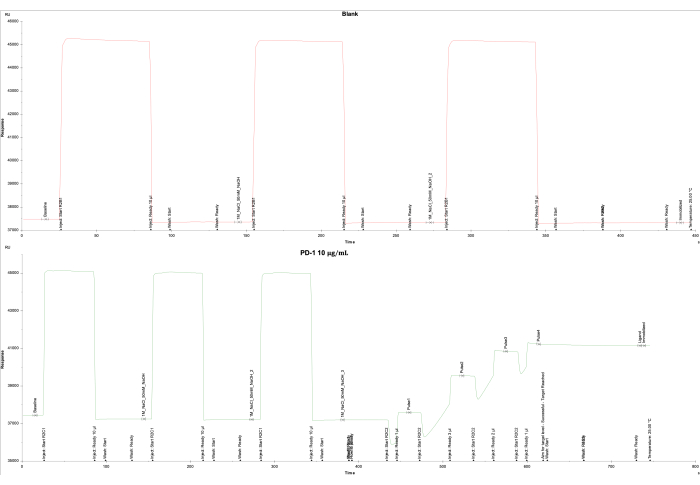
그림 2: SA 칩에서 PD-1의 고정화. (A) SA로만 코팅된 blank 플로우 셀 1 및 (B) 플로우 셀 2의 SA 칩에 있는 PD-1 단백질의 고정 곡선이 표시되어 있습니다. 블랭크(플로우 셀 1)의 경우 1M의 NaCl과 50mM의 NaOH를 3회 주입한 후 50% 이소프로판올/1M의 NaCl/50mM의 NaOH로 세척했습니다. PD-1 단백질(플로우 셀 2)의 경우 1M의 NaCl과 50mM의 NaOH를 3회 주입한 다음 PD-1 단백질의 5개 펄스를 주입하여 4000RU의 목표 수준에 도달하고 50% 이소프로판올/1M의 NaCl/50mM의 NaOH를 최종 세척했습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3: PD-1 및 PD-L1에 대한 재생 정찰. 시료 검사 전에 최적의 재생 용액을 결정하기 위해 pH 1.5, 2, 2.5 및 3에서 글리신을 사용한 4가지 재생 조건에 대해 기준선 및 샘플 반응을 얻었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 4: PD-1/PD-L1 상호 작용 검증. 스트렙타비딘 코팅된 칩 표면에서 PD-1에 다양한 농도의 PD-L1(0.037μM, 0.111μM 및 0.333μM)의 결합 동역학. PD-L1은 PD-1과 뚜렷한 연관성(0-60초) 및 해리 단계(61-120초)를 보여줍니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
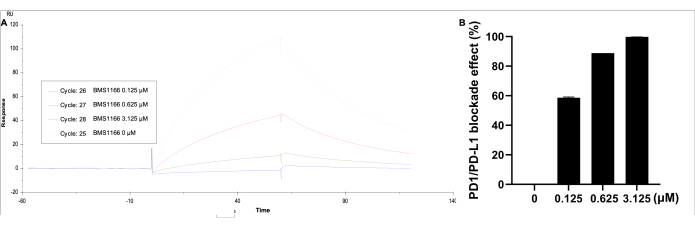
그림 5: 용액에 PD-L1(0.11μM) 및 BMS-1166(0-3.125μM)이 포함된 SA 고정 CM5 칩에 코팅된 PD-1의 SPR 분석. (A) PD-1/PD-L1과 BMS-1166 사이의 결합 반응에 대한 대표적인 실시간 SPR 응답. (B) PD-1/PD-L1 상호 작용 결합 역학에 대한 백분율 차단 효과 BMS-1166. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 6: PD-1 단백질 고정화 전략 비교. 아민 커플 링 (A) vs. 이중 단계 Streptavidin-비오틴 커플링(B). 아민 결합은 접근 가능한 결합 부위의 제한된 가용성, 결합에 대한 입체 장애, 고정 중 PD-1 결합 부위의 잠재적 변형 등의 문제를 제시합니다. 대조적으로, 이중 단계 Streptavidin-biotin 접근법은 고정된 PD-1의 자유 결합 부위의 가용성을 향상시켜 용액 내 PD-L1과의 향상된 상호 작용을 촉진합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
지난 수십 년 동안 암 백신, 면역관문억제제, CAR T세포 요법 등 다양한 면역요법 접근법이 암 치료법을 크게 발전시켰다21. 면역 관문은 병원성 반응 중 면역 세포 매개 부수적 손상을 방지하고 자가면역을 억제하는 데 중요한 역할을 합니다. 대표적인 예가 PD-L1과 PD-1의 상호작용인데, PD-1은 주요 면역 관문을 형성하여 암세포가 면역 감시를 회피할 수 있도록 합니다. 단클론 항체로 PD-1/PD-L1 경로를 표적으로 하는 것은 임상 종양학에서 놀라운 성공을 거두었습니다. 그러나 단클론 항체 치료제와 관련된 한계와 면역 관련 부작용 발생률 증가로 인해 PD-1/PD-L1을 표적으로 하는 저분자 억제제 개발에 대한 관심이 높아지고 있습니다 9,22.
저분자 PD-1/PD-L1 억제제에 대한 현재 스크리닝 전략은 주로 ELISA, 세포 기반 리포터 분석 및 T 세포 분석과 같은 생물학적 분석 기술에 중점을 두고 있습니다. SPR 및 BLI(biolayer Interferometry)를 포함한 생물물리학 기법은 결합 친화도의 특성화를 위해 널리 사용되지만, 스크리닝 도구로 사용될 가능성은 과소평가되고 있습니다23. 이 연구는 저분자 PD-1/PD-L1 억제제 발견에 적합한 고처리량 플랫폼을 제공하는 최적화된 SPR 기반 PD-1/PD-L1 차단 분석을 개발했습니다. SA는 ~2000 RU로 고정되었고, 이어서 비오틴-스트렙타비딘 상호작용을 통해 비오틴화된 PD-1(~4000 RU)이 이루어졌습니다. 이 강력한 결합은 최적의 방향으로 안전한 PD-1 코팅을 보장하여 비특이적 결합을 최소화하고 단백질 사용량(10μg/mL)을 줄였습니다. 현장 지시 고정화(site-directed immobilization)는 기존 방법에 비해 효율성을 향상시켰습니다. 글리신 완충액(pH 2.0)을 사용하여 사이클 사이에 센서 표면을 재생하여 실험적 무결성을 유지하고 비특이적 결합을 방지했습니다.
표준 면역학적 방법 및 ELISA와 비교했을 때, 이 SPR 접근법은 높은 민감도와 특이성으로 실시간 label-free 검출을 제공했습니다. 또한 120초/샘플의 런타임으로 고처리량 스크리닝을 가능하게 하고 PD-1/PD-L1 표적 화합물 및 생물학적 제제의 차단 효율을 평가할 때 생물학적 분석을 보완합니다. 달성된 고정 수준은 이전 플랫폼(3698.5RU 대 3688.5RU)과 유사했으며 PD-1/PD-L1 상호 작용 친화도(KD = 6.536 × 10-7M 대 1.295 × 10-7M )였습니다. BMS-1166 억제제는 더 낮은 농도에서 더 높은 해리율을 보였으며, 이전 플랫폼과 유사한 차단 효과(3.125μM에서 99.8% 대 94.2%)를 보였습니다. BMS-1166은 HTRF 및 세포 기반 분석에서 각각 1.4nM 및 96nM의 IC50 값이 지원하는 PD-1/PD-L1 봉쇄 속도를 보여주었습니다24. 또한, 다른 연구에서는 HTRF 및 면역 관문 봉쇄 공동 배양 분석 방법을 사용하여 3.9nM 및 1574nM의 IC50 값을 보고했습니다25. 이전 결과는 BMS-1166의 IC50 값이 85.4nM임을 보여주었으며, 이는 이러한 초기 발견과 일치합니다18. 이 스크리닝 플랫폼의 또 다른 장점은 견고함과 높은 처리량입니다. 이 방법은 384웰 플레이트 형식을 사용하는 고처리량 스크리닝에 광범위하게 적용되었으며, BMS-1166 및 BMS-202는 10개 샘플마다 양성 대조군으로 포함되었습니다. 봉쇄율 범위는 10nM(n=플레이트당 11)에서 BMS-1166의 경우 29.8%-38.1%, BMS-202의 경우 6.0%-10.4%였습니다.
DMSO는 국소 구성 및 구조에 따라 이질적인 생물학적 막에 다양한 영향을 미칠 수 있으며, 잠재적으로 막 관련 생물학적 기능에 영향을 미칠 수 있습니다. 상대적으로 낮은 농도에서 DMSO는 용액의 단백질 특성을 변경하여 변성, 응집 또는 분해를 유발할 수 있습니다. 또한, DMSO는 단백질26,27의 겉보기 결합 특성을 변형시킬 수 있습니다. 또한 DMSO 간섭에 대한 이전의 우려를 해결하기 위해 이 프로토콜에서 DMSO 농도를 0.003%(이전 플랫폼에서는 0.01% DMSO가 사용됨)로 줄였습니다.
이전 연구에서 PD-1 단백질은 EDC/NHS 화학을 사용하여 칩의 카르복시메틸기를 활성화하고 PD-1의 아민기와 공유 결합을 형성하는 아민 커플링을 사용하여 센서 칩에 고정되었습니다. PD-1의 288개 아미노산에 걸쳐 여러 아민기가 존재하기 때문에 이 방법은 본질적으로 임의의 방향을 초래합니다. 보다 구체적인 방향을 달성하기 위해 이 연구에서는 비오틴화된 인간 PD-1(Fc, Avitag)과 포획 결합 전략을 사용했습니다. Avitag의 단일 lysine 잔류물은 PD-1의 Fc 영역에서 효소를 통해 비오틴화되어 비오틴과 스트렙타비딘 간의 높은 친화성 상호 작용을 통해 정확한 고정을 가능하게 합니다(그림 6). 이론적으로 이 접근 방식은 PD-1의 가변 영역이 노출된 상태로 유지되도록 하여 버퍼에서 PD-L1과의 최적의 상호 작용을 촉진합니다. 그러나 이러한 가정을 위해서는 단백질 배향을 확인하기 위해 Cryo-EM 또는 결정학과 같은 기술을 사용한 추가 검증이 필요합니다.
글리신 2.0은 가벼운 재생 완충액으로 사용되어 고정된 PD-1을 감소시키지 않고 PD-L1을 효과적으로 제거했습니다. 그러나 단단히 결합된 억제제 또는 단백질 응집체의 경우 후속 샘플과의 간섭을 방지하기 위해 0.5% SDS 또는 50-100mM의 NaOH와 같은 더 강력한 재생 완충액이 필요할 수 있습니다.
이 연구에는 몇 가지 제한 사항이 있습니다. 첫째, SPR 계측의 높은 비용으로 인해 접근성이 제한될 수 있지만, 이 프로토콜은 종종 유사한 기능을 가진 다른 SPR 시스템에 맞게 조정될 수 있습니다. 단백질 농도 및 결합/해리 시간과 같은 매개변수는 특정 SPR 플랫폼에 맞게 조정할 수 있습니다. 또 다른 한계는 재조합 단백질에 의존한다는 것인데, 이는 네이티브 단백질 상호 작용을 완전히 복제하지 못할 수 있습니다. SPR 데이터를 해석하고 결과를 세포 기반 분석 또는 생체 내 모델과 같은 보완 기술과 비교할 때 이러한 요소를 고려해야 합니다.
이러한 한계에도 불구하고 최적화된 SPR 방법은 PD-1/PD-L1 상호 작용의 저분자 억제제를 스크리닝하기 위한 빠른 실시간, 고처리량 및 label-free 접근법을 제공합니다. PD-1/PD-L1 상호작용을 표적으로 하는 소분자 및 생물학적 제제를 특성화하기 위한 생물물리학적 기술을 크게 향상시킵니다. 민감도가 높은 이 플랫폼은 면역 관문 상호 작용 및 광범위한 단백질-단백질 상호 작용(PPI)을 연구하는 데 특히 유용하므로 PPI 약물 발견을 진전시키기 위한 강력한 도구입니다.
공개
저자는 공개할 내용이 없습니다.
감사의 말
저자들은 미국 국립보건원(NIH) 산하 기관인 국립연구자원센터(National Center for Research Resources, NCRR)의 그랜트 P20GM103430(Grant )의 지원을 받는 로드아일랜드대학교(University of Rhode Island)의 RI-INBRE 핵심 시설을 인정한다. 이 연구는 로드 아일랜드 대학교 약학 대학의 파일럿 그랜트 상, 로드 아일랜드 생명 과학 허브(RILSH)의 소규모 그랜트 상, 로드 아일랜드 재단 보조금의 지원을 받았으며, 모두 Chang Liu 박사에게 수여되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| 50 mM NaOH | Cytiva Life Sciences | 100358 | |
| 50 mM NaOH | Fisher Scientific | 905376 | |
| 96-Well Polystyrene Microplates | Cytiva Life Sciences | BR100503 | |
| Amine Coupling Kit | Cytiva Life Sciences | 35120 | |
| Biacore T200 SPR System and Evaluation Software 3.2 | Cytiva Life Sciences | 28975001 | |
| Biotinylated Human PD-1 Fc, Avitag Protein | Acro Biosystems | PD1-H82F1 | |
| BMS1166 | MedChemExpress | HY-102011 | |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | 276855 | |
| DNase Free Water | Fisher Scientific | 188506 | |
| Glycine 1.5 | Cytiva Life Sciences | BR100354 | |
| Glycine 2.0 | Cytiva Life Sciences | BR100355 | |
| Glycine 2.5 | Cytiva Life Sciences | BR100356 | |
| Glycine 3.0 | Cytiva Life Sciences | BR100357 | |
| HBS-EP+ Buffer | Cytiva Life Sciences | BR100669 | |
| Human PD-L1 Fc Tag Protein | Acro Biosystems | PD-1-H5258 | |
| Isopropanol | Fisher Scientific | BP2618-1 | |
| Microplate Foil, 96-Well | Cytiva Life Sciences | 28975816 | |
| NaCl | Sigma-Aldrich | 746398 | |
| Plastic Vials 7 mm | Cytiva Life Sciences | BR100212 | |
| Rubber Caps, Type 3 | Cytiva Life Sciences | BR100502 | |
| Series S Sensor Chip CM5 | Cytiva Life Sciences | 29149603 | |
| Sodium Acetate 4.5 | Cytiva Life Sciences | 100350 | |
| Sodium Acetate 5.0 | Cytiva Life Sciences | 100351 | |
| Streptavidin | Sigma-Aldrich | S4762 |
참고문헌
- Ribas, A., Wolchok, J. D. Cancer immunotherapy using checkpoint blockade. Science. 359 (6382), 1350-1355 (2018).
- Zhang, X., et al. Structural and functional analysis of the costimulatory receptor programmed death-1. Immunity. 20 (3), 337-347 (2004).
- Freeman, G. J., et al. Engagement of the PD-1 immunoinhibitory receptor by a novel B7 family member leads to negative regulation of lymphocyte activation. J Exp Med. 192 (7), 1027-1034 (2000).
- Han, Y., Liu, D., Li, L. PD-1/PD-L1 pathway: Current researches in cancer. Am J Cancer Res. 10 (3), 727-742 (2020).
- Lyu, N., et al. Recognition of PDL1/L2 by different induced-fit mechanisms of PD1: A comparative study of molecular dynamics simulations. Phys Chem Chem Phys. 22 (3), 1276-1287 (2020).
- Taube, J. M., et al. Colocalization of inflammatory response with B7-H1 expression in human melanocytic lesions supports an adaptive resistance mechanism of immune escape. Sci Trans Med. 4 (127), 127ra37 (2012).
- Sun, X., et al. Immune-related adverse events associated with programmed cell death protein-1 and programmed cell death ligand 1 inhibitors for non-small cell lung cancer: A PRISMA systematic review and meta-analysis. BMC Cancer. 19 (1), 558 (2019).
- Alkholifi, F. K., Alsaffar, R. M. Dostarlimab an inhibitor of PD-1/PD-L1: A new paradigm for the treatment of cancer. Medicina. 58 (11), 1572 (2022).
- Uzar, W., et al. An updated patent review on PD-1/PD-L1 antagonists (2022-present). Expert Opin Ther Pat. 34 (8), 627-650 (2024).
- Ai, L., et al. Research status and outlook of PD-1/PD-L1 inhibitors for cancer therapy. Drug Des Devel Ther. 14, 3625-3649 (2020).
- Conroy, M., Naidoo, J. Immune-related adverse events and the balancing act of immunotherapy. Nat Commun. 13 (1), 392 (2022).
- Guzik, K., et al. Small-molecule inhibitors of the Programmed Cell Death-1/Programmed Death-Ligand 1 (PD-1/PD-L1) interaction via transiently induced protein states and dimerization of PD-L1. J Med Chem. 60 (13), 5857-5867 (2017).
- Sifniotis, V., Cruz, E., Eroglu, B., Kayser, V. Current advancements in addressing key challenges of therapeutic antibody design, manufacture, and formulation. Antibodies. 8 (2), 36 (2019).
- Beck, H., Härter, M., Haß, B., Schmeck, C., Baerfacker, L. Small molecules and their impact in drug discovery: A perspective on the occasion of the 125th anniversary of the Bayer Chemical Research Laboratory. Drug Discov Today. 27 (6), 1560-1574 (2022).
- Nguyen, H. H., Park, J., Kang, S., Kim, M. Surface plasmon resonance: A Versatile technique for biosensor applications. Sensors (Basel, Switzerland). 15 (5), 10481-10510 (2015).
- Liu, C., Seeram, N. P., Ma, H. Small molecule inhibitors against PD-1/PD-L1 immune checkpoints and current methodologies for their development: A review. Cancer Cell Int. 21 (1), 239 (2021).
- Hu, K., Li, X. -. J., Asmamaw, M. D., Shi, X. -. J., Liu, H. -. M. Establishment of high-throughput screening HTRF assay for identification small molecule inhibitors of Skp2-Cks1. Sci Rep. 11 (1), 21105 (2021).
- Puopolo, T., et al. Establishment of human PD-1/PD-L1 blockade assay based on surface plasmon resonance (SPR) biosensor. Bio-protoc. 13 (15), e4765 (2023).
- Ding, M., Chen, Y., Lang, Y., Cui, L. The role of cellular prion protein in cancer biology: A potential therapeutic target. Front Oncol. 11, 742949 (2021).
- Li, H., Seeram, N. P., Liu, C., Ma, H. Further investigation of blockade effects and binding affinities of selected natural compounds to immune checkpoint PD-1/PD-L1. Front Oncol. 12, 995461 (2022).
- Kamrani, A., et al. New immunotherapeutic approaches for cancer treatment. Pathol Res Pract. 248, 154632 (2023).
- Yan, Y., Zhang, L., Zuo, Y., Qian, H., Liu, C. Immune checkpoint blockade in cancer immunotherapy: mechanisms, clinical outcomes, and safety profiles of PD-1/PD-L1 inhibitors. Arch Immunol Ther Exp. 68 (6), 36 (2020).
- Chandrasekharan, G., Unnikrishnan, M. High throughput methods to study protein-protein interactions during host-pathogen interactions. Eur J Cell Biol. 103 (2), 151393 (2024).
- Zhang, Y., et al. BMS-202, a PD-1/PD-L1 inhibitor, decelerates the profibrotic effects of fibroblasts derived from scar tissues via ERK and TGFβ1/Smad signaling pathways. Immun Inflamm Dis. 10 (1), e591 (2022).
- Surmiak, E., et al. PD-L1 inhibitors: Different classes, activities, and mechanisms of action. Int J Mol Sci. 22 (21), 11797 (2021).
- Feoli, A., Sarno, G., Castellano, S., Sbardella, G. DMSO-Related effects on ligand-binding properties of lysine methyltransferases G9a and SETD8. ChemBioChem. 25 (4), e202300809 (2024).
- Tjernberg, A., Markova, N., Griffiths, W. J., HalléN, D. DMSO-Related effects in protein characterization. SLAS Discov. 11 (2), 131-137 (2006).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유