Method Article
Identificazione degli inibitori PD-1/PD-L1 con la tecnologia di risonanza plasmonica di superficie
In questo articolo
Riepilogo
Questo protocollo descrive un test di blocco per inibitori di PD-1/PD-L1 utilizzando la tecnologia di risonanza plasmonica di superficie. Impiega una strategia di immobilizzazione in due fasi e un sistema tampone su misura per misurare con precisione le unità di risposta, facilitando la valutazione dei tassi di blocco per composti o farmaci biologici. Inoltre, supporta l'identificazione ad alto rendimento degli inibitori PD-1/PD-L1.
Abstract
L'interruzione dell'interazione PD-1/PD-L1 è una strategia promettente per l'immunoterapia del cancro. Piattaforme di screening affidabili sono essenziali per valutare l'efficacia degli inibitori di PD-1/PD-L1. Un test di blocco PD-1/PD-L1 umano precedentemente stabilito che utilizza la tecnologia di risonanza plasmonica di superficie (SPR) (piattaforma di screening SPR inibitore PD-1/PD-L1 di prima generazione) ha dimostrato risultati paragonabili a quelli ottenuti attraverso la fluorescenza omogenea risolta nel tempo (HTRF) e i saggi basati su cellule, con un potenziale per lo screening su larga scala. In questo articolo, viene presentata una versione ottimizzata di questo test (piattaforma di screening SPR con inibitore PD-1/PD-L1 di seconda generazione), caratterizzata da un processo di accoppiamento a doppia fase che combina l'accoppiamento di ammina e bio-streptavidina per migliorare il controllo dell'orientamento PD-1 sul chip e ridurre il consumo di proteina PD-1. La piattaforma aggiornata è stata convalidata con successo utilizzando l'inibitore PD-1/PD-L1 BMS-1166, mostrando effetti di blocco paragonabili al precedente metodo basato su SPR e ad altre tecniche consolidate come l'ELISA. Questi risultati confermano l'affidabilità dell'approccio. Questa piattaforma ottimizzata per lo screening SPR offre uno strumento affidabile e ad alto rendimento per identificare nuovi inibitori di PD-1/PD-L1, far progredire la ricerca sull'immunoterapia del cancro ed evidenziare il potenziale di SPR nello screening degli inibitori del checkpoint immunitario.
Introduzione
Le terapie di blocco dei checkpoint immunitari, in particolare quelle mirate alla morte cellulare programmata-1 (PD-1) e al ligando 1 della morte cellulare programmata (PD-L1), sono all'avanguardia nelle strategie di immunoterapia del cancro. Le terapie anti-PD-1/PD-L1 hanno ricevuto l'approvazione per l'uso in vari tipi di cancro, come i tumori ematologici, cutanei, polmonari, epatici, della vescica urinaria e renali1. PD-1 è una glicoproteina transmembrana appartenente alla superfamiglia delle immunoglobuline, caratterizzata da un singolo dominio immunoglobulina variabile (IgV) al N-terminale, un gambo di circa 20 amminoacidi che separa il dominio IgV dalla membrana plasmatica, un dominio transmembrana e una coda citoplasmatica contenente motivi di segnalazione a base di tirosina2. PD-L1, identificata come uno dei ligandi per PD-1, è una proteina transmembrana di tipo I caratterizzata da una regione transmembrana, due domini extracellulari - costante delle immunoglobuline (IgC) e IgV - e un dominio citoplasmatico relativamente corto che innesca le vie di segnalazione intracellulare3. La via inibitoria PD-1/PD-L1 funge da checkpoint immunitario critico che regola l'attivazione delle cellule T e l'autoimmunità4. PD-1 è espresso sulle cellule T, dove interagisce con PD-L1, inibisce la segnalazione del recettore delle cellule T e blocca la stimolazione delle molecole CD28 e CD80 sulle cellule presentanti l'antigene e sulle cellule T5. I tessuti tumorali sfruttano questo meccanismo fisiologico sovraesprimendo PD-L1 durante la fase di fuga, creando così un ambiente immunosoppressivo che promuove la crescita e la progressione del tumore6. Gli inibitori di PD-1 e PD-L1 interrompono questa interazione, consentendo al sistema immunitario di eludere la soppressione indotta dal tumore e di riavviare il processo di morte delle cellule tumorali mediato dalle cellule T7.
Basandosi sul ruolo di primo piano delle terapie di blocco dei checkpoint immunitari, lo sviluppo degli inibitori PD-1/PD-L1 ha segnato un progresso significativo nell'immunoterapia del cancro. La Food and Drug Administration (FDA) degli Stati Uniti ha approvato nove inibitori del checkpoint immunitario che mirano specificamente alla via PD-1/PD-L1. Questi includono sei inibitori PD-1 - pembrolizumab, dostarlimab, nivolumab, cemiplimab, oripalimab e tislelizumab - e tre inibitori PD-L1 - atezolizumab, avelumab e durvalumab 8,9. Queste terapie sono state efficacemente utilizzate per trattare una varietà di tumori, come il melanoma, il cancro ai polmoni, il cancro uroteliale, il cancro cervicale, il cancro gastrico o gastroesofageo e altri tumori solidi10. Nonostante la loro efficacia, le terapie a base di anticorpi monoclonali devono affrontare limitazioni significative, tra cui bassi tassi di risposta, costi elevati, emivite prolungate, gravi eventi avversi immuno-correlati e restrizioni alla somministrazione endovenosa o sottocutanea 11,12,13. Di conseguenza, la ricerca è sempre più focalizzata sullo sviluppo di inibitori di piccole molecole che hanno come bersaglio l'asse PD-1/PD-L1. Queste piccole molecole offrono vantaggi distinti, come una migliore penetrazione cellulare, la modulazione di diversi bersagli biologici, una maggiore biodisponibilità orale e costi ridotti, con l'obiettivo di ottenere risultati terapeutici comparabili con meno effetti avversi14. Tuttavia, lo sviluppo di inibitori di piccole molecole che hanno come bersaglio l'interazione PD-1/PD-L1 è nelle sue fasi iniziali, principalmente a causa della mancanza di una piattaforma di screening affidabile ad alto rendimento. Tali piattaforme sono essenziali per valutare rapidamente vaste librerie di piccole molecole e identificare composti guida per un'ulteriore convalida e ottimizzazione. Superare questa sfida è fondamentale per far progredire l'immunoterapia del cancro.
La tecnologia di risonanza plasmonica di superficie (SPR) è ampiamente impiegata nel rilevamento di varie biomolecole, tra cui antigeni anticorpali, enzimi, acidi nucleici e farmaci, ed è particolarmente efficace nello screening di farmaci di piccole molecole15,16. A differenza di altre tecniche biofisiche, SPR offre un rilevamento senza marcatura, dati cinetici in tempo reale e un ampio intervallo di rilevamento. Al contrario, la calorimetria a titolazione isotermica non dispone di informazioni cinetiche in tempo reale e richiede volumi di campione maggiori, limitando la produttività. La termoforesi su microscala è soggetta a interferenze tampone e non è in grado di fornire dati cinetici, mentre l'interferometria a biostrati presenta limitazioni specifiche per l'applicazione in base alle dimensioni e alle proprietà molecolari. La fluorescenza omogenea risolta nel tempo richiede la marcatura ed è suscettibile alle interferenze fluorescenti. Riconosciamo che l'HTRF è un'altra tecnologia adatta per esplorare gli inibitori di PD-1/PD-L1. Una limitazione intrinseca dell'HTRF, rispetto all'SPR, è l'estinzione della fluorescenza causata da interazioni esterne con il processo di eccitazione intramolecolare (ad esempio, trasferimento di elettroni, FRET e sbiancamento), la sensibilità è troppo bassa nel processo di screening dei farmaci a causa del piccolo intervallo di finestre e l'interferenza di composti di librerie fluorescenti o proteine biologiche17. Queste caratteristiche posizionano l'SPR come uno strumento superiore per la scoperta di farmaci. I nostri studi precedenti hanno dimostrato che l'SPR è in grado di determinare l'effetto di blocco di piccole molecole contro PD-1/PD-L1, il che è vantaggioso rispetto ad altre tecniche che richiedono elevati requisiti di tecnologia di etichettatura, passaggi multipli, scarsa specificità e costi elevati nel processo di scoperta di farmaci18.
Questo studio introduce una piattaforma ottimizzata basata su SPR, che integra un processo di accoppiamento a doppia fase che utilizza sia l'accoppiamento amminico che quello bio-streptavidina per migliorare l'orientamento di PD-1 sul chip e ridurre al minimo l'uso di proteine. Questo approccio aggiornato è stato convalidato con successo utilizzando l'inibitore PD-1/PD-L1 BMS-1166 come legante di controllo positivo, dimostrando effetti di blocco paragonabili sia al nostro precedente metodo SPR che ad altre tecniche consolidate come ELISA19,20. Questo non solo conferma l'affidabilità e la riproducibilità del nostro protocollo, ma illustra anche l'efficacia della nostra piattaforma modificata nel facilitare lo screening ad alto rendimento degli inibitori PD-1/PD-L1. L'incorporazione della fase di cattura della bio-streptavidina fornisce un orientamento della proteina diretto al sito piuttosto che casuale, consentendo una ridotta concentrazione di PD-1 (40 μg/mL rispetto a 10 μg/mL) e risparmi sui costi consentendo all'utente finale di immobilizzare la streptavidina (SA) su un chip CM5, un'alternativa meno costosa ai chip SA pre-immobilizzati commercializzati. Ciò lo rende vantaggioso per screening su larga scala ed economici di librerie di composti/peptidi. Sebbene ulteriori metodi di caratterizzazione, tra cui saggi in silico, in vitro e in vivo, siano essenziali per valutare il potenziale clinico degli inibitori di PD-1/PD-L1 contro il cancro, la nostra piattaforma di screening avanzata basata su SPR si distingue come uno strumento efficiente per lo screening su larga scala degli inibitori di PD-1/PD-L1.
Protocollo
I reagenti e le attrezzature sono elencati nella Tabella dei Materiali.
1. Immobilizzazione della proteina streptavidina (SA) sul chip CM5
- Imposta il metodo di immobilizzazione sullo strumento SPR: apri/ nuovo modello di procedura guidata , scegli l'immobilizzazione, imposta il tipo di chip su CM5 e fai scorrere le celle per ciclo su 1. Controllare l'immobilizzazione della cella di flusso 1 e della cella di flusso 2.
- Imposta Ammina come metodo di immobilizzazione. Obiettivo impostato per il livello immobilizzato, concentrazione del ligando: 40 μg/mL di streptavidina, livello target: 2000 RU, soluzione di lavaggio: 50 mM NaOH. Quindi, controlla l'adescamento prima di eseguire.
- Preparare le seguenti provette: R2 B1 e R2 C1 - 40 μg/mL di streptavidina; R2 B2 e R2 C2: 50 mM di NaOH; R2 B3 e R2 C3: EDC; R2 B4 e R2 C4: SSN; R2 B5 e R2 C5: vuoto; R2 B6 e R2 C6: Etanolammina.
NOTA: EDC e NHS attivano i gruppi carbossilici sul chip CM5, consentendo l'accoppiamento covalente con le ammine sul ligando. L'etanolammina viene utilizzata come tampone bloccante per prevenire il legame aspecifico durante l'immobilizzazione. - Diluire 20 mL di tampone HBS-EP+ 10× in 180 mL di acqua deionizzata (DI) per preparare 200 mL di soluzione tampone 1× HBS-EP+.
- Aggiungere 1 mL di acqua priva di DNasi a 1 mg di streptavidina e incubare a temperatura ambiente per 30 minuti. Successivamente, diluire la soluzione di streptavidina a 40 μg/mL in tampone acetato (pH 4,5). Preparare i reagenti del kit di reagenti Amine Coupling secondo le istruzioni del produttore e posizionare tutti i layout dei reagenti pertinenti come indicato al punto 1.2.
- Sostituire il chip del sensore di manutenzione con il chip CM5, quindi posizionare la provetta A nel tampone 1× HBS-EP+ preparato, riaprire il metodo di immobilizzazione e inserire le provette secondo il layout del passaggio 1.2. Eseguire il metodo e salvare il file dei risultati.
- Manutenzione post-corsa: sostituire il chip CM5 con il chip del sensore di manutenzione, posizionare il tubo A in una bottiglia piena di acqua deionizzata ed eseguire l'adescamento. Dopo aver estratto il chip CM5, lavare il chip con alcune gocce di acqua deionizzata e asciugarlo all'aria. Posizionare il chip in una provetta da 50 ml a 4 °C.
NOTA: L'SA immobilizzato consente al chip CM5 di funzionare come un chip SA.
2. Immobilizzazione della proteina PD-1 sul chip SA
- Impostare il metodo di immobilizzazione sullo strumento SPR: aprire/nuovo modello della procedura guidata , scegliere l'immobilizzazione, impostare il tipo di chip su SA e far scorrere le celle per ciclo su 1. Controllare immobilizzare la cella di flusso 1 e la cella 2.
- Impostare la cattura SA-biotina come metodo. Per la cella 1 impostare l'immobilizzazione in bianco, per la cella 2 puntare al livello immobilizzato, 10 μg/mL PD-1 come ligando, livello target: 4000 RU, quindi controllare l'indice prima di eseguire.
NOTA: Preparare i seguenti tubi - R2 B1 e R2 C1: 1 M di NaCl, 50 mM di NaOH; R2 B2 e R2 C2: 50% isopropanolo/50 mM di NaOH/1 m di NaCl; R2 C3: 10 μg/mL PD-1.
- Impostare la cattura SA-biotina come metodo. Per la cella 1 impostare l'immobilizzazione in bianco, per la cella 2 puntare al livello immobilizzato, 10 μg/mL PD-1 come ligando, livello target: 4000 RU, quindi controllare l'indice prima di eseguire.
- Sciogliere 58,44 mg di NaCl in 1 mL di 50 mM di NaOH per preparare 1 M di NaCl e 50 mM di soluzione di NaOH. Sciogliere 58,44 mg di NaCl e 4,0 mg di NaOH in 500 μL di acqua, quindi aggiungere 500 μL di isopropanolo per preparare la soluzione di isopropanolo al 50%/50 mM di NaOH/1 M di NaCl.
- Preparare la soluzione di PD-1: aggiungere 200 μL di acqua priva di DNasi a 100 μg di PD-1 umano biotinilato (Fc e Avitagged) e stabilizzare a temperatura ambiente per 30 minuti, quindi diluire a 10 μg/mL in tampone acetato (pH 5,0).
- Posizionare tutti i layout dei reagenti come descritto al punto 2.1. Posizionare la provetta A nella soluzione tampone di corsa HBS-EP+ 1×, quindi espellere il chip del sensore di manutenzione e inserire il chip SA (chip CM5 rivestito dalla proteina streptavidina del passaggio 1). Riaprire il metodo di immobilizzazione, inserire il rack dei reagenti 2 e controllare le posizioni, quindi eseguire il metodo per il tempo di esecuzione stimato.
- Ripetere il passaggio 1.6.
3. Scouting della rigenerazione per PD-1 e PD-L1
- Impostare il metodo di ricerca della rigenerazione: aprire/nuovo modello di procedura guidata , scegliere la ricerca della rigenerazione, impostare il percorso del flusso: 2-1, 4-3, tipo di chip su SA, controllare il ciclo di condizionamento dell'esecuzione, registrare la soluzione come HBS-EP+, tempo di contatto come 30 s, numero di iniezioni come 3, soluzione come PD-L1, tempo di contatto: 30 s, portata: 30 μL/min.
- Per i parametri di rigenerazione, la portata è di 30 μL/min e il periodo di stabilizzazione è di 300 s. Nel disegno sperimentale, impostare il numero di condizioni su 4 e il numero di cicli per ciascuna condizione su 2. Impostare le condizioni su 4, soluzione rigenerante: glicina 1,5, 2,0, 2,5, 3,0, tempi di contatto: 30 s, quindi controllare l'adescamento prima dell'esecuzione.
- Impostare ciascuna concentrazione di PD-L1 come posizione separata del pozzetto del campione in un layout di micropiastra a 96 pozzetti: da R1 A1 a R1 A9: 1 μM PD-L1; da R1 A10 a R1 A12: tampone HBS-EP+; R1 B1: Glicina 1,5; R1 B2: Glicina 2; R1 B3: Glicina 2,5; R1 B4: Glicina 3.
NOTA: Come tampone di rigenerazione viene utilizzato un tampone di glicina con pH diverso. - Preparare la soluzione di PD-L1: aggiungere 200 μL di acqua priva di DNasi a 100 μg di proteina Fc Tag umana PD-L1 (9,42 μM), quindi diluire a 1 μM in tampone HBS-EP+.
- Collocare tutti i reagenti pertinenti nel layout come descritto al punto 3.2. Posizionare la provetta A nel tampone HBS-EP+ da 1×, quindi sostituire il chip del sensore di manutenzione con il chip SA. Riaprire il metodo di ricerca della rigenerazione , seguire la posizione della provetta dal passaggio 3.2, inserire il rack dei reagenti, quindi eseguire il metodo per il tempo di esecuzione stimato.
- Ripetere il passaggio 1.6.
4. Validazione dell'interazione PD-1/PD-L1
NOTA: Per la convalida, è stato seguito un rapporto18 pubblicato in precedenza con piccoli aggiustamenti.
- Utilizzare gli stessi parametri del report pubblicato in Impostazioni generali, Fasi del saggio e Tipi di ciclo, impostare il tempo di contatto su 60 s, il tempo di dissociazione su 60 s e la velocità di flusso su 30 μL/min. In Variabili di metodo, Variabili di valutazione e Comandi, utilizzare la stessa impostazione, ad eccezione dell'utilizzo di Glicina 2.0 come Rigenerazione, impostare la velocità di flusso a 30 μL/min, passare attraverso il percorso del flusso di 1, 2, 3, 4.
- Quindi imposta la corsa e scegli il percorso del flusso: 2-1, 4-3. Input PD-L1 con concentrazioni da 0 μM, 0,037 μM, 0,111 μM, 0,333 μM e un peso molecolare di 51.300 Da. Controllare tutte le fasi del test per la verifica e selezionare il primo prima della corsa.
- Quindi impostare ciascuna concentrazione di PD-L1 come posizione separata del pozzetto del campione in un layout del rack reagenti 2: R2 B1: PD-L1 0 μM; R2 B2: PD-L1 0,037 μM; R2 B3: PD-L1 0,111 μM; R2 B4: PD-L1 0,333 μM; R2 A1: Glicina 2.0 come tampone di rigenerazione; R2 A2: HBS-EP+ come buffer di avvio.
- Preparare 200 mL di soluzione tampone 1× HBS-EP+. Preparare le concentrazioni di PD-L1: diluire la proteina PD-L1 a 9,42 μM a 0,333 μM, 0,111 μM e 0,037 μM in HBS-EP+. Posizionare quindi tutti i reagenti pertinenti nel layout come descritto al punto 4.1.2.
- Inserire la provetta A nella soluzione tampone di corsa HBS-EP+ da 1×, espellere il chip del sensore di manutenzione e inserire il chip SA. Riaprire il metodo di ricerca della rigenerazione , seguire il posizionamento della provetta dal passaggio 4.1, inserire il rack dei reagenti 2 ed eseguire il metodo per la durata stimata.
- Ripetere il passaggio 1.6.
5. Saggio di blocco PD-1/PD-L1 con inibitore di piccole molecole: BMS-1166
NOTA: Per il test del blocco, è stato seguito un rapporto18 precedentemente pubblicato con piccoli aggiustamenti.
- Utilizzare gli stessi parametri del report pubblicato in Impostazioni generali, Fasi del saggio e Tipi di ciclo, impostare il tempo di contatto su 60 s, il tempo di dissociazione su 60 s e la velocità di flusso su 30 μL/min. Anche le variabili di metodo, le variabili di valutazione e i comandi utilizzano la stessa impostazione, ad eccezione dell'utilizzo di glicina 2.0 come rigenerazione, impostando la velocità di flusso a 30 μL/min, passando attraverso il percorso del flusso di 1, 2, 3, 4.
- Quindi imposta la corsa e scegli il percorso del flusso: 2-1, 4-3. Inserire la soluzione del campione: PD-L1 (0,111 μM, peso molecolare di 51.300 Da) con BMS-1166 a 0 μM, 0,125 μM, 0,625 μM, 3,125 μM. Controllare tutte le fasi del test per la verifica e selezionare il primo prima della corsa.
- Quindi impostare ciascuna concentrazione di PD-L1 come posizione separata del pozzetto del campione in un layout del rack reagenti 2: R2 B1: PD-L1 (0,111 μM) + BMS-1166 0 μM R2 B2: PD-L1 (0,111 μM) + BMS-1166 0,125 μM; R2 B3: PD-L1 (0,111 μM) + BMS-1166 0,625 μM; R2 B4: PD-L1 (0,111 μM) + BMS-1166 3,125 μM; R2 A1: Glicina 2.0 per la rigenerazione.
- Preparare 200 mL di soluzione tampone 1× HBS-EP+. Preparare la miscela BMS-1166/PD-L1: Sciogliere 5 mg di BMS-1166 in 77,99 μL di dimetilsolfossido (DMSO) per preparare una soluzione madre da 100 mM. Diluire la soluzione madre con la proteina PD-L1 (0,11 μM) alle concentrazioni target di BMS-1166 a 0 μM, 0,125 μM, 0,625 μM e 3,125 μM in HBS-EP+. Posizionare tutti i reagenti pertinenti come descritto al punto 5.1.2.
- Posizionare la provetta A nella soluzione tampone di corsa HBS-EP+ da 1×, quindi espellere il chip del sensore di manutenzione e inserire il chip SA. Riaprire il metodo di ricerca della rigenerazione , seguire la posizione della provetta dal punto 5.1 e inserire il rack dei reagenti 2, quindi eseguire il metodo per il tempo di esecuzione stimato.
- Ripetere il passaggio 1.6.
Risultati
Immobilizzazione di SA su chip CM5
I dati sono stati analizzati tramite l'output dello strumento SPR e del software di analisi associato, indicando il raggiungimento dell'UR target (2000 UR) della proteina SA sulla cella di flusso 1 e sulla cella di flusso 2. Le celle di flusso 1 e 2 sono state immobilizzate con SA (40 μg/mL) sulla superficie del chip CM5 con una risposta finale di 1902,3 RU sulla cella di flusso 1 (Figura 1A) e 1900,7 RU sulla cella di flusso 2 (Figura 1B).
Immobilizzazione di PD-1 su chip SA
L'analisi dei dati basata sull'output dello strumento SPR e del software associato ha indicato un'unità di risposta (RU) bassa per la cella vuota sulla cella di flusso 1 e il raggiungimento con successo dell'UR target (4000 RU) per il ligando PD-1 sulla cella di flusso 2. La cella di flusso 1 è stata immobilizzata in bianco, producendo una risposta finale di -161,0 UR (Figura 2A), mentre la cella di flusso 2 è stata immobilizzata con la proteina PD-1 biotinilata (10 μg/mL) rivestita sul chip SA, ottenendo una risposta finale di 3698,5 UR (Figura 2B).
Scouting di rigenerazione per PD-1 e PD-L1.
Lo scouting della rigenerazione è stato condotto con PD-1 immobilizzato sulla cella di flusso 2 e PD-L1 in soluzione (0,1 μM) a vari livelli di pH della glicina (1,5, 2, 2,5 e 3) per determinare la soluzione di rigenerazione con conseguente base stabile e risposta del campione. I dati sono stati analizzati tramite l'output dello strumento SPR e del software di analisi, risultando nella glicina (pH 2,0) come condizione di rigenerazione ottimale grazie a cambiamenti minimi nella risposta sia per il basale che per la risposta del campione, indicando una perdita minima della proteina PD-1 e una rimozione riuscita della proteina PD-L1 dalla superficie del chip. A pH 2,0 della glicina, la risposta basale è rimasta relativamente costante e la risposta dell'analita è stata stabile e vicina alla risposta all'inizio dell'esperimento, indicando il tampone di rigenerazione più ottimale tra le quattro condizioni di pH della glicina testate. Un pH più alto è insufficiente e un pH più basso è troppo duro; Il pH 2,0 è identificato come la condizione di rigenerazione più adatta (Figura 3).
Validazione dell'interazione PD-1/PD-L1
I dati sono stati analizzati utilizzando il software di valutazione corrispondente. Nella sezione Cinetica/Affinità, selezionare "surface-bound", scegliere la curva 2-1, impostare il modello cinetico su un legame 1:1 e regolare il parametro RI su un adattamento costante per determinare il tasso di associazione (ka), il tasso di dissociazione (kd) e la costante di dissociazione di equilibrio (KD). L'interazione di legame di PD-L1 a concentrazioni variabili con PD-1 è stata quantificata, producendo una risposta misurabile (Figura 4). I parametri di legame analizzati includevano un tasso di associazione (ka) di 3,611 × 104 (1/Ms), un tasso di dissociazione (kd) di 0,0236 (1/s) e una costante di dissociazione di equilibrio (KD) di 6,536 × 10-7 M.
Saggio di blocco PD-1/PD-L1 con inibitore di piccole molecole stabilito: BMS-1166
I dati sono stati analizzati utilizzando il corrispondente. In Cinetica/Affinità, selezionare Limite superficie, scegliere la curva 2-1, quindi ottenere la curva visualizzata del risultato e dell'unità di risposta. L'effetto di blocco dell'interazione di legame PD-1/PD-L1 è stato osservato con BMS-1166 (0-3,125 μM) con 0,11 μM di proteina PD-L1 nel tampone HBS EP+ (Figura 5). L'unità di risposta più alta è identificata dalla sola PD-L1, mentre all'aumentare della concentrazione di BMS-1166, l'unità di risposta di legame diminuisce proporzionalmente.

Figura 1: Immobilizzazione di SA su chip CM5. Sono mostrate le curve di immobilizzazione della proteina streptavidina sulla cella di flusso 1 (A) e sulla cella di flusso 2 (B) del chip CM5. In primo luogo, cinque pre-concentrazioni della proteina SA sono fluite sulle cellule di flusso 1 e 2, seguite da un lavaggio NaOH e dalla creazione di una linea di base stabile. Successivamente, sono stati aggiunti EDC e NHS, quindi è stato condotto un lavaggio con etanolammina cloridrato. Successivamente, sono stati eseguiti cinque impulsi di PD-1 per raggiungere il livello target (2000 RU), seguiti da un lavaggio con etanolammina cloridrato per rimuovere i ligandi legati elettrostaticamente e disattivare gli esteri NHS che rimangono non reattivi. Clicca qui per visualizzare una versione più grande di questa figura.
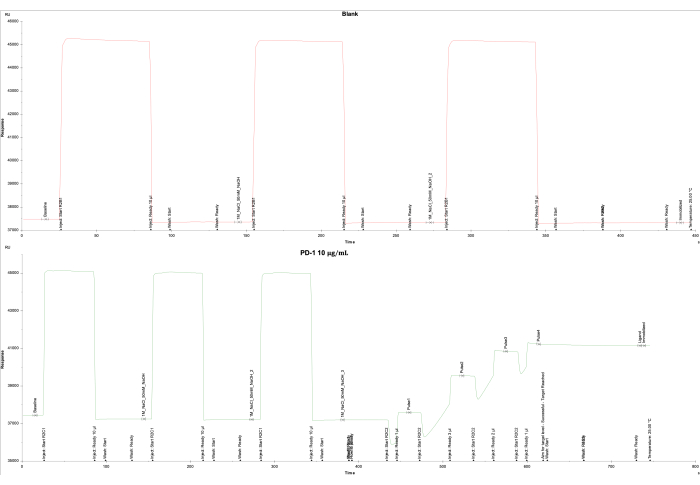
Figura 2: Immobilizzazione di PD-1 su chip SA. Sono mostrate le curve di immobilizzazione di (A) la cella di flusso vuota 1 rivestita solo con SA e (B) la proteina PD-1 sul chip SA sulla cella di flusso 2. Per il bianco (cella di flusso 1), 1 M di NaCl e 50 mM di NaOH sono stati iniettati tre volte, seguiti da un lavaggio con isopropanolo al 50%/1 M di NaCl/50 mM di NaOH. Per la proteina PD-1 (cella di flusso 2), 1 M di NaCl e 50 mM di NaOH sono stati iniettati tre volte, seguiti da cinque impulsi di proteina PD-1 per raggiungere il livello target di 4000 RU e un lavaggio finale di isopropanolo al 50%/1 M di NaCl/50 mM di NaOH. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Scouting della rigenerazione per PD-1 e PD-L1. Il basale e la risposta del campione sono stati ottenuti per quattro condizioni di rigenerazione con glicina a pH 1,5, 2, 2,5 e 3 per determinare la soluzione di rigenerazione ottimale prima del test del campione. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Validazione dell'interazione PD-1/PD-L1. Cinetica di legame di diverse concentrazioni di PD-L1 (0,037 μM, 0,111 μM e 0,333 μM) a PD-1 sulla superficie del chip rivestito di streptavidina. PD-L1 dimostra distinte fasi di associazione (0-60 s) e di dissociazione (61-120 s) con PD-1. Clicca qui per visualizzare una versione più grande di questa figura.
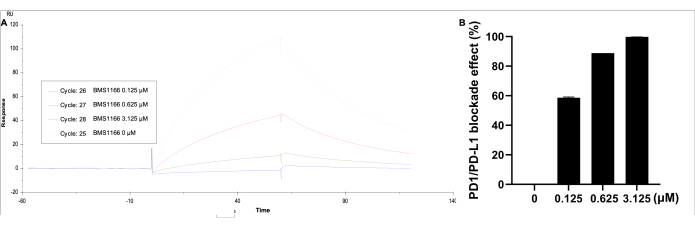
Figura 5: Analisi SPR di PD-1 rivestito su un chip CM5 immobilizzato SA, con PD-L1 (0.11 μM) e BMS-1166 (0-3.125 μM) in soluzione. (A) Una risposta SPR rappresentativa in tempo reale delle reazioni di legame tra PD-1/PD-L1 e BMS-1166. (B) L'effetto di blocco percentuale BMS-1166 sulla cinetica di legame dell'interazione PD-1/PD-L1. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 6: Confronto delle strategie di immobilizzazione della proteina PD-1. Accoppiamento amminico (A) vs. accoppiamento streptavidina-biotina a doppio stadio (B). L'accoppiamento amminico presenta sfide, tra cui la disponibilità limitata di siti di legame accessibili, l'ostacolo sterico al legame e la potenziale modifica dei siti di legame PD-1 durante l'immobilizzazione. Al contrario, l'approccio a doppia fase streptavidina-biotina migliora la disponibilità di siti di legame liberi su PD-1 immobilizzato, facilitando una migliore interazione con PD-L1 in soluzione. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Negli ultimi decenni, vari approcci immunoterapici, tra cui vaccini antitumorali, inibitori del checkpoint immunitario e terapie con cellule T CAR, hanno fatto progredire significativamente il trattamento del cancro21. I checkpoint immunitari svolgono un ruolo cruciale nella prevenzione del danno collaterale mediato dalle cellule immunitarie durante le risposte patogene e nella soppressione dell'autoimmunità. Un esempio chiave è l'interazione tra PD-L1 e PD-1, che forma un importante checkpoint immunitario, consentendo alle cellule tumorali di eludere la sorveglianza immunitaria. Il targeting della via PD-1/PD-L1 con anticorpi monoclonali ha ottenuto un notevole successo in oncologia clinica. Tuttavia, a causa delle limitazioni associate alle terapie con anticorpi monoclonali e della crescente incidenza di eventi avversi immuno-correlati, vi è un crescente interesse nello sviluppo di inibitori di piccole molecole mirati a PD-1/PD-L1 9,22.
Le attuali strategie di screening per gli inibitori PD-1/PD-L1 a piccole molecole si concentrano principalmente su tecniche di biodosaggio come ELISA, saggi reporter basati su cellule e saggi su cellule T. Le tecniche biofisiche, tra cui SPR e interferometria a biostrato (BLI) sono ampiamente utilizzate per la caratterizzazione delle affinità di legame, ma il loro potenziale di essere utilizzate come strumenti di screening è sottostimato23. Questo studio ha sviluppato un saggio di blocco PD-1/PD-L1 ottimizzato basato su SPR, che offre una piattaforma ad alto rendimento adatta per la scoperta di inibitori PD-1/PD-L1 a piccole molecole. SA è stato immobilizzato a ~2000 UR, seguito da PD-1 biotinilato (~4000 UR) tramite l'interazione biotina-streptavidina. Questo robusto legame ha garantito un rivestimento PD-1 sicuro con un orientamento ottimale, riducendo al minimo il legame aspecifico e riducendo l'uso di proteine (10 μg/mL). L'immobilizzazione sito-specifica ha migliorato l'efficienza rispetto ai metodi convenzionali. Il tampone glicina (pH 2,0) è stato utilizzato per rigenerare la superficie del sensore tra i cicli, mantenendo l'integrità sperimentale e prevenendo il legame non specifico.
Rispetto ai metodi immunologici standard e all'ELISA, questo approccio SPR offriva una rilevazione in tempo reale, senza marcatura, con elevata sensibilità e specificità. Inoltre, consente uno screening ad alto rendimento con un tempo di esecuzione di 120 s/campione e integra i saggi biologici nella valutazione dell'efficienza del blocco dei composti e dei farmaci biologici mirati a PD-1/PD-L1. Il livello di immobilizzazione raggiunto è stato paragonabile alla piattaforma precedente (3698,5 RU vs. 3688,5 RU), con un'affinità di interazione PD-1/PD-L1 simile (KD = 6,536 × 10-7 M vs. 1,295 × 10-7 M). L'inibitore BMS-1166 ha dimostrato un tasso di dissociazione più elevato a una concentrazione inferiore, con effetti di blocco paragonabili alla piattaforma precedente (99,8% vs. 94,2% a 3,125 μM). BMS-1166 ha dimostrato tassi di blocco di PD-1/PD-L1 supportati da valori IC50 di 1,4 nM e 96 nM in HTRF e saggi basati su cellule, rispettivamente24. Inoltre, altri studi hanno riportato valori IC50 di 3,9 nM e 1574 nM utilizzando HTRF e metodi di co-coltura del blocco del checkpoint immunitario25. I nostri risultati precedenti hanno mostrato che i valori IC50 di BMS-1166 erano 85,4 nM, il che è coerente con questi risultati precedenti18. Un altro vantaggio di questa piattaforma di screening è la sua robustezza e l'elevata produttività. Questo metodo è stato ampiamente applicato nello screening ad alto rendimento utilizzando un formato di piastra a 384 pozzetti, con BMS-1166 e BMS-202 inclusi come controlli positivi ogni 10 campioni. Gli intervalli di tasso di blocco sono stati del 29,8%-38,1% per BMS-1166 e del 6,0%-10,4% per BMS-202 a 10 nM (n = 11 per piastra).
È probabile che il DMSO abbia effetti variabili su membrane biologiche eterogenee, a seconda della loro composizione e struttura locale, influenzando potenzialmente le funzioni biologiche associate alla membrana. A concentrazioni relativamente basse, il DMSO può alterare le proprietà delle proteine in soluzione, portando alla denaturazione, aggregazione o degradazione. Inoltre, il DMSO può modificare le proprietà di legame apparenti delle proteine26,27. Inoltre, per risolvere le precedenti preoccupazioni relative all'interferenza del DMSO, abbiamo ridotto la concentrazione di DMSO allo 0,003% (lo 0,01% di DMSO era utilizzato nella piattaforma precedente) in questo protocollo.
In uno studio precedente, le proteine PD-1 sono state immobilizzate su chip sensore utilizzando l'accoppiamento amminico, che impiega la chimica EDC/NHS per attivare i gruppi carbossimetilici sul chip, formando legami covalenti con i gruppi amminici su PD-1. A causa della presenza di più gruppi amminici nei 288 amminoacidi di PD-1, questo metodo risulta intrinsecamente in orientamenti casuali. Per ottenere un orientamento più specifico, in questo studio abbiamo impiegato una strategia di accoppiamento di cattura con PD-1 umano biotinilato (Fc, Avitag). Il singolo residuo di lisina nell'Avitag è biotinilato enzimaticamente sulla regione Fc di PD-1, consentendo un'immobilizzazione precisa attraverso l'interazione ad alta affinità tra biotina e streptavidina (Figura 6). In teoria, questo approccio garantisce che il dominio variabile di PD-1 rimanga esposto, facilitando l'interazione ottimale con PD-L1 nel tampone. Tuttavia, questa ipotesi richiede un'ulteriore convalida utilizzando tecniche come la crio-EM o la cristallografia per confermare l'orientamento delle proteine.
La glicina 2.0 è stata utilizzata come tampone di rigenerazione lieve, rimuovendo efficacemente PD-L1 senza ridurre il PD-1 immobilizzato. Tuttavia, per gli inibitori strettamente legati o gli aggregati proteici, possono essere necessari tamponi di rigenerazione più forti, come SDS allo 0,5% o 50-100 mM di NaOH, per evitare interferenze con i campioni successivi.
Questo studio ha diverse limitazioni. In primo luogo, l'alto costo della strumentazione SPR può limitare l'accessibilità, sebbene il protocollo possa spesso essere adattato per altri sistemi SPR con capacità simili. Parametri come la concentrazione proteica e i tempi di associazione/dissociazione possono essere regolati per adattarsi a specifiche piattaforme SPR. Un'altra limitazione è la sua dipendenza dalle proteine ricombinanti, che potrebbero non replicare completamente le interazioni proteiche native. Questi fattori devono essere presi in considerazione quando si interpretano i dati SPR e si confrontano i risultati con tecniche complementari, come i saggi basati su cellule o i modelli in vivo .
Nonostante queste limitazioni, il metodo SPR ottimizzato offre un approccio rapido, in tempo reale, ad alto rendimento e privo di marcatura per lo screening di inibitori di piccole molecole dell'interazione PD-1/PD-L1. Migliora significativamente le tecniche biofisiche per la caratterizzazione di piccole molecole e farmaci biologici che mirano all'interazione PD-1/PD-L1. Con la sua elevata sensibilità, la piattaforma è particolarmente preziosa per lo studio delle interazioni dei checkpoint immunitari e delle più ampie interazioni proteina-proteina (PPI), rendendola un potente strumento per far progredire la scoperta di farmaci PPI.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori riconoscono la struttura principale RI-INBRE presso l'Università del Rhode Island, supportata da Grant P20GM103430 del National Center for Research Resources (NCRR), un componente del National Institutes of Health (NIH). Questa ricerca è stata supportata da un Pilot Grant Award dal College of Pharmacy dell'Università del Rhode Island, da un Small Grant Award dal Rhode Island Life Science Hub (RILSH) e da una borsa di studio della Rhode Island Foundation, tutti assegnati a Chang Liu, Ph.D.
Materiali
| Name | Company | Catalog Number | Comments |
| 50 mM NaOH | Cytiva Life Sciences | 100358 | |
| 50 mM NaOH | Fisher Scientific | 905376 | |
| 96-Well Polystyrene Microplates | Cytiva Life Sciences | BR100503 | |
| Amine Coupling Kit | Cytiva Life Sciences | 35120 | |
| Biacore T200 SPR System and Evaluation Software 3.2 | Cytiva Life Sciences | 28975001 | |
| Biotinylated Human PD-1 Fc, Avitag Protein | Acro Biosystems | PD1-H82F1 | |
| BMS1166 | MedChemExpress | HY-102011 | |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | 276855 | |
| DNase Free Water | Fisher Scientific | 188506 | |
| Glycine 1.5 | Cytiva Life Sciences | BR100354 | |
| Glycine 2.0 | Cytiva Life Sciences | BR100355 | |
| Glycine 2.5 | Cytiva Life Sciences | BR100356 | |
| Glycine 3.0 | Cytiva Life Sciences | BR100357 | |
| HBS-EP+ Buffer | Cytiva Life Sciences | BR100669 | |
| Human PD-L1 Fc Tag Protein | Acro Biosystems | PD-1-H5258 | |
| Isopropanol | Fisher Scientific | BP2618-1 | |
| Microplate Foil, 96-Well | Cytiva Life Sciences | 28975816 | |
| NaCl | Sigma-Aldrich | 746398 | |
| Plastic Vials 7 mm | Cytiva Life Sciences | BR100212 | |
| Rubber Caps, Type 3 | Cytiva Life Sciences | BR100502 | |
| Series S Sensor Chip CM5 | Cytiva Life Sciences | 29149603 | |
| Sodium Acetate 4.5 | Cytiva Life Sciences | 100350 | |
| Sodium Acetate 5.0 | Cytiva Life Sciences | 100351 | |
| Streptavidin | Sigma-Aldrich | S4762 |
Riferimenti
- Ribas, A., Wolchok, J. D. Cancer immunotherapy using checkpoint blockade. Science. 359 (6382), 1350-1355 (2018).
- Zhang, X., et al. Structural and functional analysis of the costimulatory receptor programmed death-1. Immunity. 20 (3), 337-347 (2004).
- Freeman, G. J., et al. Engagement of the PD-1 immunoinhibitory receptor by a novel B7 family member leads to negative regulation of lymphocyte activation. J Exp Med. 192 (7), 1027-1034 (2000).
- Han, Y., Liu, D., Li, L. PD-1/PD-L1 pathway: Current researches in cancer. Am J Cancer Res. 10 (3), 727-742 (2020).
- Lyu, N., et al. Recognition of PDL1/L2 by different induced-fit mechanisms of PD1: A comparative study of molecular dynamics simulations. Phys Chem Chem Phys. 22 (3), 1276-1287 (2020).
- Taube, J. M., et al. Colocalization of inflammatory response with B7-H1 expression in human melanocytic lesions supports an adaptive resistance mechanism of immune escape. Sci Trans Med. 4 (127), 127ra37(2012).
- Sun, X., et al. Immune-related adverse events associated with programmed cell death protein-1 and programmed cell death ligand 1 inhibitors for non-small cell lung cancer: A PRISMA systematic review and meta-analysis. BMC Cancer. 19 (1), 558(2019).
- Alkholifi, F. K., Alsaffar, R. M. Dostarlimab an inhibitor of PD-1/PD-L1: A new paradigm for the treatment of cancer. Medicina. 58 (11), 1572(2022).
- Uzar, W., et al. An updated patent review on PD-1/PD-L1 antagonists (2022-present). Expert Opin Ther Pat. 34 (8), 627-650 (2024).
- Ai, L., et al. Research status and outlook of PD-1/PD-L1 inhibitors for cancer therapy. Drug Des Devel Ther. 14, 3625-3649 (2020).
- Conroy, M., Naidoo, J. Immune-related adverse events and the balancing act of immunotherapy. Nat Commun. 13 (1), 392(2022).
- Guzik, K., et al. Small-molecule inhibitors of the Programmed Cell Death-1/Programmed Death-Ligand 1 (PD-1/PD-L1) interaction via transiently induced protein states and dimerization of PD-L1. J Med Chem. 60 (13), 5857-5867 (2017).
- Sifniotis, V., Cruz, E., Eroglu, B., Kayser, V. Current advancements in addressing key challenges of therapeutic antibody design, manufacture, and formulation. Antibodies. 8 (2), 36(2019).
- Beck, H., Härter, M., Haß, B., Schmeck, C., Baerfacker, L. Small molecules and their impact in drug discovery: A perspective on the occasion of the 125th anniversary of the Bayer Chemical Research Laboratory. Drug Discov Today. 27 (6), 1560-1574 (2022).
- Nguyen, H. H., Park, J., Kang, S., Kim, M. Surface plasmon resonance: A Versatile technique for biosensor applications. Sensors (Basel, Switzerland). 15 (5), 10481-10510 (2015).
- Liu, C., Seeram, N. P., Ma, H. Small molecule inhibitors against PD-1/PD-L1 immune checkpoints and current methodologies for their development: A review. Cancer Cell Int. 21 (1), 239(2021).
- Hu, K., Li, X. -J., Asmamaw, M. D., Shi, X. -J., Liu, H. -M. Establishment of high-throughput screening HTRF assay for identification small molecule inhibitors of Skp2-Cks1. Sci Rep. 11 (1), 21105(2021).
- Puopolo, T., et al. Establishment of human PD-1/PD-L1 blockade assay based on surface plasmon resonance (SPR) biosensor. Bio-protoc. 13 (15), e4765(2023).
- Ding, M., Chen, Y., Lang, Y., Cui, L. The role of cellular prion protein in cancer biology: A potential therapeutic target. Front Oncol. 11, 742949(2021).
- Li, H., Seeram, N. P., Liu, C., Ma, H. Further investigation of blockade effects and binding affinities of selected natural compounds to immune checkpoint PD-1/PD-L1. Front Oncol. 12, 995461(2022).
- Kamrani, A., et al. New immunotherapeutic approaches for cancer treatment. Pathol Res Pract. 248, 154632(2023).
- Yan, Y., Zhang, L., Zuo, Y., Qian, H., Liu, C. Immune checkpoint blockade in cancer immunotherapy: mechanisms, clinical outcomes, and safety profiles of PD-1/PD-L1 inhibitors. Arch Immunol Ther Exp. 68 (6), 36(2020).
- Chandrasekharan, G., Unnikrishnan, M. High throughput methods to study protein-protein interactions during host-pathogen interactions. Eur J Cell Biol. 103 (2), 151393(2024).
- Zhang, Y., et al. BMS-202, a PD-1/PD-L1 inhibitor, decelerates the profibrotic effects of fibroblasts derived from scar tissues via ERK and TGFβ1/Smad signaling pathways. Immun Inflamm Dis. 10 (1), e591(2022).
- Surmiak, E., et al. PD-L1 inhibitors: Different classes, activities, and mechanisms of action. Int J Mol Sci. 22 (21), 11797(2021).
- Feoli, A., Sarno, G., Castellano, S., Sbardella, G. DMSO-Related effects on ligand-binding properties of lysine methyltransferases G9a and SETD8. ChemBioChem. 25 (4), e202300809(2024).
- Tjernberg, A., Markova, N., Griffiths, W. J., HalléN, D. DMSO-Related effects in protein characterization. SLAS Discov. 11 (2), 131-137 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon