Method Article
Identificación de inhibidores de PD-1/PD-L1 con tecnología de resonancia de plasmón de superficie
En este artículo
Resumen
Este protocolo describe un ensayo de bloqueo para inhibidores de PD-1/PD-L1 utilizando tecnología de resonancia de plasmón de superficie. Emplea una estrategia de inmovilización de dos pasos y un sistema de amortiguación personalizado para medir con precisión las unidades de respuesta, lo que facilita la evaluación de las tasas de bloqueo de compuestos o productos biológicos. Además, admite la identificación de alto rendimiento de inhibidores de PD-1/PD-L1.
Resumen
La interrupción de la interacción PD-1/PD-L1 es una estrategia prometedora para la inmunoterapia contra el cáncer. Es esencial contar con plataformas de cribado fiables para evaluar la eficacia de los inhibidores de PD-1/PD-L1. Un ensayo de bloqueo de PD-1/PD-L1 humano previamente establecido que utiliza la tecnología de resonancia de plasmón de superficie (SPR) (plataforma de cribado SPR de inhibidor de PD-1/PD-L1 de primera generación) demostró resultados comparables a los obtenidos mediante fluorescencia homogénea resuelta en el tiempo (HTRF) y ensayos basados en células, con potencial para el cribado a gran escala. En este trabajo, se presenta una versión optimizada de este ensayo (plataforma de cribado SPR de inhibidor de PD-1/PD-L1 de segunda generación), que presenta un proceso de acoplamiento de dos pasos que combina el acoplamiento de aminas y bioestreptavidina para mejorar el control de la orientación de PD-1 en el chip y reducir el consumo de proteínas PD-1. La plataforma actualizada se validó con éxito utilizando el inhibidor de PD-1/PD-L1 BMS-1166, mostrando efectos de bloqueo comparables al método anterior basado en SPR y otras técnicas establecidas como ELISA. Estos resultados confirman la fiabilidad del enfoque. Esta plataforma optimizada de cribado de SPR ofrece una herramienta fiable y de alto rendimiento para identificar nuevos inhibidores de PD-1/PD-L1, avanzar en la investigación de la inmunoterapia contra el cáncer y destacar el potencial de la SPR en el cribado de inhibidores de puntos de control inmunitario.
Introducción
Las terapias de bloqueo de puntos de control inmunitario, en particular las dirigidas a la muerte celular programada-1 (PD-1) y al ligando de muerte celular programada 1 (PD-L1), se sitúan a la vanguardia de las estrategias de inmunoterapia contra el cáncer. Las terapias anti-PD-1/PD-L1 han recibido aprobación para su uso en varios tipos de cáncer, como el hematológico, el cutáneo, el pulmonar, el hepático, el de vejiga urinaria y el renal1. PD-1 es una glicoproteína transmembrana perteneciente a la superfamilia de las inmunoglobulinas, caracterizada por un único dominio similar a la inmunoglobulina variable (IgV) en el N-terminal, un tallo de aproximadamente 20 aminoácidos que separa el dominio IgV de la membrana plasmática, un dominio transmembrana y una cola citoplasmática que contiene motivos de señalización basados en tirosina2. PD-L1, identificada como uno de los ligandos de PD-1, es una proteína transmembrana de tipo I que presenta una región transmembrana, dos dominios extracelulares -constante de inmunoglobulina (IgC) e IgV- y un dominio citoplasmático relativamente corto que desencadena vías de señalización intracelular3. La vía inhibidora de PD-1/PD-L1 sirve como un punto de control inmunitario crítico que regula la activación de las células T y la autoinmunidad4. La PD-1 se expresa en las células T, donde interactúa con la PD-L1, inhibe la señalización del receptor de las células T y bloquea la estimulación de las moléculas CD28 y CD80 en las células presentadoras de antígenos y las células T5. Los tejidos cancerosos explotan este mecanismo fisiológico mediante la sobreexpresión de PD-L1 durante la fase de escape, creando así un entorno inmunosupresor que promueve el crecimiento y la progresión del tumor6. Los inhibidores de PD-1 y PD-L1 interrumpen esta interacción, lo que permite al sistema inmunitario evadir la supresión inducida por el tumor y reiniciar el proceso de muerte de las células tumorales mediado por las célulasT 7.
Sobre la base establecida por el papel destacado de las terapias de bloqueo de puntos de control inmunitario, el desarrollo de inhibidores de PD-1/PD-L1 ha marcado un avance significativo en la inmunoterapia contra el cáncer. La Administración de Alimentos y Medicamentos de EE. UU. (FDA, por sus siglas en inglés) ha respaldado nueve inhibidores de puntos de control inmunitario que se dirigen específicamente a la vía PD-1/PD-L1. Estos incluyen seis inhibidores de PD-1 (pembrolizumab, dostarlimab, nivolumab, cemiplimab, oripalimab y tislelizumab) y tres inhibidores de PD-L1 (atezolizumab, avelumab y durvalumab 8,9). Estasterapias se han utilizado eficazmente para tratar una variedad de cánceres, como el melanoma, el cáncer de pulmón, el cáncer urotelial, el cáncer de cuello uterino, el cáncer gástrico o gastroesofágico y otros tumores sólidos. A pesar de su eficacia, las terapias basadas en anticuerpos monoclonales enfrentan limitaciones significativas, incluyendo bajas tasas de respuesta, altos costos, vidas medias prolongadas, eventos adversos graves relacionados con el sistema inmunitario y restricciones a la administración intravenosa o subcutánea 11,12,13. En consecuencia, la investigación se centra cada vez más en el desarrollo de inhibidores de moléculas pequeñas dirigidos al eje PD-1/PD-L1. Estas pequeñas moléculas ofrecen ventajas distintivas, como una mejor penetración celular, modulación de diversas dianas biológicas, mayor biodisponibilidad oral y reducción de costos, con el objetivo de lograr resultados terapéuticos comparables con menos efectos adversos14. Sin embargo, el desarrollo de inhibidores de moléculas pequeñas dirigidos a la interacción PD-1/PD-L1 se encuentra en sus primeras etapas, principalmente debido a la falta de una plataforma de cribado fiable de alto rendimiento. Estas plataformas son esenciales para evaluar rápidamente vastas bibliotecas de moléculas pequeñas e identificar compuestos principales para su posterior validación y optimización. Superar este desafío es fundamental para avanzar en la inmunoterapia contra el cáncer.
La tecnología de resonancia de plasmón de superficie (SPR) se emplea ampliamente en la detección de diversas biomoléculas, incluidos antígenos de anticuerpos, enzimas, ácidos nucleicos y fármacos, y es particularmente eficaz en el cribado de fármacos de moléculas pequeñas15,16. A diferencia de otras técnicas biofísicas, SPR ofrece detección sin etiquetas, datos cinéticos en tiempo real y un amplio rango de detección. Por el contrario, la calorimetría de valoración isotérmica carece de información cinética en tiempo real y requiere mayores volúmenes de muestra, lo que limita el rendimiento. La termoforesis a microescala es propensa a la interferencia del tampón y no puede proporcionar datos cinéticos, mientras que la interferometría de biocapa tiene limitaciones específicas de la aplicación basadas en el tamaño y las propiedades moleculares. La fluorescencia homogénea resuelta en el tiempo requiere etiquetado y es susceptible a la interferencia fluorescente. Reconocemos que HTRF es otra tecnología adecuada para explorar los inhibidores de PD-1/PD-L1. Una limitación inherente de HTRF, en comparación con SPR, es la extinción de la fluorescencia causada por interacciones externas con el proceso de excitación intramolecular (por ejemplo, transferencia de electrones, FRET y blanqueo), la sensibilidad es demasiado baja en el proceso de selección de fármacos debido al pequeño rango de ventana y la interferencia de compuestos de biblioteca fluorescentes o proteínas biológicas17. Estas características posicionan a SPR como una herramienta superior para el descubrimiento de fármacos. Nuestros estudios previos han demostrado que SPR es capaz de determinar el efecto de bloqueo de moléculas pequeñas frente a PD-1/PD-L1, lo que es ventajoso sobre otras técnicas que requieren altos requisitos de tecnología de etiquetado, múltiples pasos, poca especificidad y alto costo en el proceso de descubrimiento de fármacos18.
Este estudio presenta una plataforma optimizada basada en SPR, que integra un proceso de acoplamiento de dos pasos que utiliza el acoplamiento de aminas y bioestreptavidina para mejorar la orientación de PD-1 en el chip y minimizar el uso de proteínas. Este enfoque actualizado se validó con éxito utilizando el inhibidor de PD-1/PD-L1 BMS-1166 como aglutinante de control positivo, demostrando efectos de bloqueo comparables tanto a nuestro método SPR anterior como a otras técnicas establecidas como ELISA19,20. Esto no solo confirma la fiabilidad y reproducibilidad de nuestro protocolo, sino que también ilustra la eficacia de nuestra plataforma modificada para facilitar el cribado de alto rendimiento de inhibidores de PD-1/PD-L1. La incorporación de la etapa de captura de bioestreptavidina proporciona una orientación de la proteína dirigida al sitio en lugar de aleatoria, lo que permite una concentración reducida de PD-1 (40 μg/mL frente a 10 μg/mL) y un ahorro de costos al permitir al usuario final inmovilizar la estreptavidina (SA) en un chip CM5, una alternativa menos costosa a los chips SA preinmovilizados comercializados. Esto lo hace ventajoso para el cribado a gran escala y rentable de bibliotecas de compuestos/péptidos. Aunque los métodos de caracterización adicionales, incluidos los ensayos in silico, in vitro e in vivo, son esenciales para evaluar el potencial clínico de los inhibidores de PD-1/PD-L1 contra el cáncer, nuestra plataforma de cribado mejorada basada en SPR se destaca como una herramienta eficaz para el cribado a gran escala de inhibidores de PD-1/PD-L1.
Protocolo
Los reactivos y equipos se enumeran en la Tabla de Materiales.
1. Inmovilización de la proteína estreptavidina (SA) en el chip CM5
- Configure el método de inmovilización en el instrumento SPR: Abrir / nueva plantilla de asistente, elegir inmovilización, establecer el tipo de chip en CM5 y las celdas de flujo por ciclo en 1. Verifique la inmovilización de la celda de flujo 1 y la celda de flujo 2.
- Establezca Amina como método de inmovilización. Establecer el objetivo para el nivel de inmovilización, concentración de ligando: 40 μg/mL de estreptavidina, nivel objetivo: 2000 RU, solución de lavado: 50 mM de NaOH. A continuación, compruebe el cebado antes de ejecutar.
- Prepare los siguientes tubos: R2 B1 y R2 C1 - 40 μg/mL de estreptavidina; R2 B2 y R2 C2: 50 mM de NaOH; R2 B3 y R2 C3: EDC; R2 B4 y R2 C4: Servicio Nacional de Salud; R2 B5 y R2 C5: vacíos; R2 B6 y R2 C6: etanolamina.
NOTA: EDC y NHS activan los grupos carboxilo en el chip CM5, lo que permite el acoplamiento covalente con aminas en el ligando. La etanolamina se utiliza como tampón de bloqueo para evitar la unión inespecífica durante la inmovilización. - Diluya 20 mL de tampón HBS-EP+ 10× en 180 mL de agua desionizada (DI) para preparar 200 mL de solución tampón HBS-EP+ 1× en funcionamiento.
- Añadir 1 mL de agua libre de DNasa a 1 mg de estreptavidina e incubar a temperatura ambiente durante 30 min. A continuación, diluir la solución de estreptavidina a 40 μg/mL en tampón de acetato (pH 4,5). Prepare los reactivos del kit de reactivos de acoplamiento de aminas de acuerdo con las instrucciones del fabricante y coloque todos los diseños de reactivos relevantes como se indica en el paso 1.2.
- Reemplace el chip del sensor de mantenimiento con el chip CM5, luego coloque el tubo A en el tampón HBS-EP+ 1× preparado, vuelva a abrir el método de inmovilización e inserte los tubos de acuerdo con el diseño del paso 1.2. Ejecute el método y guarde el archivo de resultados.
- Mantenimiento posterior a la ejecución: reemplace el chip CM5 con el chip sensor de mantenimiento, coloque el tubo A en una botella llena de agua desionizada y ejecute el cebador. Después de sacar el chip CM5, lave el chip con unas gotas de agua desionizada y séquelo al aire. Coloque el chip en un tubo de 50 ml a 4 °C.
NOTA: El SA inmovilizado permite que el chip CM5 funcione como un chip SA.
2. Inmovilización de la proteína PD-1 en el chip SA
- Establezca el método de inmovilización en el instrumento SPR: Abrir / nuevo plantilla de asistente, elegir inmovilización, establecer el tipo de chip en SA y las celdas de flujo por ciclo en 1. Verifique la inmovilización de la celda de flujo 1 y la celda 2.
- Establezca la captura de biotina SA como método. Para la celda 1 establezca la inmovilización en blanco, para la celda 2 establezca el objetivo de un nivel inmovilizado, 10 μg / mL PD-1 como ligando, nivel objetivo: 4000 RU, luego verifique el cebado antes de ejecutar.
NOTA: Prepare los siguientes tubos: R2 B1 y R2 C1: 1 M de NaCl, 50 mM de NaOH; R2 B2 y R2 C2: 50% de isopropanol/50 mM de NaOH/1 m de NaCl; R2 C3: 10 μg/mL PD-1.
- Establezca la captura de biotina SA como método. Para la celda 1 establezca la inmovilización en blanco, para la celda 2 establezca el objetivo de un nivel inmovilizado, 10 μg / mL PD-1 como ligando, nivel objetivo: 4000 RU, luego verifique el cebado antes de ejecutar.
- Disuelva 58,44 mg de NaCl en 1 mL de 50 mM de NaOH para preparar 1 M de NaCl y 50 mM de solución de NaOH. Disuelva 58,44 mg de NaCl y 4,0 mg de NaOH en 500 μL de agua, luego agregue 500 μL de isopropanol para preparar la solución de 50% de Isopropanol/50 mM de NaOH/1 M de NaCl.
- Prepare la solución de PD-1: Agregue 200 μL de agua libre de DNasa a 100 μg de PD-1 humano biotinilado (Fc y Avitagged) y estabilice a temperatura ambiente durante 30 min, luego diluya a 10 μg/mL en tampón de acetato (pH 5.0).
- Coloque todos los diseños de reactivos como se describe en el paso 2.1. Coloque el tubo A en la solución tampón de funcionamiento HBS-EP+ 1×, luego expulse el chip del sensor de mantenimiento e inserte el chip SA (chip CM5 recubierto por la proteína estreptavidina del paso 1). Vuelva a abrir el método de inmovilización, inserte el bastidor de reactivos 2 y compruebe las posiciones, luego ejecute el método para el tiempo de ejecución estimado.
- Repita el paso 1.6.
3. Exploración de regeneración para PD-1 y PD-L1
- Configure el método de exploración de regeneración: Abrir / nueva plantilla de asistente, elegir Exploración de regeneración, establecer la ruta de flujo: 2-1, 4-3, tipo de chip a SA, verificar el ciclo de acondicionamiento de ejecución, registrar la solución como HBS-EP+, tiempo de contacto como 30 s, número de inyecciones como 3, solución como PD-L1, tiempo de contacto: 30 s, caudal: 30 μL/min.
- Para los parámetros de regeneración, el caudal es de 30 μL/min y el período de estabilización es de 300 s. En el diseño experimental, establezca el número de condiciones como 4 y el número de ciclos para cada condición como 2. Establezca las condiciones como 4, solución de regeneración: glicina 1.5, 2.0, 2.5, 3.0, tiempos de contacto: 30 s, luego verifique el cebado antes de ejecutar.
- Establezca cada concentración de PD-L1 como una posición de pocillo de muestra separada en un diseño de microplaca de 96 pocillos: R1 A1 a R1 A9: 1 μM PD-L1; R1 A10 a R1 A12: tampón HBS-EP+; R1 B1: Glicina 1,5; R1 B2: Glicina 2; R1 B3: glicina 2.5; R1 B4: Glicina 3.
NOTA: El tampón de glicina con diferente pH se utiliza como tampón de regeneración. - Preparación de la solución de PD-L1: Añadir 200 μL de agua libre de DNasa a 100 μg de proteína Fc Tag PD-L1 humana (9,42 μM), luego diluir a 1 μM en tampón HBS-EP+.
- Coloque todos los reactivos relevantes en el diseño como se describe en el paso 3.2. Coloque el tubo A en el tampón HBS-EP+ 1× y, a continuación, sustituya el chip del sensor de mantenimiento por el chip SA. Vuelva a abrir el método de exploración de regeneración , siga la posición del tubo del paso 3.2, inserte la gradilla de reactivos y, a continuación, ejecute el método durante el tiempo de ejecución estimado.
- Repita el paso 1.6.
4. Validación de la interacción PD-1/PD-L1
NOTA: Para la validación, se siguió un informe18 publicado anteriormente con ajustes menores.
- Utilice los mismos parámetros que el informe publicado en Configuración general, Pasos de ensayo y Tipos de ciclo, establezca el tiempo de contacto en 60 s, el tiempo de disociación en 60 s y el caudal en 30 μL/min. En las variables de método, las variables de evaluación y los comandos, use la misma configuración, excepto para usar Glycine 2.0 como regeneración, configure el caudal a 30 μL / min, pasa por la ruta de flujo de 1, 2, 3, 4.
- A continuación, configure la ejecución y elija la ruta de flujo: 2-1, 4-3. Entrada PD-L1 con concentraciones de 0 μM, 0,037 μM, 0,111 μM, 0,333 μM y un peso molecular de 51.300 Da. Compruebe todos los pasos del ensayo para la verificación y seleccione el cebado antes de la ejecución.
- A continuación, establezca cada concentración de PD-L1 como una posición de pocillo de muestra separada en un diseño de gradilla de reactivos 2: R2 B1: PD-L1 0 μM; R2 B2: PD-L1 0,037 μM; R2 B3: PD-L1 0,111 μM; R2 B4: PD-L1 0,333 μM; R2 A1: Glicina 2.0 como tampón de regeneración; R2 A2: HBS-EP+ como búfer de inicio.
- Prepare 200 ml de solución tampón de funcionamiento HBS-EP+ al 1×. Preparar concentraciones de PD-L1: diluir la proteína PD-L1 a 9,42 μM a 0,333 μM, 0,111 μM y 0,037 μM en HBS-EP+. A continuación, coloque todos los reactivos relevantes en el diseño como se describe en el paso 4.1.2.
- Inserte el tubo A en la solución tampón de funcionamiento HBS-EP+ 1×, expulse el chip del sensor de mantenimiento e inserte el chip SA. Vuelva a abrir el método de exploración de regeneración , siga la posición del tubo del paso 4.1, inserte el bastidor de reactivos 2 y ejecute el método durante la duración estimada.
- Repita el paso 1.6.
5. Ensayo de bloqueo de PD-1 / PD-L1 con inhibidor de molécula pequeña: BMS-1166
NOTA: Para el ensayo de bloqueo, se siguió un informe18 publicado anteriormente con ajustes menores.
- Utilice los mismos parámetros que el informe publicado en Configuración general, Pasos de ensayo y Tipos de ciclo, establezca el tiempo de contacto en 60 s, el tiempo de disociación en 60 s y el caudal en 30 μL/min. En las variables de método, las variables de evaluación y los comandos también usan la misma configuración, excepto para usar Glycine 2.0 como regeneración, configura el caudal a 30 μL / min, pasa por la ruta de flujo de 1, 2, 3, 4.
- A continuación, configure la ejecución y elija la ruta de flujo: 2-1, 4-3. Introduzca la solución de muestra: PD-L1 (0,111 μM, peso molecular de 51.300 Da) con BMS-1166 a 0 μM, 0,125 μM, 0,625 μM, 3,125 μM. Compruebe todos los pasos del ensayo para la verificación y seleccione el cebado antes de la ejecución.
- A continuación, establezca cada concentración de PD-L1 como una posición de pocillo de muestra separada en un diseño de gradilla de reactivos 2: R2 B1: PD-L1 (0,111 μM) + BMS-1166 0 μM R2 B2: PD-L1 (0,111 μM) + BMS-1166 0,125 μM; R2 B3: PD-L1 (0,111 μM) + BMS-1166 0,625 μM; R2 B4: PD-L1 (0,111 μM) + BMS-1166 3,125 μM; R2 A1: Glicina 2.0 para la regeneración.
- Prepare 200 ml de solución tampón de funcionamiento HBS-EP+ al 1×. Preparación de la mezcla BMS-1166/PD-L1: Disuelva 5 mg de BMS-1166 en 77,99 μL de dimetilsulfóxido (DMSO) para preparar una solución madre de 100 mM. Diluir la solución madre con la proteína PD-L1 (0,11 μM) hasta las concentraciones objetivo de BMS-1166 a 0 μM, 0,125 μM, 0,625 μM y 3,125 μM en HBS-EP+. Coloque todos los reactivos relevantes como se describe en el paso 5.1.2.
- Coloque el tubo A en la solución tampón de funcionamiento HBS-EP+ 1×, luego expulse el chip del sensor de mantenimiento e inserte el chip SA. Vuelva a abrir el método de exploración de regeneración , siga la posición del tubo de 5.1 e inserte el bastidor de reactivos 2, luego ejecute el método durante el tiempo de ejecución estimado.
- Repita el paso 1.6.
Resultados
Inmovilización de SA en el chip CM5
Los datos se analizaron a través de los resultados del instrumento SPR y el software de análisis asociado, lo que indica el logro exitoso de la RU objetivo (2000 RU) de la proteína SA en la celda de flujo 1 y la celda de flujo 2. Las celdas de flujo 1 y 2 se inmovilizaron con SA (40 μg/mL) en la superficie del chip CM5 con una respuesta final de 1902.3 RU en la celda de flujo 1 (Figura 1A) y 1900.7 RU en la celda de flujo 2 (Figura 1B).
Inmovilización de PD-1 en chip SA
El análisis de datos basado en la salida del instrumento SPR y el software asociado indicó una unidad de respuesta (RU) baja para la celda en blanco en la celda de flujo 1 y el logro exitoso de la RU objetivo (4000 RU) para el ligando PD-1 en la celda de flujo 2. La celda de flujo 1 se inmovilizó como un blanco, produciendo una respuesta final de -161.0 RU (Figura 2A), mientras que la celda de flujo 2 se inmovilizó con proteína PD-1 biotinilada (10 μg/mL) recubierta en el chip SA, lo que resultó en una respuesta final de 3698.5 RU (Figura 2B).
Exploración de la regeneración de PD-1 y PD-L1.
La exploración de la regeneración se llevó a cabo con PD-1 inmovilizado en la celda de flujo 2 y PD-L1 en solución (0,1 μM) a varios niveles de pH de glicina (1,5, 2, 2,5 y 3) para determinar la solución de regeneración que resultó en una línea de base estable y una respuesta de la muestra. Los datos se analizaron a través de la salida del instrumento SPR y el software de análisis, lo que dio como resultado la glicina (pH 2.0) como la condición de regeneración óptima debido a cambios mínimos en la respuesta tanto para la línea de base como para la respuesta de la muestra, lo que indica una pérdida mínima de la proteína PD-1 y una eliminación exitosa de la proteína PD-L1 de la superficie del chip. A pH de glicina 2.0, la respuesta basal permaneció relativamente constante, y la respuesta del analito fue estable y cercana a la respuesta al comienzo del experimento, lo que indica el tampón de regeneración más óptimo entre las cuatro condiciones de pH de glicina probadas. Un pH más alto es insuficiente y un pH más bajo es demasiado duro; El pH 2.0 se identifica como la condición de regeneración más adecuada (Figura 3).
Validación de la interacción PD-1/PD-L1
Los datos se analizaron utilizando el software de evaluación correspondiente. En la sección Cinética/Afinidad, seleccione "Límite de superficie", elija la curva 2-1, establezca el modelo cinético en enlace 1:1 y ajuste el parámetro RI a un ajuste constante para determinar la tasa de asociación (ka), la tasa de disociación (kd) y la constante de disociación de equilibrio (KD). Se cuantificó la interacción de unión de PD-L1 a diferentes concentraciones con PD-1, produciendo una respuesta medible (Figura 4). Los parámetros de unión analizados incluyeron una tasa de asociación (ka) de 3.611 ×10 4 (1/Ms), una tasa de disociación (kd) de 0.0236 (1/s) y una constante de disociación de equilibrio (KD) de 6.536 × 10-7 M.
Ensayo de bloqueo de PD-1/PD-L1 con inhibidor de molécula pequeña establecido: BMS-1166
Los datos se analizaron mediante el uso de los datos correspondientes. En Cinética/Afinidad, seleccione Límite de superficie, elija la curva 2-1 y, a continuación, obtenga la curva visualizada del resultado y la unidad de respuesta. El efecto de bloqueo de la interacción de unión PD-1/PD-L1 con BMS-1166 (0-3,125 μM) con 0,11 μM de proteína PD-L1 en el tampón HBS EP+ (Figura 5). La unidad de respuesta más alta se identifica solo con PD-L1, mientras que con el aumento de la concentración de BMS-1166, la unidad de respuesta de unión disminuye proporcionalmente.

Figura 1: Inmovilización de SA en el chip CM5. Se muestran las curvas de inmovilización de la proteína estreptavidina en la celda de flujo 1 (A) y la celda de flujo 2 (B) del chip CM5. En primer lugar, cinco preconcentraciones de la proteína SA fluyeron sobre las celdas de flujo 1 y 2, seguidas de un lavado de NaOH y el establecimiento de una línea de base estable. A continuación, se añadieron EDC y NHS, y luego se realizó un lavado con clorhidrato de etanolamina. A continuación, se realizaron cinco pulsos de PD-1 para alcanzar el nivel objetivo (2000 RU), seguidos de un lavado con clorhidrato de etanolamina para eliminar los ligandos unidos electrostáticamente y desactivar los ésteres del NHS que no son reactivos. Haga clic aquí para ver una versión más grande de esta figura.
Figura 2: Inmovilización de PD-1 en el chip SA. Se muestran las curvas de inmovilización de (A) la celda de flujo en blanco 1 recubierta solo con SA, y (B) la proteína PD-1 en el chip SA en la celda de flujo 2. Para el blanco (celda de flujo 1), se inyectaron tres veces 1 M de NaCl y 50 mM de NaOH, seguido de un lavado con 50% de isopropanol/1 M de NaCl/50 mM de NaOH. Para la proteína PD-1 (celda de flujo 2), se inyectaron tres veces 1 M de NaCl y 50 mM de NaOH, seguidas de cinco pulsos de proteína PD-1 para alcanzar el nivel objetivo de 4000 RU y un lavado final de 50% de isopropanol/1 M de NaCl/50 mM de NaOH. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Exploración de la regeneración para PD-1 y PD-L1. La línea de base y la respuesta de la muestra se obtuvieron para cuatro condiciones de regeneración con glicina a pH 1.5, 2, 2.5 y 3 para determinar la solución de regeneración óptima antes de la prueba de la muestra. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Validación de la interacción PD-1/PD-L1. Cinética de unión de diferentes concentraciones de PD-L1 (0,037 μM, 0,111 μM y 0,333 μM) a PD-1 en la superficie del chip recubierto de estreptavidina. PD-L1 muestra una clara asociación (0-60 s) y fases de disociación (61-120 s) con PD-1. Haga clic aquí para ver una versión más grande de esta figura.
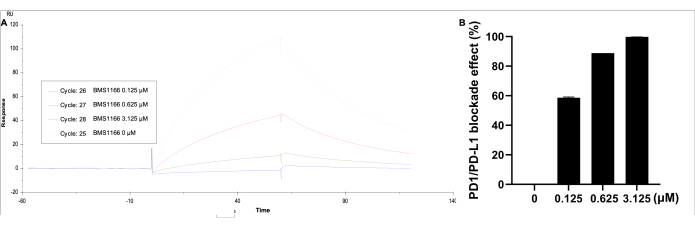
Figura 5: Análisis SPR de PD-1 recubierto en un chip CM5 inmovilizado SA, con PD-L1 (0,11 μM) y BMS-1166 (0-3,125 μM) en solución. (A) Una respuesta SPR representativa en tiempo real de las reacciones de unión entre PD-1 / PD-L1 con BMS-1166. (B) El efecto de bloqueo porcentual BMS-1166 en la cinética de unión de la interacción PD-1 / PD-L1. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Comparación de las estrategias de inmovilización de la proteína PD-1. Acoplamiento de aminas (A) vs. acoplamiento estreptavidina-biotina de doble paso (B). El acoplamiento de aminas presenta desafíos, incluida la disponibilidad limitada de sitios de unión accesibles, un obstáculo estérico para la unión y la posible modificación de los sitios de unión de PD-1 durante la inmovilización. Por el contrario, el enfoque de estreptavidina-biotina de doble paso mejora la disponibilidad de sitios de unión libres en PD-1 inmovilizado, lo que facilita una mejor interacción con PD-L1 en solución. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
En las últimas décadas, varios enfoques de inmunoterapia, incluidas las vacunas contra el cáncer, los inhibidores de puntos de control inmunitario y las terapias de células T con CAR, han avanzado significativamente en el tratamiento del cáncer21. Los puntos de control inmunitario desempeñan un papel crucial en la prevención del daño colateral mediado por las células inmunitarias durante las respuestas patógenas y en la supresión de la autoinmunidad. Un ejemplo clave es la interacción entre PD-L1 y PD-1, que forma un importante punto de control inmunitario, lo que permite a las células cancerosas evadir la vigilancia inmunitaria. El tratamiento de la vía PD-1/PD-L1 con anticuerpos monoclonales ha logrado un éxito notable en oncología clínica. Sin embargo, debido a las limitaciones asociadas con las terapias con anticuerpos monoclonales y la creciente incidencia de eventos adversos relacionados con el sistema inmunitario, existe un creciente interés en el desarrollo de inhibidores de moléculas pequeñas dirigidos a PD-1/PD-L1 9,22.
Las estrategias actuales de cribado de los inhibidores de PD-1/PD-L1 de moléculas pequeñas se centran principalmente en técnicas de bioensayo como ELISA, ensayos indicadores basados en células y ensayos de células T. Las técnicas biofísicas, como la SPR y la interferometría de biocapas (BLI), se utilizan ampliamente para la caracterización de las afinidades de unión, pero se subestima su potencial para ser utilizadas como herramientas de cribado23. Este estudio desarrolló un ensayo de bloqueo de PD-1/PD-L1 optimizado basado en SPR, que ofrece una plataforma de alto rendimiento adecuada para el descubrimiento de inhibidores de PD-1/PD-L1 de moléculas pequeñas. La SA se inmovilizó a ~2000 RU, seguida de PD-1 biotinilado (~4000 RU) a través de la interacción biotina-estreptavidina. Esta unión robusta garantizó un recubrimiento seguro de PD-1 con una orientación óptima, minimizando la unión inespecífica y reduciendo el uso de proteínas (10 μg/mL). La inmovilización dirigida al sitio mejoró la eficiencia en comparación con los métodos convencionales. Se utilizó tampón de glicina (pH 2.0) para regenerar la superficie del sensor entre ciclos, manteniendo la integridad experimental y evitando la unión inespecífica.
En comparación con los métodos inmunológicos estándar y ELISA, este enfoque SPR ofrecía una detección en tiempo real, sin marcadores, con alta sensibilidad y especificidad. Además, permite un cribado de alto rendimiento con un tiempo de ejecución de 120 s/muestra y complementa los bioensayos en la evaluación de la eficacia del bloqueo de compuestos y productos biológicos dirigidos a PD-1/PD-L1. El nivel de inmovilización alcanzado fue comparable a la plataforma anterior (3698.5 RU vs. 3688.5 RU), con una afinidad de interacción PD-1/PD-L1 similar (KD = 6.536 × 10-7 M vs. 1.295 × 10-7 M). El inhibidor de BMS-1166 demostró una mayor tasa de disociación a una concentración más baja, con efectos de bloqueo comparables a los de la plataforma anterior (99,8% frente a 94,2% a 3,125 μM). BMS-1166 demostró tasas de bloqueo de PD-1/PD-L1 respaldadas por valores de IC50 de 1,4 nM y 96 nM en ensayos HTRF y basados en células, respectivamente24. Además, otros estudios informaron valores de IC50 de 3,9 nM y 1574 nM utilizando HTRF y métodos de ensayo de cocultivo de bloqueo de puntos de control inmune25. Nuestros resultados previos mostraron que los valores de IC50 de BMS-1166 fueron de 85,4 nM, lo que es consistente con estos hallazgos anteriores18. Otra ventaja de esta plataforma de cribado es su robustez y alto rendimiento. Este método se aplicó ampliamente en el cribado de alto rendimiento utilizando un formato de placa de 384 pocillos, con BMS-1166 y BMS-202 incluidos como controles positivos para cada 10 muestras. Los rangos de tasa de bloqueo fueron de 29,8%-38,1% para BMS-1166 y de 6,0%-10,4% para BMS-202 a 10 nM (n = 11 por placa).
Es probable que el DMSO tenga efectos variables en membranas biológicas heterogéneas, dependiendo de su composición y estructura local, lo que podría influir en las funciones biológicas asociadas a la membrana. A concentraciones relativamente bajas, el DMSO puede alterar las propiedades de las proteínas en solución, lo que conduce a la desnaturalización, agregación o degradación. Además, el DMSO puede modificar las propiedades aparentes de unión de las proteínas26,27. Además, para abordar las preocupaciones anteriores con respecto a la interferencia de DMSO, redujimos la concentración de DMSO al 0,003 % (se usaba 0,01 % de DMSO en la plataforma anterior) en este protocolo.
En un estudio anterior, las proteínas PD-1 se inmovilizaron en chips sensores utilizando el acoplamiento de aminas, que emplea la química EDC/NHS para activar los grupos carboximetilo en el chip, formando enlaces covalentes con los grupos amina en PD-1. Debido a la presencia de múltiples grupos de aminas en los 288 aminoácidos de PD-1, este método da como resultado inherentemente orientaciones aleatorias. Para lograr una orientación más específica, empleamos una estrategia de acoplamiento de captura con PD-1 humano biotinilado (Fc, Avitag) en este estudio. El único residuo de lisina en el Avitag se biotinila enzimáticamente en la región Fc de PD-1, lo que permite una inmovilización precisa a través de la interacción de alta afinidad entre la biotina y la estreptavidina (Figura 6). Teóricamente, este enfoque asegura que el dominio variable de PD-1 permanezca expuesto, lo que facilita una interacción óptima con PD-L1 en el tampón. Sin embargo, esta suposición requiere una mayor validación utilizando técnicas como la crio-EM o la cristalografía para confirmar la orientación de las proteínas.
La glicina 2.0 se utilizó como un tampón de regeneración suave, eliminando eficazmente la PD-L1 sin reducir la PD-1 inmovilizada. Sin embargo, en el caso de los inhibidores estrechamente unidos o los agregados de proteínas, pueden ser necesarios tampones de regeneración más fuertes, como SDS al 0,5% o 50-100 mM de NaOH, para evitar interferencias con muestras posteriores.
Este estudio tiene varias limitaciones. En primer lugar, el alto costo de la instrumentación SPR puede limitar la accesibilidad, aunque el protocolo a menudo se puede adaptar para otros sistemas SPR con capacidades similares. Parámetros como la concentración de proteínas y los tiempos de asociación/disociación se pueden ajustar para adaptarse a plataformas SPR específicas. Otra limitación es su dependencia de las proteínas recombinantes, que pueden no replicar completamente las interacciones de las proteínas nativas. Estos factores deben tenerse en cuenta a la hora de interpretar los datos de SPR y comparar los resultados con técnicas complementarias, como los ensayos basados en células o los modelos in vivo .
A pesar de estas limitaciones, el método SPR optimizado ofrece un enfoque rápido, en tiempo real, de alto rendimiento y sin etiquetas para el cribado de inhibidores de moléculas pequeñas de la interacción PD-1/PD-L1. Mejora significativamente las técnicas biofísicas para caracterizar moléculas pequeñas y productos biológicos dirigidos a la interacción PD-1/PD-L1. Con su alta sensibilidad, la plataforma es particularmente valiosa para estudiar las interacciones de puntos de control inmunitarios y las interacciones proteína-proteína (PPI) más amplias, lo que la convierte en una poderosa herramienta para avanzar en el descubrimiento de fármacos PPI.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores reconocen la instalación central de RI-INBRE en la Universidad de Rhode Island, respaldada por Grant P20GM103430 del Centro Nacional de Recursos de Investigación (NCRR), un componente de los Institutos Nacionales de Salud (NIH). Esta investigación fue apoyada por una Subvención Piloto de la Facultad de Farmacia de la Universidad de Rhode Island, una Subvención Pequeña del Centro de Ciencias de la Vida de Rhode Island (RILSH) y una subvención de la Fundación de Rhode Island, todas otorgadas a Chang Liu, Ph.D.
Materiales
| Name | Company | Catalog Number | Comments |
| 50 mM NaOH | Cytiva Life Sciences | 100358 | |
| 50 mM NaOH | Fisher Scientific | 905376 | |
| 96-Well Polystyrene Microplates | Cytiva Life Sciences | BR100503 | |
| Amine Coupling Kit | Cytiva Life Sciences | 35120 | |
| Biacore T200 SPR System and Evaluation Software 3.2 | Cytiva Life Sciences | 28975001 | |
| Biotinylated Human PD-1 Fc, Avitag Protein | Acro Biosystems | PD1-H82F1 | |
| BMS1166 | MedChemExpress | HY-102011 | |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | 276855 | |
| DNase Free Water | Fisher Scientific | 188506 | |
| Glycine 1.5 | Cytiva Life Sciences | BR100354 | |
| Glycine 2.0 | Cytiva Life Sciences | BR100355 | |
| Glycine 2.5 | Cytiva Life Sciences | BR100356 | |
| Glycine 3.0 | Cytiva Life Sciences | BR100357 | |
| HBS-EP+ Buffer | Cytiva Life Sciences | BR100669 | |
| Human PD-L1 Fc Tag Protein | Acro Biosystems | PD-1-H5258 | |
| Isopropanol | Fisher Scientific | BP2618-1 | |
| Microplate Foil, 96-Well | Cytiva Life Sciences | 28975816 | |
| NaCl | Sigma-Aldrich | 746398 | |
| Plastic Vials 7 mm | Cytiva Life Sciences | BR100212 | |
| Rubber Caps, Type 3 | Cytiva Life Sciences | BR100502 | |
| Series S Sensor Chip CM5 | Cytiva Life Sciences | 29149603 | |
| Sodium Acetate 4.5 | Cytiva Life Sciences | 100350 | |
| Sodium Acetate 5.0 | Cytiva Life Sciences | 100351 | |
| Streptavidin | Sigma-Aldrich | S4762 |
Referencias
- Ribas, A., Wolchok, J. D. Cancer immunotherapy using checkpoint blockade. Science. 359 (6382), 1350-1355 (2018).
- Zhang, X., et al. Structural and functional analysis of the costimulatory receptor programmed death-1. Immunity. 20 (3), 337-347 (2004).
- Freeman, G. J., et al. Engagement of the PD-1 immunoinhibitory receptor by a novel B7 family member leads to negative regulation of lymphocyte activation. J Exp Med. 192 (7), 1027-1034 (2000).
- Han, Y., Liu, D., Li, L. PD-1/PD-L1 pathway: Current researches in cancer. Am J Cancer Res. 10 (3), 727-742 (2020).
- Lyu, N., et al. Recognition of PDL1/L2 by different induced-fit mechanisms of PD1: A comparative study of molecular dynamics simulations. Phys Chem Chem Phys. 22 (3), 1276-1287 (2020).
- Taube, J. M., et al. Colocalization of inflammatory response with B7-H1 expression in human melanocytic lesions supports an adaptive resistance mechanism of immune escape. Sci Trans Med. 4 (127), 127ra37 (2012).
- Sun, X., et al. Immune-related adverse events associated with programmed cell death protein-1 and programmed cell death ligand 1 inhibitors for non-small cell lung cancer: A PRISMA systematic review and meta-analysis. BMC Cancer. 19 (1), 558 (2019).
- Alkholifi, F. K., Alsaffar, R. M. Dostarlimab an inhibitor of PD-1/PD-L1: A new paradigm for the treatment of cancer. Medicina. 58 (11), 1572 (2022).
- Uzar, W., et al. An updated patent review on PD-1/PD-L1 antagonists (2022-present). Expert Opin Ther Pat. 34 (8), 627-650 (2024).
- Ai, L., et al. Research status and outlook of PD-1/PD-L1 inhibitors for cancer therapy. Drug Des Devel Ther. 14, 3625-3649 (2020).
- Conroy, M., Naidoo, J. Immune-related adverse events and the balancing act of immunotherapy. Nat Commun. 13 (1), 392 (2022).
- Guzik, K., et al. Small-molecule inhibitors of the Programmed Cell Death-1/Programmed Death-Ligand 1 (PD-1/PD-L1) interaction via transiently induced protein states and dimerization of PD-L1. J Med Chem. 60 (13), 5857-5867 (2017).
- Sifniotis, V., Cruz, E., Eroglu, B., Kayser, V. Current advancements in addressing key challenges of therapeutic antibody design, manufacture, and formulation. Antibodies. 8 (2), 36 (2019).
- Beck, H., Härter, M., Haß, B., Schmeck, C., Baerfacker, L. Small molecules and their impact in drug discovery: A perspective on the occasion of the 125th anniversary of the Bayer Chemical Research Laboratory. Drug Discov Today. 27 (6), 1560-1574 (2022).
- Nguyen, H. H., Park, J., Kang, S., Kim, M. Surface plasmon resonance: A Versatile technique for biosensor applications. Sensors (Basel, Switzerland). 15 (5), 10481-10510 (2015).
- Liu, C., Seeram, N. P., Ma, H. Small molecule inhibitors against PD-1/PD-L1 immune checkpoints and current methodologies for their development: A review. Cancer Cell Int. 21 (1), 239 (2021).
- Hu, K., Li, X. -. J., Asmamaw, M. D., Shi, X. -. J., Liu, H. -. M. Establishment of high-throughput screening HTRF assay for identification small molecule inhibitors of Skp2-Cks1. Sci Rep. 11 (1), 21105 (2021).
- Puopolo, T., et al. Establishment of human PD-1/PD-L1 blockade assay based on surface plasmon resonance (SPR) biosensor. Bio-protoc. 13 (15), e4765 (2023).
- Ding, M., Chen, Y., Lang, Y., Cui, L. The role of cellular prion protein in cancer biology: A potential therapeutic target. Front Oncol. 11, 742949 (2021).
- Li, H., Seeram, N. P., Liu, C., Ma, H. Further investigation of blockade effects and binding affinities of selected natural compounds to immune checkpoint PD-1/PD-L1. Front Oncol. 12, 995461 (2022).
- Kamrani, A., et al. New immunotherapeutic approaches for cancer treatment. Pathol Res Pract. 248, 154632 (2023).
- Yan, Y., Zhang, L., Zuo, Y., Qian, H., Liu, C. Immune checkpoint blockade in cancer immunotherapy: mechanisms, clinical outcomes, and safety profiles of PD-1/PD-L1 inhibitors. Arch Immunol Ther Exp. 68 (6), 36 (2020).
- Chandrasekharan, G., Unnikrishnan, M. High throughput methods to study protein-protein interactions during host-pathogen interactions. Eur J Cell Biol. 103 (2), 151393 (2024).
- Zhang, Y., et al. BMS-202, a PD-1/PD-L1 inhibitor, decelerates the profibrotic effects of fibroblasts derived from scar tissues via ERK and TGFβ1/Smad signaling pathways. Immun Inflamm Dis. 10 (1), e591 (2022).
- Surmiak, E., et al. PD-L1 inhibitors: Different classes, activities, and mechanisms of action. Int J Mol Sci. 22 (21), 11797 (2021).
- Feoli, A., Sarno, G., Castellano, S., Sbardella, G. DMSO-Related effects on ligand-binding properties of lysine methyltransferases G9a and SETD8. ChemBioChem. 25 (4), e202300809 (2024).
- Tjernberg, A., Markova, N., Griffiths, W. J., HalléN, D. DMSO-Related effects in protein characterization. SLAS Discov. 11 (2), 131-137 (2006).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados