Method Article
表面プラズモン共鳴技術によるPD-1/PD-L1阻害剤の同定
要約
このプロトコルは、表面プラズモン共鳴技術を使用したPD-1/PD-L1阻害剤の遮断アッセイを説明しています。デュアルステップの固定化戦略とカスタマイズされたバッファーシステムを採用して、レスポンスユニットを正確に測定し、化合物や生物製剤の封鎖率の評価を容易にします。さらに、PD-1/PD-L1阻害剤のハイスループット同定をサポートします。
要約
PD-1/PD-L1相互作用の破壊は、がん免疫療法の有望な戦略です。PD-1/PD-L1阻害剤の有効性を評価するためには、信頼性の高いスクリーニングプラットフォームが不可欠です。表面プラズモン共鳴(SPR)技術を利用した以前に確立されたヒトPD-1/PD-L1阻害アッセイ(第1世代PD-1/PD-L1阻害剤SPRスクリーニングプラットフォーム)は、均質時間分解蛍光(HTRF)および細胞ベースのアッセイで得られたものと同等の結果を示し、大規模なスクリーニングの可能性を秘めています。ここでは、このアッセイの最適化バージョン(第2世代PD-1/PD-L1阻害剤SPRスクリーニングプラットフォーム)を提示し、アミンとバイオストレプトアビジンカップリングを組み合わせてチップ上のPD-1配向制御を強化し、PD-1タンパク質消費を削減するデュアルステップカップリングプロセスを特徴としています。更新されたプラットフォームは、PD-1/PD-L1阻害剤BMS-1166を使用して検証に成功し、以前のSPRベースの方法やELISAなどの他の確立された技術に匹敵する阻害効果を示しました。これらの結果は、アプローチの信頼性を裏付けています。この最適化されたSPRスクリーニングプラットフォームは、新規のPD-1/PD-L1阻害剤を同定し、がん免疫療法研究を前進させ、免疫チェックポイント阻害剤スクリーニングにおけるSPRの可能性を強調するためのハイスループットで信頼性の高いツールを提供します。
概要
免疫チェックポイント阻害療法、特にプログラム細胞死-1(PD-1)およびプログラム細胞死リガンド1(PD-L1)を標的とする治療法は、がん免疫療法戦略の最前線に立っています。抗PD-1/PD-L1療法は、血液がん、皮膚がん、肺がん、肝臓がん、膀胱がん、腎がんなど、さまざまな種類のがんに対する使用が承認されています1。PD-1は、免疫グロブリンスーパーファミリーに属する膜貫通型糖タンパク質であり、N末端に単一の免疫グロブリン変数(IgV)様ドメイン、IgVドメインを原形質膜から分離する約20アミノ酸の茎、膜貫通ドメイン、およびチロシンベースのシグナル伝達モチーフを含む細胞質尾部を特徴とする2。PD-L1は、PD-1のリガンドの1つとして同定され、膜貫通領域、2つの細胞外ドメイン(免疫グロブリン定数(IgC)とIgV)、および細胞内シグナル伝達経路を誘発する比較的短い細胞質ドメインを特徴とするI型膜貫通タンパク質です3。PD-1/PD-L1阻害経路は、T細胞の活性化と自己免疫を調節する重要な免疫チェックポイントとして機能します4。PD-1はT細胞上に発現し、PD-L1と相互作用し、T細胞受容体シグナル伝達を阻害し、抗原提示細胞およびT細胞上のCD28およびCD80分子の刺激を阻害します5。がん組織は、脱出期にPD-L1を過剰発現させることでこの生理学的メカニズムを利用し、腫瘍の成長と進行を促進する免疫抑制環境を作り出します6。PD-1およびPD-L1の阻害剤は、この相互作用を阻害し、免疫系が腫瘍誘発性の抑制を回避し、T細胞を介した腫瘍細胞死プロセスを再開することを可能にします7。
免疫チェックポイント阻害療法の顕著な役割によって築かれた基盤の上に、PD-1/PD-L1阻害剤の開発は、がん免疫療法の大きな進歩をマークしました。米国食品医薬品局(FDA)は、PD-1/PD-L1経路を特異的に標的とする9つの免疫チェックポイント阻害剤を承認しました。これらには、6つのPD-1阻害剤-ペムブロリズマブ、ドスタリマブ、ニボルマブ、セミプリマブ、オリパリマブ、およびチスレリズマブ-、および3つのPD-L1阻害剤-アテゾリズマブ、アベルマブ、およびデュルバルマブが含まれます8,9。これらの治療法は、黒色腫、肺がん、尿路上皮がん、子宮頸がん、胃がんまたは胃食道がん、その他の固形腫瘍など、さまざまながんの治療に効果的に利用されてきました10。その有効性にもかかわらず、モノクローナル抗体ベースの治療は、低奏効率、高コスト、長期半減期、重度の免疫関連有害事象、静脈内または皮下送達の制限など、重大な制限に直面しています11,12,13。その結果、PD-1/PD-L1軸を標的とする低分子阻害剤の開発に研究がますます注力されています。これらの低分子は、細胞浸透の改善、多様な生物学的標的の調節、経口バイオアベイラビリティの向上、コストの削減などの明確な利点を提供し、副作用の少ない同等の治療結果を達成することを目標としています14。しかし、PD-1/PD-L1相互作用を標的とする低分子阻害剤の開発は、主に信頼性の高いハイスループットスクリーニングプラットフォームがないため、初期段階にあります。このようなプラットフォームは、低分子の膨大なライブラリーを迅速に評価し、さらなる検証と最適化のためにリード化合物を同定するために不可欠です。この課題を克服することは、がん免疫療法を進歩させるために重要です。
表面プラズモン共鳴(SPR)技術は、抗体抗原、酵素、核酸、薬物などのさまざまな生体分子の検出に広く採用されており、特に低分子薬物スクリーニングに効果的です15,16。他の生物物理学的手法とは異なり、SPRはラベルフリーの検出、リアルタイムのカイネティックデータ、および幅広い検出範囲を提供します。対照的に、等温滴定型カロリメトリーは、リアルタイムの速度論的洞察を欠いており、より多くのサンプル量を必要とするため、スループットが制限されます。マイクロスケールの熱泳動はバッファー干渉を受けやすく、速度論的データを提供できませんが、バイオレイヤー干渉法は分子サイズと特性に基づくアプリケーション固有の制限があります。均質な時間分解蛍光は、標識が必要であり、蛍光干渉の影響を受けやすいです。HTRFは、PD-1/PD-L1阻害剤を探索するのに適した別の技術であると認識しています。HTRFの固有の制限の1つは、SPRと比較して、分子内励起プロセス(電子移動、FRETおよび漂白など)との外部相互作用によって引き起こされる蛍光消光であり、ウィンドウ範囲が狭いため、薬物スクリーニングプロセスにおける感度が低すぎること、および蛍光ライブラリー化合物または生物学的タンパク質からの干渉17.これらの特徴により、SPRは創薬のための優れたツールとして位置付けられています。私たちの以前の研究では、SPRがPD-1 / PD-L1に対する低分子のブロック効果を決定できることが実証されており、これは、創薬プロセスで高い標識技術要件、複数のステップ、低い特異性、および高いコストを必要とする他の技術よりも有利です18。
この研究では、最適化されたSPRベースのプラットフォームを導入し、アミンカップリングとバイオストレプトアビジンカップリングの両方を利用するデュアルステップカップリングプロセスを統合して、チップ上のPD-1の配向を強化し、タンパク質の使用量を最小限に抑えます。この更新されたアプローチは、PD-1/PD-L1阻害剤BMS-1166をポジティブコントロールバインダーとして使用して検証に成功し、以前のSPR法とELISA19,20などの他の確立された技術の両方に匹敵する遮断効果が実証されました。これは、当社のプロトコールの信頼性と再現性を確認するだけでなく、PD-1/PD-L1阻害剤のハイスループットスクリーニングを容易にするための改良プラットフォームの有効性を示しています。バイオストレプトアビジン捕捉ステップを組み込むことで、ランダムなタンパク質配向ではなく部位指向性が得られるため、PD-1濃度の低下(40 μg/mL 対 10 μg/mL)が可能になり、エンドユーザーはストレプトアビジン(SA)をCM5チップに固定化することができ、商品化された固定化済みSAチップに代わる安価な代替品となるため、コストを節約できます。これにより、化合物/ペプチドライブラリーの大規模で費用対効果の高いスクリーニングに有利になります。がんに対するPD-1/PD-L1阻害剤の臨床的可能性を評価するには、in silico、in vitro、in vivoアッセイなどの追加の特性評価法が不可欠ですが、当社の強化されたSPRベースのスクリーニングプラットフォームは、PD-1/PD-L1阻害剤の大規模なスクリーニングのための効率的なツールとして際立っています。
プロトコル
試薬と機器は 、材料表に記載されています。
1. CM5チップ上のストレプトアビジン(SA)タンパク質の固定化
- SPR装置で固定化方法を設定します: オープン/新規ウィザード テンプレート、 固定化を選択し、チップタイプを CM5に設定し、サイクルあたりのフローセル数を 1に設定します。 immobilize flow cell 1 と flow cell 2 にチェックを入れます。
- 固定化方法として Amine を設定します。固定化レベル、リガンド濃度: 40 μg/mL ストレプトアビジン、目標レベル: 2000 RU、洗浄液: 50 mM NaOHの目標を設定します。次に、実行する前にプライムを確認します。
- 次のチューブを準備します:R2 B1およびR2 C1 - 40μg/ mLストレプトアビジン;R2 B2およびR2 C2:50 mMのNaOH;R2 B3 および R2 C3: EDC;R2 B4 および R2 C4: NHS;R2 B5 および R2 C5: 空。R2 B6およびR2 C6:エタノールアミン。
注:EDCとNHSはCM5チップ上のカルボキシル基を活性化し、リガンド上のアミンと共有結合を可能にします。エタノールアミンは、固定化中の非特異的結合を防ぐためのブロッキングバッファーとして使用されます。 - 20 mLのHBS-EP+バッファーを180 mLの脱イオン(DI)水で10×希釈し、200 mLの1× HBS-EP+ランニングバッファー溶液を調製します。
- 1 mLのDNaseフリー水1 mgにストレプトアビジンを加え、室温で30分間インキュベートします。次に、ストレプトアビジン溶液を酢酸緩衝液(pH 4.5)で40 μg/mLに希釈します。製造元の指示に従ってAmine Coupling試薬キットの試薬を調製し、ステップ1.2に示されているように、関連するすべての試薬のレイアウトを配置します。
- メンテナンスセンサーチップをCM5チップに交換し、用意した1× HBS-EP+バッファーにチューブAを入れ、固定化方法を再度開き、手順1.2のレイアウトに従ってチューブを挿入します。メソッド を実行し 、結果ファイルを保存します。
- 運転後のメンテナンス:CM5チップをメンテナンスセンサーチップに交換し、脱イオン水で満たされたボトルにチューブAを入れ、プライムを実行します。CM5チップを取り出した後、チップを数滴のDI水で洗い、自然乾燥させます。チップを4°Cの50 mLチューブに入れます。
注意: 固定化されたSAにより、CM5チップはSAチップとして機能します。
2. PD-1タンパク質のSAチップへの固定化
- SPR装置で固定化方法を設定します: オープン/新規ウィザード テンプレート、 固定化を選択し、チップタイプを SAに設定し、サイクルあたりのフローセル数を 1に設定します。 immobilize flow cell 1 と cell 2 にチェックを入れます。
- 方法としてSA-ビオチン捕捉を設定します。セル 1 セットのブランク固定化の場合、セル 2 セットの固定化レベル、リガンドとして 10 μg/mL PD-1、目標レベル:4000 RU を目指し、実行前にプライムを確認してください。
注:次のチューブを準備します-R2 B1およびR2 C1:1 MのNaCl、50 mMのNaOH;R2 B2およびR2 C2:50%イソプロパノール/50 mMのNaOH/1 MのNaCl;R2 C3:10 μg/mL PD-1。
- 方法としてSA-ビオチン捕捉を設定します。セル 1 セットのブランク固定化の場合、セル 2 セットの固定化レベル、リガンドとして 10 μg/mL PD-1、目標レベル:4000 RU を目指し、実行前にプライムを確認してください。
- 58.44 mgのNaClを1 mLの50 mM NaOHに溶解し、1 MのNaClおよび50 mMのNaOH溶液を調製します。58.44 mgのNaClと4.0 mgのNaOHを500 μLの水に溶解し、500 μLのイソプロパノールを加えて50%イソプロパノール/50 mM NaOH/1 M NaCl溶液を調製します。
- PD-1溶液を調製する:200 μLのDNaseフリー水1を100 μgのビオチン化ヒトPD-1(FcおよびAvitagged)に加え、室温で30分間安定させた後、酢酸緩衝液(pH 5.0)で10 μg/mLに希釈します。
- ステップ2.1で説明したように、すべての試薬レイアウトを配置します。1× HBS-EP+ランニングバッファー溶液にチューブAを入れ、メンテナンスセンサーチップを取り出してSAチップ(ステップ1のストレプトアビジンタンパク質でコーティングしたCM5チップ)を挿入します。固定化法を再度開き、試薬ラック2を挿入して位置を確認し、推定実行時間で方法を実行します。
- 手順1.6を繰り返します。
3. PD-1、PD-L1の再生スカウティング
- 再生スカウティング方法の設定: ウィザードテンプレートを開く/新規 、 再生スカウティングを選択し、流路を 2-1、4-3、チップタイプを SAに設定し、ランコンディショニングサイクルを確認し、溶液をHBS-EP+として記録し、接触時間を 30秒、注入回数を 3回、溶液を PD-L1として、接触時間: 30秒、流量: 30μL / min。
- 再生パラメータの場合、流速は30μL/min、安定化期間は300秒です。実験計画では、条件の数を4に設定し、各条件のサイクル数を2に設定します。条件を4、再生溶液:グリシン1.5、2.0、2.5、3.0、接触時間:30秒に設定し、実行する前にプライムを確認してください。
- 各 PD-L1 濃度を 96 ウェルマイクロプレートレイアウトで個別のサンプルウェル位置として設定します: R1 A1 から R1 A9: 1 μM PD-L1;R1 A10 から R1 A12: HBS-EP+ バッファー;R1 B1:グリシン1.5;R1 B2:グリシン2;R1 B3:グリシン2.5;R1 B4:グリシン3。
注:再生緩衝液として、pHの異なるグリシン緩衝液を使用します。 - PD-L1溶液を調製する:100 μgのHuman PD-L1 Fcタグタンパク質(9.42 μM)に200 μLのDNaseフリー水を加え、HBS-EP+バッファーで1 μMに希釈します。
- ステップ3.2で説明したように、関連するすべての試薬をレイアウトに配置します。チューブAを1×HBS-EP+バッファーに入れ、メンテナンスセンサーチップをSAチップに交換します。 Regeneration Scouting メソッドを再度開き、ステップ3.2のチューブ位置に従い、試薬ラックを挿入してから、推定実行時間でメソッド を実行します 。
- 手順1.6を繰り返します。
4. PD-1/PD-L1相互作用の検証
注:検証のために、以前に公開されたレポート18 に続いて、若干の調整が加えられました。
- [General Settings]、[Assay Steps]、および[Cycle Types]で公開されたレポートと同じパラメーターを使用し、接触時間を60秒、解離時間を60秒、流量を30μL/minに設定します。メソッド変数、評価変数、コマンドでは、Glycine 2.0をRegenerationとして使用する以外は、同じ設定を使用します。ただし、セットアップ流量は30μL/minで、1、2、3、4の流路を経由します。
- 次に、 runを設定し、流路( 2-1、4-3)を選択します。0 μM、0.037 μM、0.111 μM、0.333 μM、分子量51,300 Daの濃度でPD-L1をインプットします。すべてのアッセイステップで検証を行い、実行前にプライムを選択します。
- 次に、各 PD-L1 濃度を Reagent Rack 2 レイアウトの個別のサンプルウェル位置として設定します: R2 B1: PD-L1 0 μM;R2 B2: PD-L1 0.037 μM;R2 B3: PD-L1 0.111 μM;R2 B4: PD-L1 0.333 μM;R2 A1:再生バッファーとしてのグリシン2.0。R2 A2:起動バッファとしてHBS-EP +。
- 1× HBS-EP+ランニングバッファー溶液200 mLを調製します。PD-L1濃度を調製する:HBS-EP+でPD-L1タンパク質を9.42 μMから0.333 μM、0.111 μM、および0.037 μMで希釈します。次に、ステップ4.1.2で説明したように、関連するすべての試薬をレイアウトに配置します。
- チューブAを1×HBS-EP+ランニングバッファー溶液に挿入し、メンテナンスセンサーチップを排出し、SAチップを挿入します。 再生スカウティング 法を再度開き、ステップ4.1のチューブの位置決めに従い、試薬ラック2を挿入し、推定時間だけメソッド を実行します 。
- 手順1.6を繰り返します。
5. 低分子阻害剤を用いたPD-1/PD-L1阻害法:BMS-1166
注:封鎖アッセイについては、以前に発表されたレポート18 にわずかな調整を加えて追跡しました。
- [General Settings]、[Assay Steps]、および[Cycle Types]で公開されたレポートと同じパラメーターを使用し、接触時間を60秒、解離時間を60秒、流量を30μL/minに設定します。メソッド変数、評価変数、コマンドも同じ設定を使用しますが、Glycine 2.0をRegenerationとして使用する以外は、30μL/minへの設定流量は、1、2、3、4の流路を経由します。
- 次に、runを設定し、フローパス( 2-1、4-3)を選択します。サンプル溶液:PD-L1(0.111 μM、分子量51,300 Da)とBMS-1166を0 μM、0.125 μM、0.625 μM、3.125 μMで入力します。
- 次に、各 PD-L1 濃度を Reagent Rack 2 レイアウトの個別のサンプルウェル位置として設定します: R2 B1: PD-L1 (0.111 μM) + BMS-1166 0 μM R2 B2: PD-L1 (0.111 μM) + BMS-1166 0.125 μM;R2 B3: PD-L1 (0.111 μM) + BMS-1166 0.625 μM;R2 B4: PD-L1 (0.111 μM) + BMS-1166 3.125 μM;R2 A1:再生のためのグリシン2.0。
- 1× HBS-EP+ランニングバッファー溶液200 mLを調製します。BMS-1166/PD-L1混合物の調製:5 mgのBMS-1166を77.99 μLのジメチルスルホキシド(DMSO)に溶解して、100 mMのストック溶液を調製します。ストック溶液をPD-L1タンパク質(0.11 μM)でBMS-1166の目標濃度まで0 μM、0.125 μM、0.625 μM、およびHBS-EP+で希釈します。ステップ5.1.2で説明されているように、関連するすべての試薬レイアウトを配置します。
- チューブAを1×HBS-EP+ランニングバッファー溶液に入れ、メンテナンスセンサーチップを排出してSAチップを挿入します。 再生スカウティング メソッドを再度開き、5.1からのチューブ位置をたどって、試薬ラック2を挿入し、推定実行時間でメソッド を実行します 。
- 手順1.6を繰り返します。
結果
CM5チップ上のSAの固定化
SPR装置および関連する解析ソフトウェアからの出力 により 、フローセル1およびフローセル2上のSAタンパク質の目標RU(2000RU)が成功裏に達成されたことを示しました。フローセル 1 および 2 は、CM5 チップ表面に SA(40 μg/mL)を添加して固定化し、最終レスポンスはフローセル 1 で 1902.3 RU(図 1A)、フローセル 2 で 1900.7 RU(図 1B)でした。
PD-1のSAチップへの固定化
SPR 装置および関連ソフトウェアからの出力に基づくデータ解析では、フローセル 1 のブランクセルのレスポンスユニット(RU)が低く、フローセル 2 の PD-1 リガンドの目標 RU(4000 RU)の達成に成功したことが示されました。フローセル 1 をブランクとして固定化したところ、最終レスポンスは -161.0 RU (図 2A)、フローセル 2 は SA チップ上にコーティングしたビオチン化 PD-1 タンパク質 (10 μg/mL) で固定化した結果、最終レスポンスは 3698.5 RU でした (図 2B)。
PD-1とPD-L1の再生スカウティング。
再生スカウティングは、PD-1をフローセル2に固定化し、PD-L1をさまざまなグリシンpHレベル(1.5、2、2.5、および3)の溶液(0.1 μM)に溶解して実施し、安定したベースラインとサンプルレスポンスをもたらす再生溶液を決定しました。SPR装置および分析ソフトウェアからの出力 を介して データを分析した結果、ベースラインとサンプルのレスポンスの両方についてレスポンスの変化が最小限に抑えられるため、グリシン(pH 2.0)が最適な再生条件として得られ、PD-1タンパク質の損失が最小限に抑えられ、チップ表面からのPD-L1タンパク質の除去が成功したことが示されています。Glycine pH 2.0では、ベースラインレスポンスは比較的一定に保たれ、分析種のレスポンスは安定しており、実験開始時のレスポンスに近く、試験した4つのGlycine pH条件の中で最も最適な再生バッファーが示されました。pHが高いと不十分になり、pHが低いと厳しすぎます。pH 2.0は、最も適切な再生条件として特定されています(図3)。
PD-1/PD-L1相互作用の検証
データは、対応する評価ソフトウェアを使用して分析しました。[Kinetics/Affinity]セクションで、[surface-bound]を選択し、[curve 2-1]を選択し、動力学モデルを1:1バインディングに設定し、RIパラメータを定数フィットに調整して、関連付け率(ka)、解離率(kd)、および平衡解離定数(KD)を決定します。さまざまな濃度でのPD-L1とPD-1との結合相互作用を定量し、測定可能な応答が得られました(図4)。解析した結合パラメータには、3.611 × 104(1/Ms)の会合速度(ka)、0.0236(1/s)の解離率(kd)、および6.536 × 10-7 Mの平衡解離定数(KD)が含まれていました。
確立された低分子阻害剤によるPD-1/PD-L1阻害法:BMS-1166
データは、対応するものを用いて分析した。Kinetics/Affinityの下で、surface-boundを選択し、曲線2-1を選択してから、結果と応答ユニットの可視化曲線を取得します。PD-1/PD-L1結合相互作用の阻害効果は、HBS EP+バッファー中のBMS-1166(0-3.125 μM)と0.11 μM PD-L1タンパク質で観察されました(図5)。最も高い応答単位はPD-L1のみで特定されますが、BMS-1166濃度が増加すると、結合応答単位は比例して減少します。

図1:CM5チップ上のSAの固定化。 CM5チップフローセル1(A)およびフローセル2(B)上のストレプトアビジンタンパク質の固定化曲線を示す。まず、5つの前濃度のSAタンパク質をフローセル1および2に流し、続いてNaOH洗浄を行い、安定したベースラインを確立しました。次に、EDCとNHSを添加し、その後、エタノールアミン塩酸塩洗浄を行った。次に、PD-1を5パルス投与して目標レベル(2000 RU)に到達させ、続いてエタノールアミン塩酸塩洗浄を行い、静電結合したリガンドを除去し、反応性のないNHSエステルを不活性化しました。 この図の拡大版を表示するには、ここをクリックしてください。
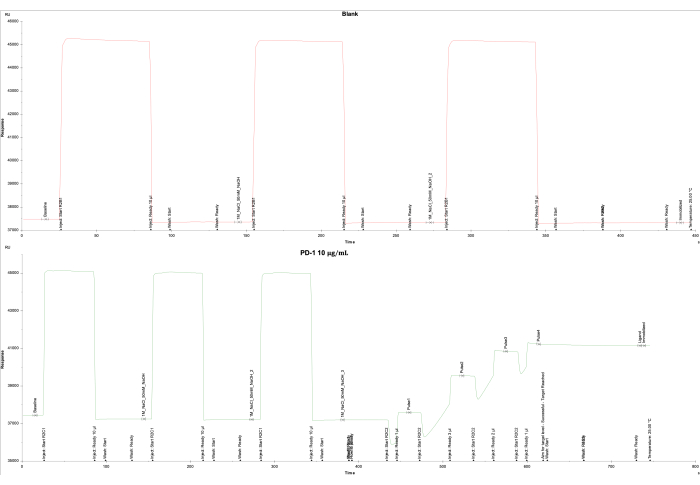
図2:SAチップ上のPD-1の固定化。 (A)ブランクフローセル1をSAのみで被覆し、(B)フローセル2上のSAチップ上のPD−1タンパク質の固定化曲線を示す。ブランク(フローセル1)については、1 MのNaClと50 mMのNaOHを3回注入した後、50%イソプロパノール/1 MのNaCl/50 mMのNaOHで洗浄しました。PD-1タンパク質(フローセル2)については、1 MのNaClと50 mMのNaOHを3回注入し、続いてPD-1タンパク質を5パルス注入して目標レベルの4000 RUに到達し、最終的に50%イソプロパノール/1 MのNaCl/50 mMのNaOHを洗浄しました。 この図の拡大版を表示するには、ここをクリックしてください。

図3:PD-1とPD-L1の再生スカウティング。ベースラインレスポンスとサンプルレスポンスは、pH 1.5、2、2.5、および 3 のグリシンを使用した 4 つの再生条件で取得し、サンプルテスト前に最適な再生ソリューションを決定しました。 この図の拡大版を表示するには、ここをクリックしてください。

図4:PD-1/PD-L1相互作用の検証。 ストレプトアビジンでコーティングされたチップ表面上のPD-1に、異なる濃度のPD-L1(0.037 μM、0.111 μM、0.333 μM)の結合速度論。PD-L1は、PD-1との明確な関連(0〜60秒)および解離相(61〜120秒)を示します。 この図の拡大版を表示するには、ここをクリックしてください。
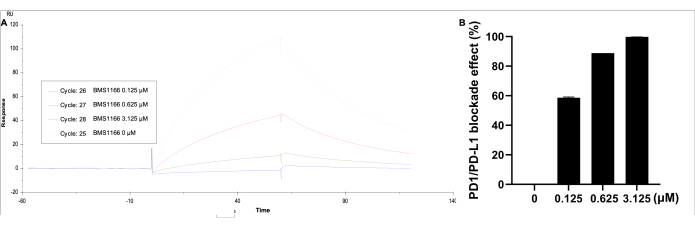
図5:溶液中のPD-L1(0.11 μM)およびBMS-1166(0-3.125 μM)を用いた、SA固定化CM5チップ上にコーティングされたPD-1のSPR分析(A)PD-1/PD-L1とBMS-1166との間の結合反応の代表的なリアルタイムSPR応答。(B)PD-1 / PD-L1相互作用結合速度論に対するBMS-1166の遮断効果の割合。この図の拡大版を表示するには、ここをクリックしてください。

図6:PD-1タンパク質固定化戦略の比較。 アミンカップリング(A) 対デュアルステップストレプトアビジン-ビオチンカップリング(B)。アミンカップリングには、アクセス可能な結合部位の利用可能性が限られている、結合に対する立体障害、固定化中のPD-1結合部位の潜在的な修飾などの課題があります。対照的に、デュアルステップのストレプトアビジン-ビオチンアプローチは、固定化されたPD-1上の遊離結合部位の利用可能性を高め、溶液中のPD-L1との相互作用を改善します。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
過去数十年にわたり、がんワクチン、免疫チェックポイント阻害剤、CAR-T細胞療法など、さまざまな免疫療法のアプローチにより、がん治療は大幅に進歩しました21。免疫チェックポイントは、病原性応答中の免疫細胞による巻き添え被害の予防や自己免疫の抑制に重要な役割を果たします。その代表的な例が、PD-L1とPD-1の相互作用で、PD-L1は主要な免疫チェックポイントを形成し、がん細胞が免疫監視を回避することを可能にします。PD-1/PD-L1経路をモノクローナル抗体で標的とすることは、臨床腫瘍学において目覚ましい成功を収めています。しかし、モノクローナル抗体療法に関連する制限や免疫関連の有害事象の発生率の増加により、PD-1/PD-L1を標的とする低分子阻害剤の開発への関心が高まっています9,22。
低分子PD-1/PD-L1阻害剤の現在のスクリーニング戦略は、主にELISA、細胞ベースのレポーターアッセイ、T細胞アッセイなどのバイオアッセイ技術に焦点を当てています。SPRやバイオレイヤーインターフェロメトリー(BLI)などの生物物理学的手法は、結合親和性の特性評価に広く使用されていますが、スクリーニングツールとして使用される可能性は過小評価されています23。この研究では、低分子PD-1/PD-L1阻害剤の探索に適したハイスループットプラットフォームを提供する、最適化されたSPRベースのPD-1/PD-L1阻害アッセイを開発しました。SAを~2000 RUに固定化し、続いてビオチン-ストレプトアビジン相互作用 を介して ビオチン化PD-1(~4000 RU)を固定化しました。この強固な結合により、最適な配向性で安全なPD-1コーティングが保証され、非特異的結合が最小限に抑えられ、タンパク質の使用量が削減されました(10 μg/mL)。サイト指向の固定化は、従来の方法と比較して効率が向上しました。グリシンバッファー(pH 2.0)を使用して、サイクル間でセンサー表面を再生し、実験の完全性を維持し、非特異的結合を防止しました。
標準的な免疫学的方法やELISAと比較して、このSPRアプローチは、高い感度と特異性を備えたリアルタイムのラベルフリー検出を提供しました。さらに、120 s/サンプルの実行時間でハイスループットスクリーニングを可能にし、PD-1/PD-L1標的化合物および生物製剤のブロック効率を評価するバイオアッセイを補完します。達成された固定化レベルは、以前のプラットフォームと同等(3698.5 RU対3688.5 RU)で、PD-1/PD-L1相互作用親和性は同程度でした(KD = 6.536 × 10-7 M 対1.295 × 10-7 M)。BMS-1166阻害剤は、低濃度でより高い解離率を示し、遮断効果は以前のプラットフォームに匹敵しました(3.125μMで99.8% 対94.2%)。BMS-1166は、HTRFおよび細胞ベースのアッセイでそれぞれ1.4nMおよび96nMのIC50 値によって支持されるPD-1/PD-L1遮断率を示しました24。さらに、他の研究では、HTRFおよび免疫チェックポイント遮断共培養アッセイ法を用いて、3.9nMおよび1574nMのIC50 値が報告された25。以前の結果では、BMS-1166のIC50 値は85.4 nMであり、これはこれらの以前の調査結果と一致していることを示しました18。このスクリーニングプラットフォームのもう一つの利点は、その堅牢性と高いスループットです。この分析法は、384ウェルプレートフォーマットを用いたハイスループットスクリーニングに広く適用され、BMS-1166およびBMS-202は10サンプルごとにポジティブコントロールとして含まれていました。封鎖率の範囲は、10 nM(n = 11 / プレート)でBMS-1166で29.8%〜38.1%、BMS-202で6.0%〜10.4%でした。
DMSOは、その局所的な組成と構造に応じて、不均一な生体膜にさまざまな影響を与える可能性が高く、膜に関連する生物学的機能に影響を与える可能性があります。比較的低濃度では、DMSOは溶液中のタンパク質特性を変化させ、変性、凝集、または分解を引き起こす可能性があります。さらに、DMSOはタンパク質の見かけの結合特性を修飾し得る26,27。さらに、DMSO干渉に関する以前の懸念に対処するために、このプロトコルではDMSO濃度を0.003%に下げました(以前のプラットフォームでは0.01%のDMSOが使用されていました)。
これまでの研究では、EDC/NHSの化学的性質を利用してチップ上のカルボキシメチル基を活性化し、PD-1上のアミン基と共有結合を形成するアミンカップリングを用いて、PD-1タンパク質をセンサーチップに固定化しました。PD-1の288アミノ酸に複数のアミン基が存在するため、この方法では本質的にランダムな配向が生じます。より特異的な方向性を達成するために、この研究では、ビオチン化ヒトPD-1(Fc、Avitag)との捕捉カップリング戦略を採用しました。Avitagの単一のリジン残基は、PD-1のFc領域で酵素的にビオチン化されており、ビオチンとストレプトアビジンの間の高親和性相互作用による正確な固定化を可能にします(図6)。理論的には、このアプローチにより、PD-1の可変ドメインが露出したままになり、バッファー内のPD-L1との最適な相互作用が容易になります。しかし、この仮定には、クライオ電子顕微鏡や結晶構造解析などの技術を使用して、タンパク質の配向を確認するためのさらなる検証が必要です。
グリシン2.0はマイルドな再生バッファーとして使用され、固定化されたPD-1を減少させることなくPD-L1を効果的に除去しました。ただし、緊密に結合した阻害剤またはタンパク質凝集体の場合、後続のサンプルとの干渉を防ぐために、0.5%SDSまたは50〜100mMのNaOHなどのより強力な再生バッファーが必要になる場合があります。
この研究にはいくつかの制限があります。まず、SPR機器はコストが高いため、アクセス性が制限される可能性がありますが、このプロトコルは、同様の機能を持つ他のSPRシステムにも適用できることがよくあります。タンパク質濃度や会合/解離時間などのパラメータは、特定のSPRプラットフォームに合わせて調整できます。また、組換えタンパク質に依存しているため、天然のタンパク質相互作用を完全には再現できないという制約もあります。SPRデータを解釈し、結果を細胞ベースのアッセイや in vivo モデルなどの補完的な手法と比較する際には、これらの要素を考慮する必要があります。
これらの制限にもかかわらず、最適化されたSPR法は、PD-1/PD-L1相互作用の低分子阻害剤をスクリーニングするための迅速、リアルタイム、ハイスループット、およびラベルフリーのアプローチを提供します。これにより、PD-1/PD-L1相互作用を標的とする低分子および生物製剤の特性評価のための生物物理学的手法が大幅に向上します。その高い感度により、このプラットフォームは免疫チェックポイント相互作用やより広範なタンパク質間相互作用(PPI)の研究に特に価値があり、PPI創薬を進めるための強力なツールとなっています。
開示事項
著者は何も開示していません。
謝辞
著者たちは、ロードアイランド大学のRI-INBRE中核施設が、国立衛生研究所(NIH)の一部門である全米研究資源センター(NCRR)のグラント・P20GM103430の支援を受けていることを認めている。この研究は、ロードアイランド大学薬学部のパイロット助成金賞、ロードアイランドライフサイエンスハブ(RILSH)のSmall Grant Award、ロードアイランド財団の助成金によって支援され、すべてChang Liu博士に授与されました。
資料
| Name | Company | Catalog Number | Comments |
| 50 mM NaOH | Cytiva Life Sciences | 100358 | |
| 50 mM NaOH | Fisher Scientific | 905376 | |
| 96-Well Polystyrene Microplates | Cytiva Life Sciences | BR100503 | |
| Amine Coupling Kit | Cytiva Life Sciences | 35120 | |
| Biacore T200 SPR System and Evaluation Software 3.2 | Cytiva Life Sciences | 28975001 | |
| Biotinylated Human PD-1 Fc, Avitag Protein | Acro Biosystems | PD1-H82F1 | |
| BMS1166 | MedChemExpress | HY-102011 | |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | 276855 | |
| DNase Free Water | Fisher Scientific | 188506 | |
| Glycine 1.5 | Cytiva Life Sciences | BR100354 | |
| Glycine 2.0 | Cytiva Life Sciences | BR100355 | |
| Glycine 2.5 | Cytiva Life Sciences | BR100356 | |
| Glycine 3.0 | Cytiva Life Sciences | BR100357 | |
| HBS-EP+ Buffer | Cytiva Life Sciences | BR100669 | |
| Human PD-L1 Fc Tag Protein | Acro Biosystems | PD-1-H5258 | |
| Isopropanol | Fisher Scientific | BP2618-1 | |
| Microplate Foil, 96-Well | Cytiva Life Sciences | 28975816 | |
| NaCl | Sigma-Aldrich | 746398 | |
| Plastic Vials 7 mm | Cytiva Life Sciences | BR100212 | |
| Rubber Caps, Type 3 | Cytiva Life Sciences | BR100502 | |
| Series S Sensor Chip CM5 | Cytiva Life Sciences | 29149603 | |
| Sodium Acetate 4.5 | Cytiva Life Sciences | 100350 | |
| Sodium Acetate 5.0 | Cytiva Life Sciences | 100351 | |
| Streptavidin | Sigma-Aldrich | S4762 |
参考文献
- Ribas, A., Wolchok, J. D. Cancer immunotherapy using checkpoint blockade. Science. 359 (6382), 1350-1355 (2018).
- Zhang, X., et al. Structural and functional analysis of the costimulatory receptor programmed death-1. Immunity. 20 (3), 337-347 (2004).
- Freeman, G. J., et al. Engagement of the PD-1 immunoinhibitory receptor by a novel B7 family member leads to negative regulation of lymphocyte activation. J Exp Med. 192 (7), 1027-1034 (2000).
- Han, Y., Liu, D., Li, L. PD-1/PD-L1 pathway: Current researches in cancer. Am J Cancer Res. 10 (3), 727-742 (2020).
- Lyu, N., et al. Recognition of PDL1/L2 by different induced-fit mechanisms of PD1: A comparative study of molecular dynamics simulations. Phys Chem Chem Phys. 22 (3), 1276-1287 (2020).
- Taube, J. M., et al. Colocalization of inflammatory response with B7-H1 expression in human melanocytic lesions supports an adaptive resistance mechanism of immune escape. Sci Trans Med. 4 (127), 127ra37 (2012).
- Sun, X., et al. Immune-related adverse events associated with programmed cell death protein-1 and programmed cell death ligand 1 inhibitors for non-small cell lung cancer: A PRISMA systematic review and meta-analysis. BMC Cancer. 19 (1), 558 (2019).
- Alkholifi, F. K., Alsaffar, R. M. Dostarlimab an inhibitor of PD-1/PD-L1: A new paradigm for the treatment of cancer. Medicina. 58 (11), 1572 (2022).
- Uzar, W., et al. An updated patent review on PD-1/PD-L1 antagonists (2022-present). Expert Opin Ther Pat. 34 (8), 627-650 (2024).
- Ai, L., et al. Research status and outlook of PD-1/PD-L1 inhibitors for cancer therapy. Drug Des Devel Ther. 14, 3625-3649 (2020).
- Conroy, M., Naidoo, J. Immune-related adverse events and the balancing act of immunotherapy. Nat Commun. 13 (1), 392 (2022).
- Guzik, K., et al. Small-molecule inhibitors of the Programmed Cell Death-1/Programmed Death-Ligand 1 (PD-1/PD-L1) interaction via transiently induced protein states and dimerization of PD-L1. J Med Chem. 60 (13), 5857-5867 (2017).
- Sifniotis, V., Cruz, E., Eroglu, B., Kayser, V. Current advancements in addressing key challenges of therapeutic antibody design, manufacture, and formulation. Antibodies. 8 (2), 36 (2019).
- Beck, H., Härter, M., Haß, B., Schmeck, C., Baerfacker, L. Small molecules and their impact in drug discovery: A perspective on the occasion of the 125th anniversary of the Bayer Chemical Research Laboratory. Drug Discov Today. 27 (6), 1560-1574 (2022).
- Nguyen, H. H., Park, J., Kang, S., Kim, M. Surface plasmon resonance: A Versatile technique for biosensor applications. Sensors (Basel, Switzerland). 15 (5), 10481-10510 (2015).
- Liu, C., Seeram, N. P., Ma, H. Small molecule inhibitors against PD-1/PD-L1 immune checkpoints and current methodologies for their development: A review. Cancer Cell Int. 21 (1), 239 (2021).
- Hu, K., Li, X. -. J., Asmamaw, M. D., Shi, X. -. J., Liu, H. -. M. Establishment of high-throughput screening HTRF assay for identification small molecule inhibitors of Skp2-Cks1. Sci Rep. 11 (1), 21105 (2021).
- Puopolo, T., et al. Establishment of human PD-1/PD-L1 blockade assay based on surface plasmon resonance (SPR) biosensor. Bio-protoc. 13 (15), e4765 (2023).
- Ding, M., Chen, Y., Lang, Y., Cui, L. The role of cellular prion protein in cancer biology: A potential therapeutic target. Front Oncol. 11, 742949 (2021).
- Li, H., Seeram, N. P., Liu, C., Ma, H. Further investigation of blockade effects and binding affinities of selected natural compounds to immune checkpoint PD-1/PD-L1. Front Oncol. 12, 995461 (2022).
- Kamrani, A., et al. New immunotherapeutic approaches for cancer treatment. Pathol Res Pract. 248, 154632 (2023).
- Yan, Y., Zhang, L., Zuo, Y., Qian, H., Liu, C. Immune checkpoint blockade in cancer immunotherapy: mechanisms, clinical outcomes, and safety profiles of PD-1/PD-L1 inhibitors. Arch Immunol Ther Exp. 68 (6), 36 (2020).
- Chandrasekharan, G., Unnikrishnan, M. High throughput methods to study protein-protein interactions during host-pathogen interactions. Eur J Cell Biol. 103 (2), 151393 (2024).
- Zhang, Y., et al. BMS-202, a PD-1/PD-L1 inhibitor, decelerates the profibrotic effects of fibroblasts derived from scar tissues via ERK and TGFβ1/Smad signaling pathways. Immun Inflamm Dis. 10 (1), e591 (2022).
- Surmiak, E., et al. PD-L1 inhibitors: Different classes, activities, and mechanisms of action. Int J Mol Sci. 22 (21), 11797 (2021).
- Feoli, A., Sarno, G., Castellano, S., Sbardella, G. DMSO-Related effects on ligand-binding properties of lysine methyltransferases G9a and SETD8. ChemBioChem. 25 (4), e202300809 (2024).
- Tjernberg, A., Markova, N., Griffiths, W. J., HalléN, D. DMSO-Related effects in protein characterization. SLAS Discov. 11 (2), 131-137 (2006).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved