Method Article
Identifizierung von PD-1/PD-L1-Inhibitoren mit der Oberflächenplasmonenresonanz-Technologie
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt einen Blockade-Assay für PD-1/PD-L1-Inhibitoren unter Verwendung der Oberflächenplasmonenresonanz-Technologie. Es verwendet eine zweistufige Immobilisierungsstrategie und ein maßgeschneidertes Puffersystem, um die Reaktionseinheiten genau zu messen und die Beurteilung der Blockaderaten für Verbindungen oder Biologika zu erleichtern. Darüber hinaus unterstützt es die Hochdurchsatz-Identifizierung von PD-1/PD-L1-Inhibitoren.
Zusammenfassung
Die Störung der PD-1/PD-L1-Interaktion ist eine vielversprechende Strategie für die Krebsimmuntherapie. Zuverlässige Screening-Plattformen sind für die Bewertung der Wirksamkeit von PD-1/PD-L1-Inhibitoren unerlässlich. Ein zuvor etablierter humaner PD-1/PD-L1-Blockade-Assay unter Verwendung der Oberflächenplasmonenresonanz (SPR)-Technologie (PD-1/PD-L1-Inhibitor-SPR-Screening-Plattform der ersten Generation) zeigte Ergebnisse, die mit denen vergleichbar sind, die durch homogene zeitaufgelöste Fluoreszenz (HTRF) und zellbasierte Assays erzielt wurden, mit Potenzial für ein groß angelegtes Screening. Darin wird eine optimierte Version dieses Assays (PD-1/PD-L1-Inhibitor-SPR-Screening-Plattform der zweiten Generation) vorgestellt, die einen zweistufigen Kopplungsprozess aufweist, der Amin- und Bio-Streptavidin-Kopplung kombiniert, um die PD-1-Orientierungskontrolle auf dem Chip zu verbessern und den PD-1-Proteinverbrauch zu reduzieren. Die aktualisierte Plattform wurde erfolgreich mit dem PD-1/PD-L1-Inhibitor BMS-1166 validiert und zeigte Blockadeeffekte, die mit der bisherigen SPR-basierten Methode und anderen etablierten Techniken wie ELISA vergleichbar sind. Diese Ergebnisse bestätigen die Zuverlässigkeit des Ansatzes. Diese optimierte SPR-Screening-Plattform bietet ein zuverlässiges Hochdurchsatzwerkzeug für die Identifizierung neuartiger PD-1/PD-L1-Inhibitoren, die Weiterentwicklung der Krebsimmuntherapieforschung und die Hervorhebung des Potenzials von SPR beim Immun-Checkpoint-Inhibitor-Screening.
Einleitung
Immun-Checkpoint-Blockade-Therapien, insbesondere solche, die auf den programmierten Zelltod-1 (PD-1) und den programmierten Zelltod-Liganden 1 (PD-L1) abzielen, stehen an der Spitze der Krebsimmuntherapie-Strategien. Anti-PD-1/PD-L1-Therapien haben die Zulassung für den Einsatz bei verschiedenen Krebsarten wie hämatologischen, Haut-, Lungen-, Leber-, Harnblasen- und Nierenkrebserhalten 1. PD-1 ist ein Transmembran-Glykoprotein, das zur Immunglobulin-Superfamilie gehört und sich durch eine einzelne Immunglobulin-variable (IgV)-ähnliche Domäne am N-Terminus, einen etwa 20-Aminosäure-Stiel, der die IgV-Domäne von der Plasmamembran trennt, eine Transmembrandomäne und einen zytoplasmatischen Schwanz mit Tyrosin-basierten Signalmotivenauszeichnet 2. PD-L1, das als einer der Liganden für PD-1 identifiziert wurde, ist ein Typ-I-Transmembranprotein mit einer Transmembranregion, zwei extrazellulären Domänen - Immunglobulinkonstante (IgC) und IgV - und einer relativ kurzen zytoplasmatischen Domäne, die intrazelluläre Signalwege auslöst3. Der PD-1/PD-L1-Hemmweg dient als kritischer Immun-Checkpoint, der die T-Zell-Aktivierung und Autoimmunität reguliert4. PD-1 wird auf T-Zellen exprimiert, wo es mit PD-L1 interagiert, die Signalübertragung von T-Zell-Rezeptoren hemmt und die Stimulation von CD28- und CD80-Molekülen auf Antigen-präsentierenden Zellen und T-Zellen blockiert5. Krebsgewebe nutzen diesen physiologischen Mechanismus, indem sie PD-L1 während der Escape-Phase überexprimieren und so ein immunsuppressives Milieu schaffen, das das Wachstum und die Progression des Tumors fördert6. Inhibitoren von PD-1 und PD-L1 stören diese Wechselwirkung und ermöglichen es dem Immunsystem, sich der tumorinduzierten Suppression zu entziehen und den T-Zell-vermittelten Tumor-Zelltod-Prozess wieder in Gang zu setzen7.
Aufbauend auf der herausragenden Rolle von Immun-Checkpoint-Blockade-Therapien hat die Entwicklung von PD-1/PD-L1-Inhibitoren einen bedeutenden Fortschritt in der Krebsimmuntherapie markiert. Die U.S. Food and Drug Administration (FDA) hat neun Immun-Checkpoint-Inhibitoren zugelassen, die speziell auf den PD-1/PD-L1-Signalweg abzielen. Dazu gehören sechs PD-1-Inhibitoren - Pembrolizumab, Dostarlimab, Nivolumab, Cemiplimab, Oripalimab und Tislelizumab - und drei PD-L1-Inhibitoren - Atezolizumab, Avelumab und Durvalumab 8,9. Diese Therapien wurden effektiv zur Behandlung einer Vielzahl von Krebsarten eingesetzt, wie z. B. Melanom, Lungenkrebs, Urothelkarzinom, Gebärmutterhalskrebs, Magen- oder Magensophaguskrebs und andere solide Tumoren10. Trotz ihrer Wirksamkeit sind monoklonale Antikörper-basierte Therapien mit erheblichen Einschränkungen konfrontiert, darunter niedrige Ansprechraten, hohe Kosten, verlängerte Halbwertszeiten, schwere immunbedingte unerwünschte Ereignisse und Einschränkungen bei der intravenösen oder subkutanen Verabreichung 11,12,13. Daher konzentriert sich die Forschung zunehmend auf die Entwicklung von niedermolekularen Inhibitoren, die auf die PD-1/PD-L1-Achse abzielen. Diese kleinen Moleküle bieten deutliche Vorteile, wie z. B. eine verbesserte zelluläre Penetration, die Modulation verschiedener biologischer Ziele, eine verbesserte orale Bioverfügbarkeit und reduzierte Kosten, mit dem Ziel, vergleichbare therapeutische Ergebnisse mit weniger Nebenwirkungen zu erzielen14. Die Entwicklung von niedermolekularen Inhibitoren, die auf die PD-1/PD-L1-Interaktion abzielen, befindet sich jedoch noch in einem frühen Stadium, was vor allem auf das Fehlen einer zuverlässigen Hochdurchsatz-Screening-Plattform zurückzuführen ist. Solche Plattformen sind unerlässlich, um große Bibliotheken kleiner Moleküle schnell zu evaluieren und Leitverbindungen für die weitere Validierung und Optimierung zu identifizieren. Die Überwindung dieser Herausforderung ist entscheidend für die Weiterentwicklung der Krebsimmuntherapie.
Die Oberflächenplasmonenresonanz (SPR)-Technologie wird in großem Umfang beim Nachweis verschiedener Biomoleküle, einschließlich Antikörperantigenen, Enzymen, Nukleinsäuren und Arzneimitteln, eingesetzt und ist besonders effektiv beim Screening kleiner molekularer Wirkstoffe15,16. Im Gegensatz zu anderen biophysikalischen Techniken bietet SPR einen markierungsfreien Nachweis, kinetische Echtzeitdaten und einen breiten Nachweisbereich. Im Gegensatz dazu fehlen der isothermen Titrationskalorimetrie kinetische Erkenntnisse in Echtzeit und sie erfordert größere Probenvolumina, was den Durchsatz einschränkt. Die mikroskalige Thermophorese ist anfällig für Pufferinterferenzen und kann keine kinetischen Daten liefern, während die Biolayer-Interferometrie anwendungsspezifische Einschränkungen aufgrund der Molekülgröße und -eigenschaften aufweist. Homogene zeitaufgelöste Fluoreszenz erfordert eine Markierung und ist anfällig für Fluoreszenzinterferenzen. Wir erkennen an, dass HTRF eine weitere geeignete Technologie zur Erforschung von PD-1/PD-L1-Inhibitoren ist. Eine inhärente Einschränkung von HTRF im Vergleich zu SPR ist die Fluoreszenzlöschung, die durch externe Wechselwirkungen mit dem intramolekularen Anregungsprozess (z. B. Elektronentransfer, FRET und Bleichen) verursacht wird, die Empfindlichkeit im Wirkstoffscreening-Prozess aufgrund des kleinen Fensterbereichs zu gering ist und Interferenzen durch fluoreszierende Bibliotheksverbindungen oder biologische Proteineverursacht werden 17. Diese Eigenschaften positionieren SPR als überlegenes Werkzeug für die Wirkstoffforschung. Unsere früheren Studien haben gezeigt, dass SPR in der Lage ist, die Blockadewirkung kleiner Moleküle gegen PD-1/PD-L1 zu bestimmen, was gegenüber anderen Techniken vorteilhaft ist, die hohe Anforderungen an die Markierungstechnologie, mehrere Schritte, schlechte Spezifität und hohe Kosten im Wirkstoffforschungsprozess erfordern18.
In dieser Studie wird eine optimierte SPR-basierte Plattform vorgestellt, die einen zweistufigen Kopplungsprozess integriert, der sowohl Amin- als auch Bio-Streptavidin-Kopplung verwendet, um die PD-1-Orientierung auf dem Chip zu verbessern und den Proteinverbrauch zu minimieren. Dieser aktualisierte Ansatz wurde erfolgreich unter Verwendung des PD-1/PD-L1-Inhibitors BMS-1166 als positives Kontrollbindemittel validiert und zeigte Blockadeeffekte, die sowohl mit unserer vorherigen SPR-Methode als auch mit anderen etablierten Techniken wie ELISA19,20 vergleichbar sind. Dies bestätigt nicht nur die Zuverlässigkeit und Reproduzierbarkeit unseres Protokolls, sondern verdeutlicht auch die Wirksamkeit unserer modifizierten Plattform bei der Erleichterung des Hochdurchsatz-Screenings von PD-1/PD-L1-Inhibitoren. Die Integration des Bio-Streptavidin-Erfassungsschritts ermöglicht eine ortsgerichtete statt zufällige Proteinorientierung, was eine reduzierte PD-1-Konzentration (40 μg/ml vs. 10 μg/ml) und Kosteneinsparungen ermöglicht, indem der Endbenutzer in die Lage versetzt wird, Streptavidin (SA) auf einem CM5-Chip zu immobilisieren, einer kostengünstigeren Alternative zu kommerzialisierten vorimmobilisierten SA-Chips. Dies macht es vorteilhaft für groß angelegte, kostengünstige Screenings von Substanz-/Peptidbibliotheken. Obwohl zusätzliche Charakterisierungsmethoden, einschließlich In-silico-, In-vitro- und In-vivo-Assays, unerlässlich sind, um das klinische Potenzial von PD-1/PD-L1-Inhibitoren gegen Krebs zu bewerten, zeichnet sich unsere verbesserte SPR-basierte Screening-Plattform als effizientes Werkzeug für das groß angelegte Screening von PD-1/PD-L1-Inhibitoren aus.
Protokoll
Die Reagenzien und Geräte sind in der Materialtabelle aufgeführt.
1. Immobilisierung des Streptavidin (SA)-Proteins auf dem CM5-Chip
- Richten Sie die Immobilisierungsmethode auf dem SPR-Gerät ein: Öffnen/Neue Assistentenvorlage , wählen Sie Immobilisierung, setzen Sie den Chiptyp auf CM5 und die Durchflusszellen pro Zyklus auf 1. Überprüfen Sie , ob die Flusszelle 1 und die Durchflusszelle 2 immobilisiert sind.
- Legen Sie Amine als Immobilisierungsmethode fest. Ziel für immobilisiertes Niveau festlegen, Ligandenkonzentration: 40 μg/ml Streptavidin, Zielwert: 2000 RU, Waschlösung: 50 mM NaOH. Überprüfen Sie als Nächstes die Prime, bevor Sie sie ausführen.
- Bereiten Sie die folgenden Röhrchen vor: R2 B1 und R2 C1 - 40 μg/ml Streptavidin; R2 B2 und R2 C2: 50 mM NaOH; R2 B3 und R2 C3: EDC; R2 B4 und R2 C4: NHS; R2 B5 und R2 C5: leer; R2 B6 und R2 C6: Ethanolamin.
HINWEIS: EDC und NHS aktivieren die Carboxylgruppen auf dem CM5-Chip und ermöglichen so eine kovalente Kopplung mit Aminen auf dem Liganden. Ethanolamin wird als Blockierungspuffer verwendet, um eine unspezifische Bindung während der Immobilisierung zu verhindern. - Verdünnen Sie 20 mL HBS-EP+ Puffer 10× in 180 mL deionisiertem (DI) Wasser, um 200 mL 1× HBS-EP+ laufende Pufferlösung herzustellen.
- 1 ml DNase-freies Wasser zu 1 mg Streptavidin geben und 30 Minuten bei Raumtemperatur inkubieren. Als nächstes verdünnen Sie die Streptavidin-Lösung auf 40 μg/ml in Acetatpuffer (pH 4,5). Bereiten Sie die Reagenzien des Amin-Kupplungs-Reagenzien-Kits gemäß den Anweisungen des Herstellers vor und platzieren Sie alle relevanten Reagenz-Layouts wie in Schritt 1.2 beschrieben.
- Ersetzen Sie den Wartungssensorchip durch den CM5-Chip, setzen Sie dann Schlauch A in den vorbereiteten 1× HBS-EP+-Puffer ein, öffnen Sie die Immobilisierungsmethode erneut und setzen Sie die Schläuche gemäß Schritt 1.2 ein. Führen Sie die Methode aus, und speichern Sie die Ergebnisdatei.
- Wartung nach dem Lauf: Ersetzen Sie den CM5-Chip durch den Wartungssensorchip, legen Sie Röhrchen A in eine mit deionisiertem Wasser gefüllte Flasche, und lassen Sie den Prime laufen. Nachdem Sie den CM5-Chip herausgenommen haben, waschen Sie den Chip mit ein paar Tropfen DI-Wasser und trocknen Sie ihn an der Luft. Legen Sie den Chip in ein 50-ml-Röhrchen bei 4 °C.
HINWEIS: Durch den immobilisierten SA kann der CM5-Chip als SA-Chip fungieren.
2. Immobilisierung des PD-1-Proteins auf dem SA-Chip
- Legen Sie die Immobilisierungsmethode auf dem SPR-Gerät fest: Assistentenvorlage öffnen/neu , wählen Sie Immobilisierung, setzen Sie den Chiptyp auf SA und die Durchflusszellen pro Zyklus auf 1. Prüfen Sie, ob die Flusszelle 1 und die Zelle 2 immobilisiert sind.
- Legen Sie SA-Biotin-Capture als Methode fest. Für Zelle 1 stellen Sie die Blind-Immobilisierung ein, für Zelle 2 setzen Sie das Ziel für den immobilisierten Pegel, 10 μg/ml PD-1 als Ligand, Zielniveau: 4000 RU, dann überprüfen Sie die Primzahl vor dem Laufen.
HINWEIS: Bereiten Sie die folgenden Röhrchen - R2 B1 und R2 C1 - vor: 1 m NaCl, 50 mM NaOH; R2 B2 und R2 C2: 50 % Isopropanol/50 mM NaOH/1 M NaCl; R2 C3: 10 µg/mL PD-1.
- Legen Sie SA-Biotin-Capture als Methode fest. Für Zelle 1 stellen Sie die Blind-Immobilisierung ein, für Zelle 2 setzen Sie das Ziel für den immobilisierten Pegel, 10 μg/ml PD-1 als Ligand, Zielniveau: 4000 RU, dann überprüfen Sie die Primzahl vor dem Laufen.
- 58,44 mg NaCl werden in 1 ml 50 mM NaOH gelöst, um 1 M NaCl und 50 mM NaOH-Lösung herzustellen. 58,44 mg NaCl und 4,0 mg NaOH in 500 μl Wasser auflösen, dann 500 μl Isopropanol hinzufügen, um die 50%ige Isopropanol/50 mM NaOH/1 M NaCl-Lösung herzustellen.
- Bereiten Sie die PD-1-Lösung vor: Geben Sie 200 μl DNase-freies Wasser zu 100 μg biotinyliertem humanem PD-1 (Fc und Avitagged) und stabilisieren Sie es 30 Minuten lang bei Raumtemperatur, dann verdünnen Sie es auf 10 μg/ml in Acetatpuffer (pH 5,0).
- Platzieren Sie alle Reagenzien-Layouts wie in Schritt 2.1 beschrieben. Geben Sie Röhrchen A in die 1× HBS-EP+ Laufpufferlösung, werfen Sie dann den Wartungssensorchip aus und setzen Sie den SA-Chip ein (CM5-Chip, der mit Streptavidin-Protein aus Schritt 1 beschichtet ist). Öffnen Sie die Immobilisierungsmethode erneut, setzen Sie Reagenziengestell 2 ein, überprüfen Sie die Positionen, und führen Sie dann die Methode für die geschätzte Laufzeit aus.
- Wiederholen Sie den Schritt 1.6.
3. Regenerations-Scouting für PD-1 und PD-L1
- Richten Sie die Regenerations-Scouting-Methode ein: Öffnen/Neue Assistentenvorlage , Wählen Sie Regenerations-Scouting, setzen Sie den Flusspfad: 2-1, 4-3, Chiptyp auf SA, überprüfen Sie den Laufkonditionierungszyklus, zeichnen Sie die Lösung als HBS-EP+ auf, die Kontaktzeit als 30 s, die Anzahl der Injektionen als 3, die Lösung als PD-L1, die Kontaktzeit: 30 s, die Durchflussrate: 30 μL/min.
- Bei den Regenerationsparametern beträgt die Durchflussrate 30 μL/min und die Stabilisierungszeit 300 s. Legen Sie im Versuchsplan die Anzahl der Bedingungen auf 4 und die Anzahl der Zyklen für jede Bedingung auf 2 fest. Stellen Sie die Bedingungen auf 4 ein, Regenerationslösung: Glycin 1,5, 2,0, 2,5, 3,0, Kontaktzeiten: 30 s, dann vor dem Ausführen die Prime prüfen.
- Stellen Sie jede PD-L1-Konzentration als separate Probenvertiefungsposition in einem 96-Well-Mikroplattenlayout ein: R1 A1 bis R1 A9: 1 μM PD-L1; R1 A10 bis R1 A12: HBS-EP+ Puffer; R1 B1: Glycin 1,5; R1 B2: Glycin 2; R1 B3: Glycin 2,5; R1 B4: Glycin 3.
HINWEIS: Als Regenerationspuffer wird Glycin-Puffer mit unterschiedlichem pH-Wert verwendet. - Bereiten Sie die PD-L1-Lösung vor: Geben Sie 200 μl DNase-freies Wasser zu 100 μg humanem PD-L1 Fc-Tag-Protein (9,42 μM) und verdünnen Sie es dann in HBS-EP+-Puffer auf 1 μM.
- Platzieren Sie alle relevanten Reagenzien im Layout, wie in Schritt 3.2 beschrieben. Setzen Sie Röhre A in den 1× HBS-EP+-Puffer ein und ersetzen Sie dann den Wartungssensorchip durch den SA-Chip. Öffnen Sie die Regenerations-Scouting-Methode erneut, befolgen Sie die Position des Röhrchens aus Schritt 3.2, setzen Sie das Reagenziengestell ein und führen Sie die Methode für die geschätzte Laufzeit aus.
- Wiederholen Sie den Schritt 1.6.
4. Validierung der PD-1/PD-L1-Wechselwirkung
HINWEIS: Zur Validierung wurde ein zuvor veröffentlichter Bericht18 mit geringfügigen Anpassungen befolgt.
- Verwenden Sie dieselben Parameter wie im veröffentlichten Bericht unter Allgemeine Einstellungen, Assay-Schritte und Zyklustypen, stellen Sie die Kontaktzeit auf 60 s, die Dissoziationszeit auf 60 s und die Flussrate auf 30 μl/min ein. Verwenden Sie unter Methodenvariablen, Auswertungsvariablen und Befehlen die gleiche Einstellung, mit der Ausnahme, dass Glycin 2.0 als Regeneration verwendet wird. Die Flussrate wird auf 30 μl/min eingestellt und durchläuft den Flusspfad von 1, 2, 3, 4.
- Richten Sie dann die Ausführung ein, und wählen Sie den Ablaufpfad aus: 2-1, 4-3. Geben Sie PD-L1 mit Konzentrationen von 0 μM, 0,037 μM, 0,111 μM, 0,333 μM und einem Molekulargewicht von 51.300 Da ein. Überprüfen Sie alle Assay-Schritte zur Verifizierung und wählen Sie vor dem Lauf Prime.
- Stellen Sie dann jede PD-L1-Konzentration als separate Probenvertiefungsposition in einem Reagenzienrack-2-Layout ein: R2 B1: PD-L1 0 μM; R2 B2: PD-L1 0,037 μM; R2 B3: PD-L1 0,111 μM; R2 B4: PD-L1 0,333 μM; R2 A1: Glycin 2.0 als Regenerationspuffer; R2 A2: HBS-EP+ als Startpuffer.
- Bereiten Sie 200 ml 1× HBS-EP+ Laufpufferlösung vor. Bereiten Sie PD-L1-Konzentrationen vor: Verdünnen Sie das PD-L1-Protein bei 9,42 μM auf 0,333 μM, 0,111 μM und 0,037 μM in HBS-EP+. Platzieren Sie dann alle relevanten Reagenzien im Layout, wie in Schritt 4.1.2 beschrieben.
- Setzen Sie Rohr A in die 1× HBS-EP+ Laufpufferlösung ein, werfen Sie den Wartungssensorchip aus und setzen Sie den SA-Chip ein. Öffnen Sie die Regenerations-Scouting-Methode erneut, befolgen Sie die Positionierung des Röhrchens aus Schritt 4.1, setzen Sie Reagenzienrack 2 ein und führen Sie die Methode für die geschätzte Dauer aus.
- Wiederholen Sie den Schritt 1.6.
5. PD-1/PD-L1-Blockade-Assay mit niedermolekularem Inhibitor: BMS-1166
HINWEIS: Für den Blockade-Assay wurde ein zuvor veröffentlichter Bericht18 mit geringfügigen Anpassungen befolgt.
- Verwenden Sie dieselben Parameter wie im veröffentlichten Bericht unter Allgemeine Einstellungen, Assay-Schritte und Zyklustypen, stellen Sie die Kontaktzeit auf 60 s, die Dissoziationszeit auf 60 s und die Flussrate auf 30 μl/min ein. Unter Methodenvariablen, Auswertungsvariablen und Befehlen wird ebenfalls die gleiche Einstellung verwendet, mit der Ausnahme, dass Glycin 2.0 als Regeneration verwendet wird, die Flussrate auf 30 μL/min einrichtet und den Flussweg von 1, 2, 3, 4 durchläuft.
- Richten Sie dann die Ausführung ein, und wählen Sie den Flusspfad aus: 2-1, 4-3. Geben Sie die Probenlösung ein: PD-L1 (0,111 μM, Molekulargewicht von 51.300 Da) mit BMS-1166 bei 0 μM, 0,125 μM, 0,625 μM, 3,125 μM. Überprüfen Sie alle Assay-Schritte zur Verifizierung und wählen Sie vor dem Lauf Prime.
- Stellen Sie dann jede PD-L1-Konzentration als separate Probenvertiefungsposition in einem Reagenzienrack-2-Layout ein: R2 B1: PD-L1 (0,111 μM) + BMS-1166 0 μM R2 B2: PD-L1 (0,111 μM) + BMS-1166 0,125 μM; R2 B3: PD-L1 (0,111 μM) + BMS-1166 0,625 μM; R2 B4: PD-L1 (0,111 μM) + BMS-1166 3,125 μM; R2 A1: Glycin 2.0 zur Regeneration.
- Bereiten Sie 200 ml 1× HBS-EP+ Laufpufferlösung vor. Bereiten Sie das Gemisch BMS-1166/PD-L1 vor: 5 mg BMS-1166 werden in 77,99 μl Dimethylsulfoxid (DMSO) gelöst, um eine 100 mM Stammlösung herzustellen. Verdünnen Sie die Stammlösung mit dem PD-L1-Protein (0,11 μM) auf die Zielkonzentrationen von BMS-1166 bei 0 μM, 0,125 μM, 0,625 μM und 3,125 μM in HBS-EP+. Platzieren Sie alle relevanten Reagenzien wie in Schritt 5.1.2 beschrieben.
- Setzen Sie Röhrchen A in die 1× HBS-EP+ Laufpufferlösung ein, werfen Sie dann den Wartungssensorchip aus und setzen Sie den SA-Chip ein. Öffnen Sie die Regenerations-Scouting-Methode erneut, folgen Sie der Röhrchenposition von 5.1, setzen Sie Reagenzien-Rack 2 ein und führen Sie die Methode für die geschätzte Laufzeit aus.
- Wiederholen Sie den Schritt 1.6.
Ergebnisse
Immobilisierung von SA auf CM5-Chip
Die Daten wurden über die Ergebnisse des SPR-Instruments und der zugehörigen Analysesoftware analysiert, was auf das erfolgreiche Erreichen des Ziels RU (2000 RU) des SA-Proteins in Flusszelle 1 und Flusszelle 2 hinweist. Die Flusszellen 1 und 2 wurden mit SA (40 μg/ml) auf der CM5-Chipoberfläche immobilisiert, mit einer endgültigen Reaktion von 1902,3 HE auf der Flusszelle 1 (Abbildung 1A) und 1900,7 HE auf der Flusszelle 2 (Abbildung 1B).
Immobilisierung von PD-1 auf SA-Chip
Die Datenanalyse auf der Grundlage der Ergebnisse des SPR-Instruments und der zugehörigen Software ergab eine niedrige Ansprecheinheit (RU) für die leere Zelle an Durchflusszelle 1 und ein erfolgreiches Erreichen der Ziel-RU (4000 RU) für den PD-1-Liganden an der Durchflusszelle 2. Flusszelle 1 wurde als Rohling immobilisiert, was zu einer endgültigen Reaktion von -161,0 RU führte (Abbildung 2A), während Flusszelle 2 mit biotinyliertem PD-1-Protein (10 μg/ml) immobilisiert wurde, das auf dem SA-Chip beschichtet war, was zu einer endgültigen Reaktion von 3698,5 RU führte (Abbildung 2B).
Regenerations-Scouting für PD-1 und PD-L1.
Das Regenerations-Scouting wurde mit PD-1 immobilisiert auf Flusszelle 2 und PD-L1 in Lösung (0,1 μM) bei verschiedenen Glycin-pH-Werten (1,5, 2, 2,5 und 3) durchgeführt, um die Regenerationslösung zu bestimmen, was zu einem stabilen Ausgangswert und einer stabilen Probenreaktion führte. Die Daten wurden über die Ausgabe des SPR-Instruments und der Analysesoftware analysiert, was zu Glycin (pH 2,0) als optimale Regenerationsbedingung führte, da sich die Reaktion sowohl auf die Ausgangslinie als auch auf die Probenreaktion minimal änderte, was auf einen minimalen PD-1-Proteinverlust und eine erfolgreiche Entfernung des PD-L1-Proteins von der Chipoberfläche hinweist. Bei Glycin pH 2,0 blieb die Ausgangsreaktion relativ konstant, und die Analytreaktion war stabil und nahe der Reaktion zu Beginn des Experiments, was auf den optimalsten Regenerationspuffer unter den vier getesteten Glycin-pH-Bedingungen hinweist. Ein höherer pH-Wert ist unzureichend und ein niedrigerer pH-Wert ist zu hart; Als die am besten geeignete Regenerationsbedingung wird ein pH-Wert von 2,0 identifiziert (Abbildung 3).
Validierung der PD-1/PD-L1-Wechselwirkung
Die Analyse der Daten erfolgte mit der entsprechenden Auswertesoftware. Wählen Sie im Abschnitt "Kinetik/Affinität" die Option "oberflächengebunden" aus, wählen Sie die Kurve 2-1, stellen Sie das Kinetikmodell auf 1:1-Bindung ein und stellen Sie den RI-Parameter auf eine konstante Anpassung ein, um die Assoziationsrate (ka), die Dissoziationsrate (kd) und die Gleichgewichtsdissoziationskonstante (KD) zu bestimmen. Die Bindungsinteraktion von PD-L1 in unterschiedlichen Konzentrationen mit PD-1 wurde quantifiziert, was zu einer messbaren Reaktion führte (Abbildung 4). Die analysierten Bindungsparameter umfassten eine Assoziationsrate (ka) von 3,611 × 104 (1/Ms), eine Dissoziationsrate (kd) von 0,0236 (1/s) und eine Gleichgewichtsdissoziationskonstante (KD) von 6,536 × 10-7 M.
PD-1/PD-L1-Blockade-Assay mit etabliertem niedermolekularem Inhibitor: BMS-1166
Die Daten wurden mit Hilfe der entsprechenden analysiert. Wählen Sie unter Kinetik/Affinität die Option oberflächengebunden aus, wählen Sie die Kurve 2-1 und rufen Sie dann die visualisierte Kurve des Ergebnisses und der Antworteinheit ab. Der Blockadeeffekt der PD-1/PD-L1-Bindungsinteraktion wurde mit BMS-1166 (0-3,125 μM) mit 0,11 μM PD-L1-Protein im HBS EP+-Puffer beobachtet (Abbildung 5). Die höchste Response-Einheit wird durch PD-L1 allein identifiziert, während mit zunehmender BMS-1166-Konzentration die Bindungsresponse-Einheit proportional abnimmt.

Abbildung 1: Immobilisierung von SA auf dem CM5-Chip. Immobilisierungskurven des Streptavidin-Proteins auf der CM5-Chip-Flusszelle 1 (A) und der Flusszelle 2 (B) sind dargestellt. Zuerst flossen fünf Vorkonzentrationen des SA-Proteins über die Flusszellen 1 und 2, gefolgt von einem NaOH-Wash und der Etablierung einer stabilen Baseline. Als nächstes wurden EDC und NHS zugegeben, und dann wurde eine Ethanolaminhydrochlorid-Wäsche durchgeführt. Anschließend wurden fünf Impulse von PD-1 durchgeführt, um das Zielniveau (2000 RU) zu erreichen, gefolgt von einer Ethanolaminhydrochlorid-Wäsche, um elektrostatisch gebundene Liganden zu entfernen und NHS-Ester zu deaktivieren, die nicht reaktiv bleiben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
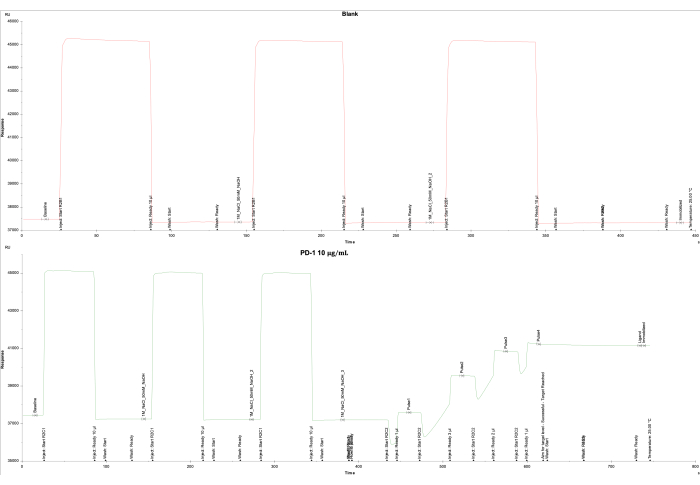
Abbildung 2: Immobilisierung von PD-1 auf dem SA-Chip. Immobilisierungskurven von (A) der blinden Flusszelle 1, die nur mit SA beschichtet ist, und (B) dem PD-1-Protein auf dem SA-Chip auf der Flusszelle 2 sind dargestellt. Für den Blindwert (Durchflusszelle 1) wurden dreimal 1 M NaCl und 50 mM NaOH injiziert, gefolgt von einer Wäsche mit 50 % Isopropanol/1 M NaCl/50 mM NaOH. Für das PD-1-Protein (Durchflusszelle 2) wurden dreimal 1 M NaCl und 50 mM NaOH injiziert, gefolgt von fünf Impulsen des PD-1-Proteins, um das Zielniveau von 4000 U zu erreichen, und einer abschließenden Wäsche von 50 % Isopropanol/1 M NaCl/50 mM NaOH. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Regenerations-Scouting für PD-1 und PD-L1. Der Ausgangswert und das Ansprechen der Probe wurden unter vier Regenerationsbedingungen mit Glycin bei pH 1,5, 2, 2,5 und 3 ermittelt, um die optimale Regenerationslösung vor der Probenuntersuchung zu bestimmen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Validierung der PD-1/PD-L1-Wechselwirkung. Bindungskinetik unterschiedlicher Konzentrationen von PD-L1 (0,037 μM, 0,111 μM und 0,333 μM) an PD-1 auf der Streptavidin-beschichteten Chipoberfläche. PD-L1 zeigt eine ausgeprägte Assoziation (0-60 s) und Dissoziationsphasen (61-120 s) mit PD-1. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
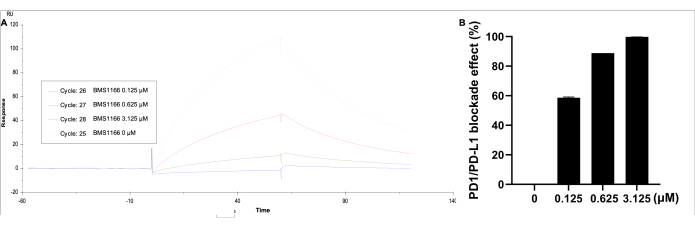
Abbildung 5: SPR-Analyse von PD-1, das auf einem SA-immobilisierten CM5-Chip beschichtet ist, mit PD-L1 (0,11 μM) und BMS-1166 (0-3,125 μM) in Lösung. (A) Eine repräsentative Echtzeit-SPR-Reaktion der Bindungsreaktionen zwischen PD-1/PD-L1 und BMS-1166. (B) Der prozentuale Blockadeeffekt von BMS-1166 auf die Bindungskinetik der PD-1/PD-L1-Wechselwirkung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 6: Vergleich von Immobilisierungsstrategien für PD-1-Proteine. Amin-Kopplung (A) vs. zweistufige Streptavidin-Biotin-Kupplung (B). Die Aminkopplung stellt Herausforderungen dar, darunter die begrenzte Verfügbarkeit zugänglicher Bindungsstellen, sterische Bindungshindernisse und mögliche Modifikationen von PD-1-Bindungsstellen während der Immobilisierung. Im Gegensatz dazu erhöht der zweistufige Streptavidin-Biotin-Ansatz die Verfügbarkeit von freien Bindungsstellen auf immobilisiertem PD-1 und ermöglicht so eine verbesserte Interaktion mit PD-L1 in Lösung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
In den letzten Jahrzehnten haben verschiedene Immuntherapieansätze – darunter Krebsimpfstoffe, Immun-Checkpoint-Inhibitoren und CAR-T-Zelltherapien – die Krebsbehandlung erheblich vorangetrieben21. Immun-Checkpoints spielen eine entscheidende Rolle bei der Verhinderung von Immunzell-vermittelten Kollateralschäden bei pathogenen Reaktionen und bei der Unterdrückung von Autoimmunität. Ein wichtiges Beispiel ist die Interaktion zwischen PD-L1 und PD-1, die einen wichtigen Immun-Checkpoint bildet, der es Krebszellen ermöglicht, sich der Immunüberwachung zu entziehen. Die Ausrichtung auf den PD-1/PD-L1-Signalweg mit monoklonalen Antikörpern hat in der klinischen Onkologie bemerkenswerte Erfolge erzielt. Aufgrund der Einschränkungen im Zusammenhang mit monoklonalen Antikörpertherapien und der zunehmenden Inzidenz immunbedingter unerwünschter Ereignisse besteht jedoch ein wachsendes Interesse an der Entwicklung niedermolekularer Inhibitoren, die auf PD-1/PD-L1 abzielen 9,22.
Aktuelle Screening-Strategien für niedermolekulare PD-1/PD-L1-Inhibitoren konzentrieren sich hauptsächlich auf Bioassay-Techniken wie ELISA, zellbasierte Reporter-Assays und T-Zell-Assays. Biophysikalische Techniken, einschließlich SPR und Biolayer-Interferometrie (BLI), werden häufig zur Charakterisierung von Bindungsaffinitäten eingesetzt, aber ihr Potenzial, als Screening-Werkzeuge eingesetzt zu werden, wird unterschätzt23. Im Rahmen dieser Studie wurde ein optimierter SPR-basierter PD-1/PD-L1-Blockade-Assay entwickelt, der eine Hochdurchsatzplattform bietet, die für die Entdeckung von niedermolekularen PD-1/PD-L1-Inhibitoren geeignet ist. SA wurde auf ~2000 U immobilisiert, gefolgt von biotinyliertem PD-1 (~4000 RU) über die Biotin-Streptavidin-Interaktion. Diese robuste Bindung gewährleistete eine sichere PD-1-Beschichtung mit optimaler Ausrichtung, minimierte unspezifische Bindungen und reduzierte den Proteinverbrauch (10 μg/ml). Die ortsgerichtete Immobilisierung verbesserte die Effizienz im Vergleich zu herkömmlichen Methoden. Glycinpuffer (pH 2,0) wurde verwendet, um die Sensoroberfläche zwischen den Zyklen zu regenerieren, die experimentelle Integrität zu erhalten und eine unspezifische Bindung zu verhindern.
Im Vergleich zu immunologischen Standardmethoden und ELISA bot dieser SPR-Ansatz einen markierungsfreien Echtzeitnachweis mit hoher Sensitivität und Spezifität. Darüber hinaus ermöglicht es ein Hochdurchsatz-Screening mit einer Laufzeit von 120 s/Probe und ergänzt Bioassays bei der Beurteilung der Blockadeeffizienz von PD-1/PD-L1-Targeting-Verbindungen und Biologika. Der erreichte Immobilisierungsgrad war vergleichbar mit der vorherigen Plattform (3698,5 RU vs. 3688,5 RU), mit einer ähnlichen PD-1/PD-L1-Interaktionsaffinität (KD = 6,536 × 10-7 M vs. 1,295 × 10-7 M). Der BMS-1166-Inhibitor zeigte eine höhere Dissoziationsrate bei niedrigerer Konzentration mit Blockadeeffekten, die mit der früheren Plattform vergleichbar waren (99,8 % vs. 94,2 % bei 3,125 μM). BMS-1166 zeigte PD-1/PD-L1-Blockaderaten, die durch IC50-Werte von 1,4 nM bzw. 96 nM in HTRF- bzw. zellbasierten Assays unterstützt wurden24. Darüber hinaus berichteten andere Studien über IC50-Werte von 3,9 nM und 1574 nM unter Verwendung von HTRF- und Immun-Checkpoint-Blockade-Co-Kultur-Assay-Methoden25. Unsere früheren Ergebnisse zeigten, dass die IC50-Werte von BMS-1166 85,4 nM betrugen, was mit diesen früheren Ergebnissen übereinstimmt18. Ein weiterer Vorteil dieser Siebplattform ist ihre Robustheit und ihr hoher Durchsatz. Diese Methode wurde ausgiebig im Hochdurchsatz-Screening mit einem 384-Well-Plattenformat angewendet, wobei BMS-1166 und BMS-202 als Positivkontrollen für jeweils 10 Proben enthalten waren. Die Blockaderatenbereiche betrugen 29,8 %-38,1 % für BMS-1166 und 6,0 %-10,4 % für BMS-202 bei 10 nM (n = 11 pro Platte).
DMSO hat wahrscheinlich unterschiedliche Auswirkungen auf heterogene biologische Membranen, abhängig von ihrer lokalen Zusammensetzung und Struktur, und beeinflusst möglicherweise membranassoziierte biologische Funktionen. Bei relativ niedrigen Konzentrationen kann DMSO die Proteineigenschaften in Lösung verändern, was zu Denaturierung, Aggregation oder Abbau führt. Darüber hinaus kann DMSO die scheinbaren Bindungseigenschaften von Proteinen modifizieren26,27. Um frühere Bedenken hinsichtlich DMSO-Interferenzen auszuräumen, haben wir die DMSO-Konzentration in diesem Protokoll auf 0,003 % reduziert (0,01 % DMSO wurde auf der früheren Plattform verwendet).
In einer früheren Studie wurden PD-1-Proteine auf Sensorchips mit Hilfe der Aminkupplung immobilisiert, bei der EDC/NHS-Chemie verwendet wird, um Carboxymethylgruppen auf dem Chip zu aktivieren und kovalente Bindungen mit Amingruppen auf PD-1 zu bilden. Aufgrund des Vorhandenseins mehrerer Amingruppen in den 288 Aminosäuren von PD-1 führt diese Methode von Natur aus zu zufälligen Orientierungen. Um eine spezifischere Orientierung zu erreichen, haben wir in dieser Studie eine Capture-Kopplungsstrategie mit biotinyliertem humanem PD-1 (Fc, Avitag) eingesetzt. Der einzelne Lysinrest im Avitag wird enzymatisch in der Fc-Region von PD-1 biotinyliert, was durch die hochaffine Wechselwirkung zwischen Biotin und Streptavidin eine präzise Immobilisierung ermöglicht (Abbildung 6). Theoretisch stellt dieser Ansatz sicher, dass die Variablendomäne von PD-1 freigelegt bleibt, was eine optimale Interaktion mit PD-L1 im Puffer ermöglicht. Diese Annahme erfordert jedoch eine weitere Validierung mit Techniken wie Kryo-EM oder Kristallographie, um die Proteinorientierung zu bestätigen.
Glycin 2.0 wurde als milder Regenerationspuffer verwendet, der PD-L1 effektiv entfernte, ohne das immobilisierte PD-1 zu reduzieren. Bei fest gebundenen Inhibitoren oder Proteinaggregaten können jedoch stärkere Regenerationspuffer wie 0,5 % SDS oder 50-100 mM NaOH erforderlich sein, um Interferenzen mit nachfolgenden Proben zu vermeiden.
Diese Studie hat mehrere Einschränkungen. Erstens können die hohen Kosten für SPR-Instrumente die Zugänglichkeit einschränken, obwohl das Protokoll oft für andere SPR-Systeme mit ähnlichen Funktionen angepasst werden kann. Parameter wie Proteinkonzentration und Assoziations-/Dissoziationszeiten können an spezifische SPR-Plattformen angepasst werden. Eine weitere Einschränkung ist die Abhängigkeit von rekombinanten Proteinen, die native Proteininteraktionen möglicherweise nicht vollständig replizieren. Diese Faktoren sollten bei der Interpretation von SPR-Daten und dem Vergleich der Ergebnisse mit ergänzenden Techniken wie zellbasierten Assays oder In-vivo-Modellen berücksichtigt werden.
Trotz dieser Einschränkungen bietet die optimierte SPR-Methode einen schnellen und markierungsfreien Echtzeit-Ansatz mit hohem Durchsatz für das Screening von niedermolekularen Inhibitoren der PD-1/PD-L1-Interaktion. Es verbessert die biophysikalischen Techniken zur Charakterisierung kleiner Moleküle und Biologika, die auf die PD-1/PD-L1-Interaktion abzielen. Mit ihrer hohen Empfindlichkeit ist die Plattform besonders wertvoll für die Untersuchung von Immun-Checkpoint-Wechselwirkungen und breiteren Protein-Protein-Wechselwirkungen (PPI), was sie zu einem leistungsstarken Werkzeug für die Weiterentwicklung der PPI-Wirkstoffforschung macht.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Die Autoren würdigen die RI-INBRE Core Facility an der University of Rhode Island, die durch Grant P20GM103430 des National Center for Research Resources (NCRR), einer Komponente der National Institutes of Health (NIH), unterstützt wird. Diese Forschung wurde durch einen Pilot Grant Award des College of Pharmacy an der University of Rhode Island, einen Small Grant Award des Rhode Island Life Science Hub (RILSH) und einen Grant der Rhode Island Foundation unterstützt, die alle an Chang Liu, Ph.D., vergeben wurden.
Materialien
| Name | Company | Catalog Number | Comments |
| 50 mM NaOH | Cytiva Life Sciences | 100358 | |
| 50 mM NaOH | Fisher Scientific | 905376 | |
| 96-Well Polystyrene Microplates | Cytiva Life Sciences | BR100503 | |
| Amine Coupling Kit | Cytiva Life Sciences | 35120 | |
| Biacore T200 SPR System and Evaluation Software 3.2 | Cytiva Life Sciences | 28975001 | |
| Biotinylated Human PD-1 Fc, Avitag Protein | Acro Biosystems | PD1-H82F1 | |
| BMS1166 | MedChemExpress | HY-102011 | |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | 276855 | |
| DNase Free Water | Fisher Scientific | 188506 | |
| Glycine 1.5 | Cytiva Life Sciences | BR100354 | |
| Glycine 2.0 | Cytiva Life Sciences | BR100355 | |
| Glycine 2.5 | Cytiva Life Sciences | BR100356 | |
| Glycine 3.0 | Cytiva Life Sciences | BR100357 | |
| HBS-EP+ Buffer | Cytiva Life Sciences | BR100669 | |
| Human PD-L1 Fc Tag Protein | Acro Biosystems | PD-1-H5258 | |
| Isopropanol | Fisher Scientific | BP2618-1 | |
| Microplate Foil, 96-Well | Cytiva Life Sciences | 28975816 | |
| NaCl | Sigma-Aldrich | 746398 | |
| Plastic Vials 7 mm | Cytiva Life Sciences | BR100212 | |
| Rubber Caps, Type 3 | Cytiva Life Sciences | BR100502 | |
| Series S Sensor Chip CM5 | Cytiva Life Sciences | 29149603 | |
| Sodium Acetate 4.5 | Cytiva Life Sciences | 100350 | |
| Sodium Acetate 5.0 | Cytiva Life Sciences | 100351 | |
| Streptavidin | Sigma-Aldrich | S4762 |
Referenzen
- Ribas, A., Wolchok, J. D. Cancer immunotherapy using checkpoint blockade. Science. 359 (6382), 1350-1355 (2018).
- Zhang, X., et al. Structural and functional analysis of the costimulatory receptor programmed death-1. Immunity. 20 (3), 337-347 (2004).
- Freeman, G. J., et al. Engagement of the PD-1 immunoinhibitory receptor by a novel B7 family member leads to negative regulation of lymphocyte activation. J Exp Med. 192 (7), 1027-1034 (2000).
- Han, Y., Liu, D., Li, L. PD-1/PD-L1 pathway: Current researches in cancer. Am J Cancer Res. 10 (3), 727-742 (2020).
- Lyu, N., et al. Recognition of PDL1/L2 by different induced-fit mechanisms of PD1: A comparative study of molecular dynamics simulations. Phys Chem Chem Phys. 22 (3), 1276-1287 (2020).
- Taube, J. M., et al. Colocalization of inflammatory response with B7-H1 expression in human melanocytic lesions supports an adaptive resistance mechanism of immune escape. Sci Trans Med. 4 (127), 127ra37 (2012).
- Sun, X., et al. Immune-related adverse events associated with programmed cell death protein-1 and programmed cell death ligand 1 inhibitors for non-small cell lung cancer: A PRISMA systematic review and meta-analysis. BMC Cancer. 19 (1), 558 (2019).
- Alkholifi, F. K., Alsaffar, R. M. Dostarlimab an inhibitor of PD-1/PD-L1: A new paradigm for the treatment of cancer. Medicina. 58 (11), 1572 (2022).
- Uzar, W., et al. An updated patent review on PD-1/PD-L1 antagonists (2022-present). Expert Opin Ther Pat. 34 (8), 627-650 (2024).
- Ai, L., et al. Research status and outlook of PD-1/PD-L1 inhibitors for cancer therapy. Drug Des Devel Ther. 14, 3625-3649 (2020).
- Conroy, M., Naidoo, J. Immune-related adverse events and the balancing act of immunotherapy. Nat Commun. 13 (1), 392 (2022).
- Guzik, K., et al. Small-molecule inhibitors of the Programmed Cell Death-1/Programmed Death-Ligand 1 (PD-1/PD-L1) interaction via transiently induced protein states and dimerization of PD-L1. J Med Chem. 60 (13), 5857-5867 (2017).
- Sifniotis, V., Cruz, E., Eroglu, B., Kayser, V. Current advancements in addressing key challenges of therapeutic antibody design, manufacture, and formulation. Antibodies. 8 (2), 36 (2019).
- Beck, H., Härter, M., Haß, B., Schmeck, C., Baerfacker, L. Small molecules and their impact in drug discovery: A perspective on the occasion of the 125th anniversary of the Bayer Chemical Research Laboratory. Drug Discov Today. 27 (6), 1560-1574 (2022).
- Nguyen, H. H., Park, J., Kang, S., Kim, M. Surface plasmon resonance: A Versatile technique for biosensor applications. Sensors (Basel, Switzerland). 15 (5), 10481-10510 (2015).
- Liu, C., Seeram, N. P., Ma, H. Small molecule inhibitors against PD-1/PD-L1 immune checkpoints and current methodologies for their development: A review. Cancer Cell Int. 21 (1), 239 (2021).
- Hu, K., Li, X. -. J., Asmamaw, M. D., Shi, X. -. J., Liu, H. -. M. Establishment of high-throughput screening HTRF assay for identification small molecule inhibitors of Skp2-Cks1. Sci Rep. 11 (1), 21105 (2021).
- Puopolo, T., et al. Establishment of human PD-1/PD-L1 blockade assay based on surface plasmon resonance (SPR) biosensor. Bio-protoc. 13 (15), e4765 (2023).
- Ding, M., Chen, Y., Lang, Y., Cui, L. The role of cellular prion protein in cancer biology: A potential therapeutic target. Front Oncol. 11, 742949 (2021).
- Li, H., Seeram, N. P., Liu, C., Ma, H. Further investigation of blockade effects and binding affinities of selected natural compounds to immune checkpoint PD-1/PD-L1. Front Oncol. 12, 995461 (2022).
- Kamrani, A., et al. New immunotherapeutic approaches for cancer treatment. Pathol Res Pract. 248, 154632 (2023).
- Yan, Y., Zhang, L., Zuo, Y., Qian, H., Liu, C. Immune checkpoint blockade in cancer immunotherapy: mechanisms, clinical outcomes, and safety profiles of PD-1/PD-L1 inhibitors. Arch Immunol Ther Exp. 68 (6), 36 (2020).
- Chandrasekharan, G., Unnikrishnan, M. High throughput methods to study protein-protein interactions during host-pathogen interactions. Eur J Cell Biol. 103 (2), 151393 (2024).
- Zhang, Y., et al. BMS-202, a PD-1/PD-L1 inhibitor, decelerates the profibrotic effects of fibroblasts derived from scar tissues via ERK and TGFβ1/Smad signaling pathways. Immun Inflamm Dis. 10 (1), e591 (2022).
- Surmiak, E., et al. PD-L1 inhibitors: Different classes, activities, and mechanisms of action. Int J Mol Sci. 22 (21), 11797 (2021).
- Feoli, A., Sarno, G., Castellano, S., Sbardella, G. DMSO-Related effects on ligand-binding properties of lysine methyltransferases G9a and SETD8. ChemBioChem. 25 (4), e202300809 (2024).
- Tjernberg, A., Markova, N., Griffiths, W. J., HalléN, D. DMSO-Related effects in protein characterization. SLAS Discov. 11 (2), 131-137 (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten