Method Article
Identification des inhibiteurs de-1/-L1 à l’aide de la technologie de résonance plasmonique de surface
Dans cet article
Résumé
Ce protocole décrit un test de blocage des inhibiteurs de-1/-L1 à l’aide de la technologie de résonance plasmonique de surface. Il utilise une stratégie d’immobilisation en deux étapes et un système tampon adapté pour mesurer avec précision les unités d’intervention, facilitant ainsi l’évaluation des taux de blocage des composés ou des produits biologiques. De plus, il prend en charge l’identification à haut débit des inhibiteurs de-1/-L1.
Résumé
La perturbation de l’interaction-1/-L1 est une stratégie prometteuse pour l’immunothérapie du cancer. Des plateformes de dépistage fiables sont essentielles pour évaluer l’efficacité des inhibiteurs de-1/-L1. Un test de blocage-1/-L1 humain précédemment établi utilisant la technologie de résonance plasmonique de surface (SPR) (plateforme de criblage SPR d’inhibiteurs de-1/-L1 de première génération) a démontré des résultats comparables à ceux obtenus par des tests de fluorescence homogène résolue dans le temps (HTRF) et des tests cellulaires, avec un potentiel de criblage à grande échelle. Dans cet article, une version optimisée de ce test (plateforme de criblage SPR d’inhibiteurs de-1/-L1 de deuxième génération) est présentée, avec un processus de couplage en deux étapes qui combine le couplage d’amines et de bio-streptavidines pour améliorer le contrôle de l’orientation de-1 sur la puce et réduire la consommation de protéines-1. La plateforme mise à jour a été validée avec succès à l’aide de l’inhibiteur de-1/-L1 BMS-1166, montrant des effets de blocage comparables à la méthode précédente basée sur la SPR et à d’autres techniques établies telles que ELISA. Ces résultats confirment la fiabilité de l’approche. Cette plateforme de dépistage SPR optimisée offre un outil fiable et à haut débit pour identifier de nouveaux inhibiteurs de-1/-L1, faire progresser la recherche sur l’immunothérapie du cancer et mettre en évidence le potentiel de la SPR dans le dépistage des inhibiteurs de point de contrôle immunitaire.
Introduction
Les thérapies par blocage des points de contrôle immunitaires, en particulier celles ciblant la mort cellulaire programmée-1 (-1) et la mort cellulaire programmée-ligand 1 (-L1), sont à l’avant-garde des stratégies d’immunothérapie du cancer. Les thérapies anti--1/-L1 ont reçu l’approbation pour une utilisation dans divers types de cancer, telsque les cancers hématologiques, cutanés, pulmonaires, hépatiques, de la vessie et du rein1. -1 est une glycoprotéine transmembranaire appartenant à la superfamille des immunoglobulines, caractérisée par un seul domaine de type variable immunoglobuline (IgV) au niveau N-terminal, une tige d’environ 20 acides aminés séparant le domaine IgV de la membrane plasmique, un domaine transmembranaire et une queue cytoplasmique contenant des motifs de signalisation basés sur la tyrosine2. -L1, identifiée comme l’un des ligands de-1, est une protéine transmembranaire de type I caractérisée par une région transmembranaire, deux domaines extracellulaires - la constante d’immunoglobuline (IgC) et l’IgV - et un domaine cytoplasmique relativement court qui déclenche les voies de signalisation intracellulaires3. La voie inhibitrice-1/-L1 sert de point de contrôle immunitaire critique qui régule l’activation des lymphocytes T et l’auto-immunité4. -1 est exprimé sur les lymphocytes T, où il interagit avec-L1, inhibe la signalisation des récepteurs des lymphocytes T et bloque la stimulation des molécules CD28 et CD80 sur les cellules présentatrices d’antigène et les lymphocytes T5. Les tissus cancéreux exploitent ce mécanisme physiologique en surexprimant-L1 pendant la phase d’échappement, créant ainsi un environnement immunosuppresseur qui favorise la croissance etla progression tumorale6. Les inhibiteurs de-1 et de-L1 perturbent cette interaction, permettant au système immunitaire d’échapper à la suppression induite par la tumeur et de relancer le processus de mort des cellules tumorales médié par les lymphocytes T7.
S’appuyant sur le rôle prépondérant des thérapies de blocage des points de contrôle immunitaires, le développement d’inhibiteurs de-1/-L1 a marqué une avancée significative dans l’immunothérapie du cancer. La Food and Drug Administration (FDA) des États-Unis a approuvé neuf inhibiteurs de point de contrôle immunitaire qui ciblent spécifiquement la voie-1/-L1. Il s’agit notamment de six inhibiteurs de-1 - pembrolizumab, dostarlimab, nivolumab, cemiplimab, oripalimab et tislelizumab - et de trois inhibiteurs de-L1 - atezolizumab, avelumab et durvalumab 8,9. Ces thérapies ont été efficacement utilisées pour traiter une variété de cancers, tels que le mélanome, le cancer du poumon, le cancer urothélial, le cancer du col de l’utérus, le cancer gastrique ou gastro-œsophagien et d’autres tumeurs solides10. Malgré leur efficacité, les traitements à base d’anticorps monoclonaux se heurtent à d’importantes limites, notamment de faibles taux de réponse, des coûts élevés, des demi-vies prolongées, des effets indésirables graves liés au système immunitaire et des restrictions à l’administration intraveineuse ou sous-cutanée 11,12,13. Par conséquent, la recherche se concentre de plus en plus sur le développement de petites molécules inhibitrices ciblant l’axe-1/-L1. Ces petites molécules offrent des avantages distincts, tels qu’une meilleure pénétration cellulaire, la modulation de diverses cibles biologiques, une biodisponibilité orale accrue et des coûts réduits, dans le but d’obtenir des résultats thérapeutiques comparables avec moins d’effets indésirables14. Cependant, le développement d’inhibiteurs de petites molécules ciblant l’interaction-1/-L1 n’en est qu’à ses débuts, principalement en raison de l’absence d’une plateforme de criblage fiable à haut débit. De telles plateformes sont essentielles pour évaluer rapidement de vastes bibliothèques de petites molécules et identifier les principaux composés en vue d’une validation et d’une optimisation plus poussées. Il est essentiel de surmonter ce défi pour faire progresser l’immunothérapie du cancer.
La technologie de résonance plasmonique de surface (SPR) est largement utilisée dans la détection de diverses biomolécules, y compris les antigènes d’anticorps, les enzymes, les acides nucléiques et les médicaments, et est particulièrement efficace dans le criblage de petites molécules15,16. Contrairement à d’autres techniques biophysiques, la SPR offre une détection sans marquage, des données cinétiques en temps réel et une large plage de détection. En revanche, la titration calorimétrie isotherme manque d’informations cinétiques en temps réel et nécessite des volumes d’échantillons plus importants, ce qui limite le débit. La thermophorèse à l’échelle microscopique est sujette aux interférences de tampon et ne peut pas fournir de données cinétiques, tandis que l’interférométrie de biocouche présente des limites spécifiques à l’application en fonction de la taille et des propriétés moléculaires. La fluorescence homogène résolue dans le temps nécessite un marquage et est sensible aux interférences fluorescentes. Nous reconnaissons que le HTRF est une autre technologie appropriée pour explorer les inhibiteurs de-1/-L1. L’une des limites inhérentes à la HTRF, par rapport à la SPR, est l’extinction de la fluorescence causée par des interactions externes avec le processus d’excitation intramoléculaire (par exemple, le transfert d’électrons, le FRET et le blanchiment), la sensibilité est trop faible dans le processus de criblage de médicaments en raison de la petite plage fenêtre et de l’interférence des composés de la bibliothèque fluorescente ou des protéines biologiques17. Ces caractéristiques positionnent la SPR comme un outil supérieur pour la découverte de médicaments. Nos études précédentes ont démontré que la SPR est capable de déterminer l’effet de blocage de petites molécules contre-1/-L1, ce qui est avantageux par rapport à d’autres techniques qui nécessitent des exigences élevées en matière de technologie de marquage, plusieurs étapes, une faible spécificité et un coût élevé dans le processus de découverte de médicaments18.
Cette étude présente une plate-forme optimisée basée sur la SPR, intégrant un processus de couplage en deux étapes qui utilise à la fois le couplage des amines et de la bio-streptavidine pour améliorer l’orientation de-1 sur la puce et minimiser l’utilisation de protéines. Cette approche mise à jour a été validée avec succès en utilisant l’inhibiteur de-1/-L1 BMS-1166 comme liant de contrôle positif, démontrant des effets de blocage comparables à la fois à notre méthode SPR précédente et à d’autres techniques établies telles que ELISA19,20. Cela confirme non seulement la fiabilité et la reproductibilité de notre protocole, mais illustre également l’efficacité de notre plateforme modifiée pour faciliter le criblage à haut débit des inhibiteurs de-1/-L1. L’incorporation de l’étape de capture de la bio-streptavidine permet une orientation protéique dirigée vers le site plutôt que de manière aléatoire, ce qui permet de réduire la concentration de-1 (40 μg/mL contre 10 μg/mL) et de réaliser des économies en permettant à l’utilisateur final d’immobiliser la streptavidine (SA) sur une puce CM5, une alternative moins coûteuse aux puces SA pré-immobilisées commercialisées. Cela le rend avantageux pour les criblages à grande échelle et rentables de banques de composés/peptides. Bien que des méthodes de caractérisation supplémentaires, y compris des essais in silico, in vitro et in vivo, soient essentielles pour évaluer le potentiel clinique des inhibiteurs de-1/-L1 contre le cancer, notre plateforme de dépistage améliorée basée sur la SPR se distingue comme un outil efficace pour le dépistage à grande échelle des inhibiteurs de-1/-L1.
Protocole
Les réactifs et l’équipement sont répertoriés dans la table des matériaux.
1. Immobilisation de la protéine streptavidine (SA) sur la puce CM5
- Configurez la méthode d’immobilisation sur l’instrument SPR : Ouvrir/Nouveau modèle d’assistant , choisir l’immobilisation, définir le type de puce sur CM5 et les cellules de flux par cycle sur 1. Vérifiez la cellule d’immobilisation 1 et la cellule d’écoulement 2.
- Définissez Amine comme méthode d’immobilisation. Objectif fixé pour le niveau immobilisé, concentration en ligand : 40 μg/mL de streptavidine, niveau cible : 2000 RU, solution de lavage : 50 mM de NaOH. Ensuite, vérifiez prime avant de courir.
- Préparez les tubes suivants : R2 B1 et R2 C1 - 40 μg/mL de streptavidine ; R2 B2 et R2 C2 : 50 mM de NaOH ; R2 B3 et R2 C3 : EDC ; R2 B4 et R2 C4 : ENM ; R2 B5 et R2 C5 : Vide ; R2 B6 et R2 C6 : Éthanolamine.
REMARQUE : EDC et NHS activent les groupes carboxyle sur la puce CM5, permettant le couplage covalent avec les amines sur le ligand. L’éthanolamine est utilisée comme tampon bloquant pour empêcher la liaison non spécifique lors de l’immobilisation. - Diluer 20 mL de tampon HBS-EP+ 10× dans 180 mL d’eau désionisée (DI) pour préparer 200 mL de solution tampon 1× de courant HBS-EP+.
- Ajouter 1 mL d’eau exempte de DNase à 1 mg de streptavidine et incuber à température ambiante pendant 30 min. Ensuite, diluez la solution de streptavidine à 40 μg/mL dans un tampon d’acétate (pH 4,5). Préparez les réactifs du kit de réactifs Amine Coupling conformément aux instructions du fabricant et placez toutes les dispositions pertinentes des réactifs comme indiqué à l’étape 1.2.
- Remplacez la puce du capteur de maintenance par la puce CM5, puis placez le tube A dans le tampon HBS-EP+ 1× préparé, rouvrez la méthode d’immobilisation et insérez les tubes conformément à la disposition de l’étape 1.2. Exécutez la méthode et enregistrez le fichier de résultats.
- Maintenance post-course : remplacez la puce CM5 par la puce du capteur de maintenance, placez le tube A dans une bouteille remplie d’eau déminéralisée et exécutez l’amorçage. Après avoir retiré la puce CM5, lavez-la avec quelques gouttes d’eau DI et séchez-la à l’air. Placez la puce dans un tube de 50 ml à 4 °C.
REMARQUE : Le SA immobilisé permet à la puce CM5 de fonctionner comme une puce SA.
2. Immobilisation de la protéine-1 sur la puce SA
- Définissez la méthode d’immobilisation sur l’instrument SPR : Ouvrir/Nouveau modèle d’assistant , choisissez l’immobilisation, définissez le type de puce sur SA et les cellules de flux par cycle sur 1. Vérifiez les cellules d’immobilisation 1 et 2.
- Définissez la capture de la biotine SA comme méthode. Pour l’ensemble de la cellule 1, l’immobilisation à blanc, pour l’ensemble de la cellule 2, visez le niveau immobilisé, 10 μg/mL de-1 comme ligand, niveau cible : 4000 RU, puis vérifiez l’amorce avant de courir.
REMARQUE : Préparez les tubes suivants - R2 B1 et R2 C1 : 1 M de NaCl, 50 mM de NaOH ; R2 B2 et R2 C2 : 50 % d’isopropanol/50 mM de NaOH/1 M de NaCl ; R2 C3 : 10 μg/mL de-1.
- Définissez la capture de la biotine SA comme méthode. Pour l’ensemble de la cellule 1, l’immobilisation à blanc, pour l’ensemble de la cellule 2, visez le niveau immobilisé, 10 μg/mL de-1 comme ligand, niveau cible : 4000 RU, puis vérifiez l’amorce avant de courir.
- Dissoudre 58,44 mg de NaCl dans 1 mL de 50 mM de NaOH pour préparer 1 M de NaCl et 50 mM de solution de NaOH. Dissoudre 58,44 mg de NaCl et 4,0 mg de NaOH dans de l’eau de 500 μL, puis ajouter 500 μL d’isopropanol pour préparer la solution d’isopropanol à 50 % d’isopropanol/50 mM de NaOH/1 M de NaCl.
- Préparez la solution de-1 : Ajoutez 200 μL d’eau exempte de DNase à 100 μg de-1 humain biotinylé (Fc et Avitagged) et stabilisez à température ambiante pendant 30 min, puis diluez à 10 μg/mL dans un tampon d’acétate (pH 5,0).
- Placez toutes les dispositions de réactifs comme décrit à l’étape 2.1. Placez le tube A dans la solution de tampon de fonctionnement HBS-EP+ 1×, puis éjectez la puce du capteur de maintenance et insérez la puce SA (puce CM5 recouverte de protéine streptavidine de l’étape 1). Rouvrez la méthode d’immobilisation, insérez le rack de réactifs 2 et vérifiez les positions, puis exécutez la méthode pendant la durée d’exécution estimée.
- Répétez l’étape 1.6.
3. Recherche de régénération pour-1 et-L1
- Configurez la méthode de dépistage de régénération : Ouvrir/Nouveau modèle d’assistant , choisissez Filtrage de régénération, définir le chemin d’écoulement : 2-1, 4-3, le type de puce sur SA, vérifier le cycle de conditionnement, enregistrer la solution en tant que HBS-EP+, le temps de contact en 30 s, le nombre d’injections en tant que 3, la solution en tant que -L1, temps de contact : 30 s, débit : 30 μL/min.
- Pour les paramètres de régénération, le débit est de 30 μL/min et la période de stabilisation est de 300 s. Dans le plan expérimental, définissez le nombre de conditions sur 4 et le nombre de cycles pour chaque condition sur 2. Réglez les conditions sur 4, solution de régénération : Glycine 1,5, 2,0, 2,5, 3,0, temps de contact : 30 s, puis vérifiez l’amorçage avant de courir.
- Définir chaque concentration de-L1 comme position de puits d’échantillon distincte dans une disposition de microplaque à 96 puits : R1 A1 à R1 A9 : 1 μM-L1 ; R1 A10 à R1 A12 : tampon HBS-EP+ ; R1 B1 : Glycine 1,5 ; R1 B2 : Glycine 2 ; R1 B3 : Glycine 2,5 ; R1 B4 : Glycine 3.
REMARQUE : Un tampon glycine avec un pH différent est utilisé comme tampon de régénération. - Préparez la solution de-L1 : Ajoutez 200 μL d’eau exempte de DNase à 100 μg de protéine humaine-L1 Fc Tag (9,42 μM), puis diluez à 1 μM dans un tampon HBS-EP+.
- Placez tous les réactifs pertinents dans la disposition décrite à l’étape 3.2. Placez le tube A dans la mémoire tampon HBS-EP+ de 1×, puis remplacez la puce du capteur de maintenance par la puce SA. Rouvrez la méthode de reconnaissance de régénération , suivez la position du tube à l’étape 3.2, insérez le rack de réactifs, puis exécutez la méthode pendant la durée d’exécution estimée.
- Répétez l’étape 1.6.
4. Validation de l’interaction-1/-L1
REMARQUE : Aux fins de validation, un rapport18 publié précédemment a été suivi de modifications mineures.
- Utilisez les mêmes paramètres que le rapport publié sous Paramètres généraux, Étapes de dosage et Types de cycles, réglez le temps de contact sur 60 s, le temps de dissociation sur 60 s et le débit sur 30 μL/min. Sous les variables de méthode, les variables d’évaluation et les commandes, utilisez le même réglage, sauf pour l’utilisation de Glycine 2.0 comme Régénération, réglez le débit à 30 μL/min, passe par le chemin d’écoulement de 1, 2, 3, 4.
- Configurez ensuite exécuter et choisissez le chemin d’écoulement : 2-1, 4-3. Entrez-L1 avec des concentrations de 0 μM, 0,037 μM, 0,111 μM, 0,333 μM et une masse moléculaire de 51 300 Da. Vérifiez toutes les étapes de test pour vérification et sélectionnez Prime avant l’exécution.
- Réglez ensuite chaque concentration de-L1 en tant que position de puits d’échantillon distincte dans une disposition de rack de réactifs 2 : R2 B1 :-L1 0 μM ; R2 B2 :-L1 0,037 μM ; R2 B3 :-L1 0,111 μM ; R2 B4 :-L1 0,333 μM ; R2 A1 : Glycine 2.0 comme tampon de régénération ; R2 A2 : HBS-EP+ comme tampon de démarrage.
- Préparez 200 ml de solution tampon courante HBS-EP+ 1×. Préparer les concentrations de-L1 : diluer la protéine-L1 à 9,42 μM à 0,333 μM, 0,111 μM et 0,037 μM dans HBS-EP+. Placez ensuite tous les réactifs pertinents dans la disposition comme décrit à l’étape 4.1.2.
- Insérez le tube A dans la solution tampon de fonctionnement HBS-EP+ 1×, éjectez la puce du capteur de maintenance et insérez la puce SA. Rouvrez la méthode de dépistage de régénération , suivez le positionnement du tube de l’étape 4.1, insérez le rack de réactifs 2 et exécutez la méthode pendant la durée estimée.
- Répétez l’étape 1.6.
5. Test de blocage de-1/-L1 avec inhibiteur de petites molécules : BMS-1166
REMARQUE : Pour l’essai de blocus, un rapport18 publié précédemment a été suivi de quelques ajustements mineurs.
- Utilisez les mêmes paramètres que le rapport publié sous Paramètres généraux, Étapes de dosage et Types de cycles, réglez le temps de contact sur 60 s, le temps de dissociation sur 60 s et le débit sur 30 μL/min. Sous les variables de méthode, les variables d’évaluation et les commandes utilisent également le même réglage, à l’exception de l’utilisation de Glycine 2.0 comme régénération, règle le débit à 30 μL/min, passe par le chemin d’écoulement de 1, 2, 3, 4.
- Configurez ensuite exécuter et choisissez le chemin d’écoulement : 2-1, 4-3. Entrez la solution d’échantillon :-L1 (0,111 μM, poids moléculaire de 51 300 Da) avec BMS-1166 à 0 μM, 0,125 μM, 0,625 μM, 3,125 μM. Vérifiez toutes les étapes de test pour la vérification et sélectionnez l’amorce avant l’analyse.
- Réglez ensuite chaque concentration de-L1 comme position de puits d’échantillon distincte dans une disposition de rack de réactifs 2 : R2 B1 :-L1 (0,111 μM) + BMS-1166 0 μM R2 B2 :-L1 (0,111 μM) + BMS-1166 0,125 μM ; R2 B3 :-L1 (0,111 μM) + BMS-1166 0,625 μM ; R2 B4 :-L1 (0,111 μM) + BMS-1166 3,125 μM ; R2 A1 : Glycine 2.0 pour la régénération.
- Préparez 200 ml de solution tampon courante HBS-EP+ 1×. Préparez le mélange BMS-1166/-L1 : Dissoudre 5 mg de BMS-1166 dans 77,99 μL de diméthylsulfoxyde (DMSO) pour préparer une solution mère de 100 mM. Diluer la solution mère avec la protéine-L1 (0,11 μM) aux concentrations cibles de BMS-1166 à 0 μM, 0,125 μM, 0,625 μM et 3,125 μM dans HBS-EP+. Placez toutes les dispositions de réactifs appropriées comme décrit à l’étape 5.1.2.
- Placez le tube A dans la solution tampon de fonctionnement HBS-EP+ 1×, puis éjectez la puce du capteur de maintenance et insérez la puce SA. Rouvrez la méthode de dépistage de régénération , suivez la position du tube à partir de la position 5.1 et, insérez le rack de réactifs 2, puis exécutez la méthode pendant la durée d’exécution estimée.
- Répétez l’étape 1.6.
Résultats
Immobilisation de SA sur puce CM5
Les données ont été analysées à l’aide des résultats de l’instrument SPR et du logiciel d’analyse associé, indiquant l’atteinte réussie de l’EF cible (2000 EF) de la protéine SA sur les cellules d’écoulement 1 et 2. Les cellules d’écoulement 1 et 2 ont été immobilisées avec de l’AS (40 μg/mL) à la surface de la puce CM5 avec une réponse finale de 1902,3 RU sur la cellule d’écoulement 1 (figure 1A) et de 1900,7 RU sur la cellule d’écoulement 2 (figure 1B).
Immobilisation de-1 sur puce SA
L’analyse des données basée sur les résultats de l’instrument SPR et du logiciel associé a indiqué une unité de réponse faible (RU) pour la cellule vierge sur la cellule d’écoulement 1 et l’obtention réussie de l’EF cible (4000 RU) pour le ligand-1 sur la cellule d’écoulement 2. La cellule d’écoulement 1 a été immobilisée à blanc, ce qui a donné une réponse finale de -161,0 RU (figure 2A), tandis que la cellule d’écoulement 2 a été immobilisée avec la protéine-1 biotinylée (10 μg/mL) enrobée sur la puce SA, ce qui a donné une réponse finale de 3698,5 RU (figure 2B).
Dépistage de régénération pour-1 et-L1.
Le dépistage de la régénération a été effectué avec-1 immobilisé sur la cellule d’écoulement 2 et-L1 en solution (0,1 μM) à divers niveaux de pH de glycine (1,5, 2, 2,5 et 3) pour déterminer la solution de régénération, ce qui a permis d’obtenir une ligne de base stable et une réponse de l’échantillon. Les données ont été analysées via les résultats de l’instrument SPR et du logiciel d’analyse, ce qui a permis de déterminer que la glycine (pH 2,0) est la condition de régénération optimale en raison de changements minimes de la réponse de base et de la réponse de l’échantillon, indiquant une perte minimale de protéine-1 et une élimination réussie de la protéine-L1 de la surface de la puce. À un pH de glycine de 2,0, la réponse de base est demeurée relativement constante, et la réponse de l’analyte était stable et proche de la réponse au début de l’expérience, indiquant le tampon de régénération le plus optimal parmi les quatre conditions de pH de glycine testées. Un pH plus élevé est insuffisant et un pH plus bas est trop dur ; Un pH de 2,0 est identifié comme la condition de régénération la plus appropriée (figure 3).
Validation de l’interaction-1/-L1
Les données ont été analysées à l’aide du logiciel d’évaluation correspondant. Dans la section Cinétique/Affinité, sélectionnez « lié à la surface », choisissez la courbe 2-1, réglez le modèle cinétique sur une liaison 1:1 et ajustez le paramètre RI sur un ajustement constant pour déterminer le taux d’association (ka), le taux de dissociation (kd) et la constante de dissociation d’équilibre (KD). L’interaction de liaison de-L1 à des concentrations variables avec-1 a été quantifiée, ce qui a donné lieu à une réponse mesurable (figure 4). Les paramètres de liaison analysés comprenaient un taux d’association (ka) de 3,611 × 104 (1/Ms), un taux de dissociation (kd) de 0,0236 (1/s) et une constante de dissociation à l’équilibre (KD) de 6,536 × 10-7 M.
Test de blocage de-1/-L1 avec inhibiteur de petites molécules établi : BMS-1166
Les données ont été analysées à l’aide des données correspondantes. Sous Cinétique/Affinité, sélectionnez Lié à la surface, choisissez la courbe 2-1, puis obtenez la courbe visualisée du résultat et l’unité de réponse. L’effet de blocage de l’interaction de liaison-1/-L1 a été observé avec BMS-1166 (0-3,125 μM) avec 0,11 μM de protéine-L1 dans le tampon HBS EP+ (Figure 5). L’unité de réponse la plus élevée est identifiée par-L1 seul, tandis qu’avec l’augmentation de la concentration de BMS-1166, l’unité de réponse de liaison diminue proportionnellement.

Figure 1 : Immobilisation de SA sur puce CM5. Les courbes d’immobilisation de la protéine streptavidine sur la cellule d’écoulement 1 (A) et la cellule d’écoulement 2 (B) de la puce CM5 sont illustrées. Tout d’abord, cinq pré-concentrations de la protéine SA ont circulé sur les cellules de flux 1 et 2, suivies d’un lavage au NaOH et de l’établissement d’une base de référence stable. Ensuite, l’EDC et le NHS ont été ajoutés, puis un lavage au chlorhydrate d’éthanolamine a été effectué. Ensuite, cinq impulsions de-1 ont été effectuées pour atteindre le niveau cible (2000 RU), suivies d’un lavage au chlorhydrate d’éthanolamine pour éliminer les ligands liés électrostatiquement et désactiver les esters NHS qui restent non réactifs. Veuillez cliquer ici pour voir une version agrandie de cette figure.
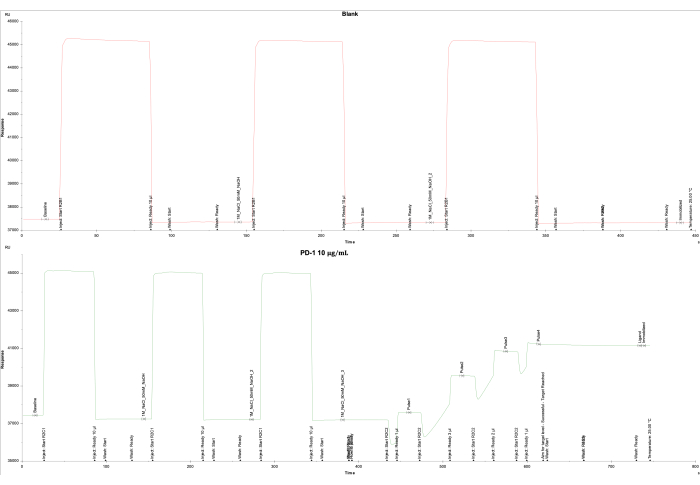
Figure 2 : Immobilisation de-1 sur puce SA. Les courbes d’immobilisation de (A) la cellule d’écoulement vierge 1 recouverte de SA uniquement, et (B) la protéine-1 sur la puce SA de la cellule d’écoulement 2 sont représentées. Pour le blanc (cellule d’écoulement 1), 1 M de NaCl et 50 mM de NaOH ont été injectés trois fois, suivis d’un lavage avec 50 % d’isopropanol/1 M de NaCl/50 mM de NaOH. Pour la protéine-1 (cellule d’écoulement 2), 1 M de NaCl et 50 mM de NaOH ont été injectés trois fois, suivis de cinq impulsions de protéine-1 pour atteindre le niveau cible de 4000 RU et d’un lavage final de 50 % d’isopropanol/1 M de NaCl/50 mM de NaOH. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Dépistage de la régénération pour-1 et-L1. La réponse de base et la réponse de l’échantillon ont été obtenues pour quatre conditions de régénération avec de la glycine à des pH de 1,5, 2, 2,5 et 3 afin de déterminer la solution de régénération optimale avant l’analyse de l’échantillon. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Validation de l’interaction-1/-L1. Cinétique de liaison de différentes concentrations de-L1 (0,037 μM, 0,111 μM et 0,333 μM) à-1 sur la surface de la puce recouverte de streptavidine. -L1 présente des phases d’association (0-60 s) et de dissociation (61-120 s) distinctes avec-1. Veuillez cliquer ici pour voir une version agrandie de cette figure.
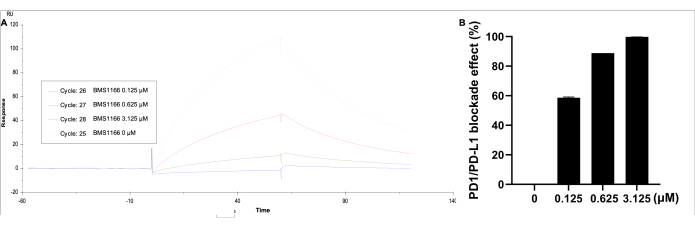
Figure 5 : Analyse SPR de-1 enrobé sur une puce CM5 immobilisée SA, avec-L1 (0,11 μM) et BMS-1166 (0-3,125 μM) en solution. (A) Une réponse SPR représentative en temps réel des réactions de liaison entre-1/-L1 et BMS-1166. (B) Le pourcentage d’effet de blocage BMS-1166 sur la cinétique de liaison de l’interaction-1/-L1. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 6 : Comparaison des stratégies d’immobilisation de la protéine-1. Couplage d’amines (A) vs. couplage streptavidine-biotine en deux étapes (B). Le couplage des amines présente des défis, notamment la disponibilité limitée de sites de liaison accessibles, l’obstacle stérique à la liaison et la modification potentielle des sites de liaison-1 pendant l’immobilisation. En revanche, l’approche en deux étapes streptavidine-biotine améliore la disponibilité des sites de liaison libres sur le-1 immobilisé, facilitant ainsi une meilleure interaction avec le-L1 en solution. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Au cours des dernières décennies, diverses approches d’immunothérapie, notamment les vaccins contre le cancer, les inhibiteurs de points de contrôle immunitaires et les thérapies à base de cellules CAR-T, ont considérablement fait progresser le traitement du cancer21. Les points de contrôle immunitaires jouent un rôle crucial dans la prévention des dommages collatéraux médiés par les cellules immunitaires lors des réponses pathogènes et dans la suppression de l’auto-immunité. Un exemple clé est l’interaction entre-L1 et-1, qui forme un point de contrôle immunitaire majeur, permettant aux cellules cancéreuses d’échapper à la surveillance immunitaire. Le ciblage de la voie-1/-L1 avec des anticorps monoclonaux a obtenu un succès remarquable en oncologie clinique. Cependant, en raison des limites associées aux thérapies par anticorps monoclonaux et de l’incidence croissante des événements indésirables liés au système immunitaire, il existe un intérêt croissant pour le développement d’inhibiteurs à petites molécules ciblant-1/-L1 9,22.
Les stratégies de dépistage actuelles des inhibiteurs de-1/-L1 à petites molécules se concentrent principalement sur des techniques d’essais biologiques telles que l’ELISA, les tests de rapporteur cellulaire et les tests de cellules T. Les techniques biophysiques, y compris la SPR et l’interférométrie de biocouche (BLI), sont largement utilisées pour la caractérisation des affinités de liaison, mais leur potentiel à être utilisé comme outil de criblage est sous-estimé23. Cette étude a mis au point un test de blocage-1/-L1 optimisé basé sur SPR, qui offre une plateforme à haut débit adaptée à la découverte de petites molécules inhibitrices de-1/-L1. L’AS a été immobilisée à ~2000 RU, suivie par la-1 biotinylée (~4000 RU) via l’interaction biotine-streptavidine. Cette liaison robuste a assuré un revêtement-1 sécurisé avec une orientation optimale, minimisant la liaison non spécifique et réduisant l’utilisation de protéines (10 μg/mL). L’immobilisation dirigée sur site a amélioré l’efficacité par rapport aux méthodes conventionnelles. Un tampon glycine (pH 2,0) a été utilisé pour régénérer la surface du capteur entre les cycles, en maintenant l’intégrité expérimentale et en empêchant la liaison non spécifique.
Par rapport aux méthodes immunologiques standard et à l’ELISA, cette approche SPR offrait une détection en temps réel, sans marquage, avec une sensibilité et une spécificité élevées. De plus, il permet un criblage à haut débit avec une durée d’exécution de 120 s/échantillon et complète les essais biologiques dans l’évaluation de l’efficacité de blocage des composés ciblant-1/-L1 et des produits biologiques. Le niveau d’immobilisation atteint était comparable à celui de la plateforme précédente (3698,5 RU contre 3688,5 RU), avec une affinité d’interaction-1/-L1 similaire (KD = 6,536 × 10-7 M contre 1,295 × 10-7 M). L’inhibiteur de BMS-1166 a démontré un taux de dissociation plus élevé à une concentration plus faible, avec des effets de blocage comparables à ceux de la plateforme précédente (99,8 % contre 94,2 % à 3,125 μM). BMS-1166 a démontré des taux de blocage-1/-L1 soutenus par des valeurs IC50 de 1,4 nM et 96 nM dans des tests HTRF et cellulaires, respectivement24. De plus, d’autres études ont rapporté des valeurs de CI50 de 3,9 nM et 1574 nM à l’aide de méthodes d’essai de co-culture HTRF et de blocage immunitaire25. Nos résultats précédents ont montré que les valeurs IC50 de BMS-1166 étaient de 85,4 nM, ce qui est cohérent avec ces résultats antérieurs18. Un autre avantage de cette plate-forme de criblage est sa robustesse et son débit élevé. Cette méthode a été largement appliquée au criblage à haut débit à l’aide d’un format de plaque de 384 puits, avec BMS-1166 et BMS-202 inclus comme témoins positifs pour 10 échantillons. Les plages de taux de blocage étaient de 29,8 % à 38,1 % pour le BMS-1166 et de 6,0 % à 10,4 % pour le BMS-202 à 10 nM (n = 11 par plaque).
Le DMSO est susceptible d’avoir des effets variables sur les membranes biologiques hétérogènes, en fonction de leur composition et de leur structure locales, influençant potentiellement les fonctions biologiques associées aux membranes. À des concentrations relativement faibles, le DMSO peut modifier les propriétés des protéines en solution, entraînant une dénaturation, une agrégation ou une dégradation. De plus, le DMSO peut modifier les propriétés de liaison apparentes des protéines26,27. De plus, pour répondre aux préoccupations précédentes concernant l’interférence du DMSO, nous avons réduit la concentration de DMSO à 0,003 % (0,01 % de DMSO était utilisé dans la plateforme précédente) dans ce protocole.
Dans une étude précédente, les protéines-1 ont été immobilisées sur des puces de capteur à l’aide d’un couplage d’amines, qui utilise la chimie EDC/NHS pour activer les groupes carboxyméthyles sur la puce, formant des liaisons covalentes avec des groupes amines sur-1. En raison de la présence de plusieurs groupes amines dans les 288 acides aminés de-1, cette méthode entraîne intrinsèquement des orientations aléatoires. Pour obtenir une orientation plus spécifique, nous avons utilisé une stratégie de couplage de capture avec des-1 humains biotinylés (Fc, Avitag) dans cette étude. Le résidu unique de lysine dans l’Avitag est biotinylé enzymatiquement sur la région Fc de-1, ce qui permet une immobilisation précise grâce à l’interaction de haute affinité entre la biotine et la streptavidine (Figure 6). Théoriquement, cette approche garantit que le domaine variable de-1 reste exposé, facilitant ainsi une interaction optimale avec-L1 dans le tampon. Cependant, cette hypothèse nécessite une validation supplémentaire à l’aide de techniques telles que la cryo-EM ou la cristallographie pour confirmer l’orientation des protéines.
La glycine 2.0 a été utilisée comme tampon de régénération doux, éliminant efficacement le-L1 sans réduire le-1 immobilisé. Cependant, pour les inhibiteurs étroitement liés ou les agrégats de protéines, des tampons de régénération plus puissants, tels que 0,5 % de SDS ou 50 à 100 mM de NaOH, peuvent être nécessaires pour éviter l’interférence avec les échantillons ultérieurs.
Cette étude présente plusieurs limites. Tout d’abord, le coût élevé de l’instrumentation SPR peut limiter l’accessibilité, bien que le protocole puisse souvent être adapté à d’autres systèmes SPR ayant des capacités similaires. Des paramètres tels que la concentration en protéines et les temps d’association/dissociation peuvent être ajustés pour s’adapter à des plateformes SPR spécifiques. Une autre limitation est sa dépendance à l’égard des protéines recombinantes, qui peuvent ne pas reproduire complètement les interactions des protéines natives. Ces facteurs doivent être pris en compte lors de l’interprétation des données SPR et de la comparaison des résultats avec des techniques complémentaires, telles que les essais cellulaires ou les modèles in vivo .
Malgré ces limitations, la méthode SPR optimisée offre une approche rapide, en temps réel, à haut débit et sans marquage pour le criblage des inhibiteurs à petites molécules de l’interaction-1/-L1. Il améliore considérablement les techniques biophysiques de caractérisation des petites molécules et des produits biologiques ciblant l’interaction-1/-L1. Grâce à sa sensibilité élevée, la plateforme est particulièrement précieuse pour étudier les interactions entre les points de contrôle immunitaires et les interactions protéine-protéine (IPP) plus larges, ce qui en fait un outil puissant pour faire progresser la découverte de médicaments IPP.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs saluent la plateforme RI-INBRE de l’Université de Rhode Island, soutenue par des P20GM103430 de subvention du National Center for Research Resources (NCRR), une composante des National Institutes of Health (NIH). Cette recherche a été soutenue par une subvention pilote du College of Pharmacy de l’Université de Rhode Island, une subvention de petite taille du Rhode Island Life Science Hub (RILSH) et une subvention de la Fondation du Rhode Island, toutes attribuées à Chang Liu, Ph.D.
matériels
| Name | Company | Catalog Number | Comments |
| 50 mM NaOH | Cytiva Life Sciences | 100358 | |
| 50 mM NaOH | Fisher Scientific | 905376 | |
| 96-Well Polystyrene Microplates | Cytiva Life Sciences | BR100503 | |
| Amine Coupling Kit | Cytiva Life Sciences | 35120 | |
| Biacore T200 SPR System and Evaluation Software 3.2 | Cytiva Life Sciences | 28975001 | |
| Biotinylated Human PD-1 Fc, Avitag Protein | Acro Biosystems | PD1-H82F1 | |
| BMS1166 | MedChemExpress | HY-102011 | |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | 276855 | |
| DNase Free Water | Fisher Scientific | 188506 | |
| Glycine 1.5 | Cytiva Life Sciences | BR100354 | |
| Glycine 2.0 | Cytiva Life Sciences | BR100355 | |
| Glycine 2.5 | Cytiva Life Sciences | BR100356 | |
| Glycine 3.0 | Cytiva Life Sciences | BR100357 | |
| HBS-EP+ Buffer | Cytiva Life Sciences | BR100669 | |
| Human PD-L1 Fc Tag Protein | Acro Biosystems | PD-1-H5258 | |
| Isopropanol | Fisher Scientific | BP2618-1 | |
| Microplate Foil, 96-Well | Cytiva Life Sciences | 28975816 | |
| NaCl | Sigma-Aldrich | 746398 | |
| Plastic Vials 7 mm | Cytiva Life Sciences | BR100212 | |
| Rubber Caps, Type 3 | Cytiva Life Sciences | BR100502 | |
| Series S Sensor Chip CM5 | Cytiva Life Sciences | 29149603 | |
| Sodium Acetate 4.5 | Cytiva Life Sciences | 100350 | |
| Sodium Acetate 5.0 | Cytiva Life Sciences | 100351 | |
| Streptavidin | Sigma-Aldrich | S4762 |
Références
- Ribas, A., Wolchok, J. D. Cancer immunotherapy using checkpoint blockade. Science. 359 (6382), 1350-1355 (2018).
- Zhang, X., et al. Structural and functional analysis of the costimulatory receptor programmed death-1. Immunity. 20 (3), 337-347 (2004).
- Freeman, G. J., et al. Engagement of the PD-1 immunoinhibitory receptor by a novel B7 family member leads to negative regulation of lymphocyte activation. J Exp Med. 192 (7), 1027-1034 (2000).
- Han, Y., Liu, D., Li, L. PD-1/PD-L1 pathway: Current researches in cancer. Am J Cancer Res. 10 (3), 727-742 (2020).
- Lyu, N., et al. Recognition of PDL1/L2 by different induced-fit mechanisms of PD1: A comparative study of molecular dynamics simulations. Phys Chem Chem Phys. 22 (3), 1276-1287 (2020).
- Taube, J. M., et al. Colocalization of inflammatory response with B7-H1 expression in human melanocytic lesions supports an adaptive resistance mechanism of immune escape. Sci Trans Med. 4 (127), 127ra37(2012).
- Sun, X., et al. Immune-related adverse events associated with programmed cell death protein-1 and programmed cell death ligand 1 inhibitors for non-small cell lung cancer: A PRISMA systematic review and meta-analysis. BMC Cancer. 19 (1), 558(2019).
- Alkholifi, F. K., Alsaffar, R. M. Dostarlimab an inhibitor of PD-1/PD-L1: A new paradigm for the treatment of cancer. Medicina. 58 (11), 1572(2022).
- Uzar, W., et al. An updated patent review on PD-1/PD-L1 antagonists (2022-present). Expert Opin Ther Pat. 34 (8), 627-650 (2024).
- Ai, L., et al. Research status and outlook of PD-1/PD-L1 inhibitors for cancer therapy. Drug Des Devel Ther. 14, 3625-3649 (2020).
- Conroy, M., Naidoo, J. Immune-related adverse events and the balancing act of immunotherapy. Nat Commun. 13 (1), 392(2022).
- Guzik, K., et al. Small-molecule inhibitors of the Programmed Cell Death-1/Programmed Death-Ligand 1 (PD-1/PD-L1) interaction via transiently induced protein states and dimerization of PD-L1. J Med Chem. 60 (13), 5857-5867 (2017).
- Sifniotis, V., Cruz, E., Eroglu, B., Kayser, V. Current advancements in addressing key challenges of therapeutic antibody design, manufacture, and formulation. Antibodies. 8 (2), 36(2019).
- Beck, H., Härter, M., Haß, B., Schmeck, C., Baerfacker, L. Small molecules and their impact in drug discovery: A perspective on the occasion of the 125th anniversary of the Bayer Chemical Research Laboratory. Drug Discov Today. 27 (6), 1560-1574 (2022).
- Nguyen, H. H., Park, J., Kang, S., Kim, M. Surface plasmon resonance: A Versatile technique for biosensor applications. Sensors (Basel, Switzerland). 15 (5), 10481-10510 (2015).
- Liu, C., Seeram, N. P., Ma, H. Small molecule inhibitors against PD-1/PD-L1 immune checkpoints and current methodologies for their development: A review. Cancer Cell Int. 21 (1), 239(2021).
- Hu, K., Li, X. -J., Asmamaw, M. D., Shi, X. -J., Liu, H. -M. Establishment of high-throughput screening HTRF assay for identification small molecule inhibitors of Skp2-Cks1. Sci Rep. 11 (1), 21105(2021).
- Puopolo, T., et al. Establishment of human PD-1/PD-L1 blockade assay based on surface plasmon resonance (SPR) biosensor. Bio-protoc. 13 (15), e4765(2023).
- Ding, M., Chen, Y., Lang, Y., Cui, L. The role of cellular prion protein in cancer biology: A potential therapeutic target. Front Oncol. 11, 742949(2021).
- Li, H., Seeram, N. P., Liu, C., Ma, H. Further investigation of blockade effects and binding affinities of selected natural compounds to immune checkpoint PD-1/PD-L1. Front Oncol. 12, 995461(2022).
- Kamrani, A., et al. New immunotherapeutic approaches for cancer treatment. Pathol Res Pract. 248, 154632(2023).
- Yan, Y., Zhang, L., Zuo, Y., Qian, H., Liu, C. Immune checkpoint blockade in cancer immunotherapy: mechanisms, clinical outcomes, and safety profiles of PD-1/PD-L1 inhibitors. Arch Immunol Ther Exp. 68 (6), 36(2020).
- Chandrasekharan, G., Unnikrishnan, M. High throughput methods to study protein-protein interactions during host-pathogen interactions. Eur J Cell Biol. 103 (2), 151393(2024).
- Zhang, Y., et al. BMS-202, a PD-1/PD-L1 inhibitor, decelerates the profibrotic effects of fibroblasts derived from scar tissues via ERK and TGFβ1/Smad signaling pathways. Immun Inflamm Dis. 10 (1), e591(2022).
- Surmiak, E., et al. PD-L1 inhibitors: Different classes, activities, and mechanisms of action. Int J Mol Sci. 22 (21), 11797(2021).
- Feoli, A., Sarno, G., Castellano, S., Sbardella, G. DMSO-Related effects on ligand-binding properties of lysine methyltransferases G9a and SETD8. ChemBioChem. 25 (4), e202300809(2024).
- Tjernberg, A., Markova, N., Griffiths, W. J., HalléN, D. DMSO-Related effects in protein characterization. SLAS Discov. 11 (2), 131-137 (2006).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon