Method Article
Capturando quebras de local frágeis comuns por γH2A nativo. X ChIP
Neste Artigo
Resumo
Apresentamos um método rápido e eficiente para detectar quebras comuns de locais frágeis por meio da imunoprecipitação nativa da cromatina γH2A.X (ChIP). Essa abordagem reduz significativamente o tempo e a mão de obra associados aos ensaios tradicionais de γH2A.X ChIP, mantendo alta reprodutibilidade e confiabilidade dos resultados.
Resumo
O estresse de replicação induzido pela exposição a agentes extrínsecos pode levar a quebras de DNA em locais frágeis comuns, que são regiões do genoma conhecidas por serem propensas à instabilidade estrutural. O ensaio de imunoprecipitação da cromatina γH2A.X (ChIP) serve como uma ferramenta poderosa em estudos de genotoxicidade, pois a fosforilação de γH2A.X é um marcador bem estabelecido para quebras de fita dupla de DNA. Os ensaios tradicionais de γH2A.X ChIP, no entanto, costumam ser trabalhosos e envolvem várias etapas demoradas. Neste estudo, apresentamos um método simplificado, mas eficaz, que combina fracionamento subcelular com ChIP nativo para isolar complexos associados a γH2A.X. Essa abordagem é particularmente adequada para analisar as interações γH2A.X-cromatina com especificidade e eficiência aprimoradas. Usando o fracionamento subcelular, os materiais não ligados à cromatina são efetivamente removidos, resultando em uma fração de cromatina purificada. A digestão subsequente da nuclease microcócica (MNase) em condições amenas permite a fragmentação da cromatina, preservando as interações fisiológicas entre γH2A.X e seus complexos proteicos associados. Essa preservação é essencial para estudar parceiros de interação nativos envolvidos nas vias de resposta a danos no DNA. Este protocolo ChIP nativo otimizado reduz substancialmente o tempo e a mão de obra associados aos ensaios convencionais de ChIP γH2A.X. O procedimento simplificado não apenas simplifica o fluxo de trabalho, mas também produz resultados altamente reprodutíveis, tornando-o particularmente vantajoso em ambientes onde é necessário o processamento de alto rendimento de várias amostras. Este método tem ampla aplicabilidade em estudos focados na estabilidade do genoma, reparo do DNA e biologia da cromatina, onde a detecção precisa e eficiente de locais de dano ao DNA é crítica. Ao empregar protocolos otimizados e etapas simplificadas, esse método permite a detecção de danos ao DNA em locais frágeis com sensibilidade aprimorada e manuseio mínimo de amostras, tornando-o uma ferramenta valiosa para estudos sobre estabilidade do genoma e resposta a danos no DNA.
Introdução
Locais frágeis comuns (CFSs) são grandes regiões cromossômicas encontradas em todos os cromossomos humanos propensos a quebrar durante a metáfase. Sob estresse de replicação, a replicação nessas regiões é significativamente atrasada, impedindo sua duplicação completa antes da entrada mitótica1, o que acaba resultando em lacunas e quebras específicas do local. Os CFSs são pontos críticos para instabilidade cromossômica e são uma das principais causas de rearranjos cromossômicos durante o desenvolvimento inicial do câncer. O estresse de replicação, que geralmente está presente em condições tumorigênicas, pode levar à perda de genes supressores de tumor e amplificação de oncogenes - coletivamente chamados de variação do número de cópias (CNV) 2 , 3 , 4 , 5 , 6 . Além disso, os CFSs são altamente propensos à integração viral, promovendo ainda mais o desenvolvimento do câncer 7,8,9,10. Múltiplas deleções homozigóticas de genes supressores de tumor foram detectadas em regiões da SFC durante análises pan-câncer de tumores primários. Os SFCs mais comumente afetados no câncer incluem FRA2F, FRA3B, FRA4F, FRA5H e FRA16D11. Os SFCs são particularmente vulneráveis à quebra na presença de agentes carcinogênicos extrínsecos12. Para avaliar os efeitos carcinogênicos prejudiciais dos contaminantes ambientais, é necessário um método rápido e confiável para quantificar a ocorrência de quebra de SFC.
Fosforilação de H2A. X no resíduo de serina 139 (γH2A.X) por Ataxia Telangiectasia e Proteína Relacionada a Rad3 (ATR) ou Ataxia Telangiectasia Mutada (ATM) é um evento chave na sinalização de travamento do garfode replicação 13. γH2A.X serve como um indicador de garfos de replicação paralisados antes da formação de quebra de fita dupla (DSB)13, criando um ambiente de cromatina favorável para facilitar o recrutamento eficiente de proteínas de reparo para locais paralisados. Além disso, γH2A.X pode ser recrutado para locais de quebra após o colapso do garfo14,15, consistente com seu papel principal no reparo de DSB. Como as quebras da SFC estão intimamente associadas a aberrações cromossômicas que impulsionam a progressão do câncer, a detecção dessas quebras pode ser fundamental para entender os estágios iniciais da tumorigênese. A presença de γH2A.X em CFSs pode ser usada como um biomarcador para detectar eventos precoces de instabilidade genômica. Essas informações podem ajudar a identificar potenciais carcinógenos e avaliar o risco associado à exposição a vários agentes extrínsecos. Ao medir quebras de DNA em CFSs induzidas por agentes extrínsecos, a cromatina IP γH2A.X (ChIP) pode fornecer informações sobre como esses agentes contribuem para os mecanismos subjacentes à tumorigênese.
No ChIP convencional (ou seja, ChIP reticulado, X-ChIP), a associação de γH2A.X com suas sequências de DNA alvo é estabilizada por reticulação reversível de formaldeído. A cromatina é subsequentemente cortada em fragmentos de aproximadamente 500 pares de bases (pb) por meio de sonicação, e a solução resultante é limpa de detritos por sedimentação 16,17,18. Um anticorpo γH2A.X de grau ChIP é então adicionado à fração de cromatina eliminada, seguido pela adição de grânulos de agarose de proteína A / G para enriquecer as regiões de cromatina ligadas a γH2A.X 16,17,18. Os complexos imunes (ou seja, complexo de DNA direcionado a grânulos-anticorpo-γH2A.X) são lavados várias vezes com tampões de lavagem rigorosos para remover fragmentos de DNA ligados não especificamente 16,17,18. Após a lavagem, o DNA especificamente ligado é eluído dos complexos imunes. As ligações cruzadas do formaldeído são então revertidas, seguidas pela digestão de proteínas usando proteinase K, após a qual o DNA enriquecido é purificado e concentrado 16,17,18. Para avaliar as regiões associadas ao γH2A.X, utiliza-se PCR, PCR quantitativo (qPCR) ou sequenciamento direto 16,17,18. A ocupação de γH2A.X em regiões específicas, como CFS, é determinada pela intensidade do sinal de PCR ou qPCR, que é proporcional à quantidade de γH2A.X ligada naquele local, fornecendo informações sobre danos ao DNA específicos do local e eventos de reparo 16,17,18.
Apesar de ser uma abordagem experimental poderosa, o X-ChIP tem várias limitações significativas: (i) requer um grande número de células, tipicamente na faixa de 1 x 107 a 5 x 107, devido à ineficiência da precipitação de anticorpos associada à fixação, o que aumenta o custo geral do experimento19; (ii) o processo de reversão das ligações cruzadas do formaldeído e subsequente purificação do DNA é demorado e trabalhoso, dificultando a manutenção da consistência e confiabilidade dos resultados; e (iii) interações γH2A.X-DNA com menor significado funcional não podem ser distinguidas daquelas com maior significado porque a etapa de reticulação pode estabilizar interações transitórias, levando à detecção de interações que podem não ser biologicamente relevantes19.
A imunoprecipitação da cromatina nativa (ChIP nativo ou N-ChIP) é uma técnica bioquímica essencial usada para estudar as interações proteína-DNA dentro de seu contexto de cromatina nativa sob condições fisiológicas de sal. Tem sido fundamental para elucidar a organização espacial e temporal da cromatina, ligação do fator de transcrição e modificações de histonas. O Native ChIP tem um papel de longa data no campo mais amplo da biologia e epigenética da cromatina, oferecendo vantagens e limitações únicas em comparação com o X-ChIP. Esse método, introduzido no final da década de 198020, envolve o isolamento da cromatina das células por métodos que preservam sua estrutura nativa, como a digestão com nuclease microcócica (MNase)21. Isso preserva os contatos proteína-DNA e histona-DNA inerentes, o que torna o ChIP nativo particularmente adequado para estudar modificações de histonas e posicionamento de nucleossomos em sua configuração natural de cromatina22. Estudos de ChIP nativo de alta resolução demonstraram o uso da digestão da MNase para reduzir a cromatina a nucleossomos individuais, o que facilita o mapeamento de modificações de histonas com maior precisão23. Além disso, como nenhuma reticulação química está envolvida, o risco de introduzir vieses ou artefatos que possam deturpar as interações proteína-DNA é minimizado24.
Em contraste com o X-ChIP, onde o formaldeído ou outros agentes de reticulação são usados para corrigir as interações proteína-DNA, o Native ChIP fornece uma visão mais realista da cromatina, evitando possíveis artefatos de reticulação. No entanto, enquanto o X-ChIP é geralmente mais adequado para detectar interações transitórias ou dinâmicas entre DNA e proteínas reguladoras25, o ChIP nativo é ideal para interações proteína-DNA estáveis, como histonas ou outras proteínas ligadas à cromatina26 , 27 . Uma das limitações observadas para o ChIP nativo é a incapacidade de capturar eventos de ligação transitórios ou de baixa afinidade, que geralmente são estabilizados por meio de reticulação no X-ChIP25.
Um corpo significativo de trabalho em epigenética alavancou o ChIP nativo para descobrir modificações de histonas em diversos ambientes biológicos28. Esses esforços têm sido cruciais na definição do código das histonas - o padrão de modificações das histonas que regulam a expressão gênica e a dinâmica da cromatina29. Embora H2A. X é uma histona ligante menos fortemente associada, a H2A nativa. O método X ChIP foi aplicado com sucesso em células-tronco embrionárias30. Neste estudo, otimizamos um procedimento de extração de cromatina para realizar ChIP nativo de γH2A.X em células 293T humanas (Figura 1). A hidroxiureia e a afidicolina são amplamente utilizadas em pesquisas para investigar o estresse, danos e instabilidade genômica da replicação do DNA31. Neste estudo, esses agentes foram aplicados às células para induzir o estresse de replicação e gerar quebras de DNA na SFC.
Usando material de partida de aproximadamente 1 x 106 a 5 x 106 células, este método pode ser dividido em quatro estágios principais: (i) fracionamento subcelular para isolar a cromatina, (ii) digestão da nuclease microcócica (MNase) para fragmentar a cromatina, (iii) imunoprecipitação e eluição e (iv) análise de DNA por PCR quantitativo (qPCR). A realização de ChIP após o fracionamento subcelular oferece vários benefícios e foi bem documentada em vários estudos 32,33,34,35. Essa abordagem permite a remoção de proteínas não ligadas à cromatina e outros detritos celulares, resultando em uma fração de cromatina altamente purificada. Ao isolar a cromatina antes da imunoprecipitação, o fracionamento subcelular ajuda a manter as interações nativas da cromatina e reduz o ruído de fundo de proteínas não associadas à cromatina, o que leva a resultados mais específicos e confiáveis, pois apenas os complexos ligados à cromatina são retidos para análise. Além disso, o fracionamento subcelular permite condições mais brandas para a digestão da cromatina, preservando assim as interações fisiológicas proteína-DNA e oferecendo uma representação mais precisa da dinâmica da cromatina no ambiente celular nativo.
O uso de ChIP nativo de γH2AX para medir o impacto de agentes extrínsecos na quebra de locais frágeis comuns tem um potencial significativo para a pesquisa do câncer. Essa técnica permite a detecção de danos ao DNA induzidos pela exposição a agentes cancerígenos ambientais, fornecendo informações sobre os mecanismos moleculares pelos quais os poluentes contribuem para a instabilidade genômica e o desenvolvimento do câncer. Ao preservar o contexto da cromatina nativa, este método facilita a avaliação precisa dos padrões de danos ao DNA associados à exposição carcinogênica, auxiliando na avaliação de riscos ambientais e no estudo da tumorigênese causada pela poluição.
Protocolo
1. Colheita de células
- Semeie cerca de 5 x 105 células HEK 293T em cada uma das quatro placas de 6 cm, cada uma contendo 4 mL de meio DMEM completo.
- Após 24 h, trate uma placa com 2 μL de solução estoque de afidicolina 1 mM (consulte a Tabela de Materiais) (concentração final de 0,5 μM) e outra placa com 20 μL de solução estoque de hidroxiureia 1 M (consulte a Tabela de Materiais) (concentração final de 5 mM) para induzir o estresse de replicação. Adicione DMSO aos dois pratos restantes para servir como controles.
- Após 24 h de tratamento, descarte os meios de cultura. Uma placa de 6 cm normalmente produz aproximadamente 2 x 106 células a 60% -70% de confluência.
- Lave cada prato 2x com 5 mL de 1x PBS gelado. Use raspadores de células para separar as células e transferir a suspensão celular para quatro tubos individuais de 1,5 mL. Pipete suavemente para cima e para baixo com uma pipeta P1000 para dissociar quaisquer aglomerados de células.
- Centrifugar as células a 500 x g durante 5 min a 4 °C e, em seguida, rejeitar o sobrenadante. Coloque as células no gelo.
2. Fracionamento subcelular
- Ressuspenda o pellet celular em 500 μL de tampão A frio recém-preparado (consulte a Tabela 1), garantindo a dissociação completa dos aglomerados de células por pipetagem suave.
- Incube os lisados no gelo por 5-10 min. Verifique a progressão da lise ao microscópio para garantir a lise celular completa.
- Pegue uma pequena alíquota do lisado (cerca de 5 - 10 μL) e coloque-a em uma lâmina de microscópio limpa. Cubra-o com uma lamínula para evitar contaminação.
- Use um microscópio óptico com uma ampliação apropriada (por exemplo, 20x - 40x) para visualizar células ou detritos. Compare com uma amostra de controle não lisada para diferenciar entre células intactas e material lisado.
NOTA: Uma amostra devidamente lisada não terá contornos celulares distintos, apenas cromatina difusa ou material celular. Ajuste o foco para observar claramente o lisado. Se necessário, aplique força mecânica durante a etapa de lise, como usar um homogeneizador Dounce, ao trabalhar com certos tipos de células.
- Centrifugar a 500 x g durante 5 min a 4 °C, uma vez que as células estejam completamente lisadas. Descarte cuidadosamente o sobrenadante. Ressuspenda o pellet de núcleos em 500 μL de tampão A frio usando pontas de pipeta de orifício largo.
NOTA: As pontas de orifício largo ajudam a minimizar as forças de cisalhamento e protegem amostras delicadas como a cromatina. Faça pontas largas cortando a extremidade das pontas padrão com uma lâmina afiada. - Centrifugue a 500 x g durante 5 min a 4 °C. Descarte cuidadosamente o sobrenadante.
- Pré-aqueça uma incubadora a 37 °C e prepare um tampão de parada (100 mM EDTA, pH 8,0; Tabela 1).
- Otimize as concentrações de nuclease microcócica (MNase, consulte a Tabela de materiais) e os tempos de incubação com antecedência.
- Divida 40 μL de amostra de cromatina de teste em várias alíquotas iguais para testar diferentes concentrações de MNase e tempos de incubação.
- Use uma variedade de concentrações de MNase (por exemplo, 0,0625 U, 0,125 U, 0,25 U, 0,5 U, 1 U, 2 U, 4 U, 8 U por reação) e teste vários tempos de incubação (por exemplo, 2, 5, 10 e 15 min).
- Adicione o tampão MNase (consulte a Tabela 1) contendo várias concentrações de MNase às alíquotas da cromatina e incube as amostras a 37 ° C pelos tempos especificados.
- Termine a reação adicionando 1/4 do volume de tampão de parada (concentração final: 20 mM EDTA) imediatamente após o tempo de incubação desejado.
- Isole o ADN das amostras de cromatina digeridas utilizando um método de extracção por fenol/clorofórmio/álcool isoamílico.
- Execute o DNA extraído em um gel de agarose a 1,5% para visualizar os padrões de digestão: A subdigestão mostrará bandas de alto peso molecular (Figura 2, pista 1-4); a digestão excessiva resultará em um esfregaço ou fragmentos muito curtos (Figura 2, pista 6-8), e a digestão ideal produzirá um padrão claro de escada nucleossômica (Figura 2, pista 5, por exemplo, mono, di, trinucleossomos).
- Identifique as condições que produzem a resolução nucleossômica desejada sem digestão excessiva.
NOTA: CaCl2 atua como um cofator para a atividade da MNase. Otimize a digestão ajustando a concentração de CaCl2 entre 1 mM e 5 mM.
- Ressuspenda suavemente os núcleos intactos com 100 μL de tampão MNase pipetando 5 a 10 vezes com pontas de orifício largo. Adicione imediatamente a quantidade pré-determinada de MNase às amostras (1,25 U MNase/100 μL de tampão MNase).
NOTA: Ao trabalhar com várias amostras, digerir cada uma individualmente para evitar a digestão excessiva. - Coloque os tubos em um rotador e incube por 5 min a 37 °C. Retorne imediatamente os tubos ao gelo e termine a digestão da MNase adicionando EDTA a uma concentração final de 20 mM e misture por vórtice.
- Adicione 500 μL de tampão B (consulte a Tabela 1) a cada amostra e misture bem pipetando para cima e para baixo 5x - 10x. Solubilize as proteínas incubando no gelo por 5 min.
NOTA: O sal e o detergente no tampão B ajudam a dissociar as proteínas fracas ligadas à cromatina e expor os epítopos para imunoprecipitação. - Granular o material insolúvel centrifugando à velocidade máxima durante 5 min a 4 °C. Transfira o sobrenadante transparente para novos tubos de 1,5 mL rotulados como a fração nativa da cromatina. As amostras podem ser armazenadas a -80 °C ou usadas para validar a eficiência da fragmentação da cromatina.
NOTA: Evite ciclos frequentes de congelamento e descongelamento, pois eles podem interromper as interações proteína-DNA de interesse. Minimize os ciclos de congelamento e descongelamento sempre que possível.
3. Verificação da fragmentação da cromatina
- Alíquota de 10 μL do sobrenadante de cada amostra em um novo tubo de 1,5 mL. Misture com 20 μL de água destilada e 30 μL de fenol/clorofórmio/álcool isoamílico (25:24:1).
- Feche bem os tubos e vortex vigorosamente por 15-30 s. Centrifugue a 20.000 x g (ou a velocidade máxima da centrífuga) por 10 min a 4 °C. Após a centrifugação, três camadas distintas serão observadas: uma camada superior clara, uma camada intermediária branca e uma camada inferior amarela.
- Transferir cuidadosamente 20 μL da fase aquosa superior (contendo ADN) para um tubo novo. Separe o DNA purificado em gel de agarose a 1,5% por 30 min a 100 V e visualize os padrões de digestão. Certifique-se de que o tamanho dos fragmentos de cromatina esteja principalmente entre 200 e 1000 pares de bases.
NOTA: Os tamanhos adequados dos fragmentos de cromatina são cruciais para o sucesso do ChIP nativo e dependem das condições de tratamento da MNase, incluindo unidades enzimáticas, tempo de incubação e concentração de CaCl2. A eficiência da digestão da MNase também pode variar de acordo com o tipo e o número de células. O padrão de fragmentação da cromatina mostrado na Figura 2 (pista 5) é recomendado para este ensaio ChIP.
4. Imunoprecipitação
- Alíquota de 20 μL de cromatina digerida de cada amostra em um tubo fresco de 1,5 mL e misture com 180 μL de tampão de eluição (consulte a Tabela 1). Rotule esses tubos como amostras de entrada e armazene a -20 °C.
- Transfira 400 μL de amostra de cromatina para outro tubo de 1,5 mL para ChIP.
- Adicione o anticorpo γH2A.X (consulte a Tabela de Material) a uma amostra tratada com DMSO, uma amostra tratada com afidicolina e uma amostra tratada com hidroxiureia. Adicione a mesma quantidade de IgG normal (consulte a Tabela de Materiais) a outra amostra tratada com DMSO como um controle negativo para o ensaio ChIP.
NOTA: Aqui, 1 μg de anticorpo primário é normalmente usado para 400 μL de cromatina (ou seja, a concentração final do anticorpo é de 2,5 μg/mL). No entanto, a quantidade ideal deve ser determinada empiricamente para diferentes anticorpos γH2A.X. - Colocar os tubos ChIP num rotador a 4 °C e incubar durante pelo menos 5 h, ou de preferência durante a noite.
- Enquanto isso, alíquota de 100 μL de grânulos magnéticos de proteína A/G de grau ChIP (consulte a Tabela de Material) em um novo tubo de 1,5 mL. Use pontas de orifício largo e pipete lentamente para garantir a medição precisa dos grânulos. Coloque o tubo em um suporte magnético por pelo menos 1 min e, em seguida, descarte cuidadosamente o líquido.
- Ressuspenda os grânulos em 1 mL de 1x PBS contendo 0,5% de BSA. Girar a 4 °C durante cerca de 4 h. Coloque o tubo em um suporte magnético por pelo menos 1 min e descarte o sobrenadante.
- Lave as esferas novamente com 1 mL de 1x PBS contendo 0.5% de BSA. Coloque o tubo no suporte magnético por 1 min para pellet as esferas magnéticas e, em seguida, descarte o sobrenadante.
NOTA: As etapas 4.5 a 4.7 são o pré-revestimento de grânulos para reduzir a ligação não específica de anticorpos aos grânulos magnéticos. - Ressuspenda os grânulos pré-revestidos em 100 μL de tampão B usando pontas de orifício largo. Adicione 25 μL da suspensão de esferas magnéticas pré-revestidas a cada tubo de amostra ChIP. Girar a 4 °C durante 2 h.
- Coloque os tubos ChIP no suporte magnético e espere até que as esferas estejam completamente presas à lateral do tubo e a solução fique transparente.
- Descarte o sobrenadante transparente sem perturbar as esferas magnéticas. Ressuspenda os grânulos com 1 mL de tampão de lavagem (consulte a Tabela 1) e gire a 4 ° C por 10 min.
- Coloque os tubos de volta no suporte magnético e espere até que a solução fique clara. Descarte o tampão de lavagem. Repita a lavagem para um total de quatro lavagens.
- Rejeitar o tampão de lavagem após a lavagem final e centrifugar brevemente os tubos a 400 x g durante 30 s a 4 °C para centrifugar qualquer líquido residual. Coloque os tubos de volta no suporte magnético e remova cuidadosamente qualquer líquido restante do fundo do tubo.
5. Eluição e precipitação de DNA
NOTA: A eficiência do anticorpo pode variar entre os diferentes lotes. É importante confirmar a afinidade de ligação de um novo anticorpo verificando as amostras imunoprecipitadas por meio da análise de Western blot.
- Verifique a eficiência de pulldown do anticorpo ChIP usando Western blot (WB) conforme descrito abaixo.
- Pegue uma pequena alíquota da amostra ChIP para análise (ou seja, geralmente 10% da amostra ChIP). Inclua cromatina de entrada (pré-imunoprecipitação) e controle negativo (por exemplo, IgG pull-down) para comparação.
- Eluir proteínas dos grânulos ligados a anticorpos aquecendo em 20 μL de tampão de carregamento 1x SDS-PAGE (consulte a Tabela de Material) a 95 ° C por 5 min.
- Carregue as amostras IP, entrada e controles em um gel SDS-PAGE de 15%. Execute o gel.
- Transfira as proteínas para uma membrana de nitrocelulose de 0,2 μm (consulte a Tabela de Materiais) ou PVDF usando um sistema de transferência úmido ou semi-seco.
- Bloqueie a membrana com 5% de leite desnatado ou BSA em TBST (consulte a Tabela 1) por 1 h em temperatura ambiente para evitar ligação inespecífica.
- Incubar a membrana com o anticorpo primário contra o γH2A.X (consulte a Tabela de Materiais) diluído em tampão de bloqueio por 1-2 h à temperatura ambiente ou durante a noite a 4 °C.
- Lave a membrana 3x com TBST para remover anticorpos não ligados. Incubar a membrana com um anticorpo secundário conjugado com HRP (consulte a Tabela de Materiais) por 1 h à temperatura ambiente. Lave a membrana novamente para remover o excesso de anticorpos secundários.
- Desenvolva a membrana usando um substrato quimioluminescente e visualize o sinal com um gerador de imagens. Compare a intensidade do sinal entre as pistas IP, de entrada e de controle para avaliar a eficiência e a especificidade do pull-down.
NOTA: Uma banda correspondente à proteína-alvo na faixa IP confirma o pull-down bem-sucedido do anticorpo. Essa abordagem garante que você possa avaliar a eficácia do anticorpo na captura da proteína-alvo durante o experimento ChIP.
- Adicione 50 μL de tampão de eluição (consulte a Tabela 1) a cada uma das amostras ChIP restantes. Coloque os tubos em um termomixer e agite por 15 min em temperatura ambiente.
- Coloque os tubos no suporte magnético por pelo menos 1 min. Colete o eluidor em novos tubos. Repita 1x e colete o eluidor nos mesmos tubos.
- Adicione 100 μL adicionais de tampão de eluição a cada amostra de eluição ChIP e 180 μL de tampão de eluição a cada amostra de entrada.
- Adicione 200 μL de fenol/clorofórmio/álcool isoamílico (25:24:1) a cada amostra e vortex vigorosamente. Centrifugue as amostras a 20.000 x g (ou velocidade máxima) por 10 min a 4 °C.
- Adicione 19 μL de acetato de sódio 3M (NaOAc, pH 5,2; consulte a Tabela 1) e 2 μL de solução de glicogênio (20 mg/mL, consulte a Tabela de Material) a cada novo tubo de centrífuga de 1,5 mL.
- Após centrifugação, transferir cuidadosamente a camada aquosa superior (cerca de 190 μl) para os tubos que contêm NaOAc e glicogénio e misturar por vórtice.
- Adicione 500 μL de etanol 100% e vórtice. Precipitar o ADN incubando as amostras a -20 °C durante, pelo menos, 2 h ou durante a noite.
- Centrifugue os tubos a 20.000 x g (ou velocidade máxima) por 10 min a 4 °C. Descarte o sobrenadante, tomando cuidado para não perturbar o pellet branco. Ressuspenda o pellet em 1 mL de etanol a 70% e vortex completamente.
- Centrifugue os tubos a 20.000 x g (ou velocidade máxima) por 5 min a 4 °C. Remova cuidadosamente o sobrenadante. Centrifugue brevemente os tubos novamente para girar qualquer etanol residual. Remova cuidadosamente o etanol usando uma pipeta P20. Seque ao ar os pellets de DNA por 2-3 min.
NOTA: Evite secar demais o pellet, pois isso pode dificultar a redissolução do DNA. - Para amostras de ChIP, ressuspenda o DNA em 400 μL de tampão TE (consulte a Tabela 1). Para DNA de entrada, ressuspenda em 1000 μL de tampão TE. As amostras eluídas agora podem ser armazenadas a -20°C.
6. Quantificação de qPCR
- Realize qPCR usando um kit comercial (consulte a Tabela de Material) com triplicados técnicos para cada amostra. Confirme a presença de um único produto específico de PCR realizando uma análise da curva de fusão para garantir a especificidade da amplificação36.
- Análise de dados
NOTA: Na análise de quantificação relativa, a amostra de teste é expressa como uma mudança de dobra em relação a uma amostra de controle (imunoprecipitada usando IgG purificada normal ou IP simulado). Os loci de DNA conhecidos por não serem ocupados pela proteína imunoprecipitada (locus negativo) podem ser usados dessa maneira como um gene de referência em comparação com loci de DNA de controle positivo conhecidoe ocupado 36.- Calcule a porcentagem de entrada para cada ChIP usando a fórmula abaixo
%Entrada = 2(-ΔCt [ChIP normalizado]) - Normalize os valores de ΔCt do locus positivo para locus negativo (ΔΔCt) subtraindo o valor de ΔCt obtido para o locus positivo do valor de ΔCt para o locus negativo usando a fórmula abaixo
(ΔΔCt = ΔCtpositivo - ΔCtnegativo) - Calcule o enriquecimento de dobras da sequência do locus positivo no DNA do ChIP sobre o locus negativo usando a fórmula abaixo
Enriquecimento de dobras =2ΔΔCt
As sequências dos primers de qPCR usados para análise são fornecidas na Tabela 2. A organização genômica de FRA3B e FRA16D37é representada na Figura 3A,B.
- Calcule a porcentagem de entrada para cada ChIP usando a fórmula abaixo
- Análise estatística
- Analise os resultados estatisticamente usando o teste t pareado de Student. Um valor de p de ≤0,05 é considerado estatisticamente significativo, indicando que as diferenças observadas provavelmente não são devidas à variação aleatória38.
Resultados
O tamanho dos fragmentos de cromatina é crucial para o sucesso do Native ChIP, pois afeta diretamente a acessibilidade das regiões de DNA para ligação de anticorpos. Para determinar a concentração ideal de MNase para fragmentação da cromatina, preparamos uma série de tubos de microcentrífuga contendo concentrações variadas de MNase (ou seja, 0,0625 U, 0,125 U, 0,25 U, 0,5 U, 1 U, 2 U, 4 U, 8 U por reação) e 40 μL de núcleos isolados. Cada reação foi incubada a 37 ° C por 5 min para atingir uma variedade de tamanhos de fragmentos de cromatina. Os resultados da digestão da MNase são apresentados na Figura 2.
Conforme mostrado na Figura 2, concentrações mais altas de MNase levaram a uma digestão mais extensa da cromatina, resultando em uma predominância de fragmentos mononucleossômicos (Figura 2, pistas 1, 2 e 3). Em contraste, em concentrações mais baixas de MNase, a maioria dos fragmentos de cromatina era maior, muitas vezes excedendo 1 kb (Figura 2, pistas 6, 7 e 8), indicando digestão insuficiente para aplicações a jusante. Com uma concentração de MNase de 1,6 unidades e um tempo de digestão de 5 min, obtivemos fragmentos de cromatina principalmente na faixa de 200 a 1000 pares de bases (Figura 2, pista 5). Esse tamanho de fragmento é ideal para ChIP nativo, pois garante o enriquecimento eficiente do DNA associado à cromatina, mantendo a acessibilidade a epítopos específicos. O tamanho apropriado do fragmento facilita a imunoprecipitação eficiente e a análise a jusante, como PCR quantitativo ou sequenciamento, tornando-o adequado para detectar interações proteína-DNA com alta resolução.
A afinidade de ligação do anticorpo é um fator crítico para o sucesso do ChIP nativo. Garantir que o anticorpo se ligue de forma eficiente e específica ao seu epítopo alvo é essencial para obter resultados confiáveis. Portanto, é crucial verificar antecipadamente a eficiência de ligação do anticorpo. Para confirmar a eficiência da imunoprecipitação γH2A.X, realizamos um ensaio de western blot (WB).
Comparamos os níveis de γH2A.X entre células tratadas com DMSO (controle), células tratadas com afidicolina e tratadas com hidroxiureia (estressadas por replicação) usando western blotting. Conforme mostrado na Figura 4A (Entrada), os níveis de γH2A.X foram significativamente mais altos nas células tratadas com afidicolina em comparação com as células tratadas com DMSO. Este aumento em γH2A.X é consistente com a indução de estresse de replicação pela afidicolina, o que leva ao aumento da formação de quebra de fita dupla e subsequente fosforilação de γH2A.X. Após a realização do ChIP, verificamos a especificidade da imunoprecipitação comparando o enriquecimento de γH2A.X nas amostras de IgG controle e γH2A.X IP. O controle de IgG não puxou para baixo nenhum γH2A.X detectável, indicando ligação mínima não específica ( Figura 4B , N-ChIP e X-ChIP ). Em contraste, a amostra γH2A.X IP enriqueceu com sucesso γH2A.X das células tratadas com afidicolina, demonstrando ligação eficiente e específica do anticorpo a γH2A.X (Figura 4, N-ChIP e X-ChIP). Esses resultados confirmam que o anticorpo γH2A.X tem afinidade de ligação e especificidade suficientes para uso em experimentos ChIP nativos, permitindo a detecção confiável e o enriquecimento de regiões de cromatina ligadas a γH2A.X. Esta etapa de validação é essencial para garantir a qualidade e a precisão das análises subsequentes de ChIP.
Usamos os locais frágeis comuns (CFSs) FRA16D e FRA3B para avaliar a especificidade da ligação de γH2A.X ao DNA em resposta ao estresse de replicação. FRA16D e FRA3B são regiões CFS bem conhecidas propensas a instabilidade sob condições de estresse de replicação, tornando-as candidatas ideais para validar interações γH2A.X/DNA. A Figura 3A, B ilustra a organização genômica desses dois CFSs, juntamente com os primers específicos de qPCR usados para análise.
Para determinar se o γH2A.X se associa a essas regiões do CFS em resposta ao estresse de replicação, realizamos a análise de ChIP-qPCR em células de controle (tratadas com DMSO), células tratadas com afidicolina e células tratadas com hidroxiureias. Os resultados do qPCR mostraram que nas células de controle tratadas com DMSO, γH2A.X não foi enriquecido em loci FRA16D ou FRA3B, sugerindo uma falta de dano significativo ao DNA ou estresse de replicação nessas regiões ( Figura 3C ). No entanto, em células tratadas com afidicolina ou hidroxiureia, que induz estresse de replicação pela inibição da DNA polimerase, observamos enriquecimento significativo de γH2A.X em FRA16D e FRA3B ( Figura 3C ). Isso indica que γH2A.X foi recrutado para essas regiões CFS especificamente em resposta ao estresse de replicação do DNA. Esses achados demonstram que a ocupação de γH2A.X em CFSs é desencadeada pelo estresse de replicação, apoiando seu papel como um marcador de resposta a danos no DNA e travamento do garfo de replicação. O recrutamento específico de γH2A.X para FRA16D e FRA3B durante o tratamento com afidicolina valida ainda mais a especificidade de nosso ensaio ChIP e confirma que γH2A.X é um indicador confiável de danos ao DNA e estresse de replicação em locais frágeis.
Para comparar a eficiência do ChIP nativo com o ChIP reticulado (X-ChIP), realizamos γH2A.X X-ChIP usando o mesmo número inicial de células, seguindo o protocolo descrito por Lyu et al.39. O recrutamento de γH2A.X para FRA16D foi aumentado pelo tratamento com afidicolina e hidroxiureia, conforme mostrado na Figura 3D. No entanto, o enriquecimento relativo de γH2A.X em FRA16D em X-ChIP foi visivelmente menor do que o observado com ChIP nativo, indicando uma eficiência reduzida de X-ChIP nessas condições. Além disso, no FRA3B, o aumento no enriquecimento de γH2A.X foi estatisticamente insignificante, fornecendo evidências adicionais de que o X-ChIP é menos eficiente quando se trabalha com um número limitado de células. Esses resultados sugerem que o ChIP nativo pode ser mais adequado para detectar o enriquecimento de γH2A.X em locais frágeis quando o tamanho da amostra é restrito. Analise os resultados estatisticamente usando o teste t pareado de Student. Um valor de p de ≤ 0,05 é considerado estatisticamente significativo.

Figura 1: Workflow para realizar um ensaio γH2A.X ChIP nativo. (i) As células são cultivadas e submetidas a tratamentos específicos (por exemplo, agentes prejudiciais ao DNA como hidroxiureia) para induzir a sinalização γH2A.X. (ii) As células tratadas são coletadas para processamento. (iii) As células são fracionadas para isolar o γH2A.X ligado à cromatina, separando-o de outros componentes celulares. (iv) A digestão da nuclease microcócica (MNase) é realizada para fragmentar a cromatina em pedaços do tamanho de nucleossomos. (v) O tamanho dos fragmentos de cromatina é validado executando uma amostra em um gel de agarose, garantindo que os fragmentos tenham o comprimento apropriado para ChIP. (vi) Um anticorpo específico para γH2A.X é usado para imunoprecipitar fragmentos de cromatina marcados por γH2A.X. (vii) As esferas de proteína A / G são usadas para puxar para baixo os complexos anticorpo-cromatina, enriquecendo o DNA ligado a γH2A.X. (viii) Os fragmentos de DNA marcados com γH2A.X são eluídos e purificados, preparando-os para análises a jusante. (ix) Análise Downstream: qPCR e sequenciamento e Análise de Dados. Clique aqui para ver uma versão maior desta figura.

Figura 2: Ensaio para otimizar a condição de digestão da MNase. Fragmentos de cromatina foram preparados a partir de células HEK 293T usando diferentes condições de digestão de MNase. O DNA purificado resultante da cromatina fragmentada foi separado em um gel de agarose a 2% e executado a 100 V por 30 min. As pistas 1 a 8 representam amostras tratadas com concentrações crescentes de MNase (ou seja, 0,0625 U, 0,125 U, 0,25 U, 0,5 U, 1 U, 2 U, 4 U, 8 U por reação), demonstrando um gradiente de digestão da cromatina. A escada de DNA (indicada por M) foi incluída para referência de tamanho. Os fragmentos de DNA foram visualizados usando a solução de coloração de DNA Midori Green Advance. A banda marcada corresponde a fragmentos de DNA do tamanho de um mononucleossomo, destacando a eficiência da digestão em cada concentração de MNase. Clique aqui para ver uma versão maior desta figura.
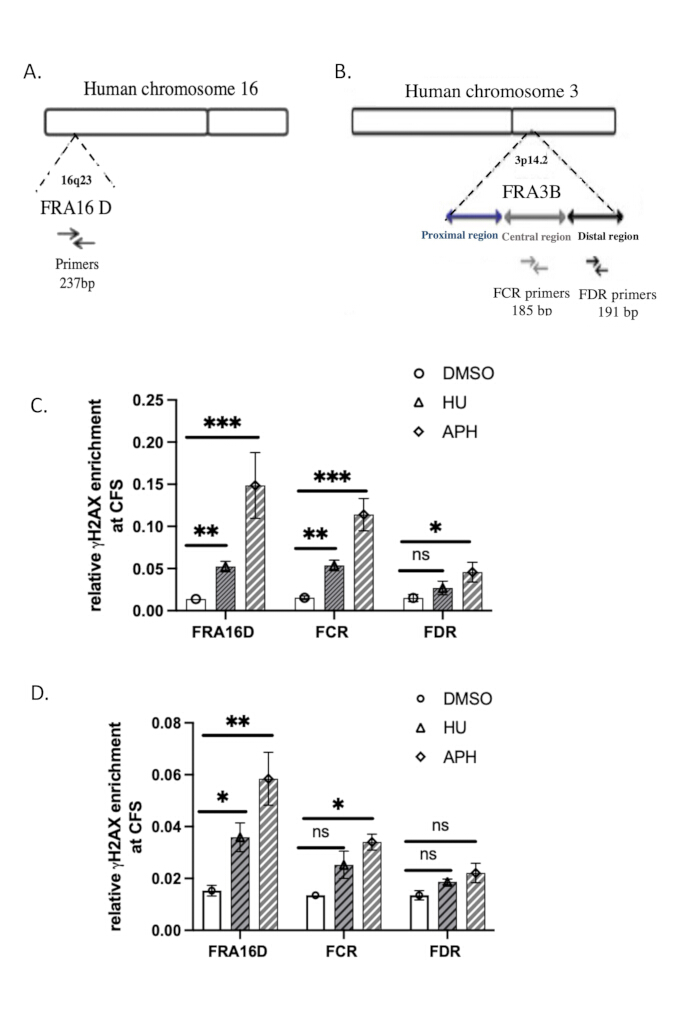
Figura 3: Verificação qPCR de γH2A.X ChIP. (A) Diagrama das organizações genômicas do FRA16D. (B) Diagrama de organizações genômicas da região FRA3B. São indicados conjuntos de primers usados para análises de qPCR das regiões FRA16D, distal (FDR) e central (FCR) dentro do locus FRA3B. (C) O enriquecimento relativo de γH2A.X em locais frágeis comuns (CFS), especificamente FRA3B e FRA16D, foi avaliado usando ChIP nativo seguido de análise de qPCR após tratamento com afidicolina e hidroxiureia. (D) O enriquecimento relativo de γH2A.X em locais frágeis comuns (CFS), especificamente FRA3B e FRA16D, foi avaliado usando X-ChIP seguido de análise de qPCR após tratamento com afidicolina e hidroxiureia. indica resultado estatisticamente significativo com p < 0,001; ** indica p < 0,01 e * indica p < 0,05, determinado por um teste t; n = 3. Clique aqui para ver uma versão maior desta figura.
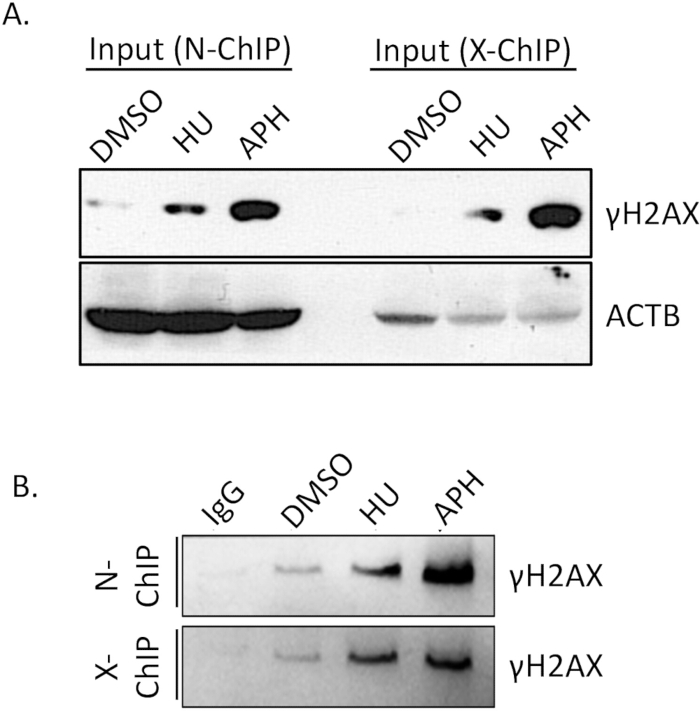
Figura 4: Verificação da eficiência de pull-down do anticorpo ChIP usando Western blot. Da amostra total, 10% da amostra ChIP, juntamente com os controles de entrada, foi separada em um gel SDS-PAGE a 15% para resolver os polipeptídeos. Após a eletroforese, as proteínas separadas foram transferidas para uma membrana de PVDF de 0,22 μm usando um protocolo de transferência padrão. A membrana foi então sondada sequencialmente com um anticorpo primário específico direcionado a γH2A.X e ACTB, seguido por um anticorpo secundário conjugado com HRP apropriado. O sinal foi posteriormente visualizado usando detecção de quimioluminescência, permitindo a avaliação da especificidade do anticorpo e da eficiência de pull-down no ensaio ChIP. (A) níveis de γH2A.X entre células tratadas com DMSO (controle), células tratadas com afidicolina e tratadas com hidroxiureia (estressadas por replicação) com ACTB como controle de carga. (B) enriquecimento γH2A.X nas amostras de controle e IP de IgG. Abreviaturas: APH = afidicalina, HU = hidroxiureia. Clique aqui para ver uma versão maior desta figura.
| Tampão A (1X) | |
| Reagente | Concentração de trabalho |
| TUBOS pH 6,8 | 10 mM |
| NaCl | 100 mM |
| MgCl2 | 3 mM |
| EGTA pH 7,6 | 1 mM |
| Armazenar a -20 °C por até 1 ano | |
| Tampão MNase (1X) | |
| Reagente | Concentração de trabalho |
| Tris-HCl, pH 7,5 | 50 mM |
| CaCl2 | 1 mM |
| MgCl2 | 4 mM |
| Armazenar a -20 °C por até 1 ano | |
| Tampão B (1X) | |
| Reagente | Concentração de trabalho |
| Fosfato de sódio pH 7. 0 | 20 mM |
| Pirofosfato de sódio10. H2O | 30 mM |
| Kcl | 250 mM |
| EDTA, pH 8,0 | 5 mM |
| Glicerol | 10% |
| Tritão X-100 | 0.10% |
| Coquetel de inibidores de protease | 1x |
| Coquetel de inibidor de fosfatase | 1x |
| TDT | 0,5 mM |
| Armazenar a -20 °C por até 1 ano | |
| Tampão de eluição | |
| Reagente | Concentração de trabalho |
| NaHCO3 | 50 mM |
| SDS | 1% |
| Preparado | |
| Tampão de lavagem | |
| Reagente | Concentração de trabalho |
| Tampão MNase | x 0,5 |
| Buffer B | x 0,5 |
| Preparado na hora | |
| Parar buffer | |
| EDTA | 100 mM, pH8.0 |
| 3 M Acetato de sódio, pH 5,2 | |
| 408.24 g Acetato de sódio | |
| ajusta o pH com ácido acético a pH 5,2 | |
| ajustando o volume para 1 L | |
| Esterilize o tampão por filtração ou autoclavagem | |
| Tampão TBST | |
| Base Tris, pH 8,0 | 10 mM |
| NaCl | 150 mM |
| Pré-adolescente 20 | 0,1% (v/v) |
| Buffer TE | |
| Tris-HCl, pH 8,0 | 10 mM |
| EDTA, pH 8,0 | 1mM |
Tabela 1: Composição do tampão.
| Loci gênicos | Primer direto | Primer reverso |
| FRA16D | TCCTGTGGAAGGGATATTTA | CCCCTCATATTCTGCTTCTA |
| FRA3B | TGTTGGAATGTTAACTCTATCCCAT | ATATCTCATCAAGACCGCTGCA |
| Referência | ||
| FRA3B | CAATGGCTTAAGCAGACATGGT | AGTGAATGGCATGGCTGGAATG |
| FDR | ||
| ACTB (controlo negativo) | GACGCAGGATGGCATGGG | ACGCCTCTGGCCGTACCAC |
Tabela 2: Sequência do primer.
| Baixa recuperação de DNA | Problema potencial | Solução possível |
| Má qualidade do anticorpo | Use anticorpo de grau ChIP. | |
| Anticorpo insuficiente | Use 1-10 μg de anticorpo ChIP por 25 μg de cromatina. | |
| Tempo de incubação insuficiente | Execute a etapa de imunoprecipitação durante a noite a 4 °C. | |
| Lavagens excessivamente rigorosas | Não use uma concentração de NaCl superior a 500 mM no tampão de lavagem. | |
| Contas de baixa qualidade | Certifique-se de que a proteína A ou G seja compatível com o anticorpo ChIP. | |
| Siga a ficha técnica do produto do grânulo para obter o volume ideal de grânulos para a proporção de anticorpos. | ||
| Arranque insuficiente | Prepare uma placa separada de células para determinar com precisão o número de células. | |
| Amostra | Aumente o número de células usadas se o alvo for de baixa abundância. | |
| Lise celular inadequada | Otimize a composição do buffer e o tempo de lise para melhorar a eficiência. | |
| Aplique força mecânica durante a etapa de lise, como usar um homogeneizador Dounce, ao trabalhar com certos tipos de células. | ||
| Ocorreu degradação dos núcleos | Certifique-se de que o isolamento dos núcleos seja suave para preservar a integridade e a acessibilidade da cromatina. | |
| Use pontas de pipeta de diâmetro largo para minimizar o cisalhamento dos núcleos durante o manuseio. | ||
| Ocorreu degradação da amostra | Execute todas as etapas no gelo ou a 4 °C. | |
| Inclua inibidores de protease em todos os tampões e certifique-se de que todos os tampões estejam recém-preparados. | ||
| Digestão insuficiente de MNase | Titular a concentração de MNase (por exemplo, 0,1–5 unidades) para determinar a quantidade ideal para um tipo de amostra específico; e comece com uma gama de concentrações para identificar as condições ideais de digestão. | |
| MNase digestão excessiva | Experimente diferentes tempos de incubação (por exemplo, 1 a 10 min) a 37 ° C e monitore o padrão de fragmentação para garantir uma digestão suficiente sem digestão excessiva. | |
| Fosforilação interrompida | Adicione o coquetel inibidor de fosfatase ao tampão de lise e certifique-se de que todos os tampões estejam preparados na hora. | |
| Elevado background em controlo negativo | Ligação inespecífica a contas | Inclua uma etapa de pré-limpeza antes da etapa de imunoprecipitação e use esferas magnéticas de grau ChIP, que geralmente exibem ligação não específica reduzida. |
| Lavagem insuficiente | Aumente o número ou rigor das lavagens ajustando a concentração de sal e detergente. | |
| Fragmentação insuficiente da cromatina | Otimize a fragmentação para obter fragmentos de 200-750 pb. | |
| A otimização é necessária para cada tipo de célula ou tecido | ||
| Solução de problemas de PCR | Sem amplificação da amostra de entrada | (1) Excessivamente fragmentado: o sinal é diminuído para amplicons de mais de 150 pb se a cromatina for fragmentada até o comprimento do mononucleossomo. |
| (2) Otimize as condições de PCR. | ||
| (3) Projete primers para amplificar uma região menor (<150 pb). | ||
| Baixa resolução | Tamanho do fragmento de DNA muito grande | Otimize a fragmentação para obter fragmentos de 200-750 pb. |
Tabela 3: Solução de problemas de imunoprecipitação da cromatina.
Discussão
A poluição ambiental é um contribuinte significativo para o câncer humano. Muitos poluentes são cancerígenos, ou seja, podem causar danos genéticos que levam ao desenvolvimento de câncer40,41. No entanto, determinar se uma determinada substância é tumorigênica é uma tarefa desafiadora. Um método rápido, confiável e econômico para identificar o potencial cancerígeno capacitaria os cientistas a rastrear com eficiência os poluentes ambientais e avaliar seu impacto na estabilidade genômica. Neste estudo, nos concentramos em uma variante de histona modificada, γH2A.X, que é amplamente reconhecida como um marcador altamente específico para DSBs. γH2A.X é formado quando a variante de histona H2A. X é fosforilada na serina 139 em resposta a danos no DNA.
Ao detectar a presença de γH2A.X por ChIP nativo em CFSs, podemos avaliar efetivamente a extensão dos danos ao DNA causados por contaminantes ambientais42. A capacidade de medir quantitativamente γH2A.X em CFSs fornece uma maneira rápida, sensível e confiável de avaliar o potencial de poluentes ambientais para induzir efeitos tumorigênicos. Em contraste com os ensaios tradicionais de genotoxicidade, que geralmente medem marcadores indiretos de danos ao DNA (como mutações, formação de micronúcleos ou transformação celular), o γH2AX ChIP nativo permite o mapeamento preciso de eventos de quebra em loci genômicos específicos42. Isso proporciona uma compreensão mais profunda da região mais vulnerável aos poluentes ambientais e auxilia na identificação de potenciais mecanismos cancerígenos.
O ChIP nativo bem-sucedido requer atenção cuidadosa a várias etapas críticas para garantir o enriquecimento efetivo das regiões-alvo. Abaixo estão as etapas críticas envolvidas no ChIP nativo de γH2AX. Preparação celular: Comece com um número adequado de células. Trate as células com um agente indutor de danos ao DNA, como a afidicolina, para induzir o estresse de replicação como um controle positivo. Incluir amostras de controlo negativo não tratadas adequadas para comparação. Isolamento da cromatina: Realize o fracionamento subcelular para isolar proteínas ligadas à cromatina, minimizando a contaminação de componentes citoplasmáticos. Otimização de MNase: Realize experimentos piloto para otimizar a concentração de nuclease microcócica (MNase) e o tempo de incubação. O objetivo é obter fragmentos de cromatina entre 200-1000 pares de bases, garantindo boa resolução para ChIP, mantendo a integridade do nucleossomo. Imunoprecipitação: Utilize um anticorpo γH2AX de grau ChIP de alta qualidade junto com a agarose da proteína A/G para garantir a redução eficiente da cromatina alvo, minimizando a ligação não específica. Lavagem: Lave as esferas com um tampão contendo a concentração apropriada de sal e detergente para remover interações não específicas, mantendo a cromatina ligada ao γH2AX. Eluição e recuperação de DNA: Recupere com eficiência a cromatina ligada por eluição e precipitação completas de DNA, garantindo perda mínima da cromatina alvo enriquecida. Em comparação com o X-ChIP convencional, existem quatro vantagens principais do γH2AX ChIP nativo. Em primeiro lugar, a integração do fracionamento subcelular reduz as interações falso-positivas e aumenta a especificidade do ChIP. Em segundo lugar, os complexos proteína-DNA são preservados em seu estado nativo, minimizando a estabilização das interações transitórias γH2AX-cromatina que podem ocorrer com agentes de reticulação. Em terceiro lugar, a omissão da fixação e as demoradas etapas de reticulação reversa simplificam o fluxo de trabalho geral. Por fim, menos material de partida é necessário e nenhum equipamento caro é necessário para o cisalhamento da cromatina, como um sonicador em banho-maria (por exemplo, Bioruptor) ou sonicador ultrassônico focalizado (por exemplo, Covaris).
Embora o ChIP nativo tenha suas vantagens, suas limitações também devem ser reconhecidas. Uma limitação é o potencial de rearranjo proteico durante a preparação da cromatina e imunoprecipitação. Esse rearranjo poderia, teoricamente, alterar as interações das proteínas ligadas à cromatina e introduzir variabilidade nos resultados. Embora não tenhamos evidências diretas para apoiar a ocorrência de tais rearranjos, é importante incluir controles experimentais apropriados para explicar esse artefato potencial. Outra limitação do ensaio ChIP nativo é o viés de clivagem da MNase, que pode levar a resultados imprecisos. A MNase cliva preferencialmente regiões ricas em A-T do genoma, tornando essas regiões mais propensas a serem fragmentadas e enriquecidas durante o processo ChIP. Esse viés pode causar super-representação de sequências ricas em A-T, e sub-representação de regiões ricas em G-C, potencialmente resultando em sinais falso-negativos para regiões genômicas que são menos acessíveis à digestão da MNase. Se os loci de interesse forem ricos em GC, é necessária uma otimização cuidadosa das condições de digestão da MNase. Alternativamente, a incorporação de controles adicionais, como fragmentação da cromatina por sonicação, pode ajudar a mitigar o impacto do viés da MNase e melhorar a confiabilidade dos resultados.
Apesar de suas certas limitações, o γH2AX ChIP nativo é particularmente útil para identificar os gens loci onde poluentes específicos induzem danos ao DNA. Ao enriquecer fragmentos de DNA ligados a γH2AX, os pesquisadores podem identificar regiões frágeis do genoma, particularmente CFSs por qPCR, que são mais propensas a quebras sob estresse de replicação induzido por poluentes. Essa técnica também permite que os pesquisadores estabeleçam uma relação direta entre a exposição a um carcinógeno específico e a formação de danos ao DNA em regiões genômicas específicas, fornecendo informações sobre o perfil genotóxico dos poluentes e seu papel potencial no início da carcinogênese43,44.
A tabela de solução de problemas (Tabela 3) fornece soluções detalhadas para problemas comuns encontrados durante o ensaio ChIP, como baixo rendimento de DNA, fragmentação insuficiente da cromatina, alto ruído de fundo ou baixo desempenho do anticorpo. Cada problema é combinado com recomendações específicas para garantir a confiabilidade e reprodutibilidade do protocolo.
Usando sistemas automatizados de manuseio de líquidos e tecnologias quantitativas de PCR ou sequenciamento, o γH2AX ChIP nativo pode identificar com eficiência agentes que induzem danos ao DNA, servindo assim como uma ferramenta valiosa em estudos toxicológicos e monitoramento ambiental. Ao empregar γH2AX ChIP nativo em amostras derivadas de populações expostas a diferentes níveis de poluentes ambientais, os pesquisadores podem identificar padrões específicos de danos ao DNA e determinar o nível de risco associado à exposição a agentes específicos. Em conclusão, o γH2AX ChIP nativo oferece vantagens significativas para avaliar o potencial carcinogênico de poluentes ambientais, fornecendo uma abordagem rápida, econômica e de alta especificidade para detectar danos ao DNA em loci genômicos específicos. Suas aplicações futuras em triagem de alto rendimento, avaliação de risco personalizada e desenvolvimento de biomarcadores o tornam uma ferramenta valiosa para avançar nossa compreensão da carcinogênese ambiental e desenvolver estratégias para prevenção e intervenção do câncer.
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Este trabalho foi apoiado pelo financiamento inicial da Universidade do Sul da China.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.2 µm nitrocellulose membrane | Amersham | 10600011 | |
| Actin B | proteintech | 20536-1-AP | |
| Aphidicolin | MedChemExpress | HY-N6733 | |
| ChIP-grade magnetic Protein A/G beads | ThermoFisher | 26162 | |
| Clarity Western ECL Substrate | Bio-Rad | #1705061 | |
| Glycogen, molecular biology grade | ThermoFisher | Cat. No. R0561 | |
| HRP-conjugated secondary antibody | proteintech | SA00001-2 | |
| hydroxyurea | MedChemExpress | HY-B0313 | |
| Micrococcal Nuclease | NEB | M0247S | |
| normal IgG | Santa Cruz | sc-2025 | |
| Taq Universal SYBR Green Supermix | BioRad | 1725120 | |
| γH2A.X antibody (for ChIP) | Sigma-Aldrich | 05-636 | |
| γH2A.X antibody (for WB) | Cell Signaling | #25955 |
Referências
- Glover, T. W., Berger, C., Coyle, J., Echo, B. DNA polymerase alpha inhibition by aphidicolin induces gaps and breaks at common fragile sites in human chromosomes. Hum Genet. 67 (2), 136-142 (1984).
- Bignell, G. R., et al. Signatures of mutation and selection in the cancer genome. Nature. 463 (7283), 893-898 (2010).
- Hellman, A., et al. A role for common fragile site induction in amplification of human oncogenes. Cancer Cell. 1 (1), 89-97 (2002).
- Kotzot, D., et al. Parental origin and mechanisms of formation of cytogenetically recognisable de novo direct and inverted duplications. J Med Genet. 37 (4), 281-286 (2000).
- Miller, C. T., et al. Genomic amplification of MET with boundaries within fragile site FRA7G and upregulation of MET pathways in esophageal adenocarcinoma. Oncogene. 25 (3), 409-418 (2006).
- Zack, T. I., et al. Pan-cancer patterns of somatic copy number alteration. Nat Genet. 45 (10), 1134-1140 (2013).
- Gao, G., et al. Common fragile sites (CFS) and extremely large CFS genes are targets for human papillomavirus integrations and chromosome rearrangements in oropharyngeal squamous cell carcinoma. Genes Chromosomes Cancer. 56 (1), 59-74 (2017).
- Thorland, E. C., Myers, S. L., Gostout, B. S., Smith, D. I. Common fragile sites are preferential targets for HPV16 integrations in cervical tumors. Oncogene. 22 (8), 1225-1377 (2003).
- Matovina, M., Sabol, I., Grubisić, G., Gasperov, N. M., Grce, M. Identification of human papillomavirus type 16 integration sites in high-grade precancerous cervical lesions. Gynecol Oncol. 113 (1), 120-127 (2009).
- Yu, T., et al. The role of viral integration in the development of cervical cancer. Cancer Genet Cytogenet. 158 (1), 27-34 (2005).
- Bignell, G. R., et al. Signatures of mutation and selection in the cancer genome. Nature. 463 (7283), 893-898 (2010).
- Thavathiru, E., Ludes-Meyers, J. H., MacLeod, M. C., Aldaz, C. M. Expression of common chromosomal fragile site genes, WWOX/FRA16D and FHIT/FRA3B is downregulated by exposure to environmental carcinogens, UV, and BPDE but not by IR. Mol Carcinog. 44 (3), 174-182 (2005).
- Sirbu, B. M., et al. Analysis of protein dynamics at active, stalled, and collapsed replication forks. Genes Dev. 25 (12), 1320-1327 (2011).
- Barlow, J. H., et al. Identification of early replicating fragile sites that contribute to genome instability. Cell. 152 (3), 620-632 (2013).
- Petermann, E., Orta, M. L., Issaeva, N., Schultz, N., Helleday, T. Hydroxyurea-stalled replication forks become progressively inactivated and require two different RAD51-mediated pathways for restart and repair. Mol Cell. 37 (4), 492-502 (2010).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. J Biol Chem. 273 (10), 5858-5868 (1998).
- Shanbhag, N. M., Rafalska-Metcalf, I. U., Balane-Bolivar, C., Janicki, S. M., Greenberg, R. A. ATM-dependent chromatin changes silence transcription in cis to DNA double-strand breaks. Cell. 141 (6), 970-981 (2010).
- Stiff, T., et al. ATR-dependent phosphorylation and activation of ATM in response to UV treatment or replication fork stalling. EMBO J. 25 (24), 5775-5782 (2006).
- Nelson, J. D., Denisenko, O., Bomsztyk, K. Fast chromatin immunoprecipitation assay. Nuc Acids Res. 34 (5), e2 (2006).
- Dorbic, T., Wittig, B. Isolation of oligonucleosomes from active chromatin using HMG17-specific monoclonal antibodies. Nuc Acids Res. 14 (8), 3363-3376 (1986).
- Dorbic, T., Wittig, B. Chromatin from transcribed genes contains HMG17 only downstream from the starting point of transcription. EMBO J. 6 (8), 2393-2399 (1987).
- Hebbes, T. R., Thorne, A. W., Crane-Robinson, C. A direct link between core histone acetylation and transcriptionally active chromatin. EMBO J. 7 (5), 1395-1402 (1988).
- Hebbes, T. R., Thorne, A. W., Clayton, A. L., Crane-Robinson, C. Histone acetylation and globin gene switching. Nuc Acids Res. 20 (5), 1017-1022 (1992).
- Hebbes, T. R., Clayton, A. L., Thorne, A. W., Crane-Robinson, C. Core histone hyperacetylation co-maps with generalized DNase I sensitivity in the chicken beta-globin chromosomal domain. EMBO J. 13 (8), 1823-1830 (1994).
- Orlando, V. Mapping chromosomal proteins in vivo by formaldehyde-crosslinked-chromatin immunoprecipitation. Trend Biochem, Sci. 25 (3), 99-104 (2000).
- Myers, F. A., Evans, D. R., Clayton, A. L., Thorne, A. W., Crane-Robinson, C. Targeted and extended acetylation of histones H4 and H3 at active and inactive genes in chicken embryo erythrocytes. J Biol Chem. 276 (23), 20197-20205 (2001).
- Litt, M. D., Simpson, M., Recillas-Targa, F., Prioleau, M. N., Felsenfeld, G. Transitions in histone acetylation reveal boundaries of three separately regulated neighboring loci. EMBO J. 20 (9), 2224-2235 (2001).
- Madisen, L., Krumm, A., Hebbes, T. R., Groudine, M. The immunoglobulin heavy chain locus control region increases histone acetylation along linked c-myc genes. Mol Cell Biol. 18 (11), 6281-6292 (1998).
- Clayton, A. L., Hebbes, T. R., Thorne, A. W., Crane-Robinson, C. Histone acetylation and gene induction in human cells. FEBS Lett. 336 (1), 23-26 (1993).
- Tseng, Z., Wu, T., Liu, Y., Zhong, M., Xiao, A. Using native chromatin immunoprecipitation to interrogate histone variant protein deposition in embryonic stem cells. Methods Mol Biol. 1176, 11-22 (2014).
- Durkin, S. G., Glover, T. W. Chromosome fragile sites. Ann Rev Genetics. 41, 169-192 (2007).
- Lee, J. B., Keung, A. J. Chromatin immunoprecipitation in human and yeast cells. Methods Mol Biol. 1767, 257-269 (2018).
- Miyamoto, R., Yokoyama, A. Protocol for fractionation-assisted native ChIP (fanChIP) to capture protein-protein/DNA interactions on chromatin. STAR Protoc. 2 (2), 100404 (2021).
- Mendez, J., Stillman, B. Chromatin association of human origin recognition complex, cdc6, and minichromosome maintenance proteins during the cell cycle: Assembly of prereplication complexes in late mitosis. Mol Cell Biol. 20 (22), 8602-8612 (2000).
- Nowak, D. E., Tian, B., Brasier, A. R. Two-step cross-linking method for identification of NF-κB gene network by chromatin immunoprecipitation. BioTechniques. 39 (5), 715-725 (2005).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nat Protoc. 3 (6), 1101-1108 (2008).
- Lu, X., Parvathaneni, S., Hara, T., Lal, A., Sharma, S. Replication stress induces specific enrichment of RECQ1 at common fragile sites FRA3B and FRA16D. Mol Cancer. 12, 29 (2013).
- Ruijter, J. M., et al. Amplification efficiency: linking baseline and bias in the analysis of quantitative PCR data. Nucl Acids Res. 37 (6), e45 (2009).
- Lyu, X., Chastain, M., Chai, W. Genome-wide mapping and profiling of γH2AX binding hotspots in response to different replication stress inducers. BMC Genomics. 20, 579 (2019).
- Farmer, P. B., et al. Molecular epidemiology studies of carcinogenic environmental pollutants. Effects of polycyclic aromatic hydrocarbons (PAHs) in environmental pollution on exogenous and oxidative DNA damage. Mutat Res. 544 (2-3), 397-402 (2003).
- Phillips, D. H., Arlt, V. M. Genotoxicity: damage to DNA and its consequences. EXS. 99, 87-110 (2009).
- Nitsch, S., Schneider, R. Native ChIP: Studying the genome-wide distribution of histone modifications in cells and tissue. Meth Mol Biol. 2846, (2024).
- Nikitina, T., Wang, D., Gomberg, M., Grigoryev, S. A., Zhurkin, V. B. Combined micrococcal nuclease and exonuclease III digestion reveals precise positions of the nucleosome core/linker junctions: implications for high-resolution nucleosome mapping. J Mol Biol. 425 (11), 1946-1960 (2013).
- Teves, S. S., Henikoff, S. Salt fractionation of nucleosomes for genome-wide profiling. Methods Mol Biol. 833, 421-432 (2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados