Method Article
ネイティブγH2Aによる一般的な脆弱サイトブレークのキャプチャ。X ChIPの
要約
私たちは、ネイティブのγH2A.Xクロマチン免疫沈降(ChIP)を通じて、一般的な脆弱部位のブレークを検出するための迅速かつ効率的な方法を提示します。このアプローチにより、従来のγH2A.X ChIPアッセイに関連する時間と労力を大幅に削減しながら、結果の高い再現性と信頼性を維持することができます。
要約
外因性物質への曝露によって誘発される複製ストレスは、構造的に不安定になりやすいことが知られているゲノム内の領域である一般的な脆弱な部位でDNA切断につながる可能性があります。γH2A.Xリン酸化はDNA二本鎖切断の確立されたマーカーであるため、γH2A.Xクロマチン免疫沈降(ChIP)アッセイは遺伝毒性試験における強力なツールとして機能します。しかし、従来のγH2A.X ChIPアッセイは、多くの場合、労働集約的で、複数の時間のかかるステップを伴います。本研究では、細胞内分画と天然ChIPを組み合わせてγH2A.X関連複合体を単離する、簡便かつ効果的な方法を紹介します。このアプローチは、特異性と効率を高めたγH2A.X-クロマチン相互作用の解析に特に適しています。細胞内分画を使用すると、クロマチン未結合の材料が効果的に除去され、精製されたクロマチン画分が得られます。その後、温和な条件下でのミクロコッカスヌクレアーゼ(MNase)消化により、γH2A.Xとその関連タンパク質複合体との間の生理学的相互作用を維持しながら、クロマチン断片化が可能になります。この保存は、DNA損傷応答経路に関与するネイティブの相互作用パートナーを研究するために不可欠です。この最適化されたネイティブChIPプロトコルにより、従来のγH2A.X ChIPアッセイに関連する時間と労力が大幅に削減されます。合理化された手順は、ワークフローを簡素化するだけでなく、再現性の高い結果をもたらすため、複数のサンプルのハイスループット処理が必要な環境で特に有利です。この方法は、DNA損傷部位の正確かつ効率的な検出が重要なゲノム安定性、DNA修復、クロマチン生物学に焦点を当てた研究に幅広く適用できます。この方法は、最適化されたプロトコルと合理化されたステップを採用することにより、感度を向上させ、サンプルの取り扱いを最小限に抑えて脆弱な部位でのDNA損傷の検出を可能にし、ゲノムの安定性とDNA損傷応答の研究に貴重なツールとなります。
概要
一般的な脆弱部位(CFS)は、中期に壊れやすいすべてのヒト染色体に見られる大きな染色体領域です。複製ストレス下では、これらの領域での複製が大幅に遅延し、有糸分裂侵入1の前に完全な複製が妨げられ、最終的には部位特異的なギャップやブレークが発生します。CFSは染色体不安定性のホットスポットであり、がんの早期発症における染色体再配列の主な原因です。腫瘍形成条件下でしばしば存在する複製ストレスは、腫瘍抑制遺伝子の喪失とがん遺伝子の増幅につながる可能性があります-総称してコピー数多型(CNV)と呼ばれます2,3,4,5,6。さらに、CFSはウイルスの統合を非常に容易にし、がんの発症をさらに促進します7,8,9,10。原発腫瘍の汎がん解析中に、CFS領域で腫瘍抑制遺伝子の複数のホモ接合性欠失が検出されています。がんで最も一般的に影響を受けるCFSには、FRA2F、FRA3B、FRA4F、FRA5H、およびFRA16D11が含まれます。CFSは、外因性発がん性物質の存在下で特に破損しやすい12。環境汚染物質の有害な発がん性影響を評価するには、CFSブレークの発生を定量化するための迅速で信頼性の高い方法が必要です。
H2Aのリン酸化。毛細血管拡張性運動失調症およびRad3関連タンパク質(ATR)または毛細血管拡張運動失調症変異(ATM)によるセリン残基139(γH2A.X)のXは、シグナル伝達の複製フォークの失速13における重要なイベントである。γH2A.Xは、二本鎖切断(DSB)形成13に先立つ失速した複製フォークの指標として機能し、停止した部位への修復タンパク質の効率的な動員を促進するための好ましいクロマチン環境を作り出します。さらに、γH2A.Xは、フォーク崩壊14,15後のサイトを壊すためにリクルートすることができ、DSB修理における主要な役割と一致しています。CFSの切断は、がんの進行を促進する染色体異常と密接に関連しているため、これらの切断を検出することは、腫瘍形成の初期段階を理解するのに役立ちます。CFSにおけるγH2A.Xの存在は、ゲノム不安定性の初期イベントを検出するためのバイオマーカーとして使用できます。この情報は、潜在的な発がん性物質を特定し、さまざまな外因性物質への曝露に関連するリスクを評価するのに役立ちます。γH2A.XクロマチンIP(ChIP)は、外因性薬剤によって誘導されたCFSのDNA切断を測定することにより、そのような薬剤が腫瘍形成の根底にあるメカニズムにどのように寄与しているかについての洞察を得ることができます。
従来のChIP(架橋ChIP、X-ChIP)では、γH2A.Xとその標的DNA配列との会合は、可逆的なホルムアルデヒド架橋によって安定化されます。続いて、クロマチンを超音波処理により約500塩基対(bp)の断片に剪断し、得られた溶液を沈殿16,17,18によって破片を除去する。次に、ChIPグレードのγH2A.X抗体を透明化したクロマチン画分に添加し、続いてProtein A/Gアガロースビーズを添加してγH2A.X結合クロマチン領域を濃縮します16,17,18。免疫複合体(すなわち、ビーズ−抗体−γH2A.X標的DNA複合体)を、非特異的に結合したDNA断片16,17,18を除去するために、厳格な洗浄バッファーで複数回洗浄する。洗浄後、特異的に結合したDNAが免疫複合体から溶出されます。次に、ホルムアルデヒド架橋を逆転させ、続いてプロテイナーゼKを使用してタンパク質を消化し、その後、濃縮されたDNAを精製して濃縮します16,17,18。γH2A.X関連領域を評価するために、PCR、定量的PCR(qPCR)、または直接シーケンシングが用いられる16,17,18。CFSなどの特定の領域におけるγH2A.Xの占有率は、PCRまたはqPCRシグナルの強度によって決定され、これはその位置に結合したγH2A.Xの量に比例し、部位特異的なDNA損傷および修復イベント16,17,18に関する洞察を提供する。
強力な実験的アプローチであるにもかかわらず、X-ChIPにはいくつかの重要な制限があります:(i)固定に関連する抗体沈殿の非効率性のために、通常は1 x 107から5 x 107の範囲の多数の細胞が必要であり、実験の全体的なコストを増加させます19。(ii)ホルムアルデヒド架橋を逆転させ、その後のDNA精製のプロセスは時間と労力を要するため、結果の一貫性と信頼性を維持することが困難である。(iii)機能的重要性が小さいγH2A.X-DNA相互作用は、交差結合ステップが一過性相互作用を安定化させ、生物学的に関連性のない相互作用の検出につながるため、有意性が高い相互作用と区別できない可能性がある19。
ネイティブクロマチン免疫沈降法(Native ChIPまたはN-ChIP)は、生理的塩条件下でネイティブクロマチンの文脈でタンパク質-DNA相互作用を研究するために使用される重要な生化学的手法です。これは、クロマチン、転写因子の結合、およびヒストン修飾の空間的および時間的組織の解明に役立っています。Native ChIPは、クロマチンバイオロジーとエピジェネティクスの幅広い分野で長年の役割を果たしており、X-ChIPと比較して独自の利点と限界を提供しています。1980年代後半に導入されたこの方法は、ミクロコッカスヌクレアーゼ(MNase)21による消化など、その天然構造を保持する方法によって細胞からクロマチンを単離することを含む。これにより、固有のタンパク質-DNAおよびヒストン-DNAの接触が維持されるため、Native ChIPは、ヒストン修飾および天然クロマチン設定におけるヌクレオソームのポジショニングの研究に特に適しています22。高分解能のNative ChIP研究では、MNase消化を使用してクロマチンを個々のヌクレオソームに還元することが実証されており、これによりヒストン修飾のマッピングがより正確に容易になることが示されています23。さらに、化学的な架橋が関与していないため、タンパク質-DNA相互作用を誤って表現する可能性のあるバイアスやアーティファクトを導入するリスクが最小限に抑えられる24。
ホルムアルデヒドやその他の架橋剤を使用してタンパク質-DNA相互作用を固定するX-ChIPとは対照的に、Native ChIPは、潜在的な架橋アーティファクトを回避することで、クロマチンをよりリアルに観察することができます。しかし、X-ChIPは一般に、DNAと調節タンパク質との間の一過性または動的相互作用の検出に適しています25が、Native ChIPは、ヒストンや他のクロマチン結合タンパク質などの安定したタンパク質間相互作用に理想的です26,27。Native ChIPの限界の1つは、X-ChIP25の架橋によって安定化されることが多い低親和性または一過性の結合イベントを捕捉できないことです。
エピジェネティクスにおける重要な研究は、Native ChIPを活用して、多様な生物学的環境におけるヒストン修飾を明らかにしてきた28。これらの努力は、ヒストンコード(遺伝子発現とクロマチンダイナミクスを調節するヒストン修飾のパターン)を定義する上で重要でした29。H2Aですが。Xは、あまり強く関連しないリンカーヒストンである天然のH2Aです。X ChIP法は、胚性幹細胞にうまく適用されています30。この研究では、ヒト293T細胞でγH2A.XのNative ChIPを実行するために、クロマチン抽出手順を最適化しました(図1)。ヒドロキシ尿素とアフィジコリンは、DNA複製ストレス、損傷、およびゲノム不安定性を調査する研究で広く使用されています31。本研究では、これらの薬剤を細胞に適用して、CFSで複製ストレスを誘導し、DNA切断を生じさせるという結果を得ました。
約1 x 106〜5 x 106細胞の出発物質を使用して、この方法は、(i)クロマチンを単離するための細胞内分画、(ii)クロマチンを分離するためのマイクロコッカスヌクレアーゼ(MNase)消化、(iii)免疫沈降と溶出、および(iv)定量PCR(qPCR)によるDNA分析の4つの主要な段階に分けることができます。細胞内分画後にChIPを実施すると、いくつかの利点が得られ、多数の研究で十分に文書化されています32,33,34,35。このアプローチにより、クロマチン未結合タンパク質やその他の細胞破片の除去が可能になり、高度に精製されたクロマチン画分が得られます。免疫沈降前にクロマチンを単離することにより、細胞内分画は天然のクロマチン相互作用を維持し、クロマチン結合していないタンパク質からのバックグラウンドノイズを低減し、クロマチン結合複合体のみが分析のために保持されるため、より特異的で信頼性の高い結果につながります。さらに、細胞内分画により、クロマチン消化の条件がより穏やかになり、生理学的タンパク質-DNA相互作用が維持され、天然の細胞環境内のクロマチンダイナミクスをより正確に表現できます。
γH2AXのネイティブChIPを使用して、一般的な脆弱部位の破損に対する外因性薬剤の影響を測定することは、がん研究にとって大きな可能性を秘めています。この技術により、環境発がん物質への曝露によって誘発されるDNA損傷の検出が可能になり、汚染物質がゲノムの不安定性とがんの発症に寄与する分子メカニズムについての洞察が得られます。この方法は、ネイティブクロマチンのコンテキストを維持することにより、発がん性曝露に関連するDNA損傷パターンの正確な評価を容易にし、環境リスクの評価と汚染による腫瘍形成の研究を支援します。
プロトコル
1.細胞回収
- 約5 x 105 HEK 293T細胞を、それぞれ4 mLの完全DMEM培地を含む4つの6 cmディッシュのそれぞれに播種します。
- 24時間後、1つのディッシュを2μLの1 mMアフィジコリン( 材料表を参照)ストック溶液(最終濃度0.5 μM)で処理し、もう1つのディッシュを20 μLの1 Mヒドロキシ尿素( 材料表を参照)ストック溶液(最終濃度5 mM)で処理して、複製ストレスを誘発します。残りの 2 つのディッシュに DMSO を追加して、コントロールとして機能します。
- 24時間処理後、培地を廃棄します。1枚の6cmプレートは、典型的には、60%〜70%のコンフルエンスで約2×106細胞を生じる。
- 各ディッシュを氷冷した5mLの1xPBSで2回すすいでください。セルスクレーパーを使用して細胞を剥離し、細胞懸濁液を4本の個別の1.5 mLチューブに移します。P1000ピペットで優しく上下にピペットで動かし、細胞の塊を解離します。
- 細胞を500 x g で5分間、4°Cで遠心分離し、上清を捨てます。細胞を氷の上に置きます。
2. 細胞内分画
- 調製したばかりの低温バッファーA( 表1参照)500 μLに細胞ペレットを再懸濁し、穏やかなピペッティングで細胞凝集が完全に解離することを確認します。
- ライセートを氷上で5〜10分間インキュベートします。顕微鏡で溶解の進行を確認し、細胞の溶解が完全であることを確認します。
- ライセートの少量アリコート(約5〜10μL)を取り、清潔な顕微鏡スライドに置きます。汚染を避けるためにカバースリップで覆います。
- 適切な倍率(20倍〜40倍など)の光学顕微鏡を使用して、細胞や破片を視覚化します。未溶解のコントロールサンプルと比較して、無傷の細胞と溶解した物質を区別します。
注:適切に溶解されたサンプルには明確な細胞輪郭はなく、拡散したクロマチンまたは細胞材料のみが含まれます。ピントを合わせると、ライセートがはっきりと観察できます。必要に応じて、特定の細胞タイプを扱う際に、Dounceホモジナイザーを使用するなど、溶解ステップ中に機械的な力を加えます。
- 細胞が完全に溶解したら、500 x g で4°Cで5分間遠心分離します。上清は慎重に捨てます。ワイドオリフィスピペットチップを使用して、核ペレットを500 μLの冷緩衝液Aに再懸濁します。
注:ワイドオリフィスチップは、せん断力を最小限に抑え、クロマチンなどの繊細なサンプルを保護するのに役立ちます。標準的な先端の端を鋭利な刃で切断することにより、ワイドボアチップを作成します。 - 500 x g で4°Cで5分間遠心分離します。 上清は慎重に捨てます。
- インキュベーターを37°Cに予温し、停止バッファー(100 mM EDTA、pH 8.0; 表 1)。
- Micrococcal Nuclease(MNase、 材料表を参照)の濃度とインキュベーション時間を事前に最適化します。
- 40 μLの試験用クロマチンサンプルをいくつかの等しい分量に分割して、異なるMNase濃度とインキュベーション時間をテストします。
- さまざまなMNase濃度(例:0.0625 U、0.125 U、0.25 U、0.5 U、1 U、2 U、4 U、8 U)を使用し、複数回のインキュベーション時間(例:2、5、10、15分)をテストします。
- さまざまな濃度のMNaseを含むMNase Buffer( 表1を参照)をクロマチンアリコートに加え、サンプルを37°Cで指定時間インキュベートします。
- 所望のインキュベーション時間の直後に1/4容量の停止バッファー(最終濃度:20 mM EDTA)を添加して反応を終了します。
- 消化したクロマチンサンプルから、フェノール/クロロホルム/イソアミルアルコール抽出法を用いてDNAを単離します。
- 抽出したDNAを1.5%アガロースゲル上で分析し、消化パターンを視覚化します:消化不足では高分子量バンドが見られます(図2、レーン1-4)。過剰消化は塗抹標本または非常に短い断片(図2、レーン6-8)をもたらし、最適な消化は明確なヌクレオソームラダーパターン(図2、レーン5、例えば、モノヌクレオソーム、ジヌクレオソーム、ジヌクレオソーム)をもたらす。
- 過度の過剰消化なしに目的のヌクレオソーム分解能が得られる条件を特定します。
注:CaCl2 はMNase活性の補因子として作用します。CaCl2濃度を1 mMから5 mMの間で調整することにより、消化を最適化します。
- 100 μL の MNase Buffer で、幅広のオリフィスチップで 5 - 10 回ピペッティングして、無傷の核を穏やかに再懸濁します。あらかじめ決めた量のMNaseを直ちにサンプルに加えます(1.25 U MNase/100 μL MNase Buffer)。
注:複数のサンプルを扱う場合は、過剰消化を避けるために、各サンプルを個別に分解してください。 - チューブをローテーターに置き、37°Cで5分間インキュベートします。 直ちにチューブを氷に戻し、EDTAを最終濃度20 mMまで添加してMNase消化を終了し、ボルテックスで混合します。
- 各サンプルに500 μLのバッファーB( 表1を参照)を加え、ピペッティングで5回から10回まで完全に混合します。氷上で5分間インキュベートすることにより、タンパク質を可溶化します。
注:バッファーBの塩と界面活性剤は、弱いクロマチン結合タンパク質を解離し、免疫沈降のためにエピトープを露出させるのに役立ちます。 - 不溶性材料を4°Cで最高速度5分間遠心分離することにより、ペレット化します。 透明な上清を、ネイティブクロマチン画分としてラベル付けされた新しい1.5 mLチューブに移します。サンプルは-80°Cで保存するか、クロマチンフラグメンテーションの効率を検証するために使用できます。
注:頻繁な凍結融解サイクルは、目的のタンパク質-DNA相互作用を混乱させる可能性があるため、避けてください。可能な限り、凍結融解サイクルを最小限に抑えます。
3. クロマチンフラグメンテーションの検証
- 各サンプルから上清10 μLを新しい1.5 mLチューブに分注します。20 μLの蒸留水と30 μLのフェノール/クロロホルム/イソアミルアルコール(25:24:1)と混合します。
- チューブをしっかりと閉じ、15〜30秒間激しく渦巻きます。20,000 x g (または遠心分離機の最高速度)で4°Cで10分間遠心分離します。 遠心分離後、透明な上層、白い中間層、黄色の底層の3つの異なる層が観察されます。
- 上部水相(DNAを含む)の20μLを新しいチューブに慎重に移します。精製したDNAを1.5%アガロースゲルで100 Vで30分間分離し、消化パターンを可視化します。クロマチンフラグメントのサイズが主に200〜1000塩基対であることを確認してください。
注:適切なクロマチンフラグメントサイズは、ネイティブChIPの成功にとって重要であり、酵素ユニット、インキュベーション時間、CaCl2濃度などのMNase処理条件に依存します。MNase消化効率は、細胞の種類や数によっても異なります。 図2 (レーン5)に示すクロマチンフラグメンテーションパターンが、このChIPアッセイに推奨されます。
4. 免疫沈降
- 各サンプルから消化したクロマチン20 μLを新鮮な1.5 mLチューブに分注し、180 μLの溶出バッファーと混合します( 表1を参照)。これらのチューブをインプットサンプルとしてラベル付けし、-20°Cで保存します。
- 400 μLのクロマチンサンプルを別の1.5 mLチューブに移し、ChIPを行ってください。
- γH2A.X抗体( 材料表参照)を、DMSO処理サンプル1個、アフィジコリン処理サンプル1個、ヒドロキシ尿素処理サンプル1個に加えます。同量の正常IgG( 材料表を参照)を、ChIPアッセイのネガティブコントロールとして別のDMSO処理サンプルに添加します。
注:ここでは、通常、1 μgの一次抗体を400 μLのクロマチンに対して使用します(つまり、抗体の最終濃度は2.5 μg/mLです)。ただし、最適な量は、さまざまなγH2A.X抗体について経験的に決定する必要があります。 - ChIPチューブを4°Cのローテーターに置き、少なくとも5時間、できれば一晩インキュベートします。
- その間に、100 μLのChIPグレードの磁性タンパク質A/Gビーズ( 材料表を参照)を新しい1.5 mLチューブに分注します。幅広のオリフィスチップを使用し、ゆっくりとピペットでビーズを正確に測定します。チューブを磁気スタンドに少なくとも1分間置き、液体を慎重に廃棄します。
- 0.5% BSAを含む1x PBSの1 mLにビーズを再懸濁します。4°Cで約4時間回転させます。チューブを磁気スタンドに少なくとも1分間置き、上清を捨てます。
- 0.5% BSAを含む1x PBS1 mLでビーズを再度洗浄します。チューブを磁気スタンドに1分間置き、磁気ビーズをペレット化してから、上清を捨てます。
注:ステップ4.5〜4.7は、磁気ビーズへの抗体の非特異的結合を減らすためのビーズのプレコーティングです。 - プレコートしたビーズを、ワイドオリフィスチップを使用して100 μLのバッファーBに再懸濁します。プレコートされた磁気ビーズ懸濁液25 μLを各ChIPサンプルチューブに加えます。4°Cで2時間回転させます。
- ChIPチューブをマグネットスタンドに置き、ビーズがチューブの側面に完全に取り付けられ、溶液が透明になるまで待ちます。
- 透明な上清は、磁気ビーズを乱さずに捨ててください。ビーズを1 mLのWash Buffer( 表1参照)で再懸濁し、4°Cで10分間回転させます。
- チューブをマグネットスタンドに戻し、溶液が透明になるまで待ちます。ウォッシュバッファーを廃棄します。洗濯を合計4回繰り返します。
- 最終洗浄後に洗浄バッファーを廃棄し、チューブを400 x g で40秒間、4°Cで短時間遠心分離して、残留液体をスピンダウンします。チューブをマグネットスタンドに戻し、チューブの底に残っている液体を慎重に取り除きます。
5. 溶出とDNA沈殿
注:抗体の効率はバッチによって異なる場合があります。新しい抗体の結合親和性を確認するためには、ウェスタンブロット解析により免疫沈降したサンプルを確認することが重要です。
- 以下に述べるウェスタンブロット(WB)を用いて、ChIP抗体のプルダウン効率を検証してください。
- 分析のためにChIPサンプルの少量を採取します(つまり、通常はChIPサンプルの10%)。インプットクロマチン(免疫沈降前)とネガティブコントロール(IgGプルダウンなど)を含めて比較します。
- 抗体結合ビーズからタンパク質を溶出するには、20 μLの1x SDS-PAGEローディングバッファー( 材料表を参照)で95°C、5分間加熱します。
- IPサンプル、インプット、コントロールを15% SDS-PAGEゲルにロードします。ジェルを走らせます。
- タンパク質を0.2 μmのニトロセルロース( 材料表を参照)またはPVDFメンブレンに、ウェットまたはセミドライトランスファーシステムを使用して移します。
- 5%無脂肪乳またはTBST( 表1参照)中のBSAでメンブレンを室温で1時間ブロックし、非特異的結合を防ぎます。
- ブロッキングバッファーで希釈したγH2A.Xに対する一次抗体( 材料表を参照)とメンブレンをインキュベートし、室温で1〜2時間、または4°Cで一晩。
- TBSTでメンブレンを3回洗浄し、結合していない抗体を除去します。メンブレンをHRP標識二次抗体( 材料表を参照)と室温で1時間インキュベートします。メンブレンを再度洗浄して、余分な二次抗体を除去します。
- 化学発光基質を用いてメンブレンを作製し、イメージャーでシグナルを可視化します。IP、入力、および制御レーン間の信号強度を比較して、プルダウンの効率と特異性を評価します。
注:IPレーンの標的タンパク質に対応するバンドは、抗体のプルダウンが成功したことを確認します。このアプローチにより、ChIP実験中に標的タンパク質を捕捉する抗体の有効性を評価できます。
- 50 μLのElution Buffer( 表1参照)を残りの各ChIPサンプルに加えます。チューブをサーモミキサーに置き、室温で15分間振とうします。
- チューブを磁気ホルダーに少なくとも1分間置きます。溶出液を新しいチューブに集めます。1回繰り返して、溶出物を同じチューブに集めます。
- 各ChIP溶出サンプルに100 μLのElution Bufferを、各インプットサンプルに180 μLのElution Bufferを追加してください。
- 各サンプルに200μLのフェノール/クロロホルム/イソアミルアルコール(25:24:1)を加え、激しくボルテックスします。サンプルを20,000 x g (または最大速度)で4°Cで10分間遠心分離します。
- 19 μL の 3M 酢酸ナトリウム (NaOAc、pH 5.2、 表 1 を参照) と 2 μL のグリコーゲン溶液 (20 mg/mL、 材料表を参照) を新しい 1.5 mL 遠心分離チューブごとに追加します。
- 遠心分離後、上部水層(約190μL)をNaOAcとグリコーゲンの入ったチューブに慎重に移し、ボルテックスして混合します。
- 500μLの100%エタノールを加え、ボルテックスします。サンプルを-20°Cで少なくとも2時間または一晩インキュベートすることにより、DNAを沈殿させます。
- チューブを20,000 x g (または最大速度)で4°Cで10分間遠心分離します。 白いペレットを乱さないように注意しながら、上清を捨てます。ペレットを1mLの70%エタノールに再懸濁し、十分にボルテックスします。
- チューブを20,000 x g (または最大速度)で4°Cで5分間遠心分離します。 上清を慎重に取り除きます。チューブを再度簡単に遠心分離して、残留エタノールをスピンダウンします。P20ピペットを使用してエタノールを慎重に取り除きます。DNAペレットを2〜3分間風乾します。
注:ペレットを過度に乾燥させると、DNAが再溶解しにくくなる可能性があるため、避けてください。 - ChIPサンプルの場合は、DNAを400 μLのTEバッファーに再懸濁します( 表1を参照)。インプットDNAについては、1000 μLのTEバッファーに再懸濁します。溶出したサンプルは、-20°Cで保存できるようになりました。
6. qPCRの定量
- 市販のキット( Table of Materialを参照)を使用して、各サンプルのテクニカルトリプリケートを使用してqPCRを実施します。増幅の特異性を確保するために融解曲線分析を行うことにより、単一の特異的PCR産物の存在を確認する36。
- データ分析
注:相対定量分析では、試験サンプルは対照サンプルに対する倍率変化として表されます(通常の精製IgGまたは模擬IPを使用して免疫沈降)。免疫沈降タンパク質によって占有されていないことが知られているDNA遺伝子座(陰性遺伝子座)は、既知、占有、陽性対照のDNA遺伝子座36と比較して、この方法で参照遺伝子として使用することができる。- 以下の式を使用して、各ChIPの入力の割合を計算します
%入力 = 2(-ΔCt [正規化されたChIP]) - 正の遺伝子座のΔCt値から正の遺伝子座のΔCt値を以下の式で減算することにより、正の遺伝子座ΔCt値を負の遺伝子座(ΔΔCt)に正規化します
(ΔΔCt = ΔCtpositive - ΔCtnegative) - ChIP DNAの陽性遺伝子座配列の負遺伝子座に対する倍濃縮度を、以下の式を用いて計算します
倍率濃縮=2ΔΔCt
分析に使用したqPCRプライマーの配列を表2に示します。FRA3BおよびFRA16D37のゲノム構成を図3A,Bに示す。
- 以下の式を使用して、各ChIPの入力の割合を計算します
- 統計分析
- スチューデントの対応のあるt検定を使用して、結果を統計的に分析します。≤0.05のp値は統計的に有意であると考えられ、観測された差がランダム変動によるものである可能性は低いことを示しています38。
結果
クロマチンフラグメントのサイズは、抗体結合のためのDNA領域のアクセシビリティに直接影響を与えるため、Native ChIPの成功にとって非常に重要です。クロマチンフラグメンテーションに最適なMNase濃度を決定するために、さまざまな濃度のMNase(つまり、反応あたり0.0625 U、0.125 U、0.25 U、0.5 U、1 U、2 U、4 U、8 U)と40 μLの単離核を含む一連の微量遠心チューブを調製しました。各反応を37°Cで5分間インキュベートし、さまざまなクロマチンフラグメントサイズを達成しました。MNase消化の結果を 図2に示します。
図2に示すように、MNaseの濃度が高いほど、クロマチンの消化がより広範囲に行われ、その結果、モノヌクレオソームフラグメントが優勢になりました(図2、レーン1、2、および3)。対照的に、MNase濃度が低い場合、クロマチンフラグメントの大部分は大きく、しばしば1 kbを超え(図2、レーン6、7、8)、ダウンストリームアプリケーションには消化が不十分であることを示しています。MNase濃度1.6ユニット、消化時間5分で、主に200〜1000塩基対の範囲のクロマチンフラグメントが得られました(図2、レーン5)。このフラグメントサイズは、特定のエピトープへのアクセス性を維持しながら、クロマチン関連DNAの効率的な濃縮を保証するため、Native ChIPに理想的です。適切なフラグメントサイズにより、効率的な免疫沈降や、定量PCRやシーケンシングなどのダウンストリーム解析が容易になり、タンパク質-DNA相互作用を高分解能で検出するのに適しています。
抗体の結合親和性は、Native ChIPの成功にとって重要な要素です。抗体が効率的かつ特異的に標的エピトープに結合することを確認することは、信頼性の高い結果を得るために不可欠です。そのため、抗体の結合効率を事前に確認することが重要です。γH2A.X免疫沈降の効率を確認するために、ウェスタンブロット(WB)アッセイを実施しました。
DMSO処理細胞(コントロール)、アフィジコリン処理細胞、ヒドロキシ尿素処理細胞(複製ストレス)のγH2A.Xレベルをウェスタンブロッティングを用いて比較した。 図4A (入力)に示すように、γH2A.Xレベルは、DMSO処理細胞と比較して、アフィジコリン処理細胞で有意に高かった。このγH2A.Xの増加は、アフィジコリンによる複製ストレスの誘導と一致しており、これにより二本鎖切断形成が増加し、その後のγH2A.Xリン酸化が増加します。ChIPを実施した後、IgGコントロールとγH2A.X IPサンプルのγH2A.X濃縮を比較することにより、免疫沈降の特異性を検証しました。IgGコントロールでは、検出可能なγH2A.Xはプルダウンされず、非特異的結合が最小限であることが示されました(図4B、N-ChIP、およびX-ChIP)。対照的に、γH2A.X IPサンプルは、アフィジコリン処理細胞からγH2A.Xを濃縮することに成功し、抗体のγH2A.Xへの効率的かつ特異的な結合を示しました(図4、N-ChIPおよびX-ChIP)。これらの結果から、γH2A.X抗体はNative ChIP実験に十分な結合親和性と特異性を有していることが確認され、γH2A.X結合クロマチン領域の信頼性の高い検出と濃縮が可能となります。この検証ステップは、その後のChIP解析の品質と精度を確保するために不可欠です。
私たちは、共通脆弱部位(CFS)FRA16DおよびFRA3Bを使用して、複製ストレスに応答したDNAへのγH2A.Xの特異性を評価しました。FRA16DおよびFRA3Bは、複製ストレス条件下で不安定になりやすいCFS領域としてよく知られており、γH2A.X/DNA相互作用の検証に理想的な候補です。 図3A、B は、これら2つのCFSのゲノム構成と、解析に使用した特定のqPCRプライマーを示しています。
γH2A.Xが複製ストレスに応答してこれらのCFS領域と会合するかどうかを調べるために、コントロール細胞(DMSO処理)、アフィジコリン処理細胞、およびヒドロキシ尿素処理細胞でChIP-qPCR解析を行いました。qPCRの結果は、DMSO処理したコントロール細胞では、γH2A.XがFRA16DまたはFRA3B遺伝子座のいずれにも濃縮されていないことを示し、これらの領域で重大なDNA損傷または複製ストレスがないことを示しました(図3C)。しかし、DNAポリメラーゼを阻害することで複製ストレスを誘導するアフィジコリンまたはヒドロキシ尿素で処理した細胞では、FRA16DとFRA3Bの両方でγH2A.Xの有意な濃縮が観察されました(図3C)。このことは、γH2A.XがDNA複製ストレスに特異的に応答してこれらのCFS領域に動員されたことを示しています。これらの知見は、CFSにおけるγH2A.Xの占有が複製ストレスによって引き起こされることを示しており、DNA損傷応答と複製フォークの失速のマーカーとしての役割を裏付けています。アフィジコリン治療中のγH2A.XのFRA16DおよびFRA3Bへの特異的動員は、当社のChIPアッセイの特異性をさらに検証し、γH2A.Xが脆弱な部位でのDNA損傷および複製ストレスの信頼できる指標であることが確認されています。
ネイティブChIPと架橋ChIP(X-ChIP)の効率を比較するために、Lyu et al.39によって記述されたプロトコルに従って、同じ開始セル数を使用してγH2A.X X-ChIPを実施しました。図 3Dに示すように、γH2A.XのFRA16Dへの動員は、アフィジコリンとヒドロキシ尿素による処理によって増加しました。しかし、X-ChIPのFRA16DにおけるγH2A.Xの相対濃縮は、ネイティブChIPで観察されたものよりも著しく低く、これらの条件下でのX-ChIPの効率が低下したことを示しています。さらに、FRA3Bでは、γH2A.X濃縮の増加は統計的に有意ではなく、X-ChIPが限られた数の細胞で作業すると効率が低下するという追加の証拠が得られました。これらの結果から、サンプルサイズに制約がある場合の脆弱な部位でのγH2A.X濃縮の検出には、ネイティブChIPがより適している可能性が示唆されています。スチューデントの対応のあるt検定を使用して、結果を統計的に分析します。p値が0.05≤は統計的に有意であると見なされます。

図1:ネイティブγH2A.X ChIPアッセイを実施するためのWオークフロー。 (i)細胞を培養し、特定の処理(例えば、ヒドロキシ尿素のようなDNA損傷剤)を施して、γH2A.Xシグナル伝達を誘導する。(ii)処理された細胞は、処理のために収集されます。(iii)細胞を分画してクロマチン結合γH2A.Xを単離し、他の細胞成分から分離します。(iv)ミクロコッカスヌクレアーゼ(MNase)消化を行い、クロマチンをヌクレオソームサイズの断片に断片化します。(v)クロマチンフラグメントのサイズは、アガロースゲル上でサンプルを泳動することにより検証し、フラグメントがChIPに適した長さであることを確認します。(vi)γH2A.Xに特異的な抗体を用いて、γH2A.Xで標識されたクロマチン断片を免疫沈降させる。(vii)プロテインA/Gビーズは、抗体-クロマチン複合体を引き下げ、γH2A.X結合DNAを濃縮するために使用されます。(viii)γH2A.XでマークされたDNA断片を溶出して精製し、ダウンストリーム分析の準備をします。(ix)ダウンストリーム解析:qPCRとシーケンシング、およびデータ解析。 この図の拡大版を表示するには、ここをクリックしてください。

図2:MNase消化条件を最適化するためのアッセイ。 クロマチンフラグメントは、さまざまなMNase消化条件を使用してHEK 293T細胞から調製しました。断片化されたクロマチンから得られた精製DNAを2%アガロースゲル上で分離し、100 Vで30分間分析しました。レーン 1 から 8 は、MNase の濃度を上げて処理したサンプル(つまり、1 反応あたり 0.0625 U、0.125 U、0.25 U、0.5 U、1 U、2 U、4 U、8 U)を表しており、クロマチン消化の勾配を示しています。DNAラダー(Mで示す)は、サイズ参照のために含めました。DNA断片は、Midori Green AdvanceDNA染色溶液を用いて可視化しました。標識されたバンドはモノヌクレオソームサイズのDNA断片に対応しており、各MNase濃度での消化効率を強調しています。 この図の拡大版を表示するには、ここをクリックしてください。
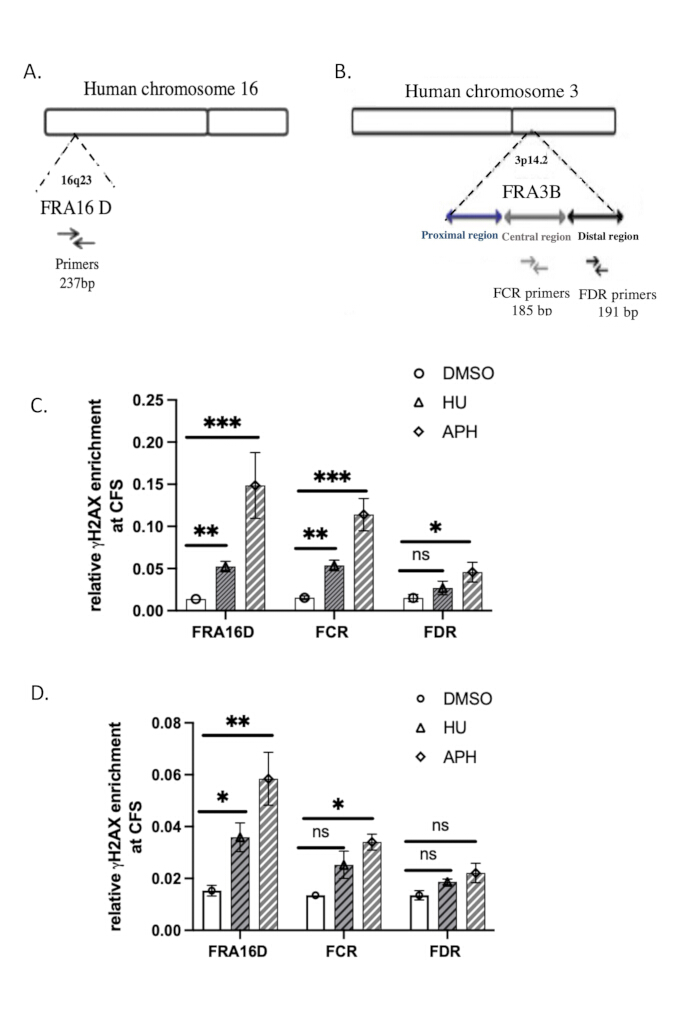
図3:γH2A.X ChIPのqPCR検証。 (A)FRA16Dのゲノム組織図。(B)FRA3B領域のゲノム組織の図。FRA3B遺伝子座内のFRA16D、遠位(FDR)、および中心(FCR)領域のqPCR解析に使用されるプライマーセットが示されています。(C)一般的な脆弱部位(CFS)、特にFRA3BおよびFRA16DにおけるγH2A.Xの相対濃縮を、ネイティブChIPを使用して評価し、その後、アフィジコリンおよびヒドロキシ尿素で処理した後のqPCR分析を行いました。(D)一般的な脆弱部位(CFS)、特にFRA3BおよびFRA16DにおけるγH2A.Xの相対濃縮度を、X-ChIPを使用して評価し、その後、アフィジコリンおよびヒドロキシ尿素で処理した後のqPCR分析を行いました。p < 0.001 の統計的に有意な結果を示します。** は p < 0.01 を示し、* は p < 0.05 を示し、t 検定によって決定されます。n = 3 です。 この図の拡大版を表示するには、ここをクリックしてください。
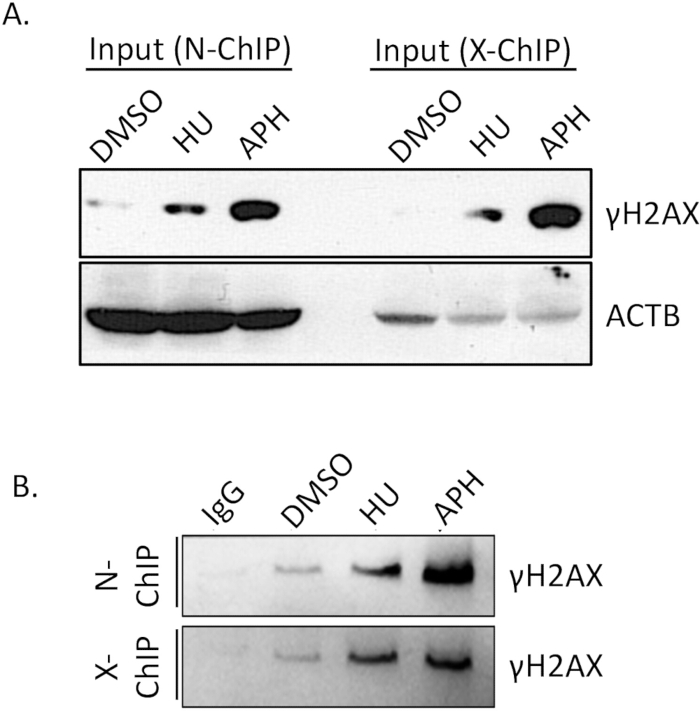
図4:ウェスタンブロットを用いたChIP抗体のプルダウン効率の検証。 全サンプルから、 ChIPサンプルの10%とインプットコントロールを15% SDS-PAGEゲル上で分離し、ポリペプチドを分離しました。電気泳動後、分離したタンパク質を標準的な転写プロトコルを用いて0.22 μm PVDFメンブレンに転写しました。次に、γH2A.XおよびACTBを標的とする特異的な一次抗体でメンブレンを順次プローブし、続いて適切なHRP標識二次抗体をプローブしました。その後、化学発光検出を用いてシグナルを可視化し、ChIPアッセイにおける抗体特異性とプルダウン効率の評価を可能にしました。(A)DMSO処理細胞(コントロール)、アフィジコリン処理細胞、およびヒドロキシ尿素処理細胞(複製ストレス)間のγH2A.Xレベル(ACTBをローディングコントロールとして)。(B)IgGコントロールおよびIPサンプルにおけるγH2A.X濃縮。略語:APH =アフィジコリン、HU =ヒドロキシ尿素。 この図の拡大版を表示するには、ここをクリックしてください。
| バッファ A (1X) | |
| 試薬 | 作業集中力 |
| パイプ pH 6.8 | 10 mM |
| NaClの | 100 mM |
| MgCl2 | 3メートル |
| EGTA pH 7.6 | 1 mM |
| -20°Cで最大1年間保存 | |
| MNase バッファー (1X) | |
| 試薬 | 作業集中力 |
| トリスHCl、pH 7.5 | 50 mM |
| CaCl2 | 1 mM |
| MgCl2 | 4メートル |
| -20°Cで最大1年間保存 | |
| バッファB(1X) | |
| 試薬 | 作業集中力 |
| リン酸ナトリウムpH 7。0 | 20 mM |
| ピロリン酸ナトリウム10。H2O | 30 mM |
| KClの | 250 mM |
| EDTA、pH 8.0 | 5メートルメートル |
| グリセロール | 10% |
| トリトンX-100 | 0.10% |
| プロテアーゼ阻害剤カクテル | 1倍 |
| ホスファターゼ阻害剤カクテル | 1倍 |
| DTTの | 0.5 mM |
| -20°Cで最大1年間保存 | |
| 溶出バッファー | |
| 試薬 | 作業集中力 |
| NaHCO3 | 50 mM |
| SDSの | 1% |
| 作りたて | |
| 洗浄バッファー | |
| 試薬 | 作業集中力 |
| MNase バッファー | ×0.5 |
| バッファB | ×0.5 |
| 作りたて | |
| ストップバッファ | |
| EDTAの | 100 mM , pH8.0 |
| 3 M酢酸ナトリウム、pH 5.2 | |
| 408.24 g 酢酸ナトリウム | |
| 酢酸によるpH5.2までのajustpH | |
| 容量を1 Lに調整する | |
| ろ過またはオートクレーブによりバッファーを滅菌します | |
| TBST バッファ | |
| トリスベース、pH 8.0 | 10 mM |
| NaClの | 150 mM |
| トゥイーン 20 | 0.1%(v/v) |
| TEバッファ | |
| トリスHCl、pH 8.0 | 10 mM |
| EDTA、pH 8.0 | 1mM |
表 1: バッファーの構成。
| 遺伝子座 | フォワードプライマー | リバースプライマー |
| FRA16Dの | TCCTGTGGAAGGGATATTTA | CCCCTCATATTCTGCTTCTA |
| FRA3Bの | TGTTGGAATGTTAACTCTATCCCAT | ATATCTCATCAAGACCGCTGCA |
| FCRの | ||
| FRA3Bの | CAATGGCTTAAGCカガキャットggt | AGTGAATGGCATGGCTGGAATG |
| ルーズベルト | ||
| ACTB(ネガティブコントロール) | GACGCAGGATGGカッコッ | ACGCCTCTGGCCGTACCAC |
表2:プライマー配列。
| DNAの回収率が低い | 潜在的な問題 | 可能な解決策 |
| 抗体の品質が悪い | ChIPグレードの抗体を使用してください。 | |
| 抗体が不十分 | クロマチン25 μgあたり1-10 μgのChIP抗体を使用してください。 | |
| インキュベーション時間が不十分 | 免疫沈降ステップを4°Cで一晩実行します。 | |
| 過度に厳しい洗浄 | 洗浄バッファーに500 mMを超える濃度のNaClを使用しないでください。 | |
| 低品質のビーズ | プロテインAまたはGがChIP抗体に適合していることを確認してください。 | |
| ビーズ製品のデータシートに従って、ビーズと抗体の比率を最適化してください。 | ||
| 不十分な始動 | セル番号を正確に決定するために、セルの別のプレートを準備します。 | |
| 見本 | ターゲットが存在量が少ない場合は、使用する細胞の数を増やします。 | |
| 細胞溶解が不十分 | バッファーの組成と溶解時間を最適化して、効率を向上させます。 | |
| 特定の細胞タイプを扱う場合は、Dounceホモジナイザーを使用するなど、溶解ステップ中に機械的な力を加えます。 | ||
| 原子核の分解が起こった | 核の単離が穏やかであることを確認して、クロマチンの完全性とアクセス性を維持します。 | |
| ワイドボアピペットチップを使用して、取り扱い中の核のせん断を最小限に抑えます。 | ||
| サンプルの劣化が発生した | すべてのステップを氷上または4°Cで実行します。 | |
| すべてのバッファーにプロテアーゼ阻害剤を含め、すべてのバッファーが新たに調製されていることを確認してください。 | ||
| 不十分なMNase消化 | MNase濃度(0.1〜5単位など)を滴定して、特定のサンプルタイプに最適な量を決定します。そして、理想的な消化条件を特定するために、さまざまな濃度から始めます。 | |
| MNaseの過剰消化 | 37°Cでさまざまなインキュベーション時間(1〜10分など)で実験し、フラグメンテーションパターンをモニターして、過剰消化せずに十分な消化を確保します。 | |
| リン酸化の乱れ | ホスファターゼ阻害剤カクテルを溶解バッファーに加え、すべてのバッファーが新たに調製されていることを確認します。 | |
| ネガティブコントロールにおける高いバックグラウンド | ビーズへの非特異的結合 | 免疫沈降ステップの前にプレクリアステップを含め、一般に非特異的結合が減少する磁性ChIPグレードのビーズを使用してください。 |
| 不十分な洗浄 | 塩と洗剤の濃度を調整することにより、洗濯の回数または硬化性を高めます。 | |
| クロマチンの不十分な断片化 | フラグメンテーションを最適化して、200-750 bp のフラグメントを実現します。 | |
| 細胞や組織の種類ごとに最適化が必要です | ||
| PCRのトラブルシューティング | インプットサンプルの増幅なし | (1)過度に断片化されている:クロマチンがモノヌクレオソーム長に断片化されている場合、150 bpを超えるアンプリコンではシグナルが減少します。 |
| (2)PCR条件を最適化します。 | ||
| (3)より小さな(<150 bp)領域を増幅するプライマーを設計します。 | ||
| 低解像度 | DNAフラグメントサイズが大きすぎる | フラグメンテーションを最適化して、200-750 bp のフラグメントを実現します。 |
表3:クロマチン免疫沈降のトラブルシューティング。
ディスカッション
環境汚染は、人間の癌の大きな原因です。多くの汚染物質は発がん性があり、癌の発症につながる遺伝的損傷を引き起こす可能性があります40,41。しかし、特定の物質が腫瘍原性であるかどうかを判断することは困難な作業です。発がん性を特定するための迅速で信頼性が高く、費用対効果の高い方法により、科学者は環境汚染物質を効率的にスクリーニングし、ゲノムの安定性への影響を評価できるようになります。本研究では、DSBの特異性の高いマーカーとして広く認識されている修飾ヒストンバリアントγH2A.Xに着目し、ヒストンバリアントH2AがH2Aを形成することでγH2A.Xが形成されます。XはDNA損傷に応答してセリン139でリン酸化されます。
CFSのNativeChIPによってγH2A.Xの存在を検出することにより、環境汚染物質42によって引き起こされるDNA損傷の程度を効果的に評価することができる。CFSでγH2A.Xを定量的に測定する能力は、環境汚染物質が腫瘍形成効果を誘発する可能性を評価するための迅速で高感度かつ信頼性の高い方法を提供します。従来の遺伝毒性アッセイでは、DNA損傷の間接的なマーカー(突然変異、小核形成、細胞形質転換など)を測定することが多いのに対し、ネイティブのγH2AX ChIPでは、特定のゲノム遺伝子座42での切断イベントを正確にマッピングすることができます。これにより、環境汚染物質に対して最も脆弱な地域をより深く理解し、潜在的な発がんメカニズムの特定に役立ちます。
ネイティブChIPを成功させるには、ターゲット領域の効果的なエンリッチメントを確実にするために、いくつかの重要なステップに細心の注意を払う必要があります。以下は、γH2AXのネイティブChIPに関連する重要なステップです。細胞調製:適切な数の細胞から始めます。アフィジコリンなどのDNA損傷誘発剤で細胞を処理し、ポジティブコントロールとして複製ストレスを誘導します。比較のために適切な未処理のネガティブコントロールサンプルを含めます。クロマチン単離:細胞内分画を行い、細胞質成分からの汚染を最小限に抑えながら、クロマチン結合タンパク質を単離します。MNaseの最適化:パイロット実験を実施して、ミクロコッカスヌクレアーゼ(MNase)の濃度とインキュベーション時間を最適化します。その目的は、200〜1000塩基対のクロマチンフラグメントを取得し、ヌクレオソームの完全性を維持しながらChIPの良好な分解能を確保することです。免疫沈降:高品質のChIPグレードのγH2AX抗体とProtein A/Gアガロースを併用することで、非特異的結合を最小限に抑えながら、標的クロマチンを効率的にプルダウンすることができます。洗浄:適切な濃度の塩と界面活性剤を含む緩衝液でビーズを洗浄し、γH2AX結合クロマチンを保持しながら非特異的な相互作用を除去します。溶出とDNA回収:DNAの徹底的な溶出と沈殿により、結合したクロマチンを効率的に回収し、濃縮された標的クロマチンの損失を最小限に抑えます。従来のX-ChIPと比較して、ネイティブのγH2AX ChIPには主に4つの利点があります。まず、細胞内分画を統合することで、偽陽性の相互作用が減少し、ChIPの特異性が向上します。次に、タンパク質-DNA複合体は天然の状態で保存されるため、架橋剤で発生する可能性のある一過性のγH2AX-クロマチン相互作用の安定化が最小限に抑えられます。第三に、固定と時間のかかる逆架橋ステップを省略することで、ワークフロー全体を合理化します。最後に、必要な出発材料が少なくて済み、クロマチンせん断のための高価な装置、例えば、ウォーターバスソニケーター(例えば、Bioruptor)または集束超音波ソニケーター(例えば、Covaris)も必要としない。
Native ChIPには利点がありますが、その制限も認識する必要があります。1つの制限は、クロマチン調製および免疫沈降中のタンパク質再配列の可能性です。この再配列は、理論的にはクロマチン結合タンパク質の相互作用を変化させ、結果にばらつきをもたらす可能性があります。このような再配列の発生を裏付ける直接的な証拠はありませんが、この潜在的なアーティファクトを説明するために適切な実験的対照を含めることが重要です。ネイティブChIPアッセイのもう一つの制限は、MNaseの切断バイアスであり、これが不正確な結果につながる可能性があります。MNaseは、ゲノムのA-Tリッチ領域を優先的に切断するため、ChIPプロセス中にこれらの領域が断片化および濃縮される可能性が高くなります。このバイアスは、A-Tリッチ配列の過剰発現やG-Cリッチ領域の過小表現を引き起こし、MNase消化にアクセスしにくいゲノム領域の偽陰性シグナルを引き起こす可能性があります。目的の遺伝子座がG-Cリッチな場合、MNase消化条件を慎重に最適化する必要があります。あるいは、超音波処理によるクロマチンフラグメンテーションなどの追加のコントロールを組み込むことで、MNaseバイアスの影響を軽減し、結果の信頼性を向上させることができます。
その一定の制限にもかかわらず、ネイティブのγH2AX ChIPは、特定の汚染物質がDNA損傷を誘発する遺伝子座を特定するのに特に有用です。γH2AXに結合したDNA断片を濃縮することにより、研究者は、汚染物質によって引き起こされる複製ストレスの下で壊れやすいゲノムの脆弱な領域、特にCFSをqPCRで特定できます。また、この技術により、研究者は特定の発がん物質への曝露と特定のゲノム領域におけるDNA損傷の形成との間に直接的な関係を確立することができ、汚染物質の遺伝毒性プロファイルと発がん開始におけるそれらの潜在的な役割についての洞察を提供することができる43,44。
トラブルシューティング表(表3)は、DNA収量の低さ、クロマチン断片化の不足、バックグラウンドノイズの高さ、抗体性能の低下など、ChIPアッセイで遭遇する一般的な問題に対する詳細な解決策を示しています。各問題は、プロトコルの信頼性と再現性を確保するために、特定の推奨事項とペアになっています。
ネイティブのγH2AX ChIPは、自動リキッドハンドリングシステムと定量的PCRまたはシーケンシング技術を使用することで、DNA損傷を誘発する薬剤を効率的に特定できるため、毒物学的研究や環境モニタリングにおける貴重なツールとして機能します。さまざまなレベルの環境汚染物質に曝露された集団から採取したサンプルにネイティブのγH2AX ChIPを使用することで、研究者は特定のDNA損傷パターンを特定し、特定の物質への曝露に関連するリスクのレベルを決定することができます。結論として、ネイティブのγH2AX ChIPは、環境汚染物質の発がん性を評価するための大きな利点を提供し、特定のゲノム遺伝子座でのDNA損傷を検出するための迅速で費用対効果が高く、高い特異性アプローチを提供します。ハイスループットスクリーニング、個別化リスク評価、バイオマーカー開発への将来の応用は、環境発がんの理解を深め、がんの予防と介入のための戦略を開発するための貴重なツールとなります。
開示事項
著者には、開示すべき利益相反はありません。
謝辞
この研究は、南中国大学のスタートアップ資金によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| 0.2 µm nitrocellulose membrane | Amersham | 10600011 | |
| Actin B | proteintech | 20536-1-AP | |
| Aphidicolin | MedChemExpress | HY-N6733 | |
| ChIP-grade magnetic Protein A/G beads | ThermoFisher | 26162 | |
| Clarity Western ECL Substrate | Bio-Rad | #1705061 | |
| Glycogen, molecular biology grade | ThermoFisher | Cat. No. R0561 | |
| HRP-conjugated secondary antibody | proteintech | SA00001-2 | |
| hydroxyurea | MedChemExpress | HY-B0313 | |
| Micrococcal Nuclease | NEB | M0247S | |
| normal IgG | Santa Cruz | sc-2025 | |
| Taq Universal SYBR Green Supermix | BioRad | 1725120 | |
| γH2A.X antibody (for ChIP) | Sigma-Aldrich | 05-636 | |
| γH2A.X antibody (for WB) | Cell Signaling | #25955 |
参考文献
- Glover, T. W., Berger, C., Coyle, J., Echo, B. DNA polymerase alpha inhibition by aphidicolin induces gaps and breaks at common fragile sites in human chromosomes. Hum Genet. 67 (2), 136-142 (1984).
- Bignell, G. R., et al. Signatures of mutation and selection in the cancer genome. Nature. 463 (7283), 893-898 (2010).
- Hellman, A., et al. A role for common fragile site induction in amplification of human oncogenes. Cancer Cell. 1 (1), 89-97 (2002).
- Kotzot, D., et al. Parental origin and mechanisms of formation of cytogenetically recognisable de novo direct and inverted duplications. J Med Genet. 37 (4), 281-286 (2000).
- Miller, C. T., et al. Genomic amplification of MET with boundaries within fragile site FRA7G and upregulation of MET pathways in esophageal adenocarcinoma. Oncogene. 25 (3), 409-418 (2006).
- Zack, T. I., et al. Pan-cancer patterns of somatic copy number alteration. Nat Genet. 45 (10), 1134-1140 (2013).
- Gao, G., et al. Common fragile sites (CFS) and extremely large CFS genes are targets for human papillomavirus integrations and chromosome rearrangements in oropharyngeal squamous cell carcinoma. Genes Chromosomes Cancer. 56 (1), 59-74 (2017).
- Thorland, E. C., Myers, S. L., Gostout, B. S., Smith, D. I. Common fragile sites are preferential targets for HPV16 integrations in cervical tumors. Oncogene. 22 (8), 1225-1377 (2003).
- Matovina, M., Sabol, I., Grubisić, G., Gasperov, N. M., Grce, M. Identification of human papillomavirus type 16 integration sites in high-grade precancerous cervical lesions. Gynecol Oncol. 113 (1), 120-127 (2009).
- Yu, T., et al. The role of viral integration in the development of cervical cancer. Cancer Genet Cytogenet. 158 (1), 27-34 (2005).
- Bignell, G. R., et al. Signatures of mutation and selection in the cancer genome. Nature. 463 (7283), 893-898 (2010).
- Thavathiru, E., Ludes-Meyers, J. H., MacLeod, M. C., Aldaz, C. M. Expression of common chromosomal fragile site genes, WWOX/FRA16D and FHIT/FRA3B is downregulated by exposure to environmental carcinogens, UV, and BPDE but not by IR. Mol Carcinog. 44 (3), 174-182 (2005).
- Sirbu, B. M., et al. Analysis of protein dynamics at active, stalled, and collapsed replication forks. Genes Dev. 25 (12), 1320-1327 (2011).
- Barlow, J. H., et al. Identification of early replicating fragile sites that contribute to genome instability. Cell. 152 (3), 620-632 (2013).
- Petermann, E., Orta, M. L., Issaeva, N., Schultz, N., Helleday, T. Hydroxyurea-stalled replication forks become progressively inactivated and require two different RAD51-mediated pathways for restart and repair. Mol Cell. 37 (4), 492-502 (2010).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. J Biol Chem. 273 (10), 5858-5868 (1998).
- Shanbhag, N. M., Rafalska-Metcalf, I. U., Balane-Bolivar, C., Janicki, S. M., Greenberg, R. A. ATM-dependent chromatin changes silence transcription in cis to DNA double-strand breaks. Cell. 141 (6), 970-981 (2010).
- Stiff, T., et al. ATR-dependent phosphorylation and activation of ATM in response to UV treatment or replication fork stalling. EMBO J. 25 (24), 5775-5782 (2006).
- Nelson, J. D., Denisenko, O., Bomsztyk, K. Fast chromatin immunoprecipitation assay. Nuc Acids Res. 34 (5), e2 (2006).
- Dorbic, T., Wittig, B. Isolation of oligonucleosomes from active chromatin using HMG17-specific monoclonal antibodies. Nuc Acids Res. 14 (8), 3363-3376 (1986).
- Dorbic, T., Wittig, B. Chromatin from transcribed genes contains HMG17 only downstream from the starting point of transcription. EMBO J. 6 (8), 2393-2399 (1987).
- Hebbes, T. R., Thorne, A. W., Crane-Robinson, C. A direct link between core histone acetylation and transcriptionally active chromatin. EMBO J. 7 (5), 1395-1402 (1988).
- Hebbes, T. R., Thorne, A. W., Clayton, A. L., Crane-Robinson, C. Histone acetylation and globin gene switching. Nuc Acids Res. 20 (5), 1017-1022 (1992).
- Hebbes, T. R., Clayton, A. L., Thorne, A. W., Crane-Robinson, C. Core histone hyperacetylation co-maps with generalized DNase I sensitivity in the chicken beta-globin chromosomal domain. EMBO J. 13 (8), 1823-1830 (1994).
- Orlando, V. Mapping chromosomal proteins in vivo by formaldehyde-crosslinked-chromatin immunoprecipitation. Trend Biochem, Sci. 25 (3), 99-104 (2000).
- Myers, F. A., Evans, D. R., Clayton, A. L., Thorne, A. W., Crane-Robinson, C. Targeted and extended acetylation of histones H4 and H3 at active and inactive genes in chicken embryo erythrocytes. J Biol Chem. 276 (23), 20197-20205 (2001).
- Litt, M. D., Simpson, M., Recillas-Targa, F., Prioleau, M. N., Felsenfeld, G. Transitions in histone acetylation reveal boundaries of three separately regulated neighboring loci. EMBO J. 20 (9), 2224-2235 (2001).
- Madisen, L., Krumm, A., Hebbes, T. R., Groudine, M. The immunoglobulin heavy chain locus control region increases histone acetylation along linked c-myc genes. Mol Cell Biol. 18 (11), 6281-6292 (1998).
- Clayton, A. L., Hebbes, T. R., Thorne, A. W., Crane-Robinson, C. Histone acetylation and gene induction in human cells. FEBS Lett. 336 (1), 23-26 (1993).
- Tseng, Z., Wu, T., Liu, Y., Zhong, M., Xiao, A. Using native chromatin immunoprecipitation to interrogate histone variant protein deposition in embryonic stem cells. Methods Mol Biol. 1176, 11-22 (2014).
- Durkin, S. G., Glover, T. W. Chromosome fragile sites. Ann Rev Genetics. 41, 169-192 (2007).
- Lee, J. B., Keung, A. J. Chromatin immunoprecipitation in human and yeast cells. Methods Mol Biol. 1767, 257-269 (2018).
- Miyamoto, R., Yokoyama, A. Protocol for fractionation-assisted native ChIP (fanChIP) to capture protein-protein/DNA interactions on chromatin. STAR Protoc. 2 (2), 100404 (2021).
- Mendez, J., Stillman, B. Chromatin association of human origin recognition complex, cdc6, and minichromosome maintenance proteins during the cell cycle: Assembly of prereplication complexes in late mitosis. Mol Cell Biol. 20 (22), 8602-8612 (2000).
- Nowak, D. E., Tian, B., Brasier, A. R. Two-step cross-linking method for identification of NF-κB gene network by chromatin immunoprecipitation. BioTechniques. 39 (5), 715-725 (2005).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nat Protoc. 3 (6), 1101-1108 (2008).
- Lu, X., Parvathaneni, S., Hara, T., Lal, A., Sharma, S. Replication stress induces specific enrichment of RECQ1 at common fragile sites FRA3B and FRA16D. Mol Cancer. 12, 29 (2013).
- Ruijter, J. M., et al. Amplification efficiency: linking baseline and bias in the analysis of quantitative PCR data. Nucl Acids Res. 37 (6), e45 (2009).
- Lyu, X., Chastain, M., Chai, W. Genome-wide mapping and profiling of γH2AX binding hotspots in response to different replication stress inducers. BMC Genomics. 20, 579 (2019).
- Farmer, P. B., et al. Molecular epidemiology studies of carcinogenic environmental pollutants. Effects of polycyclic aromatic hydrocarbons (PAHs) in environmental pollution on exogenous and oxidative DNA damage. Mutat Res. 544 (2-3), 397-402 (2003).
- Phillips, D. H., Arlt, V. M. Genotoxicity: damage to DNA and its consequences. EXS. 99, 87-110 (2009).
- Nitsch, S., Schneider, R. Native ChIP: Studying the genome-wide distribution of histone modifications in cells and tissue. Meth Mol Biol. 2846, (2024).
- Nikitina, T., Wang, D., Gomberg, M., Grigoryev, S. A., Zhurkin, V. B. Combined micrococcal nuclease and exonuclease III digestion reveals precise positions of the nucleosome core/linker junctions: implications for high-resolution nucleosome mapping. J Mol Biol. 425 (11), 1946-1960 (2013).
- Teves, S. S., Henikoff, S. Salt fractionation of nucleosomes for genome-wide profiling. Methods Mol Biol. 833, 421-432 (2012).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved