Method Article
네이티브 γH2A에 의한 일반적인 취약한 부위 파손 캡처. X 치프
요약
당사는 네이티브 γH2A.X 염색질 면역침전(ChIP)을 통해 일반적인 취약 부위 파괴를 검출하는 빠르고 효율적인 방법을 제시합니다. 이 접근법은 기존 γH2A.X ChIP 분석과 관련된 시간과 노동력을 크게 줄이면서 결과의 높은 재현성과 신뢰성을 유지합니다.
초록
외인성 물질에 대한 노출로 인한 복제 스트레스는 구조적 불안정성이 있는 것으로 알려진 게놈 영역인 일반적인 취약 부위에서 DNA 파괴로 이어질 수 있습니다. γH2A.X 인산화는 DNA 이중 가닥 절단에 대한 잘 정립된 마커이기 때문에 γH2A.X 염색질 면역침전(ChIP) 분석은 유전독성 연구에서 강력한 도구 역할을 합니다. 그러나 기존의 γH2A.X ChIP 분석은 종종 노동 집약적이며 여러 단계의 시간이 많이 소요되는 단계를 포함합니다. 이 연구에서는 γH2A.X 관련 복합체를 분리하기 위해 세포 내 분획과 네이티브 ChIP를 결합하는 간단하면서도 효과적인 방법을 제시합니다. 이 접근법은 특이성과 효율성이 향상된 γH2A.X-염색질 상호작용을 분석하는 데 특히 적합합니다. 세포 내 분획법을 사용하면 염색질이 결합되지 않은 물질이 효과적으로 제거되어 정제된 염색질 분획이 생성됩니다. 가벼운 조건에서 후속 미세구균 뉴클레아제(MNase) 분해는 γH2A.X와 관련 단백질 복합체 사이의 생리학적 상호 작용을 보존하면서 염색질 단편화를 가능하게 합니다. 이러한 보존은 DNA 손상 반응 경로에 관여하는 토착 상호 작용 파트너를 연구하는 데 필수적입니다. 이 최적화된 네이티브 ChIP 프로토콜은 기존 γH2A.X ChIP 분석과 관련된 시간과 노동력을 크게 줄여줍니다. 간소화된 절차는 워크플로우를 단순화할 뿐만 아니라 재현성이 높은 결과를 산출하므로 여러 샘플의 고처리량 처리가 필요한 환경에서 특히 유리합니다. 이 방법은 DNA 손상 부위의 정확하고 효율적인 검출이 중요한 게놈 안정성, DNA 복구 및 염색질 생물학에 중점을 둔 연구에 광범위하게 적용할 수 있습니다. 최적화된 프로토콜과 간소화된 단계를 채택한 이 방법은 감도를 개선하고 시료 처리를 최소화하면서 취약한 부위에서 DNA 손상을 감지할 수 있으므로 게놈 안정성 및 DNA 손상 반응에 대한 연구에 유용한 도구입니다.
서문
일반적인 취약 부위(Common Fragile Site, CFSs)는 모든 인간 염색체에서 발견되는 큰 염색체 영역으로, 중기 동안 파괴되기 쉽습니다. 복제 스트레스 하에서, 이 영역에서의 복제는 현저히 지연되어 유사분열진입 1 이전에 완전한 복제를 방해하며, 이는 궁극적으로 부위 특이적 갭과 단절을 초래합니다. CFS는 염색체 불안정성의 핫스팟이며 초기 암 발병 중 염색체 재배열의 주요 원인입니다. 종양 형성 조건에서 자주 나타나는 복제 스트레스는 종양 억제 유전자의 손실과 종양 유전자의 증폭으로 이어질 수 있으며, 이를 집합적으로 복제 수 변이(CNV)라고 합니다2,3,4,5,6. 또한 CFS는 바이러스 통합에 매우 취약하여 암 발병을 더욱 촉진합니다 7,8,9,10. 원발성 종양에 대한 범암 분석 중 CFS 영역에서 종양 억제 유전자의 다중 동형접합 결실이 검출되었습니다. 암에서 가장 흔하게 영향을 받는 CFS는 FRA2F, FRA3B, FRA4F, FRA5H 및 FRA16D11입니다. CFS는 외인성 발암물질이 있는 경우 파손에 특히 취약하다12. 환경 오염 물질의 유해한 발암 효과를 평가하기 위해서는 CFS 파손 발생을 정량화하는 빠르고 신뢰할 수 있는 방법이 필요합니다.
H2A의 인산화. 운동실조, Telangiectasia 및 Rad3-Related Protein (ATR) 또는 Ataxia Telangiectasia Mutated (ATM)에 의한 세린 잔기 139 (γH2A.X)에서의 X는 신호 복제 포크 스톨링13의 핵심 이벤트입니다. γH2A.X는 이중 가닥 절단(DSB) 형성13 이전에 지연된 복제 분기점의 지표 역할을 하여 지연된 부위에 복구 단백질의 효율적인 모집을 촉진하는 유리한 염색질 환경을 만듭니다. 또한, γH2A.X는 포크 붕괴14,15 후 절단 현장을 모집할 수 있으며, 이는 DSB 수리의 주요 역할과 일치합니다. CFS 단절은 암 진행을 유발하는 염색체 이상과 밀접한 관련이 있기 때문에 이러한 단절을 감지하는 것은 종양 형성의 초기 단계를 이해하는 데 도움이 될 수 있습니다. CFS에서 γH2A.X의 존재는 게놈 불안정성의 초기 이벤트를 감지하기 위한 바이오마커로 사용할 수 있습니다. 이 정보는 잠재적인 발암 물질을 식별하고 다양한 외인성 물질에 대한 노출과 관련된 위험을 평가하는 데 도움이 될 수 있습니다. 외인성 제제에 의해 유도된 CFS에서 DNA 절단을 측정함으로써 γH2A.X 염색질 IP(ChIP)는 이러한 제제가 종양 형성의 기저에 있는 메커니즘에 어떻게 기여하는지에 대한 통찰력을 제공할 수 있습니다.
종래의 ChIP(즉, 가교결합된 ChIP, X-ChIP)에서, γH2A.X와 이의 표적 DNA 염기서열의 결합은 가역적인 포름알데히드 가교결합에 의해 안정화된다. 크로마틴은 이후 초음파 처리를 통해 약 500 염기쌍(bp)의 단편으로 전단되고, 생성된 용액은 침전 16,17,18에 의해 파편을 제거합니다. 그런 다음 ChIP 등급의 γH2A.X 항체를 투명화된 염색질 분획에 첨가한 다음 단백질 A/G 아가로스 비드를 첨가하여 γH2A.X 결합 염색질 영역을 농축합니다 16,17,18. 면역 복합체(즉, 비드-항체-γH2A.X 표적 DNA 복합체)를 엄격한 세척 완충액으로 여러 번 세척하여 비특이적으로 결합된 DNA 단편을 제거합니다 16,17,18. 세척 후, 특이적으로 결합된 DNA는 면역 복합체에서 용출됩니다. 그런 다음 포름알데히드 가교를 역전시킨 다음 proteinase K를 사용하여 단백질을 분해한 후 농축된 DNA를 정제하고 농축합니다 16,17,18. γH2A.X 관련 영역을 평가하기 위해 PCR, 정량적 PCR(qPCR) 또는 직접 염기서열분석을 사용합니다 16,17,18. CFS와 같은 특정 영역에서 γH2A.X의 점유는 PCR 또는 qPCR 신호의 강도에 의해 결정되며, 이는 해당 위치에서 결합된 γH2A.X의 양에 비례하여 부위 특이적 DNA 손상 및 복구 이벤트에 대한 통찰력을 제공합니다 16,17,18.
강력한 실험적 접근법임에도 불구하고, X-ChIP는 몇 가지 중요한 한계를 가지고 있다: (i) 고정과 관련된 항체 침전의 비효율성으로 인해, 전형적으로 1 x 107 내지 5 x 107 범위의 많은 수의 세포를 필요로 하며, 이는 실험의 전체 비용을 증가시킨다19; (ii) 포름알데히드 가교 결합 및 후속 DNA 정제를 역전시키는 과정은 시간이 많이 걸리고 노동 집약적이므로 결과의 일관성과 신뢰성을 유지하는 것이 어렵습니다. (iii) 기능적 유의성이 경미한 γH2A.X-DNA 상호작용은 가교 단계가 일시적인 상호작용을 안정화시켜 생물학적으로 관련성이 없을 수 있는 상호작용을 검출할 수 있기 때문에 더 큰 유의성을 갖는 상호작용과 구별되지 않을 수 있다19.
비변성 염색질 면역침전(Native ChIP 또는 N-ChIP)은 생리학적 염 조건에서 비변성 염색질 맥락 내에서 단백질-DNA 상호 작용을 연구하는 데 사용되는 필수 생화학 기술입니다. 그것은 염색질의 공간적, 시간적 조직, 전사 인자 결합 및 히스톤 변형을 설명하는 데 중요한 역할을 했습니다. 네이티브 ChIP는 염색질 생물학 및 후성 유전학의 광범위한 분야에서 오랜 역할을 해왔으며 X-ChIP에 비해 고유한 장점과 한계를 제공합니다. 1980년대 후반에 도입된 이 방법은20 소구균 뉴클레아제(MNase)를 사용한 소화와 같이 세포의 본래 구조를 보존하는 방법으로 세포에서 염색질을 분리하는 것입니다21. 이는 고유한 단백질-DNA 및 히스톤-DNA 접촉을 보존하며, 이는 네이티브 ChIP를 자연적인 염색질 설정에서 히스톤 변형 및 뉴클레오솜 위치를 연구하는 데 특히 적합합니다22. 고분해능 네이티브 ChIP 연구는 염색질을 개별 뉴클레오솜으로 환원시키기 위해 MNase 분해를 사용하는 것을 입증했으며, 이는 더 높은 정확도로 히스톤 변형의 매핑을 용이하게 합니다23. 또한, 화학적 가교가 관련되지 않기 때문에 단백질-DNA 상호 작용을 잘못 나타낼 수 있는 편향 또는 인공물이 발생할 위험이 최소화됩니다24.
포름알데히드 또는 기타 가교제를 사용하여 단백질-DNA 상호작용을 수정하는 X-ChIP와 달리, Native ChIP는 잠재적인 가교 아티팩트를 방지하여 염색질에 대한 보다 현실적인 관점을 제공합니다. 그러나 X-ChIP는 일반적으로 DNA와 조절 단백질 간의 일시적 또는 동적 상호 작용을 감지하는 데 더 적합하지만25, Native ChIP는 히스톤 또는 기타 염색질 결합 단백질26,27과 같은 안정적인 단백질-DNA 상호 작용에 이상적입니다. Native ChIP에 대해 언급된 제한 사항 중 하나는 X-ChIP25의 교차 연결을 통해 안정화되는 경우가 많은 낮은 친화도 또는 일시적인 결합 이벤트를 캡처할 수 없다는 것입니다.
후성유전학(epigenetics)의 중요한 연구는 다양한 생물학적 환경에서 히스톤 변형을 밝히기 위해 네이티브 ChIP를 활용했습니다28. 이러한 노력은 유전자 발현과 염색질 역학을 조절하는 히스톤 변형 패턴인 히스톤 코드를 정의하는 데 결정적이었습니다29. H2A이지만. X는 덜 강하게 연관된 링커 히스톤, 즉 네이티브 H2A입니다. X ChIP 방법은 배아 줄기 세포에 성공적으로 적용되었습니다30. 본 연구에서는 인간 293T 세포에서 γH2A.X의 Native ChIP를 수행하기 위해 염색질 추출 절차를 최적화했습니다(그림 1). 하이드록시우레아(Hydroxyurea)와 아피디콜린(aphidicolin)은 DNA 복제 스트레스, 손상 및 게놈 불안정성을 조사하기 위한 연구에 널리 사용됩니다31. 이번 연구에서는 이러한 약제를 세포에 적용하여 복제 스트레스를 유발하고 CFS에서 DNA 절단을 생성했습니다.
약 1 x 106 - 5 x 106 세포의 출발 물질을 사용하는 이 방법은 (i) 염색질을 분리하기 위한 세포 내 분획, (ii) 염색질 단편화를 위한 소세포 뉴클레아제(MNase) 분해, (iii) 면역침전 및 용출, (iv) 정량적 PCR(qPCR)에 의한 DNA 분석의 4가지 주요 단계로 나눌 수 있습니다. 세포 내 분획 후 ChIP를 수행하면 여러 가지 이점이 있으며 수많은 연구에서 잘 입증되었습니다 32,33,34,35. 이 접근법을 사용하면 염색질이 결합되지 않은 단백질 및 기타 세포 파편을 제거할 수 있어 고도로 정제된 염색질 분획을 얻을 수 있습니다. 면역침전 전에 염색질을 분리함으로써 세포 내 분획은 고유한 염색질 상호작용을 유지하고 염색질 관련 단백질이 아닌 단백질의 배경 잡음을 줄이는 데 도움이 되며, 이는 분석을 위해 염색질 결합 복합체만 유지되기 때문에 보다 특이적이고 신뢰할 수 있는 결과를 얻을 수 있습니다. 또한, 세포 내 분획은 염색질 분해를 위한 보다 온화한 조건을 가능하게 하여 생리학적 단백질-DNA 상호 작용을 보존하고 네이티브 세포 환경 내에서 염색질 역학을 보다 정확하게 표현합니다.
γH2AX의 네이티브 ChIP를 사용하여 일반적인 취약한 부위 파손에 대한 외인성 제제의 영향을 측정하는 것은 암 연구에 상당한 잠재력을 가지고 있습니다. 이 기술을 사용하면 환경 발암 물질에 노출되어 유발된 DNA 손상을 감지할 수 있으며, 오염 물질이 게놈 불안정성과 암 발병에 기여하는 분자 메커니즘에 대한 통찰력을 제공합니다. 이 방법은 천연 염색질 컨텍스트를 보존함으로써 발암 물질 노출과 관련된 DNA 손상 패턴의 정확한 평가를 용이하게 하여 환경 위험 평가 및 오염 기반 종양 형성 연구를 지원합니다.
프로토콜
1. 세포 수확
- 각각 4mL의 완전한 DMEM 배지를 포함하는 4개의 6cm 접시에 약 5 x 105 HEK 293T 세포를 시드합니다.
- 24시간 후, 한 접시에 2μL의 1mM 아피디콜린( 재료 표 참조) 원액(최종 농도 0.5μM)을 처리하고 다른 접시에 20μL의 1M 하이드록시우레아( 재료 표 참조) 원액(최종 농도 5mM)을 처리하여 복제 스트레스를 유발합니다. 나머지 두 접시에 DMSO를 추가하여 대조군으로 사용합니다.
- 치료 24시간 후 배양 배지를 폐기합니다. 하나의 6cm 플레이트는 일반적으로 60%-70% 합류점에서 약 2 x 106개의 세포를 생성합니다.
- 얼음처럼 차가운 PBS 5mL로 각 접시를 2x 헹굽니다. 세포 스크레이퍼를 사용하여 세포를 분리하고 세포 현탁액을 4개의 개별 1.5mL 튜브로 옮깁니다. P1000 피펫으로 위아래로 부드럽게 피펫팅하여 세포 덩어리를 분리합니다.
- 500 x g 에서 4°C에서 5분 동안 세포를 원심분리한 다음 상층액을 버립니다. 얼음 위에 세포를 놓습니다.
2. Subcellular 분획법
- 세포 펠릿을 500 μL의 갓 준비된 저온 Buffer A( 표 1 참조)에 재현탁시켜 부드러운 피펫팅으로 세포 덩어리를 완전히 분리합니다.
- 얼음에서 용해물을 5-10분 동안 배양합니다. 현미경으로 용해 진행을 확인하여 세포 용해가 완료되었는지 확인합니다.
- 용해물의 작은 부분 표본(약 5 - 10 μL)을 깨끗한 현미경 슬라이드에 놓습니다. 오염을 방지하기 위해 커버 슬립으로 덮으십시오.
- 적절한 배율(예: 20x - 40x)의 광학 현미경을 사용하여 세포나 파편을 시각화합니다. un-lysed control 샘플과 비교하여 온전한 세포와 용해된 물질을 구별하십시오.
참고: 적절하게 용해된 샘플에는 뚜렷한 세포 윤곽이 없고 확산 염색질 또는 세포 물질만 있습니다. 용해물을 명확하게 관찰하도록 초점을 조정합니다. 필요한 경우 특정 세포 유형으로 작업할 때 Dounce 균질기를 사용하는 것과 같이 용해 단계에서 기계적 힘을 가합니다.
- 세포가 완전히 용해된 후 500 x g 에서 4°C에서 5분 동안 원심분리기. 상층액을 조심스럽게 버리십시오. 광폭 오리피스 피펫 팁을 사용하여 500μL의 저온 Buffer A에 핵 펠릿을 재현탁합니다.
참고: 와이드 오리피스 팁은 전단력을 최소화하고 염색질과 같은 섬세한 샘플을 보호하는 데 도움이 됩니다. 날카로운 칼날로 표준 팁의 끝을 잘라 넓은 구멍을 만드십시오. - 500 x g 에서 4°C에서 5분간 원심분리기. 상층액을 조심스럽게 버리십시오.
- 인큐베이터를 37°C로 예열하고 정지 버퍼(100mM EDTA, pH 8.0; 표 1).
- Micrococcal Nuclease(MNase, Table of Material 참조) 농도와 배양 시간을 미리 최적화합니다.
- 40μL의 염색질 샘플을, 서로 다른 MNase 농도와 배양 시간을 테스트하기 위해 여러 개의 동일한 부분 표본으로 나눕니다.
- 다양한 MNase 농도(예: 반응당 0.0625 U, 0.125 U, 0.25 U, 0.5 U, 1 U, 2 U, 4 U, 8 U)를 사용하고 여러 배양 시간(예: 2, 5, 10 및 15분)을 테스트합니다.
- 다양한 농도의 MNase를 포함하는 MNase Buffer( 표 1 참조)를 염색질 분취액에 추가하고 지정된 시간 동안 37°C에서 샘플을 배양합니다.
- 원하는 배양 시간 직후 1/4 부피의 정지 완충액(최종 농도: 20mM EDTA)을 추가하여 반응을 종료합니다.
- 페놀/클로로포름/이소아밀 알코올 추출 방법을 사용하여 분해된 염색질 샘플에서 DNA를 분리합니다.
- 추출된 DNA를 1.5% 아가로스 겔에 실행하여 분해 패턴을 시각화합니다: 과소 소화는 고분자량 대역을 나타냅니다(그림 2, 열 1-4). 과다 분해는 번짐 또는 매우 짧은 단편을 생성하며(그림 2, 6-8열), 최적의 절단은 명확한 뉴클레오솜 래더 패턴을 생성합니다(그림 2, 5열, 예: 모노, 디, 트리뉴클레오솜).
- 과도한 소화 없이 원하는 뉴클레오솜 분해능을 생성하는 조건을 식별합니다.
참고: CaCl2 는 MNase 활성의 보조 인자로 작용합니다. CaCl2 농도를 1mM에서 5mM 사이로 조정하여 분해를 최적화합니다.
- 넓은 오리피스 팁으로 5 - 10회 피펫팅하여 100 μL의 MNase Buffer로 손상되지 않은 핵을 부드럽게 재현탁합니다. 미리 결정된 양의 MNase를 샘플에 즉시 추가합니다(1.25 U MNase/100 μL MNase Buffer).
참고: 여러 샘플로 작업할 때 과도한 소화를 방지하기 위해 각 샘플을 개별적으로 분해하십시오. - 튜브를 회전기에 놓고 37°C에서 5분 동안 배양합니다. 즉시 튜브를 얼음으로 되돌리고 EDTA를 최종 농도 20mM에 첨가하여 MNase 분해를 종료하고 볼텍싱으로 혼합합니다.
- 각 샘플에 500 μL의 Buffer B( 표 1 참조)를 추가하고 5x - 10x 위아래로 피펫팅하여 철저히 혼합합니다. 얼음 위에서 5분 동안 배양하여 단백질을 가용화합니다.
참고: Buffer B의 소금과 세제는 약한 염색질 결합 단백질을 해리하고 면역침전을 위해 에피토프를 노출시키는 데 도움이 됩니다. - 4 °C에서 5 분 동안 최대 속도로 원심 분리하여 불용성 물질을 펠렛화합니다. 투명 상층액을 비변성 염색질 분획으로 표시된 새로운 1.5mL 튜브로 옮깁니다. 샘플은 -80°C에서 보관하거나 염색질 단편화의 효율성을 검증하는 데 사용할 수 있습니다.
참고: 잦은 동결-해동 주기는 관심 있는 단백질-DNA 상호 작용을 방해할 수 있으므로 피하십시오. 가능하면 동결-해동 주기를 최소화하십시오.
3. 염색질 단편화(chromatin fragmentation) 검증
- 각 샘플에서 10μL의 상층액을 새 1.5mL 튜브로 분주합니다. 증류수 20 μL와 페놀/클로로포름/이소아밀 알코올 30 μL(25:24:1)와 혼합합니다.
- 튜브를 단단히 닫고 15-30초 동안 격렬하게 소용돌이칩니다. 20,000 x g (또는 원심분리기의 최대 속도)에서 4°C에서 10분 동안 원심분리기. 원심분리 후, 세 개의 뚜렷한 층이 관찰됩니다: 투명한 상단 층, 흰색 중간 층 및 노란색 하단 층.
- 20 μL의 상부 수성상(DNA 포함)을 새 튜브로 조심스럽게 옮깁니다. 100V에서 30분 동안 1.5% 아가로스 젤에서 정제된 DNA를 분리하고 분해 패턴을 시각화합니다. 염색질 단편의 크기가 주로 200개에서 1000개 염기쌍 사이인지 확인합니다.
참고: 적절한 염색질 단편 크기는 native ChIP의 성공에 매우 중요하며 효소 단위, 배양 시간 및 CaCl2 농도를 포함한 MNase 처리 조건에 따라 달라집니다. MNase 분해 효율은 세포 유형과 수에 따라 달라질 수도 있습니다. 그림 2 (레인 5)에 표시된 염색질 단편화 패턴이 이 ChIP 분석에 권장됩니다.
4. 면역침전
- 각 샘플에서 분해된 염색질 20μL를 새 1.5mL 튜브에 분취하고 180μL의 용리 버퍼와 혼합합니다( 표 1 참조). 이 튜브에 입력 샘플로 라벨을 붙이고 -20 °C에서 보관합니다.
- ChIP를 위해 400μL의 염색질 샘플을 다른 1.5mL 튜브로 옮깁니다.
- DMSO 처리된 시료 1개, Aphidicolin 처리된 시료 1개, hydroxyurea 처리된 시료 1개에 γH2A.X 항체( 재료 표 참조)를 추가합니다. 동일한 양의 정상 IgG( 재료 표 참조)를 ChIP 분석에 대한 음성 대조군으로 다른 DMSO 처리된 샘플에 추가합니다.
참고: 여기서 1μg의 1차 항체는 일반적으로 400μL의 염색질에 사용됩니다(즉, 항체 최종 농도는 2.5μg/mL임). 그러나, 최적량은 상이한 γH2A.X 항체에 대해 경험적으로 결정되어야 한다. - ChIP 튜브를 4°C의 회전기에 놓고 최소 5시간 동안 또는 가급적이면 밤새 배양합니다.
- 한편, 100μL의 ChIP 등급 자성 단백질 A/G 비드( 재료 표 참조)를 새로운 1.5mL 튜브에 분취합니다. 비드의 정확한 측정을 보장하기 위해 넓은 오리피스 팁과 피펫을 천천히 사용하십시오. 튜브를 마그네틱 스탠드에 1분 이상 놓고 액체를 조심스럽게 버리십시오.
- 0.5% BSA를 함유한 1x PBS 1mL에 비드를 재현탁합니다. 4°C에서 약 4시간 동안 회전합니다. 튜브를 마그네틱 스탠드에 최소 1분 동안 놓고 상등액을 버립니다.
- 0.5% BSA가 함유된 1x PBS 1mL로 비드를 다시 세척합니다. 튜브를 마그네틱 스탠드에 1분 동안 놓고 마그네틱 비드를 펠릿화한 다음 상층액을 버립니다.
참고: 4.5-4.7단계는 자성 비드에 대한 항체의 비특이적 결합을 줄이기 위해 비드를 사전 코팅하는 것입니다. - 넓은 오리피스 팁을 사용하여 100μL의 Buffer B에 사전 코팅된 비드를 재현탁합니다. 사전 코팅된 마그네틱 비드 현탁액 25μL를 각 ChIP 시료 튜브에 추가합니다. 4°C에서 2시간 동안 회전합니다.
- ChIP 튜브를 마그네틱 스탠드에 놓고 비드가 튜브 측면에 완전히 부착되고 용액이 투명해질 때까지 기다립니다.
- 마그네틱 비드를 방해하지 않고 투명한 상등액을 버리십시오. 1mL의 세척 버퍼( 표 1 참조)로 비드를 재현탁하고 4°C에서 10분 동안 회전합니다.
- 튜브를 마그네틱 스탠드에 다시 놓고 용액이 명확해질 때까지 기다립니다. 세탁 버퍼를 버리십시오. 총 4회 세탁을 반복합니다.
- 최종 세척 후 세척 버퍼를 버리고 400 x g 에서 튜브를 30°C에서 30초 동안 잠시 원심분리하여 잔류 액체를 회전시킵니다. 튜브를 마그네틱 스탠드에 다시 놓고 튜브 바닥에 남아 있는 액체를 조심스럽게 제거합니다.
5. 용출 및 DNA 침전
참고: 항체 효율은 배치에 따라 다를 수 있습니다. 웨스턴 블롯 분석을 통해 면역침전된 시료를 확인하여 새로운 항체의 결합 친화도를 확인하는 것이 중요합니다.
- 아래와 같이 웨스턴 블롯(WB)을 사용하여 ChIP 항체 풀다운 효율을 확인합니다.
- 분석을 위해 ChIP 샘플의 작은 부분 표본(즉, 일반적으로 ChIP 샘플의 10%)을 채취합니다. 비교를 위해 입력 염색질(사전 면역침전) 및 음성 대조군(예: IgG 풀다운)을 포함합니다.
- 20μL의 1x SDS-PAGE 로딩 버퍼( 재료 표 참조)를 95°C에서 5분 동안 가열하여 항체 결합 비드에서 단백질을 용리합니다.
- IP 샘플, 입력 및 대조군을 15% SDS-PAGE 젤에 로드합니다. 젤을 실행합니다.
- 습식 또는 반건식 전달 시스템을 사용하여 단백질을 0.2μm 니트로셀룰로오스( 재료 표 참조) 또는 PVDF 멤브레인으로 옮깁니다.
- TBST( 표 1 참조)에서 5% 무지방 우유 또는 BSA로 멤브레인을 실온에서 1시간 동안 차단하여 비특이적 결합을 방지합니다.
- 차단 버퍼에 희석된 γH2A.X( 재료 표 참조)에 대한 1차 항체와 함께 멤브레인을 실온에서 1-2시간 동안 또는 4°C에서 밤새 배양합니다.
- 결합되지 않은 항체를 제거하기 위해 TBST로 멤브레인을 3x 세척합니다. 실온에서 1시간 동안 HRP 접합 2차 항체( 재료 표 참조)로 멤브레인을 배양합니다. 멤브레인을 다시 세척하여 과도한 2차 항체를 제거합니다.
- 화학발광 기질을 사용하여 멤브레인을 현상하고 이미저로 신호를 시각화합니다. IP, 입력 및 제어 레인 간의 신호 강도를 비교하여 풀다운의 효율성과 특이성을 평가합니다.
참고: IP 레인의 표적 단백질에 해당하는 밴드는 성공적인 항체 풀다운을 확인합니다. 이 접근법을 사용하면 ChIP 실험 중에 표적 단백질을 포획하는 항체의 효능을 평가할 수 있습니다.
- 나머지 ChIP 샘플 각각에 50μL의 용리 버퍼( 표 1 참조)를 추가합니다. 튜브를 써머 믹서에 놓고 실온에서 15분 동안 흔듭니다.
- 튜브를 마그네틱 홀더에 최소 1분 동안 놓습니다. 용출액을 새 튜브에 모읍니다. 1x를 반복하고 동일한 튜브에 용리액을 수집합니다.
- 각 ChIP 용리 샘플에 100μL의 용리 버퍼를 추가하고 각 입력 샘플에 180μL의 용리 버퍼를 추가합니다.
- 각 샘플에 200μL의 페놀/클로로포름/이소아밀 알코올(25:24:1)을 첨가하고 격렬하게 소용돌이칩니다. 4°C에서 10분 동안 20,000 x g (또는 최대 속도)로 시료를 원심분리합니다.
- 각각의 새로운 1.5mL 원심분리 튜브에 3M 아세트산나트륨 19μL(NaOAc, pH 5.2, 표 1 참조)와 글리코겐 용액 2μL(20mg/mL, 재료 표 참조)를 추가합니다.
- 원심분리 후 상부 수성층(약 190μL)을 NaOAc와 글리코겐이 함유된 튜브에 조심스럽게 옮기고 볼텍싱으로 혼합합니다.
- 500μL의 100% 에탄올과 와류를 추가합니다. 샘플을 -20°C에서 최소 2시간 또는 하룻밤 동안 배양하여 DNA를 침전시킵니다.
- 튜브를 20,000 x g (또는 최대 속도)로 4°C에서 10분 동안 원심분리합니다. 상등액을 버리고 흰색 펠릿을 방해하지 않도록 주의하십시오. 펠릿을 1mL의 70% 에탄올에 재현탁시키고 완전히 와류시킵니다.
- 4°C에서 5분 동안 20,000 x g (또는 최대 속도)로 튜브를 원심분리합니다. 상층액을 조심스럽게 제거하십시오. 튜브를 다시 짧게 원심분리하여 잔류 에탄올을 스핀다운합니다. P20 피펫을 사용하여 에탄올을 조심스럽게 제거합니다. DNA 펠릿을 2-3분 동안 자연 건조시킵니다.
참고: 펠릿을 과도하게 건조하면 DNA가 재용해되기 어려울 수 있으므로 피하십시오. - ChIP 샘플의 경우 400μL의 TE 버퍼에 DNA를 재현탁합니다( 표 1 참조). Input DNA의 경우 1000μL의 TE 버퍼에 재현탁합니다. 용출된 시료는 이제 -20°C에서 보관할 수 있습니다.
6. qPCR 정량화
- 각 샘플에 대해 기술적 삼중과 함께 상용 키트( 재료 표 참조)를 사용하여 qPCR을 수행합니다. 증폭의 특이성을 보장하기 위해 용융 곡선 분석을 수행하여 단일 특이적 PCR 산물의 존재를 확인합니다36.
- 데이터 분석
참고: 상대 정량화 분석에서 테스트 샘플은 대조군 샘플에 대한 접힘 변화로 표현됩니다(정상 정제된 IgG 또는 모의 IP를 사용하여 면역침전). 면역침전된 단백질(음성 유전자좌)에 의해 점유되지 않는 것으로 알려진 DNA 유전자 자리는 알려진 점유된 양성 대조군 DNA 유전자 자리와 비교하여 참조 유전자로서 이러한 방식으로 사용될 수 있습니다36.- 아래 공식을 사용하여 각 ChIP에 대한 입력 비율을 계산합니다.
%입력 = 2(-ΔCt[정규화된 ChIP]) - 아래 공식을 사용하여 음의 궤적에 대한 ΔCt 값에서 양의 궤적에 대해 얻은 ΔCt 값을 빼서 양의 궤적 ΔCt 값을 음의 궤적(ΔΔCt)으로 정규화합니다.
(ΔΔCt = ΔCt긍정적 - ΔCtnegative) - 아래 공식을 사용하여 음성 유전자 자리에 대한 ChIP DNA의 양성 유전자 자리 염기서열의 폴드 농축을 계산합니다.
폴드 농축 =2ΔΔCt
분석에 사용된 qPCR 프라이머의 염기서열은 표 2에 나와 있습니다. FRA3B 및 FRA16D37의 게놈 조직은 그림 3A, B에 나와 있습니다.
- 아래 공식을 사용하여 각 ChIP에 대한 입력 비율을 계산합니다.
- 통계 분석
- 스튜던트 쌍체 t-검정을 사용하여 결과를 통계적으로 분석합니다. p-값이 ≤0.05이면 통계적으로 유의한 것으로 간주되며, 이는 관측된 차이가 무작위 변동38에 기인하지 않을 가능성이 있음을 나타냅니다.
결과
염색질 단편의 크기는 항체 결합을 위한 DNA 영역의 접근성에 직접적인 영향을 미치기 때문에 Native ChIP의 성공에 매우 중요합니다. 염색질 단편화를 위한 최적의 MNase 농도를 결정하기 위해 다양한 농도의 MNase(즉, 반응당 0.0625 U, 0.125 U, 0.25 U, 0.5 U, 1 U, 2 U, 4 U, 8 U)와 40 μL의 분리된 핵을 포함하는 일련의 마이크로 원심분리 튜브를 준비했습니다. 각 반응은 다양한 염색질 단편 크기를 얻기 위해 37°C에서 5분 동안 배양했습니다. MNase 분해 결과는 그림 2에 나와 있습니다.
그림 2에서 볼 수 있듯이, MNase의 농도가 높을수록 염색질이 더 광범위하게 분해되어 단뉴클레오솜 단편이 우세하게 나타났습니다(그림 2, 1, 2, 3열). 대조적으로, 낮은 MNase 농도에서는 대부분의 염색질 단편이 더 커서 종종 1kb를 초과했으며(그림 2, 6, 7, 8번 열), 이는 다운스트림 응용 분야에 대한 분해가 충분하지 않음을 나타냅니다. MNase 농도가 1.6 단위이고 분해 시간이 5분인 경우, 주로 200 - 1000 염기쌍 범위의 염색질 단편을 얻었습니다(그림 2, 레인 5). 이 단편 크기는 특정 항원결정기에 대한 접근성을 유지하면서 염색질 관련 DNA의 효율적인 농축을 보장하기 때문에 Native ChIP에 이상적입니다. 적절한 단편 크기는 효율적인 면역침전 및 정량적 PCR 또는 염기서열분석과 같은 다운스트림 분석을 용이하게 하여 고해상도로 단백질-DNA 상호 작용을 검출하는 데 적합합니다.
항체의 결합 친화도는 Native ChIP의 성공에 중요한 요소입니다. 항체가 표적 항원결정기에 효율적이고 특이적으로 결합하도록 하는 것은 신뢰할 수 있는 결과를 달성하는 데 필수적입니다. 따라서 항체의 결합 효율을 사전에 확인하는 것이 중요합니다. γH2A.X 면역침전의 효과를 확인하기 위해 웨스턴 블롯(WB) 분석을 실시했습니다.
웨스턴 블로팅(western blotting)을 사용하여 DMSO 처리된 세포(대조군), 아피디콜린 처리된 세포, 하이드록시우레아 처리된 세포(복제 스트레스) 간의 γH2A.X 수준을 비교했습니다. 그림 4A (입력)에서 볼 수 있듯이, γH2A.X 수치는 DMSO 처리된 세포에 비해 아피디콜린 처리된 세포에서 유의하게 높았습니다. 이러한 γH2A.X의 증가는 아피디콜린에 의한 복제 스트레스 유도와 일치하며, 이는 이중 가닥 절단 형성 증가 및 후속 γH2A.X 인산화를 유발합니다. ChIP를 수행한 후 IgG 대조군과 γH2A.X IP 샘플에서 γH2A.X 농축을 비교하여 면역침전의 특이성을 검증했습니다. IgG 제어는 검출 가능한 γH2A.X를 풀다운하지 않았으며, 이는 최소한의 비특이적 결합을 나타냅니다(그림 4B, N-ChIP 및 X-ChIP). 대조적으로, γH2A.X IP 샘플은 aphidicolin 처리 세포에서 γH2A.X를 성공적으로 농축하여 γH2A.X에 대한 항체의 효율적이고 특이적인 결합을 입증했습니다(그림 4, N-ChIP 및 X-ChIP). 이러한 결과는 γH2A.X 항체가 Native ChIP 실험에 사용하기에 충분한 결합 친화도와 특이성을 가지고 있음을 확인시켜 γH2A.X 결합 염색질 영역의 신뢰할 수 있는 검출 및 농축을 가능하게 합니다. 이 검증 단계는 후속 ChIP 분석의 품질과 정확성을 보장하는 데 필수적입니다.
복제 스트레스에 대한 반응으로 DNA에 대한 γH2A.X 결합의 특이성을 평가하기 위해 CFS(Common Fragile Sites) FRA16D 및 FRA3B를 사용했습니다. FRA16D 및 FRA3B는 복제 스트레스 조건에서 불안정해지기 쉬운 잘 알려진 CFS 영역이므로 γH2A.X/DNA 상호 작용을 검증하는 데 이상적인 후보입니다. 그림 3A,B는 분석에 사용된 특정 qPCR 프라이머와 함께 이 두 CFS의 게놈 조직을 보여줍니다.
γH2A.X가 복제 스트레스에 대한 반응으로 이러한 CFS 영역과 연관되어 있는지 확인하기 위해 대조군(DMSO 처리), 아피디콜린 처리 세포 및 하이드록시우레아 처리 세포에서 ChIP-qPCR 분석을 수행했습니다. qPCR 결과는 DMSO로 처리된 대조군 세포에서 γH2A.X가 FRA16D 또는 FRA3B 유전자 자리에서 농축되지 않았음을 보여주었으며, 이는 이러한 영역에서 상당한 DNA 손상 또는 복제 스트레스가 없음을 시사합니다(그림 3C). 그러나 DNA 중합효소를 억제하여 복제 스트레스를 유발하는 아피디콜린(aphidicolin) 또는 하이드록시우레아(hydroxyurea)로 처리된 세포에서 FRA16D와 FRA3B 모두에서 γH2A.X의 상당한 농축이 관찰되었습니다(그림 3C). 이는 γH2A.X가 특히 DNA 복제 스트레스에 대한 반응으로 이러한 CFS 영역에 모집되었음을 나타냅니다. 이러한 결과는 CFS에서 γH2A.X의 점유가 복제 스트레스에 의해 유발된다는 것을 보여주며, DNA 손상 반응 및 복제 포크 지연의 마커로서의 역할을 뒷받침합니다. 아피디콜린 투여 중 FRA16D 및 FRA3B에 γH2A.X의 특이적 동원은 ChIP 분석의 특이성을 더욱 입증하고 γH2A.X가 취약한 부위의 DNA 손상 및 복제 스트레스에 대한 신뢰할 수 있는 지표임을 확인합니다.
네이티브 ChIP와 가교결합된 ChIP(X-ChIP)의 효율성을 비교하기 위해 Lyu et al.39에 설명된 프로토콜에 따라 동일한 시작 수의 셀을 사용하여 γH2A.X X-ChIP를 수행했습니다. FRA16D에 대한 γH2A.X 동원은 그림 3D와 같이 아피디콜린과 하이드록시우레아로 처리함으로써 증가하였다. 그러나 X-ChIP에서 FRA16D의 γH2A.X의 상대적 농축은 네이티브 ChIP에서 관찰된 것보다 현저히 낮았으며, 이는 이러한 조건에서 X-ChIP의 효율이 감소했음을 나타냅니다. 또한, FRA3B에서 γH2A.X 농축의 증가는 통계적으로 유의하지 않았으며, 이는 제한된 수의 세포로 작업할 때 X-ChIP의 효율성이 떨어진다는 추가 증거를 제공합니다. 이러한 결과는 샘플 크기가 제한될 때 native ChIP가 취약한 부위에서 γH2A.X 농축을 검출하는 데 더 적합할 수 있음을 시사합니다. 스튜던트 쌍체 t-검정을 사용하여 결과를 통계적으로 분석합니다. p-값이 ≤ 0.05이면 통계적으로 유의한 것으로 간주됩니다.

그림 1:네이티브 γH2A.X ChIP 분석을 수행하기 위한 W orkflow. (i) 세포를 배양하고 γH2A.X 신호전달을 유도하기 위해 특정 처리(예: 하이드록시우레아와 같은 DNA 손상제)를 실시합니다. (ii) 처리된 세포는 처리를 위해 수집됩니다. (iii) 세포를 분획하여 염색질 결합 γH2A.X를 분리하여 다른 세포 구성 요소와 분리합니다. (iv) 소구균 뉴클레아제(MNase) 분해를 수행하여 염색질을 뉴클레오솜 크기의 조각으로 단편화합니다. (v) 염색질 단편의 크기는 아가로스 겔에서 샘플을 실행하여 단편이 ChIP에 적합한 길이인지 확인하여 검증합니다. (vi) γH2A.X에 특이적인 항체는 γH2A.X로 표시된 염색질 단편을 면역침전시키는 데 사용됩니다. (vii) 단백질 A/G 비드는 항체-염색질 복합체를 끌어내리는 데 사용되어 γH2A.X 결합 DNA를 농축합니다. (viii) γH2A.X 표시 DNA 단편을 용출 및 정제하여 다운스트림 분석을 위해 준비합니다. (ix) 다운스트림 분석: qPCR 및 염기서열분석 및 데이터 분석. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2: MNase 분해 조건을 최적화하기 위한 분석. 염색질 단편은 다양한 MNase 분해 조건을 사용하여 HEK 293T 세포에서 준비되었습니다. 단편화된 염색질로부터 생성된 정제된 DNA를 2% 아가로스 겔에서 분리하고 100V에서 30분 동안 실행했습니다. 레인 1 - 8은 MNase의 농도가 증가하여(즉, 반응당 0.0625 U, 0.125 U, 0.25 U, 0.5 U, 1 U, 2 U, 4 U, 8 U) 처리된 샘플을 나타내며, 염색질 분해의 구배를 보여줍니다. DNA ladder(M으로 표시)는 크기 참조를 위해 포함되었습니다. DNA 단편은 Midori Green Advance DNA 염색 용액을 사용하여 시각화되었습니다. 라벨링된 밴드는 모노뉴클레오솜 크기의 DNA 단편에 해당하며, 각 MNase 농도에서 분해 효율을 강조합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
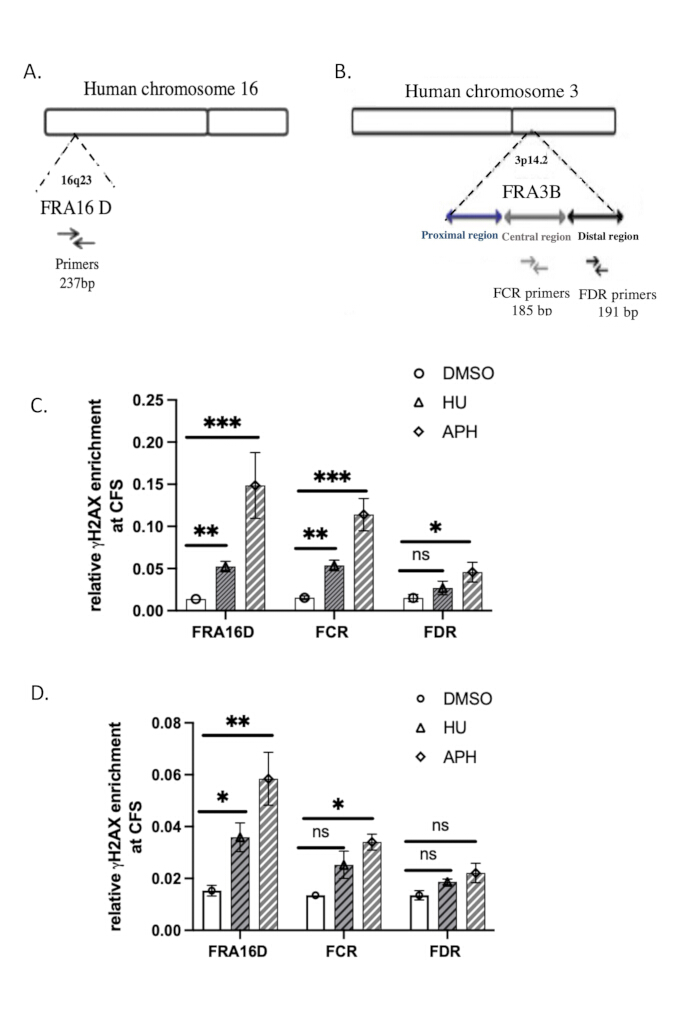
그림 3: γH2A.X ChIP의 qPCR 검증. (A) FRA16D의 게놈 조직 다이어그램. (B) FRA3B 영역의 게놈 조직 다이어그램. FRA3B 유전자 자리 내 FRA16D, 원위(FDR) 및 중심(FCR) 영역의 qPCR 분석에 사용되는 프라이머 세트가 표시됩니다. (C) 일반적인 취약 부위(CFS), 특히 FRA3B 및 FRA16D에서 γH2A.X의 상대적 농축을 네이티브 ChIP를 사용하여 평가한 후 아피디콜린 및 하이드록시우레아로 처리한 후 qPCR 분석을 수행했습니다. (D) 일반적인 취약 부위(CFS), 특히 FRA3B 및 FRA16D에서 γH2A.X의 상대적 농축을 X-ChIP를 사용하여 평가한 후 아피디콜린 및 하이드록시우레아로 처리한 후 qPCR 분석을 수행했습니다. p < 0.001로 통계적으로 유의한 결과를 나타냅니다. **는 p < 0.01을 나타내고 *는 p < 0.05를 나타내며 t-검정에 의해 결정됩니다. n = 3입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
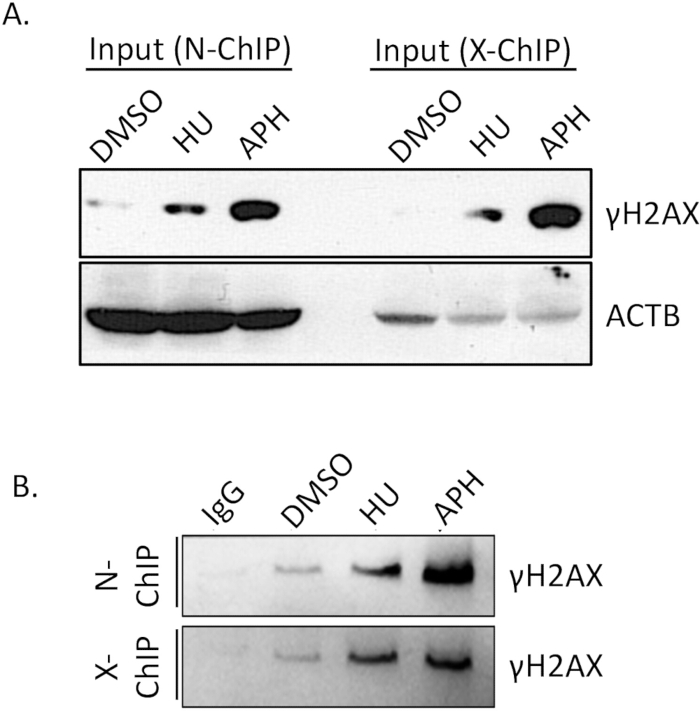
그림 4: 웨스턴 블롯을 사용한 ChIP 항체 풀다운 효율 검증. 총 샘플에서 ChIP 샘플의 10%를 입력 대조군과 함께 15% SDS-PAGE 겔에서 분리하여 폴리펩티드를 분리했습니다. 전기영동 후, 분리된 단백질을 표준 전달 프로토콜을 사용하여 0.22μm PVDF 멤브레인으로 전달했습니다. 그런 다음 γH2A.X 및 ACTB를 표적으로 하는 특정 1차 항체로 멤브레인을 순차적으로 조사한 다음 적절한 HRP 접합 2차 항체를 조사했습니다. 이후 화학발광 검출을 사용하여 신호를 시각화하여 ChIP 분석에서 항체 특이성 및 풀다운 효율을 평가할 수 있었습니다. (A) ACTB를 로딩 제어로 사용하는 DMSO 처리 세포(대조군), 아피디콜린 처리 및 하이드록시우레아 처리 세포(복제 스트레스) 사이의 γH2A.X 수준. (B) IgG 대조군 및 IP 샘플의 γH2A.X 농축. 약어 : APH = 아피디콜린, HU = 하이드록시우레아. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 버퍼 A(1X) | |
| 시약 | 작업 집중 |
| 파이프 pH 6.8 | 10 밀리미터미터 |
| 염화나트륨 | 100 밀리미터미터 |
| 재질 보기 MgCl2 | 3 밀리엠 |
| EGTA 산도 7.6 | 1 밀리엠 |
| -20°C에서 최대 1년 동안 보관 | |
| MNase 버퍼(1X) | |
| 시약 | 작업 집중 |
| 트리스-HCl, pH 7.5 | 50 밀리미터. |
| 염화칼슘2 | 1 밀리엠 |
| 재질 보기 MgCl2 | 4 밀리미터의 |
| -20°C에서 최대 1년 동안 보관 | |
| 버퍼 B(1X) | |
| 시약 | 작업 집중 |
| 인산나트륨 pH 7. 0 | 20 밀리미터미터 |
| 피로인산나트륨10. H2O | 30 밀리미터. |
| 케이클 | 250 밀리미터미터 |
| EDTA, pH 8.0 | 5 밀리미터미터 |
| 글리세롤 | 10% |
| 트리톤 X-100 | 0.10% |
| 프로테아제 억제제 칵테일 | 1배 |
| 포스파타제 억제제 칵테일 | 1배 |
| 디티(DTT) | 0.5 밀리미터. |
| -20°C에서 최대 1년 동안 보관 | |
| 용리 버퍼 | |
| 시약 | 작업 집중 |
| 나HCO3 | 50 밀리미터. |
| 증권 시세 표시기 | 1% |
| 갓 준비한 제품 | |
| 세척 버퍼 | |
| 시약 | 작업 집중 |
| MNase 버퍼 | 엑스 0.5 |
| 버퍼 B | 엑스 0.5 |
| 신선하게 준비된 | |
| 버퍼 중지 | |
| 에디타 | 100mM, pH8.0 |
| 3 M 아세트산 나트륨, pH 5.2 | |
| 408.24 g 아세트산 나트륨 | |
| pH 5.2로 아세트산을 사용한 pH를 조절합니다. | |
| 볼륨을 1L로 조정 | |
| 여과 또는 오토클레이빙으로 완충액을 살균합니다. | |
| TBST 완충액 | |
| 트리스 베이스, pH 8.0 | 10 밀리미터미터 |
| 염화나트륨 | 150 밀리미터미터 |
| 트윈 20 | 0.1%(V/V) |
| TE 버퍼 | |
| 트리스-HCl, pH 8.0 | 10 밀리미터미터 |
| EDTA, pH 8.0 | 1미분 |
표 1: 버퍼 조성.
| 유전자 위치 | 포워드 프라이머 | 리버스 프라이머 |
| FRA16D | TCCTGTGGAAGGGATATTTA | CCCCTCATATTCTCTCTA |
| FRA3B | TGTTGGAATGTTAACTCTATCCCAT | 아타트캣카아가크그트캐피 |
| 증권 시세 표시기 | ||
| FRA3B | CAATGGCTTAAGCAGACATGGT | 아가트그캣껌굿굥굥이 |
| 루즈벨트 | ||
| ACTB(음성 대조군) | GACGCG가트그캣ggg | ACGCCTCTGGCCGTACCAC |
표 2: 프라이머 서열.
| DNA의 낮은 회수율 | 잠재적인 문제 | 가능한 해결책 |
| 항체의 질이 좋지 않음 | ChIP 등급의 항체를 사용합니다. | |
| 항체 부족 | 염색질 25μg당 1-10μg의 ChIP 항체를 사용합니다. | |
| 불충분한 배양 시간 | 4°C에서 밤새 면역침전 단계를 수행합니다. | |
| 지나치게 엄격한 세척 | 세척 버퍼에서 500mM 이상의 NaCl 농도를 사용하지 마십시오. | |
| 낮은 품질의 구슬 | 단백질 A 또는 G가 ChIP 항체와 호환되는지 확인합니다. | |
| 최적의 비드 대 항체 부피 비율을 위해 비드 제품 데이터시트를 따르십시오. | ||
| 불충분한 시작 | 세포 수를 정확하게 결정하기 위해 별도의 세포 플레이트를 준비합니다. | |
| 견본 | 대상이 낮은 존재비인 경우 사용되는 세포의 수를 늘립니다. | |
| 부적절한 세포 용해 | 버퍼 조성 및 용해 시간을 최적화하여 효율성을 개선합니다. | |
| 특정 세포 유형으로 작업할 때 Dounce 균질기를 사용하는 것과 같은 용해 단계 동안 기계적 힘을 가합니다. | ||
| 핵의 분해가 발생했습니다. | 염색질 무결성과 접근성을 보존하기 위해 핵 분리가 부드럽게 이루어지도록 합니다. | |
| 취급 중 핵 전단을 최소화하기 위해 광구경 피펫 팁을 사용하십시오. | ||
| 샘플 성능 저하가 발생했습니다. | 얼음 위 또는 4°C에서 모든 단계를 수행합니다. | |
| 모든 완충액에 프로테아제 억제제를 포함시키고 모든 완충액이 신선하게 준비되었는지 확인합니다. | ||
| 불충분한 MNase 분해 | MNase 농도(예: 0.1–5 단위)를 적정하여 특정 샘플 유형에 대한 최적량을 결정합니다. 이상적인 소화 조건을 식별하기 위해 다양한 농도로 시작하십시오. | |
| MNase 과소화 | 37°C에서 다양한 배양 시간(예: 1-10분)으로 실험하고 단편화 패턴을 모니터링하여 과도한 소화 없이 충분한 분해를 보장합니다. | |
| Disrupted Phosphorylation | 용해 완충액에 인산가수분해효소 억제제 칵테일을 추가하고 모든 완충액이 새로 준비되었는지 확인합니다. | |
| 네거티브 컨트롤의 높은 배경 | 비드에 대한 비특이적 결합 | 면역침전 단계 전에 사전 투명화 단계를 포함하고, 일반적으로 감소된 비특이적 결합을 나타내는 마그네틱 ChIP 등급 비드를 사용합니다. |
| 불충분한 세척 | 소금과 세제 농도를 조정하여 세탁 횟수 또는 엄격성을 높이십시오. | |
| 염색질의 불충분한 단편화 | 200-750bp의 단편화를 달성하기 위해 단편화를 최적화합니다. | |
| 최적화는 각 세포 또는 조직 유형에 필요합니다. | ||
| PCR 문제 해결 | 입력 샘플의 증폭 없음 | (1) 과잉 단편화: 염색질이 단핵솜 길이로 단편화되면 150bp 이상의 앰플리콘에 대해 신호가 감소합니다. |
| (2) PCR 조건을 최적화합니다. | ||
| (3) 더 작은(<150bp) 영역을 증폭하도록 프라이머를 설계합니다. | ||
| 저해상도 | DNA 단편 크기가 너무 큼 | 200-750bp의 단편화를 달성하기 위해 단편화를 최적화합니다. |
표 3: 염색질 면역침전 문제 해결.
토론
환경 오염은 인간 암의 중요한 원인입니다. 많은 오염 물질은 발암성이므로 암 발병으로 이어지는 유전적 손상을 일으킬 수 있습니다40,41. 그러나 특정 물질이 종양 유발 물질인지 여부를 결정하는 것은 어려운 작업입니다. 발암 가능성을 식별하기 위한 빠르고 신뢰할 수 있으며 비용 효율적인 방법은 과학자들이 환경 오염 물질을 효율적으로 선별하고 게놈 안정성에 미치는 영향을 평가할 수 있도록 합니다. 이 연구에서는 DSB에 대한 매우 특이적인 마커로 널리 알려진 변형된 히스톤 변이체인 γH2A.X에 초점을 맞춥니다. γH2A.X는 히스톤 변이체 H2A가 형성될 때 형성됩니다. X는 DNA 손상에 대한 반응으로 serine 139에서 인산화됩니다.
CFS에서 Native ChIP에 의한 γH2A.X의 존재를 검출함으로써, 환경 오염 물질에 의한 DNA 손상의 정도를 효과적으로 평가할 수 있다42. CFSs에서 γH2A.X를 정량적으로 측정할 수 있는 기능은 종양 유발 효과를 유도하기 위한 환경 오염 물질의 잠재력을 평가할 수 있는 빠르고 민감하며 신뢰할 수 있는 방법을 제공합니다. DNA 손상(예: 돌연변이, 소핵 형성 또는 세포 형질전환)의 간접 마커를 측정하는 기존의 유전독성 분석법과 달리, native γH2AX ChIP는 특정 게놈 유전자좌42에서 파손 이벤트를 정밀하게 매핑할 수 있습니다. 이를 통해 환경 오염 물질에 가장 취약한 지역에 대한 더 깊은 이해를 제공하고 잠재적인 발암 메커니즘을 식별하는 데 도움이 됩니다.
성공적인 네이티브 ChIP를 위해서는 대상 영역의 효과적인 농축을 보장하기 위해 몇 가지 중요한 단계에 세심한 주의를 기울여야 합니다. 다음은 γH2AX의 기본 ChIP와 관련된 중요한 단계입니다. 세포 준비: 적절한 수의 세포로 시작합니다. 아피디콜린(aphidicolin)과 같은 DNA 손상 유도제로 세포를 처리하여 복제 스트레스를 양성 대조군으로 유도합니다. 비교를 위해 적절한 처리되지 않은 음성 대조군 샘플을 포함합니다. 염색질 분리: 세포질 성분의 오염을 최소화하면서 염색질 결합 단백질을 분리하기 위해 세포 내 분획을 수행합니다. MNase 최적화: 미세구균 뉴클레아제(MNase) 농도 및 배양 시간을 최적화하기 위한 파일럿 실험을 수행합니다. 목표는 200-1000 염기쌍 사이의 염색질 단편을 얻어 뉴클레오솜 무결성을 유지하면서 ChIP에 대한 우수한 분리능을 보장하는 것입니다. 면역침전: 단백질 A/G 아가로스와 함께 고품질 ChIP 등급 γH2AX 항체를 활용하여 비특이적 결합을 최소화하면서 표적 염색질의 효율적인 풀다운을 보장합니다. 세척: 적절한 농도의 소금과 세제가 함유된 완충액으로 비드를 세척하여 γH2AX 결합 염색질을 유지하면서 비특이적 상호 작용을 제거합니다. 용리 및 DNA 회수: DNA의 철저한 용출 및 침전을 통해 결합된 염색질을 효율적으로 회수하여 농축된 표적 염색질의 손실을 최소화합니다. 기존 X-ChIP와 비교하여 기본 γH2AX ChIP에는 4가지 주요 장점이 있습니다. 첫째, 세포 내 분획을 통합하면 위양성 상호 작용이 감소하고 ChIP 특이성이 향상됩니다. 둘째, 단백질-DNA 복합체가 원래 상태로 보존되어 가교제와 함께 발생할 수 있는 일시적인 γH2AX-염색질 상호 작용의 안정화를 최소화합니다. 셋째, 고정과 시간이 많이 소요되는 역방향 교차 연결 단계를 생략하면 전체 워크플로가 간소화됩니다. 마지막으로, 더 적은 출발 물질이 필요하며 수조 초음파 발생기(예: Bioruptor) 또는 집속 초음파 발생기(예: Covaris)와 같은 염색질 전단을 위한 고가의 장비가 필요하지 않습니다.
네이티브 ChIP에도 장점이 있지만 한계도 인정해야 합니다. 한 가지 한계는 염색질 준비 및 면역침전 중 단백질 재배열 가능성입니다. 이러한 재배열은 이론적으로 염색질 결합 단백질의 상호 작용을 변경하고 결과에 변동성을 유발할 수 있습니다. 이러한 재배열의 발생을 뒷받침할 직접적인 증거는 없지만 이 잠재적인 인공물을 설명하기 위해 적절한 실험 대조군을 포함하는 것이 중요합니다. 네이티브 ChIP 분석의 또 다른 한계는 MNase의 분할 편향으로 인해 부정확한 결과가 발생할 수 있다는 것입니다. MNase는 게놈의 A-T가 풍부한 영역을 우선적으로 절단하여 이러한 영역이 ChIP 프로세스 중에 단편화되고 농축될 가능성을 높입니다. 이러한 편향은 A-T가 풍부한 염기서열의 과대 표현과 G-C가 풍부한 영역의 과소 표현을 유발할 수 있으며, 잠재적으로 MNase 분해에 접근하기 어려운 게놈 영역에 대한 거짓 음성 신호를 초래할 수 있습니다. 관심 유전자 자리가 G-C가 풍부한 경우 MNase 분해 조건의 신중한 최적화가 필요합니다. 또는 초음파 처리에 의한 염색질 단편화와 같은 추가 대조군을 통합하면 MNase 편향의 영향을 완화하고 결과의 신뢰성을 향상시키는 데 도움이 될 수 있습니다.
특정 한계에도 불구하고 native γH2AX ChIP는 특정 오염 물질이 DNA 손상을 유발하는 유전자 위치를 식별하는 데 특히 유용합니다. 연구자들은 γH2AX에 결합된 DNA 단편을 농축함으로써 오염 물질로 인한 복제 스트레스로 인해 파손되기 쉬운 게놈의 취약한 영역, 특히 qPCR에 의한 CFS를 식별할 수 있습니다. 이 기술을 통해 연구자들은 특정 발암 물질에 대한 노출과 특정 게놈 영역에서의 DNA 손상 형성 사이의 직접적인 관계를 규명할 수 있으며, 오염 물질의 유전 독성 프로필과 발암 유발43,44 에 대한 잠재적 역할에 대한 통찰력을 제공합니다.
문제 해결 표(표 3)는 낮은 DNA 수율, 불충분한 염색질 단편화, 높은 백그라운드 노이즈 또는 낮은 항체 성능과 같은 ChIP 분석 중에 발생하는 일반적인 문제에 대한 자세한 솔루션을 제공합니다. 각 문제는 프로토콜의 신뢰성과 재현성을 보장하기 위한 구체적인 권장 사항과 쌍을 이룹니다.
native γH2AX ChIP는 자동화된 액체 처리 시스템과 정량적 PCR 또는 염기서열분석 기술을 사용하여 DNA 손상을 유도하는 병원체를 효율적으로 식별할 수 있으므로 독성학 연구 및 환경 모니터링에서 유용한 도구 역할을 합니다. 다양한 수준의 환경 오염 물질에 노출된 집단에서 파생된 샘플에 천연 γH2AX ChIP를 사용함으로써 연구자들은 특정 DNA 손상 패턴을 식별하고 특정 물질에 대한 노출과 관련된 위험 수준을 결정할 수 있습니다. 결론적으로, native γH2AX ChIP는 환경 오염 물질의 발암 가능성을 평가하는 데 상당한 이점을 제공하며, 특정 게놈 자리에서 DNA 손상을 검출하기 위한 빠르고 비용 효율적이며 특이도가 높은 접근 방식을 제공합니다. 고처리량 스크리닝, 맞춤형 위험 평가 및 바이오마커 개발에 대한 미래의 응용 분야는 환경 발암에 대한 이해를 발전시키고 암 예방 및 개입을 위한 전략을 개발하는 데 유용한 도구가 될 것입니다.
공개
저자는 공개할 이해 상충이 없습니다.
감사의 말
이 작업은 University of South China의 스타트업 펀딩으로 지원되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| 0.2 µm nitrocellulose membrane | Amersham | 10600011 | |
| Actin B | proteintech | 20536-1-AP | |
| Aphidicolin | MedChemExpress | HY-N6733 | |
| ChIP-grade magnetic Protein A/G beads | ThermoFisher | 26162 | |
| Clarity Western ECL Substrate | Bio-Rad | #1705061 | |
| Glycogen, molecular biology grade | ThermoFisher | Cat. No. R0561 | |
| HRP-conjugated secondary antibody | proteintech | SA00001-2 | |
| hydroxyurea | MedChemExpress | HY-B0313 | |
| Micrococcal Nuclease | NEB | M0247S | |
| normal IgG | Santa Cruz | sc-2025 | |
| Taq Universal SYBR Green Supermix | BioRad | 1725120 | |
| γH2A.X antibody (for ChIP) | Sigma-Aldrich | 05-636 | |
| γH2A.X antibody (for WB) | Cell Signaling | #25955 |
참고문헌
- Glover, T. W., Berger, C., Coyle, J., Echo, B. DNA polymerase alpha inhibition by aphidicolin induces gaps and breaks at common fragile sites in human chromosomes. Hum Genet. 67 (2), 136-142 (1984).
- Bignell, G. R., et al. Signatures of mutation and selection in the cancer genome. Nature. 463 (7283), 893-898 (2010).
- Hellman, A., et al. A role for common fragile site induction in amplification of human oncogenes. Cancer Cell. 1 (1), 89-97 (2002).
- Kotzot, D., et al. Parental origin and mechanisms of formation of cytogenetically recognisable de novo direct and inverted duplications. J Med Genet. 37 (4), 281-286 (2000).
- Miller, C. T., et al. Genomic amplification of MET with boundaries within fragile site FRA7G and upregulation of MET pathways in esophageal adenocarcinoma. Oncogene. 25 (3), 409-418 (2006).
- Zack, T. I., et al. Pan-cancer patterns of somatic copy number alteration. Nat Genet. 45 (10), 1134-1140 (2013).
- Gao, G., et al. Common fragile sites (CFS) and extremely large CFS genes are targets for human papillomavirus integrations and chromosome rearrangements in oropharyngeal squamous cell carcinoma. Genes Chromosomes Cancer. 56 (1), 59-74 (2017).
- Thorland, E. C., Myers, S. L., Gostout, B. S., Smith, D. I. Common fragile sites are preferential targets for HPV16 integrations in cervical tumors. Oncogene. 22 (8), 1225-1377 (2003).
- Matovina, M., Sabol, I., Grubisić, G., Gasperov, N. M., Grce, M. Identification of human papillomavirus type 16 integration sites in high-grade precancerous cervical lesions. Gynecol Oncol. 113 (1), 120-127 (2009).
- Yu, T., et al. The role of viral integration in the development of cervical cancer. Cancer Genet Cytogenet. 158 (1), 27-34 (2005).
- Bignell, G. R., et al. Signatures of mutation and selection in the cancer genome. Nature. 463 (7283), 893-898 (2010).
- Thavathiru, E., Ludes-Meyers, J. H., MacLeod, M. C., Aldaz, C. M. Expression of common chromosomal fragile site genes, WWOX/FRA16D and FHIT/FRA3B is downregulated by exposure to environmental carcinogens, UV, and BPDE but not by IR. Mol Carcinog. 44 (3), 174-182 (2005).
- Sirbu, B. M., et al. Analysis of protein dynamics at active, stalled, and collapsed replication forks. Genes Dev. 25 (12), 1320-1327 (2011).
- Barlow, J. H., et al. Identification of early replicating fragile sites that contribute to genome instability. Cell. 152 (3), 620-632 (2013).
- Petermann, E., Orta, M. L., Issaeva, N., Schultz, N., Helleday, T. Hydroxyurea-stalled replication forks become progressively inactivated and require two different RAD51-mediated pathways for restart and repair. Mol Cell. 37 (4), 492-502 (2010).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. J Biol Chem. 273 (10), 5858-5868 (1998).
- Shanbhag, N. M., Rafalska-Metcalf, I. U., Balane-Bolivar, C., Janicki, S. M., Greenberg, R. A. ATM-dependent chromatin changes silence transcription in cis to DNA double-strand breaks. Cell. 141 (6), 970-981 (2010).
- Stiff, T., et al. ATR-dependent phosphorylation and activation of ATM in response to UV treatment or replication fork stalling. EMBO J. 25 (24), 5775-5782 (2006).
- Nelson, J. D., Denisenko, O., Bomsztyk, K. Fast chromatin immunoprecipitation assay. Nuc Acids Res. 34 (5), e2 (2006).
- Dorbic, T., Wittig, B. Isolation of oligonucleosomes from active chromatin using HMG17-specific monoclonal antibodies. Nuc Acids Res. 14 (8), 3363-3376 (1986).
- Dorbic, T., Wittig, B. Chromatin from transcribed genes contains HMG17 only downstream from the starting point of transcription. EMBO J. 6 (8), 2393-2399 (1987).
- Hebbes, T. R., Thorne, A. W., Crane-Robinson, C. A direct link between core histone acetylation and transcriptionally active chromatin. EMBO J. 7 (5), 1395-1402 (1988).
- Hebbes, T. R., Thorne, A. W., Clayton, A. L., Crane-Robinson, C. Histone acetylation and globin gene switching. Nuc Acids Res. 20 (5), 1017-1022 (1992).
- Hebbes, T. R., Clayton, A. L., Thorne, A. W., Crane-Robinson, C. Core histone hyperacetylation co-maps with generalized DNase I sensitivity in the chicken beta-globin chromosomal domain. EMBO J. 13 (8), 1823-1830 (1994).
- Orlando, V. Mapping chromosomal proteins in vivo by formaldehyde-crosslinked-chromatin immunoprecipitation. Trend Biochem, Sci. 25 (3), 99-104 (2000).
- Myers, F. A., Evans, D. R., Clayton, A. L., Thorne, A. W., Crane-Robinson, C. Targeted and extended acetylation of histones H4 and H3 at active and inactive genes in chicken embryo erythrocytes. J Biol Chem. 276 (23), 20197-20205 (2001).
- Litt, M. D., Simpson, M., Recillas-Targa, F., Prioleau, M. N., Felsenfeld, G. Transitions in histone acetylation reveal boundaries of three separately regulated neighboring loci. EMBO J. 20 (9), 2224-2235 (2001).
- Madisen, L., Krumm, A., Hebbes, T. R., Groudine, M. The immunoglobulin heavy chain locus control region increases histone acetylation along linked c-myc genes. Mol Cell Biol. 18 (11), 6281-6292 (1998).
- Clayton, A. L., Hebbes, T. R., Thorne, A. W., Crane-Robinson, C. Histone acetylation and gene induction in human cells. FEBS Lett. 336 (1), 23-26 (1993).
- Tseng, Z., Wu, T., Liu, Y., Zhong, M., Xiao, A. Using native chromatin immunoprecipitation to interrogate histone variant protein deposition in embryonic stem cells. Methods Mol Biol. 1176, 11-22 (2014).
- Durkin, S. G., Glover, T. W. Chromosome fragile sites. Ann Rev Genetics. 41, 169-192 (2007).
- Lee, J. B., Keung, A. J. Chromatin immunoprecipitation in human and yeast cells. Methods Mol Biol. 1767, 257-269 (2018).
- Miyamoto, R., Yokoyama, A. Protocol for fractionation-assisted native ChIP (fanChIP) to capture protein-protein/DNA interactions on chromatin. STAR Protoc. 2 (2), 100404 (2021).
- Mendez, J., Stillman, B. Chromatin association of human origin recognition complex, cdc6, and minichromosome maintenance proteins during the cell cycle: Assembly of prereplication complexes in late mitosis. Mol Cell Biol. 20 (22), 8602-8612 (2000).
- Nowak, D. E., Tian, B., Brasier, A. R. Two-step cross-linking method for identification of NF-κB gene network by chromatin immunoprecipitation. BioTechniques. 39 (5), 715-725 (2005).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nat Protoc. 3 (6), 1101-1108 (2008).
- Lu, X., Parvathaneni, S., Hara, T., Lal, A., Sharma, S. Replication stress induces specific enrichment of RECQ1 at common fragile sites FRA3B and FRA16D. Mol Cancer. 12, 29 (2013).
- Ruijter, J. M., et al. Amplification efficiency: linking baseline and bias in the analysis of quantitative PCR data. Nucl Acids Res. 37 (6), e45 (2009).
- Lyu, X., Chastain, M., Chai, W. Genome-wide mapping and profiling of γH2AX binding hotspots in response to different replication stress inducers. BMC Genomics. 20, 579 (2019).
- Farmer, P. B., et al. Molecular epidemiology studies of carcinogenic environmental pollutants. Effects of polycyclic aromatic hydrocarbons (PAHs) in environmental pollution on exogenous and oxidative DNA damage. Mutat Res. 544 (2-3), 397-402 (2003).
- Phillips, D. H., Arlt, V. M. Genotoxicity: damage to DNA and its consequences. EXS. 99, 87-110 (2009).
- Nitsch, S., Schneider, R. Native ChIP: Studying the genome-wide distribution of histone modifications in cells and tissue. Meth Mol Biol. 2846, (2024).
- Nikitina, T., Wang, D., Gomberg, M., Grigoryev, S. A., Zhurkin, V. B. Combined micrococcal nuclease and exonuclease III digestion reveals precise positions of the nucleosome core/linker junctions: implications for high-resolution nucleosome mapping. J Mol Biol. 425 (11), 1946-1960 (2013).
- Teves, S. S., Henikoff, S. Salt fractionation of nucleosomes for genome-wide profiling. Methods Mol Biol. 833, 421-432 (2012).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유