Method Article
Capture des ruptures de sites fragiles communes par γH2A natif. X ChIP
Dans cet article
Résumé
Nous présentons une méthode rapide et efficace pour détecter les ruptures de sites fragiles courants grâce à l’immunoprécipitation de la chromatine (ChIP) γH2A.X native. Cette approche réduit considérablement le temps et la main-d’œuvre associés aux tests traditionnels γH2A.X ChIP tout en maintenant une reproductibilité et une fiabilité élevées des résultats.
Résumé
Le stress de réplication induit par l’exposition à des agents extrinsèques peut entraîner des cassures de l’ADN au niveau de sites fragiles communs, qui sont des régions du génome connues pour être sujettes à l’instabilité structurelle. Le test d’immunoprécipitation de la chromatine (ChIP) γH2A.X est un outil puissant dans les études de génotoxicité, car la phosphorylation de γH2A.X est un marqueur bien établi des cassures double brin de l’ADN. Cependant, les tests traditionnels de γH2A.X ChIP nécessitent souvent beaucoup de main-d’œuvre et impliquent plusieurs étapes chronophages. Dans cette étude, nous présentons une méthode simplifiée mais efficace qui combine le fractionnement subcellulaire avec la ChIP native pour isoler les complexes associés à γH2A.X. Cette approche est particulièrement adaptée à l’analyse des interactions γH2A.X-chromatine avec une spécificité et une efficacité accrues. En utilisant le fractionnement subcellulaire, les matériaux non liés à la chromatine sont efficacement éliminés, ce qui donne une fraction de chromatine purifiée. La digestion ultérieure des nucléases micrococciques (MNase) dans des conditions douces permet la fragmentation de la chromatine tout en préservant les interactions physiologiques entre γH2A.X et ses complexes protéiques associés. Cette préservation est essentielle pour l’étude des partenaires d’interaction indigènes impliqués dans les voies de réponse aux dommages de l’ADN. Ce protocole ChIP natif optimisé réduit considérablement le temps et la main-d’œuvre associés aux tests ChIP γH2A.X conventionnels. La procédure rationalisée simplifie non seulement le flux de travail, mais donne également des résultats hautement reproductibles, ce qui la rend particulièrement avantageuse dans les environnements où le traitement à haut débit de plusieurs échantillons est requis. Cette méthode a une large applicabilité dans les études axées sur la stabilité du génome, la réparation de l’ADN et la biologie de la chromatine, où une détection précise et efficace des sites de dommages à l’ADN est essentielle. En utilisant des protocoles optimisés et des étapes simplifiées, cette méthode permet de détecter les dommages à l’ADN sur les sites fragiles avec une sensibilité améliorée et une manipulation minimale des échantillons, ce qui en fait un outil précieux pour les études sur la stabilité du génome et la réponse aux dommages à l’ADN.
Introduction
Les sites fragiles communs (SFC) sont de grandes régions chromosomiques présentes sur chaque chromosome humain susceptibles de se rompre pendant la métaphase. En cas de stress de réplication, la réplication dans ces régions est considérablement retardée, ce qui empêche leur duplication complète avant l’entrée mitotique1, ce qui entraîne finalement des lacunes et des ruptures spécifiques au site. Les SFC sont des points chauds d’instabilité chromosomique et sont une cause majeure de réarrangements chromosomiques au cours du développement précoce du cancer. Le stress de réplication, qui est souvent présent dans des conditions tumorigènes, peut entraîner la perte de gènes suppresseurs de tumeurs et l’amplification d’oncogènes, collectivement appelés variation du nombre de copies (CNV)2,3,4,5,6. De plus, les SFC sont très sujets à l’intégration virale, ce qui favorise davantage le développement du cancer 7,8,9,10. De multiples délétions homozygotes de gènes suppresseurs de tumeurs ont été détectées dans les régions du SFC lors d’analyses pancancéreuses de tumeurs primaires. Les SFC les plus fréquemment touchés par le cancer sont FRA2F, FRA3B, FRA4F, FRA5H et FRA16D11. Les SFC sont particulièrement vulnérables à la casse en présence d’agents cancérigènes extrinsèques12. Pour évaluer les effets cancérogènes néfastes des contaminants environnementaux, il est nécessaire de disposer d’une méthode rapide et fiable pour quantifier l’occurrence des ruptures de SFC.
Phosphorylation de H2A. X au niveau du résidu sérine 139 (γH2A.X) par l’ataxie télangiectasie et la protéine liée à Rad3 (ATR) ou l’ataxie télangiectasie mutée (ATM) est un événement clé dans la signalisation de la réplication du blocage de la fourche13. γH2A.X sert d’indicateur de fourches de réplication bloquées avant la formation de cassures double brin (DSB)13, créant un environnement chromatinien favorable pour faciliter le recrutement efficace des protéines de réparation vers les sites bloqués. De plus, γH2A.X peut être recruté pour briser des sites après l’effondrement de la fourche14,15, conformément à son rôle principal dans la réparation des DSB. Étant donné que les cassures du SFC sont étroitement associées aux aberrations chromosomiques qui entraînent la progression du cancer, la détection de ces cassures peut être déterminante pour comprendre les premiers stades de la tumorigenèse. La présence de γH2A.X au niveau des SFC peut être utilisée comme biomarqueur pour détecter les événements précoces d’instabilité génomique. Cette information peut aider à identifier les cancérogènes potentiels et à évaluer le risque associé à l’exposition à divers agents extrinsèques. En mesurant les cassures de l’ADN au niveau des SFC induites par des agents extrinsèques, la chromatine IP γH2A.X (ChIP) peut donner un aperçu de la façon dont ces agents contribuent aux mécanismes sous-jacents à la tumorigenèse.
Dans la ChIP conventionnelle (c’est-à-dire la ChIP réticulée, X-ChIP), l’association de γH2A.X avec ses séquences d’ADN cibles est stabilisée par une réticulation réversible du formaldéhyde. La chromatine est ensuite cisaillée en fragments d’environ 500 paires de bases (pb) par sonication, et la solution résultante est débarrassée des débris par sédimentation 16,17,18. Un anticorps γH2A.X de qualité ChIP est ensuite ajouté à la fraction de chromatine éliminée, suivi de l’ajout de billes d’agarose de protéine A/G pour enrichir les régionsde chromatine liées à γH2A.X 16,17,18. Les complexes immuns (c’est-à-dire le complexe d’ADN ciblé par les billes-anticorps-γH2A.X) sont lavés plusieurs fois avec des tampons de lavage stricts pour éliminer les fragments d’ADN non spécifiquement liés 16,17,18. Après le lavage, l’ADN spécifiquement lié est élué des complexes immuns. Les réticulations du formaldéhyde sont ensuite inversées, suivies d’une digestion des protéines à l’aide de la protéinase K, après quoi l’ADN enrichi est purifié et concentré 16,17,18. Pour évaluer les régions associées à γH2A.X, la PCR, la PCR quantitative (qPCR) ou le séquençage direct sont utilisés 16,17,18. L’occupation de γH2A.X dans des régions spécifiques, telles que le SFC, est déterminée par l’intensité du signal PCR ou qPCR, qui est proportionnelle à la quantité de γH2A.X liée à cet endroit, ce qui donne un aperçu des dommages à l’ADN spécifiques au site et des événements de réparation 16,17,18.
Bien qu’il s’agisse d’une approche expérimentale puissante, la X-ChIP présente plusieurs limites importantes : (i) elle nécessite un grand nombre de cellules, généralement dans la gamme de 1 x 107 à 5 x 107, en raison de l’inefficacité de la précipitation des anticorps associée à la fixation, ce qui augmente le coût global de l’expérience19 ; (ii) le processus d’inversion des réticulations du formaldéhyde et de purification ultérieure de l’ADN prend du temps et exige beaucoup de main-d’œuvre, ce qui rend difficile le maintien de l’uniformité et de la fiabilité des résultats ; et (iii) les interactions γH2A.X-ADN ayant une signification fonctionnelle mineure ne peuvent pas être distinguées de celles ayant une signification plus grande, car l’étape de réticulation peut stabiliser les interactions transitoires, conduisant à la détection d’interactions qui peuvent ne pas être biologiquement pertinentes19.
L’immunoprécipitation de la chromatine native (Native ChIP ou N-ChIP) est une technique biochimique essentielle utilisée pour étudier les interactions protéine-ADN dans leur contexte de chromatine native dans des conditions physiologiques de sel. Il a joué un rôle déterminant dans l’élucidation de l’organisation spatiale et temporelle de la chromatine, de la liaison des facteurs de transcription et des modifications des histones. La ChIP native joue un rôle de longue date dans le domaine plus large de la biologie de la chromatine et de l’épigénétique, offrant des avantages et des limites uniques par rapport à la X-ChIP. Cette méthode, introduite à la fin des années 198020, implique l’isolement de la chromatine des cellules par des méthodes qui préservent sa structure native, comme la digestion avec des nucléases micrococciques (MNase)21. Cela préserve les contacts inhérents protéine-ADN et histone-ADN, ce qui rend la ChIP native particulièrement bien adaptée à l’étude des modifications des histones et du positionnement des nucléosomes dans leur cadre chromatien naturel22. Des études à haute résolution sur la ChIP native ont démontré l’utilisation de la digestion de la MNase pour réduire la chromatine en nucléosomes individuels, ce qui facilite la cartographie des modifications des histones avec une plus grande précision23. De plus, comme il n’y a pas de réticulation chimique, le risque d’introduire des biais ou des artefacts qui pourraient dénaturer les interactions protéine-ADN est minimisé24.
Contrairement à la X-ChIP, où le formaldéhyde ou d’autres agents de réticulation sont utilisés pour fixer les interactions protéine-ADN, la ChIP native fournit une vision plus réaliste de la chromatine en évitant les artefacts de réticulation potentiels. Cependant, alors que la X-ChIP est généralement mieux adaptée à la détection des interactions transitoires ou dynamiques entre l’ADN et les protéines régulatrices25, la ChIP native est idéale pour les interactions protéine-ADN stables, telles que les histones ou d’autres protéines liées à la chromatine 26,27. L’une des limitations notées pour le ChIP natif est l’incapacité à capturer les événements de liaison de faible affinité ou transitoires, qui sont souvent stabilisés par la réticulation dans X-ChIP25.
Un nombre important de travaux en épigénétique a tiré parti de la ChIP native pour découvrir des modifications d’histones dans divers contextes biologiques28. Ces efforts ont été cruciaux dans la définition du code des histones - le modèle de modifications des histones qui régulent l’expression des gènes et la dynamique de la chromatine29. Bien que H2A. X est une histone de liaison moins fortement associée, la H2A native. La méthode X ChIP a été appliquée avec succès dans des cellules souches embryonnaires30. Dans cette étude, nous avons optimisé une procédure d’extraction de la chromatine pour effectuer la ChIP native de γH2A.X dans les cellules 293T humaines (Figure 1). L’hydroxyurée et l’aphidicoline sont largement utilisées dans la recherche pour étudier le stress, les dommages et l’instabilité génomique de la réplication de l’ADN31. Dans cette étude, ces agents ont été appliqués aux cellules pour induire un stress de réplication et générer des cassures de l’ADN au niveau du SFC.
En utilisant du matériel de départ d’environ 1 x 106 à 5 x 106 cellules, cette méthode peut être divisée en quatre étapes principales : (i) le fractionnement subcellulaire pour isoler la chromatine, (ii) la digestion des nucléases micrococciques (MNase) pour fragmenter la chromatine, (iii) l’immunoprécipitation et l’élution, et (iv) l’analyse de l’ADN par PCR quantitative (qPCR). La réalisation de ChIP après le fractionnement subcellulaire offre plusieurs avantages et a été bien documentée dans de nombreuses études 32,33,34,35. Cette approche permet d’éliminer les protéines non liées à la chromatine et d’autres débris cellulaires, ce qui donne une fraction de chromatine hautement purifiée. En isolant la chromatine avant l’immunoprécipitation, le fractionnement subcellulaire aide à maintenir les interactions chromatiniennes natives et réduit le bruit de fond des protéines non associées à la chromatine, ce qui conduit à des résultats plus spécifiques et fiables, car seuls les complexes liés à la chromatine sont retenus pour l’analyse. De plus, le fractionnement subcellulaire permet des conditions plus douces pour la digestion de la chromatine, préservant ainsi les interactions physiologiques protéine-ADN et offrant une représentation plus précise de la dynamique de la chromatine dans l’environnement cellulaire natif.
L’utilisation de la ChIP native de γH2AX pour mesurer l’impact des agents extrinsèques sur la rupture des sites fragiles courants présente un potentiel important pour la recherche sur le cancer. Cette technique permet de détecter les dommages à l’ADN induits par l’exposition à des agents cancérigènes environnementaux, ce qui donne un aperçu des mécanismes moléculaires par lesquels les polluants contribuent à l’instabilité génomique et au développement du cancer. En préservant le contexte natif de la chromatine, cette méthode facilite l’évaluation précise des modèles de dommages à l’ADN associés à l’exposition cancérigène, ce qui facilite l’évaluation des risques environnementaux et l’étude de la tumorigenèse induite par la pollution.
Protocole
1. Prélèvement cellulaire
- Ensemez environ 5 x 10cellules 5 HEK 293T dans chacune des quatre boîtes de 6 cm, chacune contenant 4 ml de milieu DMEM complet.
- Après 24 h, traiter une boîte avec 2 μL de solution mère d’aphidicoline (voir le Tableau des matières) (concentration finale de 0,5 μM) et une autre boîte avec 20 μL de solution mère d’hydroxyurée 1 M (voir le Tableau des matières) (concentration finale de 5 mM) pour induire un stress de réplication. Ajoutez du DMSO aux deux plats restants pour servir de contrôles.
- Après 24 h de traitement, jeter le milieu de culture. Une plaque de 6 cm produit généralement environ 2 x 10cellules 6 à 60 % à 70 % de confluence.
- Rincer chaque plat 2 fois avec 5 ml de PBS glacé. À l’aide de grattoirs à cellules, détachez les cellules et transférez la suspension cellulaire dans quatre tubes individuels de 1,5 ml. Pipetez doucement de haut en bas avec une pipette P1000 pour dissocier les amas de cellules.
- Centrifugez les cellules à 500 x g pendant 5 min à 4 °C, puis jetez le surnageant. Placez les cellules sur de la glace.
2. Fractionnement subcellulaire
- Remettre la pastille cellulaire en suspension dans 500 μL de tampon A froid fraîchement préparé (voir tableau 1), en assurant la dissociation complète des amas cellulaires par pipetage doux.
- Incuber les lysats sur de la glace pendant 5 à 10 min. Vérifiez la progression de la lyse au microscope pour vous assurer que la lyse cellulaire est complète.
- Prenez une petite aliquote du lysat (environ 5 à 10 μL) et placez-la sur une lame de microscope propre. Couvrez-le d’une lamelle pour éviter toute contamination.
- Utilisez un microscope optique avec un grossissement approprié (p. ex., 20x - 40x) pour visualiser les cellules ou les débris. Comparer avec un échantillon de contrôle non lysé pour différencier les cellules intactes du matériel lysé.
REMARQUE : Un échantillon correctement lysé n’aura pas de contours cellulaires distincts, seulement de la chromatine diffuse ou du matériel cellulaire. Ajustez la mise au point pour observer clairement le lysat. Si nécessaire, appliquez une force mécanique pendant l’étape de lyse, par exemple en utilisant un homogénéisateur Dounce, lorsque vous travaillez avec certains types de cellules.
- Centrifuger à 500 x g pendant 5 min à 4 °C, une fois les cellules complètement lysées. Jetez soigneusement le surnageant. Remettre en suspension la pastille de noyaux dans 500 μL de tampon A froid à l’aide d’embouts de pipette à large orifice.
REMARQUE : Les pointes à large orifice aident à minimiser les forces de cisaillement et à protéger les échantillons délicats comme la chromatine. Fabriquez des pointes à alésage large en coupant l’extrémité des pointes standard avec une lame tranchante. - Centrifugeuse à 500 x g pendant 5 min à 4 °C. Jetez soigneusement le surnageant.
- Préchauffez l’incubateur à 37 °C et préparez un tampon d’arrêt (100 mM d’EDTA, pH 8,0 ; Tableau 1).
- Optimisez à l’avance les concentrations et les temps d’incubation des nucléases micrococciques (MNase, voir la Table des matières).
- Divisez 40 μL de l’échantillon de chromatine d’analyse en plusieurs aliquotes égales pour tester différentes concentrations de MNase et temps d’incubation.
- Utilisez une gamme de concentrations de MNase (p. ex., 0,0625 U, 0,125 U, 0,25 U, 0,5 U, 1 U, 2 U, 4 U, 8 U par réaction) et testez plusieurs temps d’incubation (p. ex., 2, 5, 10 et 15 min).
- Ajouter aux aliquotes de chromatine un tampon MNase (voir le tableau 1) contenant diverses concentrations de MNase et incuber les échantillons à 37 °C pendant les durées spécifiées.
- Terminez la réaction en ajoutant 1/4 de volume de tampon d’arrêt (concentration finale : 20 mM d’EDTA) immédiatement après le temps d’incubation souhaité.
- Isolez l’ADN des échantillons de chromatine digérés à l’aide d’une méthode d’extraction par phénol/chloroforme/alcool isoamylique.
- Analysez l’ADN extrait sur un gel d’agarose à 1,5 % pour visualiser les schémas de digestion : la sous-digestion montrera des bandes de poids moléculaire élevé (Figure 2, voies 1-4) ; une digestion excessive entraînera un frottis ou des fragments très courts (figure 2, couloirs 6-8), et une digestion optimale donnera un modèle d’échelle nucléosomique clair (figure 2, couloir 5, p. ex., mono-, di-, tri-nucléosomes).
- Identifiez les conditions qui produisent la résolution nucléosomique souhaitée sans surdigestion excessive.
REMARQUE : CaCl2 agit comme un cofacteur de l’activité MNase. Optimisez la digestion en ajustant la concentration en CaCl2 entre 1 mM et 5 mM.
- Remettez doucement en suspension les noyaux intacts avec 100 μL de tampon MNase en pipetant 5 à 10 fois avec des pointes à large orifice. Ajoutez immédiatement la quantité prédéterminée de MNase aux échantillons (1,25 U MNase/100 μL de tampon MNase).
REMARQUE : Lorsque vous travaillez avec plusieurs échantillons, digérez chacun d’eux individuellement pour éviter une digestion excessive. - Placez les tubes sur un rotateur et incubez pendant 5 min à 37 °C. Remettez immédiatement les tubes dans la glace et terminez la digestion de la MNase en ajoutant de l’EDTA à une concentration finale de 20 mM et mélangez par vortex.
- Ajouter 500 μL de tampon B (voir le tableau 1) à chaque échantillon et bien mélanger en pipetant de haut en bas de 5 à 10 fois. Solubiliser les protéines en les incubant sur de la glace pendant 5 min.
REMARQUE : Le sel et le détergent du tampon B aident à dissocier les protéines faibles liées à la chromatine et à exposer les épitopes pour l’immunoprécipitation. - Granuler la matière insoluble en la centrifugant à vitesse maximale pendant 5 min à 4 °C. Transférez le surnageant transparent dans de nouveaux tubes de 1,5 mL étiquetés comme fraction native de chromatine. Les échantillons peuvent être stockés à -80 °C ou utilisés pour valider l’efficacité de la fragmentation de la chromatine.
REMARQUE : Évitez les cycles fréquents de gel-dégel, car ils peuvent perturber les interactions protéine-ADN d’intérêt. Minimisez les cycles de gel-dégel dans la mesure du possible.
3. Vérification de la fragmentation de la chromatine
- Aliquote 10 μL du surnageant de chaque échantillon dans un nouveau tube de 1,5 mL. Mélanger avec 20 μL d’eau distillée et 30 μL d’alcool phénol/chloroforme/isoamylique (25:24:1).
- Fermez bien les tubes et agitez vigoureusement pendant 15 à 30 s. Centrifugeuse à 20 000 x g (ou la vitesse maximale de la centrifugeuse) pendant 10 min à 4 °C. Après la centrifugation, trois couches distinctes seront observées : une couche supérieure claire, une couche intermédiaire blanche et une couche inférieure jaune.
- Transférez avec précaution 20 μL de la phase aqueuse supérieure (contenant de l’ADN) dans un tube frais. Séparez l’ADN purifié dans un gel d’agarose à 1,5 % pendant 30 min à 100 V et visualisez les schémas de digestion. Assurez-vous que la taille des fragments de chromatine est principalement comprise entre 200 et 1000 paires de bases.
REMARQUE : La taille appropriée des fragments de chromatine est cruciale pour le succès de la ChIP native et dépend des conditions de traitement de la MNase, y compris les unités enzymatiques, le temps d’incubation et la concentration de CaCl2. L’efficacité de la digestion de la MNase peut également varier en fonction du type et du nombre de cellules. Le modèle de fragmentation de la chromatine illustré à la figure 2 (voie 5) est recommandé pour ce test ChIP.
4. Immunoprécipitation
- Aliquote 20 μL de chromatine digérée de chaque échantillon dans un tube frais de 1,5 mL et mélanger avec 180 μL de tampon d’élution (voir le tableau 1). Étiquetez ces tubes comme échantillons d’entrée et conservez-les à -20 °C.
- Transférez 400 μL d’échantillon de chromatine dans un autre tube de 1,5 mL pour la ChIP.
- Ajouter l’anticorps γH2A.X (voir le tableau des matières) à un échantillon traité au DMSO, à un échantillon traité à l’aphidicoline et à un échantillon traité à l’hydroxyurée. Ajouter la même quantité d’IgG normales (voir le Tableau des matières) à un autre échantillon traité au DMSO comme témoin négatif pour le dosage de la ChIP.
REMARQUE : Ici, 1 μg d’anticorps primaire est généralement utilisé pour 400 μL de chromatine (c’est-à-dire que la concentration finale de l’anticorps est de 2,5 μg/mL). Cependant, la quantité optimale doit être déterminée empiriquement pour différents anticorps γH2A.X. - Placez les tubes ChIP sur un rotateur à 4 °C et incubez pendant au moins 5 h, ou de préférence toute la nuit.
- Pendant ce temps, aliquote 100 μL de billes magnétiques de protéine A/G de qualité ChIP (voir le tableau des matériaux) dans un nouveau tube de 1,5 mL. Utilisez des pointes à large orifice et pipetez lentement pour assurer une mesure précise des billes. Placez le tube sur un support magnétique pendant au moins 1 min, puis jetez soigneusement le liquide.
- Remettre les billes en suspension dans 1 mL de 1x PBS contenant 0,5 % BSA. Tourner à 4 °C pendant environ 4 h. Placez le tube sur un support magnétique pendant au moins 1 min et jetez le surnageant.
- Lavez à nouveau les perles avec 1 ml de 1x PBS contenant 0,5 % BSA. Placez le tube sur le support magnétique pendant 1 min pour granuler les billes magnétiques, puis jetez le surnageant.
REMARQUE : Les étapes 4.5 à 4.7 consistent à pré-enrober les billes afin de réduire la liaison non spécifique des anticorps aux billes magnétiques. - Remettre en suspension les billes pré-enduites dans 100 μL de tampon B à l’aide d’embouts à large orifice. Ajoutez 25 μL de suspension à billes magnétiques pré-enrobées dans chaque tube d’échantillon ChIP. Tourner à 4 °C pendant 2 h.
- Placez les tubes ChIP sur le support magnétique et attendez que les billes soient complètement fixées sur le côté du tube et que la solution devienne claire.
- Jetez le surnageant transparent sans déranger les billes magnétiques. Remettre les billes en suspension avec 1 mL de tampon de lavage (voir le tableau 1) et les faire tourner à 4 °C pendant 10 min.
- Replacez les tubes sur le support magnétique et attendez que la solution devienne claire. Jetez le tampon de lavage. Répétez le lavage pour un total de quatre lavages.
- Jetez le tampon de lavage après le lavage final et centrifugez brièvement les tubes à 400 x g pendant 30 s à 4 °C pour faire essorer tout liquide résiduel. Replacez les tubes sur le support magnétique et retirez soigneusement tout liquide restant du fond du tube.
5. Élution et précipitation de l’ADN
REMARQUE : L’efficacité des anticorps peut varier entre les différents lots. Il est important de confirmer l’affinité de liaison d’un nouvel anticorps en vérifiant les échantillons immunoprécipités par transfert Western.
- Vérifiez l’efficacité de l’extraction de l’anticorps ChIP à l’aide du Western blot (WB) comme décrit ci-dessous.
- Prélever une petite aliquote de l’échantillon de ChIP pour analyse (c.-à-d. généralement 10 % de l’échantillon de ChIP). Inclure la chromatine d’entrée (pré-immunoprécipitation) et le contrôle négatif (par exemple, pull-down IgG) à des fins de comparaison.
- Éluer les protéines des billes liées aux anticorps en chauffant dans 20 μL de 1 tampon de chargement SDS-PAGE (voir le tableau des matières) à 95 °C pendant 5 min.
- Chargez les échantillons, les entrées et les commandes IP sur un gel SDS-PAGE à 15 %. Faites couler le gel.
- Transférez les protéines sur une membrane de nitrocellulose de 0,2 μm (voir le tableau des matériaux) ou en PVDF à l’aide d’un système de transfert humide ou semi-sec.
- Bloquer la membrane avec du lait écrémé à 5 % ou de la BSA dans du TBST (voir tableau 1) pendant 1 h à température ambiante pour éviter une liaison non spécifique.
- Incuber la membrane avec l’anticorps primaire contre le γH2A.X (voir le tableau des matériaux) dilué dans un tampon bloquant pendant 1 à 2 h à température ambiante ou toute la nuit à 4 °C.
- Lavez la membrane 3 fois avec du TBST pour éliminer les anticorps non liés. Incuber la membrane avec un anticorps secondaire conjugué à la HRP (voir le tableau des matières) pendant 1 h à température ambiante. Lavez à nouveau la membrane pour éliminer l’excès d’anticorps secondaires.
- Développez la membrane à l’aide d’un substrat chimiluminescent et visualisez le signal avec un imageur. Comparez l’intensité du signal entre les voies IP, d’entrée et de contrôle pour évaluer l’efficacité et la spécificité du pull-down.
REMARQUE : Une bande correspondant à la protéine cible dans la voie IP confirme la réussite de l’extraction de l’anticorps. Cette approche vous permet d’évaluer l’efficacité de l’anticorps à capturer la protéine cible pendant l’expérience ChIP.
- Ajouter 50 μL de tampon d’élution (voir le tableau 1) à chacun des échantillons de ChIP restants. Placez les tubes sur un thermomixeur et agitez pendant 15 min à température ambiante.
- Placez les tubes sur le support magnétique pendant au moins 1 min. Récupérez l’élu dans de nouveaux tubes. Répétez l’opération 1 fois et récupérez l’élu dans les mêmes tubes.
- Ajoutez 100 μL supplémentaires de tampon d’élution à chaque échantillon d’élution ChIP et 180 μL de tampon d’élution à chaque échantillon d’entrée.
- Ajouter 200 μL d’alcool phénol/chloroforme/isoamylique (25:24:1) à chaque échantillon et agiter vigoureusement. Centrifuger les échantillons à 20 000 x g (ou vitesse maximale) pendant 10 min à 4 °C.
- Ajouter 19 μL d’acétate de sodium 3M (NaOAc, pH 5,2 ; voir le tableau 1) et 2 μL de solution de glycogène (20 mg/mL, voir le tableau des matières) dans chaque nouveau tube à centrifuger de 1,5 mL.
- Après la centrifugation, transférez soigneusement la couche aqueuse supérieure (environ 190 μL) dans les tubes contenant du NaOAc et du glycogène et mélangez par vortex.
- Ajouter 500 μL d’éthanol à 100 % et vortex. Faites précipiter l’ADN en incubant les échantillons à -20 °C pendant au moins 2 h ou toute la nuit.
- Centrifuger les tubes à 20 000 x g (ou vitesse maximale) pendant 10 min à 4 °C. Jetez le surnageant en prenant soin de ne pas déranger la pastille blanche. Remettez la pastille en suspension dans 1 mL d’éthanol à 70 % et agitez abondamment.
- Centrifuger les tubes à 20 000 x g (ou vitesse maximale) pendant 5 min à 4 °C. Retirez délicatement le surnageant. Centrifugez brièvement les tubes à nouveau pour faire tourner l’éthanol résiduel. Prélevez délicatement l’éthanol à l’aide d’une pipette P20. Faites sécher les granulés d’ADN à l’air libre pendant 2-3 min.
REMARQUE : Évitez de trop sécher la pastille, car cela peut rendre l’ADN difficile à dissoudre. - Pour les échantillons ChIP, remettre l’ADN en suspension dans 400 μL de tampon TE (voir le tableau 1). Pour l’ADN d’entrée, remettre en suspension dans 1000 μL de tampon TE. Les échantillons élués peuvent désormais être conservés à -20°C.
6. Quantification de la qPCR
- Effectuez une qPCR à l’aide d’un kit commercial (voir le tableau des matériaux) avec des triplets techniques pour chaque échantillon. Confirmer la présence d’un seul produit de PCR spécifique en effectuant une analyse de la courbe de fusion pour garantir la spécificité de l’amplification36.
- Analyse des données
REMARQUE : Dans l’analyse de quantification relative, l’échantillon d’essai est exprimé en un changement de pli par rapport à un échantillon témoin (immunoprécipité à l’aide d’IgG purifiées normales ou d’IP fictives). Les locus d’ADN connus pour être inoccupés par la protéine immunoprécipitée (locus négatif) peuvent être utilisés de cette manière comme gène de référence par rapport aux locus d’ADN36 de contrôle positif connus, occupés et positifs.- Calculez le pourcentage d’entrée pour chaque ChIP à l’aide de la formule ci-dessous
%Entrée = 2(-ΔCt [ChIP normalisé]) - Normalisez les valeurs ΔCt du locus positif en locus négatif (ΔΔCt) en soustrayant la valeur ΔCt obtenue pour le locus positif de la valeur ΔCt pour le locus négatif à l’aide de la formule ci-dessous
(ΔΔCt = ΔCtpositif - ΔCtnégatif) - Calculez l’enrichissement du pli de la séquence du locus positif dans l’ADN ChIP sur le locus négatif à l’aide de la formule ci-dessous
Enrichissement du pli =2ΔΔCt
Les séquences des amorces de qPCR utilisées pour l’analyse sont fournies dans le tableau 2. L’organisation génomique de FRA3B et FRA16D37est illustrée à la figure 3A,B.
- Calculez le pourcentage d’entrée pour chaque ChIP à l’aide de la formule ci-dessous
- Analyse statistique
- Analysez les résultats statistiquement à l’aide du test t apparié de Student. Une valeur p de ≤0,05 est considérée comme statistiquement significative, ce qui indique qu’il est peu probable que les différences observées soient dues à une variation aléatoire38.
Résultats
La taille des fragments de chromatine est cruciale pour le succès de la ChIP native, car elle a un impact direct sur l’accessibilité des régions de l’ADN pour la liaison aux anticorps. Pour déterminer la concentration optimale de MNase pour la fragmentation de la chromatine, nous avons préparé une série de tubes de microcentrifugation contenant des concentrations variables de MNase (c’est-à-dire 0,0625 U, 0,125 U, 0,25 U, 0,5 U, 1 U, 2 U, 4 U, 8 U par réaction) et 40 μL de noyaux isolés. Chaque réaction a été incubée à 37 °C pendant 5 minutes pour obtenir une gamme de tailles de fragments de chromatine. Les résultats de la digestion de la MNase sont présentés à la figure 2.
Comme le montre la figure 2, des concentrations plus élevées de MNase ont entraîné une digestion plus étendue de la chromatine, ce qui a entraîné une prédominance de fragments de mononucléosomes (figure 2, voies 1, 2 et 3). En revanche, à des concentrations de MNase plus faibles, la majorité des fragments de chromatine étaient plus gros, dépassant souvent 1 kb (figure 2, voies 6, 7 et 8), indiquant une digestion insuffisante pour les applications en aval. Avec une concentration de MNase de 1,6 unité et un temps de digestion de 5 min, nous avons obtenu des fragments de chromatine principalement dans la gamme de 200 à 1000 paires de bases (Figure 2, voie 5). Cette taille de fragment est idéale pour le ChIP natif, car elle assure un enrichissement efficace de l’ADN associé à la chromatine tout en maintenant l’accessibilité à des épitopes spécifiques. La taille appropriée des fragments facilite l’immunoprécipitation efficace et l’analyse en aval, telle que la PCR quantitative ou le séquençage, ce qui la rend adaptée à la détection des interactions protéine-ADN avec une haute résolution.
L’affinité de liaison de l’anticorps est un facteur essentiel pour le succès de la ChIP native. Il est essentiel de s’assurer que l’anticorps se lie efficacement et spécifiquement à son épitope cible pour obtenir des résultats fiables. Par conséquent, il est crucial de vérifier à l’avance l’efficacité de liaison de l’anticorps. Pour confirmer l’efficacité de l’immunoprécipitation γH2A.X, nous avons effectué un test par western blot (WB).
Nous avons comparé les niveaux de γH2A.X entre des cellules traitées au DMSO (contrôle), des cellules traitées à l’aphidicoline et des cellules traitées à l’hydroxyurée (stressées par réplication) à l’aide du western blot. Comme le montre la figure 4A (entrée), les niveaux de γH2A.X étaient significativement plus élevés dans les cellules traitées à l’aphidicoline par rapport aux cellules traitées au DMSO. Cette augmentation de γH2A.X est cohérente avec l’induction du stress de réplication par l’aphidicoline, ce qui conduit à une augmentation de la formation de cassures double brin et à la phosphorylation ultérieure de γH2A.X. Après avoir effectué la ChIP, nous avons vérifié la spécificité de l’immunoprécipitation en comparant l’enrichissement en γH2A.X dans les échantillons de contrôle IgG et de γH2A.X IP. Le témoin IgG n’a pas permis d’extraire de γH2A.X détectable, ce qui indique une liaison non spécifique minimale (figure 4B, N-ChIP et X-ChIP). En revanche, l’échantillon γH2A.X IP a réussi à enrichir γH2A.X à partir des cellules traitées à l’aphidicoline, démontrant une liaison efficace et spécifique de l’anticorps à γH2A.X (Figure 4, N-ChIP et X-ChIP). Ces résultats confirment que l’anticorps γH2A.X a une affinité de liaison et une spécificité suffisantes pour être utilisé dans des expériences de ChIP native, permettant la détection et l’enrichissement fiables des régions de chromatine liées à γH2A.X. Cette étape de validation est essentielle pour garantir la qualité et la précision des analyses ChIP ultérieures.
Nous avons utilisé les sites fragiles communs (CFS) FRA16D et FRA3B pour évaluer la spécificité de la liaison de γH2A.X à l’ADN en réponse au stress de réplication. FRA16D et FRA3B sont des régions CFS bien connues sujettes à l’instabilité dans des conditions de stress de réplication, ce qui en fait des candidates idéales pour valider les interactions γH2A.X/ADN. Les figures 3A et B illustrent l’organisation génomique de ces deux SFC, ainsi que les amorces spécifiques de qPCR utilisées pour l’analyse.
Pour déterminer si γH2A.X s’associe à ces régions du SFC en réponse au stress de réplication, nous avons effectué une analyse ChIP-qPCR dans des cellules témoins (traitées au DMSO), des cellules traitées à l’aphidicoline et des cellules traitées à l’hydroxyuréas. Les résultats de la qPCR ont montré que dans les cellules témoins traitées au DMSO, γH2A.X n’était pas enrichi au niveau des loci FRA16D ou FRA3B, ce qui suggère une absence de dommages significatifs à l’ADN ou de stress de réplication dans ces régions (Figure 3C). Cependant, dans les cellules traitées avec de l’aphidicoline ou de l’hydroxyurée, qui induit un stress de réplication en inhibant l’ADN polymérase, nous avons observé un enrichissement significatif de γH2A.X à la fois à FRA16D et FRA3B (Figure 3C). Cela indique que γH2A.X a été recruté dans ces régions du SFC spécifiquement en réponse au stress de réplication de l’ADN. Ces résultats démontrent que l’occupation de γH2A.X au niveau des SFC est déclenchée par le stress de réplication, soutenant son rôle de marqueur de la réponse aux dommages de l’ADN et du blocage de la fourche de réplication. Le recrutement spécifique de γH2A.X pour FRA16D et FRA3B pendant le traitement par l’aphidicoline valide davantage la spécificité de notre test ChIP et confirme que γH2A.X est un indicateur fiable des dommages à l’ADN et du stress de réplication sur les sites fragiles.
Pour comparer l’efficacité de la ChIP native avec la ChIP réticulée (X-ChIP), nous avons effectué γH2A.X X-ChIP en utilisant le même nombre de cellules de départ, en suivant le protocole décrit par Lyu et al.39. Le recrutement de γH2A.X pour FRA16D a été augmenté par le traitement à l’aphidicoline et à l’hydroxyurée, comme le montre la figure 3D. Cependant, l’enrichissement relatif de γH2A.X à FRA16D dans X-ChIP était nettement inférieur à celui observé avec ChIP natif, indiquant une efficacité réduite de X-ChIP dans ces conditions. De plus, à FRA3B, l’augmentation de l’enrichissement en γH2A.X était statistiquement non significative, ce qui fournit une preuve supplémentaire que X-ChIP est moins efficace lorsqu’il travaille avec un nombre limité de cellules. Ces résultats suggèrent que la ChIP native pourrait être plus appropriée pour détecter l’enrichissement en γH2A.X dans les sites fragiles lorsque la taille de l’échantillon est limitée. Analysez les résultats statistiquement à l’aide du test t apparié de Student. Une valeur p de ≤ 0,05 est considérée comme statistiquement significative.

Figure 1 : Workflow pour la réalisation d’un test natif γH2A.X ChIP. (i) Les cellules sont cultivées et soumises à des traitements spécifiques (par exemple, des agents endommageant l’ADN comme l’hydroxyurée) pour induire la signalisation γH2A.X. (ii) Les cellules traitées sont collectées pour être traitées. (iii) Les cellules sont fractionnées pour isoler le γH2A.X lié à la chromatine, le séparant ainsi des autres composants cellulaires. (iv) La digestion des nucléases micrococciques (MNase) est effectuée pour fragmenter la chromatine en morceaux de la taille d’un nucléosome. (v) La taille des fragments de chromatine est validée en analysant un échantillon sur un gel d’agarose, en s’assurant que les fragments sont de la longueur appropriée pour la ChIP. (vi) Un anticorps spécifique de γH2A.X est utilisé pour immunoprécipiter les fragments de chromatine marqués par γH2A.X. (vii) Les billes de protéine A/G sont utilisées pour extraire les complexes anticorps-chromatine, enrichissant ainsi l’ADN lié à γH2A.X. viii) Les fragments d’ADN marqués γH2A.X sont élués et purifiés, ce qui les prépare aux analyses en aval. (ix) Analyse en aval : qPCR et séquençage et analyse des données. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Dosage pour optimiser les conditions de digestion de la MNase. Des fragments de chromatine ont été préparés à partir de cellules HEK 293T en utilisant différentes conditions de digestion de la MNase. L’ADN purifié résultant de la chromatine fragmentée a été séparé sur un gel d’agarose à 2 % et traité à 100 V pendant 30 min. Les voies 1 à 8 représentent des échantillons traités avec des concentrations croissantes de MNase (c.-à-d. 0,0625 U, 0,125 U, 0,25 U, 0,5 U, 1 U, 2 U, 4 U, 8 U par réaction), démontrant un gradient de digestion de la chromatine. L’échelle d’ADN (indiquée par M) a été incluse à titre de référence de taille. Des fragments d’ADN ont été visualisés à l’aide de la solution de coloration de l’ADN Midori Green Advance. La bande marquée correspond à des fragments d’ADN de la taille d’un mononucléosome, mettant en évidence l’efficacité de la digestion à chaque concentration de MNase. Veuillez cliquer ici pour voir une version agrandie de cette figure.
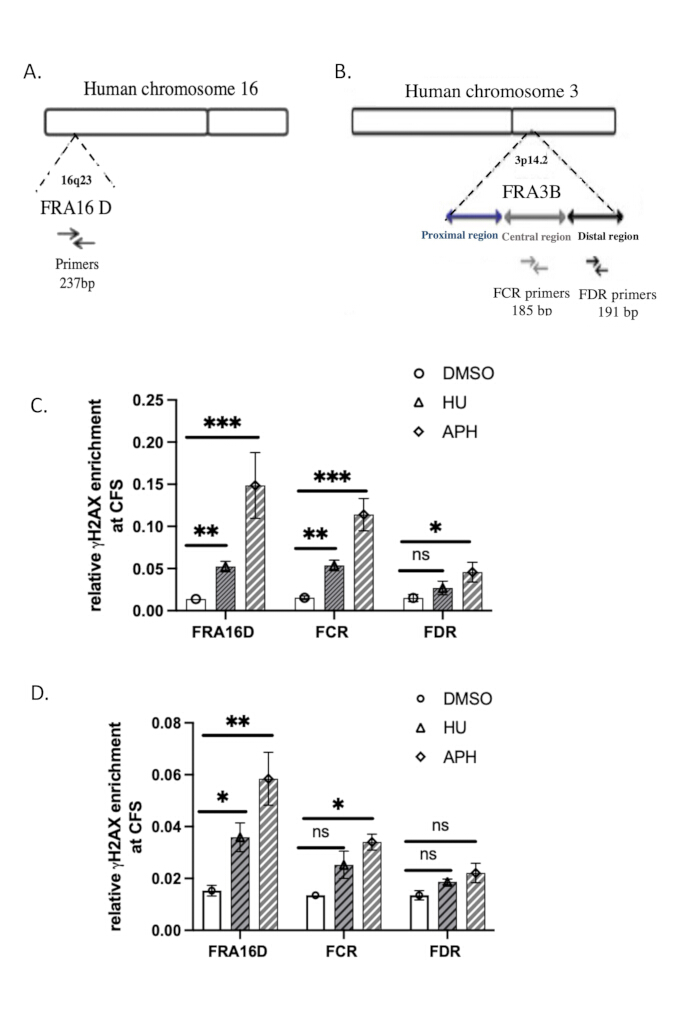
Figure 3 : Vérification qPCR de γH2A.X ChIP. (A) Schéma des organisations génomiques de la FRA16D. (B) Schéma des organisations génomiques de la région FRA3B. Les ensembles d’amorces utilisés pour les analyses qPCR des régions FRA16D, distales (FDR) et centrales (FCR) au sein du locus FRA3B sont indiqués. (C) L’enrichissement relatif en γH2A.X au niveau des sites fragiles communs (SFC), en particulier FRA3B et FRA16D, a été évalué à l’aide de la ChIP native suivie d’une analyse qPCR après traitement à l’aphidicoline et à l’hydroxyurée. (D) L’enrichissement relatif en γH2A.X au niveau des sites fragiles communs (SFC), en particulier FRA3B et FRA16D, a été évalué à l’aide de X-ChIP suivi d’une analyse qPCR après traitement à l’aphidicoline et à l’hydroxyurée. indique un résultat statistiquement significatif avec p < 0,001 ; ** indique p < 0,01 et * indique p < 0,05, déterminé par un test t ; n = 3. Veuillez cliquer ici pour voir une version agrandie de cette figure.
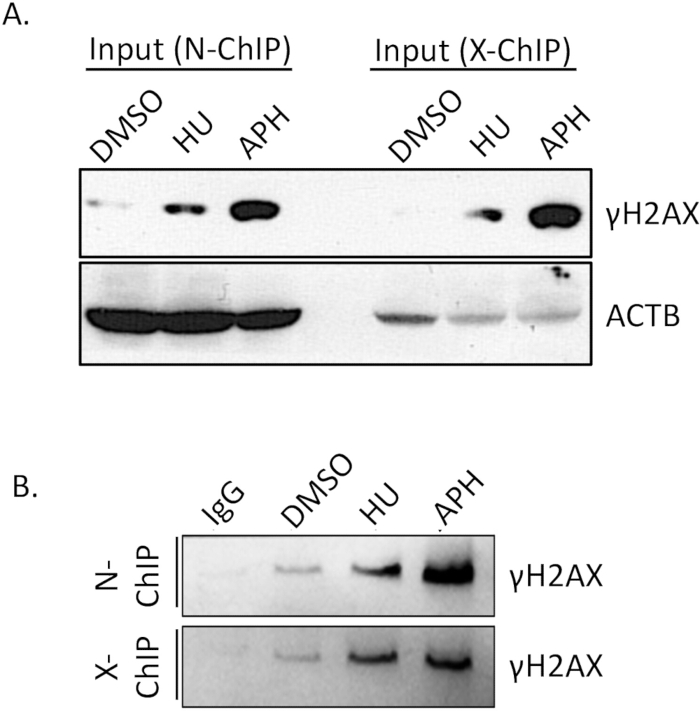
Figure 4 : Vérification de l’efficacité de l’extraction de l’anticorps ChIP à l’aide du Western blot. De l’échantillon total, 10 % de l’échantillon ChIP, ainsi que les contrôles d’entrée, ont été séparés sur un gel SDS-PAGE à 15 % pour résoudre les polypeptides. Après électrophorèse, les protéines séparées ont été transférées sur une membrane PVDF de 0,22 μm à l’aide d’un protocole de transfert standard. La membrane a ensuite été sondée séquentiellement avec un anticorps primaire spécifique ciblant γH2A.X et ACTB, suivi d’un anticorps secondaire approprié conjugué à HRP. Le signal a ensuite été visualisé à l’aide de la détection par chimiluminescence, ce qui a permis d’évaluer la spécificité de l’anticorps et l’efficacité de la traction dans le test ChIP. (A) Niveaux de γH2A.X entre les cellules traitées au DMSO (contrôle), les cellules traitées à l’aphidicoline et les cellules traitées à l’hydroxyurée (stressées par réplication) avec ACTB comme contrôle de charge. (B) Enrichissement en γH2A.X dans les échantillons de contrôle IgG et IP. Abréviations : APH = aphidicoline, HU = hydroxyurée. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Zone tampon A (1X) | |
| Réactif | Concentration de travail |
| TUYAUX pH 6,8 | 10 millions d’euros |
| NaCl | 100 millions d’euros |
| MgCl2 | 3 millions d’euros |
| EGTA pH 7,6 | 1 million d’euros |
| Conserver à -20 °C jusqu’à 1 an | |
| Tampon MNase (1X) | |
| Réactif | Concentration de travail |
| Tris-HCl, pH 7,5 | 50 millions d’euros |
| CaCl2 | 1 million d’euros |
| MgCl2 | 4 millions d’euros |
| Conserver à -20 °C jusqu’à 1 an | |
| Tampon B (1X) | |
| Réactif | Concentration de travail |
| Phosphate de sodium pH 7. 0 | 20 millions d’euros |
| Pyrophosphate de sodium10. H2O | 30 millions d’euros |
| Kcl | 250 millions d’euros |
| EDTA, pH 8,0 | 5 millions d’euros |
| Glycérol | 10% |
| Triton X-100 | 0.10% |
| Cocktail d’inhibiteurs de protéase | 1x |
| Cocktail d’inhibiteurs de la phosphatase | 1x |
| La TNT | 0,5 million d’euros |
| Conserver à -20 °C jusqu’à 1 an | |
| Tampon d’élution | |
| Réactif | Concentration de travail |
| NaHCO3 | 50 millions d’euros |
| SDS | 1% |
| Fraîchement préparé | |
| Tampon de lavage | |
| Réactif | Concentration de travail |
| Tampon MNase | x 0,5 |
| Tampon B | x 0,5 |
| Frais préparé | |
| Tampon d’arrêt | |
| EDTA | 100 mM , pH 8,0 |
| 3 M Acétate de sodium, pH 5,2 | |
| 408,24 g Acétate de sodium | |
| ajuster le pH avec de l’acide acétique à pH 5,2 | |
| ajuster le volume à 1 L | |
| Stériliser le tampon par filtration ou autoclave | |
| Tampon TBST | |
| Base Tris, pH 8,0 | 10 millions d’euros |
| NaCl | 150 millions de mètres |
| Préadolescent 20 | 0,1 % (v/v) |
| Tampon TE | |
| Tris-HCl, pH 8,0 | 10 millions d’euros |
| EDTA, pH 8,0 | 1 million d’euros |
Tableau 1 : Composition du tampon.
| Loci génétiques | Apprêt avant | Amorce inversée |
| FRA16D | TCCTGTGGAAGGGATATTTA | CCCCTCATATTCTGCTTCTA |
| FRA3B | TGTTGGAATGTTAACTCTATCCCAT | ATATCTCATCAAGACCGCTGCA |
| FCR | ||
| FRA3B | CAATGGCTTAAGCAGACATGGT | AGTGAATGGCATGGCTGGAATG |
| FDR | ||
| ACTB (contrôle négatif) | GACGCAGGATGGCATGGG | ACGCCTCTGGCCGTACCAC |
Tableau 2 : Séquence d’amorces.
| Faible récupération de l’ADN | Problème potentiel | Solution possible |
| Mauvaise qualité de l’anticorps | Utilisez un anticorps de grade ChIP. | |
| Anticorps insuffisants | Utilisez 1 à 10 μg d’anticorps ChIP pour 25 μg de chromatine. | |
| Temps d’incubation insuffisant | Effectuez l’étape d’immunoprécipitation pendant la nuit à 4 °C. | |
| Lavages trop stricts | N’utilisez pas une concentration de NaCl supérieure à 500 mM dans le tampon de lavage. | |
| Perles de mauvaise qualité | Assurez-vous que la protéine A ou G est compatible avec l’anticorps ChIP. | |
| Suivez la fiche technique du produit de billes pour un rapport volume optimal de billes / anticorps. | ||
| Démarrage insuffisant | Préparez une plaque distincte de cellules pour déterminer avec précision le numéro de cellule. | |
| Échantillon | Augmentez le nombre de cellules utilisées si la cible est une faible abondance. | |
| Lyse cellulaire inadéquate | Optimisez la composition du tampon et le temps de lyse pour améliorer l’efficacité. | |
| Appliquez une force mécanique pendant l’étape de lyse, par exemple en utilisant un homogénéisateur Dounce, lorsque vous travaillez avec certains types de cellules. | ||
| Dégradation des noyaux | Assurez-vous que l’isolation des noyaux est douce pour préserver l’intégrité et l’accessibilité de la chromatine. | |
| Utilisez des pointes de pipette à large alésage pour minimiser le cisaillement des noyaux lors de la manipulation. | ||
| Une dégradation de l’échantillon s’est produite | Effectuez toutes les étapes sur de la glace ou à 4 °C. | |
| Incluez des inhibiteurs de protéase dans tous les tampons et assurez-vous que tous les tampons sont fraîchement préparés. | ||
| Digestion insuffisante de la MNase | Titrer la concentration de MNase (p. ex., 0,1 à 5 unités) pour déterminer la quantité optimale pour un type d’échantillon spécifique ; et commencer par une gamme de concentrations pour identifier les conditions idéales de digestion. | |
| MNase surdigestion | Faites des essais avec différents temps d’incubation (p. ex., 1 à 10 min) à 37 °C et surveillez le schéma de fragmentation pour assurer une digestion suffisante sans surdigestion. | |
| Phosphorylation perturbée | Ajoutez un cocktail d’inhibiteurs de la phosphatase au tampon de lyse et assurez-vous que tous les tampons sont fraîchement préparés. | |
| Contexte élevé en contrôle négatif | Liaison non spécifique aux perles | Incluez une étape de pré-nettoyage avant l’étape d’immunoprécipitation et utilisez des billes magnétiques de qualité ChIP, qui présentent généralement une liaison non spécifique réduite. |
| Lavage insuffisant | Augmentez le nombre ou la rigueur des lavages en ajustant la concentration de sel et de détergent. | |
| Fragmentation insuffisante de la chromatine | Optimisez la fragmentation pour obtenir des fragments de 200-750 pb. | |
| L’optimisation est nécessaire pour chaque type de cellule ou de tissu | ||
| Dépannage PCR | Pas d’amplification de l’échantillon d’entrée | (1) Sur-fragmenté : le signal est diminué pour des amplicons de plus de 150 pb si la chromatine est fragmentée à la longueur d’un mononucléosome. |
| (2) Optimiser les conditions de PCR. | ||
| (3) Concevoir des amorces pour amplifier une région plus petite (<150 pb). | ||
| Basse résolution | Taille du fragment d’ADN trop grande | Optimisez la fragmentation pour obtenir des fragments de 200-750 pb. |
Tableau 3 : Dépannage de l’immunoprécipitation de la chromatine.
Discussion
La pollution de l’environnement est un contributeur important aux cancers humains. De nombreux polluants sont cancérigènes, ce qui signifie qu’ils peuvent causer des dommages génétiques qui conduisent au développement d’un cancer40,41. Cependant, déterminer si une substance particulière est tumorigène est une tâche difficile. Une méthode rapide, fiable et rentable pour identifier le potentiel cancérogène permettrait aux scientifiques de dépister efficacement les polluants environnementaux et d’évaluer leur impact sur la stabilité génomique. Dans cette étude, nous nous concentrons sur une variante d’histones modifiée, γH2A.X, qui est largement reconnue comme un marqueur hautement spécifique pour les DSBs. γH2A.X se forme lorsque la variante d’histones H2A. X est phosphorylé à la sérine 139 en réponse à des dommages à l’ADN.
En détectant la présence de γH2A.X par la ChIP native au niveau des SFC, nous pouvons évaluer efficacement l’étendue des dommages à l’ADN causés par les contaminants environnementaux42. La capacité de mesurer quantitativement γH2A.X au niveau des SFC offre un moyen rapide, sensible et fiable d’évaluer le potentiel des polluants environnementaux à induire des effets tumorigènes. Contrairement aux tests de génotoxicité traditionnels, qui mesurent souvent des marqueurs indirects de dommages à l’ADN (tels que les mutations, la formation de micronoyaux ou la transformation cellulaire), le γH2AX ChIP natif permet une cartographie précise des événements de rupture au niveau de loci génomiques spécifiques42. Cela permet de mieux comprendre la région la plus vulnérable aux polluants environnementaux et aide à identifier les mécanismes cancérogènes potentiels.
La réussite d’un ChIP natif nécessite une attention particulière à plusieurs étapes critiques pour garantir l’enrichissement efficace des régions cibles. Vous trouverez ci-dessous les étapes critiques impliquées dans le ChIP natif de γH2AX. Préparation des cellules : Commencez avec un nombre adéquat de cellules. Traitez les cellules avec un agent induisant des dommages à l’ADN, tel que l’aphidicoline, pour induire le stress de réplication en tant que contrôle positif. Inclure des échantillons de témoins négatifs non traités appropriés à des fins de comparaison. Isolement de la chromatine : Effectuez un fractionnement subcellulaire pour isoler les protéines liées à la chromatine tout en minimisant la contamination par les composants cytoplasmiques. Optimisation de la MNase : Mener des expériences pilotes pour optimiser la concentration de nucléases micrococciques (MNase) et le temps d’incubation. L’objectif est d’obtenir des fragments de chromatine entre 200 et 1000 paires de bases, assurant une bonne résolution pour la ChIP tout en préservant l’intégrité des nucléosomes. Immunoprécipitation : Utilisez un anticorps γH2AX de haute qualité de qualité ChIP avec de l’agarose protéique A/G pour assurer une réduction efficace de la chromatine cible tout en minimisant la liaison non spécifique. Lavage : Lavez les billes avec un tampon contenant la concentration appropriée de sel et de détergent pour éliminer les interactions non spécifiques tout en conservant la chromatine liée à γH2AX. Élution et récupération de l’ADN : Récupérez efficacement la chromatine liée par élution et précipitation approfondies de l’ADN, garantissant une perte minimale de la chromatine cible enrichie. Par rapport au X-ChIP conventionnel, le γH2AX ChIP natif présente quatre avantages principaux. Tout d’abord, l’intégration du fractionnement subcellulaire réduit les interactions faussement positives et améliore la spécificité de la ChIP. Deuxièmement, les complexes protéine-ADN sont conservés dans leur état natif, minimisant la stabilisation des interactions transitoires γH2AX-chromatine qui peuvent se produire avec des agents de réticulation. Troisièmement, l’omission de la fixation et les étapes chronophages de réticulation inverse rationalisent le flux de travail global. Enfin, moins de matériau de départ est nécessaire et aucun équipement coûteux n’est nécessaire pour le cisaillement de la chromatine, tel qu’un sonicateur à bain d’eau (par exemple, Bioruptor) ou un sonicateur à ultrasons focalisés (par exemple, Covaris).
Bien que le Native ChIP ait ses avantages, il faut également reconnaître ses limites. L’une des limites est le potentiel de réarrangement des protéines pendant la préparation de la chromatine et l’immunoprécipitation. Ce réarrangement pourrait théoriquement modifier les interactions des protéines liées à la chromatine et introduire de la variabilité dans les résultats. Bien que nous n’ayons pas de preuves directes à l’appui de l’existence de tels réarrangements, il est important d’inclure des contrôles expérimentaux appropriés pour tenir compte de cet artefact potentiel. Une autre limitation du test ChIP natif est le biais de clivage de la MNase, qui peut conduire à des résultats inexacts. La MNase clive préférentiellement les régions riches en A-T du génome, ce qui rend ces régions plus susceptibles d’être fragmentées et enrichies au cours du processus ChIP. Ce biais peut entraîner une surreprésentation des séquences riches en A-T et une sous-représentation des régions riches en G-C, ce qui peut entraîner des signaux faussement négatifs pour les régions génomiques qui sont moins accessibles à la digestion de la MNase. Si les loci d’intérêt sont riches en G-C, une optimisation minutieuse des conditions de digestion de la MNase est nécessaire. Par ailleurs, l’incorporation de contrôles supplémentaires, tels que la fragmentation de la chromatine par sonication, peut aider à atténuer l’impact du biais MNase et à améliorer la fiabilité des résultats.
Malgré ses limites certaines, la ChIP γH2AX native est particulièrement utile pour identifier les loci de génération où des polluants spécifiques induisent des dommages à l’ADN. En enrichissant des fragments d’ADN liés à γH2AX, les chercheurs peuvent identifier des régions fragiles du génome, en particulier les SFC par qPCR, qui sont plus sujettes à la rupture sous l’effet du stress de réplication induit par les polluants. Cette technique permet également aux chercheurs d’établir une relation directe entre l’exposition à un cancérogène spécifique et la formation de dommages à l’ADN dans des régions génomiques particulières, ce qui donne un aperçu du profil génotoxique des polluants et de leur rôle potentiel dans l’initiation de la cancérogenèse43,44.
Le tableau de dépannage (Tableau 3) fournit des solutions détaillées aux problèmes courants rencontrés lors du test ChIP, tels qu’un faible rendement en ADN, une fragmentation insuffisante de la chromatine, un bruit de fond élevé ou une mauvaise performance des anticorps. Chaque problème est associé à des recommandations spécifiques pour garantir la fiabilité et la reproductibilité du protocole.
En utilisant des systèmes automatisés de manipulation de liquides et des technologies quantitatives de PCR ou de séquençage, le γH2AX ChIP natif peut identifier efficacement les agents qui induisent des dommages à l’ADN, constituant ainsi un outil précieux dans les études toxicologiques et la surveillance environnementale. En utilisant le γH2AX ChIP natif sur des échantillons provenant de populations exposées à différents niveaux de polluants environnementaux, les chercheurs peuvent identifier des modèles spécifiques de dommages à l’ADN et déterminer le niveau de risque associé à l’exposition à des agents particuliers. En conclusion, le γH2AX ChIP natif offre des avantages significatifs pour l’évaluation du potentiel cancérogène des polluants environnementaux, fournissant une approche rapide, rentable et hautement spécifique pour détecter les dommages à l’ADN à des locus génomiques spécifiques. Ses applications futures dans le dépistage à haut débit, l’évaluation personnalisée des risques et le développement de biomarqueurs en font un outil précieux pour faire progresser notre compréhension de la cancérogenèse environnementale et développer des stratégies de prévention et d’intervention contre le cancer.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Ce travail a été soutenu par le financement de démarrage de l’Université de Chine du Sud.
matériels
| Name | Company | Catalog Number | Comments |
| 0.2 µm nitrocellulose membrane | Amersham | 10600011 | |
| Actin B | proteintech | 20536-1-AP | |
| Aphidicolin | MedChemExpress | HY-N6733 | |
| ChIP-grade magnetic Protein A/G beads | ThermoFisher | 26162 | |
| Clarity Western ECL Substrate | Bio-Rad | #1705061 | |
| Glycogen, molecular biology grade | ThermoFisher | Cat. No. R0561 | |
| HRP-conjugated secondary antibody | proteintech | SA00001-2 | |
| hydroxyurea | MedChemExpress | HY-B0313 | |
| Micrococcal Nuclease | NEB | M0247S | |
| normal IgG | Santa Cruz | sc-2025 | |
| Taq Universal SYBR Green Supermix | BioRad | 1725120 | |
| γH2A.X antibody (for ChIP) | Sigma-Aldrich | 05-636 | |
| γH2A.X antibody (for WB) | Cell Signaling | #25955 |
Références
- Glover, T. W., Berger, C., Coyle, J., Echo, B. DNA polymerase alpha inhibition by aphidicolin induces gaps and breaks at common fragile sites in human chromosomes. Hum Genet. 67 (2), 136-142 (1984).
- Bignell, G. R., et al. Signatures of mutation and selection in the cancer genome. Nature. 463 (7283), 893-898 (2010).
- Hellman, A., et al. A role for common fragile site induction in amplification of human oncogenes. Cancer Cell. 1 (1), 89-97 (2002).
- Kotzot, D., et al. Parental origin and mechanisms of formation of cytogenetically recognisable de novo direct and inverted duplications. J Med Genet. 37 (4), 281-286 (2000).
- Miller, C. T., et al. Genomic amplification of MET with boundaries within fragile site FRA7G and upregulation of MET pathways in esophageal adenocarcinoma. Oncogene. 25 (3), 409-418 (2006).
- Zack, T. I., et al. Pan-cancer patterns of somatic copy number alteration. Nat Genet. 45 (10), 1134-1140 (2013).
- Gao, G., et al. Common fragile sites (CFS) and extremely large CFS genes are targets for human papillomavirus integrations and chromosome rearrangements in oropharyngeal squamous cell carcinoma. Genes Chromosomes Cancer. 56 (1), 59-74 (2017).
- Thorland, E. C., Myers, S. L., Gostout, B. S., Smith, D. I. Common fragile sites are preferential targets for HPV16 integrations in cervical tumors. Oncogene. 22 (8), 1225-1377 (2003).
- Matovina, M., Sabol, I., Grubisić, G., Gasperov, N. M., Grce, M. Identification of human papillomavirus type 16 integration sites in high-grade precancerous cervical lesions. Gynecol Oncol. 113 (1), 120-127 (2009).
- Yu, T., et al. The role of viral integration in the development of cervical cancer. Cancer Genet Cytogenet. 158 (1), 27-34 (2005).
- Bignell, G. R., et al. Signatures of mutation and selection in the cancer genome. Nature. 463 (7283), 893-898 (2010).
- Thavathiru, E., Ludes-Meyers, J. H., MacLeod, M. C., Aldaz, C. M. Expression of common chromosomal fragile site genes, WWOX/FRA16D and FHIT/FRA3B is downregulated by exposure to environmental carcinogens, UV, and BPDE but not by IR. Mol Carcinog. 44 (3), 174-182 (2005).
- Sirbu, B. M., et al. Analysis of protein dynamics at active, stalled, and collapsed replication forks. Genes Dev. 25 (12), 1320-1327 (2011).
- Barlow, J. H., et al. Identification of early replicating fragile sites that contribute to genome instability. Cell. 152 (3), 620-632 (2013).
- Petermann, E., Orta, M. L., Issaeva, N., Schultz, N., Helleday, T. Hydroxyurea-stalled replication forks become progressively inactivated and require two different RAD51-mediated pathways for restart and repair. Mol Cell. 37 (4), 492-502 (2010).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. J Biol Chem. 273 (10), 5858-5868 (1998).
- Shanbhag, N. M., Rafalska-Metcalf, I. U., Balane-Bolivar, C., Janicki, S. M., Greenberg, R. A. ATM-dependent chromatin changes silence transcription in cis to DNA double-strand breaks. Cell. 141 (6), 970-981 (2010).
- Stiff, T., et al. ATR-dependent phosphorylation and activation of ATM in response to UV treatment or replication fork stalling. EMBO J. 25 (24), 5775-5782 (2006).
- Nelson, J. D., Denisenko, O., Bomsztyk, K. Fast chromatin immunoprecipitation assay. Nuc Acids Res. 34 (5), e2 (2006).
- Dorbic, T., Wittig, B. Isolation of oligonucleosomes from active chromatin using HMG17-specific monoclonal antibodies. Nuc Acids Res. 14 (8), 3363-3376 (1986).
- Dorbic, T., Wittig, B. Chromatin from transcribed genes contains HMG17 only downstream from the starting point of transcription. EMBO J. 6 (8), 2393-2399 (1987).
- Hebbes, T. R., Thorne, A. W., Crane-Robinson, C. A direct link between core histone acetylation and transcriptionally active chromatin. EMBO J. 7 (5), 1395-1402 (1988).
- Hebbes, T. R., Thorne, A. W., Clayton, A. L., Crane-Robinson, C. Histone acetylation and globin gene switching. Nuc Acids Res. 20 (5), 1017-1022 (1992).
- Hebbes, T. R., Clayton, A. L., Thorne, A. W., Crane-Robinson, C. Core histone hyperacetylation co-maps with generalized DNase I sensitivity in the chicken beta-globin chromosomal domain. EMBO J. 13 (8), 1823-1830 (1994).
- Orlando, V. Mapping chromosomal proteins in vivo by formaldehyde-crosslinked-chromatin immunoprecipitation. Trend Biochem, Sci. 25 (3), 99-104 (2000).
- Myers, F. A., Evans, D. R., Clayton, A. L., Thorne, A. W., Crane-Robinson, C. Targeted and extended acetylation of histones H4 and H3 at active and inactive genes in chicken embryo erythrocytes. J Biol Chem. 276 (23), 20197-20205 (2001).
- Litt, M. D., Simpson, M., Recillas-Targa, F., Prioleau, M. N., Felsenfeld, G. Transitions in histone acetylation reveal boundaries of three separately regulated neighboring loci. EMBO J. 20 (9), 2224-2235 (2001).
- Madisen, L., Krumm, A., Hebbes, T. R., Groudine, M. The immunoglobulin heavy chain locus control region increases histone acetylation along linked c-myc genes. Mol Cell Biol. 18 (11), 6281-6292 (1998).
- Clayton, A. L., Hebbes, T. R., Thorne, A. W., Crane-Robinson, C. Histone acetylation and gene induction in human cells. FEBS Lett. 336 (1), 23-26 (1993).
- Tseng, Z., Wu, T., Liu, Y., Zhong, M., Xiao, A. Using native chromatin immunoprecipitation to interrogate histone variant protein deposition in embryonic stem cells. Methods Mol Biol. 1176, 11-22 (2014).
- Durkin, S. G., Glover, T. W. Chromosome fragile sites. Ann Rev Genetics. 41, 169-192 (2007).
- Lee, J. B., Keung, A. J. Chromatin immunoprecipitation in human and yeast cells. Methods Mol Biol. 1767, 257-269 (2018).
- Miyamoto, R., Yokoyama, A. Protocol for fractionation-assisted native ChIP (fanChIP) to capture protein-protein/DNA interactions on chromatin. STAR Protoc. 2 (2), 100404 (2021).
- Mendez, J., Stillman, B. Chromatin association of human origin recognition complex, cdc6, and minichromosome maintenance proteins during the cell cycle: Assembly of prereplication complexes in late mitosis. Mol Cell Biol. 20 (22), 8602-8612 (2000).
- Nowak, D. E., Tian, B., Brasier, A. R. Two-step cross-linking method for identification of NF-κB gene network by chromatin immunoprecipitation. BioTechniques. 39 (5), 715-725 (2005).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nat Protoc. 3 (6), 1101-1108 (2008).
- Lu, X., Parvathaneni, S., Hara, T., Lal, A., Sharma, S. Replication stress induces specific enrichment of RECQ1 at common fragile sites FRA3B and FRA16D. Mol Cancer. 12, 29 (2013).
- Ruijter, J. M., et al. Amplification efficiency: linking baseline and bias in the analysis of quantitative PCR data. Nucl Acids Res. 37 (6), e45 (2009).
- Lyu, X., Chastain, M., Chai, W. Genome-wide mapping and profiling of γH2AX binding hotspots in response to different replication stress inducers. BMC Genomics. 20, 579 (2019).
- Farmer, P. B., et al. Molecular epidemiology studies of carcinogenic environmental pollutants. Effects of polycyclic aromatic hydrocarbons (PAHs) in environmental pollution on exogenous and oxidative DNA damage. Mutat Res. 544 (2-3), 397-402 (2003).
- Phillips, D. H., Arlt, V. M. Genotoxicity: damage to DNA and its consequences. EXS. 99, 87-110 (2009).
- Nitsch, S., Schneider, R. Native ChIP: Studying the genome-wide distribution of histone modifications in cells and tissue. Meth Mol Biol. 2846, (2024).
- Nikitina, T., Wang, D., Gomberg, M., Grigoryev, S. A., Zhurkin, V. B. Combined micrococcal nuclease and exonuclease III digestion reveals precise positions of the nucleosome core/linker junctions: implications for high-resolution nucleosome mapping. J Mol Biol. 425 (11), 1946-1960 (2013).
- Teves, S. S., Henikoff, S. Salt fractionation of nucleosomes for genome-wide profiling. Methods Mol Biol. 833, 421-432 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon