Method Article
通过天然 γH2A 捕获常见的脆弱位点断裂。X ChIP
摘要
我们提出了一种通过天然 γH2A.X 染色质免疫沉淀 (ChIP) 检测常见脆弱位点突破的快速有效方法。这种方法显著减少了与传统 γH2A.X ChIP 检测相关的时间和人力,同时保持了结果的高重现性和可靠性。
摘要
暴露于外源性试剂诱导的复制应激可导致常见脆弱位点的 DNA 断裂,这些位点是基因组中已知容易出现结构不稳定的区域。γH2A.X 染色质免疫沉淀 (ChIP) 检测是遗传毒性研究的有力工具,因为 γH2A.X 磷酸化是 DNA 双链断裂的公认标志物。然而,传统的 γH2A.X ChIP 检测通常是劳动密集型的,并且涉及多个耗时的步骤。在这项研究中,我们提出了一种简化但有效的方法,该方法将亚细胞分级分离与天然 ChIP 相结合,以分离 γH2A.X 相关复合物。这种方法特别适用于分析 γH2A.X-染色质相互作用,具有更高的特异性和效率。使用亚细胞组分分离,可有效去除未结合的染色质物质,从而得到纯化的染色质组分。随后在温和条件下进行微球菌核酸酶 (MNase) 消化允许染色质碎裂,同时保留 γH2A.X 与其相关蛋白质复合物之间的生理相互作用。这种保存对于研究参与 DNA 损伤反应通路的天然相互作用伴侣至关重要。这种优化的天然 ChIP 方案大大减少了与传统 γH2A.X ChIP 检测相关的时间和劳动力。简化的程序不仅简化了工作流程,而且产生了高度可重现的结果,使其在需要高通量处理多个样品的环境中特别有利。该方法在专注于基因组稳定性、DNA 修复和染色质生物学的研究中具有广泛的适用性,在这些研究中,准确有效地检测 DNA 损伤位点至关重要。通过采用优化的方案和简化的步骤,该方法能够以更高的灵敏度和最少的样品处理检测脆弱位点的 DNA 损伤,使其成为研究基因组稳定性和 DNA 损伤反应的宝贵工具。
引言
常见脆弱位点 (CFS) 是在每条人类染色体上发现的大染色体区域,在中期容易断裂。在复制应激下,这些区域的复制会显著延迟,从而阻止它们在有丝分裂进入1 之前完全复制,从而最终导致位点特异性的间隙和断裂。CFS 是染色体不稳定的热点,是癌症早期发展过程中染色体重排的主要原因。复制应激通常存在于致瘤条件下,可导致肿瘤抑制基因的丢失和癌基因的扩增,统称为拷贝数变异 (CNV)2,3,4,5,6。此外,CFS 极易发生病毒整合,进一步促进癌症发展 7,8,9,10。在原发性肿瘤的泛癌分析过程中,已在 CFS 区域检测到肿瘤抑制基因的多个纯合缺失。癌症中最常受影响的 CFS 包括 FRA2F、FRA3B、FRA4F、FRA5H 和 FRA16D11。CFS 在外源性致癌剂存在下特别容易破损12。为了评估环境污染物的有害致癌作用,需要一种快速可靠的方法来量化 CFS 破损的发生。
H2A 的磷酸化。共济失调毛细血管扩张症和 Rad3 相关蛋白 (ATR) 或共济失调毛细血管扩张症突变 (ATM) 在丝氨酸残基 139 (γH2A.X) 处的 X 是信号复制叉停滞的关键事件13。γH2A.X 可作为双链断裂 (DSB) 形成前停滞复制叉的指标13,创造有利的染色质环境,以促进修复蛋白有效募集到停滞位点。此外,γH2A.X 可以在前叉塌陷后被招募到破损部位 14,15,这与它在 DSB 修复中的主要作用一致。由于 CFS 断裂与驱动癌症进展的染色体畸变密切相关,因此检测这些断裂有助于了解肿瘤发生的早期阶段。CFS 中 γH2A.X 的存在可用作检测基因组不稳定早期事件的生物标志物。这些信息可以帮助识别潜在的致癌物并评估与接触各种外源性物质相关的风险。通过测量外源性药物诱导的 CFS 处的 DNA 断裂,γH2A.X 染色质 IP (ChIP) 可以深入了解这些药物如何促进肿瘤发生的潜在机制。
在传统的 ChIP(即交联 ChIP,X-ChIP)中,γH2A.X 与其靶 DNA 序列的结合通过可逆甲醛交联来稳定。随后通过超声处理将染色质剪切成约 500 个碱基对 (bp) 的片段,并通过沉降清除所得溶液中的碎片 16,17,18。然后将 ChIP 级 γH2A.X 抗体添加到澄清的染色质组分中,然后添加蛋白 A/G 琼脂糖珠以富集 γH2A.X 结合的染色质区域16、17、18。免疫复合物(即珠子抗体-γH2A.X 靶向 DNA 复合物)用严格的洗涤缓冲液多次洗涤,以去除非特异性结合的 DNA 片段 16,17,18。洗涤后,从免疫复合物中洗脱特异性结合的 DNA。然后逆转甲醛交联,然后使用蛋白酶 K 进行蛋白质消化,然后纯化和浓缩富集的 DNA 16,17,18。为了评估 γH2A.X 相关区域,使用 PCR、定量 PCR (qPCR) 或直接测序 16,17,18。γH2A.X 在特定区域(如 CFS)的占有率由 PCR 或 qPCR 信号的强度决定,该信号与在该位置结合的 γH2A.X 量成正比,从而提供对位点特异性 DNA 损伤和修复事件的见解 16,17,18。
尽管 X-ChIP 是一种强大的实验方法,但它有几个明显的局限性:(i) 由于与固定相关的抗体沉淀效率低下,它需要大量细胞,通常在 1 x 107 到 5 x 107 的范围内,这增加了实验的总体成本19;(ii) 逆转甲醛交联和随后的 DNA 纯化过程既耗时又费力,因此难以保持结果的一致性和可靠性;(iii) 具有次要功能意义的 γH2A.X-DNA 相互作用可能无法与具有更大意义的相互作用区分开来,因为交联步骤可以稳定瞬时相互作用,从而导致检测到可能没有生物学相关性的相互作用19。
天然染色质免疫沉淀(天然 ChIP 或 N-ChIP)是一种重要的生化技术,用于研究生理盐条件下天然染色质环境中的蛋白质-DNA 相互作用。它有助于阐明染色质的时空组织、转录因子结合和组蛋白修饰。天然 ChIP 在更广泛的染色质生物学和表观遗传学领域发挥着长期作用,与 X-ChIP 相比具有独特的优势和局限性。这种方法于 1980 年代后期推出20,涉及通过保留其天然结构的方法从细胞中分离染色质,例如用微球菌核酸酶 (MNase) 消化21。这保留了固有的蛋白质-DNA 和组蛋白-DNA 接触,这使得 Trans ChIP 特别适合研究组蛋白修饰和天然染色质环境中的核小体定位22。高分辨率天然 ChIP 研究表明,使用 MNase 消化将染色质还原为单个核小体,这有助于更准确地定位组蛋白修饰23。此外,由于不涉及化学交联,因此引入可能歪曲蛋白质-DNA 相互作用的偏差或伪影的风险被降至最低24。
与使用甲醛或其他交联剂修复蛋白质-DNA 相互作用的 X-ChIP 相比,天然 ChIP 通过避免潜在的交联伪影,提供更真实的染色质视图。然而,虽然 X-ChIP 通常更适合检测 DNA 和调节蛋白之间的瞬时或动态相互作用25,但天然 ChIP 是稳定蛋白质-DNA 相互作用的理想选择,例如组蛋白或其他染色质结合蛋白26,27。天然 ChIP 的局限性之一是无法捕获低亲和力或瞬时结合事件,这些事件通常通过 X-ChIP25 中的交联来稳定。
表观遗传学领域的重要工作利用天然 ChIP 来揭示不同生物环境中的组蛋白修饰28。这些努力对于定义组蛋白密码至关重要,组蛋白密码是调节基因表达和染色质动力学的组蛋白修饰模式29。虽然 H2A.X 是一种相关性较低的连接组蛋白,即天然 H2A。X ChIP 方法已成功应用于胚胎干细胞30.在这项研究中,我们优化了染色质提取程序,以在人 293T 细胞中进行 γH2A.X 的天然 ChIP(图 1)。羟基脲和哼虫素广泛用于研究 DNA 复制应激、损伤和基因组不稳定性31。在这项研究中,这些药物被应用于细胞以诱导复制应激并在 CFS 产生 DNA 断裂。
使用大约 1 x 106 至 5 x 106 个细胞的起始材料,该方法可分为四个主要阶段:(i) 亚细胞分级分离以分离染色质,(ii) 微球菌核酸酶 (MNase) 消化以片段化染色质,(iii) 免疫沉淀和洗脱,以及 (iv) 通过定量 PCR (qPCR) 进行 DNA 分析。在亚细胞分级分离后进行 ChIP 具有多种益处,并且已在众多研究中得到充分证明 32,33,34,35。这种方法可以去除染色质未结合的蛋白质和其他细胞碎片,从而获得高度纯化的染色质部分。通过在免疫沉淀前分离染色质,亚细胞组分分离有助于维持天然染色质相互作用并减少非染色质相关蛋白的背景噪音,从而获得更特异性和可靠的结果,因为仅保留染色质结合的复合物进行分析。此外,亚细胞组分分离为染色质消化提供了更温和的条件,从而保留了生理性蛋白质-DNA 相互作用,并提供了天然细胞环境中染色质动力学的更准确表示。
使用 γH2AX 的天然 ChIP 来测量外源性药物对常见脆弱部位破损的影响,在癌症研究中具有重要潜力。该技术能够检测因暴露于环境致癌物而诱导的 DNA 损伤,从而深入了解污染物导致基因组不稳定性和癌症发展的分子机制。通过保留天然染色质背景,该方法有助于准确评估与致癌暴露相关的 DNA 损伤模式,有助于评估环境风险和研究污染驱动的肿瘤发生。
研究方案
1. 细胞收获
- 将约 5 x 10个 5 HEK 293T 细胞接种到四个 6 cm 培养皿中,每个培养皿含有 4 mL 完全 DMEM 培养基。
- 24 小时后,用 2 μL 1 mM 哼啶素(参见 材料表)储备液(终浓度为 0.5 μM)处理一个培养皿,用 20 μL 1 M 羟基脲(参见 材料表)储备液(终浓度为 5 mM)处理另一个培养皿以诱导复制应激。将 DMSO 添加到其余两个培养皿中作为对照。
- 处理 24 小时后,丢弃培养基。一个 6 cm 板通常产生大约 2 x 10个 6 个细胞,汇合度为 60%-70%。
- 用 5 mL 冰冷的 1x PBS 冲洗每个培养皿 2 次。使用细胞刮刀分离细胞,并将细胞悬液转移到四个单独的 1.5 mL 试管中。用 P1000 移液器轻轻上下移液,以分离任何细胞团块。
- 将细胞在 4 °C 下以 500 x g 离心 5 分钟,然后弃去上清液。将细胞置于冰上。
2. 亚细胞分级分离
- 将细胞沉淀重悬于 500 μL 新鲜制备的冷缓冲液 A 中(参见 表 1),确保通过轻轻移液完全解离细胞团块。
- 将裂解物在冰上孵育 5-10 分钟。在显微镜下检查裂解过程,以确保细胞完全裂解。
- 取一小份裂解物(约 5 - 10 μL),将其放在干净的显微镜载玻片上。用盖玻片盖住它以避免污染。
- 使用具有适当放大倍率(例如 20 倍 - 40 倍)的光学显微镜来观察细胞或碎片。与未裂解的对照样品进行比较,以区分完整细胞和裂解材料。
注意:正确裂解的样品没有明显的细胞轮廓,只有弥漫的染色质或细胞材料。调整焦距以清楚地观察裂解物。如果需要,在裂解步骤中施加机械力,例如在处理某些细胞类型时使用 Dounce 匀浆器。
- 一旦细胞完全裂解,在 4 °C 下以 500 x g 离心 5 分钟。小心丢弃上清液。使用广口移液器吸头将细胞核沉淀重悬于 500 μL 冷缓冲液 A 中。
注:广口吸头有助于最大限度地减少剪切力并保护染色质等易碎样品。用锋利的刀片切割标准刀尖的末端,制作大口径刀尖。 - 在 4 °C 下以 500 x g 离心 5 分钟。 小心丢弃上清液。
- 将培养箱预热至 37 °C 并制备终止缓冲液(100 mM EDTA,pH 8.0; 表 1)。
- 提前优化微球菌核酸酶(MNase,参见 材料表)浓度和孵育时间。
- 将 40 μL 测试染色质样品分成几个相等的等分试样,以测试不同的 MNase 浓度和孵育时间。
- 使用一系列 MNase 浓度(例如,每个反应 0.0625 U、0.125 U、0.25 U、0.5 U、1 U、2 U、4 U、8 U)并测试多次孵育时间(例如,2、5、10 和 15 分钟)。
- 将含有各种浓度 MNase 的 MNase 缓冲液(参见 表 1)添加到染色质等分试样中,并在 37 °C 下孵育样品指定时间。
- 在所需的孵育时间后立即加入 1/4 体积的终止缓冲液(终浓度:20 mM EDTA)以终止反应。
- 使用苯酚/氯仿/异戊醇提取方法从消化的染色质样品中分离 DNA。
- 在 1.5% 琼脂糖凝胶上运行提取的 DNA 以观察消化模式:消化不足将显示高分子量条带(图 2,泳道 1-4);过度消化将导致弥散条带或非常短的片段(图 2,泳道 6-8),最佳消化将产生清晰的核小体阶梯模式(图 2,泳道 5,例如,单核小体、双核小体、三核小体)。
- 确定产生所需核小体分辨率而不会过度消化的条件。
注:CaCl2 充当 MNase 活性的辅因子。通过在 1 mM 和 5 mM 之间调节 CaCl2 浓度来优化消化。
- 用广口吸头移液 5 - 10 次,用 100 μL MNase 缓冲液轻轻重悬完整细胞核。立即向样品中加入预定量的 MNase(1.25 U MNase/100 μL MNase 缓冲液)。
注:处理多个样品时,请单独消化每个样品以避免过度消化。 - 将试管放在旋转器上,并在 37 °C 下孵育 5 分钟。 立即将试管放回冰中,加入 EDTA 至终浓度为 20 mM 以终止 MNase 消化,然后涡旋混合。
- 向每个样品中加入 500 μL 缓冲液 B(参见 表 1),并通过上下吹打 5 次 - 10 次充分混合。通过在冰上孵育 5 分钟来溶解蛋白质。
注:缓冲液 B 中的盐和去污剂有助于解离弱染色质结合蛋白并暴露表位以进行免疫沉淀。 - 通过在 4 °C 下以最大速度离心 5 分钟来沉淀不溶性材料。 将澄清的上清液转移到标记为天然染色质组分的新 1.5 mL 试管中。样品可以储存在 -80 °C 下,也可以用于验证染色质碎裂的效率。
注:避免频繁的冻融循环,因为它们可能会破坏感兴趣的蛋白质-DNA 相互作用。尽可能减少冻融循环。
3. 染色质片段化的验证
- 将每个样品中的 10 μL 上清液分装到新的 1.5 mL 试管中。与 20 μL 蒸馏水和 30 μL 苯酚/氯仿/异戊醇 (25:24:1) 混合。
- 紧紧关闭试管并剧烈涡旋 15-30 秒。在 4 °C 下以 20,000 x g (或离心机的最大速度) 离心 10 分钟。 离心后,将观察到三个不同的层:透明的顶层、白色的中间层和黄色的底层。
- 小心地将 20 μL 上层水相(含 DNA)转移到新试管中。在 1.5% 琼脂糖凝胶中于 100 V 下分离纯化的 DNA 30 分钟,并观察消化模式。确保染色质片段的大小主要在 200 到 1000 个碱基对之间。
注:适当的染色质片段大小对于天然 ChIP 的成功至关重要,并且取决于 MNase 处理条件,包括酶单位、孵育时间和 CaCl2 浓度。MNase 消化效率也会因细胞类型和数量而异。图 2 所示的染色质碎裂模式(泳道 5)推荐用于该 ChIP 检测。
4. 免疫沉淀
- 将每个样品中的 20 μL 消化染色质分装到新的 1.5 mL 试管中,并与 180 μL 洗脱缓冲液混合(参见 表 1)。将这些管标记为输入样品并储存在 -20 °C。
- 将 400 μL 染色质样品转移到另一个 1.5 mL 试管中进行 ChIP。
- 将 γH2A.X 抗体(参见 材料表)添加到一个 DMSO 处理的样品、一个非地霉素处理的样品和一个羟基脲处理的样品中。将等量的正常 IgG(参见 材料表)添加到另一个 DMSO 处理的样品中,作为 ChIP 检测的阴性对照。
注:此处,1 μg 一抗通常用于 400 μL 染色质(即抗体终浓度为 2.5 μg/mL)。然而,应根据经验确定不同 γH2A.X 抗体的最佳量。 - 将 ChIP 管放在 4 °C 的旋转器上,孵育至少 5 小时,或者最好过夜。
- 同时,将 100 μL ChIP 级磁性蛋白 A/G 珠(参见 材料表)分装到新的 1.5 mL 试管中。使用广口吸头并缓慢移液,以确保准确测量珠子。将试管放在磁力架上至少 1 分钟,然后小心丢弃液体。
- 将珠子重悬于 1 mL 含有 0.5% BSA 的 1x PBS 中。在 4 °C 下旋转约 4 小时。将试管放在磁力架上至少 1 分钟,然后弃去上清液。
- 用 1 mL 含有 0.5% BSA 的 1x PBS 再次洗涤珠子。将试管放在磁力架上 1 分钟以沉淀磁珠,然后弃去上清液。
注:步骤 4.5 至 4.7 是对磁珠进行预包被,以减少抗体与磁珠的非特异性结合。 - 使用广口吸头将预包被的微珠重悬于 100 μL 缓冲液 B 中。向每个 ChIP 样品管中加入 25 μL 预包被的磁珠悬浮液。在 4 °C 下旋转 2 小时。
- 将 ChIP 管放在磁力架上,等待珠子完全附着在管的侧面,溶液变得透明。
- 丢弃透明上清液,不要干扰磁珠。用 1 mL 洗涤缓冲液重悬珠子(参见 表 1),并在 4 °C 下旋转 10 分钟。
- 将试管放回磁力架上,等待溶液变清。丢弃洗涤缓冲液。重复洗涤,总共洗涤四次。
- 最终洗涤后丢弃洗涤缓冲液,并在 4 °C 下以 400 x g 短暂离心管 30 秒以旋转任何残留液体。将试管放回磁力架上,小心地从试管底部清除残留的液体。
5. 洗脱和 DNA 沉淀
注:不同批次的抗体效率可能有所不同。通过 Western blot 分析检查免疫沉淀的样品来确认新抗体的结合亲和力非常重要。
- 如下所述,使用 Western blot (WB) 验证 ChIP 抗体的沉降效率。
- 取一小份 ChIP 样品进行分析(即,通常为 ChIP 样品的 10%)。包括起始染色质(免疫前沉淀)和阴性对照(例如,IgG pull-down)以进行比较。
- 通过在 95 °C 下在 20 μL 1x SDS-PAGE 上样缓冲液(参见 材料表)中加热 5 分钟,从抗体结合的珠子中洗脱蛋白质。
- 将 IP 样品、起始量和对照品上样到 15% SDS-PAGE 凝胶上。运行凝胶。
- 使用湿式或半干式转印系统将蛋白质转印到 0.2 μm 硝酸纤维素(参见 材料表)或 PVDF 膜上。
- 在室温下用 5% 脱脂牛奶或 TBST 中的 BSA(参见 表 1)封闭膜 1 小时,以防止非特异性结合。
- 将膜与在封闭缓冲液中稀释的抗 γH 2 A.X 的一抗(参见 材料表)在室温下孵育 1-2 小时或在 4 °C 下过夜。
- 用 TBST 洗涤膜 3 次以去除未结合的抗体。在室温下将膜与 HRP 偶联的二抗(参见 材料表)孵育 1 小时。再次清洗膜以去除多余的二抗。
- 使用化学发光底物显影膜,并使用成像仪可视化信号。比较 IP、input、control 泳道之间的信号强度,以评估 pull-down 的效率和特异性。
注:与 IP 泳道中靶蛋白相对应的条带确认抗体沉降成功。这种方法可确保您可以在 ChIP 实验期间评估抗体捕获靶蛋白的功效。
- 向剩余的每个 ChIP 样品中加入 50 μL 洗脱缓冲液(参见 表 1)。将试管放在热混合器上,在室温下摇动 15 分钟。
- 将试管放在磁性支架上至少 1 分钟。将洗脱液收集到新试管中。重复 1 次并将洗脱液收集在同一管中。
- 向每个 ChIP 洗脱样品中加入额外的 100 μL 洗脱缓冲液,向每个输入样品中加入 180 μL 洗脱缓冲液。
- 向每个样品中加入 200 μL 苯酚/氯仿/异戊醇 (25:24:1) 并剧烈涡旋。将样品在 4 °C 下以 20,000 x g (或最大速度)离心 10 分钟。
- 向每个新的 1.5 mL 离心管中加入 19 μL 3M 乙酸钠(NaOAc,pH 值 5.2;参见 表 1)和 2 μL 糖原溶液(20 mg/mL,参见 材料表)。
- 离心后,小心地将上层水层(约 190 μL)转移到含有 NaOAc 和糖原的试管中,并通过涡旋混合。
- 加入 500 μL 100% 乙醇并涡旋。通过将样品在 -20 °C 下孵育至少 2 小时或过夜来沉淀 DNA。
- 将试管在 4 °C 下以 20,000 x g (或最大速度)离心 10 分钟。 弃去上清液,注意不要打扰白色沉淀。将沉淀重悬于 1 mL 70% 乙醇中并彻底涡旋。
- 将试管在 4 °C 下以 20,000 x g (或最大速度) 离心 5 分钟。 小心去除上清液。再次短暂离心试管以离心任何残留的乙醇。使用 P20 移液器小心去除乙醇。将 DNA 沉淀风干 2-3 分钟。
注:避免过度干燥沉淀,因为这会使 DNA 难以重新溶解。 - 对于 ChIP 样品,将 DNA 重悬于 400 μL TE 缓冲液中(参见 表 1)。对于输入 DNA,重悬于 1000 μL TE 缓冲液中。洗脱的样品现在可以储存在 -20°C 下。
6. qPCR 定量
- 使用商业试剂盒(参见 材料表)进行 qPCR,每个样品一式三份。通过进行熔解曲线分析来确认单个特异性 PCR 产物的存在,以确保扩增的特异性36。
- 数据分析
注:在相对定量分析中,测试样品表示为相对于对照样品的倍数变化(使用正常纯化的 IgG 或模拟 IP 进行免疫沉淀)。与已知的、被占据的阳性对照 DNA 基因座36 相比,已知未被免疫沉淀蛋白占据的 DNA 基因座(阴性基因座)可以以这种方式用作参考基因。- 使用以下公式计算每个 ChIP 的输入百分比
%起始量 = 2(-ΔCt [标准化 ChIP]) - 使用以下公式从负轨迹的 ΔCt 值中减去为正轨迹获得的 ΔCt 值,将正轨迹 ΔCt 值归一化为负轨迹 (ΔΔCt)
(ΔΔCt = ΔCtnegative - ΔCtnegative) - 使用以下公式计算 ChIP DNA 中阳性基因座序列相对于阴性基因座的倍数富集
倍数富集 =2ΔΔCt
表 2 中提供了用于分析的 qPCR 引物序列。FRA3B 和 FRA16D37的基因组组织如图 3A、B 所示。
- 使用以下公式计算每个 ChIP 的输入百分比
- 统计分析
- 使用 Student 配对 t 检验对结果进行统计分析。≤0.05 的 p 值被认为具有统计显著性,表明观察到的差异不太可能是由于随机变异38。
结果
染色质片段的大小对于天然 ChIP 的成功至关重要,因为它直接影响抗体结合的 DNA 区域的可及性。为了确定染色质碎裂的最佳 MNase 浓度,我们制备了一系列微量离心管,其中含有不同浓度的 MNase(即每个反应 0.0625 U、0.125 U、0.25 U、0.5 U、1 U、2 U、4 U、8 U)和 40 μL 分离的细胞核。每个反应应在 37 °C 下孵育 5 分钟,以获得一系列染色质片段大小。 MNase 消化的结果如图 2 所示。
如图 2 所示,较高浓度的 MNase 导致染色质的消化范围更广,导致单核小体片段占主导地位(图 2,泳道 1、2 和 3)。相比之下,在较低的 MNase 浓度下,大多数染色质片段更大,通常超过 1 kb(图 2,泳道 6、7 和 8),表明下游应用的消化不足。在 MNase 浓度为 1.6 单位且消化时间为 5 分钟的情况下,我们主要获得了 200 至 1000 个碱基对范围内的染色质片段(图 2,泳道 5)。该片段大小非常适合天然 ChIP,因为它可确保染色质相关 DNA 的有效富集,同时保持对特定表位的可及性。适当的片段大小有助于高效的免疫沉淀和下游分析,例如定量 PCR 或测序,使其适用于以高分辨率检测蛋白质-DNA 相互作用。
抗体的结合亲和力是 ChIP 成功的关键因素。确保抗体有效、特异性地结合其靶标表位对于获得可靠的结果至关重要。因此,提前验证抗体的结合效率至关重要。为了确认 γH2A.X 免疫沉淀的效率,我们进行了 western blot (WB) 测定。
我们使用 western blotting 比较了 DMSO 处理的细胞 (对照) 、蚜虫菌素处理和羟基脲处理的细胞 (复制应激) 之间的 γH2A.X 水平。如图 4A (输入)所示,与 DMSO 处理的细胞相比,aphidicolin 处理的细胞中的 γH 2 A.X 水平显着升高。γH2A.X 的这种增加与哼虫素诱导的复制应激一致,这导致双链断裂形成增加和随后的 γH2A.X 磷酸化。进行 ChIP 后,我们通过比较 IgG 对照和 γH2A.X IP 样品中的 γH2A.X 富集来验证免疫沉淀的特异性。IgG 对照没有拉下任何可检测的 γH2A.X,表明非特异性结合最小(图 4B、N-ChIP 和 X-ChIP)。相比之下,γH2A.X IP 样品成功地富集了来自蚜虫菌素处理的细胞的 γH2A.X,证明抗体与 γH2A.X 的结合有效且具有特异性(图 4,N-ChIP 和 X-ChIP)。这些结果证实,γH2A.X 抗体具有足够的结合亲和力和特异性,可用于非变性 ChIP 实验,能够可靠地检测和富集 γH2A.X 结合的染色质区域。该验证步骤对于确保后续 ChIP 分析的质量和准确性至关重要。
我们使用常见脆弱位点 (CFS) FRA16D 和 FRA3B 来评估 γH2A.X 与 DNA 结合响应复制应激的特异性。FRA16D 和 FRA3B 是众所周知的 CFS 区域,在复制应激条件下容易不稳定,使其成为验证 γH2A.X/DNA 相互作用的理想候选者。 图 3A、B 说明了这两种 CFS 的基因组组织,以及用于分析的特异性 qPCR 引物。
为了确定 γH2A.X 是否响应复制应激与这些 CFS 区域相关,我们在对照 (DMSO 处理)、哼啶菌素处理的细胞和羟基脲处理的细胞中进行了 ChIP-qPCR 分析。qPCR 结果表明,在 DMSO 处理的对照细胞中,γH2A.X 在 FRA16D 或 FRA3B 基因座上均未富集,表明这些区域没有明显的 DNA 损伤或复制应激(图 3C)。然而,在用阿非迪霉素或羟基脲处理的细胞中,通过抑制 DNA 聚合酶诱导复制应激,我们观察到 γH2A.X 在 FRA16D 和 FRA3B 处均显著富集(图 3C)。这表明 γH2A.X 被募集到这些 CFS 区域,特别是响应 DNA 复制应激。这些发现表明,γH2A.X 在 CFS 中的占据是由复制应激触发的,支持其作为 DNA 损伤反应和复制叉停滞标志物的作用。在非迪霉素治疗过程中,γH2A.X 特异性募集到 FRA16D 和 FRA3B 中,进一步验证了我们的 ChIP 检测的特异性,并证实 γH2A.X 是脆弱位点 DNA 损伤和复制应激的可靠指标。
为了比较天然 ChIP 与交联 ChIP (X-ChIP) 的效率,我们按照 Lyu 等人 39 描述的方案,使用相同的起始细胞数进行了 γH2A.X X-ChIP。用阿非迪霉素和羟基脲处理后,γH2A.X 对 FRA16D 的募集增加,如图 3D 所示。然而,X-ChIP 中 FRA16D 中 γH2A.X 的相对富集明显低于天然 ChIP 观察到的水平,表明在这些条件下 X-ChIP 的效率降低。此外,在 FRA3B 中,γH2A.X 富集的增加在统计学上不显著,这提供了额外的证据表明,当使用有限数量的细胞时,X-ChIP 的效率较低。这些结果表明,当样本量受限时,天然 ChIP 可能更适合检测脆弱部位的 γH2A.X 富集。使用 Student 配对 t 检验对结果进行统计分析。≤ 0.05 的 p 值被视为具有统计显著性。

图 1:用于进行非变性 γH2A.X ChIP 检测的 W orkflow。 (i) 培养细胞并进行特异性处理(例如,羟基脲等 DNA 损伤剂)以诱导 γH2A.X 信号传导。(ii) 收集处理过的细胞进行处理。(iii) 对细胞进行分级分离以分离染色质结合的 γH2A.X,将其与其他细胞成分分离。(iv) 进行微球菌核酸酶 (MNase) 消化,将染色质碎裂成核小体大小的片段。(v) 通过在琼脂糖凝胶上运行样品来验证染色质片段的大小,确保片段的长度适合 ChIP。(vi) γH2A.X 特异性抗体用于免疫沉淀 γH2A.X 标记的染色质片段。(vii) 蛋白 A/G 珠用于拉低抗体-染色质复合物,富集 γH2A.X 结合的 DNA。(viii) 洗脱和纯化 γH2A.X 标记的 DNA 片段,为下游分析做好准备。(ix) 下游分析:qPCR 和测序以及数据分析。 请单击此处查看此图的较大版本。

图 2:优化 MNase 消化条件的检测。 使用不同的 MNase 消化条件从 HEK 293T 细胞制备染色质片段。从 片段化染色质中纯化的 DNA 在 2% 琼脂糖凝胶上分离,并在 100 V 下运行 30 分钟。泳道 1 至 8 表示用浓度递增的 MNase 处理的样品(即每个反应 0.0625 U、0.125 U、0.25 U、0.5 U、1 U、2 U、4 U、8 U),表明染色质消化呈梯度。包括 DNA 分子量标准(由 M 表示)作为大小参考。使用 Midori Green Advance DNA 染色溶液观察 DNA 片段。标记的条带对应于单核小体大小的 DNA 片段,突出了每种 MNase 浓度下的消化效率。 请单击此处查看此图的较大版本。
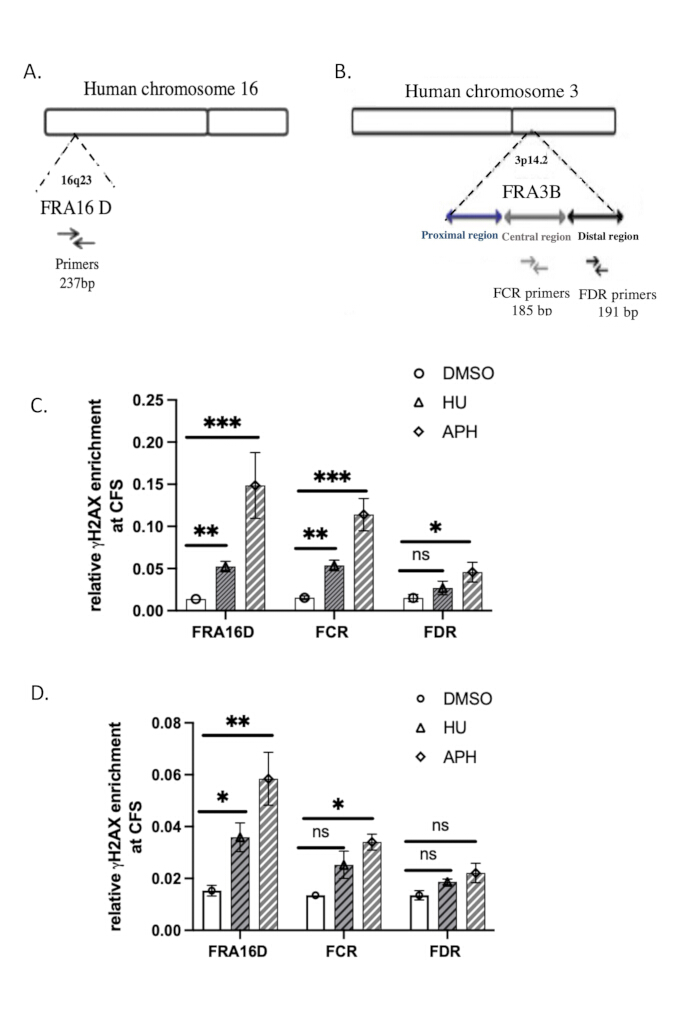
图 3:γH2A.X ChIP 的 qPCR 验证。 (A) FRA16D 的基因组组织图。(B) FRA3B 区域的基因组组织图。显示了用于 FRA3B 基因座内 FRA16D、远端 (FDR) 和中央 (FCR) 区域 qPCR 分析的引物组。(C) 使用天然 ChIP 评估 γH2A.X 在常见脆性位点 (CFS),特别是 FRA3B 和 FRA16D 的相对富集,然后在用非地霉素和羟基脲处理后进行 qPCR 分析。(D) 使用 X-ChIP 评估 γH2A.X 在常见脆性位点 (CFS),特别是 FRA3B 和 FRA16D 的相对富集,然后在用阿非迪霉素和羟基脲处理后进行 qPCR 分析。表示具有统计学意义的结果,p < 0.001;** 表示 P < 0.01,* 表示 P < 0.05,由 t 检验确定;n = 3。 请单击此处查看此图的较大版本。
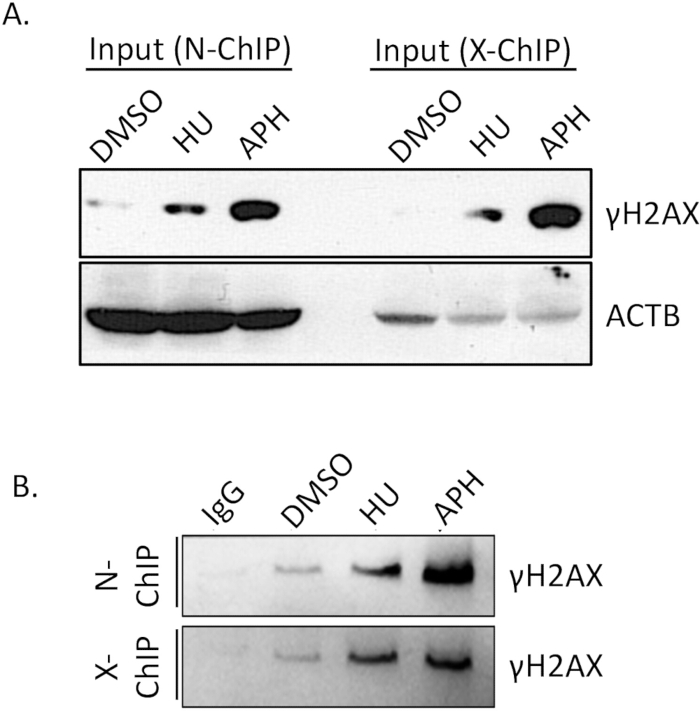
图 4:使用 Western blot 验证 ChIP 抗体的沉降效率。 从总样品中, 在 15% SDS-PAGE 凝胶上分离 10% 的 ChIP 样品以及起始对照,以分离多肽。电泳后,使用标准转印方案将分离的蛋白质转印到 0.22 μm PVDF 膜上。然后用靶向 γH2A.X 和 ACTB 的特异性一抗依次探测膜,然后用适当的 HRP 偶联二抗探测膜。随后使用化学发光检测对信号进行可视化,从而可以在 ChIP 检测中评估抗体特异性和沉降效率。(A) 以 ACTB 作为加载对照,DMSO 处理的细胞(对照)、哼啶菌素处理和羟基脲处理的细胞(复制应激)之间的 γH2A.X 水平。(B) IgG 对照和 IP 样品中的 γH2A.X 富集。缩写: APH = 蚜虫素,胡 = 羟基脲。 请单击此处查看此图的较大版本。
| 缓冲液 A (1X) | |
| 试剂 | 工作浓度 |
| 管道 pH 6.8 | 10 毫米 |
| 氯化钠 | 100 毫米 |
| 氯化镁2 | 3 毫米 |
| EGTA pH 值 7.6 | 1 毫米 |
| 在 -20 °C 下储存长达 1 年 | |
| MNase 缓冲液 (1X) | |
| 试剂 | 工作浓度 |
| Tris-HCl,pH 7.5 | 50 毫米 |
| 氯化钙2 | 1 毫米 |
| 氯化镁2 | 4 毫米 |
| 在 -20 °C 下储存长达 1 年 | |
| 缓冲液 B (1X) | |
| 试剂 | 工作浓度 |
| 磷酸钠 pH 7。0 | 20 毫米 |
| 焦磷酸钠10.端头H 2O | 30 毫米 |
| 氯化钾 | 250 毫米 |
| EDTA,pH 值 8.0 | 5 毫米 |
| 甘油 | 10% |
| 海卫一 X-100 | 0.10% |
| 蛋白酶抑制剂混合物 | 1 倍 |
| 磷酸酶抑制剂混合物 | 1 倍 |
| DTT | 0.5 毫米 |
| 在 -20 °C 下储存长达 1 年 | |
| 洗脱缓冲液 | |
| 试剂 | 工作浓度 |
| 氯化钠3 | 50 毫米 |
| 安全数据表 | 1% |
| 新鲜烹制 | |
| 洗涤缓冲液 | |
| 试剂 | 工作浓度 |
| MNase 缓冲液 | (0.5 倍) |
| 缓冲区 B | (0.5 倍) |
| 新鲜烹制 | |
| 停止缓冲区 | |
| 乙二胺四乙酸 | 100 毫米 , pH8.0 |
| 3 M 乙酸钠,pH 5.2 | |
| 408.24 g 乙酸钠 | |
| a仅 pH 值,乙酸至 pH 值 5.2 | |
| 将体积调整至 1 L | |
| 通过过滤或高压灭菌对缓冲液进行灭菌 | |
| TBST 缓冲液 | |
| Tris 碱,pH 8.0 | 10 毫米 |
| 氯化钠 | 150 毫米 |
| 补间 20 | 0.1% (垂直/垂直) |
| TE 缓冲液 | |
| Tris-HCl,pH 值 8.0 | 10 毫米 |
| EDTA,pH 值 8.0 | 1 毫米 |
表 1:缓冲液组成。
| 基因位点 | 正向引物 | 反向引物 |
| FRA16D 系列 | TCCTGTGGAAGGGATATTTA | CCCCTCATATTCTGCTTCTA |
| FRA3B 系列 | TGTTGGAATGTTAACTCTATCCCAT | ATATCTCATCAAGACCGCTGCA |
| FCR | ||
| FRA3B 系列 | CAATGGCTTAAGCAGACATGGT | AGTGAATGGCATGGCTGGAATG |
| 罗斯福 | ||
| ACTB(阴性对照) | GACGCAGGATGGCATGGG | ACGCCTCTGGCCGTACCAC |
表 2:引物序列。
| DNA 回收率低 | 潜在问题 | 可能的解决方案 |
| 抗体质量差 | 使用 ChIP 级抗体。 | |
| 抗体不足 | 每 25 μg 染色质使用 1-10 μg ChIP 抗体。 | |
| 孵育时间不足 | 在 4 °C 下过夜进行免疫沉淀步骤。 | |
| 过于严格的洗涤 | 请勿在洗涤缓冲液中使用浓度高于 500 mM 的 NaCl。 | |
| 劣质珠子 | 确保蛋白 A 或 G 与 ChIP 抗体兼容。 | |
| 请遵循磁珠产品数据表,了解磁珠体积与抗体的比例。 | ||
| 启动不足 | 准备单独的细胞板以准确测定细胞数量。 | |
| 样本 | 如果靶标丰度低,则增加使用的细胞数。 | |
| 细胞裂解不充分 | 优化缓冲液成分和裂解时间以提高效率。 | |
| 在裂解步骤中施加机械力,例如在处理某些细胞类型时使用 Dounce 匀浆器。 | ||
| 发生细胞核降解 | 确保细胞核分离温和,以保持染色质的完整性和可及性。 | |
| 使用大口径移液器吸头,以最大限度地减少处理过程中细胞核的剪切。 | ||
| 发生样品降解 | 在冰上或 4 °C 下执行所有步骤。 | |
| 在所有缓冲液中加入蛋白酶抑制剂,并确保所有缓冲液都是新鲜制备的。 | ||
| MNase 消化不足 | 滴定 MNase 浓度(例如,0.1–5 单位)以确定特定样品类型的最佳量;并从一系列浓度开始,以确定理想的消解条件。 | |
| MNase 过度消化 | 在 37 °C 下尝试不同的孵育时间(例如,1-10 分钟),并监测碎裂模式,以确保充分消化而不会过度消化。 | |
| 磷酸化被破坏 | 将磷酸酶抑制剂混合物添加到裂解缓冲液中,并确保所有缓冲液都是新鲜制备的。 | |
| 阴性对照中的高背景 | 与微珠的非特异性结合 | 在免疫沉淀步骤之前包括预透明步骤,并使用磁性 ChIP 级微珠,其通常表现出减少的非特异性结合。 |
| 洗涤不充分 | 通过调整盐和洗涤剂浓度来增加洗涤次数或严格程度。 | |
| 染色质碎裂不足 | 优化片段化以获得 200-750 bp 的片段。 | |
| 每种细胞或组织类型都需要进行优化 | ||
| PCR 故障排除 | 输入样品无扩增 | (1) 过度片段化:如果染色质片段化为单核小体长度,则超过 150 bp 的扩增子的信号会减弱。 |
| (2) 优化 PCR 条件。 | ||
| (3) 设计引物以扩增较小的 (<150 bp) 区域。 | ||
| 低分辨率 | DNA 片段大小过大 | 优化片段化以获得 200-750 bp 的片段。 |
表 3:染色质免疫沉淀疑难解答。
讨论
环境污染是导致人类癌症的重要因素。许多污染物是致癌物,这意味着它们会导致遗传损伤,从而导致癌症的发展40,41。然而,确定特定物质是否致瘤是一项具有挑战性的任务。一种快速、可靠且经济高效的识别潜在致癌物的方法将使科学家能够有效地筛选环境污染物并评估它们对基因组稳定性的影响。在这项研究中,我们专注于一种修饰的组蛋白变体 γH2A.X,它被广泛认为是 DSB 的高度特异性标志物。γH2A.X 是在组蛋白变体 H2A 时形成的。X 在丝氨酸 139 位点被磷酸化以响应 DNA 损伤。
通过在 CFS 中通过天然 ChIP 检测 γH2A.X 的存在,我们可以有效地评估环境污染物引起的 DNA 损伤程度42。在 CFS 中定量测量 γH2A.X 的能力提供了一种快速、灵敏和可靠的方法来评估环境污染物诱导致瘤作用的可能性。与传统的遗传毒性检测相比,传统的遗传毒性检测通常测量 DNA 损伤的间接标志物(如突变、微核形成或细胞转化),而天然 γH2AX ChIP 可精确定位特定基因组位点的断裂事件42。这提供了对最容易受到环境污染物影响的地区的更深入理解,并有助于识别潜在的致癌机制。
成功的天然 ChIP 需要仔细注意几个关键步骤,以确保有效富集靶标区域。以下是 γH2AX 天然 ChIP 所涉及的关键步骤。细胞制备:从足够数量的细胞开始。用 DNA 损伤诱导剂(如 aphidicolin)处理细胞,以诱导复制应激作为阳性对照。包括适当的未处理阴性对照样品进行比较。染色质分离:进行亚细胞分级分离以分离染色质结合的蛋白质,同时最大限度地减少细胞质成分的污染。MNase 优化:进行中试实验以优化微球菌核酸酶 (MNase) 浓度和孵育时间。目标是获得 200-1000 个碱基对之间的染色质片段,确保 ChIP 的良好分辨率,同时保持核小体的完整性。免疫沉淀:利用高质量的 ChIP 级 γH2AX 抗体和蛋白 A/G 琼脂糖,确保靶染色质的有效沉降,同时最大限度地减少非特异性结合。洗涤:用含有适当浓度的盐和去污剂的缓冲液洗涤珠子,以去除非特异性相互作用,同时保留 γH2AX 结合的染色质。洗脱和 DNA 回收:通过彻底洗脱和沉淀 DNA 来有效回收结合的染色质,确保富集的目标染色质损失最小。与传统的 X-ChIP 相比,天然 γH2AX ChIP 有四个主要优势。首先,整合亚细胞组分分离可减少假阳性相互作用并增强 ChIP 特异性。其次,蛋白质-DNA 复合物保持其天然状态,最大限度地减少了交联剂可能发生的瞬时 γH2AX-染色质相互作用的稳定性。第三,省略固定和耗时的反向交联步骤简化了整个工作流程。最后,需要较少的起始材料,并且不需要昂贵的设备进行染色质剪切,例如水浴超声仪(例如 Bioruptor)或聚焦超声超声仪(例如 Covaris)。
尽管天然 ChIP 有其优点,但也应承认其局限性。一个限制是在染色质制备和免疫沉淀过程中可能发生蛋白质重排。这种重排理论上可以改变染色质结合蛋白的相互作用,并在结果中引入可变性。虽然我们没有直接证据支持这种重排的发生,但重要的是要包括适当的实验对照来解释这种潜在的伪影。天然 ChIP 检测的另一个限制是 MNase 的切割偏倚,这可能导致结果不准确。MNase 优先切割基因组中富含 A-T 的区域,使这些区域在 ChIP 过程中更有可能片段化和富集。这种偏差会导致富含 A-T 的序列的过度表达和富含 G-C 的区域的表达不足,从而可能导致 MNase 消化难以接近的基因组区域出现假阴性信号。如果目标基因座富含 G-C,则需要仔细优化 MNase 消化条件。或者,结合其他对照,例如通过超声处理进行染色质碎裂,可以帮助减轻 MNase 偏倚的影响并提高结果的可靠性。
尽管存在一定的局限性,但天然 γH2AX ChIP 对于识别特定污染物诱导 DNA 损伤的基因位点特别有用。通过富集与 γH2AX 结合的 DNA 片段,研究人员可以通过 qPCR 识别基因组的脆弱区域,特别是 CFS,这些区域在污染物诱导的复制应激下更容易断裂。该技术还使研究人员能够建立暴露于特定致癌物与特定基因组区域 DNA 损伤形成之间的直接关系,从而深入了解污染物的遗传毒性特征及其在引发致癌作用中的潜在作用43,44。
故障排除表(表 3)提供了 ChIP 检测过程中遇到的常见问题的详细解决方案,例如 DNA 产量低、染色质片段化不足、背景噪音高或抗体性能差。每个问题都配有具体建议,以确保实验步骤的可靠性和可重复性。
通过使用自动化液体处理系统和定量 PCR 或测序技术,天然 γH2AX ChIP 可以有效地识别诱导 DNA 损伤的试剂,从而成为毒理学研究和环境监测的宝贵工具。通过对暴露于不同水平环境污染物的人群的样本采用天然 γH2AX ChIP,研究人员可以识别特定的 DNA 损伤模式并确定与暴露于特定试剂相关的风险水平。总之,天然 γH2AX ChIP 在评估环境污染物的致癌潜力方面具有显著优势,提供了一种快速、经济高效且高特异性的方法来检测特定基因组位点的 DNA 损伤。它在高通量筛选、个性化风险评估和生物标志物开发方面的未来应用使其成为促进我们对环境致癌作用的理解和制定癌症预防和干预策略的宝贵工具。
披露声明
作者没有需要披露的利益冲突。
致谢
这项工作得到了南华大学的启动资金支持。
材料
| Name | Company | Catalog Number | Comments |
| 0.2 µm nitrocellulose membrane | Amersham | 10600011 | |
| Actin B | proteintech | 20536-1-AP | |
| Aphidicolin | MedChemExpress | HY-N6733 | |
| ChIP-grade magnetic Protein A/G beads | ThermoFisher | 26162 | |
| Clarity Western ECL Substrate | Bio-Rad | #1705061 | |
| Glycogen, molecular biology grade | ThermoFisher | Cat. No. R0561 | |
| HRP-conjugated secondary antibody | proteintech | SA00001-2 | |
| hydroxyurea | MedChemExpress | HY-B0313 | |
| Micrococcal Nuclease | NEB | M0247S | |
| normal IgG | Santa Cruz | sc-2025 | |
| Taq Universal SYBR Green Supermix | BioRad | 1725120 | |
| γH2A.X antibody (for ChIP) | Sigma-Aldrich | 05-636 | |
| γH2A.X antibody (for WB) | Cell Signaling | #25955 |
参考文献
- Glover, T. W., Berger, C., Coyle, J., Echo, B. DNA polymerase alpha inhibition by aphidicolin induces gaps and breaks at common fragile sites in human chromosomes. Hum Genet. 67 (2), 136-142 (1984).
- Bignell, G. R., et al. Signatures of mutation and selection in the cancer genome. Nature. 463 (7283), 893-898 (2010).
- Hellman, A., et al. A role for common fragile site induction in amplification of human oncogenes. Cancer Cell. 1 (1), 89-97 (2002).
- Kotzot, D., et al. Parental origin and mechanisms of formation of cytogenetically recognisable de novo direct and inverted duplications. J Med Genet. 37 (4), 281-286 (2000).
- Miller, C. T., et al. Genomic amplification of MET with boundaries within fragile site FRA7G and upregulation of MET pathways in esophageal adenocarcinoma. Oncogene. 25 (3), 409-418 (2006).
- Zack, T. I., et al. Pan-cancer patterns of somatic copy number alteration. Nat Genet. 45 (10), 1134-1140 (2013).
- Gao, G., et al. Common fragile sites (CFS) and extremely large CFS genes are targets for human papillomavirus integrations and chromosome rearrangements in oropharyngeal squamous cell carcinoma. Genes Chromosomes Cancer. 56 (1), 59-74 (2017).
- Thorland, E. C., Myers, S. L., Gostout, B. S., Smith, D. I. Common fragile sites are preferential targets for HPV16 integrations in cervical tumors. Oncogene. 22 (8), 1225-1377 (2003).
- Matovina, M., Sabol, I., Grubisić, G., Gasperov, N. M., Grce, M. Identification of human papillomavirus type 16 integration sites in high-grade precancerous cervical lesions. Gynecol Oncol. 113 (1), 120-127 (2009).
- Yu, T., et al. The role of viral integration in the development of cervical cancer. Cancer Genet Cytogenet. 158 (1), 27-34 (2005).
- Bignell, G. R., et al. Signatures of mutation and selection in the cancer genome. Nature. 463 (7283), 893-898 (2010).
- Thavathiru, E., Ludes-Meyers, J. H., MacLeod, M. C., Aldaz, C. M. Expression of common chromosomal fragile site genes, WWOX/FRA16D and FHIT/FRA3B is downregulated by exposure to environmental carcinogens, UV, and BPDE but not by IR. Mol Carcinog. 44 (3), 174-182 (2005).
- Sirbu, B. M., et al. Analysis of protein dynamics at active, stalled, and collapsed replication forks. Genes Dev. 25 (12), 1320-1327 (2011).
- Barlow, J. H., et al. Identification of early replicating fragile sites that contribute to genome instability. Cell. 152 (3), 620-632 (2013).
- Petermann, E., Orta, M. L., Issaeva, N., Schultz, N., Helleday, T. Hydroxyurea-stalled replication forks become progressively inactivated and require two different RAD51-mediated pathways for restart and repair. Mol Cell. 37 (4), 492-502 (2010).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. J Biol Chem. 273 (10), 5858-5868 (1998).
- Shanbhag, N. M., Rafalska-Metcalf, I. U., Balane-Bolivar, C., Janicki, S. M., Greenberg, R. A. ATM-dependent chromatin changes silence transcription in cis to DNA double-strand breaks. Cell. 141 (6), 970-981 (2010).
- Stiff, T., et al. ATR-dependent phosphorylation and activation of ATM in response to UV treatment or replication fork stalling. EMBO J. 25 (24), 5775-5782 (2006).
- Nelson, J. D., Denisenko, O., Bomsztyk, K. Fast chromatin immunoprecipitation assay. Nuc Acids Res. 34 (5), e2 (2006).
- Dorbic, T., Wittig, B. Isolation of oligonucleosomes from active chromatin using HMG17-specific monoclonal antibodies. Nuc Acids Res. 14 (8), 3363-3376 (1986).
- Dorbic, T., Wittig, B. Chromatin from transcribed genes contains HMG17 only downstream from the starting point of transcription. EMBO J. 6 (8), 2393-2399 (1987).
- Hebbes, T. R., Thorne, A. W., Crane-Robinson, C. A direct link between core histone acetylation and transcriptionally active chromatin. EMBO J. 7 (5), 1395-1402 (1988).
- Hebbes, T. R., Thorne, A. W., Clayton, A. L., Crane-Robinson, C. Histone acetylation and globin gene switching. Nuc Acids Res. 20 (5), 1017-1022 (1992).
- Hebbes, T. R., Clayton, A. L., Thorne, A. W., Crane-Robinson, C. Core histone hyperacetylation co-maps with generalized DNase I sensitivity in the chicken beta-globin chromosomal domain. EMBO J. 13 (8), 1823-1830 (1994).
- Orlando, V. Mapping chromosomal proteins in vivo by formaldehyde-crosslinked-chromatin immunoprecipitation. Trend Biochem, Sci. 25 (3), 99-104 (2000).
- Myers, F. A., Evans, D. R., Clayton, A. L., Thorne, A. W., Crane-Robinson, C. Targeted and extended acetylation of histones H4 and H3 at active and inactive genes in chicken embryo erythrocytes. J Biol Chem. 276 (23), 20197-20205 (2001).
- Litt, M. D., Simpson, M., Recillas-Targa, F., Prioleau, M. N., Felsenfeld, G. Transitions in histone acetylation reveal boundaries of three separately regulated neighboring loci. EMBO J. 20 (9), 2224-2235 (2001).
- Madisen, L., Krumm, A., Hebbes, T. R., Groudine, M. The immunoglobulin heavy chain locus control region increases histone acetylation along linked c-myc genes. Mol Cell Biol. 18 (11), 6281-6292 (1998).
- Clayton, A. L., Hebbes, T. R., Thorne, A. W., Crane-Robinson, C. Histone acetylation and gene induction in human cells. FEBS Lett. 336 (1), 23-26 (1993).
- Tseng, Z., Wu, T., Liu, Y., Zhong, M., Xiao, A. Using native chromatin immunoprecipitation to interrogate histone variant protein deposition in embryonic stem cells. Methods Mol Biol. 1176, 11-22 (2014).
- Durkin, S. G., Glover, T. W. Chromosome fragile sites. Ann Rev Genetics. 41, 169-192 (2007).
- Lee, J. B., Keung, A. J. Chromatin immunoprecipitation in human and yeast cells. Methods Mol Biol. 1767, 257-269 (2018).
- Miyamoto, R., Yokoyama, A. Protocol for fractionation-assisted native ChIP (fanChIP) to capture protein-protein/DNA interactions on chromatin. STAR Protoc. 2 (2), 100404 (2021).
- Mendez, J., Stillman, B. Chromatin association of human origin recognition complex, cdc6, and minichromosome maintenance proteins during the cell cycle: Assembly of prereplication complexes in late mitosis. Mol Cell Biol. 20 (22), 8602-8612 (2000).
- Nowak, D. E., Tian, B., Brasier, A. R. Two-step cross-linking method for identification of NF-κB gene network by chromatin immunoprecipitation. BioTechniques. 39 (5), 715-725 (2005).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nat Protoc. 3 (6), 1101-1108 (2008).
- Lu, X., Parvathaneni, S., Hara, T., Lal, A., Sharma, S. Replication stress induces specific enrichment of RECQ1 at common fragile sites FRA3B and FRA16D. Mol Cancer. 12, 29 (2013).
- Ruijter, J. M., et al. Amplification efficiency: linking baseline and bias in the analysis of quantitative PCR data. Nucl Acids Res. 37 (6), e45 (2009).
- Lyu, X., Chastain, M., Chai, W. Genome-wide mapping and profiling of γH2AX binding hotspots in response to different replication stress inducers. BMC Genomics. 20, 579 (2019).
- Farmer, P. B., et al. Molecular epidemiology studies of carcinogenic environmental pollutants. Effects of polycyclic aromatic hydrocarbons (PAHs) in environmental pollution on exogenous and oxidative DNA damage. Mutat Res. 544 (2-3), 397-402 (2003).
- Phillips, D. H., Arlt, V. M. Genotoxicity: damage to DNA and its consequences. EXS. 99, 87-110 (2009).
- Nitsch, S., Schneider, R. Native ChIP: Studying the genome-wide distribution of histone modifications in cells and tissue. Meth Mol Biol. 2846, (2024).
- Nikitina, T., Wang, D., Gomberg, M., Grigoryev, S. A., Zhurkin, V. B. Combined micrococcal nuclease and exonuclease III digestion reveals precise positions of the nucleosome core/linker junctions: implications for high-resolution nucleosome mapping. J Mol Biol. 425 (11), 1946-1960 (2013).
- Teves, S. S., Henikoff, S. Salt fractionation of nucleosomes for genome-wide profiling. Methods Mol Biol. 833, 421-432 (2012).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。