Method Article
Cattura delle rotture di siti fragili comuni mediante γH2A nativo. X ChIP
In questo articolo
Riepilogo
Presentiamo un metodo rapido ed efficiente per rilevare le comuni rotture dei siti fragili attraverso l'immunoprecipitazione nativa della cromatina γH2A.X (ChIP). Questo approccio riduce significativamente sia il tempo che la manodopera associati ai tradizionali saggi ChIP γH2A.X, mantenendo al contempo un'elevata riproducibilità e affidabilità dei risultati.
Abstract
Lo stress di replicazione indotto dall'esposizione ad agenti estrinseci può portare a rotture del DNA in siti fragili comuni, che sono regioni del genoma note per essere soggette a instabilità strutturale. Il test di immunoprecipitazione della cromatina γH2A.X (ChIP) funge da potente strumento negli studi di genotossicità, poiché la fosforilazione γH2A.X è un marcatore ben consolidato per le rotture del doppio filamento del DNA. I tradizionali saggi ChIP γH2A.X, tuttavia, sono spesso laboriosi e comportano più passaggi che richiedono tempo. In questo studio, presentiamo un metodo semplificato ma efficace che combina il frazionamento subcellulare con la ChIP nativa per isolare i complessi associati a γH2A.X. Questo approccio è particolarmente adatto per analizzare le interazioni γH2A.X-cromatina con maggiore specificità ed efficienza. Utilizzando il frazionamento subcellulare, i materiali non legati alla cromatina vengono efficacemente rimossi, ottenendo una frazione di cromatina purificata. La successiva digestione della nucleasi micrococcica (MNasi) in condizioni blande consente la frammentazione della cromatina preservando le interazioni fisiologiche tra γH2A.X e i suoi complessi proteici associati. Questa conservazione è essenziale per lo studio dei partner di interazione nativi coinvolti nelle vie di risposta al danno del DNA. Questo protocollo ChIP nativo ottimizzato riduce sostanzialmente il tempo e la manodopera associati ai saggi ChIP γH2A.X convenzionali. La procedura semplificata non solo semplifica il flusso di lavoro, ma produce anche risultati altamente riproducibili, il che la rende particolarmente vantaggiosa in ambienti in cui è richiesta l'elaborazione ad alta produttività di più campioni. Questo metodo ha un'ampia applicabilità negli studi incentrati sulla stabilità del genoma, sulla riparazione del DNA e sulla biologia della cromatina, dove il rilevamento accurato ed efficiente dei siti di danno al DNA è fondamentale. Utilizzando protocolli ottimizzati e passaggi semplificati, questo metodo consente di rilevare danni al DNA in siti fragili con una migliore sensibilità e una manipolazione minima del campione, rendendolo uno strumento prezioso per gli studi sulla stabilità del genoma e sulla risposta al danno del DNA.
Introduzione
I siti fragili comuni (CFS) sono grandi regioni cromosomiche che si trovano su ogni cromosoma umano inclini a rompersi durante la metafase. Sotto stress di replicazione, la replicazione in queste regioni è significativamente ritardata, impedendo la loro completa duplicazione prima dell'ingresso mitotico1, che alla fine si traduce in lacune e rotture sito-specifiche. La CFS è un punto caldo per l'instabilità cromosomica e sono una delle principali cause di riarrangiamenti cromosomici durante lo sviluppo precoce del cancro. Lo stress di replicazione, che è spesso presente in condizioni tumorigeniche, può portare alla perdita di geni oncosoppressori e all'amplificazione di oncogeni, collettivamente indicati come variazione del numero di copie (CNV)2,3,4,5,6. Inoltre, la CFS è altamente incline all'integrazione virale, promuovendo ulteriormente lo sviluppo del cancro 7,8,9,10. Molteplici delezioni omozigoti di geni oncosoppressori sono state rilevate nelle regioni CFS durante le analisi pan-tumorali dei tumori primari. Le CFS più comunemente colpite nel cancro includono FRA2F, FRA3B, FRA4F, FRA5H e FRA16D11. I CFS sono particolarmente vulnerabili alla rottura in presenza di agenti cancerogeni estrinseci12. Per valutare gli effetti cancerogeni dannosi dei contaminanti ambientali, è necessario un metodo rapido e affidabile per quantificare l'insorgenza di rotture da CFS.
Fosforilazione di H2A. X al residuo di serina 139 (γH2A.X) da parte dell'atassia telangiectasia e della proteina correlata a Rad3 (ATR) o dell'atassia telangiectasia mutata (ATM) è un evento chiave nella segnalazione dello stallo della forcella di replicazione13. γH2A.X funge da indicatore dello stallo delle forcelle di replicazione prima della formazione del double-strand break (DSB)13, creando un ambiente cromatinico favorevole per facilitare il reclutamento efficiente delle proteine di riparazione nei siti in stallo. Inoltre, γH2A.X può essere reclutato per rompere i siti dopo il collasso della forcella14,15, coerentemente con il suo ruolo primario nella riparazione del DSB. Poiché le rotture della CFS sono strettamente associate alle aberrazioni cromosomiche che guidano la progressione del cancro, rilevare queste rotture può essere determinante per comprendere le prime fasi della tumorigenesi. La presenza di γH2A.X nella CFS può essere utilizzata come biomarcatore per rilevare eventi precoci di instabilità genomica. Queste informazioni possono aiutare a identificare potenziali agenti cancerogeni e valutare il rischio associato all'esposizione a vari agenti estrinseci. Misurando le rotture del DNA alle CFS indotte da agenti estrinseci, la cromatina γH2A.X IP (ChIP) può fornire informazioni su come tali agenti contribuiscono ai meccanismi alla base della tumorigenesi.
Nella ChIP convenzionale (i.e., Cross-linked ChIP, X-ChIP), l'associazione di γH2A.X con le sue sequenze di DNA bersaglio è stabilizzata dalla reticolazione reversibile della formaldeide. La cromatina viene successivamente tranciata in frammenti di circa 500 coppie di basi (bp) attraverso la sonicazione e la soluzione risultante viene ripulita dai detriti mediante sedimentazione 16,17,18. Un anticorpo γH2A.X di grado ChIP viene quindi aggiunto alla frazione di cromatina eliminata, seguito dall'aggiunta di perle di agarosio di proteina A/G per arricchire le regioni di cromatina legate a γH2A.X 16,17,18. I complessi immunitari (cioè perline-anticorpo-complesso di DNA mirato al γH2A.X) vengono lavati più volte con tamponi di lavaggio rigorosi per rimuovere i frammenti di DNA non specificamente legati 16,17,18. Dopo il lavaggio, il DNA specificamente legato viene eluito dai complessi immunitari. I legami incrociati della formaldeide vengono quindi invertiti, seguiti dalla digestione delle proteine utilizzando la proteinasi K, dopodiché il DNA arricchito viene purificato e concentrato 16,17,18. Per valutare le regioni associate a γH2A.X, viene utilizzata la PCR, la PCR quantitativa (qPCR) o il sequenziamento diretto 16,17,18. L'occupazione di γH2A.X in regioni specifiche, come la CFS, è determinata dall'intensità del segnale PCR o qPCR, che è proporzionale alla quantità di γH2A.X legata in quella posizione, fornendo informazioni sul danno al DNA sito-specifico e sugli eventi di riparazione 16,17,18.
Nonostante sia un potente approccio sperimentale, l'X-ChIP presenta diverse limitazioni significative: (i) richiede un gran numero di cellule, tipicamente nell'intervallo da 1 x 107 a 5 x 107, a causa dell'inefficienza della precipitazione degli anticorpi associata alla fissazione, che aumenta il costo complessivo dell'esperimento19; (ii) il processo di inversione dei legami incrociati della formaldeide e la successiva purificazione del DNA richiede tempo e lavoro, rendendo difficile mantenere la coerenza e l'affidabilità dei risultati; e (iii) le interazioni γH2A.x-DNA con minore significato funzionale potrebbero non essere distinte da quelle con maggiore significato perché la fase di cross-linking può stabilizzare le interazioni transitorie, portando al rilevamento di interazioni che potrebbero non essere biologicamente rilevanti19.
L'immunoprecipitazione nativa della cromatina (Native ChIP o N-ChIP) è una tecnica biochimica essenziale utilizzata per studiare le interazioni proteina-DNA all'interno del loro contesto nativo di cromatina in condizioni fisiologiche di sale. È stato determinante nel chiarire l'organizzazione spaziale e temporale della cromatina, il legame dei fattori di trascrizione e le modificazioni degli istoni. La ChIP nativa ha un ruolo di lunga data nel campo più ampio della biologia della cromatina e dell'epigenetica, fornendo vantaggi e limitazioni unici rispetto alla X-ChIP. Questo metodo, introdotto alla fine degli anni '8020, prevede l'isolamento della cromatina dalle cellule con metodi che ne preservano la struttura nativa, come la digestione con nucleasi micrococcica (MNase)21. Ciò preserva i contatti intrinseci proteina-DNA e istone-DNA, il che rende la ChIP nativa particolarmente adatta per studiare le modificazioni istoniche e il posizionamento dei nucleosomi nel loro ambiente naturale della cromatina22. Studi Native ChIP ad alta risoluzione hanno dimostrato l'uso della digestione MNasi per ridurre la cromatina a singoli nucleosomi, il che facilita la mappatura delle modificazioni istoniche con maggiore precisione23. Inoltre, poiché non è coinvolta alcuna reticolazione chimica, il rischio di introdurre distorsioni o artefatti che potrebbero travisare le interazioni proteina-DNA è ridotto al minimo24.
A differenza di X-ChIP, in cui la formaldeide o altri agenti reticolanti vengono utilizzati per correggere le interazioni proteina-DNA, la ChIP nativa fornisce una visione più realistica della cromatina evitando potenziali artefatti di reticolazione. Tuttavia, mentre la X-ChIP è generalmente più adatta per rilevare interazioni transitorie o dinamiche tra il DNA e le proteine regolatorie25, la ChIP nativa è ideale per interazioni stabili proteina-DNA, come gli istoni o altre proteine legate alla cromatina 26,27. Una delle limitazioni notate per Native ChIP è l'incapacità di catturare eventi di legame a bassa affinità o transitori, che sono spesso stabilizzati attraverso il cross-linking in X-ChIP25.
Un corpus significativo di lavori in epigenetica ha sfruttato la ChIP nativa per scoprire le modificazioni istoniche in diversi contesti biologici28. Questi sforzi sono stati cruciali nella definizione del codice istonico, il modello di modificazioni istoniche che regolano l'espressione genica e la dinamica della cromatina29. Sebbene H2A. X è un istone linker meno fortemente associato, l'H2A nativo. Il metodo X ChIP è stato applicato con successo nelle cellule staminali embrionali30. In questo studio, abbiamo ottimizzato una procedura di estrazione della cromatina per eseguire la ChIP nativa di γH2A.X in cellule 293T umane (Figura 1). L'idrossiurea e l'afidicolina sono ampiamente utilizzate nella ricerca per studiare lo stress da replicazione del DNA, il danno e l'instabilità genomica31. In questo studio, questi agenti sono stati applicati alle cellule per indurre lo stress di replicazione e generare rotture del DNA alla CFS.
Utilizzando materiale di partenza di circa 1 x 106 a 5 x 106 cellule, questo metodo può essere suddiviso in quattro fasi principali: (i) frazionamento subcellulare per isolare la cromatina, (ii) digestione della nucleasi micrococcica (MNasi) per frammentare la cromatina, (iii) immunoprecipitazione ed eluizione e (iv) analisi del DNA mediante PCR quantitativa (qPCR). L'esecuzione di ChIP dopo il frazionamento subcellulare offre diversi vantaggi ed è stata ben documentata in numerosi studi 32,33,34,35. Questo approccio consente la rimozione delle proteine non legate alla cromatina e di altri detriti cellulari, ottenendo una frazione di cromatina altamente purificata. Isolando la cromatina prima dell'immunoprecipitazione, il frazionamento subcellulare aiuta a mantenere le interazioni con la cromatina nativa e riduce il rumore di fondo delle proteine non associate alla cromatina, il che porta a risultati più specifici e affidabili, poiché solo i complessi legati alla cromatina vengono conservati per l'analisi. Inoltre, il frazionamento subcellulare consente condizioni più blande per la digestione della cromatina, preservando così le interazioni fisiologiche proteina-DNA e offrendo una rappresentazione più accurata della dinamica della cromatina all'interno dell'ambiente cellulare nativo.
L'utilizzo della ChIP nativa di γH2AX per misurare l'impatto degli agenti estrinseci sulla rottura di siti fragili comuni ha un potenziale significativo per la ricerca sul cancro. Questa tecnica consente di rilevare i danni al DNA indotti dall'esposizione ad agenti cancerogeni ambientali, fornendo informazioni sui meccanismi molecolari attraverso i quali gli inquinanti contribuiscono all'instabilità genomica e allo sviluppo del cancro. Preservando il contesto nativo della cromatina, questo metodo facilita la valutazione accurata dei modelli di danno al DNA associati all'esposizione ad agenti cancerogeni, aiutando nella valutazione dei rischi ambientali e nello studio della tumorigenesi causata dall'inquinamento.
Protocollo
1. Raccolta di cellule
- Seminare circa 5 x 105 cellule HEK 293T in ciascuna delle quattro piastre da 6 cm, ciascuna contenente 4 mL di terreno DMEM completo.
- Dopo 24 ore, trattare una piastra con 2 μL di 1 mM di afidicolina (fare riferimento alla Tabella dei materiali) soluzione madre (concentrazione finale di 0,5 μM) e un'altra piastra con 20 μL di 1 M idrossiurea (fare riferimento alla Tabella dei materiali) soluzione madre (concentrazione finale di 5 mM) per indurre lo stress di replicazione. Aggiungere il DMSO ai due piatti rimanenti per fungere da controlli.
- Dopo 24 ore di trattamento, eliminare il terreno di coltura. Una piastra da 6 cm produce tipicamente circa 2 x 106 celle al 60%-70% di confluenza.
- Sciacquare ogni piatto 2 volte con 5 ml di 1x PBS ghiacciato. Utilizzare raschietti per staccare le cellule e trasferire la sospensione cellulare in quattro provette singole da 1,5 mL. Pipettare delicatamente su e giù con una pipetta P1000 per dissociare eventuali grumi di cellule.
- Centrifugare le cellule a 500 x g per 5 minuti a 4 °C, quindi eliminare il surnatante. Metti le celle sul ghiaccio.
2. Frazionamento subcellulare
- Risospendere il pellet cellulare in 500 μl di tampone A freddo appena preparato (fare riferimento alla Tabella 1), garantendo la completa dissociazione dei grumi cellulari mediante pipettaggio delicato.
- Incubare i lisati su ghiaccio per 5-10 minuti. Controllare la progressione della lisi al microscopio per assicurarsi che la lisi cellulare sia completa.
- Prelevare una piccola aliquota di lisato (circa 5 - 10 μL) e posizionarla su un vetrino da microscopio pulito. Coprilo con un vetrino coprioggetti per evitare contaminazioni.
- Utilizzare un microscopio ottico con un ingrandimento appropriato (ad es. 20x - 40x) per visualizzare cellule o detriti. Confrontare con un campione di controllo non lisizzato per distinguere tra cellule intatte e materiale lisato.
NOTA: Un campione correttamente lisato non avrà contorni cellulari distinti, solo cromatina diffusa o materiale cellulare. Regolare la messa a fuoco per osservare chiaramente il lisato. Se necessario, applicare una forza meccanica durante la fase di lisi, ad esempio utilizzando un omogeneizzatore Dounce, quando si lavora con determinati tipi di cellule.
- Centrifugare a 500 x g per 5 minuti a 4 °C, una volta che le cellule sono completamente lisate. Eliminare con cautela il surnatante. Risospendere il pellet di nuclei in 500 μl di tampone A freddo utilizzando puntali per pipette a orifizio largo.
NOTA: Le punte a orifizio largo aiutano a ridurre al minimo le forze di taglio e proteggono campioni delicati come la cromatina. Realizza punte a foro largo tagliando l'estremità delle punte standard con una lama affilata. - Centrifugare a 500 x g per 5 min a 4 °C. Eliminare con cautela il surnatante.
- Preriscaldare l'incubatore a 37 °C e preparare un tampone di arresto (100 mM EDTA, pH 8,0; Tabella 1).
- Ottimizzare in anticipo le concentrazioni di nucleasi micrococcica (MNase, fare riferimento alla tabella dei materiali) e i tempi di incubazione.
- Dividere 40 μL di campione di cromatina in diverse aliquote uguali per testare diverse concentrazioni di MNasi e tempi di incubazione.
- Utilizzare un intervallo di concentrazioni di MNasi (ad esempio, 0,0625 U, 0,125 U, 0,25 U, 0,5 U, 1 U, 2 U, 4 U, 8 U per reazione) e testare più tempi di incubazione (ad esempio, 2, 5, 10 e 15 minuti).
- Aggiungere il tampone MNasi (fare riferimento alla Tabella 1) contenente varie concentrazioni di MNasi alle aliquote della cromatina e incubare i campioni a 37 °C per i tempi specificati.
- Terminare la reazione aggiungendo 1/4 di volume di tampone di arresto (concentrazione finale: 20 mM EDTA) immediatamente dopo il tempo di incubazione desiderato.
- Isolare il DNA dai campioni di cromatina digeriti utilizzando un metodo di estrazione fenolo/cloroformio/alcol isoamilico.
- Eseguire il DNA estratto su un gel di agarosio all'1,5% per visualizzare i modelli di digestione: la sottodigestione mostrerà bande di peso molecolare elevato (Figura 2, corsia 1-4); l'eccessiva digestione si tradurrà in uno striscio o in frammenti molto corti (Figura 2, corsia 6-8) e una digestione ottimale produrrà un chiaro schema a scala nucleosomica (Figura 2, corsia 5, ad es. mono-, di-, tri-nucleosomi).
- Identificare le condizioni che producono la risoluzione nucleosomica desiderata senza un'eccessiva sovradigestione.
NOTA: CaCl2 agisce come cofattore per l'attività della MNasi. Ottimizza la digestione regolando la concentrazione di CaCl2 tra 1 mM e 5 mM.
- Risospendere delicatamente i nuclei intatti con 100 μl di tampone MNase pipettandoli 5-10 volte con puntali a orifizio largo. Aggiungere immediatamente la quantità predeterminata di MNasi ai campioni (1,25 U MNasi/100 μL di tampone MNasi).
NOTA: Quando si lavora con più campioni, digerire ciascuno singolarmente per evitare una digestione eccessiva. - Posizionare le provette su un rotatore e incubare per 5 minuti a 37 °C. Rimettere immediatamente le provette nel ghiaccio e terminare la digestione della MNasi aggiungendo EDTA a una concentrazione finale di 20 mM e mescolare a vortici.
- Aggiungere 500 μl di tampone B (fare riferimento alla Tabella 1) a ciascun campione e mescolare accuratamente pipettando su e giù 5x - 10x. Solubilizzare le proteine incubandole su ghiaccio per 5 minuti.
NOTA: Il sale e il detergente nel tampone B aiutano a dissociare le proteine deboli legate alla cromatina ed espongono gli epitopi all'immunoprecipitazione. - Pellettare il materiale insolubile centrifugando alla massima velocità per 5 min a 4 °C. Trasferire il surnatante trasparente in nuove provette da 1,5 mL etichettate come frazione nativa della cromatina. I campioni possono essere conservati a -80 °C o utilizzati per convalidare l'efficienza della frammentazione della cromatina.
NOTA: Evitare frequenti cicli di gelo-disgelo, in quanto potrebbero interrompere le interazioni proteina-DNA di interesse. Ridurre al minimo i cicli di gelo-disgelo quando possibile.
3. Verifica della frammentazione della cromatina
- Aliquotare 10 μl di surnatante da ciascun campione in una nuova provetta da 1,5 mL. Miscelare con 20 μl di acqua distillata e 30 μl di fenolo/cloroformio/alcol isoamilico (25:24:1).
- Chiudere bene i tubi e vorticare energicamente per 15-30 s. Centrifugare a 20.000 x g (o la velocità massima della centrifuga) per 10 minuti a 4 °C. Dopo la centrifugazione, si osserveranno tre strati distinti: uno strato superiore chiaro, uno strato intermedio bianco e uno strato inferiore giallo.
- Trasferire con cautela 20 μl della fase acquosa superiore (contenente DNA) in una provetta nuova. Separare il DNA purificato in gel di agarosio all'1,5% per 30 minuti a 100 V e visualizzare i modelli di digestione. Assicurarsi che la dimensione dei frammenti di cromatina sia principalmente compresa tra 200 e 1000 coppie di basi.
NOTA: Le dimensioni corrette dei frammenti di cromatina sono fondamentali per il successo della ChIP nativa e dipendono dalle condizioni di trattamento con MNasi, comprese le unità enzimatiche, il tempo di incubazione e la concentrazione di CaCl2. L'efficienza della digestione della MNasi può anche variare in base al tipo e al numero di cellule. Il modello di frammentazione della cromatina mostrato nella Figura 2 (corsia 5) è raccomandato per questo test ChIP.
4. Immunoprecipitazione
- Aliquotare 20 μl di cromatina digerita da ciascun campione in una provetta fresca da 1,5 mL e miscelare con 180 μl di tampone di eluizione (fare riferimento alla Tabella 1). Etichettare queste provette come campioni di ingresso e conservarle a -20 °C.
- Trasferire 400 μL di campione di cromatina in un'altra provetta da 1,5 mL per ChIP.
- Aggiungere l'anticorpo γH2A.X (fare riferimento alla Tabella dei materiali) a un campione trattato con DMSO, a un campione trattato con afidicolina e a un campione trattato con idrossiurea. Aggiungere la stessa quantità di IgG normale (fare riferimento alla Tabella dei materiali) a un altro campione trattato con DMSO come controllo negativo per il test ChIP.
NOTA: In questo caso, 1 μg di anticorpo primario viene tipicamente utilizzato per 400 μL di cromatina (ovvero, la concentrazione finale dell'anticorpo è di 2,5 μg/mL). Tuttavia, la quantità ottimale dovrebbe essere determinata empiricamente per diversi anticorpi γH2A.X. - Posizionare le provette ChIP su un rotatore a 4 °C e incubare per almeno 5 ore, preferibilmente durante la notte.
- Nel frattempo, aliquotare 100 μL di perle di proteina magnetica A/G di grado ChIP (fare riferimento alla Tabella dei materiali) in una nuova provetta da 1,5 mL. Utilizzare puntali a orifizio largo e pipettare lentamente per garantire una misurazione accurata delle perline. Posizionare il tubo su un supporto magnetico per almeno 1 minuto, quindi scartare con cura il liquido.
- Risospendere le perle in 1 mL di 1x PBS contenente lo 0,5% di BSA. Ruotare a 4 °C per circa 4 ore. Posizionare il tubo su un supporto magnetico per almeno 1 minuto ed eliminare il surnatante.
- Lavare nuovamente le perle con 1 mL di 1x PBS contenente lo 0,5% di BSA. Posizionare il tubo sul supporto magnetico per 1 minuto per pellettare le perline magnetiche, quindi scartare il surnatante.
NOTA: I passaggi da 4.5 a 4.7 sono il pre-rivestimento delle perle per ridurre il legame non specifico degli anticorpi alle microsfere magnetiche. - Risospendere le perle pre-rivestite in 100 μL di Buffer B utilizzando punte a orifizio largo. Aggiungere 25 μl della sospensione di biglie magnetiche pre-rivestite a ciascuna provetta per campioni ChIP. Ruotare a 4 °C per 2 ore.
- Posiziona i tubi ChIP sul supporto magnetico e attendi che le perle siano completamente attaccate al lato del tubo e che la soluzione diventi limpida.
- Scartare il surnatante trasparente senza disturbare le microsfere magnetiche. Risospendere le perle con 1 mL di tampone di lavaggio (fare riferimento alla Tabella 1) e ruotare a 4 °C per 10 minuti.
- Riposizionare le provette sul supporto magnetico e attendere che la soluzione diventi limpida. Eliminare il tampone di lavaggio. Ripetere il lavaggio per un totale di quattro lavaggi.
- Eliminare il tampone di lavaggio dopo il lavaggio finale e centrifugare brevemente le provette a 400 x g per 30 s a 4 °C per eliminare il liquido residuo. Riposizionare i tubi sul supporto magnetico e rimuovere con cautela il liquido rimanente dal fondo del tubo.
5. Eluizione e precipitazione del DNA
NOTA: L'efficienza degli anticorpi può variare tra i diversi lotti. È importante confermare l'affinità di legame di un nuovo anticorpo controllando i campioni immunoprecipitati attraverso l'analisi Western blot.
- Verificare l'efficienza di pull-down degli anticorpi ChIP utilizzando Western blot (WB) come descritto di seguito.
- Prelevare una piccola aliquota del campione di ChIP per l'analisi (cioè, di solito il 10% del campione di ChIP). Includi la cromatina in ingresso (pre-immunoprecipitazione) e il controllo negativo (ad es. pull-down delle IgG) per il confronto.
- Eluire le proteine dalle microsfere legate agli anticorpi riscaldandole in 20 μL di 1 tampone di caricamento SDS-PAGE (fare riferimento alla Tabella dei materiali) a 95 °C per 5 minuti.
- Caricare i campioni IP, l'input e i controlli su un gel SDS-PAGE al 15%. Esegui il gel.
- Trasferire le proteine su una membrana di nitrocellulosa da 0,2 μm (fare riferimento alla tabella dei materiali) o PVDF utilizzando un sistema di trasferimento umido o semi-secco.
- Bloccare la membrana con latte scremato al 5% o BSA in TBST (fare riferimento alla Tabella 1) per 1 ora a temperatura ambiente per evitare legami aspecifici.
- Incubare la membrana con l'anticorpo primario contro il γH2A.X (fare riferimento alla Tabella dei materiali) diluito in tampone bloccante per 1-2 ore a temperatura ambiente o per una notte a 4 °C.
- Lavare la membrana 3 volte con TBST per rimuovere gli anticorpi non legati. Incubare la membrana con un anticorpo secondario coniugato con HRP (fare riferimento alla Tabella dei materiali) per 1 ora a temperatura ambiente. Lavare nuovamente la membrana per rimuovere gli anticorpi secondari in eccesso.
- Sviluppare la membrana utilizzando un substrato chemiluminescente e visualizzare il segnale con un imager. Confrontare l'intensità del segnale tra l'IP, le corsie di ingresso e di controllo per valutare l'efficienza e la specificità del pull-down.
NOTA: Una banda corrispondente alla proteina bersaglio nella corsia IP conferma il successo del pulldown dell'anticorpo. Questo approccio garantisce la possibilità di valutare l'efficacia dell'anticorpo nella cattura della proteina bersaglio durante l'esperimento ChIP.
- Aggiungere 50 μl di tampone di eluizione (fare riferimento alla Tabella 1) a ciascuno dei campioni ChIP rimanenti. Posizionare i tubi su un termomixer e agitare per 15 minuti a temperatura ambiente.
- Posizionare i tubi sul supporto magnetico per almeno 1 minuto. Raccogliere l'eluito in nuove provette. Ripetere 1 volta e raccogliere l'eluito nelle stesse provette.
- Aggiungere altri 100 μl di tampone di eluizione a ciascun campione di eluizione ChIP e 180 μl di tampone di eluizione a ciascun campione di input.
- Aggiungere 200 μL di fenolo/cloroformio/alcol isoamilico (25:24:1) a ciascun campione e agitare energicamente. Centrifugare i campioni a 20.000 x g (o velocità massima) per 10 minuti a 4 °C.
- Aggiungere 19 μL di acetato di sodio 3M (NaOAc, pH 5,2; fare riferimento alla Tabella 1) e 2 μL di soluzione di glicogeno (20 mg/mL, fare riferimento alla Tabella dei materiali) a ciascuna nuova provetta da centrifuga da 1,5 mL.
- Dopo la centrifugazione, trasferire con cautela lo strato acquoso superiore (circa 190 μL) nelle provette contenenti NaOAc e glicogeno e mescolare mediante vortex.
- Aggiungere 500 μl di etanolo al 100% e vortex. Far precipitare il DNA incubando i campioni a -20 °C per almeno 2 ore o durante la notte.
- Centrifugare le provette a 20.000 x g (o velocità massima) per 10 minuti a 4 °C. Scartare il surnatante, facendo attenzione a non disturbare il pallino bianco. Risospendere il pellet in 1 mL di etanolo al 70% e agitare accuratamente.
- Centrifugare le provette a 20.000 x g (o alla velocità massima) per 5 minuti a 4 °C. Rimuovere con cautela il surnatante. Centrifugare brevemente le provette per eliminare l'etanolo residuo. Rimuovere con cautela l'etanolo utilizzando una pipetta P20. Asciugare all'aria i pellet di DNA per 2-3 minuti.
NOTA: Evitare di asciugare eccessivamente il pellet, in quanto ciò può rendere difficile la ridissoluzione del DNA. - Per i campioni di ChIP, risospendere il DNA in 400 μL di tampone TE (fare riferimento alla Tabella 1). Per il DNA in ingresso, risospendere in 1000 μL di tampone TE. I campioni eluiti possono ora essere conservati a -20°C.
6. Quantificazione qPCR
- Eseguire la qPCR utilizzando un kit commerciale (fare riferimento alla Tabella dei materiali) con triplicati tecnici per ogni campione. Confermare la presenza di un singolo prodotto specifico per PCR conducendo un'analisi della curva di fusione per garantire la specificità dell'amplificazione36.
- Analisi dei dati
NOTA: Nell'analisi di quantificazione relativa, il campione di prova è espresso come variazione di piega rispetto a un campione di controllo (immunoprecipitato utilizzando IgG purificate normali o IP simulato). I loci del DNA noti per essere non occupati dalla proteina immunoprecipitata (locus negativo) possono essere utilizzati in questo modo come gene di riferimento rispetto ai loci del DNA di controllo positivo noti, occupati36.- Calcola la percentuale di input per ogni ChIP utilizzando la formula seguente
%Input = 2(-ΔCt [ChIP normalizzato]) - Normalizzare i valori ΔCt del locus positivo in locus negativo (ΔΔCt) sottraendo il valore ΔCt ottenuto per il locus positivo dal valore ΔCt per il locus negativo utilizzando la formula seguente
(ΔΔCt = ΔCtpositivo - ΔCtnegativo) - Calcolare l'arricchimento del ripiegamento della sequenza del locus positivo nel DNA ChIP rispetto al locus negativo utilizzando la formula seguente
Arricchimento delle pieghe =2ΔΔCt
Le sequenze dei primer qPCR utilizzati per l'analisi sono fornite nella Tabella 2. L'organizzazione genomica di FRA3B e FRA16D37è illustrata in Figura 3A, B.
- Calcola la percentuale di input per ogni ChIP utilizzando la formula seguente
- Analisi statistica
- Analizza i risultati statisticamente utilizzando il test t accoppiato di Student. Un valore p di ≤0,05 è considerato statisticamente significativo, indicando che è improbabile che le differenze osservate siano dovute alla variazione casuale38.
Risultati
La dimensione dei frammenti di cromatina è fondamentale per il successo del ChIP nativo, in quanto influisce direttamente sull'accessibilità delle regioni del DNA per il legame degli anticorpi. Per determinare la concentrazione ottimale di MNasi per la frammentazione della cromatina, abbiamo preparato una serie di provette per microcentrifuga contenenti concentrazioni variabili di MNasi (cioè 0,0625 U, 0,125 U, 0,25 U, 0,5 U, 1 U, 2 U, 4 U, 8 U per reazione) e 40 μL di nuclei isolati. Ogni reazione è stata incubata a 37 °C per 5 minuti per ottenere una gamma di dimensioni dei frammenti di cromatina. I risultati della digestione della MNasi sono presentati nella Figura 2.
Come mostrato nella Figura 2, concentrazioni più elevate di MNasi hanno portato a una digestione più estesa della cromatina, con conseguente predominanza di frammenti di mononucleosomi (Figura 2, corsie 1, 2 e 3). Al contrario, a concentrazioni più basse di MNasi, la maggior parte dei frammenti di cromatina erano più grandi, spesso superiori a 1 kb (Figura 2, corsie 6, 7 e 8), indicando una digestione insufficiente per le applicazioni a valle. Con una concentrazione di MNasi di 1,6 unità e un tempo di digestione di 5 minuti, abbiamo ottenuto frammenti di cromatina principalmente nell'intervallo da 200 a 1000 coppie di basi (Figura 2, corsia 5). Questa dimensione del frammento è ideale per la ChIP nativa, in quanto garantisce un arricchimento efficiente del DNA associato alla cromatina, mantenendo l'accessibilità a epitopi specifici. La dimensione appropriata del frammento facilita l'immunoprecipitazione efficiente e l'analisi a valle, come la PCR quantitativa o il sequenziamento, rendendolo adatto per rilevare le interazioni proteina-DNA ad alta risoluzione.
L'affinità di legame dell'anticorpo è un fattore critico per il successo di Native ChIP. Garantire che l'anticorpo si leghi in modo efficiente e specifico al suo epitopo bersaglio è essenziale per ottenere risultati affidabili. Pertanto, è fondamentale verificare in anticipo l'efficienza di legame dell'anticorpo. Per confermare l'efficienza dell'immunoprecipitazione γH2A.X, abbiamo condotto un saggio western blot (WB).
Abbiamo confrontato i livelli di γH2A.X tra cellule trattate con DMSO (controllo), cellule trattate con afidicolina e cellule trattate con idrossiurea (stressate dalla replicazione) utilizzando il western blotting. Come mostrato nella Figura 4A (Input), i livelli di γH2A.X erano significativamente più alti nelle cellule trattate con afidicolina rispetto alle cellule trattate con DMSO. Questo aumento di γH2A.X è coerente con l'induzione dello stress di replicazione da parte dell'afidicolina, che porta ad un aumento della formazione di rotture a doppio filamento e alla successiva fosforilazione di γH2A.X. Dopo aver eseguito ChIP, abbiamo verificato la specificità dell'immunoprecipitazione confrontando l'arricchimento di γH2A.X nei campioni di controllo IgG e γH2A.X ip. Il controllo IgG non ha abbassato alcun γH2A.X rilevabile, indicando un legame non specifico minimo (Figura 4B, N-ChIP e X-ChIP). Al contrario, il campione di γH2A.X IP ha arricchito con successo γH2A.X dalle cellule trattate con afidicolina, dimostrando un legame efficiente e specifico dell'anticorpo a γH2A.X (Figura 4, N-ChIP e X-ChIP). Questi risultati confermano che l'anticorpo γH2A.X ha un'affinità di legame e una specificità sufficienti per l'uso in esperimenti ChIP nativi, consentendo il rilevamento e l'arricchimento affidabili delle regioni della cromatina legate a γH2A.X. Questa fase di convalida è essenziale per garantire la qualità e l'accuratezza delle successive analisi ChIP.
Abbiamo utilizzato i siti fragili comuni (CFS) FRA16D e FRA3B per valutare la specificità del legame di γH2A.X al DNA in risposta allo stress di replicazione. FRA16D e FRA3B sono ben note regioni CFS soggette a instabilità in condizioni di stress di replicazione, il che le rende candidate ideali per la convalida delle interazioni γH2A.X/DNA. Le figure 3A, B illustrano l'organizzazione genomica di questi due CFS, insieme ai primer qPCR specifici utilizzati per l'analisi.
Per determinare se γH2A.X si associa a queste regioni CFS in risposta allo stress di replicazione, abbiamo eseguito l'analisi ChIP-qPCR in cellule di controllo (trattate con DMSO), trattate con afidicolina e cellule trattate con idrossiurea. I risultati della qPCR hanno mostrato che nelle cellule di controllo trattate con DMSO, γH2A.X non era arricchito né nei loci FRA16D né in FRA3B, suggerendo una mancanza di danni significativi al DNA o stress di replicazione in queste regioni (Figura 3C). Tuttavia, nelle cellule trattate con afidicolina o idrossiurea, che induce stress di replicazione inibendo la DNA polimerasi, abbiamo osservato un significativo arricchimento di γH2A.X sia a livello di FRA16D che di FRA3B (Figura 3C). Ciò indica che γH2A.X è stato reclutato in queste regioni CFS specificamente in risposta allo stress di replicazione del DNA. Questi risultati dimostrano che l'occupazione di γH2A.X nei CFS è innescata dallo stress di replicazione, supportando il suo ruolo come marcatore della risposta al danno del DNA e dello stallo della forcella di replicazione. Il reclutamento specifico di γH2A.X in FRA16D e FRA3B durante il trattamento con afidicolina convalida ulteriormente la specificità del nostro test ChIP e conferma che γH2A.X è un indicatore affidabile del danno al DNA e dello stress di replicazione nei siti fragili.
Per confrontare l'efficienza della ChIP nativa con la ChIP reticolata (X-ChIP), abbiamo condotto γH2A.X X-ChIP utilizzando lo stesso numero iniziale di cellule, seguendo il protocollo descritto da Lyu et al.39. Il reclutamento di γH2A.X in FRA16D è stato aumentato dal trattamento con afidicolina e idrossiurea, come mostrato nella Figura 3D. Tuttavia, l'arricchimento relativo di γH2A.X a FRA16D in X-ChIP era notevolmente inferiore a quello osservato con ChIP nativo, indicando una ridotta efficienza di X-ChIP in queste condizioni. Inoltre, a FRA3B, l'aumento dell'arricchimento di γH2A.X era statisticamente insignificante, fornendo ulteriori prove che X-ChIP è meno efficiente quando si lavora con un numero limitato di cellule. Questi risultati suggeriscono che la ChIP nativa potrebbe essere più adatta per rilevare l'arricchimento di γH2A.X in siti fragili quando la dimensione del campione è limitata. Analizza i risultati statisticamente utilizzando il test t accoppiato di Student. Un valore p di ≤ 0,05 è considerato statisticamente significativo.

Figura 1:Flusso di lavoro per l'esecuzione di un saggio ChIP nativo γH2A.X. (i) Le cellule vengono coltivate e sottoposte a trattamenti specifici (ad esempio, agenti dannosi per il DNA come l'idrossiurea) per indurre la segnalazione γH2A.X. (ii) Le cellule trattate vengono raccolte per la lavorazione. (iii) Le cellule vengono frazionate per isolare il γH2A.X legato alla cromatina, separandolo dagli altri componenti cellulari. (iv) La digestione della nucleasi micrococcica (MNasi) viene eseguita per frammentare la cromatina in pezzi delle dimensioni di un nucleosoma. (v) La dimensione dei frammenti di cromatina viene convalidata analizzando un campione su un gel di agarosio, assicurando che i frammenti siano della lunghezza appropriata per ChIP. vi) Un anticorpo specifico per γH2A.X viene utilizzato per immunoprecipitare frammenti di cromatina contrassegnati da γH2A.X. (vii) Le perle di proteina A/G sono utilizzate per abbattere i complessi anticorpo-cromatina, arricchendo il DNA legato a γH2A.X. viii) I frammenti di DNA marcati con γH2A.X vengono eluiti e purificati, preparandoli per le analisi a valle. (ix) Analisi a valle: qPCR e sequenziamento e analisi dei dati. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Saggio per ottimizzare la condizione di digestione della MNasi. I frammenti di cromatina sono stati preparati da cellule HEK 293T utilizzando diverse condizioni di digestione della MNasi. Il DNA purificato risultante dalla cromatina frammentata è stato separato su un gel di agarosio al 2% e fatto funzionare a 100 V per 30 minuti. Le corsie da 1 a 8 rappresentano i campioni trattati con concentrazioni crescenti di MNasi (cioè 0,0625 U, 0,125 U, 0,25 U, 0,5 U, 1 U, 2 U, 4 U, 8 U per reazione), dimostrando un gradiente di digestione della cromatina. La scala del DNA (indicata da M) è stata inclusa come riferimento per le dimensioni. I frammenti di DNA sono stati visualizzati utilizzando la soluzione di colorazione del DNA Midori Green Advance. La banda marcata corrisponde a frammenti di DNA delle dimensioni di un mononucleosoma, evidenziando l'efficienza di digestione ad ogni concentrazione di MNasi. Clicca qui per visualizzare una versione più grande di questa figura.
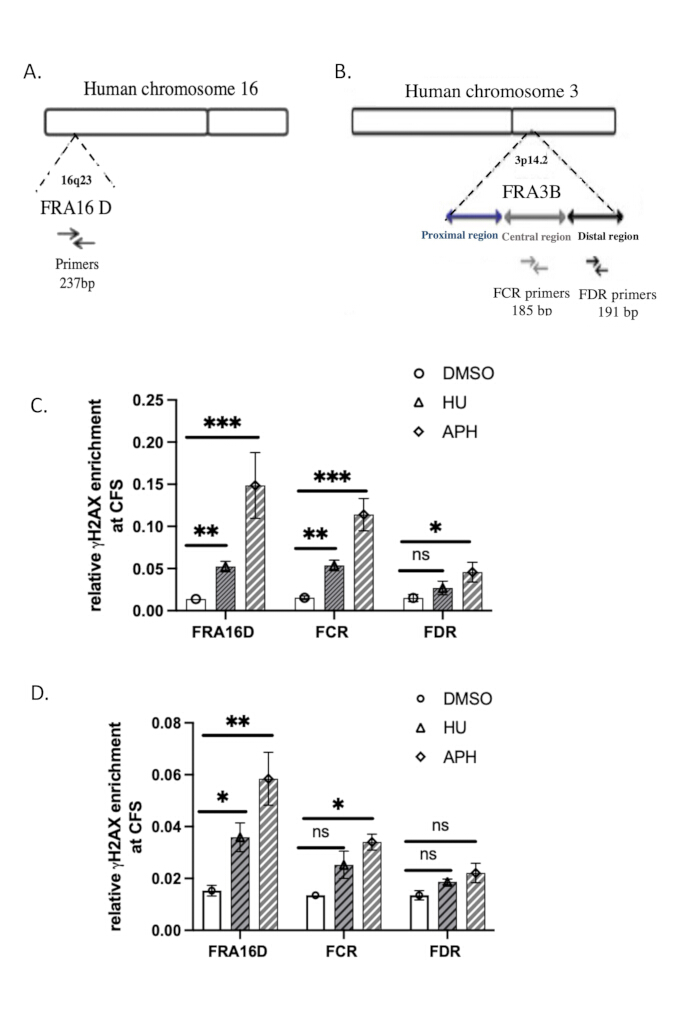
Figura 3: verifica qPCR di γH2A.X ChIP. (A) Diagramma delle organizzazioni genomiche del FRA16D. (B) Diagramma delle organizzazioni genomiche della regione FRA3B. Sono indicati i set di primer utilizzati per le analisi qPCR delle regioni FRA16D, distale (FDR) e centrale (FCR) all'interno del locus FRA3B. (C) L'arricchimento relativo di γH2A.X nei siti fragili comuni (CFS), in particolare FRA3B e FRA16D, è stato valutato utilizzando ChIP nativo seguito da analisi qPCR dopo il trattamento con afidicolina e idrossiurea. (D) L'arricchimento relativo di γH2A.X nei siti fragili comuni (CFS), in particolare FRA3B e FRA16D, è stato valutato utilizzando X-ChIP seguito da analisi qPCR dopo il trattamento con afidicolina e idrossiurea. indica un risultato statisticamente significativo con p < 0,001; ** indica p < 0,01 e * indica p < 0,05, determinato da un test t; n = 3. Clicca qui per visualizzare una versione più grande di questa figura.
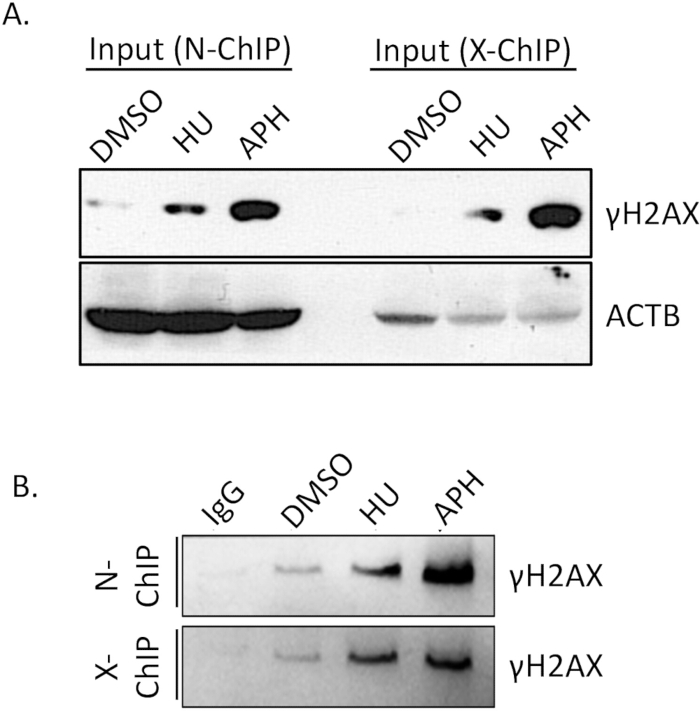
Figura 4: Verifica dell'efficienza di pull-down degli anticorpi ChIP mediante Western blot. Dal campione totale, il 10% del campione ChIP, insieme ai controlli di input, è stato separato su un gel SDS-PAGE al 15% per risolvere i polipeptidi. Dopo l'elettroforesi, le proteine separate sono state trasferite su una membrana PVDF da 0,22 μm utilizzando un protocollo di trasferimento standard. La membrana è stata quindi sondata in sequenza con un anticorpo primario specifico mirato a γH2A.X e ACTB, seguito da un anticorpo secondario coniugato HRP appropriato. Il segnale è stato successivamente visualizzato utilizzando il rilevamento a chemiluminescenza, consentendo la valutazione della specificità anticorpale e dell'efficienza di pull-down nel test ChIP. (A) Livelli di γH2A.X tra cellule trattate con DMSO (controllo), cellule trattate con afidicolina e cellule trattate con idrossiurea (stressate dalla replicazione) con ACTB come controllo del carico. (B) Arricchimento di γH2A.X nei campioni di controllo IgG e IP. Abbreviazioni: APH = afidicolina, HU = idrossiurea. Clicca qui per visualizzare una versione più grande di questa figura.
| Buffer A (1X) | |
| Reagente | Concentrazione lavorativa |
| TUBI pH 6.8 | 10 mM |
| NaCl | 100 mM |
| MgCl2 | 3 mM |
| EGTA pH 7,6 | 1 mM |
| Conservare a -20 °C per un massimo di 1 anno | |
| Tampone MNase (1X) | |
| Reagente | Concentrazione lavorativa |
| Tris-HCl, pH 7,5 | 50 mM |
| CaCl2 | 1 mM |
| MgCl2 | 4 mM |
| Conservare a -20 °C per un massimo di 1 anno | |
| Buffer B (1X) | |
| Reagente | Concentrazione lavorativa |
| Fosfato di sodio pH 7. 0 | 20 mM |
| Pirofosfato di sodio10. H2O | 30 mM |
| Kcl | 250 mM |
| EDTA, pH 8.0 | 5 mM |
| Glicerolo | 10% |
| Triton X-100 | 0.10% |
| Cocktail di inibitori della proteasi | 1x |
| Cocktail di inibitori della fosfatasi | 1x |
| DTT | 0,5 mM |
| Conservare a -20 °C per un massimo di 1 anno | |
| Tampone di eluizione | |
| Reagente | Concentrazione lavorativa |
| NaHCO3 | 50 mM |
| SDS | 1% |
| Preparata | |
| Tampone di lavaggio | |
| Reagente | Concentrazione lavorativa |
| Tampone MNase | x 0,5 |
| Buffer B | x 0,5 |
| Preparato al momento | |
| Arresta buffer | |
| EDTA | 100 mM , pH8.0 |
| 3 M Acetato di sodio , pH 5,2 | |
| 408,24 g Acetato di sodio | |
| ajust pH con acido acetico a pH 5.2 | |
| regolando il volume a 1 L | |
| Sterilizzare il tampone mediante filtrazione o autoclave | |
| Tampone TBST | |
| Tris Base, pH 8.0 | 10 mM |
| NaCl | 150 mM |
| Interpolazione 20 | 0,1% (v/v) |
| Tampone TE | |
| Tris-HCl, pH 8,0 | 10 mM |
| EDTA, pH 8.0 | 1 milione |
Tabella 1: Composizione del tampone.
| Loci genici | Innesco in avanti | Innesco inverso |
| FRA16D | TCCTGTGGAAGGGATATTTA | CCCCTCATATTCTGCTTCTA |
| FRA3B | TGTTGGAATGTTAACTCTATCCCAT | ATATCTCATCAAGACCGCTGCA |
| FCR | ||
| FRA3B | CAATGGCTTAAGCAGACATGGT | AGTGAATGGCATGGCTGGAATG |
| FDR | ||
| ACTB (controllo negativo) | GACGCAGGATGGGGG | ACGCCTCTGGCCGTACCAC |
Tabella 2: Sequenza di primer.
| Basso recupero del DNA | Potenziale problema | Possibile soluzione |
| Scarsa qualità degli anticorpi | Utilizzare anticorpi di grado ChIP. | |
| Anticorpi insufficienti | Utilizzare 1-10 μg di anticorpo ChIP per 25 μg di cromatina. | |
| Tempo di incubazione insufficiente | Eseguire la fase di immunoprecipitazione durante la notte a 4 °C. | |
| Lavaggi troppo rigorosi | Non utilizzare una concentrazione di NaCl superiore a 500 mM nel tampone di lavaggio. | |
| Perline di bassa qualità | Assicurarsi che la proteina A o G sia compatibile con l'anticorpo ChIP. | |
| Seguire la scheda tecnica del prodotto delle perline per il volume ottimale delle perle e il rapporto anticorpi. | ||
| Avviamento insufficiente | Preparare una piastra separata di celle per determinare con precisione il numero di cellule. | |
| Campione | Aumenta il numero di celle utilizzate se l'obiettivo è una bassa abbondanza. | |
| Lisi cellulare inadeguata | Ottimizza la composizione del tampone e il tempo di lisi per migliorare l'efficienza. | |
| Applicare una forza meccanica durante la fase di lisi, ad esempio utilizzando un omogeneizzatore Dounce, quando si lavora con determinati tipi di cellule. | ||
| Si è verificata la degradazione dei nuclei | Assicurarsi che l'isolamento dei nuclei sia delicato per preservare l'integrità e l'accessibilità della cromatina. | |
| Utilizzare puntali per pipette a foro largo per ridurre al minimo il taglio dei nuclei durante la manipolazione. | ||
| Si è verificata la degradazione del campione | Eseguire tutti i passaggi su ghiaccio o a 4 °C. | |
| Includere gli inibitori della proteasi in tutti i tamponi e assicurarsi che tutti i tamponi siano preparati di fresco. | ||
| Insufficiente digestione della MNasi | Titolare la concentrazione di MNasi (ad esempio, 0,1-5 unità) per determinare la quantità ottimale per un tipo di campione specifico; e iniziare con una gamma di concentrazioni per identificare le condizioni di digestione ideali. | |
| Iperdigestione della MNasi | Sperimentare diversi tempi di incubazione (ad esempio, 1-10 minuti) a 37 °C e monitorare il modello di frammentazione per garantire una digestione sufficiente senza sovradigestione. | |
| Fosforilazione interrotta | Aggiungere il cocktail di inibitori della fosfatasi al tampone di lisi e assicurarsi che tutti i tamponi siano preparati al momento. | |
| Alto background nel controllo negativo | Legatura aspecifica alle perline | Includere una fase di pre-clearing prima della fase di immunoprecipitazione e utilizzare perle magnetiche di grado ChIP, che generalmente mostrano un legame non specifico ridotto. |
| Lavaggio insufficiente | Aumentare il numero o il rigore dei lavaggi regolando la concentrazione di sale e detersivo. | |
| Insufficiente frammentazione della cromatina | Ottimizza la frammentazione per ottenere frammenti di 200-750 bp. | |
| L'ottimizzazione è necessaria per ogni tipo di cellula o tessuto | ||
| Risoluzione dei problemi relativi alla PCR | Nessuna amplificazione del campione di input | (1) Sovraframmentato: il segnale è diminuito per ampliconi superiori a 150 bp se la cromatina è frammentata alla lunghezza del mononucleosoma. |
| (2) Ottimizzare le condizioni di PCR. | ||
| (3) Progettare primer per amplificare una regione più piccola (<150 bp). | ||
| Bassa risoluzione | Dimensione del frammento di DNA troppo grande | Ottimizza la frammentazione per ottenere frammenti di 200-750 bp. |
Tabella 3: Risoluzione dei problemi di immunoprecipitazione della cromatina.
Discussione
L'inquinamento ambientale contribuisce in modo significativo ai tumori umani. Molti inquinanti sono cancerogeni, il che significa che possono causare danni genetici che portano allo sviluppo del cancro40,41. Tuttavia, determinare se una particolare sostanza è tumorigenica è un compito impegnativo. Un metodo rapido, affidabile ed economico per identificare il potenziale cancerogeno consentirebbe agli scienziati di esaminare in modo efficiente gli inquinanti ambientali e valutarne l'impatto sulla stabilità genomica. In questo studio, ci concentriamo su una variante istonica modificata, γH2A.X, che è ampiamente riconosciuta come un marcatore altamente specifico per le DSB. γH2A.X si forma quando la variante istonica H2A. X è fosforilato alla serina 139 in risposta al danno al DNA.
Rilevando la presenza di γH2A.X da parte di ChIP nativo nei CFS, possiamo valutare efficacemente l'entità del danno al DNA causato da contaminanti ambientali42. La capacità di misurare quantitativamente γH2A.X alla CFS fornisce un modo rapido, sensibile e affidabile per valutare il potenziale degli inquinanti ambientali di indurre effetti tumorigenici. A differenza dei tradizionali saggi di genotossicità, che spesso misurano marcatori indiretti di danno al DNA (come mutazioni, formazione di micronuclei o trasformazione cellulare), il ChIP nativo γH2AX consente una mappatura precisa degli eventi di rottura in specifici loci genomici42. Ciò fornisce una comprensione più approfondita della regione più vulnerabile agli inquinanti ambientali e aiuta nell'identificazione di potenziali meccanismi cancerogeni.
Il successo del ChIP nativo richiede un'attenzione particolare a diversi passaggi critici per garantire l'arricchimento efficace delle regioni target. Di seguito sono riportati i passaggi critici coinvolti nella ChIP nativa di γH2AX. Preparazione delle cellule: Iniziare con un numero adeguato di cellule. Trattare le cellule con un agente che induce danni al DNA, come l'afidicolina, per indurre lo stress di replicazione come controllo positivo. Includere campioni di controllo negativi non trattati appropriati per il confronto. Isolamento della cromatina: eseguire il frazionamento subcellulare per isolare le proteine legate alla cromatina riducendo al minimo la contaminazione da componenti citoplasmatici. Ottimizzazione della MNasi: condurre esperimenti pilota per ottimizzare la concentrazione di nucleasi micrococcica (MNase) e il tempo di incubazione. L'obiettivo è quello di ottenere frammenti di cromatina tra 200-1000 coppie di basi, garantendo una buona risoluzione per ChIP pur mantenendo l'integrità del nucleosoma. Immunoprecipitazione: utilizzare un anticorpo γH2AX di alta qualità di grado ChIP insieme alla proteina A/G agarosio per garantire un efficiente pull-down della cromatina bersaglio riducendo al minimo il legame non specifico. Lavaggio: Lavare le perle con un tampone contenente la concentrazione appropriata di sale e detersivo per rimuovere le interazioni non specifiche mantenendo la cromatina legata al γH2AX. Eluizione e recupero del DNA: Recupera in modo efficiente la cromatina legata mediante eluizione e precipitazione completa del DNA, garantendo una perdita minima della cromatina target arricchita. Rispetto all'X-ChIP convenzionale, ci sono quattro vantaggi principali del ChIP γH2AX nativo. In primo luogo, l'integrazione del frazionamento subcellulare riduce le interazioni falsi positivi e migliora la specificità della ChIP. In secondo luogo, i complessi proteina-DNA vengono preservati nel loro stato nativo, riducendo al minimo la stabilizzazione delle interazioni transitorie γH2AX-cromatina che possono verificarsi con gli agenti di reticolazione. In terzo luogo, l'omissione della fissazione e i lunghi passaggi di reticolazione inversa semplificano il flusso di lavoro complessivo. Infine, è necessario meno materiale di partenza e non sono necessarie attrezzature costose per il taglio della cromatina, come un sonicatore a bagno d'acqua (ad esempio, Bioruptor) o un sonicatore a ultrasuoni focalizzato (ad esempio, Covaris).
Sebbene il ChIP nativo abbia i suoi vantaggi, anche i suoi limiti dovrebbero essere riconosciuti. Una limitazione è il potenziale di riarrangiamento delle proteine durante la preparazione della cromatina e l'immunoprecipitazione. Questo riarrangiamento potrebbe teoricamente alterare le interazioni delle proteine legate alla cromatina e introdurre variabilità nei risultati. Sebbene non disponiamo di prove dirette a sostegno del verificarsi di tali riarrangiamenti, è importante includere controlli sperimentali appropriati per tenere conto di questo potenziale artefatto. Un'altra limitazione del test ChIP nativo è la distorsione della scissione della MNase, che può portare a risultati imprecisi. La MNasi scinde preferenzialmente le regioni ricche di A-T del genoma, rendendo queste regioni più probabilmente frammentate e arricchite durante il processo ChIP. Questa distorsione può causare una sovrarappresentazione delle sequenze ricche di A-T e una sottorappresentazione delle regioni ricche di G-C, con conseguente potenziale di segnali falsi negativi per le regioni genomiche che sono meno accessibili alla digestione della MNasi. Se i loci di interesse sono ricchi di G-C, è necessaria un'attenta ottimizzazione delle condizioni di digestione della MNasi. In alternativa, l'incorporazione di controlli aggiuntivi, come la frammentazione della cromatina mediante sonicazione, può aiutare a mitigare l'impatto della distorsione della MNasi e migliorare l'affidabilità dei risultati.
Nonostante le sue alcune limitazioni, la ChIP nativa γH2AX è particolarmente utile per identificare i loci gen in cui specifici inquinanti inducono danni al DNA. Arricchendo frammenti di DNA legati a γH2AX, i ricercatori possono identificare le regioni fragili del genoma, in particolare le CFS mediante qPCR, che sono più inclini a rompersi sotto lo stress di replicazione indotto dagli inquinanti. Questa tecnica consente inoltre ai ricercatori di stabilire una relazione diretta tra l'esposizione a uno specifico agente cancerogeno e la formazione di danni al DNA in particolari regioni genomiche, fornendo informazioni sul profilo genotossico degli inquinanti e sul loro potenziale ruolo nell'avvio della cancerogenesi43,44.
La tabella di risoluzione dei problemi (Tabella 3) fornisce soluzioni dettagliate per i problemi comuni riscontrati durante il test ChIP, come la bassa resa del DNA, l'insufficiente frammentazione della cromatina, l'elevato rumore di fondo o le scarse prestazioni degli anticorpi. Ogni problema è abbinato a raccomandazioni specifiche per garantire l'affidabilità e la riproducibilità del protocollo.
Utilizzando sistemi automatizzati di manipolazione dei liquidi e tecnologie di PCR o sequenziamento quantitativo, il ChIP nativo γH2AX è in grado di identificare in modo efficiente gli agenti che inducono danni al DNA, fungendo così da strumento prezioso negli studi tossicologici e nel monitoraggio ambientale. Utilizzando la ChIP γH2AX nativa su campioni derivati da popolazioni esposte a diversi livelli di inquinanti ambientali, i ricercatori possono identificare specifici modelli di danno al DNA e determinare il livello di rischio associato all'esposizione a particolari agenti. In conclusione, la ChIP γH2AX nativa offre vantaggi significativi per la valutazione del potenziale cancerogeno degli inquinanti ambientali, fornendo un approccio rapido, economico e ad alta specificità per rilevare il danno al DNA in specifici loci genomici. Le sue future applicazioni nello screening ad alto rendimento, nella valutazione personalizzata del rischio e nello sviluppo di biomarcatori lo rendono uno strumento prezioso per far progredire la nostra comprensione della cancerogenesi ambientale e sviluppare strategie per la prevenzione e l'intervento del cancro.
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Questo lavoro è stato supportato dai finanziamenti per le startup dell'Università della Cina meridionale.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.2 µm nitrocellulose membrane | Amersham | 10600011 | |
| Actin B | proteintech | 20536-1-AP | |
| Aphidicolin | MedChemExpress | HY-N6733 | |
| ChIP-grade magnetic Protein A/G beads | ThermoFisher | 26162 | |
| Clarity Western ECL Substrate | Bio-Rad | #1705061 | |
| Glycogen, molecular biology grade | ThermoFisher | Cat. No. R0561 | |
| HRP-conjugated secondary antibody | proteintech | SA00001-2 | |
| hydroxyurea | MedChemExpress | HY-B0313 | |
| Micrococcal Nuclease | NEB | M0247S | |
| normal IgG | Santa Cruz | sc-2025 | |
| Taq Universal SYBR Green Supermix | BioRad | 1725120 | |
| γH2A.X antibody (for ChIP) | Sigma-Aldrich | 05-636 | |
| γH2A.X antibody (for WB) | Cell Signaling | #25955 |
Riferimenti
- Glover, T. W., Berger, C., Coyle, J., Echo, B. DNA polymerase alpha inhibition by aphidicolin induces gaps and breaks at common fragile sites in human chromosomes. Hum Genet. 67 (2), 136-142 (1984).
- Bignell, G. R., et al. Signatures of mutation and selection in the cancer genome. Nature. 463 (7283), 893-898 (2010).
- Hellman, A., et al. A role for common fragile site induction in amplification of human oncogenes. Cancer Cell. 1 (1), 89-97 (2002).
- Kotzot, D., et al. Parental origin and mechanisms of formation of cytogenetically recognisable de novo direct and inverted duplications. J Med Genet. 37 (4), 281-286 (2000).
- Miller, C. T., et al. Genomic amplification of MET with boundaries within fragile site FRA7G and upregulation of MET pathways in esophageal adenocarcinoma. Oncogene. 25 (3), 409-418 (2006).
- Zack, T. I., et al. Pan-cancer patterns of somatic copy number alteration. Nat Genet. 45 (10), 1134-1140 (2013).
- Gao, G., et al. Common fragile sites (CFS) and extremely large CFS genes are targets for human papillomavirus integrations and chromosome rearrangements in oropharyngeal squamous cell carcinoma. Genes Chromosomes Cancer. 56 (1), 59-74 (2017).
- Thorland, E. C., Myers, S. L., Gostout, B. S., Smith, D. I. Common fragile sites are preferential targets for HPV16 integrations in cervical tumors. Oncogene. 22 (8), 1225-1377 (2003).
- Matovina, M., Sabol, I., Grubisić, G., Gasperov, N. M., Grce, M. Identification of human papillomavirus type 16 integration sites in high-grade precancerous cervical lesions. Gynecol Oncol. 113 (1), 120-127 (2009).
- Yu, T., et al. The role of viral integration in the development of cervical cancer. Cancer Genet Cytogenet. 158 (1), 27-34 (2005).
- Bignell, G. R., et al. Signatures of mutation and selection in the cancer genome. Nature. 463 (7283), 893-898 (2010).
- Thavathiru, E., Ludes-Meyers, J. H., MacLeod, M. C., Aldaz, C. M. Expression of common chromosomal fragile site genes, WWOX/FRA16D and FHIT/FRA3B is downregulated by exposure to environmental carcinogens, UV, and BPDE but not by IR. Mol Carcinog. 44 (3), 174-182 (2005).
- Sirbu, B. M., et al. Analysis of protein dynamics at active, stalled, and collapsed replication forks. Genes Dev. 25 (12), 1320-1327 (2011).
- Barlow, J. H., et al. Identification of early replicating fragile sites that contribute to genome instability. Cell. 152 (3), 620-632 (2013).
- Petermann, E., Orta, M. L., Issaeva, N., Schultz, N., Helleday, T. Hydroxyurea-stalled replication forks become progressively inactivated and require two different RAD51-mediated pathways for restart and repair. Mol Cell. 37 (4), 492-502 (2010).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. J Biol Chem. 273 (10), 5858-5868 (1998).
- Shanbhag, N. M., Rafalska-Metcalf, I. U., Balane-Bolivar, C., Janicki, S. M., Greenberg, R. A. ATM-dependent chromatin changes silence transcription in cis to DNA double-strand breaks. Cell. 141 (6), 970-981 (2010).
- Stiff, T., et al. ATR-dependent phosphorylation and activation of ATM in response to UV treatment or replication fork stalling. EMBO J. 25 (24), 5775-5782 (2006).
- Nelson, J. D., Denisenko, O., Bomsztyk, K. Fast chromatin immunoprecipitation assay. Nuc Acids Res. 34 (5), e2 (2006).
- Dorbic, T., Wittig, B. Isolation of oligonucleosomes from active chromatin using HMG17-specific monoclonal antibodies. Nuc Acids Res. 14 (8), 3363-3376 (1986).
- Dorbic, T., Wittig, B. Chromatin from transcribed genes contains HMG17 only downstream from the starting point of transcription. EMBO J. 6 (8), 2393-2399 (1987).
- Hebbes, T. R., Thorne, A. W., Crane-Robinson, C. A direct link between core histone acetylation and transcriptionally active chromatin. EMBO J. 7 (5), 1395-1402 (1988).
- Hebbes, T. R., Thorne, A. W., Clayton, A. L., Crane-Robinson, C. Histone acetylation and globin gene switching. Nuc Acids Res. 20 (5), 1017-1022 (1992).
- Hebbes, T. R., Clayton, A. L., Thorne, A. W., Crane-Robinson, C. Core histone hyperacetylation co-maps with generalized DNase I sensitivity in the chicken beta-globin chromosomal domain. EMBO J. 13 (8), 1823-1830 (1994).
- Orlando, V. Mapping chromosomal proteins in vivo by formaldehyde-crosslinked-chromatin immunoprecipitation. Trend Biochem, Sci. 25 (3), 99-104 (2000).
- Myers, F. A., Evans, D. R., Clayton, A. L., Thorne, A. W., Crane-Robinson, C. Targeted and extended acetylation of histones H4 and H3 at active and inactive genes in chicken embryo erythrocytes. J Biol Chem. 276 (23), 20197-20205 (2001).
- Litt, M. D., Simpson, M., Recillas-Targa, F., Prioleau, M. N., Felsenfeld, G. Transitions in histone acetylation reveal boundaries of three separately regulated neighboring loci. EMBO J. 20 (9), 2224-2235 (2001).
- Madisen, L., Krumm, A., Hebbes, T. R., Groudine, M. The immunoglobulin heavy chain locus control region increases histone acetylation along linked c-myc genes. Mol Cell Biol. 18 (11), 6281-6292 (1998).
- Clayton, A. L., Hebbes, T. R., Thorne, A. W., Crane-Robinson, C. Histone acetylation and gene induction in human cells. FEBS Lett. 336 (1), 23-26 (1993).
- Tseng, Z., Wu, T., Liu, Y., Zhong, M., Xiao, A. Using native chromatin immunoprecipitation to interrogate histone variant protein deposition in embryonic stem cells. Methods Mol Biol. 1176, 11-22 (2014).
- Durkin, S. G., Glover, T. W. Chromosome fragile sites. Ann Rev Genetics. 41, 169-192 (2007).
- Lee, J. B., Keung, A. J. Chromatin immunoprecipitation in human and yeast cells. Methods Mol Biol. 1767, 257-269 (2018).
- Miyamoto, R., Yokoyama, A. Protocol for fractionation-assisted native ChIP (fanChIP) to capture protein-protein/DNA interactions on chromatin. STAR Protoc. 2 (2), 100404 (2021).
- Mendez, J., Stillman, B. Chromatin association of human origin recognition complex, cdc6, and minichromosome maintenance proteins during the cell cycle: Assembly of prereplication complexes in late mitosis. Mol Cell Biol. 20 (22), 8602-8612 (2000).
- Nowak, D. E., Tian, B., Brasier, A. R. Two-step cross-linking method for identification of NF-κB gene network by chromatin immunoprecipitation. BioTechniques. 39 (5), 715-725 (2005).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nat Protoc. 3 (6), 1101-1108 (2008).
- Lu, X., Parvathaneni, S., Hara, T., Lal, A., Sharma, S. Replication stress induces specific enrichment of RECQ1 at common fragile sites FRA3B and FRA16D. Mol Cancer. 12, 29 (2013).
- Ruijter, J. M., et al. Amplification efficiency: linking baseline and bias in the analysis of quantitative PCR data. Nucl Acids Res. 37 (6), e45 (2009).
- Lyu, X., Chastain, M., Chai, W. Genome-wide mapping and profiling of γH2AX binding hotspots in response to different replication stress inducers. BMC Genomics. 20, 579 (2019).
- Farmer, P. B., et al. Molecular epidemiology studies of carcinogenic environmental pollutants. Effects of polycyclic aromatic hydrocarbons (PAHs) in environmental pollution on exogenous and oxidative DNA damage. Mutat Res. 544 (2-3), 397-402 (2003).
- Phillips, D. H., Arlt, V. M. Genotoxicity: damage to DNA and its consequences. EXS. 99, 87-110 (2009).
- Nitsch, S., Schneider, R. Native ChIP: Studying the genome-wide distribution of histone modifications in cells and tissue. Meth Mol Biol. 2846, (2024).
- Nikitina, T., Wang, D., Gomberg, M., Grigoryev, S. A., Zhurkin, V. B. Combined micrococcal nuclease and exonuclease III digestion reveals precise positions of the nucleosome core/linker junctions: implications for high-resolution nucleosome mapping. J Mol Biol. 425 (11), 1946-1960 (2013).
- Teves, S. S., Henikoff, S. Salt fractionation of nucleosomes for genome-wide profiling. Methods Mol Biol. 833, 421-432 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon