Method Article
Captura de roturas de sitios frágiles comunes mediante γH2A nativo. X ChIP
En este artículo
Resumen
Presentamos un método rápido y eficiente para detectar roturas comunes de sitios frágiles a través de la inmunoprecipitación de cromatina nativa γH2A.X (ChIP). Este enfoque reduce significativamente tanto el tiempo como la mano de obra asociados con los ensayos ChIP γH2A.X tradicionales, al tiempo que mantiene una alta reproducibilidad y confiabilidad de los resultados.
Resumen
El estrés de replicación inducido por la exposición a agentes extrínsecos puede provocar roturas de ADN en sitios frágiles comunes, que son regiones del genoma que se sabe que son propensas a la inestabilidad estructural. El ensayo de inmunoprecipitación de cromatina (ChIP) γH2A.X sirve como una poderosa herramienta en los estudios de genotoxicidad, ya que la fosforilación de γH2A.X es un marcador bien establecido para las roturas de doble cadena de ADN. Sin embargo, los ensayos ChIP γH2A.X tradicionales suelen requerir mucha mano de obra e implican múltiples pasos que requieren mucho tiempo. En este estudio, presentamos un método simplificado pero efectivo que combina el fraccionamiento subcelular con ChIP nativo para aislar complejos asociados a γH2A.X. Este enfoque es particularmente adecuado para analizar las interacciones de la cromatina γH2A.X con mayor especificidad y eficiencia. Mediante el fraccionamiento subcelular, los materiales no unidos a la cromatina se eliminan eficazmente, lo que da como resultado una fracción de cromatina purificada. La digestión posterior de la nucleasa microcócica (MNasa) en condiciones suaves permite la fragmentación de la cromatina al tiempo que preserva las interacciones fisiológicas entre γH2A.X y sus complejos proteicos asociados. Esta preservación es esencial para el estudio de los socios de interacción nativos involucrados en las vías de respuesta al daño del ADN. Este protocolo ChIP nativo optimizado reduce sustancialmente el tiempo y la mano de obra asociados con los ensayos ChIP γH2A.X convencionales. El procedimiento simplificado no solo simplifica el flujo de trabajo, sino que también produce resultados altamente reproducibles, lo que lo hace particularmente ventajoso en entornos donde se requiere un procesamiento de alto rendimiento de múltiples muestras. Este método tiene una amplia aplicabilidad en estudios centrados en la estabilidad del genoma, la reparación del ADN y la biología de la cromatina, donde la detección precisa y eficiente de los sitios de daño del ADN es fundamental. Mediante el empleo de protocolos optimizados y pasos simplificados, este método permite la detección de daños en el ADN en sitios frágiles con una sensibilidad mejorada y un manejo mínimo de muestras, lo que lo convierte en una herramienta valiosa para los estudios sobre la estabilidad del genoma y la respuesta al daño del ADN.
Introducción
Los sitios frágiles comunes (SFC) son grandes regiones cromosómicas que se encuentran en todos los cromosomas humanos propensos a romperse durante la metafase. Bajo estrés de replicación, la replicación en estas regiones se retrasa significativamente, evitando su duplicación completa antes de la entrada mitótica1, lo que en última instancia resulta en brechas y rupturas específicas del sitio. Los SFC son puntos calientes de inestabilidad cromosómica y son una causa importante de reordenamientos cromosómicos durante el desarrollo temprano del cáncer. El estrés de replicación, que a menudo está presente en condiciones tumorigénicas, puede conducir a la pérdida de genes supresores de tumores y a la amplificación de oncogenes, lo que se conoce colectivamente como variación del número de copias (CNV)2,3,4,5,6. Además, los SFC son muy propensos a la integración viral, lo que promueve aún más el desarrollo del cáncer 7,8,9,10. Se han detectado múltiples deleciones homocigóticas de genes supresores de tumores en las regiones del SFC durante los análisis pancancerosos de tumores primarios. Los SFC más comúnmente afectados en el cáncer incluyen FRA2F, FRA3B, FRA4F, FRA5H y FRA16D11. Los SFC son particularmente vulnerables a la rotura en presencia de agentes cancerígenos extrínsecos12. Para evaluar los efectos cancerígenos perjudiciales de los contaminantes ambientales, se necesita un método rápido y fiable para cuantificar la ocurrencia de roturas del SFC.
Fosforilación de H2A. X en el residuo de serina 139 (γH2A.X) por Ataxia Telangiectasia y Rad3-Related Protein (ATR) o Ataxia Telangiectasia Mutada (ATM) es un evento clave en la señalización del estancamiento de la horquilla de replicación13. γH2A.X sirve como indicador de horquillas de replicación estancadas antes de la formación de rotura de doble cadena (DSB)13, creando un entorno favorable para la cromatina para facilitar el reclutamiento eficiente de proteínas de reparación a los sitios estancados. Además, γH2A.X puede ser reclutado para romper sitios después del colapso de la horquilla14,15, consistente con su papel principal en la reparación de DSB. Dado que las roturas del SFC están estrechamente asociadas con las aberraciones cromosómicas que impulsan la progresión del cáncer, la detección de estas roturas puede ser fundamental para comprender las primeras etapas de la tumorigénesis. La presencia de γH2A.X en los SFC se puede utilizar como biomarcador para detectar eventos tempranos de inestabilidad genómica. Esta información puede ayudar a identificar posibles carcinógenos y evaluar el riesgo asociado con la exposición a diversos agentes extrínsecos. Al medir las roturas de ADN en los SFC inducidas por agentes extrínsecos, la cromatina γH2A.X IP (ChIP) puede proporcionar información sobre cómo dichos agentes contribuyen a los mecanismos subyacentes a la tumorigénesis.
En el ChIP convencional (es decir, ChIP reticulado, X-ChIP), la asociación de γH2A.X con sus secuencias de ADN objetivo se estabiliza mediante reticulación reversible de formaldehído. Posteriormente, la cromatina se corta en fragmentos de aproximadamente 500 pares de bases (pb) mediante sonicación, y la solución resultante se limpia de residuos mediante sedimentación 16,17,18. A continuación, se añade un anticuerpo γH2A.X de grado ChIP a la fracción de cromatina aclarada, seguido de la adición de perlas de agarosa de proteína A/G para enriquecer las regiones de cromatina unidas a γH2A.X 16,17,18. Los inmunocomplejos (es decir, el complejo de ADN dirigido a perlas-anticuerpo-γH2A.X) se lavan varias veces con tampones de lavado estrictos para eliminar los fragmentos de ADN unidos de forma no específica 16,17,18. Después del lavado, el ADN específicamente unido se eluye de los complejos inmunitarios. A continuación, se invierten los enlaces cruzados de formaldehído, seguido de la digestión de proteínas utilizando proteinasa K, tras lo cual se purifica y concentra el ADN enriquecido 16,17,18. Para evaluar las regiones asociadas a γH2A.X, se utiliza PCR, PCR cuantitativa (qPCR) o secuenciación directa 16,17,18. La ocupación de γH2A.X en regiones específicas, como el SFC, está determinada por la intensidad de la señal de PCR o qPCR, que es proporcional a la cantidad de γH2A.X unida a esa ubicación, lo que proporciona información sobre el daño y los eventos de reparación del ADN específicos del sitio 16,17,18.
A pesar de ser un poderoso enfoque experimental, el X-ChIP tiene varias limitaciones significativas: (i) requiere un gran número de células, típicamente en el rango de 1 x 107 a 5 x 107, debido a la ineficiencia de la precipitación de anticuerpos asociada con la fijación, lo que aumenta el costo total del experimento19; ii) el proceso de inversión de los enlaces cruzados de formaldehído y la posterior purificación del ADN requiere mucho tiempo y mano de obra, lo que dificulta el mantenimiento de la coherencia y la fiabilidad de los resultados; y (iii) las interacciones γH2A.X-DNA con menor significación funcional pueden no distinguirse de aquellas con mayor significación porque el paso de reticulación puede estabilizar las interacciones transitorias, lo que lleva a la detección de interacciones que pueden no ser biológicamente relevantes19.
La inmunoprecipitación de cromatina nativa (ChIP nativo o N-ChIP) es una técnica bioquímica esencial utilizada para estudiar las interacciones proteína-ADN dentro de su contexto de cromatina nativa en condiciones fisiológicas de sal. Ha sido fundamental para dilucidar la organización espacial y temporal de la cromatina, la unión de factores de transcripción y las modificaciones de histonas. Native ChIP tiene un papel de larga data en el campo más amplio de la biología y la epigenética de la cromatina, proporcionando ventajas y limitaciones únicas en comparación con X-ChIP. Este método, introducido a finales de la década de 198020, implica el aislamiento de la cromatina de las células mediante métodos que preservan su estructura nativa, como la digestión con nucleasa microcócica (MNasa)21. Esto preserva los contactos inherentes proteína-ADN e histona-ADN, lo que hace que Native ChIP sea particularmente adecuado para estudiar las modificaciones de histonas y el posicionamiento de nucleosomas en su configuración natural de cromatina22. Los estudios de ChIP nativo de alta resolución han demostrado el uso de la digestión de MNasa para reducir la cromatina a nucleosomas individuales, lo que facilita el mapeo de las modificaciones de las histonas con mayor precisión23. Además, debido a que no se trata de reticulación química, se minimiza el riesgo de introducir sesgos o artefactos que podrían tergiversar las interacciones proteína-ADN24.
A diferencia de X-ChIP, donde se utiliza formaldehído u otros agentes de reticulación para fijar las interacciones proteína-ADN, Native ChIP proporciona una visión más realista de la cromatina al evitar posibles artefactos de reticulación. Sin embargo, mientras que X-ChIP es generalmente más adecuado para detectar interacciones transitorias o dinámicas entre el ADN y las proteínas reguladoras25, Native ChIP es ideal para interacciones estables proteína-ADN, como histonas u otras proteínas unidas a la cromatina 26,27. Una de las limitaciones observadas para ChIP nativo es la incapacidad de capturar eventos de unión transitorios o de baja afinidad, que a menudo se estabilizan a través de la reticulación en X-ChIP25.
Un importante cuerpo de trabajo en epigenética ha aprovechado el ChIP nativo para descubrir modificaciones de histonas en diversos entornos biológicos28. Estos esfuerzos han sido cruciales en la definición del código de histonas, el patrón de modificaciones de histonas que regulan la expresión génica y la dinámica de la cromatina29. Aunque H2A. X es una histona enlazadora menos fuertemente asociada, la H2A nativa. El método X ChIP se ha aplicado con éxito en células madre embrionarias30. En este estudio, optimizamos un procedimiento de extracción de cromatina para realizar ChIP nativo de γH2A.X en células 293T humanas (Figura 1). La hidroxiurea y la afidicolina son ampliamente utilizadas en la investigación para investigar el estrés de replicación del ADN, el daño y la inestabilidad genómica31. En este estudio, estos agentes se aplicaron a las células para inducir estrés de replicación y generar roturas de ADN en el SFC.
Utilizando material de partida de aproximadamente 1 x 106 a 5 x 106 células, este método se puede dividir en cuatro etapas principales: (i) fraccionamiento subcelular para aislar la cromatina, (ii) digestión de la nucleasa microcócica (MNasa) para fragmentar la cromatina, (iii) inmunoprecipitación y elución, y (iv) análisis de ADN por PCR cuantitativa (qPCR). La realización de ChIP después del fraccionamiento subcelular proporciona varios beneficios y ha sido bien documentada en numerosos estudios 32,33,34,35. Este enfoque permite la eliminación de proteínas no unidas a la cromatina y otros desechos celulares, lo que da como resultado una fracción de cromatina altamente purificada. Al aislar la cromatina antes de la inmunoprecipitación, el fraccionamiento subcelular ayuda a mantener las interacciones de la cromatina nativa y reduce el ruido de fondo de las proteínas no asociadas a la cromatina, lo que conduce a resultados más específicos y confiables, ya que solo se conservan los complejos unidos a la cromatina para el análisis. Además, el fraccionamiento subcelular permite condiciones más suaves para la digestión de la cromatina, preservando así las interacciones fisiológicas proteína-ADN y ofreciendo una representación más precisa de la dinámica de la cromatina dentro del entorno celular nativo.
El uso de ChIP nativo de γH2AX para medir el impacto de los agentes extrínsecos en la rotura de sitios frágiles comunes tiene un potencial significativo para la investigación del cáncer. Esta técnica permite la detección de daños en el ADN inducidos por la exposición a carcinógenos ambientales, proporcionando información sobre los mecanismos moleculares por los cuales los contaminantes contribuyen a la inestabilidad genómica y al desarrollo del cáncer. Al preservar el contexto de la cromatina nativa, este método facilita la evaluación precisa de los patrones de daño del ADN asociados con la exposición a carcinogénicos, lo que ayuda en la evaluación de los riesgos ambientales y el estudio de la tumorigénesis impulsada por la contaminación.
Protocolo
1. Recolección de células
- Siembre alrededor de 5 x 105 células HEK 293T en cada una de las cuatro placas de 6 cm, cada una de las cuales contiene 4 mL de medio DMEM completo.
- Después de 24 h, trate un plato con 2 μL de solución madre de 1 mM de Afidicolina (consulte la Tabla de Materiales) (concentración final de 0,5 μM) y otro plato con 20 μL de solución madre de hidroxiurea 1 M (consulte la Tabla de Materiales) (concentración final de 5 mM) para inducir el estrés de replicación. Agregue DMSO a los dos platos restantes para que sirvan como controles.
- Pasadas 24 h de tratamiento, desechar los medios de cultivo. Una placa de 6 cm suele producir aproximadamente 2 x 106 celdas con una confluencia del 60%-70%.
- Enjuague cada plato 2 veces con 5 ml de PBS helado 1x. Utilice raspadores de células para separar las células y transferir la suspensión celular a cuatro tubos individuales de 1,5 ml. Pipetear suavemente hacia arriba y hacia abajo con una pipeta P1000 para disociar los grumos de células.
- Centrifugar las células a 500 x g durante 5 min a 4 °C y, a continuación, desechar el sobrenadante. Coloque las celdas en hielo.
2. Fraccionamiento subcelular
- Vuelva a suspender el pellet de células en 500 μL de tampón A frío recién preparado (consulte la Tabla 1), asegurando la disociación completa de los grupos de células mediante un pipeteo suave.
- Incubar los lisados en hielo durante 5-10 min. Verifique la progresión de la lisis bajo un microscopio para asegurarse de que la lisis celular sea completa.
- Tome una pequeña alícuota del lisado (alrededor de 5 a 10 μL) y colóquela en un portaobjetos de microscopio limpio. Cúbralo con un cubreobjetos para evitar la contaminación.
- Utilice un microscopio óptico con un aumento adecuado (por ejemplo, 20x - 40x) para visualizar células o desechos. Comparar con una muestra de control no lisada para diferenciar entre células intactas y material lisado.
NOTA: Una muestra correctamente lisada no tendrá contornos celulares distintivos, solo cromatina difusa o material celular. Ajuste el enfoque para observar claramente el lisado. Si es necesario, aplique fuerza mecánica durante el paso de lisis, como el uso de un homogeneizador Dounce, cuando trabaje con ciertos tipos de células.
- Centrifugar a 500 x g durante 5 min a 4 °C, una vez que las células estén completamente lisadas. Deseche con cuidado el sobrenadante. Vuelva a suspender el pellet de núcleos en 500 μL de tampón A frío utilizando puntas de pipeta de orificio ancho.
NOTA: Las puntas de orificio ancho ayudan a minimizar las fuerzas de cizallamiento y protegen muestras delicadas como la cromatina. Haga puntas de diámetro ancho cortando el extremo de las puntas estándar con una cuchilla afilada. - Centrifugar a 500 x g durante 5 min a 4 °C. Deseche con cuidado el sobrenadante.
- Precalentar una incubadora a 37 °C y preparar un tampón de parada (100 mM EDTA, pH 8,0; Tabla 1).
- Optimice las concentraciones de nucleasa microcócica (MNasa, consulte la Tabla de materiales) y los tiempos de incubación con anticipación.
- Divida 40 μL de la muestra de cromatina de prueba en varias alícuotas iguales para probar diferentes concentraciones de MNasa y tiempos de incubación.
- Utilice un rango de concentraciones de MNasa (p. ej., 0,0625 U, 0,125 U, 0,25 U, 0,5 U, 1 U, 2 U, 4 U, 8 U por reacción) y pruebe varios tiempos de incubación (p. ej., 2, 5, 10 y 15 min).
- Añada el tampón MNasa (consulte la Tabla 1) que contenga varias concentraciones de MNasa a las alícuotas de cromatina e incube las muestras a 37 °C durante los tiempos especificados.
- Finalice la reacción añadiendo 1/4 de volumen de tampón de parada (concentración final: 20 mM de EDTA) inmediatamente después del tiempo de incubación deseado.
- Aísle el ADN de las muestras de cromatina digeridas utilizando un método de extracción de fenol/cloroformo/alcohol isoamílico.
- Ejecute el ADN extraído en un gel de agarosa al 1,5% para visualizar los patrones de digestión: La digestión insuficiente mostrará bandas de alto peso molecular (Figura 2, carril 1-4); la digestión excesiva dará como resultado un frotis o fragmentos muy cortos (Figura 2, carril 6-8), y la digestión óptima producirá un patrón de escalera nucleosomal claro (Figura 2, carril 5, por ejemplo, mono-, di-, trinucleosomas).
- Identificar las condiciones que producen la resolución nucleosómica deseada sin una digestión excesiva.
NOTA: El CaCl2 actúa como cofactor de la actividad de la MNasa. Optimizar la digestión ajustando la concentración de CaCl2 entre 1 mM y 5 mM.
- Vuelva a suspender suavemente los núcleos intactos con 100 μL de tampón MNasa pipeteando de 5 a 10 veces con puntas de orificio ancho. Añada inmediatamente la cantidad predeterminada de MNasa a las muestras (1,25 U MNasa/100 μL de tampón MNasa).
NOTA: Cuando trabaje con varias muestras, digiera cada una individualmente para evitar la digestión excesiva. - Coloque los tubos en un rotador e incube durante 5 minutos a 37 °C. Vuelva inmediatamente los tubos a hielo y finalice la digestión de la MNasa añadiendo EDTA hasta una concentración final de 20 mM y mezcle por vórtice.
- Agregue 500 μL de tampón B (consulte la Tabla 1) a cada muestra y mezcle bien pipeteando hacia arriba y hacia abajo de 5 a 10 veces. Solubilizar las proteínas incubando en hielo durante 5 min.
NOTA: La sal y el detergente en el tampón B ayudan a disociar las proteínas débiles unidas a la cromatina y exponen los epítopos para la inmunoprecipitación. - Granular el material insoluble centrifugando a máxima velocidad durante 5 min a 4 °C. Transfiera el sobrenadante transparente a nuevos tubos de 1,5 mL marcados como la fracción de cromatina nativa. Las muestras pueden almacenarse a -80 °C o utilizarse para validar la eficacia de la fragmentación de la cromatina.
NOTA: Evite los ciclos frecuentes de congelación y descongelación, ya que pueden interrumpir las interacciones proteína-ADN de interés. Minimice los ciclos de congelación y descongelación siempre que sea posible.
3. Verificación de la fragmentación de la cromatina
- Alícuota 10 μL del sobrenadante de cada muestra en un nuevo tubo de 1,5 mL. Mezclar con 20 μL de agua destilada y 30 μL de fenol/cloroformo/alcohol isoamílico (25:24:1).
- Cierre los tubos herméticamente y en vórtice vigorosamente durante 15-30 s. Centrifugar a 20.000 x g (o la velocidad máxima de la centrífuga) durante 10 min a 4 °C. Después de la centrifugación, se observarán tres capas distintas: una capa superior transparente, una capa intermedia blanca y una capa inferior amarilla.
- Transfiera con cuidado 20 μL de la fase acuosa superior (que contiene ADN) a un tubo nuevo. Separe el ADN purificado en gel de agarosa al 1,5% durante 30 min a 100 V y visualice los patrones de digestión. Asegúrese de que el tamaño de los fragmentos de cromatina esté principalmente entre 200 y 1000 pares de bases.
NOTA: Los tamaños adecuados de los fragmentos de cromatina son cruciales para el éxito de la ChIP nativa y dependen de las condiciones de tratamiento con MNasa, incluidas las unidades de enzimas, el tiempo de incubación y la concentración de CaCl2. La eficiencia de la digestión de la MNasa también puede variar según el tipo y el número de células. Se recomienda el patrón de fragmentación de la cromatina que se muestra en la Figura 2 (carril 5) para este ensayo ChIP.
4. Inmunoprecipitación
- Alícuota 20 μL de cromatina digerida de cada muestra en un tubo nuevo de 1,5 mL y mezclar con 180 μL de tampón de elución (consultar la Tabla 1). Etiquete estos tubos como muestras de entrada y guárdelos a -20 °C.
- Transfiera 400 μL de muestra de cromatina a otro tubo de 1,5 mL para ChIP.
- Agregue el anticuerpo γH2A.X (consulte la Tabla de materiales) a una muestra tratada con DMSO, una muestra tratada con Phidicolina y una muestra tratada con hidroxiurea. Agregue la misma cantidad de IgG normal (consulte la Tabla de materiales) a otra muestra tratada con DMSO como control negativo para el ensayo ChIP.
NOTA: Aquí, 1 μg de anticuerpo primario se usa típicamente para 400 μL de cromatina (es decir, la concentración final del anticuerpo es de 2,5 μg/mL). Sin embargo, la cantidad óptima debe determinarse empíricamente para los diferentes anticuerpos γH2A.X. - Coloque los tubos ChIP en un rotador a 4 °C e incube durante al menos 5 h, o preferiblemente durante la noche.
- Mientras tanto, alicuota 100 μL de perlas magnéticas de proteína A/G de grado ChIP (consulte la Tabla de materiales) en un nuevo tubo de 1,5 mL. Utilice puntas de orificio ancho y pipetee lentamente para garantizar una medición precisa de las perlas. Coloque el tubo en un soporte magnético durante al menos 1 minuto, luego deseche el líquido con cuidado.
- Vuelva a suspender las perlas en 1 ml de 1x PBS que contenga 0,5% de BSA. Gire a 4 °C durante aproximadamente 4 h. Coloque el tubo en un soporte magnético durante al menos 1 minuto y deseche el sobrenadante.
- Lavar las perlas de nuevo con 1 mL de 1x PBS que contenga 0,5% de BSA. Coloque el tubo en el soporte magnético durante 1 minuto para granular las perlas magnéticas, luego deseche el sobrenadante.
NOTA: Los pasos 4.5 a 4.7 son el recubrimiento previo de las perlas para reducir la unión inespecífica de los anticuerpos a las perlas magnéticas. - Vuelva a suspender las perlas prerrecubiertas en 100 μL de tampón B utilizando puntas de orificio ancho. Añada 25 μL de la suspensión de perlas magnéticas prerrevestida a cada tubo de muestra de ChIP. Rotar a 4 °C durante 2 h.
- Coloque los tubos ChIP en el soporte magnético y espere hasta que las perlas estén completamente adheridas al costado del tubo y la solución se vuelva transparente.
- Deseche el sobrenadante transparente sin alterar las perlas magnéticas. Vuelva a suspender las perlas con 1 mL de tampón de lavado (consulte la Tabla 1) y gire a 4 °C durante 10 min.
- Vuelva a colocar los tubos en el soporte magnético y espere hasta que la solución se aclare. Deseche el tampón de lavado. Repita el lavado para un total de cuatro lavados.
- Deseche el tampón de lavado después del lavado final y centrifugue brevemente los tubos a 400 x g durante 30 s a 4 °C para centrifugar cualquier líquido residual. Vuelva a colocar los tubos en el soporte magnético y retire con cuidado cualquier líquido restante del fondo del tubo.
5. Elución y precipitación del ADN
NOTA: La eficacia de los anticuerpos puede variar entre los diferentes lotes. Es importante confirmar la afinidad de unión de un nuevo anticuerpo mediante el control de las muestras inmunoprecipitadas mediante el análisis de Western blot.
- Verifique la eficiencia de la extracción de anticuerpos ChIP mediante Western blot (WB) como se describe a continuación.
- Tome una pequeña alícuota de la muestra de ChIP para el análisis (es decir, generalmente el 10% de la muestra de ChIP). Incluya la cromatina de entrada (preinmunoprecipitación) y el control negativo (p. ej., reducción de IgG) para la comparación.
- Eluir las proteínas de las perlas unidas a anticuerpos calentando 20 μL de 1x tampón de carga SDS-PAGE (consulte la Tabla de materiales) a 95 °C durante 5 min.
- Cargue las muestras IP, la entrada y los controles en un gel SDS-PAGE al 15 %. Corre el gel.
- Transfiera las proteínas a una membrana de nitrocelulosa de 0,2 μm (consulte la Tabla de materiales) o PVDF utilizando un sistema de transferencia húmedo o semiseco.
- Bloquee la membrana con leche descremada al 5% o BSA en TBST (consulte la Tabla 1) durante 1 h a temperatura ambiente para evitar la unión inespecífica.
- Incubar la membrana con el anticuerpo primario contra el γH2A.X (consultar la Tabla de Materiales) diluido en tampón de bloqueo durante 1-2 h a temperatura ambiente o toda la noche a 4 °C.
- Lave la membrana 3 veces con TBST para eliminar los anticuerpos no unidos. Incubar la membrana con un anticuerpo secundario conjugado con HRP (consultar la Tabla de materiales) durante 1 h a temperatura ambiente. Vuelva a lavar la membrana para eliminar el exceso de anticuerpos secundarios.
- Desarrolle la membrana utilizando un sustrato quimioluminiscente y visualice la señal con un generador de imágenes. Compare la intensidad de la señal entre los carriles IP, de entrada y de control para evaluar la eficiencia y la especificidad del pull-down.
NOTA: Una banda correspondiente a la proteína objetivo en el carril IP confirma el éxito de la reducción de anticuerpos. Este enfoque garantiza que pueda evaluar la eficacia del anticuerpo en la captura de la proteína objetivo durante el experimento ChIP.
- Agregue 50 μL de tampón de elución (consulte la Tabla 1) a cada una de las muestras de ChIP restantes. Coloque los tubos en una termobatidora y agite durante 15 minutos a temperatura ambiente.
- Coloque los tubos en el soporte magnético durante al menos 1 minuto. Recoja el eluido en tubos nuevos. Repita 1 vez y recoja el eluto en los mismos tubos.
- Agregue 100 μL adicionales de tampón de elución a cada muestra de elución de ChIP y 180 μL de tampón de elución a cada muestra de entrada.
- Añadir 200 μL de fenol/cloroformo/alcohol isoamílico (25:24:1) a cada muestra y vórtice vigorosamente. Centrifugar las muestras a 20.000 x g (o velocidad máxima) durante 10 min a 4 °C.
- Agregue 19 μL de acetato de sodio 3M (NaOAc, pH 5.2; consulte la Tabla 1) y 2 μL de solución de glucógeno (20 mg/mL, consulte la Tabla de Materiales) a cada nuevo tubo de centrífuga de 1.5 mL.
- Después de la centrifugación, transfiera con cuidado la capa acuosa superior (aproximadamente 190 μL) a los tubos que contienen NaOAc y glucógeno y mezcle mediante vórtice.
- Agregue 500 μL de etanol 100% y vórtice. Precipitar el ADN incubando las muestras a -20 °C durante al menos 2 h o toda la noche.
- Centrifugar los tubos a 20.000 x g (o velocidad máxima) durante 10 minutos a 4 °C. Deseche el sobrenadante, teniendo cuidado de no alterar la bolita blanca. Vuelva a suspender el pellet en 1 mL de etanol al 70% y vórtice completamente.
- Centrifugar los tubos a 20.000 x g (o velocidad máxima) durante 5 min a 4 °C. Retire con cuidado el sobrenadante. Vuelva a centrifugar brevemente los tubos para centrifugar cualquier etanol residual. Retire con cuidado el etanol con una pipeta P20. Seque al aire los gránulos de ADN durante 2-3 minutos.
NOTA: Evite secar demasiado el pellet, ya que esto puede dificultar la redisolución del ADN. - En el caso de las muestras de ChIP, vuelva a suspender el ADN en 400 μL de tampón TE (consulte la Tabla 1). Para el ADN de entrada, vuelva a suspender en 1000 μL de tampón TE. Las muestras eluidas ahora se pueden almacenar a -20 °C.
6. Cuantificación de qPCR
- Realice qPCR utilizando un kit comercial (consulte la Tabla de materiales) con triplicados técnicos para cada muestra. Confirme la presencia de un único producto de PCR específico mediante la realización de un análisis de la curva de fusión para garantizar la especificidad de la amplificación36.
- Análisis de datos
NOTA: En el análisis de cuantificación relativa, la muestra de prueba se expresa como un cambio de pliegue en relación con una muestra de control (inmunoprecipitada utilizando IgG purificada normal o IP simulada). Los loci de ADN que se sabe que no están ocupados por la proteína inmunoprecipitada (locus negativo) se pueden utilizar de esta manera como un gen de referencia en comparación con los loci de ADN de control positivo conocidos, ocupados y positivos36.- Calcule el porcentaje de entrada para cada ChIP usando la siguiente fórmula
%entrada = 2(-ΔCt [ChIP normalizado]) - Normalice los valores de ΔCt del lugar geométrico positivo a un lugar geométrico negativo (ΔΔCt) restando el valor de ΔCt obtenido para el lugar geométrico positivo del valor de ΔCt para el lugar geométrico negativo utilizando la siguiente fórmula
(ΔΔCt = ΔCtpositivo - ΔCtnegativo) - Calcule el enriquecimiento de pliegues de la secuencia de locus positivo en el ADN ChIP sobre el locus negativo utilizando la siguiente fórmula
Enriquecimiento de pliegues =2ΔΔCt
Las secuencias de los cebadores de qPCR utilizados para el análisis se proporcionan en la Tabla 2. La organización genómica de FRA3B y FRA16D37se muestra en la Figura 3A,B.
- Calcule el porcentaje de entrada para cada ChIP usando la siguiente fórmula
- Análisis estadístico
- Analice los resultados estadísticamente utilizando la prueba t emparejada de Student. Un valor p de ≤0,05 se considera estadísticamente significativo, lo que indica que es poco probable que las diferencias observadas se deban a la variación aleatoria38.
Resultados
El tamaño de los fragmentos de cromatina es crucial para el éxito de la ChIP nativa, ya que afecta directamente a la accesibilidad de las regiones de ADN para la unión de anticuerpos. Para determinar la concentración óptima de MNasa para la fragmentación de la cromatina, preparamos una serie de tubos de microcentrífuga que contenían concentraciones variables de MNasa (es decir, 0,0625 U, 0,125 U, 0,25 U, 0,5 U, 1 U, 2 U, 4 U, 8 U por reacción) y 40 μL de núcleos aislados. Cada reacción se incubó a 37 °C durante 5 minutos para lograr una variedad de tamaños de fragmentos de cromatina. Los resultados de la digestión de MNase se presentan en la Figura 2.
Como se muestra en la Figura 2, las concentraciones más altas de MNasa condujeron a una digestión más extensa de la cromatina, lo que resultó en un predominio de fragmentos de mononucleosoma (Figura 2, carriles 1, 2 y 3). Por el contrario, a concentraciones más bajas de MNasa, la mayoría de los fragmentos de cromatina eran más grandes, a menudo superando 1 kb (Figura 2, carriles 6, 7 y 8), lo que indica una digestión insuficiente para aplicaciones posteriores. Con una concentración de MNasa de 1,6 unidades y un tiempo de digestión de 5 min, obtuvimos fragmentos de cromatina principalmente en el rango de 200 a 1000 pares de bases (Figura 2, carril 5). Este tamaño de fragmento es ideal para ChIP nativo, ya que garantiza un enriquecimiento eficiente del ADN asociado a la cromatina al tiempo que mantiene la accesibilidad a epítopos específicos. El tamaño adecuado del fragmento facilita la inmunoprecipitación eficiente y el análisis posterior, como la PCR cuantitativa o la secuenciación, lo que lo hace adecuado para detectar interacciones proteína-ADN con alta resolución.
La afinidad de unión del anticuerpo es un factor crítico para el éxito de ChIP nativo. Garantizar que el anticuerpo se una de manera eficiente y específica a su epítopo objetivo es esencial para lograr resultados confiables. Por lo tanto, es crucial verificar de antemano la eficacia de unión del anticuerpo. Para confirmar la eficiencia de la inmunoprecipitación γH2A.X, realizamos un ensayo de Western blot (WB).
Comparamos los niveles de γH2A.X entre células tratadas con DMSO (control), células tratadas con afidicolina y células tratadas con hidroxiurea (estresadas por replicación) mediante Western blot. Como se muestra en la Figura 4A (entrada), los niveles de γH2A.X fueron significativamente más altos en las células tratadas con afidicolina en comparación con las células tratadas con DMSO. Este aumento en γH2A.X es consistente con la inducción de estrés de replicación por aphidicolina, lo que conduce a una mayor formación de roturas de doble cadena y la posterior fosforilación de γH2A.X. Después de realizar ChIP, verificamos la especificidad de la inmunoprecipitación comparando el enriquecimiento de γH2A.X en el control de IgG y las muestras de γH2A.X IP. El control de IgG no disminuyó ningún γH2A.X detectable, lo que indica una unión no específica mínima (Figura 4B, N-ChIP y X-ChIP). Por el contrario, la muestra de γH2A.X IP enriqueció con éxito γH2A.X de las células tratadas con afidicolina, demostrando una unión eficiente y específica del anticuerpo a γH2A.X (Figura 4, N-ChIP y X-ChIP). Estos resultados confirman que el anticuerpo γH2A.X tiene suficiente afinidad de unión y especificidad para su uso en experimentos de ChIP nativo, lo que permite la detección y el enriquecimiento fiables de las regiones de cromatina unidas a γH2A.X. Este paso de validación es esencial para garantizar la calidad y la precisión de los análisis ChIP posteriores.
Utilizamos los sitios frágiles comunes (CFS) FRA16D y FRA3B para evaluar la especificidad de la unión de γH2A.X al ADN en respuesta al estrés de replicación. FRA16D y FRA3B son regiones CFS bien conocidas propensas a la inestabilidad en condiciones de estrés de replicación, lo que las convierte en candidatas ideales para validar las interacciones γH2A.X/ADN. La figura 3A,B ilustra la organización genómica de estos dos SFC, junto con los cebadores específicos de qPCR utilizados para el análisis.
Para determinar si γH2A.X se asocia con estas regiones de CFS en respuesta al estrés de replicación, realizamos un análisis ChIP-qPCR en células de control (tratadas con DMSO), tratadas con afidicolina y células tratadas con hidroxiureas. Los resultados de la qPCR mostraron que en las células de control tratadas con DMSO, γH2A.X no estaba enriquecido ni en los loci FRA16D ni en FRA3B, lo que sugiere una falta de daño significativo en el ADN o estrés de replicación en estas regiones (Figura 3C). Sin embargo, en las células tratadas con afidicolina o hidroxiurea, que induce estrés de replicación mediante la inhibición de la ADN polimerasa, observamos un enriquecimiento significativo de γH2A.X tanto en FRA16D como en FRA3B (Figura 3C). Esto indica que γH2A.X fue reclutado para estas regiones del SFC específicamente en respuesta al estrés de replicación del ADN. Estos hallazgos demuestran que la ocupación de γH2A.X en los SFC se desencadena por el estrés de replicación, lo que respalda su papel como marcador de la respuesta al daño del ADN y el estancamiento de la horquilla de replicación. El reclutamiento específico de γH2A.X para FRA16D y FRA3B durante el tratamiento con aphidicolina valida aún más la especificidad de nuestro ensayo ChIP y confirma que γH2A.X es un indicador fiable del daño en el ADN y del estrés de replicación en sitios frágiles.
Para comparar la eficiencia de ChIP nativo con ChIP reticulado (X-ChIP), realizamos γH2A.X X-ChIP utilizando el mismo número inicial de celdas, siguiendo el protocolo descrito por Lyu et al.39. El reclutamiento de γH2A.X para FRA16D aumentó con el tratamiento con afidicolina e hidroxiurea, como se muestra en la Figura 3D. Sin embargo, el enriquecimiento relativo de γH2A.X en FRA16D en X-ChIP fue notablemente menor que el observado con ChIP nativo, lo que indica una eficiencia reducida de X-ChIP en estas condiciones. Además, en FRA3B, el aumento en el enriquecimiento de γH2A.X fue estadísticamente insignificante, lo que proporciona evidencia adicional de que X-ChIP es menos eficiente cuando se trabaja con un número limitado de células. Estos resultados sugieren que la ChIP nativa puede ser más adecuada para detectar el enriquecimiento de γH2A.X en sitios frágiles cuando el tamaño de la muestra es limitado. Analice los resultados estadísticamente utilizando la prueba t emparejada de Student. Un valor p de ≤ 0,05 se considera estadísticamente significativo.

Figura 1: Flujo detrabajo para realizar un ensayo ChIP γH2A.X nativo. (i) Las células se cultivan y se someten a tratamientos específicos (por ejemplo, agentes que dañan el ADN como la hidroxiurea) para inducir la señalización de γH2A.X. (ii) Las células tratadas se recolectan para su procesamiento. (iii) Las células se fraccionan para aislar la γH2A.X unida a la cromatina, separándola de otros componentes celulares. (iv) La digestión de la nucleasa microcócica (MNasa) se realiza para fragmentar la cromatina en trozos del tamaño de un nucleosoma. (v) El tamaño de los fragmentos de cromatina se valida mediante el paso de una muestra en un gel de agarosa, asegurando que los fragmentos tengan la longitud adecuada para ChIP. (vi) Se utiliza un anticuerpo específico para γH2A.X para inmunoprecipitar fragmentos de cromatina marcados por γH2A.X. (vii) Las perlas de proteína A/G se utilizan para reducir los complejos anticuerpo-cromatina, enriqueciendo el ADN unido a γH2A.X. (viii) Los fragmentos de ADN marcados con γH2A.X se eluyen y purifican, preparándolos para los análisis posteriores. (ix) Análisis posterior: qPCR y secuenciación y análisis de datos. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Ensayo para optimizar la condición de digestión de MNasa. Los fragmentos de cromatina se prepararon a partir de células HEK 293T utilizando diferentes condiciones de digestión de MNasa. El ADN purificado resultante de la cromatina fragmentada se separó en un gel de agarosa al 2% y se ejecutó a 100 V durante 30 min. Los carriles 1 a 8 representan muestras tratadas con concentraciones crecientes de MNasa (es decir, 0,0625 U, 0,125 U, 0,25 U, 0,5 U, 1 U, 2 U, 4 U, 8 U por reacción), lo que demuestra un gradiente de digestión de la cromatina. Se incluyó la escalera de ADN (indicada por M) como referencia de tamaño. Los fragmentos de ADN se visualizaron utilizando la solución de tinción de ADN Midori Green Advance. La banda marcada corresponde a fragmentos de ADN del tamaño de un mononucleosoma, destacando la eficiencia de la digestión a cada concentración de MNasa. Haga clic aquí para ver una versión más grande de esta figura.
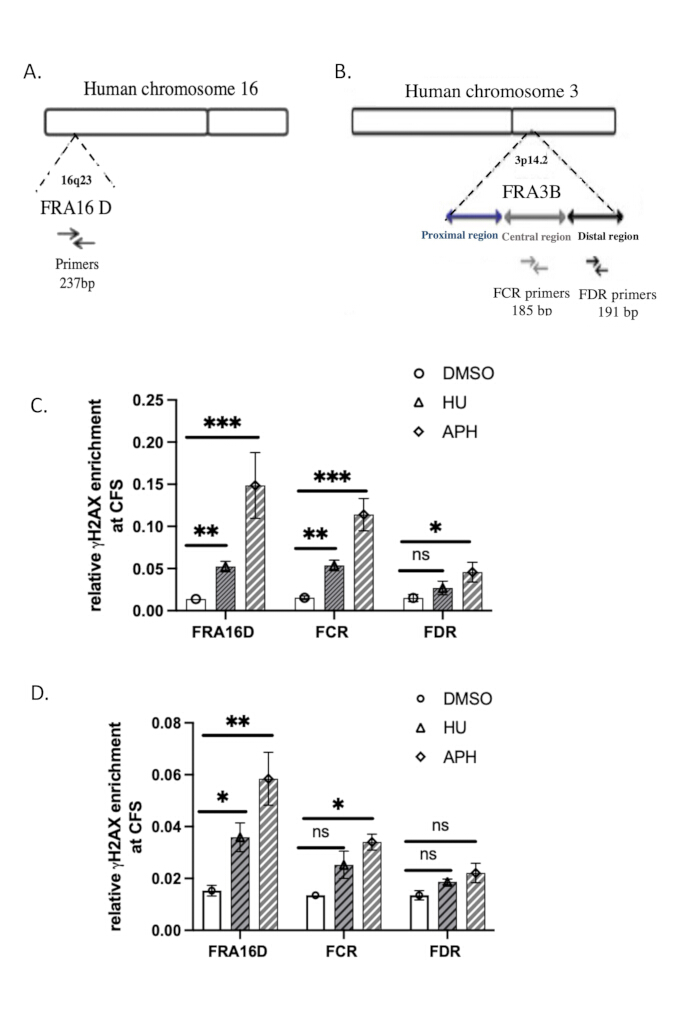
Figura 3: Verificación qPCR de γH2A.X ChIP. (A) Diagrama de las organizaciones genómicas de la FRA16D. (B) Diagrama de organizaciones genómicas de la región FRA3B. Se indican los conjuntos de cebadores utilizados para los análisis de qPCR de las regiones FRA16D, distal (FDR) y central (FCR) dentro del locus FRA3B. (C) El enriquecimiento relativo de γH2A.X en sitios frágiles comunes (SFC), específicamente FRA3B y FRA16D, se evaluó utilizando ChIP nativo seguido de análisis de qPCR después del tratamiento con afidiolina e hidroxiurea. (D) El enriquecimiento relativo de γH2A.X en sitios frágiles comunes (SFC), específicamente FRA3B y FRA16D, se evaluó utilizando X-ChIP seguido de análisis de qPCR después del tratamiento con afidiolina e hidroxiurea. indica un resultado estadísticamente significativo con p < 0,001; ** indica p < 0,01 y * indica p < 0,05, determinado por una prueba t; n = 3. Haga clic aquí para ver una versión más grande de esta figura.
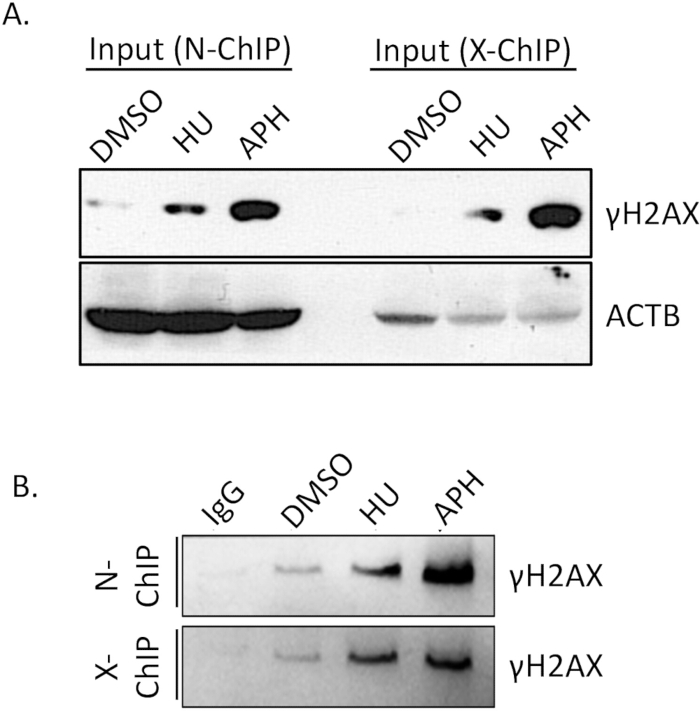
Figura 4: Verificación de la eficiencia de la reducción de anticuerpos ChIP mediante Western blot. De la muestra total, el 10% de la muestra de ChIP, junto con los controles de entrada, se separó en un gel SDS-PAGE al 15% para resolver los polipéptidos. Después de la electroforesis, las proteínas separadas se transfirieron a una membrana de PVDF de 0,22 μm utilizando un protocolo de transferencia estándar. A continuación, la membrana se probó secuencialmente con un anticuerpo primario específico dirigido a γH2A.X y ACTB, seguido de un anticuerpo secundario conjugado con HRP adecuado. Posteriormente, la señal se visualizó mediante detección de quimioluminiscencia, lo que permitió evaluar la especificidad del anticuerpo y la eficiencia de la reducción en el ensayo ChIP. (A) Niveles de γH2A.X entre células tratadas con DMSO (control), células tratadas con afidicolina y células tratadas con hidroxiurea (estresadas por replicación) con ACTB como control de carga. (B) Enriquecimiento de γH2A.X en el control de IgG y muestras de IP. Abreviaturas: APH = afidicolina, HU = hidroxiurea. Haga clic aquí para ver una versión más grande de esta figura.
| Búfer A (1X) | |
| Reactivo | Concentración de trabajo |
| TUBERÍAS pH 6.8 | 10 mM |
| NaCl | 100 mM |
| MgCl2 | 3 mM |
| EGTA pH 7.6 | 1 mM |
| Almacenar a -20 °C durante un máximo de 1 año | |
| Tampón MNase (1X) | |
| Reactivo | Concentración de trabajo |
| Tris-HCl, pH 7.5 | 50 mM |
| CaCl2 | 1 mM |
| MgCl2 | 4 mM |
| Almacenar a -20 °C durante un máximo de 1 año | |
| Búfer B (1X) | |
| Reactivo | Concentración de trabajo |
| Fosfato de sodio pH 7. 0 | 20 mM |
| Pirofosfato de sodio10. H2O | 30 mM |
| Kcl | 250 mM |
| EDTA, pH 8.0 | 5 mM |
| Glicerol | 10% |
| Tritón X-100 | 0.10% |
| Cóctel de inhibidores de la proteasa | 1 vez |
| Cóctel de inhibidores de la fosfatasa | 1 vez |
| TDT | 0,5 mM |
| Almacenar a -20 °C durante un máximo de 1 año | |
| Tampón de elución | |
| Reactivo | Concentración de trabajo |
| NaHCO3 | 50 mM |
| SDS | 1% |
| Recién preparado | |
| Tampón de lavado | |
| Reactivo | Concentración de trabajo |
| Búfer MNase | x 0,5 |
| Búfer B | x 0,5 |
| Recién preparado | |
| Búfer de parada | |
| EDTA | 100 mM , pH8.0 |
| 3 M Acetato de sodio, pH 5.2 | |
| 408.24 g Acetato de sodio | |
| pH justo con ácido acético a pH 5.2 | |
| ajustar el volumen a 1 L | |
| Esterilizar el tampón por filtración o autoclave | |
| Búfer TBST | |
| Tris Base, pH 8.0 | 10 mM |
| NaCl | 150 mM |
| Preadolescente 20 | 0.1% (v/v) |
| Búfer TE | |
| Tris-HCl, pH 8.0 | 10 mM |
| EDTA, pH 8.0 | 1 mM |
Tabla 1: Composición del tampón.
| Loci genéticos | Imprimación directa | Imprimación inversa |
| FRA16D | TCCTGTGGAAGGGATATTTA | CCCCTCATATTCTGCTTCTA |
| FRA3B | TGTTGGAATGTTAACTCTATCCCAT | ATATCTCATCAAGACCGCTGCA |
| FCR | ||
| FRA3B | CAATGGCTTAAGCAGACATGGT | AGTGAATGGCATGGCTGGAATG |
| FDR | ||
| ACTB (control negativo) | GACGCAGGATGGCATGGG | ACGCCTCTGGCCGTACCAC |
Tabla 2: Secuencia de cebadores.
| Baja recuperación de ADN | Problema potencial | Posible solución |
| Mala calidad del anticuerpo | Utilice un anticuerpo de grado ChIP. | |
| Anticuerpo insuficiente | Utilice 1-10 μg de anticuerpo ChIP por cada 25 μg de cromatina. | |
| Tiempo de incubación insuficiente | Realizar la etapa de inmunoprecipitación durante la noche a 4 °C. | |
| Lavados demasiado estrictos | No utilice una concentración de NaCl superior a 500 mM en el tampón de lavado. | |
| Cuentas de baja calidad | Asegúrese de que la proteína A o G sea compatible con el anticuerpo ChIP. | |
| Siga la hoja de datos del producto de perlas para obtener un volumen óptimo de perlas y una proporción de anticuerpos. | ||
| Arranque insuficiente | Prepare una placa separada de celdas para determinar con precisión el número de celdas. | |
| Muestra | Aumente el número de celdas utilizadas si el objetivo es de baja abundancia. | |
| Lisis celular inadecuada | Optimice la composición del tampón y el tiempo de lisis para mejorar la eficiencia. | |
| Aplique fuerza mecánica durante el paso de lisis, como el uso de un homogeneizador Dounce, cuando trabaje con ciertos tipos de células. | ||
| Se produjo la degradación de los núcleos | Asegúrese de que el aislamiento de los núcleos sea suave para preservar la integridad y la accesibilidad de la cromatina. | |
| Utilice puntas de pipeta de diámetro ancho para minimizar el cizallamiento de los núcleos durante la manipulación. | ||
| Se produjo la degradación de la muestra | Realice todos los pasos sobre hielo o a 4 °C. | |
| Incluya inhibidores de la proteasa en todos los tampones y asegúrese de que todos los tampones estén recién preparados. | ||
| Digestión insuficiente de MNasa | Valorar la concentración de MNasa (p. ej., 0,1-5 unidades) para determinar la cantidad óptima para un tipo de muestra específico; y comenzar con un rango de concentraciones para identificar las condiciones ideales de digestión. | |
| Sobredigestión de MNasa | Experimente con diferentes tiempos de incubación (por ejemplo, de 1 a 10 minutos) a 37 °C y controle el patrón de fragmentación para garantizar una digestión suficiente sin digestión excesiva. | |
| Fosforilación interrumpida | Agregue un cóctel de inhibidores de fosfatasa al tampón de lisis y asegúrese de que todos los tampones estén recién preparados. | |
| Alta experiencia en control negativo | Encuadernación inespecífica a cuentas | Incluya un paso de pre-aclarado antes del paso de inmunoprecipitación, y use perlas magnéticas de grado ChIP, que generalmente exhiben una unión inespecífica reducida. |
| Lavado insuficiente | Aumente el número o la rigurosidad de los lavados ajustando la concentración de sal y detergente. | |
| Fragmentación insuficiente de la cromatina | Optimice la fragmentación para lograr fragmentos de 200-750 pb. | |
| La optimización es necesaria para cada tipo de célula o tejido | ||
| Solución de problemas de PCR | No hay amplificación de la muestra de entrada | (1) Sobrefragmentada: la señal disminuye para amplicones de más de 150 pb si la cromatina se fragmenta a una longitud de mononucleosoma. |
| (2) Optimizar las condiciones de PCR. | ||
| (3) Diseñe cebadores para amplificar una región más pequeña (<150 pb). | ||
| Baja resolución | El tamaño del fragmento de ADN es demasiado grande | Optimice la fragmentación para lograr fragmentos de 200-750 pb. |
Tabla 3: Solución de problemas de inmunoprecipitación de cromatina.
Discusión
La contaminación ambiental contribuye significativamente a los cánceres humanos. Muchos contaminantes son cancerígenos, lo que significa que pueden causar daños genéticos que conducen al desarrollo de cáncer40,41. Sin embargo, determinar si una sustancia en particular es tumorigénica es una tarea difícil. Un método rápido, fiable y rentable para identificar el potencial carcinogénico permitiría a los científicos examinar eficazmente los contaminantes ambientales y evaluar su impacto en la estabilidad genómica. En este estudio, nos centramos en una variante modificada de la histona, γH2A.X, que es ampliamente reconocida como un marcador altamente específico para los DSB. γH2A.X se forma cuando la variante de la histona H2A. X se fosforila en la serina 139 en respuesta al daño en el ADN.
Al detectar la presencia de γH2A.X por ChIP nativo en los SFC, podemos evaluar eficazmente el alcance del daño en el ADN causado por los contaminantes ambientales42. La capacidad de medir cuantitativamente γH2A.X en los SFC proporciona una forma rápida, sensible y fiable de evaluar el potencial de los contaminantes ambientales para inducir efectos tumorigénicos. A diferencia de los ensayos de genotoxicidad tradicionales, que a menudo miden marcadores indirectos de daño en el ADN (como mutaciones, formación de micronúcleos o transformación celular), el ChIP nativo de γH2AX permite un mapeo preciso de eventos de rotura en loci genómicos específicos42. Esto proporciona una comprensión más profunda de la región más vulnerable a los contaminantes ambientales y ayuda a identificar posibles mecanismos cancerígenos.
El éxito de ChIP nativo requiere una atención cuidadosa a varios pasos críticos para garantizar el enriquecimiento efectivo de las regiones objetivo. A continuación se muestran los pasos críticos involucrados en el ChIP nativo de γH2AX. Preparación de células: Comience con un número adecuado de células. Tratar las células con un agente inductor de daño en el ADN, como la afidicolina, para inducir el estrés de replicación como un control positivo. Incluya muestras de control negativas apropiadas sin tratar para la comparación. Aislamiento de cromatina: Realice el fraccionamiento subcelular para aislar las proteínas unidas a la cromatina mientras se minimiza la contaminación de los componentes citoplasmáticos. Optimización de MNasa: Realizar experimentos piloto para optimizar la concentración de nucleasa microcócica (MNasa) y el tiempo de incubación. El objetivo es obtener fragmentos de cromatina de entre 200 y 1000 pares de bases, asegurando una buena resolución para ChIP y manteniendo la integridad de los nucleosomas. Inmunoprecipitación: Utilice un anticuerpo γH2AX de grado ChIP de alta calidad junto con la proteína A / G agarosa para garantizar una reducción eficiente de la cromatina objetivo al tiempo que minimiza la unión inespecífica. Lavado: Lave las perlas con un tampón que contenga la concentración adecuada de sal y detergente para eliminar las interacciones inespecíficas mientras conserva la cromatina unida a γH2AX. Elución y recuperación de ADN: Recupere de manera eficiente la cromatina unida mediante la elución y precipitación completas del ADN, asegurando una pérdida mínima de la cromatina objetivo enriquecida. En comparación con el X-ChIP convencional, hay cuatro ventajas principales del ChIP nativo γH2AX. En primer lugar, la integración del fraccionamiento subcelular reduce las interacciones de falsos positivos y mejora la especificidad de ChIP. En segundo lugar, los complejos proteína-ADN se conservan en su estado nativo, lo que minimiza la estabilización de las interacciones transitorias γH2AX-cromatina que pueden producirse con los agentes de reticulación. En tercer lugar, omitir la fijación y los lentos pasos de reticulación inversa agiliza el flujo de trabajo general. Por último, se requiere menos material de partida y no se necesitan equipos costosos para el cizallamiento de la cromatina, como un sonicador en baño de agua (por ejemplo, Bioruptor) o un sonicador ultrasónico enfocado (por ejemplo, Covaris).
Aunque ChIP nativo tiene sus ventajas, también hay que reconocer sus limitaciones. Una limitación es el potencial de reordenamiento de proteínas durante la preparación de la cromatina y la inmunoprecipitación. Este reordenamiento podría, en teoría, alterar las interacciones de las proteínas unidas a la cromatina e introducir variabilidad en los resultados. Aunque no tenemos evidencia directa que respalde la ocurrencia de tales reordenamientos, es importante incluir controles experimentales apropiados para dar cuenta de este posible artefacto. Otra limitación del ensayo ChIP nativo es el sesgo de escisión de la MNasa, que puede dar lugar a resultados inexactos. La MNasa escinde preferentemente las regiones ricas en A-T del genoma, lo que hace que estas regiones sean más propensas a fragmentarse y enriquecerse durante el proceso ChIP. Este sesgo puede causar una sobrerrepresentación de secuencias ricas en A-T y una infrarrepresentación de las regiones ricas en G-C, lo que puede dar lugar a señales falsas negativas para las regiones genómicas que son menos accesibles para la digestión de MNasa. Si los loci de interés son ricos en G-C, es necesaria una optimización cuidadosa de las condiciones de digestión de MNasa. Como alternativa, la incorporación de controles adicionales, como la fragmentación de la cromatina por sonicación, puede ayudar a mitigar el impacto del sesgo de la MNasa y mejorar la fiabilidad de los resultados.
A pesar de sus ciertas limitaciones, el ChIP nativo γH2AX es particularmente útil para identificar los loci genéticos donde contaminantes específicos inducen daño al ADN. Al enriquecer los fragmentos de ADN unidos a γH2AX, los investigadores pueden identificar regiones frágiles del genoma, en particular los SFC por qPCR, que son más propensos a romperse bajo el estrés de replicación inducido por los contaminantes. Esta técnica también permite a los investigadores establecer una relación directa entre la exposición a un carcinógeno específico y la formación de daños en el ADN en regiones genómicas particulares, proporcionando información sobre el perfil genotóxico de los contaminantes y su papel potencial en el inicio de la carcinogénesis43,44.
La tabla de solución de problemas (Tabla 3) proporciona soluciones detalladas para los problemas comunes encontrados durante el ensayo ChIP, como el bajo rendimiento del ADN, la fragmentación insuficiente de la cromatina, el alto ruido de fondo o el bajo rendimiento de los anticuerpos. Cada problema se combina con recomendaciones específicas para garantizar la confiabilidad y reproducibilidad del protocolo.
Mediante el uso de sistemas automatizados de manejo de líquidos y tecnologías cuantitativas de PCR o secuenciación, el ChIP nativo de γH2AX puede identificar de manera eficiente los agentes que inducen daños en el ADN, lo que sirve como una herramienta valiosa en estudios toxicológicos y monitoreo ambiental. Al emplear ChIP γH2AX nativo en muestras derivadas de poblaciones expuestas a diferentes niveles de contaminantes ambientales, los investigadores pueden identificar patrones específicos de daño en el ADN y determinar el nivel de riesgo asociado con la exposición a agentes particulares. En conclusión, el ChIP nativo γH2AX ofrece ventajas significativas para evaluar el potencial cancerígeno de los contaminantes ambientales, proporcionando un enfoque rápido, rentable y de alta especificidad para detectar daños en el ADN en loci genómicos específicos. Sus aplicaciones futuras en el cribado de alto rendimiento, la evaluación personalizada de riesgos y el desarrollo de biomarcadores lo convierten en una herramienta valiosa para avanzar en nuestra comprensión de la carcinogénesis ambiental y desarrollar estrategias para la prevención e intervención del cáncer.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Este trabajo contó con el apoyo de la financiación inicial de la Universidad del Sur de China.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.2 µm nitrocellulose membrane | Amersham | 10600011 | |
| Actin B | proteintech | 20536-1-AP | |
| Aphidicolin | MedChemExpress | HY-N6733 | |
| ChIP-grade magnetic Protein A/G beads | ThermoFisher | 26162 | |
| Clarity Western ECL Substrate | Bio-Rad | #1705061 | |
| Glycogen, molecular biology grade | ThermoFisher | Cat. No. R0561 | |
| HRP-conjugated secondary antibody | proteintech | SA00001-2 | |
| hydroxyurea | MedChemExpress | HY-B0313 | |
| Micrococcal Nuclease | NEB | M0247S | |
| normal IgG | Santa Cruz | sc-2025 | |
| Taq Universal SYBR Green Supermix | BioRad | 1725120 | |
| γH2A.X antibody (for ChIP) | Sigma-Aldrich | 05-636 | |
| γH2A.X antibody (for WB) | Cell Signaling | #25955 |
Referencias
- Glover, T. W., Berger, C., Coyle, J., Echo, B. DNA polymerase alpha inhibition by aphidicolin induces gaps and breaks at common fragile sites in human chromosomes. Hum Genet. 67 (2), 136-142 (1984).
- Bignell, G. R., et al. Signatures of mutation and selection in the cancer genome. Nature. 463 (7283), 893-898 (2010).
- Hellman, A., et al. A role for common fragile site induction in amplification of human oncogenes. Cancer Cell. 1 (1), 89-97 (2002).
- Kotzot, D., et al. Parental origin and mechanisms of formation of cytogenetically recognisable de novo direct and inverted duplications. J Med Genet. 37 (4), 281-286 (2000).
- Miller, C. T., et al. Genomic amplification of MET with boundaries within fragile site FRA7G and upregulation of MET pathways in esophageal adenocarcinoma. Oncogene. 25 (3), 409-418 (2006).
- Zack, T. I., et al. Pan-cancer patterns of somatic copy number alteration. Nat Genet. 45 (10), 1134-1140 (2013).
- Gao, G., et al. Common fragile sites (CFS) and extremely large CFS genes are targets for human papillomavirus integrations and chromosome rearrangements in oropharyngeal squamous cell carcinoma. Genes Chromosomes Cancer. 56 (1), 59-74 (2017).
- Thorland, E. C., Myers, S. L., Gostout, B. S., Smith, D. I. Common fragile sites are preferential targets for HPV16 integrations in cervical tumors. Oncogene. 22 (8), 1225-1377 (2003).
- Matovina, M., Sabol, I., Grubisić, G., Gasperov, N. M., Grce, M. Identification of human papillomavirus type 16 integration sites in high-grade precancerous cervical lesions. Gynecol Oncol. 113 (1), 120-127 (2009).
- Yu, T., et al. The role of viral integration in the development of cervical cancer. Cancer Genet Cytogenet. 158 (1), 27-34 (2005).
- Bignell, G. R., et al. Signatures of mutation and selection in the cancer genome. Nature. 463 (7283), 893-898 (2010).
- Thavathiru, E., Ludes-Meyers, J. H., MacLeod, M. C., Aldaz, C. M. Expression of common chromosomal fragile site genes, WWOX/FRA16D and FHIT/FRA3B is downregulated by exposure to environmental carcinogens, UV, and BPDE but not by IR. Mol Carcinog. 44 (3), 174-182 (2005).
- Sirbu, B. M., et al. Analysis of protein dynamics at active, stalled, and collapsed replication forks. Genes Dev. 25 (12), 1320-1327 (2011).
- Barlow, J. H., et al. Identification of early replicating fragile sites that contribute to genome instability. Cell. 152 (3), 620-632 (2013).
- Petermann, E., Orta, M. L., Issaeva, N., Schultz, N., Helleday, T. Hydroxyurea-stalled replication forks become progressively inactivated and require two different RAD51-mediated pathways for restart and repair. Mol Cell. 37 (4), 492-502 (2010).
- Rogakou, E. P., Pilch, D. R., Orr, A. H., Ivanova, V. S., Bonner, W. M. DNA double-stranded breaks induce histone H2AX phosphorylation on serine 139. J Biol Chem. 273 (10), 5858-5868 (1998).
- Shanbhag, N. M., Rafalska-Metcalf, I. U., Balane-Bolivar, C., Janicki, S. M., Greenberg, R. A. ATM-dependent chromatin changes silence transcription in cis to DNA double-strand breaks. Cell. 141 (6), 970-981 (2010).
- Stiff, T., et al. ATR-dependent phosphorylation and activation of ATM in response to UV treatment or replication fork stalling. EMBO J. 25 (24), 5775-5782 (2006).
- Nelson, J. D., Denisenko, O., Bomsztyk, K. Fast chromatin immunoprecipitation assay. Nuc Acids Res. 34 (5), e2 (2006).
- Dorbic, T., Wittig, B. Isolation of oligonucleosomes from active chromatin using HMG17-specific monoclonal antibodies. Nuc Acids Res. 14 (8), 3363-3376 (1986).
- Dorbic, T., Wittig, B. Chromatin from transcribed genes contains HMG17 only downstream from the starting point of transcription. EMBO J. 6 (8), 2393-2399 (1987).
- Hebbes, T. R., Thorne, A. W., Crane-Robinson, C. A direct link between core histone acetylation and transcriptionally active chromatin. EMBO J. 7 (5), 1395-1402 (1988).
- Hebbes, T. R., Thorne, A. W., Clayton, A. L., Crane-Robinson, C. Histone acetylation and globin gene switching. Nuc Acids Res. 20 (5), 1017-1022 (1992).
- Hebbes, T. R., Clayton, A. L., Thorne, A. W., Crane-Robinson, C. Core histone hyperacetylation co-maps with generalized DNase I sensitivity in the chicken beta-globin chromosomal domain. EMBO J. 13 (8), 1823-1830 (1994).
- Orlando, V. Mapping chromosomal proteins in vivo by formaldehyde-crosslinked-chromatin immunoprecipitation. Trend Biochem, Sci. 25 (3), 99-104 (2000).
- Myers, F. A., Evans, D. R., Clayton, A. L., Thorne, A. W., Crane-Robinson, C. Targeted and extended acetylation of histones H4 and H3 at active and inactive genes in chicken embryo erythrocytes. J Biol Chem. 276 (23), 20197-20205 (2001).
- Litt, M. D., Simpson, M., Recillas-Targa, F., Prioleau, M. N., Felsenfeld, G. Transitions in histone acetylation reveal boundaries of three separately regulated neighboring loci. EMBO J. 20 (9), 2224-2235 (2001).
- Madisen, L., Krumm, A., Hebbes, T. R., Groudine, M. The immunoglobulin heavy chain locus control region increases histone acetylation along linked c-myc genes. Mol Cell Biol. 18 (11), 6281-6292 (1998).
- Clayton, A. L., Hebbes, T. R., Thorne, A. W., Crane-Robinson, C. Histone acetylation and gene induction in human cells. FEBS Lett. 336 (1), 23-26 (1993).
- Tseng, Z., Wu, T., Liu, Y., Zhong, M., Xiao, A. Using native chromatin immunoprecipitation to interrogate histone variant protein deposition in embryonic stem cells. Methods Mol Biol. 1176, 11-22 (2014).
- Durkin, S. G., Glover, T. W. Chromosome fragile sites. Ann Rev Genetics. 41, 169-192 (2007).
- Lee, J. B., Keung, A. J. Chromatin immunoprecipitation in human and yeast cells. Methods Mol Biol. 1767, 257-269 (2018).
- Miyamoto, R., Yokoyama, A. Protocol for fractionation-assisted native ChIP (fanChIP) to capture protein-protein/DNA interactions on chromatin. STAR Protoc. 2 (2), 100404 (2021).
- Mendez, J., Stillman, B. Chromatin association of human origin recognition complex, cdc6, and minichromosome maintenance proteins during the cell cycle: Assembly of prereplication complexes in late mitosis. Mol Cell Biol. 20 (22), 8602-8612 (2000).
- Nowak, D. E., Tian, B., Brasier, A. R. Two-step cross-linking method for identification of NF-κB gene network by chromatin immunoprecipitation. BioTechniques. 39 (5), 715-725 (2005).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nat Protoc. 3 (6), 1101-1108 (2008).
- Lu, X., Parvathaneni, S., Hara, T., Lal, A., Sharma, S. Replication stress induces specific enrichment of RECQ1 at common fragile sites FRA3B and FRA16D. Mol Cancer. 12, 29 (2013).
- Ruijter, J. M., et al. Amplification efficiency: linking baseline and bias in the analysis of quantitative PCR data. Nucl Acids Res. 37 (6), e45 (2009).
- Lyu, X., Chastain, M., Chai, W. Genome-wide mapping and profiling of γH2AX binding hotspots in response to different replication stress inducers. BMC Genomics. 20, 579 (2019).
- Farmer, P. B., et al. Molecular epidemiology studies of carcinogenic environmental pollutants. Effects of polycyclic aromatic hydrocarbons (PAHs) in environmental pollution on exogenous and oxidative DNA damage. Mutat Res. 544 (2-3), 397-402 (2003).
- Phillips, D. H., Arlt, V. M. Genotoxicity: damage to DNA and its consequences. EXS. 99, 87-110 (2009).
- Nitsch, S., Schneider, R. Native ChIP: Studying the genome-wide distribution of histone modifications in cells and tissue. Meth Mol Biol. 2846, (2024).
- Nikitina, T., Wang, D., Gomberg, M., Grigoryev, S. A., Zhurkin, V. B. Combined micrococcal nuclease and exonuclease III digestion reveals precise positions of the nucleosome core/linker junctions: implications for high-resolution nucleosome mapping. J Mol Biol. 425 (11), 1946-1960 (2013).
- Teves, S. S., Henikoff, S. Salt fractionation of nucleosomes for genome-wide profiling. Methods Mol Biol. 833, 421-432 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados