Method Article
Bireysel Sıçan Nöral Hücrelerinin Lazer Kaynaklı Postiyonizasyon Kütle Spektrometresi ile Floresan Kılavuzluğunda Matris Destekli Lazer Desorpsiyon/İyonizasyon
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Bu protokol, floresan kılavuzlu, tek hücreli MALDI-2 kütle spektrometresi için mikroMS kullanımını ana hatlarıyla belirtir ve birincil sıçan nöronal hücrelerinin gelişmiş moleküler profilini çıkarmayı sağlar.
Özet
Tek hücreli ölçümler, beynin zengin uzay-kimyasal heterojenliğini anlamak için kritik öneme sahiptir. Matris destekli lazer/desorpsiyon iyonizasyon (MALDI) kütle spektrometresi (MS), tek tek hücrelerdeki endojen moleküllerin etiketsiz, yüksek verimli karakterizasyonunu yapabilir. Lazer kaynaklı post-iyonizasyon (MALDI-2) ile MALDI kütle spektrometrelerinin geliştirilmesindeki son gelişmeler, çeşitli lipitler ve diğer küçük moleküller için büyük ölçüde geliştirilmiş algılama hassasiyeti sağlar. Bununla birlikte, hücresel çözünürlükte MALDI-2 ile büyük örneklerin MS görüntülemesi çoğu uygulama için engelleyici bir şekilde yavaştır. Bu protokolde, birincil hücreler izole edilir ve iletken slaytlar üzerine dağıtılır. Bağıl hücre konumları, tam lam floresan mikroskobu ile belirlenir, ardından mikroskopi koordinatlarının MALDI-2 kütle spektrometresinin aşama koordinatlarına doğru bir şekilde birlikte kaydedilmesi sağlanır. Yalnızca hücre konumlarının hedefli MS analizi, tüm numunenin MS görüntülemesine kıyasla yüksek analit kapsamı ve azaltılmış veri boyutu ile yüksek verimli, tek hücreli ölçümler sağlar. Tek hücre hazırlama, tam slayt floresan görüntüleme, matris uygulaması ve MALDI-2 kütle spektrometresi için gerekli kritik adımları açıklıyoruz.
Giriş
Lipitler ve metabolitler, hücresel fonksiyon için temeldir ve zarların, enerji kaynaklarının ve sinyal moleküllerinin temel bileşenleri olarak hizmet eder 1,2. Bununla birlikte, bileşimleri ve bollukları, hücre tiplerindeki ve gelişimsel ve fonksiyonel durumlardaki farklılıkları yansıtarak, tek tek hücreler arasında önemli ölçüde değişebilir 3,4,5. Bu farklılıkları analiz etmek, biyolojik değişkenliği anlamak ve farklı hücre alt popülasyonlarını tanımlamak için çok önemlidir. RNA dizilimi gibi tek hücreli ölçüm teknikleri, hücreye özgü yararlı transkript profilleri sağlar6. Bununla birlikte, bu transkript düzeyindeki ölçümler, lipitlerin ve metabolitlerin gerçek hücresel miktarlarını doğrudan yansıtmaz, çünkü gen ekspresyonu her zaman bu analitlerin gerçek bolluğu ile ilişkili değildir. Bu nedenle, lipitlerin ve metabolitlerin doğrudan ölçümleri için özel yöntemler, tek hücrelerin ve popülasyonlarının kimyasal bileşiminin kapsamlı analizi için gereklidir.
Matris destekli lazer desorpsiyon/iyonizasyon (MALDI) kütle spektrometresi görüntüleme (MSI), endojen biyomoleküllerin yerindeetiketsiz uzamsal haritalaması için tercih edilen bir araçtır 7,8. Tipik olarak, MALDI ile, bir iyon ve nötr molekül tüyü oluşturan, organik bir matris ile birlikte kristalize edilmiş ince bir numune katmanından malzemeyi ablate etmek için bir UV lazeri kullanılır. Oluşan iyonlar daha sonra bir MS analizörü ile ayrılır ve bir kütle spektrometresi içinde tespit edilir. Modern kütle spektrometresi aşamalarının doğru konumlandırılması göz önüne alındığında, lazer, MSI tarafından moleküler görüntüler oluşturmak için belirli numune bölgelerini hedefleyecek şekilde konumlandırılabilir veya bölgeler arasında rasterleştirilebilir. Odaklanmış bir lazer ışını ve doğru aşama hareketi ile elde edilen hücresel veya hücre altı uzamsal çözünürlükte (< 10 μm) MSI, tek tek hücreler hakkında kimyasal bilgi elde etmek için kullanılabilir 9,10. Bununla birlikte, bu ölçekteki MSI ölçümleri, hücreler arasındaki boş bölgeleri görüntülemek için harcanan süre nedeniyle, özellikle düşük hedeflenen hücre yoğunluğuna sahip numuneler söz konusu olduğunda verimsizdir. Ayrıca, örneklenen küçük hacim nedeniyle birçok analitin saptanabilirliği sınırlıdır. Bu zorlukların üstesinden gelmek için, yüksek verimli tek hücreli analiz için görüntü kılavuzlu bir MALDI MS yaklaşımı geliştirdik11,12. Bu yaklaşımda, dağılmış hücrelerin konumları, tam slayt floresan görüntülerinden programlı olarak belirlenir ve kütle spektrometresi aşamasını, belirli parametrelere (örneğin, boyut ve şekil) sahip tek tek hücrelerin bulunduğu konumlara yönlendirmek için kullanılır ve daha sonra MALDI lazeri ile ışınlama yoluyla analiz edilir. Bu hedefli yaklaşımı kullanan önceki çalışmalar, heterojen hücre popülasyonlarında lipitleri, peptitleri ve diğer biyomolekülleri karakterize etmek için kullanılmıştır 5,13.
Tek hücrelerin kütle sınırlı doğası göz önüne alındığında, bu tür örneklerde tespit edilen analit sayısı genellikle doğrudan dokudan gözlemlenenden daha azdır. Bu nedenle, tek hücreli MS analizinde analit kapsamını artırmak için analit algılama hassasiyetinin arttırılması kritik öneme sahiptir. Bu algılama zorluğunun üstesinden gelmeye yardımcı olan yakın zamanda geliştirilen bir yaklaşım, geniş bir analit9,14,15,16 yelpazesi için hassasiyeti artırdığı gösterilen lazer post-iyonizasyonlu MALDI'dir (MALDI-2). Sonuç olarak, MALDI-2 daha kapsamlı tek hücreli veri kümeleri üretir ve izole hücreler gibi kütle sınırlı numunelerde daha derin moleküler kapsama alanı sağlar.
Bu yöntemin amacı, binlerce ayrı hücreden lipit ölçümleri elde etmektir. Bu amaçla, yüksek verimli tek hücreli MALDI-2 kütle spektrometrisini mümkün kılan ve genellikle hassas aşama kontrolü17 ile herhangi bir prob tabanlı kütle spektrometrisi yaklaşımına genişletilebilen bir iş akışını tanımlıyoruz. Bu iş akışında, ilgilenilen beyin bölge(ler)inden alınan doku parçalara ayrılır ve bir papain ayrıştırma prosedüründen sonra dokudan tek tek hücreler elde edilir. Hücreler daha sonra bir nükleer leke ile etiketlenir ve yapışmalarına izin verilen referans işaretleyicilerle kazınmış iletken cam slaytlar üzerine dağıtılır. Daha sonra, floresan mikroskobu kullanılarak tüm slayt görüntüleri alınır. Matris, tek hücreli MS analizi için tekrarlanabilir homojen bir kristal tabaka ve yüksek sinyal-gürültü üreterek süblimasyon yoluyla biriktirilir. Açık kaynaklı yazılım microMS11 kullanılarak, mikroskopi görüntüsünden hücre konumlarının göreceli koordinatları, hücrelerin biriktirildiği cam slaytlar üzerine kazınmış referans işaretleyiciler kullanılarak bir nokta seti kaydı ile kütle spektrometresi aşama koordinatları ile eşlenir ve hizalanır. Son olarak, bu bilgiler kullanılarak, her bir hücreden kesin, hedeflenmiş MS spektrumları elde edilir ve binlerce hücrenin tek bir çalışmada (<1 saat) profilinin çıkarılmasına olanak tanır12,13.
Protokol
Bu çalışmadaki tüm hayvan deneyleri, Illinois Kurumsal Hayvan Bakımı ve Kullanımı Komitesi (23228) tarafından onaylanan hayvan kullanım protokolüne uygun olarak, hayvanların etik muamelesi ve bakımı için hem ulusal hem de ARRIVE standartlarına sıkı sıkıya bağlı kalınarak yapılmıştır.
1. Malzemelerin ve çözeltilerin hazırlanması
- 1.5 mM CaCl2, 4.9 mM KCl, 0.2 mM KH2PO4, 11 mM MgCl2, 0.3 mM MgSO4, 138 mM NaCl, 27.7 mM NaHCO3, 0.8 mM Na2HPO4 ve 25 mM HEPES ile modifiye edilmiş Gey'in dengeli tuz çözeltisini (mGBSS) hazırlayın. 3 M NaOH kullanarak pH'ı 7,2'ye ayarlayın. 1 L mGBSS hazırlanması ve kullanılana kadar 4 °C'de saklanması önerilir.
- 50 mL'lik şırıngaları hazırlanan mGBSS solüsyonu ile doldurarak hazırlayın ve transkardiyal perfüzyon için buzun üzerine yerleştirin. Diseke edilen her sıçan için 1 şırınga hazırlayın.
- Worthington Papain Ayrışma Sistemi protokolünü takip ederek papain çözeltisinin alikotlarını hazırlayın. Bu sistem, aşağıda açıklandığı gibi dört şişe içerir ve bu protokol için şişe 4 kullanılmaz:
Flakon 1: bikarbonat ve fenol kırmızısı içeren steril Earle dengeli tuz çözeltisi (EBSS)
Flakon 2: L-sistein ve EDTA'lı papain
Flakon 3: deoksiribonükleaz I (DNaz)
Flakon 4: sığır serum albümini ile ovomukoid proteaz inhibitörü- Papain şişesine (şişe 2) 5 mL EBSS (şişe 1) ekleyin ve papain'i çözmek için 10 dakika boyunca 37 ° C'lik bir su banyosuna koyun.
- DNase şişesine (şişe 3) 500 μL EBSS (şişe 1) ekleyin ve hafifçe karıştırın. 250 μL seyreltilmiş DNaz şişesini (şişe 3) papain şişesine (şişe 2) ekleyin. Bu preparat, 20 birim / mL papain,% 0.005 DNaz, 0.5 mM EDTA ve 1 mM L-sistein nihai konsantrasyonunu içerir.
- Sonraki ayrışmalar için bu hazırlanmış papain çözeltisinin 250 μL'lik alikotunu dondurun (~ 20 alikot yapar). Bu çözeltiyi aşağıdaki antibiyotiklerle destekleyin: 100 birim / mL penisilin G, 100 μg / mL streptomisin ve 100 μg / mL gentamisin. Bu, kullanıcının uygulaması için değiştirilebilir.
- 150 mM'lik bir amonyum asetat çözeltisi hazırlayın ve 4 °C'de saklayın. Gliserolü hazırlanan mGBSS çözeltisine seyrelterek% 33'lük bir gliserol çözeltisi hazırlayın.
- Tungsten karbür veya elmas uçlu bir gravür kalemi veya bir CO2 lazer kazıma makinesi kullanarak indiyum kalay oksit (ITO) kaplı mikroskopi slaytlarına referanslar aşındırın. ITO cam slaytın iletken tarafında, platformlar arası (örn. mikroskop ve kütle spektrometresi) uzamsal kaydı sağlamak için lamın çevresi boyunca 20-40 X şeklinde referans işaretleyicileri aşındırın.
NOT: Numuneyi veya alanlarını çevreleyen bağlı bir çizgiyi aşındırmamaya dikkat edin, çünkü bu, alan ile bir cam kaydırma adaptörü arasındaki yüzey iletkenliğini bozar.
2. Birincil nöral hücrelerin hazırlanması
NOT: Sıçan hipokampal dokusu diseke edilir, papain ile tek tek hücrelere ayrıştırılır ve düşük yoğunlukta iletken cam slaytlar üzerine biriktirilir. Hücrelerin bu şekilde izole edilmesi, endojen lipidlerin yüksek verimli tek hücreli kütle spektrometresini mümkün kılar.
- Hayvan diseksiyonundan önce, hazırlanan papain çözeltisinin bir kısmını çözün ve CO2'nin kabın içindeki havayı değiştirmesine izin veren bir giriş ve çıkış borusu olan bir kaba yerleştirin. Bu sistemi 37 °C'ye ayarlanmış bir su banyosuna yerleştirin ve Worthington Papain Ayrışma Sistemi paketinde bulunan pH renk tablosunu kullanarak çözelti renkle belirtildiği gibi uygun pH olana kadar CO2 akışına devam edin. Hedef pH 7,2 ile 7,4 arasındadır.
NOT:CO2'yi enzimatik çözeltiye kabarcık dökmeyin, çünkü bu DNazın denatürasyonuna ve biyolojik aktivite kaybına neden olur. Kapağa yapıştırılmış iki tüplü 50 mL'lik bir santrifüj tüpü kullanın, bu da CO2'nin tüpü alttan doldurmasına ve üst çıkış tüpünden kaçmasına izin verir. - Sıçan(lar)ı kurumsal, yerel ve federal yönergeleri izleyerek IACUC protokollerine uygun olarak ötenazi yapın. Burada, 2-3 aylık erkek Sprague-Dawley sıçanları kullanılır ve CO2 boğulması ile sakrifiye edilir.
- Vasküler sistemden kanı çıkarmak ve hayvanın vücudunu hızlı bir şekilde soğutmak için sıçanı buz gibi soğuk (4 ° C) mGBSS solüsyonu ile transkardiyal olarak perfüze edin. Bu prosedürün bir örneği, We ve ark.18 tarafından hazırlanan protokolde bulunabilir.
- Beyni fareden cerrahi olarak izole edin ve ilgilenilen beyin bölgesine zarar vermemeye dikkat edin. Bu yalıtımın daha önce yayınlanmış bir genel bakışı, tek tek adımlardan bazıları hakkında daha fazla ayrıntı sağlar19.
- Bölgeyi (hipokampus, korteks veya striatum) örneklemeye maruz bırakmak için beynin bir kesitini (~ 2 mm kalınlığında) kesmek için bir doku doğrayıcı veya doku küpü kullanın.
- İlgilenilen beyin bölgesini örneklemek için 2 mm'lik bir biyopsi yumruğu kullanın ve doku parçasını papain solüsyonu şişesine bırakın.
- Bu dokuyu ve papain solüsyonunu 37 ° C'de sürekli çalkalama altında (örneğin, bir elektrikli külbütör ile) 30-90 dakika inkübe edin.
NOT: Enzimatik tedavi süresi, beyin bölgesine, hayvan suşuna, yaşa ve istenen hücre çıktısına göre optimize edilmelidir (örneğin, terminalleri olan nöronlar ve astrositlere karşı terminalleri olmayan hücrelerin maksimum verimi). Sıçan hipokampal dokusu için 60-90 dakika tipiktir. Daha kısa tedaviler bazı terminalleri koruyabilir, ancak birçok hücreyi ayrışmamış kümelerde bırakabilirken, daha uzun tedaviler verimi artırabilir ancak hücre hasarını artırabilir, bu da canlı / ölü tahliller (örneğin, CANLI / ÖLÜ canlılık / sitotoksisite kiti) kullanılarak değerlendirme gerektirir. Karşılaştırmalı çalışmalar için, tedaviler numune türleri arasında tutarlı olmalıdır. - Enzimatik aktiviteyi soğutmak ve bastırmak için bir şişe doku ve papain buzun üzerine yerleştirin. Dokunun çoğu görsel olarak parçalanana kadar P100 ayarlanabilir hacimli bir pipete bağlı kesilmiş bir pipet ucu kullanarak dokuyu ezin. Aşırı hücre hasarını önlemek için ayrışma sırasında hava kabarcıkları oluşturmayın.
NOT: Güçlü öğütme, yüksek hücre verimi ile sonuçlanır; Ancak, süreçlerinin çoğunu elinde tutmazlar. Nazik tritürasyon, hücreler üzerinde daha fazla işlem sağlar, ancak daha fazla ayrışmamış hücre ile sonuçlanır. Farklı boyutlarda pipet uçları ve diğer yöntemler de kullanılabilir ve ideal ayrışma sonuçları için optimize edilebilir20. - Hücreleri peletlemek için ayrışmış hücreleri oda sıcaklığında 5 dakika boyunca 300 x g'da santrifüjleyin. Süpernatanı çıkarın ve peleti 1 mL mGBSS'de yeniden süspanse edin. Hoechst 33342 nükleer boyasını 1 μg/mL'lik bir nihai konsantrasyona ekleyin.
- Hücre süspansiyonunun 100-500 μL'sini kazınmış ITO cam slaytlara pipetleyin ve hücrelerin çökelmesini ve slayta yapışmasını sağlamak için yaklaşık 5 dakika veya daha uzun süre bekletin.
- Hücre süspansiyonunun seyreltilmesinin ampirik olarak belirlenmesi gerekecektir. Optik bir mikroskop kullanarak, kaba yoğunluğu belirlemek için slaydın üzerine bir hücre damlası ekleyin ve hücreler arasında yeterli mesafe olduğundan emin olmak için oradan seyreltin (yani, MALDI ışın boyutundan yeterince büyük). Bu seyreltme ilk slaytta gerçekleştirilebilir ve daha sonra seyreltme, sonraki slaytlar için gerektiği gibi ayarlanabilir.
- Aşağıdakileri içeren bir vakum sistemi kullanarak (vakum hattından başlayarak sırayla bulunur) hücre süspansiyonunu sürgüden nazikçe aspire edin: 1) Sıralı gaz HEPA filtreli boru, 2) 50 mL'lik bir filtreleme şişesine takılan boru, 3) Bağlı bir pipet ucu (örn. 100 μL) ile ikinci bir filtreleme şişesi girişine bağlı boru. Kaplanmış hücreleri ileride kullanmak üzere saklıyorsanız, aspire edilmiş süspansiyonu 1-2 dakika boyunca %33 gliserol solüsyonu ile değiştirin ve çıkarın.
NOT: Bu adım hücre stabilitesini sağlayacaktır. Gliserol çözeltisini bir taraftan nazikçe aspire ederken diğer taraftan dağıtmak, hücresel yapıyı korumak için en uygunudur. İlk numunenin hazırlanması sırasında ters çevrilmiş bir mikroskop kullanarak burada açıklanan tüm adımları daima görsel olarak izleyin. - Biriken bölgelere 100-500 μL amonyum asetat uygulayın, yaklaşık 1 dakika bekletin, ardından slaytları temizlemek için aspire edin. Slaytların herhangi bir tuz veya gliserol içermediğinden emin olmak için gerektiği kadar tekrarlayın.
3. Mikroskopi
NOT: Biriken hücrelerin konumunu belirlemek için, her slayt parlak alan/floresan mikroskobu ile görüntülenir. Floresan kanalı, Hoechst lekeli hücrelerin doğru bir şekilde konumlandırılmasına izin verirken, parlak alan görüntüleme morfolojik bilgi sağlar. Döşenmiş görüntü elde edebilen herhangi bir mikroskop bu işlem için uygundur.
- Daha önce yapılmadıysa, slaytları 2-3 mL 150 mM amonyum asetat ile durulayın. Bu adım, mikroskopi gözlemine ve MALDI matris uygulamasına müdahale edebilecek gliserol ve tuz kristallerini çıkarmak için kritik öneme sahiptir. Slaytları hafif bir N2 akışı altında kurutun veya kurumaya bırakın.
- Slaytı mikroskop aşamasına yükleyin. Slayt boyunca eşit olarak dağıtılmış en az 10 destek noktası kullanarak mikroskobu odaklayın.
- DAPI (uyarma: 335-383 nm, emisyon: 420-470 nm) ve parlak alan görüntüleme için uygun filtreler kullanarak, tüm slayt üzerinde 5x-10x büyütmede döşenmiş bir floresan görüntüsü elde edin ve referans işaretleyicilerin (adım 1.5'te oluşturulan) yakalandığından emin olun.
NOT: Özellikle çok sayıda slaytın görüntülenmesi gerektiğinde, genellikle 5x büyütme yeterlidir. Daha ince hücresel morfolojiyi çözmek için, 10x veya 20x objektifler, ikinci dereceden daha uzun görüntüleme süresi ve dosya boyutlarının artması pahasına kullanılabilir. - Döşenmiş görüntüyü ZEN (Zeiss) gibi bir mikroskopi yazılımı kullanarak birleştirin. Bu adımdaki herhangi bir hata son kayıt adımına yayılarak yanlış hedeflemeye neden olacağından, doğru birleştirme çok önemlidir. Kontrol etmek için, dikey olarak bitişik döşemelerin kaydırılmadığından emin olun. Ek olarak, büyük bir döşeme çakışması (%20-%30) olan döşenmiş görüntüler almayı düşünün.
- Her görüntüyü mikroskopi yazılımında bir bigTIFF dosyası olarak işleyin ve dışa aktarın. Görüntülenen bölge nispeten küçükse ve son görüntü boyutu 2 GB'tan küçükse, bunun yerine standart bir TIFF dosya formatı kullanın.
4. Matris uygulaması
NOT: Tutarlı ve uygun MALDI matris uygulaması, kaliteli tek hücreli veriler elde etmek için kritik öneme sahiptir. Burada ticari bir aparat kullanılarak süblimasyon kullanılırken, matris uygulaması robotik bir püskürtücü12, airbrush21 veya ev yapımı süblimasyon aparatı22 kullanılarak da gerçekleştirilebilir. Tek hücreli preparatların, tipik olarak kütle spektrometresi görüntülemesi için kullanılan ince doku kesitlerinden daha az matris gerektirdiğini bulduk. Toplu etkileri azaltmak için, matrisin bir seans sırasında incelenen tüm slaytlara uygulanması ve mümkün olduğunda farklı gruplardan (örneğin, beyin bölgesi veya tedaviye karşı kontrol) hücrelerin aynı slayta bırakılması önerilir. Matris seçimi, hem geleneksel MALDI hem de MALDI-2 tek hücreli iş akışları için çok önemlidir. Tek hücreli MALDI için DHB23, 9-AA24 ve CHCA25 başarıyla kullanılmıştır. MALDI-2'de, biz ve diğerleri DHAP9 ile önemli sinyal artışı gözlemlerken, NEDC16 ve CHCA26 gibi matrisler de etkili bir şekilde uygulanmıştır.
- 20 mg 2,5-dihidroksiasetofenon (DHAP) 1.5 mL aseton içinde çözülür. Slaytları süblimasyon odasının tutucusuna yerleştirin ve süblimasyon aparatına yerleştirin.
- Çözünmüş matris çözeltisini seramik gofret üzerine pipetleyin ve asetonun buharlaşmasına izin verin. Mühürlemek için süblimasyon odasını kapatın.
- Soğutma sıvısı haznesini bir buzlu su ile doldurun ve haznenin üzerine yerleştirin.
- Vakumu açın ve sistemin 5 dakika dengelenmesine izin verin. Sistemdeki basınç 40 mbar'dan az olmalıdır.
- Hazneyi 5 dakika boyunca 200 °C'ye ısıtarak süblimleşmeye başlayın.
- Buzlu su banyosunu çıkarın, sıcaklığı 25 °C'ye getirin ve haznenin üzerine bir soğutucu yerleştirin. Yoğuşmayı önlemek için sistemin 5 dakika oda sıcaklığına ısınmasına izin verin.
NOT: Buzlu su banyosunu tutan kap, ısı emici olarak kullanılmak üzere boşaltılabilir ve sıcak su ile doldurulabilir. - Sistemi yavaşça havalandırın, hazneyi açın ve kızakları çıkarın.
5. Tek Hücreli MALDI MS
NOT: Tek hücreli MS verileri, hücreleri tespit etmek ve kütle spektrometresini yönlendirmek için açık kaynaklı microMS paketi kullanılarak bir MALDI-2 timsTOF cihazında (timsTOF flex) elde edilir. Bu, hedeflenen hücrelerin optik görüntü piksel konumlarının kütle spektrometresi aşamasının fiziksel koordinatlarına çevrilmesini gerektirir.
- MicroMS'yi açın ve hem parlak alan hem de floresan kanalları olduğundan, Görüntü Grubu seçeneğini kullanarak bigTIFF mikroskopi görüntülerini azaltın.
- Blob Seçenekleri aracını kullanarak, hedeflenen hücreler için seçilecek en uygun parametreleri belirleyin. Aşağıdaki parametreleri ayarlayın: ne kadar küçük veya büyük blobların bulunacağını seçmek için maksimum ve minimum blob boyutu; blob algılama için floresan kanalının eşiğinin nasıl alınacağını belirleyen eşik ayarı; ve tanımlanan hücrelerin dikkate alınması için ne kadar dairesel olması gerektiğini belirleyen dairesellik. Burada sunulan veriler için aşağıdaki parametre değerlerini kullandık: maksimum dairesellik: yok; min dairesellik: 0.6; eşik: 75; minimum boyut: 20; Maksimum boyut: Yok.
- Blobları bulmak için Blob bulma seçeneğini ve blob listesini istenen bir adla kaydetmek için açılır pencereyi kullanın.
- Her hücrenin birbirinden uzak olması gereken belirli bir mesafeyi seçmek için Mesafe filtresi aracını kullanın (bu veriler için 5x floresan mikroskobu kullanarak 35 piksellik bir mesafe filtresi kullandık). Bu, hücrelerin tek hücre analizi yapmak için birbirinden yeterince uzakta olmasını sağlamak içindir. Bu sayı piksel birimleri cinsindendir, bu nedenle ideal sayıyı belirlemek için mikroskobun pikselden μm'ye dönüşümünü bilin. Kayıttaki hataları hesaba katmak için lazer probu çapının boyutunu artı yaklaşık 20-30 μm kullanın.
- MTP Slayt Adaptörü II'yi kullanarak slaytları cihaza yükleyin. microMS ile bilgisayara dönün ve bir uzak masaüstü uygulaması kullanarak kütle spektrometresi bilgisayarına erişin.
- Cihaz ilk kez kullanılıyorsa, cihazın XY konumunu doğrulayın. Bunu yapmak için Araçlar ve Enstrüman Ayarları'na gidin. Açılır pencere, X ve Y konumlarıyla birlikte bir dizi koordinat görüntüler. Cihazdaki Slide Adapter II geometrisinde Bu Belirli Noktaların Her birini seçin ve microMS penceresindeki X ve Y konumlarını güncelleyin.
- Kütle spektrometresinin kamera ve sahne kontrollerini kullanarak, slayt üzerinde kolayca tanımlanabilir bir konuma gidin ve cihaz koordinatlarını kopyalayın. MicroMS'de, mikroskop görüntüsünde ilgili konumu bulun, bu konuma sağ tıklayın ve koordinatları açılır pencere metin konumuna yazın. Tüm koordinatları en yakın tamsayıya yuvarlayın ve X ve Y koordinatlarını boşlukla ayırın (örneğin, 39595 -23232).
- Adım 5.7'yi tekrarlayın. tüm slayt boyunca en az 12 referans işaretçisi kaydetmek için. Üç kayıt noktası eklendikten sonra, dairelerden biri kırmızıya dönecek ve kayıttan en kapalı konum olduğunu gösterecektir. Böyle bir durumda, shift + sağ tıklama tuşlarını kullanarak geçerli kayıt noktasını silin ve yeniden deneyin.
- Dosya'nın altında, kayıt (.msreg) dosyasını kaydetmek için Kaydet'e ve ardından Kayıt'a gidin.
- Slaytta bloblar görüntülendiğinde, Dosya'ya, ardından Kaydet'e ve ardından Araç Konumları'na giderek araç konumu (.xeo) dosyasını kaydedin. Uzak masaüstü yazılımını kullanarak bu .xeo dosyasını cihaz bilgisayarına aktarın.
- Oluşturulan özel .xeo dosyasındaki metni, cihazdaki MTP Slide Adapter II .xeo dosyasına kopyalayın ve kaydedin. Bu, bu geometri dosyasını hücrelerin konumlarıyla güncelleyecektir.
- Otomatik çalıştırmayı başlatmak için Otomasyon sekmesine tıklayın ve Yeni... 'yi seçin. Hücreleri seçmek için görüntülenen örnek bölge boyunca sürükleyin ve bunları analiz listesine eklemek için sağ tıklayın. Otomasyon çalıştırmasını kaydedin ve alımı başlatmak için Otomatik Çalıştırmayı Başlat'a tıklayın. Çalışmanın uzunluğu, hücre sayısı, lazer atışları ve lazer frekansı dahil olmak üzere çeşitli faktörlere bağlıdır, ancak genel olarak, kabaca 5-10 hücre MALDI modunda (10 kHz lazer tekrarlama oranı) analiz edilebilir ve 1-5 hücre MALDI-2 modunda (1k Hz lazer tekrarlama oranı) her saniye analiz edilebilir.
6. Veri işleme
NOT: Mevcut ticari yazılım paketleri, yüksek verimli tek hücreli kütle spektrometresi verilerini analiz etmek için pek uygun değildir. Bireysel spektrumlar görselleştirilebilirken, anlamlı biyolojik içgörüler elde etmek özel araçlar gerektirir. Bunu ele almak için, tek hücreli MALDI-2 MS veri analizini kolaylaştıran ücretsiz olarak kullanılabilen bir yazılım sunuyoruz. Güncellenen iş akışımız, tek hücreli verilerin açık kaynaklı imzML format27'ye doğrudan dönüştürülmesini kolaylaştırarak SCiLS, MVS ve diğer satıcı yazılımlarıyla uyumluluk sağlar. Daha gelişmiş veri analizi için, tam komut dosyası ayrıca Matplotlib (sürüm 3.7.3) tarafından etkinleştirilen lipid açıklama, kümeleme ve diğer görselleştirme araçları için işlevsellik içerir. Ham verilerin ayrıştırılması ve okunması, TIMSCONVERT iş akışında28 yer alan bir dizi işlev olan pyTDFSDK kitaplığı tarafından kolaylaştırılır.
- Ek Dosya 1'de sağlanan dosyayı Jupyter Notebook ile aynı çalışma yoluna kopyalayarak özel Python kitaplığı SC_MALDI2_Analysis başlatın ve ardından ilk kod hücresinde klavyede Shift + Enter tuşlarına basın. Bu, bu işlevleri daha sonra çağırmamıza ve verileri işlemek için kullanmamıza olanak tanır.
- Toplanan verilerin yolunu kopyalayın (Bruker .d dosyaları). Her dosyanın klasörlerini ve adlarını, her örnek için meta veriler oluşturmak üzere bu dosya yollarını daha sonra ayrıştırabilmemiz için açıklayıcı bir şekilde adlandırmanızı öneririz. Bruker .d Dosyalarını İçe Aktarma'yı çalıştırın ve bir liste kodu hücresine kaydedin. Bu, yüklenen toplam .d dosyası sayısını verecektir.
- Ham verileri işleme ve imzML'ye kaydetme başlıklı kod hücresini çalıştırın. Bu, sağlanan her Bruker .d dosyasını okuyacak, veriler üzerinde tepe toplama gerçekleştirecek ve ardından her tepe noktası etrafında 20 ppm veriyi .d dosyasının içine yazılmış bir .imzML dosyasına kaydedecektir.
- Kaydedilen verileri bulmak için ilk .d dosyasının konumunu açın ve imzML ve diğer meta veriler bu yola kaydedilecektir. Bu aşamada, kaydedilen imzML dosyası, SCiLS Lab MVS gibi satıcı yazılımlarına aktarılabilir. Daha gelişmiş veri analizi için komut dosyasını takip etmeye devam edin.
- Önceden kaydedilmiş imzML dosya kodu hücresinde Yükleme'yi çalıştırarak kaydedilen imzML verilerini yükleyin. Bu, kaydedilen verileri değişken verilere yükler.
- Tüm verileri ortak bir m/z ekseni boyunca gruplamak, düşük bollukta bulunan özellikleri kaldırmak (%<0,1) ve gürültüden daha düşük değerleri (100 göreli yoğunluk) kaldırmak için Verileri gruplama ve veri matrisi hücresini kaydetme hücresini çalıştırın. Bu işlev daha sonra her bölmedeki her tepe m/z değerinin ortalamasını alarak verileri yeniden birleştirir. Elde edilen karekod datacube_array olarak kaydedilir, matrisle ilişkili m/z bölmeleri valid_mz_bins olarak kaydedilir ve her bölmenin sayısı sayı olarak kaydedilir.
- Verilere açıklama eklemek için, LIPID MAPS ek açıklama hücresi için Dışa aktarma tepe noktalarını çalıştırın. Bu, veri matrisinizdeki tüm m/z tepe noktalarını panonuza kopyalayacaktır. Bunu boş bir metin dosyasına yapıştırın ve LIPID MAPS'e yüklemek için kaydedin.
- lipidmaps.org'yi açın, ardından biyolojik olarak ilgili lipid türlerini aramak için MS Data Bulk Search ve Search LMSD'yi seçin. Metin dosyasını seçerek tepe listesini yükleyin ve uygun eklentileri seçin (örneğin, [M+H]+, [M+Na]+ ve [M+K]+). Kütle spektrometreniz için uygun kütle toleransını seçin (biz +/- 0,01 m/z'yi seçtik), ilgilendiğiniz tüm lipid sınıflarını seçin ve Ara'ya basın. .tsv dosyasını indirin.
- Meta veri hücreleri için tek hücre yollarında yükleme komutunu çalıştırarak spektrum meta verilerini yükleyin. Buradaki bölünmüş kodu, dosya yolu yapılarına göre değiştirin.
- Veri Önişleme ve Lipid Açıklama hücresini çalıştırarak verileri önceden işleyin. Bu, veri kümesine 10 ppm hatasına açıklama eklemek, lipidMAPS sonuçlarından tek zincirli lipidleri ortadan kaldırmak, kalan verileri normalleştirmek ve belirli bir eşiği geçmeyen özellikleri veya hücreleri kaldırmak için lipidMAPS tsv dosyasını kullanır. Bu, yeni işlenen veri kümesini veri2 olarak ve güncelleştirilmiş spektrum meta verilerini updated_cell_types olarak kaydeder.
- Verilerin analizi scanpy tarafından gerçekleştirilir. İlgilenilen verileri seçmek için Tek Hücreler hücresi için UMAP ve Kümeleme Analizini çalıştırın (burada yalnızca MALDI-2, M2, verileri seçtik), veri kümesini ölçeklendirin ve kolay analizler için yetersiz bir AnnData nesnesine yükleyin.
NOT: Burada, veri seti üzerinde Leiden kümeleme ve boyutsallık azaltma (UMAP) işlemi gerçekleştirilmiş, hem orijinal veri sınıfları hem de UMAP üzerindeki kümeler görselleştirilmiş ve kümeler arasındaki istatistiksel olarak en anlamlı özelliklerden bazıları scanpy'den rank_genes_groups_dotplot fonksiyonu kullanılarak özetlenmiştir.
Sonuçlar
Floresan kılavuzluğunda tek hücreli MALDI-2 MS için iş akışına genel bir bakış Şekil 1'de gösterilmektedir. İlk olarak, hedeflenen beyin bölgelerinden (Şekil 1A) diseke edilen doku, tek hücrelere ayrılır ve iletken ITO kaplı mikroskopi slaytları üzerine bırakılır (Şekil 1B). Hücrelerin konumları tam slayt floresan görüntüleme (Şekil 1C), ardından MALDI matris uygulaması (Şekil 1D), microMS destekli MALDI-2 MS analizi (Şekil 1E) ve veri analizi (Şekil 1F) ile belirlenir. Bu iş akışını kullanarak, 10 ppm'den daha az bir kütle doğruluğu ile (bir MALDI-2 timsTOF kullanılarak) tek tek hücreler içinde onlarca ila yüzlerce lipit varsayılan olarak tanımlanabilir.
Kütle spektrometresi görüntülemesi, hücre hazırlığının ve matris uygulamasının kalitesini değerlendirmek için küçük bir ilgi alanı üzerinde gerçekleştirilebilir. Dağılmış hücrelerin MALDI-2 görüntülemesinin temsili bir sonucu Şekil 2A'da gösterilmiştir. Bu görüntüden, kullanıcı aynı bölgenin bir fotomikrografı ile karşılaştırarak analit yayılma derecesini değerlendirebilir. Yayılma miktarına bağlı olarak, analist, gerçek tek hücre kazanımlarını sağlamak için lazer ışını alanı boyutunun boyutunu ve mesafe filtresini ayarlayabilir. Tek hücreli MSI'da, hücresel çözünürlük (1-5 μm) elde etmek için küçük bir lazer boyutu ve raster genişliği kullanılırken, microMS için daha büyük bir lazer boyutu kullanılabilir, bu da görüntülemeye göre sinyal yoğunluğunu ve tespit edilebilir lipitlerin sayısını artırır. MALDI-2 ile elde edilen sinyal geliştirmenin temsili sonuçları Şekil 2B'de gösterilmektedir. Kolesterol, PE ve PC türlerinin artması, önceki literatürle uyumlu olarak gözlenmiştir14. Genel olarak, nispeten yüksek bir lazer gücü (%>50) ve daha az sayıda çekim (yaklaşık 10 ila 200) kullanıldığında en iyi MALDI-2 sinyal geliştirmesini elde ederiz. Çok düşük lazer gücü geliştirme sağlamayacağı ve çok fazla lazer enerjisi aşırı analit parçalanmasına neden olacağı için bir miktar optimizasyon gereklidir29. Tek hücreli analiz için uzamsal kayıt, tek tek hücrelerin doğru şekilde hedeflenmesini sağlamak için kritik bir adımdır. Kaydı doğrulamak için, bilinen noktalarda bloblar oluşturarak ve cihaz koordinatlarını microMS aracılığıyla dışa aktararak slayt etrafında küçük bir test noktaları kümesi (~ 5) oluşturulabilir. Bu test noktalarının seçildiğinde cihaz aşamasını doğru şekilde konumlandırdığının doğrulanması, sonraki hücre hedeflemesinin doğru olmasını sağlar.
MALDI MS, bireysel beyin bölgeleri arasında ve içinde hücresel heterojenliğin profilini çıkarmayı sağlar (Şekil 3). Boyutsallık azaltma teknikleri, lipid profillerindeki varyasyonları analiz ederek, hücreleri etkili bir şekilde farklı kümelere ayırır ve veri setindeki lipid bileşimlerinin çeşitliliğini vurgular (Şekil 3A). Ayrıca, Leiden kümeleme gibi kümeleme tekniklerinin uygulanması, striatum ve korteks dahil olmak üzere belirli beyin bölgelerinde benzersiz hücre alt popülasyonlarının varlığını gösterir (Şekil 3B). Ek olarak, her bir kümenin lipid profillerini birbiriyle karşılaştırarak (Şekil 3C), belirli lipidler, kümeye özgü kimlikleri tanımlamak için işaretleyici olarak kullanılabilir. Bazı lipit türleri, belirli kümelerde önemli ölçüde yukarı veya aşağı regüle edilir, bu da farklı fonksiyonel roller veya durumlar önerirken, diğerleri kümeler arasında paylaşılır, ancak göreceli sinyal yoğunlukları değişir. Sağlam aşağı akış veri analizi sağlamak için, hücrelerin çoğunda güvenilir lipid tespiti elde etmek ve potansiyel parti etkilerini değerlendirmek çok önemlidir. Şekil 3D , ayrı ITO kaplı slaytlar boyunca dağılmış altı ayrı kortikal hücreden temsili spektrumları sunar. Bireysel lipid profilleri heterojenlik sergilerken, lipitler tüm edinimlerde tutarlı bir şekilde tespit edilir. Şekil 3E , aynı koşullar altında hazırlanan ve analiz edilen farklı slaytlardan kortikal hücrelerin bir UMAP'sini göstermektedir. Slaytlar arasındaki hücrelerin önemli ölçüde örtüşmesi, toplu etkilerin biyolojik yorumları önemli ölçüde karıştırmadığını gösterir. Gerekirse, kalıntı etkilerini azaltmak için ComBat30 gibi toplu düzeltme algoritmaları uygulanabilir. Özetle, tek hücreli MALDI-2, lipid biyobelirteç tanımlaması için ilerleyen, hücresel alt popülasyonlar hakkında kritik bilgiler sunan ve beyindeki bölgesel ve fonksiyonel çeşitlilik anlayışımızı geliştiren bir tekniktir.
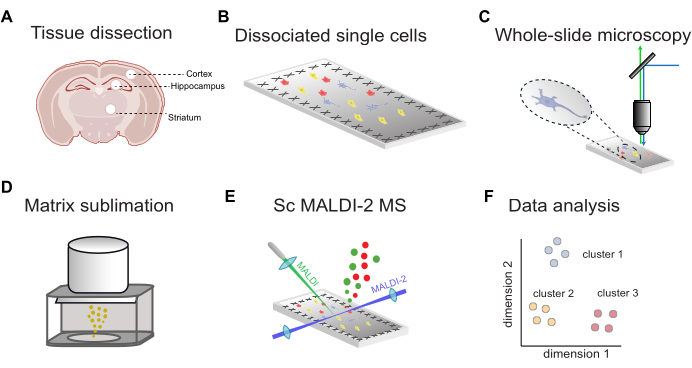
Şekil 1: scMALDI-2 MS. (A) Beyin bölgelerinin manuel doku diseksiyonu ve izolasyonu. (B) Dokunun tek hücrelere papain ayrışması ve iletken ITO kaplı slaytlar üzerine hücre birikimi. (C) Nükleer boyama (DAPI / Hoechst) yoluyla tam slayt floresan mikroskobu. (D) Numune slaytları üzerine MALDI matrisi (DHAP) süblimasyonu. (E) Görüntü kılavuzlu tek hücreli MALDI-2 MS ölçümleri. (F) Boyutsallığın azaltılması, kümeleme ve istatistiksel analiz dahil olmak üzere kapsamlı veri analizi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
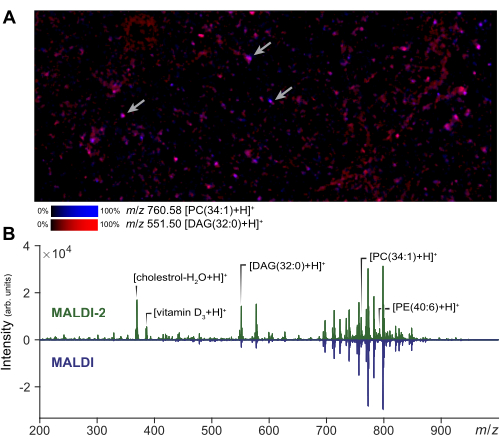
Şekil 2: Tek hücre analizi için MALDI-2 MS optimizasyonu ve görüntüleme. (A) Primer hücrelerin dağınık popülasyonlarının MALDI-2 MSI görüntülemesi. Oklar birkaç ayrı hücreyi gösterir. MSI ve küçük bir bölge gerçekleştirmek, analistin yöntem parametrelerini optimize etmesine ve numune kalitesini değerlendirmesine olanak tanır. (B) MALDI-2 ve MALDI MS edinimleri kullanılarak elde edilen sonuçların karşılaştırılması. Kolesterol, DAG (diasilgliseroller), PC (fosfatidilkolin) ve PE (fosfatidiletanolamin) dahil olmak üzere çeşitli lipitlerin sinyal geliştirmesi (MALDI 2'ye karşı MALDI), MALDI-2 spektrumunu (üstte) ve MALDI spektrumunu (altta) göstererek vurgulanmaktadır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
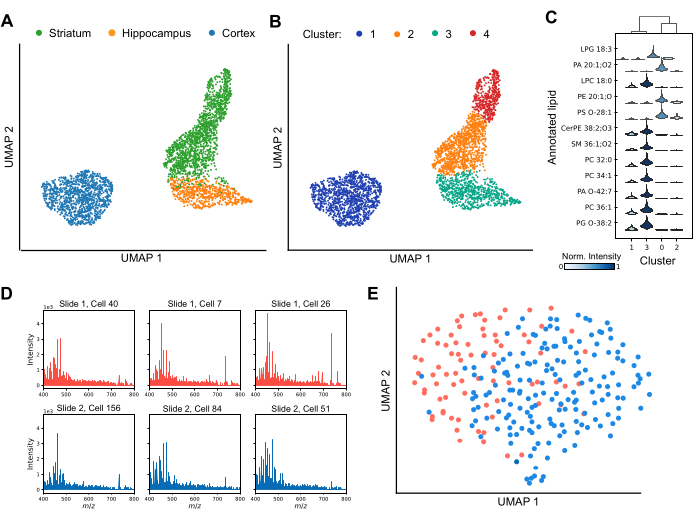
Şekil 3: MALDI-2 MS. (A) beyin orijin bölgesine göre renklendirilmiş analiz edilen hücrelerin tekdüze manifold yaklaşımı ve projeksiyon (UMAP) analizi ile üç kemirgen beyin bölgesi içinde tek hücreli lipid profili. (B) Leiden kümelenmesine tabi tutulduktan sonra aynı hücrelerin UMAP analizi, burada kümeler benzer lipid bileşimlerine sahip hücrelerin alt popülasyonlarına karşılık gelir. Buradaki renkler farklı Leiden veri kümelerini göstermektedir. (C) Kümeler arasındaki farklı lipitler için yoğunluk dağılımlarındaki farkı gösteren bir keman çizimi. (D) Altı temsili bireysel kortikal hücreden alınan spektrumlar iki ayrı slayttan analiz edildi. (E) İki ayrı slayttan analiz edilen kortikal hücrelerin UMAP analizi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Ek Dosya 1: SC-MALDI2_Analysis.py. Bu dosya, tek hücreli MALDI-2 kütle spektrometresi verilerini işlemek için geliştirilmiş bir dizi işlev içerir. Ham kütle spektrometresi verilerini okumak, gruplama yapmak ve açıklama ve aşağı akış analizleri yapmak için yöntemler içerir. Tüm veri ön işleme ve analizlerinin yükleneceği ve gerçekleştirileceği hücre seti Ek Dosya 2'de verilmiştir. Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Dosya 2: JOVE Analysis.ipynb. Bu dosya, SC-MALDI2_Analysis.py'dan özel Python işlevlerine yüklenecek ve belirtilen tüm veri ön işleme ve analizlerini gerçekleştirmek için bir dizi hücre içeren bir Jupyter not defteri içerir. Bu dosyayı indirmek için lütfen buraya tıklayın.
Tartışmalar
Yüksek verimli, görüntü kılavuzlu tek hücreli MALDI MS, tek hücre ölçeğinde kimyasal heterojenliği anlamak için değerli bir araçtır. Lazer kaynaklı post-iyonizasyon (MALDI-2) ilavesi, izole edilmiş memeli hücreleri gibi kütle ve hacim sınırlı numuneler için kritik olan daha derin analit kapsamı sağlar.
Yayınlanmış tek hücreli lipid ve metabolit MS iş akışlarının ezici çoğunluğu kültürlenmiş hücreleri kullanırken, yaklaşımımız nispeten hızlı bir şekilde izole edilen birincil hücrelere uygulanır. Bu ayrım kritiktir, çünkü kültürlenmiş hücreler kültürlemeden sadece birkaç gün sonra metabolit ve lipit içeriklerinde gösterilebilir bir değişiklik gösterir23,31. Enzimatik hücre izolasyonu, çoğu durumda, aksonlar, dendritler ve astrositik süreçler gibi ince hücresel yapıların kaybına yol açar. Bu nedenle, gözlenen analit sinyalleri esas olarak hücre somasından elde edilir. Buna rağmen, hücre tipi4 ve beyin bölgesine göre başarılı bir sınıflandırma elde edilmiştir12. Bu, farklı hücrelere özgü lipitlerin çoğunun soma içinde bulunduğunu göstermektedir.
MALDI matris uygulama yöntemindeki küçük farklılıklar, elde edilen analit sinyali üzerinde derin etkilere sahip olabilir. Homojen bir matris katmanı, yüksek sinyal-gürültü ve sınırlı analit yayılımı oluşturan ticari bir aparatla tekrarlanabilir matris süblimasyonuna dayalı bir iş akışı sunduk. Bununla birlikte, diğer yaklaşımların yanı sıra robotik bir püskürtücü veya sanatçının airbrush'ı kullanılarak matris uygulaması kullanılabilir. Yaklaşımdan bağımsız olarak, optik matris-analit oranını elde etmek için standart doku MSI'da kullanılana kıyasla tek hücreli preparatlara daha düşük bir matris yoğunluğu uygulanmalıdır. Burada uygulanan matris yoğunluğu, doku MSI için kullanılanın yaklaşık yarısı kadardır. Tabii ki, bu, hücre başına matris miktarının MSI'dan daha yüksek olduğu anlamına gelir, bu da burada kullanılan izole hücre yaklaşımında gelişmiş analit ekstraksiyonu ve tespitine katkıda bulunmaya yardımcı olur.
Önemli optimizasyon gerektiren numuneler için, Şekil 2A'da gösterildiği gibi, tek hücreli ayrışmaların küçük bir ROI'si üzerinde MSI gerçekleştirmek, optimizasyon işleminin bir parçası olarak gerçekleştirilebilir. Bu adım, analistin solvent bazlı matris uygulama yaklaşımlarında analit yayılma derecesini değerlendirmesine de izin verebilir.
Tek hücrelerin başarılı görüntü kılavuzlu MALDI'si, yüksek kaliteli floresan görüntüleri, tekrarlanabilir matris uygulaması ve hassas aşama kontrolü gerektirir. Bu deneyler, daha büyük görüntüleme alanları ve daha yüksek analit sayıları nedeniyle küçük kayıt hatalarını ve matris ve numune koşullarındaki değişkenliği tolere eden standart MSI iş akışlarından daha zahmetlidir. Bu zorluklara rağmen, geleneksel MALDI kullanarak tek organelleri başarıyla hedefledik.
Döşenmiş floresan görüntülerin doğru bir şekilde birleştirilmesi çok önemlidir, çünkü küçük hatalar sahne kontrolünü birleştirip etkileyebilir, bu da kayıt doğruluğunu kritik hale getirir. Toplu efektler de aynı slayt boyunca, slaytlar arasında ve hazırlıklar arasında gözlemlenen sinyal değişimleri ile büyük bir zorluk teşkil eder. İç standartların kullanımı ve çeyrekler arası normalleştirme de dahil olmak üzere uygun normalleştirme stratejileri bu sorunların azaltılmasına yardımcı olur. Ek olarak, ComBat30 gibi toplu düzeltme algoritmaları, teknik değişkenliği azaltmak ve gerçek biyolojik farklılıkları geliştirmek için kullanılabilir.
Tek hücrelerden güvenli moleküler açıklamalar elde etmek bir zorluk olmaya devam etmektedir. Genellikle, materyal miktarı ve tespit hassasiyeti, en bol bulunan türler dışındaki tüm türlerden tandem MS için yetersizdir. Doğrudan tek tek hücrelerden tandem MS için yeterince tespit edilemeyenler için, bir analit kütüphanesi oluşturmak için alternatif yaklaşımlar uygulanabilir. Örneğin, doku üzerinde tandem MS verileri, aynı beyin bölgesinden ve hücresel popülasyonlar oluşturmak için kullanılan hayvanlardan elde edilen ince kriyoseksiyonlardan elde edilebilir. Dokudan lipit ekstraksiyonu ve ardından LC-MS de kullanılabilir. Numune hazırlama stratejileri ve MS teknolojisi gelişmeye devam ettikçe, doğrudan tek hücrelerden giderek daha fazla ilgili yapısal bilgi elde edilebilir. Gelecekte, bu iş akışının, tek hücrelerden tandem MS verileri elde etmek için genişletileceğini ve yardımcı LC-MS/MS deneylerine olan ihtiyacı ortadan kaldıracağını tahmin ediyoruz. Ayrıca, bu yaklaşımın, benzersiz hücre tipleri, memeli organelleri, tozlar ve mikroplastikler dahil olmak üzere biyoloji ve ötesinde mikro ölçekte çok sayıda örneğe genişletilebileceğini öngörüyoruz.
Açıklamalar
Yazarların ifşa etmek için rekabet eden çıkarları yoktur.
Teşekkürler
SWC, Peixin He ve Xiaoming Chen PhD4 Bursu ve Illinois Üniversitesi Block Grant Bursu tarafından sağlanan desteği kabul eder. Bu çalışma aynı zamanda Ulusal Uyuşturucu Bağımlılığı Enstitüsü tarafından da ödül No. P30DA018310, Ulusal Yaşlanma Enstitüsü Ödül No. R01AG078797 ve Ulusal Sağlık Enstitüleri Direktörlük Ofisi tarafından S10OD032242 Numaralı Ödül altında.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 2',5'-dihydroxyacetophenone | Sigma Aldrich | D107603 | DHAP, 97% purity |
| Ammonium acetate | Sigma Aldrich | 238074 | ACS reagent, ≥97% |
| Axio M2 Imager | Zeiss | N/A | N/A |
| Biopsy punch, 2 mm | Fisher Scientific | 12-460-399 | integra miltex standard biopsy punch, 2mm |
| Calcium chloride | Sigma Aldrich | C4901 | anhydrous, powder ≥97% |
| Eppendorf Centrifuge | Sigma Aldrich | EP5405000441 | centrifuge 5425 with rotor FA-24x2 |
| Gentamicin | Sigma Aldrich | G1272 | liquid, BioReagent |
| Glass etching pen | Sigma Aldrich | Z225568 | carbide time, pkg of 1 |
| Glycerol | Sigma Aldrich | G7893 | ACS reagent, ≥99.5% |
| HEPES buffer | Sigma Aldrich | H3375 | ≥99.5% (titration) |
| Hoechst 33258 Solution | Sigma Aldrich | 94403 | 1 mg/mL in H2O, ≥98.0% (HPLC) |
| In line HEPA Filter | Sigma Aldrich | WHA67225001 | VACU-GUARD 60 mm disc, 0.45 PFTE housing |
| ITO-Coated Microscopy Slides | Delta Technologies | CG-90IN-S115 | 70-100Ω resistance |
| Magnesium chloride | Sigma Aldrich | M8266 | anhydrous, ≥98% |
| Magnesium sulfate | Sigma Aldrich | 208094 | anhydrous, ≥97% |
| Microcentrifuge tubes | Sigma Aldrich | HS4323K | tube capacity 1.5 mL, pack of 500 |
| Papain dissociation system | Worthington Biochemical | LK003150 | one box, 5 single use vials |
| Penicillin-Streptomycin | Sigma Aldrich | P4458 | liquid, BioReagent |
| Potassium chloride | Sigma Aldrich | 529552 | Molecular biology grade |
| Potassium phosphate monobasic | Sigma Aldrich | P5379 | Reagent Plus |
| Sodium biocarbonate | Sigma Aldrich | S6014 | ACS reagent, ≥99.7% |
| Sodium chloride | Sigma Aldrich | S9888 | ACS reagent, ≥99% |
| Sodium hydroxide | Sigma Aldrich | 221465 | ACS reagent, ≥97%, pellets |
| Sodium phosphate dibasic | Sigma Aldrich | S9763 | ACS reagent, ≥99% |
| Sublimate | HTX | N/A | N/A |
| timsTOF FleX MALDI-2 | Bruker | N/A | microGRID enabled |
| Vacuum tubing | Thermo Scientific | 8701-0080 | Nalgene Non-phthalate PVC Tubing |
Referanslar
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır