Method Article
Desorción/ionización láser asistida por matriz guiada por fluorescencia con espectrometría de masas de posionización inducida por láser de células neuronales de rata individuales
* Estos autores han contribuido por igual
En este artículo
Resumen
Este protocolo describe el uso de microMS para la espectrometría de masas MALDI-2 unicelular guiada por fluorescencia, lo que permite un mejor perfil molecular de las células neuronales primarias de rata.
Resumen
Las mediciones de una sola célula son fundamentales para comprender la rica heterogeneidad espacioquímica del cerebro. La espectrometría de masas (MS) de ionización por láser/desorción asistida por matriz (MALDI) es capaz de caracterizar moléculas endógenas sin etiquetas y de alto rendimiento en células individuales. Los recientes avances en el desarrollo de espectrómetros de masas MALDI con postionización inducida por láser (MALDI-2) proporcionan una sensibilidad de detección muy mejorada para una variedad de lípidos y otras moléculas pequeñas. Sin embargo, la obtención de imágenes de MS de muestras grandes con MALDI-2 a resolución celular es prohibitivamente lenta para la mayoría de las aplicaciones. En este protocolo, las células primarias se aíslan y se dispersan en portaobjetos conductores. Las ubicaciones relativas de las células se determinan mediante microscopía de fluorescencia de portaobjetos completos, seguida de un registro conjunto preciso de las coordenadas de la microscopía con las coordenadas de la etapa del espectrómetro de masas MALDI-2. El análisis de MS dirigido solo de ubicaciones de celdas proporciona mediciones de alto rendimiento de una sola celda con una alta cobertura de analito y un tamaño de datos reducido en comparación con las imágenes de MS de toda la muestra. Describimos los pasos críticos necesarios para la preparación de una sola célula, la obtención de imágenes de fluorescencia de portaobjetos completos, la aplicación de matrices y la espectrometría de masas MALDI-2.
Introducción
Los lípidos y metabolitos son fundamentales para la función celular y sirven como componentes esenciales de las membranas, fuentes de energía y moléculas de señalización 1,2. Sin embargo, su composición y abundancia pueden variar significativamente entre las células individuales, lo que refleja las diferencias en los tipos de células y los estados funcionales y de desarrollo 3,4,5. El análisis de estas diferencias es crucial para comprender la variabilidad biológica e identificar distintas subpoblaciones celulares. Las técnicas de medición de una sola célula, como la secuenciación de ARN, proporcionan perfiles de transcripción específicos de célulasútiles 6. Sin embargo, estas mediciones a nivel de transcripción no reflejan directamente las cantidades celulares reales de lípidos y metabolitos, ya que la expresión génica no siempre se correlaciona con la abundancia real de estos analitos. Por lo tanto, se requieren métodos especializados para mediciones directas de lípidos y metabolitos para el análisis exhaustivo de la composición química de células individuales y sus poblaciones.
Las imágenes de espectrometría de masas (MSI) de desorción/ionización láser asistidas por matriz (MALDI) son una herramienta de elección para el mapeo espacial sin etiquetas de biomoléculas endógenas in situ 7,8. Por lo general, con MALDI, se usa un láser UV para extirpar material de una capa de muestra delgada cocristalizada con una matriz orgánica, formando una columna de iones y moléculas neutras. A continuación, los iones formados se separan mediante un analizador MS y se detectan dentro de un espectrómetro de masas. Dado el posicionamiento preciso de las etapas de los espectrómetros de masas modernos, el láser se puede posicionar para apuntar a regiones específicas de la muestra o se puede estirar a través de regiones para generar imágenes moleculares por MSI. La MSI con una resolución espacial celular o subcelular (< 10 μm), lograda con un rayo láser enfocado y un movimiento preciso de la etapa, se puede utilizar para obtener información química sobre células individuales 9,10. Sin embargo, las mediciones de MSI a esta escala son ineficientes, especialmente en el caso de muestras con bajas densidades de células objetivo, debido a la cantidad de tiempo dedicado a obtener imágenes de regiones vacías entre células. Además, la detectabilidad de muchos analitos es limitada debido al pequeño volumen muestreado. Para superar estos desafíos, hemos desarrollado un enfoque de MS MALDI guiado por imágenes para el análisis de células individuales de alto rendimiento11,12. En este enfoque, las ubicaciones de las células dispersas se determinan mediante programación a partir de imágenes de fluorescencia de portaobjetos completos y se utilizan para guiar la etapa del espectrómetro de masas a las posiciones donde se encuentran las células individuales con parámetros específicos (por ejemplo, tamaño y forma) y luego se analizan a través de la irradiación con el láser MALDI. Trabajos previos que utilizan este enfoque dirigido se han utilizado para caracterizar lípidos, péptidos y otras biomoléculas en poblaciones celulares heterogéneas 5,13.
Dada la naturaleza limitada de la masa de las células individuales, el número de analitos detectados en dichas muestras es generalmente menor que el observado directamente en el tejido. Por lo tanto, para aumentar la cobertura del analito en el análisis de EM de una sola célula, es fundamental aumentar la sensibilidad de detección del analito. Un enfoque desarrollado recientemente que ayuda a superar este desafío de detección es MALDI con postionización láser (MALDI-2), que se ha demostrado que mejora la sensibilidad para una amplia gama de analitos 9,14,15,16. Como resultado, MALDI-2 genera conjuntos de datos más completos de una sola célula y proporciona una cobertura molecular más profunda en muestras de masa limitada, como células aisladas.
El objetivo de este método es obtener mediciones de lípidos de miles de células individuales. Con este fin, describimos un flujo de trabajo que permite la espectrometría de masas MALDI-2 de celda única de alto rendimiento y generalmente se puede extender a cualquier enfoque de espectrometría de masas basado en sondas con control de etapa preciso17. En este flujo de trabajo, se disecciona el tejido de las regiones cerebrales de interés y se obtienen células individuales del tejido después de un procedimiento de disociación de papaína. Luego, las células se etiquetan con una tinción nuclear y se dispersan en portaobjetos de vidrio conductor grabados con marcadores de referencia, donde se les permite adherirse. A continuación, se toman imágenes de portaobjetos completos mediante microscopía de fluorescencia. La matriz se deposita por sublimación, lo que genera una capa de cristal homogénea repetible y una alta relación señal-ruido para el análisis de MS de una sola célula. Utilizando el software de código abierto microMS11, las coordenadas relativas de las ubicaciones de las células de la imagen de microscopía se mapean y alinean con las coordenadas de la etapa del espectrómetro de masas mediante un registro de conjuntos de puntos utilizando marcadores de referencia grabados en los portaobjetos de vidrio donde se depositaron las células. Por último, utilizando esta información, se adquieren espectros de MS precisos y específicos de cada célula individual, lo que permite perfilar miles de células en una sola corrida (<1 h)12,13.
Protocolo
Todos los experimentos con animales en este estudio se realizaron de acuerdo con el protocolo de uso animal aprobado por el Comité Institucional de Cuidado y Uso de Animales de Illinois (23228) con estricto cumplimiento de los estándares nacionales y ARRIVE para el tratamiento ético y el cuidado de los animales.
1. Preparación de materiales y soluciones
- Prepare la solución salina balanceada de Gey modificada (mGBSS) con 1,5 mM de CaCl2, 4,9 mM de KCl, 0,2 mM de KH2PO4, 11 mM de MgCl2, 0,3 mM de MgSO4, 138 mM de NaCl, 27,7 mM de NaHCO3, 0,8 mM deNa2HPO4 y 25 mM de HEPES. Ajuste el pH a 7.2 usando 3 M de NaOH. Se recomienda preparar 1 L de mGBSS y almacenar a 4 °C hasta su uso.
- Prepare jeringas de 50 mL llenándolas con la solución de mGBSS preparada y colóquelas en hielo para la perfusión transcárdica. Prepare 1 jeringa por cada rata disecada.
- Prepare las alícuotas de la solución de papaína siguiendo el protocolo del sistema de disociación de papaína de Worthington. Este sistema contiene cuatro viales como se describe a continuación, y el vial 4 no se utiliza para este protocolo:
Vial 1: solución salina equilibrada de Earle estéril (EBSS) con bicarbonato y rojo de fenol
Vial 2: papaína con L-cisteína y EDTA
Vial 3: desoxirribonucleasa I (DNasa)
Vial 4: inhibidor de la proteasa ovomucoide con albúmina sérica bovina- Añadir 5 mL de EBSS (vial 1) al vial de papaína (vial 2) y colocarlo en un baño de agua a 37 °C durante 10 min para disolver la papaína.
- Añadir 500 μL de EBSS (vial 1) en el vial de DNasa (vial 3) y mezclar suavemente. Añadir 250 μL del vial de DNasa diluido (vial 3) en el vial de papaína (vial 2). Esta preparación contiene una concentración final de 20 unidades/mL de papaína, 0,005% de DNasa, 0,5 mM de EDTA y 1 mM de L-cisteína.
- Congele 250 μL de alícuotas de esta solución de papaína preparada para disociaciones posteriores (hace ~ 20 alícuotas). Complemente esta solución con los siguientes antibióticos: 100 unidades/mL de penicilina G, 100 μg/mL de estreptomicina y 100 μg/mL de gentamicina. Esto se puede modificar para la aplicación del usuario.
- Prepare una solución de acetato de amonio de 150 mM y guárdela a 4 °C. Prepare una solución de glicerol al 33% diluyendo glicerol en la solución de mGBSS preparada.
- Grabe fiduciales en portaobjetos de microscopía recubiertos de óxido de indio y estaño (ITO) utilizando un bolígrafo de grabado con punta de carburo de tungsteno o diamante o un grabador láser de CO2 . En el lado conductor del portaobjetos de vidrio ITO, grabe 20-40 marcadores de referencia en forma de X a lo largo de la periferia del portaobjetos para permitir el registro espacial multiplataforma (por ejemplo, microscopio y espectrómetro de masas).
NOTA: Tenga cuidado de no grabar en una línea conectada alrededor de la muestra o sus áreas, ya que esto interrumpe la conductividad de la superficie entre el área y un adaptador de portaobjetos de vidrio.
2. Preparación de células neuronales primarias
NOTA: El tejido del hipocampo de rata se disecciona, se disocia en células individuales con papaína y se deposita en portaobjetos de vidrio conductor a baja densidad. El aislamiento de las células de esta manera permite la espectrometría de masas de células individuales de alto rendimiento de lípidos endógenos.
- Antes de la disección del animal, descongele una alícuota de la solución de papaína preparada y colóquela en un recipiente con un tubo de entrada y salida que permita que el CO2 desplace el aire dentro del recipiente. Coloque este sistema en un baño de agua ajustado a 37 °C y continúe el flujo de CO2 hasta que la solución tenga el pH adecuado, como lo indica el color, utilizando la tabla de colores de pH incluida en el paquete del sistema de disociación Worthington Papain. El pH objetivo está entre 7,2 y 7,4.
NOTA: No burbujee CO2 en la solución enzimática, ya que esto provocará la desnaturalización de la DNasa y la pérdida de la actividad biológica. Utilice un tubo de centrífuga de 50 ml con dos tubos pegados en la tapa, lo que permite que el CO2 llene el tubo desde la parte inferior y escape por el tubo de salida superior. - Sacrificar ratas de acuerdo con los protocolos de IACUC siguiendo las pautas institucionales, locales y federales. Aquí, se utilizan ratas macho Sprague-Dawley de 2 a 3 meses de edad y se sacrifican por asfixia con CO2 .
- Perfundir transcardialmente la rata con una solución de mGBSS helada (4 °C) para eliminar la sangre del sistema vascular y enfriar rápidamente el cuerpo del animal. Un ejemplo de este procedimiento se puede encontrar en el protocolo de We et al.18.
- Aísla quirúrgicamente el cerebro de la rata, asegurándote de no dañar la región cerebral de interés. Una visión general de este aislamiento publicada anteriormente proporciona más detalles sobre algunos de los pasos individuales19.
- Utilice un picador de tejido o un cubo de tejido para cortar una sección transversal (~2 mm de grosor) del cerebro para exponer la región (hipocampo, corteza o cuerpo estriado) a la muestra.
- Use un punzón de biopsia de 2 mm para tomar muestras de la región cerebral de interés y deposite el trozo de tejido en el frasco de solución de papaína.
- Incubar esta solución de tejido y papaína a 37 °C bajo agitación constante (por ejemplo, con un balancín eléctrico) durante 30-90 min.
NOTA: La duración del tratamiento enzimático debe optimizarse en función de la región del cerebro, la cepa del animal, la edad y la producción celular deseada (por ejemplo, neuronas y astrocitos con terminales frente al rendimiento máximo de células sin terminales). Para el tejido del hipocampo de rata, 60-90 min es típico. Los tratamientos más cortos pueden preservar algunas terminales pero dejar muchas células en grupos no disociados, mientras que los tratamientos más largos pueden mejorar el rendimiento pero aumentar el daño celular, lo que requiere una evaluación mediante ensayos vivos/muertos (p. ej., kit de viabilidad/citotoxicidad LIVE/DEAD). Para los estudios comparativos, los tratamientos deben ser consistentes en todos los tipos de muestras. - Coloque un frasco de pañuelo y papaína sobre hielo para enfriar y suprimir la actividad enzimática. Triture el tejido con una punta de pipeta cortada conectada a una pipeta de volumen ajustable P100 hasta que la mayor parte del tejido se rompa visualmente. No forme burbujas de aire durante la disociación para evitar un daño celular excesivo.
NOTA: La trituración vigorosa da como resultado un alto rendimiento de células; sin embargo, no conservan la mayoría de sus procesos. La trituración suave proporciona más procesos en las células, pero da como resultado más células no disociadas. También se pueden utilizar puntas de pipeta de diferentes tamaños y otros métodos, que se pueden optimizar para obtener resultados de disociación ideales20. - Centrifugar las células disociadas a 300 x g durante 5 min a temperatura ambiente para granular las células. Retirar el sobrenadante y volver a suspender el pellet en 1 mL de mGBSS. Añadir la tinción nuclear Hoechst 33342 hasta una concentración final de 1 μg/mL.
- Pipetee 100-500 μL de la suspensión celular en portaobjetos de vidrio ITO grabados y déjelos reposar durante aproximadamente 5 minutos o más para permitir que las células se sedimenten y se adhieran al portaobjetos.
- Será necesario determinar empíricamente la dilución de la suspensión celular. Usando un microscopio óptico, agregue una gota de las células en el portaobjetos para determinar la densidad aproximada y diluya a partir de ahí para asegurarse de que haya suficiente distancia entre las células (es decir, lo suficientemente grande que el tamaño del haz MALDI). Esta dilución se puede realizar en el primer portaobjetos y, a continuación, la dilución se puede ajustar según sea necesario para los portaobjetos posteriores.
- Aspire suavemente la suspensión de la celda fuera del portaobjetos utilizando un sistema de vacío que contenga (ubicado en orden a partir de la línea de vacío): 1) Tubo con un filtro HEPA de gas en línea, 2) Tubo instalado en un matraz filtrante de 50 mL, 3) Tubo conectado a una segunda entrada de matraz filtrante con una punta de pipeta adjunta (por ejemplo, 100 μL). Si almacena las celdas plateadas para uso futuro, reemplace la suspensión aspirada con una solución de glicerol al 33% durante 1-2 minutos y retírela.
NOTA: Este paso asegurará la estabilidad de la celda. Dispensar la solución de glicerol por un lado mientras se aspira suavemente por el otro lado funciona es óptimo para mantener la estructura celular. Supervise visualmente siempre todos los pasos descritos aquí utilizando un microscopio invertido durante la preparación de la primera muestra. - Aplique 100-500 μL de acetato de amonio en las regiones depositadas, deje reposar durante aproximadamente 1 minuto, luego aspire para limpiar los portaobjetos. Repita tantas veces como sea necesario para asegurarse de que los portaobjetos estén libres de sal o glicerol.
3. Microscopía
NOTA: Para determinar la ubicación de las células depositadas, cada portaobjetos se visualiza mediante microscopía de campo claro/fluorescencia. El canal de fluorescencia permite localizar con precisión las células teñidas con Hoechst, mientras que las imágenes de campo claro proporcionan información morfológica. Cualquier microscopio capaz de adquirir imágenes en mosaico es adecuado para este proceso.
- Si no lo ha hecho previamente, enjuague los portaobjetos con 2-3 mL de acetato de amonio de 150 mM. Este paso es fundamental para eliminar el glicerol y los cristales de sal, que pueden interferir con la observación de la microscopía y la aplicación de la matriz MALDI. Seque los portaobjetos bajo un suave chorro de N2 o déjelos secar al aire.
- Cargue el portaobjetos en la platina del microscopio. Enfoque el microscopio utilizando al menos 10 puntos de apoyo distribuidos uniformemente por el portaobjetos.
- Utilizando filtros adecuados para DAPI (excitación: 335-383 nm, emisión: 420-470 nm) e imágenes de campo claro, obtenga una imagen de fluorescencia en mosaico con un aumento de 5x-10x en toda la diapositiva, asegurando que se capturen los marcadores de referencia (creados en el paso 1.5).
NOTA: Un aumento de 5x suele ser suficiente, especialmente cuando es necesario obtener imágenes de muchas diapositivas. Para resolver una morfología celular más fina, se pueden utilizar objetivos de 10x o 20x a costa de un tiempo de imagen cuadráticamente más largo y un aumento del tamaño de los archivos. - Cose la imagen en mosaico con un software de microscopía, como ZEN (Zeiss). La vinculación precisa es fundamental, ya que cualquier error en este paso se propagará al paso de registro final, lo que dará como resultado una orientación inexacta. Para comprobarlo, asegúrese de que las baldosas adyacentes verticalmente no estén desplazadas. Además, considere la posibilidad de adquirir imágenes en mosaico con una gran superposición de mosaicos (20%-30%).
- Procese y exporte cada imagen como un archivo bigTIFF en el software de microscopía. Si la región de la que se muestra la imagen es relativamente pequeña y da como resultado un tamaño de imagen final de menos de 2 GB, utilice un formato de archivo TIFF estándar en su lugar.
4. Aplicación matricial
NOTA: La aplicación coherente y adecuada de la matriz MALDI es fundamental para obtener datos de calidad de una sola célula. Mientras que aquí se utiliza la sublimación con un aparato comercial, la aplicación de la matriz también se puede realizar con un pulverizador robótico12, un aerógrafo21 o un aparato de sublimación casero22. Hemos descubierto que las preparaciones unicelulares requieren menos matriz que las secciones de tejido delgado que se utilizan normalmente para la obtención de imágenes de espectrometría de masas. Para reducir los efectos de lote, se recomienda aplicar la matriz a todos los portaobjetos en estudio durante una sesión y depositar células de diferentes grupos (por ejemplo, región del cerebro o tratamiento vs. control) en el mismo portaobjetos siempre que sea posible. La selección de matrices es crucial para los flujos de trabajo tradicionales de una sola celda MALDI y MALDI-2. Para MALDI de una sola celda, se han utilizado con éxito DHB23, 9-AA24 y CHCA25 . En MALDI-2, nosotros y otros hemos observado una mejora significativa de la señal con DHAP9, mientras que matrices como NEDC16 y CHCA26 también se han aplicado de manera efectiva.
- Disolver 20 mg de 2,5-dihidroxiacetofenona (DHAP) en 1,5 mL de acetona. Coloque las diapositivas en el soporte de la cámara de sublimación y colóquelas en el aparato de sublimación.
- Pipete la solución de matriz disuelta en la oblea de cerámica y deja que la acetona se evapore. Cierre la cámara de sublimación para sellarla.
- Llene la cámara de refrigerante con un aguanieve de agua helada y colóquelo encima de la cámara.
- Encienda la aspiradora y deje que el sistema se equilibre durante 5 minutos. La presión en el sistema debe ser inferior a 40 mbar.
- Comience la sublimación calentando la cámara a 200 °C durante 5 min.
- Retire el baño de agua helada, baje la temperatura a 25 °C y coloque un disipador de calor en la parte superior de la cámara. Deje que el sistema se caliente a temperatura ambiente durante 5 minutos para evitar la condensación.
NOTA: El recipiente que contiene el baño de agua helada se puede vaciar y llenar con agua caliente para que sirva como disipador de calor. - Ventile lentamente el sistema, abra la cámara y retire las correderas.
5. MALDI MS de una sola célula
NOTA: Los datos de MS de una sola célula se obtienen en un instrumento timsTOF MALDI-2 (timsTOF flex) utilizando el paquete microMS de código abierto para detectar células y guiar el espectrómetro de masas. Esto requiere que las ubicaciones de los píxeles de la imagen óptica de las células objetivo se trasladen a las coordenadas físicas de la etapa del espectrómetro de masas.
- Abra microMS y diezme las imágenes de microscopía bigTIFF usando la opción Grupo de imágenes, ya que hay canales de campo claro y fluorescencia.
- Con la herramienta Opciones de blobs, identifique los parámetros óptimos que se deben seleccionar para las celdas de destino. Ajuste los siguientes parámetros: el tamaño máximo y mínimo de los blobs para seleccionar el tamaño pequeño o grande de los blobs que desea encontrar; el ajuste de umbral, que determina cómo umbralizar el canal de fluorescencia para la detección de blobs; y la circularidad, que determina qué tan circulares deben ser las celdas identificadas para su consideración. Para los datos aquí presentados, utilizamos los siguientes valores de parámetros: circularidad máxima: ninguna; circularidad mínima: 0,6; umbral: 75; tamaño mínimo: 20; Tamaño máximo: Ninguno.
- Use la opción Buscar blobs para buscar blobs y la ventana emergente para guardar la lista de blobs con el nombre que desee.
- Utilice la herramienta de filtro de distancia para seleccionar una distancia específica a la que cada celda debe estar alejada entre sí (para estos datos, utilizando microscopía de fluorescencia 5x, utilizamos un filtro de distancia de 35 píxeles). Esto es para garantizar que las células estén lo suficientemente alejadas unas de otras para realizar análisis de una sola célula. Este número se expresa en unidades de píxel, por lo que debes conocer la conversión de píxeles a μm para que el microscopio determine el número ideal. Utilice el tamaño del diámetro de la sonda láser más alrededor de 20-30 μm para tener en cuenta los errores en el registro.
- Cargue las correderas en el instrumento utilizando el adaptador de diapositivas MTP II. Regrese a la computadora con microMS y, mediante una aplicación de escritorio remoto, acceda a la computadora del espectrómetro de masas.
- Si es la primera vez que se utiliza el instrumento, verifique la posición X-Y del instrumento. Para ello, vaya a Configuración de herramientas e instrumentos. La ventana emergente mostrará un conjunto de coordenadas con sus posiciones X e Y. Seleccione cada uno de estos puntos específicos en la geometría del adaptador de diapositiva II en el instrumento y actualice las posiciones X e Y en la ventana de microMS.
- Usando la cámara y los controles de la platina del espectrómetro de masas, navegue hasta una ubicación fácilmente identificable en el portaobjetos y copie las coordenadas del instrumento. En microMS, busque la ubicación correspondiente en la imagen del microscopio, haga clic con el botón derecho en esa posición y escriba las coordenadas en la ubicación del texto de la ventana emergente. Redondee todas las coordenadas al entero más cercano y separe las coordenadas X e Y con espacio (es decir, 39595 -23232).
- Repita el paso 5.7. para registrar al menos 12 marcadores de referencia en toda la diapositiva. Después de que se hayan agregado tres puntos de registro, uno de los círculos se volverá rojo, lo que indica que es la posición más alejada del registro. Si esto ocurre, elimine el punto de registro actual usando Mayús + clic derecho y vuelva a intentarlo.
- En Archivo, vaya a Guardar y, a continuación, a Registro para guardar el archivo de registro (.msreg).
- Con los blobs mostrados en la diapositiva, guarde el archivo de posición del instrumento (.xeo) yendo a Archivo, luego a Guardar y, a continuación, a Posiciones del instrumento. Usando el software de escritorio remoto, transfiera este archivo .xeo a la computadora del instrumento.
- Copie el texto del archivo .xeo personalizado generado en el archivo .xeo de MTP Slide Adapter II en el instrumento y guárdelo. Esto actualizará este archivo de geometría con las ubicaciones de las celdas.
- Haga clic en la pestaña Automatización y seleccione Nuevo... para iniciar una ejecución automática. Arrastre por la región de muestra mostrada para seleccionar las celdas y haga clic con el botón derecho para agregarlas a la lista de análisis. Guarde la ejecución de automatización y haga clic en Iniciar ejecución automática para comenzar la adquisición. La duración de la ejecución depende de una variedad de factores, incluido el número de celdas, los disparos láser y la frecuencia del láser, pero en general, se pueden analizar aproximadamente 5-10 celdas en modo MALDI (tasa de repetición láser de 10 kHz) y 1-5 celdas se pueden analizar en modo MALDI-2 (tasa de repetición láser de 1k Hz) cada segundo.
6. Tratamiento de datos
NOTA: Los paquetes de software comercial existentes no son adecuados para analizar datos de espectrometría de masas de célula única de alto rendimiento. Si bien se pueden visualizar espectros individuales, la extracción de información biológica significativa requiere herramientas especializadas. Para abordar esto, proporcionamos software disponible gratuitamente que facilita el análisis de datos de MALDI-2 MS de una sola celda. Nuestro flujo de trabajo actualizado facilita la conversión directa de datos de una sola celda al formato imzML de código abierto27, lo que permite la compatibilidad con SCiLS MVS y software de otros proveedores. Para un análisis de datos más avanzado, el script completo también incluye funcionalidad para la anotación de lípidos, la agrupación y otras herramientas de visualización habilitadas por Matplotlib (versión 3.7.3). El análisis y la lectura de los datos sin procesar se facilitan mediante la biblioteca pyTDFSDK, un conjunto de funciones incluidas en el flujo de trabajo TIMSCONVERT28.
- Inicialice la biblioteca personalizada de Python SC_MALDI2_Analysis copiando el archivo proporcionado en el archivo complementario 1 en la misma ruta de trabajo que Jupyter Notebook y, a continuación, presione Mayús + Entrar en el teclado en la primera celda de código. Esto nos permitirá llamar a estas funciones más adelante y usarlas para procesar los datos.
- Copie la ruta de acceso a los datos recopilados (archivos .d de Bruker). Se recomienda asignar un nombre descriptivo a las carpetas y los nombres de cada archivo, de modo que podamos analizar estas rutas de acceso de archivo más adelante para establecer los metadatos de cada ejemplo. Ejecute la importación de los archivos .d de Bruker y guárdelos en una celda de código de lista. Esto generará el número total de archivos .d cargados.
- Ejecute la celda de código titulada Procesamiento de los datos sin procesar y guardado en imzML. Esto leerá cada archivo .d de Bruker proporcionado, realizará la selección de picos en los datos y luego guardará 20 ppm de datos alrededor de cada pico en un archivo .imzML escrito dentro del archivo .d.
- Para encontrar los datos guardados, abra la ubicación del primer archivo .d y el imzML y otros metadatos se guardarán en esta ruta. En esta etapa, el archivo imzML guardado se puede importar al software del proveedor, como SCiLS Lab MVS. Para un análisis de datos más avanzado, continúe siguiendo el script.
- Cargue los datos de imzML guardados ejecutando la carga en la celda de código de archivo imzML guardada anteriormente. De este modo, se cargan los datos guardados en los datos variables.
- Ejecute la celda Agrupar los datos y guardar la matriz de datos para agrupar todos los datos a lo largo de un eje m/z común, eliminar las entidades que están presentes en baja abundancia (<0,1%) y eliminar los valores inferiores al ruido (intensidad relativa 100). A continuación, esta función vuelve a agrupar los datos promediando cada valor m/z máximo dentro de cada agrupación. La matriz de datos resultante se guarda como datacube_array, las agrupaciones m/z asociadas a la matriz se guardan como valid_mz_bins y un recuento de cada ubicación se guarda como recuento.
- Para anotar los datos, ejecute la celda de anotación Exportación de picos para MAPAS LIPID. Esto copiará todos los picos m/z dentro de su matriz de datos a su portapapeles. Pega esto en un archivo de texto vacío y guárdalo para subirlo a LIPID MAPS.
- Abra lipidmaps.org y, a continuación, seleccione Búsqueda masiva de MS Data y Búsqueda LMSD para buscar especies de lípidos biológicamente relevantes. Cargue la lista de picos eligiendo el archivo de texto y seleccionando los aductos apropiados (por ejemplo, [M+H]+, [M+Na]+ y [M+K]+). Seleccione la tolerancia de masa adecuada para su espectrómetro de masas (elegimos +/- 0,01 m/z), seleccione todas las clases de lípidos de interés y pulse Buscar. Descargue el archivo .tsv.
- Cargue los metadatos de los espectros ejecutando la carga en las rutas de acceso de una sola celda para las celdas de metadatos . Modifique el código dividido aquí en función de las estructuras de ruta de acceso del archivo.
- Preprocese los datos ejecutando la celda Preprocesamiento de datos y anotación de lípidos . Esto usará el archivo lipidMAPS tsv para anotar el conjunto de datos con un error de 10 ppm, eliminar los lípidos de cadena impares de los resultados de lipidMAPS, normalizar los datos restantes y eliminar las características o celdas que no superen un umbral específico. Esto guardará el nuevo conjunto de datos procesado como data2 y los metadatos de espectros actualizados como updated_cell_types.
- El análisis de los datos se realiza mediante scanpy. Ejecute la celda UMAP y Clustering Analysis for Single Cells para seleccionar los datos de interés (aquí seleccionamos solo los datos MALDI-2, M2), escale el conjunto de datos y cárguelo en un objeto AnnData pequeño para facilitar los análisis.
NOTA: Aquí, se realizó la agrupación de Leiden y la reducción de la dimensionalidad (UMAP) en el conjunto de datos, se visualizaron tanto las clases de datos originales como los clústeres en el UMAP, y algunas de las características estadísticamente más significativas entre los clústeres se resumieron utilizando la función rank_genes_groups_dotplot de scanpy.
Resultados
En la Figura 1 se muestra una descripción general del flujo de trabajo para la EM MALDI-2 de una sola célula guiada por fluorescencia. En primer lugar, el tejido diseccionado de las regiones cerebrales objetivo (Figura 1A) se disocia en células individuales y se deposita en portaobjetos de microscopía conductores recubiertos de ITO (Figura 1B). Las ubicaciones de las células se determinan mediante imágenes de fluorescencia de portaobjetos completos (Figura 1C), seguidas de la aplicación de la matriz MALDI (Figura 1D), el análisis de MALDI-2 MS asistido por microMS (Figura 1E) y el análisis de datos (Figura 1F). Con este flujo de trabajo, se pueden identificar de forma putativa de decenas a cientos de lípidos dentro de células individuales con una precisión de masa de menos de 10 ppm (utilizando un timsTOF de MALDI-2).
Las imágenes de espectrometría de masas se pueden realizar en una pequeña región de interés para evaluar la calidad de la preparación celular y la aplicación de la matriz. En la Figura 2A se muestra un resultado representativo de las imágenes de células dispersas con MALDI-2. A partir de esta imagen, el usuario puede evaluar el grado de dispersión del analito en comparación con una fotomicrografía de la misma región. En función de la cantidad de propagación, el analista puede ajustar el tamaño del campo del rayo láser y el filtro de distancia para garantizar verdaderas adquisiciones de una sola célula. En MSI de una sola célula, se utilizará un tamaño de láser pequeño y un ancho de trama para obtener una resolución celular (1-5 μm), mientras que un tamaño de láser más grande se puede usar para microMS, lo que aumenta la intensidad de la señal y el número de lípidos detectables en relación con las imágenes. En la Figura 2B se muestran los resultados representativos de la mejora de la señal obtenida con MALDI-2. Se observa un aumento de las especies de colesterol, PE y PC en concordancia con la literatura previa14. En general, obtenemos la mejor mejora de la señal MALDI-2 cuando utilizamos una potencia láser relativamente alta (>50%) y un número menor de disparos (alrededor de 10 a 200). Es necesaria cierta optimización, ya que una potencia láser demasiado baja no proporcionará una mejora, mientras que una energía láser excesiva provocará una fragmentación excesiva del analito29. Para el análisis de una sola célula, el registro espacial es un paso crítico para garantizar una orientación precisa de las células individuales. Para validar el registro, se puede generar un pequeño conjunto de puntos de prueba (~5) alrededor del portaobjetos creando blobs en puntos conocidos y exportando las coordenadas del instrumento a través de microMS. La verificación de que estos puntos de prueba colocan correctamente la etapa del instrumento cuando se seleccionan garantiza que la orientación posterior de las celdas sea precisa.
MALDI MS permite perfilar la heterogeneidad celular tanto entre como dentro de las regiones cerebrales individuales (Figura 3). Al analizar las variaciones en los perfiles lipídicos, las técnicas de reducción de dimensionalidad separan eficazmente las células en grupos distintos, destacando la diversidad de las composiciones lipídicas en todo el conjunto de datos (Figura 3A). Además, la aplicación de técnicas de agrupamiento, como el agrupamiento de Leiden, muestra la presencia de subpoblaciones únicas de células dentro de regiones específicas del cerebro, incluidos el cuerpo estriado y la corteza (Figura 3B). Además, al comparar los perfiles lipídicos de cada conglomerado entre sí (Figura 3C), los lípidos específicos se pueden utilizar como marcadores para definir las identidades específicas del conglomerado. Varias especies de lípidos están significativamente reguladas al alza o a la baja en grupos específicos, lo que sugiere roles funcionales o estados distintos, mientras que otras se comparten entre grupos pero varían en su intensidad de señal relativa. Para garantizar un análisis sólido de los datos posteriores, es esencial lograr una detección fiable de lípidos en la mayoría de las células y evaluar los posibles efectos de los lotes. La Figura 3D presenta espectros representativos de seis células corticales individuales dispersas en portaobjetos separados recubiertos de ITO. Si bien los perfiles lipídicos individuales muestran heterogeneidad, los lípidos se detectan de manera consistente en todas las adquisiciones. La Figura 3E muestra un UMAP de células corticales de diferentes portaobjetos, preparado y analizado en condiciones idénticas. La superposición sustancial de células entre los portaobjetos indica que los efectos de lote no confunden significativamente las interpretaciones biológicas. Si es necesario, se pueden aplicar algoritmos de corrección de lotes como ComBat30 para mitigar los efectos residuales. En resumen, MALDI-2 unicelular es una técnica avanzada para la identificación de biomarcadores lipídicos, que ofrece información crítica sobre las subpoblaciones celulares y mejora nuestra comprensión de la diversidad regional y funcional en el cerebro.
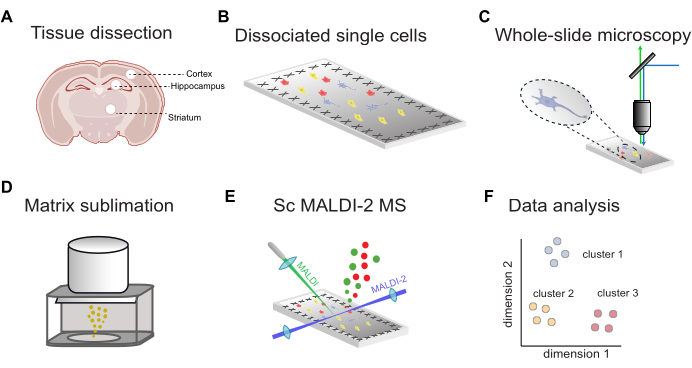
Figura 1: Flujo de trabajo general para scMALDI-2 MS. (A) Disección manual de tejidos y aislamiento de regiones cerebrales. (B) Disociación de papaína del tejido en células individuales y deposición de células en portaobjetos conductores recubiertos de ITO. (C) Microscopía de fluorescencia de portaobjetos completos mediante tinción nuclear (DAPI/Hoechst). (D) Sublimación de matriz MALDI (DHAP) en portaobjetos de muestra. (E) Mediciones de MS de MALDI-2 de una sola célula guiadas por imágenes. (F) Análisis integral de datos, incluida la reducción de la dimensionalidad, la agrupación y el análisis estadístico. Haga clic aquí para ver una versión más grande de esta figura.
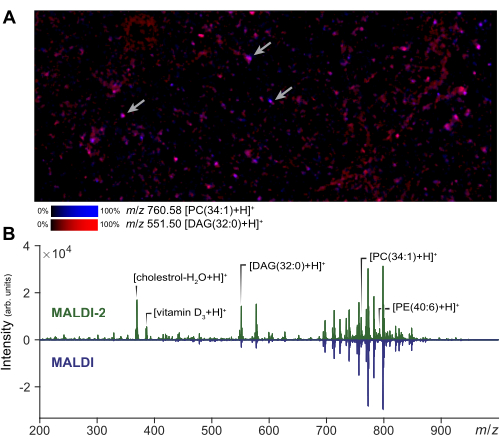
Figura 2: Optimización de MALDI-2 MS e imágenes para análisis de células individuales. (A) Imágenes de MALDI-2 MSI de poblaciones dispersas de células primarias. Las flechas indican algunas celdas individuales. La realización de MSI y una región pequeña permite al analista optimizar los parámetros del método y evaluar la calidad de la muestra. (B) Comparación de los resultados obtenidos utilizando las adquisiciones de MALDI-2 y MALDI MS. La mejora de la señal (MALDI 2 vs MALDI) de varios lípidos, incluidos el colesterol, DAG (diacilgliceroles), PC (fosfatidilcolina) y PE (fosfatidiletanolamina), se destaca mostrando el espectro MALDI-2 (arriba) y el espectro MALDI (abajo). Haga clic aquí para ver una versión más grande de esta figura.
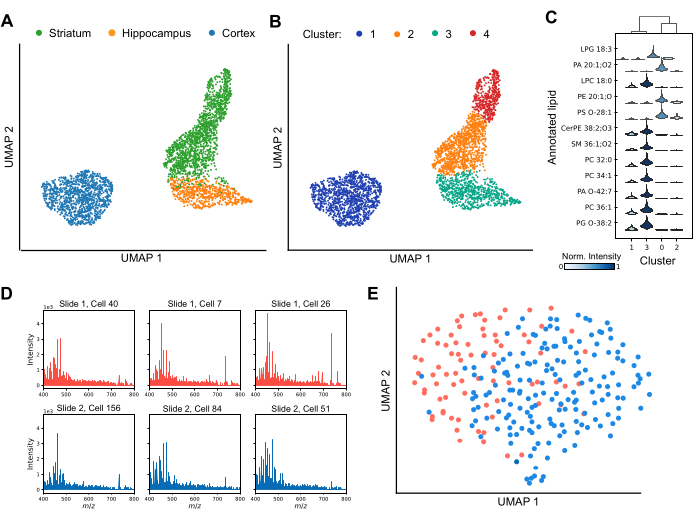
Figura 3: Perfil lipídico de una sola célula dentro de tres regiones cerebrales de roedores mediante MALDI-2 MS. (A) análisis de aproximación y proyección de variedades uniformes (UMAP) de las células analizadas coloreadas por región cerebral de origen. (B) Análisis UMAP de las mismas células después de someterse a la agrupación de Leiden, donde los grupos corresponden a subpoblaciones de células con composiciones lipídicas similares. Los colores aquí muestran diferentes clústeres de datos de Leiden. (C) Un diagrama de violín que muestra la diferencia en las distribuciones de intensidad para diferentes lípidos entre grupos. (D) Los espectros de seis células corticales individuales representativas se analizaron a partir de dos portaobjetos separados. (E) Análisis UMAP de células corticales analizadas a partir de dos portaobjetos separados. Haga clic aquí para ver una versión más grande de esta figura.
Legajo Complementario 1: SC-MALDI2_Analysis.py. Este archivo contiene un conjunto de funciones desarrolladas para el procesamiento de datos de espectrometría de masas MALDI-2 de una sola celda. Incluye métodos para leer datos brutos de espectrometría de masas, realizar agrupaciones y realizar anotaciones y análisis posteriores. El conjunto de celdas para cargar y realizar todos los preprocesamientos y análisis de datos se proporciona en el Archivo Complementario 2. Haga clic aquí para descargar este archivo.
Fichero complementario 2: JOVE Analysis.ipynb. Este archivo contiene un cuaderno de Jupyter Notebook que contiene un conjunto de celdas para cargar en las funciones personalizadas de Python desde SC-MALDI2_Analysis.py y realizar todos los análisis y preprocesamiento de datos mencionados. Haga clic aquí para descargar este archivo.
Discusión
La EM MALDI de una sola célula guiada por imágenes de alto rendimiento es una herramienta valiosa para comprender la heterogeneidad química a escala de una sola célula. La adición de postionización inducida por láser (MALDI-2) proporciona una cobertura de analito más profunda, que es fundamental para muestras de masa y volumen limitados, como las células de mamíferos aisladas.
Si bien la gran mayoría de los flujos de trabajo publicados de EM de lípidos y metabolitos de una sola célula utilizan células cultivadas, nuestro enfoque se aplica a células primarias aisladas con relativa rapidez. Esta distinción es fundamental, ya que las células cultivadas muestran un cambio demostrable en su contenido de metabolitos y lípidos en tan solo unos días de cultivo23,31. El aislamiento enzimático de las células, en la mayoría de los casos, conduce a la pérdida de estructuras celulares finas como axones, dendritas y procesos astrocíticos. Por lo tanto, las señales de analito observadas se obtienen principalmente del soma celular. A pesar de esto, se logra una clasificación exitosa por tipo de célula4 y región cerebral12. Esto sugiere que muchos de los lípidos que son exclusivos de distintas células se encuentran en el soma.
Pequeñas diferencias en el método de aplicación de la matriz MALDI pueden tener efectos profundos en la señal de analito resultante. Hemos presentado un flujo de trabajo basado en la sublimación de matriz reproducible con un aparato comercial, que genera una capa de matriz homogénea, alta relación señal-ruido y dispersión limitada del analito. Sin embargo, se puede utilizar la aplicación matricial mediante un pulverizador robótico o un aerógrafo de artista, entre otros enfoques. Independientemente del enfoque, se debe aplicar una densidad de matriz más baja a las preparaciones de una sola célula en comparación con la utilizada en la MSI de tejido estándar para lograr la relación matriz óptica / analito. La densidad de la matriz aplicada aquí es aproximadamente la mitad de la utilizada para la MSI de tejido. Por supuesto, esto implica que la cantidad de matriz por célula es mayor que en MSI, lo que ayuda a contribuir a la mejora de la extracción y detección de analitos en el enfoque de células aisladas utilizado aquí.
En el caso de las muestras que requieren una optimización significativa, la realización de MSI en un pequeño ROI de disociaciones de una sola celda, como se muestra en la Figura 2A, se puede realizar como parte del proceso de optimización. Este paso también puede permitir al analista evaluar el grado de dispersión del analito en los enfoques de aplicación de matrices basadas en solventes.
El éxito de la MALDI guiada por imágenes de células individuales requiere imágenes de fluorescencia de alta calidad, una aplicación de matriz reproducible y un control preciso de la etapa. Estos experimentos son más exigentes que los flujos de trabajo estándar de MSI, que toleran errores de registro menores y variabilidad en las condiciones de la matriz y la muestra debido a áreas de imagen más grandes y recuentos de analitos más altos. A pesar de estos desafíos, hemos atacado con éxito orgánulos individuales utilizando MALDI tradicional.
La unión precisa de las imágenes de fluorescencia en mosaico es esencial, ya que los pequeños errores pueden agravarse y afectar el control de la etapa, lo que hace que la precisión del registro sea crítica. Los efectos de lote también presentan un desafío importante, ya que se observan variaciones de señal en el mismo portaobjetos, entre portaobjetos y entre preparaciones. Las estrategias de normalización adecuadas, incluido el uso de estándares internos y la normalización intercuartílica, ayudan a mitigar estos problemas. Además, los algoritmos de corrección de lotes, como ComBat30 , se pueden utilizar para reducir la variabilidad técnica y mejorar las diferencias biológicas reales.
La obtención de anotaciones moleculares fiables a partir de células individuales sigue siendo un reto. A menudo, la cantidad de material y la sensibilidad de la detección son insuficientes para la EM en tándem de todas las especies, excepto las más abundantes. Para aquellos que no son lo suficientemente detectables para la EM en tándem directamente desde células individuales, se pueden aplicar enfoques alternativos para crear una biblioteca de analitos. Por ejemplo, los datos de EM en tándem en tejido se pueden obtener a partir de criosecciones delgadas de la misma región del cerebro y animales utilizados para generar poblaciones celulares. También se puede emplear la extracción de lípidos del tejido seguida de LC-MS. A medida que las estrategias de preparación de muestras y la tecnología de EM continúan mejorando, se puede obtener información estructural cada vez más relevante directamente de células individuales. En el futuro, anticipamos que este flujo de trabajo se extenderá para obtener datos de MS en tándem de celdas individuales, eliminando la necesidad de experimentos auxiliares de LC-MS/MS. Además, prevemos que este enfoque podría extenderse a numerosas muestras a microescala en biología y más allá, incluidos tipos de células únicos, orgánulos de mamíferos, polvos y microplásticos.
Divulgaciones
Los autores no tienen intereses contrapuestos que revelar.
Agradecimientos
S.W.C agradece el apoyo brindado por la beca Peixin He y Xiaoming Chen PhD4 y la beca Block Grant de la Universidad de Illinois. Este trabajo también fue apoyado por el Instituto Nacional sobre el Abuso de Drogas en virtud del laudo No. P30DA018310, el Instituto Nacional sobre el Envejecimiento (National Institute on Aging) R01AG078797, y por la Oficina del Director de los Institutos Nacionales de Salud bajo el Premio Número S10OD032242.
Materiales
| Name | Company | Catalog Number | Comments |
| 2',5'-dihydroxyacetophenone | Sigma Aldrich | D107603 | DHAP, 97% purity |
| Ammonium acetate | Sigma Aldrich | 238074 | ACS reagent, ≥97% |
| Axio M2 Imager | Zeiss | N/A | N/A |
| Biopsy punch, 2 mm | Fisher Scientific | 12-460-399 | integra miltex standard biopsy punch, 2mm |
| Calcium chloride | Sigma Aldrich | C4901 | anhydrous, powder ≥97% |
| Eppendorf Centrifuge | Sigma Aldrich | EP5405000441 | centrifuge 5425 with rotor FA-24x2 |
| Gentamicin | Sigma Aldrich | G1272 | liquid, BioReagent |
| Glass etching pen | Sigma Aldrich | Z225568 | carbide time, pkg of 1 |
| Glycerol | Sigma Aldrich | G7893 | ACS reagent, ≥99.5% |
| HEPES buffer | Sigma Aldrich | H3375 | ≥99.5% (titration) |
| Hoechst 33258 Solution | Sigma Aldrich | 94403 | 1 mg/mL in H2O, ≥98.0% (HPLC) |
| In line HEPA Filter | Sigma Aldrich | WHA67225001 | VACU-GUARD 60 mm disc, 0.45 PFTE housing |
| ITO-Coated Microscopy Slides | Delta Technologies | CG-90IN-S115 | 70-100Ω resistance |
| Magnesium chloride | Sigma Aldrich | M8266 | anhydrous, ≥98% |
| Magnesium sulfate | Sigma Aldrich | 208094 | anhydrous, ≥97% |
| Microcentrifuge tubes | Sigma Aldrich | HS4323K | tube capacity 1.5 mL, pack of 500 |
| Papain dissociation system | Worthington Biochemical | LK003150 | one box, 5 single use vials |
| Penicillin-Streptomycin | Sigma Aldrich | P4458 | liquid, BioReagent |
| Potassium chloride | Sigma Aldrich | 529552 | Molecular biology grade |
| Potassium phosphate monobasic | Sigma Aldrich | P5379 | Reagent Plus |
| Sodium biocarbonate | Sigma Aldrich | S6014 | ACS reagent, ≥99.7% |
| Sodium chloride | Sigma Aldrich | S9888 | ACS reagent, ≥99% |
| Sodium hydroxide | Sigma Aldrich | 221465 | ACS reagent, ≥97%, pellets |
| Sodium phosphate dibasic | Sigma Aldrich | S9763 | ACS reagent, ≥99% |
| Sublimate | HTX | N/A | N/A |
| timsTOF FleX MALDI-2 | Bruker | N/A | microGRID enabled |
| Vacuum tubing | Thermo Scientific | 8701-0080 | Nalgene Non-phthalate PVC Tubing |
Referencias
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados