Method Article
امتصاص / تأين الليزر بمساعدة المصفوفة المضان مع قياس الطيف الكتلي للتأين الناجم عن الليزر للخلايا العصبية الفردية للفئران
* These authors contributed equally
In This Article
Summary
يحدد هذا البروتوكول استخدام microMS لقياس الطيف الكتلي MALDI-2 أحادي الخلية الموجه بالفلورة ، مما يتيح التنميط الجزيئي المعزز للخلايا العصبية الأولية للفئران.
Abstract
تعد قياسات الخلية المفردة أمرا بالغ الأهمية لفهم عدم التجانس الكيميائي المكاني الغني للدماغ. قياس الطيف الكتلي بالليزر / التأين بمساعدة المصفوفة (MALDI) قادر على توصيف الجزيئات الداخلية في الخلايا الفردية الخالية من الملصقات وعالية الإنتاجية. توفر التطورات الحديثة في تطوير مطياف الكتلة MALDI مع ما بعد التأين الناجم عن الليزر (MALDI-2) حساسية محسنة بشكل كبير للكشف عن مجموعة متنوعة من الدهون والجزيئات الصغيرة الأخرى. ومع ذلك ، فإن تصوير التصلب العصبي المتعدد للعينات الكبيرة باستخدام MALDI-2 بدقة خلوية بطيء للغاية بالنسبة لمعظم التطبيقات. في هذا البروتوكول ، يتم عزل الخلايا الأولية وتشتيتها على شرائح موصلة. يتم تحديد مواقع الخلايا النسبية عن طريق الفحص المجهري الفلوري كامل الشريحة ، متبوعا بتسجيل مشترك دقيق لإحداثيات الفحص المجهري لإحداثيات المرحلة لمطياف الكتلة MALDI-2. يوفر تحليل MS المستهدف لمواقع الخلايا فقط قياسات عالية الإنتاجية وحيدة الخلية مع تغطية تحليلية عالية وحجم بيانات أقل مقارنة بتصوير MS للعينة بأكملها. نصف الخطوات الحاسمة اللازمة لإعداد الخلية الواحدة ، والتصوير الفلوري للشريحة الكاملة ، وتطبيق المصفوفة ، وقياس الطيف الكتلي MALDI-2.
Introduction
تعتبر الدهون والمستقلبات أساسية للوظيفة الخلوية وتعمل كمكونات أساسية للأغشية ومصادر الطاقة وجزيئات الإشارات1،2. ومع ذلك ، يمكن أن يختلف تكوينها ووفرة اختلافها بشكل كبير بين الخلايا الفردية ، مما يعكس الاختلافات في أنواع الخلايا والحالات التنمويةوالوظيفية 3،4،5. يعد تحليل هذه الاختلافات أمرا بالغ الأهمية لفهم التباين البيولوجي وتحديد مجموعات الخلايا الفرعية المتميزة. توفر تقنيات قياس الخلية المفردة ، مثل تسلسل الحمض النووي الريبي ، ملفات تعريف نسخ مفيدة خاصة بالخلية6. ومع ذلك ، فإن هذه القياسات على مستوى النسخ لا تعكس بشكل مباشر الكميات الخلوية الفعلية للدهون والمستقلبات ، حيث لا يرتبط التعبير الجيني دائما بالوفرة الفعلية لهذه التحليلات. لذلك ، هناك حاجة إلى طرق متخصصة للقياسات المباشرة للدهون والمستقلبات للتحليل الشامل للتركيب الكيميائي للخلايا المفردة وأعدادها.
يعد التصوير اللطيفي الكتلي (MSI) لامتصاص / تأين الليزر بمساعدة المصفوفة (MALDI) أداة مفضلة لرسم الخرائط المكانية الخالية من الملصقات للجزيئات الحيوية الداخلية في الموقع7،8. عادة ، مع MALDI ، يتم استخدام ليزر الأشعة فوق البنفسجية لاستئصال المواد من طبقة عينة رقيقة متبلورة مع مصفوفة عضوية ، وتشكيل عمود من الأيونات والجزيئات المحايدة. ثم يتم فصل الأيونات المتكونة بواسطة محلل MS واكتشافها داخل مطياف الكتلة. نظرا لتحديد المواقع الدقيقة لمراحل مطياف الكتلة الحديثة ، يمكن وضع الليزر لاستهداف مناطق عينة معينة أو تنقيطه عبر المناطق لإنشاء صور جزيئية بواسطة MSI. يمكن استخدام MSI بدقة مكانية خلوية أو تحت خلوية (< 10 ميكرومتر) ، والتي يتم تحقيقها باستخدام شعاع ليزر مركز وحركة مرحلية دقيقة ، للحصول على معلومات كيميائية حول الخلايا الفردية9،10. ومع ذلك ، فإن قياسات MSI على هذا المقياس غير فعالة ، خاصة في حالة العينات ذات الكثافة الخلوية المستهدفة المنخفضة ، بسبب مقدار الوقت المستغرق في تصوير المناطق الفارغة بين الخلايا. علاوة على ذلك ، فإن قابلية الكشف عن العديد من التحليلات محدودة بسبب الحجم الصغير الذي تم أخذ عينات منه. للتغلب على هذه التحديات ، قمنا بتطوير نهج MALDI MS الموجه بالصور لتحليل الخلية المفردة عاليالإنتاجية 11،12. في هذا النهج ، يتم تحديد مواقع الخلايا المشتتة برمجيا من صور مضان الشريحة الكاملة واستخدامها لتوجيه مرحلة مطياف الكتلة إلى المواضع التي توجد فيها الخلايا الفردية ذات المعلمات المحددة (على سبيل المثال ، الحجم والشكل) ثم يتم تحليلها عن طريق التشعيع باستخدام ليزر MALDI. تم استخدام العمل السابق باستخدام هذا النهج المستهدف لتوصيف الدهون والببتيدات والجزيئات الحيوية الأخرى في مجموعات الخلايا غير المتجانسة5،13.
نظرا للطبيعة المحدودة للكتلة للخلايا المفردة ، فإن عدد التحليلات المكتشفة في هذه العينات أقل بشكل عام من تلك التي لوحظت مباشرة من الأنسجة. لذلك ، لزيادة تغطية التحليل في تحليل MS أحادي الخلية ، تعد زيادة حساسية الكشف عن التحليل أمرا بالغ الأهمية. أحد الأساليب التي تم تطويرها مؤخرا والتي تساعد في التغلب على تحدي الكشف هذا هو MALDI مع الليزر بعد التأين (MALDI-2) ، والذي ثبت أنه يعزز الحساسية لمجموعة واسعة من التحليلات9،14،15،16. نتيجة لذلك ، يولد MALDI-2 مجموعات بيانات أكثر شمولا من خلية واحدة ويوفر تغطية جزيئية أعمق في العينات المحدودة الكتلة ، مثل الخلايا المعزولة.
الهدف من هذه الطريقة هو الحصول على قياسات الدهون من آلاف الخلايا الفردية. تحقيقا لهذه الغاية ، نصف سير العمل الذي يتيح قياس الطيف الكتلي أحادي الخلية MALDI-2 عالي الإنتاجية ويمكن تمديده بشكل عام إلى أي نهج قياس الطيف الكتلي القائم على المسبار مع التحكم الدقيق في المرحلة17. في سير العمل هذا ، يتم تشريح الأنسجة من منطقة (مناطق) الدماغ ذات الأهمية ، ويتم الحصول على الخلايا الفردية من الأنسجة بعد إجراء تفكك غراء. ثم يتم تمييز الخلايا ببقعة نووية ويتم تشتيتها على شرائح زجاجية موصلة محفورة بعلامات إيمانية ، حيث يسمح لها بالالتصاق. بعد ذلك ، يتم التقاط صور الشريحة الكاملة باستخدام المجهر الفلوري. يتم ترسيب المصفوفة عن طريق التسامي ، مما يولد طبقة بلورية متجانسة قابلة للتكرار وإشارة عالية إلى ضوضاء لتحليل MS أحادي الخلية. باستخدام البرنامج مفتوح المصدر microMS11 ، يتم تعيين الإحداثيات النسبية لمواقع الخلايا من صورة الفحص المجهري ومحاذاتها مع إحداثيات مرحلة مطياف الكتلة عن طريق تسجيل مجموعة النقاط باستخدام علامات إمانية محفورة على الشرائح الزجاجية حيث تم إيداع الخلايا. أخيرا ، باستخدام هذه المعلومات ، يتم الحصول على أطياف MS الدقيقة والمستهدفة من كل خلية على حدة ، مما يسمح بتحديد الآلاف من الخلايا في تشغيل واحد (<1 ساعة) 12،13.
Protocol
أجريت جميع التجارب على في هذه الدراسة وفقا لبروتوكول استخدام المعتمد من قبل لجنة إلينوي المؤسسية لرعاية واستخدامه (23228) مع الالتزام الصارم بالمعايير الوطنية ومعايير ARRIVE للمعاملة الأخلاقية للحيوانات ورعايتها.
1. تحضير المواد والحلول
- تحضير محلول الملح المتوازن Gey المعدل (mGBSS) مع 1.5 ملي CaCl2 ، 4.9 ملي كلوريد كلوريد ، 0.2 ملي كلوريد كلوريد2 PO4 ، 11 ملي ملي كلوريد2 ، 0.3 ملي مللي كلوريدالصوديوم 4 ، 138 ملي كلوريد الصوديوم ، 27.7 ملي هيدروكسيدالصوديوم 3 ، 0.8 ملي سنوميوم2HPO4 ، و 25 ملي هيبس. اضبط الرقم الهيدروجيني إلى 7.2 باستخدام 3 M هيدروكسيد الصوديوم. يوصى بتحضير 1 لتر من mGBSS وتخزينه في درجة حرارة 4 درجات مئوية حتى الاستخدام.
- قم بإعداد محاقن سعة 50 مل عن طريق ملئها بمحلول mGBSS المحضر ووضعها على الثلج من أجل نضح القلب. تحضير 1 حقنة لكل فأر تشريح.
- قم بإعداد حصص من محلول غراء باتباع بروتوكول نظام تفكك غراء ورثينجتون. يحتوي هذا النظام على أربع قوارير كما هو موضح أدناه ، مع عدم استخدام القارورة 4 لهذا البروتوكول:
قارورة 1: محلول ملح إيرل المتوازن المعقم (EBSS) مع البيكربونات والفينول الأحمر
قارورة 2: غراء مع L-cysteine و EDTA
قارورة 3: ديوكسيربونوكلاز I (DNase)
قارورة 4: مثبط البروتياز الإبيضي مع ألبومين مصل الأبقار- أضف 5 مل من EBSS (قارورة 1) إلى قارورة غراء (قارورة 2) وضعها في حمام مائي 37 درجة مئوية لمدة 10 دقائق لإذابة الغراء.
- أضف 500 ميكرولتر من EBSS (قارورة 1) في قارورة DNase (قارورة 3) واخلطها برفق. أضف 250 ميكرولتر من قارورة DNase المخففة (قارورة 3) في قارورة الغراء (القارورة 2). يحتوي هذا المستحضر على تركيز نهائي يبلغ 20 وحدة / مل من غراء ، و 0.005٪ DNase ، و 0.5 ملي مولار EDTA ، و 1 ملي مولار L-cysteine.
- قم بتجميد 250 ميكرولتر من محلول غراء المحضر هذا للتفكك اللاحق (يجعل ~ 20 حصة). استكمل هذا المحلول بالمضادات الحيوية التالية: 100 وحدة / مل بنسلين G ، 100 ميكروغرام / مل ستربتومايسين ، و 100 ميكروغرام / مل جنتاميسين. يمكن تعديل هذا لتطبيق المستخدم.
- قم بإعداد محلول أسيتات الأمونيوم 150 ملي مولار وتخزينه عند 4 درجات مئوية. قم بإعداد محلول الجلسرين بنسبة 33٪ عن طريق تخفيف الجلسرين في محلول mGBSS المحضر.
- قم بحفر الحسابات على شرائح الفحص المجهري المطلي بأكسيد القصدير الإنديوم (ITO) باستخدام قلم نقش مع كربيد التنجستن أو طرف الماس أو آلة نقش ليزر CO2 . على الجانب الموصل من الشريحة الزجاجية ITO ، قم بحفر 20-40 علامة إيمانية على شكل X على طول محيط الشريحة لتمكين التسجيل المكاني عبر الأنظمة الأساسية (على سبيل المثال ، المجهر ومطياف الكتلة).
ملاحظة: احرص على عدم الحفر في خط متصل واحد يحيط بالعينة أو مناطقها لأن هذا يعطل توصيل السطح بين المنطقة ومحول الشريحة الزجاجية.
2. تحضير الخلايا العصبية الأولية
ملاحظة: يتم تشريح أنسجة الحصين في الفئران ، وتفكيكها إلى خلايا فردية مع غراء ، وترسبها على شرائح زجاجية موصلة بكثافة منخفضة. يتيح عزل الخلايا بهذه الطريقة قياس طيف كتلة الخلية المفردة عالي الإنتاجية للدهون الداخلية.
- قبل تشريح ، قم بإذابة جزء من محلول غراء المحضر وضعه في وعاء به أنبوب مدخل ومخرج يسمح لثاني أكسيد الكربون2 بإزاحة الهواء داخل الحاوية. ضع هذا النظام في حمام مائي مضبوطة على 37 درجة مئوية واستمر في تدفق ثاني أكسيد الكربون2 حتى يصبح المحلول هو الأس الهيدروجيني المناسب ، كما هو موضح في اللون ، باستخدام مخطط ألوان الأس الهيدروجيني المضمن في حزمة نظام تفكك غراء ورثينجتون. يتراوح الرقم الهيدروجيني المستهدف بين 7.2 و 7.4.
ملاحظة: لا تضع ثاني أكسيد الكربون2 في المحلول الأنزيمي ، لأن هذا سيؤدي إلى تمسخ DNase وفقدان النشاط البيولوجي. استخدم أنبوب طرد مركزي سعة 50 مل مع أنبوبين ملتصقين في الغطاء ، مما يسمح لثاني أكسيد الكربون2 بملء الأنبوب من الأسفل والهروب من أنبوب المخرج العلوي. - القتل الرحيم للفئران (الفئران) وفقا لبروتوكولات IACUC باتباع المبادئ التوجيهية المؤسسية والمحلية والفيدرالية. هنا ، يتم استخدام ذكور فئران Sprague-Dawley البالغة من العمر 2 إلى 3 أشهر والتضحية بها عن طريق اختناق ثاني أكسيد الكربون2 .
- قم بنشر الفئران عبر القلب بمحلول مثلج بارد (4 درجات مئوية) mGBSS لإزالة الدم من الأوعية الدموية وتبريد جسم بسرعة. يمكن العثور على أحد الأمثلة على هذا الإجراء في البروتوكول بواسطة We et al.18.
- عزل الدماغ جراحيا عن الفئران ، مع التأكد من عدم إتلاف منطقة الدماغ ذات الاهتمام. توفر نظرة عامة منشورة سابقا على هذه العزل مزيدا من التفاصيل حول بعض الخطوات الفردية19.
- استخدم مفرمة الأنسجة أو مكعب الأنسجة لقطع مقطع عرضي (~ 2 مم) من الدماغ لكشف المنطقة (الحصين أو القشرة أو المخطط) لأخذ العينات.
- استخدم لكمة خزعة 2 مم لأخذ عينات من منطقة الدماغ ذات الاهتمام وإيداع قطعة الأنسجة في قارورة محلول غراء.
- احتضان هذا النسيج ومحلول غراء عند 37 درجة مئوية تحت التحريك المستمر (على سبيل المثال ، بواسطة هزاز كهربائي) لمدة 30-90 دقيقة.
ملاحظة: يجب تحسين مدة العلاج الأنزيمي بناء على منطقة الدماغ ، وسلالة ، والعمر ، وإخراج الخلية المطلوب (على سبيل المثال ، الخلايا العصبية والخلايا النجمية ذات الأطراف مقابل أقصى إنتاجية للخلايا بدون أطراف). بالنسبة لأنسجة الحصين في الفئران ، تكون 60-90 دقيقة نموذجية. قد تحافظ العلاجات الأقصر على بعض الأطراف ولكنها تترك العديد من الخلايا في مجموعات غير منفصلة ، في حين أن العلاجات الأطول يمكن أن تحسن المحصول ولكنها تزيد من تلف الخلايا ، مما يتطلب تقييما باستخدام فحوصات حية / ميتة (على سبيل المثال ، مجموعة البقاء / السمية الخلوية LIVE / DEAD). بالنسبة للدراسات المقارنة ، يجب أن تكون العلاجات متسقة عبر أنواع العينات. - ضع قارورة من الأنسجة والغراء على الثلج لتبريد وقمع النشاط الأنزيمي. قم بتقطيع الأنسجة باستخدام طرف ماصة مقطوع متصل بماصة حجم قابلة للتعديل P100 حتى يتم تفتيت معظم الأنسجة بصريا. لا تشكل فقاعات هواء أثناء التفكك لتجنب تلف الخلايا المفرط.
ملاحظة: ينتج عن التجربة القوية إنتاجية عالية من الخلايا. ومع ذلك ، فإنهم لا يحتفظون بمعظم عملياتهم. يوفر التخلي اللطيف المزيد من العمليات على الخلايا ولكنه ينتج عنه المزيد من الخلايا غير المنفصلة. يمكن أيضا استخدام أطراف الماصات ذات الأحجام المختلفة والطرق الأخرى وتحسينها للحصول على نتائج تفكك مثالية20. - الطرد المركزي للخلايا المنفصلة عند 300 × جم لمدة 5 دقائق في درجة حرارة الغرفة لحبيبات الخلايا. قم بإزالة المادة الطافية وأعد تعليق الحبيبات في 1 مل من mGBSS. أضف صبغة Hoechst 33342 النووية إلى تركيز نهائي قدره 1 ميكروغرام / مل.
- الماصة 100-500 ميكرولتر من تعليق الخلية على شرائح زجاجية محفورة ITO والسماح لها بالجلوس لمدة 5 دقائق تقريبا أو أكثر للسماح للخلايا بالترسيب والالتصاق بالشريحة.
- يجب تحديد تخفيف تعليق الخلية تجريبيا. باستخدام المجهر البصري ، أضف قطرة من الخلايا إلى الشريحة لتحديد الكثافة الخشنة وتخفيفها من هناك للتأكد من وجود مسافة كافية بين الخلايا (أي أكبر بما فيه الكفاية من حجم شعاع MALDI). يمكن إجراء هذا التخفيف على الشريحة الأولى ، ومن ثم يمكن ضبط التخفيف حسب الضرورة للشرائح اللاحقة.
- قم بسحب تعليق الخلية برفق من الشريحة باستخدام نظام تفريغ يحتوي على (يقع بالترتيب بدءا من خط التفريغ): 1) أنبوب مزود بمرشح HEPA غاز مدمج ، 2) أنبوب مثبت على قارورة ترشيح سعة 50 مل ، 3) أنبوب متصل بمدخل قارورة ترشيح ثان بطرف ماصة متصل (على سبيل المثال ، 100 ميكرولتر). في حالة تخزين الخلايا المطلية للاستخدام في المستقبل ، استبدل المعلق المستنشق بمحلول جلسرين بنسبة 33٪ لمدة 1-2 دقيقة وقم بإزالته.
ملاحظة: ستضمن هذه الخطوة استقرار الخلية. يعد الاستغناء عن محلول الجلسرين من جانب واحد مع الشفط اللطيف من الجانب الآخر مثاليا للحفاظ على البنية الخلوية. راقب دائما بصريا جميع الخطوات الموضحة هنا باستخدام مجهر مقلوب أثناء تحضير العينة الأولى. - ضع 100-500 ميكرولتر من أسيتات الأمونيوم على المناطق المترسبة ، واتركها لمدة دقيقة واحدة تقريبا ، ثم استنشق لتنظيف الشرائح. كرر حسب الضرورة للتأكد من خلو الشرائح من أي ملح أو جلسرين.
3. الفحص المجهري
ملاحظة: لتحديد موقع الخلايا المترسبة ، يتم تصوير كل شريحة بواسطة المجهر الساطع / الفلور. تسمح قناة التألق بتحديد موقع الخلايا الملطخة ب Hoechst بدقة ، بينما يوفر التصوير الساطع معلومات مورفولوجية. أي مجهر قادر على الحصول على الصور المبلطة مناسب لهذه العملية.
- إذا لم يتم القيام به مسبقا ، اشطف الشرائح ب 2-3 مل من أسيتات الأمونيوم 150 مل. هذه الخطوة ضرورية لإزالة بلورات الجلسرين والملح ، والتي يمكن أن تتداخل مع المراقبة المجهرية وتطبيق مصفوفة MALDI. ينزلق التجفيف تحت تيار لطيف من N2 أو يترك ليجف في الهواء.
- تحميل الشريحة في مرحلة المجهر. قم بتركيز المجهر باستخدام 10 نقاط دعم على الأقل موزعة بالتساوي عبر الشريحة.
- باستخدام مرشحات مناسبة ل DAPI (الإثارة: 335-383 نانومتر ، الانبعاث: 420-470 نانومتر) والتصوير الساطع ، احصل على صورة مضان مبلطة بتكبير 5x-10x على الشريحة بأكملها ، مما يضمن التقاط العلامات الإيمانية (التي تم إنشاؤها في الخطوة 1.5).
ملاحظة: عادة ما يكون التكبير بمقدار 5x كافيا، خاصة عندما تحتاج العديد من الشرائح إلى التصوير. لحل التشكل الخلوي الدقيق ، يمكن استخدام أهداف 10x أو 20x على حساب وقت تصوير أطول بشكل تربيعي وزيادة أحجام الملفات. - قم بربط الصورة المبلطة باستخدام برنامج الفحص المجهري ، مثل ZEN (Zeiss). يعد الخياطة الدقيقة أمرا بالغ الأهمية ، حيث ستنتشر أي أخطاء في هذه الخطوة إلى خطوة التسجيل النهائية ، مما يؤدي إلى استهداف غير دقيق. للتحقق، تأكد من عدم إزاحة المربعات المتجاورة رأسيا. بالإضافة إلى ذلك ، ضع في اعتبارك الحصول على صور متجانبة مع تداخل كبير في البلاط (20٪ -30٪).
- قم بمعالجة وتصدير كل صورة كملف bigTIFF في برنامج الفحص المجهري. إذا كانت المنطقة المصورة صغيرة نسبيا وينتج عنها حجم صورة نهائي أقل من 2 غيغابايت، فاستخدم تنسيق ملف TIFF قياسي بدلا من ذلك.
4. تطبيق المصفوفة
ملاحظة: يعد تطبيق مصفوفة MALDI المتسق والمناسب أمرا بالغ الأهمية للحصول على بيانات عالية الجودة أحادية الخلية. أثناء استخدام التسامي باستخدام جهاز تجاري هنا ، يمكن أيضا إجراء تطبيق المصفوفة باستخدام بخاخ آلي12 أو البخاخة21 أو جهاز التسامي محلي الصنع22. لقد وجدنا أن المستحضرات أحادية الخلية تتطلب مصفوفة أقل من أقسام الأنسجة الرقيقة المستخدمة عادة في تصوير قياس الطيف الكتلي. لتقليل تأثيرات الدفعات ، يوصى بتطبيق المصفوفة على جميع الشرائح قيد الدراسة خلال جلسة واحدة وإيداع الخلايا من مجموعات مختلفة (على سبيل المثال ، منطقة الدماغ أو العلاج مقابل التحكم) على نفس الشريحة كلما أمكن ذلك. يعد اختيار المصفوفة أمرا بالغ الأهمية لكل من سير العمل أحادي الخلية MALDI و MALDI-2 التقليدي. بالنسبة إلى MALDI أحادية الخلية ، تم استخدام DHB23 و 9-AA24 و CHCA25 بنجاح. في MALDI-2 ، لاحظنا نحن وآخرون تحسنا كبيرا في الإشارة مع DHAP9 ، بينما تم أيضا تطبيق المصفوفات مثل NEDC16 و CHCA26 بشكل فعال.
- قم بإذابة 20 مجم من 2,5-ثنائي هيدروكسي أسيتوفينون (DHAP) في 1.5 مل من الأسيتون. ضع الشرائح في حامل غرفة التسامي وضعها في جهاز التسامي.
- قم بسحب محلول المصفوفة المذاب على رقاقة السيراميك والسماح للأسيتون بالتبخر. أغلق غرفة التسامي لإغلاقها.
- املأ غرفة التبريد بطين من الماء المثلج وضعه فوق الغرفة.
- قم بتشغيل المكنسة الكهربائية واسمح للنظام بالتوازن لمدة 5 دقائق. يجب أن يكون الضغط في النظام أقل من 40 ملي بار.
- ابدأ التسامي عن طريق تسخين الغرفة إلى 200 درجة مئوية لمدة 5 دقائق.
- قم بإزالة حمام الماء المثلج ، وقم بتحويل درجة الحرارة إلى 25 درجة مئوية ، ثم ضع غرفة التبريد فوق الغرفة. اترك النظام يسخن إلى درجة حرارة الغرفة لمدة 5 دقائق لتجنب التكثيف.
ملاحظة: يمكن إفراغ الحاوية التي تحتوي على حمام الماء المثلج وتعبئتها بالماء الساخن لتكون بمثابة المشتت الحراري. - قم بتنفيس النظام ببطء ، وافتح الغرفة ، وقم بإزالة الشرائح.
5. خلية واحدة MALDI MS
ملاحظة: يتم الحصول على بيانات MS أحادية الخلية على جهاز MALDI-2 timsTOF (timsTOF flex) باستخدام حزمة microMS مفتوحة المصدر لاكتشاف الخلايا وتوجيه مطياف الكتلة. يتطلب ذلك ترجمة مواقع بكسل الصورة البصرية للخلايا المستهدفة إلى الإحداثيات الفيزيائية لمرحلة مطياف الكتلة.
- افتح microMS وقم بالقضاء على صور الفحص المجهري bigTIFF باستخدام خيار Image Group ، حيث توجد قنوات brightfield و fluorescence.
- باستخدام أداة خيارات الكائن الثنائي كبير الحجم، حدد المعلمات المثلى لتحديدها للخلايا المستهدفة. اضبط المعلمات التالية: الحد الأقصى والحد الأدنى لحجم الكائن الثنائي كبير الحجم لتحديد مدى صغر أو حجم الكائنات الثنائية كبيرة الحجم التي يجب العثور عليها. إعداد العتبة، الذي يحدد كيفية عتبة قناة التألق للكشف عن الكائن الثنائي كبير الحجم؛ والدائرية ، التي تحدد مدى دائرية الخلايا المحددة للنظر فيها. بالنسبة للبيانات المعروضة هنا ، استخدمنا قيم المعلمات التالية: الحد الأقصى للدائرية: لا شيء. الحد الأدنى للدائرية: 0.6 ؛ العتبة: 75; الحد الأدنى للحجم: 20 ؛ الحد الأقصى للحجم: لا شيء.
- استخدم خيار البحث عن الكائن الثنائي كبير الحجم للبحث عن الكائنات الثنائية كبيرة الحجم والنافذة المنبثقة لحفظ قائمة الكائن الثنائي كبير الحجم تحت الاسم المطلوب.
- استخدم أداة تصفية المسافة لتحديد مسافة محددة تحتاجها كل خلية لتكون بعيدة عن بعضها البعض (بالنسبة لهذه البيانات ، باستخدام الفحص المجهري الفلوري 5x ، استخدمنا مرشح مسافة يبلغ 35 بكسل). هذا للتأكد من أن الخلايا بعيدة بما يكفي عن بعضها البعض لإجراء تحليل خلية واحدة. هذا الرقم بوحدات البكسل ، لذا تعرف على تحويل البكسل إلى ميكرومتر للمجهر لتحديد الرقم المثالي. استخدم حجم قطر مسبار الليزر بالإضافة إلى حوالي 20-30 ميكرومتر لحساب الأخطاء في التسجيل.
- قم بتحميل الشرائح في الجهاز باستخدام محول الشريحة MTP II. ارجع إلى الكمبيوتر باستخدام microMS ، وباستخدام تطبيق سطح المكتب البعيد ، قم بالوصول إلى كمبيوتر مطياف الكتلة.
- إذا كانت هذه هي المرة الأولى التي يتم فيها استخدام الأداة ، فتحقق من موضع XY للأداة. للقيام بذلك ، انتقل إلى إعدادات الأدوات والأداة. ستعرض النافذة المنبثقة مجموعة من الإحداثيات مع موضعيها X و Y. حدد كل نقطة من هذه النقاط المحددة في هندسة محول الشرائح II على الجهاز وقم بتحديث مواضع X و Y في نافذة microMS.
- باستخدام الكاميرا وعناصر التحكم في المسرح لمقياس طيف الكتلة ، انتقل إلى موقع يمكن التعرف عليه بسهولة على الشريحة وانسخ إحداثيات الأداة. في microMS ، ابحث عن الموقع المقابل في صورة المجهر ، وانقر بزر الماوس الأيمن على هذا الموضع ، واكتب الإحداثيات في موقع نص النافذة المنبثقة. قم بتقريب جميع الإحداثيات لأقرب عدد صحيح ، وافصل إحداثيات X و Y مع المسافة (على سبيل المثال ، 39595 -23232).
- كرر الخطوة 5.7. لتسجيل ما لا يقل عن 12 علامة إيمانية عبر الشريحة بأكملها. بعد إضافة ثلاث نقاط تسجيل ، ستتحول إحدى الدوائر إلى اللون الأحمر ، مما يشير إلى أنها الموضع الأكثر انبعادا من التسجيل. إذا حدث ذلك، فاحذف نقطة التسجيل الحالية باستخدام shift + انقر بزر الماوس الأيمن وحاول مرة أخرى.
- ضمن ملف، انتقل إلى حفظ ثم التسجيل لحفظ ملف التسجيل (.msreg).
- مع عرض الكائنات الثنائية كبيرة الحجم على الشريحة، احفظ ملف موضع الأداة (.xeo) بالانتقال إلى ملف، ثم حفظ، ثم مواضع الأداة. باستخدام برنامج سطح المكتب البعيد ، انقل ملف .xeo هذا إلى كمبيوتر الأجهزة.
- انسخ النص من ملف .xeo المخصص الذي تم إنشاؤه في ملف MTP Slide Adapter II .xeo على الأداة واحفظه. سيؤدي هذا إلى تحديث هذا الملف الهندسي بمواقع الخلايا.
- انقر فوق علامة التبويب التنفيذ التلقائي وحدد جديد... لبدء التشغيل التلقائي. اسحب عبر منطقة العينة المعروضة لتحديد الخلايا، ثم انقر بزر الماوس الأيمن لإضافتها إلى قائمة التحليل. احفظ تشغيل التشغيل التلقائي وانقر فوق بدء التشغيل التلقائي لبدء الاستحواذ. يعتمد طول الجري على مجموعة متنوعة من العوامل ، بما في ذلك عدد الخلايا ولقطات الليزر وتردد الليزر ، ولكن بشكل عام ، يمكن تحليل ما يقرب من 5-10 خلايا في وضع MALDI (معدل تكرار الليزر 10 كيلو هرتز) ، ويمكن تحليل 1-5 خلايا في وضع MALDI-2 (معدل تكرار الليزر 1 كيلو هرتز) كل ثانية.
6. معالجة البيانات
ملاحظة: حزم البرامج التجارية الحالية ليست مناسبة تماما لتحليل بيانات قياس الطيف الكتلي أحادي الخلية عالية الإنتاجية. بينما يمكن تصور الأطياف الفردية ، فإن استخراج رؤى بيولوجية ذات مغزى يتطلب أدوات متخصصة. لمعالجة هذه المشكلة ، نقدم برنامجا متاحا مجانا يسهل تحليل بيانات MALDI-2 MS أحادية الخلية. يسهل سير العمل المحدث لدينا التحويل المباشر للبيانات أحادية الخلية إلى تنسيق imzMLمفتوح المصدر 27 ، مما يسمح بالتوافق مع SCiLS MVS وبرامج البائعين الأخرى. لتحليل البيانات الأكثر تقدما ، يتضمن البرنامج النصي الكامل أيضا وظائف للتعليق التوضيحي للدهون والتجميع وأدوات التصور الأخرى التي تم تمكينها بواسطة Matplotlib (الإصدار 3.7.3). يتم تسهيل تحليل البيانات الأولية وقراءتها من خلال مكتبة pyTDFSDK ، وهي مجموعة من الوظائف المضمنة في سير عمل TIMSCONVERT28.
- قم بتهيئة مكتبة Python المخصصة SC_MALDI2_Analysis عن طريق نسخ الملف المتوفر في الملف التكميلي 1 إلى نفس مسار العمل مثل Jupyter Notebook ثم اضغط على Shift + Enter على لوحة المفاتيح في خلية التعليمات البرمجية الأولى. سيسمح لنا ذلك باستدعاء هذه الوظائف لاحقا واستخدامها لمعالجة البيانات.
- انسخ المسار إلى البيانات التي تم جمعها (ملفات Bruker .d). نوصي بتسمية المجلدات والأسماء الخاصة بكل ملف بشكل وصفي حتى نتمكن من تحليل مسارات الملفات هذه لاحقا لإنشاء بيانات وصفية لكل عينة. قم بتشغيل استيراد ملفات Bruker .d واحفظها في خلية رمز القائمة. سيؤدي هذا إلى إخراج العدد الإجمالي لملفات .d التي تم تحميلها.
- قم بتشغيل خلية التعليمات البرمجية بعنوان معالجة البيانات الأولية وحفظها في imzML. سيؤدي هذا إلى قراءة كل ملف Bruker .d المقدم ، وإجراء التقاط الذروة للبيانات ، ثم حفظ 20 صفحة في الدقيقة من البيانات حول كل ذروة إلى ملف .imzML مكتوب داخل ملف .d.
- للعثور على البيانات المحفوظة ، افتح موقع ملف .d الأول وسيتم حفظ imzML والبيانات الوصفية الأخرى في هذا المسار. في هذه المرحلة ، يمكن استيراد ملف imzML المحفوظ إلى برنامج المورد ، مثل SCiLS Lab MVS. لمزيد من تحليل البيانات المتقدمة، استمر في اتباع البرنامج النصي.
- قم بتحميل بيانات imzML المحفوظة عن طريق تشغيل التحميل في خلية التعليمات البرمجية لملف imzML المحفوظة مسبقا. يؤدي هذا إلى تحميل البيانات المحفوظة في البيانات المتغيرة.
- قم بتشغيل تجميع البيانات وحفظ خلية مصفوفة البيانات لتجميع جميع البيانات على طول محور m/z مشترك، وإزالة الميزات الموجودة بكثرة منخفضة (<0.1٪)، وإزالة القيم الأقل من الضوضاء (100 كثافة نسبية). تقوم هذه الوظيفة بعد ذلك بإعادة تجميع البيانات عن طريق حساب متوسط كل قيمة ذروة m / z داخل كل سلة. يتم حفظ مصفوفة البيانات الناتجة على أنها datacube_array ، ويتم حفظ الصناديق m / z المرتبطة بالمصفوفة على أنها valid_mz_bins ، ويتم حفظ عدد كل حاوية كعدد.
- للتعليق على البيانات، قم بتشغيل تصدير ذروة الخلية للتعليق التوضيحي لخرائط LIPID . سيؤدي هذا إلى نسخ جميع قمم m / z داخل مصفوفة البيانات الخاصة بك إلى الحافظة الخاصة بك. الصق هذا في ملف نصي فارغ واحفظه لتحميله على خرائط LIPID.
- افتح lipidmaps.org ، ثم حدد MS Data Bulk Search and Search LMSD للبحث عن أنواع الدهون ذات الصلة بيولوجيا. قم بتحميل قائمة الذروة عن طريق اختيار الملف النصي وحدد المقاربات المناسبة (على سبيل المثال ، [M + H] + و [M + Na] + و [M + K] +). حدد تفاوت الكتلة المناسب لمطياف الكتلة الخاص بك (اخترنا +/- 0.01 م / ز) ، وحدد جميع فئات الدهون ذات الأهمية ، واضغط على بحث. قم بتنزيل ملف .tsv.
- قم بتحميل بيانات تعريف الأطياف عن طريق تشغيل التحميل في مسارات الخلية المفردة لخلايا بيانات التعريف . قم بتغيير التعليمات البرمجية المنقسمة هنا بناء على هياكل مسار الملف.
- المعالجة المسبقة للبيانات عن طريق تشغيل خلية المعالجة المسبقة للبيانات والتعليقات التوضيحية للدهون . سيستخدم هذا ملف lipidMAPS tsv للتعليق على مجموعة البيانات إلى خطأ 10 صفحات في المليون ، وإزالة دهون السلسلة الفردية من نتائج lipidMAPS ، وتطبيع البيانات المتبقية ، وإزالة الميزات أو الخلايا التي لا تتجاوز حدا محددا. سيؤدي هذا إلى حفظ مجموعة البيانات الجديدة المعالجة كبيانات2 وبيانات تعريف الأطياف المحدثة ك updated_cell_types.
- يتم إجراء تحليل البيانات بواسطة Scanpy. قم بتشغيل UMAP و تحليل المجموعات لخلية الخلايا المفردة لتحديد البيانات ذات الأهمية (هنا اخترنا بيانات MALDI-2 و M2 فقط) ، وتوسيع نطاق مجموعة البيانات ، وتحميلها في كائن AnnData ضئيل لإجراء تحليلات سهلة.
ملاحظة: هنا ، تم إجراء تجميع Leiden وتقليل الأبعاد (UMAP) على مجموعة البيانات ، وتم تصور كل من فئات البيانات الأصلية والمجموعات الموجودة على UMAP ، وتم تلخيص بعض الميزات الأكثر دلالة إحصائيا بين المجموعات باستخدام وظيفة rank_genes_groups_dotplot من scanpy.
النتائج
تظهر نظرة عامة على سير العمل ل MALDI-2 MS أحادية الخلية الموجهة بالفلورة في الشكل 1. أولا ، يتم فصل الأنسجة التي تم تشريحها من مناطق الدماغ المستهدفة (الشكل 1 أ) إلى خلايا مفردة وترسبها على شرائح الفحص المجهري الموصلة المطلية ب ITO (الشكل 1 ب). يتم تحديد مواقع الخلايا عن طريق التصوير الفلوري للشريحة الكاملة (الشكل 1 ج) ، متبوعا بتطبيق مصفوفة MALDI (الشكل 1 د) ، وتحليل MALDI-2 MS بمساعدة microMS (الشكل 1E) ، وتحليل البيانات (الشكل 1F). باستخدام سير العمل هذا ، يمكن تحديد عشرات إلى مئات الدهون داخل الخلايا الفردية بدقة كتلة أقل من 10 جزء في المليون (باستخدام MALDI-2 timsTOF).
يمكن إجراء تصوير قياس الطيف الكتلي في منطقة صغيرة ذات أهمية لتقييم جودة تحضير الخلية وتطبيق المصفوفة. تظهر النتيجة التمثيلية لتصوير MALDI-2 للخلايا المشتتة في الشكل 2 أ. من هذه الصورة ، يمكن للمستخدم تقييم مدى انتشار المادة التحليلية بالمقارنة مع صورة مجهرية لنفس المنطقة. بناء على مقدار الانتشار ، يمكن للمحلل ضبط حجم مجال شعاع الليزر ومرشح المسافة لضمان عمليات الاستحواذ الحقيقية على الخلية الواحدة. في MSI أحادي الخلية ، سيتم استخدام حجم ليزر صغير وعرض نقطي للحصول على دقة خلوية (1-5 ميكرومتر) ، بينما يمكن استخدام حجم ليزر أكبر ل microMS ، مما يزيد من شدة الإشارة وعدد الدهون التي يمكن اكتشافها بالنسبة للتصوير. تظهر النتائج التمثيلية لتحسين الإشارة التي تم الحصول عليها باستخدام MALDI-2 في الشكل 2 ب. لوحظ زيادة أنواع الكوليسترول و PE و PC بالاتفاق مع الأدبيات السابقة14. بشكل عام ، نحصل على أفضل تحسين لإشارة MALDI-2 عند استخدام طاقة ليزر عالية نسبيا (>50٪) وعدد أقل من اللقطات (حوالي 10 إلى 200). بعض التحسين ضروري ، لأن الانخفاض الشديد في طاقة الليزر لن يوفر تعزيزا ، في حين أن الكثير من طاقة الليزر سيؤدي إلى تجزئة مفرطة للتحليل29. بالنسبة لتحليل الخلية الواحدة ، يعد التسجيل المكاني خطوة حاسمة لضمان الاستهداف الدقيق للخلايا الفردية. للتحقق من صحة التسجيل ، يمكن إنشاء مجموعة صغيرة من نقاط الاختبار (~ 5) حول الشريحة عن طريق إنشاء كائنات كبيرة الحجم في أماكن معروفة وتصدير إحداثيات الأداة عبر microMS. يضمن التحقق من أن نقاط الاختبار هذه تحدد مرحلة الأداة بشكل صحيح عند تحديدها أن استهداف الخلايا اللاحق سيكون دقيقا.
يتيح MALDI MS تحديد التنميط بين مناطق الدماغ الفردية وداخلها (الشكل 3). من خلال تحليل الاختلافات في ملفات تعريف الدهون ، تفصل تقنيات تقليل الأبعاد الخلايا بشكل فعال إلى مجموعات متميزة ، مما يسلط الضوء على تنوع تركيبات الدهون عبر مجموعة البيانات (الشكل 3 أ). علاوة على ذلك ، يظهر تطبيق تقنيات التجميع ، مثل تجميع ليدن ، وجود مجموعات فرعية فريدة من الخلايا داخل مناطق معينة من الدماغ ، بما في ذلك المخطط والقشرة (الشكل 3 ب). بالإضافة إلى ذلك ، من خلال مقارنة ملفات تعريف الدهون لكل مجموعة ببعضها البعض (الشكل 3 ج) ، يمكن استخدام دهون محددة كعلامات لتحديد الهويات الخاصة بالمجموعة. يتم تنظيم العديد من أنواع الدهون بشكل كبير في مجموعات محددة ، مما يشير إلى أدوار أو حالات وظيفية مميزة ، بينما يتم مشاركة أنواع أخرى عبر مجموعات ولكنها تختلف في شدة الإشارة النسبية. لضمان تحليل قوي للبيانات النهائية ، من الضروري تحقيق اكتشاف موثوق للدهون في غالبية الخلايا وتقييم تأثيرات الدفعات المحتملة. يعرض الشكل 3D أطياف تمثيلية من ست خلايا قشرية فردية منتشرة عبر شرائح منفصلة مطلية ب ITO. في حين أن ملامح الدهون الفردية تظهر عدم التجانس ، يتم الكشف عن الدهون باستمرار في جميع عمليات الاستحواذ. يعرض الشكل 3E UMAP للخلايا القشرية من شرائح مختلفة ، تم إعدادها وتحليلها في ظل ظروف متطابقة. يشير التداخل الكبير للخلايا بين الشرائح إلى أن تأثيرات الدفعات لا تربك بشكل كبير التفسيرات البيولوجية. إذا لزم الأمر ، يمكن تطبيق خوارزميات تصحيح الدفعات مثل ComBat30 للتخفيف من أي تأثيرات متبقية. باختصار ، MALDI-2 أحادي الخلية هو تقنية متقدمة لتحديد المؤشرات الحيوية للدهون ، وتقدم رؤى مهمة حول المجموعات السكانية الفرعية الخلوية وتعزز فهمنا للتنوع الإقليمي والوظيفي في الدماغ.
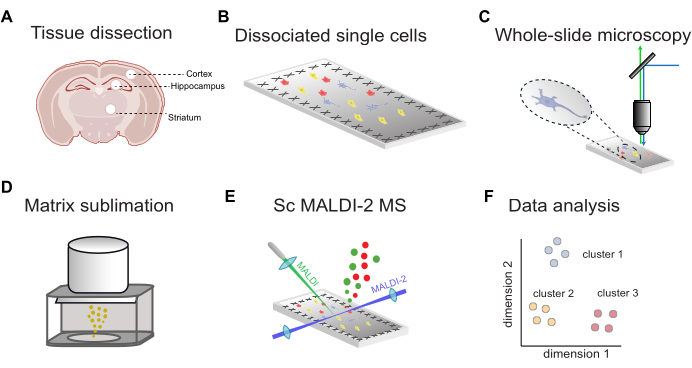
الشكل 1: سير العمل العام ل scMALDI-2 MS. (أ) تشريح الأنسجة اليدوي وعزل مناطق الدماغ. (ب) تفكك غراء الأنسجة إلى خلايا مفردة وترسب الخلية على شرائح موصلة مغلفة ب ITO. (ج) الفحص المجهري الفلوري كامل الشريحة عن طريق التلوين النووي (DAPI / Hoechst). (د) تسامي مصفوفة MALDI (DHAP) على شرائح العينة. (ه) قياسات MALDI-2 MS أحادية الخلية الموجهة بالصور. (و) تحليل شامل للبيانات، بما في ذلك تخفيض الأبعاد، والتجميع، والتحليل الإحصائي. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
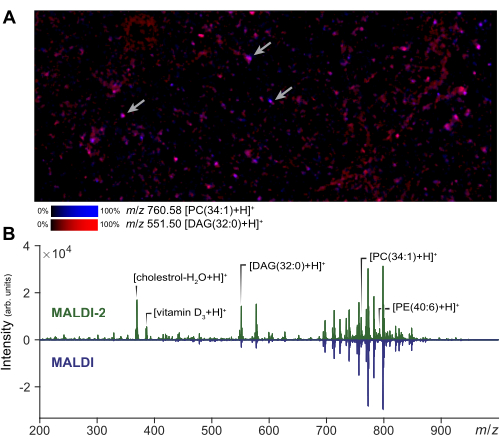
الشكل 2: تحسين MALDI-2 MS والتصوير لتحليل الخلية الواحدة. (أ) تصوير MALDI-2 MSI للمجموعات المتفرقة من الخلايا الأولية. تشير الأسهم إلى عدد قليل من الخلايا الفردية. يسمح أداء MSI ومنطقة صغيرة للمحلل بتحسين معلمات الطريقة وتقييم جودة العينة. (ب) مقارنة النتائج التي تم الحصول عليها باستخدام اقتناء MALDI-2 و MALDI MS. يتم تسليط الضوء على تحسين الإشارة (MALDI 2 مقابل MALDI) لمختلف الدهون ، بما في ذلك الكوليسترول ، DAG (دياسيل الجلسرين) ، PC (فوسفاتيديل كولين) ، و PE (فوسفاتيديل إيثانولامين) ، من خلال إظهار طيف MALDI-2 (أعلى) وطيف MALDI (أسفل). الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
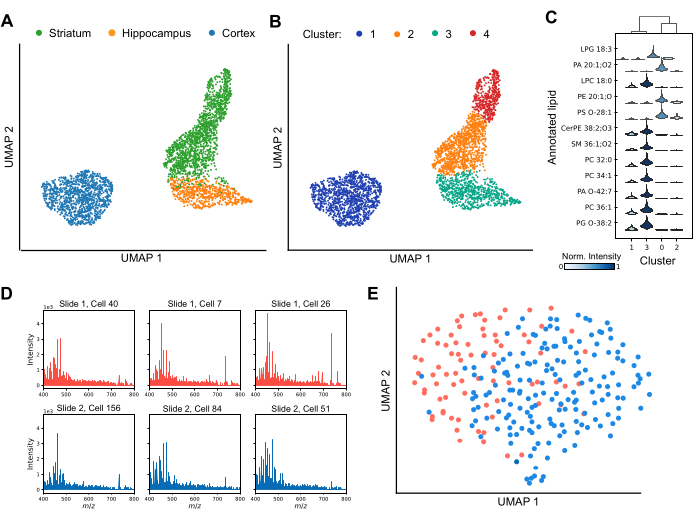
الشكل 3: التنميط الدهني أحادي الخلية داخل ثلاث مناطق في دماغ القوارض بواسطة MALDI-2 MS. (أ) تحليل التقريب والإسقاط المتشعب المنتظم (UMAP) للخلايا التي تم تحليلها الملونة حسب منطقة المنشأ الدماغية. (ب) تحليل UMAP لنفس الخلايا بعد الخضوع لتجميع ليدن ، حيث تتوافق المجموعات مع مجموعات سكانية فرعية من الخلايا ذات التركيبات الدهنية المتشابهة. تظهر الألوان هنا مجموعات مختلفة من بيانات Leiden. (ج) مخطط كمان يوضح الفرق في توزيعات الشدة للدهون المختلفة بين المجموعات. (د) تم تحليل الأطياف من ست خلايا قشرية فردية تمثيلية من شريحتين منفصلتين. (ه) تحليل UMAP للخلايا القشرية التي تم تحليلها من شريحتين منفصلتين. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
الملف التكميلي 1: SC-MALDI2_Analysis.py. يحتوي هذا الملف على مجموعة من الوظائف التي تم تطويرها لمعالجة بيانات قياس الطيف الكتلي MALDI-2 أحادية الخلية. يتضمن طرقا لقراءة بيانات قياس الطيف الكتلي الخام ، وإجراء التجميع ، وإجراء التعليقات التوضيحية وتحليلات المصب. يتم توفير مجموعة الخلايا لتحميل وتنفيذ جميع المعالجة المسبقة للبيانات والتحليلات في الملف التكميلي 2. الرجاء النقر هنا لتنزيل هذا الملف.
الملف التكميلي 2: JOVE Analysis.ipynb. يحتوي هذا الملف على دفتر ملاحظات Jupyter يحتوي على مجموعة من الخلايا لتحميلها في وظائف Python المخصصة من SC-MALDI2_Analysis.py وإجراء جميع المعالجة المسبقة للبيانات والتحليلات المذكورة. الرجاء النقر هنا لتنزيل هذا الملف.
Discussion
يعد MALDI MS أحادي الخلية عالي الإنتاجية والموجه بالصور أداة قيمة لفهم عدم التجانس الكيميائي على نطاق خلية واحدة. توفر إضافة ما بعد التأين الناجم عن الليزر (MALDI-2) تغطية تحليلية أعمق ، وهو أمر بالغ الأهمية للعينات محدودة الكتلة والحجم مثل خلايا الثدييات المعزولة.
في حين أن الغالبية العظمى من تدفقات عمل التصلب العصبي المتعدد أحادية الخلية وحيدة الخلية المنشورة تستخدم الخلايا المستنبتة ، يتم تطبيق نهجنا على الخلايا الأولية المعزولة بسرعة نسبيا. هذا التمييز أمر بالغ الأهمية ، حيث تظهر الخلايا المستنبتة تغيرا واضحا في محتوى مستقلبها والدهون في غضون أيام قليلة من زراعة23،31. يؤدي عزل الخلايا الأنزيمية ، في معظم الحالات ، إلى فقدان الهياكل الخلوية الدقيقة مثل المحاور والتشعبات والعمليات النجمية. لذلك ، يتم الحصول على إشارات التحليل المرصودة بشكل أساسي من سوما الخلية. على الرغم من ذلك ، تم تحقيق تصنيف ناجح حسب نوع الخلية4 ومنطقةالدماغ 12. يشير هذا إلى أن العديد من الدهون الفريدة للخلايا المتميزة تقع في سوما.
يمكن أن يكون للاختلافات الصغيرة في طريقة تطبيق مصفوفة MALDI تأثيرات عميقة على إشارة التحليل الناتجة. لقد قدمنا سير عمل يعتمد على تسامي المصفوفة القابلة للتكرار باستخدام جهاز تجاري ، والذي يولد طبقة مصفوفة متجانسة ، وإشارة عالية إلى ضوضاء ، وانتشار محدود للتحليل. ومع ذلك ، يمكن استخدام تطبيق المصفوفة باستخدام بخاخ آلي أو بخاخة الفنان ، من بين طرق أخرى. بغض النظر عن النهج ، يجب تطبيق كثافة مصفوفة أقل على مستحضرات الخلية المفردة مقارنة بتلك المستخدمة في MSI القياسي للأنسجة من أجل تحقيق نسبة المصفوفة البصرية إلى التحليل. كثافة المصفوفة المطبقة هنا هي ما يقرب من نصف تلك المستخدمة في MSI الأنسجة. بالطبع ، هذا يعني أن كمية المصفوفة لكل خلية أعلى مما هي عليه في MSI ، مما يساعد على المساهمة في استخراج التحليل المعزز والكشف عنه في نهج الخلية المعزولة المستخدم هنا.
بالنسبة للعينات التي تتطلب تحسينا كبيرا ، يمكن إجراء إجراء MSI على عائد استثمار صغير لتفكك الخلية المفردة ، كما هو موضح في الشكل 2 أ ، كجزء من عملية التحسين. يمكن أن تسمح هذه الخطوة أيضا للمحلل بتقييم مدى انتشار التحليل في مناهج تطبيق المصفوفة القائمة على المذيبات.
يتطلب MALDI الناجح الموجه بالصور للخلايا المفردة صورا مضان عالية الجودة ، وتطبيق مصفوفة قابلة للتكرار ، وتحكما دقيقا في المرحلة. هذه التجارب أكثر تطلبا من مهام سير عمل MSI القياسية ، والتي تتسامح مع أخطاء التسجيل الطفيفة والتباين في ظروف المصفوفة والعينة بسبب مناطق التصوير الأكبر وعدد التحليلات الأعلى. على الرغم من هذه التحديات ، نجحنا في استهداف عضيات مفردة باستخدام MALDI التقليدي.
يعد الخياطة الدقيقة للصور الفلورية المبلطة أمرا ضروريا ، حيث يمكن أن تتفاقم الأخطاء الصغيرة وتؤثر على التحكم في المرحلة ، مما يجعل دقة التسجيل أمرا بالغ الأهمية. تمثل تأثيرات الدفعات أيضا تحديا كبيرا ، حيث لوحظت اختلافات الإشارة عبر نفس الشريحة وبين الشرائح وعبر الاستعدادات. تساعد استراتيجيات التطبيع المناسبة، بما في ذلك استخدام المعايير الداخلية والتطبيع بين الربيعات، في التخفيف من هذه المسائل. بالإضافة إلى ذلك ، يمكن استخدام خوارزميات تصحيح الدفعات مثل ComBat30 لتقليل التباين التقني وتعزيز الاختلافات البيولوجية الحقيقية.
لا يزال الحصول على التعليقات التوضيحية الجزيئية الواثقة من الخلايا المفردة يمثل تحديا. في كثير من الأحيان ، تكون كمية المواد وحساسية الكشف غير كافية لمرض التصلب العصبي المتعدد الترادفي من جميع الأنواع باستثناء الأنواع الأكثر وفرة. بالنسبة لأولئك الذين لا يمكن اكتشافهم بما يكفي لمرض التصلب العصبي المتعدد الترادفي مباشرة من الخلايا الفردية ، يمكن تطبيق طرق بديلة لإنشاء مكتبة تحليلية. على سبيل المثال ، يمكن الحصول على بيانات التصلب العصبي المتعدد الترادفي على الأنسجة من عمليات القطع بالتبريد الرقيقة من نفس منطقة الدماغ المستخدمة لتوليد مجموعات خلوية. يمكن أيضا استخدام استخراج الدهون من الأنسجة متبوعا ب LC-MS. مع استمرار تحسن استراتيجيات تحضير العينات وتقنية التصلب العصبي المتعدد ، يمكن الحصول على المزيد والمزيد من المعلومات الهيكلية ذات الصلة مباشرة من الخلايا المفردة. في المستقبل ، نتوقع أن يتم توسيع سير العمل هذا للحصول على بيانات MS الترادفية من الخلايا المفردة ، مما يلغي الحاجة إلى تجارب LC-MS / MS الإضافية. نتصور أيضا أن هذا النهج يمكن أن يمتد إلى العديد من العينات على النطاق الجزئي في علم الأحياء وما بعده ، بما في ذلك أنواع الخلايا الفريدة ، وعضيات الثدييات ، والمساحيق ، واللدائن الدقيقة.
Disclosures
ليس لدى المؤلفين مصالح متنافسة للإفصاح عنها.
Acknowledgements
تعترف SWC بالدعم المقدم من زمالة Peixin He و Xiaoming Chen PhD4 وزمالة جامعة إلينوي Block Grant. وحظي هذا العمل بدعم من المعهد الوطني لتعاطي المخدرات أيضا بموجب الجائزة رقم P30DA018310 ، المعهد الوطني للشيخوخة بموجب الجائزة رقم. R01AG078797 ، ومن قبل مكتب مدير ، المعاهد الوطنية للصحة بموجب الجائزة رقم S10OD032242.
Materials
| Name | Company | Catalog Number | Comments |
| 2',5'-dihydroxyacetophenone | Sigma Aldrich | D107603 | DHAP, 97% purity |
| Ammonium acetate | Sigma Aldrich | 238074 | ACS reagent, ≥97% |
| Axio M2 Imager | Zeiss | N/A | N/A |
| Biopsy punch, 2 mm | Fisher Scientific | 12-460-399 | integra miltex standard biopsy punch, 2mm |
| Calcium chloride | Sigma Aldrich | C4901 | anhydrous, powder ≥97% |
| Eppendorf Centrifuge | Sigma Aldrich | EP5405000441 | centrifuge 5425 with rotor FA-24x2 |
| Gentamicin | Sigma Aldrich | G1272 | liquid, BioReagent |
| Glass etching pen | Sigma Aldrich | Z225568 | carbide time, pkg of 1 |
| Glycerol | Sigma Aldrich | G7893 | ACS reagent, ≥99.5% |
| HEPES buffer | Sigma Aldrich | H3375 | ≥99.5% (titration) |
| Hoechst 33258 Solution | Sigma Aldrich | 94403 | 1 mg/mL in H2O, ≥98.0% (HPLC) |
| In line HEPA Filter | Sigma Aldrich | WHA67225001 | VACU-GUARD 60 mm disc, 0.45 PFTE housing |
| ITO-Coated Microscopy Slides | Delta Technologies | CG-90IN-S115 | 70-100Ω resistance |
| Magnesium chloride | Sigma Aldrich | M8266 | anhydrous, ≥98% |
| Magnesium sulfate | Sigma Aldrich | 208094 | anhydrous, ≥97% |
| Microcentrifuge tubes | Sigma Aldrich | HS4323K | tube capacity 1.5 mL, pack of 500 |
| Papain dissociation system | Worthington Biochemical | LK003150 | one box, 5 single use vials |
| Penicillin-Streptomycin | Sigma Aldrich | P4458 | liquid, BioReagent |
| Potassium chloride | Sigma Aldrich | 529552 | Molecular biology grade |
| Potassium phosphate monobasic | Sigma Aldrich | P5379 | Reagent Plus |
| Sodium biocarbonate | Sigma Aldrich | S6014 | ACS reagent, ≥99.7% |
| Sodium chloride | Sigma Aldrich | S9888 | ACS reagent, ≥99% |
| Sodium hydroxide | Sigma Aldrich | 221465 | ACS reagent, ≥97%, pellets |
| Sodium phosphate dibasic | Sigma Aldrich | S9763 | ACS reagent, ≥99% |
| Sublimate | HTX | N/A | N/A |
| timsTOF FleX MALDI-2 | Bruker | N/A | microGRID enabled |
| Vacuum tubing | Thermo Scientific | 8701-0080 | Nalgene Non-phthalate PVC Tubing |
References
- Sunshine, H., Iruela-Arispe, M. L. Membrane lipids and cell signaling. Curr Opin Lipidol. 28 (5), 408-413 (2017).
- Van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nat Rev Mol Cell Biol. 9 (2), 112-124 (2008).
- Cheng, K. W., Su, P. R., Feller, K. J. A., Chien, M. P., Hsu, C. C. Investigating the Metabolic Heterogeneity of Cancer Cells Using Functional Single-Cell Selection and nLC Combined with Multinozzle Emitter Mass Spectrometry. Anal Chem. 96 (2), 624-629 (2024).
- Neumann, E. K., Comi, T. J., Rubakhin, S. S., Sweedler, J. V. Lipid Heterogeneity between Astrocytes and Neurons Revealed by Single-Cell MALDI-MS Combined with Immunocytochemical Classification. Ange Chem. 131 (18), 5971-5975 (2019).
- Bhaduri, A., Neumann, E. K., Kriegstein, A. R., Sweedler, J. V. Identification of Lipid Heterogeneity and Diversity in the Developing Human Brain. JACS Au. 1 (12), 2261-2270 (2021).
- Saliba, A. E., Westermann, A. J., Gorski, S. A., Vogel, J. Single-cell RNA-seq: advances and future challenges. Nuc Acid Res. 42 (14), 8845-8860 (2014).
- Karas, M., Hillenkamp, F. Laser desorption ionization of proteins with molecular masses exceeding 10,000 daltons. Anal Chem. 60 (20), 2299-2301 (1988).
- Croslow, S. W., Trinklein, T. J., Sweedler, J. V. Advances in multimodal mass spectrometry for single-cell analysis and imaging enhancement. FEBS Lett. 598 (6), 591-601 (2024).
- Bien, T., Bessler, S., Dreisewerd, K., Soltwisch, J. Transmission-Mode MALDI Mass Spectrometry Imaging of Single Cells: Optimizing Sample Preparation Protocols. Anal Chem. 93 (10), 4513-4520 (2021).
- Rappez, L., et al. SpaceM reveals metabolic states of single cells. Nat Methods. 18 (7), 799-805 (2021).
- Comi, T. J., Neumann, E. K., Do, T. D., Sweedler, J. V. microMS: A Python Platform for Image-Guided Mass Spectrometry Profiling. J Am Soc Mass Spec. 28 (9), 1919-1928 (2017).
- Xie, Y. R., et al. Multiscale biochemical mapping of the brain through deep-learning-enhanced high-throughput mass spectrometry. Nat Methods. 21 (3), 521-530 (2024).
- Neumann, E. K., Ellis, J. F., Triplett, A. E., Rubakhin, S. S., Sweedler, J. V. Lipid Analysis of 30 000 Individual Rodent Cerebellar Cells Using High-Resolution Mass Spectrometry. Anal Chem. 91 (12), 7871-7878 (2019).
- Soltwisch, J., et al. Mass spectrometry imaging with laser-induced postionization. Science. 348 (6231), 211-215 (2015).
- Barré, F. P. Y., et al. Enhanced Sensitivity Using MALDI Imaging Coupled with Laser Postionization (MALDI-2) for Pharmaceutical Research. Anal Chem. 91 (16), 10840-10848 (2019).
- McKinnon, J. C., et al. MALDI-2-Enabled Oversampling for the Mass Spectrometry Imaging of Metabolites at Single-Cell Resolution. J Am Soc Mass Spec. 35 (11), 2729-2742 (2024).
- Comi, T. J., Makurath, M. A., Philip, M. C., Rubakhin, S. S., Sweedler, J. V. MALDI MS Guided Liquid Microjunction Extraction for Capillary Electrophoresis-Electrospray Ionization MS Analysis of Single Pancreatic Islet Cells. Anal Chem. 89 (14), 7765-7772 (2017).
- Wu, J., et al. Transcardiac Perfusion of the Mouse for Brain Tissue Dissection and Fixation. Bio Protoc. 11 (5), e3988(2021).
- Aboghazleh, R., et al. Rodent brain extraction and dissection: A comprehensive approach. MethodsX. 12, 102516(2024).
- Jager, L. D., et al. Effect of enzymatic and mechanical methods of dissociation on neural progenitor cells derived from induced pluripotent stem cells. Adv Med Sci. 61 (1), 78-84 (2016).
- Chan-Andersen, P. C., Romanova, E. V., Rubakhin, S. S., Sweedler, J. V. Profiling 26,000 Aplysia californica neurons by single cell mass spectrometry reveals neuronal populations with distinct neuropeptide profiles. J Biol Chem. 298 (8), 102254(2022).
- Castro, D. C., Xie, Y. R., Rubakhin, S. S., Romanova, E. V., Sweedler, J. V. Image-guided MALDI mass spectrometry for high-throughput single-organelle characterization. Nat Methods. 18 (10), 1233-1238 (2021).
- Castro, D. C., et al. Single-Cell and Subcellular Analysis Using Ultrahigh Resolution 21 T MALDI FTICR Mass Spectrometry. Anal Chem. 95 (17), 6980-6988 (2023).
- Ibáñez, A. J., et al. Mass spectrometry-based metabolomics of single yeast cells. Proc Natl Acad Sci. 110 (22), 8790-8794 (2013).
- Krestensen, K. K., et al. Molecular Profiling of Glioblastoma Patient-Derived Single Cells Using Combined MALDI-MSI and MALDI-IHC. Anal Chem. 97 (7), 3846-3854 (2025).
- Niehaus, M., Soltwisch, J., Belov, M. E., Dreisewerd, K. Transmission-mode MALDI-2 mass spectrometry imaging of cells and tissues at subcellular resolution. Nat Methods. 16 (9), 925-931 (2019).
- Xie, Y. R., et al. Data-Driven and Machine Learning-Based Framework for Image-Guided Single-Cell Mass Spectrometry. J Proteome Res. 22 (2), 491-500 (2023).
- Luu, G. T., et al. TIMSCONVERT: a workflow to convert trapped ion mobility data to open data formats. Bioinformatics. 38 (16), 4046-4047 (2022).
- Vandergrift, G. W., et al. Experimental and Computational Evaluation of Lipidomic In-Source Fragmentation as a Result of Postionization with Matrix-Assisted Laser Desorption/Ionization. Anal Chem. 96 (41), 16127-16133 (2024).
- Zhang, Y., Parmigiani, G., Johnson, W. E. ComBat-seq: batch effect adjustment for RNA-seq count data. NAR Genom Bioinfo. 2 (3), lqaa078(2020).
- Nemes, P., Knolhoff, A. M., Rubakhin, S. S., Sweedler, J. V. Single-Cell Metabolomics: Changes in the Metabolome of Freshly Isolated and Cultured Neurons. ACS Chem Neurosci. 3 (10), 782-792 (2012).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
ISSN 1940-087X
Copyright © 2025 MyJoVE Corporation. All rights reserved
We use cookies to enhance your experience on our website.
By continuing to use our website or clicking “Continue”, you are agreeing to accept our cookies.