Method Article
Desorbimento/ionizzazione laser assistita da matrice guidata da fluorescenza con spettrometria di massa di postionizzazione indotta da laser di singole cellule neurali di ratto
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo protocollo delinea l'uso della microMS per la spettrometria di massa MALDI-2 a cellula singola guidata dalla fluorescenza, consentendo una migliore profilazione molecolare delle cellule neuronali primarie di ratto.
Abstract
Le misurazioni di singole cellule sono fondamentali per comprendere la ricca eterogeneità spaziochimica del cervello. La spettrometria di massa (MS) con laser/desorbimento assistita da matrice (MALDI) è in grado di caratterizzare molecole endogene in singole cellule senza marcatura e ad alto rendimento. I recenti progressi nello sviluppo degli spettrometri di massa MALDI con post-ionizzazione indotta da laser (MALDI-2) forniscono una sensibilità di rivelazione notevolmente migliorata per una varietà di lipidi e altre piccole molecole. Tuttavia, l'imaging MS di campioni di grandi dimensioni con MALDI-2 a risoluzione cellulare è proibitivo per la maggior parte delle applicazioni. In questo protocollo, le cellule primarie vengono isolate e disperse su vetrini conduttivi. Le posizioni relative delle cellule sono determinate mediante microscopia a fluorescenza a vetrino intero, seguita da un'accurata coregistrazione delle coordinate della microscopia con le coordinate dello stadio dello spettrometro di massa MALDI-2. L'analisi MS mirata delle sole posizioni cellulari fornisce misurazioni ad alta produttività, su singole cellule, con un'elevata copertura dell'analita e una dimensione dei dati ridotta rispetto all'imaging MS dell'intero campione. Descriviamo i passaggi critici necessari per la preparazione di singole cellule, l'imaging a fluorescenza su vetrino, l'applicazione della matrice e la spettrometria di massa MALDI-2.
Introduzione
I lipidi e i metaboliti sono fondamentali per la funzione cellulare e fungono da componenti essenziali delle membrane, delle fonti di energia e delle molecole di segnalazione 1,2. Tuttavia, la loro composizione e abbondanza possono variare in modo significativo tra le singole cellule, riflettendo le differenze nei tipi di cellule e negli stati di sviluppo e funzionali 3,4,5. L'analisi di queste differenze è fondamentale per comprendere la variabilità biologica e identificare sottopopolazioni cellulari distinte. Le tecniche di misurazione di singole cellule, come il sequenziamento dell'RNA, forniscono utili profili di trascrizione specifici per cellula6. Tuttavia, queste misurazioni a livello di trascritto non riflettono direttamente le effettive quantità cellulari di lipidi e metaboliti, poiché l'espressione genica non sempre è correlata all'effettiva abbondanza di questi analiti. Sono quindi necessari metodi specializzati per la misurazione diretta di lipidi e metaboliti per un'analisi completa della composizione chimica delle singole cellule e delle loro popolazioni.
L'imaging per spettrometria di massa (MSI) con desorbimento/ionizzazione laser assistita da matrice (MALDI) è uno strumento di scelta per la mappatura spaziale senza marcatura di biomolecole endogene in situ 7,8. Tipicamente, con MALDI, un laser UV viene utilizzato per ablare il materiale da un sottile strato di campione co-cristallizzato con una matrice organica, formando un pennacchio di ioni e molecole neutre. Gli ioni formati vengono quindi separati da un analizzatore MS e rilevati all'interno di uno spettrometro di massa. Dato il posizionamento accurato dei moderni stadi dello spettrometro di massa, il laser può essere posizionato per mirare a specifiche regioni del campione o rasterizzato tra le regioni per generare immagini molecolari da MSI. L'MSI con risoluzione spaziale cellulare o subcellulare (< 10 μm), ottenuta con un raggio laser focalizzato e un accurato movimento dello stadio, può essere utilizzato per ottenere informazioni chimiche sulle singole cellule 9,10. Tuttavia, le misurazioni MSI su questa scala sono inefficienti, soprattutto nel caso di campioni con basse densità di cellule mirate, a causa della quantità di tempo impiegato per l'imaging delle regioni vuote tra le cellule. Inoltre, la rilevabilità di molti analiti è limitata a causa del piccolo volume campionato. Per superare queste sfide, abbiamo sviluppato un approccio MALDI MS guidato da immagini per l'analisi di singole cellule ad alto rendimento11,12. In questo approccio, le posizioni delle cellule disperse sono determinate programmaticamente da immagini a fluorescenza dell'intero vetrino e utilizzate per guidare lo stadio dello spettrometro di massa verso le posizioni in cui si trovano le singole cellule con parametri specifici (ad esempio, dimensioni e forma) e vengono quindi analizzate tramite irradiazione con il laser MALDI. Lavori precedenti che utilizzano questo approccio mirato sono stati utilizzati per caratterizzare lipidi, peptidi e altre biomolecole in popolazioni cellulari eterogenee 5,13.
Data la natura di massa limitata delle singole cellule, il numero di analiti rilevati in tali campioni è generalmente inferiore a quello osservato direttamente dai tessuti. Pertanto, per aumentare la copertura dell'analita nell'analisi della SM su singola cellula, è fondamentale aumentare la sensibilità di rilevamento dell'analita. Un approccio sviluppato di recente che aiuta a superare questa sfida di rilevamento è MALDI con post-ionizzazione laser (MALDI-2), che ha dimostrato di migliorare la sensibilità per un'ampia gamma di analiti 9,14,15,16. Di conseguenza, MALDI-2 genera set di dati più completi su singole cellule e fornisce una copertura molecolare più profonda in campioni a massa limitata, come le cellule isolate.
L'obiettivo di questo metodo è ottenere misurazioni dei lipidi da migliaia di singole cellule. A tal fine, descriviamo un flusso di lavoro che consente la spettrometria di massa MALDI-2 a singola cellula ad alto rendimento ed è generalmente estendibile a qualsiasi approccio di spettrometria di massa basato su sonda con controllo preciso dello stadio17. In questo flusso di lavoro, il tessuto della regione o delle regioni cerebrali di interesse viene sezionato e le singole cellule vengono ottenute dal tessuto dopo una procedura di dissociazione della papaina. Le cellule vengono quindi etichettate con una colorazione nucleare e vengono disperse su vetrini conduttivi incisi con marcatori fiduciali, dove possono aderire. Successivamente, vengono acquisite immagini di vetrini interi utilizzando la microscopia a fluorescenza. La matrice viene depositata per sublimazione, generando uno strato cristallino omogeneo ripetibile e un elevato rapporto segnale/rumore per l'analisi MS su singola cellula. Utilizzando il software open-source microMS11, le coordinate relative delle posizioni delle cellule dall'immagine microscopica vengono mappate e allineate con le coordinate dello stadio dello spettrometro di massa mediante una registrazione di set di punti utilizzando marcatori fiduciali incisi sui vetrini in cui sono state depositate le cellule. Infine, utilizzando queste informazioni, vengono acquisiti spettri MS precisi e mirati da ogni singola cellula, consentendo di profilare migliaia di cellule in una singola corsa (<1 h)12,13.
Protocollo
Tutti gli esperimenti sugli animali in questo studio sono stati eseguiti in conformità con il protocollo di utilizzo degli animali approvato dall'Illinois Institutional Animal Care and Use Committee (23228) con il rigoroso rispetto degli standard nazionali e ARRIVE per il trattamento e la cura etica degli animali.
1. Preparazione dei materiali e delle soluzioni
- Preparare la soluzione salina bilanciata di Gey modificata (mGBSS) con 1,5 mM di CaCl2, 4,9 mM di KCl, 0,2 mM di KH2PO4, 11 mM di MgCl2, 0,3 mM di MgSO4, 138 mM di NaCl, 27,7 mM di NaHCO3, 0,8 mM di Na2HPO4 e 25 mM di HEPES. Regolare il pH a 7,2 utilizzando 3 M NaOH. Si consiglia di preparare 1 L di mGBSS e di conservarlo a 4 °C fino al momento dell'uso.
- Preparare siringhe da 50 ml riempiendole con la soluzione mGBSS preparata e metterle su ghiaccio per la perfusione transcardica. Preparare 1 siringa per ogni ratto sezionato.
- Preparare aliquote di soluzione di papaina seguendo il protocollo del sistema di dissociazione della papaina di Worthington. Questo sistema contiene quattro fiale come descritto di seguito, con la fiala 4 non utilizzata per questo protocollo:
Fiala 1: soluzione salina bilanciata di Earle sterile (EBSS) con bicarbonato e rosso fenolo
Fiala 2: papaina con L-cisteina e EDTA
Fiala 3: desossiribonucleasi I (DNasi)
Fiala 4: inibitore della proteasi ovomucoide con albumina sierica bovina- Aggiungere 5 mL di EBSS (flaconcino 1) al flaconcino di papaina (flaconcino 2) e metterlo a bagnomaria a 37 °C per 10 minuti per sciogliere la papaina.
- Aggiungere 500 μl di EBSS (flaconcino 1) nel flaconcino di DNasi (flaconcino 3) e mescolare delicatamente. Aggiungere 250 μl del flaconcino di DNasi diluito (flaconcino 3) nel flaconcino di papaina (flaconcino 2). Questo preparato contiene una concentrazione finale di 20 unità/mL di papaina, 0,005% di DNasi, 0,5 mM di EDTA e 1 mM di L-cisteina.
- Congelare 250 μL di aliquote di questa soluzione di papaina preparata per le successive dissociazioni (si ottiene ~20 aliquote). Integrare questa soluzione con i seguenti antibiotici: 100 unità/mL di penicillina G, 100 μg/mL di streptomicina e 100 μg/mL di gentamicina. Questa opzione può essere modificata per l'applicazione dell'utente.
- Preparare una soluzione di acetato di ammonio da 150 mM e conservarla a 4 °C. Preparare una soluzione di glicerolo al 33% diluendo il glicerolo nella soluzione mGBSS preparata.
- Incidere i fiduciali su vetrini per microscopia rivestiti di ossido di indio e stagno (ITO) utilizzando una penna per incisione con punta in carburo di tungsteno o diamante o un incisore laser a CO2 . Sul lato conduttivo del vetrino ITO, incidere 20-40 marcatori fiduciali a forma di X lungo la periferia del vetrino per consentire la registrazione spaziale cross-platform (ad esempio, microscopio e spettrometro di massa).
NOTA: Fare attenzione a non incidere in una linea collegata che circonda il campione o le sue aree, poiché ciò interrompe la conduttività superficiale tra l'area e un adattatore per vetrini.
2. Preparazione delle cellule neurali primarie
NOTA: Il tessuto ippocampale di ratto viene sezionato, dissociato in singole cellule con papaina e depositato su vetrini conduttivi a bassa densità. L'isolamento delle cellule in questo modo consente la spettrometria di massa a singola cellula ad alto rendimento di lipidi endogeni.
- Prima della dissezione animale, scongelare un'aliquota della soluzione di papaina preparata e metterla in un contenitore con un tubo di ingresso e di uscita che consenta alla CO2 di spostare l'aria all'interno del contenitore. Immergere questo sistema in un bagnomaria impostato a 37 °C e continuare il flusso di CO2 fino a quando la soluzione non raggiunge il pH corretto, come indicato dal colore, utilizzando la tabella dei colori del pH inclusa nella confezione del sistema di dissociazione della papaina Worthington. Il pH target è compreso tra 7,2 e 7,4.
NOTA: Non far bollire CO2 nella soluzione enzimatica, poiché ciò comporterebbe la denaturazione della DNasi e la perdita di attività biologica. Utilizzare una provetta da centrifuga da 50 ml con due provette incollate nel coperchio, in modo che la CO2 riempia la provetta dal basso e fuoriesca dalla provetta di uscita superiore. - Eutanasia dei ratti in conformità con i protocolli IACUC seguendo le linee guida istituzionali, locali e federali. Qui, i ratti maschi di Sprague-Dawley di 2 o 3 mesi vengono utilizzati e sacrificati per asfissia da CO2 .
- Perfondere transcardialmente il ratto con una soluzione di mGBSS ghiacciata (4 °C) per rimuovere il sangue dal sistema vascolare e raffreddare rapidamente il corpo dell'animale. Un esempio di questa procedura può essere trovato nel protocollo di We et al.18.
- Isolare chirurgicamente il cervello dal ratto, facendo attenzione a non danneggiare la regione cerebrale di interesse. Una panoramica pubblicata in precedenza di questo isolamento fornisce ulteriori dettagli su alcuni dei singoli passaggi19.
- Utilizzare un tritatutto o un cubo di tessuto per tagliare una sezione trasversale (~2 mm di spessore) del cervello per esporre la regione (ippocampo, corteccia o striato) al campione.
- Utilizzare un punzone per biopsia da 2 mm per campionare la regione cerebrale di interesse e depositare il pezzo di tessuto nella fiala di soluzione di papaina.
- Incubare questa soluzione di tessuto e papaina a 37 °C sotto costante agitazione (ad esempio, con un bilanciere elettrico) per 30-90 minuti.
NOTA: La durata del trattamento enzimatico deve essere ottimizzata in base alla regione del cervello, al ceppo animale, all'età e alla produzione cellulare desiderata (ad esempio, neuroni e astrociti con terminali rispetto alla resa massima di cellule senza terminali). Per il tessuto ippocampale di ratto, 60-90 minuti è tipico. Trattamenti più brevi possono preservare alcuni terminali ma lasciare molte cellule in cluster non dissociati, mentre trattamenti più lunghi possono migliorare la resa ma aumentare il danno cellulare, richiedendo una valutazione utilizzando saggi vivi/morti (ad esempio, kit di vitalità/citotossicità LIVE/DEAD). Per gli studi comparativi, i trattamenti devono essere coerenti tra i tipi di campione. - Metti una fiala di tessuto e papaina sul ghiaccio per raffreddare e sopprimere l'attività enzimatica. Triturare il tessuto utilizzando una punta di pipetta tagliata collegata a una pipetta a volume regolabile P100 fino a quando la maggior parte del tessuto non è visivamente spezzata. Non formare bolle d'aria durante la dissociazione per evitare danni eccessivi alle cellule.
NOTA: Una triturazione vigorosa si traduce in un'elevata resa di cellule; Tuttavia, non conservano la maggior parte dei loro processi. La triturazione delicata fornisce più processi sulle cellule, ma si traduce in più cellule non dissociate. È inoltre possibile utilizzare puntali per pipette di diverse dimensioni e altri metodi e ottimizzarli per ottenere risultati di dissociazione ideali20. - Centrifugare le celle dissociate a 300 x g per 5 minuti a temperatura ambiente per pellettare le cellule. Rimuovere il surnatante e risospendere il pellet in 1 mL di mGBSS. Aggiungere la colorazione nucleare Hoechst 33342 a una concentrazione finale di 1 μg/mL.
- Pipettare 100-500 μl della sospensione cellulare su vetrini ITO incisi e lasciare riposare per circa 5 minuti o più per consentire alle cellule di sedimentare e aderire al vetrino.
- La diluizione della sospensione cellulare dovrà essere determinata empiricamente. Utilizzando un microscopio ottico, aggiungere una gocciolina delle cellule sul vetrino per determinare la densità approssimativa e diluire da lì per assicurarsi che ci sia una distanza sufficiente tra le cellule (cioè sufficientemente grande della dimensione del fascio MALDI). Questa diluizione può essere eseguita sul primo vetrino, quindi la diluizione può essere regolata secondo necessità per i vetrini successivi.
- Aspirare delicatamente la sospensione cellulare dal vetrino utilizzando un sistema a vuoto contenente (situato nell'ordine a partire dalla linea del vuoto): 1) Tubo con filtro HEPA per gas in linea, 2) Tubo montato su un pallone filtrante da 50 mL, 3) Tubo collegato a un secondo ingresso del pallone filtrante con un puntale per pipetta collegato (ad es. 100 μL). Se si conservano le celle piastrate per un uso futuro, sostituire la sospensione aspirata con una soluzione di glicerolo al 33% per 1-2 minuti e rimuoverla.
NOTA: Questo passaggio garantirà la stabilità della cella. L'erogazione della soluzione di glicerolo da un lato mentre si aspira delicatamente dall'altro lato è ottimale per mantenere la struttura cellulare. Durante la preparazione del primo campione, monitorare sempre visivamente tutti i passaggi qui descritti utilizzando un microscopio invertito. - Applicare 100-500 μL di acetato di ammonio sulle regioni depositate, lasciare riposare per circa 1 minuto, quindi aspirare per pulire i vetrini. Ripetere se necessario per assicurarsi che i vetrini siano privi di sale o glicerolo.
3. Microscopia
NOTA: Per determinare la posizione delle cellule depositate, ogni vetrino viene ripreso mediante microscopia a campo chiaro/fluorescenza. Il canale di fluorescenza consente di localizzare con precisione le cellule colorate con Hoechst, mentre l'imaging in campo chiaro fornisce informazioni morfologiche. Qualsiasi microscopio in grado di acquisire immagini affiancate è adatto a questo processo.
- Se non è stato fatto in precedenza, risciacquare i vetrini con 2-3 mL di acetato di ammonio 150 mM. Questo passaggio è fondamentale per rimuovere i cristalli di glicerolo e sale, che possono interferire con l'osservazione al microscopio e l'applicazione della matrice MALDI. Asciugare gli scivoli sotto un leggero getto di N2 o lasciare asciugare all'aria.
- Caricare il vetrino nel tavolino del microscopio. Mettere a fuoco il microscopio utilizzando almeno 10 punti di supporto distribuiti uniformemente sul vetrino.
- Utilizzando filtri adatti per DAPI (eccitazione: 335-383 nm, emissione: 420-470 nm) e imaging in campo chiaro, è possibile ottenere un'immagine a fluorescenza affiancata con un ingrandimento di 5x-10x sull'intero vetrino, assicurandosi che i marcatori fiduciali (creati nel passaggio 1.5) vengano acquisiti.
NOTA: Un ingrandimento 5x è solitamente sufficiente, soprattutto quando è necessario visualizzare molte diapositive. Per risolvere la morfologia cellulare più fine, è possibile utilizzare obiettivi 10x o 20x al costo di un tempo di imaging quadraticamente più lungo e di un aumento delle dimensioni dei file. - Unisci l'immagine piastrellata utilizzando un software di microscopia, come ZEN (Zeiss). L'unione accurata è fondamentale, poiché eventuali errori in questa fase si propagheranno alla fase di registrazione finale, con conseguente targeting impreciso. Per verificare, assicurarsi che le tessere adiacenti verticalmente non siano sfalsate. Inoltre, prendi in considerazione l'acquisizione di immagini affiancate con una grande sovrapposizione di porzioni (20%-30%).
- Elabora ed esporta ogni immagine come file bigTIFF nel software di microscopia. Se l'area di cui viene eseguita l'immagine è relativamente piccola e le dimensioni dell'immagine finale sono inferiori a 2 GB, utilizzare un formato di file TIFF standard.
4. Applicazione a matrice
NOTA: L'applicazione coerente e corretta della matrice MALDI è fondamentale per ottenere dati di qualità a cella singola. Mentre qui viene utilizzata la sublimazione con un apparato commerciale, l'applicazione della matrice può essere eseguita anche con uno spruzzatore robotizzato12, un aerografo21 o un apparecchio di sublimazione fatto in casa22. Abbiamo scoperto che le preparazioni a singola cellula richiedono meno matrice rispetto alle sezioni sottili di tessuto tipicamente utilizzate per l'imaging con spettrometria di massa. Per ridurre gli effetti batch, si consiglia di applicare la matrice a tutti i vetrini in studio durante una sessione e di depositare cellule di gruppi diversi (ad esempio, regione del cervello o trattamento o controllo) sullo stesso vetrino quando possibile. La selezione della matrice è fondamentale sia per i flussi di lavoro tradizionali MALDI che per quelli MALDI-2 a singola cellula. Per MALDI a cella singola, DHB23, 9-AA24 e CHCA25 sono stati utilizzati con successo. In MALDI-2, noi e altri abbiamo osservato un significativo miglioramento del segnale con DHAP9, mentre matrici come NEDC16 e CHCA26 sono state applicate in modo efficace.
- Sciogliere 20 mg di 2,5-diidrossiacetofenone (DHAP) in 1,5 mL di acetone. Posizionare i vetrini nel supporto della camera di sublimazione e posizionarli nell'apparecchio di sublimazione.
- Pipett la soluzione di matrice disciolta sul wafer di ceramica e lasciare evaporare l'acetone. Chiudere la camera di sublimazione per sigillarla.
- Riempire la camera del refrigerante con una granita di acqua ghiacciata e posizionarla sopra la camera.
- Accendere il vuoto e lasciare che il sistema si equilibri per 5 minuti. La pressione nel sistema deve essere inferiore a 40 mbar.
- Iniziare la sublimazione riscaldando la camera a 200 °C per 5 minuti.
- Togliete il bagnomaria con acqua ghiacciata, portate la temperatura a 25 °C e posizionate un dissipatore di calore sopra la camera. Lasciare che il sistema si riscaldi a temperatura ambiente per 5 minuti per evitare la formazione di condensa.
NOTA: Il contenitore che contiene il bagno di acqua ghiacciata può essere svuotato e riempito con acqua calda per fungere da dissipatore di calore. - Sfiatare lentamente il sistema, aprire la camera e rimuovere i vetrini.
5. MALDI MS a cellula singola
NOTA: I dati MS a cella singola sono ottenuti su uno strumento MALDI-2 timsTOF (timsTOF flex) utilizzando il pacchetto microMS open-source per rilevare le cellule e guidare lo spettrometro di massa. Ciò richiede che le posizioni dei pixel dell'immagine ottica delle cellule bersaglio siano tradotte nelle coordinate fisiche dello stadio dello spettrometro di massa.
- Apri microMS e decima le immagini al microscopio bigTIFF utilizzando l'opzione Gruppo immagini, poiché sono presenti sia canali di campo chiaro che di fluorescenza.
- Utilizzando lo strumento Opzioni BLOB, identificare i parametri ottimali da selezionare per le celle di destinazione. Regolare i parametri seguenti: la dimensione massima e minima del BLOB per selezionare la dimensione piccola o grande dei BLOB da trovare; l'impostazione della soglia, che determina come soglie il canale di fluorescenza per il rilevamento dei blob; e la circolarità, che determina quanto circolari devono essere le celle identificate per essere prese in considerazione. Per i dati qui presentati, abbiamo utilizzato i seguenti valori dei parametri: circolarità massima: nessuna; circolarità minima: 0,6; soglia: 75; Dimensione minima: 20; Dimensione massima: Nessuna.
- Utilizzare l'opzione Trova BLOB per trovare i BLOB e la finestra popup per salvare l'elenco BLOB con il nome desiderato.
- Usa lo strumento filtro Distanza per selezionare una distanza specifica che ogni cella deve essere lontana l'una dall'altra (per questi dati, utilizzando la microscopia a fluorescenza 5x, abbiamo utilizzato un filtro distanza di 35 pixel). Questo per garantire che le cellule siano sufficientemente lontane l'una dall'altra per eseguire l'analisi di singole cellule. Questo numero è espresso in unità pixel, quindi conosci la conversione da pixel a μm per il microscopio per determinare il numero ideale. Utilizzare le dimensioni del diametro della sonda laser più circa 20-30 μm per tenere conto degli errori nella registrazione.
- Caricare le diapositive nello strumento utilizzando l'MTP Slide Adapter II. Torna al computer con microMS e, utilizzando un'applicazione desktop remoto, accedi al computer dello spettrometro di massa.
- Se è la prima volta che lo strumento viene utilizzato, verificare la posizione X-Y dello strumento. Per fare ciò, vai su Strumenti e impostazioni dello strumento. La finestra popup mostrerà una serie di coordinate con le loro posizioni X e Y. Selezionare ciascuno di questi punti specifici sulla geometria dell'adattatore per diapositive II sullo strumento e aggiornare le posizioni X e Y nella finestra microMS.
- Utilizzando la fotocamera e i controlli del tavolino dello spettrometro di massa, navigare in una posizione facilmente identificabile sul vetrino e copiare le coordinate dello strumento. In microMS, trova la posizione corrispondente nell'immagine del microscopio, fai clic con il pulsante destro del mouse su quella posizione e digita le coordinate nella posizione del testo della finestra popup. Arrotonda tutte le coordinate all'intero più vicino e separa le coordinate X e Y con lo spazio (ad esempio, 39595 -23232).
- Ripetere il passaggio 5.7. registrare almeno 12 marcatori fiduciali nell'intero vetrino. Dopo che sono stati aggiunti tre punti di registrazione, uno dei cerchi diventerà rosso, indicando che è la posizione più lontana dalla registrazione. In questo caso, eliminare il punto di registrazione corrente utilizzando Maiusc + clic destro e riprovare.
- In File passare a Salva e quindi a Registrazione per salvare il file di registrazione (con estensione msreg).
- Con i blob visualizzati nella diapositiva, salvare il file di posizione dello strumento (con estensione xeo) passando a File, quindi a Salva e infine a Posizioni strumento. Utilizzando il software desktop remoto, trasferire questo file .xeo sul computer dello strumento.
- Copiare il testo dal file .xeo personalizzato generato nel file .xeo MTP Slide Adapter II sullo strumento e salvarlo. Questo aggiornerà questo file di geometria con le posizioni delle celle.
- Fare clic sulla scheda Automazione e selezionare Nuovo... per avviare un'esecuzione automatica. Trascinare il puntatore del mouse sulla regione campione visualizzata per selezionare le celle e fare clic con il pulsante destro del mouse per aggiungerle all'elenco di analisi. Salvare l'esecuzione dell'automazione e fare clic su Avvia esecuzione automatica per iniziare l'acquisizione. La durata della corsa dipende da una varietà di fattori, tra cui il numero di celle, i colpi laser e la frequenza del laser, ma in generale, circa 5-10 celle possono essere analizzate in modalità MALDI (frequenza di ripetizione laser di 10 kHz) e 1-5 celle possono essere analizzate in modalità MALDI-2 (frequenza di ripetizione laser di 1k Hz) ogni secondo.
6. Trattamento dei dati
NOTA: I pacchetti software commerciali esistenti non sono adatti per l'analisi dei dati di spettrometria di massa a cella singola ad alto rendimento. Mentre i singoli spettri possono essere visualizzati, l'estrazione di intuizioni biologiche significative richiede strumenti specializzati. Per risolvere questo problema, forniamo un software disponibile gratuitamente che facilita l'analisi dei dati MALDI-2 MS a cella singola. Il nostro flusso di lavoro aggiornato facilita la conversione diretta dei dati a cella singola nel formato imzML27 open source, consentendo la compatibilità con SCiLS MVS e altri software dei fornitori. Per un'analisi dei dati più avanzata, lo script completo include anche funzionalità per l'annotazione dei lipidi, il clustering e altri strumenti di visualizzazione abilitati da Matplotlib (versione 3.7.3). L'analisi e la lettura dei dati grezzi sono facilitate dalla libreria pyTDFSDK, un insieme di funzioni incluse nel flusso di lavoro TIMSCONVERT28.
- Inizializzare la libreria Python personalizzata SC_MALDI2_Analysis copiando il file fornito nel file supplementare 1 nello stesso percorso di lavoro di Jupyter Notebook e quindi premere MAIUSC + INVIO sulla tastiera nella prima cella di codice. Questo ci permetterà di chiamare queste funzioni in un secondo momento e di utilizzarle per elaborare i dati.
- Copiare il percorso dei dati raccolti (file Bruker .d). Si consiglia di denominare le cartelle e i nomi di ogni file in modo descrittivo in modo da poter analizzare questi percorsi di file in un secondo momento per stabilire i metadati per ogni esempio. Esegui l'importazione dei file Bruker .d e salva in una cella di codice elenco. Questo produrrà il numero totale di file .d caricati.
- Eseguire la cella di codice intitolata Elaborazione dei dati non elaborati e salvataggio in imzML. In questo modo verrà letto ogni file Bruker .d fornito, verrà eseguito il prelievo dei picchi sui dati, quindi verranno salvati 20 ppm di dati attorno a ciascun picco in un file .imzML scritto all'interno del file .d.
- Per trovare i dati salvati, apri la posizione del primo file .d e l'imzML e altri metadati verranno salvati in questo percorso. In questa fase, il file imzML salvato può essere importato nel software del fornitore, come SCiLS Lab MVS. Per un'analisi dei dati più avanzata, continua a seguire lo script.
- Caricare i dati imzML salvati eseguendo la cella di codice Caricamento nel file imzML salvato in precedenza. In questo modo i dati salvati vengono caricati nei dati variabili.
- Eseguire la creazione di contenitori dei dati e salvare la cella della matrice di dati per raggruppare tutti i dati lungo un asse m/z comune, rimuovere le funzioni presenti in bassa abbondanza (<0,1%) e rimuovere i valori inferiori al disturbo (100 intensità relativa). Questa funzione quindi esegue il rebins dei dati calcolando la media di ogni valore m/z di picco all'interno di ogni bin. La matrice di dati risultante viene salvata come datacube_array, i contenitori m/z associati alla matrice vengono salvati come valid_mz_bins e un conteggio di ciascun contenitore viene salvato come conteggio.
- Per annotare i dati, eseguire la cella di annotazione Esportazione dei picchi per le MAPPE LIPIDICHE . In questo modo verranno copiati tutti i picchi m/z all'interno della matrice di dati negli appunti. Incollalo in un file di testo vuoto e salvalo per caricarlo su LIPID MAPS.
- Apri lipidmaps.org, quindi seleziona MS Data Bulk Search e Search LMSD per cercare specie lipidiche biologicamente rilevanti. Caricare l'elenco dei picchi scegliendo il file di testo e selezionare gli addotti appropriati (ad esempio, [M+H]+, [M+Na]+ e [M+K]+). Seleziona la tolleranza di massa appropriata per il tuo spettrometro di massa (abbiamo scelto +/- 0,01 m/z), seleziona tutte le classi lipidiche di interesse e premi Cerca. Scarica il file .tsv.
- Caricare i metadati degli spettri eseguendo il comando Caricamento nei percorsi delle singole celle per le celle dei metadati . Modifica il codice diviso qui in base alle strutture del percorso del file.
- Pre-elaborare i dati eseguendo la cella Pre-elaborazione dati e annotazione lipidica . Questo utilizzerà il file tsv di lipidMAPS per annotare il set di dati con un errore di 10 ppm, eliminare i lipidi a catena dispari dai risultati di lipidMAPS, normalizzare i dati rimanenti e rimuovere le caratteristiche o le celle che non superano una soglia specifica. In questo modo il nuovo set di dati elaborato verrà salvato come dati2 e i metadati spettrali aggiornati come updated_cell_types.
- L'analisi dei dati viene eseguita da scanpy. Esegui la cella UMAP e Clustering Analysis for Single Cells per selezionare i dati di interesse (qui abbiamo selezionato solo i dati MALDI-2, M2), ridimensionare il set di dati e caricarlo in un oggetto AnnData scanpy per facilitare le analisi.
NOTA: Qui, il clustering di Leiden e la riduzione della dimensionalità (UMAP) sono stati eseguiti sul set di dati, sono state visualizzate sia le classi di dati originali che i cluster sull'UMAP e alcune delle caratteristiche statisticamente più significative tra i cluster sono state riassunte utilizzando la funzione rank_genes_groups_dotplot di scanpy.
Risultati
Una panoramica del flusso di lavoro per la MS MALDI-2 a cellula singola guidata dalla fluorescenza è mostrata nella Figura 1. In primo luogo, il tessuto sezionato da regioni cerebrali mirate (Figura 1A) viene dissociato in singole cellule e depositato su vetrini per microscopia rivestiti di ITO conduttivo (Figura 1B). Le posizioni delle cellule sono determinate mediante imaging a fluorescenza su vetrino intero (Figura 1C), seguito dall'applicazione della matrice MALDI (Figura 1D), dall'analisi MALDI-2 MS assistita da microMS (Figura 1E) e dall'analisi dei dati (Figura 1F). Utilizzando questo flusso di lavoro, è possibile identificare da decine a centinaia di lipidi all'interno di singole cellule con un'accuratezza di massa inferiore a 10 ppm (utilizzando un MALDI-2 timsTOF).
L'imaging con spettrometria di massa può essere eseguito su una piccola regione di interesse per valutare la qualità della preparazione cellulare e l'applicazione della matrice. Un risultato rappresentativo dell'imaging MALDI-2 di cellule disperse è mostrato nella Figura 2A. Da questa immagine, l'utente può valutare l'entità della diffusione dell'analita rispetto a una micrografia fotografica della stessa regione. In base alla quantità di diffusione, l'analista può regolare la dimensione del campo del raggio laser e il filtro della distanza per garantire vere acquisizioni di singole cellule. Nell'MSI a singola cellula, per ottenere una risoluzione cellulare (1-5 μm) verrà utilizzata una dimensione laser e una larghezza raster ridotte, mentre per la microMS è possibile utilizzare un laser di dimensioni maggiori, aumentando l'intensità del segnale e il numero di lipidi rilevabili rispetto all'imaging. I risultati rappresentativi dell'aumento del segnale ottenuto con MALDI-2 sono mostrati nella Figura 2B. L'aumento delle specie di colesterolo, EP e PC è stato osservato in accordo con la letteratura precedente14. In generale, otteniamo il miglior miglioramento del segnale MALDI-2 quando si utilizza una potenza laser relativamente elevata (>50%) e un numero inferiore di scatti (da 10 a 200 circa). È necessaria una certa ottimizzazione, poiché una potenza laser troppo bassa non fornirà un miglioramento, mentre un'energia laser eccessiva causerà un'eccessiva frammentazione dell'analita29. Per l'analisi di singole cellule, la registrazione spaziale è un passaggio fondamentale per garantire un targeting accurato delle singole cellule. Per convalidare la registrazione, è possibile generare un piccolo set di punti di prova (~5) attorno al vetrino creando blob in punti noti ed esportando le coordinate dello strumento tramite microMS. La verifica che questi punti di prova posizionino correttamente lo stadio dello strumento, quando selezionati, garantisce che il successivo puntamento delle cellule sia accurato.
La MALDI MS consente di profilare l'eterogeneità cellulare sia tra le singole regioni cerebrali che all'interno di esse (Figura 3). Analizzando le variazioni nei profili lipidici, le tecniche di riduzione della dimensionalità separano efficacemente le cellule in cluster distinti, evidenziando la diversità delle composizioni lipidiche nel set di dati (Figura 3A). Inoltre, l'applicazione di tecniche di clustering, come il clustering di Leiden, mostra la presenza di sottopopolazioni uniche di cellule all'interno di specifiche regioni cerebrali, tra cui lo striato e la corteccia (Figura 3B). Inoltre, confrontando i profili lipidici di ciascun cluster tra loro (Figura 3C), lipidi specifici possono essere utilizzati come marcatori per definire le identità specifiche del cluster. Diverse specie lipidiche sono significativamente sovraregolate in cluster specifici, suggerendo ruoli funzionali o stati distinti, mentre altre sono condivise tra i cluster ma variano nella loro intensità di segnale relativa. Per garantire una solida analisi dei dati a valle, è essenziale ottenere un rilevamento affidabile dei lipidi nella maggior parte delle cellule e valutare i potenziali effetti dei batch. La Figura 3D presenta spettri rappresentativi di sei singole cellule corticali disperse su vetrini separati rivestiti di ITO. Mentre i singoli profili lipidici mostrano eterogeneità, i lipidi vengono rilevati in modo coerente in tutte le acquisizioni. La Figura 3E mostra un UMAP di cellule corticali provenienti da diversi vetrini, preparati e analizzati in condizioni identiche. La sostanziale sovrapposizione di cellule tra i vetrini indica che gli effetti batch non confondono significativamente le interpretazioni biologiche. Se necessario, è possibile applicare algoritmi di correzione batch come ComBat30 per mitigare eventuali effetti residui. In sintesi, MALDI-2 a cellula singola è una tecnica avanzata per l'identificazione dei biomarcatori lipidici, che offre informazioni critiche sulle sottopopolazioni cellulari e migliora la nostra comprensione della diversità regionale e funzionale nel cervello.
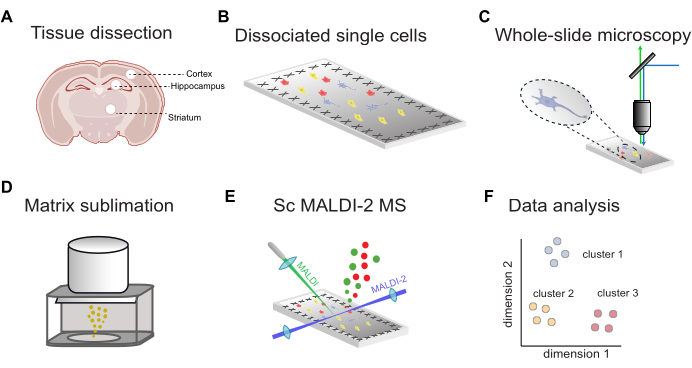
Figura 1: Flusso di lavoro generale per scMALDI-2 MS. (A) Dissezione manuale dei tessuti e isolamento delle regioni cerebrali. (B) Dissociazione della papaina del tessuto in singole cellule e deposizione cellulare su vetrini conduttivi rivestiti di ITO. (C) Microscopia a fluorescenza su vetrino intero tramite colorazione nucleare (DAPI/Hoechst). (D) Sublimazione della matrice MALDI (DHAP) su vetrini campione. (E) Misure MALDI-2 MS a cella singola guidate da immagini. (F) Analisi completa dei dati, compresa la riduzione della dimensionalità, il clustering e l'analisi statistica. Clicca qui per visualizzare una versione più grande di questa figura.
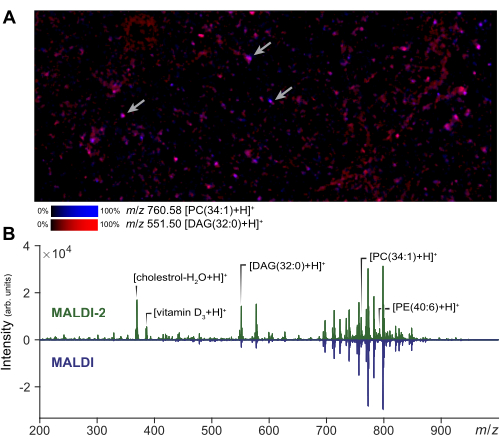
Figura 2: Ottimizzazione e imaging MALDI-2 MS per l'analisi di singole cellule. (A) Imaging MALDI-2 MSI di popolazioni disperse di cellule primarie. Le frecce indicano alcune singole celle. L'esecuzione di MSI e di una piccola regione consente all'analista di ottimizzare i parametri del metodo e valutare la qualità del campione. (B) Confronto dei risultati ottenuti utilizzando le acquisizioni MALDI-2 e MALDI MS. L'aumento del segnale (MALDI 2 vs MALDI) di vari lipidi, tra cui colesterolo, DAG (diacilgliceroli), PC (fosfatidilcolina) e PE (fosfatidiletanolammina), si sta evidenziando mostrando lo spettro MALDI-2 (in alto) e lo spettro MALDI (in basso). Clicca qui per visualizzare una versione più grande di questa figura.
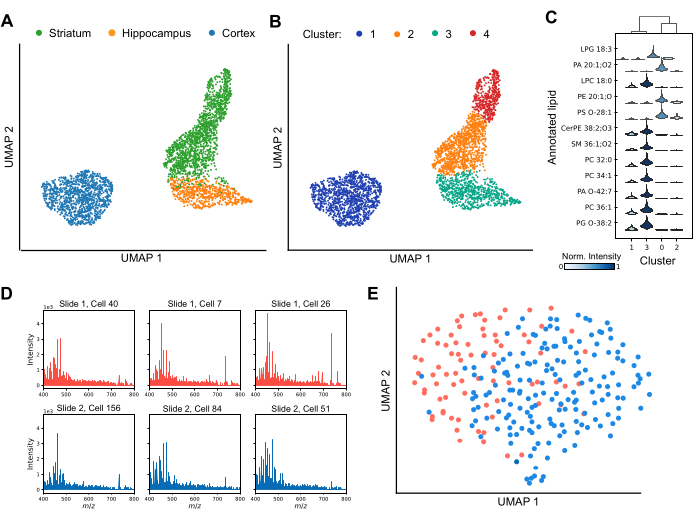
Figura 3: Profilazione lipidica a singola cellula all'interno di tre regioni cerebrali di roditori mediante MALDI-2 MS. (A) analisi UMAP (Uniform Manifold Approximation and Projection) delle cellule analizzate colorate in base alla regione cerebrale di origine. (B) Analisi UMAP delle stesse cellule dopo essere state sottoposte a clustering di Leiden, dove i cluster corrispondono a sottopopolazioni di cellule con composizioni lipidiche simili. I colori qui mostrano diversi cluster di dati di Leida. (C) Un grafico a violino che mostra la differenza nelle distribuzioni di intensità per diversi lipidi tra cluster. (D) Gli spettri di sei singole cellule corticali rappresentative sono stati analizzati da due vetrini separati. (E) Analisi UMAP di cellule corticali analizzate da due vetrini separati. Clicca qui per visualizzare una versione più grande di questa figura.
Fascicolo supplementare 1: SC-MALDI2_Analysis.py. Questo file contiene una serie di funzioni sviluppate per l'elaborazione dei dati di spettrometria di massa MALDI-2 a cella singola. Include metodi per la lettura dei dati grezzi della spettrometria di massa, l'esecuzione del binning e l'esecuzione di annotazioni e analisi a valle. Il set di celle per caricare ed eseguire tutta la pre-elaborazione e l'analisi dei dati è fornito nel file supplementare 2. Clicca qui per scaricare questo file.
File supplementare 2: JOVE Analysis.ipynb. Questo file contiene un notebook Jupyter che contiene un set di celle da caricare nelle funzioni Python personalizzate da SC-MALDI2_Analysis.py ed eseguire tutte le operazioni di pre-elaborazione e analisi dei dati menzionate. Clicca qui per scaricare questo file.
Discussione
La MALDI MS a singola cellula ad alto rendimento e guidata da immagini è uno strumento prezioso per comprendere l'eterogeneità chimica su scala di singola cellula. L'aggiunta della post-ionizzazione indotta da laser (MALDI-2) fornisce una copertura più profonda dell'analita, che è fondamentale per campioni con massa e volume limitati come le cellule isolate di mammifero.
Mentre la stragrande maggioranza dei flussi di lavoro pubblicati per la SM di lipidi e metaboliti a cellula singola utilizza cellule in coltura, il nostro approccio viene applicato a cellule primarie isolate in tempi relativamente brevi. Questa distinzione è fondamentale, poiché le cellule in coltura mostrano un cambiamento dimostrabile nel loro contenuto di metaboliti e lipidi in pochi giorni di coltura23,31. L'isolamento enzimatico delle cellule, nella maggior parte dei casi, porta alla perdita di strutture cellulari fini come assoni, dendriti e processi astrocitici. Pertanto, i segnali dell'analita osservati sono ottenuti principalmente dal soma cellulare. Nonostante ciò, si ottiene una classificazione di successo in base al tipo di cellula4 e alla regione del cervello12. Ciò suggerisce che molti dei lipidi che sono unici per cellule distinte si trovano nel soma.
Piccole differenze nel metodo di applicazione della matrice MALDI possono avere effetti profondi sul segnale dell'analita risultante. Abbiamo presentato un flusso di lavoro basato sulla sublimazione di matrice riproducibile con un apparato commerciale, che genera uno strato di matrice omogeneo, un elevato rapporto segnale/rumore e una diffusione limitata dell'analita. Tuttavia, è possibile utilizzare l'applicazione della matrice utilizzando uno spruzzatore robotico o un aerografo per artisti, tra gli altri approcci. Indipendentemente dall'approccio, una densità di matrice inferiore dovrebbe essere applicata alle preparazioni a singola cellula rispetto a quella utilizzata nell'MSI tissutale standard al fine di ottenere il rapporto matrice ottica/analita. La densità della matrice applicata qui è circa la metà di quella utilizzata per l'MSI tissutale. Naturalmente, ciò implica che la quantità di matrice per cellula è superiore a quella dell'MSI, il che contribuisce a migliorare l'estrazione e il rilevamento dell'analita nell'approccio delle cellule isolate utilizzato in questo caso.
Per i campioni che richiedono un'ottimizzazione significativa, l'esecuzione di MSI su un piccolo ROI di dissociazioni di singole cellule, come dimostrato nella Figura 2A, può essere eseguita come parte del processo di ottimizzazione. Questo passaggio può anche consentire all'analista di valutare l'entità della diffusione dell'analita negli approcci applicativi di matrici a base di solventi.
Il successo del MALDI guidato da immagini di singole cellule richiede immagini a fluorescenza di alta qualità, un'applicazione riproducibile della matrice e un controllo preciso del tavolino. Questi esperimenti sono più impegnativi dei flussi di lavoro MSI standard, che tollerano errori di registrazione minori e variabilità nelle condizioni della matrice e del campione a causa di aree di imaging più grandi e conteggi di analiti più elevati. Nonostante queste sfide, abbiamo individuato con successo singoli organelli utilizzando MALDI tradizionale.
L'unione accurata delle immagini a fluorescenza piastrellate è essenziale, poiché piccoli errori possono aggravarsi e influire sul controllo del tavolino, rendendo fondamentale l'accuratezza della registrazione. Anche gli effetti batch rappresentano una sfida importante, con variazioni di segnale osservate sullo stesso vetrino, tra i vetrini e tra le preparazioni. Strategie di normalizzazione adeguate, tra cui l'uso di standard interni e la normalizzazione interquartile, aiutano a mitigare questi problemi. Inoltre, gli algoritmi di correzione dei lotti come ComBat30 possono essere utilizzati per ridurre la variabilità tecnica e migliorare le differenze biologiche reali.
Ottenere annotazioni molecolari sicure da singole cellule rimane una sfida. Spesso, la quantità di materiale e la sensibilità di rilevamento sono insufficienti per la SM tandem di tutte le specie, tranne le più abbondanti. Per coloro che non sono sufficientemente rilevabili per la SM tandem direttamente dalle singole cellule, è possibile applicare approcci alternativi per creare una libreria di analiti. Ad esempio, i dati sulla SM tandem sui tessuti possono essere ottenuti da sottili criosezioni della stessa regione del cervello e da animali utilizzati per generare popolazioni cellulari. Può essere impiegata anche l'estrazione dei lipidi dai tessuti seguita da LC-MS. Man mano che le strategie di preparazione dei campioni e la tecnologia MS continuano a migliorare, è possibile ottenere informazioni strutturali sempre più rilevanti direttamente dalle singole cellule. In futuro, prevediamo che questo flusso di lavoro sarà esteso per ottenere dati MS tandem da singole cellule, eliminando la necessità di esperimenti LC-MS/MS accessori. Prevediamo inoltre che questo approccio potrebbe essere esteso a numerosi campioni su microscala in biologia e oltre, inclusi tipi di cellule uniche, organelli di mammiferi, polveri e microplastiche.
Divulgazioni
Gli autori non hanno interessi concorrenti da rivelare.
Riconoscimenti
S.W.C riconosce il sostegno fornito dalla Peixin He and Xiaoming Chen PhD4 Fellowship e dalla University of Illinois Block Grant Fellowship. Questo lavoro è stato sostenuto anche dal National Institute on Drug Abuse nell'ambito del premio n. P30DA018310, l'Istituto Nazionale sull'Invecchiamento con il Premio n. R01AG078797, e dall'Ufficio del Direttore, del National Institutes of Health con il numero di premio S10OD032242.
Materiali
| Name | Company | Catalog Number | Comments |
| 2',5'-dihydroxyacetophenone | Sigma Aldrich | D107603 | DHAP, 97% purity |
| Ammonium acetate | Sigma Aldrich | 238074 | ACS reagent, ≥97% |
| Axio M2 Imager | Zeiss | N/A | N/A |
| Biopsy punch, 2 mm | Fisher Scientific | 12-460-399 | integra miltex standard biopsy punch, 2mm |
| Calcium chloride | Sigma Aldrich | C4901 | anhydrous, powder ≥97% |
| Eppendorf Centrifuge | Sigma Aldrich | EP5405000441 | centrifuge 5425 with rotor FA-24x2 |
| Gentamicin | Sigma Aldrich | G1272 | liquid, BioReagent |
| Glass etching pen | Sigma Aldrich | Z225568 | carbide time, pkg of 1 |
| Glycerol | Sigma Aldrich | G7893 | ACS reagent, ≥99.5% |
| HEPES buffer | Sigma Aldrich | H3375 | ≥99.5% (titration) |
| Hoechst 33258 Solution | Sigma Aldrich | 94403 | 1 mg/mL in H2O, ≥98.0% (HPLC) |
| In line HEPA Filter | Sigma Aldrich | WHA67225001 | VACU-GUARD 60 mm disc, 0.45 PFTE housing |
| ITO-Coated Microscopy Slides | Delta Technologies | CG-90IN-S115 | 70-100Ω resistance |
| Magnesium chloride | Sigma Aldrich | M8266 | anhydrous, ≥98% |
| Magnesium sulfate | Sigma Aldrich | 208094 | anhydrous, ≥97% |
| Microcentrifuge tubes | Sigma Aldrich | HS4323K | tube capacity 1.5 mL, pack of 500 |
| Papain dissociation system | Worthington Biochemical | LK003150 | one box, 5 single use vials |
| Penicillin-Streptomycin | Sigma Aldrich | P4458 | liquid, BioReagent |
| Potassium chloride | Sigma Aldrich | 529552 | Molecular biology grade |
| Potassium phosphate monobasic | Sigma Aldrich | P5379 | Reagent Plus |
| Sodium biocarbonate | Sigma Aldrich | S6014 | ACS reagent, ≥99.7% |
| Sodium chloride | Sigma Aldrich | S9888 | ACS reagent, ≥99% |
| Sodium hydroxide | Sigma Aldrich | 221465 | ACS reagent, ≥97%, pellets |
| Sodium phosphate dibasic | Sigma Aldrich | S9763 | ACS reagent, ≥99% |
| Sublimate | HTX | N/A | N/A |
| timsTOF FleX MALDI-2 | Bruker | N/A | microGRID enabled |
| Vacuum tubing | Thermo Scientific | 8701-0080 | Nalgene Non-phthalate PVC Tubing |
Riferimenti
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon