Method Article
Fluoreszenzgesteuerte matrixgestützte Laserdesorption/Ionisation mit laserinduzierter Postionisations-Massenspektrometrie einzelner Nervenzellen von Ratten
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die Verwendung von microMS für die fluoreszenzgesteuerte Einzelzell-MALDI-2-Massenspektrometrie, die ein verbessertes molekulares Profiling von primären neuronalen Zellen der Ratte ermöglicht.
Zusammenfassung
Einzelzellmessungen sind entscheidend für das Verständnis der reichhaltigen raumchemischen Heterogenität des Gehirns. Die Matrix-gestützte Laser-/Desorptionsionisations-Massenspektrometrie (MALDI) ist in der Lage, endogene Moleküle in einzelnen Zellen markierungsfrei und mit hohem Durchsatz zu charakterisieren. Die jüngsten Fortschritte bei der Entwicklung von MALDI-Massenspektrometern mit laserinduzierter Postionisation (MALDI-2) bieten eine stark verbesserte Empfindlichkeit der Detektion für eine Vielzahl von Lipiden und anderen kleinen Molekülen. Die MS-Bildgebung großer Proben mit MALDI-2 in zellulärer Auflösung ist jedoch für die meisten Anwendungen unerschwinglich langsam. Bei diesem Protokoll werden Primärzellen isoliert und auf leitfähigen Objektträgern dispergiert. Die relativen Zellpositionen werden durch Fluoreszenzmikroskopie des gesamten Objektträgers bestimmt, gefolgt von einer genauen Coregistrierung der Mikroskopiekoordinaten mit den Tischkoordinaten des MALDI-2-Massenspektrometers. Die gezielte MS-Analyse nur von Zellstandorten ermöglicht Einzelzellmessungen mit hohem Durchsatz und hoher Analytabdeckung und reduzierter Datengröße im Vergleich zur MS-Bildgebung der gesamten Probe. Wir beschreiben die kritischen Schritte, die für die Einzelzellpräparation, die Fluoreszenzbildgebung des gesamten Objektträgers, die Matrixapplikation und die MALDI-2-Massenspektrometrie erforderlich sind.
Einleitung
Lipide und Metaboliten sind grundlegend für die zelluläre Funktion und dienen als wesentliche Bestandteile von Membranen, Energiequellen und Signalmolekülen 1,2. Ihre Zusammensetzung und Häufigkeit kann jedoch zwischen den einzelnen Zellen erheblich variieren, was die Unterschiede in den Zelltypen und Entwicklungs- und Funktionszuständen widerspiegelt 3,4,5. Die Analyse dieser Unterschiede ist entscheidend für das Verständnis der biologischen Variabilität und die Identifizierung unterschiedlicher Zellsubpopulationen. Einzelzell-Messtechniken, wie z. B. die RNA-Sequenzierung, liefern nützliche zellspezifische Transkriptprofile6. Diese Messungen auf Transkriptebene spiegeln jedoch nicht direkt die tatsächlichen zellulären Mengen an Lipiden und Metaboliten wider, da die Genexpression nicht immer mit der tatsächlichen Häufigkeit dieser Analyten korreliert. Für eine umfassende Analyse der chemischen Zusammensetzung einzelner Zellen und ihrer Populationen sind daher spezialisierte Methoden zur direkten Messung von Lipiden und Metaboliten erforderlich.
Die matrixgestützte Laserdesorption/Ionisation (MALDI) Massenspektrometrie-Bildgebung (MSI) ist ein Werkzeug der Wahl für die markierungsfreie räumliche Kartierung endogener Biomoleküle in situ 7,8. Typischerweise wird bei MALDI ein UV-Laser verwendet, um Material aus einer dünnen Probenschicht abzutragen, die mit einer organischen Matrix cokristallisiert ist und eine Wolke aus Ionen und neutralen Molekülen bildet. Die gebildeten Ionen werden dann mit einem MS-Analysator getrennt und in einem Massenspektrometer detektiert. Aufgrund der genauen Positionierung moderner Massenspektrometertische kann der Laser so positioniert werden, dass er auf bestimmte Probenregionen abzielt, oder über Regionen hinweg gerastert werden, um molekulare Bilder durch MSI zu erzeugen. MSI mit zellulärer oder subzellulärer Ortsauflösung (< 10 μm), die mit einem fokussierten Laserstrahl und einer genauen Tischbewegung erreicht wird, kann verwendet werden, um chemische Informationen über einzelne Zellenzu erhalten 9,10. MSI-Messungen in dieser Größenordnung sind jedoch ineffizient, insbesondere bei Proben mit geringer Zielzelldichte, da viel Zeit für die Bildgebung leerer Bereiche zwischen den Zellen aufgewendet wird. Darüber hinaus ist die Nachweisbarkeit vieler Analyten aufgrund des geringen Probenvolumens eingeschränkt. Um diese Herausforderungen zu meistern, haben wir einen bildgesteuerten MALDI-MS-Ansatz für die Hochdurchsatz-Einzelzellanalyse entwickelt11,12. Bei diesem Ansatz werden die Positionen dispergierter Zellen programmatisch aus Ganzdia-Fluoreszenzbildern bestimmt und dazu verwendet, den Massenspektrometertisch an Positionen zu führen, an denen sich einzelne Zellen mit bestimmten Parametern (z. B. Größe und Form) befinden und dann durch Bestrahlung mit dem MALDI-Laser analysiert werden. Frühere Arbeiten mit diesem gezielten Ansatz wurden zur Charakterisierung von Lipiden, Peptiden und anderen Biomolekülen in heterogenen Zellpopulationen verwendet 5,13.
Angesichts der begrenzten Masse einzelner Zellen ist die Anzahl der in solchen Proben nachgewiesenen Analyten im Allgemeinen geringer als die Anzahl der direkt aus Gewebe beobachteten. Um die Analytabdeckung in der Einzelzell-MS-Analyse zu erhöhen, ist es daher entscheidend, die Empfindlichkeit der Analytdetektion zu erhöhen. Ein kürzlich entwickelter Ansatz, der bei der Überwindung dieser Detektionsherausforderung hilft, ist MALDI mit Laser-Nachionisation (MALDI-2), der nachweislich die Empfindlichkeit für ein breites Spektrum von Analytenerhöht 9,14,15,16. Infolgedessen generiert MALDI-2 umfassendere Einzelzelldatensätze und bietet eine tiefere molekulare Abdeckung in massenbegrenzten Proben, wie z. B. isolierten Zellen.
Ziel dieser Methode ist es, Lipidmessungen von Tausenden von einzelnen Zellen zu erhalten. Zu diesem Zweck beschreiben wir einen Arbeitsablauf, der eine Hochdurchsatz-Einzelzell-MALDI-2-Massenspektrometrie ermöglicht und im Allgemeinen auf jeden sondenbasierten Massenspektrometrie-Ansatz mit präziser Tischkontrolle erweitert werdenkann 17. In diesem Arbeitsablauf wird Gewebe aus der/den interessierenden Hirnregion(en) präpariert, und einzelne Zellen werden nach einem Papain-Dissoziationsverfahren aus dem Gewebe gewonnen. Die Zellen werden dann mit einem Kernfleck markiert und auf leitfähige Objektträger dispergiert, die mit Passermarken geätzt sind, wo sie haften bleiben. Als nächstes werden ganze Objektträgerbilder mit Hilfe der Fluoreszenzmikroskopie aufgenommen. Die Matrix wird durch Sublimation abgeschieden, wodurch eine wiederholbare homogene Kristallschicht und ein hohes Signal-Rausch-Verhältnis für die Einzelzell-MS-Analyse erzeugt werden. Mit der Open-Source-Software microMS11 werden die relativen Koordinaten der Zellstandorte aus dem Mikroskopiebild kartiert und mit den Koordinaten des Massenspektrometertisches abgeglichen, indem eine Punktsatzregistrierung verwendet wird, bei der Passermarkenmarker verwendet werden, die auf die Objektträger geätzt werden, in denen die Zellen abgelegt wurden. Schließlich werden unter Verwendung dieser Informationen präzise, zielgerichtete MS-Spektren von jeder einzelnen Zelle erfasst, so dass Tausende von Zellen in einem einzigen Lauf (<1 h) profiliert werden können12,13.
Protokoll
Alle Tierversuche in dieser Studie wurden in Übereinstimmung mit dem Tierverwendungsprotokoll durchgeführt, das vom Illinois Institutional Animal Care and Use Committee (23228) genehmigt wurde, unter strikter Einhaltung sowohl der nationalen als auch der ARRIVE-Standards für die ethische Behandlung und Pflege von Tieren.
1. Vorbereitung von Materialien und Lösungen
- Bereiten Sie modifizierte Gey's balanced salt solution (mGBSS) mit 1,5 mM CaCl2, 4,9 mM KCl, 0,2 mM KH2PO4, 11 mM MgCl2, 0,3 mM MgSO4, 138 mM NaCl, 27,7 mM NaHCO3, 0,8 mM Na2HPO4 und 25 mM HEPES vor. Stellen Sie den pH-Wert mit 3 M NaOH auf 7,2 ein. Es wird empfohlen, 1 l mGBSS vorzubereiten und bis zur Verwendung bei 4 °C zu lagern.
- Bereiten Sie 50-ml-Spritzen vor, indem Sie sie mit der vorbereiteten mGBSS-Lösung füllen und für die transkardiale Perfusion auf Eis legen. Bereiten Sie 1 Spritze für jede sezierte Ratte vor.
- Bereiten Sie Aliquots der Papain-Lösung nach dem Protokoll des Worthington Papain-Dissoziationssystems vor. Dieses System enthält vier Fläschchen, wie unten beschrieben, wobei Fläschchen 4 für dieses Protokoll nicht verwendet wird:
Fläschchen 1: sterile Earle's Balanced Salt Solution (EBSS) mit Bikarbonat und Phenolrot
Fläschchen 2: Papain mit L-Cystein und EDTA
Fläschchen 3: Desoxyribonuklease I (DNase)
Fläschchen 4: Ovomucoid-Proteasehemmer mit Rinderserumalbumin- Geben Sie 5 mL EBSS (Fläschchen 1) in das Papain-Fläschchen (Fläschchen 2) und legen Sie es für 10 Minuten in ein 37 °C heißes Wasserbad, um das Papain aufzulösen.
- 500 μl EBSS (Fläschchen 1) in das DNase-Fläschchen (Fläschchen 3) geben und vorsichtig mischen. 250 μl der verdünnten DNase-Durchstechflasche (Durchstechflasche 3) in die Durchstechflasche Papain (Durchstechflasche 2) geben. Dieses Präparat enthält eine Endkonzentration von 20 Einheiten/ml Papain, 0,005 % DNase, 0,5 mM EDTA und 1 mM L-Cystein.
- Frieren Sie 250 μl Aliquote dieser vorbereiteten Papainlösung für nachfolgende Dissoziationen ein (ergibt ~20 Aliquote). Ergänzen Sie diese Lösung mit den folgenden Antibiotika: 100 Einheiten/ml Penicillin G, 100 μg/ml Streptomycin und 100 μg/ml Gentamicin. Dies kann für die Anwendung des Benutzers geändert werden.
- Bereiten Sie eine 150 mM Ammoniumacetatlösung vor und lagern Sie sie bei 4 °C. Bereiten Sie eine 33%ige Glycerinlösung vor, indem Sie Glycerin in die vorbereitete mGBSS-Lösung verdünnen.
- Ätzen Sie Passermarken auf mit Indiumzinnoxid (ITO) beschichtete Objektträger, indem Sie entweder einen Gravierstift mit einer Wolframkarbid- oder Diamantspitze oder einen CO2 -Lasergravierer verwenden. Ätzen Sie auf der leitfähigen Seite des ITO-Objektträgers 20-40 X-förmige Passermarkenmarker entlang der Peripherie des Objektträgers, um eine plattformübergreifende (z. B. Mikroskop und Massenspektrometer) räumliche Registrierung zu ermöglichen.
HINWEIS: Achten Sie darauf, dass Sie nicht in eine verbundene Leitung ätzen, die die Probe oder ihre Bereiche umgibt, da dies die Oberflächenleitfähigkeit zwischen dem Bereich und einem Glasschiebeadapter stört.
2. Präparation der primären Nervenzellen
HINWEIS: Das Hippocampusgewebe der Ratte wird präpariert, mit Papain in einzelne Zellen dissoziiert und bei geringer Dichte auf leitfähigen Glasobjektträgern abgelagert. Die Isolierung von Zellen auf diese Weise ermöglicht die Hochdurchsatz-Einzelzell-Massenspektrometrie von endogenen Lipiden.
- Vor der Sektion von Tieren wird ein Aliquot der vorbereiteten Papainlösung aufgetaut und in einen Behälter mit einem Ein- und Auslassrohr gegeben, so dass CO2 die Luft im Behälter verdrängen kann. Stellen Sie dieses System in ein auf 37 °C eingestelltes Wasserbad und setzen Sie den CO2 -Fluss fort, bis die Lösung den richtigen pH-Wert hat, wie durch die Farbe angezeigt, unter Verwendung der pH-Farbkarte, die in der Verpackung des Worthington Papain Dissoziationssystems enthalten ist. Der angestrebte pH-Wert liegt zwischen 7,2 und 7,4.
HINWEIS: Blasen Sie CO2 nicht in die enzymatische Lösung, da dies zu einer Denaturierung der DNase und zum Verlust der biologischen Aktivität führt. Verwenden Sie ein 50-ml-Zentrifugenröhrchen mit zwei Röhrchen, die in den Deckel eingeklebt sind, so dass CO2 das Röhrchen von unten füllen und aus dem oberen Auslassröhrchen entweichen kann. - Euthanasieren Sie Ratten(n) in Übereinstimmung mit den IACUC-Protokollen gemäß den institutionellen, lokalen und bundesstaatlichen Richtlinien. Hier werden 2 bis 3 Monate alte männliche Sprague-Dawley-Ratten verwendet und durch CO2 -Erstickung getötet.
- Die Ratte wird transkardial mit eiskalter (4 °C) mGBSS-Lösung perfundiert, um Blut aus dem Gefäßsystem zu entfernen und den Körper des Tieres schnell zu kühlen. Ein Beispiel für dieses Vorgehen findet sich im Protokoll von We et al.18.
- Isolieren Sie das Gehirn chirurgisch von der Ratte und achten Sie darauf, die interessierende Gehirnregion nicht zu beschädigen. Eine zuvor veröffentlichte Übersicht über diese Isolation enthält weitere Details zu einigen der einzelnen Schritte19.
- Verwenden Sie einen Gewebehacker oder einen Gewebewürfel, um einen Querschnitt (~2 mm dick) des Gehirns zu schneiden, um die Region (Hippocampus, Kortex oder Striatum) für die Probe freizulegen.
- Verwenden Sie einen 2-mm-Biopsiestempel, um die gewünschte Gehirnregion zu entnehmen, und geben Sie das Gewebestück in das Fläschchen mit Papainlösung.
- Inkubieren Sie dieses Gewebe und die Papainlösung bei 37 °C unter ständigem Rühren (z. B. durch eine elektrische Wippe) für 30-90 Minuten.
HINWEIS: Die enzymatische Behandlungsdauer sollte auf der Grundlage der Gehirnregion, des Tierstamms, des Alters und der gewünschten Zellleistung optimiert werden (z. B. Neuronen und Astrozyten mit Terminals im Vergleich zur maximalen Ausbeute von Zellen ohne Terminals). Für Hippocampusgewebe der Ratte sind 60-90 min typisch. Kürzere Behandlungen können einige Endterminals erhalten, aber viele Zellen in undissoziierten Clustern belassen, während längere Behandlungen die Ausbeute verbessern, aber die Zellschädigung erhöhen können, was eine Bewertung mit Lebend-/Tot-Assays erfordert (z. B. LIVE/DEAD-Viabilitäts-/Zytotoxizitätskit). Für vergleichende Studien müssen die Behandlungen über alle Stichprobentypen hinweg konsistent sein. - Legen Sie ein Fläschchen mit Taschentuch und Papain auf Eis, um die enzymatische Aktivität zu kühlen und zu unterdrücken. Zerreiben Sie das Gewebe mit einer abgeschnittenen Pipettenspitze, die an einer P100-Pipette mit einstellbarem Volumen befestigt ist, bis der größte Teil des Gewebes visuell aufgebrochen ist. Bilden Sie während der Dissoziation keine Luftblasen, um übermäßige Zellschäden zu vermeiden.
HINWEIS: Eine kräftige Verreibung führt zu einer hohen Ausbeute an Zellen; Die meisten ihrer Prozesse behalten sie jedoch nicht bei. Die sanfte Verreibung sorgt für mehr Prozesse an den Zellen, führt aber zu mehr undissoziierten Zellen. Auch Pipettenspitzen unterschiedlicher Größe und andere Methoden können verwendet und für ein optimales Dissoziationsergebnis optimiert werden20. - Zentrifugieren Sie die dissoziierten Zellen bei 300 x g für 5 min bei Raumtemperatur, um die Zellen zu pelletieren. Entfernen Sie den Überstand und resuspendieren Sie das Pellet in 1 mL mGBSS. Hoechst 33342 Kernfärbung bis zu einer Endkonzentration von 1 μg/ml zugeben.
- Pipettieren Sie 100-500 μl der Zellsuspension auf geätzte ITO-Objektträger und lassen Sie sie etwa 5 Minuten oder länger ruhen, damit die Zellen sedimentieren und am Objektträger haften können.
- Die Verdünnung der Zellsuspension muss empirisch bestimmt werden. Geben Sie mit einem optischen Mikroskop ein Tröpfchen der Zellen auf den Objektträger, um die grobe Dichte zu bestimmen, und verdünnen Sie von dort aus, um sicherzustellen, dass genügend Abstand zwischen den Zellen vorhanden ist (d. h. ausreichend größer als die MALDI-Strahlgröße). Diese Verdünnung kann am ersten Objektträger durchgeführt werden, und dann kann die Verdünnung bei Bedarf für nachfolgende Objektträger angepasst werden.
- Saugen Sie die Zellsuspension vorsichtig mit einem Vakuumsystem vom Objektträger ab, das Folgendes enthält (in der Reihenfolge von der Vakuumleitung aus): 1) Schlauch mit einem Inline-Gas-HEPA-Filter, 2) Schlauch auf einem 50-ml-Filterkolben, 3) Schlauch, der mit einem zweiten Filterkolbeneinlass mit einer angebrachten Pipettenspitze (z. B. 100 μl) verbunden ist. Wenn Sie die plattierten Zellen für die zukünftige Verwendung aufbewahren, ersetzen Sie die aspirierte Suspension 1-2 Minuten lang durch 33%ige Glycerinlösung und entfernen Sie sie.
HINWEIS: Dieser Schritt stellt die Zellstabilität sicher. Die Abgabe von Glycerinlösung von der einen Seite bei sanftem Aspirieren von der anderen Seite ist optimal für die Aufrechterhaltung der Zellstruktur. Überwachen Sie alle hier beschriebenen Schritte während der Vorbereitung der ersten Probe immer visuell mit einem inversen Mikroskop. - Tragen Sie 100-500 μl Ammoniumacetat auf die abgelagerten Bereiche auf, lassen Sie es etwa 1 Minute einwirken und saugen Sie dann ab, um die Objektträger zu reinigen. Wiederholen Sie den Vorgang bei Bedarf, um sicherzustellen, dass die Objektträger frei von Salz oder Glycerin sind.
3. Mikroskopie
HINWEIS: Um die Position der abgelagerten Zellen zu bestimmen, wird jeder Objektträger durch Hellfeld-/Fluoreszenzmikroskopie abgebildet. Der Fluoreszenzkanal ermöglicht die genaue Lokalisierung von Hoechst-gefärbten Zellen, während die Hellfeld-Bildgebung morphologische Informationen liefert. Jedes Mikroskop, das in der Lage ist, gekachelte Bilder zu erfassen, ist für diesen Prozess geeignet.
- Falls noch nicht geschehen, spülen Sie die Objektträger mit 2-3 ml 150 mM Ammoniumacetat aus. Dieser Schritt ist entscheidend, um Glycerin- und Salzkristalle zu entfernen, die die Mikroskopie und die Anwendung der MALDI-Matrix beeinträchtigen können. Trocknen Sie die Folie unter einem sanften Strahl N2 oder lassen Sie sie an der Luft trocknen.
- Laden Sie den Objektträger in den Mikroskoptisch. Fokussieren Sie das Mikroskop mit mindestens 10 Stützpunkten, die gleichmäßig über den Objektträger verteilt sind.
- Unter Verwendung von Filtern, die für DAPI (Anregung: 335-383 nm, Emission: 420-470 nm) und Hellfeld-Bildgebung geeignet sind, erhalten Sie ein gekacheltes Fluoreszenzbild mit 5-facher bis 10-facher Vergrößerung über den gesamten Objektträger, wodurch sichergestellt wird, dass die (in Schritt 1.5 erstellten Passermarken) erfasst werden.
HINWEIS: Eine 5-fache Vergrößerung ist in der Regel ausreichend, insbesondere wenn viele Dias abgebildet werden müssen. Um eine feinere zelluläre Morphologie aufzuklären, können 10x- oder 20x-Objektive verwendet werden, was zu einer quadratisch längeren Bildgebungszeit und einer Zunahme der Dateigrößen führt. - Fügen Sie das gekachelte Bild mit einer Mikroskopiesoftware wie ZEN (Zeiss) zusammen. Ein genaues Stitching ist von entscheidender Bedeutung, da alle Fehler in diesem Schritt auf den letzten Registrierungsschritt übertragen werden, was zu einem ungenauen Targeting führt. Stellen Sie zur Überprüfung sicher, dass vertikal benachbarte Kacheln nicht versetzt sind. Darüber hinaus sollten Sie gekachelte Bilder mit einer großen Kachelüberlappung (20 % bis 30 %) erwerben.
- Verarbeiten und exportieren Sie jedes Bild als bigTIFF-Datei in der Mikroskopie-Software. Wenn der abgebildete Bereich relativ klein ist und zu einer endgültigen Bildgröße von weniger als 2 GB führt, verwenden Sie stattdessen ein Standard-TIFF-Dateiformat.
4. Matrix-Anwendung
HINWEIS: Eine konsistente und korrekte Anwendung der MALDI-Matrix ist entscheidend für die Erzielung qualitativ hochwertiger Einzelzelldaten. Während hier die Sublimation unter Verwendung einer kommerziellen Vorrichtung verwendet wird, kann das Auftragen der Matrix auch unter Verwendung eines Robotersprühgeräts12, einer Airbrush21 oder einer selbstgebauten Sublimationsvorrichtung22 durchgeführt werden. Wir haben festgestellt, dass Einzelzellpräparate weniger Matrix benötigen als dünne Gewebeschnitte, die typischerweise für die massenspektrometrische Bildgebung verwendet werden. Um Batch-Effekte zu reduzieren, wird empfohlen, die Matrix während einer Sitzung auf alle zu untersuchenden Objektträger anzuwenden und Zellen aus verschiedenen Gruppen (z. B. Hirnregion oder Behandlung vs. Kontrolle) nach Möglichkeit auf demselben Objektträger zu deponieren. Die Auswahl der Matrix ist sowohl für traditionelle MALDI- als auch für MALDI-2-Einzelzell-Workflows von entscheidender Bedeutung. Für einzelliges MALDI wurden DHB23, 9-AA24 und CHCA25 erfolgreich eingesetzt. In MALDI-2 haben wir und andere eine signifikante Signalverstärkung mit DHAP9 beobachtet, während Matrizen wie NEFZ16 und CHCA26 ebenfalls effektiv angewendet wurden.
- Löse 20 mg 2,5-Dihydroxyacetophenon (DHAP) in 1,5 ml Aceton auf. Legen Sie die Objektträger in die Halterung der Sublimationskammer und legen Sie sie in die Sublimationsapparatur.
- Pipettieren Sie die gelöste Matrixlösung auf den Keramikwafer und lassen Sie das Aceton verdampfen. Schließen Sie die Sublimationskammer, um sie zu verschließen.
- Füllen Sie die Kühlmittelkammer mit einem Eiswasser-Slush und legen Sie ihn auf die Kammer.
- Schalten Sie das Vakuum ein und lassen Sie das System 5 Minuten lang äquilibrieren. Der Druck im System sollte weniger als 40 mbar betragen.
- Beginnen Sie die Sublimation, indem Sie die Kammer 5 Minuten lang auf 200 °C erhitzen.
- Entfernen Sie das Eiswasserbad, drehen Sie die Temperatur auf 25 °C und stellen Sie einen Kühlkörper auf die Kammer. Lassen Sie das System 5 Minuten lang auf Raumtemperatur erwärmen, um Kondensation zu vermeiden.
HINWEIS: Der Behälter mit dem Eiswasserbad kann geleert und mit heißem Wasser gefüllt werden, um als Kühlkörper zu dienen. - Entlüften Sie das System langsam, öffnen Sie die Kammer und entfernen Sie die Objektträger.
5. Einzelzellige MALDI MS
HINWEIS: Einzelzell-MS-Daten werden mit einem MALDI-2 timsTOF-Gerät (timsTOF flex) unter Verwendung des Open-Source-microMS-Pakets zur Erkennung von Zellen und zur Führung des Massenspektrometers gewonnen. Dies erfordert, dass die optischen Bildpixelpositionen der Zielzellen in die physikalischen Koordinaten des Massenspektrometertisches übersetzt werden.
- Öffnen Sie microMS und dezimieren Sie die bigTIFF-Mikroskopiebilder mit der Option Bildgruppe, da es sowohl Hellfeld- als auch Fluoreszenzkanäle gibt.
- Identifizieren Sie mit dem Tool Blob-Optionen die optimalen Parameter, die für die Zielzellen ausgewählt werden sollen. Passen Sie die folgenden Parameter an: die maximale und minimale Blob-Größe, um auszuwählen, wie klein oder groß die Blobs gefunden werden sollen. die Schwellenwerteinstellung, die bestimmt, wie der Schwellenwert des Fluoreszenzkanals für die Blob-Erkennung festgelegt wird; und die Zirkularität, die bestimmt, wie kreisförmig die identifizierten Zellen sein müssen, um in Betracht gezogen zu werden. Für die hier vorgestellten Daten haben wir die folgenden Parameterwerte verwendet: maximale Zirkularität: keine; Min. Zirkularität: 0,6; Schwelle: 75; Mindestgröße: 20; Maximale Größe: Keine.
- Verwenden Sie die Option Blob suchen, um Blobs zu suchen, und das Popup-Fenster, um die Blobliste unter einem gewünschten Namen zu speichern.
- Verwenden Sie das Werkzeug Entfernungsfilter, um einen bestimmten Abstand auszuwählen, der für jede Zelle voneinander entfernt sein muss (für diese Daten haben wir mit Hilfe der 5-fachen Fluoreszenzmikroskopie einen Entfernungsfilter von 35 Pixeln verwendet). Dadurch soll sichergestellt werden, dass die Zellen weit genug voneinander entfernt sind, um eine Einzelzellanalyse durchzuführen. Diese Zahl wird in Pixeleinheiten angegeben, also kennen Sie die Umrechnung von Pixel in μm für das Mikroskop, um die ideale Zahl zu bestimmen. Verwenden Sie die Größe des Lasersondendurchmessers plus ca. 20-30 μm, um Fehler bei der Registrierung zu berücksichtigen.
- Legen Sie die Objektträger mit dem MTP Slide Adapter II in das Gerät ein. Kehren Sie mit microMS zum Computer zurück und greifen Sie mit einer Remote-Desktop-Anwendung auf den Massenspektrometer-Computer zu.
- Wenn das Instrument zum ersten Mal verwendet wird, überprüfen Sie die X-Y-Position des Instruments. Gehen Sie dazu zu Tools und Instrumenteneinstellungen. Das Popup-Fenster zeigt einen Satz von Koordinaten mit ihren X- und Y-Positionen an. Wählen Sie jeden dieser spezifischen Punkte auf der Schiebeadapter-II-Geometrie am Gerät aus und aktualisieren Sie die X- und Y-Positionen im microMS-Fenster.
- Navigieren Sie mit der Kamera und den Tischsteuerungen des Massenspektrometers zu einer leicht erkennbaren Stelle auf dem Objektträger und kopieren Sie die Koordinaten des Geräts. Suchen Sie in microMS die entsprechende Position im Mikroskopbild, klicken Sie mit der rechten Maustaste auf diese Position und geben Sie die Koordinaten in die Textposition des Popup-Fensters ein. Runden Sie alle Koordinaten auf die nächste ganze Zahl, und trennen Sie die X- und Y-Koordinaten durch Leerzeichen (z. B. 39595 -23232).
- Wiederholen Sie Schritt 5.7. um mindestens 12 Passermarken auf der gesamten Folie zu registrieren. Nachdem drei Registrierungspunkte hinzugefügt wurden, färbt sich einer der Kreise rot, was darauf hinweist, dass er die am weitesten von der Registrierung abweichende Position ist. Löschen Sie in diesem Fall den aktuellen Registrierungspunkt mit Umschalt + Rechtsklick, und versuchen Sie es erneut.
- Wechseln Sie unter Datei zu Speichern und dann zu Registrierung, um die Registrierungsdatei (MSREG) zu speichern.
- Wenn die Blobs auf der Folie angezeigt werden, speichern Sie die Datei mit der Instrumentenposition (.xeo), indem Sie zu Datei, dann zu Speichern und dann zu Instrumentenpositionen wechseln. Übertragen Sie diese .xeo-Datei mit Hilfe der Remote-Desktop-Software auf den Gerätecomputer.
- Kopieren Sie den Text aus der benutzerdefinierten .xeo-Datei, die in die .xeo-Datei des MTP Slide Adapter II auf dem Instrument generiert wurde, und speichern Sie sie. Dadurch wird diese Geometriedatei mit den Positionen der Zellen aktualisiert.
- Klicken Sie auf die Registerkarte Automatisierung und wählen Sie Neu..., um eine automatische Ausführung zu starten. Ziehen Sie über den angezeigten Probenbereich, um die Zellen auszuwählen, und klicken Sie mit der rechten Maustaste, um sie der Analyseliste hinzuzufügen. Speichern Sie den Automatisierungslauf und klicken Sie auf Automatischen Lauf starten , um die Erfassung zu starten. Die Länge des Laufs hängt von einer Vielzahl von Faktoren ab, darunter die Anzahl der Zellen, die Laserschüsse und die Laserfrequenz, aber im Allgemeinen können im MALDI-Modus (10 kHz Laserwiederholrate) etwa 5-10 Zellen analysiert werden, und 1-5 Zellen können im MALDI-2-Modus (1 kHz Laserwiederholrate) pro Sekunde analysiert werden.
6. Datenverarbeitung
HINWEIS: Bestehende kommerzielle Softwarepakete eignen sich nicht gut für die Analyse von Einzelzell-Massenspektrometriedaten mit hohem Durchsatz. Während einzelne Spektren visualisiert werden können, erfordert die Extraktion aussagekräftiger biologischer Erkenntnisse spezielle Werkzeuge. Um dieses Problem zu lösen, stellen wir frei verfügbare Software zur Verfügung, die die Analyse von Einzelzell-MALDI-2-MS-Daten ermöglicht. Unser aktualisierter Workflow erleichtert die direkte Konvertierung von Einzelzelldaten in das Open-Source-imzML-Format27 und ermöglicht so die Kompatibilität mit SCiLS MVS und anderer Anbietersoftware. Für eine erweiterte Datenanalyse enthält das vollständige Skript auch Funktionen für Lipidannotation, Clustering und andere Visualisierungswerkzeuge, die von Matplotlib (Version 3.7.3) aktiviert werden. Das Parsen und Lesen der Rohdaten wird durch die pyTDFSDK-Bibliothek erleichtert, eine Reihe von Funktionen, die im TIMSCONVERT-Workflow28 enthalten sind.
- Initialisieren Sie die benutzerdefinierte Python-Bibliothek SC_MALDI2_Analysis, indem Sie die in der Zusatzdatei 1 bereitgestellte Datei in denselben Arbeitspfad wie Jupyter Notebook kopieren und dann UMSCHALT+EINGABETASTE auf der Tastatur in der ersten Codezelle drücken. Dies ermöglicht es uns, diese Funktionen später aufzurufen und zur Verarbeitung der Daten zu verwenden.
- Kopieren Sie den Pfad zu den gesammelten Daten (Bruker .d-Dateien). Es wird empfohlen, die Ordner und Namen der einzelnen Dateien beschreibend zu benennen, damit wir diese Dateipfade später analysieren können, um Metadaten für jedes Beispiel zu erstellen. Führen Sie das Importieren der Bruker-.d-Dateien aus, und speichern Sie es in einer Listencodezelle. Dadurch wird die Gesamtzahl der geladenen .d-Dateien ausgegeben.
- Führen Sie die Codezelle mit dem Titel Verarbeiten der Rohdaten und Speichern in imzML aus. Dadurch wird jede bereitgestellte Bruker-.d-Datei gelesen, ein Peak-Picking für die Daten durchgeführt und dann 20 ppm Daten um jeden Peak herum in einer .imzML-Datei gespeichert, die in die .d-Datei geschrieben wird.
- Um die gespeicherten Daten zu finden, öffnen Sie den Speicherort der ersten .d-Datei und die imzML und andere Metadaten werden in diesem Pfad gespeichert. Zu diesem Zeitpunkt kann die gespeicherte imzML-Datei in Herstellersoftware, wie z. B. SCiLS Lab MVS, importiert werden. Für eine erweiterte Datenanalyse fahren Sie mit dem Skript fort.
- Laden Sie die gespeicherten imzML-Daten, indem Sie den Befehl Laden in der zuvor gespeicherten imzML-Dateicodezelle ausführen. Dadurch werden die gespeicherten Daten in die variablen Daten geladen.
- Führen Sie die Zelle Binning der Daten und Speichern der Data Matrix aus, um alle Daten entlang einer gemeinsamen m/z-Achse zu sortieren, Features zu entfernen, die in geringer Häufigkeit (<0,1 %) vorhanden sind, und Werte zu entfernen, die kleiner als Rauschen sind (100 relative Intensität). Diese Funktion ordnet die Daten dann neu, indem sie jeden m/z-Spitzenwert innerhalb jedes Abschnitts mittelt. Die resultierende Datenmatrix wird als datacube_array gespeichert, die der Matrix zugeordneten m/z-Klassen werden als valid_mz_bins gespeichert, und die Zählung jeder Klasse wird als Anzahl gespeichert.
- Um die Daten mit Anmerkungen zu versehen, führen Sie die Annotationszelle Peaks für LIPID MAPS exportieren aus. Dadurch werden alle m/z-Peaks innerhalb Ihrer Datenmatrix in Ihre Zwischenablage kopiert. Fügen Sie diese in eine leere Textdatei ein und speichern Sie sie, um sie auf LIPID MAPS hochzuladen.
- Öffnen Sie lipidmaps.org, und wählen Sie dann MS Data Bulk Search und Search LMSD aus, um nach biologisch relevanten Lipidspezies zu suchen. Laden Sie die Peakliste hoch, indem Sie die Textdatei auswählen und die entsprechenden Addukte auswählen (z. B. [M+H]+, [M+Na]+ und [M+K]+). Wählen Sie die für Ihr Massenspektrometer geeignete Massentoleranz aus (wir haben +/- 0,01 m/z gewählt), wählen Sie alle gewünschten Lipidklassen aus und klicken Sie auf Suchen. Laden Sie die TSV-Datei herunter.
- Laden Sie die Spektrenmetadaten, indem Sie das Laden in den einzelnen Zellpfaden für Metadatenzellen ausführen. Ändern Sie hier den geteilten Code basierend auf den Dateipfadstrukturen.
- Verarbeiten Sie die Daten vor, indem Sie die Zelle Datenvorverarbeitung und Lipidannotation ausführen. Dabei wird die lipidMAPS-TSV-Datei verwendet, um den Datensatz mit einem Fehler von 10 ppm zu annotieren, ungerade Lipide aus den lipidMAPS-Ergebnissen zu entfernen, die verbleibenden Daten zu normalisieren und Merkmale oder Zellen zu entfernen, die einen bestimmten Schwellenwert nicht überschreiten. Dadurch wird der neu verarbeitete Datensatz als Daten2 und die aktualisierten Spektrenmetadaten als updated_cell_types gespeichert.
- Die Analyse der Daten erfolgt durch scanpy. Führen Sie die Zelle UMAP- und Clustering-Analyse für einzelne Zellen aus, um relevante Daten auszuwählen (hier haben wir nur MALDI-2-, M2-Daten ausgewählt), skalieren Sie das Dataset und laden Sie es in ein scanpy AnnData-Objekt, um die Analyse zu vereinfachen.
HINWEIS: Hier wurde Leiden Clustering and Dimensionality Reduction (UMAP) für den Datensatz durchgeführt, sowohl die ursprünglichen Datenklassen als auch die Cluster auf dem UMAP wurden visualisiert, und einige der statistisch signifikantesten Merkmale unter den Clustern wurden mit der Funktion rank_genes_groups_dotplot von scanpy zusammengefasst.
Ergebnisse
Abbildung 1 gibt einen Überblick über den Arbeitsablauf für die fluoreszenzgesteuerte einzellige MALDI-2-MS. Zunächst wird das Gewebe, das aus den Zielregionen des Gehirns präpariert wird (Abbildung 1A), in einzelne Zellen dissoziiert und auf leitfähigen, ITO-beschichteten Mikroskopieobjektträgern abgelagert (Abbildung 1B). Die Positionen der Zellen werden durch Fluoreszenzbildgebung des gesamten Objektträgers (Abbildung 1C) bestimmt, gefolgt von der Anwendung der MALDI-Matrix (Abbildung 1D), der microMS-gestützten MALDI-2-MS-Analyse (Abbildung 1E) und der Datenanalyse (Abbildung 1F). Mit diesem Arbeitsablauf können mutmaßlich Dutzende bis Hunderte von Lipiden in einzelnen Zellen mit einer Massengenauigkeit von weniger als 10 ppm (unter Verwendung eines MALDI-2 timsTOF) identifiziert werden.
Die massenspektrometrische Bildgebung kann an einer kleinen Region von Interesse durchgeführt werden, um die Qualität der Zellvorbereitung und der Matrixanwendung zu beurteilen. Ein repräsentatives Ergebnis der MALDI-2-Bildgebung von dispergierten Zellen ist in Abbildung 2A dargestellt. Anhand dieses Bildes kann der Benutzer das Ausmaß der Ausbreitung des Analyten im Vergleich zu einer Mikroaufnahme derselben Region beurteilen. Basierend auf dem Ausmaß der Streuung kann der Analyst die Größe des Laserstrahlfelds und den Abstandsfilter anpassen, um eine echte Einzelzellerfassung zu gewährleisten. Bei der Einzelzell-MSI wird eine kleine Lasergröße und Rasterbreite verwendet, um eine zelluläre Auflösung (1-5 μm) zu erhalten, während bei microMS eine größere Lasergröße verwendet werden kann, wodurch die Signalintensität und die Anzahl der nachweisbaren Lipide im Vergleich zur Bildgebung erhöht werden. Repräsentative Ergebnisse der mit MALDI-2 erzielten Signalverstärkung sind in Abbildung 2B dargestellt. Eine Erhöhung von Cholesterin-, PE- und PC-Spezies wird in Übereinstimmung mit der bisherigen Literatur beobachtet14. Im Allgemeinen erzielen wir die beste MALDI-2-Signalverstärkung, wenn wir eine relativ hohe Laserleistung (>50%) und eine geringere Anzahl von Schüssen (etwa 10 bis 200) verwenden. Einige Optimierungen sind erforderlich, da eine zu geringe Laserleistung keine Verbesserung bewirkt, während zu viel Laserenergie zu einer übermäßigen Analytfragmentierung führt29. Bei der Einzelzellanalyse ist die räumliche Registrierung ein entscheidender Schritt, um eine genaue Ausrichtung einzelner Zellen zu gewährleisten. Um die Registrierung zu validieren, kann ein kleiner Satz von Testpunkten (~5) um den Objektträger herum generiert werden, indem Blobs an bekannten Stellen erstellt und die Instrumentenkoordinaten über microMS exportiert werden. Die Überprüfung, ob diese Testpunkte den Instrumententisch bei der Auswahl korrekt positionieren, stellt sicher, dass die nachfolgende Zellenausrichtung genau ist.
MALDI MS ermöglicht die Profilierung der zellulären Heterogenität sowohl zwischen als auch innerhalb einzelner Hirnregionen (Abbildung 3). Durch die Analyse von Variationen in den Lipidprofilen trennen Techniken zur Dimensionalitätsreduktion die Zellen effektiv in unterschiedliche Cluster, wodurch die Vielfalt der Lipidzusammensetzungen im gesamten Datensatz hervorgehoben wird (Abbildung 3A). Darüber hinaus zeigt die Anwendung von Clustering-Techniken, wie z. B. dem Leiden-Clustering, das Vorhandensein einzigartiger Subpopulationen von Zellen innerhalb bestimmter Hirnregionen, einschließlich des Striatums und des Kortex (Abbildung 3B). Darüber hinaus können durch den Vergleich der Lipidprofile der einzelnen Cluster miteinander (Abbildung 3C) spezifische Lipide als Marker zur Definition clusterspezifischer Identitäten verwendet werden. Einige Lipidspezies sind in bestimmten Clustern signifikant hoch- oder herunterreguliert, was auf unterschiedliche funktionelle Rollen oder Zustände hindeutet, während andere über Cluster verteilt sind, aber in ihrer relativen Signalintensität variieren. Um eine robuste nachgelagerte Datenanalyse zu gewährleisten, ist es wichtig, einen zuverlässigen Lipidnachweis in den meisten Zellen zu erreichen und mögliche Batch-Effekte zu bewerten. Abbildung 3D zeigt repräsentative Spektren von sechs einzelnen kortikalen Zellen, die über separate ITO-beschichtete Objektträger verteilt sind. Während einzelne Lipidprofile Heterogenität aufweisen, werden Lipide über alle Aufnahmen hinweg konsistent nachgewiesen. Abbildung 3E zeigt eine UMAP von kortikalen Zellen aus verschiedenen Objektträgern, die unter identischen Bedingungen präpariert und analysiert wurden. Die erhebliche Überlappung von Zellen zwischen den Objektträgern deutet darauf hin, dass Batch-Effekte die biologischen Interpretationen nicht signifikant verwirren. Bei Bedarf können Batch-Korrekturalgorithmen wie ComBat30 angewendet werden, um Resteffekte zu mildern. Zusammenfassend lässt sich sagen, dass Einzelzell-MALDI-2 eine fortschrittliche Technik zur Identifizierung von Lipid-Biomarkern ist, die wichtige Einblicke in zelluläre Subpopulationen bietet und unser Verständnis der regionalen und funktionellen Vielfalt im Gehirn verbessert.
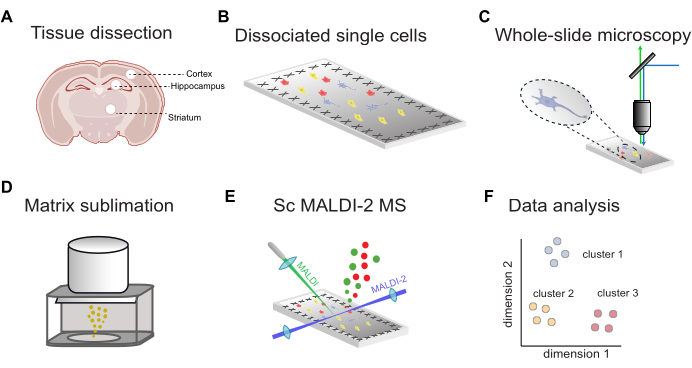
Abbildung 1: Allgemeiner Arbeitsablauf für scMALDI-2 MS. (A) Manuelle Gewebedissektion und Isolierung von Hirnregionen. (B) Papain-Dissoziation von Gewebe in einzelne Zellen und Zellablagerung auf leitfähigen ITO-beschichteten Objektträgern. (C) Fluoreszenzmikroskopie des gesamten Objektträgers mittels Kernfärbung (DAPI/Hoechst). (D) Sublimation der MALDI-Matrix (DHAP) auf Objektträger. (E) Bildgeführte einzellige MALDI-2 MS-Messungen. (F) Umfassende Datenanalyse, einschließlich Dimensionalitätsreduzierung, Clustering und statistischer Analyse. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
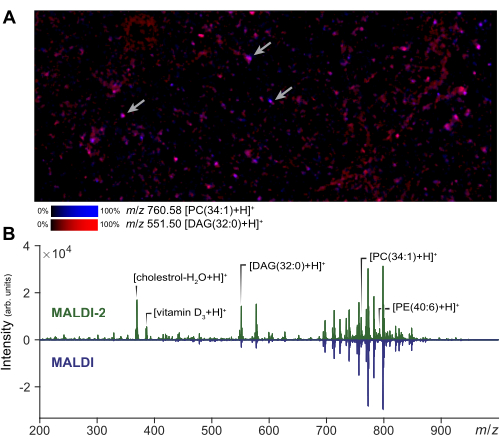
Abbildung 2: MALDI-2 MS-Optimierung und Bildgebung für die Einzelzellanalyse. (A) MALDI-2 MSI-Bildgebung von dispergierten Populationen von Primärzellen. Die Pfeile zeigen einige einzelne Zellen an. Die Durchführung von MSI und einer kleinen Region ermöglicht es dem Analytiker, die Methodenparameter zu optimieren und die Probenqualität zu beurteilen. (B) Vergleich der Ergebnisse, die mit MALDI-2- und MALDI-MS-Erfassungen erzielt wurden. Die Signalverstärkung (MALDI 2 vs. MALDI) verschiedener Lipide, einschließlich Cholesterin, DAG (Diacylglycerine), PC (Phosphatidylcholin) und PE (Phosphatidylethanolamin), wird durch die Darstellung des MALDI-2-Spektrums (oben) und des MALDI-Spektrums (unten) hervorgehoben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
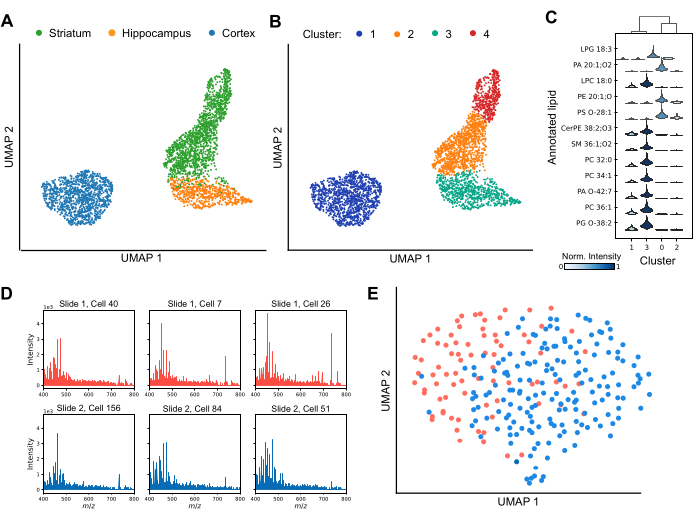
Abbildung 3: Einzelzell-Lipid-Profiling in drei Nagetier-Hirnregionen mittels MALDI-2 MS. (A) Uniform Manifold Approximation and Projection (UMAP) Analyse der analysierten Zellen, eingefärbt nach Hirnregion, aus der das Gehirn stammt. (B) UMAP-Analyse derselben Zellen nach Leiden-Clustering, wobei die Cluster Subpopulationen von Zellen mit ähnlicher Lipidzusammensetzung entsprechen. Die Farben hier zeigen verschiedene Leiden-Datencluster. (C) Ein Violin-Diagramm, das den Unterschied in den Intensitätsverteilungen für verschiedene Lipide zwischen Clustern zeigt. (D) Spektren von sechs repräsentativen einzelnen kortikalen Zellen wurden von zwei separaten Objektträgern analysiert. (E) UMAP-Analyse von kortikalen Zellen, die von zwei separaten Objektträgern analysiert wurden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Akte 1: SC-MALDI2_Analysis.py. Diese Datei enthält eine Reihe von Funktionen, die für die Verarbeitung von Einzelzell-MALDI-2-Massenspektrometriedaten entwickelt wurden. Es umfasst Methoden zum Lesen von Massenspektrometrie-Rohdaten, zum Durchführen von Binning sowie zum Durchführen von Annotationen und nachgelagerten Analysen. Die Zellen zum Laden und Durchführen aller Datenvorverarbeitungen und -analysen sind in der Zusatzdatei 2 enthalten. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 2: JOVE Analysis.ipynb. Diese Datei enthält ein Jupyter-Notebook, das eine Reihe von Zellen enthält, die in den benutzerdefinierten Python-Funktionen aus SC-MALDI2_Analysis.py geladen und alle genannten Datenvorverarbeitungen und -analysen durchgeführt werden sollen. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Die bildgesteuerte Einzelzell-MALDI-MS mit hohem Durchsatz ist ein wertvolles Werkzeug für das Verständnis der chemischen Heterogenität auf Einzelzellebene. Die Zugabe von laserinduzierter Nachionisation (MALDI-2) ermöglicht eine tiefere Analytabdeckung, was für massen- und volumenbegrenzte Proben, wie z. B. isolierte Säugetierzellen, von entscheidender Bedeutung ist.
Während die überwiegende Mehrheit der veröffentlichten einzelligen Lipid- und Metaboliten-MS-Arbeitsabläufe kultivierte Zellen verwendet, wird unser Ansatz auf relativ schnell isolierte Primärzellen angewendet. Diese Unterscheidung ist von entscheidender Bedeutung, da kultivierte Zellen innerhalb weniger Tage nach der Kultivierung eine nachweisbare Veränderung ihres Metaboliten- und Lipidgehalts aufweisen23,31. Die enzymatische Zellisolierung führt in den meisten Fällen zum Verlust feiner zellulärer Strukturen wie Axone, Dendriten und astrozytärer Prozesse. Daher werden die beobachteten Analytsignale hauptsächlich aus dem Zellsoma gewonnen. Trotzdem gelingt eine erfolgreiche Klassifikation nach Zelltyp4 und Hirnregion12. Dies deutet darauf hin, dass sich viele der Lipide, die für verschiedene Zellen einzigartig sind, im Soma befinden.
Kleine Unterschiede in der Applikationsmethode der MALDI-Matrix können tiefgreifende Auswirkungen auf das resultierende Analytsignal haben. Wir haben einen Arbeitsablauf vorgestellt, der auf reproduzierbarer Matrixsublimation mit einem kommerziellen Gerät basiert, das eine homogene Matrixschicht, ein hohes Signal-Rausch-Verhältnis und eine begrenzte Analytausbreitung erzeugt. Es kann aber auch das Matrix-Auftragen unter anderem mit einem Robotersprühgerät oder einer Künstlerairbrush zum Einsatz kommen. Unabhängig vom Ansatz sollte bei Einzelzellpräparaten eine geringere Matrixdichte angewendet werden als bei Standardgewebe-MSI, um das Verhältnis von optischer Matrix zu Analyt zu erreichen. Die hier angewandte Matrixdichte ist etwa halb so hoch wie die für Gewebe-MSI verwendete. Dies bedeutet natürlich, dass die Matrixmenge pro Zelle höher ist als bei MSI, was zu einer verbesserten Analytextraktion und -detektion bei dem hier verwendeten isolierten Zellansatz beiträgt.
Bei Proben, die eine signifikante Optimierung erfordern, kann die MSI bei einem kleinen ROI von Einzelzelldissoziaten, wie in Abbildung 2A gezeigt, als Teil des Optimierungsprozesses durchgeführt werden. Dieser Schritt kann es dem Analytiker auch ermöglichen, das Ausmaß der Analytausbreitung bei lösungsmittelbasierten Matrixapplikationsansätzen zu beurteilen.
Eine erfolgreiche bildgesteuerte MALDI einzelner Zellen erfordert qualitativ hochwertige Fluoreszenzbilder, eine reproduzierbare Matrixapplikation und eine präzise Tischsteuerung. Diese Experimente sind anspruchsvoller als Standard-MSI-Workflows, die aufgrund größerer Bildgebungsbereiche und höherer Analytzahlen geringfügige Registrierungsfehler und Variabilität der Matrix- und Probenbedingungen tolerieren. Trotz dieser Herausforderungen haben wir mit traditionellem MALDI erfolgreich auf einzelne Organellen abzielen können.
Genaues Stitching von gekachelten Fluoreszenzbildern ist unerlässlich, da kleine Fehler die Tischsteuerung verstärken und beeinträchtigen können, was die Registrierungsgenauigkeit entscheidend macht. Batch-Effekte stellen ebenfalls eine große Herausforderung dar, da Signalvariationen über denselben Objektträger, zwischen Objektträgern und über Präparate hinweg beobachtet werden. Geeignete Normalisierungsstrategien, einschließlich der Verwendung interner Standards und der Normalisierung des Interquartils, tragen dazu bei, diese Probleme zu mildern. Darüber hinaus können Chargenkorrekturalgorithmen wie ComBat30 verwendet werden, um die technische Variabilität zu reduzieren und echte biologische Unterschiede zu verbessern.
Es bleibt eine Herausforderung, sichere molekulare Annotationen von einzelnen Zellen zu erhalten. Oft sind die Menge des Materials und die Empfindlichkeit des Nachweises für Tandem-MS bei allen außer den am häufigsten vorkommenden Spezies unzureichend. Für diejenigen, die nicht ausreichend für Tandem-MS direkt aus einzelnen Zellen nachweisbar sind, können alternative Ansätze zur Erstellung einer Analytbibliothek angewendet werden. Zum Beispiel können Tandem-MS-Daten am Gewebe aus dünnen Kryoschnitten aus derselben Gehirnregion und von Tieren gewonnen werden, die zur Erzeugung von Zellpopulationen verwendet wurden. Eine Lipidextraktion aus Gewebe mit anschließender LC-MS kann ebenfalls eingesetzt werden. Mit der Weiterentwicklung der Probenvorbereitungsstrategien und der MS-Technologie können immer mehr relevante Strukturinformationen direkt aus einzelnen Zellen gewonnen werden. Wir gehen davon aus, dass dieser Workflow in Zukunft erweitert wird, um Tandem-MS-Daten von einzelnen Zellen zu erhalten, wodurch zusätzliche LC-MS/MS-Experimente überflüssig werden. Wir stellen uns außerdem vor, dass dieser Ansatz auf zahlreiche Proben auf der Mikroskala in der Biologie und darüber hinaus ausgeweitet werden könnte, einschließlich einzigartiger Zelltypen, Säugetierorganellen, Pulver und Mikroplastik.
Offenlegungen
Die Autoren haben keine konkurrierenden Interessen offenzulegen.
Danksagungen
S.W.C dankt der Unterstützung durch das Peixin He and Xiaoming Chen PhD4 Fellowship und das University of Illinois Block Grant Fellowship. Diese Arbeit wurde auch vom National Institute on Drug Abuse unter der Preis-Nr. P30DA018310, das National Institute on Aging unter der Auszeichnung Nr. R01AG078797 und vom Büro des Direktors der National Institutes of Health unter der Auszeichnungsnummer S10OD032242.
Materialien
| Name | Company | Catalog Number | Comments |
| 2',5'-dihydroxyacetophenone | Sigma Aldrich | D107603 | DHAP, 97% purity |
| Ammonium acetate | Sigma Aldrich | 238074 | ACS reagent, ≥97% |
| Axio M2 Imager | Zeiss | N/A | N/A |
| Biopsy punch, 2 mm | Fisher Scientific | 12-460-399 | integra miltex standard biopsy punch, 2mm |
| Calcium chloride | Sigma Aldrich | C4901 | anhydrous, powder ≥97% |
| Eppendorf Centrifuge | Sigma Aldrich | EP5405000441 | centrifuge 5425 with rotor FA-24x2 |
| Gentamicin | Sigma Aldrich | G1272 | liquid, BioReagent |
| Glass etching pen | Sigma Aldrich | Z225568 | carbide time, pkg of 1 |
| Glycerol | Sigma Aldrich | G7893 | ACS reagent, ≥99.5% |
| HEPES buffer | Sigma Aldrich | H3375 | ≥99.5% (titration) |
| Hoechst 33258 Solution | Sigma Aldrich | 94403 | 1 mg/mL in H2O, ≥98.0% (HPLC) |
| In line HEPA Filter | Sigma Aldrich | WHA67225001 | VACU-GUARD 60 mm disc, 0.45 PFTE housing |
| ITO-Coated Microscopy Slides | Delta Technologies | CG-90IN-S115 | 70-100Ω resistance |
| Magnesium chloride | Sigma Aldrich | M8266 | anhydrous, ≥98% |
| Magnesium sulfate | Sigma Aldrich | 208094 | anhydrous, ≥97% |
| Microcentrifuge tubes | Sigma Aldrich | HS4323K | tube capacity 1.5 mL, pack of 500 |
| Papain dissociation system | Worthington Biochemical | LK003150 | one box, 5 single use vials |
| Penicillin-Streptomycin | Sigma Aldrich | P4458 | liquid, BioReagent |
| Potassium chloride | Sigma Aldrich | 529552 | Molecular biology grade |
| Potassium phosphate monobasic | Sigma Aldrich | P5379 | Reagent Plus |
| Sodium biocarbonate | Sigma Aldrich | S6014 | ACS reagent, ≥99.7% |
| Sodium chloride | Sigma Aldrich | S9888 | ACS reagent, ≥99% |
| Sodium hydroxide | Sigma Aldrich | 221465 | ACS reagent, ≥97%, pellets |
| Sodium phosphate dibasic | Sigma Aldrich | S9763 | ACS reagent, ≥99% |
| Sublimate | HTX | N/A | N/A |
| timsTOF FleX MALDI-2 | Bruker | N/A | microGRID enabled |
| Vacuum tubing | Thermo Scientific | 8701-0080 | Nalgene Non-phthalate PVC Tubing |
Referenzen
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten