Method Article
Identificando interacção proteína-proteína em
Neste Artigo
Resumo
Drosophila é famoso por sua poderosa manipulação genética, mas não para a sua adequação de análises bioquímicas em profundidade. Aqui apresentamos um procedimento baseado em TAP para identificar interagindo parceiros de qualquer proteína de interesse a partir do cérebro da mosca. Este procedimento pode potencialmente levar a novos caminhos de pesquisa.
Resumo
Telas genéticas realizadas utilizando Drosophila melanogaster (mosca da fruta) fizeram numerosas descobertas marco no avanço das ciências biológicas. No entanto, o uso de telas bioquímicas destinadas a estender o conhecimento adquirido a partir da análise genética foi explorada apenas recentemente. Aqui nós descrevemos um método para purificar o complexo de proteínas que se associa com qualquer proteína de interesse a partir de cabeças de moscas adultas. Este método tira vantagem do sistema de Drosophila GAL4/UAS para expressar uma proteína isco fundido com um tag de afinidade em tandem de Purificação (TAP) em neurónios mosca in vivo, e, em seguida, executa duas rondas de purificação utilizando um procedimento de TAP semelhante à originalmente estabelecido em levedura 1 para purificar o complexo de proteína que interage. No final deste procedimento, uma mistura de vários complexos de proteína é obtido cujas identidades molecular pode ser determinada por espectrometria de massa. Validação das proteínas candidatos serão beneficiados com a resource e facilidade de realizar estudos de perda de função em moscas. Abordagens semelhantes podem ser aplicados a outros tecidos de moscas. Acreditamos que a combinação de manipulações genéticas e esta abordagem proteômica no sistema modelo mosca tem um tremendo potencial para a resolução dos problemas fundamentais no campo da neurobiologia e além.
Introdução
Definindo as vias moleculares ou redes que mediam um processo biológico em particular é um dos objetivos finais da pesquisa biomédica. Fly geneticistas têm dependido fortemente de genética para a frente, especialmente modificador telas genéticos (tanto Enhancer e telas supressores), para identificar os fatores que trabalham em conjunto, em paralelo com, ou a montante ou a jusante de um gene de interesse. No entanto, as telas de genética para a frente muitas vezes não conseguem identificar genes essenciais que, quando mutado, causar letalidade em estágios iniciais de desenvolvimento, ou genes com redundância funcional e compensação cuja perda de função só causar defeitos subtis que são difíceis de marcar. Uma maneira de superar essa dificuldade é para triagem de interações proteína-proteína diretos. Por mais de uma década, uma lista crescente de métodos bioquímicos, incluindo levedura de dois híbridos, display de fagos, cross-linking química, Co-IP, Tandem Affinity Purification (TAP), etc. têm sido utilizados para investigar proteína-proteína interações. Cada uma dessas abordagens tem seu próprio conjunto de pontos fortes e fracos em relação à sensibilidade e especificidade. Entre eles, o método permite que a TAP para detecção da interacção física sob condições quase fisiológicas, preserva a especificidade e consistência 2 e inclui a capacidade de estender a análise de alto rendimento de 3,4.
O método TAP foi originalmente desenvolvido na levedura por colegas Rigautand 1. Neste método, uma proteína de interesse é expressa com uma etiqueta TAP. A etiqueta TAP abriga dois domínios de ligação de afinidade independentes: uma proteína de um domínio que se liga a IgG e um domínio de ligação à calmodulina. Os dois domínios estão separados por um local de clivagem de TEV (Tobacco Etch Vírus). Essa combinação permite a duas rodadas independentes de purificações de afinidade para reduzir suficientemente ligações inespecíficas e enriquecer vinculações específicas 1. Para este exemplo, o método TAP é um poderoso metodod para identificar interacções in vivo de uma determinada proteína, embora o sobre-expressam a proteína exógena pode torná-lo mais propenso a associar-se com proteínas que normalmente não fazem complexo com o seu homólogo endógeno. Uma vez que o seu desenvolvimento, o método da TAP foi aplicado em muitos outros sistemas, incluindo--base de cultura de células e outros sistemas de 5,6 in vivo, os sistemas modelo de 6-9. Aqui nós descrevemos a adaptação do método TAP em Drosophila. O primeiro gerar pUAST-NTAP e pUAST-CTAP vectores para facilitar a clonagem e a fusão da etiqueta TAP ou o N-ou C-terminal do gene de interesse. A-marcado com TAP UAS transgene é, então, expresso no sistema nervoso sob o controlo de um controlador 10 de GAL4 neuronal. Em seguida, um grande número de cabeças de moscas adultas serão recolhidos, que tem elevado teor de células neuronais e são fáceis de separar a partir de outras partes do corpo, após a congelação com base em diferenças de tamanho. As cabeças adultas são homogeneizados e limpou bcentrifugações sequenciais y, e o sobrenadante é sujeito a um procedimento de TAP descrito abaixo.
Protocolo
1. Gerar UAS-marcado pela TAP Moscas transgênicas
- Gerar pUAST marcadas pela TAP construções de DNA.
- Decidir qual o lado (N-ou C-terminal) da proteína isco a etiqueta TAP deverá ser fundido a, com base na estrutura / função da proteína. Veja a discussão para mais detalhes.
- Subclonar a região de cDNA codificadora do gene de interesse para os múltiplos locais de clonagem (MCS) dos vectores de pUAST-NTAP ou pUAST-CTAP para gerar N-ou C-terminal marcadas transgenes UAS-TAP, respectivamente. Veja na Figura 1 mapas detalhados e sítios de restrição utilizáveis e quadros de leitura.
- Gerar UAS-marcado-TAP voa transgênicos.
- Gerar moscas transgênicas seguindo protocolos padrão, utilizando P-mediada pelo elemento de inserção 11. Um certo número de serviços de injecção estão disponíveis comercialmente.
- Atravessar neuronal condutor de GAL4 (ou seja BG380-GAL4) para cada linha transgénica individual e determinar os níveis de expressão de proteínasde cada linha por Western blot (com anticorpo anti-peroxidase Peroxidase) e / ou a imunomarcação (com anticorpo anti-TAP). Em geral, recomenda-se uma linha transgénica com um nível de expressão de proteína que é próximo da proteína endógena para procedimentos TAP. Veja a discussão para mais detalhes.
- PerformGAL4/UAS-based experiências de socorro para confirmar a funcionalidade dos transgenes marcadas com TAP, se a perda de função dos genes mutantes de interesse estão disponíveis. Escolha um transgene que pode salvar substancialmente os fenótipos mutantes para os seguintes experimentos TAP.
2. Preparar amostras para a TAP Procedimento
- Criar um stock de mosca que transporta um controlador neuronal GAL4 (por exemplo BG380-Gal4) e o transgene marcado com TAP escolhido, a fim de facilitar a ampliação de amostras de moscas. Recolha as progênies F1 do piloto GAL4 ea cruz UAS-transgene em casos raros, quando a combinação acima faz com que a sobrevivência eo crescimento desvantagem.
- Coletar amostras de pequena escala e otimizar a condição de lise para solubilizar a proteína marcadas-TAP.
- Fazer uma série de tampões de lise usando uma combinação de detergentes não iónicos de NP-40 (0,1-1%), NaDOC (0,1-1%) e Triton X-100 (0,05-0,5%). Ver Tabela 1 e discussão para mais informações.
- Em cima de um bloco de CO2, use # 5 fórceps para dissecar 20 cabeças adultas do transgene TAP expressar moscas e recolher em um tubo de 1,5 ml para testar cada condição de tampão de lise.
- Adicionar tampão teste de lise 100 ul para o tubo e homogeneizar as cabeças acariciando cima e para baixo com um pilão de plástico, em seguida, adicione um outro tampão teste 100 ul.
- Girar o lisado cabeça em 21.500 xg durante 10 minutos (4 ° C), e separar do sobrenadante e sedimento, após a centrifugação. Adicionar 25 ul de tampão de carregamento 2 x SDS ao precipitado e tampão de carregamento de 10 ul 4x SDS a 30 ul de sobrenadante, respectivamente.
- Ferver as duas amostras durante 5 minutos e analisam-los lado-Lado utilizando SDS-PAGE e subsequente Western Blot com anticorpo PAP. Determinar a solubilidade pela relação dos níveis de proteína-TAP em sobrenadante contra o sedimento.
- Prepare amostra em grande escala
- Expandir e recolher moscas adultas.
- Expandir as ações neuronal-GAL4/UAS-TAP-transgene em garrafas e virar as garrafas a cada 3 dias até acumulativas 250 garrafas são usados para a coleção.
- Colete de 1-3 dias de idade adulta voa para 50 ml tubos cônicos, coloque o tubo em nitrogênio líquido imediatamente para congelador as moscas. Armazenar as moscas em um congelador a -80 ° C. Note-se que o volume das moscas não deve ser superior a 2/3 do tubo de 50 ml.
- Colete cabeças mosca (executar este passo em cima de gelo seco em pó).
- Retire as peneiras prechilled eo almofariz e pilão do freezer -80 ° C e colocá-los em gelo seco, de preferência dentro de um grande balde de gelo. Empilhe duas peneiras de teste padrão dos EUA com um No. 25 no the superior e n ° 40, na parte inferior.
- Pegue as moscas congelados para fora e deixá-los em nitrogênio líquido e manter as moscas lá por cerca de 10 min. Vortex ou agitar os tubos vigorosamente para quebrar as cabeças, pernas e asas dos corpos.
- Despeje a mistura para o peneiro superior e, em seguida, agitar as peneiras vigorosamente, mantendo ambas as peneiras juntos. Depois de peneirado, os corpos vão ficar no topo peneira, voar cabeças serão retidos na peneira de fundo, e as pernas, asas e outros detritos vão cair para o gelo seco. Separe as duas peneiras e transferir cuidadosamente as cabeças de voar para a argamassa frio.
- Homogeneizar as cabeças de mosca
- Em cima do gelo seco, moer a cabeça com a argamassa eo pilão para partículas de pó, em seguida, transferir o pó a 15 ml de vidro Dounce Grinder tecido que foi previamente resfriada em gelo.
- Meça os pesos do moedor de antes e depois da amostra cabeça foi derramado nele, e em seguida, calcular o quanto o samp cabeçale pesa. Um total de 6-15 gramas de cabeças mosca será suficiente para cada experimento TAP ajuste de acordo com os níveis de expressão da proteína. Adicionar 15 ml de tampão de homogeneização gelada (lise otimizado no passo 2.2) para o pó e, em seguida, derrame com o grande pilão folga até que seja fácil para os pilão para ir para cima e para baixo. Manter o moinho de vidro em gelo em todos os momentos.
- Prepare o sobrenadante para a TAP
- Transferir o homogeneizado para um tubo de centrifugação de alta velocidade de rotação e durante 20 min a ~ 50.000 xg (4 ° C). Transferir o sobrenadante para um novo tubo de centrifugação de alta velocidade e repetir a centrifugação mais uma vez.
- Transferir o sobrenadante para um tubo de ultracentrifugação e executar um 40 min ~ 250.000 xg rotação para limpar ainda mais o sobrenadante. O sobrenadante está pronto para os procedimentos de purificação por afinidade em tandem após ultracentrifugação.
- Expandir e recolher moscas adultas.
3. TAP Purificação
As secções que se seguem foram obtidos a partir do laboratório de TAP Séraphin protocol 12 (http://web.as.uky.edu/Biology/faculty/rymond/BIO%20510/Bertran%20Seraphin%27s%20TAP%20page.pdf )
- Realize IgG talão purificação por afinidade
- Prepare IgG Sepharose talão enquanto as amostras são centrifugadas. Lavar 3x 400 ul de contas de IgG num tubo Falcon de 15 ml com 10 ml de tampão de lavagem frio IgG. Para cada lavagem, balançar suavemente o tubo para 2 min, e depois girar as contas a 1.000 xg por mais 2 min. No final da terceira lavagem, remover o tampão e deixar apenas as esferas no tubo.
- Transferir cuidadosamente o sobrenadante clarificado (~ 15 ml) para o tubo de 15 ml contendo as contas de IgG. Incubar os grânulos e mistura lisado cérebro, a 4 º C em um nutator durante 2 horas.
- Configurar uma coluna micro limpo e vazio com cerca de 15 ml de volume total na câmara fria. Coloque a mistura do grânulo IgG por constantemente despejando a mistura para a coluna, não tentar prender qualqueras bolhas de ar no interior da coluna. Permitir que as contas para se instalar na coluna e no tampão para drenar lentamente por fluxo de gravidade.
- Lavar cuidadosamente a coluna com 10 ml de tampão de lavagem frio IgG depois de todo o lisado cérebro fluiu através da coluna de IgG resolvido. Repita o 2x lavagem. Nota: nunca deixar secar o talão no ar.
- Realize TEV clivagem
- Após a terceira lavagem, lava-se a coluna outra vez com 10 ml de tampão de clivagem de TEV. Esta etapa prepara o talão IgG que seqüestra a complexa isca para TEV clivagem.
- Imediatamente antes da última gota de tampão de TEV é sobre a escorrer para fora, colocar uma tampa na parte inferior da coluna para bloquear o fluxo, adicionar 1,3 ml de tampão de TEV contendo 130 unidades da enzima de TEV para a coluna, e em seguida tampão de forma segura a parte superior do a coluna. Certifique-se de que a coluna é bem fechadas em ambas as extremidades.
- Rodar a coluna a 18 º C durante 2 horas para permitir que a enzima de TEV para clivar o péptido no local da TEV e libertar a proteína whi complexole deixando para trás a proteína de um péptido de domínio ligado a esferas de sefarose da IgG.
- Realize Calmodulina talão purificação por afinidade
- Prepare os grânulos de calmodulina, enquanto os grânulos de IgG foram incubadas com a enzima de TEV. Lavar 200 ul grânulos calmodulina em um Falcon de 15 ml de tubos de 3x, de cada vez com 10 ml de tampão de ligação frio calmodulina. Para cada lavagem, agitar suavemente o tubo durante 2 min num nutator, e depois girar as esferas a 1000 xg durante 2 min. No final da terceira lavagem, retirar todo o tampão e deixar apenas as esferas no tubo.
- No final da incubação TEV (passo 3.2.3), o retorno da coluna IgG de volta para a sala fria e coloque-o em linha reta. Vamos as contas se contentar com 10 min.
- Retire a tampa superior e, em seguida, a tampa inferior e, em seguida, recolher o produto de clivagem 1,3 ml TEV em um tubo Falcon de 15 ml. Deixe o tampão drenar completamente. Adicionar um buffer de 200 mL TEV adicional para a coluna para empurrar para fora o volume morto da coluna, recolher o fbaixa no mesmo tubo.
- Adicionar 4,5 ml de tampão de calmodulina e 4,5 mL de 1 M de CaCl 2 de ligação para o produto de clivagem de 1,5 ml de TEV recolhidos acima. O CaCl 2 serve para dosear o EDTA no buffer de TEV. Transferir a mistura de 6 ml para o tubo contendo as esferas de calmodulina e rodar o tubo a 4 ° C num nutator durante 1 hora.
- Configure outra coluna micro limpo e vazio com cerca de 10 ml de volume total na câmara fria. Coloque a mistura de pérolas para calmodulina para a coluna e permitir que a drenar pela força da gravidade.
- Quando toda a solução fluiu através da coluna de calmodulina resolvido, lava-se a coluna duas vezes, cada uma com 10 ml de tampão de ligação frio calmodulina. Nota: evitar perturbar as contas calmodulina e tentar manter a superfície dos grânulos de tão plana quanto possível, durante a lavagem.
- Eluir o complexo de isca de coluna calmodulina.
Logo após a lavagem, eluição da coluna de calmodulina com cinco frações de 200 mL frio Calmodutampão de eluição lin. Para cada fracção, adiciona cuidadosamente 200 ul de tampão de eluição à coluna e recolher o eluato com um marcado tubo Eppendorf de 1,5 ml. Repita este 4x. - Analisar o complexo de proteína por SDS-PAGE
- Tomar uma pequena alíquota de cada um dos cinco fracções (aproximadamente 30 ul) e adicionar tampão de carga de SDS. Ferver as amostras durante 5 minutos e carregar as amostras de lado-a-lado com os marcadores moleculares de proteínas em um gradiente (4-15%) em gel de SDS-PAGE.
- Após as amostras terem totalmente resolvidas no gel, parar a eletroforese e manchar o gel com todos os procedimentos de coloração à base de L-250 sensíveis Coomassie coloidal como "prata azul 'manchando 13. Coloração prata é opcional, mas não preferível, porque não é totalmente compatível com a análise de espectrometria de massa subseqüente. Armazenar o resto do eluato num congelador a -80 ° C para posterior análise, tal como a espectrometria de massa para descobrir as identidades moleculares do complexo de proteína purificada. See discussão.
Resultados
Aqui demonstramos o nosso esforço na identificação de proteínas Highwire-que interagem no cérebro da mosca. Highwire (Hiw) e seus homólogos de vertebrados e invertebrados são enormes ligases de ubiquitina que regulam o desenvolvimento e reparação do sistema nervoso 14. Eles compartilham um número de domínios funcionais altamente conservadas. No entanto, suas ações moleculares não são totalmente claras. Trabalho feito em verme, mosca e do rato levou à do modelo de trabalho actual que funciona como uma ligase HIW E3 e como uma proteína de andaimes para facilitar a formação de um complexo multi-subunidade de ubiquitinação, que regula as funções neuronais específicas do tipo de célula e de tempo-a através combinação de interagir com diferentes co-fatores e visando diferentes substratos ubiquitina. Para identificar o complexo ubiquitination Hiw associada, primeiro gerou um marcado transgene UAS-TAP-Hiw N-terminal que é totalmente funcional em resgatar o fenótipo mutante hiw 15. Cerca de 10 g de fl adultocabeças y que expressam TAP só ou proteínas transgénicas TAP-HIW foram colhidos e sujeitos a procedimentos de TAP, lado a lado, tal como descrito acima. Eluatos final de ambas as purificações foram analisadas por SDS-PAGE e coloração com prata, em seguida. A espectrometria de massa identificou uma lista de proteínas apenas na amostra TAP-Hiw, incluindo Drosophila FSN (DFSN, uma proteína F-box) e rae1 (Figura 2). Análises genéticas e bioquímicas subsequentes revelaram que Hiw e DFSN trabalhar juntos como SCF-like complexo ubiquitina ligase E3 para regular a estrutura e função sináptica 16, e associados rae1 com Hiw in vivo e restringe o crescimento excessivo sináptica 17. Esta função de rae1 é pelo menos parcialmente alcançados através da sua capacidade para promover a estabilidade da proteína Hiw através Hiw proteger da degradação autophagic, que revela um novo mecanismo que controla selectivamente Hiw abundância de proteínas durante o desenvolvimento sináptica 17.
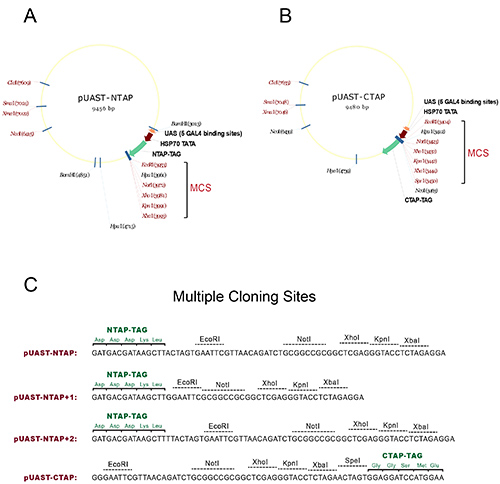
Figura 1. Vetores de geração de moscas transgênicas TAP-tag. (A) O mapa da pUAST-NTAP construir. (B) O mapa da pUAST-CTAP construir. Os locais de clonagem múltiplos (MCS) em ambas as construções são marcadas com um suporte, em que os locais de restrição de corte único são apresentados com a cor vermelha. (C) Sequência Primária mostrando os múltiplos locais de clonagem dos vectores da torneira com o quadro de leitura da etiqueta TAP. Para facilitar a subclonagem, mais duas construções NTAP são gerados a partir do original pUAST-NTAP: pUAST-NTAP +1 e pUAST-NTAP +2. Juntos, os três NTAP constrói abrange todas as três possíveis fases de leitura para acomodar a utilização de MCS. Todos os vectores são construídos usando a NTAP ou fragmentos CTAP originalmente gerada emSeraphin Lab 1. Clique aqui para ver imagem ampliada.

. Figura 2 Purificação de Highwire-interagindo proteínas do cérebro da mosca por purificação TAP cabeças adultas de moscas que expressam TAP. (BG380-GAL4; UAS-TAP) ou TAP-Hiw (BG380-GAL4; UAS-TAP-Hiw) no sistema nervoso são colhidos, homogeneizados, e submetida a purificação TAP, respectivamente. Os eluatos finais foram analisados por unidimensional em gel de SDS-PAGE seguida por coloração com prata. A cabeça de seta indica a Hiw proteína isca eo asterisco indica uma proteína truncada TAP devido à TEV clivagem. Na amostra TAP-Hiw, uma lista de proteínas são identificadas por massaespectrometria, incluindo HSC-70, β-tubulina, rae1 e DFSN (indicado por setas). Este valor é modificado a partir da Figura 1-A de Tian et al. 17 Clique aqui para ver imagem ampliada.
| Amortecedor | Composição | Comentários |
| O tampão de lise | 50 mM Tris-HCl pH 7,5 | NOTA: 1) Adicionar a TDT e os inibidores da protease e proteassomas imediatamente antes da utilização. |
| NaCl 125 mM | 2) mostrando aqui é um tampão de lise com 0,5% NP40. Ver discussão para modificações em detergentes não-iônicos. | |
| 5% de Glicerol | ||
| 0,5% de NP40 | ||
| 1,5 mM de MgCl2 | ||
| NaF 25 mM | ||
| DTT 0,2 mM | ||
| (Os seguintes são inibidores da protease e do proteassoma) | ||
| 1 mM Na 3 VO 4 | ||
| 0,05 mM MG-115 | ||
| 1 mM de PMSF | ||
| Mistura de inibidores da protease (Sigma P8340) | ||
| Inibidor da protease cocktail (Roche 04693159001) | ||
| Tampão de lavagem IgG | 10 mM Tris-Cl pH 8,0 (0,5 mL de 2 M de estoque) | |
| NaCl 150 mM (3 ml de 5 M de ações) | ||
| 0,1% NP40 (1,0 ml de 10% de ações) | ||
| H 2 0 a 100 mL finais | ||
| Tampão clivagem TEV | 10 mM Tris-Cl pH 8,0 (0,5 mL de 2 M de estoque) | Nota: Adicione TDT imediatamente antes da utilização. |
| NaCl 150 mM (3 ml de 5 M de ações) | ||
| 0,1% NP40 (1,0 ml de 10% de ações) | ||
| EDTA 0,5 mM (100 ul de 0,5 M de estoque) | ||
| DTT 1 mM (100 ul de 1 M) | ||
| H2O para 100 ml finais | ||
| Tampão de ligação a calmodulina | 10 mM β-mercaptoetanol (69,7 l de estoque) | |
| 10 mM Tris-Cl pH 8,0 (0,5 mL de 2 M de estoque) | ||
| NaCl 150 mM (3 ml de 5 M de ações) | ||
| 1 mM de acetato de magnésio (100 mL de 1 M) | ||
| 1 imidazol mM (100 ml de 1 M) | ||
| 2 mM de CaCl 2 (200 mL de 1 M) | ||
| NP40 a 0,1% (1 ml de 10% de estoque) | ||
| H2O para 100 ml finais | ||
| Tampão calmodulina eluição | 10 mM β-mercaptoetanol (69,7 l de estoque) | |
| 10 mM Tris-Cl pH 8,0 (0,5 mL de 2 M de estoque) | ||
| NaCl 150 mM (3 ml de 5 M de ações) | ||
| 1 mM de acetato de magnésio (100 mL de 1 M) | ||
| 1 imidazol mM (100 ml de 1 M) | ||
| EGTA 10 mM (2 mL de 0,5 M de estoque) | ||
| NP40 a 0,1% (1 ml de 10% de estoque) | ||
| H2O para 100 ml finais |
Tabela 1. Composição de buffers.
Discussão
Método de purificação por afinidade em tandem (TAP) oferece um protocolo de purificação dupla que permite o isolamento e enriquecimento de complexos de proteínas por meio de duas etapas independentes de purificação de afinidade. O design da marca TAP não se restringe ao que é apresentado neste protocolo, outros domínios de ligação de proteínas e motivos também são aplicáveis se as condições do tampão são ajustados em conformidade. Um bom exemplo de outras tags TAP é a marca GS-TAP, uma combinação de uma proteína G e um motivo de ligação de estreptavidina, concebido pelo grupo de Giulio Superti-Furga visa complexo de proteínas purificadora em linhagens de células de mamíferos em cultura 18. O GS-TAP foi depois adaptado para estudar a interacção proteína-proteína em células de Drosophila S2 e de embriões 19. Uma publicação recente JoVE por Bailey et al. Demonstrou como o procedimento de GS-TAP foi realizada com o lisado de cultura de células 20. Aqui apresentamos o uso de TAP-tag em tecidos neurais Drosophila (adcabeças ult) para triagem proteômica em larga escala. Este protocolo pode ser potencialmente adaptado para outros tecidos que permitem a coleta de amostras de grande escala, tais como embriões. Os organismos adultos recolhidos no peneiro de cima (passo 2.3.2.3) também pode ser utilizado para TAP, que pode ser uma tarefa muito difícil. O tampão de lise deve ser modificado e condicionado para inibir as proteases maciças presentes no tracto digestivo, no máximo, e o primeiro processo de purificação ser significativamente encurtado, a fim de reduzir o tempo de exposição das proteínas para as proteases. Apesar de diferentes marcas TAP exigem diferentes manejos e condições de buffer para purificações de afinidade, os princípios gerais da TAP são os mesmos. Abaixo discutimos assuntos que afetam a qualidade dos resultados da TAP.
O sucesso da abordagem TAP depende da geração do transgene direita. Primeiro, a própria tag TAP não deve alterar a conformação global da proteína isca, o que é fundamental para manter a sua capacidadepara interagir com os seus parceiros de ligação fisiológicas. Assim, é crucial para decidir para onde fundem a etiqueta TAP para o gene de interesse. A decisão pode ser feita com base em estudos anteriores da proteína, especialmente informações sobre a sua estrutura ou transgenes marcado com epitode funcionais. No caso de essa informação não estiver disponível, a marcação da proteína isco no N-ou C-terminal em paralelo é recomendada. Em seguida, as experiências de socorro funcionais podem ser realizadas para determinar qual do transgene deve ser usado. Em segundo lugar, os níveis de proteína transgénica de expressão deve ser próxima da da proteína endógena. Isto é particularmente importante porque o sistema Gal4/UAS normalmente dirige a expressão do transgene em diferentes horários e em um nível muito mais elevado em comparação com as proteínas endógenas, o que pode aumentar ou falsos positivos ou provocar consequências inesperadas que alteram o perfil da interacção de proteínas redes das células. Por exemplo, as proteínas andaimes superexpressão pode cause os efeitos negativos dominantes e que sobre-expressam proteínas que possuam as actividades enzimáticas, tais como cinases, muitas vezes causar ganho-de-função, os efeitos tanto da qual pode por sua vez, afectam a capacidade dos seus parceiros de ligação endógenos para interagir com elas. Assim, o sistema GAL4/UAS devem ser evitados quando o tempo eo nível de expressão do gene de interesse é crítica para a preservação das interacções endógenas que apenas apresentam sob condições normais. Em vez disso, a expressão do transgene marcado com TAP deve ser controlado de acordo com o seu próprio promotor. Isto pode ser conseguido quer através da substituição da sequência UAS com a sequência do promotor endógeno, ou por fusão da sequência de TAP em moldura com a sequência de exonal em um fragmento de ADN genómico que contém todo o locus do gene de interesse 21. Em alguns casos, o experimento TAP pode ser realizada em uma perda de fundo função mutante do gene de interesse para reduzir a competição da proteína endógena para incorporating em complexos multiproteicos. Para este exemplo, um fundo nulo proteína, se disponível, fornece a condição ideal para eliminar completamente a proteína endógena. Usando o fundo mutante genético sempre irá aumentar a chance de produzir resultados com maior qualidade.
Solubilidade da proteína isca é outro fator crucial para o sucesso. Detergentes não iónicos são normalmente usados no tampão de lise para dissolver a membrana plasmática e manter o complexo de proteína, solúvel e intacta, num estado que está perto o ambiente fisiológico. No entanto, dependendo se ou não as proteínas de interesse são proteínas ou associados integrada à membrana, a composição e concentração de detergentes não-iônicos podem ser cruciais para solubilizar a proteína isca. 0,1-0,3% de NP40 é muitas vezes suficiente para solubilizar proteínas citosólicas. Para as proteínas da membrana, de uma combinação de NP40 (0,1-1%), NaDOC (0,1-1%), e Triton X-100 (0,05-0,5%), é por vezes necessária a solubiLize e enriquecer bastante proteínas para o experimento TAP. No entanto, uma vez que um tampão de lise rigorosas tendem a perturbar interacções proteína-proteína mais fracas, o equilíbrio tem de ser alcançado em que as proteínas solúveis são isca suficiente presente na amostra, enquanto ao mesmo tempo a maioria das interacções proteína-proteína são preservados. Um princípio geral é que sempre tentar usar o menor número de tipos de detergentes com a menor concentração.
Para facilitar a resolução de problemas, é altamente recomendável para salvar uma pequena alíquota da amostra de cada passo das duas purificações. Posteriormente por SDS-PAGE e coloração de proteína / análise de Western sobre as alíquotas de permitir que os níveis da proteína de isco e o enriquecimento sequencial de certas espécies de proteína a ser monitorizado. Por exemplo, se foi detectada uma redução súbita da proteína isco em aliquota do fluxo de saída, após clivagem TEV (passo 3.3.3), é provável que a proteína isco foram perdidos durante a IgG talão de lavagem (passo 3.1.4) . A possvel causa pode ser a composição detergente inadequada do tampão de lavagem de IgG. Há uma queda acentuada na composição e concentração dos detergentes no tampão de lavagem de IgG (0,1% de NP-40 só) em comparação com o tampão de lise. A proteína isco pode ser sensível a essas mudanças e desassociar a partir da porção de IgG nas esferas. Ajustar a lavagem IgG para tampão de lise pode ajudar a resolver o problema. Alternativamente, embora menos provável, a clivagem de TEV (passo 3.2) pode não ter funcionado suficientemente para libertar o complexo contendo o isco a partir das pérolas de IgG. Os passos a pena uma segunda inspecção são o equilíbrio de contas de IgG com o buffer de TEV (passo 3.2.1) e da atividade enzimática, manuseio incorreto pode amortecer a enzima.
Tenha em mente que, mesmo com a purificação de duas etapas, haverá ainda falsos positivos e proteínas que são puxados para baixo inespecífica presente no complexo purificado-TAP final. Uma maneira de identificar essas interacções não específicas, empelo menos parcialmente, é a de incluir um controlo usando o TAP TAP-transgene apenas em paralelo com marcado com TAP-transgene nas experiências de purificação de TAP. As proteínas identificadas no procedimento só-TAP deve ser removido da lista identificado na purificação de TAP-transgene.
Um TAP sucedido irá purificar uma mistura de proteínas que são potencialmente associados com a proteína de interesse in vivo. O próximo passo é a identificação molecular destas proteínas, e espectrometria de massa é utilizada para esta finalidade. Dependendo dos resultados da SDS-PAGE e as necessidades do experimento, as identidades molecular do complexo proteína purificada pode ser determinada de duas maneiras: 1) cada banda proteína individual pode ser cortado do gel e submetido a uma baixa complexidade LC-MS/MS, ou 2) a fracção de eluído que contém o teor de proteínas de pico (normalmente a segunda eluição) pode ser directamente submetido a complexidade moderada LC-MS/MS. Tal shot-gun proteomic abordagem potencialmente identificar todas as proteínas do complexo, em função da gama dinâmica do equipamento MS 22.
Em resumo, apresentamos um método para identificar as interações proteína-proteína em tecidos neurais de um organismo modelo genético - a mosca da fruta. Há um número de vantagens de se aplicar o método de TAP para a mosca: 1) as moscas da fruta tem um curto ciclo de vida, de modo que é relativamente rápido e fácil de gerar moscas transgénicas e para obter tecidos em grande quantidade; ambos são cruciais para o TAP abordagem, 2) o genoma da mosca é totalmente anotada e contém 70% dos genes causadores de doenças humanas, e 3) mais importante, que é fácil para validar funcionalmente e caracterizar o candidato interagir proteínas em moscas. Existem recursos abrangentes disponíveis na comunidade de pesquisa da mosca para a caracterização de um determinado gene, tais como a recolha RNAi baseada transgênico útil para estudar tecido específico de perda de função de um maior parte do genes, os alelos mutantes e de deficiência da maioria dos loci de genes, assim como os clones e os transgenes úteis para estudos de ganho-de-função de cDNA. Acreditamos que o método fly TAP vai ser uma adição valiosa para a caixa de ferramenta utilizada em laboratórios de voar. Na perspectiva futura, este método pode ser combinado com mosca paradigmas comportamentais e modelos de doenças humanas para examinar a mudança na rede de interacção proteína-proteína em resposta a estímulos específicos e condições.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Agradecemos por nos enviar EUROSARF levedura TAP plasmídeos de expressão. Nós também estamos gratos pela ajuda editorial de Ryan Labadens. Este trabalho foi apoiado por uma bolsa do NIH / NINDS (R01NS070962) para CW
Materiais
| Name | Company | Catalog Number | Comments |
| U.S.A. standard test sieve No. 25 | Fisher Scientific | 04-881-18 | |
| U.S.A. standard test sieve No. 40 | Fisher Scientific | 04-881-21 | |
| Kontes Dounce Tissue Grinders 15 ml | Kimble Chase | 885300-0015 | |
| IgG sepharose beads | Pharmacia | 17-0969-01 | |
| Econo-column 0.7 cm x 20 cm | Bio-Rad | 737-4721 | |
| Econo-column 0.5 cm x 15 cm | Bio-Rad | 737-4716 | |
| Calmodulin beads | Stratagene | 214303 | |
| Coors Mortar and Pestle | CoorsTek | 60311 | |
| AcTEV Protease | Invitrogen | 12575-015 | |
| Protease Inhibitor Cocktail | Roche | 11836153001 | |
| Protease Inhibitor Mix | Sigma | P8340 |
Referências
- Rigaut, G., et al. A generic protein purification method for protein complex characterization and proteome exploration. Nat. Biotechnol. 17, 1030-1032 (1999).
- Collins, S. R., et al. Toward a comprehensive atlas of the physical interactome of Saccharomyces cerevisiae. Mol. Cell. Proteomics. 6, 439-450 (1074).
- Gavin, A. C., et al. Proteome survey reveals modularity of the yeast cell machinery. Nature. 440, 631-636 (2006).
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440, 637-643 (2006).
- Forler, D., et al. An efficient protein complex purification method for functional proteomics in higher eukaryotes. Nat. Biotechnol. 21, 89-92 (2003).
- Veraksa, A., Bauer, A., Artavanis-Tsakonas, S. Analyzing protein complexes in Drosophila with tandem affinity purification-mass spectrometry. Dev. Dyn. 232, 827-834 (2005).
- Li, Y. The tandem affinity purification technology: an overview. Biotechnol. Lett. 33, 1487-1499 (2011).
- Volkel, P., Le Faou, P., Angrand, P. O. Interaction proteomics: characterization of protein complexes using tandem affinity purification-mass spectrometry. Biochem. Soc. Trans. 38, 883-887 (2010).
- Xu, X., et al. The tandem affinity purification method: an efficient system for protein complex purification and protein interaction identification. Protein Expr. Purif. 72, 149-156 (2010).
- Brand, A. H., Perrimon, N. Targeted gene expression as a means of altering cell fates and generating dominant phenotypes. Development. 118, 401-415 (1993).
- Bachmann, A., Knust, E. The use of P-element transposons to generate transgenic flies. Methods Mol. Biol. 420, 61-77 (2008).
- Puig, O., et al. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Candiano, G., et al. Blue silver: a very sensitive colloidal Coomassie G-250 staining for proteome analysis. Electrophoresis. 25, 1327-1333 (2004).
- Tian, X., Wu, C. The role of ubiquitin-mediated pathways in regulating synaptic development, axonal degeneration and regeneration: insights from fly and worm. J. Physiol. , (2013).
- Wu, C., Wairkar, Y. P., Collins, C. A., DiAntonio, A. Highwire function at the Drosophila neuromuscular junction: spatial, structural, and temporal requirements. J. Neurosci. 25, 9557-9566 (2005).
- Wu, C., Daniels, R. W., DiAntonio, A. DFsn collaborates with Highwire to down-regulate the Wallenda/DLK kinase and restrain synaptic terminal growth. Neural Dev. 2, 16 (2007).
- Tian, X., Li, J., Valakh, V., DiAntonio, A., Wu, C. Drosophila Rae1 controls the abundance of the ubiquitin ligase Highwire in post-mitotic neurons. Nat. Neurosci. 14, 1267-1275 (2011).
- Burckstummer, T., et al. An efficient tandem affinity purification procedure for interaction proteomics in mammalian cells. Nat. Methods. 3, 1013-1019 (2006).
- Kyriakakis, P., Tipping, M., Abed, L., Veraksa, A. Tandem affinity purification in Drosophila: the advantages of the GS-TAP system. Fly. 2, 229-235 (2008).
- Bailey, D., Urena, L., Thorne, L., Goodfellow, I. Identification of protein interacting partners using tandem affinity purification. J. Vis. Exp. (60), e3643 (2012).
- Wu, Y., et al. A Drosophila model for Angelman syndrome. Proc. Natl. Acad. Sci. U.S.A. 105, 12399-12404 (2008).
- Liao, L., McClatchy, D. B., Yates, J. R. Shotgun proteomics in neuroscience. Neuron. 63, 12-26 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados