Method Article
Identificare proteina-proteina interazione in
In questo articolo
Riepilogo
Drosophila è famosa per la sua manipolazione genetica potente, ma non per la sua idoneità di analisi biochimica in profondità. Qui vi presentiamo una procedura basata TAP-per identificare interagiscono partner di qualsiasi proteina di interesse da parte del cervello della mosca. Questa procedura può potenzialmente portare a nuove vie di ricerca.
Abstract
Schermi genetici condotti utilizzando Drosophila melanogaster (moscerino della frutta) hanno fatto numerose scoperte pietra miliare nel progresso delle scienze biologiche. Tuttavia, l'uso di schermi biochimiche finalizzate ad estendere le conoscenze acquisite da analisi genetica è stata esplorata solo di recente. Qui si descrive un metodo per purificare il complesso proteico che si associa con qualsiasi proteina di interesse da teste di mosca adulta. Questo metodo sfrutta il sistema Drosophila GAL4/UAS per esprimere una proteina esca fuso con un Tandem Affinity Purification (TAP) tag in neuroni di topo in vivo e quindi implementa due cicli di purificazione utilizzando una procedura TAP simile a quello inizialmente stabilito in lievito 1 per purificare il complesso proteina interagente. Al termine di questa procedura, una miscela di diversi complessi proteici si ottiene la cui identità molecolare può essere determinato mediante spettrometria di massa. Validazione delle proteine candidati beneficeranno del reSource e facilità di esecuzione di perdita-di-funzione studi di mosche. Approcci simili possono essere applicati ad altri tessuti mosca. Crediamo che la combinazione di manipolazioni genetiche e questo approccio proteomico nel sistema modello fly detiene un enorme potenziale per affrontare i problemi fondamentali nel campo della neurobiologia e oltre.
Introduzione
Definizione dei meccanismi molecolari o reti che mediano un particolare processo biologico è uno degli obiettivi finali della ricerca biomedica. Fly genetisti hanno dipendeva pesantemente sulla genetica in avanti, soprattutto modificatore schermi genetici (sia enhancer e schermi soppressori), per identificare i fattori che lavorano insieme, in parallelo, o monte oa valle di un gene di interesse. Tuttavia, gli schermi genetica in avanti spesso non riescono a identificare i geni essenziali che, se mutato, causa letalità nelle fasi precoci dello sviluppo, oi geni con ridondanza funzionale e compensazione la cui perdita di funzione solo causare difetti sottili che sono difficili da segnare. Un modo per superare questa difficoltà è quello di schermo per interazioni dirette proteina-proteina. Per più di un decennio, un crescente elenco di metodi biochimici, tra cui due ibridi di lievito, esposizione su fago, reticolazione chimica, Co-IP, Tandem Affinity Purification (TAP), etc. sono stati utilizzati per studiare proteine-proteina interazioni. Ognuno di questi approcci ha una propria serie di punti di forza e di debolezza in materia di sensibilità e specificità. Tra questi, il metodo TAP consente il rilevamento di interazione fisica in condizioni quasi fisiologiche, conserva specificità e consistenza 2 e include la possibilità di estendere a high-throughput analisi 3,4.
Il metodo TAP è stato originariamente sviluppato nel lievito da colleghi Rigautand 1. In questo metodo, una proteina di interesse è espressa con un tag TAP. Il tag TAP ospita due affinità di legame domini indipendenti: una proteina Un dominio che si lega a IgG e un dominio-calmodulina vincolante. I due domini sono separati da una TEV (Tobacco Etch Virus) sito di clivaggio. Tale combinazione permette per due turni indipendenti di purificazioni affinità per ridurre sufficientemente le associazioni aspecifici e arricchiscono le associazioni specifiche 1. Per questo esempio, il metodo TAP è un potente metodologiad identificare interazioni in vivo di una data proteina, sebbene sovraesprimono la proteina esogena può rendere più inclini ad associare proteine che normalmente non complesso con la sua controparte endogena. Dal suo sviluppo, il metodo TAP è stato applicato in molti altri sistemi, inclusi-coltura a base di cellule sistemi 5,6 e altri sistemi modello in vivo 6-9. Qui si descrive l'adattamento del metodo TAP in Drosophila. Per prima cosa generiamo pUAST-NTAP e pUAST-CTAP vettori per facilitare la clonazione e la fusione del tag TAP sia alla N-o C-terminale del gene di interesse. Il UAS-TAP-tag transgene viene poi espressa nel sistema nervoso sotto il controllo di un driver GAL4 neuronale 10. Successivamente, verrà raccolto un gran numero di teste mosca adulta, che hanno un alto contenuto di cellule neurali e sono facili da separare da altre parti del corpo dopo il congelamento sulla base delle differenze di dimensioni. Le teste adulti sono omogeneizzati e liquidati bcentrifugazioni sequenziali y, e il surnatante è soggetto a una procedura di TAP descritta di seguito.
Protocollo
1. Generare UAS-TAP-tag mosche transgeniche
- Generare pUAST-TAP-tagged costrutti di DNA.
- Decidere quale lato (N-o C-terminale) della proteina esca il tag TAP deve essere fuso a, sulla base della struttura / funzione della proteina. Si veda la discussione per ulteriori dettagli.
- Subclone la regione codificante del cDNA del gene di interesse nei siti di clonazione multipla (MCS) dei vettori pUAST-NTAP o pUAST-CTAP per generare N-o C-terminale con targhetta transgeni UAS-TAP, rispettivamente. Vedere la Figura 1 per le mappe dettagliate e siti di restrizione utilizzabili e cornici di lettura.
- Generare UAS-TAP-tagged mosche transgeniche.
- Generare mosche transgeniche seguenti protocolli standard utilizzando P-elemento-mediata di inserimento 11. Un certo numero di servizi di iniezione sono disponibili in commercio.
- Attraversare neuronale autista GAL4 (cioè BG380-GAL4) per ogni singola linea transgenica e determinare i livelli di espressione della proteinadi ogni riga mediante western blot (con anticorpi perossidasi anti-perossidasi) e / o immunostaining (con anticorpo anti-TAP). In generale, una linea transgenica con un livello di espressione della proteina che si trova vicino alla proteina endogena è raccomandato per procedure TAP. Si veda la discussione per ulteriori dettagli.
- PerformGAL4/UAS-based esperimenti di salvataggio per confermare la funzionalità dei transgeni TAP-tag se sono disponibili la perdita di funzione mutanti dei geni di interesse. Scegliere un transgene che può salvare la sostanza dei fenotipi mutanti per i seguenti esperimenti TAP.
2. Preparare i campioni per TAP procedura
- Generare un titolo mosca che porta sia un driver neuronale GAL4 (es. BG380-Gal4) e il transgene TAP-tag scelti in modo da facilitare l'espansione dei campioni mosca. Raccogliere le progenie F1 del conducente GAL4 e la croce UAS-transgene nei rari casi in cui la combinazione di cui sopra determina la sopravvivenza e la crescita di svantaggio.
- Raccogliere campioni di piccole dimensioni e ottimizzare le condizioni di lisi per solubilizzare la proteina TAP-tag.
- Effettuare una serie di buffer di lisi utilizzando una combinazione dei detergenti non ionici NP-40 (0,1-1%), NaDOC (0,1-1%) e Triton X-100 (0,05-0,5%). Vedi Tabella 1 e di discussione per maggiori informazioni.
- Sulla cima di un pad CO2, utilizzare # 5 pinze per sezionare 20 capi adulti dal transgene TAP esprimere mosche e raccoglierli in una provetta da 1,5 ml a testare ogni condizione di buffer di lisi.
- Aggiungere 100 ul di buffer test di lisi al tubo e omogeneizzare le testine segnando su e giù con un pestello in plastica, poi aggiungere un altro buffer test 100 ul.
- Centrifugare il lisato testa a 21.500 xg per 10 min (4 ° C), e separare il surnatante e pellet dopo centrifugazione. Aggiungere 25 ul 2x SDS tampone di caricamento per il pellet e tampone di caricamento 10 ul 4x SDS a 30 ul di surnatante, rispettivamente.
- Bollire i due campioni per 5 min e analizzarli side-byLato usando SDS-PAGE e successivo Western Blot con l'anticorpo PAP. Determinare la solubilità dal rapporto tra livelli TAP-proteina nelle supernatante vs il pellet.
- Preparare campione in larga scala
- Espandere e raccogliere mosche adulte.
- Espandere lo stock neuronal-GAL4/UAS-TAP-transgene in bottiglie e capovolgere le bottiglie ogni 3 giorni fino cumulative di 250 bottiglie vengono utilizzati per la raccolta.
- Raccogliere 1-3 giorni di età adulta vola in 50 ml provette coniche, mettere il tubo in azoto liquido immediatamente Deep Freeze linea. Conservare le mosche in una -80 ° C freezer. Si noti che il volume di linea non deve superare i 2/3 del tubo da 50 ml.
- Raccogliere teste fly (eseguire questo passaggio sulla cima di ghiaccio secco in polvere).
- Estrarre i setacci prechilled e il mortaio e pestello da -80 ° C freezer e metterli su ghiaccio secco, idealmente all'interno di un grande secchio di ghiaccio. Stack due setacci di prova standard USA con un No. 25 su °e superiore e No. 40 in basso.
- Prendere le mosche congelati fuori e farli cadere in azoto liquido e conservare in linea in là per circa 10 min. Vortex o agitare le provette energicamente per rompere le teste, le gambe e le ali dai corpi.
- Versare il composto al setaccio superiore, e quindi scuotere i setacci energicamente tenendo setacci insieme. Dopo la setacciatura, i corpi rimarranno sul setaccio superiore, volare teste saranno conservati sul setaccio di fondo, e le gambe, ali, e altri detriti cadranno al ghiaccio secco. Separare i due setacci e con attenzione trasferire i capi mosca al mortaio freddo.
- Omogeneizzare le teste fly
- In cima a ghiaccio secco, macinare i capi con il mortaio e il pestello di particelle in polvere, poi trasferire la polvere a 15 ml di vetro Dounce Tissue Grinder che è stato prechilled sul ghiaccio.
- Misurare i pesi della smerigliatrice prima e dopo il campione testa è stata versata in esso, e quindi calcolare qual è il samp testale pesa. Un totale di 6-15 grammi di teste fly sarà sufficiente per ciascun esperimento TAP regolando di conseguenza per i livelli di espressione della proteina. Aggiungere 15 ml di tampone di omogeneizzazione ghiacciato (tampone di lisi ottimizzata nel passaggio 2.2) per la polvere e poi corsa con il grande pestello pallone fino a che è facile per il pestello per andare su e giù. Mantenere la smerigliatrice vetro su ghiaccio in ogni momento.
- Preparare il surnatante per TAP
- Trasferire l'omogeneizzato in una provetta da centrifuga ad alta velocità e centrifuga per 20 minuti a 50.000 xg ~ (4 ° C). Trasferire il surnatante in una nuova provetta da centrifuga ad alta velocità e ripetere la centrifugazione ancora una volta.
- Trasferire il supernatante in un tubo ultracentrifuga ed eseguire un 40 min ~ 250.000 xg centrifuga per eliminare ulteriormente il surnatante. Il supernatante è pronto per le procedure di purificazione per affinità tandem dopo ultracentrifugazione.
- Espandere e raccogliere mosche adulte.
3. TAP Purificazione
Le sezioni che seguono sono stati ricavati dal laboratorio TAP Séraphin protocol 12 (http://web.as.uky.edu/Biology/faculty/rymond/BIO%20510/Bertran%20Seraphin%27s%20TAP%20page.pdf )
- Eseguire IgG tallone purificazione per affinità
- Preparare IgG Sepharose tallone mentre i campioni vengono centrifugati. Lavare 400 microlitri 3x perline IgG in un tubo Falcon da 15 ml con 10 ml di freddo tampone di lavaggio IgG. Per ogni lavaggio, scuotere delicatamente il tubo per 2 minuti, e poi girare le perle a 1.000 xg per altri 2 min. Alla fine del terzo lavaggio, rimuovere il tampone e lasciare solo le perline nel tubo.
- Trasferire con cautela il surnatante eliminato (~ 15 ml) nel tubo da 15 ml contenente le perline IgG. Incubare le perline e mix lisato cervello a 4 º C su una nutator per 2 ore.
- Impostare un micro colonna pulito e vuoto con circa 15 ml di volume totale nella stanza fredda. Caricare la miscela sferette IgG dalla costante versare la miscela nella colonna, non cercare di intrappolare qualsiasibolle d'aria all'interno della colonna. Lasciare le perle di stabilirsi nella colonna e il buffer per drenare lentamente flusso di gravità.
- Lavare la colonna a fondo con 10 ml di freddo tampone di lavaggio IgG dopo tutto il lisato cervello ha fluito attraverso la colonna IgG risolta. Ripetere il lavaggio 2x. Nota: non lasciare mai che il tallone asciugare all'aria.
- Eseguire TEV scissione
- Dopo il terzo lavaggio, lavare nuovamente la colonna con 10 ml di tampone TEV scissione. Questo passaggio prepara il tallone IgG che sequestranti il complesso esca per TEV scissione.
- Destra prima dell'ultima goccia di tampone TEV sta per gocciolare, mettere un tappo nella parte inferiore della colonna di bloccare il flusso, aggiungere 1,3 ml di tampone TEV contenente 130 unità di enzima TEV alla colonna, e quindi tappare saldamente cima colonna. Assicurarsi che la colonna è sigillato bene ad entrambe le estremità.
- Ruotare la colonna a 18 ° C per 2 ore per consentire l'enzima TEV per fendere il peptide al sito TEV e rilasciare il complesso proteico WHIle lasciandosi alle spalle la proteina A peptide dominio legata alle perle del IgG Sepharose.
- Eseguire Calmodulina tallone purificazione per affinità
- Preparare le perline calmodulina mentre le perle IgG vengono incubate con l'enzima TEV. Lavare 200 microlitri calmodulina perline in 15 ml di 3x tubo Falcon, ogni volta con 10 ml di freddo Calmodulin binding buffer. Per ogni lavaggio, scuotere delicatamente il tubo per 2 minuti su un nutator, e poi girare le perle a 1.000 xg per 2 minuti. Alla fine del terzo lavaggio, estrarre tutto il buffer e lasciare solo le perline nel tubo.
- Al termine dell'incubazione TEV (fase 3.2.3), riportare la colonna IgG torna alla camera fredda e impostare verso l'alto. Lasciate le perline accontentarsi di 10 min.
- Togliere il tappo superiore e poi il tappo inferiore, e poi raccogliere il 1.3 ml TEV prodotto di scissione in un tubo Falcon 15 ml. Lasciate che il buffer di scarico completamente. Aggiungere un ulteriore buffer di 200 microlitri TEV alla colonna per spingere fuori il volume morto della colonna, raccogliere la fbasso nello stesso tubo.
- Aggiungere 4,5 ml di tampone di legame Calmodulin e 4,5 ml 1 M CaCl 2 per il prodotto di scissione 1,5 ml TEV raccolti sopra. Il CaCl 2 serve per titolare l'EDTA nel buffer TEV. Trasferire la miscela 6 ml nella provetta contenente le perline calmodulina e ruotare il tubo a 4 ° C su una nutator per 1 ora.
- Impostare un altro micro colonna pulito e vuoto con circa 10 ml di volume totale nella stanza fredda. Caricare la miscela sferette Calmodulina alla colonna e lasciarlo scolare per gravità.
- Quando tutta la soluzione è fluito attraverso la colonna Calmodulina costante, lavare la colonna due volte, ciascuna con 10 ml di freddo Calmodulin binding buffer. Nota: evitare di disturbare le perline calmodulina e cercare di mantenere la superficie delle perle più piatta possibile durante il lavaggio.
- Eluire il complesso esca dalla colonna Calmodulin.
Subito dopo il lavaggio, eluire la colonna Calmodulina con cinque frazioni di 200 microlitri freddo Calmodulin tampone di eluizione. Per ciascuna frazione, aggiungere lentamente 200 ml di tampone di eluizione alla colonna e raccogliere l'eluato con una marcata 1,5 ml provetta Eppendorf. Ripetere questa 4x. - Analizzare il complesso proteico mediante SDS-PAGE
- Prendere una piccola aliquota da ciascuna delle cinque frazioni (circa 30 mL) e aggiungere tampone di caricamento SDS. Bollire i campioni per 5 min e caricare i campioni side-by-side con marcatori molecolari di proteine in un gradiente (4-15%) SDS-PAGE gel.
- Dopo che i campioni sono completamente risolti nel gel, fermare la elettroforesi e macchiare il gel con qualsiasi sensibili colloidali procedure di colorazione Coomassie basate G-250 come 'argento blu' colorazione 13. Argento colorazione è opzionale ma non preferibile perché non è completamente compatibile con la successiva analisi di spettrometria di massa. Conservare il resto l'eluato in un congelatore -80 ° C per ulteriori analisi quali spettrometria di massa per scoprire le identità molecolare del complesso proteina purificata. See discussione.
Risultati
Qui mostriamo il nostro sforzo per identificare le proteine Highwire interagenti nel cervello della mosca. Highwire (HIW) e le sue vertebrati e invertebrati omologhi sono enormi ubiquitina ligasi che regolano lo sviluppo e la riparazione del sistema nervoso 14. Essi condividono un certo numero di domini funzionali altamente conservate. Tuttavia, le loro azioni molecolari non sono del tutto chiare. Lavoro fatto in verme, vola e mouse hanno portato a modello di lavoro corrente che funzioni hiw come ligasi E3 e come una proteina ponteggio per facilitare la formazione di un complesso ubiquitinazione multi-subunità, che regola le funzioni neuronali specifici del tipo a tempo e di cella attraverso la combinazione di interagire con diversi cofattori e il targeting diversi substrati ubiquitina. Per identificare il complesso ubiquitination hiw-associata, in primo luogo abbiamo generato un N-terminale tagged transgene UAS-TAP-hiw che è pienamente funzionale nel salvataggio del hiw fenotipo mutante 15. Circa 10 g di fl adultiteste y che esprimono TAP solo o proteine transgeniche TAP-hiw stati raccolti e sottoposti a procedure TAP lato all'altro come descritto sopra. Eluati finale di entrambe le purificazioni sono stati analizzati mediante SDS-PAGE e poi colorazione argentica. La spettrometria di massa ha identificato un elenco di proteine in soltanto il campione TAP-hiw, compresi Drosophila FSN (DFSN, una proteina F-box) e Rae1 (Figura 2). Analisi genetiche e biochimiche successivi hanno rivelato che hiw e DFSN lavorare insieme come SCF-like complesso ubiquitina ligasi E3 per regolare struttura e funzione 16 sinaptica, e associa Rae1 con hiw in vivo e frena la crescita eccessiva sinaptica 17. Questa funzione di Rae1 è almeno parzialmente raggiunto dalla sua capacità di promuovere hiw stabilità della proteina tramite hiw proteggere dalla degradazione autophagic, che rivela un nuovo meccanismo che controlla selettivamente hiw proteina abbondanza durante lo sviluppo sinaptico 17.
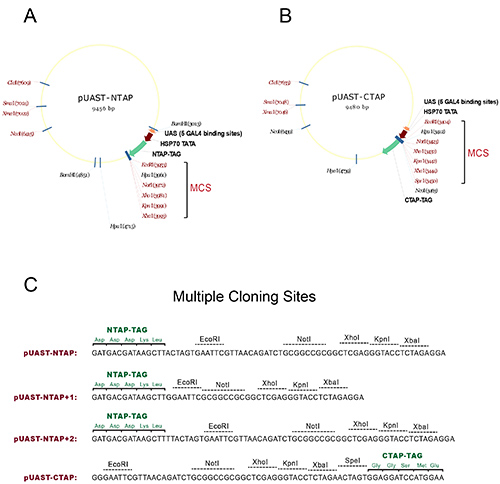
Figura 1. Vettori per la generazione di mosche transgeniche TAP-tag. (A) La mappa del pUAST-NTAP costrutto. (B) La mappa del pUAST-CTAP costrutto. I molteplici siti di clonaggio (MCS) in entrambi i costrutti sono contrassegnati con una staffa, dove i siti di restrizione singolo taglio sono rappresentati con il colore rosso. (C) sequenza primaria che mostra i diversi siti di clonaggio dei vettori rubinetto con la cornice di lettura del tag TAP. Per facilitare subcloning, altri due costrutti NTAP vengono generati dall'originale pUAST-NTAP: pUAST-NTAP +1 e pUAST-NTAP +2. Insieme i tre NTAP costruisce copre tutte le tre cornici di lettura per accomodare l'uso della MCS. Tutti i vettori sono costruiti utilizzando la NTAP o frammenti CTAP originariamente generato inSeraphin Lab 1. Clicca qui per vedere l'immagine ingrandita.

. Figura 2 Purificazione di Highwire interagenti proteine da mosca cervello di purificazione TAP capi adulti di mosche che esprimono TAP. (BG380-GAL4, UAS-TAP) o TAP-hiw (BG380-GAL4, UAS-TAP-HIW) nel sistema nervoso sono raccolti, omogeneizzati, e sottoposto a purificazione TAP, rispettivamente. Gli eluati finali sono stati analizzati mediante unidimensionale gel SDS-PAGE seguita da colorazione con argento. La punta della freccia indica l'esca proteica hiw e l'asterisco indica una proteina TAP troncata a causa di TEV scissione. Nel campione TAP-hiw, una lista di proteine sono identificati in massaspettrometria, compresi HSC-70, β-tubulina, Rae1 e DFSN (indicato dalle frecce). Questa figura è modificato dalla figura 1a di Tian et al. 17 Clicca qui per vedere l'immagine ingrandita.
| Buffer | Composizione | Commenti |
| Tampone di lisi | 50 mM Tris-HCl pH 7.5 | NOTE: 1) Aggiungere DTT e le proteasi e inibitori del proteasoma appena prima dell'uso. |
| 125 mM NaCl | 2) Risultati: ecco un tampone di lisi con lo 0,5% NP40. Si veda la discussione per le modifiche relative ai detergenti non ionici. | |
| 5% Glicerina | ||
| 0,5% NP40 | ||
| 1,5 mM MgCl 2 | ||
| 25 mM NaF | ||
| 0.2 mM DTT | ||
| (Di seguito sono inibitori della proteasi e del proteasoma) | ||
| 1 mM Na 3 VO 4 | ||
| 0,05 mm MG-115 | ||
| 1 mM PMSF | ||
| Protease mix inibitore (Sigma P8340) | ||
| Inibitori della proteasi cocktail (Roche 04693159001) | ||
| Tampone di lavaggio IgG | 10 mM Tris-Cl pH 8,0 (0,5 ml di 2 M magazzino) | |
| 150 mM NaCl (3 ml di 5 m stock) | ||
| 0,1% NP40 (1,0 ml di 10% stock) | ||
| H 2 0 a 100 ml finale | ||
| Buffer di scissione TEV | 10 mM Tris-Cl pH 8,0 (0,5 ml di 2 M magazzino) | NOTA: aggiungere DTT poco prima dell'uso. |
| 150 mM NaCl (3 ml di 5 m stock) | ||
| 0,1% NP40 (1,0 ml di 10% stock) | ||
| EDTA 0,5 mm (100 ml di 0,5 M magazzino) | ||
| 1 mM DTT (100 ml di 1 M magazzino) | ||
| H 2 O a 100 ml finale | ||
| Calmodulin binding buffer | 10 mM β-mercaptoetanolo (69,7 ml di magazzino) | |
| 10 mM Tris-Cl pH 8,0 (0,5 ml di 2 M magazzino) | ||
| 150 mM NaCl (3 ml di 5 m stock) | ||
| 1 mM Mg-acetato (100 ml di 1 M magazzino) | ||
| 1 imidazolo mm (100 ml di 1 M magazzino) | ||
| 2 mM CaCl 2 (200 ml di 1 M magazzino) | ||
| 0,1% NP40 (1 ml di 10% stock) | ||
| H 2 O a 100 ml finale | ||
| Tampone di eluizione Calmodulin | 10 mM β-mercaptoetanolo (69,7 ml di magazzino) | |
| 10 mM Tris-Cl pH 8,0 (0,5 ml di 2 M magazzino) | ||
| 150 mM NaCl (3 ml di 5 m stock) | ||
| 1 mM Mg-acetato (100 ml di 1 M magazzino) | ||
| 1 imidazolo mm (100 ml di 1 M magazzino) | ||
| EGTA 10 mm (2 ml di 0,5 M magazzino) | ||
| 0,1% NP40 (1 ml di 10% stock) | ||
| H 2 O a 100 ml finale |
Tabella 1. Composizione di buffer.
Discussione
Metodo Tandem purificazione per affinità (TAP) offre un protocollo dual purificazione che permette l'isolamento e l'arricchimento di complessi proteici attraverso due passaggi di purificazione di affinità indipendenti. Il design del tag TAP non è limitata a ciò che viene presentato in questo protocollo, le altre proteine domini di legame e motivi sono applicabili anche se le condizioni del buffer vengono adeguati di conseguenza. Un buon esempio di altri tag TAP è il tag GS-TAP, una combinazione di una proteina G e un motivo-streptavidina vincolante, progettata dal gruppo di Giulio Superti Furga, volto a complesso proteico purificante in colture di linee cellulari di mammiferi 18. Il GS-TAP è stato successivamente adattato per studiare l'interazione proteina-proteina in cellule di Drosophila S2 e embrioni 19. Una recente pubblicazione JoVE da Bailey et al. Ha dimostrato come la procedura di GS-TAP è stata effettuata con colture di cellule lisato 20. Qui abbiamo presentato l'uso del TAP-tag nei tessuti neurali Drosophila (adteste ULT) per lo screening proteomica su larga scala. Questo protocollo può essere potenzialmente adattato ad altri tessuti che permettono la raccolta del campione larga scala, come gli embrioni. I corpi adulti raccolti sul setaccio superiore (fase 2.3.2.3) possono essere utilizzati anche per TAP, che può essere un compito molto impegnativo. Il tampone di lisi deve essere modificato e condizionato per inibire le proteasi massicce presenti nel tratto digestivo al massimo e la prima procedura di purificazione notevolmente abbreviati al fine di ridurre il tempo di esposizione delle proteine alle proteasi. Anche se diversi tag TAP richiedono diversi maneggi e le condizioni di buffer per purificazioni di affinità, i principi generali per TAP sono la stessa cosa. Qui di seguito discutiamo questioni che influenzano la qualità dei risultati TAP.
Il successo dell'approccio TAP dipende generazione del transgene destra. In primo luogo, il tag TAP stessa non deve modificare la conformazione complessiva della proteina esca, che è fondamentale per mantenere la sua capacitàper interagire con i partner di legame fisiologiche. Pertanto, è fondamentale decidere dove fondere il tag TAP al gene di interesse. La decisione può essere basata su studi precedenti della proteina, in particolare informazioni sulla sua struttura o funzionali transgeni epitode-tag. Nel caso in cui tale informazione non è disponibile, la codifica la proteina esca a N-o C-terminale in parallelo è raccomandato. Poi esperimenti soccorso funzionali possono essere eseguite per determinare quale transgene deve essere utilizzato. In secondo luogo, i livelli di espressione della proteina transgenica dovrebbero essere vicina a quella della proteina endogena. Ciò è particolarmente importante in quanto il sistema Gal4/UAS guidare normalmente l'espressione del transgene in diversi tempi e ad un livello molto più elevato rispetto alle proteine endogene, che possono sia aumentare falsi positivi o provocare conseguenze inaspettate che alterano il profilo dell'interazione proteina reti nelle cellule. Ad esempio, che iperesprimono proteine ponteggi possono Cause gli effetti negativi dominanti e proteine iperesprimenti che possiedono attività enzimatiche, quali le chinasi, spesso causano guadagno di funzione effetti, entrambi i quali possono a loro volta influenzare la capacità dei loro partner di legame endogeni per interagire con essi. Così, il sistema GAL4/UAS dovrebbe essere evitato quando la sincronizzazione e livello di espressione del gene di interesse è fondamentale per la tutela delle interazioni endogene che presentano solo in condizioni normali. Invece, l'espressione del transgene TAP-tag deve essere controllata con il proprio promotore. Ciò può essere ottenuto o sostituendo la sequenza UAS con la sequenza promotore endogeno, o fondendo la sequenza TAP in frame con la sequenza exonal in un frammento di DNA genomico che contiene l'intero loci del gene di interesse 21. In alcuni casi, l'esperimento TAP può essere eseguita in una perdita di funzione mutante sfondo del gene di interesse per ridurre la concorrenza dalla proteina endogena per incorporating in complessi multiproteici. Per questo esempio, una proteina sfondo nullo, se disponibile, fornisce la condizione ideale per eliminare completamente la proteina endogena. Utilizzando lo sfondo mutante genetico sarà sempre aumentare le possibilità di produrre risultati di qualità superiore.
Solubilità della proteina esca è un altro fattore fondamentale per il successo. Detergenti non ionici sono normalmente utilizzati in tampone di lisi per sciogliere la membrana plasmatica e trattenere il complesso proteico, solubile e intatta, in uno stato che è vicino l'ambiente fisiologico. Tuttavia a seconda se le proteine di interesse sono proteine o associate alla membrana integrata, la composizione e la concentrazione di detergenti non ionici possono essere cruciale per solubilizzare la proteina esca. 0,1-0,3% NP40 è spesso sufficiente a solubilizzare le proteine citosoliche. Per proteine di membrana, una combinazione di NP40 (0,1-1%), NaDOC (0,1-1%) e Triton X-100 (0,05-0,5%) è talvolta richiesto di solubilitàLize e arricchire abbastanza proteine per l'esperimento TAP. Tuttavia, poiché un tampone di lisi rigorosa tendono a disturbare deboli interazioni proteina-proteina, un equilibrio deve essere raggiunto in cui sono presenti nel campione proteine esca abbastanza solubili mentre allo stesso tempo la maggior parte delle interazioni proteina-proteina sono conservati. Un principio generale è che cerca sempre di utilizzare i tipi minor numero di detersivi con la concentrazione più bassa.
Per facilitare la risoluzione dei problemi, si consiglia vivamente di salvare una piccola aliquota di campione da ogni passo dei due purificazioni. Successivi SDS-PAGE e colorazione delle proteine / analisi Western delle aliquote consentire i livelli della proteina esca e l'arricchimento sequenziale di alcune specie proteiche da monitorare. Ad esempio, se una improvvisa riduzione della proteina esca è stata rilevata in aliquota del flusso dopo scissione TEV (passo 3.3.3), è probabile che la proteina esca stati persi durante la IgG tallone lavaggio (passo 3.1.4) . A possbile causa potrebbe essere la composizione detergente inadeguato del tampone di lavaggio IgG. C'è una forte riduzione sulla composizione e concentrazione dei detergenti nel tampone di lavaggio IgG (0,1% NP-40), rispetto al tampone di lisi. La proteina esca può essere sensibile a questi cambiamenti e dissociare dalla frazione IgG sulle perline. Regolazione del lavaggio IgG verso tampone di lisi può aiutare a risolvere il problema. In alternativa, anche se meno probabile, la scissione TEV (passo 3.2) può non essere sufficientemente lavorato per rilasciare il complesso contenente esca dalle perline IgG. I passi che meritano una seconda ispezione sono il raggiungimento dell'equilibrio di perline IgG con il buffer di TEV (fase 3.2.1) e l'attività enzimatica, maltrattamento può attutire l'enzima.
Tenete a mente che anche con la purificazione due fasi, ci saranno ancora falsi positivi e proteine che sono tirati giù non specifico presente nel complesso TAP-purificato finale. Un modo per identificare le interazioni aspecifiche, aalmeno in parte, è quello di includere un TAP controllo utilizzando il TAP-only transgene in parallelo con TAP-tag-transgene negli esperimenti di purificazione TAP. Le proteine individuate nella procedura solo TAP-devono essere rimossi dall'elenco individuato nella purificazione TAP-transgene.
Un TAP successo purificherà una miscela di proteine che sono potenzialmente associati con la proteina di interesse in vivo. Il passo successivo è l'identificazione molecolare di queste proteine, e la spettrometria di massa è comunemente utilizzato per questo scopo. A seconda dei risultati della SDS-PAGE e le esigenze dell'esperimento, le identità molecolari nel complesso proteina purificata può essere determinato nei seguenti due modi: 1) ciascuna banda proteica individuo può essere tagliata dal gel e sottoposto a una bassa complessità LC-MS/MS o 2) la frazione eluire che contiene il contenuto proteico di picco (normalmente la seconda eluizione) può essere direttamente sottoposto a moderata complessità LC-MS/MS. Tale fucile da caccia papproccio roteomic potenzialmente identificare tutte le proteine nel complesso, a seconda della gamma dinamica dell'apparecchiatura MS 22.
In sintesi, vi presentiamo un metodo per identificare le interazioni proteina-proteina nei tessuti neurali di un organismo modello genetico - la mosca della frutta. Ci sono una serie di vantaggi dell'applicazione del metodo TAP al fly: 1) i moscerini della frutta hanno un ciclo di vita breve, quindi è relativamente semplice e veloce per generare le mosche transgeniche e di ottenere tessuti in grande quantità, entrambi sono cruciali per il TAP approccio, 2) la mosca genoma è completamente annotato e contiene il 70% dei geni che causano malattie umane, e 3) più importante, è facile da validare e caratterizzare funzionalmente il candidato proteine interagenti in mosche. Ci sono risorse globali disponibili nella comunità della ricerca mosca per la caratterizzazione di un dato gene, come ad esempio a base di transgenici raccolta utile RNAi per studiare tessuto-specifica la perdita di funzione di una maggioranza del genes, alleli mutanti e carenza per la maggior parte del gene loci, nonché cloni di cDNA e transgeni utili per guadagno di funzione studi. Noi crediamo che il metodo mosca TAP sarà una preziosa aggiunta alla casella di strumento utilizzato nei laboratori di mosca. In prospettiva futura, questo metodo può essere combinato con fly paradigmi comportamentali e modelli di malattia umana per schermare la variazione proteina-proteina rete interazione in risposta a condizioni e stimoli specifici.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo EUROSARF per averci inviato il lievito TAP plasmidi di espressione. Siamo anche grati per l'aiuto editoriale da Ryan Labadens. Questo lavoro è stato sostenuto da una borsa NIH / NINDS (R01NS070962) per CW
Materiali
| Name | Company | Catalog Number | Comments |
| U.S.A. standard test sieve No. 25 | Fisher Scientific | 04-881-18 | |
| U.S.A. standard test sieve No. 40 | Fisher Scientific | 04-881-21 | |
| Kontes Dounce Tissue Grinders 15 ml | Kimble Chase | 885300-0015 | |
| IgG sepharose beads | Pharmacia | 17-0969-01 | |
| Econo-column 0.7 cm x 20 cm | Bio-Rad | 737-4721 | |
| Econo-column 0.5 cm x 15 cm | Bio-Rad | 737-4716 | |
| Calmodulin beads | Stratagene | 214303 | |
| Coors Mortar and Pestle | CoorsTek | 60311 | |
| AcTEV Protease | Invitrogen | 12575-015 | |
| Protease Inhibitor Cocktail | Roche | 11836153001 | |
| Protease Inhibitor Mix | Sigma | P8340 |
Riferimenti
- Rigaut, G., et al. A generic protein purification method for protein complex characterization and proteome exploration. Nat. Biotechnol. 17, 1030-1032 (1999).
- Collins, S. R., et al. Toward a comprehensive atlas of the physical interactome of Saccharomyces cerevisiae. Mol. Cell. Proteomics. 6, 439-450 (1074).
- Gavin, A. C., et al. Proteome survey reveals modularity of the yeast cell machinery. Nature. 440, 631-636 (2006).
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440, 637-643 (2006).
- Forler, D., et al. An efficient protein complex purification method for functional proteomics in higher eukaryotes. Nat. Biotechnol. 21, 89-92 (2003).
- Veraksa, A., Bauer, A., Artavanis-Tsakonas, S. Analyzing protein complexes in Drosophila with tandem affinity purification-mass spectrometry. Dev. Dyn. 232, 827-834 (2005).
- Li, Y. The tandem affinity purification technology: an overview. Biotechnol. Lett. 33, 1487-1499 (2011).
- Volkel, P., Le Faou, P., Angrand, P. O. Interaction proteomics: characterization of protein complexes using tandem affinity purification-mass spectrometry. Biochem. Soc. Trans. 38, 883-887 (2010).
- Xu, X., et al. The tandem affinity purification method: an efficient system for protein complex purification and protein interaction identification. Protein Expr. Purif. 72, 149-156 (2010).
- Brand, A. H., Perrimon, N. Targeted gene expression as a means of altering cell fates and generating dominant phenotypes. Development. 118, 401-415 (1993).
- Bachmann, A., Knust, E. The use of P-element transposons to generate transgenic flies. Methods Mol. Biol. 420, 61-77 (2008).
- Puig, O., et al. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Candiano, G., et al. Blue silver: a very sensitive colloidal Coomassie G-250 staining for proteome analysis. Electrophoresis. 25, 1327-1333 (2004).
- Tian, X., Wu, C. The role of ubiquitin-mediated pathways in regulating synaptic development, axonal degeneration and regeneration: insights from fly and worm. J. Physiol. , (2013).
- Wu, C., Wairkar, Y. P., Collins, C. A., DiAntonio, A. Highwire function at the Drosophila neuromuscular junction: spatial, structural, and temporal requirements. J. Neurosci. 25, 9557-9566 (2005).
- Wu, C., Daniels, R. W., DiAntonio, A. DFsn collaborates with Highwire to down-regulate the Wallenda/DLK kinase and restrain synaptic terminal growth. Neural Dev. 2, 16 (2007).
- Tian, X., Li, J., Valakh, V., DiAntonio, A., Wu, C. Drosophila Rae1 controls the abundance of the ubiquitin ligase Highwire in post-mitotic neurons. Nat. Neurosci. 14, 1267-1275 (2011).
- Burckstummer, T., et al. An efficient tandem affinity purification procedure for interaction proteomics in mammalian cells. Nat. Methods. 3, 1013-1019 (2006).
- Kyriakakis, P., Tipping, M., Abed, L., Veraksa, A. Tandem affinity purification in Drosophila: the advantages of the GS-TAP system. Fly. 2, 229-235 (2008).
- Bailey, D., Urena, L., Thorne, L., Goodfellow, I. Identification of protein interacting partners using tandem affinity purification. J. Vis. Exp. (60), e3643 (2012).
- Wu, Y., et al. A Drosophila model for Angelman syndrome. Proc. Natl. Acad. Sci. U.S.A. 105, 12399-12404 (2008).
- Liao, L., McClatchy, D. B., Yates, J. R. Shotgun proteomics in neuroscience. Neuron. 63, 12-26 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon