Method Article
Identifier les interactions protéine-protéine dans
Dans cet article
Résumé
Drosophile est célèbre pour sa manipulation génétique puissant, mais pas pour son aptitude d'analyse biochimique en profondeur. Ici, nous présentons une procédure basée TAP à identifier les partenaires d'interaction de toute protéine d'intérêt à partir du cerveau de la mouche. Cette procédure peut potentiellement conduire à de nouvelles pistes de recherche.
Résumé
Écrans génétiques effectuées à l'aide Drosophila melanogaster (de la mouche des fruits) ont fait de nombreuses découvertes d'étape dans le progrès des sciences biologiques. Cependant, l'utilisation d'écrans biochimiques visant à étendre les connaissances acquises à partir de l'analyse génétique a été explorée que récemment. Ici, nous décrivons une méthode pour purifier le complexe de protéine qui s'associe avec n'importe quelle protéine d'intérêt à partir des têtes de mouches adultes. Cette méthode tire parti du système Drosophila GAL4/UAS d'exprimer une protéine appât fusionnée avec une étiquette Tandem Affinity Purification (TAP) dans les neurones de la mouche in vivo, puis met en œuvre deux cycles de purification en utilisant une procédure de TAP similaire à celui établi à l'origine en une levure pour purifier le complexe protéique interagissant. A la fin de cette procédure, un mélange de plusieurs complexes protéiques dont l'identité est obtenue moléculaire peut être déterminée par spectrométrie de masse. Validation des protéines candidates bénéficiera de la resource et la facilité d'effectuer des études de perte de fonction chez les mouches. Des approches similaires peuvent être appliqués à d'autres tissus de braguette. Nous pensons que la combinaison de manipulations génétiques et cette approche protéomique dans le système de modèle de la mouche détient un énorme potentiel pour résoudre les problèmes fondamentaux dans le domaine de la neurobiologie et au-delà.
Introduction
Définir les voies moléculaires ou des réseaux qui interviennent dans un processus biologique particulier est l'un des buts ultimes de la recherche biomédicale. Fly généticiens ont beaucoup compté sur la génétique à terme, en particulier modificateur écrans génétiques (deux amplificateurs et écrans suppresseurs), d'identifier les facteurs qui travaillent ensemble, en parallèle avec, ou en amont ou en aval d'un gène d'intérêt. Cependant, les écrans de génétique à terme arrive souvent ne parviennent pas à identifier les gènes essentiels qui, lorsqu'ils sont mutés, provoquent létalité à des stades précoces du développement, ou des gènes avec redondance fonctionnelle et d'indemnisation dont la perte de fonction ne causer des anomalies subtiles qui sont difficiles à marquer. Une façon de surmonter cette difficulté consiste à cribler des interactions protéine-protéine directe. Pour plus d'une décennie, un nombre croissant de méthodes biochimiques, y compris les deux hybrides de levure, phage display, réticulation chimique, Co-IP, Tandem Affinity Purification (TAP), etc. ont été utilisées pour étudier la protéine-les interactions protéiques. Chacune de ces approches a ses propres forces et faiblesses en ce qui concerne la sensibilité et la spécificité. Parmi eux, la méthode TAP permet la détection de l'interaction physique dans des conditions quasi-physiologiques, préserve la spécificité et la cohérence 2 et comprend la possibilité d'étendre à haut débit analyses 3,4.
La méthode de TAP a été développé à l'origine dans la levure par des collègues Rigautand 1. Dans ce procédé, une protéine d'intérêt est exprimé avec une étiquette de TAP. La balise TAP abrite deux domaines d'affinité de liaison indépendants: une protéine un domaine qui se lie à l'IgG et un domaine liant la calmoduline. Les deux domaines sont séparés par une TEV (Tobacco Etch Virus) site de clivage. Une telle combinaison permet à deux tours indépendantes de purifications d'affinité pour réduire suffisamment les liaisons non spécifiques et enrichissent fixations spécifiques 1. Pour ce cas, la méthode de TAP est une métho très puissantd pour identifier d'interactions in vivo d'une protéine donnée, bien que la surexpression de la protéine exogène, il peut être plus enclins à associer avec des protéines qui font normalement pas complexe avec son homologue endogène. Depuis son développement, la méthode de TAP a été appliquée dans de nombreux autres systèmes, y compris à base de culture de cellules et d'autres systèmes 5,6 vivo systèmes modèles à 6-9. Nous décrivons ici l'adaptation de la méthode de TAP chez la drosophile. On génère premier pUAST-NTAP et pUAST-CTAP vecteurs pour faciliter le clonage et la fusion de l'étiquette TAP à l'extrémité N-ou C-terminale du gène d'intérêt. Le SAMU-TAP-étiqueté transgène est ensuite exprimé dans le système nerveux sous le contrôle d'un chauffeur de GAL4 neuronale 10. Ensuite, un grand nombre de têtes de mouches adultes sera recueillie, qui ont une teneur élevée en cellules neurales et sont faciles à séparer des autres parties du corps après congélation en fonction des différences de taille. Les têtes d'adultes sont homogénéisés et dégagé bcentrifugations successives y et le surnageant est soumis à une procédure de TAP décrit ci-dessous.
Protocole
Une. Générer SAMU-TAP-étiqueté mouches transgéniques
- Générer pUAST-TAP-étiqueté constructions d'ADN.
- Décider de quel côté (N-ou C-terminale) de la protéine d'appât pour la balise TAP devrait être fusionné à, sur la base de la structure / fonction de la protéine. Voir la discussion pour plus de détails.
- Sous-cloner la région codante de l'ADNc du gène d'intérêt dans les sites de clonage multiple (MCS) des vecteurs pUAST-PAEEN ou pUAST-CTAP pour générer N-ou C-terminal-marquées transgènes UAS-TAP, respectivement. Voir la Figure 1 pour des cartes détaillées et des sites de restriction utilisables et cadres de lecture.
- Générer SAMU-TAP-étiqueté mouches transgéniques.
- Générer des mouches transgéniques suivant des protocoles standard à l'aide de l'élément P médiation insertion 11. Un certain nombre de services d'injection sont disponibles dans le commerce.
- Traverser neuronale pilote de GAL4 (c.-à-BG380-GAL4) pour chaque lignée transgénique individu et déterminer les niveaux d'expression de protéinesde chaque ligne par western blot (avec les anticorps de la peroxydase anti-peroxydase) et / ou immuno-coloration (avec l'anticorps anti-TAP). D'une manière générale, une lignée transgénique avec un niveau d'expression de protéine qui est proche de la protéine endogène est recommandé pour les procédures de la TAP. Voir la discussion pour plus de détails.
- PerformGAL4/UAS-based expériences de récupération de confirmer la fonctionnalité des transgènes TAP-marqué si mutants des gènes d'intérêt avec perte de fonction sont disponibles. Choisissez un transgène qui peut sensiblement sauver les phénotypes mutants pour les expériences de TAP suivants.
2. Préparer les échantillons pour TAP procédure
- Générer une mouche stock qui porte à la fois un pilote neuronale GAL4 (par exemple BG380-Gal4) et le transgène TAP marqués choisi afin de faciliter l'expansion des échantillons de mouches. Recueillir les descendances F1 du pilote GAL4 et la croix de la SAMU transgène dans les rares cas où la combinaison ci-dessus entraîne la survie et la croissance inconvénient.
- Prélever de petits échantillons échelle et d'optimiser l'état de lyse pour solubiliser la protéine TAP-étiqueté.
- Faire une série de tampons de lyse en utilisant une combinaison des détergents non ioniques NP-40 (0,1 à 1%), NADOC (0,1-1%) et du Triton X-100 (0,05 à 0,5%). Voir le tableau 1 et la discussion pour plus d'informations.
- Au sommet d'une garniture de CO2, utilisez # 5 pinces à disséquer 20 chefs d'adultes du transgène exprimant vol TAP et les recueillir dans un tube de 1,5 ml pour tester chaque condition de tampon de lyse.
- Ajouter tests tampon de lyse 100 ul dans le tube et homogénéiser les têtes en caressant de haut en bas avec un pilon en plastique, puis ajouter un autre tampon d'essai de 100 ul.
- Faites tourner la lysat tête à 21 500 g pendant 10 min (4 ° C), et séparer le surnageant et le culot après centrifugation. Ajouter 25 ul de tampon de charge SDS 2x au culot et le tampon de chargement 10 ul 4x SDS à 30 ul de surnageant respectivement.
- Faire bouillir les deux échantillons pendant 5 min et les analyser côteCôté en utilisant SDS-PAGE et Western Blot subséquente avec l'anticorps PAP. Déterminer la solubilité par le rapport entre les niveaux TAP-protéine dans le surnageant par rapport à la pastille.
- Préparer l'échantillon à grande échelle
- Développer et recueillir des mouches adultes.
- Développer le stock neuronal-GAL4/UAS-TAP-transgene en bouteilles et retourner les bouteilles tous les 3 jours jusqu'à cumulés 250 bouteilles sont utilisés pour la collecte.
- Vieux collecter 1-3 jours mouches adultes dans 50 ml tubes coniques, mettre le tube dans de l'azote liquide immédiatement au congélateur les mouches. Conserver les mouches dans un congélateur à -80 ° C. A noter que le volume de vol ne doit pas dépasser 2/3 du tube de 50 ml.
- Recueillir têtes de mouches (effectuer cette étape au-dessus de la glace sèche en poudre).
- Sortez les tamis préalablement refroidie et le mortier et le pilon du congélateur à -80 ° C et les mettre sur la glace sèche, idéalement dans un grand seau à glace. Empilez deux tamis de test standard USA avec un n ° 25 sur ee haut et n ° 40 en bas.
- Prenez vol congelés sur et déposez-les dans de l'azote liquide et de garder les mouches là pendant environ 10 min. Vortex ou secouer les tubes vigoureusement pour briser les têtes, les jambes et les ailes des organes.
- Verser le mélange sur le tamis supérieur, puis secouer les tamis vigoureusement tout en maintenant les deux tamis ensemble. Après tamisage, les corps restent sur le tamis supérieur, voler les têtes seront retenus sur le tamis de fond, et les jambes, ailes et autres débris vont tomber sur la glace sèche. Séparer les deux tamis et soigneusement transférer les têtes de mouches au mortier froid.
- Homogénéiser les têtes de mouches
- En plus de la glace sèche, broyer les têtes avec le mortier et le pilon de particules de poudre, puis transférer la poudre à un Dounce broyeur de tissus de 15 ml en verre qui a été préalablement refroidi sur glace.
- Mesurer le poids de la meuleuse avant et après l'échantillon de la tête a été versé dans, puis calculer combien la samp têtele pèse. Un total de 15.6 grammes de têtes de mouches suffira pour chaque expérience TAP ajustant en conséquence les niveaux de la protéine d'expression. Ajouter 15 ml de tampon d'homogénéisation glacé (tampon de lyse optimisé à l'étape 2.2) à la poudre, puis la course avec le grand pilon en jeu jusqu'à ce qu'il soit facile pour les pilon pour monter et descendre. Gardez le broyeur de verre sur la glace en tout temps.
- Préparer le surnageant pour TAP
- Transférer le broyat dans un tube de centrifugeuse à grande vitesse et un essorage pendant 20 min à ~ 50 000 x g (4 ° C). Transférer le surnageant dans un nouveau tube de centrifugation à grande vitesse et répéter la centrifugation une fois de plus.
- Transférer le surnageant dans un tube d'ultracentrifugation et de réaliser un 40 min ~ 250 000 xg rotation pour dégager davantage le surnageant. Le surnageant est prêt pour les opérations de purification d'affinité en tandem après ultracentrifugation.
- Développer et recueillir des mouches adultes.
3. TAP Purification
Les sections suivantes ont été tirées du laboratoire TAP Séraphin protocol 12 (http://web.as.uky.edu/Biology/faculty/rymond/BIO%20510/Bertran%20Seraphin%27s%20TAP%20page.pdf )
- Effectuer IgG perle purification d'affinité
- Préparer IgG sépharose perles tandis que les échantillons sont centrifugés. Laver 3x 400 ul d'IgG de billes dans un tube Falcon de 15 ml avec 10 ml de tampon de lavage froid IgG. Pour chaque lavage, secouez doucement le tube pendant 2 min, puis centrifuger les perles à 1000 g pendant 2 min. A la fin du troisième lavage, éliminer le tampon et ne laisser que les billes dans le tube.
- Soigneusement transférer le surnageant clarifié (~ 15 ml) dans le tube de 15 ml contenant les billes IgG. Incuber les billes et le cerveau lysat mélange à 4 ° C sur une nutator pendant 2 heures.
- Mettre en place un micro colonne vide et propre avec environ 15 ml de volume total en chambre froide. Chargez le mélange de perles IgG en versant régulièrement le mélange dans la colonne; essayer de ne pas piéger toutles bulles d'air à l'intérieur de la colonne. Laisser les perles de s'installer dans la colonne et le tampon de drainer lentement par gravité.
- Laver la colonne à fond avec 10 ml de tampon de lavage froid IgG après tout du lysat de cerveau s'est écoulé à travers la colonne d'IgG réglée. Répétez l'2x de lavage. Remarque: ne laissez jamais le cordon sécher à l'air.
- Effectuer TEV clivage
- Après le troisième lavage, laver de nouveau la colonne avec 10 ml de tampon de clivage TEV. Cette étape prépare le cordon IgG que sequestrates le complexe d'appât pour TEV clivage.
- Juste avant la dernière goutte de tampon TEV est sur le point de s'écouler, mettre un bouchon au bas de la colonne de bloquer la circulation, ajouter 1,3 ml de tampon de TEV contenant 130 unités de TEV enzyme de la colonne, et puis boucher solidement le haut de la colonne. Assurez-vous que la colonne est bien scellé aux deux extrémités.
- Tournez la colonne à 18 ° C pendant 2 heures pour permettre à l'enzyme TEV pour cliver le peptide sur le site de TEV et libérer la protéine WHI complexele laissant derrière la protéine A peptide de domaine lié aux perles de l'IgG.
- Effectuer calmoduline perle purification d'affinité
- Préparer les billes calmoduline tandis que les billes sont incubées avec des IgG de l'enzyme TEV. Lavez 200 pi perles calmoduline dans un 15 ml Falcon 3x tube, chaque fois avec 10 ml de calmoduline froid de tampon de liaison. Pour chaque lavage, secouez légèrement le tube pendant 2 minutes sur un nutator, puis centrifuger les perles à 1000 g pendant 2 min. A la fin du troisième lavage, retirer toute la mémoire tampon et ne laisser que les perles dans le tube.
- A la fin de l'incubation TEV (étape 3.2.3), retourner la colonne IgG retour à la chambre froide et mettez-le vers le haut. Laissez les perles se contenter de 10 min.
- Retirez le couvercle supérieur et le couvercle inférieur, puis recueillir le produit de clivage de 1,3 ml de TEV dans un tube Falcon de 15 ml. Laissez le tampon vider complètement. Ajouter un tampon de 200 TEV ul supplémentaire à la colonne de pousser le volume mort de la colonne, recueillir les ffaible dans le même tube.
- Ajouter 4,5 ml de calmoduline et 4,5 ul de tampon de 1 M de CaCl2 de liaison au produit de clivage de 1,5 ml TEV recueillis ci-dessus. Le CaCl 2 sert à titrer l'EDTA dans le tampon TEV. Transférer le mélange dans 6 ml au tube contenant les billes calmoduline et faire tourner le tube à 4 ° C sur une nutator pendant 1 heure.
- Mettre en place un autre micro colonne vide et propre avec environ 10 ml de volume total en chambre froide. Chargez le mélange de perles calmoduline à la colonne et lui permettre de s'écouler par gravité.
- Lorsque toute la solution a traversé la colonne calmoduline constante, laver la colonne deux fois, chacune avec 10 ml de calmoduline froid de tampon de liaison. Remarque: éviter de déranger les billes calmoduline et essayer de garder la surface des perles plates que possible pendant le lavage.
- Éluer le complexe de l'appât de la colonne calmoduline.
Juste après le lavage, l'élution de la colonne calmoduline avec cinq fractions de 200 ul Calmodu froidlin tampon d'élution. Pour chaque fraction, ajouter doucement 200 ul de tampon d'élution de la colonne et recueillir l'éluat avec un marqué 1.5 ml tube Eppendorf. Répétez cette 4x. - Analyser le complexe de protéines par SDS-PAGE,
- Prenez une petite aliquote de chacune des cinq fractions (environ 30 pi) et ajouter un tampon de charge SDS. Faire bouillir les échantillons pendant 5 min et charger les échantillons côte-à-côte avec des marqueurs moléculaires de protéines dans un gradient (4-15%) sur gel de SDS-PAGE.
- Après les échantillons ont entièrement résolu dans le gel, arrêter le électrophorèse et tacher le gel avec toutes les procédures sensibles base-G-250 colloïdales Coomassie comme «argent bleu 'coloration 13. Coloration à l'argent est facultative, mais pas préférable car il n'est pas compatible avec l'analyse par spectrométrie de masse subséquente. Stocker le reste de l'éluat dans un congélateur C -80 ° pour une analyse ultérieure telle que la spectrométrie de masse pour découvrir les identités moléculaires du complexe de protéine purifiée. Sediscussion de e.
Résultats
Ici, nous démontrons notre effort pour identifier les protéines Highwire-qui interagissent dans le cerveau de la mouche. Highwire (Hiw) et ses homologues vertébrés et d'invertébrés sont énormes ubiquitine ligases qui régulent le développement et la réparation du système nerveux 14. Ils partagent un certain nombre de domaines fonctionnels hautement conservées. Cependant, leurs actions moléculaires ne sont pas tout à fait clair. Les travaux effectués dans le ver, la mouche et de la souris a conduit au modèle de travail courant qui fonctionne Hiw comme une ligase E3 et en tant que protéine d'échafaudage pour faciliter la formation d'un complexe d'ubiquitination sous-unités multiples, qui régule les fonctions neuronales spécifiques de type dans le temps et de cellule à travers le combinaison d'interagir avec différents cofacteurs et ciblant différents substrats de l'ubiquitine. Pour identifier le complexe d'ubiquitination Hiw associée, nous avons généré un premier marqué transgène N-terminal UAS-TAP-Hiw qui est entièrement fonctionnelle dans le sauvetage du phénotype mutant hiw 15. Environ 10 g de fl adultey têtes qui expriment uniquement ou TAP TAP-protéines transgéniques DID ont été recueillies et soumises à des procédures de TAP côte à côte comme décrit ci-dessus. Éluats provenant des deux purifications finales ont été analysés par SDS-PAGE, puis coloration à l'argent. La spectrométrie de masse a identifié une liste de protéines dans l'échantillon TAP-Hiw seulement, y compris chez la drosophile FSN (DFSN, une protéine F-box) et Rae1 (Figure 2). Analyses génétiques et biochimiques ultérieures ont révélé que Hiw et DFSN travaillent ensemble comme SCF-comme complexe ubiquitine ligase E3 pour réguler la structure et la fonction synaptique 16, et associés Rae1 avec Hiw in vivo et réprime ses prolifération synaptique 17. Cette fonction de Rae1 est au moins partiellement atteint par sa capacité à promouvoir la stabilité des protéines Hiw via Hiw protéger de la dégradation autophagic, ce qui révèle un nouveau mécanisme qui contrôle sélectivement Hiw abondance de la protéine synaptique au cours du développement 17.
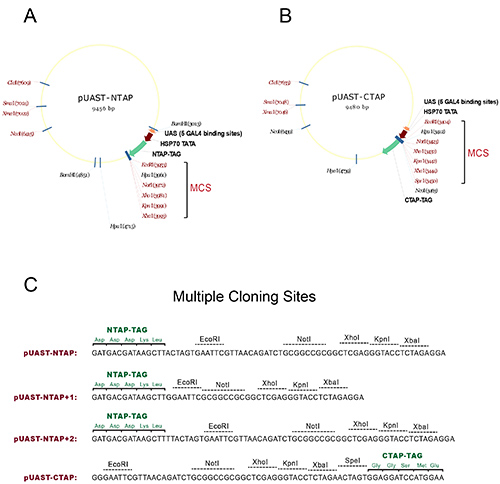
Figure 1. Vecteurs pour générer des mouches transgéniques TAP marqué. (A) La carte de la pUAST-PAEEN construire. (B) La carte de la pUAST-CTAP construire. Les sites de clonage multiple (MCS) dans les deux constructions sont marqués avec un support, où les sites de restriction unique coupées sont présentées par la couleur rouge. (C) la séquence primaire montrant les sites de clonage multiples des vecteurs de robinet avec le cadre de lecture de l'étiquette TAP. Pour faciliter le sous-clonage, deux autres constructions PAEEN sont générés à partir de l'original pUAST-NTAP: pUAST-PAEEN 1 et pUAST-PAEEN 2. Ensemble, les trois PAEEN construit couvre les trois cadres de lecture possibles pour permettre l'utilisation de la MCS. Tous les vecteurs sont construits en utilisant la NTAP ou fragments CTAP originairement réalisé surSéraphin Lab 1. Cliquez ici pour agrandir l'image.

. Figure 2: Purification de Highwire-interaction des protéines de volée cerveau par purification TAP têtes d'adultes de mouches exprimant TAP. (BG380-GAL4; SAMU-TAP) ou TAP-Hiw (BG380-GAL4; SAMU-TAP-Hiw) dans le système nerveux sont rassemblés, homogénéisés et soumis à une purification de TAP, respectivement. Les éluats finaux ont été analysés par SDS-PAGE sur gel à une dimension, suivie par coloration à l'argent. La tête de flèche indique l'appât protéine Hiw et l'astérisque indique une protéine TAP tronquée en raison de TeV clivage. Dans l'échantillon TAP-Hiw, une liste de protéines sont identifiées en massespectrométrie, y compris HSC-70, β-tubuline, Rae1 et DFSN (indiqué par les flèches). Ce chiffre est modifié à partir de la figure 1a de Tian et al. 17 Cliquez ici pour agrandir l'image.
| Tampon | Composition | Commentaires |
| tampon de lyse | 50 mM Tris-HCl pH 7,5 | REMARQUE: 1) Ajouter la TNT et les inhibiteurs de la protéase et protéasome juste avant utilisation. |
| 125 mM NaCl | 2) Montrer ici est un tampon de lyse avec 0,5% de NP40. Voir Discussion des modifications sur les détergents non ioniques. | |
| 5% Glycérine | ||
| 0,5% NP40 | ||
| 1,5 mM de MgCl2 | ||
| MM NaF 25 | ||
| 0,2 mM de DTT | ||
| (Ce qui suit sont des inhibiteurs de la protéase et protéasome) | ||
| 1 mM Na 3 VO 4 | ||
| 0,05 mM MG-115 | ||
| 1 mM de PMSF | ||
| Inhibiteur de la protéase mélange (Sigma P8340) | ||
| Inhibiteur de la protéase cocktail (Roche 04693159001) | ||
| Tampon de lavage IgG | 10 mM de Tris-Cl pH 8,0 (0,5 ml de 2 m stock) | |
| NaCl 150 mM (3 ml 5 m stock) | ||
| 0,1% de NP40 (1,0 ml de 10% stock) | ||
| H 2 0 à 100 ml final | ||
| Tampon de clivage TEV | 10 mM de Tris-Cl pH 8,0 (0,5 ml de 2 m stock) | REMARQUE: ajouter la TNT juste avant utilisation. |
| NaCl 150 mM (3 ml 5 m stock) | ||
| 0,1% de NP40 (1,0 ml de 10% stock) | ||
| EDTA 0,5 mM (100 ul de 0,5 M actions) | ||
| 1 mM de DTT (100 pi de 1 M stocks) | ||
| H 2 O et 100 ml final | ||
| un tampon de liaison de la calmoduline | 10 mM β-mercaptoéthanol (69,7 pi d'actions) | |
| 10 mM de Tris-Cl pH 8,0 (0,5 ml de 2 m stock) | ||
| NaCl 150 mM (3 ml 5 m stock) | ||
| 1 mM de Mg-acétate (100 pi de 1 M stocks) | ||
| 1 mM d'imidazole (100 pi de 1 M stocks) | ||
| 2 mM de CaCl2 (200 pi de 1 M stocks) | ||
| 0,1% de NP40 (1 ml de 10% stock) | ||
| H 2 O et 100 ml final | ||
| Tampon d'élution calmoduline | 10 mM β-mercaptoéthanol (69,7 pi d'actions) | |
| 10 mM de Tris-Cl pH 8,0 (0,5 ml de 2 m stock) | ||
| NaCl 150 mM (3 ml 5 m stock) | ||
| 1 mM de Mg-acétate (100 pi de 1 M stocks) | ||
| 1 mM d'imidazole (100 pi de 1 M stocks) | ||
| 10 mM d'EGTA (2 ml de 0,5 M actions) | ||
| 0,1% de NP40 (1 ml de 10% stock) | ||
| H 2 O et 100 ml final |
Tableau 1. Composition des tampons.
Discussion
Procédé de purification par affinité en tandem (TAP) propose un protocole de double purification permettant l'isolement et l'enrichissement des complexes de protéines à travers deux étapes indépendantes de purification d'affinité. La conception de l'étiquette TAP n'est pas limitée à ce qui est présenté dans ce protocole, d'autres domaines et motifs de liaison de protéines sont également applicables si les conditions tampons sont ajustés en conséquence. Un bon exemple d'autres balises TAP est la balise GS-TAP, une combinaison d'une protéine G et un motif de liaison à la streptavidine, conçu par le groupe de Giulio Superti-Furga visant à complexe protéique de purification dans des lignées cellulaires de mammifères en culture 18. Le GS-TAP a ensuite été adapté pour étudier l'interaction protéine-protéine dans les cellules et d'embryons de drosophile S2 19. Une récente publication JoVE par Bailey et al. Démontré comment la procédure GS-TAP a été réalisée avec la culture de lysat cellulaire 20. Ici, nous avons présenté l'utilisation de TAP-tag dans les tissus neuronaux chez la drosophile (adtêtes ult) pour le dépistage protéomique à grande échelle. Ce protocole peut être potentiellement adaptée à d'autres tissus qui permettent la collecte d'échantillons à grande échelle, tels que des embryons. Les organes adultes recueillies sur le tamis supérieur (étape 2.3.2.3) peuvent également être utilisés pour TAP, qui peut être une tâche très difficile. Le tampon de lyse doit être modifié et conditionné pour inhiber les protéases massives présentes dans le tractus digestif au maximum et la première opération de purification sera sensiblement raccourci afin de réduire le temps d'exposition des protéines pour les proteases. Bien que les étiquettes de TAP nécessitent des manipulations et des conditions de tampon pour les purifications d'affinité, les principes généraux de la TAP sont les mêmes. Ci-dessous, nous discutons de questions qui auront une incidence sur la qualité des résultats de la TAP.
Le succès de l'approche de TAP dépend de la génération de la droite transgène. Tout d'abord, la balise elle-même TAP doit pas modifier la conformation globale de la protéine appât, ce qui est essentiel pour conserver son aptituded'interagir avec ses partenaires de liaison physiologiques. Ainsi, il est essentiel de décider où faire fondre l'étiquette TAP pour le gène d'intérêt. La décision peut être prise sur la base des études antérieures de la protéine, en particulier des informations sur sa structure ou de transgènes epitode marqués fonctionnels. Si une telle information n'est pas disponible, le marquage de la protéine appât à N-ou C-terminale en parallèle est recommandée. Ensuite, les expériences de sauvetage fonctionnels peuvent être effectuées pour déterminer quelle transgène doit être utilisé. Deuxièmement, les niveaux de la protéine transgénique d'expression devrait être proche de celle de la protéine endogène. Ceci est particulièrement important que le système d'entraînement GAL4/UAS normalement l'expression du transgène à un instant différent et à un niveau beaucoup plus élevé par rapport aux protéines endogènes, qui peuvent soit augmenter les faux positifs ou de provoquer des conséquences inattendues qui altèrent le profil de l'interaction de la protéine les réseaux dans les cellules. Par exemple, les protéines d'échafaudage peuvent surexprimant Cause effets négatifs dominants et des protéines surexprimant qui possèdent des activités enzymatiques, comme les kinases, il arrive souvent entraîner un gain de fonction effets, qui peuvent à leur tour affecter la capacité de leurs partenaires de liaison endogènes d'interagir avec eux. Ainsi, il convient d'éviter au système de GAL4/UAS lorsque le calage et le niveau de l'expression du gène d'intérêt est essentielle pour la préservation des interactions endogènes qui présentent seulement dans des conditions normales. Au lieu de cela, l'expression du transgène de TAP-étiqueté doit être contrôlée en vertu de son propre promoteur. Ceci peut être réalisé soit par le remplacement de la séquence UAS avec la séquence de promoteur endogène, ou en fusionnant la séquence d'TAP dans le cadre avec la séquence exonal dans un fragment d'ADN génomique qui contient la totalité du locus du gène d'intérêt 21. Dans certains cas, l'expérience de TAP peut être effectuée dans une perte de la fonction de fond mutant du gène d'intérêt à réduire la compétition de la protéine endogène pour incorporating dans des complexes multiprotéiques. Pour ce cas, une protéine fond null, le cas échéant, fournit les conditions idéales pour éliminer complètement la protéine endogène. Utilisation du fond mutant génétique sera toujours augmenter les chances de produire des résultats de qualité supérieure.
Solubilité de la protéine appât est un autre facteur crucial pour le succès. Les détergents non ioniques sont normalement utilisés dans le tampon de lyse pour dissoudre la membrane plasmique et de retenir le complexe protéique, soluble et intact, dans un état qui est proche de l'environnement physiologique. Toutefois en fonction de si oui ou non les protéines d'intérêt sont des protéines membranaires ou associées intégré, la composition et la concentration de détergents non-ioniques peuvent être cruciale pour la solubilisation de la protéine appât. 0,1 à 0,3% de NP40 est souvent suffisante pour solubiliser les protéines cytosoliques. Pour les protéines de la membrane, une combinaison de NP40 (0,1-1%), NADOC (0,1-1%), et du Triton X-100 (0,05 à 0,5%) est parfois nécessaire de solubiliséliser et d'enrichir suffisamment de protéines pour l'expérience de TAP. Toutefois, étant donné un tampon de lyse strictes ont tendance à perturber les interactions protéine-protéine les plus faibles, un équilibre doit être atteint dans lequel protéines appâts sont suffisamment solubles présents dans l'échantillon tandis que dans le même temps la plupart des interactions protéine-protéine sont conservés. Un principe général est que toujours essayer d'utiliser le moins de types de détergents avec la concentration la plus basse.
Pour faciliter le dépannage, il est fortement recommandé de sauvegarder une petite aliquote de l'échantillon de chaque étape des deux purifications. Subséquentes SDS-PAGE et coloration des protéines / analyse Western sur les aliquotes permettre aux niveaux de la protéine appât et l'enrichissement séquentiel de certaines espèces de protéines à surveiller. Par exemple, si une réduction soudaine de la protéine appât a été détectée dans la fraction aliquote de l'écoulement après le clivage TEV (étape 3.3.3), il est probable que la protéine appât ont été perdus au cours de l'IgG bourrelet lavage (étape 3.1.4) . Un possible pourrait être la cause de la composition détergente insuffisante du tampon de lavage IgG. Il existe une forte baisse de la composition et la concentration des produits de lavage dans le tampon de lavage d'IgG (0,1% de NP-40 seulement) par rapport au tampon de lyse. La protéine appât peut être sensible à ces changements et de dissocier la fraction IgG sur les billes. Réglage de la lave IgG vers un tampon de lyse peut aider à résoudre le problème. Sinon, bien que moins probable, le clivage TEV (étape 3.2) peut ne pas avoir travaillé suffisamment pour libérer le complexe contenant l'appât des billes IgG. Les étapes d'une valeur d'une seconde inspection sont l'équilibrage de billes d'IgG avec le tampon TEV (étape 3.2.1) et l'activité enzymatique, une mauvaise manipulation peut amortir l'enzyme.
Gardez à l'esprit que, même avec la purification en deux étapes, il y aura toujours des faux positifs et des protéines qui sont tirés vers le bas de manière non spécifique présent dans le complexe TAP purifié finale. Une façon d'identifier les interactions non spécifiques, àmoins en partie, est d'inclure un TAP de commande en utilisant le TAP-transgène seulement en parallèle avec TAP-tagged-transgène dans les expériences de purification de TAP. Les protéines identifiées dans la procédure de TAP-ne doivent être retirées de la liste identifiée dans la purification TAP-transgène.
Un TAP retenu purifier un mélange de protéines qui sont potentiellement associés à la protéine d'intérêt in vivo. L'étape suivante est l'identification moléculaire de ces protéines, et la spectrométrie de masse est couramment utilisé à cette fin. Selon les résultats de la SDS-PAGE et les besoins de l'expérience, les identités moléculaires dans le complexe de protéine purifiée peuvent être déterminées dans les deux façons suivantes: 1) chaque bande de protéine individuelle peut être coupé à partir du gel et soumis à une faible LC-MS/MS complexité, ou 2) la fraction de l'éluat qui contient la teneur en protéines de crête (normalement la seconde élution) peut être directement soumis à une complexité modérée LC-MS/MS. Un tel fusil papproche roteomic sera potentiellement identifier toutes les protéines dans le complexe, en fonction de la plage dynamique de l'équipement de MS 22.
En résumé, nous présentons une méthode pour identifier les interactions protéine-protéine dans les tissus neuronaux d'un organisme modèle génétique - la mouche des fruits. Il ya un certain nombre d'avantages de l'application de la méthode de TAP à la volée: 1) les mouches des fruits ont un cycle de vie court, il est donc relativement rapide et facile de générer des mouches transgéniques et à obtenir des tissus en grande quantité, les deux sont essentiels pour la TAP approche; 2) le génome de la mouche est entièrement annoté et contient 70% de gènes responsables de maladies humaines, et 3) le plus important, il est facile de valider fonctionnellement et caractériser le candidat protéines interagissant en vol. Il existe des ressources globales disponibles dans la communauté de la recherche à la mouche pour la caractérisation d'un gène donné, telles que la collecte de l'ARNi basée transgénique-utile d'étudier tissu-spécifique de perte de fonction de la majorité du gènes, allèles mutants et une carence de la plupart des loci de gènes, ainsi que des clones et des transgènes utiles pour des études de gain de fonction des ADNc. Nous croyons que la méthode mouche TAP sera un ajout précieux à la boîte à outils utilisée dans les laboratoires de mouches. En perspective avenir, cette méthode peut être combinée à la mouche paradigmes comportementaux et des modèles de maladies humaines, pour cribler le changement de réseau d'interactions protéine-protéine en réponse à des stimuli et des conditions spécifiques.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Nous remercions EUROSARF de nous envoyer la levure TAP plasmides d'expression. Nous sommes également reconnaissants de l'aide éditoriale de Ryan Labadens. Ce travail a été soutenu par une subvention du NIH / NINDS (R01NS070962) à CW
matériels
| Name | Company | Catalog Number | Comments |
| U.S.A. standard test sieve No. 25 | Fisher Scientific | 04-881-18 | |
| U.S.A. standard test sieve No. 40 | Fisher Scientific | 04-881-21 | |
| Kontes Dounce Tissue Grinders 15 ml | Kimble Chase | 885300-0015 | |
| IgG sepharose beads | Pharmacia | 17-0969-01 | |
| Econo-column 0.7 cm x 20 cm | Bio-Rad | 737-4721 | |
| Econo-column 0.5 cm x 15 cm | Bio-Rad | 737-4716 | |
| Calmodulin beads | Stratagene | 214303 | |
| Coors Mortar and Pestle | CoorsTek | 60311 | |
| AcTEV Protease | Invitrogen | 12575-015 | |
| Protease Inhibitor Cocktail | Roche | 11836153001 | |
| Protease Inhibitor Mix | Sigma | P8340 |
Références
- Rigaut, G., et al. A generic protein purification method for protein complex characterization and proteome exploration. Nat. Biotechnol. 17, 1030-1032 (1999).
- Collins, S. R., et al. Toward a comprehensive atlas of the physical interactome of Saccharomyces cerevisiae. Mol. Cell. Proteomics. 6, 439-450 (1074).
- Gavin, A. C., et al. Proteome survey reveals modularity of the yeast cell machinery. Nature. 440, 631-636 (2006).
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440, 637-643 (2006).
- Forler, D., et al. An efficient protein complex purification method for functional proteomics in higher eukaryotes. Nat. Biotechnol. 21, 89-92 (2003).
- Veraksa, A., Bauer, A., Artavanis-Tsakonas, S. Analyzing protein complexes in Drosophila with tandem affinity purification-mass spectrometry. Dev. Dyn. 232, 827-834 (2005).
- Li, Y. The tandem affinity purification technology: an overview. Biotechnol. Lett. 33, 1487-1499 (2011).
- Volkel, P., Le Faou, P., Angrand, P. O. Interaction proteomics: characterization of protein complexes using tandem affinity purification-mass spectrometry. Biochem. Soc. Trans. 38, 883-887 (2010).
- Xu, X., et al. The tandem affinity purification method: an efficient system for protein complex purification and protein interaction identification. Protein Expr. Purif. 72, 149-156 (2010).
- Brand, A. H., Perrimon, N. Targeted gene expression as a means of altering cell fates and generating dominant phenotypes. Development. 118, 401-415 (1993).
- Bachmann, A., Knust, E. The use of P-element transposons to generate transgenic flies. Methods Mol. Biol. 420, 61-77 (2008).
- Puig, O., et al. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Candiano, G., et al. Blue silver: a very sensitive colloidal Coomassie G-250 staining for proteome analysis. Electrophoresis. 25, 1327-1333 (2004).
- Tian, X., Wu, C. The role of ubiquitin-mediated pathways in regulating synaptic development, axonal degeneration and regeneration: insights from fly and worm. J. Physiol. , (2013).
- Wu, C., Wairkar, Y. P., Collins, C. A., DiAntonio, A. Highwire function at the Drosophila neuromuscular junction: spatial, structural, and temporal requirements. J. Neurosci. 25, 9557-9566 (2005).
- Wu, C., Daniels, R. W., DiAntonio, A. DFsn collaborates with Highwire to down-regulate the Wallenda/DLK kinase and restrain synaptic terminal growth. Neural Dev. 2, 16 (2007).
- Tian, X., Li, J., Valakh, V., DiAntonio, A., Wu, C. Drosophila Rae1 controls the abundance of the ubiquitin ligase Highwire in post-mitotic neurons. Nat. Neurosci. 14, 1267-1275 (2011).
- Burckstummer, T., et al. An efficient tandem affinity purification procedure for interaction proteomics in mammalian cells. Nat. Methods. 3, 1013-1019 (2006).
- Kyriakakis, P., Tipping, M., Abed, L., Veraksa, A. Tandem affinity purification in Drosophila: the advantages of the GS-TAP system. Fly. 2, 229-235 (2008).
- Bailey, D., Urena, L., Thorne, L., Goodfellow, I. Identification of protein interacting partners using tandem affinity purification. J. Vis. Exp. (60), e3643 (2012).
- Wu, Y., et al. A Drosophila model for Angelman syndrome. Proc. Natl. Acad. Sci. U.S.A. 105, 12399-12404 (2008).
- Liao, L., McClatchy, D. B., Yates, J. R. Shotgun proteomics in neuroscience. Neuron. 63, 12-26 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon