Method Article
Identifizierung von Protein-Protein-Wechselwirkung in
In diesem Artikel
Zusammenfassung
Drosophila ist berühmt für seine leistungsstarken genetischen Manipulation, aber nicht für die Eignung der eingehenden biochemischen Analyse. Hier präsentieren wir eine TAP-basierte Verfahren, um Interaktionspartner eines Proteins von Interesse aus dem Fliegenhirn zu identifizieren. Dieser Vorgang kann möglicherweise zu neue Wege der Forschung führen.
Zusammenfassung
Genetische Screens durchgeführt unter Verwendung von Drosophila melanogaster (Fruchtfliege) haben zahlreiche Meilenstein Entdeckungen im Vorfeld der biologischen Wissenschaften gemacht. Jedoch wurde die Verwendung von biochemischen Screening auf die sich die Kenntnis der genetischen Analyse gewonnenen Ziel erst kürzlich entdeckt. Hier beschreiben wir eine Methode, um den Proteinkomplex zu reinigen, der sich an jedem Protein von Interesse von erwachsenen Fliegenköpfe. Diese Methode nutzt die Drosophila GAL4/UAS System, um eine Köderprotein mit einem Tandem Affinity Purification (TAP)-Tag in fly Neuronen in vivo verschmolzen Ausdruck zu bringen, und dann implementiert zwei Runden der Reinigung mit einem TAP-Verfahren, ähnlich dem ursprünglich fest Hefe ein, um die Interaktion Protein-Komplex zu reinigen. Am Ende dieses Verfahrens wird ein Gemisch aus mehreren Protein-Komplexe erhalten wird, dessen Molekular Identitäten durch Massenspektrometrie bestimmt werden. Validierung der Kandidatenproteine aus der r profitierenesource und einfache Durchführung loss-of-function-Studien in Fliegen. Ähnliche Ansätze können auf andere Flug Gewebe angewendet werden. Wir glauben, dass die Kombination von genetischen Manipulationen und proteomischen Ansatz dieser in der Flugmodellsystem birgt ein enormes Potenzial für die Bekämpfung der grundlegenden Probleme im Bereich der Neurobiologie und darüber hinaus.
Einleitung
Definieren der molekularen Signalwege und Netzwerke, die einen bestimmten biologischen Prozess zu vermitteln ist eines der obersten Ziele der biomedizinischen Forschung. Fly Genetiker haben massiv auf zukunfts Genetik abhängig, vor allem Modifikator genetischen Bildschirme (beide Enhancer und Suppressor-Screens), um Faktoren, die zusammenarbeiten, parallel, oder stromaufwärts oder stromabwärts eines Gens von Interesse zu identifizieren. , Vorwärts Genetik Bildschirme oft scheitern jedoch zu wesentlichen Gene zu identifizieren, die, wenn mutiert, verursachen Letalität in frühen Entwicklungsstadien, oder Gene mit funktionellen Redundanz und Entschädigung, deren Funktionsverlust nur subtile Defekte, die schwer zu erzielen sind, verursachen. Eine Möglichkeit, diese Schwierigkeit zu überwinden, ist für die direkte Protein-Protein-Interaktionen zu screenen. Seit mehr als einem Jahrzehnt eine wachsende Liste von biochemischen Methoden, einschließlich Hefe-Zwei-Hybrid-, Phagen-Display, chemische Vernetzung, Co-IP, Tandem Affinity Purification (TAP), usw. wurden verwendet, um Protein zu untersuchen-Protein-Wechselwirkungen. Jeder dieser Ansätze hat ihre eigenen Stärken und Schwächen im Hinblick auf die Empfindlichkeit und Spezifität. Unter ihnen kann der TAP-Verfahren zum Nachweis von physikalischen Wechselwirkung unter nahezu physiologischen Bedingungen bewahrt Spezifität und Konsistenz 2 und schließt die Fähigkeit zum Hochdurchsatz-Analyse 3,4 erstrecken.
Die TAP-Methode wurde ursprünglich in der Hefe durch Rigautand Kollegen 1 entwickelt. In diesem Verfahren wird ein Protein von Interesse mit einem TAP-Tag exprimiert. Der TAP-Tag birgt zwei unabhängige Affinität bindende Domänen: eine Protein A-Domäne, die an IgG bindet und eine Calmodulin-Bindungsdomäne. Die beiden Bereiche sind durch eine TEV (Tabak-Etch-Virus)-Spaltstelle getrennt sind. Eine solche Kombination ermöglicht zwei unabhängige Runden der Affinitätsreinigungen ausreichend unspezifische Bindungen zu reduzieren und bereichern spezifischen Bindungen ein. Für diesen Fall ist die TAP-Methode ein sehr leistungsfähiges method in vivo Interaktionen eines bestimmten Proteins zu identifizieren, obwohl Überexpression des exogenen Proteins kann es die sich bei Proteinen, die normalerweise nicht mit den komplexen endogenen Gegen Verbindung zu bringen. Seit seiner Entwicklung hat die TAP-Methode in vielen anderen Systemen, einschließlich Zellkultur-basierten Systemen 5,6 und in vivo Modellsystemen 9.6 angewendet. Hier beschreiben wir die Anpassung der TAP-Methode in Drosophila. Zunächst erzeugen pUAST NTAP-und pUAST CTAP-Vektoren Klonierung und Fusion der TAP-Tag entweder am N-oder C-Terminus des Gens von Interesse zu erleichtern. Die FH-TAP-markierten Transgen wird dann im Nervensystem unter der Kontrolle eines neuronalen GAL4 Treiber 10 ausgedrückt. Als nächstes wird eine große Anzahl von erwachsenen Fliege Köpfe erfasst werden, die hohen Gehalt an Nervenzellen und sind leicht von anderen Körperteilen nach dem Einfrieren auf Größe Differenzen zu trennen. Die erwachsenen Köpfe sind homogenisiert und gelöscht by aufeinanderfolgenden Zentrifugationen und der Überstand wird unter einer unten beschriebenen Prozedur TAP.
Protokoll
1. Generieren UAS-TAP-markierten Transgene Fliegen
- Generieren pUAST-TAP-markierten DNA-Konstrukte.
- Entscheiden Sie, welche Seite (N-oder C-Terminus) der Köder-Protein die TAP-Tag sollte die fusioniert werden sollen, aufgrund der Struktur / Funktion des Proteins. Siehe Diskussion für weitere Details.
- Subklonierung der cDNA-codierenden Region des Gens von Interesse in die multiple Klonierungsstellen (MCS) der pUAST NTAP-oder pUAST CTAP-Vektoren, um N-oder C-terminalen-markierten UAS-TAP-Transgene zu erzeugen sind. Siehe Abbildung 1 für detaillierte Karten und nutzbar Restriktionsstellen und Leserahmen.
- Generieren UAS-TAP-markierten transgenen Fliegen.
- Generieren transgenen Fliegen nach Standardprotokollen mit P-Element-vermittelte Insertion 11. Eine Reihe von Einspritzleistungen sind im Handel erhältlich.
- Überqueren neuronalen GAL4 Treiber (BG380-GAL4) zu jedem einzelnen transgenen Linie und bestimmen die Proteinexpressionsniveausjeder Zeile durch Western-Blot (mit Peroxidase-Anti-Peroxidase-Antikörper) und / oder die Immunfärbung (mit Anti-TAP-Antikörper). In der Regel wird eine transgene Linie mit einer Proteinexpression Wert in der Nähe des endogenen Proteins ist für TAP Verfahren empfohlen. Siehe Diskussion für weitere Details.
- PerformGAL4/UAS-based Rettungsversuche, um die Funktionalität der TAP-markierten Transgene bestätigen, wenn loss-of-function Mutanten der Gene von Interesse sind. Wählen Sie ein Transgen, die die mutierten Phänotypen für die folgenden Experimente wesentlich TAP retten können.
2. Bereiten Proben für die TAP-Verfahren
- Generieren Sie eine Fliege stock, die sowohl eine neuronale GAL4 Treiber (zB BG380-Gal4) und der gewählten TAP-markierten Transgen um Ausbau der Fliege trägt Proben erleichtern. Sammeln Sie die F1 Nachkommen des GAL4-Treiber und dem UAS-Transgen Kreuz in seltenen Fällen, wenn die oben genannte Kombination bewirkt, dass das Überleben und Wachstum Nachteil.
- Sammeln kleinen Proben und optimieren Lyse Bedingung für die Solubilisierung des TAP-markierten Protein.
- Stellen eine Reihe von Lysepuffer mit einer Kombination der nichtionischen Detergenzien NP-40 (0,1-1%), NaDOC (0,1-1%) und Triton X-100 (0,05-0,5%). Siehe Tabelle 1 und Diskussion für weitere Informationen.
- Auf einem CO2-Pad, verwenden Nr. 5 Pinzette aus der TAP-Transgen exprimiert Fliegen sezieren 20 Erwachsenen Köpfe und sammeln sie in einem 1,5-ml-Tube, um jeden Lysepuffer Zustand zu testen.
- Geben Sie 100 ul Lysepuffer Prüfung an das Rohr und homogenisieren die Köpfe durch Streicheln auf und ab mit einer Kunststoff-Stößel, fügen Sie dann weitere 100 ul Testpuffer.
- Drehen Sie den Kopf Lysat bei 21.500 g für 10 min (4 ° C), und trennen Sie den Überstand und Pellet nach Zentrifugation. In 25 ul 2x SDS-Ladepuffer zum Pellet und 10 ul 4x SDS-Ladepuffer zu 30 ul des Überstandes auf.
- Man kocht die beiden Proben für 5 min und zu analysieren nebenSeitige Verwendung von SDS-PAGE und anschließendem Western-Blot mit dem PAP-Antikörper. Bestimmung der Löslichkeit durch das Verhältnis der TAP-Proteinspiegel im Überstand der Pellet-vs.
- Bereiten Probe in großem Maßstab
- Erweitern und sammeln erwachsenen Fliegen.
- Erweitern Sie den neuronal-GAL4/UAS-TAP-transgene Lager in Flaschen und drehen Sie die Flaschen alle 3 Tage bis kumulative 250 Flaschen werden für die Sammlung verwendet.
- Sammeln 1-3 Tage alten Erwachsenen in 50 ml konische Röhrchen fliegt, legte das Rohr sofort in flüssigem Stickstoff tiefgefroren, um die Fliegen. Bewahren Sie die Fliegen in einem -80 ° C Gefrierschrank. Beachten Sie, dass das Volumen der Fliegen muß 2/3 der 50-ml-Röhrchen nicht überschreiten.
- Sammeln Fliegenköpfe (führen Sie diesen Schritt auf der Oberseite des pulverförmigen Trockeneis).
- Nehmen Sie die vorgekühlten Siebe und der Mörser und Stößel aus dem -80 ° C Gefrierschrank und legte sie auf Trockeneis, idealerweise in einem großen Eiskübel. Stapeln beiden USA-Standardtestsiebe mit Nr. 25 auf the oben und Nr. 40 an der Unterseite.
- Nehmen Sie die gefrorenen Fliegen aus und legen Sie sie in flüssigem Stickstoff und halten die Fliegen dort für ca. 10 min. Vortex oder kräftig schütteln, die Rohre, um die Köpfe, Beine und Flügel aus dem Körper zu brechen.
- Gießen Sie die Mischung auf das obere Sieb, und dann schütteln die Siebe kräftig, während die beiden Siebe zusammenhält. Nach dem Sieben werden die Körper auf dem oberen Sieb bleiben, fliegen Köpfe werden auf der Unterseite Sieb zurückgehalten werden, und die Beine, Flügel und andere Verunreinigungen werden dem Trockeneis fallen. Trennen Sie die beiden Siebe und sorgfältig übertragen Sie die Fliege Köpfe der kalten Mörtel.
- Homogenisieren Fliegenköpfe
- Oben auf Trockeneis, mahlen die Köpfe mit dem Mörser und Stößel, um die Pulverpartikel, dann übertragen Sie das Pulver in ein 15-ml-Glas Dounce Tissue Grinder, die auf Eis vorgekühlt wurde.
- Messen Sie die Gewichte der Mühle vor und nach der Kopf-Probe wurde in sie gegossen, und dann berechnen, wie viel der Kopf sample wiegt. Eine Gesamtzahl von 6-15 Gramm Fliegenköpfen für jeden TAP Experiment entsprechend eingestellt, um den Expressionsspiegel des Proteins ausreichen. 15 ml eiskaltem Homogenisierungspuffer (Lyse-Puffer in Schritt 2.2 optimiert) auf das Pulver und dann Schlag mit dem großen Abstand Stößel, bis es einfach für die Stößel zu gehen auf und ab. Halten Sie das Glas Mühle auf Eis zu allen Zeiten.
- Bereiten Sie den Überstand für TAP
- Übertragen des Homogenats, um eine Hochgeschwindigkeits-Spin-Zentrifugenröhrchen und für 20 Minuten bei ~ 50.000 xg (4 º C). Den Überstand in ein neues Hochgeschwindigkeits-Zentrifugenröhrchen und wiederholen Sie die Zentrifugation noch einmal.
- Den Überstand in ein Ultrazentrifugenröhrchen und führen Sie eine 40 min ~ 250.000 xg Spin um den Überstand weiter zu löschen. Der Überstand ist bereit für die Tandem-Affinitätsreinigung Verfahren nach Ultrazentrifugation.
- Erweitern und sammeln erwachsenen Fliegen.
3. TAP Reinigung
Die folgenden Abschnitte wurden aus dem Labor Séraphin TAP abgeleitet protocol 12 (http://web.as.uky.edu/Biology/faculty/rymond/BIO%20510/Bertran%20Seraphin%27s%20TAP%20page.pdf )
- Führen IgG Wulst Affinitätsreinigung
- Bereiten IgG Sepharose Wulst während die Proben werden zentrifugiert. Waschen 400 ul IgG Wulst 3x in einem 15 ml Falcon-Röhrchen mit 10 ml kaltem Waschpuffer IgG. Für jede Wäsche, rocken das Rohr vorsichtig für 2 min, und dann Spin-Down der Kügelchen bei 1.000 g für weitere 2 min. Am Ende des dritten waschen, entfernen Sie den Puffer und lassen Sie nur die Perlen in die Röhre.
- Die geklärte Überstand (~ 15 ml) vorsichtig in den 15-ml-Tube mit den IgG-Perlen zu übertragen. Die Perlen und Gehirn Lysat Mix Inkubation bei 4 º C auf einem Kolbenpendel für 2 Stunden.
- Richten Sie eine sauber und leer Mikro-Säule mit etwa 15 ml Gesamtvolumen im Kühlraum. Laden Sie die IgG Wulst Mischung durch stetig Gießen Sie die Mischung in der Kolonne; versuchen Sie nicht, abzufangen,Luftblasen in der Säule. Lassen Sie die Perlen in der Spalte und der Puffer absetzen, langsam durch Schwerkraft ablaufen lassen.
- Gründlich die Säule mit 10 ml kaltem Waschpuffer IgG schließlich des Gehirns Lysat durch die Säule ständiger IgG floss. Wiederholen Sie den Wasch 2x. Hinweis: nie den Wulst an der Luft trocknen lassen.
- Führen TEV-Spaltung
- Nach dem dritten Waschen, waschen Sie die Spalte wieder mit 10 ml TEV-Spaltpuffer. Dieser Schritt bereitet die IgG-Perle, die die Köder-Komplex für TEV-Spaltung sequestrates.
- Kurz bevor der letzte Tropfen TEV-Puffer ist im Begriff, heraustropfen, legte eine Kappe am unteren Ende der Säule, um den Fluss zu blockieren, fügen Sie 1,3 ml TEV-Puffer mit 130 Einheiten der TEV-Enzym an der Säule, und dann sicher Kappe die Spitze die Säule. Sicherzustellen, dass die Säule und an beiden Enden verschlossen.
- Drehen Sie die Säule bei 18 º C für 2 Stunden, damit der TEV-Enzym, um das Peptid an der TEV-Website zu spalten und lassen Sie die Proteinkomplex while und hinterließ den Protein A-Domäne des Peptids an IgG Sepharose-Kügelchen gebunden.
- Führen Calmodulin Wulst Affinitätsreinigung
- Bereiten Sie die Calmodulin-Perlen, während die IgG-Perlen werden mit dem TEV Enzym inkubiert. In einem 15 ml Falcon-Röhrchen 3x mit 10 ml kaltem Calmodulin-Bindungspuffer waschen 200 ul Calmodulin-Perlen, jedes Mal. Für jede Wäsche, schaukeln das Rohr für 2 min auf einem Kolbenpendel, und Spin-Down der Kügelchen bei 1.000 g für 2 min. Am Ende des dritten waschen, nehmen Sie alle den Puffer und lassen Sie nur die Perlen in die Röhre.
- Am Ende der TEV-Inkubation (Schritt 3.2.3), kehren die IgG-Säule zurück in den Kühlraum und legen Sie sie gerade nach oben. Lassen Sie die Perlen für 10 Minuten absetzen.
- Entfernen Sie die obere Abdeckung und dann die untere Kappe, und sammeln Sie die 1,3 ml TEV-Spaltprodukt in einem 15 ml Falcon-Röhrchen dann. Lassen Sie den Puffer vollständig ablaufen lassen. Fügen Sie eine zusätzliche 200 ul TEV-Puffer auf die Säule, um das Totvolumen der Säule schieben, sammeln die fNieder in derselben Röhre.
- In 4,5 ml Calmodulin-Bindungspuffer und 4,5 ul 1 M CaCl 2 bis 1,5 ml TEV-Spaltprodukt oben gesammelt. Der CaCl 2 dient dazu, die EDTA in TEV-Puffer titriert. Übertragen Sie die 6 ml Mischung auf das Rohr, das die Calmodulin-Perlen und drehen Sie die Röhrchen bei 4 º C auf einem Kolbenpendel für 1 Stunde.
- Richten Sie einen sauberen und leeren Mikro-Säule mit etwa 10 ml Gesamtvolumen im Kühlraum. Laden Sie das Calmodulin Wulst Mischung auf die Säule und lassen Sie es durch die Schwerkraft entleeren.
- Wenn die gesamte Lösung durch das Siedlungs Spalte Calmodulin floss, waschen Sie die Säule zweimal mit je 10 ml kaltem Calmodulin-Bindungspuffer. Hinweis: vermeiden Sie die Calmodulin-Perlen stören und versuchen, die Oberfläche der Perlen so flach wie möglich während des Wasch halten.
- Eluiere das Köder-Komplex aus Calmodulin Spalte.
Gleich nach dem Waschen der Säule eluieren Calmodulin mit fünf Fraktionen von 200 ul kalten Calmodulin Elutionspuffer. Für jede Fraktion, vorsichtig mit 200 &mgr; l Elutionspuffer, um die Säule und Sammeln des Eluats mit einem markierten 1,5 ml Eppendorf-Röhrchen. Wiederholen Sie diesen 4x. - Analyse der Proteinkomplex durch SDS-PAGE
- Einen kleinen Aliquots von jedem der fünf Fraktionen (etwa 30 ul) hinzugefügt und SDS-Ladepuffer. Kochen Sie die Proben für 5 min und laden Sie die Proben Seite-an-Seite mit Protein molekulare Marker in einem Gradienten (4-15%) SDS-PAGE-Gel.
- Nachdem die Proben vollständig in das Gel aufgelöst, stoppen Sie die Elektrophorese und färben das Gel mit allen G-250-basierte empfindlich kolloidale Coomassie-Färbung Verfahren wie "blue silver"-Färbung 13. Silberfärbung ist optional, aber nicht bevorzugt, da es nicht kompatibel mit der anschließenden Massenspektrometrie-Analyse. Speichern den Rest des Eluats in einem -80 ° C Gefrierschrank für die weitere Analyse, wie der Massenspektrometrie für die Aufdeckung der molekularen Identitäten der gereinigten Proteinkomplex. See Diskussion.
Ergebnisse
Hier zeigen wir unsere Anstrengungen bei der Identifizierung von Highwire-interagierenden Proteinen im Fliegenhirn. Highwire (Hiw) und seine Wirbeltieren und wirbellosen Homologen sind riesig Ubiquitin-Ligasen, die die Entwicklung und Reparatur des Nervensystems 14 zu regeln. Sie teilen sich eine Reihe von hoch konservierten funktionellen Domänen. Allerdings sind ihre molekularen Aktionen nicht ganz klar. Arbeiten im Schnecken getan, Fliegen und Maus führte zu der aktuellen Arbeitsmodell, dass Hiw Funktionen als E3-Ligase und als Gerüstprotein zur Bildung eines Multi-Untereinheiten-Ubiquitinierung Komplex, der Zeit-und Zelltyp-spezifische neuronale Funktionen durch regelt erleichtern Kombination der Interaktion mit verschiedenen Cofaktoren und an die verschiedenen Ubiquitin-Substraten. Um die Ubiquitinierung Hiw-assoziierten Komplex zu identifizieren, haben wir zuerst einen N-terminalen getaggt UAS-TAP-Hiw Transgen, das in die Rettung der HIW Mutantenphänotyps 15 voll funktionsfähig ist. Etwa 10 g der Erwachsenen fly Köpfe, die TAP-TAP oder nur Hiw transgenen Proteine exprimieren, wurden gesammelt und an TAP Verfahren nebeneinander unterzogen, wie oben beschrieben. Abschluss Eluate aus beiden Reinigungen wurden durch SDS-PAGE und Silberfärbung analysiert. Massenspektrometrie identifiziert eine Liste von Proteinen nur in der TAP-Hiw Probe, einschließlich Drosophila FSN (DFSN, einem F-Box-Protein) und RAE1 (Abbildung 2). Spätere genetische und biochemische Analysen zeigten, dass Hiw und DFSN zusammen als SCF-ähnlichen E3-Ubiquitin-Ligase-Komplex synaptische Struktur und Funktion 16 zu regeln, und RAE1 assoziiert mit Hiw in vivo und hält synaptischen Überwucherung 17. Diese Funktion RAE1 zumindest teilweise durch ihre Fähigkeit, die Proteinstabilität über Hiw Schutz Hiw vom autophagic Abbau, einen neuartigen Mechanismus, der selektiv steuert Hiw Proteinmenge während der synaptischen Entwicklungs 17 zeigt Förderung erreicht.
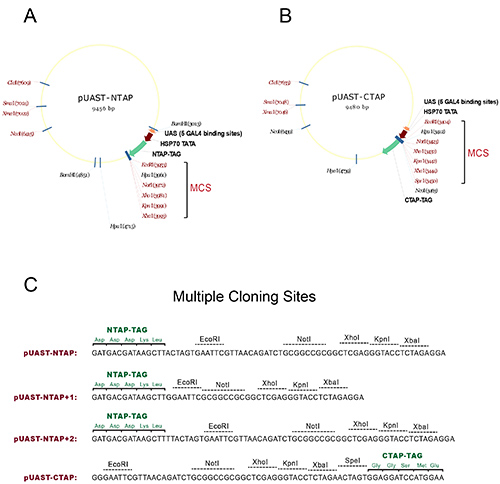
Abbildung 1. Vektoren zur Erzeugung von TAP-markierten transgenen Fliegen. (A) Die Karte des pUAST-NTAP zu konstruieren. (B) Die Karte des pUAST-CTAP zu konstruieren. Die Mehrfachklonierungsstellen (MCS) in beiden Konstrukte werden mit einer Halterung, in der Single-Cut-Restriktionsstellen sind mit rot markiert dargestellt. (C) Primärsequenz, welche die multiplen Klonierungsstellen der TAP-Vektoren mit dem Leserahmen der TAP-Tag. PUAST NTAP-1 und pUAST-NTAP 2: Zur Subklonierung zu erleichtern, werden zwei weitere NTAP Konstrukte aus dem Original-pUAST NTAP erzeugt. Zusammen bilden die drei NTAP-Konstrukte umfasst alle drei möglichen Leserahmen, um die Verwendung des MCS aufzunehmen. Alle Vektoren werden mit dem NTAP oder CTAP Fragmente ursprünglich erzeugt aufgebautSeraphin Lab ein. Klicken Sie hier für eine größere Ansicht.

. Abbildung 2: Reinigung von Highwire-interagierenden Proteinen aus Fliegenhirn von TAP Reinigung Erwachsener Köpfe von Fliegen TAP ausdrückt. (BG380-GAL4, UAS-TAP) oder TAP-Hiw (BG380-GAL4, UAS-TAP-Hiw) im Nervensystem werden gesammelt, homogenisiert und auf TAP Reinigung unterworfen sind. Die endgültigen Eluate wurden durch eindimensionale SDS-PAGE-Gel, gefolgt von Silberfärbung analysiert. Die Pfeilspitze zeigt die Köderprotein Hiw und der Stern zeigt ein verkürztes Protein TAP durch Spaltung TEV. In der TAP-Hiw Probe werden eine Liste von Proteinen, die durch Masse identifiziertSpektrometrie, einschließlich HSC-70, β-Tubulin, RAE1 und DFSN (durch Pfeile angedeutet). Diese Zahl wird in Abbildung 1a Tian et al. Modifizierten 17 Klicken Sie hier für eine größere Ansicht.
| Puffer | Zusammensetzung | Kommentare |
| Lyse-Puffer | 50 mM Tris-HCl pH 7,5 | HINWEIS: 1) In DTT und die Protease-und Proteasom-Inhibitoren kurz vor der Verwendung. |
| 125 mM NaCl | 2) Es werden hier eine Lyse-Puffer mit 0,5% NP40. Siehe Diskussion für Modifikationen an nichtionischen Reinigungsmitteln. | |
| 5% Glycerol | ||
| 0,5% NP40 | ||
| 1,5 mM MgCl 2 | ||
| 25 mM NaF | ||
| 0,2 mM DTT | ||
| (Die folgenden sind Protease und Proteasom-Inhibitoren) | ||
| 1 mM Na 3 VO 4 | ||
| 0,05 mM MG-115 | ||
| 1 mM PMSF | ||
| Protease-Inhibitor-Mix (Sigma P8340) | ||
| Protease-Inhibitor-Cocktail (Roche 04693159001) | ||
| IgG-Waschpuffer | 10 mM Tris-Cl pH 8,0 (0,5 ml 2 M Lager) | |
| 150 mM NaCl (3 ml 5 M Lager) | ||
| 0,1% NP40 (1,0 ml 10% Aktien) | ||
| H 2 0 auf 100 ml final | ||
| TEV-Spaltpuffer | 10 mM Tris-Cl pH 8,0 (0,5 ml 2 M Lager) | HINWEIS: add DTT kurz vor der Verwendung. |
| 150 mM NaCl (3 ml 5 M Lager) | ||
| 0,1% NP40 (1,0 ml 10% Aktien) | ||
| 0,5 mM EDTA (100 ul 0,5 M Lager) | ||
| 1 mM DTT (100 ul 1 M Lager) | ||
| H 2 O auf 100 ml final | ||
| Calmodulin-Bindungspuffer | 10 mM β-Mercaptoethanol (69,7 ul Lager) | |
| 10 mM Tris-Cl pH 8,0 (0,5 ml 2 M Lager) | ||
| 150 mM NaCl (3 ml 5 M Lager) | ||
| 1 mM Mg-Acetat (100 ul 1 M Lager) | ||
| 1 mM Imidazol (100 ul 1 M Lager) | ||
| 2 mM CaCl 2 (200 ul 1 M Lager) | ||
| 0,1% NP40 (1 ml 10% Aktien) | ||
| H 2 O auf 100 ml final | ||
| Calmodulin-Elutionspuffer | 10 mM β-Mercaptoethanol (69,7 ul Lager) | |
| 10 mM Tris-Cl pH 8,0 (0,5 ml 2 M Lager) | ||
| 150 mM NaCl (3 ml 5 M Lager) | ||
| 1 mM Mg-Acetat (100 ul 1 M Lager) | ||
| 1 mM Imidazol (100 ul 1 M Lager) | ||
| 10 mM EGTA (2 ml 0,5 M Lager) | ||
| 0,1% NP40 (1 ml 10% Aktien) | ||
| H 2 O auf 100 ml final |
Tabelle 1. Zusammensetzung der Puffer.
Diskussion
Tandem-Affinitätsreinigung (TAP)-Methode bietet eine Dual-Reinigungsprotokoll, das die Isolierung und Anreicherung von Proteinkomplexen durch zwei unabhängige Affinität Reinigungsschritte ermöglicht. Das Design des TAP-Tag nicht zu dem, was in diesem Protokoll Beispiel beschränkt, andere Proteinbindungsdomänen und Motive auch anwendbar, wenn Pufferbedingungen entsprechend angepasst. Ein gutes Beispiel für andere TAP-Tags ist die GS-TAP-Tag, eine Kombination aus einem G-Protein und einem Streptavidin-Bindungsmotiv, von Giulio Superti-Furga Gruppe an Reinigungs Protein-Komplex in kultivierten Säugerzelllinien 18 gerichtet ausgelegt. Der GS-TAP später angepasst Protein-Protein-Wechselwirkung in Drosophila S2 Zellen und Embryonen 19 zu studieren. Eine kürzlich JoVE Veröffentlichung von Bailey et al. Gezeigt, wie das GS-TAP-Verfahren wurde mit Zellkultur-Lysat 20 durchgeführt. Hier haben wir die Verwendung von TAP-Tag in Drosophila Nervengewebe (adult Köpfe) für große Proteom-Screening. Dieses Protokoll kann möglicherweise zu anderen Geweben, die groß angelegte Probenentnahme zu ermöglichen, wie Embryonen angepasst werden. Die erwachsenen Körper auf dem oberen Sieb (Schritt 2.3.2.3) gesammelt werden, können auch für TAP verwendet werden, was eine sehr schwierige Aufgabe sein kann. Der Lysepuffer modifiziert und konditioniert, um die massiven im Verdauungstrakt bei den meisten gegenwärtigen und dem ersten Reinigungsverfahren wesentlich, um die Belichtungszeit der Proteine an den Proteasen senken verkürzt werden Proteasen zu hemmen. Obwohl unterschiedliche TAP-Tags erfordern unterschiedliche Handhabungen und Pufferbedingungen für Affinitätsreinigungen sind die allgemeinen Grundsätze für die TAP die gleichen. Im Folgenden diskutieren wir Themen, die die Qualität der TAP Ergebnisse beeinflussen.
Der Erfolg der TAP-Ansatz hängt die Erzeugung der richtigen Transgen. Erstens muß die TAP-Tag selbst die Gesamt Konformation des Köder-Proteins, die entscheidend für seine Fähigkeit beibehalten wird, nicht ändernmit seinen physiologischen Bindungspartnern zu interagieren. Daher ist es von entscheidender Bedeutung, um zu entscheiden, wo die TAP-Tag, um das Gen von Interesse zu verschmelzen. Die Entscheidung kann auf der Basis früherer Studien des Proteins, insbesondere Informationen über die Struktur-oder Funktions epitode-markierten Transgenen hergestellt werden. Falls solche Informationen nicht verfügbar ist, Markieren des Köder-Proteins im N-oder C-terminalen parallel empfohlen. Dann können funktionelle Rettungsexperimente durchgeführt, um zu bestimmen, welche Transgens verwendet werden sollten. Zweitens sollte die Expressionsniveaus des transgenen Proteins in der Nähe von der des endogenen Proteins sein. Dies ist besonders wichtig, da die GAL4/UAS System normalerweise fahren die Expression des Transgens zu unterschiedlichen Zeitpunkten und in einem viel höheren Niveau im Vergleich zu den körpereigenen Proteinen, die entweder erhöhen können falsch-positive oder zu unerwarteten Konsequenzen, die das Profil der Protein-Interaktion zu verändern Netzwerke in den Zellen. Zum Beispiel kann Gerüstproteine überexprimieren Dermatitise dominant negative Effekte und Überexpression von Proteinen, die Enzymaktivitäten, wie Kinasen besitzen, oftmals verursachen gain-of-function-Effekte, die beide wiederum Auswirkungen auf die Fähigkeit ihrer endogenen Bindungspartner, mit ihnen zu interagieren. Somit sollte die GAL4/UAS System vermieden werden, wenn der Zeitpunkt und die Höhe der Expression des Gens von Interesse ist für die Erhaltung der endogenen Wechselwirkungen, die nur unter normalen Bedingungen zu präsentieren. Stattdessen sollte die Expression des TAP-markierten Transgen unter seinem eigenen Promotor kontrolliert werden. Dies kann entweder durch Ersetzen der UAS-Sequenz mit dem endogenen Promotor-Sequenz oder durch Verschmelzen des TAP-Sequenz im Leserahmen mit der Sequenz exonal in einem genomischen DNA-Fragment, das die gesamte Loci des Gens von Interesse enthält 21 erreicht werden. In einigen Fällen kann der TAP-Experiment in einem Funktionsverlust-Mutante Hintergrund des Gens von Interesse durchgeführt, um Konkurrenz des endogenen Proteins zu reduzieren inc.orporating in Multiproteinkomplexen. Für diesen Fall, ein Protein null Hintergrund, wenn verfügbar, bietet die ideale Voraussetzung, um die endogene Protein vollständig zu beseitigen. Mit der genetischen Mutanten-Hintergrund wird immer die Chance erhöhen, zu Ergebnissen mit höherer Qualität zu produzieren.
Die Löslichkeit des Köderprotein ist ein weiterer entscheidender Faktor für den Erfolg. Nichtionische Detergentien sind in der Regel im Lysepuffer verwendet, um die Plasmamembran zu lösen und den Proteinkomplex, löslich und intakt, in einem Status, der in der Nähe der physiologischen Umgebung zu halten. Jedoch abhängig davon, ob oder ob nicht die Proteine von Interesse sind membranintegrierte oder-assoziierten Proteinen, die Zusammensetzung und die Konzentration der nichtionischen Reinigungsmitteln entscheidender Bedeutung, das Köderprotein Solubilisierung sein könnte. 0,1-0,3% NP40 ist oft ausreichend, um cytosolische Proteine löslich. Für Membranproteine ist eine Kombination von NP40 (0,1-1%), NaDOC (0,1-1%), und Triton X-100 (0,05-0,5%) manchmal erforderlich ist, um LöseverhaltenLize und bereichern genug Proteine, die für die TAP-Experiment. Da eine strenge Lysepuffer eher schwächer Protein-Protein-Wechselwirkungen stören, muss jedoch ein Gleichgewicht erreicht werden, in denen ausreichend löslich Köder-Proteine in der Probe vorhanden sind, während zur gleichen Zeit die Mehrzahl von Protein-Protein-Wechselwirkungen werden beibehalten. Ein allgemeiner Grundsatz ist, dass immer versuchen, die wenigsten Arten von Detergenzien mit der niedrigsten Konzentration zu verwenden.
Um die Fehlerbehebung zu erleichtern, ist es sehr empfehlenswert, um eine kleine Teilmenge der Probe von jedem Schritt der beiden Reinigungen speichern. Nachfolgende SDS-PAGE und Proteinfärbung / Western-Analyse auf die Aliquots damit die Ebenen der Köderprotein und das sequentielle Anreicherung bestimmter Proteinarten überwacht werden. Zum Beispiel, wenn eine plötzliche Verminderung der Köderprotein wurde in Aliquote des Ausfließen nach TEV-Spaltung (Schritt 3.3.3) detektiert, ist es wahrscheinlich, dass das Köderprotein wurden im IgG Wulst Waschen (Schritt 3.1.4) verloren . Ein möglible Ursache könnte die unzureichende Reinigungszusammensetzung IgG Waschpuffer sein. Es gibt einen starken Rückgang der Zusammensetzung und Konzentration der Reinigungsmittel in der IgG-Waschpuffer (0,1% nur NP-40) im Vergleich zu dem Lysepuffer. Der Köder-Protein kann empfindlich auf diese Veränderungen und distanzieren von der IgG-Einheit auf den Perlen. Einstellen der Wasch IgG gegen Lysepuffer kann helfen, das Problem zu lösen. Alternativ, wenn auch weniger wahrscheinlich, die TEV-Spaltung (Schritt 3.2) nicht ausreichend gearbeitet haben, um den Köder-haltigen Komplexes von den IgG-Perlen lösen. Die Schritte, lohnt sich ein zweiter Inspektion sind die Gleichgewichtseinstellung von IgG-Kügelchen mit dem TEV-Puffer (Schritt 3.2.1) und der Enzymaktivität können falsche Handhabung, die das Enzym zu betäuben.
Denken Sie daran, dass auch mit der Zwei-Schritt-Reinigung, es wird immer noch Fehlalarme und Proteine, die unspezifisch unten in der letzten TAP-Komplex vorhanden gereinigt gezogen werden können. Ein Weg, um die unspezifische Wechselwirkungen zu identifizieren, beidest teilweise, eine Steuer TAP mit der TAP-Transgen nur parallel mit TAP-tagged-Transgens in der TAP-Reinigung Experimente enthalten. Proteine, die in der TAP-Verfahren identifiziert nur von der Liste in TAP-Transgen Reinigung identifiziert entfernt werden.
Eine erfolgreiche TAP wird eine Mischung von Proteinen, die potentiell mit dem Protein von Interesse in vivo assoziiert sind, zu reinigen. Der nächste Schritt ist die molekulare Identifizierung dieser Proteine und Massenspektrometrie wird häufig für diesen Zweck verwendet. Abhängig von den Ergebnissen der SDS-PAGE und das Experiment Bedürfnissen kann das Molekular Identitäten in dem gereinigten Proteinkomplex in den folgenden zwei Arten bestimmt werden: 1) jede einzelne Proteinbande kann aus dem Gel ausgeschnitten und mit einem Nieder unterworfen werden Komplexität LC-MS/MS, oder 2) das Eluat Fraktion, die die Spitzenproteingehalt (in der Regel die zweite Elution) enthält, kann direkt mit mäßiger Komplexität LC-MS/MS unterzogen werden. Solch eine Schrotflinte proteomic Ansatz wird möglicherweise identifizieren alle Proteine in der Anlage, je nach der Dynamikbereich der MS-Geräten 22.
Zusammenfassend stellen wir eine Methode, um Protein-Protein-Interaktionen in neuronalen Geweben eines genetischen Modellorganismus zu identifizieren - der Fruchtfliege. Es gibt eine Anzahl von Vorteilen der Anwendung des Verfahrens auf die TAP-fly: 1) Fruchtfliegen haben eine kurze Lebensdauer, so dass es relativ einfach und schnell zu transgenen Fliegen zu erzeugen und Geweben in einer großen Menge zu erhalten, die beide entscheidend für die TAP Ansatz, 2) die Fliege Genom ist vollständig kommentiert und enthält 70% der menschlichen krankheitsverursachenden Gene und 3) am wichtigsten ist, ist es einfach, funktionell zu validieren und zu charakterisieren, den Kandidaten interagierenden Proteinen in Fliegen. Es gibt umfangreiche Ressourcen in der Flug Forschung zur Charakterisierung eines bestimmten Gens, wie transgenen RNAi-basierte Sammlung nützlich zum Studium gewebespezifische Verlust-Funktion von einer Mehrheit der Genes,-Mangel-Mutante und für die meisten Allele des Gen-Loci, sowie cDNA-Klone und Transgene nützlich gain-of-function-Studien. Wir glauben, dass die Fliege TAP Methode wird eine wertvolle Ergänzung zu den Werkzeugkasten in der Fliege Labors verwendet wird. In Zukunft perspektivische kann dieses Verfahren mit Fliegenverhaltensparadigmen und Mensch-Krankheitsmodellen kombiniert werden, um die Veränderung in Protein-Protein-Interaktionsnetzwerk in Reaktion auf bestimmte Bedingungen und Stimuli zu screenen.
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Wir danken für die Zusendung EUROSARF Hefe TAP Expressionsplasmide. Wir sind auch dankbar für redaktionelle Hilfe von Ryan Labadens. Diese Arbeit wurde durch ein NIH / NINDS Zuschuss (R01NS070962) auf CW unterstützt
Materialien
| Name | Company | Catalog Number | Comments |
| U.S.A. standard test sieve No. 25 | Fisher Scientific | 04-881-18 | |
| U.S.A. standard test sieve No. 40 | Fisher Scientific | 04-881-21 | |
| Kontes Dounce Tissue Grinders 15 ml | Kimble Chase | 885300-0015 | |
| IgG sepharose beads | Pharmacia | 17-0969-01 | |
| Econo-column 0.7 cm x 20 cm | Bio-Rad | 737-4721 | |
| Econo-column 0.5 cm x 15 cm | Bio-Rad | 737-4716 | |
| Calmodulin beads | Stratagene | 214303 | |
| Coors Mortar and Pestle | CoorsTek | 60311 | |
| AcTEV Protease | Invitrogen | 12575-015 | |
| Protease Inhibitor Cocktail | Roche | 11836153001 | |
| Protease Inhibitor Mix | Sigma | P8340 |
Referenzen
- Rigaut, G., et al. A generic protein purification method for protein complex characterization and proteome exploration. Nat. Biotechnol. 17, 1030-1032 (1999).
- Collins, S. R., et al. Toward a comprehensive atlas of the physical interactome of Saccharomyces cerevisiae. Mol. Cell. Proteomics. 6, 439-450 (1074).
- Gavin, A. C., et al. Proteome survey reveals modularity of the yeast cell machinery. Nature. 440, 631-636 (2006).
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440, 637-643 (2006).
- Forler, D., et al. An efficient protein complex purification method for functional proteomics in higher eukaryotes. Nat. Biotechnol. 21, 89-92 (2003).
- Veraksa, A., Bauer, A., Artavanis-Tsakonas, S. Analyzing protein complexes in Drosophila with tandem affinity purification-mass spectrometry. Dev. Dyn. 232, 827-834 (2005).
- Li, Y. The tandem affinity purification technology: an overview. Biotechnol. Lett. 33, 1487-1499 (2011).
- Volkel, P., Le Faou, P., Angrand, P. O. Interaction proteomics: characterization of protein complexes using tandem affinity purification-mass spectrometry. Biochem. Soc. Trans. 38, 883-887 (2010).
- Xu, X., et al. The tandem affinity purification method: an efficient system for protein complex purification and protein interaction identification. Protein Expr. Purif. 72, 149-156 (2010).
- Brand, A. H., Perrimon, N. Targeted gene expression as a means of altering cell fates and generating dominant phenotypes. Development. 118, 401-415 (1993).
- Bachmann, A., Knust, E. The use of P-element transposons to generate transgenic flies. Methods Mol. Biol. 420, 61-77 (2008).
- Puig, O., et al. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Candiano, G., et al. Blue silver: a very sensitive colloidal Coomassie G-250 staining for proteome analysis. Electrophoresis. 25, 1327-1333 (2004).
- Tian, X., Wu, C. The role of ubiquitin-mediated pathways in regulating synaptic development, axonal degeneration and regeneration: insights from fly and worm. J. Physiol. , (2013).
- Wu, C., Wairkar, Y. P., Collins, C. A., DiAntonio, A. Highwire function at the Drosophila neuromuscular junction: spatial, structural, and temporal requirements. J. Neurosci. 25, 9557-9566 (2005).
- Wu, C., Daniels, R. W., DiAntonio, A. DFsn collaborates with Highwire to down-regulate the Wallenda/DLK kinase and restrain synaptic terminal growth. Neural Dev. 2, 16 (2007).
- Tian, X., Li, J., Valakh, V., DiAntonio, A., Wu, C. Drosophila Rae1 controls the abundance of the ubiquitin ligase Highwire in post-mitotic neurons. Nat. Neurosci. 14, 1267-1275 (2011).
- Burckstummer, T., et al. An efficient tandem affinity purification procedure for interaction proteomics in mammalian cells. Nat. Methods. 3, 1013-1019 (2006).
- Kyriakakis, P., Tipping, M., Abed, L., Veraksa, A. Tandem affinity purification in Drosophila: the advantages of the GS-TAP system. Fly. 2, 229-235 (2008).
- Bailey, D., Urena, L., Thorne, L., Goodfellow, I. Identification of protein interacting partners using tandem affinity purification. J. Vis. Exp. (60), e3643 (2012).
- Wu, Y., et al. A Drosophila model for Angelman syndrome. Proc. Natl. Acad. Sci. U.S.A. 105, 12399-12404 (2008).
- Liao, L., McClatchy, D. B., Yates, J. R. Shotgun proteomics in neuroscience. Neuron. 63, 12-26 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten