Method Article
La identificación de la interacción proteína-proteína en
En este artículo
Resumen
Drosophila es famoso por su gran alcance de la manipulación genética, pero no por su idoneidad del análisis bioquímico en profundidad. Aquí presentamos un procedimiento basado en TAP para identificar socios que interactúan de cualquier proteína de interés a partir de cerebro de la mosca. Este procedimiento puede potencialmente conducir a nuevas vías de investigación.
Resumen
Pantallas genéticos realizados con Drosophila melanogaster (mosca de la fruta) han hecho numerosos descubrimientos hito en el avance de las ciencias biológicas. Sin embargo, el uso de pantallas bioquímicos destinados a ampliar los conocimientos adquiridos en el análisis genético fue explorado recientemente. Aquí se describe un método para purificar el complejo de la proteína que se asocia con cualquier proteína de interés a partir de cabezas de moscas adultas. Este método toma ventaja del sistema de Drosophila GAL4/UAS para expresar una proteína cebo fusionado con una etiqueta tándem Purificación por Afinidad (TAP) en las neuronas de la mosca in vivo, y luego implementa dos rondas de purificación utilizando un procedimiento de punción similar a la establecida originalmente en de levadura 1 para purificar el complejo de proteína de interacción. Al final de este procedimiento, se obtiene una mezcla de varios complejos de proteínas cuyas identidades molecular se puede determinar por espectrometría de masas. Validación de las proteínas candidatas se beneficiará de la resource y la facilidad de la realización de estudios de pérdida de función en las moscas. Enfoques similares se pueden aplicar a otros tejidos de la mosca. Creemos que la combinación de la manipulación genética y este enfoque proteómico en el sistema modelo de la mosca tiene un tremendo potencial para abordar los problemas fundamentales en el campo de la neurobiología y más allá.
Introducción
Definición de las vías moleculares o redes que median un proceso biológico en particular es uno de los objetivos fundamentales de la investigación biomédica. Fly genetistas han dependido en gran medida de la genética a plazo, especialmente modificador pantallas genéticos (tanto potenciadoras y pantallas supresoras), para identificar los factores que trabajan juntos, en paralelo con, o aguas arriba o aguas abajo de un gen de interés. Sin embargo, las pantallas de genética hacia adelante muchas veces fallan en identificar los genes esenciales que, cuando muta, causa la letalidad en las etapas tempranas del desarrollo, o los genes con redundancia funcional y una indemnización, cuya pérdida de la función única causa defectos sutiles que son difíciles de marcar. Una forma de superar esta dificultad es la detección de interacciones directas proteína-proteína. Durante más de una década, una creciente lista de métodos bioquímicos, incluyendo la levadura de dos híbridos, visualización de fagos, entrecruzamiento químico, Co-IP, Tandem Affinity purificación (TAP), etc. se han utilizado para investigar proteínas-proteína interacciones. Cada uno de estos enfoques tiene su propio conjunto de fortalezas y debilidades en cuanto a sensibilidad y especificidad. Entre ellos, el método de TAP permite la detección de la interacción física bajo condiciones casi fisiológicas, conserva la especificidad y la consistencia y 2 incluye la capacidad de extender al análisis de alto rendimiento de 3,4.
El método TAP fue desarrollado originalmente en levadura por colegas Rigautand 1. En este método, una proteína de interés se expresa con una etiqueta de TAP. La etiqueta TAP alberga dos dominios de unión por afinidad independientes: una proteína de un dominio que se une a IgG y un dominio de unión a calmodulina. Los dos dominios están separados por una TEV (Tobacco Etch Virus) sitio de escisión. Tal combinación permite dos rondas independientes de purificaciones de afinidad para reducir suficientemente enlaces no específicos y enriquecen enlaces específicos 1. Para este ejemplo, el método TAP es una muy poderosa method para identificar en las interacciones in vivo de una proteína dada, aunque la sobreexpresión de la proteína exógena puede hacer que sea más propensos a asociarse con proteínas que normalmente no complejo con su homólogo endógeno. Desde su desarrollo, el método TAP se ha aplicado en muchos otros sistemas, incluidos los sistemas celulares basados en la cultura y otros 5,6 sistemas modelo in vivo en 6-9. Aquí se describe la adaptación del método TAP en Drosophila. En primer lugar, generar vectores pUAST-NTAP y pUAST-CTAP para facilitar la clonación y la fusión de la etiqueta TAP para el extremo N-o C-terminal del gen de interés. El TAP-etiquetados-UAS transgén se expresa entonces en el sistema nervioso bajo el control de un controlador de GAL4 neuronal 10. A continuación, se recogerá un gran número de cabezas de moscas adultas, que tienen un alto contenido de células neuronales y son fáciles de separar de otras partes del cuerpo después de la congelación sobre la base de las diferencias de tamaño. Las cabezas de los adultos se homogeneizan y se aclaró bcentrifugaciones secuenciales y, y el sobrenadante está sujeto a un procedimiento de punción se describe a continuación.
Protocolo
1. Generar UAS-TAP-etiquetados moscas transgénicas
- Generar pUAST-TAP-etiquetados construcciones de ADN.
- Decidir qué lado (N-o C-terminal) de la proteína cebo la etiqueta grifo deberá estar fusionada a, basada en la estructura / función de la proteína. Véase la discusión para más detalles.
- Subclónese la región de codificación de cDNA del gen de interés en los sitios de clonación múltiple (MCS) de los vectores pUAST-NTAP o pUAST-CTAP para generar N-o C-terminal de etiquetado transgenes UAS-TAP, respectivamente. Consulte la Figura 1 para los mapas detallados y sitios de restricción utilizables y marcos de lectura.
- Generar UAS-TAP-etiquetados moscas transgénicas.
- Generar moscas transgénicas siguiendo protocolos estándar utilizando P-elemento de inserción mediada por 11. Un número de servicios de inyección se encuentran disponibles comercialmente.
- Cruce GAL4 conductor neuronal (es decir BG380-GAL4) a cada línea transgénica individual y determinar los niveles de expresión de proteínasde cada línea por transferencia de western (con anticuerpo anti-peroxidasa de peroxidasa) y / o la inmunotinción (con anticuerpo anti-TAP). En general, se recomienda una línea transgénica con un nivel de expresión de la proteína que se encuentra cerca de la proteína endógena para los procedimientos de TAP. Véase la discusión para más detalles.
- PerformGAL4/UAS-based experimentos de rescate para confirmar la funcionalidad de los transgenes TAP-etiquetados si la pérdida de función de los mutantes de los genes de interés están disponibles. Elija un transgén que puede rescatar a la veracidad de los fenotipos mutantes para los siguientes experimentos TAP.
2. Preparar muestras de Procedimiento TAP
- Generar una acción mosca que transmite tanto un controlador neuronal GAL4 (por ejemplo, BG380-Gal4) y el transgén de TAP-etiquetados elegido con el fin de facilitar la expansión de las muestras de la mosca. Recoger las progenies F1 de la GAL4 conductor y la cruz-UAS transgenes en los casos raros en que la combinación anterior hace que la supervivencia y el crecimiento de desventaja.
- Recoger muestras a pequeña escala y optimizar condiciones lisis para solubilizar la proteína TAP-etiquetados.
- Hacer una serie de tampones de lisis utilizando una combinación de los detergentes no iónicos NP-40 (0,1-1%), NaDOC (0,1-1%) y Triton X-100 (0,05-0,5%). Véase la Tabla 1 y la discusión para más información.
- En la parte superior de una almohadilla de CO2, utilice # 5 pinzas para diseccionar 20 cabezas adultas desde el transgén que expresa TAP moscas y recoger en un tubo de 1,5 ml para probar cada condición de tampón de lisis.
- Añadir 100 ul de tampón de lisis para las pruebas del tubo y homogeneizar las cabezas acariciando arriba y abajo con una mano de mortero de plástico, a continuación, añadir otro búfer pruebas de 100 ul.
- Girar la cabeza lisado en 21.500 xg durante 10 min (4 ° C), y separar el sobrenadante y el sedimento después de la centrifugación. Añadir 25 ul de tampón de carga 2x SDS al sedimento y tampón de carga de 10 ul 4x SDS a 30 ul de sobrenadante respectivamente.
- Hervir las dos muestras durante 5 min y analizarlos lado a-Lado utilizando SDS-PAGE y posterior transferencia de Western con el anticuerpo PAP. Determinar la solubilidad por la relación de los niveles de proteína PAT en sobrenadante frente a la pastilla.
- Preparar la muestra en gran escala
- Expandir y recoger las moscas adultas.
- Ampliar las acciones neuronal-GAL4/UAS-TAP-transgene en botellas y voltear las botellas cada 3 días hasta 250 botellas acumulativos se utilizan para la recolección.
- Recoger 1-3 días de edad moscas adultas en 50 ml tubos cónicos, coloque el tubo en nitrógeno líquido inmediatamente a lo profundo del congelamiento de las moscas. Guarde las moscas en un -80 ° C congelador. Tenga en cuenta que el volumen de las moscas no debe superar los 2/3 del tubo de 50 ml.
- Recoger las cabezas de mosca (realizar este paso en la parte superior del hielo seco en polvo).
- Saque los tamices preenfriado y el mortero y la maja de la -80 ° C congelador y ponerlos en hielo seco, idealmente dentro de un gran cubo de hielo. Apila dos tamices de ensayo estándar USA con un número 25 en el the superior y N º 40 en la parte inferior.
- Tome las moscas congeladas y soltarlos en nitrógeno líquido y mantener las moscas allí por unos 10 minutos. Vortex o agitar los tubos vigorosamente para romper las cabezas, piernas y alas de los cuerpos.
- Vierta la mezcla en el tamiz superior, y luego sacudir los tamices vigorosamente mientras sujeta ambos tamices juntos. Después de cribado, los cuerpos se quedan en el tamiz superior, volar cabezas serán retenidos en el tamiz de fondo, y las piernas, alas, y otros escombros caerán al hielo seco. Separar los dos tamices y cuidadosamente transferir las cabezas de mosca al mortero frío.
- Homogeneizar las cabezas de mosca
- En la parte superior del hielo seco, moler las cabezas con el mortero y la mano del mortero a las partículas en polvo, y luego transferir el polvo a una de 15 ml de vidrio Dounce triturador de tejidos que se preenfriar en hielo.
- Medir el peso de la amoladora antes y después de la muestra de la cabeza se vertió en él, y luego calcular cuánto samp cabezale pesa. Un total de 6-15 gramos de cabezas de moscas será suficiente para cada experimento TAP ajustar en consecuencia a los niveles de expresión de la proteína. Añadir 15 ml de tampón de homogeneización enfriado con hielo (tampón de lisis optimizado en el paso 2.2) para el polvo y luego golpe con la mano de mortero de gran holgura hasta que sea fácil para los maja que suben y bajan. Mantenga el triturador de vidrio en el hielo en todo momento.
- Preparar el sobrenadante de TAP
- Transferir el homogeneizado a un tubo de centrífuga de alta velocidad y efecto durante 20 minutos a 50.000 xg ~ (4 ° C). Transferir el sobrenadante a un nuevo tubo de centrífuga de alta velocidad y repetir la centrifugación una vez más.
- Transferir el sobrenadante a un tubo de ultracentrífuga y realizar una 40 min ~ 250.000 xg de centrifugado para eliminar aún más el sobrenadante. El sobrenadante está listo para los procedimientos de purificación por afinidad en tándem después de ultracentrifugación.
- Expandir y recoger las moscas adultas.
3. Purificación TAP
En las siguientes secciones se derivaron de la TAP laboratorio Séraphin protocol 12 (http://web.as.uky.edu/Biology/faculty/rymond/BIO%20510/Bertran%20Seraphin%27s%20TAP%20page.pdf )
- Realice IgG grano purificación por afinidad
- Preparar cordón de sefarosa de IgG, mientras que las muestras se centrifugaron. Lave 400 l 3x perlas de IgG en un tubo Falcon de 15 ml con 10 ml de tampón de lavado frío IgG. Para cada lavado, sacudir el tubo suavemente durante 2 minutos, y luego de girar las bolas a 1.000 xg durante 2 min. Al final del tercer lavado, eliminar el tampón y dejar sólo las perlas en el tubo.
- Transferir con cuidado el sobrenadante clarificado (~ 15 ml) en el tubo de 15 ml que contiene las perlas de IgG. Incubar las cuentas y mezcla lisado cerebral a 4 º C en un nutator durante 2 horas.
- Configurar una columna micro limpio y vacío con aproximadamente 15 ml de volumen total en el cuarto frío. Cargue la mezcla de glóbulos IgG vertiendo constantemente la mezcla en la columna, no trate de atrapar a cualquierburbujas de aire en el interior de la columna. Permitir que las perlas se asientan en la columna y el tampón para drenar lentamente por gravedad.
- Lavar la columna a fondo con 10 ml de tampón de lavado IgG fría después de todo el lisado cerebral ha fluido a través de la columna de IgG resuelto. Repita el lavado 2x. Nota: nunca deje que el cordón seco en el aire.
- Realice TEV división
- Después del tercer lavado, lavar la columna de nuevo con 10 ml de tampón de disociación TEV. Este paso se prepara el cordón de IgG que secuestra el complejo de cebo para TEV división.
- Justo antes de la última gota de tampón de TEV está a punto de gotear, poner un tapón en la parte inferior de la columna para bloquear el flujo, añadir 1,3 ml de tampón de TEV que contiene 130 unidades de enzima TEV a la columna, y a continuación, tape de forma segura la parte superior de la columna. Asegúrese de que la columna se sella bien en ambos extremos.
- Gire la columna a 18 º C durante 2 horas para permitir que la enzima TEV para escindir el péptido en el lugar de TEV y suelte el WHI complejo proteicole dejando atrás la proteína Un péptido de dominio vinculado a las perlas de la IgG sepharose.
- Realice Calmodulina grano purificación por afinidad
- Preparar las perlas Calmodulin mientras que las perlas de IgG se incubaron con la enzima de TEV. Lave 200 l perlas Calmodulin en unos 15 ml 3x tubo Falcon, cada vez con 10 ml de frío Calmodulina de tampón de unión. Para cada lavado, agite con cuidado el tubo durante 2 minutos en un nutator, y luego de girar las bolas a 1.000 xg durante 2 min. Al final del tercer lavado, saque todo el buffer y dejar sólo las perlas en el tubo.
- Al final de la incubación TEV (paso 3.2.3), devuelva la columna de la IgG de nuevo a la cámara frigorífica y déjelo a un derecho para arriba. Deje que los granos se conforman con 10 min.
- Quite la tapa superior y luego la tapa inferior y, a continuación, recoger el producto de escisión ml TEV 1,3 en un tubo Falcon de 15 ml. Deje que el buffer se drene completamente. Añadir un tampón de 200 l de TEV adicional a la columna para empujar hacia fuera el volumen muerto de la columna, recoger el fbaja en el mismo tubo.
- Añadir 4,5 ml de tampón de unión de calmodulina y 4,5 l 1 M de CaCl2 al producto de escisión 1,5 ml de TEV recogido anteriormente. El CaCl2 sirve para valorar el EDTA en el tampón de TEV. Transferir la mezcla de 6 ml al tubo que contenía las perlas de calmodulina y girar el tubo a 4 º C en un nutator durante 1 hora.
- Crear otra columna micro limpio y vacío con aproximadamente 10 ml de volumen total en el cuarto frío. Cargar la mezcla de glóbulos de calmodulina a la columna y dejar que se drene por gravedad.
- Cuando toda la solución ha fluido a través de la columna de calmodulina asentado, lavar la columna dos veces, cada una con 10 ml de calmodulina de tampón de unión frío. Nota: no molestar a los granos de calmodulina y tratar de mantener la superficie de las perlas tan planas como sea posible durante el lavado.
- Eluir el complejo de cebo de la columna de calmodulina.
Inmediatamente después del lavado, eluir la columna de la calmodulina con cinco fracciones de 200 l frío Calmodutampón de elución Lin. Para cada fracción, agregar suavemente 200 l de tampón de elución a la columna y recoger el eluido con una marcada 1,5 ml tubo Eppendorf. Repita este 4x. - Analizar el complejo de proteínas por SDS-PAGE
- Tomar una pequeña alícuota de cada una de las cinco fracciones (unos 30 l) y añadir tampón de carga SDS. Hervir las muestras durante 5 min y la carga de las muestras de lado a lado con la proteína de marcadores moleculares en un gradiente (4-15%) en gel de SDS-PAGE.
- Después de que las muestras han resuelto por completo en el gel, detener la electroforesis y teñir el gel con cualquier procedimiento coloidales sensibles basados-G-250 de Coomassie de tinción tales como 'plata azul' tinción 13. La tinción con plata es opcional, pero no es preferible debido a que no es totalmente compatible con el análisis de espectrometría de masas posterior. Almacenar el resto del eluido en un congelador -80 ° C para su posterior análisis, tales como la espectrometría de masas para el descubrimiento de las identidades moleculares del complejo de proteína purificada. See discusión.
Resultados
Aquí se demuestra nuestro esfuerzo en la identificación de proteínas Highwire interaccionan en el cerebro de mosca. Highwire (Hiw) y sus homólogos de vertebrados e invertebrados son enormes ubiquitina ligasas que regulan el desarrollo y la reparación del sistema nervioso 14. Ellos comparten una serie de dominios funcionales altamente conservadas. Sin embargo, sus acciones moleculares no están del todo claras. El trabajo realizado en gusano, mosca y el ratón condujo al modelo de trabajo actual que HIW funciona como una ligasa E3 y como un andamio de proteínas para facilitar la formación de un complejo de ubiquitinación de múltiples subunidades, que regula las funciones neuronales de tipo específico de tiempo-y de células a través de la combinación de la interacción con diferentes cofactores y dirigidos a diferentes sustratos ubiquitina. Para identificar el complejo de ubiquitinación asociada a Hiw, primero generamos un N-terminal etiquetados transgén UAS-TAP-Hiw que es completamente funcional en el rescate del fenotipo mutante hiw 15. Acerca de 10 g de fl adultosY cabezas que expresan sólo TAP o proteínas transgénicas de TAP-HIW se recogieron y se sometieron a procedimientos de TAP lado a lado como se describe anteriormente. Eluatos finales de ambos purificaciones se analizaron por SDS-PAGE y tinción con plata continuación. La espectrometría de masas identificó una lista de las proteínas en la muestra sólo TAP-Hiw, incluyendo Drosophila FSN (DFSN, una proteína F-box) y Rae1 (Figura 2). Análisis genéticos y bioquímicos posteriores revelaron que Hiw y DFSN trabajan juntos como SCF-como E3 ubiquitina ligase complejo de regular la estructura y función sináptica 16, y se asocia con Rae1 Hiw en vivo y frena el crecimiento excesivo sináptica 17. Esta función de Rae1 es al menos parcialmente alcanzado por su capacidad para promover la estabilidad de la proteína Hiw a través de la protección de Hiw de la degradación de la autofagia, que revela un nuevo mecanismo que controla selectivamente Hiw abundancia de proteínas durante el desarrollo sináptica 17.
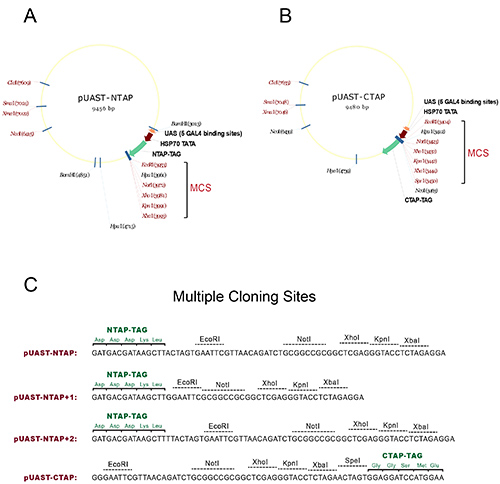
Figura 1. Los vectores para la generación de moscas transgénicas-TAP etiquetado. (A) El mapa de la pUAST-NTAP construir. (B) El mapa de la pUAST-CTAP construir. Los sitios de clonación múltiple (MCS) en ambas construcciones están marcados con un soporte, donde los sitios de restricción de un solo corte se presentan con el rojo. (C) la secuencia primaria que muestra los sitios de clonación múltiple de los vectores de TAP con el marco de lectura de la etiqueta TAP. Para facilitar la subclonación, otras dos construcciones NTAP se generan a partir del original pUAST-NTAP: pUAST-NTAP 1 y pUAST-NTAP +2. Juntos las tres NTAP construye cubre todos los tres posibles marcos de lectura para acomodar el uso de la MCS. Todos los vectores se construyeron utilizando el NTAP o fragmentos de CTAP originalmente generada en losSeraphin Lab 1. Haga clic aquí para ver la imagen más grande.

. Figura 2 Purificación de Highwire-que interactúan las proteínas del cerebro de la mosca de la purificación TAP cabezas adultas de moscas que expresan TAP. (BG380-GAL4; UAS-TAP) o TAP-Hiw (BG380-GAL4; UAS-TAP-Hiw) en el sistema nervioso se recogen, se homogeneizaron, y se sometió a purificación TAP, respectivamente. Los eluatos finales se analizaron por gel de SDS-PAGE unidimensional seguido por tinción con plata. La punta de la flecha indica la proteína Hiw cebo y el asterisco indica una proteína TAP truncada debido al TEV división. En la muestra de TAP-Hiw, una lista de las proteínas se identifican en masaespectrometría, incluyendo HSC-70, β-tubulina, Rae1 y DFSN (indicado por las flechas). Esta cifra se modificó a partir de la figura 1a de Tian y col. 17 Haga clic aquí para ver la imagen más grande.
| Buffer | Composición | Comentarios |
| El tampón de lisis | MM de Tris-HCl pH 7,5 50 | NOTA: 1) Añadir la TDT y los inhibidores de la proteasa y de proteasoma justo antes de su uso. |
| NaCl 125 mM | 2) Mostrando aquí es un tampón de lisis con 0,5% de NP40. Ver discusión de las modificaciones relativas a los detergentes no iónicos. | |
| 5% de glicerol | ||
| 0,5% de NP40 | ||
| 1,5 mM de MgCl 2 | ||
| 25 mM NaF | ||
| TDT 0,2 mM | ||
| (Los siguientes son los inhibidores de la proteasa y de proteasoma) | ||
| 1 mM Na 3 VO 4 | ||
| 0,05 mM de MG-115 | ||
| 1 mM de PMSF | ||
| Mezcla de inhibidores de proteasas (Sigma P8340) | ||
| Cóctel inhibidor de proteasa (Roche 04693159001) | ||
| Tampón de lavado IgG | 10 mM de Tris-Cl, pH 8,0 (0,5 ml de 2 M de stock) | |
| NaCl 150 mM (3 ml de 5 M de valores) | ||
| 0,1% NP40 (1,0 ml de 10% en acciones) | ||
| H 2 0 a 100 ml final | ||
| División de amortiguación TEV | 10 mM de Tris-Cl, pH 8,0 (0,5 ml de 2 M de stock) | NOTA: añadir TDT justo antes de su uso. |
| NaCl 150 mM (3 ml de 5 M de valores) | ||
| 0,1% NP40 (1,0 ml de 10% en acciones) | ||
| EDTA 0,5 mM (100 l de 0,5 M de valores) | ||
| DTT 1 mM (100 l de 1 M) | ||
| H 2 O a 100 ml final | ||
| Tampón de unión de calmodulina | 10 mM β-mercaptoetanol (69,7 l de existencias) | |
| 10 mM de Tris-Cl, pH 8,0 (0,5 ml de 2 M de stock) | ||
| NaCl 150 mM (3 ml de 5 M de valores) | ||
| 1 mM Mg-acetato (100 l de 1 M) | ||
| Imidazol 1 mM (100 l de 1 M) | ||
| 2 mM CaCl 2 (200 l de 1 M) | ||
| 0,1% NP40 (1 ml de 10% en acciones) | ||
| H 2 O a 100 ml final | ||
| Tampón de elución calmodulina | 10 mM β-mercaptoetanol (69,7 l de existencias) | |
| 10 mM de Tris-Cl, pH 8,0 (0,5 ml de 2 M de stock) | ||
| NaCl 150 mM (3 ml de 5 M de valores) | ||
| 1 mM Mg-acetato (100 l de 1 M) | ||
| Imidazol 1 mM (100 l de 1 M) | ||
| 10 mM EGTA (2 ml de 0,5 M de valores) | ||
| 0,1% NP40 (1 ml de 10% en acciones) | ||
| H 2 O a 100 ml final |
Tabla 1. Composición de tampones.
Discusión
Método de purificación por afinidad en tándem (PAT) ofrece un protocolo de purificación dual que permite el aislamiento y enriquecimiento de complejos de proteínas a través de dos etapas de purificación de afinidad independientes. El diseño de la etiqueta TAP no se limita a lo que se presenta en este protocolo, otros dominios y motivos de unión de proteínas también son aplicables si las condiciones del tampón se ajustan en consecuencia. Un buen ejemplo de otras etiquetas de TAP es la etiqueta GS-TAP, una combinación de una proteína G y un motivo de unión a estreptavidina, diseñado por el grupo de Giulio Superti-Furga destinado a purificar complejo de proteínas en líneas celulares de mamífero en cultivo 18. El GS-TAP fue posteriormente adaptada para estudiar la interacción proteína-proteína en las células S2 de Drosophila y de los embriones 19. Una publicación reciente del Jove por Bailey et al. Demuestra cómo se realizó el procedimiento GS-TAP con lisado de cultivo celular 20. Aquí presentamos el uso de PAT-etiqueta de los tejidos neurales Drosophila (adcabezas ULT) para el cribado proteómico a gran escala. Este protocolo se puede potencialmente adaptado a otros tejidos que permiten la recogida de muestras a gran escala, tales como embriones. Los cuerpos de los adultos recogidos en el tamiz superior (etapa 2.3.2.3) también se pueden utilizar para TAP, que puede ser una tarea muy difícil. El tampón de lisis debe ser modificado y acondicionado para inhibir las proteasas masivas presentes en el tracto digestivo como máximo, y el primer procedimiento de purificación acortarse significativamente con el fin de reducir el tiempo de exposición de las proteínas a las proteasas. Aunque diferentes etiquetas TAP requieren diferentes manejos y condiciones de amortiguación para las purificaciones de afinidad, los principios generales de TAP son los mismos. A continuación se discuten los asuntos que afectan a la calidad de los resultados de TAP.
El éxito del enfoque de TAP depende de la generación de la transgén derecha. En primer lugar, la propia etiqueta TAP no debe cambiar la conformación global de la proteína cebo, que es fundamental para mantener su capacidadpara interactuar con sus compañeros de unión fisiológicas. Por lo tanto, es crucial para decidir dónde fundir la etiqueta TAP para el gen de interés. La decisión puede ser tomada sobre la base de estudios previos de la proteína, en especial información sobre su estructura o transgenes epitode-etiquetados funcionales. En caso de que tal información no está disponible, el etiquetado de la proteína cebo en N-o C-terminal en paralelo se recomienda. Entonces experimentos de rescate funcionales se pueden realizar para determinar qué transgén se debe utilizar. En segundo lugar, los niveles de expresión de la proteína transgénica deben estar cerca de la de la proteína endógena. Esto es particularmente importante ya que el sistema Gal4/UAS normalmente conducir la expresión del transgén en diferentes momentos y en un nivel mucho más alto en comparación con las proteínas endógenas, que pueden o bien aumentar falsos positivos o causar consecuencias inesperadas que alteran el perfil de la interacción de proteínas redes en las células. Por ejemplo, las proteínas de andamiaje que sobreexpresan pueden cause efectos negativos dominantes y las proteínas que sobreexpresan que poseen actividades enzimáticas, tales como quinasas, muchas veces causan aumento de la función de los efectos, los cuales a su vez puede afectar a la capacidad de sus compañeros de unión endógenas para interactuar con ellos. Por lo tanto, el sistema GAL4/UAS debe evitarse cuando el momento y el nivel de la expresión del gen de interés es crítico para la preservación de las interacciones endógenas que sólo está presente en condiciones normales. En lugar de ello, la expresión del transgén de TAP-etiquetados debe ser controlado en virtud de su propio promotor. Esto se puede lograr ya sea mediante la sustitución de la secuencia UAS con la secuencia de promotor endógeno, o mediante la fusión de la secuencia de TAP en el marco con la secuencia de exonal en un fragmento de ADN genómico que contiene todo el locus del gen de interés 21. En algunos casos, el experimento de TAP se puede realizar en una pérdida de fondo función mutante del gen de interés para reducir la competencia de la proteína endógena para Incorporating en complejos multiproteicos. Para este ejemplo, un fondo nula proteína, si está disponible, proporciona la condición ideal para eliminar por completo la proteína endógena. Usando el fondo mutante genético siempre aumentará la posibilidad de producir resultados con mayor calidad.
La solubilidad de la proteína cebo es otro factor crucial para el éxito. Detergentes no iónicos se utilizan normalmente en el tampón de lisis para disolver la membrana plasmática y retener el complejo de proteínas, solubles e intacto, en un estado que está cerca del entorno fisiológico. Sin embargo, dependiendo de si o no las proteínas de interés son proteínas asociadas a la membrana o integrado, la composición y la concentración de los detergentes no iónicos podrían ser cruciales para la solubilización de la proteína cebo. 0,1-0,3% de NP40 es a menudo suficiente para solubilizar proteínas citosólicas. Para las proteínas de membrana, una combinación de NP40 (0,1-1%), NaDOC (0,1-1%), y Triton X-100 (0,05-0,5%) a veces es necesario para solubilize y enriquecer suficientes proteínas para el experimento TAP. Sin embargo, desde un tampón de lisis estrictas tienden a perturbar más débiles interacciones proteína-proteína, un equilibrio debe ser alcanzado en el que las proteínas de cebo lo suficientemente solubles están presentes en la muestra, mientras que al mismo tiempo se conservan la mayoría de las interacciones proteína-proteína. Un principio general es que siempre trato de utilizar los tipos de menor cantidad de detergentes con la concentración más baja.
Para facilitar la resolución de problemas, se recomienda guardar una pequeña alícuota de muestra de cada paso de los dos purificaciones. Subsiguientes SDS-PAGE y tinción de proteína / análisis de Western en las alícuotas de permitir que los niveles de la proteína cebo y el enriquecimiento secuencial de ciertas especies de proteínas a ser monitoreados. Por ejemplo, si se detectó una reducción repentina de la proteína cebo en la parte alícuota de flujo de salida después de TEV división (etapa 3.3.3), es probable que la proteína cebo se perdieron durante el cordón de lavado de IgG (etapa 3.1.4) . A possble causa podría ser la composición de detergente inadecuada del tampón de lavado de IgG. Hay una fuerte caída en la composición y concentración de los detergentes en el tampón de lavado de IgG (0,1% de NP-40 solamente) en comparación con el tampón de lisis. El cebo de proteína puede ser sensible a estos cambios y desvincularse de la fracción IgG en las cuentas. Ajuste del lavado de IgG hacia tampón de lisis puede ayudar a resolver el problema. Como alternativa, aunque menos probable, el TEV división (paso 3.2) puede no haber trabajado lo suficiente para liberar el complejo que contiene cebo de las perlas de IgG. Los pasos valor de un segundo de inspección son el equilibrado de perlas de IgG con el tampón de TEV (etapa 3.2.1) y la actividad de la enzima, mal manejo puede amortiguar la enzima.
Tenga en cuenta que incluso con la purificación de dos pasos, todavía habrá falsos positivos y las proteínas que se extraen hacia abajo de manera no específica presente en el complejo TAP-purificado final. Una manera de identificar estas interacciones no específicas, enmenos en parte, es incluir un TAP control utilizando el sólo TAP transgén en paralelo con TAP-etiquetados transgén en los experimentos de purificación de TAP. Las proteínas identificadas en el procedimiento-TAP sólo deben ser removidos de la lista identificada en la purificación de TAP-transgén.
Un TAP éxito va a purificar una mezcla de proteínas que podrían estar asociadas con la proteína de interés in vivo. El siguiente paso es la identificación molecular de estas proteínas, y espectrometría de masas se utiliza comúnmente para este propósito. Dependiendo de los resultados de la SDS-PAGE y las necesidades del experimento, las identidades moleculares en el complejo de la proteína purificada se pueden determinar en las siguientes dos maneras: 1) cada banda de proteína individual puede ser cortado del gel y se sometió a un mínimo de LC-MS/MS complejidad, o 2) la fracción de elución que contiene el contenido de proteína de pico (normalmente la segunda elución) puede someterse directamente a LC-MS/MS moderada complejidad. Tal p escopetaroteomic enfoque potencialmente identificar todas las proteínas en el complejo, dependiendo de la gama dinámica del equipo MS 22.
En resumen, presentamos un método para identificar las interacciones proteína-proteína en los tejidos neuronales de un organismo modelo genético - la mosca de la fruta. Hay una serie de ventajas de la aplicación del método TAP de la marcha: 1) moscas de la fruta tienen un ciclo de vida corto, por lo que es relativamente rápida y fácil de generar moscas transgénicas y obtener tejidos en gran cantidad, ambos son cruciales para el TAP enfoque; 2) el genoma de la mosca está totalmente anotado y contiene 70% de los genes causantes de enfermedades humanas, y 3) que es más importante, es fácil para validar funcionalmente y caracterizar el candidato proteínas que interactúan en las moscas. Hay amplios recursos disponibles en la comunidad de investigación de la mosca para la caracterización de un gen determinado, como la recogida de RNAi basada en transgénicos útil para estudiar los tejidos específicos de la pérdida de la función de la mayoría de los geness, alelos mutantes y la deficiencia de la mayor parte de los loci de genes, así como clones y los transgenes útiles para los estudios de ganancia de la función de cDNA. Creemos que el método TAP mosca será una valiosa adición a la caja de herramientas utilizadas en los laboratorios de la mosca. En perspectiva de futuro, este método se puede combinar con los paradigmas de comportamiento de moscas y modelos de enfermedades humanas para detectar el cambio en la red de interacción proteína-proteína en respuesta a las condiciones y estímulos específicos.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos EUROSARF por enviarnos plásmidos de expresión de TAP levadura. También estamos agradecidos por la ayuda editorial de Ryan Labadens. Este trabajo fue apoyado por una beca NIH / NINDS (R01NS070962) a CW
Materiales
| Name | Company | Catalog Number | Comments |
| U.S.A. standard test sieve No. 25 | Fisher Scientific | 04-881-18 | |
| U.S.A. standard test sieve No. 40 | Fisher Scientific | 04-881-21 | |
| Kontes Dounce Tissue Grinders 15 ml | Kimble Chase | 885300-0015 | |
| IgG sepharose beads | Pharmacia | 17-0969-01 | |
| Econo-column 0.7 cm x 20 cm | Bio-Rad | 737-4721 | |
| Econo-column 0.5 cm x 15 cm | Bio-Rad | 737-4716 | |
| Calmodulin beads | Stratagene | 214303 | |
| Coors Mortar and Pestle | CoorsTek | 60311 | |
| AcTEV Protease | Invitrogen | 12575-015 | |
| Protease Inhibitor Cocktail | Roche | 11836153001 | |
| Protease Inhibitor Mix | Sigma | P8340 |
Referencias
- Rigaut, G., et al. A generic protein purification method for protein complex characterization and proteome exploration. Nat. Biotechnol. 17, 1030-1032 (1999).
- Collins, S. R., et al. Toward a comprehensive atlas of the physical interactome of Saccharomyces cerevisiae. Mol. Cell. Proteomics. 6, 439-450 (1074).
- Gavin, A. C., et al. Proteome survey reveals modularity of the yeast cell machinery. Nature. 440, 631-636 (2006).
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440, 637-643 (2006).
- Forler, D., et al. An efficient protein complex purification method for functional proteomics in higher eukaryotes. Nat. Biotechnol. 21, 89-92 (2003).
- Veraksa, A., Bauer, A., Artavanis-Tsakonas, S. Analyzing protein complexes in Drosophila with tandem affinity purification-mass spectrometry. Dev. Dyn. 232, 827-834 (2005).
- Li, Y. The tandem affinity purification technology: an overview. Biotechnol. Lett. 33, 1487-1499 (2011).
- Volkel, P., Le Faou, P., Angrand, P. O. Interaction proteomics: characterization of protein complexes using tandem affinity purification-mass spectrometry. Biochem. Soc. Trans. 38, 883-887 (2010).
- Xu, X., et al. The tandem affinity purification method: an efficient system for protein complex purification and protein interaction identification. Protein Expr. Purif. 72, 149-156 (2010).
- Brand, A. H., Perrimon, N. Targeted gene expression as a means of altering cell fates and generating dominant phenotypes. Development. 118, 401-415 (1993).
- Bachmann, A., Knust, E. The use of P-element transposons to generate transgenic flies. Methods Mol. Biol. 420, 61-77 (2008).
- Puig, O., et al. The tandem affinity purification (TAP) method: a general procedure of protein complex purification. Methods. 24, 218-229 (2001).
- Candiano, G., et al. Blue silver: a very sensitive colloidal Coomassie G-250 staining for proteome analysis. Electrophoresis. 25, 1327-1333 (2004).
- Tian, X., Wu, C. The role of ubiquitin-mediated pathways in regulating synaptic development, axonal degeneration and regeneration: insights from fly and worm. J. Physiol. , (2013).
- Wu, C., Wairkar, Y. P., Collins, C. A., DiAntonio, A. Highwire function at the Drosophila neuromuscular junction: spatial, structural, and temporal requirements. J. Neurosci. 25, 9557-9566 (2005).
- Wu, C., Daniels, R. W., DiAntonio, A. DFsn collaborates with Highwire to down-regulate the Wallenda/DLK kinase and restrain synaptic terminal growth. Neural Dev. 2, 16 (2007).
- Tian, X., Li, J., Valakh, V., DiAntonio, A., Wu, C. Drosophila Rae1 controls the abundance of the ubiquitin ligase Highwire in post-mitotic neurons. Nat. Neurosci. 14, 1267-1275 (2011).
- Burckstummer, T., et al. An efficient tandem affinity purification procedure for interaction proteomics in mammalian cells. Nat. Methods. 3, 1013-1019 (2006).
- Kyriakakis, P., Tipping, M., Abed, L., Veraksa, A. Tandem affinity purification in Drosophila: the advantages of the GS-TAP system. Fly. 2, 229-235 (2008).
- Bailey, D., Urena, L., Thorne, L., Goodfellow, I. Identification of protein interacting partners using tandem affinity purification. J. Vis. Exp. (60), e3643 (2012).
- Wu, Y., et al. A Drosophila model for Angelman syndrome. Proc. Natl. Acad. Sci. U.S.A. 105, 12399-12404 (2008).
- Liao, L., McClatchy, D. B., Yates, J. R. Shotgun proteomics in neuroscience. Neuron. 63, 12-26 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados