Method Article
Spatially Compact Arrangement of Larval Zebrafish Sections for Spatial Transcriptomic Analysis
요약
여기에서는 여러 제브라피시(Danio rerio) 유충 샘플을 정렬 및 동결 절제하고 공간 전사체 분석을 위해 단일 슬라이드에 수집하는 방법을 제시합니다.
초록
공간 전사체 기법은 공간적으로 등록된 유전자 발현 패턴을 시각화하기 위한 생물의학 연구의 정교한 도구입니다. 공간 이미징 플랫폼을 사용한 여러 샘플의 이미징 및 분석은 비용이 많이 들 수 있습니다. 개발 연구에서 볼 수 있듯이 여러 실험 조건에서 이러한 테스트를 수행하면 비용이 더욱 증가합니다. 비용을 줄이기 위해 이 연구는 발달 연구를 위한 공간 전사체 표본 배열의 기술과 전략을 최적화하고자 했습니다. 여기에서 이 연구는 발달 중에 투명하고 잘 정립된 발달 척추동물 모델인 제브라피시를 활용했으며, 인간과 ~70%의 유전적 상동성을 가지고 있으며, 전사체 분석에 이상적인 고도로 주석이 달린 게놈을 사용했습니다. 크기가 작기 때문에 제브라피시를 개발하면 여러 생물학적 복제물에 걸쳐 연속적인 단면을 조밀하게 배치할 수 있습니다. 여기서, 우리는 멀티플렉스 현장 혼성화 공간 이미징 플랫폼의 이미징 영역 내에서 여러 물고기 샘플의 최적화된 고정, 동결 절편 및 신뢰할 수 있는 정렬을 보고합니다. 이 방법을 사용하면 최소 4개의 서로 다른 주형과 최대 174개의 절편에서 최소 15일 후의 제브라피시를 성공적으로 동결 절제하고 22mm 10.5mm( 현장 공간 전사체 슬라이드의 경우)의 이미징 영역 내에서 수집하고 동시에 처리할 수 있습니다. 섹션 품질, 샘플 정렬 및 슬라이드당 샘플 크기를 기반으로 하는 제브라피시의 이 방법은 공간 전사체 기술의 출력 및 샘플당 비용을 최적화합니다.
서문
조직에서 공간적으로 구별되는 발현 패턴에 대한 평가는 발달, 암 및 질병에 대한 게놈 영향에 대한 이해에 여전히 중요합니다 1,2,3. 공간 전사체학(spatial transcriptomics)은 다중화 발현 기법과 조직 내 발현의 공간 등록을 결합합니다. "공간 전사체학(spatial transcriptomics)"은 Ståhl과 동료들4에 의해 처음 만들어졌으며, 이 용어에서는 현장 차세대 염기서열분석을 사용하여 장착된 암 표본을 조사했습니다. 그 이후로 "spatial transcriptomics"는 공간 정합과 결합된 고처리량 발현 연구를 위한 포괄적으로 사용되었습니다. 이러한 도구들은 강력한 도구이지만, 데이터를 생성하기 전에 대규모 기관 투자와 실험실 비용을 필요로 하는 비용이 많이 드는 작업이기도 합니다5. 고품질 데이터를 보존하면서 비용을 최소화하는 전략에 대한 수요가 높습니다.
제브라피시( Danio rerio)는 발달 생물학을 연구하기 위한 중요한 모델 시스템이 되었으며 제한된 공간에서 척추동물의 전체 장기(및 유기체) 분석을 증식시키는 수단을 제공합니다. 제브라피쉬는 작고(유충의 경우 4-6mm, 성충의 경우 2-3cm) 한 번에 수백 개의 투명한 알을 낳을 수 있습니다6. 제브라피시 배아는 외부에서 수정되고 빠르게 발달하기 때문에 연구자들은 발달 초기 단계에서 이식유전자를 도입하여 이득 및 기능 상실 대립유전자를 쉽게 생성할 수 있습니다7. 단일 슬라이드에 여러 시편을 장착하는 것은 비용을 절감할 수 있는 매력적인 전략입니다. 제브라피시는 높은 번식력과 작은 크기로 인해 표본을 위한 공간이 제한된 다중화 공간 전사체 분석에 이상적인 후보입니다8.
제브라피시 유충을 냉동 절제하는 것은 어려운 기술입니다. 많은 spatial transcriptomic 플랫폼은 제브라피시 파라핀 절편에 최적화되지 않았으며, 조직 구조를 보존하고 RNA 전사체를 유지하기 위해 제브라피시를 모델 유기체로 사용할 때 cryosection이 필요합니다. 또한 제브라피쉬의 크기가 작기 때문에 고품질 냉동 절편을 얻고 여러 샘플을 효과적으로 분석하기가 어렵습니다. 이 작업은 성체 유충보다 작고 연약한 제브라피시 유충으로 작업할 때 더 어려워집니다. 이러한 문제를 극복하기 위해 여러 샘플을 안정적으로 정렬하고 공간 이미징 플랫폼의 이미징 영역을 효율적으로 활용하여 단일 슬라이드에서 많은 고품질 섹션을 얻은 다음 공간 이미징 플랫폼에서 이미지화하고 분석할 수 있는 방법을 설명합니다(그림 1). 이 경우 이 방법은 공간 전사체 이미징 플랫폼에 적용됩니다.
프로토콜
이 프로토콜은 다트머스 대학의 기관 동물 관리 및 사용 위원회의 지침을 따릅니다.
1. 저온 유지 장치 준비
- 저온 유지 장치를 -22°C로 냉각하고 용기에 파편을 솔질하여 저온 유지 장치의 내부 표면을 청소합니다. 필요한 모든 브러시와 도구를 챔버 내부에 놓습니다.
2. 일회용 기본 금형 준비
- 샘플 정렬을 위한 기준점으로 사용할 영구 마커가 있는 기본 몰드 내부를 가로질러 직선을 그려 샘플 정렬을 위한 기본 몰드(37mm, 24mm, 5mm 일회용 플라스틱 몰드)를 준비합니다. 최상의 결과를 위해 선을 그리기 전에 직선이 있어야 하는 베이스 몰드 내부에 실험실 테이프 조각을 놓습니다(그림 2A).
- 바닥 몰드 내부의 왼쪽 또는 오른쪽 벽 근처에 점을 그려 동결 절편 중 적절한 시료 방향을 확인합니다(그림 2B).
- 각도기로 원하는 절단 각도를 측정하고 각 샘플에 대한 기본 금형 내부에 이를 표시합니다(그림 2C).
- 준비된 기본 주형에 얕은 층의 동결(광간섭 단층촬영[OCT]) 매체를 적용합니다. 샘플을 덮을 수 있을 만큼 충분한 냉동 매체가 있는지 확인하십시오.
- 매체가 전체 표면에 고르게 분포되도록 기본 금형의 수평면을 이동하기 전에 중간 병의 노즐을 프라이밍하고 기본 금형의 한쪽 모서리에 필요한 양의 매체를 추가하여 동결 매체를 적용할 때 기포를 방지합니다.
- 냉동 매체를 분배할 때 병을 천천히 짜십시오.
- 1L 비커에 얼음을 넣고 얼음 수조에 냉동 매체가 있는 기본 금형을 놓고 매체를 냉각시키기 위해 최소 10분 동안 배양합니다.
3. 드라이 아이스 준비 : 100 % 에탄올 목욕
- 얼음 양동이에 드라이 아이스 한 부분에 100% 에탄올 한 부분을 추가하여 흄 후드에 드라이 아이스와 100% 에탄올 수조를 준비합니다.
- 일회용 알루미늄 접시를 사용하거나 알루미늄 호일을 일회용 기본 금형에 맞을 만큼 큰 보트에 접습니다. 접시나 보트가 기본 틀이 완전히 평평하게 놓일 수 있을 만큼 충분히 큰지 확인하십시오.
- 접시나 보트를 욕조에 넣고 양동이를 덮습니다. 샘플을 얼리기 전에 버킷이 식을 때까지 5-10분 정도 기다립니다.
4. 안락사 샘플
- 단면화를 위해 제브라피시를 무작위로 선택합니다. 샘플의 크기가 다양한 경우 정확한 정렬을 더 쉽게 할 수 있도록 상대적 크기에 따라 그룹으로 분리하십시오(자세한 내용은 설명 참조). 큰 물고기와 작은 물고기를 별도의 접시에 담습니다.
- 비커에 물고기 시스템의 물을 채우고 비커를 얼음 양동이에 넣습니다. 비커를 얼음으로 둘러쌉니다.
- 온도계로 물의 온도를 모니터링하십시오. 온도를 2-4 °C 사이에서 안정화시킵니다.
- 그물이나 거름망을 사용하여 한 그룹의 물고기를 4°C의 물에 넣습니다. 물고기는 물에 완전히 잠겨야 하며 얼음과 접촉하지 않아야 합니다. 수술 운동이 멈추면 물에 얼음을 넣어 4°C 미만으로 유지되도록 합니다. 물고기를 4°C의 물에 10분 동안 그대로 두십시오.
참고: 다음 그룹을 안락사시키기 전에 안락사된 샘플의 각 그룹의 삽입, 정렬 및 급속 동결을 계속합니다. 매번 물을 교체하십시오.
5. 임베딩 및 정렬
- 안락사된 물고기를 4°C의 물에 10분 동안 담근 후 수집합니다. 꼬리 지느러미를 잡고 끝이 가는 집게로 물고기를 물에서 꺼내고 흡수성이 있고 보푸라기가 없는 물티슈로 부드럽게 눌러 말립니다.
참고: 고품질 섹션의 경우 4°C의 물에서 물고기를 꺼낸 것과 급속 동결 사이의 시간을 제한하는 것이 중요합니다 - 준비된 기본 주형으로 실체 현미경(10배 배율)으로 얼음 수조에서 작업하고 각 샘플을 올바른 방향으로 기준점을 따라 기본 주형에 놓고 샘플을 다른 얇은 동결 매체 층으로 부드럽게 덮습니다.
알림: 전체 금형을 냉동 매체로 채우지 마십시오. 대신 모든 샘플을 덮을 수 있을 만큼만 얇은 층을 사용하십시오. - 해부학적 기준점을 사용하여 물고기를 기본 금형 내부에 표시된 선에 정확하게 정렬합니다. 끝이 가는 집게를 사용하여 각 물고기의 방향을 조정하여 정렬되고 동일한 방향이 되도록 합니다. 천천히 움직여 동결 매체에 거품이 생기지 않도록 하십시오.
- 샘플 아래의 기본 몰드 바닥에 드라이 아이스 조각을 국부적으로 동결 될 때까지 바릅니다. 드라이 아이스로 제자리로 얼기 전에 샘플이 기준점에서 벗어나지 않도록 기본 금형 레벨의 수평면을 유지하십시오.
- 샘플이 든 기본 금형을 알루미늄 보트에 놓거나 드라이 아이스에 접시 : 100 % 에탄올 욕조에 담습니다. 보트가 수조 표면에 떠 있고 기본 곰팡이가 건조한 상태로 유지되는지 확인하십시오. 목욕을 덮고 샘플을 10분 동안 띄우십시오.
- 냉동 베이스 몰드를 호일로 싸서 절단할 준비가 될 때까지 -80°C 냉동고에 보관합니다. 나머지 그룹에 대해 4.2-5.6단계를 반복합니다.
6. 냉동 절편
- 동결 절편을 위해 동결된 베이스 몰드를 예냉된 저온 유지 장치에 넣고 저온 유지 챔버에 놓습니다. 얼어붙은 블록은 해동을 방지하기 위해 드라이아이스와 함께 상자에 넣어 운반하십시오.
- 스토리지에서 현장 공간 이미징 슬라이드를 제거하고 미리 칠해진 슬라이드 홀더에 넣습니다. 관심 영역에서 절편을 수집할 준비가 될 때까지 -22°C 저온 유지 장치 챔버에 공간 이미징 슬라이드가 있는 슬라이드 홀더를 보관하십시오.
- 기본 금형에서 냉동 샘플을 제거한 다음 새 냉동 매체를 사용하여 척에 얼립니다. 절단면이 칼날을 향하도록 척에 고정하십시오.
- 신선하고 미세한 마이크로톰 블레이드를 저온 유지 장치에 놓습니다.
- 금형을 블레이드에 정렬하고 관심 영역을 다듬습니다(권장 트림 두께 20-50μm). 금형 내부에 만들어진 표시가 샘플 내의 위치를 식별하는 데 도움이 되도록 얼어붙은 샘플 블록으로 전송되었는지 확인합니다.
- 트리밍 단계에서 금형을 조정하여 절단 표면이 동결 매체의 참조 표시와 평행이 되도록 합니다.
- 표준 양전하를 띤 현미경 슬라이드에 절편을 모으고 명시야로 확인하여 트리밍이 더 이상 필요하지 않은 시기를 확인합니다.
- 저온 유지 장치 챔버에서 공간 이미징 슬라이드를 제거하고 4°C 얼음 수조에 넣습니다. 슬라이드를 슬라이드 홀더에 보관하십시오. 슬라이드가 젖지 않도록 하십시오.
- 동결 절편 시작(권장 10-14 μm; 그림 3) 샘플의 관심 영역에 도달할 때까지 양전하를 띤 슬라이드에 절편을 수집합니다. 명시야별 섹션을 확인하여 관심 영역이 금형의 다음 섹션이 될 것인지 확인합니다.
- single-cell spatial imaging 슬라이드를 저온 유지 챔버로 다시 가져옵니다. 슬라이드 홀더에서 슬라이드를 제거하고 섹션이 굴러가지 않도록 끝이 가는 페인트 브러시를 사용하여 정확한 관심 영역에서 공간 이미징 슬라이드로 한 줄씩 섹션을 수집합니다.
- 붓 뒷면으로 비어 있고 얼어붙은 매체의 모서리를 칼 스테이지로 눌러 수집을 위해 슬라이드를 잡을 때 섹션이 평평하게 유지되도록 합니다.
- 슬라이드 상단을 피벗 포인트로 사용하고, 슬라이드를 섹션 위로 천천히 내리고, 슬라이드를 나이프 스테이지에서 들어 올리기 전에 섹션이 슬라이드에 3초 동안 부착되도록 합니다.
- 슬라이드의 이미징 영역에서 섹션을 수집할 때는 왼쪽에서 오른쪽으로 작업하고 가능한 경우 비어 있고 동결된 매체의 레이어를 겹칩니다.
- 조직을 보기 어려운 경우 슬라이드의 이미징 영역 내에 섹션을 배치하기 위해 색종이 테두리를 참조로 사용합니다.
- 관심 영역에서 섹션을 수집한 후 슬라이드를 슬라이드 홀더에 다시 놓습니다. 공간 이미징 슬라이드에 더 이상 수집할 샘플이 없는 경우, 현장 공간 이미징 플랫폼을 사용하여 기기를 분석할 준비가 될 때까지 최대 2주 동안 슬라이드를 -80°C에서 보관하십시오. 여러 금형에서 단면을 수집해야 하는 경우 공간 이미징 슬라이드를 4°C 얼음 수조에 다시 넣고 다음 금형에 대해 6.3-6.11 단계를 반복합니다.
- 헤마톡실린 및 에오신(HE) 염색을 위한 표준 양전하 현미경 슬라이드에서 각 금형의 관심 영역 절편 전후를 수집하여 분석을 진행하기 전에 샘플 정렬과 절편 품질이 충분한지 확인합니다.
7. 샘플 고정
- 저온 유지 장치에서 기준 슬라이드를 제거하고 RT에서 30분 동안 자연 건조하여 슬라이드에 섹션을 부착합니다.
- 4% 파라포름알데히드(표 1)가 포함된 슬라이드 용기에 20분 동안 섹션을 넣어 수정합니다.
- 증류수와 함께 슬라이드 용기에 3분 동안 넣어 섹션을 세척합니다.
- HE 염색을 계속하거나 건조시키고 향후 염색을 위해 슬라이드를 -80°C에서 보관하십시오.
8. 단면도의 HE 염색
- 슬라이드를 100% 에탄올에 2분, 95%(표 2) 에탄올에 2분, 수돗물을 1분 동안 배양하여 섹션을 탈수하고 청소합니다. 현미경 슬라이드 염색 랙을 사용하여 수조에서 수조로 슬라이드를 옮깁니다.
- 핵을 염색하고 슬라이드를 Hematoxylin에서 2분 45초, 수돗물을 1분, 0.3% 산성화 알코올(표 3)에서 1분 동안 배양한 다음 수돗물을 1분 동안 흘려서 구별합니다. 현미경 슬라이드 염색 랙을 사용하여 수조에서 수조로 슬라이드를 옮깁니다.
- Eosin Y 1%에서 45초, 50% 에탄올(표 4)에서 1분, 95% 에탄올을 1분, 100% 에탄올에서 1분 동안 배양하여 세포질 성분을 염색하고 탈수합니다. 현미경 슬라이드 염색 랙을 사용하여 수조에서 수조로 슬라이드를 옮깁니다.
- Xylene에서 슬라이드를 1분 동안 배양하여 섹션을 지웁니다. 전사 피펫으로 슬라이드의 상단 1/3에 장착 매체 한 방울을 바르고 집게로 장착 매체 상단의 커버슬립을 천천히 내려 슬라이드를 장착하고 덮습니다.
9. 단면도의 공간 transcriptomic 화상 진찰 그리고 분석
- -80 °C 저장소에서 이미징 슬라이드를 제거하고 공간 전사체 분석을 위한 현장 공간 이미징 플랫폼으로 이미지화합니다.
참고: 이미징과 관련된 정확한 단계는 공간 이미징 플랫폼에 의해 결정됩니다. - spatial transcriptomic platform의 품질 관리 메트릭을 검토하십시오. 확인해야 할 중요한 지표는 검출된 세포의 수, 세포당 중앙값 전사체, 100μm당 핵 전사체2, 프로브 세트에 있는 각 유전자의 총 고품질 디코딩된 전사체입니다.
참고: 이러한 품질 관리 지표에는 보편적인 임계값이 없으며 이러한 임계값에 대한 기대치는 사용하는 샘플 및 유전자 패널에 따라 달라집니다. - 검출 가능한 RNA 전사체의 데이터 출력을 읽고, 실험의 품질 관리 지표를 기반으로 어떤 전사체가 품질이 낮은지 결정하고, 품질이 낮은 전사체를 필터링합니다. 섹션 내의 공간 배열과 관련하여 나머지 고품질 대본을 분석합니다.
- 실험적 관심 분야를 기반으로 한 절편과 클러스터 세포의 세포 분할을 볼 수 있습니다. 동일한 슬라이드에서 서로 다른 연령대의 제브라피시 그룹에 걸쳐 관심 영역의 세포 클러스터 내 RNA 전사체를 비교합니다.
결과
이 방법(그림 1)에서 제브라피쉬는 공간적으로 분해된 유전자 발현 패턴을 조사하기 위한 동물 모델로 사용됩니다. 공간 이미징을 위해 유충 제브라피시를 효율적으로 냉동 절편하는 것은 어려운 일입니다. 절편은 조직 구조와 검출 가능한 유전자를 유지하기 위해 고품질이어야 합니다(그림 4). 공간적으로 효율적인 이미징을 위해 여러 샘플이 포함된 섹션은 모든 샘플에서 관심 영역을 분석할 수 있도록 정확하게 정렬되어야 합니다(그림 2 및 그림 5). 마지막으로, 공간 전사체학 슬라이드의 이미징 영역 내에서 잠재적인 데이터 포인트의 수를 최대화하기 위해 섹션을 효율적으로 수집하고 간격을 지정해야 합니다(그림 3 및 그림 6). 실험은 성공적인지 최적이 아닌지를 결정하기 위해 이 세 가지 매개변수를 사용하여 측정되었습니다.
많은 프로토콜 수정은 공간 전사체 이미징에 적합한 후보인 고품질 절편을 수집하는 능력에 기여했습니다. 안락사, 고정 및 삽입에 대한 프로토콜을 조정하여 단면 품질을 개선했습니다(그림 4). 첫 번째 프로토콜(그림 4, 프로토콜 1)에서 단면 품질은 최적이 아니었고 조직은 구조를 유지하지 못했습니다. 이를 바로잡기 위해 두 번째 프로토콜(그림 4, 프로토콜 2)에서는 슬라이드에 절편을 수집한 후 샘플과 고정 조직을 삽입한 후 급속 동결 단계를 도입했습니다. 이로 인해 절편의 전반적인 조직 구조가 개선되었지만 공간 전사체학에 대해 허용 가능한 품질이라고 생각되는 수준에는 도달하지 못했습니다. 마지막으로, 세 번째 프로토콜(그림 4, 프로토콜 3)에서는 물고기를 안락사시키는 데 걸리는 시간을 단축하고 물고기를 급속 냉동 전에 얼음 위에 박아 넣고 채취 후 절단하고 고정함으로써 안락사 기술을 개선했습니다. 그 결과 공간 전사체 이미징에 적합한 최고 품질의 샘플을 얻을 수 있었습니다.
여러 샘플을 관심 영역에 정렬하려면 프로토콜에 대한 약간의 수정도 필요했습니다. 샘플 간의 정렬을 가장 많이 개선한 수정 사항 중 하나는 제브라피시가 내장된 기본 금형에 기준점을 도입하는 것이었습니다(그림 2). 이 연구에서 수행된 실험에서 샘플은 동일한 절단의 여러 섹션에 걸쳐 구조를 비교하여 정렬되었습니다(그림 5).
효율적인 공간 전사체학을 위한 방법을 개발하는 데 있어 마지막으로 중요한 단계는 이미징을 위해 서로 다른 연령의 정렬된 고품질 샘플을 동일한 슬라이드에 최대한 많이 맞추는 것이었습니다. 경험을 쌓고 기술을 향상시키면 수집되는 샘플의 수를 늘릴 수 있습니다. 이러한 개선은 또한 프로토콜의 임베딩 및 수집 단계에 대한 약간의 조정에 주로 기인합니다. 그림 6 의 이러한 패널은 시간이 지남에 따라 각 슬라이드의 이미징 영역에 수집된 섹션의 수를 강조합니다. 그림 6A (15dpf 물고기의 40개 섹션)는 절편 중에 하나의 주형을 사용하는 초기 실행입니다. 그림 6B (15 dpf 물고기의 90개 섹션 및 26 dpf 물고기의 47개 섹션)는 두 번째 주형을 소개하고 포매딩 중 샘플 사이의 공간을 줄여 공간 배열의 개선을 보여줍니다. 그림 6C (15 dpf 물고기의 54개 섹션, 19 dpf 물고기의 80개 섹션, 23 dpf 물고기의 24개 섹션, 26 dpf 물고기의 21개 섹션)는 세 번째 및 네 번째 주형을 소개하고 샘플 주위의 블록의 절단면을 트리밍하고 비어 있는 동결 매체를 겹쳐서 공간 배치의 지속적인 개선을 보여줍니다. 이러한 개선을 통해 공간 전사체 슬라이드의 이미징 영역에 179개의 섹션을 배치하고 4가지 다른 연령의 물고기를 테스트할 수 있었습니다.
프로토콜이 완성되었을 때, 우리는 기성품 다중화 현장 소프트웨어와 맞춤형 방법 모두에서 쉽게 분석할 수 있는 샘플을 갖게 되었습니다. 여기에 사용된 멀티플렉스 소프트웨어는 염기서열분석 데이터9에서 phred 품질 점수를 기반으로 하는 유사 phred 점수를 사용했습니다. 점수는 59%-70% 사이였으며 60%는 낮은 품질의 판독에 대한 경고의 컷오프였습니다. 검사 결과, 낮은 pseudo-phred 점수는 섹션 사이의 빈 공간에서 낮은 신호 복잡성 때문이었습니다. 슬라이드 영역은 효율적으로 사용되었지만 슬라이드 영역은 여전히 >50%가 비어 있었습니다. 조직이 차지하는 슬라이드 면적의 비율을 고려할 때, 신호가 충분히 복잡한 경우 신호 품질이 우수했습니다.
저강도, 비특이적 신호는 슬라이드의 두 가지 일반적인 영역, 즉 표본 외부와 빈 노토코드 내부에서도 관찰되었습니다(그림 7A). 단면 외부에는 세척 인공물과 일치하는 패턴이 있었는데, 이는 표본에서 멀어지는 것을 감소시킬 것입니다(그림 7B). 비특이적 신호는 노토코드(notochord)가 차지하고 있는 표본의 빈 영역 내에서도 관찰되었다. 이것은 증폭 시약이 갇힐 수 있는 세척 아티팩트일 가능성이 있는 것으로 간주되었습니다. 두 영역의 전사체는 주로 세포질(즉, 리보솜 단백질 L3 및 L4) 또는 미토콘드리아(아스파르테이트 전이효소 및 이소시트레이트 탈수소효소)였습니다. 이러한 영역이 핵10 주위의 전사체의 국소화 및 클러스터링을 기반으로 세포를 식별하는 Baysor segmentation에 포함되었을 때, 분할 분석에는 세포 경계가 포함되었으며, 여기에는 때때로 이러한 경계가 포함되었습니다. 이 신호는 표본과 쉽게 구별할 수 있을 만큼 낮았습니다.
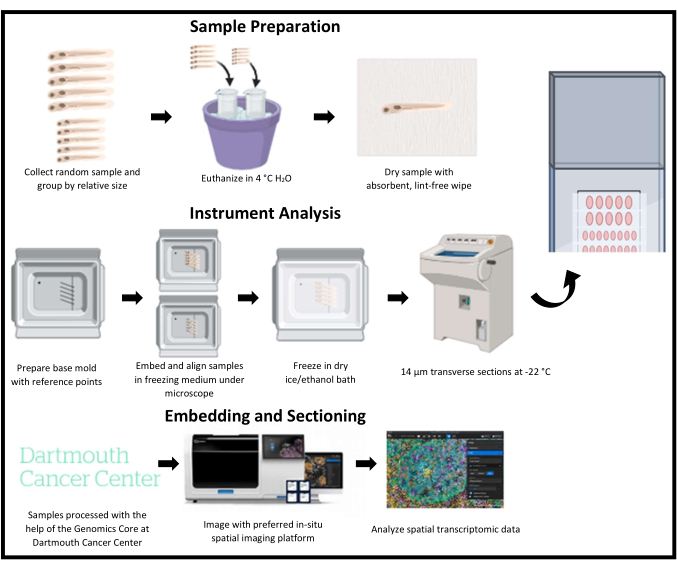
그림 1: 제브라피시의 공간 전사체학에 대한 냉동 절편의 개략도. 어린 제브라피쉬는 무작위로 수집된 다음 상대적인 크기별로 그룹화됩니다. 그런 다음 샘플은 동결 매체에 삽입되고 관심 영역에 해당하는 기본 금형의 기준점에 의해 정렬됩니다. 각 금형은 급속 동결, 동결 절편 및 동일한 슬라이드에서 한 번에 한 줄씩 수집됩니다. 슬라이드는 절편 후 -80°C에서 보관하고 공간 전사체 이미징 플랫폼을 사용하여 이미징 및 분석합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2: 정확한 절편을 위한 기준점 설정. 일련의 이미지는 냉동 절편을 위해 여러 샘플을 정렬하기 위해 일회용 기본 금형에 기준점을 추가하는 과정을 보여줍니다. 첫 번째 단계(A)는 실험실 테이프를 사용하여 모든 샘플이 정렬을 위한 참조로 사용할 금형 내부에 선을 표시하는 것입니다. (B) 동결 절편 중 적절한 시료 방향을 알리기 위해 왼쪽 또는 오른쪽 벽 근처의 금형 내부에 점을 배치한 다음 (C) 원하는 각도로 각 시료에 대한 선을 표시합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
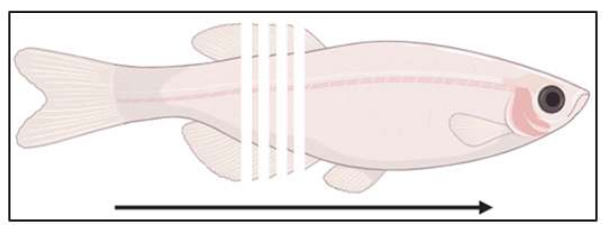
그림 3: 단면 위치 개략도. 물고기는 꼬리에서 꼬리까지 14μm 간격으로 관상으로 절단됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
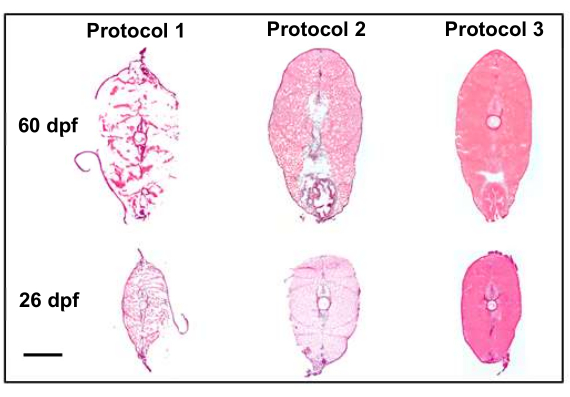
그림 4: 프로토콜 수정을 통한 섹션 품질 개선(10배). 이 그림은 최적이 아닌 조직 품질을 초래한 처음 두 프로토콜과 공간 전사체학에 적합한 후보인 고품질 절편(형태학을 유지함)을 도출한 최종 프로토콜을 비교합니다. 프로토콜 1은 Penn State bio-atlas의 비교 분석8을 활용하며, 절편 전에 4%의 파라포름알데히드 고정으로 강조됩니다. 프로토콜 2는 안락사 후 드라이아이스/에탄올 급속 동결 및 절편 후 고정을 도입하여 절편 전체에 걸쳐 인공물이 얼어붙는다는 경고와 함께 조직 구조를 훨씬 더 잘 보존합니다. 프로토콜 3은 안락사 기법을 개선하고, 급속 냉동 전과 채취 후 절편 및 고정 전 얼음 위에 포매하는 방법을 개선하여 최고 품질의 샘플을 얻을 수 있었습니다. 눈금 막대는 500μm입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 5: 다중 섹션 정렬 확인(20x). 26개의 dpf 절편의 대표적인 HE 이미지로, 한 번의 절단에서 여러 시료 간의 정렬을 강조합니다. 눈금 막대는 500μm입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 6: 표본의 공간 배치 개선. 이 패널은 시간이 지남에 따라 각 슬라이드의 이미징 영역에 수집된 섹션의 수를 강조 표시합니다. 경험을 쌓고 기술을 향상시키면 수집되는 샘플의 수를 늘릴 수 있습니다. 이러한 개선은 또한 프로토콜의 임베딩 및 수집 단계에 대한 약간의 조정에 주로 기인합니다. 패널 A (15dpf 물고기의 40개 섹션)는 단면 중에 하나의 금형을 사용하는 초기 실행입니다. 패널 B (15 dpf 물고기의 90개 섹션과 26 dpf 물고기의 47개 섹션)는 두 번째 곰팡이를 도입하고 포매딩 중 샘플 사이의 공간을 줄여 공간 배열의 개선을 보여줍니다. 패널 C (15 dpf 물고기의 54 섹션, 19 dpf 물고기의 80 섹션, 23 dpf 물고기의 24 섹션, 26 dpf 물고기의 21 섹션)는 세 번째와 네 번째 주형을 소개하고 샘플 주위의 블록의 절단면을 다듬고 비어 있고 얼어 있는 매체를 겹쳐서 공간 배치의 지속적인 개선을 보여줍니다. 각 패널의 브래킷은 단면 중에 사용되는 다양한 금형을 나타냅니다. 단면은 10.5mm 22mm 영역에 있습니다. 눈금 막대는 4mm입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 7: 공간 전사체 실험의 대표적인 결과. (A) 그림 6C에서 볼 수 있듯이 프로토콜로 처리된 슬라이드의 단일 이미지. (B) 공간 전사체 이미징 후 15dpf에서 제브라피쉬의 14μm 단면은 단면에 인접하고 노토코드 내에서 비특이적 신호를 표시합니다. 패널 A에서 눈금 막대는 4mm입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 파라포름알데히드 4% | 재고 집중도 | 분량 | 최종 집중 |
| 파라포름알데히드(분말) | 95% w/w | 40 지 | 4% w/v |
| 나오 | 1 N | Dropwise (분말이 녹을 때까지) | |
| 염산(HCl) | 1 N | 드롭와이즈(pH 6.9까지) | |
| PBS 1x 주식 | 1배 | ~1000 mL (총 부피 1000 mL 도달) | 1배 |
| 합계 | 1000 밀리리터 |
표 1: 4% 파라포름알데히드 공식. 샘플을 고정하기 위한 4% 파라포름알데히드 용액의 조성.
| 95% 에탄올 | 재고 집중도 | 분량 | 최종 집중 |
| 200 프루프 에탄올 | 100% V/V | 190 밀리리터 | 95% V/V |
| RO 물 | 100% V/V | 10 밀리리터 | 5% V/V |
| 합계 | 200 밀리리터 |
표 2: 95% 에탄올 공식. H&E 염색 중 단면을 탈수하기 위한 95% 에탄올 용액의 구성.
| 0.3% 산성화 알코올 | 재고 집중도 | 분량 | 최종 집중 |
| 빙초산 | 100% V/V | 300 마이크로L | 0.3% V/V |
| RO 물 | 100% V/V | 99.7 밀리리터 | 99.7% V/V |
| 합계 | 100 밀리리터 |
표 3: 0.3% 산성화 알코올 공식. HE 염색 시 구별하기 위한 0.3% 산성화 알코올의 조성.
| 50% 에탄올 | 재고 집중도 | 분량 | 최종 집중 |
| 200 프루프 에탄올 | 100% V/V | 100 밀리리터 | 50% V/V |
| RO 물 | 100% V/V | 100 밀리리터 | 50% V/V |
| 합계 | 200 밀리리터 |
표 4: 50% 에탄올 공식. HE 염색 중 단면을 탈수하기 위한 50% 에탄올 조성.
토론
이 보고서는 개발 중 공간 전사체 분석에서 모델 유기체로서의 제브라피시와 관련된 많은 기술적 문제에 대한 자세한 솔루션을 제공합니다. 이러한 문제를 해결하기 위해 당사의 소형 검체 배열은 새로운 공간 전사체 플랫폼1에서 비용을 최적화합니다. 공간 이미징을 위해 제브라피시 유충을 냉동 절편하는 것은 어려운 일입니다. 절편은 만족스러운 실험 실행 및 다운스트림 공간 분석을 위해 충분한 조직 구조와 전사체 품질을 유지해야 합니다8. 공간적으로 효율적인 이미징을 위해 여러 샘플을 포함하는 슬라이스는 모든 샘플에서 공통 관심 영역을 분석하기 위해 정확하게 정렬되어야 합니다. 마지막으로, 이미징 슬라이드의 지정된 영역 내에서 잠재적인 데이터 포인트의 수를 최대화하기 위해 섹션을 효율적으로 수집하고 간격을 지정해야 합니다.
이 프로토콜은 단일 세포 공간 이미징 플랫폼에 대한 훌륭한 후보인 고품질 절편을 수집할 수 있도록 합니다. 첫 번째 수정은 제브라피시를 급속 동결한 다음 슬라이드에 수집한 후 4% 파라포름알데히드로 단면을 고정하는 것이었습니다. 원래 제브라피쉬는 4°C에서 하룻밤 동안 4%의 파라포름알데히드로 고정하고 그 후에 포매하고 절편화하였다11. 샘플을 절편한 후 고정하기로 선택하면 전체 샘플을 고정할 때 효과적으로 보존되지 않는 내부 조직을 보존하는 데 도움이 되었습니다(그림 4).
두 번째 중요한 수정은 조직의 무결성을 보존하기 위해 안락사 방법을 개선하는 것이었습니다. 제브라피쉬는 원래 충분히 안락사될 수 있도록 4°C의 물에 20분 동안 두었습니다. 이는 IACUC 가이드라인의 최소 요구 사항을 10분 초과한 것입니다. 4°C의 물에서 이러한 추가 시간은 표본이 분해되는 데 더 많은 시간을 제공했으며 매우 미세하거나 섬세한 내부 구조의 이상적인 단면 품질을 낮추는 데 기여했습니다. 4°C의 물에 담기는 시간을 최소 요구 사항인 10분으로 줄임으로써 이전의 절편 시도에서 누락된 섬세한 내부 구조를 보존하는 데 도움이 되었습니다. 그림 4 에 표시되지 않은 다른 수정 사항으로는 안락사와 같은 날에 절편화, 안락사와 급속 냉동 사이의 시간을 15분에서 5분 미만으로 단축, 포매 시 사전 냉각된 냉동 매체를 사용하는 작업 등이 있습니다. 절편 전에 샘플을 -80°C에서 오래 보관할수록 절편의 조직 품질이 더 나빠졌습니다. 그 차이는 미묘했지만, 안락사와 블록 생성과 같은 날에 절편하는 데 이상적입니다. 안락사 후 및 급속 동결 전에 샘플을 정렬하는 데 걸리는 시간을 5분 이하로 단축하는 것은 절편 품질에 매우 중요합니다.
여러 샘플을 관심 영역에 정렬하려면 프로토콜에 대한 약간의 수정도 필요했습니다. 달성된 정렬의 정밀도(그림 5)는 상대적 크기별로 샘플을 그룹화하고 효율적인 공간 배열에 가장 큰 기여를 제공했습니다. 샘플을 정렬하기 위한 기본 전략은 제브라피시가 포함되어 있는 기본 주형에 그려진 기준점에 제브라피시를 오버레이하는 것입니다. 표본은 제브라피시 표본 내의 해부학적 구조에 의해 이러한 기준점에 정렬되며, 이는 원하는 관심 영역에 따라 달라질 수 있습니다. 같은 나이의 발달 중인 물고기는 크기가 최대 50%12까지 다양할 수 있습니다. 크기가 크게 다른 샘플은 해부학적 기준점과 관심 영역 사이의 거리가 다르기 때문에 잘못 정렬될 수 있습니다. 동일한 연령의 샘플을 상대적 크기별로 그룹화하고 각 크기를 금형에 포함함으로써 금형 내에서 여러 샘플을 정렬하는 능력이 향상되었습니다. 이 방법은 관심 영역과 해부학적 기준점의 선택에서 허용하는 창에 따라 샘플의 약 2/3를 안정적으로 정렬할 수 있습니다.
포매딩 및 정렬할 때 샘플 사이의 공간을 줄이면 슬라이드당 샘플 수가 더 많아집니다. 그러나 이러한 긴밀한 구성은 이미징 영역의 기준 한계에 의해 더욱 제한됩니다. 저온 유지 장치(cryostat)에 대한 경험 외에도 이를 조정하기 위한 몇 가지 전략이 설명되어 있습니다. 첫째, 절편 사이에 겹치는 매립 매체를 정밀하게 배치하고 얼어붙은 주형을 트리밍하면 특정 이미징 영역에 들어갈 수 있는 절편 수에 큰 영향을 미칠 수 있습니다. 그림 6 은 임베딩 배열을 최적화하기 전과 후에 얻은 섹션의 수를 강조합니다. 둘째, 이미징 슬라이드에 적절하게 배치하는 데 도움이 되도록 임베딩 중 및 동결 전에 가장 바깥쪽 샘플의 외부에 경계를 배치했습니다. 셋째, 유충 제브라피쉬의 크기와 투명한 특성으로 인해 샘플이 슬라이드 내의 어디에 있는지 파악하기 어려워 슬라이드에 정확하게 수집할 수 있습니다. 우리는 색종이를 사용하여 흰색 냉동 매체를 대조하여 가장 바깥쪽 샘플의 바깥쪽에 경계를 만드는 것이 슬라이드에 섹션을 훨씬 더 정확하게 배치할 수 있다는 것을 발견했습니다.
현지 온도의 뉘앙스는 절단 과정에서 샘플을 보존하는 데 도움이 됩니다. 여러 금형의 샘플이 단일 슬라이드에 수집되면 이미징 슬라이드는 4°C에서 보관되고 다음 금형은 다시 수집하기 전에 원하는 관심 영역으로 트리밍됩니다. 이미징 슬라이드는 슬라이드 표면에 제대로 부착되도록 섹션보다 약간 더 따뜻해야 합니다. 따라서 이미징 슬라이드를 -80°C에서 보관하거나 -20°C의 저온 유지 장치에 보관하는 것은 다른 금형에서 샘플을 채취하는 것이 이상적이지 않습니다. 또한 이미징 슬라이드에서 수집된 샘플이 여러 번의 동결/해동 주기를 거치지 않는 것이 중요한데, 이는 각 샘플 내 RNA 전사체의 품질에 부정적인 영향을 미칠 수 있기 때문입니다. 이전에 수집된 절편이 있는 이미징 슬라이드를 4°C로 유지하면 RNA 전사체를 적절하게 보존하는 동시에 동결/해동 주기와 관련된 부정적인 부작용 없이 적절한 준수를 위해 더 낮은 온도를 유지할 수 있습니다13.
만족스러운 결과를 얻었지만 이 방법의 설계에는 가능한 제한 사항이 있습니다. 첫 번째 한계는 공간 전사체 슬라이드의 이미징 영역에 제약을 받는다는 것인데, 이는 동일한 슬라이드에서 이미징할 수 있는 총 섹션 수에 상한을 둡니다. 섹션 사이에 여전히 빈 공간이 있다는 점을 감안할 때 이 방법이 가능한 범위에 아직 도달하지 않았을 가능성이 높지만 이미징 슬라이드에 더 이상 섹션을 맞출 수 없는 지점이 있음을 인식합니다. 더 나이가 많은 나이에 더 큰 물고기로 작업하면 전체 샘플 크기가 제한되지만 슬라이드의 이미징 영역에 들어갈 수 있는 조직의 총량에 영향을 주어서는 안 됩니다. 공간 이미징 플랫폼에 의한 기술적 개선은 이미징 슬라이드의 이미징 영역을 확대하여 이미징할 수 있는 조직의 총량을 증가시킬 수 있습니다. 이 방법의 또 다른 제한 사항은 프로토콜이 15dpf 미만의 물고기에 대해 최적화되지 않았다는 것입니다. 물고기는 어릴수록 점점 더 연약해지며, 15 dpf 미만의 물고기는 다루기가 매우 어렵고 포매 과정에서 쉽게 손상되는 것으로 밝혀졌습니다. 이로 인해 안락사와 급속 냉동 사이의 시간이 늘어났고, 이는 조직의 질에 해로웠습니다. 이는 또한 냉동 절편을 위해 남겨진 생존 가능한 샘플의 비율을 감소시켰고, 절편을 위해 남아 있는 몇 안 되는 생존 가능한 물고기는 품질 분석을 위해 조직 구조를 유지할 만큼 충분히 단단하지 않았습니다. 더 연약한 물고기를 다루고 밀도가 낮은 동결 매체로 작업하는 데 더 적합한 도구를 찾고 절단하는 동안 샘플의 강성을 증가시키면 이 방법을 15dpf 미만의 물고기에 적용할 수 있습니다. 이 방법의 마지막 주요 한계는 매우 작은 관심 영역에 대한 샘플 정렬입니다. 우리는 샘플에서 관심 영역이 작을수록 적절하게 정렬할 수 있는 가능성이 적다는 것을 발견했습니다. 우리는 약 50μm 크기의 관심 영역으로 작업했으며, 그 결과 섹션의 약 2/3가 적절하게 정렬되었습니다. 관심 영역이 50μm보다 작은 샘플은 정렬되는 절편의 2/3 미만을 초래할 수 있습니다. 잘못 정렬된 샘플의 대부분은 프로토콜의 임베딩 단계에서 발생합니다. 이미 정렬된 다른 샘플을 이동하지 않고 동결 매체에서 하나의 샘플을 조정하기가 어렵습니다. 또한 연구자가 조직의 품질에 부정적인 영향을 미치기 전에 이러한 샘플을 정렬해야 하는 시간이 제한되어 있습니다. 마지막으로, 주형은 실체현미경에서 급속냉동조로 이동하며, 샘플이 완전히 얼기 전에 주형에서 이동할 수 있습니다. 더 차가운 동결 매체로 작업하면 연구자가 이미 정렬된 다른 샘플에 영향을 주지 않고 하나의 샘플을 더 쉽게 이동할 수 있습니다. 정렬 후 금형을 이동할 필요 없이 금형을 급속 동결하면 제자리에 완전히 얼기 전에 시료 이동을 방지할 수 있습니다. 우리는 표본 외부에서 비특이적 신호를 식별했습니다.
최종 결과물과 초기 사후 분석은 추가적인 교훈을 제시했습니다. 비특이적 신호가 존재하여 고려가 필요했습니다. 제조업체에 따르면, 일부 낮은 신호 전사체는 알려진 인공물이며, 분석 후 관리할 수 있는 단일 세포 RNA-seq에서 주변 RNA를 볼 수 있습니다14. 여기에 사용된 프로토콜은 표본 배치 후 고정이 발생했기 때문에 주변 전사체에 기여했을 수 있습니다. 그러나 이른 고착은 이 어린 나이에 대해 좋지 않고 일관성 없는 형태를 초래하는 것으로 밝혀졌습니다. 대안적으로, 풀링된 증폭 시약이 이러한 효과를 생성할 수 있습니다. 질적으로는, 노토코드(notochord)에서 볼 수 있는 비특이적 신호가 가장 두드러졌다. 처리 과정에서 노토코드는 인접한 노토코드 내벽 조직의 세포질 및 미토콘드리아 프로파일과 유사한 마운팅 배지 및 전사체 신호를 포함하지 않았기 때문에 유일하게 진정으로 비어 있는 영역이었습니다. 이는 물리적 "지지"(조직 또는 장착 매체에 의한)가 이 신호를 줄이는 데 도움이 된다는 것을 시사합니다. 궁극적으로, 우리는 후처리에서 신호를 제외하는 것이 만족스러운 전략이라고 추론합니다. 비특이적 신호 특징은 실험 후에 쉽게 식별할 수 있었고 예를 들어 표본 외부 영역의 비특이적 신호를 수동으로 배제하여 다운스트림 분석 중에 완화할 수 있었습니다.
최소 4개의 서로 다른 주형(및 연령)에서 15dpf의 어린 제브라피시를 공간 전사체 이미징 영역 내에서 성공적으로 동결 절제하고 수집할 수 있으며 최대 179개의 절편을 동시에 처리할 수 있습니다. 이미징 중 RNA 전사체의 절편 품질, 시료 정렬, 시료 크기 및 품질을 기반으로 이 방법이 효율적인 공간 전사체 분석을 수행하기 위한 효과적인 프로토콜이라는 결론을 내렸습니다. 연구원들은 이 방법을 사용하여 공간 이미징과 관련된 비용을 절감하고, 공간 전사체 분석 중에 샘플 크기를 늘리고, 한 번의 실행으로 대규모 실험을 수행할 수도 있습니다.
공개
저자는 이 보고서와 관련하여 어떠한 공개나 이해 상충도 없습니다.
감사의 말
절편 및 이미징은 NCI 암 센터 지원 보조금 5P30CA023108 및 다트머스 대학의 정량 생물학 센터(NIGMS COBRE)의 자금 지원을 받는 다트머스 암 센터의 공유 리소스에서 제공하는 기기를 사용하여 수행되었습니다.
자료
| Name | Company | Catalog Number | Comments |
| 1 L Beaker | Pyrex | 1003 | |
| 200 proof pure ethanol | Koptec | V1001 | |
| Acetic acid, glacial | VWR | 0714 | acidified alcohol |
| Aluminum foil | |||
| Cover slips | Epredia | 24X50-1.5-001G | |
| Disposable base mold | Fisher HealthCare | 22-363-556 | |
| Distilled water | |||
| DPX mountant | Sigma-Aldrich | 06522 | mountant for histology |
| Dry ice pellets | |||
| Dumont #5SF Forceps | Fine Science Tools | 11252-00 | |
| Eosin-Y Alcoholic | Epredia | 71204 | Eosin Y 1% |
| Gill 1 Hematoxylin | Epredia | 72411 | Hematoxylin |
| Kimwipe | Kimberly-Clark Professional | 34120 | absorbent, lint-free wipe |
| Lab labelling tape | VWR | 89097-934 | |
| Microtome blade MX35 Ultra | Epredia | 3053835 | |
| Microtome Cryostat | Thermo Scientific | Microme HM 525 | |
| O.C.T. Compound | Fisher HealthCare | 23-730-571 | freezing medium |
| Paraformaldehyde | Sigma-Aldrich | 158127 | PFA |
| Permanent Marker | VWR | 52877-886 | |
| Protractor | |||
| SafeClear Xylene Substitute | Fisherbrand | 68551-16-6 | Xylene substitute |
| Single Edge Blades | American Line | 66-0407 | |
| Steriomicroscope | Zeiss | 4350639000 | Stemi 305 w/ double spot LED (4355259020) and Stand K lab (4354259010) |
| Superfrost Plus Micro Slides | VWR | 48311-703 | |
| Transfer pipet | |||
| Xenium V1 slide | 10X/Xenium | 3000941 | spatial transcriptomic imaging slide |
참고문헌
- Rao, A., Barkley, D., França, G. S., Yanai, I. Exploring tissue architecture using spatial transcriptomics. Nature. 596 (7871), 211-220 (2021).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet. 22, 627-644 (2021).
- Duhan, L., et al. Single-cell transcriptomics: background, technologies, applications, and challenges. Mol Biol Rep. 51, 600 (2024).
- Ståhl, P. L., et al. Visualization and analysis of gene expression in tissue sections by spatial transcriptomics. Science. 353 (6294), 78-82 (2016).
- Colman, R. E., et al. Whole-genome and targeted sequencing of drug-resistant Mycobacterium tuberculosis on the iSeq100 and MiSeq: A performance, ease-of-use, and cost evaluation. PLOS Med. 16 (4), e1002194 (2019).
- Marques, I. J., Lupi, E., Mercader, N. Model systems for regeneration: zebrafish. Development. 146 (18), dev167692 (2019).
- Mohideen, M. P. K., et al. Histology-based screen for zebrafish mutants with abnormal cell differentiation. Dev Dyn. 228 (3), 414-423 (2003).
- Copper, J. E., et al. Comparative analysis of fixation and embedding techniques for optimized histological preparation of zebrafish. Comp Biochem Physiol C Toxicol Pharmacol. 208, 38-46 (2018).
- Stock, R. J., Labudovich, M., Ducatman, B. Asymptomatic first-trimester liver cell adenoma: diagnosis by fine-needle aspiration cytology with cytochemical and ultrastructural study. Obstet Gynecol. 66 (2), 287-290 (1985).
- Petukhov, V., et al. Cell segmentation in imaging-based spatial transcriptomics. Nat Biotechnol. 40 (3), 345-354 (2022).
- Meyer, T., Tiburcy, M., Zimmermann, W. H. Cardiac macrotissues-on-a-plate models for phenotypic drug screens. Adv Drug Deliv Rev. 140, 93-100 (2019).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Dev Dyn. 203 (3), 253-310 (1995).
- Yu, K., Xing, J., Zhang, J., Zhao, R., Zhang, Y., Zhao, L. Effect of multiple cycles of freeze-thawing on the RNA quality of lung cancer tissues. Cell Tissue Bank. 18 (3), 433-440 (2017).
- Yang, S., et al. Decontamination of ambient RNA in single-cell RNA-seq with DecontX. Genome Biol. 21 (1), 57 (2020).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유