Method Article
Agencement spatialement compact de coupes de larves de poisson-zèbre pour l’analyse transcriptomique spatiale
Dans cet article
Résumé
Ici, nous présentons une méthode pour aligner et cryosectionner plusieurs échantillons de larves de poisson-zèbre (Danio rerio) et les collecter sur une seule lame pour une analyse transcriptomique spatiale.
Résumé
Les techniques transcriptomiques spatiales sont un outil sophistiqué dans la recherche biomédicale pour visualiser des modèles d’expression génique enregistrés dans l’espace. L’imagerie et l’analyse de plusieurs échantillons à l’aide de plateformes d’imagerie spatiale peuvent être coûteuses. La réalisation de ces tests dans plusieurs conditions expérimentales, comme on le voit dans les études de développement, augmente encore les coûts. Afin de réduire les coûts, cette étude a cherché à optimiser les techniques et les stratégies d’arrangement transcriptomique spatial des échantillons pour les études de développement. Ici, l’étude a utilisé le poisson zèbre, qui est un modèle de vertébré de développement bien établi qui est transparent pendant le développement, a ~70 % d’homologie génétique avec les humains et un génome hautement annoté idéal pour l’analyse transcriptomique. En raison de leur petite taille, le développement du poisson-zèbre permet également de placer de manière compacte des sections en série sur plusieurs réplicats biologiques. Dans cet article, nous rapportons l’optimisation de la fixation, de la cryosection et de l’alignement fiable de plusieurs échantillons de poissons dans la zone d’imagerie d’une plateforme d’imagerie spatiale d’hybridation in situ multiplexe. Avec cette méthode, des poissons-zèbres aussi jeunes que 15 jours après la fécondation (dpf) d’au moins 4 moules différents et jusqu’à 174 sections peuvent être cryosectionnés avec succès, collectés dans la zone d’imagerie de 22 mm 10,5 mm (pour une lame transcriptomique spatiale in situ ) et traités simultanément. Basée sur la qualité de la section, l’alignement de l’échantillon et la taille de l’échantillon par lame, cette méthode chez le poisson zèbre optimise le rendement et le coût par échantillon des techniques de transcriptomique spatiale.
Introduction
L’évaluation des modèles d’expression spatialement distincts dans les tissus reste essentielle pour notre compréhension des influences génomiques dans le développement, le cancer et la maladie 1,2,3. La transcriptomique spatiale combine des techniques d’expression multiplexées avec l’enregistrement spatial de l’expression dans les tissus. La « transcriptomique spatiale » a été inventée pour la première fois par Ståhl et ses collègues4, où des échantillons de cancer montés ont été sondés à l’aide d’un séquençage in situ de nouvelle génération. Depuis lors, la « transcriptomique spatiale » a été utilisée comme un fourre-tout pour les études d’expression à haut débit combinées à l’enregistrement spatial. Bien qu’il s’agisse d’outils puissants, ils sont également coûteux et nécessitent souvent des investissements institutionnels importants et des coûts de laboratoire avant que les données puissent être générées5. Les stratégies visant à minimiser les coûts tout en préservant des données de haute qualité sont très demandées.
Le poisson-zèbre, Danio rerio, est devenu un système modèle important pour l’étude de la biologie du développement et offre un moyen de multiplier les analyses d’organes entiers (et d’organismes) de vertébrés dans un espace limité. Les poissons-zèbres sont petits (4 à 6 mm en tant que larves et 2 à 3 cm en tant qu’adultes) et peuvent pondre des centaines d’œufs transparents à la fois6. Les embryons de poisson-zèbre sont fécondés à l’extérieur et se développent rapidement, ce qui permet aux chercheurs d’introduire des transgènes aux premiers stades de développement pour générer facilement des allèles de gain et de perte de fonction7. L’ajustement de plusieurs échantillons sur une seule lame est une stratégie attrayante pour réduire les coûts. Leur grande fécondité et leur petite taille font du poisson-zèbre un candidat idéal pour le multiplexage des tests transcriptomiques spatiaux qui ont restreint l’espace pour les spécimens8.
La cryosection des larves de poisson-zèbre est une technique difficile. De nombreuses plateformes transcriptomiques spatiales n’ont pas été optimisées pour les coupes de paraffine du poisson-zèbre et nécessitent des cryosections lorsqu’on travaille avec le poisson-zèbre en tant qu’organisme modèle pour préserver la structure tissulaire et conserver les transcrits d’ARN. De plus, la petite taille du poisson-zèbre rend difficile l’obtention de cryosections de qualité et l’analyse efficace de plusieurs échantillons. Cette tâche devient plus difficile lorsque l’on travaille avec des larves de poisson-zèbre qui sont plus petites et plus fragiles que leurs homologues adultes. Pour surmonter ces défis, nous décrivons une méthode qui aligne de manière fiable plusieurs échantillons et utilise efficacement la zone d’imagerie des plateformes d’imagerie spatiale pour obtenir de nombreuses sections de haute qualité sur une seule lame qui peuvent ensuite être imagées et analysées par des plateformes d’imagerie spatiale (Figure 1). Dans ce cas, cette méthode est appliquée à une plateforme d’imagerie transcriptomique spatiale.
Protocole
Ce protocole suit les lignes directrices du comité institutionnel de soin et d’utilisation des animaux du Dartmouth College.
1. Préparation du cryostat
- Refroidissez le cryostat à -22 °C et nettoyez les surfaces intérieures du cryostat en brossant les débris dans le récipient. Placez toutes les brosses et tous les outils nécessaires à l’intérieur de la chambre.
2. Préparation du moule de base jetable
- Préparez un moule de base (moule en plastique jetable de 37 mm, 24 mm, 5 mm) pour l’alignement de l’échantillon en traçant une ligne droite à l’intérieur d’un moule de base avec un marqueur permanent à utiliser comme point de référence pour l’alignement de l’échantillon. Placez un morceau de ruban adhésif de laboratoire à l’intérieur du moule de base, à l’endroit où la ligne droite doit se trouver, avant de tracer une ligne pour de meilleurs résultats (Figure 2A).
- Tracez un point à l’intérieur du moule de base, près de la paroi gauche ou droite, pour assurer une bonne orientation de l’échantillon pendant la cryosection (Figure 2B).
- Mesurez l’angle de coupe souhaité à l’aide d’un rapporteur et marquez-le à l’intérieur du moule de base pour chaque échantillon (Figure 2C).
- Appliquez une couche peu profonde de milieu de congélation (tomographie par cohérence optique [OCT]) sur le moule de base préparé. Assurez-vous qu’il y a juste assez de milieu de congélation pour couvrir les échantillons.
- Évitez les bulles d’air lors de l’application du produit de congélation en amorçant la buse de la bouteille de fluide et en ajoutant la quantité nécessaire de fluide dans un coin du moule de base avant de déplacer le plan horizontal du moule de base afin que le milieu soit réparti uniformément sur toute la surface.
- Pressez lentement le flacon lors de la distribution du fluide de congélation.
- Ajoutez de la glace dans un bécher de 1 L, placez le moule de base avec le milieu de congélation dans le bain de glace et incubez pendant au moins 10 minutes pour refroidir le milieu.
3. Préparation de la glace sèche : bain à 100 % éthanol
- Préparez un bain de glace sèche et d’éthanol à 100 % dans une hotte en ajoutant une partie d’éthanol à une partie de glace carbonique dans un seau à glace.
- Utilisez un plat en aluminium jetable ou pliez du papier d’aluminium dans un bateau suffisamment grand pour contenir un moule de base jetable. Assurez-vous que le plat ou le bateau est suffisamment grand pour que le moule de base soit complètement à plat.
- Placez le plat ou le bateau dans le bain et couvrez le seau. Attendez 5 à 10 minutes pour que le seau refroidisse avant de congeler les échantillons.
4. Euthanasie des échantillons
- Sélectionnez au hasard le poisson-zèbre pour la section. Si les échantillons varient en taille, séparez-les en groupes par taille relative pour faciliter un alignement précis (voir la discussion pour plus de détails). Placez les gros poissons et les petits poissons dans des plats séparés.
- Remplissez un bécher avec de l’eau du système de poisson et placez le bécher dans un seau à glace. Entourez le bécher de glace.
- Surveillez la température de l’eau à l’aide d’un thermomètre. Laissez la température se stabiliser entre 2 et 4 °C.
- À l’aide d’un filet ou d’une passoire, mettez un groupe de poissons dans l’eau à 4 °C. Les poissons doivent être complètement immergés dans l’eau et ne pas être en contact avec la glace. Une fois que le mouvement de l’opercule a cessé, ajoutez de la glace à l’eau pour vous assurer qu’elle reste en dessous de 4 °C. Laissez le poisson dans de l’eau à 4 °C pendant 10 min.
REMARQUE : Poursuivre l’enrobage, l’alignement et la congélation instantanée de chaque groupe d’échantillons euthanasiés avant d’euthanasier le groupe suivant. Remplacez l’eau à chaque fois.
5. Intégration et alignement
- Récupérez les poissons euthanasiés après qu’ils aient été immergés dans de l’eau à 4 °C pendant 10 min. Retirez les poissons de l’eau à l’aide d’une pince à pointe fine en les saisissant par la nageoire caudale et séchez-les en les pressant doucement contre une lingette absorbante et non pelucheuse.
REMARQUE : Pour des sections de haute qualité, il est essentiel de limiter le temps entre le retrait du poisson de l’eau à 4 °C et la congélation instantanée - En travaillant avec le moule de base préparé dans un bain de glace sous un stéréomicroscope (grossissement 10x), placez chaque échantillon dans le moule de base le long des points de référence dans la bonne orientation et recouvrez doucement les échantillons d’une autre fine couche de produit de congélation.
REMARQUE : Ne remplissez pas tout le moule avec du produit de congélation. Au lieu de cela, n’utilisez qu’une couche mince, juste assez pour couvrir tous les échantillons. - Utilisez un point de référence anatomique pour aligner précisément le poisson sur les lignes marquées à l’intérieur du moule de base. À l’aide d’une pince à pointe fine, ajustez l’orientation de chaque poisson afin qu’ils soient alignés et dans la même orientation. Évitez de créer des bulles dans le milieu de congélation en vous déplaçant lentement.
- Appliquez un morceau de glace sèche au fond du moule de base sous les échantillons jusqu’à ce qu’ils soient localement gelés en position. Maintenez le plan horizontal du moule de base à niveau pour éviter de déplacer les échantillons de leurs points de référence avant de les geler en position avec de la glace sèche.
- Placez le moule de base avec les échantillons sur le bateau ou le plat en aluminium dans le bain de glace carbonique : 100 % éthanol. Assurez-vous que le bateau flotte à la surface du bain et que le moule de base reste sec. Couvrez le bain et laissez les échantillons flotter pendant 10 min.
- Enveloppez le moule de base congelé dans du papier d’aluminium et conservez-le au congélateur à -80 °C jusqu’au moment de le sectionner. Répétez les étapes 4.2 à 5.6 pour les groupes restants.
6. Cryosectionnement
- Apportez les moules de base congelés dans le cryostat pré-refroidi pour la cryosection et placez-les dans la chambre du cryostat. Transportez les blocs congelés dans une boîte avec de la glace sèche pour éviter la décongélation.
- Retirez la lame d’imagerie spatiale in situ de son stockage et placez-la dans un support de lame pré-refroidi. Rangez le support de lames avec la lame d’imagerie spatiale dans la chambre du cryostat à -22 °C jusqu’à ce qu’il soit prêt à collecter des coupes dans la région d’intérêt.
- Retirez les échantillons congelés du moule de base, puis congelez-les dans un mandrin avec un produit de congélation frais. Congelez-les sur le mandrin afin que la surface de coupe fasse face à la lame.
- Placez une lame de microtome fraîche et fine dans le cryostat.
- Alignez le moule sur la lame et coupez la région d’intérêt (épaisseur de coupe recommandée 20-50 μm). Assurez-vous que les marques faites à l’intérieur du moule sont transférées sur le bloc d’échantillons congelé pour aider à identifier l’emplacement dans les échantillons.
- Ajustez le moule pendant la phase de coupe de manière à ce que la surface de coupe soit parallèle aux repères de référence dans le produit de congélation.
- Collectez des coupes sur une lame de microscope standard chargée positivement et vérifiez-les en fond clair pour confirmer quand le rognage n’est plus nécessaire.
- Retirez la lame d’imagerie spatiale de la chambre du cryostat et placez-la dans un bain de glace à 4 °C. Gardez la diapositive dans le support de diapositive. Assurez-vous que la diapositive ne soit pas mouillée.
- Commencer la cryosection (recommandé 10-14 μm ; Graphique 3) et collecter des sections sur des lames chargées positivement jusqu’à ce que la région d’intérêt des échantillons soit atteinte. Vérifiez les sections par fond clair pour confirmer que la région d’intérêt sera la section suivante du moule.
- Ramenez la diapositive d’imagerie spatiale à cellule unique dans la chambre du cryostat. Retirez la diapositive du support de diapositive et collectez des sections de la zone d’intérêt exacte sur la diapositive d’imagerie spatiale, rangée par rangée, à l’aide d’un pinceau à pointe fine pour empêcher les sections de s’enrouler.
- Appuyez sur un coin du médium vide et congelé dans l’étape du couteau avec le dos du pinceau afin que la section reste plate lorsque vous saisissez la lame pour la collecte.
- Utilisez le haut de la glissière comme point de pivot, abaissez lentement la glissière sur la section et laissez les sections adhérer à la glissière pendant 3 secondes avant de soulever la glissière de la platine du couteau.
- Travaillez de gauche à droite lors de la collecte de sections dans la zone d’imagerie de la lame et superposez des couches de support vide et figé lorsque cela est possible.
- Utilisez des bordures en papier coloré comme référence pour placer des sections dans la zone d’imagerie de la lame si le tissu est difficile à voir.
- Après avoir collecté des sections de la région d’intérêt, replacez la diapositive dans le support de diapositive. S’il n’y a plus d’échantillons à collecter sur la lame d’imagerie spatiale, conservez la lame à -80 °C jusqu’à 2 semaines jusqu’à ce qu’elle soit prête pour l’analyse de l’instrument avec la plateforme d’imagerie spatiale in situ . Si des sections doivent être prélevées à partir de plusieurs moules, remettez la diapositive d’imagerie spatiale dans le bain de glace à 4 °C et répétez les étapes 6.3 à 6.11 avec le moule suivant.
- Collectez des coupes avant et après les sections de la région d’intérêt de chaque moule sur une lame de microscope standard chargée positivement pour la coloration à l’hématoxyline et à l’éosine (HE) afin de vérifier que l’alignement de l’échantillon et la qualité de la section sont suffisants avant de procéder à l’analyse.
7. Fixation de l’échantillon
- Retirez les lames de référence du cryostat et faites-les sécher à l’air libre à RT pendant 30 minutes pour faire adhérer les sections à la lame.
- Fixez les sections en les plaçant dans un récipient à lames avec 4 % de paraformaldéhyde (tableau 1) pendant 20 min.
- Lavez les sections en les plaçant dans un récipient à lames avec de l’eau distillée pendant 3 min.
- Poursuivez la coloration HE ou séchez-les et stockez les lames à -80 °C pour une coloration future.
8. Coloration HE des sections
- Déshydratez et nettoyez les sections en incubant les lames dans de l’éthanol à 100 % pendant 2 min, de l’éthanol à 95 % (tableau 2) pendant 2 min, puis de l’eau du robinet pendant 1 min. Utilisez un support de coloration de lames de microscope pour transférer les lames d’un bain à l’autre.
- Colorer les noyaux et différencier en incubant les lames dans de l’hématoxyline pendant 2 min 45 s, de l’eau du robinet pendant 1 min, de l’alcool acidifié à 0,3 % (tableau 3) pendant 1 min, puis de l’eau du robinet pendant 1 min. Utilisez un support de coloration de lames de microscope pour transférer les lames d’un bain à l’autre.
- Colorer les composants cytoplasmiques et déshydrater en incubant les lames dans de l’éosine Y 1 % pendant 45 s, de l’éthanol à 50 % (tableau 4) pendant 1 min, de l’éthanol à 95 % pendant 1 min et de l’éthanol à 100 % pendant 1 min. Utilisez un support de coloration de lames de microscope pour transférer les lames d’un bain à l’autre.
- Nettoyez les sections en incubant les lames dans du xylène pendant 1 min. Montez et couvrez les lames en appliquant une goutte de support de montage sur le tiers supérieur de la lame à l’aide d’une pipette de transfert et en abaissant lentement une lamelle sur le dessus du support de montage à l’aide d’une pince.
9. Imagerie transcriptomique spatiale et analyse des coupes
- Retirez les lames d’imagerie du stockage à -80 °C et imagez avec une plate-forme d’imagerie spatiale in situ pour l’analyse transcriptomique spatiale.
REMARQUE : Les étapes exactes de l’imagerie seront déterminées par la plate-forme d’imagerie spatiale. - Examinez les mesures de contrôle de la qualité de la plateforme transcriptomique spatiale. Les paramètres importants à vérifier sont le nombre de cellules détectées, les transcrits médians par cellule, les transcrits nucléaires par 100 μm2 et le total des transcrits décodés de haute qualité de chaque gène dans l’ensemble de sondes.
REMARQUE : Ces paramètres de contrôle de la qualité n’ont pas de seuils universels, et les attentes à l’égard de ces seuils varient en fonction de l’échantillon et du panel de gènes utilisés. - Lisez les données de sortie des transcrits d’ARN détectables, déterminez quels transcrits sont de mauvaise qualité en fonction des mesures de contrôle de la qualité de l’expérience et filtrez les transcrits de mauvaise qualité. Analysez les transcriptions de haute qualité restantes par rapport à leur disposition spatiale dans la section.
- Visualisez la segmentation cellulaire des sections et des cellules de cluster en fonction des intérêts expérimentaux. Comparez les transcrits d’ARN au sein des groupes de cellules de la région d’intérêt à travers des groupes de poissons zèbres d’âges différents sur la même lame.
Résultats
Dans cette méthode (Figure 1), le poisson-zèbre est utilisé comme modèle animal pour sonder les modèles d’expression génique spatialement résolus. La cryosection efficace des larves de poisson-zèbre pour l’imagerie spatiale est un défi. Les coupes doivent être de haute qualité pour conserver la structure tissulaire et les gènes détectables (Figure 4). Les sections contenant plusieurs échantillons pour une imagerie spatialement efficace doivent être alignées avec précision pour analyser les régions d’intérêt dans tous les échantillons (Figure 2 et Figure 5). Enfin, les sections doivent être collectées et espacées efficacement pour maximiser le nombre de points de données potentiels dans la zone d’imagerie d’une lame de transcriptomique spatiale (Figure 3 et Figure 6). Les expériences ont été mesurées à l’aide de ces trois paramètres pour déterminer si elles étaient réussies ou sous-optimales.
De nombreuses modifications du protocole ont contribué à notre capacité à collecter des coupes de haute qualité qui sont de bons candidats pour l’imagerie transcriptomique spatiale. L’ajustement des protocoles d’euthanasie, de fixation et d’intégration a permis d’améliorer la qualité des sections (figure 4). Dans le premier protocole (Figure 4, protocole 1), la qualité de la section était sous-optimale et les tissus ne conservaient pas leur structure. Pour corriger cela, dans le deuxième protocole (Figure 4, protocole 2), une étape de congélation instantanée a été introduite après l’enrobage des échantillons et la fixation des tissus après le prélèvement de coupes sur des lames. Cela a amélioré la structure tissulaire globale des coupes, mais n’a pas permis d’atteindre ce que l’on pensait être une qualité acceptable pour la transcriptomique spatiale. Enfin, dans le troisième protocole (figure 4, protocole 3), les techniques d’euthanasie ont été affinées en raccourcissant le temps nécessaire à l’euthanasie du poisson et en l’enrobant sur de la glace avant la congélation instantanée et en la sectionnant et en fixant après la collecte. Cela a permis d’obtenir des échantillons de la plus haute qualité qui étaient de bons candidats pour l’imagerie transcriptomique spatiale.
L’alignement de plusieurs échantillons sur une région d’intérêt a également nécessité quelques modifications du protocole. L’une des modifications qui a le plus amélioré l’alignement entre les échantillons a été l’introduction de points de référence dans les moules de base dans lesquels les poissons-zèbres sont intégrés (Figure 2). Dans les expériences menées dans le cadre de cette étude, les échantillons ont été alignés en comparant les structures de plusieurs sections de la même coupe (figure 5).
La dernière étape importante dans le développement de la méthode pour une transcriptomique spatiale efficace a été d’ajuster le plus grand nombre possible de ces échantillons alignés de haute qualité d’âges différents sur la même lame pour l’imagerie. L’acquisition d’expérience et l’amélioration des compétences techniques contribuent à l’augmentation du nombre d’échantillons collectés. Ces améliorations étaient également dues en grande partie à de légers ajustements aux étapes d’intégration et de collecte dans le protocole. Les panneaux de la figure 6 mettent en évidence le nombre de sections collectées dans la zone d’imagerie de chaque lame au fil du temps. La figure 6A (40 sections de poissons de 15 dpf) est une première course qui utilise un moule pendant la section. La figure 6B (90 sections de poissons de 15 dpf et 47 sections de poissons de 26 dpf) introduit un deuxième moule et montre une amélioration de l’agencement spatial en réduisant la quantité d’espace entre les échantillons lors de l’encastrement. La figure 6C (54 sections de poissons de 15 dpf, 80 sections de 19 poissons de dpf, 24 sections de poissons de 23 dpf et 21 sections de 26 poissons de dpf) présente un troisième et un quatrième moule et montre l’amélioration continue de l’agencement spatial en coupant la surface de coupe du bloc autour des échantillons et en chevauchant le milieu de congélation vide. Ces améliorations nous ont permis de placer 179 sections dans la zone d’imagerie de notre lame transcriptomique spatiale et de tester des poissons de 4 âges différents.
À la fin du protocole, nous disposions d’échantillons facilement analysables à la fois avec le logiciel in situ multiplexé prêt à l’emploi et avec des méthodes personnalisées. Le logiciel multiplex utilisé ici a utilisé un score pseudo-phred, vaguement basé sur les scores de qualité phred dans les données de séquençage9. Les scores variaient de 59 % à 70 %, 60 % étant le seuil pour un avertissement de lectures de qualité inférieure. Lors de l’inspection, le faible score pseudo-phred était dû à la faible complexité du signal dans l’espace vide entre les sections. Bien que la zone de glissement ait été utilisée efficacement, la zone de glissement était encore vide à >50 %. Si l’on tient compte de la proportion de la surface de la lame occupée par des tissus, où le signal était suffisamment complexe, la qualité du signal était excellente.
Des signaux non spécifiques de faible intensité ont également été observés dans deux régions générales de la lame : à l’extérieur des échantillons et à l’intérieur de la notocorde vide (figure 7A). À l’extérieur des sections, il y avait un motif compatible avec un artefact de lavage, qui diminuait à mesure que l’on s’éloignait des spécimens (figure 7B). Un signal non spécifique a également été observé dans les régions vides des spécimens occupés par la notochorde. Cela a été considéré comme un artefact de lavage probable où les réactifs d’amplification pouvaient être piégés. Dans les deux régions, les transcrits étaient principalement cytoplasmiques (c’est-à-dire les protéines ribosomales L3 et L4) ou mitochondriales (aspartate transférase et isocitrate déshydrogénase). Lorsque ces régions ont été incluses dans la segmentation de Baysor, qui identifie les cellules en fonction de la localisation et du regroupement des transcrits autour des noyaux10, l’analyse de segmentation a inclus les limites des cellules, qui les incluaient parfois. Ce signal était suffisamment faible pour qu’il soit facile de le distinguer de l’échantillon.
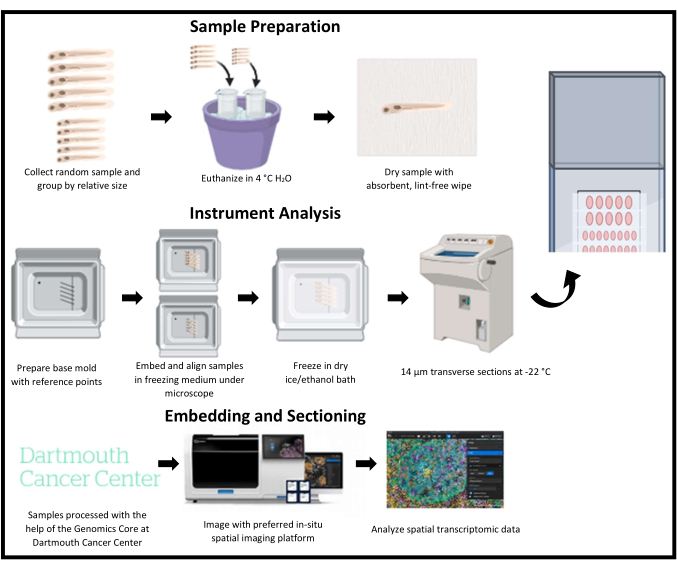
Figure 1 : Schéma de la cryosection pour la transcriptomique spatiale chez le poisson zèbre. Les poissons-zèbres juvéniles sont collectés au hasard, puis regroupés par taille relative. Les échantillons sont ensuite noyés dans un milieu de congélation et alignés par des points de référence sur le moule de base correspondant à une région d’intérêt. Chaque moule est surgelé, cryosectionné et collecté une rangée à la fois sur la même lame. Les lames sont stockées à -80 °C après sectionnement, puis imagées et analysées à l’aide d’une plateforme d’imagerie transcriptomique spatiale. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Établissement de points de référence pour un sectionnement précis. Une série d’images illustre le processus d’ajout de points de référence à un moule de base jetable pour aligner plusieurs échantillons pour la cryosection. La première étape (A) consiste à utiliser du ruban adhésif de laboratoire pour marquer une ligne à l’intérieur du moule que tous les échantillons utiliseront comme référence pour l’alignement. (B) Placez un point à l’intérieur du moule près de la paroi gauche ou droite pour informer de l’orientation correcte de l’échantillon pendant la cryosection, puis (C) marquez des lignes pour chaque échantillon à l’angle souhaité. Veuillez cliquer ici pour voir une version agrandie de cette figure.
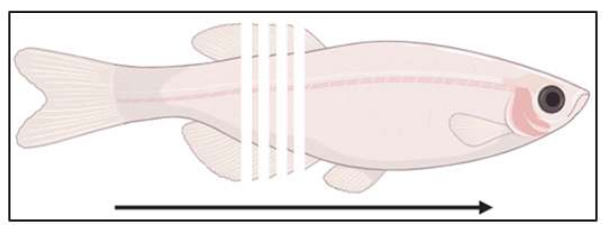
Figure 3 : Schéma de l’emplacement de la section. Les poissons sont sectionnés coronalement, de la caudale au rostral, à des intervalles de 14 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
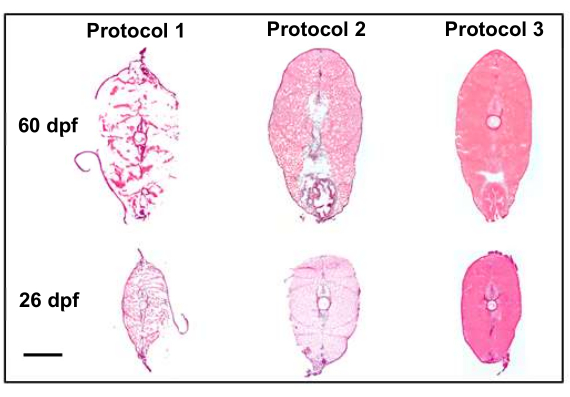
Figure 4 : Amélioration de la qualité de la section avec modifications de protocole (10x). La figure compare les deux premiers protocoles, qui ont entraîné une qualité tissulaire sous-optimale, avec le protocole final, qui a abouti à des coupes de haute qualité (qui ont conservé la morphologie) qui sont de bons candidats pour la transcriptomique spatiale. Le protocole 1 utilise l’analyse comparative 8 du bio-atlasde Penn State, mise en évidence par une fixation de 4 % de paraformaldéhyde avant la section. Le protocole 2 introduit la congélation instantanée de la glace sèche et de l’éthanol après l’euthanasie et la fixation après la section, ce qui préserve beaucoup mieux la structure des tissus avec la mise en garde de la congélation des artefacts tout au long de la section. Le protocole 3 a permis de perfectionner les techniques d’euthanasie et d’enrobage sur glace avant la congélation instantanée, puis de sectionnement et de fixation après la collecte, ce qui a permis d’obtenir des échantillons de la plus haute qualité. La barre d’échelle est de 500 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.

Figure 5 : Vérification de l’alignement de plusieurs sections (20x). Images HE représentatives de 26 sections dpf qui mettent en évidence l’alignement entre plusieurs échantillons en une seule coupe. La barre d’échelle est de 500 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.

Figure 6 : Amélioration de la disposition spatiale des échantillons. Ces panneaux mettent en évidence le nombre de sections collectées dans la zone d’imagerie de chaque lame au fil du temps. L’acquisition d’expérience et l’amélioration des compétences techniques contribuent à l’augmentation du nombre d’échantillons collectés. Ces améliorations étaient également dues en grande partie à de légers ajustements aux étapes d’intégration et de collecte dans le protocole. Le panneau A (40 sections de poissons de 15 dpf) est un premier passage qui utilise un moule lors de la section. Le panneau B (90 sections de poissons de 15 dpf et 47 sections de poissons de 26 dpf) introduit un deuxième moule et montre une amélioration de l’agencement spatial en réduisant la quantité d’espace entre les échantillons lors de l’encastrement. Le panneau C (54 sections de poissons de 15 dpf, 80 sections de 19 poissons de dpf, 24 sections de poissons de 23 dpf et 21 sections de poissons de 26 dpf) introduit un troisième et un quatrième moule et montre une amélioration continue de l’agencement spatial en coupant la surface de coupe du bloc autour des échantillons et en chevauchant le milieu de congélation vide. Les supports de chaque panneau représentent différents moules utilisés lors du sectionnement. Les sections sont sur une zone de 10,5 mm 22 mm. La barre d’échelle est de 4 mm. Veuillez cliquer ici pour voir une version plus grande de cette figure.

Figure 7 : Résultats représentatifs de notre expérience de transcriptomique spatiale. (A) Une seule image d’une diapositive traitée avec le protocole comme le montre la figure 6C. (B) Sections de 14 μm d’un poisson-zèbre à 15 dpf après imagerie transcriptomique spatiale montrant un signal non spécifique adjacent aux sections et à l’intérieur de la notochorde. La barre d’échelle est de 4 mm dans le panneau A. Veuillez cliquer ici pour voir une version plus grande de cette figure.
| 4 % de paraformaldéhyde | Concentration des actions | Quantité | Concentration finale |
| Paraformaldéhyde (poudre) | 95 % p/p | 40 grammes | 4 % p/v |
| NaOH | 1 N | Goutte à goutte (jusqu’à dissolution de la poudre) | |
| Hcl | 1 N | Goutte à goutte (jusqu’à 6,9 pH) | |
| PBS 1x Stock | 1x | ~1000 mL (atteindre un volume total de 1000 mL) | 1x |
| Total | 1000 ml |
Tableau 1 : Formule de paraformaldéhyde à 4 %. Composition d’une solution de paraformaldéhyde à 4 % pour fixer les échantillons.
| 95 % d’éthanol | Concentration des actions | Quantité | Concentration finale |
| Éthanol à 200 degrés | 100 % v/v | 190 ml | 95 % v/v |
| Eau osmosée | 100 % v/v | 10 ml | 5 % v/v |
| Total | 200 ml |
Tableau 2 : Formule à 95 % d’éthanol. Composition d’une solution d’éthanol à 95 % pour déshydrater les sections lors de la coloration H&E.
| 0,3 % d’alcool acidifié | Concentration des actions | Quantité | Concentration finale |
| Acide acétique glacial | 100 % v/v | 300 μL | 0,3 % v/v |
| Eau osmosée | 100 % v/v | 99,7 ml | 99,7 % v/v |
| Total | 100 ml |
Tableau 3 : Formule d’alcool acidifié à 0,3 %. Composition d’alcool acidifié à 0,3 % pour différencier lors de la coloration HE.
| 50 % d’éthanol | Concentration des actions | Quantité | Concentration finale |
| Éthanol à 200 degrés | 100 % v/v | 100 ml | 50 % v/v |
| Eau osmosée | 100 % v/v | 100 ml | 50 % v/v |
| Total | 200 ml |
Tableau 4 : Formule à 50 % d’éthanol. Composition de 50 % d’éthanol pour déshydrater les sections lors de la coloration HE.
Discussion
Ce rapport fournit des solutions détaillées à de nombreux défis techniques associés au poisson-zèbre en tant qu’organisme modèle dans l’analyse transcriptomique spatiale au cours du développement. Pour relever ces défis, notre arrangement compact des échantillons optimise les coûts sur les plateformes transcriptomiques spatiales émergentes1. La cryosection des larves de poisson-zèbre à des fins d’imagerie spatiale est un défi. Les coupes doivent conserver une structure tissulaire et une qualité de transcrit suffisantes pour une exécution expérimentale satisfaisante et une analyse spatiale en aval8. Les coupes contenant plusieurs échantillons pour une imagerie spatialement efficace doivent être alignées avec précision pour analyser les régions d’intérêt communes à tous les échantillons. Enfin, les sections doivent être collectées et espacées efficacement pour maximiser le nombre de points de données potentiels dans la zone désignée d’une lame d’imagerie.
Le protocole permet de collecter des coupes de haute qualité qui sont d’excellents candidats pour les plates-formes d’imagerie spatiale à cellule unique. La première modification a consisté à congeler rapidement le poisson-zèbre, puis à fixer des sections dans du paraformaldéhyde à 4 % après les avoir recueillies sur une lame. À l’origine, les poissons-zèbres étaient fixés avec 4 % de paraformaldéhyde à 4 °C pendant la nuit, puis nâtés et sectionnés après11 °C. Le choix de fixer les échantillons après leur section a permis de préserver les tissus internes qui n’étaient pas efficacement préservés lors de la fixation d’un échantillon entier (Figure 4).
La deuxième modification critique a été d’affiner la méthode d’euthanasie pour préserver l’intégrité des tissus. Les poissons-zèbres ont d’abord été placés dans de l’eau à 4 °C pendant 20 minutes pour s’assurer qu’ils étaient suffisamment euthanasiés. C’est 10 minutes au-delà des exigences minimales des directives de l’IACUC. Ce temps supplémentaire dans une eau à 4 °C a donné aux échantillons plus de temps pour se décomposer et a contribué à une qualité de section moins qu’idéale des structures internes très fines ou délicates. La réduction du temps passé dans une eau à 4 °C au minimum requis de 10 minutes a permis de préserver les structures internes délicates qui manquaient lors des tentatives de coupe précédentes. D’autres modifications non illustrées à la figure 4 comprennent l’épilation le même jour que l’euthanasie, la réduction du temps entre l’euthanasie et la congélation instantanée de 15 minutes à moins de 5 minutes, et l’utilisation d’un produit de congélation pré-réfrigéré lors de l’enrobage. Plus les échantillons étaient conservés longtemps à -80 °C avant la section, plus la qualité des tissus de la section finissait par se détériorer. Les différences étaient subtiles, mais il est idéal pour la sectionnement le même jour que l’euthanasie et la création de blocs. Réduire le temps nécessaire à l’alignement des échantillons après l’euthanasie et avant la congélation éclair à un maximum de 5 minutes est essentiel à la qualité de la section.
L’alignement de plusieurs échantillons sur une région d’intérêt a également nécessité quelques modifications du protocole. La précision de l’alignement obtenue (figure 5) a permis de regrouper les échantillons par taille relative et a fourni la plus grande contribution à une organisation spatiale efficace. La principale stratégie d’alignement des échantillons consiste à superposer le poisson-zèbre sur des points de référence qui sont dessinés sur les moules de base dans lesquels ils sont intégrés. Les spécimens sont alignés sur ces points de référence par des structures anatomiques à l’intérieur des spécimens du poisson-zèbre qui peuvent varier en fonction de la région d’intérêt souhaitée. La taille des poissons en développement du même âge peut varier jusqu’à 50 %12. Des échantillons de tailles radicalement différentes peuvent se désaligner en raison des distances variables entre le point de référence anatomique et la région d’intérêt. Le regroupement d’échantillons du même âge par taille relative et l’intégration de chaque taille dans leurs moules ont amélioré la capacité d’aligner plusieurs échantillons dans leur moule. Cette méthode permet d’aligner de manière fiable environ 2/3 des échantillons en fonction de la fenêtre permise par la région d’intérêt et le choix du point de référence anatomique.
La réduction de l’espace entre les échantillons lors de l’enrobage et de l’alignement permet d’obtenir un plus grand nombre d’échantillons par lame. Cependant, cette configuration étroite est encore limitée par les limites de repère sur la zone d’imagerie. En plus de l’expérience au cryostat, plusieurs stratégies pour les concilier sont décrites. Tout d’abord, le placement précis du milieu d’enrobage qui se chevauche entre les sections et le découpage du moule congelé peuvent avoir des impacts profonds sur le nombre de sections pouvant tenir dans une zone d’imagerie spécifique. La figure 6 met en évidence le nombre de sections obtenues avant et après l’optimisation de l’agencement d’encastrement. Deuxièmement, une limite a été placée à l’extérieur des échantillons les plus externes pendant l’enrobage et avant la congélation pour faciliter le placement correct sur la lame d’imagerie. Troisièmement, la taille et la nature transparente des larves de poisson-zèbre peuvent rendre difficile de savoir où se trouvent les échantillons dans une lame afin qu’ils puissent être collectés avec précision sur une lame. Nous avons constaté que l’utilisation de papier coloré pour contraster le médium de congélation blanc afin de créer une limite à l’extérieur des échantillons les plus externes conduisait à un placement beaucoup plus précis des sections sur les lames.
Les nuances de la température locale aident à la conservation de l’échantillon pendant le processus de découpe. Une fois que les échantillons de plusieurs moules sont collectés sur une seule lame, la lame d’imagerie est stockée à 4 °C, le moule suivant est coupé à la région d’intérêt souhaitée avant d’être collecté à nouveau. La lame d’imagerie doit être légèrement plus chaude que les sections pour qu’elle adhère correctement à la surface de la lame. Ainsi, le stockage de la lame d’imagerie à -80 °C ou dans le cryostat à -20 °C entre le prélèvement d’échantillons dans différents moules n’est pas idéal. Il est également essentiel que les échantillons collectés sur la lame d’imagerie ne subissent pas plusieurs cycles de congélation/décongélation, car cela peut avoir un impact négatif sur la qualité des transcrits d’ARN dans chaque échantillon. Le fait de maintenir la lame d’imagerie avec les sections précédemment collectées à 4 °C permet de préserver adéquatement les transcrits d’ARN tout en maintenant une température plus basse pour une bonne adhérence sans effets secondaires négatifs associés aux cycles de gel/dégel13.
Bien que des résultats satisfaisants aient été obtenus, la conception de cette méthode comporte des limites possibles. La première limitation est que nous sommes limités à la zone d’imagerie d’une lame transcriptomique spatiale, ce qui limite le nombre total de sections pouvant être imagées sur la même lame. Étant donné que nous avons encore un espace vide entre les sections, il est probable que nous n’ayons pas encore atteint les limites de ce qui est possible pour cette méthode, mais nous reconnaissons qu’il y a un point où il n’est plus possible d’insérer d’autres sections sur la lame d’imagerie. Travailler avec des poissons plus gros à un âge plus avancé limitera également la taille totale de l’échantillon, mais ne devrait pas affecter la quantité totale de tissu qui peut tenir dans la zone d’imagerie d’une lame. Les améliorations techniques apportées par les plates-formes d’imagerie spatiale pourraient éventuellement augmenter la quantité totale de tissus qui pourraient être imagés en élargissant la zone d’imagerie sur les lames d’imagerie. Une autre limitation de cette méthode est que le protocole n’a pas été optimisé pour les poissons de moins de 15 dpf. Il a été constaté que les poissons sont de plus en plus fragiles à mesure qu’ils sont jeunes et que les poissons de moins de 15 dpf étaient extrêmement difficiles à manipuler et étaient facilement endommagés pendant le processus d’encastrement. Cela a entraîné une augmentation du temps entre l’euthanasie et la congélation éclair, ce qui a nui à la qualité des tissus. Cela a également réduit le pourcentage d’échantillons viables laissés pour la cryosection, et les quelques poissons viables qui sont restés pour la section n’étaient pas assez rigides pour conserver la structure tissulaire pour une analyse de qualité. Trouver des outils mieux adaptés à la manipulation de poissons plus fragiles et travailler avec un milieu de congélation moins dense qui augmente également la rigidité des échantillons lors de la section pourrait permettre d’appliquer cette méthode aux poissons de moins de 15 dpf. La dernière limitation majeure de cette méthode est l’alignement de l’échantillon pour de très petites régions d’intérêt. Nous avons constaté que plus la région d’intérêt de l’échantillon était petite, moins il était probable que nous puissions les aligner correctement. Nous avons travaillé avec une région d’intérêt d’environ 50 μm, ce qui a permis d’aligner correctement environ 2/3 des sections. Les échantillons dont la région d’intérêt est inférieure à 50 μm peuvent entraîner l’alignement de moins de 2/3 des sections. La majorité des échantillons mal alignés proviennent de l’étape d’intégration dans le protocole. Il est difficile d’ajuster un échantillon dans le milieu de congélation sans déplacer d’autres échantillons déjà alignés. Le chercheur dispose également d’un temps limité pour aligner ces échantillons avant d’avoir un impact négatif sur la qualité des tissus. Enfin, les moules sont déplacés du stéréomicroscope au bain de congélation instantanée, et il est possible que les échantillons se déplacent dans le moule avant d’être complètement congelés. Il est possible que le travail avec un milieu de congélation plus froid permette au chercheur de déplacer un échantillon plus facilement sans affecter les autres échantillons déjà alignés. La congélation instantanée du moule sans avoir à le déplacer après l’alignement pourrait éliminer le déplacement de l’échantillon avant qu’il ne soit complètement figé en place. Nous avons identifié des signaux non spécifiques à l’extérieur des échantillons.
Le produit final et la post-analyse initiale ont présenté des leçons supplémentaires. Des signaux non spécifiques étaient présents, qui nécessitaient une prise en compte. Selon le fabricant, certains transcrits à faible signal sont un artefact connu et l’ARN ambiant peut être observé dans le séquençage de l’ARN unicellulaire qui peut être géré après l’analyse14. Le protocole utilisé ici peut avoir contribué à l’obtention de transcrits à l’air ambiant, car la fixation s’est produite après la mise en place des échantillons. Cependant, il a été constaté qu’une fixation précoce conduisait à une morphologie médiocre et incohérente pour ces jeunes âges. Alternativement, les réactifs d’amplification qui se regroupent pourraient créer un tel effet. Qualitativement, le signal non spécifique observé dans la notocorde était le plus prononcé. Pendant le traitement, la notocorde était la seule région vraiment vide car elle ne contenait pas de milieux de montage et de signaux de transcription, qui ressemblaient aux profils cytoplasmiques et mitochondriaux des tissus adjacents de la paroi de la notochorde. Cela suggère que le « support » physique (par des tissus ou des supports de montage) est utile pour réduire ce signal. En fin de compte, nous pensons que l’exclusion du signal en post-traitement est une stratégie satisfaisante. Les caractéristiques des signaux non spécifiques étaient facilement identifiables après les expériences et pouvaient être atténuées lors de l’analyse en aval, par exemple par l’exclusion manuelle des signaux non spécifiques de régions situées à l’extérieur de l’échantillon.
Des poissons-zèbres aussi jeunes que 15 dpf provenant d’au moins 4 moules différents (et âges) peuvent être cryosectionnés et collectés avec succès dans la zone d’imagerie transcriptomique spatiale, et jusqu’à 179 sections peuvent être traitées simultanément. Sur la base de la qualité de la section, de l’alignement de l’échantillon, de la taille de l’échantillon et de la qualité des transcrits d’ARN pendant l’imagerie, nous concluons que cette méthode est un protocole efficace pour réaliser des tests transcriptomiques spatiaux efficaces. Les chercheurs peuvent utiliser cette méthode pour réduire les coûts associés à l’imagerie spatiale, augmenter la taille de l’échantillon lors des tests transcriptomiques spatiaux et même réaliser de grandes expériences en une seule fois.
Déclarations de divulgation
Les auteurs n’ont aucune divulgation ou conflit d’intérêts concernant ce rapport.
Remerciements
La coupe et l’imagerie ont été réalisées à l’aide d’instruments fournis par des ressources partagées du Dartmouth Cancer Center, financées par la subvention de soutien du NCI Cancer Center 5P30CA023108, et du Center for Quantitative Biology du Dartmouth College (NIGMS COBRE).
matériels
| Name | Company | Catalog Number | Comments |
| 1 L Beaker | Pyrex | 1003 | |
| 200 proof pure ethanol | Koptec | V1001 | |
| Acetic acid, glacial | VWR | 0714 | acidified alcohol |
| Aluminum foil | |||
| Cover slips | Epredia | 24X50-1.5-001G | |
| Disposable base mold | Fisher HealthCare | 22-363-556 | |
| Distilled water | |||
| DPX mountant | Sigma-Aldrich | 06522 | mountant for histology |
| Dry ice pellets | |||
| Dumont #5SF Forceps | Fine Science Tools | 11252-00 | |
| Eosin-Y Alcoholic | Epredia | 71204 | Eosin Y 1% |
| Gill 1 Hematoxylin | Epredia | 72411 | Hematoxylin |
| Kimwipe | Kimberly-Clark Professional | 34120 | absorbent, lint-free wipe |
| Lab labelling tape | VWR | 89097-934 | |
| Microtome blade MX35 Ultra | Epredia | 3053835 | |
| Microtome Cryostat | Thermo Scientific | Microme HM 525 | |
| O.C.T. Compound | Fisher HealthCare | 23-730-571 | freezing medium |
| Paraformaldehyde | Sigma-Aldrich | 158127 | PFA |
| Permanent Marker | VWR | 52877-886 | |
| Protractor | |||
| SafeClear Xylene Substitute | Fisherbrand | 68551-16-6 | Xylene substitute |
| Single Edge Blades | American Line | 66-0407 | |
| Steriomicroscope | Zeiss | 4350639000 | Stemi 305 w/ double spot LED (4355259020) and Stand K lab (4354259010) |
| Superfrost Plus Micro Slides | VWR | 48311-703 | |
| Transfer pipet | |||
| Xenium V1 slide | 10X/Xenium | 3000941 | spatial transcriptomic imaging slide |
Références
- Rao, A., Barkley, D., França, G. S., Yanai, I. Exploring tissue architecture using spatial transcriptomics. Nature. 596 (7871), 211-220 (2021).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet. 22, 627-644 (2021).
- Duhan, L., et al. Single-cell transcriptomics: background, technologies, applications, and challenges. Mol Biol Rep. 51, 600 (2024).
- Ståhl, P. L., et al. Visualization and analysis of gene expression in tissue sections by spatial transcriptomics. Science. 353 (6294), 78-82 (2016).
- Colman, R. E., et al. Whole-genome and targeted sequencing of drug-resistant Mycobacterium tuberculosis on the iSeq100 and MiSeq: A performance, ease-of-use, and cost evaluation. PLOS Med. 16 (4), e1002194 (2019).
- Marques, I. J., Lupi, E., Mercader, N. Model systems for regeneration: zebrafish. Development. 146 (18), dev167692 (2019).
- Mohideen, M. P. K., et al. Histology-based screen for zebrafish mutants with abnormal cell differentiation. Dev Dyn. 228 (3), 414-423 (2003).
- Copper, J. E., et al. Comparative analysis of fixation and embedding techniques for optimized histological preparation of zebrafish. Comp Biochem Physiol C Toxicol Pharmacol. 208, 38-46 (2018).
- Stock, R. J., Labudovich, M., Ducatman, B. Asymptomatic first-trimester liver cell adenoma: diagnosis by fine-needle aspiration cytology with cytochemical and ultrastructural study. Obstet Gynecol. 66 (2), 287-290 (1985).
- Petukhov, V., et al. Cell segmentation in imaging-based spatial transcriptomics. Nat Biotechnol. 40 (3), 345-354 (2022).
- Meyer, T., Tiburcy, M., Zimmermann, W. H. Cardiac macrotissues-on-a-plate models for phenotypic drug screens. Adv Drug Deliv Rev. 140, 93-100 (2019).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Dev Dyn. 203 (3), 253-310 (1995).
- Yu, K., Xing, J., Zhang, J., Zhao, R., Zhang, Y., Zhao, L. Effect of multiple cycles of freeze-thawing on the RNA quality of lung cancer tissues. Cell Tissue Bank. 18 (3), 433-440 (2017).
- Yang, S., et al. Decontamination of ambient RNA in single-cell RNA-seq with DecontX. Genome Biol. 21 (1), 57 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon