Method Article
Disposición espacialmente compacta de secciones de larvas de pez cebra para análisis transcriptómico espacial
En este artículo
Resumen
Aquí, presentamos un método para alinear y crioseccionar múltiples muestras de larvas de pez cebra (Danio rerio) y recolectarlas en un solo portaobjetos para el análisis transcriptómico espacial.
Resumen
Las técnicas transcriptómicas espaciales son una herramienta sofisticada en la investigación biomédica para visualizar patrones de expresión génica registrados espacialmente. La obtención de imágenes y el análisis de múltiples muestras con plataformas de imágenes espaciales pueden ser costosos. La realización de estas pruebas en múltiples condiciones experimentales, como se ha visto en los estudios de desarrollo, aumenta aún más los costos. Para reducir costos, este estudio buscó optimizar las técnicas y estrategias de disposición de muestras transcriptómicas espaciales para estudios de desarrollo. Aquí, el estudio utilizó peces cebra, que son un modelo de vertebrados en desarrollo bien establecido que es transparente durante el desarrollo, tienen ~ 70% de homología genética con los humanos y un genoma altamente anotado ideal para el análisis transcriptómico. Debido a su pequeño tamaño, el pez cebra en desarrollo también permite la colocación compacta de secciones en serie a través de varias réplicas biológicas. En este artículo, informamos sobre la fijación optimizada, la criosección y la alineación confiable de múltiples muestras de peces dentro del área de imágenes de una plataforma de imágenes espaciales de hibridación in situ múltiple . Con este método, el pez cebra de tan solo 15 días después de la fertilización (dpf) de al menos 4 moldes diferentes y hasta 174 secciones puede crioseccionarse con éxito, recolectarse dentro del área de imagen de 22 mm 10,5 mm (para un portaobjetos transcriptómico espacial in situ ) y procesarse simultáneamente. En función de la calidad de la sección, la alineación de la muestra y el tamaño de la muestra por portaobjetos, este método en el pez cebra optimiza la salida y el coste por muestra de las técnicas transcriptómicas espaciales.
Introducción
La evaluación de patrones de expresión espacialmente distintos en los tejidos sigue siendo fundamental para nuestra comprensión de las influencias genómicas en el desarrollo, el cáncer y la enfermedad 1,2,3. La transcriptómica espacial combina técnicas de expresión multiplexada con el registro espacial de la expresión en tejidos. La "transcriptómica espacial" fue acuñada por primera vez por Ståhl y sus colegas4, donde los especímenes de cáncer montados se sondearon utilizando secuenciación de próxima generación in situ. Desde entonces, la "transcriptómica espacial" se ha utilizado como un cajón de sastre para los estudios de expresión de alto rendimiento combinados con el registro espacial. Si bien se trata de herramientas poderosas, también son empresas costosas que a menudo requieren grandes inversiones institucionales y costos de laboratorio antes de que se puedan generar datos5. Las estrategias para minimizar los costos y preservar los datos de alta calidad tienen una gran demanda.
El pez cebra, Danio rerio, se ha convertido en un importante sistema modelo para el estudio de la biología del desarrollo y ofrece un medio para multiplicar los análisis de órganos (y organismos) completos de vertebrados en un espacio limitado. Los peces cebra son pequeños (4-6 mm como larvas y 2-3 cm como adultos) y pueden poner cientos de huevos transparentes a la vez6. Los embriones de pez cebra se fertilizan externamente y se desarrollan rápidamente, lo que permite a los investigadores introducir transgenes en etapas tempranas de desarrollo para generar fácilmente alelos de ganancia y pérdida de función7. Colocar varios especímenes en un solo portaobjetos es una estrategia atractiva para reducir costos. Su alta fecundidad y pequeño tamaño hacen del pez cebra un candidato ideal para ensayos transcriptómicos espaciales multiplexados que tienen un espacio restringido para los especímenes8.
La criosección de larvas de pez cebra es una técnica desafiante. Muchas plataformas transcriptómicas espaciales no se han optimizado para las secciones de parafina del pez cebra y requieren criosecciones cuando se trabaja con el pez cebra como organismo modelo para preservar la estructura del tejido y retener las transcripciones de ARN. Además, el pequeño tamaño del pez cebra dificulta la obtención de criosecciones de calidad y el análisis eficaz de múltiples muestras. Esta tarea se vuelve más difícil cuando se trabaja con larvas de pez cebra que son más pequeñas y frágiles que sus contrapartes adultas. Para superar estos desafíos, describimos un método que alinea de manera confiable múltiples muestras y utiliza el área de imágenes de las plataformas de imágenes espaciales de manera eficiente para obtener muchas secciones de alta calidad en un solo portaobjetos que luego pueden ser visualizadas y analizadas por plataformas de imágenes espaciales (Figura 1). En este caso, este método se aplica a una plataforma de imágenes transcriptómicas espaciales.
Protocolo
Este protocolo sigue las pautas del comité institucional de cuidado y uso de animales de Dartmouth College.
1. Preparación del criostato
- Enfríe el criostato a -22 °C y limpie las superficies interiores del criostato cepillando los residuos en el receptáculo. Coloque todos los cepillos y herramientas necesarios dentro de la cámara.
2. Preparación del molde base desechable
- Prepare un molde base (molde de plástico desechable de 37 mm, 24 mm, 5 mm) para la alineación de muestras dibujando una línea recta a través del interior de un molde base con un marcador permanente para usarlo como punto de referencia para la alineación de muestras. Coloque un pedazo de cinta de laboratorio en el interior del molde base donde debe estar la línea recta antes de dibujar una línea para obtener mejores resultados (Figura 2A).
- Dibuje un punto en el interior del molde base cerca de la pared izquierda o derecha para garantizar la orientación adecuada de la muestra durante la criosección (Figura 2B).
- Mida el ángulo de corte deseado con un transportador y márquelo en el interior del molde base para cada muestra (Figura 2C).
- Aplique una capa superficial de medio de congelación (tomografía de coherencia óptica [OCT]) al molde base preparado. Asegúrese de que haya suficiente medio de congelación para cubrir las muestras.
- Evite las burbujas de aire al aplicar el medio de congelación cebando la boquilla de la botella mediana y agregando la cantidad necesaria de medio a una esquina del molde base antes de cambiar el plano horizontal del molde base para que el medio se distribuya uniformemente por toda la superficie.
- Apriete el frasco lentamente cuando dispense el medio de congelación.
- Agregue hielo a un vaso de precipitados de 1 L, coloque el molde base con medio de congelación en el baño de hielo e incube durante al menos 10 minutos para enfriar el medio.
3. Preparación de hielo seco: baño de etanol 100%
- Prepare un baño de hielo seco y etanol 100% en una campana extractora agregando una parte de etanol 100% a una parte de hielo seco en un balde de hielo.
- Use un plato de aluminio desechable o doble el papel de aluminio en un bote lo suficientemente grande como para caber en un molde de base desechable. Asegúrese de que el plato o bote sea lo suficientemente grande para que el molde base quede completamente plano.
- Coloque el plato o el bote en la bañera y cubra el balde. Espere de 5 a 10 minutos para que el balde se enfríe antes de congelar las muestras.
4. Eutanasia de muestras
- Seleccione al azar el pez cebra para seccionar. Si las muestras varían en tamaño, sepárelas en grupos por tamaño relativo para facilitar la alineación precisa (consulte la discusión para obtener más detalles). Coloque los peces más grandes y los peces más pequeños en platos separados.
- Llene un vaso de precipitados con agua del sistema de peces y colóquelo en un cubo de hielo. Rodea el vaso de precipitados con hielo.
- Controle la temperatura del agua con un termómetro. Deje que la temperatura se estabilice entre 2-4 °C.
- Use una red o un colador para poner un grupo de peces en el agua a 4 ° C. Los peces deben estar completamente sumergidos en el agua y no en contacto con el hielo. Una vez que haya cesado el movimiento opercular, agregue hielo al agua para asegurarse de que permanezca por debajo de los 4 ° C. Deje los peces en agua a 4 °C durante 10 min.
NOTA: Continúe con la inclusión, alineación y congelación instantánea de cada grupo de muestras eutanasiadas antes de sacrificar al siguiente grupo. Reemplace el agua cada vez.
5. Incrustación y alineación
- Recoja los peces sacrificados después de haberlos sumergido en agua a 4 °C durante 10 minutos. Retire los peces del agua con pinzas de punta fina agarrándolos por la aleta caudal y séquelos presionándolos suavemente contra una toallita absorbente que no suelte pelusa.
NOTA: En el caso de las secciones de alta calidad, es fundamental limitar el tiempo entre la extracción del pescado del agua a 4 °C y la congelación instantánea - Trabajando con el molde base preparado en un baño de hielo bajo un microscopio estereoscópico (aumento de 10x), coloque cada muestra en el molde base a lo largo de los puntos de referencia en la orientación correcta y cubra suavemente las muestras con otra capa delgada de medio de congelación.
NOTA: No llene todo el molde con medio de congelación. En su lugar, use solo una capa delgada, lo suficiente para cubrir todas las muestras. - Use un punto de referencia anatómico para alinear con precisión el pez con las líneas marcadas en el interior del molde base. Use pinzas de punta fina para ajustar la orientación de cada pez de modo que estén alineados y en la misma orientación. Evite crear burbujas en el medio de congelación moviéndose lentamente.
- Aplique un trozo de hielo seco en el fondo del molde base debajo de las muestras hasta que se congelen localmente en su posición. Mantenga el plano horizontal del molde base nivelado para evitar que las muestras se desvíen de sus puntos de referencia antes de congelarlas en su posición con hielo seco.
- Coloque el molde base con las muestras en el bote o plato de aluminio en el baño de hielo seco: 100% etanol. Asegúrese de que el bote esté flotando en la superficie del baño y que el molde base permanezca seco. Cubra el baño y deje que las muestras floten durante 10 minutos.
- Envuelva el molde base congelado en papel de aluminio y guárdelo en el congelador a -80 °C hasta que esté listo para seccionar. Repita los pasos 4.2 a 5.6 para los grupos restantes.
6. Crioseccionamiento
- Lleve los moldes base congelados al criostato preenfriado para la crisección y colóquelos en la cámara de criostato. Transporta los bloques congelados en una caja con hielo seco para evitar que se descongelen.
- Retire el portaobjetos de imágenes espaciales in situ del almacenamiento y colóquelo en un soporte de portaobjetos preenfriado. Guarde el portaobjetos con portaobjetos de imágenes espaciales en la cámara de criostato a -22 °C hasta que esté listo para recoger secciones de la región de interés.
- Retire las muestras congeladas del molde base y luego congélelas en un mandril con un medio de congelación fresco. Congélelos en el mandril de modo que la superficie de corte quede orientada hacia la hoja.
- Coloque una cuchilla de micrótomo nueva y fina en el criostato.
- Alinee el molde con la cuchilla y recorte la región de interés (espesor de corte recomendado 20-50 μm). Asegúrese de que las marcas hechas dentro del molde se transfieran al bloque de muestras congeladas para ayudar a identificar la ubicación dentro de las muestras.
- Ajuste el molde durante la fase de recorte para que la superficie de corte quede paralela a las marcas de referencia en el medio de congelación.
- Recoja secciones en un portaobjetos de microscopio estándar con carga positiva y compruébelas mediante campo claro para confirmar cuándo ya no es necesario recortar.
- Retire el portaobjetos de imágenes espaciales de la cámara de criostato y colóquelo en un baño de hielo a 4 °C. Mantenga la corredera en el soporte de la corredera. Asegúrese de que el tobogán no se moje.
- Iniciar la criosección (recomendado 10-14 μm; Figura 3) y recoja secciones en portaobjetos cargados positivamente hasta que se alcance la región de interés en las muestras. Verifique las secciones por campo claro para confirmar que la región de interés será la siguiente sección del molde.
- Lleve el portaobjetos de imágenes espaciales de una sola célula a la cámara de criostato. Retire la diapositiva del soporte de diapositivas y recoja secciones del área exacta de interés en la diapositiva de imágenes espaciales fila por fila con un pincel de punta fina para evitar que las secciones se enrollen.
- Presione una esquina del medio vacío y congelado en la etapa del cuchillo con la parte posterior del pincel para que la sección permanezca plana al agarrar el portaobjetos para recogerlo.
- Use la parte superior de la corredera como punto de pivote, baje lentamente la corredera sobre la sección y permita que las secciones se adhieran a la corredera durante 3 segundos antes de levantar la corredera de la etapa de la cuchilla.
- Trabaje de izquierda a derecha cuando recoja secciones en el área de imagen del portaobjetos y superponga capas de medio vacío y congelado cuando sea posible.
- Use bordes de papel de colores como referencia para colocar secciones dentro del área de imagen del portaobjetos si el tejido es difícil de ver.
- Después de recoger secciones de la región de interés, vuelva a colocar la corredera en el soporte de la corredera. Si no hay más muestras para recolectar en el portaobjetos de imágenes espaciales, almacene el portaobjetos a -80 °C durante un máximo de 2 semanas hasta que esté listo para el análisis instrumental con la plataforma de imágenes espaciales in situ . Si es necesario recoger secciones de varios moldes, vuelva a colocar el portaobjetos de imágenes espaciales en el baño de hielo a 4 °C y repita los pasos 6.3-6.11 con el siguiente molde.
- Recoja las secciones antes y después de las secciones de la región de interés de cada molde en un portaobjetos de microscopio estándar con carga positiva para la tinción de hematoxilina y eosina (HE) para verificar que la alineación de la muestra y la calidad de la sección sean suficientes antes de continuar con el análisis.
7. Fijación de la muestra
- Retire los portaobjetos de referencia del criostato y séquelos al aire en RT durante 30 minutos para que se adhieran las secciones al portaobjetos.
- Fije las secciones colocándolas en un recipiente con 4% de paraformaldehído (Tabla 1) durante 20 min.
- Lave las secciones colocándolas en un recipiente deslizante con agua destilada durante 3 min.
- Continúe con la tinción HE o seque y guarde los portaobjetos a -80 °C para futuras tinciones.
8. Tinción HE de las secciones
- Deshidrate y limpie las secciones incubando los portaobjetos en etanol al 100% durante 2 minutos, etanol al 95% (Tabla 2) durante 2 minutos y luego agua del grifo durante 1 minuto. Use una gradilla de tinción de portaobjetos de microscopio para transferir los portaobjetos de un baño a otro.
- Tiñir los núcleos y diferenciar incubando los portaobjetos en hematoxilina durante 2 min 45 s, agua del grifo durante 1 min, alcohol acidificado al 0,3% (Tabla 3) durante 1 min, y luego agua corriente del grifo durante 1 min. Use una gradilla de tinción de portaobjetos de microscopio para transferir los portaobjetos de un baño a otro.
- Tiñir los componentes citoplasmáticos y deshidratar incubando los portaobjetos en eosina Y al 1% durante 45 s, etanol al 50% (Tabla 4) durante 1 min, etanol al 95% durante 1 min y etanol al 100% durante 1 min. Use una gradilla de tinción de portaobjetos de microscopio para transferir los portaobjetos de un baño a otro.
- Limpie las secciones incubando portaobjetos en xileno durante 1 min. Monte y cubra los portaobjetos aplicando una gota de medio de montaje en el tercio superior del portaobjetos con una pipeta de transferencia y bajando lentamente un cubreobjetos sobre el medio de montaje con pinzas.
9. Imágenes transcriptómicas espaciales y análisis de las secciones
- Retire las diapositivas de imagen del almacenamiento a -80 °C y obtenga imágenes con una plataforma de imágenes espaciales in situ para el análisis transcriptómico espacial.
NOTA: Los pasos exactos involucrados en la obtención de imágenes serán determinados por la plataforma de imágenes espaciales. - Revise las métricas de control de calidad de la plataforma transcriptómica espacial. Las métricas importantes que se deben comprobar son el número de células detectadas, la mediana de transcripciones por célula, las transcripciones nucleares por 100 μm2 y el total de transcripciones decodificadas de alta calidad de cada gen en el conjunto de sondas.
NOTA: Estas métricas de control de calidad no tienen umbrales universales, y las expectativas para estos umbrales variarán según la muestra y el panel de genes que se utilicen. - Lea la salida de datos de las transcripciones de ARN detectables, determine qué transcripciones son de baja calidad en función de las métricas de control de calidad del experimento y filtre las transcripciones de baja calidad. Analice las transcripciones de alta calidad restantes en relación con su disposición espacial dentro de la sección.
- Vea la segmentación celular de las secciones y agrupe las células en función de los intereses experimentales. Compare las transcripciones de ARN dentro de los grupos de células de la región de interés en grupos de diferentes edades de pez cebra en el mismo portaobjetos.
Resultados
En este método (Figura 1), el pez cebra se utiliza como modelo animal para sondear los patrones de expresión génica resueltos espacialmente. La criosección eficiente de larvas de pez cebra para la obtención de imágenes espaciales es un desafío. Las secciones deben ser de alta calidad para conservar la estructura del tejido y los genes detectables (Figura 4). Las secciones que contienen múltiples muestras para obtener imágenes espacialmente eficientes deben alinearse con precisión para analizar las regiones de interés en todas las muestras (Figura 2 y Figura 5). Finalmente, las secciones deben recopilarse y espaciarse de manera eficiente para maximizar el número de puntos de datos potenciales dentro del área de imagen de un portaobjetos de transcriptómica espacial (Figura 3 y Figura 6). Los experimentos se midieron utilizando estos tres parámetros para determinar si eran exitosos o subóptimos.
Muchas modificaciones del protocolo contribuyeron a nuestra capacidad para recopilar secciones de alta calidad que son buenas candidatas para la obtención de imágenes transcriptómicas espaciales. Ajustar los protocolos de eutanasia, fijación e inclusión para mejorar la calidad de la sección (Figura 4). En el primer protocolo (Figura 4, protocolo 1), la calidad de la sección fue subóptima y los tejidos no conservaron su estructura. Para corregir esto, en el segundo protocolo (Figura 4, protocolo 2), se introdujo un paso de congelación instantánea después de incrustar muestras y tejido fijo después de recolectar secciones en portaobjetos. Esto mejoró la estructura tisular general de las secciones, pero no logró alcanzar lo que se pensaba que era una calidad aceptable para la transcriptómica espacial. Finalmente, en el tercer protocolo (Figura 4, protocolo 3), las técnicas de eutanasia se refinaron acortando la cantidad de tiempo que se tardaba en sacrificar peces e incorporándolos en hielo antes de la congelación instantánea y el posterior seccionamiento y fijación posteriores a la recolección. Esto dio como resultado muestras de la más alta calidad que eran buenas candidatas para la obtención de imágenes transcriptómicas espaciales.
La alineación de varias muestras con una región de interés también requirió algunas modificaciones en el protocolo. Una de las modificaciones que más mejoró la alineación entre muestras fue la introducción de puntos de referencia a los moldes base en los que se incrustan los peces cebra (Figura 2). En los experimentos realizados en este estudio, las muestras se alinearon comparando estructuras a través de múltiples secciones en el mismo corte (Figura 5).
El último paso importante en el desarrollo del método para una transcriptómica espacial eficiente fue encajar la mayor cantidad posible de estas muestras alineadas de alta calidad de diferentes edades en el mismo portaobjetos para la obtención de imágenes. La adquisición de experiencia y la mejora de las habilidades técnicas contribuyen a un mayor número de muestras que se recogen. Estas mejoras también se debieron en gran medida a ligeros ajustes en los pasos de incrustación y recopilación en el protocolo. Estos paneles de la Figura 6 resaltan el número de secciones recopiladas en el área de imagen de cada portaobjetos a lo largo del tiempo. La Figura 6A (40 secciones de peces de 15 dpf) es una tirada temprana que utiliza un molde durante el corte. La Figura 6B (90 secciones de peces de 15 dpf y 47 secciones de peces de 26 dpf) introduce un segundo molde y muestra una mejora en la disposición espacial al reducir la cantidad de espacio entre las muestras durante la incrustación. La Figura 6C (54 secciones de peces de 15 dpf, 80 secciones de peces de 19 dpf, 24 secciones de peces de 23 dpf y 21 secciones de peces de 26 dpf) presenta un tercer y cuarto molde y muestra una mejora continua en una disposición espacial mediante el recorte de la superficie de corte del bloque alrededor de las muestras y la superposición de medio vacío y congelado. Estas mejoras nos dieron la oportunidad de colocar 179 secciones en el área de imágenes de nuestro portaobjetos transcriptómicos espaciales y probar peces de 4 edades diferentes.
Al finalizar el protocolo, disponíamos de muestras que eran fácilmente analizables tanto con el software multiplexado in situ disponible en el mercado como con métodos personalizados. El software multiplex utilizado aquí empleó una puntuación pseudo-phred, basada vagamente en las puntuaciones de calidad phred en los datos de secuenciación9. Las puntuaciones oscilaron entre el 59% y el 70%, siendo el 60% el límite para una advertencia de lecturas de menor calidad. Tras la inspección, la baja puntuación de pseudo-phred se debió a la baja complejidad de la señal en el espacio vacío entre las secciones. Aunque el área de deslizamiento se utilizó de manera eficiente, el área de deslizamiento todavía estaba vacía en un >50%. Al tener en cuenta la proporción del área de deslizamiento ocupada por el tejido, donde la señal era lo suficientemente compleja, la calidad de la señal fue excelente.
También se observaron señales inespecíficas de baja intensidad en dos regiones generales del portaobjetos: fuera de los especímenes y dentro de la notocorda vacía (Figura 7A). Fuera de las secciones, había un patrón consistente con un artefacto de lavado, que disminuiría al alejarse de los especímenes (Figura 7B). También se observó una señal inespecífica dentro de las regiones vacías de los especímenes ocupadas por la notocorda. Esto se consideró un probable artefacto de lavado en el que los reactivos de amplificación podrían quedar atrapados. Las transcripciones en ambas regiones fueron en gran medida citoplasmáticas (es decir, proteínas ribosómicas L3 y L4) o mitocondriales (aspartato transferasa e isocitrato deshidrogenasa). Cuando estas regiones se incluyeron en la segmentación de Baysor, que identifica las células en función de la localización y el agrupamiento de las transcripciones alrededor de los núcleos10, el análisis de segmentación incluyó los bordes de las células, que a veces los incluían. Esta señal era lo suficientemente baja como para que se distinguiera fácilmente del espécimen.
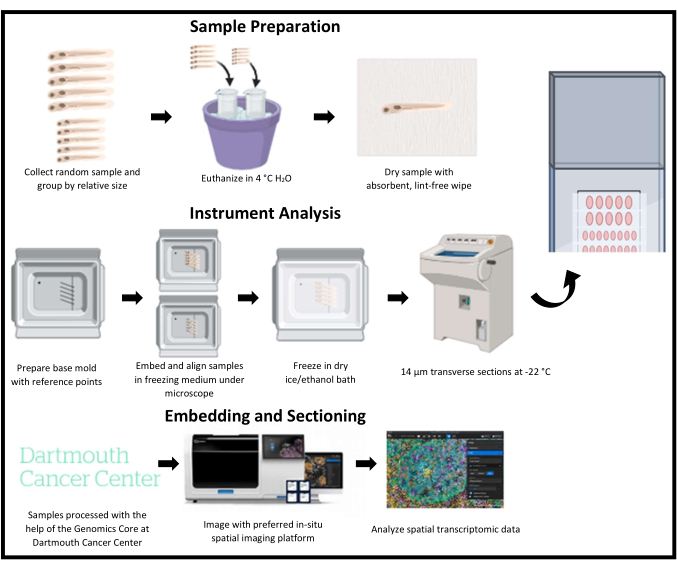
Figura 1: Esquema esquemático de la criosección para transcriptómica espacial en pez cebra. Los peces cebra juveniles se recolectan al azar y luego se agrupan por tamaño relativo. A continuación, las muestras se incrustan en un medio de congelación y se alinean mediante puntos de referencia en el molde base correspondientes a una región de interés. Cada molde se congela rápidamente, se criocorta y se recoge una fila a la vez en el mismo portaobjetos. Los portaobjetos se almacenan a -80 °C después de la sección y se obtienen imágenes y analizan utilizando una plataforma de imágenes transcriptómicas espaciales. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Establecimiento de puntos de referencia para un corte preciso. Una serie de imágenes muestra el proceso de agregar puntos de referencia a un molde de base desechable para alinear múltiples muestras para la criosección. El primer paso (A) es usar cinta de laboratorio para marcar una línea en el interior del molde que todas las muestras usarán como referencia para la alineación. (B) Coloque un punto en el interior del molde cerca de la pared izquierda o derecha para informar la orientación adecuada de la muestra durante la criosección, y luego (C) marque líneas para cada muestra en el ángulo deseado. Haga clic aquí para ver una versión más grande de esta figura.
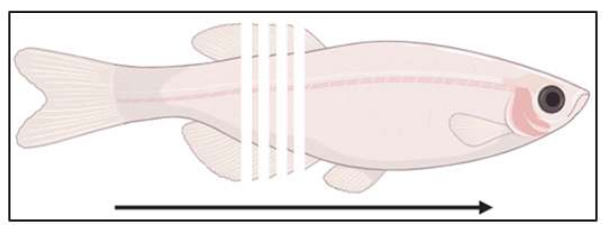
Figura 3: Esquema de la ubicación de la sección. Los peces se seccionan coronalmente, desde la caudal hasta la rostra, a intervalos de 14 μm. Haga clic aquí para ver una versión más grande de esta figura.
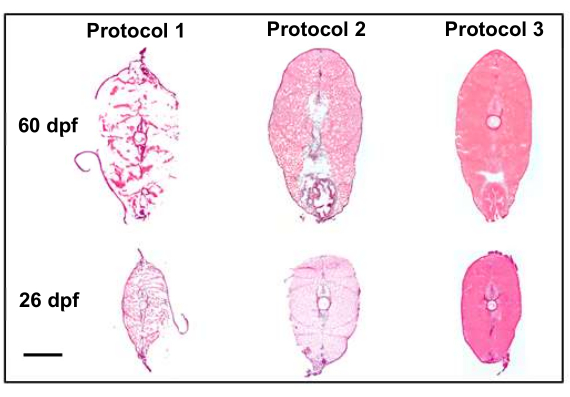
Figura 4: Mejora de la calidad de la sección con modificaciones de protocolo (10x). La figura compara los dos primeros protocolos, que resultaron en una calidad de tejido subóptima, con el protocolo final, que resultó en secciones de alta calidad (que conservaron la morfología) que son buenos candidatos para la transcriptómica espacial. El protocolo 1 utiliza el análisis comparativo 8 del bio-atlasde la Universidad Estatal de Pensilvania, destacado por la fijación de paraformaldehído al 4% antes del corte. El Protocolo 2 introduce la congelación rápida de hielo seco/etanol después de la eutanasia y la fijación después del corte, que preserva la estructura del tejido mucho mejor con la advertencia de artefactos de congelación en toda la sección. El Protocolo 3 perfeccionó las técnicas de eutanasia y la incrustación en hielo antes de la congelación instantánea y el posterior seccionamiento y fijación posteriores a la recolección, lo que dio como resultado muestras de la más alta calidad. La barra de escala es de 500 μm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Verificación de la alineación de múltiples secciones (20x). Imágenes HE representativas de secciones de 26 dpf que resaltan la alineación entre múltiples muestras en un solo corte. La barra de escala es de 500 μm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Mejora de la disposición espacial de los especímenes. Estos paneles resaltan el número de secciones recopiladas en el área de imagen de cada portaobjetos a lo largo del tiempo. La adquisición de experiencia y la mejora de las habilidades técnicas contribuyen a un mayor número de muestras que se recogen. Estas mejoras también se debieron en gran medida a ligeros ajustes en los pasos de incrustación y recopilación en el protocolo. El panel A (40 secciones de peces de 15 dpf) es una tirada temprana que utiliza un molde durante el corte. El panel B (90 secciones de peces de 15 dpf y 47 secciones de peces de 26 dpf) introduce un segundo molde y muestra una mejora en la disposición espacial al reducir la cantidad de espacio entre las muestras durante la incrustación. El panel C (54 secciones de peces de 15 dpf, 80 secciones de peces de 19 dpf, 24 secciones de peces de 23 dpf y 21 secciones de peces de 26 dpf) introduce un tercer y cuarto molde y muestra una mejora continua en una disposición espacial mediante el recorte de la superficie de corte del bloque alrededor de las muestras y la superposición de medio vacío y congelado. Los soportes de cada panel representan los diferentes moldes utilizados durante el seccionado. Las secciones están en un área de 10,5 mm y 22 mm. La barra de escala es de 4 mm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 7: Resultados representativos de nuestro experimento transcriptómico espacial. (A) Una sola imagen de una diapositiva procesada con el protocolo como se ve en la Figura 6C. (B) Secciones de 14 μm de un pez cebra a 15 dpf después de imágenes transcriptómicas espaciales que muestran una señal inespecífica adyacente a las secciones y dentro de la notocorda. La barra de escala es de 4 mm en el panel A. Haga clic aquí para ver una versión más grande de esta figura.
| 4% de paraformaldehído | Concentración de existencias | Importe | Concentración final |
| Paraformaldehído (polvo) | 95 % p/p | 40 g | 4 % p/v |
| NaOH | 1 N | Gota a gota (hasta que el polvo se disuelva) | |
| Hcl | 1 N | Gota a gota (hasta 6,9 pH) | |
| PBS 1x Stock | 1 vez | ~1000 mL (alcanza un volumen total de 1000 mL) | 1 vez |
| Total | 1000 mL |
Tabla 1: Fórmula de paraformaldehído al 4%. Composición de solución de paraformaldehído al 4% para fijar muestras.
| 95% Etanol | Concentración de existencias | Importe | Concentración final |
| Etanol de 200 grados | 100% v/v | 190 mL | 95% v/v |
| Agua de ósmosis inversa | 100% v/v | 10 mL | 5% v/v |
| Total | 200 mL |
Tabla 2: Fórmula de etanol al 95%. Composición de una solución de etanol al 95% para deshidratar secciones durante la tinción con H&E.
| 0,3% Alcohol acidificado | Concentración de existencias | Importe | Concentración final |
| Ácido acético glacial | 100% v/v | 300 μL | 0.3% v/v |
| Agua de ósmosis inversa | 100% v/v | 99.7 mL | 99.7% v/v |
| Total | 100 mL |
Tabla 3: Fórmula de alcohol acidificado al 0,3%. Composición de alcohol acidificado al 0,3% para diferenciar durante la tinción HE.
| 50% Etanol | Concentración de existencias | Importe | Concentración final |
| Etanol de 200 grados | 100% v/v | 100 mL | 50% v/v |
| Agua de ósmosis inversa | 100% v/v | 100 mL | 50% v/v |
| Total | 200 mL |
Tabla 4: Fórmula de etanol al 50%. Composición de etanol al 50% para deshidratar secciones durante la tinción HE.
Discusión
Este informe proporciona soluciones detalladas a muchos de los desafíos técnicos asociados con el pez cebra como organismo modelo en el análisis transcriptómico espacial durante el desarrollo. Al abordar estos desafíos, nuestra disposición compacta de muestras optimiza los costos en las plataformas transcriptómicas espaciales emergentes1. La criosección de larvas de pez cebra para la obtención de imágenes espaciales es un desafío. Las secciones deben conservar una estructura de tejido y una calidad de transcripción suficientes para una ejecución experimental satisfactoria y un análisis espacial posterior8. Los cortes que contienen varias muestras para obtener imágenes espacialmente eficientes deben alinearse con precisión para analizar las regiones comunes de interés en todas las muestras. Finalmente, las secciones deben recopilarse y espaciarse de manera eficiente para maximizar el número de puntos de datos potenciales dentro del área designada de un portaobjetos de imágenes.
El protocolo permite la recolección de secciones de alta calidad que son excelentes candidatas para plataformas de imágenes espaciales de una sola célula. La primera modificación fue congelar rápidamente el pez cebra y luego fijar las secciones en paraformaldehído al 4% después de recogerlas en un portaobjetos. Originalmente, el pez cebra se fijó con 4% de paraformaldehído a 4 °C durante la noche y se incrustó y seccionó después11. La elección de fijar las muestras después de que se seccionaron ayudó a preservar los tejidos internos que no se conservaron de manera efectiva al fijar una muestra completa (Figura 4).
La segunda modificación crítica fue el perfeccionamiento del método de eutanasia para preservar la integridad de los tejidos. Originalmente, los peces cebra se colocaban en agua a 4 °C durante 20 minutos para asegurarse de que estaban suficientemente eutanasiados. Esto es 10 minutos más allá de los requisitos mínimos de las pautas de IACUC. Este tiempo adicional en agua a 4 °C dio a los especímenes más tiempo para descomponerse y contribuyó a una calidad de sección menos que ideal de estructuras internas muy finas o delicadas. La reducción de la cantidad de tiempo en agua a 4 °C al requisito mínimo de 10 minutos ayudó a preservar las delicadas estructuras internas que faltaban en los intentos de corte anteriores. Otras modificaciones que no se muestran en la Figura 4 incluyen el corte el mismo día de la eutanasia, la reducción de la cantidad de tiempo entre la eutanasia y la congelación instantánea de 15 minutos a menos de 5 minutos, y el trabajo con un medio de congelación preenfriado al incorporar. Cuanto más tiempo se almacenaban las muestras a -80 °C antes de la sección, peor terminaba siendo la calidad del tejido de la sección. Las diferencias fueron sutiles, pero es ideal para seccionar el mismo día que la eutanasia y la creación de bloques. Acortar la cantidad de tiempo que se tarda en alinear las muestras después de la eutanasia y antes de la congelación instantánea a no más de 5 minutos es fundamental para la calidad de la sección.
La alineación de varias muestras con una región de interés también requirió algunas modificaciones en el protocolo. La precisión de la alineación que se logró (Figura 5) agrupó las muestras por tamaño relativo y proporcionó la mayor contribución a una disposición espacial eficiente. La estrategia principal para alinear las muestras es superponer el pez cebra en puntos de referencia que se dibujan en los moldes base en los que están incrustados. Los especímenes se alinean a estos puntos de referencia mediante estructuras anatómicas dentro de los especímenes del pez cebra que pueden variar según la región de interés deseada. Los peces en desarrollo de la misma edad pueden variar en tamaño hasta un 50%12. Las muestras con tamaños drásticamente diferentes pueden desalinearse debido a las diferentes distancias entre el punto de referencia anatómico y la región de interés. Agrupar muestras de la misma edad por tamaño relativo e incrustar cada tamaño en sus moldes mejoró la capacidad de alinear múltiples muestras dentro de su molde. Este método puede alinear de forma fiable aproximadamente 2/3 de las muestras, dependiendo de la ventana permitida por la región de interés y la elección del punto de referencia anatómico.
La reducción del espacio entre muestras al incrustar y alinear conduce a un mayor número de muestras por portaobjetos. Sin embargo, esta configuración ajustada se ve aún más limitada por los límites fiduciales en el área de imagen. Además de la experiencia en el criostato, se describen varias estrategias para conciliarlas. En primer lugar, la ubicación precisa del medio de inclusión superpuesto entre las secciones y el recorte del molde congelado pueden tener un profundo impacto en el número de secciones que pueden caber en un área de imagen específica. En la Figura 6 se destaca el número de secciones obtenidas antes y después de optimizar la disposición de la incrustación. En segundo lugar, se colocó un límite en el exterior de las muestras más externas durante la inclusión y antes de la congelación para ayudar a la colocación adecuada en el portaobjetos de imagen. En tercer lugar, el tamaño y la naturaleza transparente de las larvas de pez cebra pueden dificultar saber dónde se encuentran las muestras dentro de un portaobjetos para que puedan recogerse con precisión en un portaobjetos. Descubrimos que el uso de papel de color para contrastar el medio blanco de congelación para crear un límite en el exterior de las muestras más externas condujo a una colocación mucho más precisa de las secciones en los portaobjetos.
Los matices de la temperatura local ayudan a la conservación de la muestra durante el proceso de corte. Una vez que las muestras de varios moldes se recogen en un solo portaobjetos, el portaobjetos de imagen se almacena a 4 °C, el siguiente molde se recorta a la región de interés deseada antes de volver a recogerlo. El portaobjetos de imagen debe estar ligeramente más caliente que las secciones para que se adhiera correctamente a la superficie del portaobjetos. Por lo tanto, no es ideal almacenar el portaobjetos de imagen a -80 °C o en el criostato a -20 °C entre la recolección de muestras de diferentes moldes. También es fundamental que las muestras recogidas en el portaobjetos de imagen no se sometan a múltiples ciclos de congelación/descongelación, ya que esto puede afectar negativamente a la calidad de las transcripciones de ARN dentro de cada muestra. Mantener el portaobjetos de imagen con secciones previamente recolectadas a 4 °C preserva adecuadamente las transcripciones de ARN mientras mantiene una temperatura más baja para una adherencia adecuada sin efectos secundarios negativos asociados con los ciclos de congelación/descongelación13.
Aunque se obtuvieron resultados satisfactorios, el diseño de este método presenta posibles limitaciones. La primera limitación es que estamos limitados al área de imagen de una diapositiva transcriptómica espacial, lo que pone un techo al número total de secciones que se pueden visualizar en la misma diapositiva. Dado que todavía tenemos espacio vacío entre las secciones, es probable que aún no hayamos alcanzado los límites de lo que es posible para este método, pero reconocemos que hay un punto en el que ya no es posible colocar más secciones en el portaobjetos de imagen. Trabajar con peces más grandes a edades más avanzadas también limitará el tamaño total de la muestra, pero no debería afectar la cantidad total de tejido que puede caber en el área de imagen de un portaobjetos. Las mejoras técnicas introducidas por las plataformas de imágenes espaciales podrían aumentar la cantidad total de tejido del que se podrían obtener imágenes ampliando el área de imagen en los portaobjetos. Otra limitación de este método es que el protocolo no ha sido optimizado para peces menores de 15 dpf. Se descubrió que los peces son cada vez más frágiles cuanto más jóvenes son y que los peces menores de 15 dpf eran extremadamente difíciles de manejar y se dañaban fácilmente durante el proceso de incrustación. Esto resultó en un aumento de la cantidad de tiempo entre la eutanasia y la congelación instantánea, lo que fue perjudicial para la calidad del tejido. Esto también redujo el porcentaje de muestras viables que quedaban para la criosección, y los pocos peces viables que quedaban para la sección no eran lo suficientemente rígidos como para retener la estructura del tejido para el análisis de calidad. Encontrar herramientas que sean más adecuadas para manipular peces más frágiles y trabajar con un medio de congelación menos denso que también aumente la rigidez de las muestras durante el corte podría permitir que este método se aplique a peces menores de 15 dpf. La última limitación importante de este método es la alineación de muestras para regiones de interés muy pequeñas. Descubrimos que cuanto más pequeña era la región de interés en la muestra, menos probable era que pudiéramos alinearlas correctamente. Trabajamos con una región de interés que tiene un tamaño aproximado de 50 μm, y esto dio como resultado que aproximadamente 2/3 de las secciones estuvieran correctamente alineadas. Las muestras con una región de interés más pequeña que 50 μm podrían resultar en menos de 2/3 de las secciones alineadas. La mayoría de las muestras desalineadas provienen del paso de incrustación en el protocolo. Es difícil ajustar una muestra en el medio de congelación sin mover otras muestras que ya se han alineado. También hay una cantidad limitada de tiempo que el investigador tiene para alinear estas muestras antes de que afecte negativamente a la calidad del tejido. Por último, los moldes se mueven del microscopio estereoscópico al baño de congelación instantánea, y es posible que las muestras se desplacen en el molde antes de que se congelen por completo. Es posible que trabajar con un medio de congelación más frío pueda permitir al investigador mover una muestra más fácilmente sin afectar a otras muestras que ya están alineadas. La congelación instantánea del molde sin tener que moverlo después de la alineación podría eliminar el desplazamiento de las muestras antes de que se congelen por completo en su lugar. Identificamos señales inespecíficas fuera de los especímenes.
El producto final y el post-análisis inicial presentaron lecciones adicionales. Había señales inespecíficas presentes, que requerían consideración. Según el fabricante, algunas transcripciones de señal baja son un artefacto conocido y el ARN ambiental se puede ver en la secuenciación de ARN de una sola célula que se puede gestionar después del análisis14. El protocolo utilizado aquí puede haber contribuido a las transcripciones ambientales, ya que la fijación se produjo después de la colocación de las muestras. Sin embargo, se encontró que la fijación temprana condujo a una morfología pobre e inconsistente para estas edades tempranas. Alternativamente, los reactivos de amplificación que se agrupan podrían crear tal efecto. Cualitativamente, la señal inespecífica observada en la notocorda fue la más pronunciada. Durante el procesamiento, la notocorda fue la única región verdaderamente vacía, ya que no contenía medios de montaje ni señales de transcripción, que se asemejaban a los perfiles citoplasmáticos y mitocondriales de los tejidos adyacentes del revestimiento de la notocorda. Esto sugiere que el "respaldo" físico (por tejido o medios de montaje) es útil para reducir esta señal. En última instancia, razonamos que la exclusión de la señal en el post-procesamiento es una estrategia satisfactoria. Las características de las señales no específicas eran fácilmente identificables después de los experimentos y podían mitigarse durante el análisis posterior, por ejemplo, mediante la exclusión manual de señales no específicas de regiones fuera de la muestra.
El pez cebra de tan solo 15 dpf de al menos 4 moldes (y edades) diferentes se puede crioseccionar y recolectar con éxito dentro del área de imágenes transcriptómicas espaciales, y se pueden procesar hasta 179 secciones simultáneamente. Basándonos en la calidad de la sección, la alineación de la muestra, el tamaño de la muestra y la calidad de las transcripciones de ARN durante la obtención de imágenes, concluimos que este método es un protocolo eficaz para realizar ensayos transcriptómicos espaciales eficientes. Los investigadores pueden usar este método para reducir los costos asociados con las imágenes espaciales, aumentar el tamaño de la muestra durante los ensayos transcriptómicos espaciales e incluso lograr grandes experimentos en una sola ejecución.
Divulgaciones
Los autores no tienen divulgaciones ni conflictos de intereses con respecto a este informe.
Agradecimientos
El seccionamiento y las imágenes se realizaron con instrumentos proporcionados por recursos compartidos en el Centro Oncológico de Dartmouth, financiado por la Subvención de Apoyo del Centro Oncológico del NCI 5P30CA023108, y el Centro de Biología Cuantitativa del Colegio de Dartmouth (NIGMS COBRE).
Materiales
| Name | Company | Catalog Number | Comments |
| 1 L Beaker | Pyrex | 1003 | |
| 200 proof pure ethanol | Koptec | V1001 | |
| Acetic acid, glacial | VWR | 0714 | acidified alcohol |
| Aluminum foil | |||
| Cover slips | Epredia | 24X50-1.5-001G | |
| Disposable base mold | Fisher HealthCare | 22-363-556 | |
| Distilled water | |||
| DPX mountant | Sigma-Aldrich | 06522 | mountant for histology |
| Dry ice pellets | |||
| Dumont #5SF Forceps | Fine Science Tools | 11252-00 | |
| Eosin-Y Alcoholic | Epredia | 71204 | Eosin Y 1% |
| Gill 1 Hematoxylin | Epredia | 72411 | Hematoxylin |
| Kimwipe | Kimberly-Clark Professional | 34120 | absorbent, lint-free wipe |
| Lab labelling tape | VWR | 89097-934 | |
| Microtome blade MX35 Ultra | Epredia | 3053835 | |
| Microtome Cryostat | Thermo Scientific | Microme HM 525 | |
| O.C.T. Compound | Fisher HealthCare | 23-730-571 | freezing medium |
| Paraformaldehyde | Sigma-Aldrich | 158127 | PFA |
| Permanent Marker | VWR | 52877-886 | |
| Protractor | |||
| SafeClear Xylene Substitute | Fisherbrand | 68551-16-6 | Xylene substitute |
| Single Edge Blades | American Line | 66-0407 | |
| Steriomicroscope | Zeiss | 4350639000 | Stemi 305 w/ double spot LED (4355259020) and Stand K lab (4354259010) |
| Superfrost Plus Micro Slides | VWR | 48311-703 | |
| Transfer pipet | |||
| Xenium V1 slide | 10X/Xenium | 3000941 | spatial transcriptomic imaging slide |
Referencias
- Rao, A., Barkley, D., França, G. S., Yanai, I. Exploring tissue architecture using spatial transcriptomics. Nature. 596 (7871), 211-220 (2021).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet. 22, 627-644 (2021).
- Duhan, L., et al. Single-cell transcriptomics: background, technologies, applications, and challenges. Mol Biol Rep. 51, 600(2024).
- Ståhl, P. L., et al. Visualization and analysis of gene expression in tissue sections by spatial transcriptomics. Science. 353 (6294), 78-82 (2016).
- Colman, R. E., et al. Whole-genome and targeted sequencing of drug-resistant Mycobacterium tuberculosis on the iSeq100 and MiSeq: A performance, ease-of-use, and cost evaluation. PLOS Med. 16 (4), e1002194(2019).
- Marques, I. J., Lupi, E., Mercader, N. Model systems for regeneration: zebrafish. Development. 146 (18), dev167692(2019).
- Mohideen, M. P. K., et al. Histology-based screen for zebrafish mutants with abnormal cell differentiation. Dev Dyn. 228 (3), 414-423 (2003).
- Copper, J. E., et al. Comparative analysis of fixation and embedding techniques for optimized histological preparation of zebrafish. Comp Biochem Physiol C Toxicol Pharmacol. 208, 38-46 (2018).
- Stock, R. J., Labudovich, M., Ducatman, B. Asymptomatic first-trimester liver cell adenoma: diagnosis by fine-needle aspiration cytology with cytochemical and ultrastructural study. Obstet Gynecol. 66 (2), 287-290 (1985).
- Petukhov, V., et al. Cell segmentation in imaging-based spatial transcriptomics. Nat Biotechnol. 40 (3), 345-354 (2022).
- Meyer, T., Tiburcy, M., Zimmermann, W. H. Cardiac macrotissues-on-a-plate models for phenotypic drug screens. Adv Drug Deliv Rev. 140, 93-100 (2019).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Dev Dyn. 203 (3), 253-310 (1995).
- Yu, K., Xing, J., Zhang, J., Zhao, R., Zhang, Y., Zhao, L. Effect of multiple cycles of freeze-thawing on the RNA quality of lung cancer tissues. Cell Tissue Bank. 18 (3), 433-440 (2017).
- Yang, S., et al. Decontamination of ambient RNA in single-cell RNA-seq with DecontX. Genome Biol. 21 (1), 57(2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados