Method Article
空間トランスクリプトーム解析のためのゼブラフィッシュ幼生切片の空間コンパクト配置
要約
ここでは、複数のゼブラフィッシュ(Danio rerio)の幼生サンプルを整列させて凍結切片化し、それらを1枚のスライドに集めて空間的トランスクリプトーム解析を行う方法を紹介します。
要約
空間的トランスクリプトーム技術は、空間的に登録された遺伝子発現パターンを視覚化するための生物医学研究における洗練されたツールです。空間イメージングプラットフォームを使用した複数のサンプルのイメージングと分析には、コストがかかる場合があります。これらの試験を複数の実験条件で実施すると、発生研究で見られるように、さらにコストが増加します。コストを削減するために、この研究では、発生研究のための空間的トランスクリプトーム標本配置の技術と戦略を最適化しようとしました。本研究では、発生時に透明である確立された発生脊椎動物モデルであり、ヒトと~70%の遺伝的相同性を持ち、トランスクリプトーム解析に最適な高アノテーションゲノムを持つゼブラフィッシュを利用しました。ゼブラフィッシュはサイズが小さいため、発育中のゼブラフィッシュは、いくつかの生物学的複製に連続切片をコンパクトに配置することもできます。ここでは、マルチプレックス in situハイブリダイゼーション空間イメージングプラットフォームのイメージングエリア内での複数の魚サンプルの最適化された固定、凍結切片、および信頼性の高いアライメントを報告します。この方法では、受精後15日齢(dpf)のゼブラフィッシュを、少なくとも4つの異なる型と最大174の切片から凍結切片化し、22 mm 10.5 mmのイメージング領域内( in situ 空間トランスクリプトームスライド用)で採取し、同時に処理することができます。ゼブラフィッシュのこの方法は、切片の品質、サンプルのアライメント、スライドあたりのサンプルサイズに基づいて、空間トランスクリプトーム法のアウトプットとサンプルあたりのコストを最適化します。
概要
組織における空間的に異なる発現パターンの評価は、発生、がん、および疾患におけるゲノムの影響を理解するために依然として重要です1,2,3。空間的トランスクリプトミクスは、マルチプレックス発現技術と組織における発現の空間的レジストレーションを組み合わせたものです。「空間的トランスクリプトミクス」は、Ståhl らによって最初に造られた4、マウントされたがん検体がin situ次世代シーケンシングを使用してプローブされました。それ以来、「空間的トランスクリプトミクス」は、空間的レジストレーションと組み合わせたハイスループット発現研究のキャッチオールとして使用されてきました。これらは強力なツールである一方で、データを生成する前に多額の機関投資やラボのコストを必要とすることが多い高価な事業でもあります5。高品質のデータを保持しながらコストを最小限に抑える戦略は、高い需要があります。
ゼブラフィッシュ(Danio rerio)は、発生生物学を研究するための重要なモデルシステムとなり、限られたスペースで脊椎動物の全臓器(および生物)の分析を倍増させる手段を提供しています。ゼブラフィッシュは小型(幼虫は4-6 mm、成魚は2-3 cm)で、一度に数百個の透明な卵を産むことができる6。ゼブラフィッシュの胚は外部から受精し、急速に発達するため、研究者は発生の初期段階で導入遺伝子を導入し、機能獲得対立遺伝子と機能喪失対立遺伝子を容易に生成することができます7。1枚のスライドに複数の試料をはめ込むことは、コスト削減のための魅力的な戦略です。ゼブラフィッシュは繁殖力が高く、サイズが小さいため、標本のスペースが限られている空間トランスクリプトーミクスアッセイのマルチプレックス化に理想的な候補です8。
ゼブラフィッシュの幼生の凍結切片は、難しい技術です。多くの空間的トランスクリプトームプラットフォームは、ゼブラフィッシュのパラフィン切片用に最適化されておらず、ゼブラフィッシュをモデル生物として扱う際には、組織構造を保存し、RNA転写物を保持するために凍結切片が必要です。さらに、ゼブラフィッシュはサイズが小さいため、高品質の凍結切片を入手し、複数のサンプルを効果的に分析することが困難です。この作業は、成魚よりも小さくて壊れやすいゼブラフィッシュの幼生を扱う場合、より困難になります。これらの課題を克服するために、複数のサンプルを確実にアライメントし、空間イメージングプラットフォームのイメージング領域を効率的に利用して、1枚のスライド上に多くの高品質な切片を取得し、それを空間イメージングプラットフォームでイメージングおよび分析できる方法について説明します(図1)。この例では、この方法を空間トランスクリプトームイメージングプラットフォームに適用します。
プロトコル
このプロトコルは、ダートマス大学の施設内動物管理および使用委員会のガイドラインに従っています。
1. クライオスタットの準備
- クライオスタットを-22°Cに冷却し、破片をレセプタクルにブラッシングしてクライオスタットの内面を清掃します。必要なすべてのブラシとツールをチャンバー内に配置します。
2.使い捨てベースモールドの準備
- ベースモールドの内側に油性マーカーで直線を描き、サンプルアライメント用のベースモールド(37mm、24mm、5mm使い捨てプラスチックモールド)を準備し、サンプルアライメントの基準点として使用します。最良の結果を得るには、線を引く前に、直線があるべきベースモールドの内側にラボテープを置きます(図2A)。
- ベースモールドの内側の左壁または右壁の近くに点を描き、クライオセクショニング中にサンプルの適切な向きを確保します(図2B)。
- 分度器で目的の切断角度を測定し、これを各サンプルのベースモールドの内側に印を付けます(図2C)。
- 準備したベース型に、凍結(光干渉断層撮影[OCT])媒体の浅い層を塗布します。サンプルを覆うのに十分な凍結媒体があることを確認してください。
- 凍結媒体を塗布するときは、媒体ボトルのノズルを下塗りし、ベース金型の1つのコーナーに必要な量の媒体を追加してから、ベース金型の水平面をずらして、媒体が全面に均一に分布するようにして、気泡を避けてください。
- 凍結媒体を分配するときは、ボトルをゆっくりと絞ってください。
- 1Lビーカーに氷を加え、ベース型を凍結培地と一緒に氷浴に入れ、少なくとも10分間インキュベートして培地を冷却します。
3.ドライアイスの準備:100%エタノールバス
- アイスバケツのドライアイス1部に100%エタノール1部を加えて、ドラフトにドライアイスと100%エタノール浴を準備します。
- 使い捨てのアルミ皿を使用するか、アルミホイルを折りたたんで、使い捨てのベース型に収まるほどの大きさのボートに入れます。皿またはボートが、ベースモールドが完全に平らになるのに十分な大きさであることを確認してください。
- 皿やボートをお風呂に入れ、バケツを覆います。バケツが冷えるまで5〜10分待ってから、サンプルを凍結します。
4.サンプルの安楽死
- ゼブラフィッシュをランダムに選択して切片化します。サンプルのサイズが異なる場合は、正確な位置合わせを容易にするために、相対的なサイズでグループに分けます(詳細については、ディスカッションを参照してください)。大きな魚と小さな魚を別々の皿に入れます。
- ビーカーに魚系の水を入れ、ビーカーを氷のバケツに入れます。ビーカーを氷で囲みます。
- 温度計で水の温度を監視します。温度を2〜4°Cの間で安定させます。
- ネットまたはストレーナーを使用して、1つの魚のグループを4°Cの水に入れます。魚は氷と接触せず、完全に水に浸す必要があります。手術の動きが止まったら、水に氷を加えて、水が4°C未満に保たれるようにします。 魚を4°Cの水に10分間置いておきます。
注:次のグループを安楽死させる前に、安楽死させたサンプルの各グループの埋め込み、位置合わせ、および瞬間凍結を続行してください。毎回水を交換してください。
5. 埋め込みとアライメント
- 安楽死させた魚を4°Cの水に10分間沈めた後、魚を採取します。尾びれで魚をつかんで先端の細い鉗子で魚を水から取り出し、吸収性のある糸くずの出ないワイプにそっと押し付けて乾かします。
注:高品質のセクションでは、4°Cの水から魚を取り出してから瞬間冷凍するまでの時間を制限することが重要です - 実体顕微鏡(10倍倍)の氷浴で準備したベース型を操作し、各サンプルを基準点に沿って正しい向きでベース型に配置し、サンプルを別の薄い凍結媒体の層で静かに覆います。
注意: 金型全体に凍結媒体を充填しないでください。代わりに、すべてのサンプルを覆うのに十分な薄い層のみを使用してください。 - 解剖学的な基準点を使用して、魚をベースモールドの内側にマークされた線に正確に位置合わせします。先端の細い鉗子を使用して、各魚の向きを調整し、同じ向きになるようにします。ゆっくりと移動して、凍結媒体に気泡を作らないようにしてください。
- サンプルが所定の位置に局所的に凍結されるまで、サンプルの下のベースモールドの底にドライアイスを塗布します。ベースモールドの水平面を水平に保ち、ドライアイスで所定の位置に凍結する前にサンプルを基準点からずらさないようにします。
- サンプルを入れたベースモールドを、ドライアイス:100%エタノールバスのアルミボートまたは皿に置きます。ボートがバスの表面に浮いており、ベースモールドが乾いたままであることを確認してください。浴に蓋をして、サンプルを10分間浮かべます。
- 凍結したベース型をホイルで包み、切断する準備ができるまで-80°Cの冷凍庫で保管します。残りのグループに対して手順 4.2 から 5.6 を繰り返します。
6. クライオセクショニング
- 凍結したベース型を予冷したクライオスタットに持ち込んでクライオセクショニングを行い、クライオスタットチャンバーに入れます。凍結したブロックは、解凍を防ぐためにドライアイスを入れた箱に入れて輸送します。
- in situ空間イメージングスライドを保管庫から取り出し、事前に冷却したスライドホルダーに入れます。空間イメージングスライド付きのスライドホルダーは、関心領域から切片を収集する準備ができるまで、-22°Cのクライオスタットチャンバーに保管します。
- 凍結したサンプルをベースモールドから取り出し、新鮮な凍結培地を入れたチャックで凍結します。それらをチャックに凍結して、切断面がブレードに面するようにします。
- 新鮮で細かいミクロトームブレードをクライオスタットに入れます。
- 金型をブレードに合わせ、対象領域をトリムします(推奨されるトリム厚さ20〜50μm)。金型内で行われたマーキングが凍結したサンプルブロックに転写されていることを確認して、サンプル内の位置を特定するのに役立ちます。
- トリミング段階では、切断面が凍結媒体の参照マーキングと平行になるように金型を調整します。
- 切片を標準的な正電荷の顕微鏡スライドに集め、明視野でチェックして、トリミングが不要になったかどうかを確認します。
- クライオスタットチャンバーから空間イメージングスライドを取り出し、4°Cの氷浴に入れます。スライドはスライドホルダーに入れておきます。スライドが濡れないように注意してください。
- 凍結切片を開始します(推奨10-14μm; 図3)そして、サンプルの関心領域に達するまで、正に帯電したスライド上に切片を収集します。明視野でセクションをチェックして、関心領域が金型の次のセクションになることを確認します。
- シングルセル空間イメージングスライドをクライオスタットチャンバーに戻します。スライドホルダーからスライドを取り外し、先端の細いペイントブラシを使用して、正確な関心領域からセクションを空間イメージングスライドに行ごとに収集し、セクションがロールアップしないようにします。
- 空の凍結媒体の角を絵筆の裏側でナイフステージに押し込み、スライドをつかんで収集するときにセクションが平らになるようにします。
- スライドの上部をピボットポイントとして使用し、スライドをゆっくりとセクションに下げ、セクションがスライドに3秒間付着するまで待ってから、スライドをナイフステージから持ち上げます。
- スライドのイメージング領域で切片を収集するときは左から右に作業し、可能な場合は空の凍結媒体の層を重ねます。
- 色付きの紙の境界線を基準にして、組織が見にくい場合はスライドのイメージング領域内にセクションを配置します。
- 対象領域から切片を収集した後、スライドをスライドホルダーに戻します。空間イメージングスライドに収集するサンプルがなくなった場合は、スライドを-80°Cで最大2週間保存し、 in situ 空間イメージングプラットフォームでの装置分析の準備が整います。複数の型から切片を収集する必要がある場合は、空間イメージングスライドを4°Cの氷浴に戻し、次の型で手順6.3〜6.11を繰り返します。
- ヘマトキシリンおよびエオシン(HE)染色用の標準的な正電荷顕微鏡スライド上で、各型から関心領域切片の前後の切片を収集し、サンプルのアライメントと切片の品質が十分であることを確認してから、分析を進めます。
7. サンプルの固定
- 参照スライドをクライオスタットから取り出し、RTで30分間風乾して切片をスライドに接着します。
- 切片を4%パラホルムアルデヒド(表1)の入ったスライド容器に20分間入れて固定します。
- 切片を蒸留水を入れたスライド容器に3分間入れて洗浄します。
- HE染色を続けるか、乾燥させてスライドを-80°Cで保存し、将来の染色に備えてください。
8. 切片のHE染色
- スライドを100%エタノールで2分間、95%(表2)エタノールで2分間、次に水道水で1分間インキュベートすることにより、切片を脱水して洗浄します。顕微鏡スライド染色ラックを使用して、スライドを浴から浴に移します。
- スライドをヘマトキシリンで2分間45秒間、水道水で1分間、0.3%酸性アルコール(表3)で1分間インキュベートし、水道水を1分間流して、核を染色し、分化させます。顕微鏡スライド染色ラックを使用して、スライドを浴から浴に移します。
- 細胞質成分を染色し、スライドをEosin Y 1%で45秒間、50%エタノール(表4)で1分間、95%エタノールで1分間、および100%エタノールで1分間インキュベートして脱水します。顕微鏡スライド染色ラックを使用して、スライドを浴から浴に移します。
- スライドをキシレンで1分間インキュベートして、切片を透明にします。転写ピペットでスライドの上部3分の1に封入剤を滴下し、鉗子で封入剤の上にカバースリップをゆっくりと下げて、スライドを取り付けて覆います。
9. 切片の空間的トランスクリプトームイメージングと解析
- -80°Cの保管からイメージングスライドを取り出し、空間トランスクリプトーム解析用の in situ 空間イメージングプラットフォームでイメージングします。
注:イメージングに関連する正確な手順は、空間イメージングプラットフォームによって決定されます。 - 空間トランスクリプトームプラットフォームの品質管理指標を確認します。チェックすべき重要な指標は、検出された細胞数、細胞あたりの転写産物の中央値、100 μm2あたりの核転写産物、およびプローブセット内の各遺伝子の高品質なデコード転写産物の合計です。
注:これらの品質管理指標には普遍的な閾値がなく、これらの閾値に対する期待値は、使用するサンプルと遺伝子パネルによって異なります。 - 検出可能なRNA転写産物のデータ出力を読み取り、実験の品質管理指標に基づいてどの転写産物が低品質であるかを判断し、低品質の転写産物を除外します。残りの高品質のトランスクリプトを、セクション内の空間配置に関連して分析します。
- 実験的な関心に基づいて、切片とクラスター細胞の細胞セグメンテーションを表示します。関心領域の細胞クラスター内のRNA転写産物を、同じスライド上の異なる年齢のゼブラフィッシュのグループ間で比較します。
結果
この方法(図1)では、ゼブラフィッシュを動物モデルとして用い、空間的に分解された遺伝子発現パターンを探索します。ゼブラフィッシュの幼生を効率的に凍結切片化して空間イメージングを行うことは困難です。切片は、組織構造と検出可能な遺伝子を保持するために高品質でなければなりません(図4)。空間効率の高いイメージングのためには、複数のサンプルを含む切片を正確に位置合わせして、すべてのサンプルの関心領域を解析する必要があります(図2および図5)。最後に、空間トランスクリプトミクススライドのイメージング領域内の潜在的なデータポイントの数を最大化するために、セクションを効率的に収集し、間隔を空ける必要があります(図3および図6)。これら3つのパラメータを使用して実験を測定し、成功したか最適でないかを判断しました。
多くのプロトコルの変更により、空間トランスクリプトームイメージングの有力な候補となる高品質な切片を収集することができました。安楽死、固定、および埋め込みのためのプロトコルの調整により、切片の品質が向上しました(図4)。最初のプロトコル(図4、プロトコル1)では、切片の品質は最適ではなく、組織はその構造を保持していませんでした。これを修正するために、2番目のプロトコル(図4、 プロトコル2)では、サンプルを埋め込み、切片をスライドに収集した後に組織を固定した後にフラッシュフリーズステップを導入しました。これにより、切片の全体的な組織構造は改善されましたが、空間トランスクリプトミクスで許容できると考えられていた品質には達しませんでした。最後に、第3のプロトコル(図4、 プロトコル3)では、魚を安楽死させるのにかかる時間を短縮し、急速凍結前の氷上に魚を埋め込み、その後の収集後の切片化と固定を行うことで、安楽化技術が改良されました。その結果、空間トランスクリプトームイメージングに適した候補となる最高品質のサンプルが得られました。
複数のサンプルを関心領域にアライメントするには、プロトコールにもいくつかの変更を加える必要がありました。サンプル間のアライメントを最も改善した変更の1つは、ゼブラフィッシュが埋め込まれているベース型に基準点を導入したことです(図2)。本研究で実施した実験では、同じカット内の複数のセクションにわたる構造を比較することにより、サンプルを整列させました(図5)。
効率的な空間トランスクリプトミクスの手法を開発するための最後の重要なステップは、これらの高品質で整列した異なる年代のサンプルをできるだけ多く同じスライドに収めてイメージングすることでした。経験を積み、技術力を向上させることで、サンプルの採取数の増加に貢献しています。これらの改善は、主にプロトコルの埋め込みと収集のステップのわずかな調整によるものでした。 図 6 のこれらのパネルは、各スライドのイメージング領域に収集されたセクションの数を経時的に強調表示しています。 図6A (15dpf魚の40セクション)は、切片化中に1つの金型を使用する初期のランです。 図6B (15 dpf魚の90セクションと26 dpf魚の47セクション)は、2番目の型を導入し、埋め込み中のサンプル間のスペースの量を減らすことにより空間配置の改善を示しています。 図6C (15 dpf魚54セクション、19 dpf魚80セクション、23 dpf魚24セクション、および26 dpf魚21セクション)は、3番目と4番目の型を導入し、サンプルの周りのブロックの切断面をトリミングし、空の凍結媒体を重ねることにより、空間配置の継続的な改善を示しています。これらの改善により、4つの異なる年齢の空間トランスクリプトームスライドとテストフィッシュのイメージングエリアに179のセクションを配置する機会を得ることができました。
プロトコールの完了時には、既製のマルチプレックス in situ ソフトウェアとカスタムメソッドの両方で容易に分析できるサンプルが得られました。ここで使用したマルチプレックスソフトウェアは、シーケンシングデータ9のphred品質スコアに大まかに基づいて、擬似phredスコアを採用した。スコアは59%から70%の範囲であり、60%は低品質の読み取りに対する警告のカットオフ値でした。調査の結果、疑似フレドスコアが低いのは、セクション間の空きスペースの信号の複雑さが低いためでした。スライドエリアは効率的に利用されていましたが、スライドエリアはまだ>50%空いていました。シグナルが十分に複雑である組織によって占めるスライド領域の割合を考慮すると、シグナル品質は優れていました。
低強度の非特異的なシグナルは、スライド上の2つの一般的な領域、つまり標本の外側と空の脊索の内側でも見られました(図7A)。切片の外側には、洗浄アーチファクトと一致するパターンがあり、これにより試料から離れる動きが減少しました(図7B)。非特異的なシグナルは、脊索が占める標本の空の領域内にも見られました。これは、増幅試薬が閉じ込められる可能性のある洗浄アーティファクトである可能性が高いと考えられていました。両領域の転写産物は、主に細胞質(すなわち、リボソームタンパク質L3およびL4)またはミトコンドリア(アスパラギン酸トランスフェラーゼおよびイソクエン酸デヒドロゲナーゼ)であった。これらの領域が、核10の周りの転写産物の局在化とクラスタリングに基づいて細胞を識別するBaysorセグメンテーションに含まれていた場合、セグメンテーション分析には細胞境界が含まれ、細胞境界が含まれることもありました。この信号は十分に低かったため、標本と簡単に区別できました。
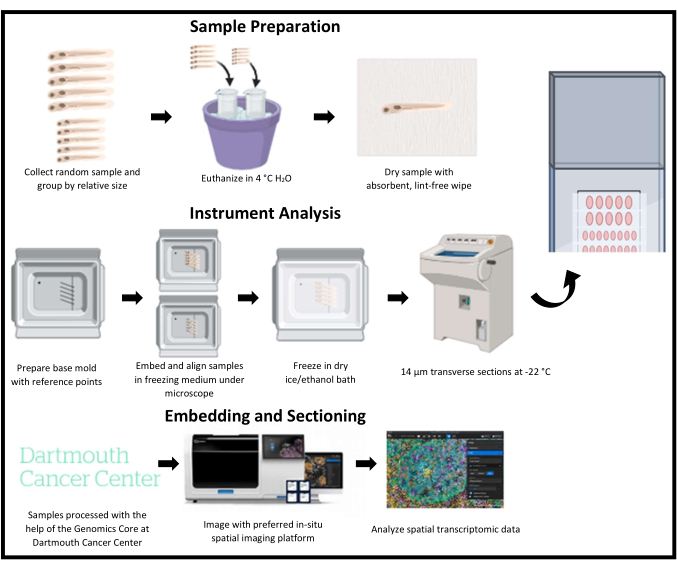
図1:ゼブラフィッシュの空間トランスクリプトミクスのためのクライオセクショニングの概略図。 ゼブラフィッシュの稚魚をランダムに採集し、相対的なサイズでグループ化します。次に、サンプルを凍結媒体に埋め込み、関心領域に対応するベースモールド上の基準点によって位置合わせします。各型は急速冷凍され、凍結切片化され、同じスライド上に一度に1行ずつ収集されます。スライドは切片化後-80°Cで保存され、空間トランスクリプトームイメージングプラットフォームを使用して画像化および分析されます。 この図の拡大版を表示するには、ここをクリックしてください。

図2:正確なセクショニングのための基準点の確立。 一連の画像は、クライオセクショニングのために複数のサンプルを整列させるために、使い捨てのベースモールドに基準点を追加するプロセスを示しています。最初のステップ(A)は、ラボテープを使用して、すべてのサンプルが位置合わせの基準として使用する金型の内側の線をマークすることです。(B)クライオセクショニング中の適切なサンプルの向きを知らせるために、金型の内側の左側または右側の壁の近くにドットを配置し、次に(C)各サンプルの線を希望の角度でマークします。 この図の拡大版を表示するには、ここをクリックしてください。
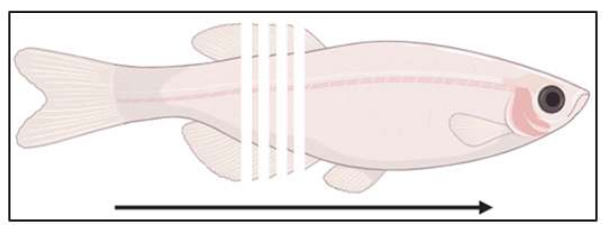
図3:セクショニング位置の概略図。 魚は、尾側から吻側まで、14μm間隔で冠状に切片化されます。 この図の拡大版を表示するには、ここをクリックしてください。
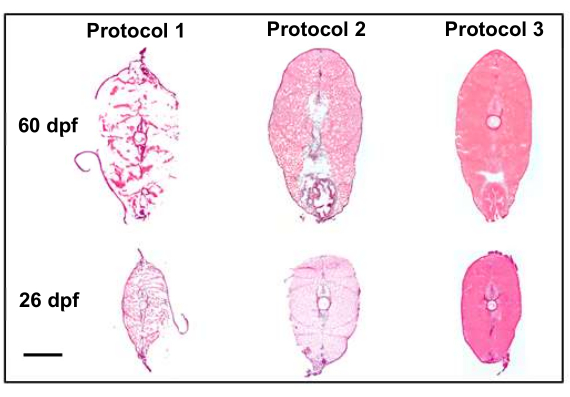
図4:プロトコルの変更によるセクションの品質向上(10倍)。 この図は、組織の品質が最適ではなかった最初の 2 つのプロトコルと、空間トランスクリプトミクスの候補として優れた高品質の切片 (形態が保持されている) が得られた最終的なプロトコルを比較したものです。プロトコル1は、ペンシルベニア州立大学バイオアトラスの比較分析8を利用しており、切片化前の4%パラホルムアルデヒド固定によって強調されています。プロトコル2は、安楽死後のドライアイス/エタノール瞬間凍結と切片化後の固定を導入し、切片全体でアーティファクトを凍結するという注意点により、組織構造をはるかによく保存します。プロトコル3は、安楽死技術と氷への包埋を急速凍結前に改良し、その後の収集後の切片化と固定により、最高品質のサンプルが得られました。スケールバーは500μmです。 この図の拡大版を表示するには、ここをクリックしてください。

図5:複数セクションのアライメントの検証(20倍)。 26 dpfセクションの代表的なHE画像で、1回のカットで複数のサンプル間のアライメントを強調しています。スケールバーは500μmです。 この図の拡大版を表示するには、ここをクリックしてください。

図6:標本の空間配置の改善。 これらのパネルは、各スライドのイメージング領域に収集されたセクションの数を経時的に強調表示します。経験を積み、技術力を向上させることで、サンプルの採取数の増加に貢献しています。これらの改善は、主にプロトコルの埋め込みと収集のステップのわずかな調整によるものでした。パネル A (15dpf魚の40セクション)は、切片化中に1つの金型を使用する初期ランです。パネル B (15dpf魚90セクション、26dpf魚47セクション)は、第2の型を導入し、埋め込み中のサンプル間のスペースを減らすことにより、空間配置の改善を示しています。パネル C (15dpf魚54セクション、19dpf魚80セクション、23dpf魚24セクション、26dpf魚21セクション)は、第3および第4の型を導入し、サンプルの周りのブロックの切断面をトリミングし、空の凍結媒体を重ねることにより、空間配置の継続的な改善を示しています。各パネルのブラケットは、セクショニング中に使用されるさまざまな金型を表しています。セクションは10.5 mm 22 mmの領域にあります。スケールバーは4 mmです。 この図の拡大版を表示するには、ここをクリックしてください。

図7:空間トランスクリプトーム実験の代表的な結果。 (A) 図6Cに見られるようなプロトコルで処理されたスライドの単一の画像。(B)空間的トランスクリプトームイメージング後の15 dpfでのゼブラフィッシュの14 μm切片で、切片に隣接し、脊索内に非特異的なシグナルを示す。パネルAのスケールバーは4mmです。 この図の拡大版を表示するには、ここをクリックしてください。
| 4%パラホルムアルデヒド | ストック濃度 | 量 | 最終濃度 |
| パラホルムアルデヒド(粉末) | 95 % w/w | 40グラム | 4 % w/v |
| NaOHの | 1 N | ドロップワイズ(粉末が溶けるまで) | |
| HCl | 1 N | ドロップワイズ(6.9 pHまで) | |
| PBS 1xストック | 1倍 | ~1000 mL (総容量 1000 mL に達する) | 1倍 |
| トータル | 1000ミリリットル |
表1:4%パラホルムアルデヒド式。 サンプルを固定するための4%パラホルムアルデヒド溶液の組成。
| 95%エタノール | ストック濃度 | 量 | 最終濃度 |
| 200プルーフエタノール | 100% V/V | 190ミリリットル | 95% v/v |
| ROウォーター | 100% V/V | 10ミリリットル | 5% v/v |
| トータル | 200ミリリットル |
表2:95%エタノール式。 H&E染色中の切片を脱水するための95%エタノール溶液の組成。
| 0.3%酸性アルコール | ストック濃度 | 量 | 最終濃度 |
| 氷酢酸 | 100% V/V | 300μL | 0.3%V/V |
| ROウォーター | 100% V/V | 99.7ミリリットル | 99.7%/v/v |
| トータル | 100ミリリットル |
表3:0.3%酸性化アルコール処方。 HE染色中に区別するための0.3%酸性アルコールの組成。
| 50%エタノール | ストック濃度 | 量 | 最終濃度 |
| 200プルーフエタノール | 100% V/V | 100ミリリットル | 50% V/V |
| ROウォーター | 100% V/V | 100ミリリットル | 50% V/V |
| トータル | 200ミリリットル |
表4:50%エタノール式。 HE染色中に切片を脱水するための50%エタノールの組成。
ディスカッション
本稿では、ゼブラフィッシュをモデル生物として持つ開発中の空間トランスクリプトーム解析における多くの技術的課題について、詳細な解決策を提供します。これらの課題に対処するために、当社のコンパクトな試料配置は、新しい空間トランスクリプトームプラットフォーム1のコストを最適化します。ゼブラフィッシュの幼生を凍結切片で空間イメージングすることは困難です。切片は、満足のいく実験の実行と下流の空間分析のために十分な組織構造と転写産物の品質を保持する必要があります8。空間効率の高いイメージングのためには、複数のサンプルを含むスライスを正確に位置合わせして、すべてのサンプルに共通の関心領域を解析する必要があります。最後に、イメージングスライドの指定された領域内の潜在的なデータポイントの数を最大化するために、セクションを効率的に収集し、間隔を空ける必要があります。
このプロトコルにより、シングルセル空間イメージングプラットフォームの優れた候補である高品質の切片を収集することができます。最初の変更は、ゼブラフィッシュを急速冷凍し、スライド上に集めた後、4%パラホルムアルデヒドで切片を固定することでした。もともとゼブラフィッシュは、4%パラホルムアルデヒドで4°Cで一晩固定され、その後、埋め込まれて切片化されていました11。サンプルを切片化した後にサンプルを固定することを選択することで、サンプル全体を固定する際に効果的に保存できなかった内部組織を保存することができました(図4)。
2番目の重要な変更は、組織の完全性を維持するための安楽死法の改良でした。ゼブラフィッシュはもともと、十分に安楽死させるために、4°Cの水に20分間置かれていました。これは、IACUC ガイドラインの最小要件を 10 分超えています。この4°Cの水での余分な時間により、試料が分解する時間が長くなり、非常に微細なまたは繊細な内部構造の理想的とは言えない切片品質に貢献しました。4°Cの水で過ごす時間を最小要件の10分に短縮することで、以前の切片化の試みでは欠けていた繊細な内部構造を保存することができました。 図4 に示されていないその他の変更には、安楽死と同じ日にセクショニングすること、安楽死から瞬間凍結までの時間を15分から5分未満に短縮すること、埋め込み時に事前に冷却した凍結媒体を使用することが含まれます。切片化する前にサンプルを-80°Cで保存する時間が長いほど、切片の組織品質は悪化します。微妙な違いはありましたが、安楽死やブロック作成と同じ日にセクショニングを行うのに最適です。安楽死後、瞬間凍結前のサンプルの整列にかかる時間を5分以内に短縮することは、切片の品質にとって重要です。
複数のサンプルを関心領域にアライメントするには、プロトコールにもいくつかの変更を加える必要がありました。達成されたアライメントの精度(図5)は、サンプルを相対的なサイズでグループ化し、効率的な空間配置に最も貢献しました。サンプルを位置合わせするための主な方法は、ゼブラフィッシュが埋め込まれているベース型に描かれた基準点にゼブラフィッシュをオーバーレイすることです。標本は、ゼブラフィッシュの標本内の解剖学的構造によってこれらの基準点に整列されますが、これは目的の関心領域によって異なります。同じ年齢の発育中の魚は、サイズが最大50%12まで異なることがあります。サイズが大きく異なるサンプルは、解剖学的基準点と関心領域の間の距離が異なるため、位置がずれる可能性があります。同じ年代のサンプルを相対的なサイズでグループ化し、各サイズを型に埋め込むことで、型内で複数のサンプルを位置合わせする能力が向上しました。この方法では、関心領域によって許容されるウィンドウと解剖学的基準点の選択に応じて、サンプルの約2/3を確実に整列させることができます。
埋め込みやアライメント時にサンプル間のスペースを減らすと、スライドあたりのサンプル数が多くなります。ただし、このタイトな構成は、イメージング領域の基準制限によってさらに制約されます。クライオスタットでの経験に加えて、これらを調整するためのいくつかの戦略について説明します。まず、セクション間に重なり合う埋め込み媒体の正確な配置と、凍結した金型のトリミングは、特定のイメージング領域に収まるセクションの数に大きな影響を与える可能性があります。 図6 は、埋込み配置を最適化する前と後で取得したセクションの数を示しています。次に、イメージングスライドへの適切な配置を支援するために、埋め込み中および凍結前に、最も外側のサンプルの外側に境界を配置しました。第三に、ゼブラフィッシュの幼生のサイズと透明性により、サンプルがスライド内のどこにあるかを知ることが難しくなり、サンプルをスライド上に正確に収集できます。色紙を使用して白い凍結媒体を対比し、最も外側のサンプルの外側に境界を作成すると、スライド上のセクションをより正確に配置できることがわかりました。
局所的な温度の微妙な違いは、切断プロセス中のサンプル保存に役立ちます。複数の金型からのサンプルを1枚のスライドに集めたら、イメージングスライドを4°Cで保存し、次の型を目的の関心領域にトリミングしてから再度採取します。イメージングスライドがスライドの表面に適切に付着するには、切片よりもわずかに暖かくなければなりません。そのため、異なる型からのサンプル採取の合間に、イメージングスライドを-80°Cで保存したり、クライオスタットで-20°Cに保管したりすることは理想的ではありません。また、イメージングスライド上に収集されたサンプルが、各サンプル内のRNA転写産物の品質に悪影響を与える可能性があるため、複数回の凍結融解サイクルを経ないことも重要です。以前に採取した切片を含むイメージングスライドを4°Cに保つことで、凍結融解サイクル13に関連する悪影響なしに適切な接着のために低温を維持しながら、RNA転写産物を十分に保存することができる。
満足のいく結果が得られましたが、この方法の設計には制限が伴います。1つ目の制限は、空間的なトランスクリプトームスライドのイメージング領域に制約されているため、同じスライドでイメージングできるセクションの総数に上限が設けられることです。セクション間にまだ空きスペースがあることを考えると、この方法で可能な範囲にはまだ達していない可能性がありますが、イメージングスライドにこれ以上セクションを収めることがもはや不可能になるポイントがあることを認識しています。高齢で大型の魚を扱う場合も、総サンプルサイズが制限されますが、スライドのイメージング領域に収まる組織の総量に影響を与えることはありません。空間イメージングプラットフォームによる技術的改善により、イメージングスライド上のイメージング領域を拡大することにより、イメージング可能な組織の総量が増加する可能性があります。この方法の別の制限は、プロトコルが15 dpf未満の魚に最適化されていないことです。その結果、魚は若ければ若いほど壊れやすくなり、15dpf未満の魚は取り扱いが非常に難しく、埋め込みプロセス中に簡単に損傷を受けることがわかりました。その結果、安楽死から瞬間凍結までの時間が長くなり、組織の質に悪影響を及ぼしました。これにより、凍結切片に残された生存可能なサンプルの割合も減少し、切片化のために残った数匹の生存魚は、品質分析のために組織構造を保持するのに十分な硬さではありませんでした。より壊れやすい魚の取り扱いに適したツールを見つけ、セクショニング中のサンプルの剛性を高める密度の低い凍結媒体で作業することで、この方法を15dpf未満の魚に適用できる可能性があります。この方法の最後の主要な制限は、非常に小さな関心領域のサンプルアライメントです。サンプルの関心領域が小さいほど、それらを適切に整列させることができた可能性が低くなることがわかりました。約50μmの関心領域で作業した結果、切片の約2/3が適切に整列しました。関心領域が50μmより小さいサンプルでは、整列する切片が2/3未満になる可能性があります。ミスアライメントされたサンプルの大部分は、プロトコルの埋め込みステップから来ています。凍結媒体内の1つのサンプルを調整することは、すでに位置合わせされている他のサンプルを移動せずに困難です。また、研究者がこれらのサンプルを整列させなければならない時間も限られており、組織の品質に悪影響を与える可能性があります。最後に、型を実体顕微鏡から瞬間凍結槽に移し、サンプルが完全に凍結する前に型内を移動することができます。より冷たい凍結媒体を使用すると、研究者は、すでに整列している他のサンプルに影響を与えることなく、1つのサンプルをより簡単に移動できる可能性があります。位置合わせ後に金型を動かさずに瞬間凍結することで、サンプルが完全に固定される前にサンプルがずれるのを防ぐことができます。標本の外側に非特異的なシグナルを同定しました。
最終製品と最初の事後分析では、追加の教訓が得られました。非特異的なシグナルが存在し、検討が必要であった。メーカーによると、一部の低シグナル転写産物は既知のアーティファクトであり、アンビエントRNAは解析後に管理できるシングルセルRNA-seqで見ることができます14。ここで使用されたプロトコルは、標本の配置後に固定が行われたため、周囲の転写産物に寄与した可能性があります。しかし、早期の固定は、これらの若い年齢の形態が貧弱で一貫性のないものにつながったことがわかりました。あるいは、プールされた増幅試薬がそのような効果を生み出す可能性があります。定性的には、脊索に見られる非特異的なシグナルが最も顕著でした。プロセシング中、脊索は、隣接する脊索内層組織の細胞質およびミトコンドリアプロファイルに類似した封入媒体および転写シグナルを含んでいなかったため、唯一の真に空の領域でした。これは、物理的な「バッキング」(組織または封入剤による)がこのシグナルの減少に役立つことを示唆しています。最終的には、後処理で信号を排除することが満足のいく戦略であると推論します。非特異的なシグナルの特徴は、実験後に容易に識別でき、例えば、試料の外側の領域の非特異的シグナルを手動で除外するなど、ダウンストリーム分析中に軽減することができました。
少なくとも4つの異なる型(および年齢)から得られた15 dpfの若さのゼブラフィッシュを、空間トランスクリプトームイメージングエリア内で首尾よく凍結切片化して収集することができ、最大179の切片を同時に処理することができます。イメージング中の切片の品質、サンプルのアライメント、サンプルサイズ、およびRNA転写産物の品質に基づいて、この方法は効率的な空間トランスクリプトームアッセイを実施するための効果的なプロトコルであると結論付けています。研究者は、この方法を使用して、空間イメージングに関連するコストを削減し、空間トランスクリプトームアッセイ中のサンプルサイズを大きくし、さらには1回の実行で大規模な実験を達成することができます。
開示事項
著者は、このレポートに関して開示または利益相反を持っていません。
謝辞
切片化とイメージングは、NCI Cancer Center Support Grant 5P30CA023108、およびCenter for Quantitative Biology at Dartmouth College(NIGMS COBRE)の資金提供を受けたDartmouth Cancer Centerの共有リソースから提供された機器を使用して実施されました。
資料
| Name | Company | Catalog Number | Comments |
| 1 L Beaker | Pyrex | 1003 | |
| 200 proof pure ethanol | Koptec | V1001 | |
| Acetic acid, glacial | VWR | 0714 | acidified alcohol |
| Aluminum foil | |||
| Cover slips | Epredia | 24X50-1.5-001G | |
| Disposable base mold | Fisher HealthCare | 22-363-556 | |
| Distilled water | |||
| DPX mountant | Sigma-Aldrich | 06522 | mountant for histology |
| Dry ice pellets | |||
| Dumont #5SF Forceps | Fine Science Tools | 11252-00 | |
| Eosin-Y Alcoholic | Epredia | 71204 | Eosin Y 1% |
| Gill 1 Hematoxylin | Epredia | 72411 | Hematoxylin |
| Kimwipe | Kimberly-Clark Professional | 34120 | absorbent, lint-free wipe |
| Lab labelling tape | VWR | 89097-934 | |
| Microtome blade MX35 Ultra | Epredia | 3053835 | |
| Microtome Cryostat | Thermo Scientific | Microme HM 525 | |
| O.C.T. Compound | Fisher HealthCare | 23-730-571 | freezing medium |
| Paraformaldehyde | Sigma-Aldrich | 158127 | PFA |
| Permanent Marker | VWR | 52877-886 | |
| Protractor | |||
| SafeClear Xylene Substitute | Fisherbrand | 68551-16-6 | Xylene substitute |
| Single Edge Blades | American Line | 66-0407 | |
| Steriomicroscope | Zeiss | 4350639000 | Stemi 305 w/ double spot LED (4355259020) and Stand K lab (4354259010) |
| Superfrost Plus Micro Slides | VWR | 48311-703 | |
| Transfer pipet | |||
| Xenium V1 slide | 10X/Xenium | 3000941 | spatial transcriptomic imaging slide |
参考文献
- Rao, A., Barkley, D., França, G. S., Yanai, I. Exploring tissue architecture using spatial transcriptomics. Nature. 596 (7871), 211-220 (2021).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet. 22, 627-644 (2021).
- Duhan, L., et al. Single-cell transcriptomics: background, technologies, applications, and challenges. Mol Biol Rep. 51, 600(2024).
- Ståhl, P. L., et al. Visualization and analysis of gene expression in tissue sections by spatial transcriptomics. Science. 353 (6294), 78-82 (2016).
- Colman, R. E., et al. Whole-genome and targeted sequencing of drug-resistant Mycobacterium tuberculosis on the iSeq100 and MiSeq: A performance, ease-of-use, and cost evaluation. PLOS Med. 16 (4), e1002194(2019).
- Marques, I. J., Lupi, E., Mercader, N. Model systems for regeneration: zebrafish. Development. 146 (18), dev167692(2019).
- Mohideen, M. P. K., et al. Histology-based screen for zebrafish mutants with abnormal cell differentiation. Dev Dyn. 228 (3), 414-423 (2003).
- Copper, J. E., et al. Comparative analysis of fixation and embedding techniques for optimized histological preparation of zebrafish. Comp Biochem Physiol C Toxicol Pharmacol. 208, 38-46 (2018).
- Stock, R. J., Labudovich, M., Ducatman, B. Asymptomatic first-trimester liver cell adenoma: diagnosis by fine-needle aspiration cytology with cytochemical and ultrastructural study. Obstet Gynecol. 66 (2), 287-290 (1985).
- Petukhov, V., et al. Cell segmentation in imaging-based spatial transcriptomics. Nat Biotechnol. 40 (3), 345-354 (2022).
- Meyer, T., Tiburcy, M., Zimmermann, W. H. Cardiac macrotissues-on-a-plate models for phenotypic drug screens. Adv Drug Deliv Rev. 140, 93-100 (2019).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Dev Dyn. 203 (3), 253-310 (1995).
- Yu, K., Xing, J., Zhang, J., Zhao, R., Zhang, Y., Zhao, L. Effect of multiple cycles of freeze-thawing on the RNA quality of lung cancer tissues. Cell Tissue Bank. 18 (3), 433-440 (2017).
- Yang, S., et al. Decontamination of ambient RNA in single-cell RNA-seq with DecontX. Genome Biol. 21 (1), 57(2020).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved