Method Article
Räumlich kompakte Anordnung von Zebrafischlarvenschnitten für die räumliche Transkriptomanalyse
In diesem Artikel
Zusammenfassung
Hier stellen wir eine Methode vor, mit der mehrere Zebrafisch-Larvenproben (Danio rerio) ausgerichtet und kryosektioniert und auf einem einzigen Objektträger für die räumliche transkriptomische Analyse gesammelt werden können.
Zusammenfassung
Räumliche Transkriptomtechniken sind ein ausgeklügeltes Werkzeug in der biomedizinischen Forschung, um räumlich registrierte Genexpressionsmuster sichtbar zu machen. Die Bildgebung und Analyse mehrerer Proben mit räumlichen Bildgebungsplattformen kann kostspielig sein. Die Durchführung dieser Tests unter mehreren experimentellen Bedingungen, wie sie in Entwicklungsstudien zu sehen sind, erhöht die Kosten weiter. Um die Kosten zu senken, wurde in dieser Studie versucht, die Techniken und Strategien der räumlichen transkriptomischen Probenanordnung für Entwicklungsstudien zu optimieren. Hier wurden in der Studie Zebrafische verwendet, die ein gut etabliertes Entwicklungswirbeltiermodell sind, das während der Entwicklung transparent ist, ~70% genetische Homologie zum Menschen aufweist und ein hochgradig annotiertes Genom hat, das sich ideal für die transkriptomische Analyse eignet. Aufgrund ihrer geringen Größe ermöglicht der sich entwickelnde Zebrafisch auch eine kompakte Platzierung von Serienabschnitten über mehrere biologische Replikate. Darin berichten wir über eine optimierte Fixierung, Kryosektion und zuverlässige Ausrichtung mehrerer Fischproben innerhalb des Bildgebungsbereichs einer räumlichen Multiplex-in-situ-Hybridisierungs-Bildgebungsplattform. Mit dieser Methode können Zebrafische bereits 15 Tage nach der Befruchtung (dpf) von mindestens 4 verschiedenen Schimmelpilzen und bis zu 174 Schnitten erfolgreich kryoschnittiert, innerhalb des Bildgebungsbereichs von 22 mm 10,5 mm (für einen räumlichen Transkriptom-Objektträger in situ ) entnommen und gleichzeitig verarbeitet werden. Basierend auf der Schnittqualität, der Probenausrichtung und der Probengröße pro Objektträger optimiert diese Methode beim Zebrafisch die Leistung und die Kosten pro Probe für räumliche Transkriptomtechniken.
Einleitung
Die Bewertung räumlich unterschiedlicher Expressionsmuster im Gewebe ist nach wie vor entscheidend für unser Verständnis genomischer Einflüsse auf Entwicklung, Krebs und Krankheit 1,2,3. Die räumliche Transkriptomik kombiniert gemultiplexte Expressionstechniken mit der räumlichen Registrierung der Expression in Geweben. "Räumliche Transkriptomik" wurde zuerst von Ståhl und Kollegengeprägt 4, bei denen montierte Krebsproben mit Hilfe von in situ Next-Generation-Sequencing untersucht wurden. Seitdem wird die "räumliche Transkriptomik" als Sammelbegriff für Hochdurchsatz-Expressionsstudien in Kombination mit räumlicher Registrierung verwendet. Dies sind zwar leistungsstarke Werkzeuge, aber auch teure Unternehmungen, die oft hohe institutionelle Investitionen und Laborkosten erfordern, bevor Daten generiert werden können5. Strategien zur Kostenminimierung bei gleichzeitiger Beibehaltung qualitativ hochwertiger Daten sind sehr gefragt.
Der Zebrafisch, Danio rerio, ist zu einem wichtigen Modellsystem für die Erforschung der Entwicklungsbiologie geworden und bietet die Möglichkeit, Analysen ganzer Organe (und Organismen) von Wirbeltieren auf begrenztem Raum zu vervielfachen. Zebrafische sind klein (4-6 mm als Larven und 2-3 cm als erwachsene Tiere) und können Hunderte von durchsichtigen Eiern auf einmal legen6. Zebrafischembryonen werden extern befruchtet und entwickeln sich schnell, was es den Forschern ermöglicht, Transgene in frühen Entwicklungsstadien einzuführen, um leicht Funktionsgewinn- und -verlustallele zu erzeugen7. Das Anpassen mehrerer Proben auf einem einzigen Objektträger ist eine attraktive Strategie, um Kosten zu senken. Ihre hohe Fruchtbarkeit und geringe Größe machen Zebrafische zu einem idealen Kandidaten für das Multiplexing von räumlichen Transkriptom-Assays, bei denen nur wenig Platz für Proben zur Verfügung steht8.
Die Kryosektion von Zebrafischlarven ist eine anspruchsvolle Technik. Viele räumliche transkriptomische Plattformen sind nicht für Zebrafisch-Paraffinschnitte optimiert und erfordern Kryosektionen, wenn mit Zebrafischen als Modellorganismus gearbeitet wird, um die Gewebestruktur zu erhalten und RNA-Transkripte zu erhalten. Darüber hinaus macht es die geringe Größe des Zebrafisches schwierig, qualitativ hochwertige Kryoschnitte zu erhalten und mehrere Proben effektiv zu analysieren. Schwieriger wird diese Aufgabe, wenn man mit Zebrafischlarven arbeitet, die kleiner und zerbrechlicher sind als ihre erwachsenen Artgenossen. Um diese Herausforderungen zu meistern, beschreiben wir eine Methode, die mehrere Proben zuverlässig ausrichtet und den Bildgebungsbereich von räumlichen Bildgebungsplattformen effizient nutzt, um viele hochwertige Schnitte auf einem einzigen Objektträger zu erhalten, die dann von räumlichen Bildgebungsplattformen abgebildet und analysiert werden können (Abbildung 1). In diesem Fall wird diese Methode auf eine räumliche transkriptomische Bildgebungsplattform angewendet.
Protokoll
Dieses Protokoll folgt den Richtlinien des institutionellen Ausschusses für Tierpflege und -nutzung des Dartmouth College.
1. Vorbereitung des Kryostaten
- Kühlen Sie den Kryostaten auf -22 °C ab und reinigen Sie die Innenflächen des Kryostaten, indem Sie Schmutz in den Behälter bürsten. Platzieren Sie alle notwendigen Bürsten und Werkzeuge in der Kammer.
2. Vorbereitung der Einweg-Basisform
- Bereiten Sie eine Basisform (37 mm, 24 mm, 5 mm Einweg-Kunststoffform) für die Probenausrichtung vor, indem Sie mit einem Permanentmarker, der als Referenzpunkt für die Probenausrichtung verwendet wird, eine gerade Linie über die Innenseite einer Basisform zeichnen. Legen Sie ein Stück Laborband auf die Innenseite der Grundform, wo die gerade Linie sein sollte, bevor Sie eine Linie zeichnen, um optimale Ergebnisse zu erzielen (Abbildung 2A).
- Zeichnen Sie einen Punkt auf die Innenseite der Grundform in der Nähe der linken oder rechten Wand, um die richtige Ausrichtung der Probe während der Kryosektion sicherzustellen (Abbildung 2B).
- Messen Sie den gewünschten Schnittwinkel mit einem Winkelmesser und markieren Sie diesen für jede Probe auf der Innenseite der Grundform (Abbildung 2C).
- Tragen Sie eine flache Schicht Gefriermedium (optische Kohärenztomographie [OCT]) auf die vorbereitete Grundform auf. Stellen Sie sicher, dass gerade genügend Gefriermedium vorhanden ist, um die Proben zu bedecken.
- Vermeiden Sie Luftblasen beim Auftragen des Gefriermediums, indem Sie die Düse der Mediumflasche grundieren und die erforderliche Menge Medium in eine Ecke der Grundform geben, bevor Sie die horizontale Ebene der Basisform so verschieben, dass das Medium gleichmäßig über die gesamte Oberfläche verteilt wird.
- Drücken Sie die Flasche langsam zusammen, wenn Sie das Gefriermedium ausgeben.
- Geben Sie Eis in ein 1-Liter-Becherglas, stellen Sie die Grundform mit dem Gefriermedium in das Eisbad und inkubieren Sie es mindestens 10 Minuten lang, um das Medium abzukühlen.
3. Vorbereitung von Trockeneis: 100% Ethanolbad
- Bereiten Sie ein Bad mit Trockeneis und 100 % Ethanol in einem Abzug vor, indem Sie einen Teil 100 % Ethanol zu einem Teil Trockeneis in einem Eiskübel hinzufügen.
- Verwenden Sie eine Einweg-Aluminiumschale oder falten Sie Alufolie zu einem Boot, das groß genug ist, um eine Einweg-Basisform aufzunehmen. Stellen Sie sicher, dass die Schale oder das Schiffchen groß genug ist, damit die Bodenform vollständig flach aufliegt.
- Stellen Sie die Schale oder das Boot in die Badewanne und decken Sie den Eimer ab. Lassen Sie den Eimer 5-10 Minuten abkühlen, bevor Sie die Proben einfrieren.
4. Einschläfern von Proben
- Wählen Sie zufällig Zebrafische für die Sektion aus. Wenn die Größe der Proben variiert, teilen Sie sie nach relativer Größe in Gruppen auf, um die genaue Ausrichtung zu erleichtern (siehe Diskussion für Details). Größere und kleinere Fische in getrennte Schalen geben.
- Füllen Sie ein Becherglas mit Wasser aus dem Fischsystem und stellen Sie das Becherglas in einen Eiskübel. Den Becher mit Eis umgeben.
- Überwachen Sie die Temperatur des Wassers mit einem Thermometer. Lassen Sie die Temperatur zwischen 2-4 °C stabilisieren.
- Verwenden Sie ein Netz oder Sieb, um eine Gruppe Fische in das 4 °C warme Wasser zu setzen. Fische sollten vollständig in Wasser getaucht werden und nicht mit Eis in Berührung kommen. Sobald die Bewegung der Opercularität aufgehört hat, gib Eis in das Wasser, um sicherzustellen, dass es unter 4 °C bleibt. Fisch 10 min in 4 °C heißem Wasser ziehen lassen.
HINWEIS: Fahren Sie mit dem Einbetten, Ausrichten und Schockfrosten jeder Gruppe euthanasierter Proben fort, bevor Sie die nächste Gruppe einschläfern. Ersetzen Sie das Wasser jedes Mal.
5. Einbettung und Ausrichtung
- Sammeln Sie die eingeschläferten Fische, nachdem sie 10 Minuten lang in 4 °C heißes Wasser getaucht wurden. Nehmen Sie die Fische mit einer feinen Pinzette aus dem Wasser, indem Sie sie an der Schwanzflosse packen und trocknen Sie sie, indem Sie sie vorsichtig gegen ein saugfähiges, fusselfreies Tuch drücken.
HINWEIS: Bei hochwertigen Abschnitten ist es wichtig, die Zeit zwischen der Entnahme der Fische aus dem 4 °C warmen Wasser und dem Schockfrosten zu begrenzen - Arbeiten Sie mit der vorbereiteten Basisform in einem Eisbad unter einem Stereomikroskop (10-fache Vergrößerung), legen Sie jede Probe entlang der Referenzpunkte in der richtigen Ausrichtung in die Basisform und bedecken Sie die Proben vorsichtig mit einer weiteren dünnen Schicht Gefriermedium.
HINWEIS: Füllen Sie nicht die gesamte Form mit Gefriermedium. Verwenden Sie stattdessen nur eine dünne Schicht, gerade genug, um alle Proben zu bedecken. - Verwenden Sie einen anatomischen Referenzpunkt, um den Fisch genau an den Linien auszurichten, die auf der Innenseite der Grundform markiert sind. Verwenden Sie eine Pinzette mit feiner Spitze, um die Ausrichtung jedes Fisches so einzustellen, dass sie ausgerichtet und in der gleichen Ausrichtung sind. Vermeiden Sie die Bildung von Blasen im Gefriermedium, indem Sie sich langsam bewegen.
- Tragen Sie ein Stück Trockeneis auf den Boden der Basisform unter den Proben auf, bis sie lokal eingefroren sind. Halten Sie die horizontale Ebene der Grundform waagerecht, um zu vermeiden, dass die Proben vor dem Einfrieren mit Trockeneis von ihren Referenzpunkten verschoben werden.
- Legen Sie die Basisform mit den Proben auf das Aluminiumschiffchen oder die Aluminiumschale in das Trockeneis: 100% Ethanolbad. Stellen Sie sicher, dass das Boot an der Oberfläche des Bades schwimmt und die Grundform trocken bleibt. Decken Sie das Bad ab und lassen Sie die Proben 10 Minuten lang schwimmen.
- Wickeln Sie die gefrorene Grundform in Folie ein und lagern Sie sie bis zum Schnitt bei -80 °C im Gefrierschrank. Wiederholen Sie die Schritte 4.2 bis 5.6 für alle verbleibenden Gruppen.
6. Kryosektion
- Bringen Sie gefrorene Basisformen zur Kryosektion in den vorgekühlten Kryostaten und legen Sie sie in die Kryostatenkammer. Transportieren Sie gefrorene Blöcke in einer Box mit Trockeneis, um ein Auftauen zu verhindern.
- Nehmen Sie den in situ Spatial Imaging Dia aus dem Lager und legen Sie ihn in einen vorgekühlten Objektträgerhalter. Lagern Sie den Objektträgerhalter mit dem Objektträger für die räumliche Bildgebung in der -22 °C heißen Kryostatkammer, bis Sie bereit sind, Schnitte aus dem interessierenden Bereich zu entnehmen.
- Nehmen Sie die gefrorenen Proben aus der Grundform und frieren Sie sie dann in einem Chuck mit einem frischen Gefriermedium ein. Frieren Sie sie auf dem Spannfutter ein, so dass die Schnittfläche zur Klinge zeigt.
- Legen Sie eine frische, feine Mikrotomklinge in den Kryostaten.
- Richten Sie die Form an der Klinge aus und trimmen Sie den interessierenden Bereich (empfohlene Beschnittstärke 20-50 μm). Stellen Sie sicher, dass die in der Form vorgenommenen Markierungen auf den gefrorenen Probenblock übertragen werden, um die Position in den Proben zu identifizieren.
- Stellen Sie die Form während der Trimmphase so ein, dass die Schnittfläche parallel zu den Referenzmarkierungen im Gefriermedium verläuft.
- Sammeln Sie Schnitte auf einem positiv geladenen Standard-Objektträger und überprüfen Sie sie mit Hellfeld, um zu bestätigen, wann ein Trimmen nicht mehr erforderlich ist.
- Nehmen Sie den Objektträger aus der Kryostatenkammer und legen Sie ihn in ein 4 °C heißes Eisbad. Bewahren Sie die Folie im Folienhalter auf. Achten Sie darauf, dass die Rutsche nicht nass wird.
- Beginn der Kryosektion (empfohlen 10-14 μm; Abbildung 3) und sammeln Sie Schnitte auf positiv geladenen Objektträgern, bis der Bereich von Interesse in den Proben erreicht ist. Überprüfen Sie die Schnitte nach Hellfeld, um sicherzustellen, dass der interessierende Bereich der nächste Abschnitt aus der Form ist.
- Bringen Sie den Einzelzell-Objektträger zurück in die Kryostatenkammer. Nehmen Sie das Objektträger aus dem Objektträgerhalter und sammeln Sie mit einem feinen Pinsel Reihe für Reihe Abschnitte aus dem genauen Interessenbereich auf dem Objektträger für die räumliche Bildgebung, um zu verhindern, dass sich die Abschnitte aufrollen.
- Drücken Sie mit der Rückseite des Pinsels eine Ecke des leeren, gefrierenden Mediums in den Messertisch, so dass der Abschnitt flach bleibt, wenn Sie den Objektträger zum Sammeln greifen.
- Verwenden Sie die Oberseite des Schlittens als Drehpunkt, senken Sie den Schlitten langsam auf den Abschnitt ab und lassen Sie die Teile 3 s lang am Schlitten haften, bevor Sie den Schlitten vom Messertisch abheben.
- Arbeiten Sie beim Sammeln von Abschnitten im Bildbereich des Objektträgers von links nach rechts und überlappen Sie nach Möglichkeit Schichten aus leerem, gefrierendem Medium.
- Verwenden Sie farbige Papierränder als Referenz, um Abschnitte innerhalb des Bildgebungsbereichs des Objektträgers zu platzieren, wenn das Gewebe schwer zu erkennen ist.
- Nachdem Sie Abschnitte aus dem interessierenden Bereich gesammelt haben, setzen Sie die Folie wieder in den Folienhalter ein. Wenn keine Proben mehr auf dem Objektträger für die räumliche Bildgebung entnommen werden müssen, lagern Sie den Objektträger bis zu 2 Wochen lang bei -80 °C, bis er für die Instrumentenanalyse mit der In-situ-Plattform für die räumliche Bildgebung bereit ist. Wenn Schnitte aus mehreren Formen entnommen werden müssen, legen Sie den Objektträger wieder in das 4 °C heiße Eisbad und wiederholen Sie die Schritte 6.3-6.11 mit der nächsten Form.
- Entnehmen Sie Schnitte vor und nach den "Region of Interest"-Schnitten von jeder Form auf einem positiv geladenen Standard-Objektträger für die Hämatoxylin- und Eosin (HE)-Färbung, um zu überprüfen, ob die Probenausrichtung und die Schnittqualität ausreichend sind, bevor Sie mit der Analyse fortfahren.
7. Fixieren der Probe
- Nehmen Sie die Referenzobjektträger aus dem Kryostaten und trocknen Sie sie 30 Minuten lang an der Luft, um die Abschnitte auf dem Objektträger zu kleben.
- Fixieren Sie die Abschnitte, indem Sie sie für 20 Minuten in einen Objektträgerbehälter mit 4 % Paraformaldehyd (Tabelle 1) legen.
- Waschen Sie die Abschnitte, indem Sie sie 3 Minuten lang in einen Schiebebehälter mit destilliertem Wasser legen.
- Fahren Sie mit der HE-Färbung fort oder trocknen Sie sie und lagern Sie die Objektträger bei -80 °C für zukünftige Färbungen.
8. HE-Färbung der Sektionen
- Dehydrieren und reinigen Sie die Abschnitte, indem Sie die Objektträger 2 Minuten lang in 100 % Ethanol, 2 Minuten lang in 95 % (Tabelle 2) Ethanol und dann 1 Minute lang in Leitungswasser inkubieren. Verwenden Sie ein Färbegestell für Objektträger, um Objektträger von Bad zu Bad zu übertragen.
- Färben Sie die Zellkerne und differenzieren Sie, indem Sie die Objektträger 2 min 45 s lang in Hämatoxylin inkubieren, 1 min lang in Leitungswasser, 1 min mit 0,3 % angesäuertem Alkohol (Tabelle 3) und dann 1 min lang mit fließendem Leitungswasser inkubieren. Verwenden Sie ein Färbegestell für Objektträger, um Objektträger von Bad zu Bad zu übertragen.
- Färben Sie die zytoplasmatischen Komponenten und dehydrieren Sie, indem Sie die Objektträger in Eosin Y 1% für 45 s, 50% Ethanol (Tabelle 4) für 1 min, 95% Ethanol für 1 min und 100% Ethanol für 1 min inkubieren. Verwenden Sie ein Färbegestell für Objektträger, um Objektträger von Bad zu Bad zu übertragen.
- Reinigen Sie die Abschnitte, indem Sie die Objektträger 1 Minute lang in Xylol inkubieren. Montieren und bedecken Sie die Objektträger, indem Sie mit einer Transferpipette einen Tropfen Eindeckmedium auf das obere Drittel des Objektträgers auftragen und ein Deckglas langsam mit einer Pinzette auf das Eindeckmedium absenken.
9. Räumliche Transkriptomik und Analyse der Schnitte
- Entnehmen Sie die Objektträger aus der Lagerung bei -80 °C und bilden Sie sie mit einer In-situ-Plattform für die räumliche Transkriptomanalyse ab.
HINWEIS: Die genauen Schritte der Bildgebung werden von der Spatial Imaging-Plattform bestimmt. - Überprüfen Sie die Qualitätskontrollmetriken der räumlichen Transkriptomik-Plattform. Wichtige Metriken, die überprüft werden müssen, sind die Anzahl der nachgewiesenen Zellen, die medianen Transkripte pro Zelle, die Kerntranskripte pro 100μm 2 und die Gesamtzahl der qualitativ hochwertigen dekodierten Transkripte jedes Gens im Sondensatz.
HINWEIS: Für diese Qualitätskontrollmetriken gibt es keine universellen Schwellenwerte, und die Erwartungen an diese Schwellenwerte variieren je nach verwendeter Probe und Genpanel. - Lesen Sie die Datenausgabe von nachweisbaren RNA-Transkripten, bestimmen Sie anhand der Qualitätskontrollmetriken des Experiments, welche Transkripte von geringer Qualität sind, und filtern Sie Transkripte von geringer Qualität heraus. Analysieren Sie die verbleibenden hochwertigen Transkripte in Bezug auf ihre räumliche Anordnung innerhalb des Abschnitts.
- Sehen Sie sich die zelluläre Segmentierung der Schnitte und Clusterzellen basierend auf experimentellen Interessen an. Vergleichen Sie die RNA-Transkripte innerhalb von Zellclustern der interessierenden Region über Gruppen unterschiedlichen Alters von Zebrafischen auf demselben Objektträger.
Ergebnisse
Bei dieser Methode (Abbildung 1) wird der Zebrafisch als Tiermodell verwendet, um nach räumlich aufgelösten Genexpressionsmustern zu suchen. Die effiziente Kryosektion von Zebrafischlarven für die räumliche Bildgebung ist eine Herausforderung. Die Schnitte müssen von hoher Qualität sein, um die Gewebestruktur und die nachweisbaren Gene zu erhalten (Abbildung 4). Schnitte mit mehreren Proben für eine räumlich effiziente Bildgebung müssen präzise ausgerichtet werden, um die interessierenden Bereiche aller Proben zu analysieren (Abbildung 2 und Abbildung 5). Schließlich müssen die Schnitte gesammelt und effizient verteilt werden, um die Anzahl potenzieller Datenpunkte innerhalb des Bildgebungsbereichs eines räumlichen Transkriptomik-Objektträgers zu maximieren (Abbildung 3 und Abbildung 6). Die Experimente wurden anhand dieser drei Parameter gemessen, um festzustellen, ob sie erfolgreich oder suboptimal waren.
Viele Protokollmodifikationen trugen dazu bei, dass wir in der Lage waren, qualitativ hochwertige Schnitte zu sammeln, die gute Kandidaten für die räumliche transkriptomische Bildgebung sind. Die Anpassung der Protokolle für Euthanasie, Fixierung und Einbettung führte zu einer Verbesserung der Schnittqualität (Abbildung 4). Im ersten Protokoll (Abbildung 4, Protokoll 1) war die Schnittqualität suboptimal, und das Gewebe behielt seine Struktur nicht bei. Um dies zu korrigieren, wurde im zweiten Protokoll (Abbildung 4, Protokoll 2) ein Schockfrostschritt nach dem Einbetten von Proben und fixiertem Gewebe nach der Entnahme von Schnitten auf Objektträger eingeführt. Dadurch wurde die Gewebestruktur der Schnitte insgesamt verbessert, aber nicht die Qualität erreicht, die für die räumliche Transkriptomik als akzeptabel angesehen wurde. Im dritten Protokoll (Abbildung 4, Protokoll 3) wurden die Euthanasietechniken schließlich verfeinert, indem die Zeit verkürzt wurde, die für die Einschläferung der Fische und das Einbetten der Fische auf Eis vor dem Schockfrosten und der anschließenden Sektion und Fixierung nach der Entnahme benötigt wurde. Das Ergebnis waren Proben von höchster Qualität, die sich gut für die räumliche transkriptomische Bildgebung eigneten.
Die Ausrichtung mehrerer Proben auf eine Region of Interest erforderte ebenfalls einige Änderungen am Protokoll. Eine der Modifikationen, die die Ausrichtung zwischen den Proben am meisten verbesserte, war die Einführung von Referenzpunkten in die Basisformen, in die der Zebrafisch eingebettet ist (Abbildung 2). In den Experimenten, die in dieser Studie durchgeführt wurden, wurden die Proben ausgerichtet, indem Strukturen über mehrere Schnitte im selben Schnitt verglichen wurden (Abbildung 5).
Der letzte wichtige Schritt bei der Entwicklung der Methode für eine effiziente räumliche Transkriptomik bestand darin, möglichst viele dieser hochwertigen, ausgerichteten Proben unterschiedlichen Alters für die Bildgebung auf denselben Objektträger zu bringen. Das Sammeln von Erfahrungen und die Verbesserung der technischen Fähigkeiten tragen dazu bei, dass die Anzahl der entnommenen Proben verbessert wird. Diese Verbesserungen sind vor allem auch auf geringfügige Anpassungen der Einbettungs- und Erfassungsschritte im Protokoll zurückzuführen. Diese Felder in Abbildung 6 zeigen die Anzahl der Schnitte, die im Zeitverlauf im Bildgebungsbereich jedes Objektträgers erfasst wurden. Abbildung 6A (40 Schnitte von 15 dpf Fischen) ist ein früher Lauf, bei dem eine Form während des Schnitts verwendet wird. Abbildung 6B (90 Schnitte von 15 dpf Fischen und 47 Schnitte von 26 dpf Fischen) führt eine zweite Form ein und zeigt eine Verbesserung der räumlichen Anordnung durch Reduzierung des Abstands zwischen den Proben während der Einbettung. Abbildung 6C (54 Schnitte von 15 dpf Fischen, 80 Schnitte von 19 dpf Fischen, 24 Schnitte von 23 dpf Fischen und 21 Schnitte von 26 dpf Fischen) führt eine dritte und vierte Form ein und zeigt eine kontinuierliche Verbesserung der räumlichen Anordnung, indem die Schnittfläche des Blocks um die Proben herum abgeschnitten und leeres, gefrierendes Medium überlappt wird. Diese Verbesserungen gaben uns die Möglichkeit, 179 Schnitte im Bildgebungsbereich unseres räumlichen Transkriptom-Objektträgers zu platzieren und Fische in 4 verschiedenen Altersstufen zu testen.
Nach Fertigstellung des Protokolls hatten wir Proben, die sowohl mit der handelsüblichen Multiplex-In-situ-Software als auch mit kundenspezifischen Methoden leicht analysiert werden konnten. Die hier verwendete Multiplex-Software verwendete einen Pseudo-phred-Score, der lose auf phred-Qualitätswerten in Sequenzierungsdaten basiert9. Die Punktzahlen reichten von 59 % bis 70 %, wobei 60 % der Grenzwert für eine Warnung vor Lesevorgängen mit geringerer Qualität waren. Bei der Inspektion war der niedrige Pseudo-Phred-Score auf die geringe Signalkomplexität im leeren Raum zwischen den Abschnitten zurückzuführen. Obwohl der Folienbereich effizient genutzt wurde, war der Rutschenbereich immer noch zu >50 % leer. Unter Berücksichtigung des Anteils der Objektträgerfläche, die von Gewebe eingenommen wurde, wo das Signal ausreichend komplex war, war die Signalqualität ausgezeichnet.
Schwache, unspezifische Signale wurden auch in zwei allgemeinen Regionen auf dem Objektträger beobachtet: außerhalb der Proben und innerhalb der leeren Chorda (Abbildung 7A). Außerhalb der Schnitte gab es ein Muster, das mit einem Waschartefakt übereinstimmte, das die Entfernung von den Proben verringerte (Abbildung 7B). Ein unspezifisches Signal wurde auch in den leeren Bereichen der Exemplare beobachtet, die von der Chorda besetzt sind. Dies wurde als wahrscheinliches Waschartefakt angesehen, bei dem Amplifikationsreagenzien eingeschlossen werden könnten. Die Transkripte in beiden Regionen waren weitgehend zytoplasmatisch (d. h. ribosomale Proteine L3 und L4) oder mitochondrial (Aspartattransferase und Isocitrat-Dehydrogenase). Wenn diese Regionen in die Baysor-Segmentierung einbezogen wurden, die Zellen anhand der Lokalisierung und Clusterbildung von Transkripten um den Zellkern10 identifiziert, umfasste die Segmentierungsanalyse die Zellgrenzen, die manchmal auch diese einschlossen. Dieses Signal war so gering, dass es leicht von der Probe unterschieden werden konnte.
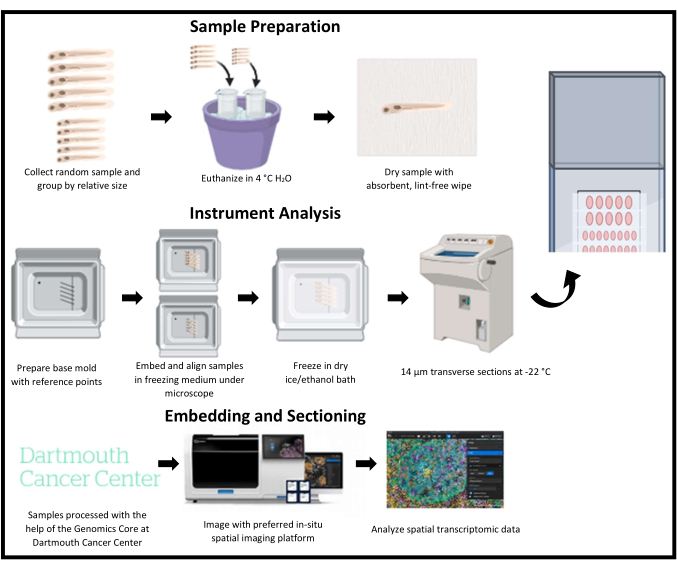
Abbildung 1: Schematische Darstellung der Kryosektion für die räumliche Transkriptomik im Zebrafisch. Jungliche Zebrafische werden nach dem Zufallsprinzip gesammelt und dann nach relativer Größe gruppiert. Die Proben werden dann in ein Gefriermedium eingebettet und durch Referenzpunkte auf der Basisform ausgerichtet, die einem interessierenden Bereich entsprechen. Jede Form wird schockgefroren, kryosektioniert und reihenweise auf demselben Objektträger gesammelt. Die Objektträger werden nach dem Schnitt bei -80 °C gelagert und mit einer räumlichen Transkriptom-Bildgebungsplattform abgebildet und analysiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Festlegen von Referenzpunkten für eine präzise Schnitteinteilung. Eine Reihe von Bildern zeigt den Prozess des Hinzufügens von Referenzpunkten zu einer Einweg-Basisform, um mehrere Proben für die Kryosektion auszurichten. Der erste Schritt (A) besteht darin, mit Laborklebeband eine Linie auf der Innenseite der Form zu markieren, die alle Proben als Referenz für die Ausrichtung verwenden. (B) Platzieren Sie einen Punkt auf der Innenseite der Form in der Nähe der linken oder rechten Wand, um die richtige Probenausrichtung während der Kryosektion zu gewährleisten, und markieren Sie dann (C) Linien für jede Probe in einem gewünschten Winkel. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
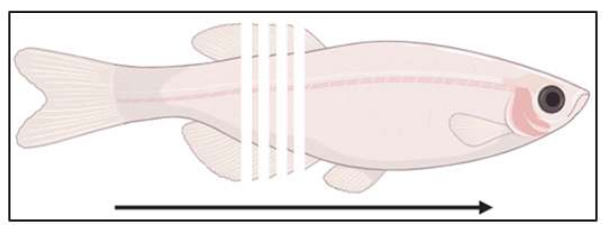
Abbildung 3: Schematische Darstellung der Schnittstelle. Die Fische werden koronal, von kaudal bis rostral, im Abstand von 14 μm geschnitten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
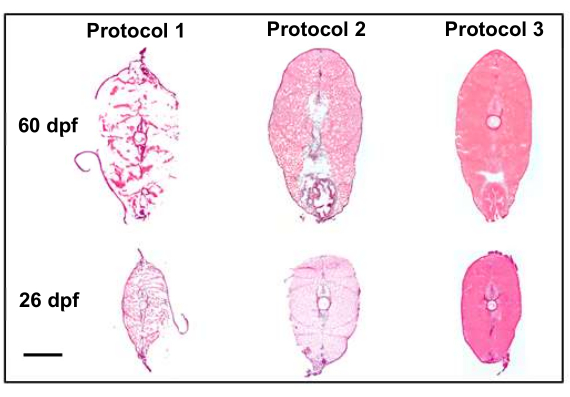
Abbildung 4: Verbesserung der Schnittqualität durch Protokollmodifikationen (10x). Die Abbildung vergleicht die ersten beiden Protokolle, die zu einer suboptimalen Gewebequalität führten, mit dem endgültigen Protokoll, das zu qualitativ hochwertigen Schnitten führte (die die Morphologie beibehielten), die gute Kandidaten für die räumliche Transkriptomik sind. Protokoll 1 verwendet die vergleichende Analyse8 des Penn State Bioatlas, die durch eine 4%ige Paraformaldehyd-Fixierung vor der Schnittierung hervorgehoben wird. Protokoll 2 führt das Trockeneis/Ethanol-Schockfrosten nach der Euthanasierung und die Fixierung nach dem Schnitt ein, wodurch die Gewebestruktur viel besser erhalten bleibt, mit dem Vorbehalt des Einfrierens von Artefakten während des gesamten Schnitts. Protokoll 3 verfeinerte die Euthanasietechniken und die Einbettung auf Eis vor dem Schockfrosten und der anschließenden Sektion und Fixierung nach der Entnahme, was zu den qualitativ hochwertigsten Proben führte. Der Maßstabsbalken beträgt 500 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Überprüfung der Ausrichtung mehrerer Abschnitte (20x). Repräsentative HE-Bilder von 26 dph-Schnitten, die die Ausrichtung zwischen mehreren Proben in einem einzigen Schnitt hervorheben. Der Maßstabsbalken beträgt 500 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 6: Verbesserte räumliche Anordnung der Proben. In diesen Fenstern wird die Anzahl der Abschnitte hervorgehoben, die im Laufe der Zeit im Bildbereich jedes Objektträgers erfasst wurden. Das Sammeln von Erfahrungen und die Verbesserung der technischen Fähigkeiten tragen dazu bei, dass die Anzahl der entnommenen Proben verbessert wird. Diese Verbesserungen sind vor allem auch auf geringfügige Anpassungen der Einbettungs- und Erfassungsschritte im Protokoll zurückzuführen. Panel A (40 Abschnitte mit 15 dpf Fischen) ist ein früher Lauf, bei dem während des Schneidens eine Form verwendet wird. Panel B (90 Schnitte von 15 dpf Fischen und 47 Schnitte von 26 dpf Fischen) führt einen zweiten Schimmel ein und zeigt eine Verbesserung der räumlichen Anordnung, indem der Abstand zwischen den Proben während der Einbettung reduziert wird. Panel C (54 Schnitte mit 15 dpf Fischen, 80 Schnitte mit 19 dpf Fischen, 24 Schnitte mit 23 dpf Fischen und 21 Schnitte mit 26 dpf Fischen) führt eine dritte und vierte Form ein und zeigt eine kontinuierliche Verbesserung der räumlichen Anordnung, indem die Schnittfläche des Blocks um die Proben herum beschnitten und das leere, gefrierende Medium überlappt wird. Die Klammern in den einzelnen Paneelen stellen unterschiedliche Formen dar, die beim Trennen verwendet werden. Die Abschnitte befinden sich auf einer Fläche von 10,5 mm und 22 mm. Der Maßstabsbalken beträgt 4 mm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 7: Repräsentative Ergebnisse unseres räumlichen Transkriptom-Experiments. (A) Ein einzelnes Bild eines Objektträgers, das mit dem Protokoll verarbeitet wurde, wie in Abbildung 6C zu sehen. (B) 14-μm-Schnitte eines Zebrafisches bei 15 dpf nach räumlicher Transkriptombildgebung, die ein unspezifisches Signal in der Nähe von Schnitten und innerhalb der Chorda zeigten. Die Maßstabsleiste beträgt 4 mm in Feld A. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| 4% Paraformaldehyd | Bestandskonzentration | Menge | Endkonzentration |
| Paraformaldehyd (Pulver) | 95 % w/w | 40 g | 4 % w/v |
| NaOH | 1 N | Tropfen (bis sich das Pulver aufgelöst hat) | |
| Hcl | 1 N | Tropfen (bis 6,9 pH) | |
| PBS 1x Aktie | 1x | ~1000 mL (1000 mL Gesamtvolumen erreichen) | 1x |
| Gesamt | ca. 1000 mL |
Tabelle 1: Formel mit 4 % Paraformaldehyd. Zusammensetzung aus 4%iger Paraformaldehydlösung zum Fixieren von Proben.
| 95% Ethanol | Bestandskonzentration | Menge | Endkonzentration |
| 200 Proof Ethanol | 100% V/V | ca. 190 mL | 95 % v/v |
| RO-Wasser | 100% V/V | 10 mL | 5 % v/v |
| Gesamt | 200 mL |
Tabelle 2: 95% Ethanol-Formel. Zusammensetzung einer 95%igen Ethanollösung zur Dehydrierung von Schnitten während der H&E-Färbung.
| 0,3% Säuerlicher Alkohol | Bestandskonzentration | Menge | Endkonzentration |
| Eisessig | 100% V/V | 300 μL | 0,3 % v/v |
| RO-Wasser | 100% V/V | 99,7 mL | 99,7 % v/v |
| Gesamt | 100 mL |
Tabelle 3: 0,3 % Säuerungsmittel Alkoholformel. Zusammensetzung von 0,3 % angesäuertem Alkohol zur Unterscheidung während der HE-Färbung.
| 50% Ethanol | Bestandskonzentration | Menge | Endkonzentration |
| 200 Proof Ethanol | 100% V/V | 100 mL | 50 % v/v |
| RO-Wasser | 100% V/V | 100 mL | 50 % v/v |
| Gesamt | 200 mL |
Tabelle 4: 50 % Ethanol-Formel. Zusammensetzung von 50% Ethanol zur Dehydrierung von Schnitten während der HE-Färbung.
Diskussion
Dieser Bericht bietet detaillierte Lösungen für viele der technischen Herausforderungen, die mit dem Zebrafisch als Modellorganismus in der räumlichen Transkriptomanalyse während der Entwicklung verbunden sind. Um diese Herausforderungen zu bewältigen, optimiert unsere kompakte Probenanordnung die Kosten auf den neu entstehenden räumlichen Transkriptomplattformen1. Die Kryosektion von Zebrafischlarven für die räumliche Bildgebung ist eine Herausforderung. Die Schnitte sollten eine ausreichende Gewebestruktur und Transkriptqualität aufweisen, um eine zufriedenstellende experimentelle Durchführung und nachgelagerte räumliche Analyse zu ermöglichen8. Schnitte, die mehrere Proben enthalten, müssen für eine räumlich effiziente Bildgebung präzise ausgerichtet werden, um gemeinsame Bereiche von Interesse über alle Proben hinweg zu analysieren. Schließlich müssen die Schnitte effizient erfasst und angeordnet werden, um die Anzahl potenzieller Datenpunkte innerhalb des vorgesehenen Bereichs eines Objektträgers zu maximieren.
Das Protokoll ermöglicht die Sammlung von qualitativ hochwertigen Schnitten, die sich hervorragend für räumliche Einzelzellbildgebungsplattformen eignen. Die erste Modifikation bestand darin, Zebrafische schockgefroren und dann Abschnitte in 4 % Paraformaldehyd zu fixieren, nachdem sie auf einem Objektträger gesammelt worden waren. Ursprünglich wurden Zebrafische über Nacht bei 4 °C mit 4% Paraformaldehyd fixiert und anschließend eingebettet und geschnitten11. Die Entscheidung, Proben nach der Sektion zu fixieren, trug dazu bei, internes Gewebe zu erhalten, das bei der Fixierung einer ganzen Probe nicht effektiv konserviert wurde (Abbildung 4).
Die zweite entscheidende Modifikation war die Verfeinerung der Euthanasiemethode, um die Integrität des Gewebes zu erhalten. Zebrafische wurden ursprünglich für 20 Minuten in 4 °C heißes Wasser gelegt, um sicherzustellen, dass sie ausreichend eingeschläfert wurden. Dies liegt 10 Minuten über den Mindestanforderungen der IACUC-Richtlinien. Diese zusätzliche Zeit in 4 °C heißem Wasser gab den Proben mehr Zeit zur Zersetzung und trug zu einer nicht idealen Schnittqualität von sehr feinen oder empfindlichen inneren Strukturen bei. Die Reduzierung der Zeit in 4 °C heißem Wasser auf die Mindestanforderung von 10 Minuten trug dazu bei, empfindliche innere Strukturen zu erhalten, die bei früheren Schnittversuchen fehlten. Zu den weiteren Modifikationen, die in Abbildung 4 nicht dargestellt sind, gehören das Schneiden am selben Tag wie die Einschläferung, die Verkürzung der Zeit zwischen der Einschläferung und dem Schockfrosten von 15 Minuten auf unter 5 Minuten und die Arbeit mit einem vorgekühlten Gefriermedium beim Einbetten. Je länger die Proben vor dem Schnitt bei -80 °C gelagert wurden, desto schlechter war die Gewebequalität des Schnitts. Die Unterschiede waren subtil, aber es ist ideal für die Sektion am selben Tag wie die Euthanasie und die Blockbildung. Die Verkürzung der Zeit, die zum Ausrichten der Proben nach der Einschläferung und vor dem Schockfrosten benötigt wird, auf nicht mehr als 5 Minuten ist entscheidend für die Schnittqualität.
Die Ausrichtung mehrerer Proben auf eine Region of Interest erforderte ebenfalls einige Änderungen am Protokoll. Die erreichte Ausrichtungsgenauigkeit (Abbildung 5) gruppierte die Stichproben nach relativer Größe und leistete den größten Beitrag zu einer effizienten räumlichen Anordnung. Die primäre Strategie zum Ausrichten der Proben besteht darin, den Zebrafisch auf Referenzpunkte zu legen, die auf die Basisformen gezeichnet werden, in die er eingebettet ist. Die Proben werden durch anatomische Strukturen innerhalb der Exemplare des Zebrafisches an diesen Referenzpunkten ausgerichtet, die je nach gewünschter Region variieren können. Sich entwickelnde Fische im gleichen Alter können in der Größe bis zu 50 % variieren12. Proben mit drastisch unterschiedlichen Größen können aufgrund unterschiedlicher Abstände zwischen dem anatomischen Referenzpunkt und der interessierenden Region falsch ausgerichtet werden. Das Gruppieren von Proben gleichen Alters nach relativer Größe und das Einbetten jeder Größe in ihre Formen verbesserte die Fähigkeit, mehrere Proben innerhalb ihrer Form auszurichten. Mit dieser Methode können etwa 2/3 der Proben zuverlässig ausgerichtet werden, abhängig von dem Fenster, das die interessierende Region und die Wahl des anatomischen Referenzpunkts zulassen.
Die Reduzierung des Abstands zwischen den Proben beim Einbetten und Ausrichten führt zu einer größeren Anzahl von Proben pro Objektträger. Diese enge Konfiguration wird jedoch durch die Passermarkengrenzen im Bildgebungsbereich weiter eingeschränkt. Neben den Erfahrungen am Kryostaten werden verschiedene Strategien beschrieben, um diese in Einklang zu bringen. Erstens kann die präzise Platzierung des überlappenden Einbettmediums zwischen den Abschnitten und das Trimmen der gefrorenen Form tiefgreifende Auswirkungen auf die Anzahl der Abschnitte haben, die in einen bestimmten Bildgebungsbereich passen. Abbildung 6 zeigt die Anzahl der Schnitte, die vor und nach der Optimierung der Einbettungsanordnung erhalten wurden. Zweitens wurde während der Einbettung und vor dem Einfrieren eine Grenze an der Außenseite der äußersten Proben platziert, um die korrekte Platzierung auf dem Objektträger zu unterstützen. Drittens kann es aufgrund der Größe und Transparenz von Zebrafischlarven schwierig sein, zu wissen, wo sich Proben innerhalb eines Objektträgers befinden, damit sie präzise auf einem Objektträger gesammelt werden können. Wir stellten fest, dass die Verwendung von farbigem Papier als Kontrast zum weißen Gefriermedium, um eine Begrenzung an der Außenseite der äußersten Proben zu schaffen, zu einer viel präziseren Platzierung der Abschnitte auf den Objektträgern führte.
Die Nuancen der lokalen Temperatur helfen bei der Konservierung der Probe während des Schneidprozesses. Sobald Proben aus mehreren Formen auf einem einzigen Objektträger gesammelt wurden, wird der Objektträger bei 4 °C gelagert, die nächste Form wird auf den gewünschten Interessenbereich getrimmt, bevor sie erneut entnommen wird. Der Objektträger muss etwas wärmer sein als die Schnitte, damit er richtig auf der Oberfläche des Objektträgers haftet. Daher ist die Lagerung des Objektträgers bei -80 °C oder im Kryostaten bei -20 °C zwischen der Probenentnahme aus verschiedenen Formen nicht ideal. Es ist auch wichtig, dass die gesammelten Proben auf dem Objektträger nicht mehreren Gefrier-/Auftauzyklen unterzogen werden, da sich dies negativ auf die Qualität der RNA-Transkripte in jeder Probe auswirken kann. Wenn der Objektträger mit zuvor entnommenen Schnitten bei 4 °C gehalten wird, bleiben die RNA-Transkripte angemessen erhalten, während eine niedrigere Temperatur für eine ordnungsgemäße Adhärenz ohne negative Nebenwirkungen im Zusammenhang mit Gefrier-/Auftauzyklen beibehaltenwird 13.
Obwohl zufriedenstellende Ergebnisse erzielt wurden, ist das Design dieser Methode mit möglichen Einschränkungen verbunden. Die erste Einschränkung besteht darin, dass wir auf den Bildgebungsbereich eines räumlichen Transkripto-Objektträgers beschränkt sind, was eine Obergrenze für die Gesamtzahl der Abschnitte festlegt, die auf demselben Objektträger abgebildet werden können. Angesichts der Tatsache, dass wir immer noch leeren Raum zwischen den Schnitten haben, ist es wahrscheinlich, dass wir die Grenzen dessen, was mit dieser Methode möglich ist, noch nicht erreicht haben, aber wir erkennen, dass es einen Punkt gibt, an dem es nicht mehr möglich ist, weitere Schnitte auf den Objektträger einzupassen. Die Arbeit mit größeren Fischen in höherem Alter begrenzt auch die Gesamtprobengröße, sollte aber nicht die Gesamtmenge an Gewebe beeinflussen, die in den Bildgebungsbereich eines Objektträgers passt. Technische Verbesserungen durch räumliche Bildgebungsplattformen könnten möglicherweise die Gesamtmenge an Gewebe erhöhen, die möglicherweise abgebildet werden könnte, indem der Bildgebungsbereich auf den Objektträgern vergrößert wird. Eine weitere Einschränkung dieser Methode besteht darin, dass das Protokoll nicht für Fische unter 15 dpf optimiert wurde. Es wurde festgestellt, dass Fische umso zerbrechlicher werden, je jünger sie sind, und dass Fische, die jünger als 15 dpf sind, extrem schwer zu handhaben sind und während des Einbettungsprozesses leicht beschädigt werden können. Dies führte zu einer Verlängerung der Zeitspanne zwischen der Euthanasierung und dem Schockfrosten, was sich nachteilig auf die Gewebequalität auswirkte. Dadurch verringerte sich auch der Prozentsatz lebensfähiger Proben, die für die Kryosektion übrig blieben, und die wenigen lebensfähigen Fische, die für die Schnitte übrig blieben, waren nicht steif genug, um die Gewebestruktur für eine qualitativ hochwertige Analyse zu erhalten. Die Suche nach Werkzeugen, die besser für den Umgang mit empfindlicheren Fischen geeignet sind, und die Arbeit mit einem weniger dichten Gefriermedium, das auch die Steifigkeit der Proben während des Schneidens erhöht, könnte es ermöglichen, diese Methode auf Fische unter 15 dpf anzuwenden. Die letzte große Einschränkung dieser Methode ist die Probenausrichtung für sehr kleine Bereiche von Interesse. Wir stellten fest, dass je kleiner der interessierende Bereich in der Stichprobe war, desto unwahrscheinlicher war es, dass wir sie richtig ausrichten konnten. Wir arbeiteten mit einem Bereich of Interest, der etwa 50 μm groß ist, und dies führte dazu, dass etwa 2/3 der Abschnitte richtig ausgerichtet waren. Proben mit einem kleineren Bereich von mehr als 50 μm können dazu führen, dass weniger als 2/3 der Schnitte ausgerichtet werden. Ein Großteil der falsch ausgerichteten Proben stammt aus dem Einbettungsschritt im Protokoll. Es ist schwierig, eine Probe im Gefriermedium zu justieren, ohne andere Proben zu bewegen, die bereits ausgerichtet wurden. Es gibt auch eine begrenzte Zeitspanne, die der Forscher hat, um diese Proben auszurichten, bevor er sich negativ auf die Gewebequalität auswirkt. Schließlich werden die Formen vom Stereomikroskop in das Schockfrostbad gebracht, und es ist möglich, dass sich die Proben in der Form verschieben, bevor sie vollständig eingefroren sind. Es ist möglich, dass die Arbeit mit einem kälteren Gefriermedium es dem Forscher ermöglicht, eine Probe leichter zu bewegen, ohne andere Proben zu beeinträchtigen, die bereits ausgerichtet sind. Durch das Schockfrosten der Form, ohne dass sie nach dem Ausrichten bewegt werden muss, kann eine Verschiebung der Proben vermieden werden, bevor sie vollständig an Ort und Stelle eingefroren sind. Wir identifizierten unspezifische Signale außerhalb der Proben.
Das Endprodukt und die erste Nachanalyse enthielten zusätzliche Lehren. Es waren unspezifische Signale vorhanden, die eine Berücksichtigung erforderten. Nach Angaben des Herstellers sind einige Transkripte mit niedrigem Signalgehalt ein bekanntes Artefakt, und Umgebungs-RNA kann in Einzelzell-RNA-Seq gesehen werden, die nach der Analyse behandelt werden kann14. Das hier verwendete Protokoll könnte zu den Umgebungstranskripten beigetragen haben, da die Fixierung nach der Platzierung der Proben erfolgte. Es wurde jedoch festgestellt, dass eine frühe Fixierung zu einer schlechten und inkonsistenten Morphologie für diese jungen Altersgruppen führte. Alternativ könnten Amplifikationsreagenzien, die gepoolt werden, einen solchen Effekt erzeugen. Qualitativ war das unspezifische Signal, das in der Chorda zu sehen war, am ausgeprägtesten. Während der Prozessierung war die Notochord die einzige wirklich leere Region, da sie keine Einhängemedien und Transkriptsignale enthielt, die den zytoplasmatischen und mitochondrialen Profilen benachbarter Notochordauskleidungsgewebe ähnelten. Dies deutet darauf hin, dass eine physikalische "Rückseite" (durch Gewebe oder Einhängemedien) hilfreich ist, um dieses Signal zu reduzieren. Letztendlich sind wir der Meinung, dass der Ausschluss des Signals in der Nachbearbeitung eine zufriedenstellende Strategie ist. Die unspezifischen Signalmerkmale waren nach Experimenten leicht erkennbar und konnten in der nachgelagerten Analyse gemildert werden, z. B. durch manuellen Ausschluss unspezifischer Signale von Regionen außerhalb der Probe.
Zebrafische mit einem Alter von nur 15 dpf aus mindestens 4 verschiedenen Formen (und Altersgruppen) können erfolgreich kryoschnittiert und im räumlichen transkriptomischen Bildgebungsbereich entnommen werden, und bis zu 179 Schnitte können gleichzeitig verarbeitet werden. Basierend auf der Schnittqualität, der Probenausrichtung, der Probengröße und der Qualität der RNA-Transkripte während der Bildgebung schließen wir, dass diese Methode ein effektives Protokoll für die Durchführung effizienter räumlicher Transkriptom-Assays ist. Forscher können diese Methode nutzen, um die mit der räumlichen Bildgebung verbundenen Kosten zu senken, die Probengröße bei räumlichen Transkriptom-Assays zu erhöhen und sogar große Experimente in einem einzigen Lauf durchzuführen.
Offenlegungen
Die Autoren haben keine Offenlegungen oder Interessenkonflikte in Bezug auf diesen Bericht.
Danksagungen
Schnitte und Bildgebung wurden mit Instrumenten durchgeführt, die von gemeinsamen Ressourcen des Dartmouth Cancer Center zur Verfügung gestellt wurden, finanziert durch den NCI Cancer Center Support Grant 5P30CA023108, und des Center for Quantitative Biology am Dartmouth College (NIGMS COBRE).
Materialien
| Name | Company | Catalog Number | Comments |
| 1 L Beaker | Pyrex | 1003 | |
| 200 proof pure ethanol | Koptec | V1001 | |
| Acetic acid, glacial | VWR | 0714 | acidified alcohol |
| Aluminum foil | |||
| Cover slips | Epredia | 24X50-1.5-001G | |
| Disposable base mold | Fisher HealthCare | 22-363-556 | |
| Distilled water | |||
| DPX mountant | Sigma-Aldrich | 06522 | mountant for histology |
| Dry ice pellets | |||
| Dumont #5SF Forceps | Fine Science Tools | 11252-00 | |
| Eosin-Y Alcoholic | Epredia | 71204 | Eosin Y 1% |
| Gill 1 Hematoxylin | Epredia | 72411 | Hematoxylin |
| Kimwipe | Kimberly-Clark Professional | 34120 | absorbent, lint-free wipe |
| Lab labelling tape | VWR | 89097-934 | |
| Microtome blade MX35 Ultra | Epredia | 3053835 | |
| Microtome Cryostat | Thermo Scientific | Microme HM 525 | |
| O.C.T. Compound | Fisher HealthCare | 23-730-571 | freezing medium |
| Paraformaldehyde | Sigma-Aldrich | 158127 | PFA |
| Permanent Marker | VWR | 52877-886 | |
| Protractor | |||
| SafeClear Xylene Substitute | Fisherbrand | 68551-16-6 | Xylene substitute |
| Single Edge Blades | American Line | 66-0407 | |
| Steriomicroscope | Zeiss | 4350639000 | Stemi 305 w/ double spot LED (4355259020) and Stand K lab (4354259010) |
| Superfrost Plus Micro Slides | VWR | 48311-703 | |
| Transfer pipet | |||
| Xenium V1 slide | 10X/Xenium | 3000941 | spatial transcriptomic imaging slide |
Referenzen
- Rao, A., Barkley, D., França, G. S., Yanai, I. Exploring tissue architecture using spatial transcriptomics. Nature. 596 (7871), 211-220 (2021).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet. 22, 627-644 (2021).
- Duhan, L., et al. Single-cell transcriptomics: background, technologies, applications, and challenges. Mol Biol Rep. 51, 600 (2024).
- Ståhl, P. L., et al. Visualization and analysis of gene expression in tissue sections by spatial transcriptomics. Science. 353 (6294), 78-82 (2016).
- Colman, R. E., et al. Whole-genome and targeted sequencing of drug-resistant Mycobacterium tuberculosis on the iSeq100 and MiSeq: A performance, ease-of-use, and cost evaluation. PLOS Med. 16 (4), e1002194 (2019).
- Marques, I. J., Lupi, E., Mercader, N. Model systems for regeneration: zebrafish. Development. 146 (18), dev167692 (2019).
- Mohideen, M. P. K., et al. Histology-based screen for zebrafish mutants with abnormal cell differentiation. Dev Dyn. 228 (3), 414-423 (2003).
- Copper, J. E., et al. Comparative analysis of fixation and embedding techniques for optimized histological preparation of zebrafish. Comp Biochem Physiol C Toxicol Pharmacol. 208, 38-46 (2018).
- Stock, R. J., Labudovich, M., Ducatman, B. Asymptomatic first-trimester liver cell adenoma: diagnosis by fine-needle aspiration cytology with cytochemical and ultrastructural study. Obstet Gynecol. 66 (2), 287-290 (1985).
- Petukhov, V., et al. Cell segmentation in imaging-based spatial transcriptomics. Nat Biotechnol. 40 (3), 345-354 (2022).
- Meyer, T., Tiburcy, M., Zimmermann, W. H. Cardiac macrotissues-on-a-plate models for phenotypic drug screens. Adv Drug Deliv Rev. 140, 93-100 (2019).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Dev Dyn. 203 (3), 253-310 (1995).
- Yu, K., Xing, J., Zhang, J., Zhao, R., Zhang, Y., Zhao, L. Effect of multiple cycles of freeze-thawing on the RNA quality of lung cancer tissues. Cell Tissue Bank. 18 (3), 433-440 (2017).
- Yang, S., et al. Decontamination of ambient RNA in single-cell RNA-seq with DecontX. Genome Biol. 21 (1), 57 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten