Method Article
Generazione di organoidi apicali intestinali e valutazione del tasso di proliferazione degli organoidi mediante marcatura EdU
In questo articolo
Riepilogo
Qui, descriviamo un protocollo per la generazione di organoidi intestinali apicali da colture di organoidi standard incorporate in matrice. Delinea inoltre la successiva incorporazione di EdU in cellule che proliferano attivamente e la quantificazione semiautomatica di cellule EdU-positive.
Abstract
Qui, descriviamo la generazione di colture galleggianti di organoidi intestinali apicali da colture di organoidi intestinali inclusi in idrogel. Contemporaneamente, vengono stabilite colture di organoidi basali galleggianti per il confronto diretto tra organoidi apical-out e basal-out. Gli organoidi apicali e basali vengono successivamente sottoposti all'analogo della timidina 5-etinil-2'-desossiuridina (EdU), che viene integrato nel DNA di nuova sintesi durante la fase S della divisione cellulare. Questa incorporazione nel DNA può essere visualizzata in organoidi morfologicamente intatti utilizzando la microscopia confocale a scansione laser. Le cellule marcate con Hoechst33342 ed EdU vengono quindi quantificate in modo semiautomatico utilizzando un software di analisi delle immagini. Il calcolo della percentuale di cellule EdU-positive sul numero totale di cellule consente l'analisi della proliferazione cellulare in organoidi tridimensionali (3D). Nonostante sia utilizzato qui per l'analisi della proliferazione negli organoidi intestinali, il protocollo è applicabile anche all'analisi di colorazioni nucleo-specifiche di vario tipo in altri organoidi o colture cellulari bidimensionali.
Introduzione
Gli organoidi intestinali sono modelli tridimensionali in vitro che ricapitolano l'epitelio intestinale comprendente diversi tipi di cellule. Questi organoidi possono essere facilmente stabiliti da cellule staminali adulte isolate da cripte intestinali1. Poiché gli organoidi sono molto più vicini all'epitelio in vivo, stanno diventando sempre più importanti nella ricerca biomedica. Gli organoidi dell'intestino non sono utilizzati solo per l'analisi dei meccanismi fisiologici (ad esempio, la segnalazione della nicchia intestinale 2,3 e la differenziazione cellulare 4,5) ma anche per la ricerca sulle malattie infettive 6,7. Tuttavia, la crescita di cellule polarizzate in 3D che racchiudono un lume centrale è impegnativa, poiché la superficie cellulare apicale finisce per essere inaccessibile all'interno del lume dell'organoide. L'esame delle differenze tra le superfici cellulari apicali e basolaterali può essere importante negli studi metabolici, come esemplificato dalle differenze nell'assorbimento degli acidi grassi8 e dalla ricerca sulle malattie infettive 9,10,11,12.
La generazione dei cosiddetti organoidi apical-out è un'opzione facile per superare questo problema. Rimuovendo la matrice extracellulare da colture di organoidi standard (cioè organoidi basali, incorporati nella matrice) e seminando questi organoidi in un mezzo privo di matrice, è possibile indurre un interruttore di polarità9.
Come abbiamo precedentemente pubblicato, la maggior parte degli organoidi inverte la propria polarità entro 12 ore. Tuttavia, occorrono 48-72 ore per ottenere una coltura con più del 90% di organoidi apicali. Nonostante il loro vantaggio di consentire l'accesso alla superficie cellulare apicale, gli organoidi apicali mostrano una proliferazione significativamente ridotta dopo l'inversione di polarità, mentre i tassi di morte cellulare aumentano13. Il fattore dell'attività proliferativa può rappresentare una variabile confondente in varie analisi e dovrebbe essere tenuto a mente quando si progetta un esperimento.
Qui, presentiamo un protocollo dettagliato per la creazione di colture di organoidi apicali intestinali e di organoidi di controllo basali galleggianti per le analisi a valle. Inoltre, descriviamo la marcatura con 5-etinil-2'-deossiuridina (EdU), incorporata nel DNA di nuova sintesi e quindi marcante le cellule che proliferano attivamente. Descriviamo ulteriormente l'analisi semiautomatica del tasso di proliferazione degli organoidi utilizzando il software arivis Pro (Zeiss) mediante quantificazione di cellule EdU+ . Uno schema del processo è delineato nella Figura 1.
Protocollo
Sono stati utilizzati organoidi intestinali adulti derivati da cellule staminali di cani, stabiliti secondo Kramer et al., 202014 . Sulla base delle linee guida del comitato etico istituzionale, l'uso del materiale tissutale raccolto durante l'escissione terapeutica o post mortem è incluso nel "consenso del proprietario per il trattamento" dell'università, che è stato firmato da tutti i proprietari dei pazienti.
1. Coltura di organoidi
- Coltivare organoidi intestinali derivati da cellule staminali adulte incorporati nella matrice di estratto della membrana basale (BME) di scelta in piastre a 24 pozzetti e dividerli meccanicamente utilizzando pipette di vetro, come descritto in dettaglio in Pleguezuelos et al., 202015.
NOTA: Per il protocollo qui descritto, Geltrex è stato utilizzato come BME. - Rimuovere con cautela il terreno raffinato (Tabella 1) dagli organoidi incorporati nel BME.
- Aggiungere 500 μl di tripsina-EDTA allo 0,05% per pozzetto e staccare tutte le cupole della matrice dal pozzetto pipettando ripetutamente su e giù. Trasferire gli organoidi in una provetta da 15 ml e risospendere bene per dissociare completamente la matrice di idrogel.
- Incubare gli organoidi con tripsina-EDTA allo 0,05 % a 37 °C in un bagno d'acqua fino a quando tutti gli organoidi sono dissociati in singole cellule o piccoli gruppi di cellule.
- Diluire la sospensione tripsina/cellulare con terreno basale (Tabella 1). Utilizzare almeno il doppio del terreno basale rispetto alla tripsina.
- Centrifugare le celle a 8 °C a 420 × g per 5 min. Rimuovere quanto più surnatante possibile.
- Cellule di semi incorporate nel BME in tanti pozzetti quanti sono stati utilizzati per la tripsinizzazione nel passaggio 1.3. e fornire loro un terreno raffinato (Tabella 1).
- Incubare le cellule per 3 giorni in un incubatore per colture cellulari e procedere con il passaggio 2.1.
2. Induzione dell'inversione di polarità e marcatura EdU
- Rimuovere con cautela il terreno dagli organoidi incorporati nel BME.
- Aggiungere 500 μl di soluzione per la raccolta di organoidi a ciascun pozzetto e staccare tutte le cupole della matrice dal pozzetto pipettando ripetutamente su e giù. Trasferire gli organoidi in una provetta da 15 ml e risospendere bene per dissociare completamente la matrice di idrogel.
- Incubare la provetta da 15 mL con ghiaccio per 1,5 ore. Agitare bene la provetta ogni 10 minuti per evitare la formazione di grumi degli organoidi e garantire una dissociazione uniforme dei restanti componenti dell'idrogel.
- Durante l'incubazione, rivestire una piastra a 96 pozzetti con una soluzione antiaderente. Per ogni pozzetto di una piastra a 24 pozzetti tripsinizzata al punto 1.3., rivestire 8 pozzetti di una piastra a 96 pozzetti. Incubare per almeno 1 ora a temperatura ambiente (RT).
- Aggiungere almeno il doppio della quantità di PBS utilizzata per la soluzione di raccolta degli organoidi nella provetta da 15 mL e centrifugare a 8 °C a 150 × g per 5 minuti.
- Rimuovere il surnatante e risospendere gli organoidi in 1 mL di PBS.
- Trasferire 500 μl di sospensione organoide/PBS in una provetta separata da 15 mL e centrifugare nuovamente entrambe le provette a 8 °C a 150 × g per 5 minuti.
- Rimuovere tutta la soluzione antiaderente dalla piastra a 96 pozzetti.
- Rimuovere quanto più surnatante possibile dopo la centrifugazione.
- Utilizzare una delle provette per la generazione di organoidi basali galleggianti (BO) e l'altra provetta per gli organoidi apical-out (AO).
- Per gli organoidi BO, aggiungere 100 μl di terreno raffinato contenente il 7,5% di BME per pozzetto della piastra a 96 pozzetti in una provetta. Mescolare bene e disperdere gli organoidi in modo uniforme sul numero desiderato di pozzetti della piastra a 96 pozzetti pre-rivestita.
- Per gli organoidi AO, aggiungere 100 μl di terreno semplice raffinato (senza l'aggiunta di BME) per pozzetto della piastra a 96 pozzetti all'altra provetta. Mescolare bene e disperdere gli organoidi in modo uniforme sul numero desiderato di pozzetti della piastra a 96 pozzetti pre-rivestita.
- Incubare gli organoidi a 37 °C e 5% di CO2 per 3 giorni.
NOTA: Le differenze morfologiche inizieranno ad essere visibili dopo il giorno 1. Tuttavia, i risultati pubblicati in precedenza mostrano che la stragrande maggioranza degli organoidi impiega circa 3 giorni per presentare la polarità apicale13. - In punti temporali definiti, ad esempio 24 ore/48 ore/72 ore dopo l'induzione dell'inversione di polarità, aggiungere 50 μL di 3 μM di EdU diluito in terreno raffinato a tutti i pozzetti degli organoidi da marcare, per ricevere una concentrazione finale di 1 μM di EdU in ciascun pozzetto.
- Incubare a 37 °C e 5% di CO2 per 1,5 h.
- Raccogliere gli organoidi marcati con EdU utilizzando puntali a foro largo e trasferirli in una provetta da 15 ml.
- Aggiungere una quantità uguale di PFA al 4% in ogni provetta (organoidi BO e AO; concentrazione finale di PFA = 2%) e mescolare accuratamente.
- Incubare a RT per 15 min.
- Aggiungere almeno il doppio della quantità di PBS in base al volume della provetta da 15 mL e centrifugare a 8 °C a 80 × g per 5 minuti.
- Rimuovere il surnatante e risospendere gli organoidi in 1 mL di PBS.
- Centrifugare nuovamente a 8 °C a 80 × g per 5 min.
- Rimuovere il surnatante, risospendere gli organoidi in 1 mL di PBS e trasferirli in una provetta da 1,5 mL. Conservare gli organoidi a 4 °C fino all'esecuzione della reazione di colorazione EdU (vedere paragrafo 3).
- Ripetere i passaggi 2.14-2.22 per ogni punto temporale da analizzare.
3. Reazione di colorazione EdU Click-it e colorazione Hoechst 33342
- Centrifugare gli organoidi fissati (dal punto 2.22) in una provetta da 1,5 mL a 4 °C a 50 × g per 5 min.
- Rimuovere il surnatante e risospendere gli organoidi in 1 mL di albumina sierica bovina (BSA) al 3% diluita in PBS utilizzando punte a foro largo.
- Ripetere i passaggi 3.1. e 3.2 (fasi di lavaggio).
- Rimuovere il surnatante e risospendere gli organoidi in 1 mL di Triton X-100 allo 0,5% diluito in PBS.
- Incubare a RT per 20 min.
- Centrifugare gli organoidi a RT a 50 × g per 5 min.
- In una provetta nuova, preparare 1x additivo tampone mescolando 45 μl di H2O con 5 μl di additivo tampone 10x.
- In un'altra provetta, preparare il cocktail di reazione mescolando i seguenti componenti: 385,8 μl di H2O, 43 μl di tampone di reazione 10x, 20 μl di CuSO, 4,1,2 μl di azoturo Alexa Fluor 647 e 50 μl o 1x di additivo tampone (dal passaggio 3.7.).
NOTA: Questa miscela è sufficiente per cinque reazioni e può essere scalata su e giù in base al numero richiesto di reazioni di colorazione. - Ripetere i passaggi 3.1. e 3.2. due volte (fasi di lavaggio).
- Rimuovere il surnatante e aggiungere 100 μL di Cocktail di Reazione (passaggio 3.8.) in ogni provetta con organoidi.
- Incubare a RT per 30 minuti al buio.
- Ripetere i passaggi 3.1. e 3.2 (fasi di lavaggio).
- Centrifugare gli organoidi a 50 × g per 5 minuti a RT.
- Risospendere organoidi in 1 mL da 10 μg/mL Hoechst33342 diluito in PBS.
- Incubare a RT per 45 minuti al buio.
- Ripetere i passaggi 3.1. e 3.2 (fasi di lavaggio).
- Centrifugare gli organoidi a 50 × g per 5 min a 4 °C.
- Organoidi di semi nella matrice BME di scelta in un vetrino compatibile con la microscopia confocale (ad es. μ-Slide 18 pozzetti)
- Immagine di organoidi al microscopio confocale.
4. Analisi semiautomatica delle immagini
NOTA: Per questa analisi è stata utilizzata la versione 4.2.2 di arivis Pro (software di analisi delle immagini).
- Importare le immagini del microscopio confocale nel software di analisi delle immagini e definire una cartella in cui verrà salvato il file di analisi. Utilizzare l'opzione Immagini come punti temporali per importare più immagini (immagini replicate, punti temporali diversi, trattamenti diversi, ecc.). Ogni immagine verrà quindi trattata come un singolo punto temporale, consentendo un facile passaggio da un'immagine all'altra.
- Aprite il pannello di analisi e importate la pipeline di analisi fornita come File supplementare 1 (arivis_EdU_pipeline).
- Usa lo strumento Posiziona Nuovi Oggetti e Sfera con il tag Organoide per circondare facilmente tutti gli organoidi ben separati.
- Passa all'immagine successiva, cioè al punto temporale successivo, e procedi alla marcatura degli organoidi per tutte le immagini.
- Per gli organoidi che sono molto vicini tra loro e quindi non possono essere separati utilizzando la modalità Sfera , apri l'elenco degli oggetti e attiva il tag Organoide.
- Continuate a utilizzare lo strumento Disegna oggetti in modalità Poligono per marcare a mano i contorni degli organoidi.
NOTA: Nell'elenco degli oggetti, tutti gli organoidi sono ora contrassegnati e assegnati a un punto temporale specifico. Questo punto temporale corrisponde all'ordine delle immagini importate nel passaggio 4.1. Questi punti temporali possono ora essere rinominati in modo che corrispondano al nome dell'immagine originale, se lo si preferisce. - Nell'elenco degli oggetti, contrassegnare tutti gli oggetti (=organoidi) e fare clic con il pulsante destro del mouse, quindi fare clic su Rimuovi tag per rimuovere il tag Manuale da tutti gli oggetti.
- Disattiva il tag Organoide nell'elenco degli oggetti e contrassegna tutte le aree all'interno degli oggetti precedentemente definiti che devono essere escluse dall'analisi utilizzando lo strumento Disegna Oggetti in modalità Poligono . Queste aree possono includere cellule morte all'interno del lume dell'organoide o aree sfocate, che potrebbero distorcere l'analisi.
- Quando apri l'elenco degli oggetti, assicurati che tutti gli organoidi siano elencati con il tag Organoid e che tutte le aree che devono essere escluse abbiano il tag Manual.
- Avvia la pipeline di analisi facendo clic sulla freccia nel pannello in alto a sinistra.
NOTA: Il software ora segmenta tutti i nuclei nei canali Hoechst33342 e EdU. Questa pipeline di analisi considera solo i nuclei segmentati di Hoechst33342+ ed EdU+ di dimensioni superiori a 15 μm2 (cioè l'area nel segmento del nucleo) per garantire che i piccoli nuclei, molto probabilmente provenienti da cellule morte, siano esclusi dall'analisi. - Trovare i risultati dell'analisi nell'elenco degli oggetti facendo clic sul tag Colonne funzionalità .
- Passa alla vista Dettagli master e seleziona il tag Organoidi nel pannello superiore e la funzione Primo punto temporale.
- Nel pannello inferiore, selezionare le funzioni da visualizzare per organoide. Usa l'area di proiezione (x/y/z) (voxel), l'intensità media # 1 (canale EdU) e le intensità SD #1.
- Esporta i risultati (Master Detail Report) utilizzando la funzione di esportazione Excel .
- Salvare il file di analisi e chiudere il software.
5. Determinazione dei tassi di proliferazione degli organoidi
- Aprire il file del report dettagli master esportato nel passaggio 4.14.
- C'è una scheda separata per ogni immagine analizzata. Calcola la somma area/voxel per i nuclei e la colorazione EdU per ogni immagine. Quindi, copia tutti i dati risultanti in una nuova scheda collettiva e raggruppa tutti i dati che si riferiscono allo stesso punto temporale.
- Quindi, calcola l'area totale dei nuclei cellulari (cioè la somma dell'area di Hoechst33342+ e dell'area EdU+ ) per ogni immagine.
- Successivamente, dividere l'area totale per se stessa (= 100%) e l'area media di EdU+ per l'area totale (=percentuale di DNA EdU+ ).
- Tracciare la percentuale di DNA proliferativo degli organoidi BO e AO sull'asse y e i diversi punti temporali sull'asse x.
Risultati
Gli organoidi coltivati per 3 giorni dopo la tripsinizzazione dovrebbero essere idealmente compresi tra 50 e 250 μm, come illustrato nella Figura 2A. Gli organoidi che sono considerevolmente più grandi di questo potrebbero non invertire la loro polarità in modo efficiente. Anche gli organoidi più grandi possono iniziare a germogliare e abbiamo notato che questi organoidi possono avere problemi anche con un'efficiente inversione di polarità. Gli organoidi basal-out e apical-out presentano evidenti differenze morfologiche già nell'imaging in campo chiaro. Mentre gli organoidi basali mantengono il loro ampio lume (Figura 2B) dopo 3 giorni di coltura in sospensione, gli organoidi apicali (Figura 2C) sembrano più compatti. Una caratteristica molto specifica delle colture in sospensione apical-out è il numero di cellule morte che galleggiano intorno agli organoidi. Ciò è dovuto agli organoidi apicali che esculdono cellule morte e differenziate terminali nel mezzo circostante, mentre gli organoidi basali accumulano cellule morte nel lume dell'organoide.
Gli organoidi intestinali che hanno incorporato l'EdU nel loro DNA appena sintetizzato prima della divisione cellulare possono essere visualizzati utilizzando un microscopio confocale. Gli organoidi basal-out mostrano segnali EdU molto più elevati nel tempo rispetto alle loro controparti apical-out (Figura 3). È importante notare che non tutti gli organoidi mostrano cellule EdU+ nonostante crescano di dimensioni. Ciò è dovuto all'assenza di cellule EdU+ nello specifico strato di imaging.
Queste immagini confocali possono essere analizzate quantitativamente utilizzando il software di analisi delle immagini e la pipeline di analisi fornita (File supplementare 1). Durante l'esecuzione del protocollo di analisi, gli organoidi devono essere prima circondati (Figura 4A) prima di escludere alcune aree dall'analisi vera e propria (Figura 4B). Queste aree di esclusione possono riferirsi a cellule morte all'interno del lume dell'organoide, che non sono di interesse per la quantificazione dei segnali EdU. La successiva esecuzione della pipeline di analisi automatica porta alla segmentazione di tutti i nuclei rilevati, differenziando tra nuclei Hoechst33342+ e EdU+ . Inoltre, tutti i nuclei al di sotto di un cut-off di 15 μm2 sono esclusi in quanto molto probabilmente si tratta di cellule morte o nuclei che non vengono mostrati per intero in questo strato di immagine (Figura 4C).
Dopo l'analisi delle immagini utilizzando il software ed esportando le misure di quantificazione, questi dati vengono ulteriormente calcolati per analizzare la percentuale del segnale EdU+ rispetto alla massa totale del DNA (Figura 5). Nel caso presentato all'interno di questo manoscritto, gli organoidi intestinali apicali mostrano livelli di proliferazione drasticamente ridotti rispetto agli organoidi basali galleggianti.

Figura 1: Panoramica schematica del protocollo descritto. Lo schema include la coltivazione di organoidi, l'induzione dell'inversione di polarità, la marcatura EdU e l'analisi semiautomatica delle immagini. Figura creata con Biorender.com. Clicca qui per visualizzare una versione più grande di questa figura.
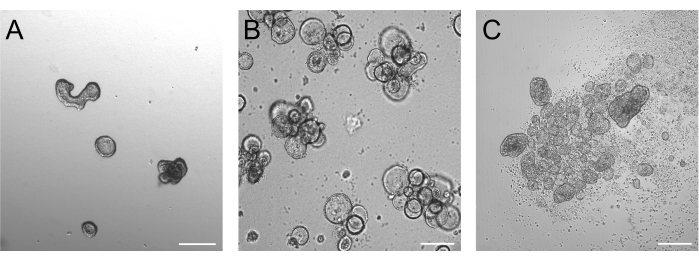
Figura 2: Organoidi prima e dopo l'induzione dell'inversione di polarità. (A) Organoidi intestinali incorporati nella matrice tre giorni dopo che le cellule tripsinizzate sono state seminate in un mezzo di crescita. (B) Organoidi basali galleggianti tre giorni dopo la semina in coltura in sospensione. (C) Organoidi apicali 3 giorni dopo la semina in coltura in sospensione senza BME per l'induzione dell'inversione di polarità. Barra della scala = 200 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
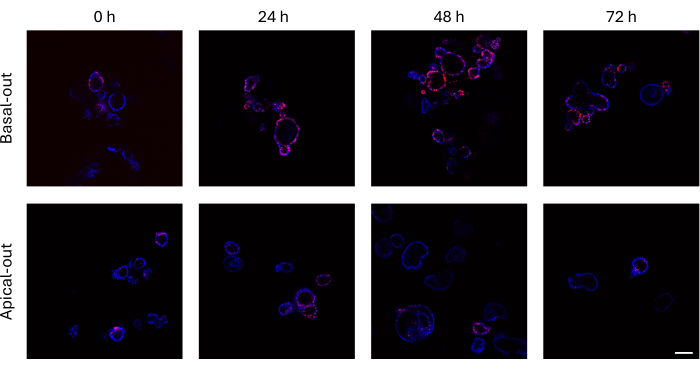
Figura 3: Organoidi intestinali basal-out e apical-out marcati con EdU. Il pannello superiore che mostra le immagini confocali degli organoidi basali nel tempo presenta molte più cellule EdU+ rispetto agli organoidi apicali (pannello inferiore) negli stessi punti temporali. Barra della scala = 100 μm. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Analisi quantitativa dell'immagine di organoidi intestinali colorati con EdU. (A) Organoidi circondati utilizzando le modalità Sfera e Poligono . (B) L'area da escludere dall'analisi è contrassegnata in bianco (indicata dalla freccia). Quest'area segna il lume dell'organoide, che contiene un numero di cellule morte, che saranno escluse dall'analisi successiva. (C) L'immagine dopo l'analisi mostra tutti i nuclei segmentati. I nuclei Hoechst33342 + sono contrassegnati in ciano, i nuclei EdU+ sono contrassegnati in giallo e i nuclei che non raggiungono i 15 μm2 sono contrassegnati in verde (Hoechst33342) e arancione (EdU). Barra di scala = 50 μm. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 5: Tasso di proliferazione degli organoidi. La percentuale di DNA EdU+ sul DNA totale che funge da proxy per il tasso di proliferazione degli organoidi che mostra una proliferazione più elevata negli organoidi basali rispetto agli organoidi apicali. I dati sono presentati come SEM medio. Clicca qui per visualizzare una versione più grande di questa figura.
| Medio basale | |
| Componenti multimediali | Concentrazione finale |
| DMEM-F12 | |
| Integratore 100X di penicillina-streptomicina per Media | 1x |
| Integratore GlutaMAX | 2 mM |
| Soluzione tampone HEPES 1 M, liquido | 10 mM |
| Medium raffinato | |
| B27 Integratore Senza Siero (50x), liquido | 1x |
| N-acetil-L-cisteina, testata in coltura cellulare | 1 mM |
| [Leu15]-gastrina I umana | 10 nM |
| A 83-01 | 500 nM |
| HGF umano | 50 ng/mL |
| NOGGIN Umano (Mammifero) | 100 ng/mL |
| IGF-I umano | 100 ng/mL |
| FGF-basic umano | 50 ng/mL |
| Mezzo condizionato con R-spondina | 10 % (v/v) |
| Wnt-3a fluido condizionato | 50 % (v/v) |
| Medio basale | volume residuo per la diluizione dei fattori di crescita sopra menzionati/terreno condizionato |
Tabella 1: Composizione dei media basale e raffinata.
Discussione
Questo protocollo descrive in dettaglio come indurre l'inversione di polarità in colture standard di organoidi intestinali derivati da cellule staminali adulte. Gli organoidi apicali hanno lo scopo di accedere alla superficie cellulare apicale, che di solito è orientata verso il lume dell'organoide. Essere in grado di sondare la superficie apicale può essere di grande importanza per alcune applicazioni, in quanto questa è la porzione della membrana cellulare che è esposta a tutto il contenuto del tratto digestivo in condizioni fisiologiche in vivo. Altri metodi per sfidare la superficie apicale in modo specifico sono la microiniezione16, l'uso di monostrati derivati da organoidi17 e la frammentazione degli organoidi18. Tuttavia, ciascuno di questi metodi presenta svantaggi e svantaggi specifici, come noi e altri abbiamo esaminato più dettagliatamente in precedenza19,20.
Il metodo di generazione di organoidi apical-out presenta diversi vantaggi: 1) La generazione di organoidi apical-out è relativamente semplice rispetto ad altri metodi e non richiede alcuna strumentazione o attrezzatura specializzata. Pertanto, gli organoidi apicali sono un modo economico per accedere alla superficie cellulare apicale; 2) gli organoidi basali galleggianti possono essere coltivati in parallelo e utilizzati come organoidi di controllo significativi, in particolare per analizzare gli effetti specifici della polarità; 3) gli organoidi BO e AO galleggianti possono essere sottoposti ad applicazioni standard a valle, come esemplificato dall'incorporazione di EdU all'interno di questo rapporto, ma anche dalla colorazione immuno(isto)chimica, nonché da saggi basati sulla luminescenza e sulla fluorescenza, come precedentemente riportato13 e dall'imaging diretto di cellule vive12. Le analisi a valle, come l'analisi dell'incorporazione di EdU o le colorazioni immuno(isto)chimiche, consentono anche una certa flessibilità in termini di tempo, poiché gli organoidi possono essere conservati a 4 °C fino a un nuovo utilizzo.
Nonostante la facilità di inversione di polarità, esistono alcuni punti critici nel nostro protocollo che dovrebbero essere considerati quando si applica questo metodo. Innanzitutto, gli organoidi utilizzati nel passaggio 2.1. dovrebbe essere di una certa dimensione. Nella nostra esperienza, l'uso di organoidi 3 giorni dopo la tripsinizzazione funziona bene. Tuttavia, questo tempo può variare con gli organoidi di altri animali a causa delle differenze nella proliferazione cellulare o a seconda della densità di semina dei cluster di singole cellule. Inoltre, la dissociazione del BME utilizzando la soluzione di raccolta degli organoidi è incrementale. Come descritto da Co et al., 20199, concentrazioni di BME fino al 2,5% potrebbero essere sufficienti per mantenere gli organoidi in una morfologia basale in coltura in sospensione. Pertanto, è necessario rimuovere la maggior quantità possibile di BME per garantire un'efficiente inversione di polarità. Un altro punto importante è l'uso corretto di piastre di coltura tissutale non trattate per ridurre al minimo l'adesione degli organoidi durante la coltura in sospensione. In base alla nostra esperienza, il prerivestimento delle piastre con soluzione antiaderente (fase 2.4) consente l'uso di praticamente qualsiasi piastra per la coltura di organoidi apicali. Tuttavia, possono essere esplorate altre opzioni che rendono superflue le lastre di pre-rivestimento.
L'importanza di valutare gli effetti sulla superficie cellulare basolaterale e apicale delle cellule epiteliali polarizzate diventa evidente in molti casi. Ad esempio, abbiamo precedentemente analizzato gli effetti delle tossine del batterio anaerobio Clostridioides difficile sugli organoidi BO e AO. I risultati di questo studio mostrano che la tossina B di C. difficile (TcdB) danneggia l'integrità della barriera epiteliale intestinale solo negli organoidi BO dell'intestino tenue, ma non negli organoidi AO. Diversi altri rapporti che mostrano reazioni dominio-specifiche di vari microrganismi patogeni evidenziano l'importanza di valutare entrambi i lati delle cellule epiteliali 9,16,21,22,23,24,25.
Uno dei principali svantaggi degli organoidi apicali è il loro comportamento alterato per quanto riguarda la proliferazione cellulare. Come descritto in precedenza, gli organoidi AO mostrano tassi di proliferazione cellulare drasticamente ridotti rispetto alle loro controparti BO. Abbiamo precedentemente riportato che questa mancanza di proliferazione va di pari passo con un leggero aumento dei livelli di morte cellulare13. In definitiva, ciò non consente una coltura a lungo termine di organoidi AO a meno che non si trovi un modo per prolungare la durata degli organoidi AO. Tuttavia, riteniamo che gli organoidi AO siano uno strumento molto utile per accedere facilmente alla superficie cellulare apicale, a condizione che i ricercatori prestino attenzione alle conclusioni che potrebbero essere influenzate dalla diminuzione della proliferazione cellulare.
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Questa ricerca è stata supportata utilizzando le risorse della VetImaging Core Facility (VetCore, Vetmeduni, Austria). Ringraziamo Ursula Reichart per il suo supporto nell'analisi semiquantitativa delle immagini. GC ha ricevuto una borsa di studio DOC (numero di sovvenzione 26349) dell'Accademia Austriaca delle Scienze (ÖAW) presso la Divisione di Medicina Interna dei Piccoli Animali di Vetmeduni.
Materiali
| Name | Company | Catalog Number | Comments |
| 24 well plates | Biologix | 07-6024 | |
| [Leu15]-Gastrin I human, ≥95% (HPLC) | Sigma-Aldrich | G-9145 | |
| µ-Slide 18 Well Glass Bottom | ibidi | 81817 | |
| 100X Penicillin-Streptomycin supplement for Media | Gibco/Thermo | 15140122 | |
| A 83-01 | Tocris Bioscience | 2939/10 | |
| Anti-Adherence Rinsing Solution | StemCell Technologies | 7010 | |
| arivis Pro | Zeiss | Version 4.2.2 | |
| B27 SerumFree Supplement (50X), liquid | Gibco/Thermo | 17504044 | |
| Bisbenzimide H 33342 (Hoechst 33342) | Abcam | ab145597 | |
| Bovine Serum Albumin | Sigma-Aldrich | A7906-100G | |
| Click-iT EdU Cell Proliferation Kit for Imaging, Alexa Fluor 647 dye | Thermo Scientific | C10340 | |
| DMEM-F12 | Gibco/Thermo | 11320033 | |
| Geltrex | Gibco/Thermo | A1413202 | |
| GlutaMAX Supplement | Gibco/Thermo | 35050061 | |
| HEPES Buffer Solution 1 M, liquid | Gibco/Thermo | 15630056 | |
| Human FGF-basic | PeproTech | 100-18B-100µG | |
| Human HGF | PeproTech | 100-39-100µG | |
| Human IGF-I | PeproTech | 100-11-100µG | |
| Human NOGGIN (Mammalian) | PeproTech | 120-10C-200µG | |
| N-Acetyl-L-cysteine,cell culture tested, BioReagent | Sigma-Aldrich | A9165-5G | |
| Organoid Harvesting Solution | Thermo | C10340 | |
| Triton(TM) X-100,for molecular biology | Sigma-Aldrich | T8787-250ML | |
| Trypsin-EDTA (0.05%), phenol red | Gibco/Thermo | 25300054 |
Riferimenti
- Sato, T., et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature. 459, 262-265 (2009).
- Li, Y., et al. BMP suppresses Wnt signaling via the Bcl11b-regulated NuRD complex to maintain intestinal stem cells. EMBO J. 43 (23), 6032-6051 (2024).
- Gaowa, A., Leangpanich, S., Park, E. J., Kawamoto, E., Shimaoka, M. Irisin promotes intestinal epithelial cell proliferation via Wnt/β-catenin and focal adhesion kinase signaling pathways. Sci Rep. 14, 25702 (2024).
- Basak, O., et al. Induced quiescence of Lgr5+ stem cells in intestinal organoids enables differentiation of hormone-producing enteroendocrine cells. Cell Stem Cell. 20 (2), 177-190 (2017).
- Ludikhuize, M. C., et al. Mitochondria define intestinal stem cell differentiation downstream of a FOXO/Notch axis. Cell Metab. 32 (5), 889-900.e7 (2020).
- Ettayebi, K., et al. Replication of human noroviruses in stem cell-derived human enteroids. Science (1979). 353 (6306), 1387-1393 (2016).
- Zhou, J., et al. Infection of bat and human intestinal organoids by SARS-CoV-2. Nat Med. 26 (7), 1077-1083 (2020).
- Joo, S. -. S., et al. Porcine intestinal apical-out organoid model for gut function study. Animals. 12 (3), 372 (2022).
- Co, J. Y., et al. Controlling epithelial polarity: A human enteroid model for host-pathogen interactions. Cell Rep. 26, 2509-2520.e4 (2019).
- Sasaki, N., et al. Development of a scalable coculture system for gut anaerobes and human colon epithelium. Gastroenterology. 159 (1), 388-390.e5 (2020).
- Mayorgas, A., et al. A novel strategy to study the invasive capability of adherent-invasive escherichia coli by using human primary organoid-derived epithelial monolayers. Front Immunol. 12, 646906 (2021).
- Csukovich, G., et al. Neutralising effects of different antibodies on clostridioides difficile toxins TcdA and TcdB in a translational approach. Int J Mol Sci. 24 (4), 3867 (2023).
- Csukovich, G., et al. Polarity reversal of canine intestinal organoids reduces proliferation and increases cell death. Cell Prolif. 57 (2), e13544 (2023).
- Kramer, N., et al. Generation of differentiating and long-living intestinal organoids reflecting the cellular diversity of canine intestine. Cells. 9 (4), 822 (2020).
- Pleguezuelos-Manzano, C., et al. Establishment and culture of human intestinal organoids derived from adult stem cells. Curr Protoc Immunol. 130 (1), e106 (2020).
- Dutta, D., Heo, I., O'Connor, R. Studying infection in 3D tissue-derived human organoid culture systems by microinjection. J Vis Exp. (151), e59610 (2019).
- Kumar, A., et al. Decreased SLC26A3 expression and function in intestinal epithelial cells in response to Cryptosporidium parvum infection. Am J Physiol Cell Physiol. 317 (6), C1205-C1212 (2019).
- Dutta, D., Clevers, H. Organoid culture systems to study host-pathogen interactions. Curr Opin Immunol. 48, 15-22 (2017).
- Csukovich, G., Pratscher, B., Burgener, I. A. The world of organoids: Gastrointestinal disease modelling in the age of 3R and one health with specific relevance to dogs and cats. Animals. 12 (18), 2461 (2022).
- Han, X., et al. Creating a more perfect union: Modeling intestinal bacteria-epithelial interactions using organoids. Cell Mol Gastroenterol Hepatol. 12 (2), 769-782 (2021).
- Li, Y., et al. Next-generation porcine intestinal organoids: an apical-out organoid model for swine enteric virus infection and immune response investigations. J Virol. 94 (21), e01006-e01020 (2020).
- Smith, D., et al. The development of ovine gastric and intestinal organoids for studying ruminant host-pathogen interactions. Front Cell Infect Microbiol. 11, 733811 (2021).
- Nash, T. J., Morris, K. M., Mabbott, N. A., Vervelde, L. Inside-out chicken enteroids with leukocyte component as a model to study host-pathogen interactions. Commun Biol. 4 (1), 377 (2021).
- Hanson, K. I., Weiss, A. A. Intestinal tissue response to Shiga toxin exposure. mBio. 15 (9), e0123224 (2024).
- Hovhannisyan, P., et al. Infection of human organoids supports an intestinal niche for Chlamydia trachomatis. PLoS Pathog. 20 (8), e1012144 (2024).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon