Method Article
Generación de organoides intestinales apicales y evaluación de la tasa de proliferación de organoides mediante el marcaje EdU
En este artículo
Resumen
Aquí, describimos un protocolo para generar organoides intestinales apicales a partir de cultivos de organoides estándar incrustados en matriz. También describe la posterior incorporación de EdU en células en proliferación activa y la cuantificación semiautomática de células EdU positivas.
Resumen
Aquí, describimos la generación de cultivos flotantes de organoides intestinales apicales a partir de cultivos de organoides intestinales incrustados en hidrogel. Al mismo tiempo, se establecen cultivos flotantes de organoides basales para la comparación directa entre organoides apicales y basales. Los organoides apicales y basales se someten posteriormente al análogo de timidina 5-etinil-2'-desoxiuridina (EdU), que se integra en el ADN recién sintetizado durante la fase S de la división celular. Esta incorporación al ADN se puede visualizar en organoides morfológicamente intactos utilizando microscopía confocal de barrido láser. A continuación, las células marcadas con Hoechst33342 y EdU se cuantifican de forma semiautomática utilizando un software de análisis de imágenes. El cálculo del porcentaje de células EdU positivas del número total de células permite el análisis de la proliferación celular en organoides tridimensionales (3D). A pesar de que se utiliza aquí para el análisis de la proliferación en organoides intestinales, el protocolo es aplicable al análisis de tinciones específicas de núcleos de varios tipos en otros organoides o también en cultivos celulares bidimensionales.
Introducción
Los organoides intestinales son modelos tridimensionales in vitro que recapitulan el epitelio intestinal compuesto por diferentes tipos de células. Estos organoides pueden establecerse fácilmente a partir de células madre adultas aisladas de criptas intestinales1. Dado que los organoides están mucho más cerca del epitelio in vivo, son cada vez más importantes en la investigación biomédica. Los organoides del intestino no solo se utilizan para el análisis de mecanismos fisiológicos (por ejemplo, la señalización del nicho intestinal 2,3 y la diferenciación celular 4,5), sino también para la investigación de enfermedades infecciosas 6,7. Sin embargo, el crecimiento de células polarizadas en 3D que encierran un lumen central es un desafío, ya que la superficie de la célula apical termina siendo inaccesible dentro del lumen del organoide. El examen de las diferencias entre las superficies celulares apicales y basolaterales puede ser importante en los estudios metabólicos, como lo demuestran las diferencias en la absorción de ácidos grasos8 y la investigación de enfermedades infecciosas 9,10,11,12.
La generación de los llamados organoides apicales es una opción fácil para superar este problema. Al eliminar la matriz extracelular de los cultivos de organoides estándar (es decir, organoides basales e incrustados en la matriz) y sembrar estos organoides en un medio libre de matriz, se puede inducir un cambio de polaridad9.
Como hemos publicado anteriormente, la mayoría de los organoides invierten su polaridad en 12 h. Sin embargo, se necesitan 48-72 h para obtener un cultivo con más del 90% de organoides apicales. A pesar de su ventaja de permitir el acceso a la superficie de la célula apical, los organoides apicales muestran una proliferación significativamente disminuida después de la inversión de la polaridad, mientras que las tasas de muerte celular aumentan13. El factor de la actividad proliferativa puede representar una variable de confusión en varios análisis y debe tenerse en cuenta al diseñar un experimento.
Aquí, presentamos un protocolo detallado para establecer cultivos de organoides apicales intestinales y organoides de control basales flotantes para análisis posteriores. Además, describimos el marcaje con 5-etinil-2'-desoxiuridina (EdU), incorporado en el ADN recién sintetizado y, por lo tanto, marca las células en proliferación activa. Describimos además el análisis semiautomático de la tasa de proliferación de los organoides utilizando el software arivis Pro (Zeiss) mediante la cuantificación de células EdU+ . En la Figura 1 se describe un esquema del proceso.
Protocolo
Se utilizaron organoides intestinales derivados de células madre adultas de perros, establecidos según Kramer et al., 202014 . De acuerdo con las directrices del comité de ética institucional, el uso de material tisular recogido durante la escisión terapéutica o la autopsia se incluye en el "consentimiento del propietario para el tratamiento" de la universidad, que fue firmado por todos los pacientes propietarios.
1. Cultivo de organoides
- Cultivo de organoides intestinales derivados de células madre adultas incrustados en la matriz de extracto de membrana basal (BME) de elección en placas de 24 pocillos y división mecánica con pipetas de vidrio como se describe en detalle en Pleguezuelos et al., 202015.
NOTA: Para el protocolo descrito aquí, se utilizó Geltrex como BME. - Retire con cuidado el medio refinado (Tabla 1) de los organoides incluidos en BME.
- Añada 500 μl de tripsina-EDTA al 0,05 % por pocillo y separe todas las cúpulas de la matriz del pocillo pipeteando hacia arriba y hacia abajo repetidamente. Transfiera los organoides a un tubo de 15 mL y vuelva a suspender bien para disociar completamente la matriz de hidrogel.
- Incubar organoides con tripsina-EDTA al 0,05 % a 37 °C en un baño de agua hasta que todos los organoides se disocien en células individuales o pequeños grupos de células.
- Diluir la suspensión de tripsina/célula con medio basal (Tabla 1). Use al menos el doble de medio basal que tripsina.
- Centrifugar las celdas a 8 °C a 420 × g durante 5 min. Retire la mayor cantidad posible de sobrenadante.
- Células semilla incrustadas en BME en tantos pocillos como se utilizaron para la tripsinización en el paso 1.3. y suministrarles medio refinado (Tabla 1).
- Incubar las células durante 3 días en una incubadora de cultivo celular y continuar con el paso 2.1.
2. Inducción de inversión de polaridad y marcaje de EdU
- Retire con cuidado el medio de los organoides incluidos en BME.
- Agregue 500 μL de solución de recolección de organoides a cada pocillo y separe todas las cúpulas de la matriz del pocillo pipeteando hacia arriba y hacia abajo repetidamente. Transfiera los organoides a un tubo de 15 mL y vuelva a suspender bien para disociar completamente la matriz de hidrogel.
- Incubar el tubo de 15 mL en hielo durante 1,5 h. Agite bien el tubo cada 10 minutos para evitar la aglomeración de los organoides y asegurar una disociación uniforme de los componentes restantes del hidrogel.
- Durante la incubación, cubra una placa de 96 pocillos con una solución antiadherente. Para cada pocillo de una placa de 24 pocillos tripsinizada en el paso 1.3., cubra 8 pocillos de una placa de 96 pocillos. Incubar durante al menos 1 h a temperatura ambiente (RT).
- Agregue al tubo de 15 mL al menos el doble de PBS que la solución de recolección de organoides utilizada y centrifugue a 8 °C a 150 × g durante 5 min.
- Retirar el sobrenadante y resuspender los organoides en 1 mL de PBS.
- Transfiera 500 μL de la suspensión de organoide/PBS en un tubo separado de 15 mL y centrifugue ambos tubos nuevamente a 8 °C a 150 × g durante 5 min.
- Retire toda la solución antiadherente de la placa de 96 pocillos.
- Elimine la mayor cantidad posible de sobrenadante después de la centrifugación.
- Utilice uno de los tubos para la generación de organoides basales flotantes (BO) y el otro tubo para organoides apicales (AO).
- En el caso de los organoides BO, añadir 100 μL de medio refinado que contenga un 7,5% de BME por pocillo de la placa de 96 pocillos a un tubo. Mezcle bien y disperse los organoides de manera uniforme en el número deseado de pocillos de la placa de 96 pocillos prerrecubierta.
- En el caso de los organoides AO, añada 100 μL de medio refinado simple (sin ningún BME añadido) por pocillo de la placa de 96 pocillos al otro tubo. Mezcle bien y disperse los organoides de manera uniforme en el número deseado de pocillos de la placa de 96 pocillos prerrecubierta.
- Incubar los organoides a 37 °C y 5% de CO2 durante 3 días.
NOTA: Las diferencias morfológicas comenzarán a ser visibles después del día 1. Sin embargo, los resultados publicados anteriormente muestran que la gran mayoría de los organoides tardan alrededor de 3 días en presentar la polaridad apical13. - En puntos de tiempo definidos, por ejemplo, 24 h/48 h/72 h después de la inducción de la inversión de polaridad, agregue 50 μL de 3 μM de EdU diluido en medio refinado a todos los pocillos de organoides que se van a marcar, para recibir una concentración final de 1 μM de EdU en cada pocillo.
- Incubar a 37 °C y 5% de CO2 durante 1,5 h.
- Recoja los organoides marcados con EdU con puntas de diámetro ancho y transfiérelos a un tubo de 15 ml.
- Agregue una cantidad igual de 4% de PFA a cada tubo (organoides BO y AO; concentración final de PFA = 2%) y mezcle cuidadosamente.
- Incubar en RT durante 15 min.
- Agregue al menos el doble de la cantidad de PBS según el volumen en el tubo de 15 ml y centrifugue a 8 °C a 80 × g durante 5 minutos.
- Retirar el sobrenadante y resuspender los organoides en 1 mL de PBS.
- Centrifugar de nuevo a 8 °C a 80 × g durante 5 min.
- Retire el sobrenadante, vuelva a suspender los organoides en 1 mL de PBS y transfiéralos a un tubo de 1,5 mL. Almacene los organoides a 4 °C hasta que se realice la reacción de tinción de EdU (ver sección 3).
- Repita los pasos 2.14-2.22 para cada punto de tiempo que se vaya a analizar.
3. Reacción de tinción EdU click-it y tinción Hoechst 33342
- Centrifugar los organoides fijos (a partir del paso 2.22.) en un tubo de 1,5 mL a 4 °C a 50 × g durante 5 min.
- Retirar el sobrenadante y resuspender los organoides en 1 mL de albúmina sérica bovina (BSA) al 3% diluida en PBS utilizando puntas de diámetro ancho.
- Repita los pasos 3.1. y 3.2 (etapas de lavado).
- Retirar el sobrenadante y resuspender organoides en 1 mL de Triton X-100 al 0,5% diluido en PBS.
- Incubar en RT durante 20 min.
- Centrifugar los organoides a RT a 50 × g durante 5 min.
- En un tubo nuevo, prepare 1x Aditivo tampón mezclando 45 μL de H2O con 5 μL de 10x Aditivo tampón.
- En otro tubo, prepare el cóctel de reacción mezclando los siguientes componentes: 385,8 μL de H2O, 43 μL de 10x Tampón de Reacción, 20 μL de CuSO, 4,1,2 μL de azida Alexa Fluor 647 y 50 μL o 1x Aditivo tampón (del paso 3.7.).
NOTA: Esta mezcla es suficiente para cinco reacciones y se puede escalar hacia arriba y hacia abajo de acuerdo con el número requerido de reacciones de tinción. - Repita los pasos 3.1. y 3.2. dos veces (pasos de lavado).
- Retirar el sobrenadante y añadir 100 μL de Reaction Cocktail (paso 3.8.) en cada tubo con organoides.
- Incubar en RT durante 30 minutos en la oscuridad.
- Repita los pasos 3.1. y 3.2 (etapas de lavado).
- Centrifugar organoides a 50 × g durante 5 min en RT.
- Resuspender organoides en 1 mL de 10 μg/mL Hoechst33342 diluido en PBS.
- Incubar en RT durante 45 min en la oscuridad.
- Repita los pasos 3.1. y 3.2 (etapas de lavado).
- Centrifugar organoides a 50 × g durante 5 min a 4 °C.
- Siembre los organoides de la matriz BME elegida en un portaobjetos compatible con la microscopía confocal (p. ej., pocillo μ-Slide 18)
- Imagen de organoides bajo un microscopio confocal.
4. Análisis semiautomático de imágenes
NOTA: Para este análisis se utilizó arivis Pro versión 4.2.2 (software de análisis de imágenes).
- Importe las imágenes del microscopio confocal en el software de análisis de imágenes y defina una carpeta en la que se guardará el archivo de análisis. Utilice la opción Imágenes como puntos de tiempo para importar varias imágenes (replicar imágenes, diferentes puntos de tiempo, diferentes tratamientos, etc.). Cada imagen se tratará como un único punto de tiempo, lo que permite cambiar fácilmente entre las imágenes individuales.
- Abra el panel de análisis e importe la canalización de análisis proporcionada como Archivo complementario 1 (arivis_EdU_pipeline).
- Utilice la herramienta Colocar nuevos objetos y la Esfera con la etiqueta Organoide para rodear fácilmente todos los organoides bien separados.
- Cambie a la siguiente imagen, es decir, al siguiente punto de tiempo, y proceda a marcar organoides para todas las imágenes.
- Para los organoides que están muy cerca unos de otros y, por lo tanto, no se pueden separar usando el modo Esfera , abra la lista de objetos y active la etiqueta Organoide.
- Continúe utilizando la herramienta Dibujar objetos en el modo Polígono para marcar los contornos de los organoides a mano.
NOTA: En la lista de objetos, todos los organoides ahora están marcados y asignados a un punto de tiempo específico. Este punto de tiempo corresponde al orden de las imágenes importadas en el paso 4.1. Ahora se puede cambiar el nombre de estos puntos de tiempo para que coincidan con el nombre de la imagen original si se prefiere. - En la lista de objetos, marque todos los objetos (=organoides) y haga clic con el botón derecho, luego haga clic en Eliminar etiquetas para eliminar la etiqueta Manual de todos los objetos.
- Desactive la etiqueta Organoide en la lista de objetos y marque todas las áreas dentro de los objetos previamente definidos que deben excluirse del análisis utilizando la herramienta Dibujar objetos en el modo Polígono . Estas áreas pueden incluir células muertas dentro de la luz del organoide o áreas borrosas, lo que podría sesgar el análisis.
- Al abrir la lista de objetos, asegúrese de que todos los organoides estén enumerados con la etiqueta Organoide y que todas las áreas que deben excluirse tengan la etiqueta Manual.
- Inicie la canalización de análisis haciendo clic en la flecha en el panel superior izquierdo.
NOTA: El software ahora segmenta todos los núcleos en los canales Hoechst33342 y EdU. Esta canalización de análisis solo tiene en cuenta los núcleos segmentados de Hoechst33342+ y EdU+ de más de 15μm2 de tamaño (es decir, el área en el segmento del núcleo) para garantizar que los núcleos pequeños, muy probablemente procedentes de células muertas, se excluyan del análisis. - Busque los resultados del análisis en la lista de objetos haciendo clic en la etiqueta Columnas de características .
- Cambie a la vista de detalles maestra y seleccione la etiqueta Organoides en el panel superior y la función Primer punto de tiempo.
- En el panel inferior, seleccione las características que desea mostrar por organoide. Utilice el área de proyección (x/y/z) (vóxel), la intensidad media # 1 (canal EdU) y las intensidades SD # 1.
- Exporte los resultados (Informe de detalle maestro) utilizando la función de exportación de Excel .
- Guarde el archivo de análisis y cierre el software.
5. Determinación de las tasas de proliferación de organoides
- Abra el archivo de informe de detalle maestro exportado en el paso 4.14.
- Hay una pestaña separada para cada imagen analizada. Calcule la suma de área/vóxel para los núcleos y la tinción de EdU para cada imagen. A continuación, copie todos los datos resultantes en una nueva pestaña colectiva y agrupe todos los datos que se refieran al mismo punto temporal.
- A continuación, calcule el área total de los núcleos celulares (es decir, la suma del área de Hoechst33342+ y el área de EdU+ ) para cada imagen.
- Posteriormente, divida el área total por sí misma (= 100%) y el área media de EdU+ por el área total (=porcentaje de ADN de EdU+ ).
- Grafique el porcentaje de ADN proliferativo de los organoides BO y AO en el eje y y los diferentes puntos de tiempo en el eje x.
Resultados
Los organoides cultivados durante 3 días después de la tripsinización deben estar idealmente entre 50-250 μm, como se muestra en la Figura 2A. Es posible que los organoides que son considerablemente más grandes que esto no inviertan su polaridad de manera eficiente. Los organoides más grandes también pueden comenzar a brotar, y hemos notado que estos organoides también pueden tener problemas con la inversión eficiente de la polaridad. Los organoides basales y apicales presentan diferencias morfológicas obvias ya en las imágenes de campo claro. Mientras que los organoides basales retienen su gran lumen (Figura 2B) después de 3 días de cultivo en suspensión, los organoides apicales (Figura 2C) parecen más compactos. Una característica muy específica de los cultivos en suspensión apical es el número de células muertas que flotan alrededor de los organoides. Esto se debe a que los organoides apicales expulsan células terminales diferenciadas y muertas en el medio circundante, mientras que los organoides basales acumulan células muertas en la luz del organoide.
Los organoides intestinales que han incorporado EdU a su ADN recién sintetizado antes de la división celular se pueden obtener mediante un microscopio confocal. Los organoides basal-out muestran señales de EdU mucho más altas a lo largo del tiempo que sus contrapartes apical-out (Figura 3). Es importante tener en cuenta que no todos los organoides muestran células EdU+ a pesar de crecer en tamaño. Esto se debe a que no hay células EdU+ presentes en la capa de imagen específica.
Estas imágenes confocales se pueden analizar cuantitativamente utilizando el software de análisis de imágenes y la canalización de análisis proporcionada (Archivo complementario 1). Al llevar a cabo el protocolo de análisis, primero se deben rodear los organoides (Figura 4A) antes de excluir ciertas áreas del análisis real (Figura 4B). Estas áreas de exclusión pueden referirse a células muertas dentro de la luz del organoide, que no son de interés para la cuantificación de las señales de EdU. La ejecución posterior de la canalización de análisis automático conduce a la segmentación de todos los núcleos detectados, diferenciando entre los núcleos Hoechst33342+ y EdU+ . Además, se excluyen todos los núcleos por debajo de un límite de 15 μm2, ya que lo más probable es que se trate de células muertas o núcleos que no se muestran en su totalidad en esta capa de imagen (Figura 4C).
Después del análisis de la imagen con el software y la exportación de las mediciones de cuantificación, estos datos se calculan aún más para analizar el porcentaje de señal de EdU+ de la masa total de ADN (Figura 5). En el caso que se presenta en este manuscrito, los organoides intestinales apicales muestran niveles de proliferación drásticamente reducidos en comparación con los organoides basales flotantes.

Figura 1: Resumen esquemático del protocolo descrito. El esquema incluye el cultivo de organoides, la inducción de la inversión de polaridad, el etiquetado de EdU y el análisis semiautomático de imágenes. Figura creada con Biorender.com. Haga clic aquí para ver una versión más grande de esta figura.
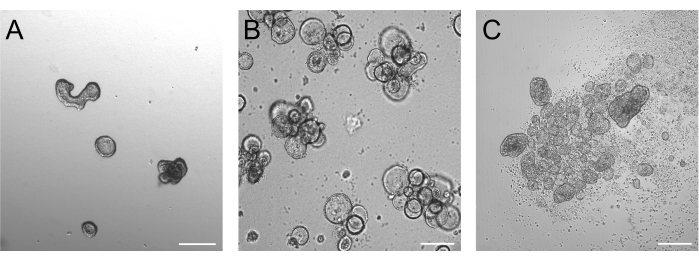
Figura 2: Organoides antes y después de la inducción de la inversión de polaridad. (A) Organoides intestinales incrustados en la matriz tres días después de que las células tripsinizadas se hayan sembrado en un medio de crecimiento. (B) Organoides basales flotantes tres días después de la siembra en cultivo en suspensión. (C) Organoides apicales 3 días después de la siembra en cultivo en suspensión sin BME para la inducción de inversión de polaridad. Barra de escala = 200 μm. Haga clic aquí para ver una versión más grande de esta figura.
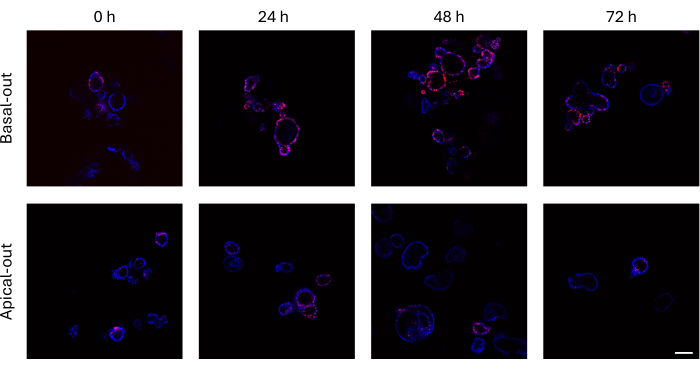
Figura 3: Organoides intestinales basales y apicales marcados con EdU. El panel superior, que muestra imágenes confocales de organoides basales a lo largo del tiempo, presenta muchas más células EdU+ en comparación con los organoides apicales (panel inferior) en los mismos puntos de tiempo. Barra de escala = 100 μm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Análisis cuantitativo de imágenes de organoides intestinales teñidos con EdU. (A) Organoides rodeados usando los modos Esfera y Polígono. (B) El área que se va a excluir del análisis está marcada en blanco (indicada por la flecha). Esta área marca la luz del organoide, que contiene una serie de células muertas, que se excluirán del análisis posterior. (C) La imagen después del análisis muestra todos los núcleos segmentados. Los núcleos de Hoechst33342+ están marcados en cian, los núcleos de EdU+ están marcados en amarillo y los núcleos que no alcanzan 15 μm2 están marcados en verde (Hoechst33342) y naranja (EdU). Barra de escala = 50 μm. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Tasa de proliferación de organoides. El porcentaje de ADN EdU+ del ADN total que sirve como proxy de la tasa de proliferación de organoides, mostrando una mayor proliferación en los organoides basales en comparación con los organoides apicales. Los datos se presentan como media SEM. Haga clic aquí para ver una versión más grande de esta figura.
| Medio basal | |
| Componentes de medios | Concentración final |
| DMEM-F12 | |
| Suplemento 100X de penicilina-estreptomicina para los medios de comunicación | 1 vez |
| Suplemento GlutaMAX | 2 mM |
| HEPES Solución Tampón 1 M, líquida | 10 mM |
| Medio refinado | |
| B27 SerumFree Supplement (50x), líquido | 1 vez |
| N-acetil-L-cisteína, cultivo celular probado | 1 mM |
| [Leu15]-Gastrina I humana | 10 nM |
| Un 83-01 | 500 nM |
| HGF humano | 50 ng/mL |
| NOGGIN Humano (Mamífero) | 100 ng/mL |
| IGF-I humano | 100 ng/mL |
| FGF humano-básico | 50 ng/mL |
| Medio acondicionado R-Spondin | 10 % (v/v) |
| Medio condicionado Wnt-3a | 50 % (v/v) |
| Medio basal | Volumen restante para la dilución de los factores de crecimiento/medio acondicionado mencionados anteriormente |
Tabla 1: Composición de medios basales y refinados.
Discusión
Este protocolo describe en detalle cómo inducir la inversión de polaridad en cultivos estándar de organoides intestinales derivados de células madre adultas. Los organoides apicales cumplen el propósito de acceder a la superficie de la célula apical, que generalmente está orientada hacia la luz del organoide. Ser capaz de sondear la superficie apical puede ser de gran importancia para ciertas aplicaciones, ya que esta es la porción de la membrana celular que está expuesta a todo el contenido del tracto digestivo en condiciones fisiológicas in vivo. Otros métodos para desafiar específicamente la superficie apical son la microinyección16, el uso de monocapas derivadas de organoides17 y la fragmentación de organoides18. Sin embargo, cada uno de estos métodos tiene inconvenientes y desventajas específicas, como hemos revisado con más detalle anteriormente19,20.
El método de generación de organoides apicales tiene varias ventajas: 1) La generación de organoides apicales es relativamente fácil en comparación con otros métodos y no requiere ninguna instrumentación o equipo especializado. Por lo tanto, los organoides apicales son una forma rentable de acceder a la superficie de las células apicales; 2) los organoides basales flotantes pueden cultivarse en paralelo y utilizarse como organoides de control significativos, especialmente para analizar los efectos específicos de la polaridad; 3) los organoides flotantes BO y AO pueden someterse a aplicaciones estándar posteriores, como se ejemplifica con la incorporación de EdU en este informe, pero también a la tinción inmuno(histo)química, así como a los ensayos basados en luminiscencia y fluorescencia, como se ha informado anteriormente13 , y a la obtención directa de imágenes de células vivas12. Los análisis posteriores, como el análisis de la incorporación de EdU o las tinciones inmuno(histo)químicas, también permiten cierta flexibilidad en términos de tiempo, ya que los organoides pueden almacenarse a 4 °C hasta su uso posterior.
A pesar de la facilidad de inversión de polaridad, existen algunos puntos críticos en nuestro protocolo que deben tenerse en cuenta al aplicar este método. En primer lugar, los organoides utilizados en el paso 2.1. debe ser de un cierto tamaño. En nuestra experiencia, el uso de organoides 3 días después de la tripsinización funciona bien. Sin embargo, este tiempo puede variar con organoides de otros animales debido a diferencias en la proliferación celular o dependiendo de la densidad de siembra de grupos unicelulares. Además, la disociación de BME utilizando la solución de recolección de organoides es incremental. Como se describe en Co et al., 20199, concentraciones de BME tan bajas como el 2,5% podrían ser suficientes para mantener los organoides en una morfología basal en cultivo en suspensión. Por lo tanto, se debe eliminar la mayor cantidad posible de BME para garantizar una inversión de polaridad eficiente. Otro punto importante es el uso correcto de las placas de cultivo de tejidos no tratadas para minimizar la adhesión de organoides durante el cultivo en suspensión. En nuestra experiencia, el recubrimiento previo de placas con solución antiadherente (paso 2.4.) permite el uso de prácticamente cualquier placa para el cultivo de organoides apicales. Sin embargo, se pueden explorar otras opciones que hagan que las placas de prerrecubrimiento sean redundantes.
La importancia de evaluar los efectos sobre la superficie celular basolateral y apical de las células epiteliales polarizadas se hace evidente en muchos casos. Por ejemplo, hemos analizado previamente los efectos de las toxinas de la bacteria anaerobia Clostridioides difficile sobre los organoides BO y AO. Los resultados de este estudio muestran que la toxina B de C. difficile (TcdB) solo daña la integridad de la barrera epitelial intestinal en los organoides BO del intestino delgado, pero no en los organoides AO. Varios otros informes que muestran reacciones específicas de dominio de varios microorganismos patógenos destacan la importancia de evaluar ambos lados de las células epiteliales 9,16,21,22,23,24,25.
Uno de los principales inconvenientes de los organoides apicales es su comportamiento alterado con respecto a la proliferación celular. Como se describió anteriormente, los organoides AO muestran tasas drásticamente reducidas de proliferación celular en comparación con sus contrapartes BO. Hemos reportado previamente que esta falta de proliferación va de la mano con un ligero aumento de los niveles de muerte celular13. En última instancia, esto no permite un cultivo a largo plazo de organoides AO a menos que se encuentre una manera de prolongar la vida útil de los organoides AO. Sin embargo, consideramos que los organoides AO son una herramienta muy útil para acceder fácilmente a la superficie celular apical, siempre y cuando los investigadores tengan cuidado con las conclusiones que podrían estar influenciadas por la disminución de la proliferación celular.
Divulgaciones
Los autores declaran no tener ningún conflicto de intereses.
Agradecimientos
Esta investigación fue apoyada utilizando recursos del VetImaging Core Facility (VetCore, Vetmeduni, Austria). Queremos agradecer a Ursula Reichart por su apoyo con el análisis semicuantitativo de imágenes. GC es beneficiario de una beca DOC (subvención número 26349) de la Academia Austriaca de Ciencias (ÖAW) en la División de Medicina Interna de Pequeños Animales de Vetmeduni.
Materiales
| Name | Company | Catalog Number | Comments |
| 24 well plates | Biologix | 07-6024 | |
| [Leu15]-Gastrin I human, ≥95% (HPLC) | Sigma-Aldrich | G-9145 | |
| µ-Slide 18 Well Glass Bottom | ibidi | 81817 | |
| 100X Penicillin-Streptomycin supplement for Media | Gibco/Thermo | 15140122 | |
| A 83-01 | Tocris Bioscience | 2939/10 | |
| Anti-Adherence Rinsing Solution | StemCell Technologies | 7010 | |
| arivis Pro | Zeiss | Version 4.2.2 | |
| B27 SerumFree Supplement (50X), liquid | Gibco/Thermo | 17504044 | |
| Bisbenzimide H 33342 (Hoechst 33342) | Abcam | ab145597 | |
| Bovine Serum Albumin | Sigma-Aldrich | A7906-100G | |
| Click-iT EdU Cell Proliferation Kit for Imaging, Alexa Fluor 647 dye | Thermo Scientific | C10340 | |
| DMEM-F12 | Gibco/Thermo | 11320033 | |
| Geltrex | Gibco/Thermo | A1413202 | |
| GlutaMAX Supplement | Gibco/Thermo | 35050061 | |
| HEPES Buffer Solution 1 M, liquid | Gibco/Thermo | 15630056 | |
| Human FGF-basic | PeproTech | 100-18B-100µG | |
| Human HGF | PeproTech | 100-39-100µG | |
| Human IGF-I | PeproTech | 100-11-100µG | |
| Human NOGGIN (Mammalian) | PeproTech | 120-10C-200µG | |
| N-Acetyl-L-cysteine,cell culture tested, BioReagent | Sigma-Aldrich | A9165-5G | |
| Organoid Harvesting Solution | Thermo | C10340 | |
| Triton(TM) X-100,for molecular biology | Sigma-Aldrich | T8787-250ML | |
| Trypsin-EDTA (0.05%), phenol red | Gibco/Thermo | 25300054 |
Referencias
- Sato, T., et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature. 459, 262-265 (2009).
- Li, Y., et al. BMP suppresses Wnt signaling via the Bcl11b-regulated NuRD complex to maintain intestinal stem cells. EMBO J. 43 (23), 6032-6051 (2024).
- Gaowa, A., Leangpanich, S., Park, E. J., Kawamoto, E., Shimaoka, M. Irisin promotes intestinal epithelial cell proliferation via Wnt/β-catenin and focal adhesion kinase signaling pathways. Sci Rep. 14, 25702 (2024).
- Basak, O., et al. Induced quiescence of Lgr5+ stem cells in intestinal organoids enables differentiation of hormone-producing enteroendocrine cells. Cell Stem Cell. 20 (2), 177-190 (2017).
- Ludikhuize, M. C., et al. Mitochondria define intestinal stem cell differentiation downstream of a FOXO/Notch axis. Cell Metab. 32 (5), 889-900.e7 (2020).
- Ettayebi, K., et al. Replication of human noroviruses in stem cell-derived human enteroids. Science (1979). 353 (6306), 1387-1393 (2016).
- Zhou, J., et al. Infection of bat and human intestinal organoids by SARS-CoV-2. Nat Med. 26 (7), 1077-1083 (2020).
- Joo, S. -. S., et al. Porcine intestinal apical-out organoid model for gut function study. Animals. 12 (3), 372 (2022).
- Co, J. Y., et al. Controlling epithelial polarity: A human enteroid model for host-pathogen interactions. Cell Rep. 26, 2509-2520.e4 (2019).
- Sasaki, N., et al. Development of a scalable coculture system for gut anaerobes and human colon epithelium. Gastroenterology. 159 (1), 388-390.e5 (2020).
- Mayorgas, A., et al. A novel strategy to study the invasive capability of adherent-invasive escherichia coli by using human primary organoid-derived epithelial monolayers. Front Immunol. 12, 646906 (2021).
- Csukovich, G., et al. Neutralising effects of different antibodies on clostridioides difficile toxins TcdA and TcdB in a translational approach. Int J Mol Sci. 24 (4), 3867 (2023).
- Csukovich, G., et al. Polarity reversal of canine intestinal organoids reduces proliferation and increases cell death. Cell Prolif. 57 (2), e13544 (2023).
- Kramer, N., et al. Generation of differentiating and long-living intestinal organoids reflecting the cellular diversity of canine intestine. Cells. 9 (4), 822 (2020).
- Pleguezuelos-Manzano, C., et al. Establishment and culture of human intestinal organoids derived from adult stem cells. Curr Protoc Immunol. 130 (1), e106 (2020).
- Dutta, D., Heo, I., O'Connor, R. Studying infection in 3D tissue-derived human organoid culture systems by microinjection. J Vis Exp. (151), e59610 (2019).
- Kumar, A., et al. Decreased SLC26A3 expression and function in intestinal epithelial cells in response to Cryptosporidium parvum infection. Am J Physiol Cell Physiol. 317 (6), C1205-C1212 (2019).
- Dutta, D., Clevers, H. Organoid culture systems to study host-pathogen interactions. Curr Opin Immunol. 48, 15-22 (2017).
- Csukovich, G., Pratscher, B., Burgener, I. A. The world of organoids: Gastrointestinal disease modelling in the age of 3R and one health with specific relevance to dogs and cats. Animals. 12 (18), 2461 (2022).
- Han, X., et al. Creating a more perfect union: Modeling intestinal bacteria-epithelial interactions using organoids. Cell Mol Gastroenterol Hepatol. 12 (2), 769-782 (2021).
- Li, Y., et al. Next-generation porcine intestinal organoids: an apical-out organoid model for swine enteric virus infection and immune response investigations. J Virol. 94 (21), e01006-e01020 (2020).
- Smith, D., et al. The development of ovine gastric and intestinal organoids for studying ruminant host-pathogen interactions. Front Cell Infect Microbiol. 11, 733811 (2021).
- Nash, T. J., Morris, K. M., Mabbott, N. A., Vervelde, L. Inside-out chicken enteroids with leukocyte component as a model to study host-pathogen interactions. Commun Biol. 4 (1), 377 (2021).
- Hanson, K. I., Weiss, A. A. Intestinal tissue response to Shiga toxin exposure. mBio. 15 (9), e0123224 (2024).
- Hovhannisyan, P., et al. Infection of human organoids supports an intestinal niche for Chlamydia trachomatis. PLoS Pathog. 20 (8), e1012144 (2024).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados