Method Article
Génération d’organoïdes intestinaux apical-out et évaluation du taux de prolifération des organoïdes à l’aide du marquage EdU
Dans cet article
Résumé
Ici, nous décrivons un protocole pour générer des organoïdes intestinaux apical-out à partir de cultures d’organoïdes standard enrobées de matrice. Il décrit également l’incorporation ultérieure de l’EdU dans les cellules à prolifération active et la quantification semi-automatique des cellules positives à l’EdU.
Résumé
Ici, nous décrivons la génération de cultures flottantes d’organoïdes intestinaux apical-out à partir de cultures d’organoïdes intestinaux enrobés d’hydrogel. En même temps, des cultures d’organoïdes basal-out flottants sont établies pour une comparaison directe entre les organoïdes apicaux et basaux. Les organoïdes apical-out et basal-out sont ensuite soumis à l’analogue de la thymidine 5-éthynyl-2'-désoxyuridine (EdU), qui est intégrée dans l’ADN nouvellement synthétisé pendant la phase S de la division cellulaire. Cette incorporation dans l’ADN peut être visualisée dans des organoïdes morphologiquement intacts à l’aide de la microscopie confocale à balayage laser. Les cellules marquées avec Hoechst33342 et EdU sont ensuite quantifiées de manière semi-automatique à l’aide d’un logiciel d’analyse d’images. Le calcul du pourcentage de cellules EdU-positives par rapport au nombre total de cellules permet d’analyser la prolifération cellulaire dans les organoïdes tridimensionnels (3D). Bien qu’il soit utilisé ici pour l’analyse de la prolifération dans les organoïdes intestinaux, le protocole est applicable à l’analyse de colorations spécifiques au noyau de divers types dans d’autres organoïdes ou des cultures cellulaires bidimensionnelles.
Introduction
Les organoïdes intestinaux sont des modèles in vitro tridimensionnels récapitulant l’épithélium intestinal comprenant différents types de cellules. Ces organoïdes peuvent être facilement établis à partir de cellules souches adultes isolées dans des cryptes intestinales1. Les organoïdes étant beaucoup plus proches de l’épithélium in vivo, ils prennent de plus en plus d’importance dans la recherche biomédicale. Les organoïdes de l’intestin ne sont pas seulement utilisés pour l’analyse des mécanismes physiologiques (par exemple, la signalisation de la niche intestinale 2,3 et la différenciation cellulaire 4,5), mais aussi pour la recherche sur les maladies infectieuses 6,7. Cependant, la croissance de cellules polarisées en 3D entourant une lumière centrale est un défi, car la surface de la cellule apicale finit par être inaccessible dans la lumière organoïde. L’examen des différences entre les surfaces cellulaires apicales et basolatérales peut être important dans les études métaboliques, comme en témoignent les différences dans l’absorption des acides gras8 et la recherche sur les maladies infectieuses 9,10,11,12.
La génération d’organoïdes dits apicals est une option facile pour surmonter ce problème. En retirant la matrice extracellulaire des cultures d’organoïdes standard (c’est-à-dire les organoïdes basal-out, intégrés dans la matrice) et en ensemenceant ces organoïdes dans un milieu sans matrice, un changement de polarité peut être induit9.
Comme nous l’avons déjà publié, la plupart des organoïdes inversent leur polarité en 12 heures. Cependant, il faut 48 à 72 h pour obtenir une culture avec plus de 90 % d’organoïdes apicals. Malgré leur avantage de permettre l’accès à la surface cellulaire apicale, les organoïdes apical-out montrent une prolifération significativement réduite après l’inversion de polarité, tandis que les taux de mort cellulaire augmentent de13. Le facteur de l’activité proliférative peut représenter une variable confondante dans diverses analyses et doit être gardé à l’esprit lors de la conception d’une expérience.
Nous présentons ici un protocole détaillé pour l’établissement de cultures d’organoïdes apical-out intestinaux et d’organoïdes de contrôle basal-out flottants pour les analyses en aval. De plus, nous décrivons le marquage avec la 5-éthynyl-2'-désoxyuridine (EdU), incorporée dans l’ADN nouvellement synthétisé et marque ainsi les cellules en prolifération active. Nous décrivons plus en détail l’analyse semi-automatique d’images du taux de prolifération des organoïdes à l’aide du logiciel arivis Pro (Zeiss) par quantification des cellules EdU+ . Un schéma du processus est présenté à la figure 1.
Protocole
Des organoïdes intestinaux dérivés de cellules souches adultes de chiens, établis selon Kramer et al., 202014 ont été utilisés. Selon les directives du comité d’éthique de l’établissement, l’utilisation de matériel tissulaire prélevé lors de l’excision thérapeutique ou de l’autopsie est incluse dans le « consentement du propriétaire pour le traitement » de l’université, qui a été signé par tous les patients propriétaires.
1. Culture d’organoïdes
- Cultivez des organoïdes intestinaux dérivés de cellules souches adultes intégrés dans la matrice d’extrait de membrane basale (BME) de votre choix dans des plaques de 24 puits et divisez-les mécaniquement à l’aide de pipettes en verre, comme décrit en détail dans Pleguezuelos et al., 202015.
REMARQUE : Pour le protocole décrit ici, Geltrex a été utilisé comme BME. - Retirer avec précaution le milieu raffiné (Tableau 1) des organoïdes inclus dans le BME.
- Ajouter 500 μL de trypsine-EDTA à 0,05 % par puits et détacher tous les dômes matriciels du puits en pipetant de haut en bas à plusieurs reprises. Transférez les organoïdes dans un tube de 15 mL et remettez-les bien en suspension pour dissocier complètement la matrice d’hydrogel.
- Incuber des organoïdes avec 0,05 % de trypsine-EDTA à 37 °C dans un bain-marie jusqu’à ce que tous les organoïdes soient dissociés en cellules uniques ou en petits amas de cellules.
- Diluer la trypsine/suspension cellulaire avec un milieu basal (tableau 1). Utilisez au moins deux fois plus de milieu de base que de trypsine.
- Cellules de centrifugation à 8 °C à 420 × g pendant 5 min. Retirez autant de surnageant que possible.
- Cellules germes intégrées dans BME dans autant de puits que ceux utilisés pour la trypsinisation à l’étape 1.3. et leur fournir un milieu raffiné (tableau 1).
- Incuber des cellules pendant 3 jours dans un incubateur de culture cellulaire et passer à l’étape 2.1.
2. Induction de l’inversion de polarité et marquage EdU
- Retirez délicatement le milieu des organoïdes incrustés dans le BME.
- Ajoutez 500 μL de solution de récolte d’organoïdes dans chaque puits et détachez tous les dômes matriciels du puits en pipetant de haut en bas à plusieurs reprises. Transférez les organoïdes dans un tube de 15 mL et remettez-les bien en suspension pour dissocier complètement la matrice d’hydrogel.
- Incuber le tube de 15 mL sur de la glace pendant 1,5 h. Bien agiter le tube toutes les 10 minutes pour éviter l’agglutination des organoïdes et assurer une dissociation uniforme des composants restants de l’hydrogel.
- Pendant l’incubation, enduire une plaque de 96 puits d’une solution anti-adhérence. Pour chaque puits d’une plaque de 24 puits trypsinisée à l’étape 1.3., enduire 8 puits d’une plaque de 96 puits. Incuber pendant au moins 1 h à température ambiante (RT).
- Ajouter au tube de 15 mL au tube de 15 mL au moins deux fois plus de PBS que la solution de prélèvement d’organoïdes a été utilisée et centrifuger à 8 °C à 150 × g pendant 5 min.
- Retirer le surnageant et remettre les organoïdes en suspension dans 1 mL de PBS.
- Transvaser 500 μL de la suspension organoïde/PBS dans un tube séparé de 15 mL et centrifuger à nouveau les deux tubes à 8 °C à 150 × g pendant 5 min.
- Retirez toute la solution anti-adhérence de la plaque à 96 puits.
- Éliminer autant de surnageant que possible après la centrifugation.
- Utilisez l’un des tubes pour la génération d’organoïdes basaux flottants (BO) et l’autre tube pour les organoïdes apical-out (AO).
- Pour les organoïdes BO, ajouter 100 μL de milieu raffiné contenant 7,5 % de BME par puits de la plaque de 96 puits dans un tube. Mélangez bien et répartissez uniformément les organoïdes sur le nombre souhaité de puits de la plaque pré-enduite de 96 puits.
- Pour les organoïdes AO, ajouter 100 μL de milieu raffiné ordinaire (sans aucun BME ajouté) par puits de la plaque de 96 puits dans l’autre tube. Mélangez bien et répartissez uniformément les organoïdes sur le nombre souhaité de puits de la plaque pré-enduite de 96 puits.
- Incuber les organoïdes à 37 °C et 5 % de CO2 pendant 3 jours.
REMARQUE : Les différences morphologiques commenceront à être visibles après le jour 1. Cependant, les résultats publiés précédemment montrent qu’il faut environ 3 jours pour que la grande majorité des organoïdes présentent une polarité apicale13. - À des moments définis, par exemple 24 h/48 h/72 h après l’induction de l’inversion de polarité, ajouter 50 μL de 3 μM d’EdU dilués dans un milieu raffiné à tous les puits d’organoïdes à étiqueter, pour obtenir une concentration finale de 1 μM EdU dans chaque puits.
- Incuber à 37 °C et 5 % de CO2 pendant 1,5 h.
- Prélever les organoïdes marqués EdU à l’aide d’embouts à gros calibre et les transférer dans un tube de 15 ml.
- Ajouter une quantité égale de 4 % de PFA dans chaque tube (organoïdes BO et AO ; concentration finale de PFA = 2 %) et mélanger soigneusement.
- Incuber à RT pendant 15 min.
- Ajouter au moins deux fois la quantité de PBS qu’il y a de volume dans le tube de 15 mL et centrifuger à 8 °C à 80 × g pendant 5 min.
- Retirer le surnageant et remettre les organoïdes en suspension dans 1 mL de PBS.
- Centrifuger à nouveau à 8 °C à 80 × g pendant 5 min.
- Retirez le surnageant, remettez les organoïdes en suspension dans 1 mL de PBS et transférez-les dans un tube de 1,5 mL. Stockez les organoïdes à 4 °C jusqu’à ce que la réaction de coloration EdU soit effectuée (voir rubrique 3).
- Répétez les étapes 2.14 à 2.22 pour chaque point temporel à analyser.
3. Réaction de coloration Click-it EdU et coloration Hoechst 33342
- Centrifuger les organoïdes fixés (à partir de l’étape 2.22) dans un tube de 1,5 mL à 4 °C à 50 × g pendant 5 min.
- Retirer le surnageant et remettre en suspension les organoïdes dans 1 mL d’albumine sérique bovine (BSA) à 3 % diluée dans du PBS à l’aide d’embouts à large diamètre.
- Répétez les étapes 3.1. et 3.2 (étapes de lavage).
- Retirer le surnageant et remettre les organoïdes en suspension dans 1 mL de Triton X-100 à 0,5 % dilué dans du PBS.
- Incuber à RT pendant 20 min.
- Centrifuger les organoïdes à RT à 50 × g pendant 5 min.
- Dans un tube frais, préparez 1x additif tampon en mélangeant 45 μL de H2O avec 5 μL d’additif tampon 10x.
- Dans un autre tube, préparez le cocktail réactionnel en mélangeant les composants suivants : 385,8 μL de H2O, 43 μL de 10x tampon de réaction, 20 μL de CuSO, 4,1,2 μL d’azoture Alexa Fluor 647 et 50 μL ou 1x additif tampon (à partir de l’étape 3.7.).
REMARQUE : Ce mélange est suffisant pour cinq réactions et peut être augmenté ou réduit en fonction du nombre requis de réactions de coloration. - Répétez les étapes 3.1. et 3.2. deux fois (étapes de lavage).
- Retirer le surnageant et ajouter 100 μL de cocktail de réaction (étape 3.8.) dans chaque tube contenant des organoïdes.
- Incuber à RT pendant 30 min dans l’obscurité.
- Répétez les étapes 3.1. et 3.2 (étapes de lavage).
- Centrifuger les organoïdes à 50 × g pendant 5 min à RT.
- Mettre les organoïdes en suspension dans 1 mL de 10 μg/mL de Hoechst33342 dilué dans du PBS.
- Incuber à RT pendant 45 min dans l’obscurité.
- Répétez les étapes 3.1. et 3.2 (étapes de lavage).
- Centrifuger les organoïdes à 50 × g pendant 5 min à 4 °C.
- Intégrer les organoïdes de la matrice BME de votre choix dans une lame compatible avec la microscopie confocale (p. ex., puits μ-Slide 18)
- Imagez des organoïdes au microscope confocal.
4. Analyse d’image semi-automatique
REMARQUE : Pour cette analyse, la version 4.2.2 d’arivis Pro (logiciel d’analyse d’images) a été utilisée.
- Importez les images du microscope confocal dans le logiciel d’analyse d’images et définissez un dossier dans lequel le fichier d’analyse sera enregistré. Utilisez l’option Images comme points temporels pour importer plusieurs images (répliquer des images, différents points temporels, différents traitements, etc.). Chaque image sera alors traitée comme un point temporel unique, ce qui permettra de passer facilement d’une image à l’autre.
- Ouvrez le panneau d’analyse et importez le pipeline d’analyse fourni en tant que Fichier supplémentaire 1 (arivis_EdU_pipeline).
- Utilisez l’outil Placer de nouveaux objets et Sphère avec la balise Organoïde pour encercler facilement tous les organoïdes bien séparés.
- Passez à l’image suivante, c’est-à-dire au prochain point temporel, et procédez au marquage des organoïdes pour toutes les images.
- Pour les organoïdes qui sont très proches les uns des autres et qui ne peuvent donc pas être séparés à l’aide du mode Sphère , ouvrez la liste d’objets et activez la balise Organoïde.
- Continuez à utiliser l’outil Dessiner des objets en mode Polygone pour marquer les contours des organoïdes à la main.
REMARQUE : Dans la liste d’objets, tous les organoïdes sont maintenant marqués et attribués à un point temporel spécifique. Ce point temporel correspond à l’ordre des images importées à l’étape 4.1. Ces points temporels peuvent désormais être renommés pour correspondre au nom de l’image d’origine si vous le souhaitez. - Dans la liste des objets, marquez tous les objets (=organoïdes) et cliquez avec le bouton droit de la souris, puis cliquez sur Supprimer les balises pour supprimer la balise Manuel de tous les objets.
- Désactivez la balise Organoïde dans la liste d’objets et marquez toutes les zones des objets précédemment définis qui doivent être exclues de l’analyse à l’aide de l’outil Dessiner des objets en mode Polygone . Ces zones peuvent inclure des cellules mortes dans la lumière organoïde ou des zones floues, ce qui peut fausser l’analyse.
- Lorsque vous ouvrez la liste d’objets, assurez-vous que tous les organoïdes sont répertoriés avec l’étiquette Organoïde et que toutes les zones qui doivent être exclues ont l’étiquette Manuel.
- Démarrez le pipeline d’analyse en cliquant sur la flèche dans le panneau supérieur gauche.
REMARQUE : Le logiciel segmente désormais tous les noyaux dans les canaux Hoechst33342 et EdU. Ce pipeline d’analyse ne prend en compte que les noyaux segmentés de Hoechst33342+ et EdU+ de plus de 15 μm2 (c’est-à-dire l’aire dans le segment du noyau) afin de s’assurer que les petits noyaux, très probablement provenant de cellules mortes, sont exclus de l’analyse. - Recherchez les résultats de l’analyse dans la liste d’objets en cliquant sur la balise Feature Columns .
- Passez à la vue détaillée principale et sélectionnez la balise Organoids dans le panneau supérieur et la fonction Premier point de temps.
- Dans le panneau inférieur, sélectionnez les fonctionnalités à afficher par organoïde. Utilisez la zone de projection (x/y/z) (voxel), l’intensité moyenne # 1 (canal EdU) et les intensités SD #1.
- Exportez les résultats (Master Detail Report) à l’aide de la fonction d’exportation Excel .
- Enregistrez le fichier d’analyse et fermez le logiciel.
5. Détermination des taux de prolifération des organoïdes
- Ouvrez le fichier de rapport détaillé principal exporté à l’étape 4.14.
- Il y a un onglet distinct pour chaque image analysée. Calculez la somme aire/voxel pour les noyaux et la coloration EdU pour chaque image. Ensuite, copiez toutes les données résultantes dans un nouvel onglet collectif et regroupez toutes les données faisant référence au même point temporel.
- Ensuite, calculez l’aire totale des noyaux cellulaires (c’est-à-dire la somme de l’aire de Hoechst33342+ et de l’aire EdU+ ) pour chaque image.
- Ensuite, divisez la surface totale par elle-même (= 100 %) et la surface moyenne EdU+ par la surface totale (=pourcentage d’ADN EdU+ ).
- Tracez le pourcentage d’ADN prolifératif des organoïdes BO et AO sur l’axe des y et les différents points temporels sur l’axe des x.
Résultats
Les organoïdes cultivés pendant 3 jours après la trypsinisation doivent idéalement avoir une taille comprise entre 50 et 250 μm, comme le montre la figure 2A. Les organoïdes qui sont considérablement plus grands que cela peuvent ne pas inverser efficacement leur polarité. Les organoïdes plus grands peuvent également commencer à bourgeonner, et nous avons remarqué que ces organoïdes peuvent également avoir des problèmes d’inversion efficace de la polarité. Les organoïdes basal-out et apical-out présentent déjà des différences morphologiques évidentes en imagerie en fond clair. Alors que les organoïdes basaux conservent leur grande lumière (Figure 2B) après 3 jours de culture en suspension, les organoïdes apicals (Figure 2C) semblent plus compacts. Une caractéristique très spécifique des cultures en suspension apicales est le nombre de cellules mortes flottant autour des organoïdes. Cela est dû au fait que les organoïdes apical-out extrudent des cellules mortes et différenciées en phase terminale dans le milieu environnant, tandis que les organoïdes basal-out accumulent des cellules mortes dans la lumière organoïde.
Les organoïdes intestinaux qui ont incorporé EdU dans leur ADN nouvellement synthétisé avant la division cellulaire peuvent être imagés à l’aide d’un microscope confocal. Les organoïdes basaux-out montrent des signaux EdU beaucoup plus élevés au fil du temps que leurs homologues apical-out (Figure 3). Il est important de noter que tous les organoïdes ne présentent pas de cellules EdU+ malgré leur croissance. Cela est dû au fait qu’aucune cellule EdU+ n’est présente au niveau de la couche d’imagerie spécifique.
Ces images confocales peuvent être analysées quantitativement à l’aide d’un logiciel d’analyse d’images et du pipeline d’analyse fourni (Fichier supplémentaire 1). Lors de la mise en œuvre du protocole d’analyse, les organoïdes doivent d’abord être encerclés (Figure 4A) avant d’exclure certaines zones de l’analyse proprement dite (Figure 4B). Ces zones d’exclusion peuvent faire référence à des cellules mortes à l’intérieur de la lumière organoïde, qui ne présentent pas d’intérêt pour la quantification des signaux EdU. L’exécution ultérieure du pipeline d’analyse automatique conduit à la segmentation de tous les noyaux détectés, en différenciant les noyaux Hoechst33342+ et EdU+ . De plus, tous les noyaux situés en dessous d’un seuil de 15 μm2 sont exclus, car il s’agit très probablement de cellules mortes ou de noyaux qui ne sont pas représentés en entier dans cette couche d’image (figure 4C).
Après l’analyse de l’image à l’aide du logiciel et l’exportation des mesures de quantification, ces données sont calculées pour analyser le pourcentage de signal EdU+ par rapport à la masse totale d’ADN (Figure 5). Dans le cas présenté dans ce manuscrit, les organoïdes intestinaux apical-out présentent des niveaux de prolifération considérablement réduits par rapport aux organoïdes basaux-out flottants.

Figure 1 : Vue d’ensemble schématique du protocole décrit. Le schéma comprend la culture d’organoïdes, l’induction de l’inversion de polarité, l’étiquetage EdU et l’analyse semi-automatique des images. Figurine créée avec Biorender.com. Veuillez cliquer ici pour voir une version agrandie de cette figure.
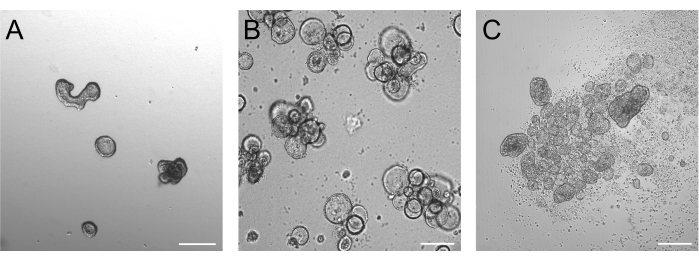
Figure 2 : Organoïdes avant et après l’induction de l’inversion de polarité. (A) Organoïdes intestinaux enrobés de matrice trois jours après que les cellules trypsinisées ont été ensemencées dans un milieu de croissance. (B) Organoïdes basal-out flottants trois jours après l’ensemencement en culture en suspension. (C) Organoïdes apicals 3 jours après l’ensemencement en culture en suspension sans BME pour l’induction de l’inversion de polarité. Barre d’échelle = 200 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
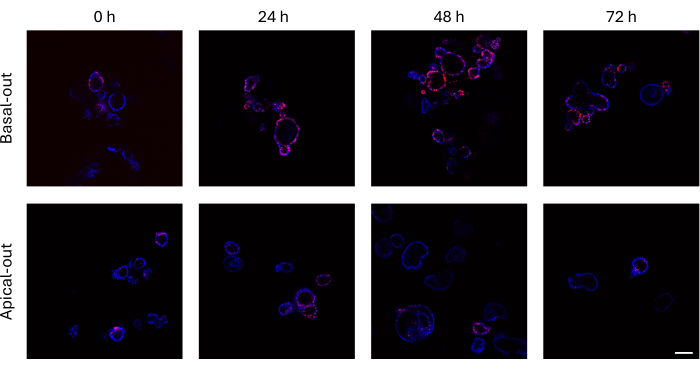
Figure 3 : Organoïdes intestinaux basal-out et apical-out marqués avec EdU. Le panneau supérieur, qui représente des images confocales d’organoïdes basaux-out au fil du temps, présente beaucoup plus de cellules EdU+ que d’organoïdes apical-out (panneau inférieur) aux mêmes points temporels. Barre d’échelle = 100 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.

Figure 4 : Analyse d’images quantitatives d’organoïdes intestinaux colorés à l’EdU. (A) Organoïdes encerclés à l’aide des modes Sphère et Polygone. (B) La zone à exclure de l’analyse est marquée en blanc (indiquée par la flèche). Cette zone marque la lumière organoïde, qui contient un certain nombre de cellules mortes, qui seront exclues de l’analyse ultérieure. (C) L’image après analyse montre tous les noyaux segmentés. Les noyaux de Hoechst33342 + sont marqués en cyan, les noyaux EdU+ sont marqués en jaune et les noyaux n’atteignant pas 15 μm2 sont marqués en vert (Hoechst33342) et en orange (EdU). Barre d’échelle = 50 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Taux de prolifération des organoïdes. Le pourcentage d’ADN EdU+ par rapport à l’ADN total servant de proxy pour le taux de prolifération des organoïdes montrant une prolifération plus élevée dans les organoïdes basaux par rapport aux organoïdes apicals. Les données sont présentées sous forme de SEM moyen. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Milieu basal | |
| Composants multimédias | Concentration finale |
| DMEM-F12 | |
| 100X supplément de pénicilline-streptomycine pour les milieux | 1x |
| Supplément GlutaMAX | 2 millions d’euros |
| HEPES Buffer Solution 1 M, liquide | 10 millions d’euros |
| Médium raffiné | |
| B27 Supplément sans sérum (50x), liquide | 1x |
| N-acétyl-L-cystéine, testé en culture cellulaire | 1 million d’euros |
| [Leu15]-Gastrin I humain | 10 nM |
| A 83-01 | 500 nM |
| EFC humain | 50 ng/mL |
| NOGGIN humain (mammifère) | 100 ng/mL |
| IGF-I humain | 100 ng/mL |
| FGF humain de base | 50 ng/mL |
| Milieu conditionné R-Spondin | 10 % (v/v) |
| Milieu conditionné Wnt-3a | 50 % (v/v) |
| Milieu basal | Volume restant pour la dilution des facteurs de croissance/milieu conditionné mentionnés ci-dessus |
Tableau 1 : Composition des milieux de base et raffinés.
Discussion
Ce protocole décrit en détail comment induire l’inversion de polarité dans des cultures d’organoïdes intestinaux standard dérivés de cellules souches adultes. Les organoïdes apicals servent à accéder à la surface de la cellule apicale, qui est généralement orientée vers la lumière organoïde. Être capable de sonder la surface apicale peut être d’une grande importance pour certaines applications, car il s’agit de la partie de la membrane cellulaire qui est exposée à tout le contenu du tube digestif dans des conditions physiologiques in vivo. D’autres méthodes pour défier spécifiquement la surface apicale sont la micro-injection16, l’utilisation de monocouches dérivées d’organoïdes17 et la fragmentation organoïde18. Cependant, chacune de ces méthodes présente des inconvénients et des inconvénients spécifiques, comme nous et d’autres l’avons examiné plus en détail précédemment19,20.
La méthode de génération d’organoïdes apical-out présente plusieurs avantages : 1) La génération d’organoïdes apical-out est relativement facile par rapport à d’autres méthodes et ne nécessite aucune instrumentation ou équipement spécialisé. Par conséquent, les organoïdes apicals sont un moyen rentable d’accéder à la surface de la cellule apicale ; 2) les organoïdes basaux flottants peuvent être cultivés en parallèle et utilisés comme organoïdes de contrôle significatifs, en particulier pour analyser les effets spécifiques à la polarité ; 3) les organoïdes flottants BO et AO peuvent être soumis à des applications standard en aval, comme en témoigne l’incorporation d’EdU dans ce rapport, mais aussi la coloration immuno(histo)chimique, ainsi que les tests basés sur la luminescence et la fluorescence, comme indiqué précédemment13 , et l’imagerie directe des cellules vivantes12. Les analyses en aval, telles que l’analyse de l’incorporation d’EdU ou les colorations immuno(histo)chimiques, permettent également une certaine flexibilité en termes de temps, car les organoïdes peuvent être stockés à 4 °C jusqu’à une utilisation ultérieure.
Malgré la facilité d’inversion de polarité, il existe certains points critiques dans notre protocole qui doivent être pris en compte lors de l’application de cette méthode. Tout d’abord, les organoïdes utilisés à l’étape 2.1. doit être d’une certaine taille. D’après notre expérience, l’utilisation d’organoïdes 3 jours après la trypsinisation fonctionne bien. Cependant, ce temps peut varier avec celui des organoïdes d’autres animaux en raison des différences de prolifération cellulaire ou en fonction de la densité d’ensemencement des grappes unicellulaires. De plus, la dissociation de l’EMB à l’aide de la solution de récolte d’organoïdes est progressive. Comme décrit par Co et al., 20199, des concentrations de BME aussi faibles que 2,5 % pourraient être suffisantes pour maintenir les organoïdes dans une morphologie basale en culture en suspension. Par conséquent, la plus grande partie possible de BME doit être éliminée pour assurer une inversion efficace de la polarité. Un autre point important est l’utilisation correcte des plaques de culture tissulaire non traitées pour minimiser la fixation des organoïdes pendant la culture en suspension. D’après notre expérience, le pré-revêtement des plaques avec une solution anti-adhérence (étape 2.4.) permet d’utiliser pratiquement n’importe quelle plaque pour la culture d’organoïdes apicals. Cependant, d’autres options rendant les plaques de pré-revêtement redondantes peuvent être explorées.
L’importance d’évaluer les effets sur la surface des cellules basolatérales et apicales des cellules épithéliales polarisées devient évidente dans de nombreux cas. Par exemple, nous avons précédemment analysé les effets des toxines de la bactérie anaérobie Clostridioides difficile sur les organoïdes BO et AO. Les résultats de cette étude montrent que la toxine B de C. difficile (TcdB) n’endommage l’intégrité de la barrière épithéliale intestinale que dans les organoïdes BO de l’intestin grêle mais pas dans les organoïdes AO. Plusieurs autres rapports montrant des réactions spécifiques à un domaine de divers micro-organismes pathogènes soulignent l’importance d’évaluer les deux côtés des cellules épithéliales 9,16,21,22,23,24,25.
L’un des principaux inconvénients des organoïdes apicals est leur comportement altéré en ce qui concerne la prolifération cellulaire. Comme décrit ci-dessus, les organoïdes AO présentent des taux de prolifération cellulaire considérablement réduits par rapport à leurs homologues BO. Nous avons déjà signalé que cette absence de prolifération va de pair avec une légère augmentation des niveaux de mort cellulaire13. En fin de compte, cela ne permet pas une culture à long terme des organoïdes AO à moins qu’un moyen de prolonger la durée de vie des organoïdes AO ne soit trouvé. Cependant, nous considérons que les organoïdes AO sont un outil très utile pour accéder facilement à la surface des cellules apicales, à condition que les chercheurs soient prudents avec les conclusions qui pourraient être influencées par une diminution de la prolifération cellulaire.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Cette recherche a été financée à l’aide des ressources de la plateforme VetImaging (VetCore, Vetmeduni, Autriche). Nous tenons à remercier Ursula Reichart pour son soutien dans l’analyse semi-quantitative des images. GC est récipiendaire d’une bourse DOC (numéro de subvention 26349) de l’Académie autrichienne des sciences (ÖAW) à la Division de médecine interne des petits animaux de Vetmeduni.
matériels
| Name | Company | Catalog Number | Comments |
| 24 well plates | Biologix | 07-6024 | |
| [Leu15]-Gastrin I human, ≥95% (HPLC) | Sigma-Aldrich | G-9145 | |
| µ-Slide 18 Well Glass Bottom | ibidi | 81817 | |
| 100X Penicillin-Streptomycin supplement for Media | Gibco/Thermo | 15140122 | |
| A 83-01 | Tocris Bioscience | 2939/10 | |
| Anti-Adherence Rinsing Solution | StemCell Technologies | 7010 | |
| arivis Pro | Zeiss | Version 4.2.2 | |
| B27 SerumFree Supplement (50X), liquid | Gibco/Thermo | 17504044 | |
| Bisbenzimide H 33342 (Hoechst 33342) | Abcam | ab145597 | |
| Bovine Serum Albumin | Sigma-Aldrich | A7906-100G | |
| Click-iT EdU Cell Proliferation Kit for Imaging, Alexa Fluor 647 dye | Thermo Scientific | C10340 | |
| DMEM-F12 | Gibco/Thermo | 11320033 | |
| Geltrex | Gibco/Thermo | A1413202 | |
| GlutaMAX Supplement | Gibco/Thermo | 35050061 | |
| HEPES Buffer Solution 1 M, liquid | Gibco/Thermo | 15630056 | |
| Human FGF-basic | PeproTech | 100-18B-100µG | |
| Human HGF | PeproTech | 100-39-100µG | |
| Human IGF-I | PeproTech | 100-11-100µG | |
| Human NOGGIN (Mammalian) | PeproTech | 120-10C-200µG | |
| N-Acetyl-L-cysteine,cell culture tested, BioReagent | Sigma-Aldrich | A9165-5G | |
| Organoid Harvesting Solution | Thermo | C10340 | |
| Triton(TM) X-100,for molecular biology | Sigma-Aldrich | T8787-250ML | |
| Trypsin-EDTA (0.05%), phenol red | Gibco/Thermo | 25300054 |
Références
- Sato, T., et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature. 459, 262-265 (2009).
- Li, Y., et al. BMP suppresses Wnt signaling via the Bcl11b-regulated NuRD complex to maintain intestinal stem cells. EMBO J. 43 (23), 6032-6051 (2024).
- Gaowa, A., Leangpanich, S., Park, E. J., Kawamoto, E., Shimaoka, M. Irisin promotes intestinal epithelial cell proliferation via Wnt/β-catenin and focal adhesion kinase signaling pathways. Sci Rep. 14, 25702 (2024).
- Basak, O., et al. Induced quiescence of Lgr5+ stem cells in intestinal organoids enables differentiation of hormone-producing enteroendocrine cells. Cell Stem Cell. 20 (2), 177-190 (2017).
- Ludikhuize, M. C., et al. Mitochondria define intestinal stem cell differentiation downstream of a FOXO/Notch axis. Cell Metab. 32 (5), 889-900.e7 (2020).
- Ettayebi, K., et al. Replication of human noroviruses in stem cell-derived human enteroids. Science (1979). 353 (6306), 1387-1393 (2016).
- Zhou, J., et al. Infection of bat and human intestinal organoids by SARS-CoV-2. Nat Med. 26 (7), 1077-1083 (2020).
- Joo, S. -. S., et al. Porcine intestinal apical-out organoid model for gut function study. Animals. 12 (3), 372 (2022).
- Co, J. Y., et al. Controlling epithelial polarity: A human enteroid model for host-pathogen interactions. Cell Rep. 26, 2509-2520.e4 (2019).
- Sasaki, N., et al. Development of a scalable coculture system for gut anaerobes and human colon epithelium. Gastroenterology. 159 (1), 388-390.e5 (2020).
- Mayorgas, A., et al. A novel strategy to study the invasive capability of adherent-invasive escherichia coli by using human primary organoid-derived epithelial monolayers. Front Immunol. 12, 646906 (2021).
- Csukovich, G., et al. Neutralising effects of different antibodies on clostridioides difficile toxins TcdA and TcdB in a translational approach. Int J Mol Sci. 24 (4), 3867 (2023).
- Csukovich, G., et al. Polarity reversal of canine intestinal organoids reduces proliferation and increases cell death. Cell Prolif. 57 (2), e13544 (2023).
- Kramer, N., et al. Generation of differentiating and long-living intestinal organoids reflecting the cellular diversity of canine intestine. Cells. 9 (4), 822 (2020).
- Pleguezuelos-Manzano, C., et al. Establishment and culture of human intestinal organoids derived from adult stem cells. Curr Protoc Immunol. 130 (1), e106 (2020).
- Dutta, D., Heo, I., O'Connor, R. Studying infection in 3D tissue-derived human organoid culture systems by microinjection. J Vis Exp. (151), e59610 (2019).
- Kumar, A., et al. Decreased SLC26A3 expression and function in intestinal epithelial cells in response to Cryptosporidium parvum infection. Am J Physiol Cell Physiol. 317 (6), C1205-C1212 (2019).
- Dutta, D., Clevers, H. Organoid culture systems to study host-pathogen interactions. Curr Opin Immunol. 48, 15-22 (2017).
- Csukovich, G., Pratscher, B., Burgener, I. A. The world of organoids: Gastrointestinal disease modelling in the age of 3R and one health with specific relevance to dogs and cats. Animals. 12 (18), 2461 (2022).
- Han, X., et al. Creating a more perfect union: Modeling intestinal bacteria-epithelial interactions using organoids. Cell Mol Gastroenterol Hepatol. 12 (2), 769-782 (2021).
- Li, Y., et al. Next-generation porcine intestinal organoids: an apical-out organoid model for swine enteric virus infection and immune response investigations. J Virol. 94 (21), e01006-e01020 (2020).
- Smith, D., et al. The development of ovine gastric and intestinal organoids for studying ruminant host-pathogen interactions. Front Cell Infect Microbiol. 11, 733811 (2021).
- Nash, T. J., Morris, K. M., Mabbott, N. A., Vervelde, L. Inside-out chicken enteroids with leukocyte component as a model to study host-pathogen interactions. Commun Biol. 4 (1), 377 (2021).
- Hanson, K. I., Weiss, A. A. Intestinal tissue response to Shiga toxin exposure. mBio. 15 (9), e0123224 (2024).
- Hovhannisyan, P., et al. Infection of human organoids supports an intestinal niche for Chlamydia trachomatis. PLoS Pathog. 20 (8), e1012144 (2024).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon