Method Article
Generierung apikaler Darmorganoide und Bestimmung der Organoid-Proliferationsrate mittels EdU-Markierung
In diesem Artikel
Zusammenfassung
Hier beschreiben wir ein Protokoll zur Generierung von apikal-out-Darmorganoiden aus Standard-Matrix-eingebetteten Organoidkulturen. Es skizziert auch den anschließenden Einbau von EdU in aktiv proliferierende Zellen und die halbautomatische Quantifizierung von EdU-positiven Zellen.
Zusammenfassung
Hier beschreiben wir die Erzeugung von Schwimmkulturen von apikal-out-Darmorganoiden aus Hydrogel-eingebetteten Darmorganoidkulturen. Gleichzeitig werden schwimmende Basal-Out-Organoidkulturen für den direkten Vergleich zwischen apikal-out- und Basal-out-Organoiden etabliert. Apical-out- und Basal-out-Organoide werden anschließend dem Thymidin-Analogon 5-Ethinyl-2'-Desoxyuridin (EdU) ausgesetzt, das während der S-Phase der Zellteilung in die neu synthetisierte DNA integriert wird. Dieser Einbau in die DNA kann in morphologisch intakten Organoiden mittels konfokaler Laserscanning-Mikroskopie sichtbar gemacht werden. Zellen, die mit Hoechst33342 und EdU markiert sind, werden dann halbautomatisch mit Hilfe einer Bildanalysesoftware quantifiziert. Die Berechnung des prozentualen Anteils der EdU-positiven Zellen an der Gesamtzahl der Zellen ermöglicht die Analyse der Zellproliferation in dreidimensionalen (3D) Organoiden. Obwohl das Protokoll hier für die Analyse der Proliferation in Darmorganoiden verwendet wird, ist es auch auf die Analyse von kernspezifischen Färbungen verschiedener Art in anderen Organoiden oder zweidimensionalen Zellkulturen anwendbar.
Einleitung
Darm-Organoide sind dreidimensionale In-vitro-Modelle, die das Darmepithel aus verschiedenen Zelltypen rekapitulieren. Diese Organoide lassen sich leicht aus adulten Stammzellen etablieren, die aus Darmkrypten isoliert wurden1. Da Organoide viel näher am In-vivo-Epithel liegen, gewinnen sie in der biomedizinischen Forschung zunehmend an Bedeutung. Organoide des Darms werden nicht nur für die Analyse physiologischer Mechanismen (z.B. intestinale Nischensignalwege 2,3 und Zelldifferenzierungen 4,5) verwendet, sondern auch für die Erforschung von Infektionskrankheiten 6,7. Die Züchtung polarisierter Zellen in 3D, die ein zentrales Lumen umschließen, ist jedoch eine Herausforderung, da die apikale Zelloberfläche innerhalb des organoiden Lumens unzugänglich ist. Die Untersuchung der Unterschiede zwischen apikalen und basolateralen Zelloberflächen kann in metabolischen Studien wichtig sein, wie z. B. Unterschiede in der Fettsäureaufnahme8 und die Erforschung von Infektionskrankheiten 9,10,11,12 zeigen.
Die Erzeugung sogenannter apikaler Organoide ist eine einfache Möglichkeit, dieses Problem zu lösen. Durch Entfernen der extrazellulären Matrix aus Standard-Organoidkulturen (d. h. basal-out, matrixeingebetteten Organoiden) und Aussäen dieser Organoide in einem matrixfreien Medium kann ein Polaritätswechsel induziert werden9.
Wie wir bereits veröffentlicht haben, kehren die meisten Organoide ihre Polarität innerhalb von 12 h um. Es dauert jedoch 48-72 Stunden, um eine Kultur mit mehr als 90% apikalen Organoiden zu erhalten. Trotz ihres Vorteils, den Zugang zur apikalen Zelloberfläche zu ermöglichen, zeigen apikal-out-Organoide eine signifikant verringerte Proliferation nach Polaritätsumkehr, während die Zelltodraten steigen13. Der Faktor der proliferativen Aktivität kann in verschiedenen Analysen eine Störvariable darstellen und sollte bei der Planung eines Experiments berücksichtigt werden.
Hier stellen wir ein detailliertes Protokoll für die Etablierung von intestinalen apikalen Organoidkulturen und schwimmenden basalen Kontrollorganoiden für nachgelagerte Analysen vor. Des Weiteren beschreiben wir die Markierung mit 5-Ethinyl-2'-Desoxyuridin (EdU), das in neu synthetisierte DNA eingebaut wird und somit aktiv proliferierende Zellen markiert. Des Weiteren beschreiben wir die halbautomatische Bildanalyse der Proliferationsrate der Organoide mit der Software arivis Pro (Zeiss) durch Quantifizierung von EdU+ Zellen. Ein Schema des Prozesses ist in Abbildung 1 dargestellt.
Protokoll
Es wurden adulte Stammzell-abgeleitete Darmorganoide von Hunden verwendet, die nach Kramer et al., 202014 etabliert wurden. Basierend auf den Richtlinien der institutionellen Ethikkommission ist die Verwendung von Gewebematerial, das während der therapeutischen Exzision oder post mortem entnommen wurde, in der "Einverständniserklärung des Eigentümers zur Behandlung" der Universität enthalten, die von allen Patientenbesitzern unterzeichnet wurde.
1. Organoid-Kultur
- Züchten Sie aus adulten Stammzellen gewonnene Darmorganoide, die in die Basalmembranextrakt-Matrix (BME) Ihrer Wahl eingebettet sind, in 24-Well-Platten und spalten Sie sie mechanisch mit Glaspipetten, wie ausführlich beschrieben in Pleguezuelos et al., 202015.
HINWEIS: Für das hier beschriebene Protokoll wurde Geltrex als BME verwendet. - Entfernen Sie vorsichtig das raffinierte Medium (Tabelle 1) von den BME-eingebetteten Organoiden.
- Geben Sie 500 μl 0,05 % Trypsin-EDTA pro Vertiefung hinzu und lösen Sie alle Matrixkuppeln aus der Vertiefung, indem Sie wiederholt auf und ab pipettieren. Übertragen Sie die Organoide in ein 15-ml-Röhrchen und resuspendieren Sie sie gut, um die Hydrogelmatrix vollständig zu dissoziieren.
- Organoide mit 0,05 % Trypsin-EDTA bei 37 °C im Wasserbad inkubieren, bis alle Organoide in einzelne Zellen oder kleine Zellcluster dissoziiert sind.
- Verdünnen Sie die Trypsin/Zellsuspension mit Basalmedium (Tabelle 1). Verwenden Sie mindestens doppelt so viel Basalmedium wie Trypsin.
- Zellen bei 8 °C bei 420 × g 5 min zentrifugieren. So viel Überstand wie möglich entfernen.
- Keimzellen, die in BME eingebettet sind, in so vielen Vertiefungen, wie in Schritt 1.3 für die Trypsinisierung verwendet wurden. und versorgen sie mit raffiniertem Medium (Tabelle 1).
- Inkubieren Sie die Zellen 3 Tage lang in einem Zellkultur-Inkubator und fahren Sie mit Schritt 2.1 fort.
2. Induktion der Polaritätsumkehr und EdU-Beschriftung
- Entfernen Sie das Medium vorsichtig von den BME-eingebetteten Organoiden.
- Geben Sie 500 μl Organoid-Erntelösung in jede Vertiefung und lösen Sie alle Matrixkuppeln aus der Vertiefung, indem Sie wiederholt auf und ab pipettieren. Übertragen Sie die Organoide in ein 15-ml-Röhrchen und resuspendieren Sie sie gut, um die Hydrogelmatrix vollständig zu dissoziieren.
- Inkubieren Sie das 15-ml-Röhrchen 1,5 h lang auf Eis. Schütteln Sie das Röhrchen alle 10 Minuten gut, um ein Verklumpen der Organoide zu verhindern und eine gleichmäßige Dissoziation der verbleibenden Hydrogelkomponenten zu gewährleisten.
- Beschichten Sie während der Inkubation eine 96-Well-Platte mit Anti-Adhärenzlösung. Für jede Vertiefung einer 24-Well-Platte, die in Schritt 1.3 trypsinisiert wurde, werden 8 Wells einer 96-Well-Platte beschichtet. Mindestens 1 h bei Raumtemperatur (RT) inkubieren.
- Geben Sie mindestens doppelt so viel PBS wie die Organoid-Erntelösung in das 15-ml-Röhrchen gegeben und zentrifugieren Sie 5 Minuten lang bei 8 °C bei 150 × g .
- Entfernen Sie den Überstand und resuspendieren Sie die Organoide in 1 ml PBS.
- 500 μl der Organoid-/PBS-Suspension in ein separates 15-ml-Röhrchen überführen und beide Röhrchen erneut bei 8 °C bei 150 × g für 5 min zentrifugieren.
- Entfernen Sie alle Anti-Adhärenz-Lösungen von der 96-Well-Platte.
- Nach der Zentrifugation so viel Überstand wie möglich entfernen.
- Verwenden Sie eine der Röhren für die Erzeugung von schwimmenden Basal-out-Organoiden (BO) und die andere Röhre für apikale (AO) Organoide.
- Bei BO-Organoiden geben Sie 100 μl raffiniertes Medium mit 7,5 % BME pro Vertiefung der 96-Well-Platte in ein Röhrchen. Gut mischen und die Organoide gleichmäßig über die gewünschte Anzahl von Vertiefungen der vorbeschichteten 96-Well-Platte verteilen.
- Bei AO-Organoiden geben Sie 100 μl raffiniertes Normalmedium (ohne Zugabe von BME) pro Vertiefung der 96-Well-Platte in das andere Röhrchen. Gut mischen und die Organoide gleichmäßig über die gewünschte Anzahl von Vertiefungen der vorbeschichteten 96-Well-Platte verteilen.
- Inkubieren Sie die Organoide 3 Tage lang bei 37 °C und 5 % CO2.
HINWEIS: Morphologische Unterschiede werden nach Tag 1 sichtbar. Zuvor veröffentlichte Ergebnisse zeigen jedoch, dass es etwa 3 Tage dauert, bis die überwiegende Mehrheit der Organoide eine apikale Polarität13 aufweist. - Zu definierten Zeitpunkten, z. B. 24 h/48 h/72 h nach Induktion der Polaritätsumkehr, werden 50 μL 3 μM EdU, verdünnt in raffiniertem Medium, in alle Vertiefungen der zu markierenden Organoide gegeben, um eine Endkonzentration von 1 μM EdU in jeder Vertiefung zu erhalten.
- Bei 37 °C und 5 % CO2 1,5 h inkubieren.
- Sammeln Sie EdU-markierte Organoide mit breiten Spitzen und überführen Sie sie in ein 15-ml-Röhrchen.
- In jedes Röhrchen (BO- und AO-Organoide; Endkonzentration von PFA = 2 %) wird die gleiche Menge PFA gegeben und vorsichtig gemischt.
- 15 Minuten bei RT inkubieren.
- Fügen Sie mindestens die doppelte Menge PBS hinzu, da das Volumen im 15-ml-Röhrchen vorhanden ist, und zentrifugieren Sie bei 8 °C bei 80 × g für 5 min.
- Entfernen Sie den Überstand und resuspendieren Sie die Organoide in 1 ml PBS.
- Erneut bei 8 °C bei 80 × g 5 min zentrifugieren.
- Entfernen Sie den Überstand, resuspendieren Sie die Organoide in 1 ml PBS und überführen Sie sie in ein 1,5 ml-Röhrchen. Lagern Sie die Organoide bei 4 °C, bis die EdU-Färbereaktion durchgeführt wird (siehe Abschnitt 3).
- Wiederholen Sie die Schritte 2.14-2.22 für jeden zu analysierenden Zeitpunkt.
3. Click-it EdU-Färbereaktion und Hoechst 33342 Färbereaktion
- Die fixierten Organoide (aus Schritt 2.22.) werden in einem 1,5 mL Röhrchen bei 4 °C bei 50 × g für 5 min zentrifugiert.
- Entfernen Sie den Überstand und resuspendieren Sie die Organoide in 1 ml 3 % Rinderserumalbumin (BSA), verdünnt in PBS, unter Verwendung von Spitzen mit breiter Bohrung.
- Wiederholen Sie die Schritte 3.1. und 3.2 (Waschschritte).
- Entfernen Sie den Überstand und resuspendieren Sie Organoide in 1 ml 0,5 % Triton X-100, verdünnt in PBS.
- 20 Minuten bei RT inkubieren.
- Zentrifugieren Sie die Organoide bei RT bei 50 × g für 5 min.
- Bereiten Sie in einem frischen Röhrchen 1x Pufferadditiv vor, indem Sie 45 μl H2O mit 5 μl 10x Pufferadditiv mischen.
- Bereiten Sie den Reaktionscocktail in einem anderen Röhrchen zu, indem Sie die folgenden Komponenten mischen: 385,8 μl H2O, 43 μl 10x Reaktionspuffer, 20 μl CuSO, 4,1,2 μl Alexa Fluor 647 azid und 50 μl oder 1x Pufferadditiv (aus Schritt 3.7.).
HINWEIS: Diese Mischung ist für fünf Reaktionen ausreichend und kann entsprechend der erforderlichen Anzahl von Färbereaktionen nach oben und unten skaliert werden. - Wiederholen Sie die Schritte 3.1. und 3.2. zweimal (Waschschritte).
- Den Überstand entfernen und 100 μl Reaktionscocktail (Schritt 3.8.) in jedes Röhrchen mit Organoiden geben.
- Inkubieren Sie bei RT für 30 min im Dunkeln.
- Wiederholen Sie die Schritte 3.1. und 3.2 (Waschschritte).
- Organoide bei 50 × g für 5 min bei RT zentrifugieren.
- Resuspendieren Sie Organoide in 1 ml von 10 μg/ml, Hoechst33342, verdünnt in PBS.
- Inkubieren Sie bei RT für 45 Minuten im Dunkeln.
- Wiederholen Sie die Schritte 3.1. und 3.2 (Waschschritte).
- Organoide bei 50 × g 5 min bei 4 °C zentrifugieren.
- Seed-Organoide in der BME-Matrix der Wahl in einen Objektträger, der mit der konfokalen Mikroskopie kompatibel ist (z. B. μ-Slide 18 Well)
- Organoide unter einem konfokalen Mikroskop abbilden.
4. Halbautomatische Bildanalyse
HINWEIS: Für diese Analyse wurde arivis Pro Version 4.2.2 (Bildanalysesoftware) verwendet.
- Importieren Sie die konfokalen Mikroskopbilder in die Bildanalysesoftware und definieren Sie einen Ordner, in dem die Analysedatei gespeichert wird. Verwenden Sie die Option Bilder als Zeitpunkte , um mehrere Bilder zu importieren (Bilder replizieren, verschiedene Zeitpunkte, unterschiedliche Behandlungen usw.). Jedes Bild wird dann als ein einzelner Zeitpunkt behandelt, was ein einfaches Wechseln zwischen den einzelnen Bildern ermöglicht.
- Öffnen Sie den Analysebereich, und importieren Sie die Analysepipeline, die als ergänzende Datei 1 (arivis_EdU_pipeline) bereitgestellt wird.
- Verwenden Sie das Werkzeug "Neue Objekte platzieren " und "Kugel " mit dem Tag "Organoid ", um alle gut getrennten Organoide einfach einzukreisen.
- Wechseln Sie zum nächsten Bild, d.h. zum nächsten Zeitpunkt, und fahren Sie mit dem Markieren von Organoiden für alle Bilder fort.
- Für Organoide, die sehr nahe beieinander liegen und daher nicht mit dem Kugelmodus getrennt werden können, öffnen Sie die Objektliste und aktivieren Sie den Tag Organoid.
- Verwenden Sie weiterhin das Werkzeug "Objekte zeichnen " im Polygon-Modus , um organoide Umrisse von Hand zu markieren.
HINWEIS: In der Objektliste sind nun alle Organoide markiert und einem bestimmten Zeitpunkt zugeordnet. Dieser Zeitpunkt entspricht der Reihenfolge der Bilder, die in Schritt 4.1 importiert wurden. Diese Zeitpunkte können jetzt bei Bedarf so umbenannt werden, dass sie mit dem ursprünglichen Bildnamen übereinstimmen. - Markieren Sie in der Objektliste alle Objekte (=Organoide) und klicken Sie mit der rechten Maustaste, dann klicken Sie auf Tags entfernen , um das manuelle Tag von allen Objekten zu entfernen.
- Deaktivieren Sie das Organoid-Tag in der Objektliste und markieren Sie alle Bereiche innerhalb zuvor definierter Objekte, die von der Analyse ausgeschlossen werden sollen, mit dem Werkzeug Objekte zeichnen im Polygon-Modus . Zu diesen Bereichen können abgestorbene Zellen innerhalb des Organoidlumens oder verschwommene Bereiche gehören, die die Analyse verzerren könnten.
- Achten Sie beim Öffnen der Objektliste darauf, dass alle Organoide mit dem Tag Organoid aufgelistet sind und alle Bereiche, die ausgeschlossen werden sollen, das Tag Manuell haben.
- Starten Sie die Analysepipeline, indem Sie auf den Pfeil im oberen linken Bereich klicken.
HINWEIS: Die Software segmentiert jetzt alle Kerne in den Hoechst33342- und EdU-Kanälen. Diese Analysepipeline berücksichtigt nur segmentierte Hoechst33342+ - und EdU+ -Kerne, die größer als 15 μm2 sind (d. h. Fläche im Zellkernsegment), um sicherzustellen, dass kleine Zellkerne, die höchstwahrscheinlich aus toten Zellen stammen, von der Analyse ausgeschlossen werden. - Suchen Sie die Ergebnisse der Analyse in der Objektliste, indem Sie auf das Feld Feature-Spalten klicken.
- Wechseln Sie in die Master-Detailansicht und wählen Sie im oberen Bereich das Tag Organoide und das Feature Erster Zeitpunkt.
- Wählen Sie im unteren Bereich die Merkmale aus, die pro Organoid angezeigt werden sollen. Verwenden Sie Projektionsbereich (x/y/z) (Voxel), mittlere Intensität # 1 (EdU-Kanal) und SD-Intensitäten #1.
- Exportieren Sie die Ergebnisse (Master Detail Report) mit der Excel-Export-Funktion .
- Speichern Sie die Analysedatei, und schließen Sie die Software.
5. Bestimmung der Proliferationsraten von Organoiden
- Öffnen Sie die in Schritt 4.14 exportierte Master-Detailberichtsdatei.
- Für jedes analysierte Bild gibt es eine separate Registerkarte. Berechnen Sie die Flächen-/Voxelsumme für die Kerne und den EdU-Farbstoff für jedes Bild. Kopieren Sie dann alle resultierenden Daten in eine neue kollektive Registerkarte und gruppieren Sie alle Daten, die sich auf denselben Zeitpunkt beziehen.
- Berechnen Sie dann die Gesamtfläche der Zellkerne (d. h. die Summe aus Hoechst33342+ Fläche und EdU+ Fläche) für jedes Bild.
- Teilen Sie anschließend die Gesamtfläche durch sich selbst (= 100%) und die mittlere EdU+ -Fläche durch die Gesamtfläche (=% der EdU+ -DNA).
- Plotten Sie den prozentualen Anteil der proliferativen DNA von BO- und AO-Organoiden auf der y-Achse und die verschiedenen Zeitpunkte auf der x-Achse.
Ergebnisse
Organoide, die 3 Tage nach der Trypsinisierung gezüchtet werden, sollten idealerweise zwischen 50 und 250 μm liegen, wie in Abbildung 2A dargestellt. Organoide, die deutlich größer sind, kehren ihre Polarität möglicherweise nicht effizient um. Größere Organoide können auch anfangen zu knospen, und wir haben festgestellt, dass diese Organoide auch Probleme mit einer effizienten Polaritätsumkehr haben können. Basal-out- und apikal-out-Organoide weisen bereits in der Hellfeld-Bildgebung offensichtliche morphologische Unterschiede auf. Während basal-out-Organoide nach 3 Tagen Suspensionskultur ihr großes Lumen behalten (Abbildung 2B), erscheinen apikal-out-Organoide (Abbildung 2C) kompakter. Ein sehr spezifisches Merkmal von apikalen Suspensionskulturen ist die Anzahl der toten Zellen, die um die Organoide herum schweben. Dies ist darauf zurückzuführen, dass apikal-out-Organoide terminale differenzierte und tote Zellen in das umgebende Medium extrudieren, während basal-out-Organoide tote Zellen im Organoidlumen ansammeln.
Darmorganoide, die vor der Zellteilung EdU in ihre neu synthetisierte DNA eingebaut haben, können mit einem konfokalen Mikroskop abgebildet werden. Basal-out-Organoide zeigen im Laufe der Zeit viel höhere EdU-Signale als ihre apikal-out-Gegenstücke (Abbildung 3). Es ist wichtig zu beachten, dass nicht alle Organoide trotz zunehmender Größe EdU+-Zellen aufweisen. Dies liegt daran, dass keine EdU+ -Zellen in der spezifischen Bildgebungsschicht vorhanden sind.
Diese konfokalen Bilder können mit Hilfe einer Bildanalysesoftware und der bereitgestellten Analysepipeline quantitativ analysiert werden (Ergänzende Datei 1). Bei der Durchführung des Analyseprotokolls müssen Organoide zunächst eingekreist werden (Abbildung 4A), bevor dann bestimmte Bereiche aus der eigentlichen Analyse ausgeschlossen werden (Abbildung 4B). Diese Ausschlussbereiche können sich auf tote Zellen im Organoidlumen beziehen, die für die Quantifizierung von EdU-Signalen nicht von Interesse sind. Die anschließende Ausführung der automatischen Analysepipeline führt zu einer Segmentierung aller detektierten Zellkerne, wobei zwischen Hoechst33342+- und EdU+ -Kernen unterschieden wird. Zusätzlich werden alle Zellkerne unterhalb eines Cut-offs von 15μm 2 ausgeschlossen, da es sich höchstwahrscheinlich um tote Zellen oder Kerne handelt, die in dieser Bildschicht nicht vollständig dargestellt werden (Abbildung 4C).
Nach der Bildanalyse mit der Software und dem Exportieren der Quantifizierungsmessungen werden diese Daten weiter berechnet, um den prozentualen Anteil des EdU+ -Signals an der Gesamtmasse der DNA zu analysieren (Abbildung 5). In dem in diesem Manuskript vorgestellten Fall zeigen apikal-out-Darm-Organoide eine drastisch reduzierte Proliferation im Vergleich zu floatenden basal-out-Organoiden.

Abbildung 1: Schematische Übersicht des beschriebenen Protokolls. Das Schema umfasst die Kultivierung von Organoiden, die Induktion der Polaritätsumkehr, die EdU-Markierung und die halbautomatische Bildanalyse. Mit Biorender.com erstellte Figur. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
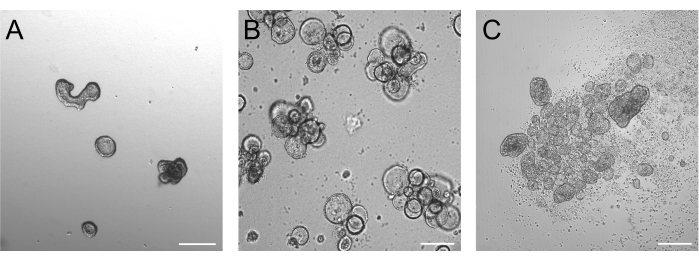
Abbildung 2: Organoide vor und nach Induktion der Polaritätsumkehr. (A) In die Matrix eingebettete Darmorganoide drei Tage nachdem trypsinisierte Zellen in ein Wachstumsmedium ausgesät wurden. (B) Floatende Basal-out-Organoide drei Tage nach der Aussaat in Suspensionskultur. (C) Apical-out-Organoide 3 Tage nach der Aussaat in Suspensionskultur ohne BME zur Induktion der Polaritätsumkehr. Maßstabsleiste = 200 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
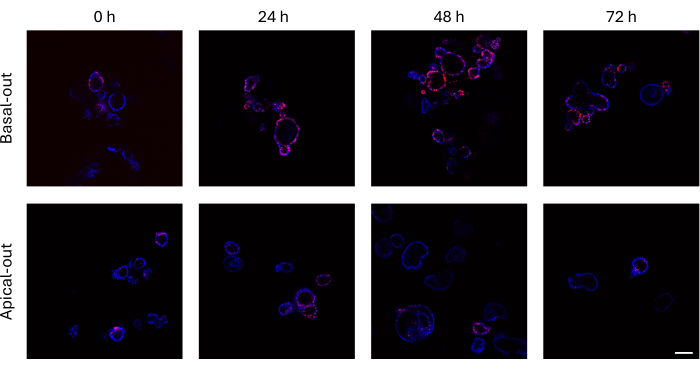
Abbildung 3: Basal-out- und apikal-out-Darmorganoide, die mit EdU markiert sind. Das obere Bild, das konfokale Bilder von basal-out-Organoiden im Zeitverlauf darstellt, zeigt viel mehr EdU+ -Zellen im Vergleich zu apikal-out-Organoiden (unteres Bild) zu gleichen Zeitpunkten. Maßstabsleiste = 100 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Quantitative Bildanalyse von EdU-gefärbten Darmorganoiden. (A) Einkreisende Organoide mit den Modi Kugel und Polygon . (B) Der Bereich, der von der Analyse ausgeschlossen werden soll, ist weiß markiert (gekennzeichnet durch den Pfeil). Dieser Bereich markiert das Organoid-Lumen, das eine Reihe von toten Zellen enthält, die von der anschließenden Analyse ausgeschlossen werden. (C) Das Bild nach der Analyse zeigt alle segmentierten Kerne. Hoechst33342 + Kerne sind in Cyan markiert, EdU+ Kerne sind gelb markiert und Kerne mit einer Größe von nicht 15μm 2 sind grün (Hoechst33342) und orange (EdU) markiert. Maßstabsleiste = 50 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Proliferationsrate von Organoiden. Der prozentuale Anteil der EdU+ -DNA an der Gesamt-DNA, der als Proxy für die Organoid-Proliferationsrate dient, zeigt eine höhere Proliferation bei basal-out-Organoiden im Vergleich zu apikal-out-Organoiden. Die Daten sind als mittlere REM dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Basales Medium | |
| Medienkomponenten | Endkonzentration |
| DMEM-F12 | |
| 100X Penicillin-Streptomycin-Ergänzung für Medien | 1x |
| GlutaMAX Ergänzung | ca. 2 mM |
| HEPES Pufferlösung 1 M, flüssig | ca. 10 mM |
| Raffiniertes Medium | |
| B27 SerumFree Supplement (50x), flüssig | 1x |
| N-Acetyl-L-Cystein, Zellkultur getestet | 1 mM |
| [Leu15]-Gastrin I Mensch | 10 nm |
| A 83-01 | 500 nM |
| Menschliches HGF | 50 ng/ml |
| Mensch NOGGIN (Säugetier) | 100 ng/ml |
| Menschliches IGF-I | 100 ng/ml |
| Mensch FGF-basic | 50 ng/ml |
| R-Spondin konditioniertes Medium | 10 % (v/v) |
| Wnt-3a konditioniertes Medium | 50 % (v/v) |
| Basales Medium | Restvolumen zur Verdünnung der oben genannten Wachstumsfaktoren/konditioniertes Medium |
Tabelle 1: Zusammensetzung der basalen und raffinierten Medien.
Diskussion
Dieses Protokoll beschreibt im Detail, wie eine Polaritätsumkehr in Standardkulturen von adulten Stammzellen aus Darmorganoiden induziert wird. Apikale Organoide dienen dem Zweck, Zugang zur apikalen Zelloberfläche zu erhalten, die in der Regel zum Organoidlumen hin orientiert ist. Die Möglichkeit, die apikale Oberfläche zu sondieren, kann für bestimmte Anwendungen von großer Bedeutung sein, da dies der Teil der Zellmembran ist, der unter physiologischen Bedingungen in vivo dem gesamten Inhalt des Verdauungstrakts ausgesetzt ist. Andere Verfahren zur spezifischen Herausforderung der apikalen Oberfläche sind die Mikroinjektion16, die Verwendung von Organoid-abgeleiteten Monoschichten17 und die Organoidfragmentierung18. Jede dieser Methoden hat jedoch spezifische Nachteile und Nachteile, wie wir und andere zuvor ausführlicher untersucht haben19,20.
Die Methode zur Erzeugung von apikalen Organoiden hat mehrere Vorteile: 1) Die Erzeugung von apikalen Organoiden ist im Vergleich zu anderen Methoden relativ einfach und erfordert keine speziellen Instrumente oder Geräte. Daher sind apikale Organoide eine kostengünstige Möglichkeit, an die apikale Zelloberfläche zu gelangen. 2) schwimmende Basal-out-Organoide können parallel kultiviert und als sinnvolle Kontrollorganoide verwendet werden, insbesondere zur Analyse polaritätsspezifischer Effekte; 3) Floating-BO- und AO-Organoide können Standard-Downstream-Anwendungen unterzogen werden, wie z. B. die EdU-Integration in diesem Bericht, aber auch immun(histo)chemische Färbung sowie Lumineszenz- und Fluoreszenz-basierte Assays, wie bereits berichtet13 , und direkte Lebendzellbildgebung12. Nachgelagerte Analysen, wie z. B. die Analyse des EdU-Einbaus oder immun(histo)chemischer Färbungen, ermöglichen ebenfalls eine gewisse zeitliche Flexibilität, da Organoide bis zur weiteren Verwendung bei 4 °C gelagert werden können.
Trotz der Leichtigkeit der Polaritätsumkehr gibt es einige kritische Punkte in unserem Protokoll, die bei der Anwendung dieser Methode berücksichtigt werden sollten. Zunächst die in Schritt 2.1 verwendeten Organoide. sollte eine bestimmte Größe haben. Nach unserer Erfahrung funktioniert die Verwendung von Organoiden 3 Tage nach der Trypsinisierung gut. Diese Zeit kann jedoch bei Organoiden anderer Tiere aufgrund von Unterschieden in der Zellproliferation oder abhängig von der Aussaatdichte von Einzelzellclustern variieren. Darüber hinaus ist die Dissoziation von BME mit der Organoid-Harvesting-Lösung inkrementell. Wie von Co et al., 20199 beschrieben, könnten BME-Konzentrationen von nur 2,5 % ausreichen, um Organoide in einer basal-out-Morphologie in Suspensionskultur zu halten. Daher muss so viel BME wie möglich entfernt werden, um eine effiziente Polaritätsumkehr zu gewährleisten. Ein weiterer wichtiger Punkt ist die richtige Verwendung von unbehandelten Gewebekulturplatten, um die Anheftung von Organoiden während der Suspensionskultur zu minimieren. Nach unserer Erfahrung ermöglicht die Vorbeschichtung von Platten mit Anti-Adhärenz-Lösung (Schritt 2.4.) die Verwendung praktisch jeder Platte für die Kultivierung von apikal-out-Organoiden. Es können jedoch auch andere Optionen geprüft werden, um Vorbeschichtungsplatten überflüssig zu machen.
Die Bedeutung der Beurteilung von Effekten auf die basolaterale sowie die apikale Zelloberfläche polarisierter Epithelzellen wird in vielen Fällen deutlich. So haben wir bereits die Auswirkungen von Toxinen des anaeroben Bakteriums Clostridioides difficile auf BO- und AO-Organoide analysiert. Die Ergebnisse dieser Studie zeigen, dass C. difficile Toxin B (TcdB) die Integrität der Darmepithelbarriere nur in BO-Organoiden des Dünndarms schädigt, nicht aber in AO-Organoiden. Mehrere andere Berichte, die domänenspezifische Reaktionen verschiedener pathogener Mikroorganismen zeigen, unterstreichen die Bedeutung der Bewertung beider Seiten von Epithelzellen 9,16,21,22,23,24,25.
Ein großer Nachteil von apikal-out-Organoiden ist ihr verändertes Verhalten in Bezug auf die Zellproliferation. Wie oben beschrieben, zeigen AO-Organoide im Vergleich zu ihren BO-Pendants drastisch reduzierte Raten der Zellproliferation. Wir haben bereits berichtet, dass dieser Mangel an Proliferation mit einem leicht steigenden Zelltod einhergeht13. Letztendlich ermöglicht dies keine langfristige Kultivierung von AO-Organoiden, es sei denn, es wird ein Weg gefunden, die Lebensdauer von AO-Organoiden zu verlängern. Wir betrachten AO-Organoide jedoch als ein sehr nützliches Werkzeug, um leicht auf die apikale Zelloberfläche zugreifen zu können, solange die Forscher vorsichtig mit Schlussfolgerungen sind, die durch eine verminderte Zellproliferation beeinflusst werden könnten.
Offenlegungen
Die Autoren erklären, dass kein Interessenkonflikt besteht.
Danksagungen
Diese Forschung wurde mit Mitteln der VetImaging Core Facility (VetCore, Vetmeduni, Österreich) unterstützt. Wir danken Ursula Reichart für ihre Unterstützung bei der semiquantitativen Bildanalyse. GC ist Stipendiat eines DOC-Stipendiums (Förderkennzeichen 26349) der Österreichischen Akademie der Wissenschaften (ÖAW) an der Abteilung für Innere Medizin der Kleintiere der Vetmeduni.
Materialien
| Name | Company | Catalog Number | Comments |
| 24 well plates | Biologix | 07-6024 | |
| [Leu15]-Gastrin I human, ≥95% (HPLC) | Sigma-Aldrich | G-9145 | |
| µ-Slide 18 Well Glass Bottom | ibidi | 81817 | |
| 100X Penicillin-Streptomycin supplement for Media | Gibco/Thermo | 15140122 | |
| A 83-01 | Tocris Bioscience | 2939/10 | |
| Anti-Adherence Rinsing Solution | StemCell Technologies | 7010 | |
| arivis Pro | Zeiss | Version 4.2.2 | |
| B27 SerumFree Supplement (50X), liquid | Gibco/Thermo | 17504044 | |
| Bisbenzimide H 33342 (Hoechst 33342) | Abcam | ab145597 | |
| Bovine Serum Albumin | Sigma-Aldrich | A7906-100G | |
| Click-iT EdU Cell Proliferation Kit for Imaging, Alexa Fluor 647 dye | Thermo Scientific | C10340 | |
| DMEM-F12 | Gibco/Thermo | 11320033 | |
| Geltrex | Gibco/Thermo | A1413202 | |
| GlutaMAX Supplement | Gibco/Thermo | 35050061 | |
| HEPES Buffer Solution 1 M, liquid | Gibco/Thermo | 15630056 | |
| Human FGF-basic | PeproTech | 100-18B-100µG | |
| Human HGF | PeproTech | 100-39-100µG | |
| Human IGF-I | PeproTech | 100-11-100µG | |
| Human NOGGIN (Mammalian) | PeproTech | 120-10C-200µG | |
| N-Acetyl-L-cysteine,cell culture tested, BioReagent | Sigma-Aldrich | A9165-5G | |
| Organoid Harvesting Solution | Thermo | C10340 | |
| Triton(TM) X-100,for molecular biology | Sigma-Aldrich | T8787-250ML | |
| Trypsin-EDTA (0.05%), phenol red | Gibco/Thermo | 25300054 |
Referenzen
- Sato, T., et al. Single Lgr5 stem cells build crypt-villus structures in vitro without a mesenchymal niche. Nature. 459, 262-265 (2009).
- Li, Y., et al. BMP suppresses Wnt signaling via the Bcl11b-regulated NuRD complex to maintain intestinal stem cells. EMBO J. 43 (23), 6032-6051 (2024).
- Gaowa, A., Leangpanich, S., Park, E. J., Kawamoto, E., Shimaoka, M. Irisin promotes intestinal epithelial cell proliferation via Wnt/β-catenin and focal adhesion kinase signaling pathways. Sci Rep. 14, 25702 (2024).
- Basak, O., et al. Induced quiescence of Lgr5+ stem cells in intestinal organoids enables differentiation of hormone-producing enteroendocrine cells. Cell Stem Cell. 20 (2), 177-190 (2017).
- Ludikhuize, M. C., et al. Mitochondria define intestinal stem cell differentiation downstream of a FOXO/Notch axis. Cell Metab. 32 (5), 889-900.e7 (2020).
- Ettayebi, K., et al. Replication of human noroviruses in stem cell-derived human enteroids. Science (1979). 353 (6306), 1387-1393 (2016).
- Zhou, J., et al. Infection of bat and human intestinal organoids by SARS-CoV-2. Nat Med. 26 (7), 1077-1083 (2020).
- Joo, S. -. S., et al. Porcine intestinal apical-out organoid model for gut function study. Animals. 12 (3), 372 (2022).
- Co, J. Y., et al. Controlling epithelial polarity: A human enteroid model for host-pathogen interactions. Cell Rep. 26, 2509-2520.e4 (2019).
- Sasaki, N., et al. Development of a scalable coculture system for gut anaerobes and human colon epithelium. Gastroenterology. 159 (1), 388-390.e5 (2020).
- Mayorgas, A., et al. A novel strategy to study the invasive capability of adherent-invasive escherichia coli by using human primary organoid-derived epithelial monolayers. Front Immunol. 12, 646906 (2021).
- Csukovich, G., et al. Neutralising effects of different antibodies on clostridioides difficile toxins TcdA and TcdB in a translational approach. Int J Mol Sci. 24 (4), 3867 (2023).
- Csukovich, G., et al. Polarity reversal of canine intestinal organoids reduces proliferation and increases cell death. Cell Prolif. 57 (2), e13544 (2023).
- Kramer, N., et al. Generation of differentiating and long-living intestinal organoids reflecting the cellular diversity of canine intestine. Cells. 9 (4), 822 (2020).
- Pleguezuelos-Manzano, C., et al. Establishment and culture of human intestinal organoids derived from adult stem cells. Curr Protoc Immunol. 130 (1), e106 (2020).
- Dutta, D., Heo, I., O'Connor, R. Studying infection in 3D tissue-derived human organoid culture systems by microinjection. J Vis Exp. (151), e59610 (2019).
- Kumar, A., et al. Decreased SLC26A3 expression and function in intestinal epithelial cells in response to Cryptosporidium parvum infection. Am J Physiol Cell Physiol. 317 (6), C1205-C1212 (2019).
- Dutta, D., Clevers, H. Organoid culture systems to study host-pathogen interactions. Curr Opin Immunol. 48, 15-22 (2017).
- Csukovich, G., Pratscher, B., Burgener, I. A. The world of organoids: Gastrointestinal disease modelling in the age of 3R and one health with specific relevance to dogs and cats. Animals. 12 (18), 2461 (2022).
- Han, X., et al. Creating a more perfect union: Modeling intestinal bacteria-epithelial interactions using organoids. Cell Mol Gastroenterol Hepatol. 12 (2), 769-782 (2021).
- Li, Y., et al. Next-generation porcine intestinal organoids: an apical-out organoid model for swine enteric virus infection and immune response investigations. J Virol. 94 (21), e01006-e01020 (2020).
- Smith, D., et al. The development of ovine gastric and intestinal organoids for studying ruminant host-pathogen interactions. Front Cell Infect Microbiol. 11, 733811 (2021).
- Nash, T. J., Morris, K. M., Mabbott, N. A., Vervelde, L. Inside-out chicken enteroids with leukocyte component as a model to study host-pathogen interactions. Commun Biol. 4 (1), 377 (2021).
- Hanson, K. I., Weiss, A. A. Intestinal tissue response to Shiga toxin exposure. mBio. 15 (9), e0123224 (2024).
- Hovhannisyan, P., et al. Infection of human organoids supports an intestinal niche for Chlamydia trachomatis. PLoS Pathog. 20 (8), e1012144 (2024).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten