Method Article
Manipolazione spazio-temporale di attività GTPasi Piccolo a livello subcellulare e su scala temporale di secondi nelle cellule viventi
In questo articolo
Riepilogo
Un metodo per spazio-temporale controllo delle attività di piccola GTPasi dalla luce viene descritta. Questo metodo si basa sulla rapamicina indotta FKBP-FRB eterodimerizzazione e foto-gabbie sistemi. Ottimizzazione di irradiazione di luce permette l'attivazione spazio-temporalmente controllato delle piccole GTPasi a livello subcellulare.
Abstract
Regolazione dinamica della famiglia di Rho triphosphatases guanosina piccole (GTPasi) con grande precisione spazio-temporale è essenziale per varie funzioni cellulari ed eventi 1, 2. La loro natura dinamica spatiotemporally è stato rivelato attraverso la visualizzazione della loro attività e localizzazione in 3 tempo reale. Al fine di ottenere una più profonda comprensione del loro ruolo in diverse funzioni cellulari a livello molecolare, il passo successivo dovrebbe essere perturbazione delle attività della proteina in una posizione precisa subcellulare e la tempistica.
Per raggiungere questo obiettivo, abbiamo sviluppato un metodo per indotto dalla luce, spazio-temporalmente l'attivazione controllata di piccole GTPasi combinando due tecniche: (1) rapamicina indotta FKBP-FRB eterodimerizzazione e (2) una foto-ingabbiamento metodo di rapamicina. Con l'uso di rapamicina-mediata FKBP-FRB eterodimerizzazione, abbiamo sviluppato un metodo per l'attivazione rapida inducibile o inattivazione di piccole GTPasi including Rac 4, Cdc42 4, RhoA 4 e Ras 5, in cui rapamicina induce traslocazione di FKBP-fusi GTPasi, o loro attivatori, alla membrana plasmatica, dove è ancorato FRB. Per l'accoppiamento con questo sistema eterodimerizzazione, abbiamo anche sviluppato un sistema di foto-ingabbiamento di analoghi della rapamicina. Un composto foto-gabbia è una piccola molecola la cui attività è soppresso con un gruppo di photocleavable protezione noto come gruppo di ingabbiamento. Per sopprimere l'attività eterodimerizzazione completamente, abbiamo progettato un rapamicina gabbia che è legato ad una macromolecola tale che il complesso risultante grande non può attraversare la membrana plasmatica, portando a alcuna attività di sfondo come dimerizer chimico nelle cellule 6. La figura 1 illustra uno schema di nostra sistema. Con la combinazione di questi due sistemi, a livello locale abbiamo reclutato un attivatore Rac alla membrana plasmatica su una scala temporale di secondi e ha raggiunto indotto dalla luce l'attivazione Rac al subcellulare Level 6.
Protocollo
1. La trasfezione di DNA plasmidico
- Aggiungi DNA plasmide, 0,5 mg di membrana-tethered FRB (di seguito denominato LDR) e 0,5 mcg FKBP-Tiam1 (T-cell linfoma invasione e metastasi che inducono proteina 1: Rac attivatore) codificata con una proteina fluorescente a 37,5 ul dH 2 O. Aggiungere 1 ml Fugene HD.
- Vortex e incubare a temperatura ambiente per 20 minuti. Nel frattempo, procedere ai passaggi 1,3-1,8.
- Lavare ciascun pozzetto di una camera 8-bene con 50 microlitri poli-D-Lisina.
- Lavare il centro di ciascun pozzetto con dH 2 O.
- Tripsinizzare 80-85% confluenti NIH-3T3 e diluire a 10 ml di terreno di coltura (DMEM con 10% FBS).
- Rimuovere 375 microlitri di sospensione e centrifugare a 1750 rpm per 3 minuti.
- Aspirare i media, facendo attenzione a non disturbare il pellet.
- Aggiungere 750 microlitri di FBS DMEM w / 10% e risospendere.
- Aggiungi sospensione cellulare al dH 2 O / DNA / Fugene soluzione HD e mescolare delicatamente.
- Aggiungere 250microlitri di cellule / DNA sospensione a ciascun pozzetto (si riempirà fino a 3 pozzetti).
- Incubare le cellule transfettate a 37 ° C e 5% CO 2 per 15-18 ore.
2. Preparazione del CRB-A Solution
- Sintetizzare in gabbia rapamicina-biotina addotto (CRB). Vedi "Schema sintetico del CRB" per i dettagli. In breve, frazioni di ingabbiamento e biotina vengono elaborati separatamente. La porzione di gabbia è poi coniugata rapamicina (LC lab), successivamente il prodotto accoppiato alla porzione biotina con un mezzo di reazione click 7 ottenendo CRb.
- Preparare una soluzione stock 1 uM del CRB in DMSO.
- Sciogliere 1mg avidina in 250 microlitri di PBS in una provetta Eppendorf da 0,5 ml.
- Aggiungere 1 ml di soluzione madre crb di avidina dissolto. Mescolare agitando parecchie volte.
- Incubare a temperatura ambiente per 15 minuti per ottenere CRB-coniugato avidina (CRB-A) Soluzione.
- Escludi molecole non legate piccole e purificare crb-A utilizzando una dimensione di esclusione della colonna.
3. Microscopia e illuminazione luce a livello subcellulare
- Siero fame cellule trasfettate in DMEM senza FBS per 12 ore.
- Aspirare delicatamente i media al largo delle celle e aggiungere 250 microlitri della preparato 400 nM crb-A soluzione (dal punto 2.6).
- Per visualizzare dinamica della proteina in cellule viventi a risoluzione subcellulare prima e dopo il trattamento con luce, microscopia disco rotante è stata utilizzata (Figura 2a). Illuminazione UV è stato ottenuto per accoppiamento ottica UV LED (365 nm) con epi-fluorescenza luce (Figura 2b).
- MetaMorph software è stato utilizzato per catturare le immagini di fluorescenza di cellule trasfettate ogni 15 secondi.
- Definite le coordinate e la dimensione di un punto di illuminazione utilizzando una lastra di vetro rivestita con coloranti fluorescenti eccitato dalla luce UV. La dimensione puòessere variata applicando lente obiettivo con diversi ingrandimenti e scegliendo fibre ottiche con diverse dimensioni del nucleo.
- Dopo la determinazione di livelli di fondo arruffarsi membrana, irradiare luce UV alla periferia delle cellule. Le seguenti due passaggi sono precedenti per contrastare gli effetti localizzati.
- Globalmente irradiare le cellule con la luce a 365 nm da una lampada di mercurio.
- Sciogliere Uncaged, così rapamicina originale in DMSO e poi in PBS (400 nM finale), e aggiungere la soluzione alla camera di imaging per indurre effetti globali.
4. Risultati rappresentativi
Un esempio dei risultati rappresentativi è mostrato in Figura 3. Irradiazione di luce UV in una piccola regione in prossimità di un bordo cellulare rapidamente indotto la formazione di volant localizzata solo in e vicino alla zona irradiata. Per confermare che questo era effettivamente l'attività locale, quindi a livello globale le cellule irradiate con raggi UV, o aggiunti rapamicina ai media di massa, sia di WHIch indotto formazione globale balza in tutta la membrana. Per un'analisi quantitativa, abbiamo diviso le cellule bersaglio in quattro quadranti, e contava la frequenza della formazione volant in ogni quadrante sul locale così come in seguito a stimolazione globale in condizioni diverse, inclusi i controlli (Figura 4). E 'solo questa combinazione di irradiazione UV, la CRB-A, e adeguate FKBP-FRB costrutti che induce arruffarsi locale.

Figura 1. Schematica di dimerizzazione proteina spazialmente confinata utilizzando gabbia rapamicina-coniugato avidina (CRB-A) e alla luce UV. Irradiazione UV scinde il linker tra rapamicina e biotina, con conseguente rilascio di chimica dimerizer (HE-Rapa, C40-O-(2-idrossietil) rapamicina) e il suo sottoprodotto. Il dimerizer rilasciato diffonde nelle cellule e induce dimerizzazione tra FKBP-POI (proteina di interesse) e plasma membRane ancorata FRB solo in prossimità della regione irradiata (cerchio blu). Senza l'irradiazione UV, FKBP e FRB non associato (croce rossa), producendo quindi alcun effetto. (Copyright @ ACS2011) 6

Figura 2. Immagine della misura microscopio confocale a fluorescenza. (A) vista frontale del microscopio confocale (invertito Axiovert 200, Zeiss). (B) primo piano, vista dall'alto di un dispositivo di accoppiamento per EPI fluorescenza e illuminazione UV.
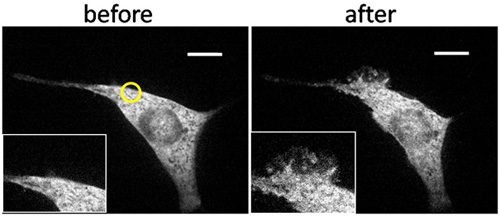
Figura 3. Formazione ruffle spazialmente confinati in una trasfettato NIH3T3 cellulare indotta da radiazioni UV localizzato. Confocale immagini di fluorescenza di un cellulare di fibroblasti NIH3T3 che è trasfettata con YFP-tagged FKBP-Tiam1 (Rac attivatore) e LDR (membrana ancorata FRB) ha subito l'irradiazione in una zona limitata in prossimità del suo bordo con LED luce a 365 nm per 15 secondi a partire da 210 sec in presenza di CRB-A coniugato nei mezzi di bulk. Questo trattamento è stato seguito da illuminazione globale con lampada al mercurio per 1 sec a oltre 1.063 sec e globale della rapamicina finali 400 Nm a 1.560 sec. La cella ha mostrato quasi alcuna attività scompigliava all'inizio (a), ma ha mostrato spazialmente limitata scompigliava dopo l'illuminazione localizzata (cerchio giallo) (b). La cella ha mostrato formazione globale balza dopo l'illuminazione globale e l'aggiunta della rapamicina (cd), indicando che la formazione balza locale è stata causata dall'attivazione di Rac spazialmente limitata. Scale bar: 10 pm. (Copyright @ ACS2011) 6
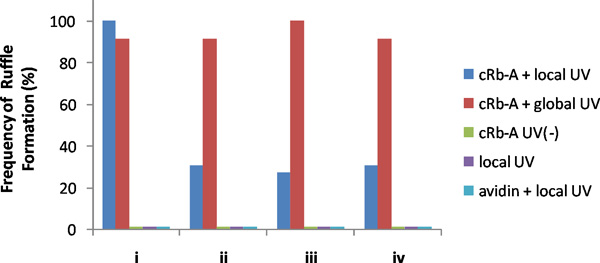
Figura 4. Frequenza di formazione balza in quadranti di trasfettate cellule NIH3T3. Irradiazione UV locale con CRB-A e dopo irradiazione UV globale sono stati eseguiti su cellule 13. CRB-A (no irradiazione UV), irradiazione UV locale con o senza avidina stati attuati su cellule 13, 5 e 6, rispettivamente. Nessuna delle cellule osservate mostrato formazione crespo con CRb-A senza irradiazione UV, o irradiazione UV locale con o senza mezzi di avidina in bulk. Abbiamo raccolto i dati da cellule con la formazione di bassa ruffle sfondo, ma che ha avuto la capacità di rapamicina indotta la formazione di volant, che abbiamo controllato dopo ogni esperimento confermando la formazione globale balza a seguito di aggiunta di rapamicina (finale 400 nm). Questi risultati hanno dimostrato che CRb-A e UV irradiazione locale sono necessari per la formazione ruffle locale nelle cellule. (Copyright @ ACS2011) 6
Abbreviazioni:
FKBP: FK506-binding protein
FRB: FKBP-rapamicina-binding protein
Tiam1: T-cell linfoma invasione e metastasi-proteina che induce 1 (Rac GEF)
LDR: Lyn DSAG-linker-FRB (Lyn: N-terminale di 11 aminoacidi chinasi Lyn)
HE-Rapa: C40-O - (2-idrossietil) rapamicina
CRB: rapamicina in gabbia con la frazione biotina
CRb-A: CRb coniugato avidina
Discussione
Abbiamo descritta una tecnica che utilizza un nuovo composto gabbia insieme FKBP-FRB eterodimerizzazione sistema per manipolare Rho GTPasi attività in una posizione precisa subcellulare su una scala di tempo in secondi.
Ci sono tre limiti dell'approccio attuale. Innanzitutto, poiché il metodo si basa sulla prevenzione o consentire la diffusione del dimerizer attraverso la membrana plasmatica, la posizione bersaglio cellulare deve essere membrana plasmatica o nelle sue vicinanze. Ulteriore ottimizzazione della struttura chimica della CRB può permettere al prodotto di entrare nelle cellule senza indurre dimerizzazione fino irradiazione di luce. Con tale dimerizer, potremmo in teoria manipolare attività di molecole di segnalazione ovunque all'interno delle cellule. In secondo luogo, la diffusione molecolare di dimerizers chimici, FKBP-FRB complessi dimerizzazione o attivati molecole endogene di segnalazione possono influenzare notevolmente la reclusione precisa degli effetti previsti. Questo effetto è particolarmente pronunciatoin momenti successivi, e dipende parametri associati con irradiazione UV (come larghezza del fascio, l'energia, e la modalità fascio (vs continuo pulsato)) ed i coefficienti di diffusione dei composti coinvolti. Ulteriore ottimizzazione empirica di questi parametri consente di reclusione più rigorosa delle attività di segnale. In terzo luogo, una luce UV presenta non banale fototossicità alle cellule. Un modo semplice per migliorare questo problema è di utilizzare una lunghezza d'onda come 405 nm a uncage gruppi ingabbiamento nitrobenzile. Two-photon illuminazione per uncaging consente illuminazione a lunghezza d'onda ancora più nonché al regione estremamente localizzata delle cellule. Recentemente, altri ricercatori hanno segnalato un approccio elegante per attivare Rac in una posizione subcellulare con sensibili alla luce proteine vegetali 8-11. Finora, la sua applicazione è limitata a solo poche proteine. Rispetto alle loro strategie, la nostra tecnica attuale è più facilmente adattabile per espandere il numero di proteine bersaglio, senza ampia e time dispendio di re-engineering. Noi e gli altri hanno già sviluppato una vasta gamma di sonde dimerizzazione che possono manipolare l'attività di diverse molecole di segnalazione a livello della membrana plasmatica 5, 6, 12-14. Eventi cellulari Spatiotemporally dinamici sono ora direttamente verificabile.
Divulgazioni
Non ci sono conflitti di interesse dichiarati.
Riconoscimenti
Questo studio è stato sostenuto da NIH finanziamento della ricerca (DK090868 e GM092930 a TI). C'è un brevetto per un analogo rapamicina in gabbia.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente / apparecchiatura | Azienda | Numero di catalogo | |
| Rapamicina | Tecoland | RAPA99 | |
| Fugene HD | Roche | 04709691001 | |
| Avidina | Sigma | A9275-10MG | |
| DMSO | Sigma | D2650-5X5ML | |
| Size-esclusione colonna | GE Healthcare | Spin Trappola G-25 | |
| Con fondo di vetro da 8 pozzetti camera | Thermo Scientific | 12-565-47 | |
| Inverted Microscopio a fluorescenza | Zeiss | Axiovert200M | |
| UV sorgente luminosa a LED | Rapp Opto Elettronica | UVILED | |
| Confocale disco rotante | Yokogawa Electric Corp. | CSU10 | |
| CCD della fotocamera | Hamamatsu Photonics | ORCA-ER | |
| 100x lente dell'obiettivo | Zeiss | Piano Apochromat |
Schema sintetico del CRB (Copyright ACS2011) 6

Condizioni sintetici: (a) BnBr, K 2 CO 3, DMF (b) f.HNO 3, AcOH (c) TFA (d) Propargyl-Br, K 2 CO 3, DMF, (e) glicerolo, cat. PTSA, toluene (f) NaBH 3 CN, TiCl 4, MeCN (g) Tf 2 O, piridin (h) metanolo HCl, (i) LiAlH 4, THF (j) TsCl, piridina, CH 2 Cl 2 (k) NaN 3, DMF (L) 8, 2,6-di-t-butilpiridina, CH 2 Cl 2 (m) 13, CuSO 4, ascorbato, 2-propanolo, H 2 O, CH 2 Cl 2.
Riferimenti
- Etienne-Manneville, S., Hall, A. Rho GTPases in cell biology. Nature. 420, 629-635 (2002).
- Takai, Y., Sasaki, T., Matozaki, T. Small GTP-binding proteins. Physiol. Rev. 81, 153-208 (2001).
- Kiyokawa, E., Aoki, K., Nakamura, T., Matsuda, M. Spatiotemporal regulation of small GTPases as revealed by probes based on the principle of Forster Resonance Energy Transfer (FRET): Implications for signaling and pharmacology. Annu. Rev. Pharmacol. Toxicol. 51, 337-358 (2011).
- Inoue, T., Heo, W. D., Grimley, J. S., Wandless, T. J., Meyer, T. An inducible translocation strategy to rapidly activate and inhibit small GTPase signaling pathways. Nat. Methods. 2, 415-418 (2005).
- Komatsu, T. Organelle-specific, rapid induction of molecular activities and membrane tethering. Nat. Methods. 7, 206-208 (2010).
- Umeda, N., Ueno, T., Pohlmeyer, C., Nagano, T., Inoue, T. A photocleavable rapamycin conjugate for spatiotemporal control of small GTPase activity. J. Am .Chem. Soc. 133, 12-14 (2011).
- Kolb, H. C., Finn, M. G., Sharpless, K. B. Click Chemistry: Diverse Chemical Function from a Few Good Reactions. Angew. Chem. Int. Ed. Engl. 40, 2004-2021 (2001).
- Kennedy, M. J. Rapid blue-light-mediated induction of protein interactions in living cells. Nat. Methods. 7, 973-975 (2010).
- Levskaya, A., Weiner, O. D., Lim, W. A., Voigt, C. A. Spatiotemporal control of cell signalling using a light-switchable protein interaction. Nature. 461, 997-1001 (2009).
- Wu, Y. I. A genetically encoded photoactivatable Rac controls the motility of living cells. Nature. 461, 104-108 (2009).
- Yazawa, M., Sadaghiani, A. M., Hsueh, B., Dolmetsch, R. E. Induction of protein-protein interactions in live cells using light. Nat. Biotechnol. 27, 941-945 (2009).
- Fegan, A., White, B., Carlson, J. C., Wagner, C. R. Chemically controlled protein assembly: techniques and applications. Chem. Rev. 110, 3315-3336 (2010).
- Suh, B. C., Inoue, T., Meyer, T., Hille, B. Rapid chemically induced changes of PtdIns(4,5)P2 gate KCNQ ion channels. Science. 314, 1454-1457 (2006).
- Varnai, P., Thyagarajan, B., Rohacs, T., Balla, T. Rapidly inducible changes in phosphatidylinositol 4,5-bisphosphate levels influence multiple regulatory functions of the lipid in intact living cells. J. Cell. Biol. 175, 377-382 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon