Method Article
Lentiviral CRISPR/Cas9-Vermittelte Genombearbeitung zur Untersuchung hämatopoetischer Zellen in Krankheitsmodellen
In diesem Artikel
Zusammenfassung
Beschrieben werden Protokolle für die hocheffiziente Genombearbeitung von murinen hämatopoetischen Stamm- und Vorläuferzellen (HSPC) durch das CRISPR/Cas9-System zur schnellen Entwicklung von Mausmodellsystemen mit hämatopoetischen systemspezifischen Genmodifikationen.
Zusammenfassung
Die Manipulation von Genen in hämatopoetischen Stammzellen mit konventionellen Transgeneseansätzen kann zeitaufwändig, teuer und herausfordernd sein. Durch fortschritte In der Genom-Editing-Technologie und lentivirusvermittelten Transgen-Bereitstellungssystemen wird hier eine effiziente und wirtschaftliche Methode beschrieben, die Mäuse etabliert, bei denen Gene speziell in hämatopoetischen Stammzellen manipuliert werden. Lentiviren werden verwendet, um Cas9-extierende Linien-negative Knochenmarkzellen mit einer Guide-RNA (gRNA) zu transduzieren, die auf bestimmte Gene und ein rotes Fluoreszenz-Reporter-Gen (RFP) abzielt, dann werden diese Zellen in tödlich bestrahlte C57BL/6-Mäuse transplantiert. Mäuse, die mit Lentivirus transplantiert werden, die nicht zielgerichtete gRNA exdrücken, werden als Kontrollen verwendet. Die Transplantation von transduzierten hämatopoetischen Stammzellen wird durch zytometrische Durchflussanalyse von RFP-positiven Leukozyten peripheren Blutes bewertet. Mit dieser Methode können 90 % Transduktion von myeloischen Zellen und 70 % der Lymphzellen nach 4 Wochen nach der Transplantation erreicht werden. Genomische DNA wird aus RFP-positiven Blutzellen isoliert, und Teile der Ziel-Site-DNA werden durch PCR verstärkt, um die Genombearbeitung zu validieren. Dieses Protokoll bietet eine hochdurchsatzhohe Auswertung von hämatopoese-regulatorischen Genen und kann auf eine Vielzahl von Mauskrankheitsmodellen mit hämatopoetischer Zellbeteiligung erweitert werden.
Einleitung
Viele Studien in Hämatologie und Immunologie basieren auf der Verfügbarkeit von genetisch veränderten Mäusen, einschließlich konventioneller und bedingter transgener/knock-out-Mäuse, die hämatopoetische systemspezifische Cre-Treiber wie Mx1-Cre, Vav-Cre und andere verwenden. 1,2,3,4,5. Diese Strategien erfordern die Etablierung neuer Mausstämme, die zeitaufwändig und finanziell belastend sein können. Während revolutionäre Fortschritte in der Genom-Editing-Technologie die Erzeugung neuer Mausstämme in nur 3-4 Monaten mit dem entsprechenden technischen Know-how ermöglicht haben6,7,8,9 , viel mehr Zeit ist erforderlich, um die Mauskolonie zu verstärken, bevor Experimente durchgeführt werden. Darüber hinaus sind diese Verfahren kostspielig. Beispielsweise listet Jackson Laboratory den aktuellen Preis für Dieerzeugungsdienste für Knock-out-Mäuse auf 16.845 US-Dollar pro Stamm (Stand Dezember 2018) auf. Daher sind Methoden, die wirtschaftlicher und effizienter sind als herkömmliche murintransgene Ansätze, vorteilhafter.
Die clusterierte, regelmäßig interspacete kurze palindromische Wiederholungen/CRISPR-assoziierte Protein-9-Technologie (CRISPR/Cas9) hat zur Entwicklung neuer Werkzeuge für eine schnelle und effiziente RNA-basierte, sequenzspezifische Genombearbeitung geführt. Ursprünglich als bakterieller adaptiver Immunmechanismus zur Zerstörung der eindringenden Pathogen-DNA entdeckt, wurde das CRISPR/Cas9-System als Werkzeug zur Steigerung der Wirksamkeit der Genombearbeitung in eukaryotischen Zellen und Tiermodellen eingesetzt. Eine Reihe von Ansätzen wurden verwendet, um CRISPR/Cas9-Maschinen in hämatopoetische Stammzellen zu übertragen (d. h. Elektroporation, Nukleofektion, Lipofektion, virale Abgabe und andere).
Hier wird ein Lentivirus-System eingesetzt, um Zellen zu transduzieren, da es in der Lage ist, Cas9-exezierende murine hämatopoetische Stammzellen effektiv zu infizieren und das Führungs-RNA-Expressionskonstrukt, Promotoren, regulatorische Sequenzen und Gene, die kodieren, zusammenzupacken. fluoreszierende Reporterproteine (z. B. GFP, RFP). Mit dieser Methode wurde eine Ex-vivo-Genbearbeitung von hämatopoetischen Stammzellen der Maus erreicht, gefolgt von einer erfolgreichen Rekonstitution des Knochenmarks bei tödlich bestrahlten Mäusen10. Der für diese Studie verwendete Lentivirus-Vektor drückt die Cas9- und GFP-Reportergene aus dem gemeinsamen Kern-EF1a-Promotor mit einer internen ribosomalen Einstiegsstelle vor dem Reportergen aus. Die Rna-Führungssequenz wird von einem separaten U6-Promotor exprimiert. Dieses System wird dann verwendet, um Insertions- und Deletionsmutationen in den Kandidaten-Klonhämatopoese-Treibergen Tet2 und Dnmt3a10zu erzeugen. Die Transduktionseffizienz bei dieser Methode ist jedoch relativ gering (ca. 5%-10%) aufgrund der großen Größe des Vektoreinsatzes (13 Kbp), die die Transduktionseffizienz und die Virentiter während der Produktion reduziert.
In anderen Studien wurde gezeigt, dass eine größere virale RNA-Größe sowohl die Virusproduktion als auch die Transduktionseffizienz negativ beeinflusst. Beispielsweise wird eine Erhöhung der Insert-Größe um 1 kb gemeldet, um die Virusproduktion um 50 % zu verringern, und die Transduktionseffizienz wird bei hämatopoetischen Stammzellen der Maus auf mehr als 50 % sinken11. Daher ist es vorteilhaft, die Größe des viralen Einsatzes so weit wie möglich zu reduzieren, um die Effizienz des Systems zu verbessern.
Dieses Manko kann durch den Einsatz von Transgenmäusen von Cas9 überwunden werden, bei denen das Cas9-Protein entweder konstitutiv oder induzierbar exprimiert wird12. Die konstitutiven CRISPR/Cas9-Knock-in-Mäuse drücken Cas9 Endonuklease und EGFP aus dem CAG-Promotor am Rosa26-Ort auf allgegenwärtige Weise aus. So kann ein Konstrukt mit sgRNA unter der Kontrolle des U6-Promoters und des RFP-Reportergens unter der Kontrolle des EF1a-Kernpromotors mit dem Lentivirus-Vektor geliefert werden, um eine Genombearbeitung zu erreichen. Mit diesem System wurden die Gene hämatopoetischer Stammzellen erfolgreich bearbeitet, was eine Transduktionseffizienz von 90 % zeigt. Somit bietet dieses Protokoll eine schnelle und effektive Methode, um Mäuse zu erstellen, bei denen gezielte Genmutationen in das hämatopoetische System eingeführt werden. Während unser Labor diese Art von Technologie hauptsächlich verwendet, um die Rolle der klonalen Hämatopoese in Herz-Kreislauf-Erkrankungen13,14,15zu untersuchen, ist es auch auf Studien hämatologischer bösartig16. Darüber hinaus kann dieses Protokoll auf die Analyse erweitert werden, wie DNA-Mutationen in HSPC andere Krankheits- oder Entwicklungsprozesse im hämatopoetischen System beeinflussen.
Um ein robustes Lentivirus-Vektorsystem zu etablieren, sind hohe Tikern-Virenbestände und optimierte Bedingungen für die Transduktion und Transplantation hämatopoetischer Zellen erforderlich. Im Protokoll werden Anweisungen zur Herstellung eines Hochtiter-Virusbestands in Abschnitt 1, zur Optimierung der Kulturbedingungen von murinen hämatopoetischen Stammzellen in Abschnitt 2, Methoden zur Knochenmarktransplantation in Abschnitt 3 und zur Bewertung der Engraftment in Abschnitt 4.
Protokoll
Alle Verfahren, die tierische Themen betreffen, wurden vom Institutional Animal Care and Use Committee (IACUC) der University of Virginia genehmigt.
1. Erzeugung und Reinigung von Lentiviruspartikeln
HINWEIS: Lentivirus-Partikel, die die optimierte Guide-RNA enthalten, können durch die detaillierten Protokolle von Addgene erzeugt werden: . Optimierte Methoden für High-Titer Lentivirus Vorbereitung und Lagerung werden an anderer Stelle diskutiert17,18. Kurz gesagt, Lentiviren werden durch Ko-Transfektion eines Lentivirus-Vektorplasmids, psPAX2 und pMD2.G in HEK 293T-Zellen produziert. Kulturüberstand wird bei 48 h nach der Transfektion gesammelt und durch Ultrazentrifugation konzentriert. Lentiviraltiter wird durch einen kommerziell erhältlichen qPCR-basierten Assay bestimmt. Dieses Verfahren sollte in einem Schrank der Biosicherheitsklasse II durchgeführt werden.
- Bereiten Sie eine 1:200 Lösung von Kollagen (0.0005%) in 1x PBS.
- Beschichten Sie eine 6-Well-Platte mit Kollagenlösung und brüten bei 37 °C, 5%CO2 für 30 min.
- Samen 293T-Zellen mit einer Dichte von 1 x 106 Zellen pro Brunnen und inkubieren bei 37 °C, 5%CO2 für 2 h.
- Um die Mischung aus drei Transfektionsplasmiden für einen Brunnen vorzubereiten, kombinieren Sie 0,9 g Lentivirus-Vektor, 0,6 g psPAX2 und 0,3 g pMD2.G, und erreichen Dann ein Gesamtvolumen von 10 l durch Zugabe von deionisiertem Wasser. Passen Sie die Beträge entsprechend an die Anzahl der Brunnen an. Die Menge und das Verhältnis jedes Plasmids müssen möglicherweise weiter optimiert werden, um den Anforderungen der Forscher gerecht zu werden.
- 50 l 1x PBS und 5 l des verdünnten PEI MAX (1,0 mg/ml) vorsichtig in das Plasmidgemisch geben und 15 min bei Raumtemperatur (RT) inkubieren (Tabelle 1).
- 1 ml DMEM in die Mischung geben.
- Aspirieren Sie Medien aus der 6-Well-Platte, fügen Sie 1 ml Plasmid-Mischung hinzu und brüten bei 37 °C, 5%CO2 für 3 h.
- Ersetzen Sie die Medien durch 2 ml frisches DMEM und brüten Bei 37 °C, 5%CO2 für 24 h.
- 1 ml frisches DMEM hinzufügen und bei 37 °C, 5%CO2 für weitere 24 h inkubieren (Gesamtinkubationszeit 48 h).
- Übertragen Sie den Kulturüberstand in ein 50 ml Rohr und zentrifugieren bei 3.000 x g für 15 min, um frei schwebende Zellen zu entfernen.
- Filtern Sie den Überstand durch einen 0,45-m-Filter.
- Das Filtrat in Polypropylenzentrifugenrohre übertragen.
- Ultrazentrifuge bei 4 °C und 72.100 x g bei rmax für 3 h.
- Den Überstand vorsichtig ansaugen und das weiße Pellet zurücklassen.
- Setzen Sie das Pellet mit 100 L serumfreiem hämatopoetischem Zellausdehnungsmedium ohne Belüftung aus.
- Bewahren Sie ein 10 L Aliquot auf, um den viralen Titer zu messen, und lagern Sie alle verbleibenden Aliquots bei -80 °C, bis dies erforderlich ist.
- Titrate das Virus mit einem qPCR-basierten Assay nach den Anweisungen des Herstellers mit dem 10 L viralen Aliquot.
2. Isolierung und Transduktion von Linien-negativen Zellen aus Demmausknochenmark (Abbildung 1A)
HINWEIS: In der Regel werden, um genügend Zellen zu isolieren, Paare von Tibias, Oberschenkelknochen und Humeri von jeder Maus geerntet werden. Becken- und Wirbelsäulenknochen können auch als Quelle von Linien-negativen Zellen geerntet werden.
- Isolierung von Knochenmarkzellen
- Euthanisieren Sie 8-10 Wochen alte männliche CRISPR/Cas9 Knock-in-Mäuse um 5% Isofluran, gefolgt von zervikalen Dislokation, dann desinfizieren sie ihre Haut mit 70% Ethanol.
- Mit sezierender Schere, machen Sie einen Querschnitt in der Haut direkt unter dem Brustkorb und schälen Sie die Haut in beide Richtungen, um die Beine und Arme auszusetzen.
- Trennen Sie vorsichtig die unteren Gliedmaßen vom Hüftknochen, indem Sie das Hüftgelenk verziehen. Schneiden Sie entlang des Oberschenkelknochenkopfes, um den Oberschenkelknochen vollständig aus der Hüfte zu entfernen. Dislozieren Sie das Knie und schneiden Sie am Gelenk, um den Oberschenkelknochen und die Tibia zu trennen, während die Knochenepiphyse intakt bleibt. Dislozieren Sie das Sprunggelenk und schälen Sie den Fuß und zusätzlichen Muskel.
- Mit einer Schere über die Schulter schneiden, um die oberen Gliedmaßen zu lösen. Dislozieren Sie die Schulter, dann am Ellenbogengelenk schneiden, um den Humerusknochen zu ernten.
- Verwenden Sie Zellulose-Faser-Tücher, um Muskeln vorsichtig von den Oberschenkelknochen, Tibias und Humeri zu entfernen. Ergreifen Sie zusätzliche Vorsichtsmaßnahmen, um sicherzustellen, dass die Knochen während dieses Prozesses nicht brechen.
- Legen Sie die isolierten Knochen in ein 50 ml konisches Rohr mit RPMI und legen Sie sie auf Eis.
HINWEIS: Die folgenden Schritte sollten in einem Schrank der Biosicherheitsklasse II durchgeführt werden. - Übertragen Sie die Knochen in eine sterile, 100 mm Kulturschale.
- Greifen Sie den Knochen mit stumpfer Zange, und mit sezierender Schere, sorgfältig schneiden beide Epiphysen.
HINWEIS: Ein unzureichendes Schneiden führt zu einer unvollständigen Bündigung des Knochenmarks, während übermäßig aggressives Schneiden zu Zellverlust führt. - Füllen Sie eine 10 ml Spritze mit eiskaltem RPMI und spülen Sie mit einer 22 G Nadel das Knochenmark aus dem Schaft in eine neue 100-mm-Kulturschale.
HINWEIS: Knochen werden weiß und durchscheinend, wenn die Knochenwelle gut gespült wurde. Wenn nicht, schneiden Sie die Knochenenden erneut und spülen Sie sie erneut. - Nachdem das Knochenmark gesammelt wurde, machen Sie eine einzellige Suspension, indem Sie das Knochenmark mehrmals durch eine 10 ml Spritze mit einer 18 G Nadel passieren. Wiederholen Sie 10x, um eine einzellige Suspension zu gewährleisten.
- Filtern Sie die Zellsuspension durch ein 70-m-Zellsieb in ein 50 ml konisches Rohr.
- Zentrifuge bei 310 x g für 10 min bei 4 °C.
- Saugen Sie den Überstand an und setzen Sie die Zellpellets in einem geeigneten Volumen optimierten Trennpuffers für den folgenden Zelltrennungsprozess wieder aus.
- Isolierung und Lentivirus-Transduktion von Linien-negativen Zellen
HINWEIS: Maus-Linie-negative Zellen werden aus dem Knochenmark von Cas9 transgene Mäuse3 oderanderen Stämmen von Mäusen isoliert, mit einem Lineage-Depletion-Kit nach den Anweisungen des Herstellers. Typischerweise machen Linien-negative Zellen 2%-5% der ganzen Knochenmarkzellen aus, und die Reinheit ist in der Regel größer als 90% nach der Isolierung. Die isolierten Linien-negativen Zellen werden in serumfreiem hämatopoetischem Zellexpansionsmedium kultiviert, ergänzt mit 20 ng/ml rekombinantem murineminem tPO und 50 ng/mL rekombinantem murinen SCF, dann mit dem Lentivirus-Vektor für 16 h bei einer Vielzahl von Infektion (MOI) = 100.- Um Liniennegative Zellen zu isolieren, verwenden Sie das Futterzellen-Erschöpfungskit gemäß den Anweisungen des Herstellers.
- Nach der Isolierung setzen Sie die Linien-negativen Zellen in 1 ml serumfreiem hämatopoetischen Zellausdehnungsmedium wieder aus.
- Säen Sie die Zellen in eine 6-Well-Platte mit einer Dichte von 1,5 x 106 Zellen/ml (5 x 105 Lineage-negative Zellen/Maus).)
- Fügen Sie rekombinante murine TPO und SCF in Brunnen bei Endkonzentrationen von 20 ng/ml bzw. 50 ng/ml hinzu.
- Vorinkubationszellen bei 37 °C in 5%CO2 für 2 h.
- Lentivirus bei MOI = 100, 4 g/ml Polybren und Penicillin/Streptomycin in die Brunnen geben und bei 37 °C, 5%CO2 für 16-20 h(Abbildung 1B)brüten.
- Sammeln Sie am nächsten Tag die lentivirus-transduzierten Zellen in ein 15 ml kegelförmiges Rohr und Zentrifuge bei 300 g für 10 min.
- Sorgsam den Überstand ansaugen und das Pellet in 200 L RPMI pro Maus wieder aufsetzen. Bewahren Sie die Zellen bei RT bis zur Transplantation in Mäuse auf (Abschnitt 3).
3. Transplantation von transduzierten Zellen in tödlich bestrahlte Mäuse
- Legen Sie die Empfängermäuse am Tag der Knochenmarktransplantation in einen Acht-Scheiben-Kuchenkäfig und setzen Sie sie zwei Dosen Ganzkörperbestrahlung aus (550 Rad/Dosis, Gesamtdosis = 1100 Rad), wobei zwischen jeder Bestrahlungssitzung etwa 4 h liegen.
- Nach der zweiten Bestrahlungssitzung projizieren Sie transduzierte Liniennegative Zellen über den retroorbitalen Venenplexus (insgesamt 200 l) mit einer Insulinspritze in jede anästhesierte Empfängermaus (Abbildung 1C).
- Nach der Bestrahlung sollten Mäuse in sterilisierten Käfigen untergebracht und mit einer weichen Ernährung und Trinkwasser versorgt werden, das mit Antibiotika für 14 d ergänzt wird.
- Nach 3-4 Wochen nach der Knochenmarktransplantation analysieren Sie peripheres Blut, um die Transplantation von transducierten Spenderzellen zu überprüfen (Abschnitt 4).
4. Bewertung der Chimäre des peripheren Blutes
- Anästhesisieren Sie Mäuse mit 5% Isofluran und erhalten Sie eine Blutprobe aus einer retro-orbitalen Vene mit Kapillarröhren, und sammeln Sie sie in K2EDTA-Röhren (das Volumen in einem Kapillarrohr ist ausreichend für den folgenden Test).
- 20 L Blut aus den K2EDTA-Röhrchen in die 5 ml runden Polystyrol-Reagenzgläser übertragen und auf Eis legen.
- Fügen Sie 1,5 ml RBC-Lysepuffer hinzu, um rote Blutkörperchen zu lyse. 5 min auf Eis inkubieren.
- Um den Lysepuffer zu neutralisieren, waschen Sie Proben mit FACS-Puffer (1,5 ml/Probe).
- Zentrifuge bei 609 x g beir max für 5 min bei 4 °C. Entsorgen Sie den Überstand.
- Inkubieren Sie die Zellen mit einem Cocktail von monoklonalen Antikörpern (verdünnt in 100 L FACS Puffer/Probe) bei RT für 20 min im Dunkeln. Eine vollständige Liste der Antikörper finden Sie im Abschnitt Materialien oben.
- Waschen Sie die Zellen einmal mit FACS-Puffer (2 ml/Probe). Zentrifuge bei 609 x g beir max (1.800 U/min) für 5 min bei 4 °C. Entsorgen Sie den Überstand vollständig.
- Fixieren Sie die Zellen mit paraformaldehydhaltigem Fixationspuffer (100 l/Tube) für 10 min bei 4 °C.
- Einmal Zellen mit FACS-Puffer (3 ml/Probe) waschen. Zentrifuge bei 609 x g beir max (1.800 U/min) für 5 min bei 4 °C. Entsorgen Sie den Überstand vollständig.
- Setzen Sie das Pellet in 400 L FACS-Puffer aus.
- Halten Sie die Proben bei 4 °C bis zur Analyse durch Durchflusszytometrie.
Ergebnisse
Mit dem oben beschriebenen Protokoll wurden ca. 0,8-1,0 x 108 Knochenmarkzellen pro Maus erhalten. Die Anzahl der Linien-negativen Zellen, die wir erhalten, beträgt ungefähr 3 x 106 Zellen pro Maus. Typischerweise beträgt die Ausbeute von Knochenmark-Linien-negativen Zellen 4%-5% der Gesamtknochenmark-Kernzellen.
Der Chimäre der transduzierten Zellen (RFP-positiv) wird durch die Durchflusszytometrie des peripheren Blutes bewertet (Abbildung 2A,B). Blut wird aus der retroorbitalen Vene isoliert und geeignete Marker werden verwendet, um die Identität jeder hämatopoetischen Zellpopulation zu bestimmen (d. h. Neutrophile, Monozyten, T-Zellen usw.) (Abbildung 3A,B). Genomische DNA kann aus RFP-positiven Blutzellen isoliert werden, und Abschnitte der Ziel-Site-DNA können durch PCR verstärkt und zur Sequenzanalyse in TA-Klonvektoren subkloniert werden. Diese Plasmide werden in E. coli transduziert und die Ziel-Standortsequenzen werden durch Sanger-Sequenzierung bestimmt (Abbildung 4). Alternativ können Zielstandortsequenzen durch andere Methoden bestimmt werden, wie z. B. Sanger-Sequenzierung des gepoolten Genoms, gefolgt von der Verfolgung von Indels durch Zersetzungsanalyse (TIDE)10. Für den Kontrollzustand werden Mäuse in der Regel mit Zellen transplantiert, die mit einem Lentivirus transduziert werden, das nicht zielgerichtete Führungs-RNA exdrückt.
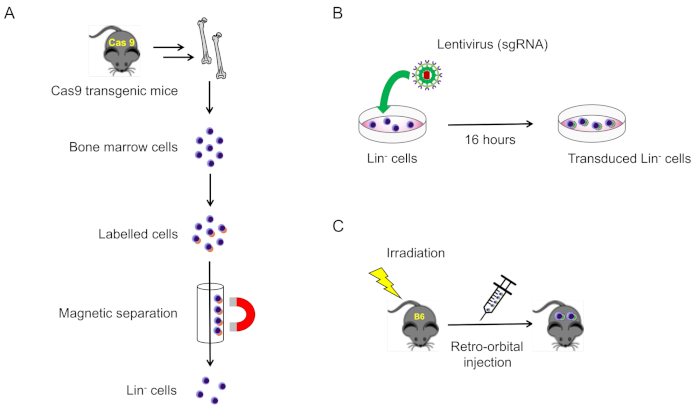
Abbildung 1: Schematische Darstellung dieses Protokolls. (A) Isolierung von liniennegativen Knochenmarkzellen von Cas9-exezierenden Mäusen (Abschnitt 2.1). (B) Lentivirus-Transduktion von Linien-negativen Zellen (Abschnitt 2.2). (C) Retroorbitale Injektion von transduzierten Zellen in tödlich bestrahlte Wildtypmäuse (Abschnitt 3). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
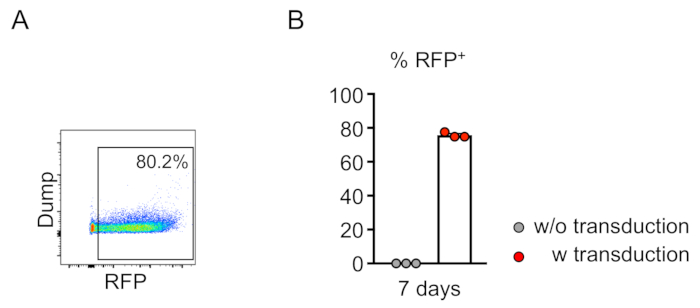
Abbildung 2:Effiziente lentivirale Transduktion von Mausknochenmarklinien-negativen Zellen in vitro. (A) Die Analyse der Durchflusszytometrie zeigt eine erfolgreiche Transduktion von Linien-negativen Zellen. Die Analyse wurde nach 7 Tagen In-vitro-Kultur durchgeführt. (B) Im Durchschnitt wurden 75,7 % der Zellen in diesem Test transduziert (n = 3). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
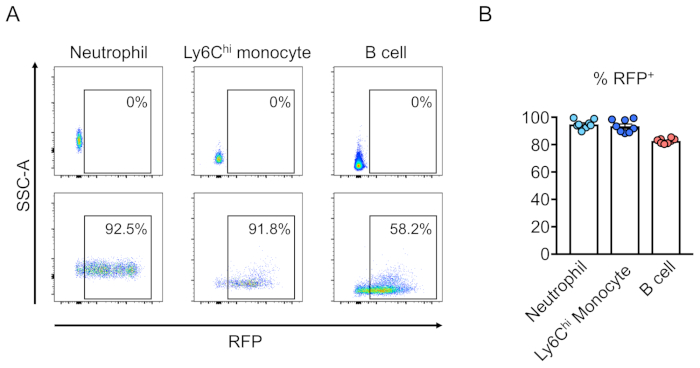
Abbildung 3:Rekonstitution von tödlich bestrahltem Mausknochenmark durch transduzierte Liniennegative Zellen. (A) Durchflusszytometrieanalyse von mausperipherem Blut nach Rekonstitution durch hämatopoetische Stammzellen, die (unten) oder nicht (oben) mit Lentivirus exzessizierendem RFP transduziert wurden. Neutrophile sind definiert als Ly6G+ und Ly6Chi monocytes als Ly6G- und Ly6C+, und B-Zellen als CD45R+. (B) Bei diesen Assays sind durchschnittlich 94,8 %, 93,5 % und 82,7 % der Zellen RFP+ in den Neutrophilen, Ly6C-Chi-Monozyten- bzw. B-Zellpopulationen (n = 8). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
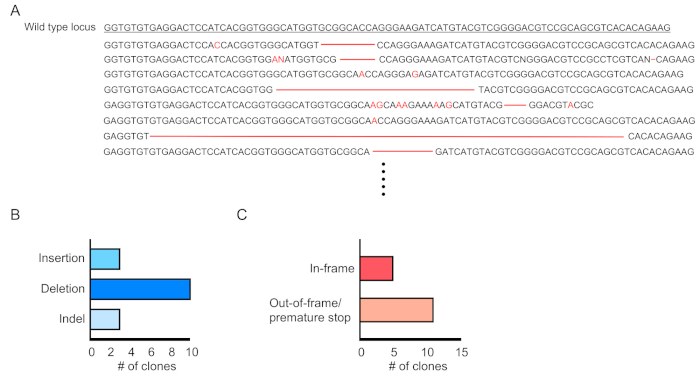
Abbildung 4:Bewertungder Genbearbeitung in transduzierten Blutkörperchen. (A) Beispiel für die Genbearbeitung, die Sequenzierungsergebnisse mutierter Dnmt3a-Lokus in RPF-positiven Blutzellen zeigt. Löschungen werden als rote Striche und Einfügungen mit roten Buchstaben bezeichnet. (B) Zusammenfassung der festgestellten Mutationen. (C) 69% (11/16 Klone) zeigten Out-of-Frame/vorzeitige Stop-Mutationen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Plasmid | Größe (bp) | Betrag pro Brunnen (g) | verhältnis |
| pLKO5.0 | 7700 | 0.9 | 2 |
| psPAX2 | 10668 | 0.6 | 1 |
| pMD2.G | 5822 | 0.3 | 1 |
| PEI-max (Lager: 100 mg/ml) | 5 l/well | ||
Tabelle 1: Für die Transfektion verwendete Plasmid- und PEI-max-Mengen.
Diskussion
Der Vorteil dieses Protokolls ist die Schaffung von Tiermodellen, die spezifische Mutationen in hämatopoetischen Zellen in einer schnellen und äußerst kostengünstigen Weise im Vergleich zu herkömmlichen transgenen Ansätzen der Maus beherbergen. Es wurde festgestellt, dass diese Methode die Erzeugung von Mäusen mit hämatopoetischen Zellgenmanipulationen innerhalb eines Monats ermöglicht. Es gibt mehrere wichtige Schritte in diesem Protokoll, die einer weiteren Prüfung bedürfen.
Screening der gRNA-Sequenz
Es wird empfohlen, gRNAs in vitro zu testen, um die Bearbeitungseffizienz zu bewerten, bevor in vivo-Experimente durchgeführt werden. Die Effizienz von gRNAs wird mit einem zellfreien In-vitro-Transkriptions- und Screening-System getestet. Die transkribierte gRNA wird validiert, indem sie ihre Effizienz bei der Verkleben der Vorlagen-DNA in Gegenwart von rekombinantem Cas9-Protein mit Agarose-Gel-Elektrophorese misst. Zu diesem Zweck sind handelsübliche Kits erhältlich.
Hier sind Indelmutationen durch das TA-Klonen von PCR-Produkten gekennzeichnet, die aus der bearbeiteten Region verstärkt werden, bakterielle Zellen mit diesen Plasmiden transformieren und einzelne Kolonien für die Sanger-Sequenzierung aufnehmen. Diese Methode ist jedoch mühsam und zeitaufwändig. Alternativ kann eine Sequenzierung der nächsten Generation (NGS) oder eine gepoolte DNA-Sequenzierung gefolgt von einer TIDE-Analyse durchgeführt werden19. Der TIDE-Algorithmus wurde erstellt, um Sanger-Sequenzspuren zu analysieren, die aus komplexen Samples generiert wurden. Es hat sich gezeigt, dass Indel-Schätzungen mit TIDE in der Regel mit denen von NGS20nicht zielgerichtet sind. Die Analysesoftware ist online unter verfügbar.
Generierung von High-Titer-Lentivirus-Partikeln
Das virale vesikuläre Stomatitis-Virus G-Protein, das für eine Zellinfektion unerlässlich ist, ist hoch pH-empfindlich. Daher ist es wichtig, das Kulturmedium innerhalb eines akzeptablen pH-Bereichs zu halten, und es sollte kein gelbliches Aussehen entwickeln. Kollagenbeschichtete Gerichte für die Virusgenerierung werden eingesetzt, weil sie die Befestigung von HEK293T-Zellen beschleunigt und die Durchführung der Transfektion innerhalb weniger Stunden ermöglicht, anstatt über Nacht zu warten. Je nach Versuchsplan kann jedoch auch eine inkubation über Nacht in Betracht gezogen werden.
Reinigung von Lentiviruspartikeln
Um eine effiziente Transduktion hämatopoetischer Stammzellen zu erreichen, ist es notwendig, High-Titer-Lentivirus zu erzeugen. Die Optimierung der Zentrifugationsgeschwindigkeit ist ein Schlüsselmerkmal. Während die Konzentration des Lentivirus in der Regel bei 90.000 x gdurchgeführt wird, haben mehrere Berichte gezeigt, dass die Viruswiederherstellung zunimmt, wenn das Material mit der niedrigeren Geschwindigkeit von 20.000 x g18zentrifugiert wird. Die Produktion von High-Titer-Lentivirus-Präparat ohne Ultrazentrifugation wurde ebenfalls vorgeschlagen17. Es sollte beachtet werden, dass es wichtig ist, das Viruszentrifugationspellet auszusetzen und gleichzeitig eine kräftige Pipettion zu vermeiden, um die Belüftung zu minimieren und die Virusintegrität aufrechtzuerhalten. Für eine effiziente Transduktion hämatopoetischer Stammzellen werden Hochtiter-Lentivirus-Partikel benötigt11. Pilotversuche ergaben, dass ein MOI von 100 optimal in Bezug auf Dieduktionseffizienz und Zelllebensfähigkeit ist. Es wird empfohlen, die Lentivirusbestände auf der Grundlage der Zelllebensfähigkeit und der Transduktionseffizienz zu bewerten.
Lagerung von Lentivirus-Partikeln
Der Lentivirus-Titer ist hochgradig temperaturempfindlich, und der Titer kann durch unangemessene Lagerbedingungen und wiederholte Frost-Tau-Zyklen drastisch reduziert werden. Es wurde festgestellt, dass die Transduktionseffizienz des Lentivirus bei 4 °C [t(1/2) = 1,3 Tage] oder mehreren Frost-Tau-Zyklen [t(1/2) = 1,1 Runden] rapide abnimmt. Es wird empfohlen, die Viruspräparate in flüssigem Stickstoff oder zerkleinertem Trockeneis kurz nach dem Suspendierten des Viruspellets zu einfrieren. Die Virusbestände sollten bei -80 °C gehalten und auf Eis auf RT kurz vor dem Ausgleich aufgetaut werden und11verwenden.
Es sollten mehrere potenzielle Einschränkungen beachtet werden. Erstens wird die Einführung von Off-Target-Indelmutationen durch CRISPR/Cas9 seit langem geschätzt. Es wurde auch gezeigt, dass CRISPR/Cas9 Off-Target-Mutationen in vivo21induzieren kann. In der Praxis können Off-Target-Indelmutationen vermieden werden, indem gRNA-Sequenzen verwendet werden, die eng mit Denkgenom-Standorten übereinstimmen und mehr als vier Inkongruenzen zu vorhergesagten sekundären Standorten aufweisen. Ein solches Design kann mit vorhandenen silico Werkzeugen22durchgeführt werden. Andere Rechenwerkzeuge zur Vorhersage von gRNA mit minimierten Off-Target-Aktionen sind verfügbar ( oder ). Es kann auch von Vorteil sein, ein Tiermodell mit zwei oder mehr verschiedenen gRNAs zu analysieren, um den Phänotyp zu bestätigen und die Möglichkeit zu minimieren, dass der beobachtete Phänotyp durch einen Off-Target-Effekt einer bestimmten gRNA vermittelt wird.
Zusätzlich zu den herkömmlichen Indel-Mutationen, die von CRISPR/Cas9 erzeugt wurden, wurden größere Deletionen berichtet, die über Kilobasen hinausgehen. Dies kann Studien verwirren; jedoch, diese größeren Löschungen werden berichtet, dass viel niedrigere Häufigkeit im Vergleich zu Indels23. Ein weiteres potenzielles Problem ist die genetische Kompensation. Es wurde berichtet, dass mutierte RNA mit einem vorzeitigen Beendigungscodon (PTC) zur Upregulation verwandter Gene mit Sequenzähnlichkeit durch COMPASS-Komplex-vermittelte Aktivierung der Transkription24,25führen kann. Dieses Ereignis wurde als ein Mechanismus vorgeschlagen, der zu phänotypischen Unterschieden zwischen Knock-out- und Knockdown-Ansätzen der Genablation führen kann. Da die CRISPR/Cas9-vermittelte Genombearbeitung stark auf der stochastischen Einführung von Frame-Shift-Mutationen beruht, die zur Erzeugung von PTC führen, kann die genetische Kompensation den Phänotyp verändern. Um eine genetische Kompensation zu vermeiden, können Experimente in Betracht gezogen werden, bei denen die regulatorischen Sequenzen eines Gens von CRISPR/Cas9 oder durch die Einführung epigenetischer Modifikatoren mit Cas9 als RNA-geführte DNA-Erkennungsplattform ins Visier genommen werden.
Schließlich sollte anerkannt werden, dass Hämatopoese aus Zellen, die in tödlich bestrahlte Mäuse eingraviert sind, von den nativen Bedingungen der Hämatopoese abweichen können. Darüber hinaus kann die Bestrahlung systemische Auswirkungen auf den Organismus haben, die die Interpretation von Experimenten verwirren können, die die Folgen von Genmutationen in hämatopoetischen Zellen untersuchen.
Forscher haben katalytisch inaktive Cas9 -Proteine (dCas9) als "RNA-geführte DNA-Erkennungsplattform" genutzt und dCas9-Fusionsproteine verwendet, um Effektordomänen zu bestimmten DNA-Sequenzen zu lokalisieren, um entweder zu repressen (CRISPRi) oder (CRISPRa) zu aktivieren. Transkription off-target Gene26,27. Während dieses Protokoll katalytisch aktive Transgenmäuse von Cas9 verwendet, um dsDNA-Spaltung in die genomische DNA-Sequenz einzuführen, ist die epigenetische Modifikation zur Repress- oder Aktivierung bestimmter Gene anwendbar, indem dCas9 mit Chromatin-Modifikator-Domänen wie dCas9-KRAB bzw. dCas9-VP64. Alternativ kann dCas9 als Transkriptionsrepressor als sein eigener verwendet werden, indem Transkriptionsmaschinen für den Zugriff auf die Gen-Site blockiert werden27. In jüngerer Zeit haben Zhou et al. transgene DCas9-SunTag-p65-HSF1 (SPH) transgene Mäuse etabliert, die eine modifizierte Version eines mit dCas9 verschmolzenen epigenetischen Aktivators ausdrücken und gezeigt haben, dass dieses CRISPRa-System in vivo28funktionsfähig ist.
Unser Labor nutzt diese Technologie vorwiegend, um die Rolle der klonalen Hämatopoese in Herz-Kreislauf-Erkrankungen zu untersuchen. Im wuchernden Gewebe können somatische Mutationen in Krebstreibergenen einen zellulären Wachstumsvorteil verschaffen und zu abnormen klonalen Expansionen führen. Im hämatopoetischen System wird dieser Prozess als "klonale Hämatopoese" bezeichnet und führt zu Situationen, in denen ein wesentlicher Teil der Leukozyten eines Individuums durch mutierte Klone ersetzt wird. Es gibt eine wachsende Wertschätzung, dass abnorme klonale Expansionen Herz-Kreislauf-Erkrankungen wie Arteriosklerose und Herzinsuffizienz beschleunigen und zur Morbidität und Gesamtmortalität beitragen15,29.
Kürzlich wurde ein kausaler Zusammenhang zwischen mehreren dieser somatischen Mutationen und Herz-Kreislauf-Erkrankungen dokumentiert, und Aspekte der zugrunde liegenden Mechanismen wurden10,13,14erläutert. Diese somatischen Mutationen stellen jedoch wahrscheinlich die "Spitze des Eisbergs" dar, da epidemiologische Studien gezeigt haben, dass viele zusätzliche Kandidatengene mit klonaler Hämatopoese und potenziell erhöhter Kardiovaskulär-Krankheitssterblichkeit in Verbindung gebracht werden. Daher ist eine systematische, höhere Durchsatzauswertung der klonalen Hämatopoese-Treibergene erforderlich. Aktuelle Studien über den kausalen Zusammenhang von klonaler Hämatopoese und Herz-Kreislauf-Erkrankungen basieren auf der Analyse von Mäusen mit hämatopoetischen systemspezifischen bedingten transgenen (Mx1-Cre, Vav-Cre, etc.) oder Mäusen nach Knochenmarktransplantation. Diese Strategien müssen jedoch neue Mauskolonien errichten und können zu einer finanziellen und physischen Belastung für Forscher werden. Daher ist eine billigere und schnellere Methode als der in der Vergangenheit angewandte konventionelle murintransgene/Knock-out-Ansatz gerechtfertigt. Lentivirale Vektoren zur Transduce von HSPC- und CRISPR-Technologien zur Entwicklung von Mutationen, wie in diesem Manuskript beschrieben, erleichtern die Untersuchung von klonaler Hämatopoese und Herz-Kreislauf-Erkrankungen.
Neben der Erzeugung konventioneller Knock-out-Lokus ist diese Methode auf die Produktion von abgeschnittenen mutierten Proteinen anwendbar. Zum Beispiel haben Forscher erfolgreich eine hämatopoetische-Ppm1d-Abschneidung erzeugt, die häufig bei Patienten mit klonaler Hämatopoese beobachtet wird, indem sie Frameshift-Mutationen mit einer gRNA-Targeting exon 6 des Ppm1d-Gens30einführten.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
S. S. wurde von einem Postdoktorandenstipendium der American Heart Association 17POST33670076 unterstützt. K. W. wurde durch die NIH-Zuschüsse R01 HL138014, R01 HL141256 und R01 HL139819 unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| RPMI Medium 1640 (1X) | Gibco | 11875-093 | Medium |
| Sulfamethoxazole and Trimethoprim injection | TEVA | 0703-9526-01 | |
| 1/2 cc LO-DOSE INSULIN SYRINGE | EXELINT | 26028 | general supply |
| 293T cells | ATCC | CRL-3216-- | Cell line |
| APC-anti-mouse Ly6C (Clone AL-21) | BD Biosciences | 560599 | Antibodies |
| APC-Cy7-anti-mouse CD45R (RA3-6B2) | BD Biosciences | 552094 | Antibodies |
| BD Luer-Lok disposable syringes, 10 ml | BD | 309604 | general supply |
| BD Microtainer blood collection tubes, K2EDTA added | BD Bioscience | 365974 | general supply |
| BD Precisionglide needle, 18 G | BD | 305195 | general supply |
| BD Precisionglide needle, 22 G | BD | 305155 | general supply |
| BV510-anti-mouse CD8a (Clone 53-6.7) | Biolegend | 100752 | Antibodies |
| BV711-anti-mouse CD3e (Clone 145-2C11) | Biolegend | 100349 | Antibodies |
| Collagen from calf skin | Sigma-Aldrich | 9007-34-5 | general supply |
| Corning Costar Ultra-Low Attachment Multiple Well Plate, 6 well | Millipore Sigma | CLS3471 | general supply |
| CRISPR/Cas9 knock-in mice | The Jackson Laboratory | 028555 | mouse |
| DietGel 76A | Clear H2O | 70-01-5022 | general supply |
| Dulbecco’s Modified Eagle’s Medium (DMEM) - high glucose | Sigma Aldrich | D6429 | Medium |
| eBioscience 1X RBC Lysis Buffer | Thermo fisher Scientific | 00-4333-57 | Solution |
| Falcon 100 mm TC-Treated Cell Culture Dish | Life Sciences | 353003 | general supply |
| Falcon 5 mL round bottom polystyrene test tube | Life Sciences | 352054 | general supply |
| Falcon 50 mL Conical Centrifuge Tubes | Fisher Scientific | 352098 | general supply |
| Falcon 6 Well Clear Flat Bottom TC-Treated Multiwell Cell Culture Plate | Life Science | 353046 | general supply |
| Fisherbrand microhematocrit capillary tubes | Thermo Fisher Scientific | 22-362566 | general supply |
| Fisherbrand sterile cell strainers, 70 μm | Fisher Scientific | 22363548 | general supply |
| FITC-anti-mouse CD4 (Clone RM4-5) | Invitrogen | 11-0042-85 | Antibodies |
| Fixation Buffer | BD Bioscience | 554655 | Solution |
| Guide-it Compete sgRNA Screening Systems | Clontech | 632636 | Kit |
| Isothesia (Isoflurane) solution | Henry Schein | 29404 | Solution |
| Lenti-X qRT-PCR Titration Kit | Takara | 631235 | Kit |
| Lineage Cell Depletion Kit, mouse | Miltenyi Biotec | 130-090-858 | Kit |
| Millex-HV Syringe Filter Unit, 0.45 mm | Millipore Sigma | SLHV004SL | general supply |
| PBS pH7.4 (1X) | Gibco | 10010023 | Solution |
| PE-Cy7-anti-mouse CD115 (Clone AFS98) | eBioscience | 25-1152-82 | Antibodies |
| PEI MAX | Polysciences | 24765-1 | Solution |
| Penicillin-Streptomycin Mixture | Lonza | 17-602F | Solution |
| PerCP-Cy5.5-anti-mouse Ly6G (Clone 1A8) | BD Biosciences | 560602 | Antibodies |
| pLKO5.sgRNA.EFS.tRFP | Addgene | 57823 | Plasmid |
| pMG2D | Addgene | 12259 | Plasmid |
| Polybrene Infection/Transfection Reagent | Sigma Aldrich | TR-1003-G | Solution |
| Polypropylene Centrifuge Tubes | BECKMAN COULTER | 326823 | general supply |
| psPAX2 | Addgene | 12260 | Plasmid |
| RadDisk – Rodent Irradiator Disk | Braintree Scientific | IRD-P M | general supply |
| Recombinant Murine SCF | Peprotech | 250-03 | Solution |
| Recombinant Murine TPO | Peprotech | 315-14 | Solution |
| StemSpan SFEM | STEMCELL Technologies | 09600 | Solution |
| TOPO TA cloning kit for sequencing with One Shot TOP10 Chemically Competent E.coli | Thermo fisher Scientific | K457501 | Kit |
| Zombie Aqua Fixable Viability Kit | BioLegend | 423102 | Solution |
Referenzen
- Sasaki, Y., et al. Defective immune responses in mice lacking LUBAC-mediated linear ubiquitination in B cells. EMBO Journal. 32, 2463-2476 (2013).
- Ogilvy, S., et al. Transcriptional regulation of vav, a gene expressed throughout the hematopoietic compartment. Blood. 91, 419-430 (1998).
- Meyer, S. E., et al. DNMT3A Haploinsufficiency Transforms FLT3ITD Myeloproliferative Disease into a Rapid, Spontaneous, and Fully Penetrant Acute Myeloid Leukemia. Cancer Discovery. 6, 501-515 (2016).
- Kuhn, R., Schwenk, F., Aguet, M., Rajewsky, K. Inducible gene targeting in mice. Science. 269, 1427-1429 (1995).
- Cole, C. B., et al. PML-RARA requires DNA methyltransferase 3A to initiate acute promyelocytic leukemia. Journal of Clinical Investigation. 126, 85-98 (2016).
- Yang, H., et al. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell. 154, 1370-1379 (2013).
- Wang, H., et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153, 910-918 (2013).
- Quadros, R. M., et al. Easi-CRISPR: a robust method for one-step generation of mice carrying conditional and insertion alleles using long ssDNA donors and CRISPR ribonucleoproteins. Genome Biology. 18, 92 (2017).
- Miura, H., Quadros, R. M., Gurumurthy, C. B., Ohtsuka, M. Easi-CRISPR for creating knock-in and conditional knock-out mouse models using long ssDNA donors. Nature Protocols. 13, 195-215 (2018).
- Sano, S., et al. CRISPR-Mediated Gene Editing to Assess the Roles of Tet2 and Dnmt3a in Clonal Hematopoiesis and Cardiovascular Disease. Circulation Research. 123, 335-341 (2018).
- Cante-Barrett, K., et al. Lentiviral gene transfer into human and murine hematopoietic stem cells: size matters. BMC Research Notes. 9 (312), (2016).
- Chu, V. T., et al. Efficient CRISPR-mediated mutagenesis in primary immune cells using CrispRGold and a C57BL/6 Cas9 transgenic mouse line. Proceedings of the National Academy of Sciences of the United States of America. 113, 12514-12519 (2016).
- Fuster, J. J., et al. Clonal hematopoiesis associated with TET2 deficiency accelerates atherosclerosis development in mice. Science. 355, 842-847 (2017).
- Sano, S., et al. Tet2-Mediated Clonal Hematopoiesis Accelerates Heart Failure Through a Mechanism Involving the IL-1beta/NLRP3 Inflammasome. Journal of the American College of Cardiology. 71, 875-886 (2018).
- Sano, S., Wang, Y., Walsh, K. Clonal Hematopoiesis and Its Impact on Cardiovascular Disease. Circulatory Journal. 71, 875-886 (2018).
- Heckl, D., et al. Generation of mouse models of myeloid malignancy with combinatorial genetic lesions using CRISPR-Cas9 genome editing. Nature Biotechnology. 32, 941-946 (2014).
- Jiang, W., et al. An optimized method for high-titer lentivirus preparations without ultracentrifugation. Scientific Reports. 5, 13875 (2015).
- Cribbs, A. P., Kennedy, A., Gregory, B., Brennan, F. M. Simplified production and concentration of lentiviral vectors to achieve high transduction in primary human T cells. BMC Biotechnology. 13, 98 (2013).
- Brinkman, E. K., Chen, T., Amendola, M., van Steensel, B. Easy quantitative assessment of genome editing by sequence trace decomposition. Nucleic Acids Research. 42, e168 (2014).
- Sentmanat, M. F., Peters, S. T., Florian, C. P., Connelly, J. P., Pruett-Miller, S. M. A Survey of Validation Strategies for CRISPR-Cas9 Editing. Scientific Reports. 8, 888 (2018).
- Akcakaya, P., et al. In vivo CRISPR editing with no detectable genome-wide off-target mutations. Nature. 561, 416-419 (2018).
- Bae, S., Park, J., Kim, J. S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics. 30, 1473-1475 (2014).
- Kosicki, M., Tomberg, K., Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nature Biotechnology. 36, 765-771 (2018).
- El-Brolosy, M. A., et al. Genetic compensation triggered by mutant mRNA degradation. Nature. 568, 193-197 (2019).
- Ma, Z., et al. PTC-bearing mRNA elicits a genetic compensation response via Upf3a and COMPASS components. Nature. 568, 259-263 (2019).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154, 442-451 (2013).
- Qi, L. S., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152, 1173-1183 (2013).
- Zhou, H., et al. In vivo simultaneous transcriptional activation of multiple genes in the brain using CRISPR-dCas9-activator transgenic mice. Nature Neuroscience. 21, 440-446 (2018).
- Fuster, J. J., Walsh, K. Somatic Mutations and Clonal Hematopoiesis: Unexpected Potential New Drivers of Age-Related Cardiovascular Disease. Circulatory Research. 122, 523-532 (2018).
- Kahn, J. D., et al. PPM1D truncating mutations confer resistance to chemotherapy and sensitivity to PPM1D inhibition in hematopoietic cells. Blood. , (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten