Method Article
2D-HELS MS Seq: Farklı Nükleotid Modifikasyonlarına Sahip RNA Karışımlarının Doğrudan ve de novo Dizilimi için Genel LC-MS Tabanlı Bir Yöntem
Bu Makalede
Özet
Burada, bir cDNA ara ürünü olmadan kısa RNA'yı (çalışma başına <35 nt) dizilemek için doğrudan bir yöntem olarak ve tek bir çalışmada tek baz hassasiyetinde farklı nükleotid modifikasyonlarını dizilemek için genel bir yöntem olarak kullanılabilen LC-MS tabanlı bir dizileme yöntemi için ayrıntılı bir protokol açıklıyoruz.
Özet
Kütle spektrometresi (MS) tabanlı dizileme yaklaşımlarının, tamamlayıcı bir DNA (cDNA) ara ürününe ihtiyaç duymadan doğrudan dizileme RNA'sında yararlı olduğu gösterilmiştir. Bununla birlikte, bu tür yaklaşımlar nadiren bir de novo RNA dizileme yöntemi olarak uygulanır, ancak esas olarak saflaştırılmış tek sarmallı RNA örneklerinin bilinen dizilerini doğrulamak için kalite güvencesine yardımcı olabilecek bir araç olarak kullanılır. Son zamanlarda, 2 boyutlu kütle tutma süresi hidrofobik uç etiketleme stratejisini MS tabanlı dizilemeye (2D-HELS MS Seq) entegre ederek doğrudan bir RNA dizileme yöntemi geliştirdik. Bu yöntem, tek RNA dizilerinin yanı sıra 12 adede kadar farklı RNA dizisi içeren karışımları doğru bir şekilde dizileme yeteneğine sahiptir. Dört kanonik ribonükleotidi (A, C, G ve U) ek olarak, yöntem, modifiye edilmiş nükleotidler içeren RNA oligonükleotidlerini dizileme kapasitesine sahiptir. Bu mümkündür, çünkü modifiye edilmiş nükleobaz ya tanımlanmasına ve RNA dizisindeki konumuna yardımcı olabilecek özünde benzersiz bir kütleye sahiptir ya da benzersiz bir kütleye sahip bir ürüne dönüştürülebilir. Bu çalışmada, tek bir RNA oligonükleotidinin yanı sıra her biri farklı bir diziye ve/veya modifiye nükleotidlere sahip bir RNA oligonükleotid karışımının de novo dizilimi için yöntemin uygulanmasını göstermek için iki temsili modifiye nükleotid (psödouridin (Ψ) ve 5-metilsitozin (m5C)) içeren RNA'yı kullandık. Bu model RNA'ları sıralamak için burada açıklanan prosedürler ve protokoller, standart bir yüksek çözünürlüklü LC-MS sistemi kullanıldığında diğer kısa RNA örneklerine (<35 nt) uygulanabilir olacaktır ve ayrıca modifiye edilmiş terapötik RNA oligonükleotidlerinin dizi doğrulaması için de kullanılabilir. Gelecekte, daha sağlam algoritmaların geliştirilmesi ve daha iyi aletlerle, bu yöntem daha karmaşık biyolojik örneklerin dizilenmesine izin verebilir.
Giriş
RNA'nın doğrudan dizilenmesi için yukarıdan aşağıya MS ve tandem MS 1,2,3,4 dahil olmak üzere kütle spektrometresi (MS) tabanlı dizileme yöntemleri geliştirilmiştir. Bununla birlikte, kütle spektrometrelerinde yüksek kaliteli RNA merdivenlerini etkili bir şekilde oluşturmak için yerinde parçalanma teknikleri şu anda de novo dizileme 5,6'ya uygulanamamaktadır. Ayrıca, tek bir saflaştırılmış RNA dizisinin bile de novo dizilimi için geleneksel tek boyutlu (1D) MS verilerini analiz etmek çok önemsiz değildir ve karışık RNA örneklerinin MS dizilimi için daha da zor olacaktır 7,8. Bu nedenle, iki boyutlu (2D) sıvı kromatografisi (LC)-MS tabanlı bir RNA dizileme yöntemi geliştirilmiştir ve 1D kütle merdivenlerinin yerini almak için 2D kütle tutma süresi (tR) merdivenlerinin üretimini içerir ve RNA'ların de novo dizilimi için gerekli merdiven bileşenlerini tanımlamayı çok daha kolay hale getirir8. Bununla birlikte, 2D LC-MS tabanlı RNA dizileme yöntemi, yalnızca tek bir merdivene dayalı tam bir diziyi okuyamadığından, ancak bir arada var olan iki bitişik merdivene (5' ve 3' merdiven) dayanması gerektiğinden, esas olarak saflaştırılmış sentetik kısa RNA ile sınırlıdır8. Daha spesifik olarak, bu yaklaşım, düşük kütleli bölge8'deki terminal nükleobazları okumak için çift yönlü çift uçlu okumalar gerektirir. Eşleştirilmiş uç okumanın eklenen karmaşıklığı, bu yöntemin RNA karışımlarının dizilimi için savunulamaz olmasına neden olur, çünkü bilinmeyen numuneler için hangi merdiven parçasının hangi merdivene ait olduğu konusunda kafa karışıklığı ortaya çıkar.
MS tabanlı RNA dizileme yaklaşımlarında yukarıda belirtilen engellerin üstesinden gelmek ve doğrudan RNA dizilemede bu tür uygulamaları genişletmek için iki konunun ele alınması gerekir: 1) bir RNA zincirindeki ilk nükleotidden sonuncusuna kadar tam bir diziyi okumak için kullanılabilecek yüksek kaliteli bir kütle merdiveninin nasıl oluşturulacağı ve 2) karmaşık bir MS veri setindeki her bir RNA/kütle merdiveninin nasıl etkili bir şekilde tanımlanacağı. İyi kontrol edilen asit bozunması ile birlikte, MS tabanlı dizileme tekniğine bir hidrofobik uç etiketleme stratejisi (HELS) ekleyerek yeni bir dizileme yöntemi geliştirdik vedizilenecek RNA'ların 5' ve/veya 3' ucuna hidrofobik bir etiket ekleyerek bu iki sorunu başarıyla ele aldık 9. Bu yöntem, RNA'dan "ideal" bir dizi merdiveni oluşturur - her merdiven parçası, yalnızca her bir fosfodiester bağındaki bölgeye özgü RNA bölünmesinden türetilir ve iki bitişik merdiven parçası arasındaki kütle farkı, bu konumdaki nükleotidin veya nükleotid modifikasyonunun tam kütlesidir 8,9,10. Bu mümkündür, çünkü RNA'yı cihaza enjekte edilmeden önce molekül başına ortalama bir kez parçalayan yüksek kontrollü bir asidik hidroliz adımı dahil ediyoruz. Sonuç olarak, her bir bozunma parçası ürünü kütle spektrometresinde tespit edilir ve tüm parçalar birlikte bir sıralama merdiveni 8,9,10 oluşturur. Bu yeni strateji, bir RNA zincirinin tek bir merdiveninden bir RNA dizisinin, RNA'nın diğer merdiveninden eşleştirilmiş uç okuması olmadan tam olarak okunmasını sağlar ve ayrıca kombinatoryal nükleotid modifikasyonları içeren birden fazla farklı iplikçikli RNA karışımlarının MS dizilenmesine izin verir9. RNA'nın 5' ve/veya 3' ucuna bir etiket ekleyerek, etiketli merdiven parçaları, iki kütle merdivenini birbirinden ve ayrıca gürültülü düşük kütleli bölgeden ayırt etmeye yardımcı olabilecek önemli bir tR gecikmesi gösterir. Hidrofobik etiketin eklenmesinden kaynaklanan kütle-tR kayması, kütle merdiveni tanımlamasını kolaylaştırır ve dizi oluşturma için veri analizini basitleştirir. Ayrıca, hidrofobik etiketin eklenmesi, etiketin neden olduğu kütle ve hidrofobiklik artışı nedeniyle karşılık gelen merdiven parçasının gürültülü düşük kütleli tR bölgesinde olmasını önleyerek iplikçikteki terminal tabanın tanımlanmasına yardımcı olabilir, böylece tek bir merdivenden bir RNA'nın tam dizisinin tanımlanmasına izin verir; Eşleştirilmiş uç okumaları gerekli değildir. Sonuç olarak, daha önce herhangi bir gelişmiş dizileme algoritması9 kullanmadan 12 adede kadar RNA farklı zincirinden oluşan karmaşık bir karışımın başarılı bir şekilde dizilenmesini gösterdik, bu da hem kanonik hem de modifiye nükleotidler içeren RNA'nın de novo MS dizilimi için kapıyı açar ve karışık ve daha karmaşık RNA örneklerinin dizilenmesi için daha uygun hale getirir. Aslında, 2D-HELS MS Seq kullanarak, karışık bir tRNA örneğipopülasyonunu 10 başarıyla diziledik ve uygulamasını diğer karmaşık RNA örneklerine aktif olarak genişletiyoruz.
2D-HELS MS Seq'in daha geniş bir RNA örneği yelpazesini doğrudan dizilemesini kolaylaştırmak için, burada bu dizileme yaklaşımının teknik yönlerine odaklanacağız ve tekniği RNA örneklerinin doğrudan dizilenmesine uygularken gereken tüm temel adımları kapsayacağız. Sentetik tek RNA dizileri, birden fazla farklı RNA dizisinin karışımları ve psödouridin (ψ) ve 5-metilsitozin (m5C) gibi hem kanonik hem de modifiye nükleotidleri içeren modifiye RNA'lar dahil olmak üzere dizileme tekniğini göstermek için spesifik örnekler kullanılacaktır. RNA'ların tümü fosfodiester bağları içerdiğinden, her tür RNA, optimum koşullar altında 2D-HELS MS Seq için ideal bir dizi merdiveni oluşturmak üzere asitle hidrolize edilebilir 8,9. Bununla birlikte, belirli bir RNA'nın tüm merdiven parçalarının tespiti alete bağlıdır. Standart bir yüksek çözünürlüklü LC-MS'de (40K), saflaştırılmış bir kısa RNA örneğinin (<35 nt) dizilenmesi için minimum yükleme miktarı, çalışma başına 100 pmol'dür. Bununla birlikte, ek deneylerin yapılması gerektiğinde (örneğin, aynı kütleleri paylaşan izomerik baz modifikasyonlarını ayırt etmek için) daha fazla materyal gereklidir (RNA örneği başına 400 pmol'e kadar). Model sentetik modifiye RNA'ların dizilenmesinde kullanılan protokol, bilinmeyen baz modifikasyonlarına sahip biyolojik RNA örnekleri de dahil olmak üzere daha geniş RNA örneklerinin dizilenmesi için de geçerli olacaktır. Bununla birlikte, standart bir LC-MS cihazı kullanılarak tRNA'yı dizilemek için 1000 pmol (~ 76 nt) gibi daha da büyük bir numune miktarı, tüm modifikasyonlarla birlikte tam tRNA'yı dizilemek için gereklidir ve de novo dizileme10 için gelişmiş bir algoritma geliştirilmelidir.
Protokol
1. RNA oligonükleotidlerini tasarlayın
- Hem kanonik hem de modifiye nükleotidlere sahip bir (RNA #6) dahil olmak üzere farklı uzunluklarda (19 nt, 20 nt ve 21 nt) sentetik RNA oligonükleotidleri tasarlayın. ψ, kütle değiştirmeyen modifikasyonlar için bir model olarak kullanılır, bu da MS dizilemesi için zordur çünkü U. m5C, yaklaşımın sağlamlığını göstermek için kütle değiştiren modifikasyonlar için bir model olarak seçilmiştir.
RNA #1: 5'-HO-CGCAUCUGACUGACCAAAA-OH-3'
RNA #2: 5'-HO-AUAGCCCAGUCAGUCUACGC-OH-3'
RNA #3: 5'-HO-AAACCGUUACCAUUACUGAG-OH-3'
RNA #4: 5'-HO-GCGUACAUCUUCCCCUUUAU-OH-3'
RNA #5: 5'-HO-GCGGAUUUAGCUCAGUUGGGA-OH-3'
RNA #6: 5'-HO-AAACCGUm:5CUGAG-OH-3'
- 100 mM'lik bir RNA stok çözeltisi elde etmek için her bir sentetik RNA'yı nükleaz içermeyen dietil pirokarbonat (DEPC) ile muamele edilmiş suda (aksi belirtilmedikçe DEPC ile muamele edilmiş H2Oolarak ifade edilir) çözün. Stok çözeltileri -20 °C'de uzun süreli olarak saklanır.
- Olası RNA numunesi bozulmasını önlemek için, DEPC ile arıtılmış su, mikrosantrifüj tüpleri ve pipet uçları dahil olmak üzere RNaz içermeyen deneysel malzemeler kullanın. RNase eliminasyon mendilleri kullanarak laboratuvar malzemelerinin yüzeylerini sık sık silin.
2. RNA'ların 3' ucunu biyotin ile etiketleyin
- İki aşamalı reaksiyon protokolü (adenilasyon ve ligasyon)
- 50 mM sodyum asetat, pH 6.0, 10 mM MgCl2, 5 mM diklorodifeniltrikloroetan (DTT), 0.1 mM etilendiamintetraasetik asit (EDTA), 1 μL 1 mM ATP, 1 μL 100 μM biyotinillenmiş sitidin bifosfat (pCp-biotin), 1 μL 50 μM Mth RNA ligaz ve 6 μL DEPC ile muamele edilmiş H2O (toplam hacim 10 μL) içeren 1 μL 10x adenilasyon reaksiyonu tamponunu RNaz içermeyen ince duvarlı 0.2 mL PCR tüpüne ekleyin.
NOT: İki aşamalı reaksiyondan önce reaktifleri -20 °C'de saklayın. Reaktifleri oda sıcaklığında çözün ve reaksiyona eklemeden önce girdap ve santrifüjleme ile iyice karıştırın. - Reaksiyonu bir PCR makinesinde 65 °C'de 1 saat inkübe edin ve reaksiyonu 85 °C'de 5 dakika inaktive edin.
- Ligasyon adımını, 50 mM tris (hidroksimetil) aminometan (Tris) -HCl, pH 7.8, 10 mM MgCl2, 1 mM DTT, 1.5 μL içeren 10 μL 10x T4 RNA ligaz reaksiyon tamponu ekleyerek önceki adımdan 10 μL reaksiyon çözeltisi içeren RNaz içermeyen, ince duvarlı 0.2 mL PCR tüpünde gerçekleştirin. %10 (h/v), 1 μL T4 RNA ligaz (10 birim/μL) ve 11.5 μL DEPC ile muamele edilmiş H2O'ya (toplam 30 mL hacim) ulaşmak için 3 μL susuz dimetil sülfoksit (DMSO). Reaksiyonu gece boyunca bir PCR makinesinde 16 °C'de inkübe edin.
NOT: DMSO'nun yüksek donma noktası (18.45 °C) nedeniyle reaksiyon bileşenlerini oda sıcaklığında birleştirin. - Reaksiyonu gece boyunca 16 °C'de inkübe edin.
- Oligo Clean & Concentrator (Zymo Research, Irvine, CA, ABD) kullanarak enzimleri ve serbest pCp-biyotini uzaklaştırmak için kolon saflaştırma ile reaksiyonu söndürün ve saflaştırın. Kit içerisinde Oligo Bağlayıcı Tampon, DNA Yıkama Tamponu, spin kolonları ve toplama tüpleri bulunmaktadır. Bağlayıcı Tamponu eklemeden önce 50 mL'lik bir numune hacmine ulaşmak için reaksiyon çözeltisine 20 mL DEPC ile muamele edilmiş H2O ekleyin.
- Her reaksiyon çözeltisine 100 mL bağlayıcı tampon ekleyin. 400 μL etanol ekleyin, pipetleyerek karıştırın ve karışımı kolona aktarın. 30 saniye boyunca 10.000 x g'da santrifüjleyin. Akışı atın.
- Kolona 750 μL DNA Yıkama Tamponu ekleyin. Sırasıyla 10.000 x g'da ve maksimum hızda 30 s ve 1 dakika santrifüjleyin.
- Kolonu 1.5 mL'lik bir mikrosantrifüj tüpüne aktarın. Kolona 15 μL DEPC ile muamele edilmiş H2O ekleyin ve RNA ürününü elüte etmek için 30 saniye boyunca 10.000 x g'da santrifüjleyin.
NOT: Numuneler bu aşamada bir sonraki adım gerçekleştirilene kadar -20 °C'de saklanabilir.
- 50 mM sodyum asetat, pH 6.0, 10 mM MgCl2, 5 mM diklorodifeniltrikloroetan (DTT), 0.1 mM etilendiamintetraasetik asit (EDTA), 1 μL 1 mM ATP, 1 μL 100 μM biyotinillenmiş sitidin bifosfat (pCp-biotin), 1 μL 50 μM Mth RNA ligaz ve 6 μL DEPC ile muamele edilmiş H2O (toplam hacim 10 μL) içeren 1 μL 10x adenilasyon reaksiyonu tamponunu RNaz içermeyen ince duvarlı 0.2 mL PCR tüpüne ekleyin.
- Tek adımlı reaksiyon protokolü
- 2 μL 150 μM adenosin-5'-5'-difosfat-{5'-(sitidin-2'-O-metil-3'-fosfat-TEG}C-biyotin (AppCp-biotin), 3 μL 10x ligaz reaksiyon tamponu, dizilenecek RNA'nın 100 mM'lik numune stoğunun 1.5 μL'si, %10'a (h/h) ulaşmak için 3 μL susuz DMSO, 1 μL T4 RNA ligaz (10 birim/μL), ve 1.5 mL RNaz içermeyen bir mikrosantrifüj tüpünde 19.5 μL DEPC ile muamele edilmiş H2O (toplam 30 mL hacim için).
- Reaksiyonu gece boyunca bir PCR makinesinde 16 °C'de inkübe edin.
- Yukarıda 2.1.5-2.1.8 adımlarında açıklandığı gibi sütun saflaştırması gerçekleştirin.
NOT: Her RNA örneği için ayrı/özel bir reaksiyon tüpü hazırlayın (150 pmol RNA ölçeği). RNA'ların 5' ucunun sülfo-Siyanin3 (Cy3) veya Cy3 ile etiketlenmesi gerekebilir (örneğin, çift yönlü sıralama doğrulaması için). Yöntem, 3'-biyotinilasyondan farklıdır ve önceki bir yayın9'da açıklanmıştır.
3. Streptavidin boncukları üzerinde biyotinile RNA örneği yakalayın
- 1,5 mL RNaz içermeyen bir mikrosantrifüj tüpüne 200 μL 1x S&B tampon (5 mM Tris-HCl, pH 7.5, 0.5 mM EDTA, 1 M NaCl) ekleyerek 200 μL streptavidin C1 mıknatıs boncuklarını aktive edin. Bu çözeltiyi girdaplayın ve 2 dakika boyunca bir mıknatıs standına yerleştirin. Ardından, çözeltiyi dikkatlice pipetleyerek süpernatanı atın.
- Boncukları iki kez 200 μL Çözelti A (DEPC ile muamele edilmiş 0.1 M NaOH ve DEPC ile muamele edilmiş 0.05 M NaCl) ve 200 μL Çözelti B'de bir kez (DEPC ile muamele edilmiş 0.1 M NaCl) yıkayın. Her yıkama adımı için, çözeltiyi girdaplayın ve 2 dakika boyunca bir mıknatıs standına yerleştirin, ardından süpernatantı atın. Daha sonra 100 μL 2x S&B tampon (10 mM Tris-HCl, pH 7.5, 1 mM EDTA, 2 M NaCl) ekleyin.
- Hacim 100 μL olana kadar biyotinile edilmiş RNA örneğine 1x S&B tamponu ekleyin. Daha sonra bu çözeltiyi 100 μL 2x S&B tamponda saklanan yıkanmış boncuklara ekleyin. 100 rpm'de sallanan bir platform çalkalayıcı üzerinde oda sıcaklığında 30 dakika inkübe edin. Tüpü 2 dakika boyunca bir mıknatıs standına yerleştirin ve süpernatanı atın.
- Kaplanmış boncukları 1x S&B tamponda 3 kez yıkayın ve hedef RNA moleküllerinin boncuklar üzerinde kaldığını doğrulamak için geri kazanım analizi için Nanodrop ile her yıkama adımında nihai süpernatant konsantrasyonunu ölçün.
- Boncukları 10 mM EDTA, pH 8.2'de% 95 formamid ile 65 ° C'de bir PCR makinesinde 5 dakika inkübe edin. Tüpü mıknatıs standında 2 dakika tutun ve süpernatanı (streptavidin boncuklarından salınan biyotinile RNA'ları içeren) pipetle toplayın.
NOT: Asit bozunmasından önceki bu fiziksel ayırma adımı, yalnızca Şekil 1c'deki RNA # 1'in dizilimi için kullanılır ve hidrofobik biyotin etiketi, 3' etiketli merdiven parçalarının LC-MS ölçümü sırasında önemli ölçüde gecikmeli bir tR'ye sahip olmasına neden olabileceğinden, etiketli 3' merdiven parçalarını 2B kütle-tR grafiğindeki etiketsiz 5' merdiven parçalarından açıkça ayırt edebilir.
4. Dizileme için MS merdivenleri oluşturmak için RNA'nın asit hidrolizi
- Her RNA örneğini üç eşit alikota bölün. Örneğin, hacmi 15 μL olan bir RNA örneğini 5 μL'lik üç alikota bölün.
- Reaksiyon karışımında %50 (h/h) formik asit elde etmek için eşit hacimde formik asit ekleyin 8,9.
- Reaksiyonu bir PCR makinesinde 40 ° C'de, bir reaksiyon sırasıyla 2 dakika, biri 5 dakika ve diğeri 15 dakika boyunca çalışacak şekilde inkübe edin.
- Her reaksiyon bittikten sonra numuneyi kuru buz üzerinde hemen dondurarak asit bozunmasını söndürün.
- Numuneyi kurutmak için bir santrifüj vakum yoğunlaştırıcı kullanın. Numune tipik olarak 30 dakika içinde tamamen kurutulur ve kurutma işlemi sırasında formik asit H 2O ilebirlikte çıkarılır çünkü formik asit H2O'nunkine (100 ° C) benzer bir kaynama noktasına (100.8 ° C) sahiptir.
- LC-MS ölçümü için 20 μL DEPC ile muamele edilmiş H2O'da toplam üç kurutulmuş numuneyi askıya alın ve birleştirin.
NOT: Numuneler bu aşamada LC-MS ölçümü beklenirken -20 °C'de saklanabilir.
5. ψ CMC-ψ eklentisine dönüştürün
- 0.0141 g N-sikloheksil-Nʹ- (2-morfolinoetil)-karbodiimid meto-p-toluensülfonat (CMC) ve 0.07 g üre içeren 1.5 mL RNaz içermeyen bir mikrosantrifüj tüpüne 80 μL DEPC ile muamele edilmiş H2O ekleyin. Dizilenecek RNA'nın 100 μM'lik numune stoğunun 10 μL'sini, 8 μL'sini 1 M bikain tamponunu (pH 8.3) ve 1.28 μL'sini 0.5 M EDTA'yı ekleyin. Toplam 160 μL hacme ulaşmak için DEPC ile muamele edilmiş H2O ekleyin. Nihai konsantrasyonlar 50 mM bicine (pH 8.3) içinde 0.17 M CMC, 7 M üre ve 4 mM EDTA'dır11.
NOT: Bu protokol, tek bir sentetik RNA dizisine veya RNA karışımlarına uygulanabilir. - 160 μL reaksiyon çözeltisini RNaz içermeyen, ince duvarlı 0.2 mL PCR tüplerinde dört eşit alikota bölün ve bir PCR makinesinde 20 dakika boyunca 37 ° C'de inkübe edin.
NOT: Tüp başına 50 μL, bir PCR makinesinde kullanılabilecek maksimum reaksiyon hacmidir. - Her reaksiyonu 10 μL 1.5 M sodyum asetat ve 0.5 mM EDTA (pH 5.6) ile söndürün.
- Adım 2.1.5-2.1.8'de açıklanan prosedüre göre aşırı reaktanları uzaklaştırmak için dört paralel döndürme sütunu ile kolon saflaştırması gerçekleştirin. Saflaştırılmış ürünü, her 1.5 mL RNaz içermeyen mikrosantrifüj tüpünde 15 μL DEPC ile muamele edilmiş H2O içinde çözün.
- Saflaştırılmış ürünü dört RNaz içermeyen, ince duvarlı 0.2 mL PCR tüpüne aktarın. Her 15 μL saflaştırılmış ürüne 20 μL 0.1 MNa2CO3tamponu (pH 10.4) ekleyin ve her reaksiyon tüpü için 40 μL'lik bir nihai hacim elde etmek için DEPC ile muamele edilmiş H2O ekleyin (toplam dört tüp). Reaksiyonu bir PCR makinesinde 37 ° C'de 2 saat inkübe edin.
- Adım 2.1.5'te açıklandığı gibi dört paralel spin sütunu ile kolon saflaştırarak reaksiyonu söndürün ve saflaştırın. CMC-ψ dönüştürülmüş ürünü, her biri 15 μL DEPC ile muamele edilmiş H2O içeren 1,5 mL RNaz içermeyen bir mikrosantrifüj tüpüne boşaltın.
- Saflaştırılmış CMC-ψ dönüştürülmüş numuneyi dört toplama tüpünden tek bir tüpte birleştirin. Dizileme için MS merdivenleri oluşturmak için adım 4.1-4.6'da açıklanan prosedürlere göre formik asit bozunmasını %50 (h/h) gerçekleştirin.
6. LC-MS ölçümü
- LC-MS ölçümü için mobil fazları hazırlayın. Mobil faz A, LC-MS dereceli suda 10 mM diizopropilamin ile 25 mM heksafloro-2-propanoldür; mobil faz B metanoldür.
- Numuneyi analiz için LC-MS numune şişesine aktarın. Her numune enjeksiyon hacmi, 100-400 pmol RNA içeren 20 μL'dir.
- Aşağıdaki LC koşullarını kullanın: 35 °C kolon sıcaklığı, 0,3 mL/dk akış hızı; 15 dakika boyunca %2-20 mobil faz B'den doğrusal bir gradyan ve ardından %90 mobil faz B ile 2 dakikalık bir yıkama adımı.
NOT: Bölüm 2'de belirtildiği gibi Cy3 ve sülfo-Cy3 gibi daha hidrofobik uç etiketler için, numune elüsyonu için daha yüksek bir organik çözücü yüzdesi gerekli olabilir (yani, benzer bir gradyan kullanılabilir, ancak mobil faz B'nin artan bir yüzde aralığı ile). Örneğin, %90 mobil faz B ile 2 dakikalık bir yıkama adımı ile 30 dakika boyunca %2-38 mobil faz B. - Bir otomatik numune alma cihazı ve bir MS HPLC (Yüksek Performanslı Sıvı Kromatografisi) sistemi ile donatılmış bir LC sistemine bağlı bir Agilent Q-TOF (Dört Kutuplu Uçuş Süresi) kütle spektrometresinde numuneleri ayırın ve analiz edin. LC kolonu, partikül boyutu 1,7 μm olan 50 mm x 2,1 mm C18 kolonudur. Aşağıdaki MS ayarlarını kullanın: negatif iyon modu; aralık, 350 m/z ila 3200 m/z; tarama hızı, 2 spektrum/sn; kurutma gazı akışı, 17 L/dak; kurutma gazı sıcaklığı, 250 °C; nebulizatör basıncı, 30 psig; kılcal gerilim, 3500 V; ve parçalayıcı voltajı, 365 V. Lütfen bu parametrelerin kullanılan kütle spektrometresinin tipine veya modeline özgü olduğunu unutmayın.
- Agilent MassHunter edinme yazılımı ile veri elde edin. Kütle, alıkonma süresi, hacim (ilgili iyon türleri için MFE bolluğu) ve kalite puanı vb. dahil olmak üzere bileşik bilgilerini çıkarmak için Agilent moleküler özellik ekstraksiyonu (MFE) iş akışını kullanın. Aşağıdaki MFE ayarlarını kullanın: "centroid veri formatı, küçük moleküller (kromatografik), yüksekliği 100≥ olan tepe, maksimum 1000'e kadar, kalite puanı ≥ 50".
NOT: MFE ayarlarını, en ≥ 50 kalite puanlarıyla maksimum 1000'e kadar mümkün olduğunca çok sayıda potansiyel bileşik elde edecek şekilde optimize edin.
7. Hesaplamalı bir algoritma ile RNA dizisi oluşturmayı otomatikleştirin
NOT: Bu prosedür yalnızca Şekil 1c'de RNA #1 için gösterilmiştir.
- MFE ekstrakte edilen bileşikleri azalan hacim (tepe yoğunluğu) ve tR sırasına göre sıralayın. 1) biyotin tarafından etiketlenen RNA fragmanlarını seçmek için tR'yi 4 ila 10 dakika arasında ayarlayarak veri ön seçimini gerçekleştirin, çünkü biyotin etiketli kütle merdiveni bileşenlerinin tR'leribu tR penceresine kaydırılır (4 dakika ila 10 dakika) ve 2) hacme dayalı veri miktarını azaltmak için algoritma hesaplaması için merdiven parçalarının sayısından daha yüksek bir büyüklük sırası daha yüksek bir girdi bileşiği kullanarak. Örneğin, 20 nt'lik bir RNA için, 20 nt'lik RNA'nın dizilimi için 20 etiketli kütle-tR merdiven bileşeni gerekecektir ve bu nedenle, MFE veri dosyasından 200 bileşik hacme dayalı olarak seçilecektir. Farklı tipte veya model bir kütle spektrometresi kullanıldığında tR penceresinin farklı olabileceğini lütfen unutmayın.
- Yayınlanmış bir algoritmanın gözden geçirilmiş bir sürümünü kullanarak RNA #1'in veri işlemesini ve dizi üretimini gerçekleştirin8. Gözden geçirilmiş algoritmanın kaynak kodları daha önce açıklanmıştır (https://academic-oup-com.remotexs.ntu.edu.sg/nar/article/47/20/e125/5558343#supplementary-data)9.
- Algoritmayı kullanarak dizi oluşturmayı otomatikleştirmenin yanı sıra, temel arama için iki bitişik merdiven bileşeni arasındaki kütle farklarını manuel olarak hesaplayın. RNA'daki tüm bazlar manuel olarak çağrılabilir ve RNA nükleotid ve modifikasyon veri tabanındaki teorik olanlarla eşleştirilebilir8; bu nedenle, RNA zincirinin tam dizisi manuel olarak doğru bir şekilde okunabilir, bu da algoritma tarafından bildirilen dizi okumasının doğruluğunu doğrulamak için kullanılır. RNA modifikasyon veritabanlarında12 daha fazla RNA modifikasyon yapısı bulunabilir ve bunlara karşılık gelen teorik kütleler ChemBioDraw ile elde edilir. Tablo S1–S2'de, gözlemlenen kütle belirli bir merdiven bileşeni için teorik kütlesi ile karşılaştırıldığında ppm (milyonda parça) kütle farkı gösterilir ve 10 ppm'den küçük bir değer, her bir temel çağrı için iyi bir eşleşme olarak kabul edilir.
8. Dizileme RNA karışımları
- Beş RNA zincirinin (RNA # 1 ila # 5) bir karışımını 3' uçlarında A (5 ') pp (5 ') Cp-TEG-biyotin ile adım 2.2'de açıklanan tek adımlı bir protokol kullanarak etiketleyin. Toplam 150 μL reaksiyon çözeltisi hacminde, 15 μL 10x T4 RNA ligaz reaksiyon tamponu, her RNA zincirinden 1.5 μL (toplam 7.5 μL hacim için sırasıyla 100 μM RNA # 1 ila # 5 stoğu), 10 μL 150 μM A (5 ') pp (5 ') Cp-TEG-biotin, 15 μL susuz DMSO, 5 μL T4 RNA ligaz (10 birim / μL) ve 97.5 μL DEPC ile muamele edilmiş H2O. Reaksiyon çözeltisini beş alikota eşit olarak dağıtın. Her RNaz içermeyen mikrosantrifüj tüpü 30 μL reaksiyon çözeltisi içerir.
- Reaksiyonu gece boyunca bir PCR makinesinde 16 °C'de inkübe edin.
- Beş paralel döndürme sütunu ile 2.1.5-2.1.8 adımlarında açıklanan prosedüre göre sütun saflaştırmasını gerçekleştirin. 3'-biyotinile edilmiş 5 RNA ipliğinden (RNA #1 ila #5 karışımı) oluşan bir karışım örneğini, her biri 15 μL DEPC ile muamele edilmiş H2O içeren 1.5 mL RNaz içermeyen bir mikrosantrifüj tüpüne boşaltın.
- Beş toplama tüpünden saflaştırılmış karışım örneklerini tek bir tüpte birleştirin. Formik asit bozunmasını Bölüm 4'te açıklanan prosedüre göre gerçekleştirin.
- Numuneleri Bölüm 6'da açıklandığı gibi LC-MS ile ölçün ve adım 6.5'te açıklandığı gibi kütle, tR ve hacim içeren verileri çıkarmak için optimize edilmiş MFE ayarlarına sahip veri analiz yazılımını kullanarak verileri analiz edin. Tipik işleme ve temel çağırma algoritması, karışımdan kaynaklanan önemli ölçüde artan veri karmaşıklığı nedeniyle uygulanmaz. Karışık numunenin RNA'sındaki tüm bazlar, Bölüm 7.3'e benzer bir yöntemle manuel olarak çağrılır ve RNA nükleotidi ve modifikasyon veritabanı8'deki teorik olanlarla iyi bir şekilde eşleşir, böylece karışık numunedeki beş RNA zincirinin tümünün tam dizileri doğru bir şekilde okunur. Tablo S7-S11'de, gözlenen kütle, tR, hacim, kalite puanı ve ppm kütle farkı dahil olmak üzere tüm bilgiler listelenmiştir.
Sonuçlar
Kolayca tanımlanabilen kütle-tR merdivenleri üretmek için RNA'nın 3' ucuna bir biyotin etiketi eklemek. 2D-HELS MS Seq yaklaşımının iş akışı Şekil 1a'da gösterilmiştir. RNA'nın 3' ucuna eklenen hidrofobik biyotin etiketi (bakınız Bölüm 2), 3' etiketli merdiven bileşenlerinin kütlelerini ve tR'lerini, etiketlenmemiş muadillerine kıyasla arttırır. Böylece, 3' merdiven eğrisi, 2B kütle-tR grafiğinde daha büyük y ekseni değerlerine (tRs'deki artış nedeniyle) kaydırılır ve daha büyük x ekseni değerlerine (kütlelerdeki artış nedeniyle) kaydırılır. Şekil 1b, 2D-HELS MS Seq için RNA'nın 3'-ucuna bir biyotin etiketinin eklenmesi de dahil olmak üzere numune hazırlama protokolünü göstermektedir. Şekil 1c, RNA #1'in 3'-biyotin etiketli kütle-tR merdiven fragmanlarının tR'lerindekisistematik değişikliklere dayalı olarak bir 2D kütle-tR grafiği üzerinde 3'-merdivenin 5'-merdivenden ve diğer istenmeyen parçalardan ayrılmasını göstermektedir. 3' merdiven eğrisi tek başına tam bir RNA #1 dizisi verir ve tR kayması göstermeyen 5' merdiven eğrisi ters diziyi sağlar, ancak terminal tabanı8'i okumak için son eşleştirme gerektirir. Bu 2D-HELS stratejisiyle, daha önce bildirildiği gibi son eşleştirme gerekli değildir ve tüm RNA dizisi yalnızca bir etiketli merdiven eğrisinden8 tamamen okunabilir. Bu nedenle, birden fazla RNA içeren karışık numuneleri, örneğin farklı uzunluklarda iki RNA ipliği (sırasıyla RNA #1 ve RNA #2, 19 nt ve 20 nt) ve her RNA'da 5'-biyotin etiketi ile dizilemek mümkündür (Şekil 1d).
ψ 2D-HELS MS Seq için CMC-ψ eklentisine dönüştürme. ψ, üridin (U) ile aynı kütleye sahip olduğu için MS tabanlı dizileme için zor bir nükleotid modifikasyonudur. Bu iki bazı birbirinden ayırt etmek için, RNA'yı bir ψ CMC-ψ eklentisine dönüştüren CMC ile tedavi ediyoruz (bakınız Bölüm 5). Eklenti, sizden farklı bir kütleye sahiptir ve 2D-HELS MS Seq'de ayırt edilebilir. Şekil 2a, ψ RNA #6'daki CMC eklentisine dönüştüren reaksiyonun ham ürününün HPLC profilini göstermektedir. UV tepe noktalarını entegre ederek, dönüşüm yüzdesini hesapladık ve Bölüm 5'te gösterilen işlemden sonra ψ %42'si CMC-ψ eklentisine dönüştürülür. Asit bozunması ve LC-MS ölçümünden sonra, algoritma tarafından işlenen verilerden 8,9 tanımlanan hem CMC'ye dönüştürülmemiş merdivenlere hem de CMC'ye dönüştürülmüş merdivenlere dayalı olarak diziyi manuel olarak elde ettik. RNA # 6'da (Şekil 2b) 8. pozisyondan başlayarak ψ'dan başlayarak gri eğriden kırmızı bir eğri dallanır, çünkü ψ CMC-ψ eklentisine kısmi dönüşümü nedeniyle. CMC'nin kütlesi ve hidrofobikliği nedeniyle, bu dönüşüm, dönüştürülmemiş muadili ile karşılaştırıldığında, her bir CMC-ψ eklenti içeren merdiven bileşeni için kütlede 252.2076 Dalton'luk bir artışa ve tR'de önemli bir artışa neden olur. Bu nedenle, RNA #6'da 8. pozisyondan başlayan dramatik bir kayma, 2D kütle-tR grafiğinde gözlemlenebilir, bu da 8. pozisyonun gerçekten de RNA #6'da bir ψ olduğunu gösterir.
Dizileme RNA karışımları. Beş farklı RNA zincirinin bir karışımı, 3' uçlu etiketleme ile 2D-HELS MS Seq yaklaşımı ile sıralanır (bkz. Bölüm 8). Karışık RNA'ların dizilenmesiyle ilgili endişe, 2B kütle-tR grafiğindeki çoklu merdiven eğrilerinin, hepsi aynı başlangıç noktalarını paylaştığında (2B kütle-tR grafiğindeki hidrofobik etiket) birbiriyle örtüşebilmesidir. Bununla birlikte, temel çağrı, her biri MFE verilerindeki iki bitişik merdiven parçası arasındaki kütle farkına dayalı olarak tek tek yapılır. Her bir kütle farkı, veri havuzundaki 8,9 kanonik veya modifiye edilmiş nükleotidlerin teorik kütlelerinden biriyle iyi bir şekilde eşleştiği sürece (bir PPM MS farkı < 10) doğru temel çağrı yapılabilir. Çoğullanmış RNA örneklerinin analizinde, Şekil 1 ve 2'de kullanılan tipik işleme ve baz çağırma algoritması, esas olarak karışımdan kaynaklanan önemli ölçüde artan veri karmaşıklığı nedeniyle kullanılmamaktadır. Bu diziler, iki bitişik kütle merdiveni parçası arasındaki kütle farkının hesaplanması ve bunun veri havuzundaki9 nükleotidin teorik kütlesi ile karşılaştırılması yoluyla manuel olarak çağrılır. Kütle PPM <10 ile eşleşen herhangi bir taban, bu konumda temel kimlik olarak seçilir. Baz arama için bu baz bazında manuel hesaplama ile karışımdaki tüm diziler doğru bir şekilde sıralanır. OriginLab yazılımı, beş farklı RNAdizisini daha iyi görselleştirmek için her dizi için başlangıç tR'nin sistematik olarak normalleştirildiği bir 2B kütle-t R grafiğini yeniden oluşturmak için kullanılır (Şekil 3). Böyle bir normalizasyon olmadan, beş RNA'nın tümünün dizileri için harf kodları (yani, A, C, G ve U) çizim üzerinde bir araya getirilecektir (Şekil S1), bu da Şekil 3'te bildirilenlere kıyasla daha az görselleştirme kolaylığı ile sonuçlanacaktır. Dizileme sonuçları, 2D-HELS MS Seq yaklaşımının sadece saflaştırılmış tek sarmallı RNA'ların dizilimi ile sınırlı olmadığını, aynı zamanda daha da önemlisi, çoklu RNA sarmallı RNA karışımları ile sınırlı olduğunu göstermektedir. Temel arama ve dizi oluşturma sürecini otomatikleştirmek için algoritmalar şu anda geliştirilme aşamasındadır.
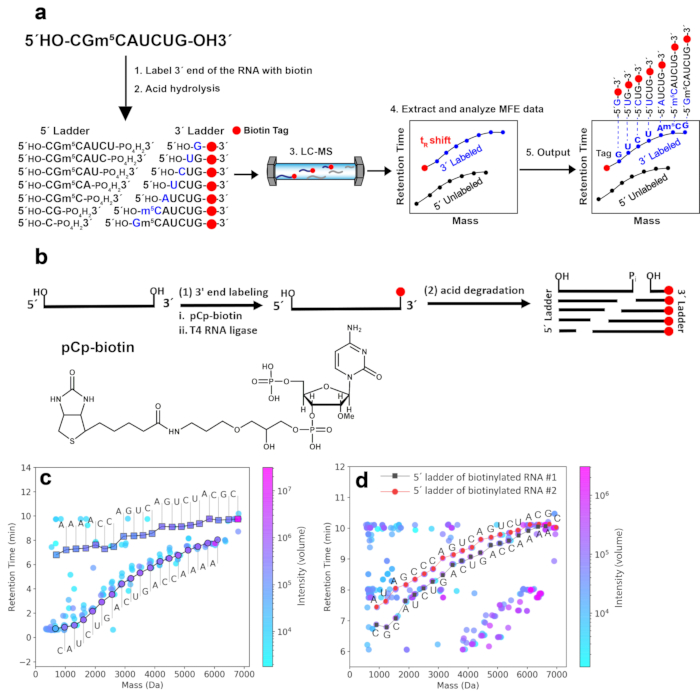
Şekil 1. Temsili RNA örneklerinin 2D-HELS MS Seq'i. (a) 2D-HELS MS Seq için İş Akışı. Başlıca adımlar arasında 1) dizilenecek RNA'nın hidrofobik etiket etiketlemesi, 2) asit hidrolizi, 3) LC-MS ölçümü, 4) MFE verilerinin çıkarılması ve analizi ve 5) algoritmalar veya manuel hesaplama yoluyla dizi oluşturma yer alır. (b) 2D-HELS MS Seq. için RNA'nın 3'-ucuna bir biyotin etiketi eklenmesini içeren numune hazırlama protokolü. (c) RNA #1'in (19 nt) 3'-biyotin etiketli kütle-tR merdiven fragmanlarının tR'lerindekisistematik değişikliklere dayalı olarak 2D kütle tutma süresi (tR) grafiğinde 3'-merdivenin 5'-merdivenden ve diğer istenmeyen parçalardan ayrılması. Diziler de novo ve otomatik olarak doğrudan bir temel çağırma algoritması9 tarafından okunur. (d) Sırasıyla RNA # 1 ve RNA # 2, 19 nt ve 20 nt etiketli 5'-biyotinin eşzamanlı dizilimi. RNA'nın 5'-ucuna bir biyotin etiketi ekleme yöntemleri, 3'-biyotinilasyondan farklıdır ve daha önce yayınlanan protokol9'da bulunabilir. İki RNA'nın (RNA # 1 ve RNA # 2) 5' ucu biyotinile edilir ve 5'-biyotinillenmiş merdivenleri kolayca tanımlanabilir; her iki 5'-biyotinile merdiven, LC-MS'den sonra 2D kütle-tR grafiğindeki etiketlenmemiş 3'-merdivenlerinden kolayca ayrılır, çünkü biyotinile merdiven bileşenleri, biyotinin hidrofobikliği nedeniyle daha büyük tR kaymalarına sahipken, etiketlenmemiş merdiven bileşenleri daha düşük tR bölgesindedir. 5' merdivenler ve 3' merdivenler bir arada bulunmalarına rağmen, iki karışık RNA zincirinin dizi yorumuna müdahale etmezler. Bu iki RNA'nın her bir dizisi, hesaplama algoritması ile işlenmiş verileredayalı olarak 5'-biyotinillenmiş merdivenlerden manuel olarak elde edilir 8,9. Bu rakam Zhang ve ark.9'dan değiştirilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 2. Psödouridin (ψ) 2D-HELS MS Seq için eklentisine dönüştürülmesi. (a) Bir ψ içeren 20 nt RNA'da (RNA #6) ψ CMC eklentisine dönüştüren reaksiyonun ham ürününün HPLC profili. (b) ψ içeren bir RNA #6'nın dizilimi. ψ CMC-ψ eklentilerine (ψ*) dönüştürülmesi, kütlesi ve CMC'nin hidrofobikliği nedeniyle kütlede 252.2076 Dalton'luk bir artışa ve tR'de önemli bir artışa neden olur. Böylece, kütle-tR grafiğinde 8 konumundan başlayan dramatik bir kayma gözlemlenebilir, bu da bunun RNA dizisinde 8 konumunda bir ψ olduğunu gösterir. Diziler, hesaplama algoritması tarafından işlenen verilere dayalı olarak manuel olarak elde edilir 8,9. Bu rakam Zhang ve ark.9'dan değiştirilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 3. Beş farklı RNA içeren dizileme RNA karışımları. Her RNA'yı 2D-HELS MS Seq'ten önce 3' ucunda etiketlemek için bir biyotin kullanılır. Her sekans için, başlangıç tR değerleri, görselleştirmeyi kolaylaştırmak için 7 dakikalık aralıklarla başlayacak şekilde sistematik olarak normalleştirilir. Başlangıç tR değeri ile sonraki tRdeğerleri arasındaki mutlak farklar, beş RNA'nın her biri için değişmeden kalır ve bu nedenle her birini aynı grafikte görselleştirmek daha kolaydır. Tüm bazlar, iki bitişik merdiven bileşeninin kütle farklarının manuel olarak hesaplanması ve bunların RNA nükleotidi ve modifikasyon veri tabanı8'deki teorik kütle farkları ile eşleştirilmesiyle tanımlanır; Şekil 3 için çizimler, manuel baz çağırma ve sıralama verilerine dayalı olarak OriginLab kullanılarak yeniden oluşturulur (Temsili Sonuçlardaki RNA karışımlarını Sıralama Bölümüne bakın). T Rnormalizasyonu olmayan beş karışık RNA'nın 2D kütle-tR şekli Şekil S1'de gösterilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tartışmalar
Tandem tabanlı MS fragmantasyonundan farklı olarak, bir kütle spektrometresi9,10 ile analizden önce RNA'yı parçalamak için 2D-HELS MS Seq yaklaşımında yüksek kontrollü asidik hidroliz kullanılır. Sonuç olarak, asitle bozunmuş her parça, bir sıralama merdiveninin eşdeğerini oluşturan cihaz tarafından tespit edilebilir. Optimal koşullar altında, bu yöntem, yalnızca bir fosfodiester bağında 8,9,10, molekül başına bir bölgeye özgü RNA bölünmesi yoluyla RNA'dan "ideal" bir dizi merdiveni oluşturur. Her bozulmuş parça, kütle spektrometresi tarafından tek bir çalışmada ölçüldükten sonra, iki bitişik merdiven parçası arasındaki kütle farkı, RNA nükleotidinin tam kütlesine veya o konumdaki modifikasyona karşılık gelir. Her RNA modifikasyonu, onu RNA'da tanımlamaya ve konumlandırmaya yardımcı olabilecek içsel benzersiz bir kütleye sahiptir veya benzersiz bir kütleye sahip bir kütleye dönüştürülebilir. Bu nedenle, teorik olarak, bu yöntem, herhangi bir RNA'nın de novo ve doğrudan dizilimi için hem kanonik hem de modifiye nükleotidlerin kimliğini ve konumunu rapor edebilir. Bununla birlikte, farklı dizi merdivenleri birbiriyle örtüşebilir, bu da MS veri analizini karmaşıklaştırabilir ve pratikte MS tarafından RNA dizilemesi için zorlaştırabilir.
3'-hidrofobik etiketin faydalarından biri, herhangi bir parçalanma yöntemindeki büyük bir zorluğun üstesinden gelmesidir, yani her RNA molekülünün tam olarak iki parçaya bölünmesi gerekir (ve ideal olarak daha fazla değil): bir parça orijinal 5'-ucunu içerir ve diğeri RNA'nın orijinal 3'-ucunu içerir. Bu nedenle, her bölünme olayı iki parça üretir ve biri 5' ucundan ve diğeri 3' uçtan ölçülen iki merdiven üretir. Hangi MS zirvesinin hangi merdivene ait olduğunun belirlenmesinde her zaman belirsizlik vardır. Bu, çok sayıda örtüşen dizi merdiveninin oluşması nedeniyle birkaç farklı RNA'nın karışımında daha sorunlu hale gelir. Bununla birlikte, 3' uçlarından gelen tüm merdiven parçaları hidrofobik bir etiketle etiketlendiğinden, çok daha uzun tRs sergilerler (Şekil 1a). Sonuç olarak, yalnızca 3' etiketli RNA'dan türetilen 2D kütle-tR verilerinde açık ve net merdivenler elde edebiliriz. Özellikle, farklı kimyasal konjugasyon yöntemleri kullanarak herhangi bir RNA'nın 5'- veya 3'-ucunu seçici olarak etiketlemek için yaklaşımları optimize ediyoruz9. Ayrıca, burada terminal taban(lar)ını belirlemek için kullanılmayan, ancak hem 5' hem de 3' yönden okurken (yani, çift yönlü sıralama doğrulaması) aynı dizi bilgisini iki kez sağlamak için kullanılan çift yönlü sıralama da gerçekleştirebiliriz ve böylece sıralamanın doğruluğunu daha da artırırız.
Bilinmeyen RNA örneklerinin, özellikle karmaşık biyolojik örnekler için de novo dizilimi için, dizi oluşturma için büyük miktarda LC-MS verisini doğru ve verimli bir şekilde işlemek için genel ve sağlam bir algoritma gereklidir ve bu, yakın zamanda diğer yayınlanmış çalışmalar aracılığıyla kullanıma sunulmuştur10. Bu algoritmalar daha karmaşık örneklerin10 dizilenmesi için kullanılmış olsa da, bu çalışmada aksi belirtilmedikçe dizi oluşturma için manuel temel arama gerçekleştirdik. 2D-HELS MS Seq'deki tüm önemli adımları kapsamayı amaçlıyoruz ve ek dizileme algoritmaları kullanmadan bile, dizilenecek RNA'nın dizilerini manuel olarak okuyabildiğimiz süreci göstermek istiyoruz. Görselleştirmeyi kolaylaştırmak ve 2B kütle-tR grafiğinde sıralama için gereken merdiven parçalarını daha hızlı tanımlamak için, her LC-MS çalışmasının MFE dosyaları, aksi belirtilmedikçe, dizilerini okumadan önce yayınlanmış bir algoritmanın8 gözden geçirilmiş bir sürümü tarafından işlenir. Yayınlanan algoritma, LC-MS verilerinden dizileri okumak için doğrudan kullanılamaz, ancak işlevinin bir kısmı verileri işlemek için hala kullanılabilir - bu algoritma aracılığıyla hiyerarşik olarak kümelenmiş kütle eklentileri, her bir merdiven bileşeninin yoğunluğunu artıracak ve bu da veri karmaşıklığını azaltacaktır, özellikle dizi okumalarının oluşturulduğu kritik bölgede8, 9.
2D-HELS MS Seq için numune hazırlama sırasındaki en önemli adımlardan biri, RNA hidrofobik etiket sonu etiketleme verimliliğinin iyileştirilmesiyle sonuçlanır. Yüksek bir etiketleme verimliliği, dizi verilerinin dayandığı MS sinyallerini üretmek için gereken RNA örneği miktarını azaltmaya yardımcı olabilir. Etiketleme verimliliğini artırmak için, RNA'nın 3' ucunu etiketlerken adenilasyon adımından kaçınmak için aktive edilmiş AppCp-biyotin kullanmak da dahil olmak üzere yeni etiketleme stratejileri kullanıyoruz. 19 nt RNA'nın 3'-ucunu biyotin ile etiketlemek için reaksiyonun verimi (bkz. adım 2.2), bu tek adımlı yöntem kullanılarak %60'tan ~%95'e9 yükseltilebilir. Etkili etiketleme ile, daha önce açıklandığı gibi 12 farklı RNA'ya kadar içeren karışık bir numuneyidizileyebiliyoruz 9. Bu çalışmada, dizileme sürecini göstermek için temsili bir örnek olarak beş RNA'nın bir karışımını kullanıyoruz. Ayrıca, doğru dizileme için gereken tüm merdiven parçalarını tespit ediyoruz ve karışımdaki beş RNA dizisinin her birinin tam dizilerini okuyoruz. Daha yüksek etiketleme verimliliği, yalnızca numune yükleme miktarını en aza indirmeye yardımcı olmakla kalmaz, aynı zamanda dizi oluşturma için aşağı akış veri analizi sırasında veri karmaşıklığının önemli ölçüde azaltılmasına da yardımcı olur. RNA'ları hem 5' hem de 3' uçlarda etiketlemede kantitatif verim elde etmek için yeni reaksiyonlar şu anda geliştirilme aşamasındadır.
Şekil 1c'de gösterildiği gibi RNA #1'i sıralarken, asit bozunmasından önce biyotinile RNA #1'i fiziksel olarak ayırmak için streptavidin yakalama ve serbest bırakma adımları kullanılır (bkz. Bölüm 3). Bu, etiketlenmemiş RNA'nın küçük bir kısmını çıkarır ve daha sonra 2B kütle-tR grafiğindeki etiketli kütle merdivenlerinin görsel olarak tanımlanmasında daha kolay bir yol açar. Bununla birlikte, fiziksel ayırma aşaması zorunlu değildir, çünkü biyotinile edilmiş RNA merdiven fragmanları, etiketlenmemiş muadillerine kıyasla biyotin etiketinden gelen hidrofobiklik nedeniyle gecikmiş/daha uzun tR'leresahiptir. Ek olarak, baz çağrısı fiziksel ayırmaya dayanmaz, ancak bitişik kütle merdiveni bileşenlerinin kütle farklılıklarına dayanır, bu nedenle, iki bitişik merdiven bileşeninin kütle farkları, belirli bir nükleotidin karşılık gelen kütleleri ile iyi bir şekilde eşleştiği sürece doğru temel çağrısı elde edilebilir veya RNA nükleotidi ve modifikasyon tarih tabanı8. Temel aramayı ve sıra oluşturmayı otomatikleştirmek için şu anda bir hesaplama algoritması geliştirilme aşamasındadır.
Orijinal LC-MS verilerinin (.d dosya türünde) elektronik tablo dosyalarına aktarılması sırasındaki MFE ayarları, veri işleme ve müteakip dizi oluşturma için son derece önemlidir (bkz. Bölüm 6.5). Örneğin, MFE ayarını 100 ila 1000 arasında bir aralıkta "yükseklikle zirve" olarak test ettik ve 100 ayarının bize 1000 ayarından 2 kat daha fazla bileşik sağlayabileceğini fark ettik. Herhangi bir merdiven bileşeninin eksik olmasını önlemek için, sıralama iş akışı sırasında MFE ayarını yapabiliriz. Bu ayar büyük olasılıkla cihaz kütle çözünürlüğüne, kütle merdiveni parçalarının miktarına ve veri karmaşıklığına bağlıdır. Ek olarak, küçük moleküller için centroid veri setini ve kromatografik tip ayarını kullanmak önemlidir. Kalite puanı, veri kalitesine bağlı olarak %50 ile %100 arasında değişebilir.
Çalışmada kullandığımız LC-MS cihazı, ~ 40K'lık bir üst kütle çözünürlüğüne sahiptir ve yöntemi yalnızca 35 bazdan daha kısa RNA'yı dizileme ile sınırlar. Bununla birlikte, bu yöntemin tam okuma uzunluğu cihaza bağlıdır; Daha yüksek çözme gücüne sahip daha gelişmiş cihazlar, daha uzun okuma uzunluğuna neden olabilir. Benzer şekilde, verim, yani tek bir LC-MS çalışmasında aynı anda kaç RNA dizisinin dizilenebileceği araştırılmaya devam ediyor, ancak herhangi bir algoritma9 kullanmadan bile 12 farklı RNA dizisine kadar bir RNA örneği karışımını manuel olarak diziledik. Mevcut iş akışında, her LC-MS çalışması için ~100 pmol kısa RNA (<35 nt) gereklidir. Ek deneylere ihtiyaç duyulduğunda yükleme miktarı artar: izomerik nükleotid modifikasyonlarını ayırt etmek için tipik olarak 400 pmol'e kadar RNA gereklidir. tRNAPhe gibi spesifik tRNA'yı dizilemek için, dizileme ve modifikasyon analizi için ~1000 pmol numune gerekebilir. Bununla birlikte, LC-MS cihazlarında daha yüksek hassasiyetle gerekli numune yükleme miktarlarının azaltılmasını bekliyoruz. Numune etiketleme verimliliği, sıralama algoritması ve cihaz hassasiyeti ve çözünürlüğündeki iyileştirmelerle, yöntemimizin, özellikle çeşitli RNA modifikasyonlarına sahip olanlar olmak üzere daha geniş bir RNA numunesi yelpazesine uygulanabilir olmasını bekliyoruz.
Açıklamalar
Yazarlar, bu el yazmasında tartışılan teknolojiyle ilgili geçici bir patent başvurusunda bulunmuşlardır.
Teşekkürler
Yazarlar, Ulusal Sağlık Enstitüleri'nden (1R21HG009576) S. Z. ve W. L.'ye R21 hibesini ve bu çalışmayı destekleyen New York Teknoloji Enstitüsü (NYIT) Araştırma ve Yaratıcılık için Kurumsal Destek hibelerini S. Z.'ye kabul etmektedir. Yazarlar, figür yapımına yardımcı olduğu için doktora öğrencisi Xuanting Wang'a (Columbia Üniversitesi) teşekkür eder ve Prof. Michael Hadjiargyrou (NYIT), Prof. Jingyue Ju (Columbia Üniversitesi), Dr. James Russo, Shiv Kumar, Xiaoxu Li, Steffen Jockusch ve Ju laboratuvarının diğer üyeleri (Columbia Üniversitesi), Dr. Yongdong Wang (Cerno Bioscience), Meina Aziz (NYIT) ve Wenhao Ni'ye (NYIT) makalemiz için yararlı tartışmalar ve öneriler için teşekkür eder.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 5' DNA Adenylation kit | New England Biolabs | E2610S | 50uM concentration |
| 6550 Q-TOF mass spectrometer | Agilent Technologies | 5991-2116EN | Coupled to a 1290 Infinity LC system |
| A(5´)pp(5´)Cp-TEG-biotin-3´ | ChemGenes | 91718 | HPLC purified |
| ATPγS | Sigma-Aldrich | 11162306001 | Lithium salt |
| Bicine | Sigma-Aldrich | B8660 | BioXtra, ≥99% (titration) |
| Biotin maleimide | Vector Laboratories | SP-1501 | Long arm |
| C18 column | Waters | 186003532 | 50 mm × 2.1 mm Xbridge C18 column with a particle size of 1.7 μm |
| Centrifugal Vacuum Concentrator | Labconco | Refrig 115v/60hz 7310022 | Labconco CentriVap |
| ChemBioDraw | PerkinElmer | ChemDraw Prime | Generate a chemical structure and property data of structures & fragments |
| CMC (N-cyclohexyl-N?-(2-morpholinoethyl)-carbodiimide metho-p-toluenesulfonate) | Sigma-Aldrich | 2491-17-0 | 95% Purifiy |
| Cyanine3 maleimide (Cy3) | Lumiprobe | 11080 | Water insoluble |
| DEPC-treated water | Thermo Fisher Scientific | AM9906 | Autoclaved, certified nuclease-free |
| Diisopropylamine (DIPA) | Thermo Fisher Scientific | 108-18-9 | 99% Alfa Aesar |
| DMSO | Sigma-Aldrich | 276855 | Anhydrous dimethyl sulfoxide, 99.9% |
| EDTA | Sigma-Aldrich | E6758 | Anhydrous, crystalline, BioReagent, suitable for cell culture |
| Formic acid | Merck | 64-18-6 | 98-100%, ACS reag, Ph Eur |
| Hexafluoro-2-propanol (HFIP) | Thermo Fisher Scientific | 920-66-1 | 99% Acros Organics |
| LC-MS sample vials | Thermo Fisher Scientific | C4000-11 | Plastic screw thread vials |
| LC-MS vial caps | Thermo Fisher Scientific | C5000-54A | Autosampler vial screw thread caps |
| Na2CO3 buffer | Sigma-Aldrich | 88975 | BioUltra, >0.1 M Na2CO3, >0.2 M NaHCO3 |
| Oligo Clean & Concentrator | Zymo Research | D4060 | Spin column |
| OriginLab | OriginLab | OriginPro | Data analysis and graphing software |
| pCp-biotin | TriLink BioTechnologies | NU-1706-BIO | 20 ul (1 mM) |
| RNA #1--#6 | Integrated DNA Technologies | Custom RNA oligos | 19nt-21nt single-stranded RNAs, used without further purification |
| Rocking platform shaker | VWR | Orbital Shaker Standard 1000 | Speed Range 40 to 300 rpm |
| Streptavidin magnetic beads | Thermo Fisher Scientific | 88816 | Binding approx. 55ug biotinylated rabbit lgG per mg of beads |
| Sulfonated Cyanine3 maleimide | Lumiprobe | 11380 | Water soluble |
| T4 DNA ligase 1 | New England Biolabs | M0202S | 400 units/uL |
| T4 polynucleotide kinase | Sigma-Aldrich | T4PNK-RO | From phage T4 am N81 pse T1 infected Escherichia coli BB |
| Tris-HCl buffer | Sigma-Aldrich | T6455 | Tris-HCl Buffer, pH 10, 10×, Antigen Retriever |
| Urea | Sigma-Aldrich | 81871 | Urea for synthesis. CAS No. 57-13-6, EC Number 200-315-5. |
Referanslar
- Addepalli, B., Venus, S., Thakur, P., Limbach, P. A. Novel ribonuclease activity of cusativin from Cucumis sativus for mapping nucleoside modifications in RNA. Analytical and Bioanalytical Chemistry. 409 (24), 5645-5654 (2017).
- Gao, H., Liu, Y., Rumley, M., Yuan, H., Mao, B. Sequence confirmation of chemically modified RNAs using exonuclease digestion and matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 23 (21), 3423-3430 (2009).
- McLuckey, S. A., Van Berkel, G. J., Glish, G. L. Tandem mass spectrometry of small, multiply charged oligonucleotides. Journal of The American Society for Mass Spectrometry. 3 (1), 60-70 (1992).
- Fountain, K. J., Gilar, M., Gebler, J. C. Analysis of native and chemically modified oligonucleotides by tandem ion-pair reversed-phase high-performance liquid chromatography/electrospray ionization mass spectrometry. Rapid Communications in Mass Spectrometry. 17 (7), 646-653 (2003).
- Taucher, M., Breuker, K. Characterization of modified RNA by top-down mass spectrometry. Angewandte Chemie International Edition in English. 51 (45), 11289-11292 (2012).
- Kellner, S., Burhenne, J., Helm, M. Detection of RNA modifications. RNA Biology. 7 (2), 237-247 (2010).
- Thomas, B., Akoulitchev, A. V. Mass spectrometry of RNA. Trends in Biochemical Sciences. 31 (3), 173-181 (2006).
- Bjorkbom, A., et al. Bidirectional direct sequencing of noncanonical RNA by two-dimensional analysis of mass chromatograms. Journal of the American Chemical Society. 137 (45), 14430-14438 (2015).
- Zhang, N., et al. A general LC-MS-based RNA sequencing method for direct analysis of multiple-base modifications in RNA mixtures. Nucleic Acids Research. 47 (20), 125(2019).
- Zhang, N., et al. Direct sequencing of tRNA by 2D-HELS-AA MS Seq reveals its different isoforms and dynamic base modifications. ACS Chemical Biology. 15 (6), 1464-1472 (2020).
- Bakin, A., Ofengand, J. Four newly located pseudouridylate residues in Escherichia coli 23S ribosomal RNA are all at the peptidyltransferase center: analysis by the application of a new sequencing technique. Biochemistry. 32 (37), 9754-9762 (1993).
- Cantara, W. A., et al. The RNA Modification Database, RNAMDB: 2011 update. Nucleic Acids Research. 39 (Database issue), D195-D201 (2011).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır