Method Article
2D-HELS MS Seq : une méthode générale basée sur LC-MS pour le séquençage direct et de novo de mélanges d’ARN avec différentes modifications nucléotidiques
Dans cet article
Résumé
Ici, nous décrivons un protocole détaillé pour une méthode de séquençage basée sur la LC-MS qui peut être utilisée comme méthode directe pour séquencer l’ARN court (<35 nt par exécution) sans intermédiaire d’ADNc, et comme méthode générale pour séquencer différentes modifications nucléotidiques dans une seule étude avec une précision de base unique.
Résumé
Les approches de séquençage basées sur la spectrométrie de masse (MS) se sont révélées utiles pour le séquençage direct de l’ARN sans avoir besoin d’un intermédiaire d’ADN complémentaire (ADNc). Cependant, de telles approches sont rarement appliquées comme méthode de séquençage de novo de l’ARN, mais utilisées principalement comme un outil pouvant aider à l’assurance de la qualité pour confirmer les séquences connues d’échantillons d’ARN simple brin purifiés. Récemment, nous avons développé une méthode de séquençage direct de l’ARN en intégrant une stratégie de marquage hydrophobe à 2 dimensions dans le séquençage basé sur la MS (2D-HELS MS Seq). Cette méthode est capable de séquencer avec précision des séquences d’ARN uniques ainsi que des mélanges contenant jusqu’à 12 séquences d’ARN distinctes. En plus des quatre ribonucléotides canoniques (A, C, G et U), la méthode a la capacité de séquencer des oligonucléotides d’ARN contenant des nucléotides modifiés. Cela est possible parce que la nucléobase modifiée a soit une masse intrinsèquement unique qui peut aider à son identification et à son emplacement dans la séquence d’ARN, soit peut être convertie en un produit avec une masse unique. Dans cette étude, nous avons utilisé l’ARN, incorporant deux nucléotides modifiés représentatifs (pseudouridine (Ψ) et 5-méthylcytosine (m5C)), pour illustrer l’application de la méthode pour le séquençage de novo d’un seul oligonucléotide d’ARN ainsi que d’un mélange d’oligonucléotides d’ARN, chacun avec une séquence différente et/ou des nucléotides modifiés. Les procédures et les protocoles décrits ici pour séquencer ces ARN modèles seront applicables à d’autres échantillons d’ARN courts (<35 nt) lors de l’utilisation d’un système LC-MS standard à haute résolution, et peuvent également être utilisés pour la vérification de séquence d’oligonucléotides d’ARN thérapeutiques modifiés. À l’avenir, avec le développement d’algorithmes plus robustes et de meilleurs instruments, cette méthode pourrait permettre le séquençage d’échantillons biologiques plus complexes.
Introduction
Des méthodes de séquençage basées sur la spectrométrie de masse (MS), y compris la MS descendante et la MSen tandem 1,2,3,4, ont été développées pour le séquençage direct de l’ARN. Cependant, les techniques de fragmentation in situ permettant de générer efficacement des échelles d’ARN de haute qualité dans les spectromètres de masse ne peuvent actuellement pas être appliquées au séquençage de novo 5,6. De plus, il n’est pas très trivial d’analyser les données MS unidimensionnelles (1D) traditionnelles pour le séquençage de novo d’une seule séquence d’ARN purifié, et ce serait encore plus difficile pour le séquençage MS d’échantillons d’ARN mixtes 7,8. Par conséquent, une méthode de séquençage de l’ARN bidimensionnelle (2D) basée sur la chromatographie liquide (LC)-MS a été développée, intégrant la production d’échelles de temps de rétention de masse (tR) 2D pour remplacer les échelles de masse 1D, ce qui facilite grandement l’identification des composants de l’échelle nécessaires au séquençage de novo des ARN8. Cependant, la méthode de séquençage de l’ARN basée sur la LC-MS 2D est principalement limitée à l’ARN court synthétique purifié, car elle ne peut pas lire une séquence complète uniquement sur la base d’une seule échelle, mais doit s’appuyer sur deux échelles adjacentes coexistantes (échelles 5' et 3')8. Plus précisément, cette approche nécessite des lectures bidirectionnelles à extrémités appariées pour la lecture des nucléobases terminales dans la région de faible masse8. La complexité supplémentaire de la lecture des extrémités appariées fait que cette méthode est intenable pour le séquençage des mélanges d’ARN, car la confusion est soulevée quant à savoir quel fragment d’échelle appartient à quelle échelle pour les échantillons inconnus.
Pour surmonter les obstacles mentionnés ci-dessus dans les approches de séquençage de l’ARN basées sur la SEP et pour élargir ces applications au séquençage direct de l’ARN, deux questions doivent être abordées : 1) comment générer une échelle de masse de haute qualité qui peut être utilisée pour lire une séquence complète, du premier nucléotide au dernier dans un brin d’ARN, et 2) comment identifier efficacement chaque échelle ARN/masse dans un ensemble de données MS complexe. Parallèlement à une dégradation acide bien contrôlée, nous avons développé une nouvelle méthode de séquençage en introduisant une stratégie de marquage hydrophobe (HELS) dans la technique de séquençage basée sur la MS, et nous avons résolu avec succès ces deux problèmes en ajoutant un marqueur hydrophobe à l’extrémité 5' et/ou 3' des ARN à séquencer9. Cette méthode crée une échelle de séquence « idéale » à partir de l’ARN : chaque fragment d’échelle dérive d’un clivage d’ARN spécifique au site exclusivement au niveau de chaque liaison phosphodiester, et la différence de masse entre deux fragments d’échelle adjacents est la masse exacte du nucléotide ou de la modification du nucléotide à cette position 8,9,10. Cela est possible parce que nous incluons une étape d’hydrolyse acide hautement contrôlée, qui fragmente l’ARN, en moyenne, une fois par molécule, avant qu’il ne soit injecté dans l’instrument. En conséquence, chaque produit de fragment de dégradation est détecté sur le spectromètre de masse et tous les fragments forment ensemble une échelle de séquençage 8,9,10. Cette nouvelle stratégie permet une lecture complète d’une séquence d’ARN à partir d’une seule échelle d’un brin d’ARN sans lecture d’extrémité appariée de l’autre échelle de l’ARN, et permet en outre le séquençage par MS de mélanges d’ARN avec plusieurs brins différents contenant des modifications nucléotidiques combinatoires9. En ajoutant une étiquette à l’extrémité 5' et/ou 3' de l’ARN, les fragments d’échelle marqués présentent un retard significatif de tR, ce qui peut aider à distinguer les deux échelles de masse l’une de l’autre et aussi de la région bruyante de faible masse. Le décalage mass-tR provoqué par l’ajout de l’étiquette hydrophobe facilite l’identification de l’échelle de masse et simplifie l’analyse des données pour la génération de séquences. De plus, l’ajout de l’étiquette hydrophobe peut aider à identifier la base terminale dans le brin en empêchant son fragment d’échelle correspondant d’être dans la région bruyante de faible masseR en raison de l’augmentation de la masse et de l’hydrophobie causée par l’étiquette, permettant ainsi l’identification de la séquence complète d’un ARN à partir d’une seule échelle ; Aucune lecture d’extrémité appariée n’est requise. En conséquence, nous avons déjà démontré le séquençage réussi d’un mélange complexe de jusqu’à 12 brins d’ARN distincts sans l’utilisation d’un algorithme de séquençage avancé9, ce qui ouvre la porte au séquençage MS de novo d’ARN contenant à la fois des nucléotides canoniques et modifiés et rend plus réalisable le séquençage d’échantillons d’ARN mixtes et plus complexes. En fait, en utilisant 2D-HELS MS Seq, nous avons même réussi à séquencer une population mixte d’échantillons d’ARNt10 et nous étendons activement son application à d’autres échantillons d’ARN complexes.
Pour faciliter le séquençage direct d’un plus large éventail d’échantillons d’ARN par 2D-HELS MS Seq, nous nous concentrerons ici sur les aspects techniques de cette approche de séquençage et couvrirons toutes les étapes essentielles nécessaires lors de l’application de la technique au séquençage direct d’échantillons d’ARN. Des exemples spécifiques seront utilisés pour illustrer la technique de séquençage, y compris des séquences d’ARN uniques synthétiques, des mélanges de plusieurs séquences d’ARN distinctes et des ARN modifiés contenant à la fois des nucléotides canoniques et modifiés tels que la pseudouridine (ψ) et la 5-méthylcytosine (m5C). Étant donné que les ARN contiennent tous des liaisons phosphodiester, tout type d’ARN peut être hydrolysé à l’acide pour générer une échelle de séquence idéale pour le séquençage 2D-HELS MS Seq dans des conditions optimales 8,9. Cependant, la détection de tous les fragments d’échelle d’un ARN donné dépend de l’instrument. Sur un LC-MS haute résolution standard (40K), la quantité de charge minimale pour le séquençage d’un échantillon d’ARN court purifié (<35 nt) est de 100 pmol par passage. Cependant, il faut plus de matériel (jusqu’à 400 pmol par échantillon d’ARN) lorsque des expériences supplémentaires doivent être menées (p. ex., pour distinguer les modifications de bases isomères qui partagent des masses identiques). Le protocole utilisé pour le séquençage du modèle d’ARN modifiés synthétiques sera également applicable au séquençage d’échantillons d’ARN plus larges, y compris des échantillons d’ARN biologiques avec des modifications de base inconnues. Cependant, une quantité d’échantillon encore plus importante, telle que 1000 pmol pour le séquençage de l’ARNt (~76 nt) à l’aide d’un instrument LC-MS standard, est nécessaire pour séquencer l’ARNt complet avec toutes les modifications, et un algorithme avancé doit être développé pour son séquençage de novo 10.
Protocole
1. Concevoir des oligonucléotides d’ARN
- Concevoir des oligonucléotides d’ARN synthétiques de différentes longueurs (19 nt, 20 nt et 21 nt), dont un (ARN #6) avec des nucléotides canoniques et modifiés. ψ est utilisé comme modèle pour les modifications non altérant la masse, ce qui est difficile pour le séquençage MS car il a une masse identique à U. m5C est choisi comme modèle pour les modifications altérant la masse afin de démontrer la robustesse de l’approche.
ARN #1 : 5'-HO-CGCAUCUGACUGACCIAAA-OH-3'
ARN #2 : 5'-HO-AUAGCCCAGUCAGUCUACGC-OH-3'
ARN #3 : 5'-HO-AAACCGUUACCAUACUGAG-OH-3'
ARN #4 : 5'-HO-GCGUACAUCUUCCCCUUUAU-OH-3'
ARN #5 : 5'-HO-HO-GCGGAUUUAGCUCAGUUGGGA-OH-3'
ARN #6 : 5'-HO-AAACCGUψACCAUUAm5CUGAG-OH-3'
- Dissoudre chaque ARN synthétique dans de l’eau traitée au pyrocarbonate de diéthyle sans nucléases (exprimée en H2O traitée au DEPC, sauf indication contraire) pour obtenir une solution mère d’ARN de 100 mM. Les solutions mères sont stockées à long terme à -20 °C.
- Pour éviter une éventuelle dégradation de l’échantillon d’ARN, utilisez des fournitures expérimentales exemptes de RNase, notamment de l’eau traitée au DEPC, des tubes de microcentrifugation et des pointes de pipette. Essuyez fréquemment les surfaces des fournitures de laboratoire à l’aide de lingettes d’élimination de la RNase.
2. Étiqueter l’extrémité 3' des ARN avec de la biotine
- Protocole de réaction en deux étapes (adénylation et ligature)
- Ajouter 1 μL de tampon de réaction d’adénylation 10x contenant 50 mM d’acétate de sodium, pH 6,0, 10 mM de MgCl2, 5 mM de dichlorodiphényltrichloroéthane (DTT), 0,1 mM d’acide éthylènediaminetétraacétique (EDTA), 1 μL de 1 mM d’ATP, 1 μL de 100 μM de bisphosphate de cytidine biotinylé (pCp-biotine), 1 μL de 50 μM d’ARN ligase et 6 μL de H2O traité au DEPC (un volume total de 10 μL) dans un tube PCR à paroi mince de 0,2 mL sans RNase.
REMARQUE : Stockez les réactifs à -20 °C avant la réaction en deux étapes. Décongelez les réactifs à température ambiante et mélangez bien par vortex et centrifugation avant de les ajouter à la réaction. - Incuber la réaction dans une machine PCR à 65 °C pendant 1 h et inactiver la réaction à 85 °C pendant 5 min.
- Effectuer l’étape de ligature dans un tube PCR de 0,2 mL sans RNase, à paroi mince, contenant 10 μL de solution réactionnelle de l’étape précédente en ajoutant 3 μL de tampon de réaction 10x T4 RNA ligase contenant 50 mM de tris(hydroxyméthyl)aminométhane (Tris)-HCl, pH 7,8, 10 mM MgCl2, 1 mM DTT, 1,5 μL du stock d’échantillon de 100 mM de l’ARN à séquencer, 3 μL de diméthylsulfoxyde anhydre (DMSO) pour atteindre 10 % (v/v), 1 μL d’ARN ligase T4 (10 unités/μL) et 11,5 μL de H2O traité au DEPC (pour un volume total de 30 mL). Incuber la réaction pendant une nuit à 16 °C dans un appareil de PCR.
REMARQUE : Combinez les composants de la réaction à température ambiante en raison du point de congélation élevé du DMSO (18,45 °C). - Incuber la réaction pendant la nuit à 16 °C.
- Tremper et purifier la réaction par purification de la colonne pour éliminer les enzymes et la pCp-biotine libre à l’aide d’Oligo Clean & Concentrator (Zymo Research, Irvine, CA, USA). Le tampon de liaison à l’oligo, le tampon de lavage de l’ADN, les colonnes d’essorage et les tubes de collecte sont fournis dans le kit. Ajouter 20 mL de H2O traité au DEPC à la solution réactionnelle pour atteindre un volume d’échantillon de 50 mL avant d’ajouter le tampon de liaison.
- Ajouter 100 mL de tampon de liaison à chaque solution réactionnelle. Ajouter 400 μL d’éthanol, mélanger par pipetage et transférer le mélange dans la colonne. Centrifugeuse à 10 000 x g pendant 30 s. Jetez le flux continu.
- Ajoutez 750 μL de tampon de lavage d’ADN dans la colonne. Centrifugeuse à 10 000 x g et vitesse maximale pendant 30 s et 1 minute, respectivement.
- Transférez la colonne dans un tube de microcentrifugation de 1,5 ml. Ajouter 15 μL de H2O traité au DEPC dans la colonne et centrifuger à 10 000 x g pendant 30 s pour éluer le produit ARN.
REMARQUE : Les échantillons peuvent être stockés à -20 °C à ce stade jusqu’à ce que l’étape suivante soit effectuée.
- Ajouter 1 μL de tampon de réaction d’adénylation 10x contenant 50 mM d’acétate de sodium, pH 6,0, 10 mM de MgCl2, 5 mM de dichlorodiphényltrichloroéthane (DTT), 0,1 mM d’acide éthylènediaminetétraacétique (EDTA), 1 μL de 1 mM d’ATP, 1 μL de 100 μM de bisphosphate de cytidine biotinylé (pCp-biotine), 1 μL de 50 μM d’ARN ligase et 6 μL de H2O traité au DEPC (un volume total de 10 μL) dans un tube PCR à paroi mince de 0,2 mL sans RNase.
- Protocole de réaction en une étape
- Effectuer une réaction de marquage en une seule étape en combinant 2 μL d’adénosine-5'-5'-diphosphate-{5'-(cytidine-2'-O-méthyl-3'-phosphate-TEG}C-biotine (AppCp-biotine), 3 μL de tampon de réaction 10x ligase, 1,5 μL du stock d’échantillon de 100 mM de l’ARN à séquencer, 3 μL de DMSO anhydre pour atteindre 10 % (v/v), 1 μL d’ARN ligase T4 (10 unités/μL), et 19,5 μL de H2O traité au DEPC (pour un volume total de 30 mL) dans un tube de microcentrifugation sans RNase de 1,5 mL.
- Incuber la réaction pendant une nuit à 16 °C dans un appareil de PCR.
- Effectuez la purification de la colonne comme décrit ci-dessus aux étapes 2.1.5 à 2.1.8.
REMARQUE : Préparez un tube réactionnel séparé/exclusif pour chaque échantillon d’ARN (échelle de 150 pmol d’ARN). Le marquage de l’extrémité 5' de l’ARN avec de la sulfo-cyanine3 (Cy3) ou du Cy3 peut être nécessaire (par exemple, pour la vérification du séquençage bidirectionnel). La méthode est différente de celle de la 3'-biotinylation et est décrite dans une publication précédente9.
3. Capturer un échantillon d’ARN biotinylé sur des billes de streptavidine
- Activez 200 μL de billes magnétiques de streptavidine C1 en ajoutant 200 μL de 1 tampon N&B (5 mM de Tris-HCl, pH 7,5, 0,5 mM d’EDTA, 1 M de NaCl) dans un tube de microcentrifugation sans RNase de 1,5 mL. Vartex cette solution et placez-la sur un support magnétique pendant 2 min. Ensuite, jetez le surnageant en pipetant soigneusement la solution.
- Lavez les billes deux fois avec 200 μL de solution A (NaOH 0,1 M traité au DEPC et 0,05 M NaCl traité au DEPC) et une fois dans 200 μL de solution B (NaCl 0,1 M traité au DEPC). Pour chaque étape de lavage, agitez la solution et placez-la sur un support magnétique pendant 2 min, puis jetez le surnageant. Ajouter ensuite 100 μL de tampon 2x N&B (10 mM de Tris-HCl, pH 7,5, 1 mM d’EDTA, 2 M de NaCl).
- Ajouter 1 tampon N&B à l’échantillon d’ARN biotinylé jusqu’à ce que le volume atteigne 100 μL. Ajoutez ensuite cette solution aux billes lavées stockées dans 100 μL de tampon 2x N&B. Incuber pendant 30 min à température ambiante sur une plate-forme basculante, agiter à 100 tr/min. Placez le tube sur un support magnétique pendant 2 min et jetez le surnageant.
- Laver les billes enrobées 3 fois dans 1x tampon N&B et mesurer la concentration finale de surnageant à chaque étape de lavage par Nanodrop pour l’analyse de récupération, afin de confirmer que les molécules d’ARN cibles restent sur les billes.
- Incuber les billes dans 10 mM d’EDTA, pH 8,2 avec 95 % de formamide à 65 °C pendant 5 min dans une machine PCR. Maintenez le tube sur le support magnétique pendant 2 min et récupérez le surnageant (contenant les ARN biotinylés libérés par les billes de streptavidine) à l’aide d’une pipette.
REMARQUE : Cette étape de séparation physique avant la dégradation acide n’est utilisée que pour le séquençage de l’ARN#1 dans la figure 1c, et n’est pas obligatoire pour le 2D-HELS MS Seq car le marquage hydrophobe à la biotine peut faire en sorte que les fragments de l’échelle marqués 3' aient un tR significativement retardé pendant la mesure LC-MS, ce qui permet de distinguer clairement les fragments de l’échelle 3' marqués des fragments de l’échelle 5' non marqués dans le graphique 2D mass-tR .
4. Hydrolyse acide de l’ARN pour générer des échelles MS pour le séquençage
- Divisez chaque échantillon d’ARN en trois aliquotes égales. Par exemple, divisez un échantillon d’ARN d’un volume de 15 μL en trois aliquotes de 5 μL.
- Ajouter un volume égal d’acide formique pour obtenir 50 % (v/v) d’acide formique dans le mélange réactionnel 8,9.
- Incuber la réaction à 40 °C dans un appareil de PCR, avec une réaction pendant 2 min, une pendant 5 min et une pendant 15 min, respectivement.
- Éteindre la dégradation acide en congelant immédiatement l’échantillon sur de la glace sèche après la fin de chaque réaction.
- Utilisez un concentrateur centrifuge sous vide pour sécher l’échantillon. L’échantillon est généralement complètement séché en 30 minutes, et l’acide formique est éliminé avec le H2O pendant le processus de séchage, car l’acide formique a un point d’ébullition (100,8 °C) similaire à celui du H2O (100 °C).
- Mettez en suspension et combinez un total de trois échantillons séchés dans 20 μL de H2O traité au DEPC pour la mesure LC-MS.
REMARQUE : Les échantillons peuvent être stockés à -20 °C à ce stade en attendant la mesure LC-MS.
5. Convertir ψ en adduction CMC-ψ
- Ajouter 80 μL de H2O traité au DEPC dans un tube de microcentrifugation sans RNase de 1,5 mL contenant 0,0141 g de N-cyclohexyl-Nʹ-(2-morpholinoéthyl)-carbodiimide métho-p-toluènesulfonate (CMC) et 0,07 g d’urée. Ajouter 10 μL de l’échantillon de 100 μM de l’ARN à séquencer, 8 μL de tampon de bicine 1 M (pH 8,3) et 1,28 μL d’EDTA 0,5 M. Ajouter du H2O traité au DEPC pour atteindre un volume total de 160 μL. Les concentrations finales sont de 0,17 M CMC, 7 M d’urée et 4 mM d’EDTA dans 50 mM de bicine (pH 8,3)11.
REMARQUE : Ce protocole s’applique soit à une seule séquence d’ARN synthétique, soit à des mélanges d’ARN. - Divisez la solution réactionnelle de 160 μL en quatre aliquotes égales dans des tubes PCR de 0,2 mL sans RNase et à paroi mince et incubez à 37 °C pendant 20 min dans un appareil de PCR.
REMARQUE : 50 μL par tube est le volume de réaction maximal pouvant être utilisé dans une machine PCR. - Tremper chaque réaction avec 10 μL d’acétate de sodium 1,5 M et 0,5 mM d’EDTA (pH 5,6).
- Effectuer la purification de la colonne à l’aide de quatre colonnes de centrifugation parallèles pour éliminer les réactifs en excès conformément à la procédure décrite aux étapes 2.1.5 à 2.1.8. Dissoudre le produit purifié dans 15 μL de H2O traité au DEPC dans chaque tube de microcentrifugation sans RNase de 1,5 mL.
- Transférez le produit purifié dans quatre tubes PCR de 0,2 mL sans RNase et à paroi mince. Ajouter 20 μL de tampon CO3 0,1 M Na2(pH 10,4) dans chaque 15 μL de produit purifié et ajouter du H2O traité au DEPC pour obtenir un volume final de 40 μL pour chaque tube de réaction (au total quatre tubes). Incuber la réaction à 37 °C pendant 2 h dans un appareil de PCR.
- Tremper et purifier la réaction par purification sur colonne à l’aide de quatre colonnes de centrifugation parallèles, comme décrit à l’étape 2.1.5. Éluer le produit converti CMC-ψ en un tube de microcentrifugation sans RNase de 1,5 mL contenant chacun 15 μL de H2O traité au DEPC.
- Combinez l’échantillon converti en CMC-ψ purifié de quatre tubes de collecte en un seul tube. Effectuer la dégradation de l’acide formique à 50 % (v/v) conformément aux procédures décrites aux étapes 4.1 à 4.6 pour générer des échelles MS pour le séquençage.
6. Mesure LC-MS
- Préparer les phases mobiles pour la mesure LC-MS. La phase mobile A est de 25 mM d’hexafluoro-2-propanol avec 10 mM de diisopropylamine dans de l’eau de qualité LC-MS ; la phase B mobile est le méthanol.
- Transférez l’échantillon dans un flacon d’échantillon LC-MS pour analyse. Chaque volume d’injection d’échantillon est de 20 μL et contient 100 à 400 pmol d’ARN.
- Utilisez les conditions LC suivantes : température de la colonne de 35 °C, débit de 0,3 mL/min ; une pente linéaire de 2 à 20 % de phase B mobile sur 15 min, suivie d’une étape de lavage de 2 min avec 90 % de phase B mobile.
REMARQUE : Pour les étiquettes finales plus hydrophobes telles que Cy3 et sulfo-Cy3, comme mentionné à la section 2, un pourcentage plus élevé de solvant organique peut être nécessaire pour l’élution de l’échantillon (c.-à-d. qu’un gradient similaire peut être utilisé, mais avec une plage de pourcentage accrue de la phase B mobile). Par exemple, 2 à 38 % de phase B mobile sur 30 min avec une étape de lavage de 2 min avec 90 % de phase B mobile. - Séparez et analysez les échantillons à l’aide d’un spectromètre de masse Agilent Q-TOF (Quadrupole Time-of-Flight) couplé à un système LC équipé d’un passeur d’échantillons et d’un système MS HPLC (High Performance Liquid Chromatography). La colonne LC est une colonne C18 de 50 mm x 2,1 mm avec une taille de particule de 1,7 μm. Utilisez les paramètres MS suivants : mode ions négatifs ; portée, 350 m/z à 3200 m/z ; vitesse de balayage, 2 spectres/s ; débit de gaz de séchage, 17 L/min ; température des gaz de séchage, 250 °C ; pression du nébuliseur, 30 psig ; tension capillaire, 3500 V ; et tension de fragmenteur, 365 V. Veuillez noter que ces paramètres sont spécifiques au type ou au modèle de spectromètre de masse utilisé.
- Acquérez des données avec le logiciel d’acquisition Agilent MassHunter. Utilisez le flux de travail d’extraction de caractéristiques moléculaires (MFE) d’Agilent pour extraire des informations sur les composés, notamment la masse, le temps de rétention, le volume (l’abondance de MFE pour les espèces d’ions respectives) et le score de qualité, etc. Utilisez les paramètres MFE suivants : « format de données centroïde, petites molécules (chromatographique), pic d’une hauteur ≥ 100, jusqu’à un maximum de 1000, score de qualité ≥ 50 ».
REMARQUE : Optimisez les paramètres MFE pour extraire autant de composés potentiels que possible, jusqu’à un maximum de 1000, avec des scores de qualité de ≥ 50.
7. Automatiser la génération de séquences d’ARN à l’aide d’un algorithme de calcul
REMARQUE : Cette procédure n’est illustrée que pour l’ARN #1 dans la figure 1c.
- Trier les composés extraits par MFE par ordre décroissant de volume (intensité maximale) et tR. Effectuez une présélection des données via 1) le réglage de tR de 4 à 10 min pour sélectionner les fragments d’ARN marqués par la biotine, puisque les tRs des composants de l’échelle de masse marqués à la biotine sont décalés vers cette fenêtre tR (4 min à 10 min), et 2) en utilisant un ordre de grandeur supérieur des composés d’entrée au nombre de fragments d’échelle pour le calcul de l’algorithme afin de réduire la quantité de données en fonction du volume. Par exemple, pour un ARN de 20 nt, 20 composants de l’échelle mass-tR marqués seront nécessaires pour le séquençage de l’ARN de 20 nt, et donc, 200 composés du fichier de données MFE seront sélectionnés en fonction du volume. Veuillez noter que la fenêtre tR peut être différente lorsqu’un type ou un modèle de spectromètre de masse différent est utilisé.
- Effectuer le traitement des données et la génération de séquences de l’ARN #1 à l’aide d’une version révisée d’un algorithme publié8. Les codes sources de l’algorithme révisé sont décrits précédemment (https://academic-oup-com.remotexs.ntu.edu.sg/nar/article/47/20/e125/5558343#supplementary-data)9.
- En plus d’automatiser la génération de séquences à l’aide de l’algorithme, calculez manuellement les différences de masse entre deux composants d’échelle adjacents pour l’appel de base. Toutes les bases de l’ARN peuvent être appelées manuellement et appariées aux bases théoriques de la base de données de nucléotides et de modifications de l’ARN8 ; ainsi, la séquence complète du brin d’ARN peut être lue manuellement avec précision, ce qui est utilisé pour confirmer la précision de la lecture de séquence rapportée par l’algorithme. D’autres structures de modifications de l’ARN peuvent être trouvées dans les bases de données de modification de l’ARN12, et leurs masses théoriques correspondantes sont obtenues par ChemBioDraw. Dans les tableaux S1 et S2, la différence de masse en ppm (parties par million) est indiquée en comparant la masse observée à sa masse théorique pour un composant d’échelle spécifique, et une valeur inférieure à 10 ppm est considérée comme une bonne correspondance pour chaque appel de base.
8. Séquençage des mélanges d’ARN
- Marquez un mélange de cinq brins d’ARN (ARN #1 à #5) à leurs extrémités 3' avec A(5')pp(5')Cp-TEG-biotine en utilisant un protocole en une étape décrit à l’étape 2.2. Dans un volume total de 150 μL de solution réactionnelle, ajouter 15 μL de tampon de réaction 10x T4 RNA ligase, 1,5 μL de chaque brin d’ARN (100 μM de stock d’ARN #1 à #5, respectivement, pour un volume total de 7,5 μL), 10 μL de 150 μM A(5')pp(5')Cp-TEG-biotine, 15 μL de DMSO anhydre, 5 μL d’ARN ligase T4 (10 unités/μL) et 97,5 μL de H2O traité au DEPC. Répartissez également la solution réactionnelle en cinq aliquotes. Chaque tube de microcentrifugation sans RNase contient 30 μL de solution réactionnelle.
- Incuber la réaction pendant une nuit à 16 °C dans un appareil de PCR.
- Effectuez la purification de la colonne conformément à la procédure décrite aux étapes 2.1.5 à 2.1.8 avec cinq colonnes de centrifugation parallèles. Éluer un échantillon de mélange de 5 brins d’ARN biotinylés 3' (mélange d’ARN #1 à #5) dans un tube de microcentrifugation sans RNase de 1,5 mL contenant chacun 15 μL de H2O traité au DEPC.
- Combinez les échantillons de mélange purifié des cinq tubes de collecte en un seul tube. Effectuer la dégradation à l’acide formique conformément à la procédure décrite à la section 4.
- Mesurez les échantillons par LC-MS comme décrit à la section 6 et analysez les données à l’aide du logiciel d’analyse de données avec des paramètres MFE optimisés pour extraire les données contenant la masse, le tR et le volume, comme décrit à l’étape 6.5. L’algorithme typique de traitement et d’appel de base n’est pas appliqué en raison de la complexité considérablement accrue des données résultant du mélange. Toutes les bases de l’ARN de l’échantillon mixte sont appelées manuellement selon une méthode similaire à la section 7.3 et correspondent bien aux bases théoriques de la base de données8 des nucléotides et des modifications de l’ARN, de sorte que les séquences complètes des cinq brins d’ARN de l’échantillon mixte sont lues avec précision. Dans les tableaux S7 à S11, tous les renseignements sont énumérés, y compris la masse observée, tR, le volume, le score de qualité et la différence de masse en ppm.
Résultats
Introduire une étiquette de biotine à l’extrémité 3' de l’ARN pour produire des échelles de masse-tR facilement identifiables. Le flux de travail de l’approche 2D-HELS MS Seq est illustré à la figure 1a. Le marquage hydrophobe à la biotine introduit à l’extrémité 3' de l’ARN (voir section 2) augmente les masses et les tRs des composants de l’échelle marqués 3' par rapport à ceux de leurs homologues non marqués. Ainsi, la courbe de l’échelle 3' est décalée vers des valeurs plus grandes de l’axe des y (en raison de l’augmentation des tRs) et décalée vers des valeurs plus grandes de l’axe des x (en raison de l’augmentation des masses) dans le graphique 2D masse-tR . La figure 1b montre le protocole de préparation de l’échantillon, y compris l’introduction d’un marqueur de biotine à l’extrémité 3' de l’ARN pour le séquençage de la MS 2D-HELS. La figure 1c montre la séparation de l’échelle 3' de l’échelle 5' et d’autres fragments indésirables sur un graphique 2D mass-tR basé sur des changements systématiques de tRdes fragments de l’échelle masse-tR marqués à la biotine 3' de l’ARN #1. La courbe à échelle 3' seule donne une séquence complète de l’ARN #1, et la courbe à échelle 5' qui ne montre pas de décalage tR fournit la séquence inverse, mais elle nécessite un appariement final pour lire la base terminale8. Avec cette stratégie de 2D-HELS, l’appariement final n’est pas nécessaire comme indiqué précédemment et la séquence entière de l’ARN peut être lue complètement à partir d’une seule courbe d’échelle8 marquée. Ainsi, il est possible de séquencer des échantillons mixtes contenant plusieurs ARN, par exemple deux brins d’ARN de longueurs différentes (ARN #1 et ARN #2, 19 nt et 20 nt, respectivement) avec un marqueur de 5'-biotine à chaque ARN (figure 1d).
Conversion de ψ à son adduction CMC-ψ pour 2D-HELS MS Seq. ψ est une modification nucléotidique difficile pour le séquençage basé sur la MS car elle a la même masse que l’uridine (U). Pour différencier ces deux bases l’une de l’autre, nous traitons l’ARN avec CMC, qui convertit un ψ en adduit CMC-ψ (voir Section 5). L’adduit a une masse différente de la vôtre et peut être différencié dans le 2D-HELS MS Seq. La figure 2a montre le profil HPLC du produit brut de la réaction convertissant ψ en son adduit CMC dans l’ARN #6. En intégrant leurs pics UV, nous avons calculé le pourcentage de conversion et 42 % ψ est converti en adduction CMC-ψ après le processus illustré à la section 5. Après la dégradation acide et la mesure LC-MS, nous avons acquis manuellement la séquence basée sur les échelles non converties en CMC et les échelles converties en CMC identifiées à partir des données traitées par algorithme 8,9. Une courbe rouge se ramifie à partir de la courbe grise à partir de ψ à la position 8 dans l’ARN #6 (figure 2b) en raison de la conversion partielle de ψ en adduit CMC-ψ. En raison de la masse et de l’hydrophobicité du CMC, cette conversion entraîne une augmentation de masse de 252,2076 Dalton et une augmentation significative de tR pour chaque composant d’échelle contenant un adduit CMC-ψ par rapport à son homologue non converti. Ainsi, un changement spectaculaire commençant à la position 8 dans l’ARN #6 peut être observé dans le graphique 2D masse-tR, indiquant que la position 8 est bien une ψ dans l’ARN #6.
Séquençage de mélanges d’ARN. Un mélange de cinq brins d’ARN différents est séquencé par l’approche 2D-HELS MS Seq avec marquage à 3' (voir section 8). Le problème pour le séquençage des ARN mixtes est que plusieurs courbes en échelle dans le graphique 2D mass-tR peuvent se chevaucher lorsqu’elles partagent toutes les mêmes points de départ (l’étiquette hydrophobe dans le graphique 2D mass-tR). Cependant, l’appel de base est effectué un par un, chacun basé sur une différence de masse entre deux fragments d’échelle adjacents dans les données MFE. L’appel de base correct peut être effectué tant que chaque différence de masse correspond bien (une différence PPM MS < 10) avec l’une des masses théoriques des nucléotides canoniques ou modifiés dans le pool de données 8,9. Dans l’analyse des échantillons d’ARN multiplexés, l’algorithme typique de traitement et d’appel de base utilisé dans les figures 1 et 2 n’est pas utilisé, principalement en raison de la complexité considérablement accrue des données résultant du mélange. Ces séquences sont appelées manuellement en calculant la différence de masse entre deux fragments d’échelle de masse adjacents et en la comparant à la masse théorique du nucléotide dans le pool de données9. Toute base appariée avec une masse PPM <10 est choisie comme identité de base à cette position. Grâce à ce calcul manuel base par base pour l’appel de base, toutes les séquences du mélange sont séquencées avec précision. Le logiciel OriginLab est utilisé pour reconstruire un graphique 2D mass-tR, dans lequel le tR de départ de chaque séquence est normalisé systématiquement pour mieux visualiser cinq séquences d’ARN différentes (Figure 3). Sans une telle normalisation, les codes alphabétiques (c’est-à-dire A, C, G et U) pour les séquences des cinq ARN seraient regroupés sur le graphique (figure S1), ce qui entraînerait une moins grande facilité de visualisation par rapport à celle rapportée dans la figure 3. Les résultats du séquençage démontrent que l’approche 2D-HELS MS Seq ne se limite pas seulement au séquençage d’ARN simple brin purifiés, mais aussi, plus important encore, à des mélanges d’ARN avec plusieurs brins d’ARN. Des algorithmes sont actuellement en cours de développement pour automatiser le processus d’appel de base et de génération de séquences.
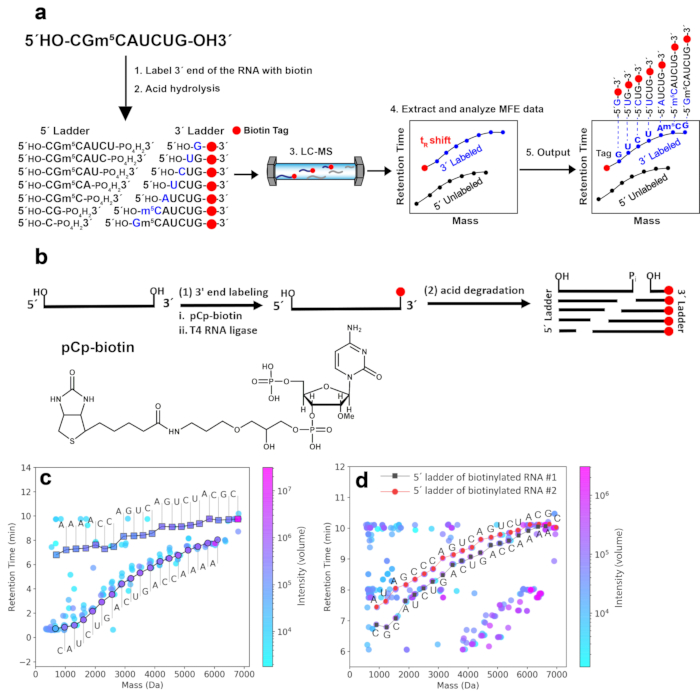
Figure 1. 2D-HELS MS Séquençage d’échantillons d’ARN représentatifs. (a) Flux de travail pour 2D-HELS MS Seq. Les principales étapes comprennent 1) le marquage hydrophobe de l’ARN à séquencer, 2) l’hydrolyse acide, 3) la mesure LC-MS, 4) l’extraction et l’analyse des données MFE, et 5) la génération de séquences via des algorithmes ou un calcul manuel. (b) Protocole de préparation de l’échantillon, y compris l’introduction d’une étiquette de biotine à l’extrémité 3' de l’ARN pour le séquestrage 2D-HELS MS (C) Séparation de l’échelle 3' de l’échelle 5' et d’autres fragments indésirables dans un graphique 2D du temps de rétention de la masse (tR) basé sur des changements systématiques de tRdes fragments del’échelle de l’ARN #1 marqués à la biotine 3' (19 nt). Les séquences sont de novo et lues automatiquement directement par un algorithme d’appel de base9. (d) Séquençage simultané de l’ARN #1 et de l’ARN #2 marqués à la 5'-biotine, 19 nt et 20 nt, respectivement. Les méthodes d’introduction d’un marqueur de biotine à l’extrémité 5' de l’ARN sont différentes de celles de la biotinylation 3' et peuvent être trouvées dans le protocole9 précédemment publié. L’extrémité 5' de deux ARN (ARN #1 et ARN #2) est biotinylée et leurs échelles 5'-biotinylées peuvent être facilement identifiées ; les deux échelles biotinylées 5' sont facilement séparées de leurs échelles 3' non marquées dans le graphique 2D mass-tR après LC-MS, car les composants de l’échelle biotinylés ont les plus grands décalages tR en raison de l’hydrophobicité de la biotine, tandis que les composants de l’échelle non marqués sont dans la région inférieure tR. Bien que les échelles 5' et 3' coexistent, elles n’interfèrent pas avec l’interprétation de la séquence de deux brins d’ARN mixtes. Chaque séquence de ces deux ARN est acquise manuellement à partir d’échelles biotinylées 5' sur la base des données traitées par algorithme de calcul 8,9. Cette figure a été modifiée de Zhang et al.9. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Graphique 2. Conversion de la pseudouridine (ψ) en son adduit pour 2D-HELS MS Seq. (a) Profil HPLC du produit brut de la réaction convertissant ψ en son adduit CMC dans un ARN de 20 nt (ARN #6) qui contient un ψ. (b) Séquençage d’un ARN contenant du ψ #6. La conversion du ψ en adduits CMC-ψ (ψ*) entraîne une augmentation de masse de 252,2076 Dalton et une augmentation significative de tR en raison de sa masse et de son hydrophobicité du CMC. Ainsi, un décalage spectaculaire commençant à la position 8 peut être observé dans le graphique mass-tR, indiquant qu’il s’agit d’un ψ à la position 8 dans la séquence d’ARN. Les séquences sont acquises manuellement sur la base des données traitées par algorithme de calcul 8,9. Cette figure a été modifiée de Zhang et al.9. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Graphique 3. Séquençage de mélanges d’ARN contenant cinq ARN distincts. Une biotine est utilisée pour marquer chaque ARN à son extrémité 3' avant le séquençage 2D-HELS MS. Pour chaque séquence, les valeurs tR de départ sont normalisées systématiquement pour commencer à des intervalles de 7 minutes pour faciliter la visualisation. Les différences absolues entre la valeur de départ de tR et lest Rsuivantes restent inchangées pour chacun des cinq ARN, et il est donc plus facile de visualiser chacun d’entre eux dans le même graphique. Toutes les bases sont identifiées en calculant manuellement les différences de masse de deux composants de l’échelle adjacents et en les faisant correspondre avec les différences de masse théoriques dans la base de données de nucléotides et de modifications de l’ARN8 ; les tracés de la figure 3 sont reconstruits à l’aide d’OriginLab sur la base de données manuelles d’appel de base et de séquençage (voir la section sur le séquençage des mélanges d’ARN dans Résultats représentatifs). La figureR de masse 2D des cinq ARN mixtes sans normalisation tR est illustrée à la figure S1. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Graphique S1. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Contrairement à la fragmentation MS en tandem, l’hydrolyse acide hautement contrôlée est utilisée dans l’approche 2D-HELS MS Seq pour fragmenter l’ARN avant l’analyse avec un spectromètrede masse 9,10. En conséquence, chaque fragment dégradé à l’acide peut être détecté par l’instrument, formant l’équivalent d’une échelle de séquençage. Dans des conditions optimales, cette méthode crée une échelle de séquence « idéale » à partir de l’ARN via, en moyenne, un clivage d’ARN spécifique au site de la molécule exclusivement au niveau d’une liaison phosphodiester 8,9,10. Une fois que chaque fragment dégradé a été mesuré par le spectromètre de masse en une seule fois, la différence de masse entre deux fragments en échelle adjacents correspond à la masse exacte du nucléotide d’ARN ou de la modification à cette position. Chaque modification de l’ARN a soit une masse intrinsèque unique qui peut aider à l’identifier et à la localiser dans l’ARN, soit peut être convertie en une modification avec une masse unique. Ainsi, en théorie, cette méthode peut rapporter l’identité et l’emplacement des nucléotides canoniques et modifiés pour le séquençage de novo et direct de tout ARN. Cependant, différentes échelles de séquences peuvent se chevaucher, ce qui complique l’analyse des données de MS et rend difficile le séquençage de l’ARN par MS dans la pratique.
L’un des avantages de l’étiquette 3'-hydrophobe est qu’elle surmonte un défi majeur dans toute méthode de fragmentation, c’est-à-dire que chaque molécule d’ARN doit être clivée en exactement deux fragments (et idéalement pas plus) : un fragment contenant l’extrémité 5' d’origine, et l’autre contenant l’extrémité 3' d’origine de l’ARN. Par conséquent, chaque événement de clivage produit deux fragments, produisant deux échelles, l’une mesurée à partir de l’extrémité 5' et l’autre à partir de l’extrémité 3'. Il y a toujours une ambiguïté dans la détermination de quel pic de MS appartient à quelle échelle. Cela devient plus problématique dans un mélange de plusieurs ARN différents, en raison de la génération d’un grand nombre d’échelles de séquences qui se chevauchent. Cependant, comme tous les fragments d’échelle des extrémités 3' sont marqués d’une étiquette hydrophobe, ils présentent des tRs beaucoup plus longs (figure 1a). En conséquence, nous pouvons obtenir des échelles claires et sans ambiguïté dans les données 2D mass-tR exclusivement dérivées de l’ARN marqué en 3'. Notamment, nous optimisons les approches pour marquer sélectivement l’extrémité 5' ou 3' de tout ARN en utilisant différentes méthodes de conjugaison chimique9. Nous pouvons également effectuer un séquençage bidirectionnel, qui n’est pas utilisé pour déterminer la ou les bases terminales ici, mais est utilisé pour fournir des informations de séquence identiques deux fois lors de la lecture à partir des directions 5' et 3' (c’est-à-dire la vérification du séquençage bidirectionnel), améliorant ainsi encore la précision du séquençage.
Pour le séquençage de novo d’échantillons d’ARN inconnus, en particulier pour les échantillons biologiques complexes, un algorithme général et robuste est nécessaire pour traiter une grande quantité de données LC-MS pour la génération de séquences d’une manière précise et efficace, ce qui est récemment devenu disponible via d’autres travaux publiés10. Bien que ces algorithmes aient été utilisés pour le séquençage d’échantillons plus complexes10, dans cette étude, nous avons effectué un appel manuel de base pour la génération de séquences, sauf indication contraire. Notre objectif est de couvrir toutes les étapes clés du 2D-HELS MS Seq, et nous aimerions illustrer le processus au cours duquel, même sans utiliser d’algorithmes de séquençage supplémentaires, nous pouvons toujours lire manuellement les séquences de l’ARN à séquencer. Pour faciliter la visualisation et pour identifier plus rapidement les fragments d’échelle nécessaires au séquençage dans le tracé 2D mass-tR , les fichiers MFE de chaque exécution LC-MS sont traités par une version révisée d’un algorithme publié8 avant de lire leurs séquences, sauf indication contraire. L’algorithme publié ne peut pas être utilisé directement pour lire les séquences à partir des données LC-MS, mais une partie de sa fonction peut toujours être utilisée pour traiter les données - le regroupement hiérarchique des adduits de masse à travers cet algorithme augmentera l’intensité de chaque composant de l’échelle, ce qui à son tour réduira la complexité des données, en particulier dans la région cruciale où les lectures de séquences sont générées8, 9. Planche à billets
L’une des étapes cruciales de la préparation des échantillons pour le MS Seq 2D-HELS permet d’améliorer l’efficacité de l’étiquetage hydrophobe de l’ARN. Une efficacité de marquage élevée peut aider à réduire la quantité d’échantillon d’ARN nécessaire pour générer des signaux MS sur lesquels reposent les données de séquence. Afin d’augmenter l’efficacité du marquage, nous employons de nouvelles stratégies de marquage, notamment l’utilisation de la biotine AppCp-activée pour éviter l’étape d’adénylation lors du marquage de l’extrémité 3' de l’ARN. Le rendement de la réaction de marquage de l’extrémité 3' d’un ARN de 19 nt avec de la biotine (voir étape 2.2) peut être amélioré de 60 % à ~95 %9 en utilisant cette méthode en une étape. Grâce à un marquage efficace, nous sommes en mesure de séquencer un échantillon mixte contenant jusqu’à 12 ARN distincts, comme décrit précédemment9. Dans cette étude, nous utilisons un mélange de cinq ARN comme exemple représentatif pour illustrer le processus de séquençage. Nous détectons également tous les fragments d’échelle nécessaires à un séquençage précis et lisons les séquences complètes de chacune des cinq séquences d’ARN du mélange. Une efficacité d’étiquetage plus élevée permet non seulement de minimiser la quantité de charge d’échantillons, mais aussi de réduire considérablement la complexité des données lors de l’analyse des données en aval pour la génération de séquences. De nouvelles réactions sont actuellement en cours de développement pour obtenir un rendement quantitatif dans le marquage des ARN aux extrémités 5' et 3'.
Lors du séquençage de l’ARN #1 comme le montre la figure 1c, les étapes de capture et de libération de la streptavidine sont utilisées pour séparer physiquement l’ARN biotinylé #1 avant la dégradation acide (voir la section 3). Cela permet d’éliminer une petite partie de l’ARN non marqué, ce qui permet par la suite une plus grande facilité d’identification visuelle des échelles de masse marquées dans le graphique 2D mass-tR . Cependant, l’étape de séparation physique n’est pas obligatoire car les fragments de l’échelle d’ARN biotinylé ont des tRretardés/plus longs en raison de l’hydrophobicité de l’étiquette de biotine par rapport à leurs homologues non marqués. De plus, l’appel de base ne repose pas sur la séparation physique, mais sur les différences de masse des composants adjacents de l’échelle de masse, ainsi, l’appel de base correct peut être obtenu tant que les différences de masse de deux composants adjacents de l’échelle correspondent bien aux masses correspondantes d’un nucléotide particulier ou à la modification du nucléotide d’ARN et à la date de modification8. Un algorithme de calcul est actuellement en cours de développement pour automatiser les appels de base et la génération de séquences.
Les paramètres MFE lors de l’exportation des données LC-MS d’origine (dans le type de fichier .d) dans des fichiers tableur sont très importants pour le traitement des données et la génération ultérieure de séquences (voir section 6.5). Par exemple, nous avons testé le réglage MFE « pic avec hauteur » dans une plage de 100 à 1000 et avons remarqué que le réglage de 100 peut nous fournir 2 fois plus de composés que ceux du réglage 1000. Afin d’éviter de manquer des composants de l’échelle, nous pouvons ajuster le paramètre MFE pendant le flux de travail de séquençage. Ce paramètre dépend probablement de la résolution de masse de l’instrument, de la quantité de fragments d’échelle de masse et de la complexité des données. De plus, il est important d’utiliser l’ensemble de données du centroïde et le paramètre de type chromatographique pour les petites molécules. Le score de qualité peut varier de 50 % à 100 % en fonction de la qualité des données.
L’instrument LC-MS que nous utilisons dans l’étude a une résolution de masse supérieure de ~40K, limitant la méthode au séquençage de l’ARN de moins de 35 bases. Cependant, la longueur de lecture exacte de cette méthode dépend de l’instrument ; Des instruments plus avancés avec un pouvoir de résolution plus élevé peuvent conduire à une longueur de lecture plus longue. De même, le débit, c’est-à-dire le nombre de séquences d’ARN pouvant être séquencées simultanément en une seule exécution LC-MS, reste à explorer, bien que nous ayons séquencé manuellement un mélange d’échantillons d’ARN jusqu’à 12 brins d’ARN distincts, même sans utiliser d’algorithme9. Avec le flux de travail actuel, ~100 pmol d’ARN court (<35 nt) sont nécessaires pour chaque exécution LC-MS. La quantité de charge augmente lorsque des expériences supplémentaires sont nécessaires : pour différencier les modifications de nucléotides isomères, jusqu’à 400 pmol d’ARN sont généralement nécessaires. Pour le séquençage d’ARNt spécifiques comme l’ARNtPhe, ~1000 pmol d’échantillon peuvent être nécessaires pour le séquençage et l’analyse de modification. Cependant, nous nous attendons à ce que les quantités de charge d’échantillon requises soient réduites sur les instruments LC-MS ayant une plus grande sensibilité. Grâce à l’amélioration de l’efficacité du marquage des échantillons, de l’algorithme de séquençage, de la sensibilité et de la résolution des instruments, nous nous attendons à ce que notre méthode soit applicable à un plus large éventail d’échantillons d’ARN, en particulier ceux présentant diverses modifications de l’ARN.
Déclarations de divulgation
Les auteurs ont déposé un brevet provisoire lié à la technologie discutée dans ce manuscrit.
Remerciements
Les auteurs reconnaissent la subvention R21 des National Institutes of Health (1R21HG009576) à S. Z. et W. L. et les subventions de soutien institutionnel à la recherche et à la créativité du New York Institute of Technology (NYIT) à S. Z., qui ont soutenu ce travail. Les auteurs tiennent à remercier le doctorant Xuanting Wang (Université Columbia) pour son aide à la création de figures, et remercient le professeur Michael Hadjiargyrou (NYIT), le professeur Jingyue Ju (Université Columbia), les Drs James Russo, Shiv Kumar, Xiaoxu Li, Steffen Jockusch et d’autres membres du laboratoire Ju (Université Columbia), le Dr Yongdong Wang (Cerno Bioscience), Meina Aziz (NYIT) et Wenhao Ni (NYIT) pour les discussions et suggestions utiles pour notre manuscrit.
matériels
| Name | Company | Catalog Number | Comments |
| 5' DNA Adenylation kit | New England Biolabs | E2610S | 50uM concentration |
| 6550 Q-TOF mass spectrometer | Agilent Technologies | 5991-2116EN | Coupled to a 1290 Infinity LC system |
| A(5´)pp(5´)Cp-TEG-biotin-3´ | ChemGenes | 91718 | HPLC purified |
| ATPγS | Sigma-Aldrich | 11162306001 | Lithium salt |
| Bicine | Sigma-Aldrich | B8660 | BioXtra, ≥99% (titration) |
| Biotin maleimide | Vector Laboratories | SP-1501 | Long arm |
| C18 column | Waters | 186003532 | 50 mm × 2.1 mm Xbridge C18 column with a particle size of 1.7 μm |
| Centrifugal Vacuum Concentrator | Labconco | Refrig 115v/60hz 7310022 | Labconco CentriVap |
| ChemBioDraw | PerkinElmer | ChemDraw Prime | Generate a chemical structure and property data of structures & fragments |
| CMC (N-cyclohexyl-N?-(2-morpholinoethyl)-carbodiimide metho-p-toluenesulfonate) | Sigma-Aldrich | 2491-17-0 | 95% Purifiy |
| Cyanine3 maleimide (Cy3) | Lumiprobe | 11080 | Water insoluble |
| DEPC-treated water | Thermo Fisher Scientific | AM9906 | Autoclaved, certified nuclease-free |
| Diisopropylamine (DIPA) | Thermo Fisher Scientific | 108-18-9 | 99% Alfa Aesar |
| DMSO | Sigma-Aldrich | 276855 | Anhydrous dimethyl sulfoxide, 99.9% |
| EDTA | Sigma-Aldrich | E6758 | Anhydrous, crystalline, BioReagent, suitable for cell culture |
| Formic acid | Merck | 64-18-6 | 98-100%, ACS reag, Ph Eur |
| Hexafluoro-2-propanol (HFIP) | Thermo Fisher Scientific | 920-66-1 | 99% Acros Organics |
| LC-MS sample vials | Thermo Fisher Scientific | C4000-11 | Plastic screw thread vials |
| LC-MS vial caps | Thermo Fisher Scientific | C5000-54A | Autosampler vial screw thread caps |
| Na2CO3 buffer | Sigma-Aldrich | 88975 | BioUltra, >0.1 M Na2CO3, >0.2 M NaHCO3 |
| Oligo Clean & Concentrator | Zymo Research | D4060 | Spin column |
| OriginLab | OriginLab | OriginPro | Data analysis and graphing software |
| pCp-biotin | TriLink BioTechnologies | NU-1706-BIO | 20 ul (1 mM) |
| RNA #1--#6 | Integrated DNA Technologies | Custom RNA oligos | 19nt-21nt single-stranded RNAs, used without further purification |
| Rocking platform shaker | VWR | Orbital Shaker Standard 1000 | Speed Range 40 to 300 rpm |
| Streptavidin magnetic beads | Thermo Fisher Scientific | 88816 | Binding approx. 55ug biotinylated rabbit lgG per mg of beads |
| Sulfonated Cyanine3 maleimide | Lumiprobe | 11380 | Water soluble |
| T4 DNA ligase 1 | New England Biolabs | M0202S | 400 units/uL |
| T4 polynucleotide kinase | Sigma-Aldrich | T4PNK-RO | From phage T4 am N81 pse T1 infected Escherichia coli BB |
| Tris-HCl buffer | Sigma-Aldrich | T6455 | Tris-HCl Buffer, pH 10, 10×, Antigen Retriever |
| Urea | Sigma-Aldrich | 81871 | Urea for synthesis. CAS No. 57-13-6, EC Number 200-315-5. |
Références
- Addepalli, B., Venus, S., Thakur, P., Limbach, P. A. Novel ribonuclease activity of cusativin from Cucumis sativus for mapping nucleoside modifications in RNA. Analytical and Bioanalytical Chemistry. 409 (24), 5645-5654 (2017).
- Gao, H., Liu, Y., Rumley, M., Yuan, H., Mao, B. Sequence confirmation of chemically modified RNAs using exonuclease digestion and matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 23 (21), 3423-3430 (2009).
- McLuckey, S. A., Van Berkel, G. J., Glish, G. L. Tandem mass spectrometry of small, multiply charged oligonucleotides. Journal of The American Society for Mass Spectrometry. 3 (1), 60-70 (1992).
- Fountain, K. J., Gilar, M., Gebler, J. C. Analysis of native and chemically modified oligonucleotides by tandem ion-pair reversed-phase high-performance liquid chromatography/electrospray ionization mass spectrometry. Rapid Communications in Mass Spectrometry. 17 (7), 646-653 (2003).
- Taucher, M., Breuker, K. Characterization of modified RNA by top-down mass spectrometry. Angewandte Chemie International Edition in English. 51 (45), 11289-11292 (2012).
- Kellner, S., Burhenne, J., Helm, M. Detection of RNA modifications. RNA Biology. 7 (2), 237-247 (2010).
- Thomas, B., Akoulitchev, A. V. Mass spectrometry of RNA. Trends in Biochemical Sciences. 31 (3), 173-181 (2006).
- Bjorkbom, A., et al. Bidirectional direct sequencing of noncanonical RNA by two-dimensional analysis of mass chromatograms. Journal of the American Chemical Society. 137 (45), 14430-14438 (2015).
- Zhang, N., et al. A general LC-MS-based RNA sequencing method for direct analysis of multiple-base modifications in RNA mixtures. Nucleic Acids Research. 47 (20), 125 (2019).
- Zhang, N., et al. Direct sequencing of tRNA by 2D-HELS-AA MS Seq reveals its different isoforms and dynamic base modifications. ACS Chemical Biology. 15 (6), 1464-1472 (2020).
- Bakin, A., Ofengand, J. Four newly located pseudouridylate residues in Escherichia coli 23S ribosomal RNA are all at the peptidyltransferase center: analysis by the application of a new sequencing technique. Biochemistry. 32 (37), 9754-9762 (1993).
- Cantara, W. A., et al. The RNA Modification Database, RNAMDB: 2011 update. Nucleic Acids Research. 39 (Database issue), D195-D201 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon