Method Article
2D-HELS MS Seq: Eine allgemeine LC-MS-basierte Methode zur direkten und de novo Sequenzierung von RNA-Gemischen mit unterschiedlichen Nukleotidmodifikationen
In diesem Artikel
Zusammenfassung
Hier beschreiben wir ein detailliertes Protokoll für eine LC-MS-basierte Sequenzierungsmethode, die als direkte Methode zur Sequenzierung kurzer RNA (<35 nt pro Lauf) ohne cDNA-Zwischenprodukt und als allgemeine Methode zur Sequenzierung verschiedener Nukleotidmodifikationen in einer einzigen Studie mit Einzelbasenpräzision verwendet werden kann.
Zusammenfassung
Massenspektrometrie (MS)-basierte Sequenzierungsansätze haben sich bei der direkten Sequenzierung von RNA als nützlich erwiesen, ohne dass ein komplementäres DNA-Zwischenprodukt (cDNA) erforderlich ist. Solche Ansätze werden jedoch selten als De-novo-RNA-Sequenzierungsmethode angewendet, sondern hauptsächlich als Werkzeug verwendet, das bei der Qualitätssicherung zur Bestätigung bekannter Sequenzen von gereinigten einzelsträngigen RNA-Proben helfen kann. Vor kurzem haben wir eine direkte RNA-Sequenzierungsmethode entwickelt, indem wir eine 2-dimensionale hydrophobe Endmarkierungsstrategie mit Massenretentionszeit in die MS-basierte Sequenzierung (2D-HELS MS Seq) integriert haben. Diese Methode ist in der Lage, sowohl einzelne RNA-Sequenzen als auch Mischungen mit bis zu 12 verschiedenen RNA-Sequenzen genau zu sequenzieren. Zusätzlich zu den vier kanonischen Ribonukleotiden (A, C, G und U) ist das Verfahren in der Lage, RNA-Oligonukleotide zu sequenzieren, die modifizierte Nukleotide enthalten. Dies ist möglich, weil die modifizierte Nukleobase entweder eine intrinsisch einzigartige Masse hat, die bei ihrer Identifizierung und ihrer Position in der RNA-Sequenz helfen kann, oder in ein Produkt mit einer einzigartigen Masse umgewandelt werden kann. In dieser Studie haben wir RNA verwendet, die zwei repräsentative modifizierte Nukleotide (Pseudouridin (Ψ) und 5-Methylcytosin (m5C)) enthält, um die Anwendung der Methode zur De-novo-Sequenzierung eines einzelnen RNA-Oligonukleotids sowie einer Mischung von RNA-Oligonukleotiden mit jeweils unterschiedlicher Sequenz und/oder modifizierten Nukleotiden zu veranschaulichen. Die hier beschriebenen Verfahren und Protokolle zur Sequenzierung dieser Modell-RNAs sind auf andere kurze RNA-Proben (<35 nt) anwendbar, wenn ein hochauflösendes Standard-LC-MS-System verwendet wird, und können auch zur Sequenzverifizierung von modifizierten therapeutischen RNA-Oligonukleotiden verwendet werden. In Zukunft könnte diese Methode mit der Entwicklung robusterer Algorithmen und besserer Instrumente die Sequenzierung komplexerer biologischer Proben ermöglichen.
Einleitung
Massenspektrometrie (MS)-basierte Sequenzierungsmethoden, einschließlich Top-down-MS und Tandem-MS 1,2,3,4, wurden für die direkte Sequenzierung von RNA entwickelt. In-situ-Fragmentierungstechniken zur effektiven Erzeugung hochwertiger RNA-Leitern in Massenspektrometern können jedoch derzeit nicht auf die De-novo-Sequenzierung angewendet werden 5,6. Darüber hinaus ist es nicht sehr trivial, die traditionellen eindimensionalen (1D) MS-Daten für die De-novo-Sequenzierung auch nur einer gereinigten RNA-Sequenz zu analysieren, und es wäre eine noch größere Herausforderung für die MS-Sequenzierung von gemischten RNA-Proben 7,8. Aus diesem Grund wurde eine zweidimensionale (2D) Flüssigchromatographie (LC)-MS-basierte RNA-Sequenzierungsmethode entwickelt, die die Herstellung von 2D-Massenretentionszeitleitern (tR) umfasst, um 1D-Massenleitern zu ersetzen, was die Identifizierung von Leiterkomponenten, die für die De-novo-Sequenzierung von RNAs benötigt werden, erheblich erleichtert8. Die 2D-LC-MS-basierte RNA-Sequenzierungsmethode ist jedoch hauptsächlich auf gereinigte synthetische Kurz-RNA beschränkt, da sie keine vollständige Sequenz allein auf der Grundlage einer einzigen Leiter lesen kann, sondern sich auf zwei nebeneinander existierende Leitern (5'- und 3'-Leitern) stützen muss8. Genauer gesagt erfordert dieser Ansatz bidirektionale Paired-End-Reads zum Lesen von terminalen Nukleobasen in der Region mit geringer Masse8. Die zusätzliche Komplexität des Paired-End-Readings führt dazu, dass diese Methode für die Sequenzierung von RNA-Gemischen unhaltbar ist, da bei den unbekannten Proben Verwirrung darüber entsteht, welches Leiterfragment zu welcher Leiter gehört.
Um die oben genannten Barrieren bei MS-basierten RNA-Sequenzierungsansätzen zu überwinden und solche Anwendungen in der direkten RNA-Sequenzierung zu erweitern, müssen zwei Fragen angegangen werden: 1) wie kann eine qualitativ hochwertige Massenleiter generiert werden, mit der eine vollständige Sequenz vom ersten bis zum letzten Nukleotid in einem RNA-Strang gelesen werden kann, und 2) wie kann jede RNA/Massenleiter in einem komplexen MS-Datensatz effektiv identifiziert werden. Zusammen mit einem gut kontrollierten Säureabbau haben wir eine neue Sequenzierungsmethode entwickelt, indem wir eine hydrophobe Endmarkierungsstrategie (HELS) in die MS-basierte Sequenzierungstechnik eingeführt haben, und diese beiden Probleme erfolgreich gelöst, indem wir entweder am 5'- und/oder 3'-Ende der zu sequenzierenden RNAs einen hydrophoben Tag hinzugefügt haben9. Diese Methode erzeugt eine "ideale" Sequenzleiter aus RNA – jedes Leiterfragment stammt ausschließlich an jeder Phosphodiesterbindung aus der ortsspezifischen RNA-Spaltung, und die Massendifferenz zwischen zwei benachbarten Leiterfragmenten ist die exakte Masse des Nukleotids oder der Nukleotidmodifikation an dieser Position 8,9,10. Dies ist möglich, weil wir einen hochgradig kontrollierten sauren Hydrolyseschritt einbauen, bei dem die RNA durchschnittlich einmal pro Molekül fragmentiert wird, bevor sie in das Instrument injiziert wird. Als Ergebnis wird jedes Abbaufragmentprodukt auf dem Massenspektrometer detektiert und alle Fragmente zusammen bilden eine Sequenzierungsleiter 8,9,10. Diese neue Strategie ermöglicht das vollständige Lesen einer RNA-Sequenz von einer einzigen Leiter eines RNA-Strangs ohne Paired-End-Lesen von der anderen Leiter der RNA und ermöglicht zusätzlich die MS-Sequenzierung von RNA-Gemischen mit mehreren verschiedenen Strängen, die kombinatorische Nukleotidmodifikationen enthalten9. Durch Hinzufügen eines Tags am 5'- und/oder 3'-Ende der RNA weisen die markierten Leiterfragmente eine signifikante Verzögerung von tR auf, was dazu beitragen kann, die beiden Massenleitern voneinander und auch von der verrauschten Region mit geringer Masse zu unterscheiden. Die durch das Hinzufügen des hydrophoben Tags verursachte Verschiebung von mass-tR erleichtert die Identifizierung der Massenleiter und vereinfacht die Datenanalyse für die Sequenzgenerierung. Darüber hinaus kann die Hinzufügung des hydrophoben Tags dazu beitragen, die terminale Basis in dem Strang zu identifizieren, indem verhindert wird, dass sich das entsprechende Leiterfragment aufgrund der durch den Tag verursachten Massen- und Hydrophobizitätszunahme in der verrauschten R-Region mit geringer Masse befindet, wodurch die Identifizierung der vollständigen Sequenz einer RNA von einer einzigen Leiter ermöglicht wird; Es sind keine Lesevorgänge mit gekoppelten Enden erforderlich. Als Ergebnis haben wir bereits die erfolgreiche Sequenzierung einer komplexen Mischung von bis zu 12 unterschiedlichen RNA-Strängen ohne den Einsatz eines fortschrittlichen Sequenzierungsalgorithmus9 demonstriert, was die Tür für die De-novo-MS-Sequenzierung von RNA öffnet, die sowohl kanonische als auch modifizierte Nukleotide enthält, und sie für die Sequenzierung von gemischten und komplexeren RNA-Proben praktikabler macht. Mit 2D-HELS MS Seq haben wir sogar erfolgreich eine gemischte Population von tRNA-Probensequenziert 10 und weiten die Anwendung aktiv auf andere komplexe RNA-Proben aus.
Um 2D-HELS MS Seq die direkte Sequenzierung eines breiteren Spektrums von RNA-Proben zu erleichtern, konzentrieren wir uns hier auf die technischen Aspekte dieses Sequenzierungsansatzes und decken alle wesentlichen Schritte ab, die bei der Anwendung der Technik zur direkten Sequenzierung von RNA-Proben erforderlich sind. Zur Veranschaulichung der Sequenzierungstechnik werden spezifische Beispiele verwendet, einschließlich synthetischer Einzel-RNA-Sequenzen, Mischungen aus mehreren unterschiedlichen RNA-Sequenzen und modifizierter RNAs, die sowohl kanonische als auch modifizierte Nukleotide wie Pseudouridin (ψ) und 5-Methylcytosin (m5C) enthalten. Da alle RNAs Phosphodiesterbindungen enthalten, kann jede Art von RNA säurehydrolysiert werden, um eine ideale Sequenzleiter für 2D-HELS MS Seq unter optimalen Bedingungen zu erzeugen 8,9. Der Nachweis aller Leiterfragmente einer gegebenen RNA ist jedoch instrumentenabhängig. Bei einer standardmäßigen hochauflösenden LC-MS (40 K) beträgt die minimale Lademenge für die Sequenzierung einer gereinigten kurzen RNA-Probe (<35 nt) 100 pmol pro Lauf. Es wird jedoch mehr Material benötigt (bis zu 400 pmol pro RNA-Probe), wenn zusätzliche Experimente durchgeführt werden müssen (z. B. zur Unterscheidung von isomeren Basenmodifikationen mit identischen Massen). Das Protokoll, das bei der Sequenzierung der synthetischen modifizierten RNAs des Modells verwendet wird, wird auch für die Sequenzierung breiterer RNA-Proben anwendbar sein, einschließlich biologischer RNA-Proben mit unbekannten Basenmodifikationen. Es ist jedoch eine noch größere Probenmenge, wie z. B. 1000 pmol für die Sequenzierung von tRNA (~76 nt) mit einem Standard-LC-MS-Instrument, erforderlich, um die vollständige tRNA mit allen Modifikationen zu sequenzieren, und für die De-novo-Sequenzierung muss ein fortschrittlicher Algorithmus entwickelt werden10.
Protokoll
1. Entwerfen Sie RNA-Oligonukleotide
- Entwicklung synthetischer RNA-Oligonukleotide mit unterschiedlichen Längen (19 nt, 20 nt und 21 nt), darunter eines (RNA #6) mit kanonischen und modifizierten Nukleotiden. ψ wird als Modell für nicht-massenverändernde Modifikationen verwendet, was für die MS-Sequenzierung eine Herausforderung darstellt, da es eine identische Masse wieU. m 5C hat, wird als Modell für massenverändernde Modifikationen gewählt, um die Robustheit des Ansatzes zu demonstrieren.
RNA #1: 5'-HO-CGCAUCUGACUGACCAAAA-OH-3'
RNA #2: 5'-HO-AUAGCCCAGUCAGUCUACGC-OH-3'
RNA #3: 5'-HO-AAACCGUUACCAUUACUGAG-OH-3'
RNA #4: 5'-HO-GCGUACAUCUUCCCCUUUAU-OH-3'
RNA #5: 5'-HO-GCGGAUUUAGCUCAGUUGGGA-OH-3'
RNA #6: 5'-HO-AAACCGUψACCAUUAm5CUGAG-OH-3'
- Jede synthetische RNA wird in nukleasefreiem Diethylpyrocarbonat (DEPC)-behandeltem Wasser (ausgedrückt als DEPC-behandeltes H2O, sofern nicht anders angegeben) gelöst, um eine 100 mM RNA-Stammlösung zu erhalten. Stammlösungen werden langfristig bei -20 °C gelagert.
- Um einen möglichen Abbau der RNA-Probe zu vermeiden, verwenden Sie RNase-freies experimentelles Verbrauchsmaterial, einschließlich DEPC-behandeltem Wasser, Mikrozentrifugenröhrchen und Pipettenspitzen. Wischen Sie die Oberflächen von Laborbedarf häufig mit RNase-Eliminierungstüchern ab.
2. Markieren Sie das 3'-Ende von RNAs mit Biotin
- Zweistufiges Reaktionsprotokoll (Adenylierung und Ligation)
- Geben Sie 1 μl 10x Adenylierungsreaktionspuffer mit 50 mM Natriumacetat, pH 6,0, 10 mM MgCl2, 5 mM Dichlordiphenyltrichlorethan (DTT), 0,1 mM Ethylendiamintetraessigsäure (EDTA), 1 μl 1 mM ATP, 1 μl 100 μM biotinyliertes Cytidinbisphosphat (pCp-Biotin), 1 μl 50 μM Mth RNA-Ligase und 6 μl DEPC-behandeltes H2O (ein Gesamtvolumen von 10 μl) in ein RNase-freies, dünnwandiges 0,2 mL PCR-Röhrchen.
HINWEIS: Lagern Sie die Reagenzien vor der zweistufigen Reaktion bei -20 °C. Tauen Sie die Reagenzien bei Raumtemperatur auf und mischen Sie sie gut, indem Sie sie vortexen und zentrifugieren, bevor Sie sie der Reaktion hinzufügen. - Inkubieren Sie die Reaktion in einem PCR-Gerät bei 65 °C für 1 h und inaktivieren Sie die Reaktion bei 85 °C für 5 min.
- Führen Sie den Ligationsschritt in einem RNase-freien, dünnwandigen 0,2-ml-PCR-Röhrchen durch, das 10 μl Reaktionslösung aus dem vorherigen Schritt enthält, indem Sie 3 μl 10x T4-RNA-Ligase-Reaktionspuffer hinzufügen, der 50 mM Tris(hydroxymethyl)aminomethan (Tris)-HCl, pH 7,8, 10 mM MgCl2, 1 mM DTT, 1,5 μl des 100 mM-Probenbestands der zu sequenzierenden RNA enthält. 3 μl wasserfreies Dimethylsulfoxid (DMSO) zum Erreichen von 10 % (v/v), 1 μl T4-RNA-Ligase (10 Einheiten/μl) und 11,5 μl DEPC-behandeltes H2O (für ein Gesamtvolumen von 30 ml). Inkubieren Sie die Reaktion über Nacht bei 16 °C in einer PCR-Maschine.
HINWEIS: Kombinieren Sie die Reaktionskomponenten aufgrund des hohen Gefrierpunkts von DMSO (18,45 °C) bei Raumtemperatur. - Die Reaktion wird über Nacht bei 16 °C inkubiert.
- Quenden und reinigen Sie die Reaktion durch Säulenreinigung, um Enzyme und freies pCp-Biotin mit dem Oligo Clean & Concentrator (Zymo Research, Irvine, CA, USA) zu entfernen. Oligo-Bindungspuffer, DNA-Waschpuffer, Spin-Säulen und Sammelröhrchen sind im Kit enthalten. Geben Sie 20 mL DEPC-behandeltes H2O in die Reaktionslösung, um ein Probenvolumen von 50 mL zu erreichen, bevor Sie den Bindungspuffer hinzufügen.
- Geben Sie 100 ml Bindungspuffer zu jeder Reaktionslösung. Fügen Sie 400 μl Ethanol hinzu, mischen Sie durch Pipettieren und geben Sie die Mischung in die Säule. Zentrifugieren Sie bei 10.000 x g für 30 s. Verwerfen Sie den Durchfluss.
- Geben Sie 750 μl DNA Wash Buffer in die Säule. Zentrifugieren Sie bei 10.000 x g und maximaler Geschwindigkeit für 30 s bzw. 1 Minute.
- Übertragen Sie die Säule in ein 1,5-ml-Mikrozentrifugenröhrchen. Geben Sie 15 μl DEPC-behandeltes H2O in die Säule und zentrifugieren Sie bei 10.000 x g für 30 s, um das RNA-Produkt zu eluieren.
HINWEIS: Die Proben können zu diesem Zeitpunkt bei -20 °C gelagert werden, bis der nächste Schritt durchgeführt wird.
- Geben Sie 1 μl 10x Adenylierungsreaktionspuffer mit 50 mM Natriumacetat, pH 6,0, 10 mM MgCl2, 5 mM Dichlordiphenyltrichlorethan (DTT), 0,1 mM Ethylendiamintetraessigsäure (EDTA), 1 μl 1 mM ATP, 1 μl 100 μM biotinyliertes Cytidinbisphosphat (pCp-Biotin), 1 μl 50 μM Mth RNA-Ligase und 6 μl DEPC-behandeltes H2O (ein Gesamtvolumen von 10 μl) in ein RNase-freies, dünnwandiges 0,2 mL PCR-Röhrchen.
- Einstufiges Reaktionsprotokoll
- Führen Sie eine einstufige Markierungsreaktion durch, indem Sie 2 μl 150 μM Adenosin-5'-5'-diphosphat-{5'-(cytidin-2'-O-methyl-3'-phosphat-TEG}C-biotin (AppCp-biotin), 3 μl 10x Ligase-Reaktionspuffer, 1,5 μl des 100 mM Probenbestands der zu sequenzierenden RNA, 3 μl wasserfreies DMSO zum Erreichen von 10 % (v/v), 1 μl T4-RNA-Ligase (10 Einheiten/μl) und 19,5 μl DEPC-behandeltes H2O (für ein Gesamtvolumen von 30 ml) in einem 1,5 mL RNase-freien Mikrozentrifugenröhrchen.
- Inkubieren Sie die Reaktion über Nacht bei 16 °C in einer PCR-Maschine.
- Führen Sie die Säulenreinigung wie oben in den Schritten 2.1.5 bis 2.1.8 beschrieben durch.
HINWEIS: Bereiten Sie für jede RNA-Probe ein separates/exklusives Reaktionsröhrchen vor (RNA-Skala von 150 pmol). Eine Markierung des 5'-Endes der RNA(s) mit Sulfo-Cyanin3 (Cy3) oder Cy3 kann erforderlich sein (z. B. für die bidirektionale Sequenzierungsverifizierung). Die Methode unterscheidet sich von der der 3'-Biotinylierung und wird in einer früheren Veröffentlichungbeschrieben 9.
3. Erfassen Sie biotinylierte RNA-Proben auf Streptavidin-Kügelchen
- Aktivieren Sie 200 μl Streptavidin C1-Magnetkügelchen durch Zugabe von 200 μl 1x B&W-Puffer (5 mM Tris-HCl, pH 7,5, 0,5 mM EDTA, 1 M NaCl) in ein 1,5 mL RNase-freies Mikrozentrifugenröhrchen. Ziehen Sie diese Lösung vor und legen Sie sie für 2 Minuten auf einen Magnetständer. Entsorgen Sie dann den Überstand, indem Sie die Lösung vorsichtig herauspipettieren.
- Waschen Sie die Kügelchen zweimal mit 200 μl Lösung A (DEPC-behandelt 0,1 M NaOH und DEPC-behandelt 0,05 M NaCl) und einmal in 200 μl Lösung B (DEPC-behandelt 0,1 M NaCl). Für jeden Waschschritt die Lösung vortexen und 2 Minuten lang auf einen Magnetständer legen, gefolgt von der Entsorgung des Überstands. Geben Sie dann 100 μL 2x B&W Puffer (10 mM Tris-HCl, pH 7,5, 1 mM EDTA, 2 M NaCl) hinzu.
- Geben Sie 1x B&W Puffer in die biotinylierte RNA-Probe, bis das Volumen 100 μl beträgt. Geben Sie diese Lösung dann zu den gewaschenen Kügelchen, die in 100 μl 2x B&W Puffer gelagert sind. 30 min bei Raumtemperatur auf einer Wippplattform bei 100 U/min inkubieren. Stellen Sie das Röhrchen für 2 Minuten auf einen Magnetständer und entsorgen Sie den Überstand.
- Waschen Sie die beschichteten Kügelchen 3 Mal in 1x S/W-Puffer und messen Sie die endgültige Konzentration des Überstands in jedem Waschschritt mit Nanodrop für die Rückgewinnungsanalyse, um zu bestätigen, dass die Ziel-RNA-Moleküle auf den Kügelchen verbleiben.
- Inkubieren Sie die Kügelchen in 10 mM EDTA, pH 8,2 mit 95% Formamid bei 65 °C für 5 min in einer PCR-Maschine. Lassen Sie das Röhrchen 2 Minuten lang auf dem Magnetständer und sammeln Sie den Überstand (der die biotinylierten RNAs enthält, die von den Streptavidin-Kügelchen freigesetzt werden) per Pipetto.
HINWEIS: Dieser physikalische Trennschritt vor dem Säureabbau wird nur für die Sequenzierung von RNA#1 in Abbildung 1c verwendet und ist für den 2D-HELS MS Seq nicht obligatorisch, da die hydrophobe Biotinmarkierung dazu führen kann, dass die 3'-markierten Leiterfragmente während der LC-MS-Messung einen signifikant verzögerten tR aufweisen, wodurch die markierten 3'-Leiterfragmente im 2D-mass-t R-Diagramm deutlich von den unmarkierten 5'-Leiterfragmenten unterschieden werden können.
4. Saure Hydrolyse von RNA zur Erzeugung von MS-Leitern für die Sequenzierung
- Teilen Sie jede RNA-Probe in drei gleiche Aliquots auf. Teilen Sie beispielsweise eine RNA-Probe mit einem Volumen von 15 μl RNA-Probe in drei Aliquots von 5 μl auf.
- Ein gleiches Volumen Ameisensäure wird zugegeben, um 50 % (v/v) Ameisensäure in dem Reaktionsgemischzu erreichen 8,9.
- Inkubieren Sie die Reaktion bei 40 °C in einer PCR-Maschine, wobei eine Reaktion für 2 Minuten, eine für 5 Minuten und eine für 15 Minuten läuft.
- Beenden Sie den Säureabbau, indem Sie die Probe nach Beendigung jeder Reaktion sofort auf Trockeneis einfrieren.
- Verwenden Sie einen Zentrifugal-Vakuumkonzentrator, um die Probe zu trocknen. Die Probe wird in der Regel innerhalb von 30 min vollständig getrocknet, und Ameisensäure wird während des Trocknungsprozesses zusammen mit H2O entfernt, da Ameisensäure einen Siedepunkt (100,8 °C) hat, der dem von H2O (100 °C) ähnelt.
- Suspendieren und kombinieren Sie insgesamt drei getrocknete Proben in 20 μl DEPC-behandeltem H2O für die LC-MS-Messung.
HINWEIS: Zu diesem Zeitpunkt können Proben bei -20 °C gelagert werden, während auf die LC-MS-Messung gewartet wird.
5. Konvertieren Sie ψ in CMC-ψ Addukt
- 80 μl DEPC-behandeltes H2O in ein 1,5 mL RNase-freies Mikrozentrifugenröhrchen geben, das 0,0141 g N-Cyclohexyl-Nʹ-(2-morpholinoethyl)-carbodiimid-metho-p-toluolsulfonat(CMC) und 0,07 g Harnstoff enthält. Fügen Sie 10 μl des 100 μM Probenbestands der zu sequenzierenden RNA, 8 μl 1 M Bicinpuffer (pH 8,3) und 1,28 μl 0,5 M EDTA hinzu. Fügen Sie DEPC-behandeltes H2O hinzu, um ein Gesamtvolumen von 160 μl zu erreichen. Die Endkonzentrationen betragen 0,17 M CMC, 7 M Harnstoff und 4 mM EDTA in 50 mM Bicin (pH 8,3)11.
HINWEIS: Dieses Protokoll ist entweder auf eine einzelne synthetische RNA-Sequenz oder auf RNA-Mischungen anwendbar. - Die 160 μl Reaktionslösung in RNase-freien, dünnwandigen 0,2 mL PCR-Röhrchen aufteilen und 20 min lang bei 37 °C in einer PCR-Maschine inkubieren.
HINWEIS: 50 μl pro Röhrchen ist das maximale Reaktionsvolumen, das in einer PCR-Maschine verwendet werden kann. - Jede Reaktion wird mit 10 μl 1,5 M Natriumacetat und 0,5 mM EDTA (pH 5,6) abgeschreckt.
- Führen Sie die Säulenreinigung mit vier parallelen Spin-Säulen durch, um überschüssige Reaktanten gemäß dem in den Schritten 2.1.5 bis 2.1.8 beschriebenen Verfahren zu entfernen. Das gereinigte Produkt wird in 15 μl DEPC-behandeltem H2O in jedem 1,5 mL RNase-freien Mikrozentrifugenröhrchen gelöst.
- Übertragen Sie das gereinigte Produkt in vier RNase-freie, dünnwandige 0,2-ml-PCR-Röhrchen. Geben Sie 20 μl 0,1 M Na2CO3 Puffer (pH 10,4) in jeweils 15 μl gereinigtes Produkt und fügen Sie DEPC-behandeltes H2O hinzu, um ein Endvolumen von 40 μl für jedes Reaktionsröhrchen (insgesamt vier Röhrchen) zu erhalten. Inkubieren Sie die Reaktion bei 37 °C für 2 h in einer PCR-Maschine.
- Die Reaktion wird durch Säulenreinigung mit vier parallelen Spinsäulen wie in Schritt 2.1.5 beschrieben abgekühlt und gereinigt. Eluieren Sie das CMC-ψ umgewandelte Produkt in ein 1,5 mL RNase-freies Mikrozentrifugenröhrchen mit jeweils 15 μl DEPC-behandeltem H2O.
- Kombinieren Sie die aufgereinigte CMC-ψ umgewandelte Probe aus vier Sammelröhrchen in einem Röhrchen. Führen Sie einen Ameisensäureabbau von 50 % (v/v) gemäß den in den Schritten 4.1-4.6 beschriebenen Verfahren durch, um MS-Leitern für die Sequenzierung zu generieren.
6. LC-MS-Messung
- Bereiten Sie mobile Phasen für die LC-MS-Messung vor. Mobile Phase A besteht aus 25 mM Hexafluor-2-propanol mit 10 mM Diisopropylamin in Wasser in LC-MS-Qualität; Die mobile Phase B ist Methanol.
- Übertragen Sie die Probe zur Analyse in ein LC-MS-Probenfläschchen. Jedes Probeninjektionsvolumen beträgt 20 μl und enthält 100-400 pmol RNA.
- Verwenden Sie die folgenden LC-Bedingungen: Säulentemperatur von 35 °C, Durchflussrate von 0,3 mL/min; ein linearer Gradient von 2–20 % mobiler Phase B über 15 Minuten, gefolgt von einem 2 minütigen Waschschritt mit 90 % mobiler Phase B.
HINWEIS: Für hydrophobe Endmarkierungen wie Cy3 und Sulfo-Cy3, wie in Abschnitt 2 erwähnt, kann ein höherer Anteil an organischem Lösungsmittel für die Probenelution erforderlich sein (d. h. ein ähnlicher Gradient kann verwendet werden, jedoch mit einem größeren prozentualen Bereich der mobilen Phase B). Zum Beispiel 2–38 % mobile Phase B über 30 min mit einem 2-minütigen Waschschritt mit 90 % mobiler Phase B. - Trennen und analysieren Sie Proben mit einem Agilent Q-TOF-Massenspektrometer (Quadrupole Time-of-Flight), das mit einem LC-System gekoppelt ist, das mit einem Autosampler und einem MS HPLC-System (High Performance Liquid Chromatography) ausgestattet ist. Bei der LC-Säule handelt es sich um eine 50 mm x 2,1 mm große C18-Säule mit einer Partikelgröße von 1,7 μm. Verwenden Sie die folgenden MS-Einstellungen: negativer Ionenmodus; Reichweite, 350 m/z bis 3200 m/z; Abtastrate, 2 Spektren/s; Durchfluss des Trocknungsgases, 17 l/min; Temperatur des Trocknungsgases, 250 °C; Verneblerdruck, 30 psig; Kapillarspannung, 3500 V; und Fragmentorspannung, 365 V. Bitte beachten Sie, dass diese Parameter spezifisch für den Typ oder das Modell des verwendeten Massenspektrometers sind.
- Erfassen Sie Daten mit der Erfassungssoftware Agilent MassHunter. Verwenden Sie den Agilent Workflow zur Extraktion molekularer Merkmale (MFE), um Verbindungsinformationen wie Masse, Retentionszeit, Volumen (die MFE-Häufigkeit für die jeweilige Ionenspezies) und Qualitätsfaktor usw. zu extrahieren. Verwenden Sie die folgenden MFE-Einstellungen: "Schwerpunktdatenformat, kleine Moleküle (chromatographisch), Peak mit Höhe ≥ 100, bis zu einem Maximum von 1000, Qualitätsfaktor ≥ 50".
HINWEIS: Optimieren Sie die MFE-Einstellungen, um so viele potenzielle Verbindungen wie möglich zu extrahieren, bis zu einem Maximum von 1000, mit Qualitätswerten von ≥ 50.
7. Automatisieren Sie die Generierung von RNA-Sequenzen durch einen Berechnungsalgorithmus
HINWEIS: Dieses Verfahren ist nur für RNA #1 in Abbildung 1c dargestellt.
- Sortieren Sie die MFE-extrahierten Verbindungen in der Reihenfolge des abnehmenden Volumens (Spitzenintensität) und tR. Führen Sie eine Datenvorauswahl durch, indem Sie 1)t R von 4 bis 10 min einstellen, um die vom Biotin markierten RNA-Fragmente auszuwählen, da diet Rs der Biotin-markierten Massenleiterkomponenten in dieses tR-Fenster (4 min bis 10 min) verschoben werden, und 2) eine Größenordnung verwenden, die höher ist als die Anzahl der Leiterfragmente für die Algorithmusberechnung, um die Datenmenge basierend auf dem Volumen zu reduzieren. Zum Beispiel werden für eine 20-nt-RNA 20 markierteMassen-t-R-Leiterkomponenten für die Sequenzierung der 20-nt-RNA benötigt, und daher werden 200 Verbindungen aus der MFE-Datendatei basierend auf dem Volumen ausgewählt. Bitte beachten Sie, dass das tR-Fenster unterschiedlich sein kann, wenn ein anderer Typ oder ein anderes Modell eines Massenspektrometers verwendet wird.
- Führen Sie die Datenverarbeitung und Sequenzgenerierung von RNA #1 mit einer überarbeiteten Version eines veröffentlichten Algorithmusdurch 8. Die Quellcodes des überarbeiteten Algorithmus sind bereits beschrieben (https://academic-oup-com.remotexs.ntu.edu.sg/nar/article/47/20/e125/5558343#supplementary-data)9.
- Zusätzlich zur Automatisierung der Sequenzgenerierung mithilfe des Algorithmus können Sie die Massenunterschiede zwischen zwei benachbarten Kontaktplankomponenten für den Basisaufruf manuell berechnen. Alle Basen in der RNA können manuell aufgerufen und mit den theoretischen Basen in der RNA-Nukleotid- und Modifikationsdatenbankabgeglichen werden 8; Auf diese Weise kann die vollständige Sequenz des RNA-Strangs manuell genau ausgelesen werden, was zur Bestätigung der Genauigkeit der vom Algorithmus gemeldeten Sequenz verwendet wird. Weitere Strukturen von RNA-Modifikationen finden sich in RNA-Modifikationsdatenbanken12, und die entsprechenden theoretischen Massen werden mit ChemBioDraw ermittelt. In den Tabellen S1 bis S2 wird der Massenunterschied in ppm (parts per million) angezeigt, wenn die beobachtete Masse mit ihrer theoretischen Masse für eine bestimmte Kontaktplankomponente verglichen wird, und ein Wert von weniger als 10 ppm wird für jeden Basenaufruf als gute Übereinstimmung angesehen.
8. Sequenzierung von RNA-Gemischen
- Markieren Sie ein Gemisch aus fünf RNA-Strängen (RNA #1 bis #5) an ihren 3'-Enden mit A(5')pp(5')Cp-TEG-biotin unter Verwendung eines einstufigen Protokolls, das in Schritt 2.2 beschrieben ist. In einem Gesamtvolumen von 150 μl Reaktionslösung werden 15 μl 10x T4-RNA-Ligase-Reaktionspuffer, 1,5 μl von jedem RNA-Strang (100 μM Stamm RNA #1 bis #5 für ein Gesamtvolumen von 7,5 μl), 10 μl 150 μM A(5')pp(5')Cp-TEG-biotin, 15 μl wasserfreies DMSO hinzugefügt. 5 μl T4-RNA-Ligase (10 Einheiten/μl) und 97,5 μl DEPC-behandeltes H2O. Die Reaktionslösung wird gleichmäßig auf fünf Aliquots verteilt. Jedes RNase-freie Mikrozentrifugenröhrchen enthält 30 μl Reaktionslösung.
- Inkubieren Sie die Reaktion über Nacht bei 16 °C in einer PCR-Maschine.
- Führen Sie die Säulenreinigung gemäß dem in den Schritten 2.1.5 bis 2.1.8 beschriebenen Verfahren mit fünf parallelen Spin-Säulen durch. Eine Mischprobe aus 3'-biotinylierten 5 RNA-Strängen (Mischung aus RNA #1 bis #5) wird auf ein 1,5 mL RNase-freies Mikrozentrifugenröhrchen mit jeweils 15 μl DEPC-behandeltem H2O eluiert.
- Kombinieren Sie die gereinigten Gemischproben aus den fünf Sammelröhrchen in einem Röhrchen. Der Abbau von Ameisensäure ist nach dem in Abschnitt 4 beschriebenen Verfahren durchzuführen.
- Messen Sie Proben mittels LC-MS, wie in Abschnitt 6 beschrieben, und analysieren Sie die Daten mit der Datenanalysesoftware mit optimierten MFE-Einstellungen, um Daten mit Masse, tR und Volumen zu extrahieren, wie in Schritt 6.5 beschrieben. Der typische Verarbeitungs- und Base-Calling-Algorithmus wird aufgrund der deutlich erhöhten Datenkomplexität, die sich aus der Mischung ergibt, nicht angewendet. Alle Basen in der RNA der gemischten Probe werden manuell in einer Methode ähnlich wie Abschnitt 7.3 aufgerufen und stimmen gut mit den theoretischen Basen in der RNA-Nukleotid- und Modifikationsdatenbank8 überein, so dass die vollständigen Sequenzen aller fünf RNA-Stränge in der gemischten Probe genau ausgelesen werden. In den Tabellen S7–S11 sind alle Informationen aufgeführt, einschließlich der beobachteten Masse, tR, Volumen, Qualitätsfaktor und ppm-Massendifferenz.
Ergebnisse
Einführung eines Biotin-Tags am 3'-Ende der RNA, um leicht identifizierbare Leiterleitern der Stärke tR herzustellen. Der Arbeitsablauf des 2D-HELS MS Seq-Ansatzes ist in Abbildung 1a dargestellt. Die hydrophobe Biotinmarkierung, die an das 3'-Ende der RNA eingeführt wird (siehe Abschnitt 2), erhöht die Massen undt Rs der 3'-markierten Leiterkomponenten im Vergleich zu denen ihrer unmarkierten Gegenstücke. Somit wird die 3'-Kontaktkurve im 2D-Masse-tR-Diagramm auf größere Werte der y-Achse verschoben (aufgrund der Zunahme dert Rs) und auf größere Werte der x-Achse (aufgrund der Zunahme der Massen). Abbildung 1b zeigt das Probenvorbereitungsprotokoll einschließlich der Einführung eines Biotin-Tags an das 3'-Ende der RNA für 2D-HELS MS Seq. Abbildung 1c zeigt die Trennung der 3'-Leiter von der 5'-Leiter und anderer unerwünschter Fragmente in einem 2D-Mass-tR-Diagramm basierend auf systematischen Änderungen dert Rs der 3'-Biotin-markierten Massen-t R-Leiterfragmente von RNA #1. Die 3'-Leiterkurve allein ergibt eine vollständige Sequenz von RNA #1, und die 5'-Leiterkurve, die keine tR-Verschiebung zeigt, liefert die umgekehrte Sequenz, erfordert jedoch eine Endpaarung zum Lesen der terminalen Basis8. Mit dieser Strategie des 2D-HELS ist keine Endpaarung erforderlich, wie zuvor berichtet, und die gesamte RNA-Sequenz kann vollständig aus nur einer markierten Kontaktleiterkurve8 ausgelesen werden. Auf diese Weise ist es möglich, gemischte Proben, die mehrere RNAs enthalten, zu sequenzieren, z. B. zwei RNA-Stränge unterschiedlicher Länge (RNA #1 und RNA #2, 19 nt bzw. 20 nt) mit einer 5'-Biotin-Markierung an jeder RNA (Abbildung 1d).
Konvertierung ψ in sein CMC-ψ Addukt für 2D-HELS MS Seq. ψ ist eine schwierige Nukleotidmodifikation für die MS-basierte Sequenzierung, da sie die gleiche Masse wie Uridin (U) hat. Um diese beiden Basen voneinander zu unterscheiden, behandeln wir die RNA mit CMC, wodurch ein ψ in ein CMC-ψ Addukt umgewandelt wird (siehe Abschnitt 5). Das Addukt hat eine andere Masse als U und kann in der 2D-HELS MS Seq unterschieden werden. Abbildung 2a zeigt das HPLC-Profil des Rohprodukts der Reaktion, das ψ in RNA #6 in sein CMC-Addukt umwandelt. Durch die Integration ihrer UV-Peaks berechneten wir die prozentuale Umwandlung und 42 % ψ werden nach dem in Abschnitt 5 dargestellten Prozess in sein CMC-ψ-Addukt umgewandelt. Nach dem Säureabbau und der LC-MS-Messung erfassten wir die Sequenz manuell sowohl auf der Grundlage von nicht-CMC-konvertierten Leitern als auch von CMC-konvertierten Leitern, die aus den algorithmusverarbeiteten Daten identifiziert wurden 8,9. Eine rote Kurve verzweigt sich von der grauen Kurve ab ψ an Position 8 in RNA #6 (Abbildung 2b) aufgrund der teilweisen Umwandlung von ψ in das CMC-ψ-Addukt. Aufgrund der Masse und Hydrophobizität des CMC führt diese Umwandlung zu einer Zunahme der Masse um 252,2076 Dalton und einer signifikanten Zunahme von tR für jede CMC-ψ addukthaltige Leiterkomponente im Vergleich zu seinem nicht umgesetzten Gegenstück. Daher kann eine dramatische Verschiebung ab Position 8 in RNA #6 im 2D-mass-tR-Diagramm beobachtet werden, was darauf hindeutet, dass Position 8 tatsächlich eine ψ in RNA #6 ist.
Sequenzierung von RNA-Mischungen. Eine Mischung aus fünf verschiedenen RNA-Strängen wird durch den 2D-HELS MS Seq-Ansatz mit 3'-End-Markierung sequenziert (siehe Abschnitt 8). Das Problem bei der Sequenzierung gemischter RNAs besteht darin, dass sich mehrere Leiterkurven im2D-Mass-t-R-Diagramm überlappen können, wenn sie alle dieselben Startpunkte haben (das hydrophobe Tag im 2D-Massen-tR-Diagramm). Der Basisaufruf erfolgt jedoch nacheinander, jeweils basierend auf einer Massendifferenz zwischen zwei benachbarten Kontaktplanfragmenten in den MFE-Daten. Der korrekte Basenaufruf kann durchgeführt werden, solange jede Massendifferenz gut übereinstimmt (eine PPM-MS-Differenz < 10) mit einer der theoretischen Massen kanonischer oder modifizierter Nukleotide im Datenpool 8,9. Bei der Analyse der gemultiplexten RNA-Proben wird der in Abbildung 1 und 2 verwendete typische Verarbeitungs- und Basenaufrufalgorithmus vor allem aufgrund der deutlich erhöhten Datenkomplexität, die sich aus der Mischung ergibt, nicht verwendet. Diese Sequenzen werden manuell als Basis aufgerufen, indem die Massendifferenz zwischen zwei benachbarten Massenleiterfragmenten berechnet und mit der theoretischen Masse des Nukleotids im Datenpool9 verglichen wird. Jede übereinstimmende Basis mit einem Massen-PPM <10 wird an dieser Position als Basisidentität ausgewählt. Mit dieser manuellen Base-by-Base-Berechnung für das Base-Calling werden alle Sequenzen in der Mischung genau sequenziert. Die OriginLab-Software wird verwendet, um ein 2D-Mass-t-R-Diagramm zu rekonstruieren, in dem das Anfangs-tR für jede Sequenz systematisch normalisiert wird, um fünf verschiedene RNA-Sequenzen besser sichtbar zu machen (Abbildung 3). Ohne eine solche Normalisierung würden die Buchstabencodes (d. h. A, C, G und U) für die Sequenzen aller fünf RNAs im Diagramm zusammengedrängt (Abbildung S1), was zu einer geringeren Visualisierung im Vergleich zu der in Abbildung 3 berichteten führt. Die Sequenzierungsergebnisse zeigen, dass der 2D-HELS MS Seq-Ansatz nicht nur auf die Sequenzierung von gereinigten einzelsträngigen RNAs beschränkt ist, sondern vor allem auch auf RNA-Mischungen mit mehreren RNA-Strängen. Derzeit befinden sich Algorithmen in der Entwicklung, um den Prozess des Base-Aufrufs und der Sequenzgenerierung zu automatisieren.
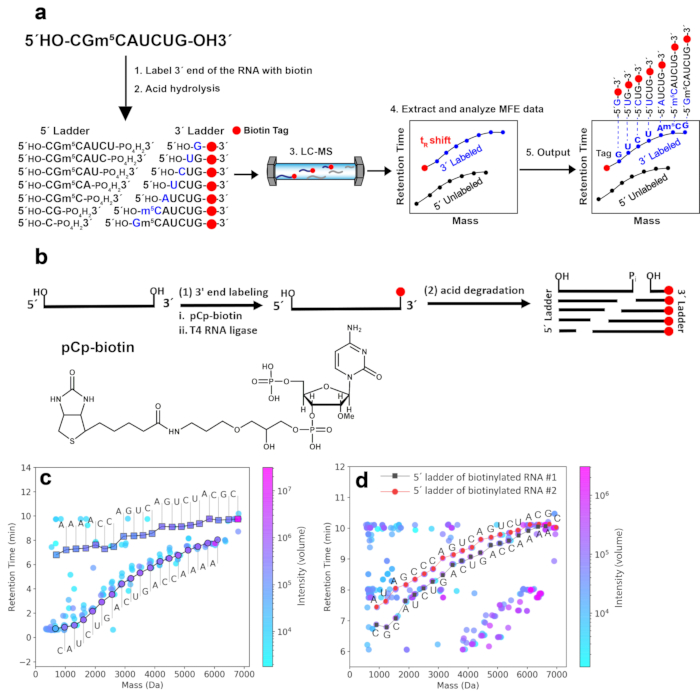
Abbildung 1. 2D-HELS MS Seq von repräsentativen RNA-Proben. (a) Arbeitsablauf für 2D-HELS MS Seq. Zu den wichtigsten Schritten gehören 1) die hydrophobe Markierung der zu sequenzierenden RNA, 2) die saure Hydrolyse, 3) die LC-MS-Messung, 4) die Extraktion und Analyse von MFE-Daten und 5) die Sequenzgenerierung über Algorithmen oder manuelle Berechnung. (b) Probenvorbereitungsprotokoll einschließlich des Einführens eines Biotin-Tags an das 3'-Ende der RNA für 2D-HELS MS Seq. (c) Trennung der 3'-Leiter von der 5'-Leiter und anderer unerwünschter Fragmente in einem 2D-Plot mit Massenretentionszeit (tR) auf der Grundlage systematischer Änderungen dert Rs von 3'-Biotin-markiertenMasse-t R-Leiterfragmenten der RNA #1 (19 nt). Die Sequenzen sind de novo und werden automatisch direkt durch einen basenaufrufenden Algorithmus9 ausgelesen. (d) Gleichzeitige Sequenzierung von 5'-Biotin-markierter RNA #1 und RNA #2, 19 nt bzw. 20 nt. Methoden zum Einbringen eines Biotin-Tags an das 5'-Ende der RNA unterscheiden sich von denen der 3'-Biotinylierung und sind im zuvor veröffentlichten Protokoll9 zu finden. Das 5'-Ende von zwei RNAs (RNA #1 und RNA #2) ist biotinyliert und ihre 5'-biotinylierten Leitern können leicht identifiziert werden; Beide 5'-biotinylierten Leitern lassen sich im 2D-mass-t R-Plot nach LC-MS leicht von ihren unmarkierten 3'-Leitern trennen, da die biotinylierten Leiterkomponenten aufgrund der Hydrophobizität des Biotins die größeren tR-Verschiebungen aufweisen, während sich unmarkierte Leiterkomponenten im unteren tR-Bereich befinden. Obwohl die 5'-Leitern und 3'-Leitern nebeneinander existieren, beeinträchtigen sie die Sequenzinterpretation zweier gemischter RNA-Stränge nicht. Jede Sequenz dieser beiden RNAs wird manuell von 5'-biotinylierten Leitern auf der Grundlage der durch den Computeralgorithmus verarbeiteten Daten 8,9 gewonnen. Diese Abbildung wurde von Zhang et al.9 modifiziert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2. Umwandlung von Pseudouridin (ψ) in sein Addukt für 2D-HELS MS Seq. (a) HPLC-Profil des Rohprodukts der Reaktion, das ψ in sein CMC-Addukt in einer 20-nt-RNA (RNA #6) umwandelt, die eine ψ enthält. (b) Sequenzierung einer ψ-haltigen RNA #6. Die Umwandlung des ψ in die CMC-ψ-Addukte (ψ*) führt zu einer Zunahme der Masse um 252,2076 Dalton und einer signifikanten Zunahme von tR aufgrund seiner Masse und Hydrophobie des CMC. Daher kann immass-t R-Diagramm eine dramatische Verschiebung ab der Position von 8 beobachtet werden, was darauf hindeutet, dass es sich um eine ψ an der Position von 8 in der RNA-Sequenz handelt. Die Sequenzen werden manuell auf der Grundlage der vom Berechnungsalgorithmus verarbeiteten Daten 8,9 erfasst. Diese Abbildung wurde von Zhang et al.9 modifiziert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3. Sequenzierung von RNA-Gemischen, die fünf verschiedene RNAs enthalten. Ein Biotin wird verwendet, um jede RNA an ihrem 3'-Ende vor 2D-HELS MS Seq zu markieren. Für jede Sequenz werden dietR-Startwerte systematisch so normalisiert, dass sie in 7-Minuten-Intervallen beginnen, um die Visualisierung zu erleichtern. Die absoluten Differenzen zwischen dem anfänglichen tR-Wert und den nachfolgenden tRs bleiben für jede der fünf RNAs unverändert, so dass es einfacher ist, jede von ihnen im selben Diagramm zu visualisieren. Alle Basen werden identifiziert, indem die Massenunterschiede zweier benachbarter Leiterkomponenten manuell berechnet und mit den theoretischen Massenunterschieden in der RNA-Nukleotid- und Modifikationsdatenbank8 abgeglichen werden; Die Diagramme für Abbildung 3 wurden mit OriginLab auf der Grundlage manueller Basenaufruf- und Sequenzierungsdaten rekonstruiert (siehe Abschnitt Sequenzierung von RNA-Mischungen in Repräsentative Ergebnisse). Die 2D-Masse-tR-Abbildung der fünf gemischten RNAs ohnetR-Normalisierung ist in Abbildung S1 dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Abbildung S1. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Im Gegensatz zur tandembasierten MS-Fragmentierung wird beim 2D-HELS MS Seq-Ansatz eine hochgradig kontrollierte saure Hydrolyse verwendet, um die RNA vor der Analyse mit einem Massenspektrometerzu fragmentieren 9,10. Dadurch kann jedes säureabgebaute Fragment mit dem Instrument detektiert werden, was einer Sequenzierungsleiter entspricht. Unter optimalen Bedingungen erzeugt dieses Verfahren eine "ideale" Sequenzleiter aus RNA über eine durchschnittlich ein-pro-Molekül-ortsspezifische RNA-Spaltung ausschließlich an einer Phosphodiesterbindung 8,9,10. Nachdem jedes degradierte Fragment mit dem Massenspektrometer in einem einzigen Lauf gemessen wurde, entspricht die Massendifferenz zwischen zwei benachbarten Leiterfragmenten der exakten Masse des RNA-Nukleotids oder der RNA-Modifikation an dieser Position. Jede RNA-Modifikation hat entweder eine intrinsische einzigartige Masse, die helfen kann, sie in der RNA zu identifizieren und zu lokalisieren, oder sie kann in eine mit einer einzigartigen Masse umgewandelt werden. Theoretisch kann diese Methode also die Identität und den Ort sowohl von kanonischen als auch von modifizierten Nukleotiden für die de novo und direkte Sequenzierung jeder RNA melden. Verschiedene Sequenzleitern können sich jedoch überlappen, was die Datenanalyse von MS erschwert und die RNA-Sequenzierung mit MS in der Praxis erschwert.
Einer der Vorteile des 3'-hydrophoben Tags besteht darin, dass er eine große Herausforderung bei jeder Fragmentierungsmethode überwindet, d.h. dass jedes RNA-Molekül in genau zwei Fragmente (und idealerweise nicht mehr) gespalten werden muss: ein Fragment, das das ursprüngliche 5'-Ende enthält, und das andere, das das ursprüngliche 3'-Ende der RNA enthält. Daher erzeugt jedes Spaltungsereignis zwei Fragmente, die zwei Leitern erzeugen – eine gemessen vom 5'-Ende und die andere vom 3'-Ende. Es gibt immer Unklarheiten bei der Bestimmung, welcher MS-Peak zu welcher Leiter gehört. Problematischer wird dies bei einer Mischung aus mehreren verschiedenen RNAs, da eine große Anzahl von überlappenden Sequenzleitern entsteht. Da jedoch alle Leiterfragmente von den 3'-Enden mit einem hydrophoben Tag markiert sind, weisen sie deutlich längere tRs auf (Abbildung 1a). Als Ergebnis können wir klare und eindeutige Leitern in den 2D-mass-t-R-Daten erhalten, die ausschließlich aus der 3'-markierten RNA stammen. Insbesondere optimieren wir Ansätze, um entweder das 5'- oder das 3'-Ende einer RNA selektiv mit verschiedenen chemischen Konjugationsmethoden zu markieren9. Wir können auch eine bidirektionale Sequenzierung durchführen, die hier nicht zur Bestimmung der terminalen(n) Base(s) verwendet wird, sondern verwendet wird, um beim Lesen aus 5'- und 3'- Richtungen zweimal identische Sequenzinformationen zu liefern (d.h. bidirektionale Sequenzierungsverifizierung), und so die Genauigkeit der Sequenzierung weiter zu verbessern.
Für die De-novo-Sequenzierung unbekannter RNA-Proben, insbesondere für komplexe biologische Proben, ist ein allgemeiner und robuster Algorithmus erforderlich, um eine große Menge an LC-MS-Daten für die Sequenzgenerierung genau und effizient zu verarbeiten, der kürzlich durch andere veröffentlichte Arbeiten verfügbar geworden ist10. Obwohl diese Algorithmen für die Sequenzierung komplizierterer Proben10 verwendet wurden, führten wir in dieser Studie, sofern nicht anders angegeben, manuelle Basenaufrufe für die Sequenzgenerierung durch. Unser Ziel ist es, alle wichtigen Schritte im 2D-HELS MS Seq abzudecken und möchte den Prozess veranschaulichen, bei dem wir auch ohne den Einsatz zusätzlicher Sequenzierungsalgorithmen Sequenzen der zu sequenzierenden RNA manuell auslesen können. Zur Vereinfachung der Visualisierung und zur schnelleren Identifizierung von Kontaktplanfragmenten, die für die Sequenzierung im 2D-mass-tR-Diagramm benötigt werden, werden die MFE-Dateien jedes LC-MS-Laufs vor dem Lesen ihrer Sequenzen durch eine überarbeitete Version eines veröffentlichten Algorithmus8 verarbeitet, sofern nicht anders angegeben. Der veröffentlichte Algorithmus kann nicht direkt zum Auslesen der Sequenzen aus den LC-MS-Daten verwendet werden, aber ein Teil seiner Funktion kann dennoch zur Verarbeitung der Daten verwendet werden – die hierarchische Clusterung von Massenaddukten durch diesen Algorithmus erhöht die Intensität jeder Kontaktplankomponente, was wiederum die Datenkomplexität reduziert, insbesondere in dem entscheidenden Bereich, in dem Sequenz-Reads generiert werden8, 9. Urheberrecht
Einer der entscheidenden Schritte während der Probenvorbereitung für 2D-HELS MS Seq führt zu einer Verbesserung der Effizienz der hydrophoben Markierung von RNA-Markierungen. Eine hohe Markierungseffizienz kann dazu beitragen, die Menge an RNA-Proben zu reduzieren, die für die Erzeugung von MS-Signalen benötigt werden, auf die die Sequenzdaten angewiesen sind. Um die Markierungseffizienz zu erhöhen, setzen wir neue Markierungsstrategien ein, einschließlich der Verwendung von aktiviertem AppCp-Biotin, um den Adenylierungsschritt bei der Markierung des 3'-Endes der RNA zu vermeiden. Die Ausbeute der Reaktion zur Markierung des 3'-Endes einer 19 nt-RNA mit Biotin (siehe Schritt 2.2) kann mit dieser einstufigen Methode von 60 % auf ~95 %9 verbessert werden. Mit der effizienten Markierung sind wir in der Lage, eine gemischte Probe mit bis zu 12 verschiedenen RNAs zu sequenzieren, wie zuvor beschrieben9. In dieser Studie verwenden wir eine Mischung aus fünf RNAs als repräsentatives Beispiel, um den Sequenzierungsprozess zu veranschaulichen. Wir detektieren auch alle Leiterfragmente, die für eine genaue Sequenzierung benötigt werden, und lesen die vollständigen Sequenzen jeder der fünf RNA-Sequenzen in der Mischung aus. Eine höhere Etikettierungseffizienz trägt nicht nur zur Minimierung der Probenlademenge bei, sondern trägt auch zu einer deutlichen Reduzierung der Datenkomplexität bei der nachgelagerten Datenanalyse für die Sequenzgenerierung bei. Neuartige Reaktionen befinden sich derzeit in der Entwicklung, um eine quantitative Ausbeute bei der Markierung von RNAs sowohl am 5'- als auch am 3'-Ende zu erzielen.
Bei der Sequenzierung von RNA #1, wie in Abbildung 1c gezeigt, werden Streptavidin-Capture- und -Release-Schritte verwendet, um biotinylierte RNA #1 vor dem Säureabbau physikalisch zu trennen (siehe Abschnitt 3). Dadurch wird ein kleiner Teil der unmarkierten RNA entfernt, was in der Folge zu einer einfacheren visuellen Identifizierung der markierten Massenleitern im2D-mass-t-R-Diagramm führt. Der physikalische Trennschritt ist jedoch nicht obligatorisch, da die biotinylierten RNA-Leiterfragmente aufgrund der Hydrophobizität des Biotin-Tags im Vergleich zu ihren unmarkierten Gegenstücken verzögerte/längere tRs aufweisen. Darüber hinaus beruht das Base-Calling nicht auf einer physikalischen Trennung, sondern auf den Massenunterschieden benachbarter Massenleiterkomponenten, so dass der korrekte Basenaufruf erreicht werden kann, solange die Massenunterschiede zweier benachbarter Leiterkomponenten gut mit den entsprechenden Massen eines bestimmten Nukleotids oder einer Modifikation in der RNA-Nukleotid- und Modifikationsdatumsbasis8 übereinstimmen. Ein Berechnungsalgorithmus befindet sich derzeit in der Entwicklung, um das Aufrufen von Basen und die Generierung von Sequenzen zu automatisieren.
Die MFE-Einstellungen beim ursprünglichen LC-MS-Datenexport (in der Dateityp .d) in Tabellenkalkulationsdateien sind für die Datenverarbeitung und die anschließende Sequenzgenerierung von entscheidender Bedeutung (siehe Abschnitt 6.5). Zum Beispiel haben wir die MFE-Einstellung "Spitze mit Höhe" in einem Bereich von 100 bis 1000 getestet und festgestellt, dass wir mit einer Einstellung von 100 2-fach mehr Verbindungen erhalten können als mit der Einstellung 1000. Um zu vermeiden, dass Kontaktplankomponenten fehlen, können wir die MFE-Einstellung während des Sequenzierungs-Workflows anpassen. Diese Einstellung hängt wahrscheinlich von der Massenauflösung des Instruments, der Anzahl der Massenleiterfragmente und der Datenkomplexität ab. Darüber hinaus ist es wichtig, den Schwerpunktdatensatz und die chromatographische Typeinstellung für kleine Moleküle zu verwenden. Der Qualitätsfaktor kann je nach Datenqualität von 50 % bis 100 % variiert werden.
Das LC-MS-Instrument, das wir in der Studie verwenden, hat eine obere Massenauflösung von ~40K, was die Methode auf die Sequenzierung von RNA mit einer Länge von weniger als 35 Basen beschränkt. Die genaue Leselänge dieser Methode ist jedoch instrumentenabhängig. Fortschrittlichere Geräte mit höherem Auflösungsvermögen können zu einer längeren Leselänge führen. Ebenso muss der Durchsatz, d.h. wie viele RNA-Sequenzen gleichzeitig in einem einzigen LC-MS-Lauf sequenziert werden können, noch erforscht werden, obwohl wir eine Mischung aus RNA-Proben bis zu 12 verschiedenen RNA-Strängen auch ohne Verwendung eines Algorithmus manuell sequenziert haben9. Mit dem aktuellen Workflow sind ~100 pmol kurze RNA (<35 nt) für jeden LC-MS-Lauf erforderlich. Die Beladungsmenge nimmt zu, wenn zusätzliche Experimente erforderlich sind: Für die Differenzierung isomerer Nukleotidmodifikationen sind typischerweise bis zu 400 pmol RNA erforderlich. Für die Sequenzierung spezifischer tRNA wie tRNAPhe können ~1000 pmol Probe für die Sequenzierung und Modifikationsanalyse benötigt werden. Wir gehen jedoch davon aus, dass die erforderlichen Probenbeladungsmengen bei LC-MS-Instrumenten mit höherer Empfindlichkeit verringert werden. Mit Verbesserungen bei der Effizienz der Probenmarkierung, dem Sequenzierungsalgorithmus sowie der Empfindlichkeit und Auflösung des Geräts erwarten wir, dass unsere Methode auf ein breiteres Spektrum von RNA-Proben anwendbar sein wird, insbesondere auf solche mit verschiedenen RNA-Modifikationen.
Offenlegungen
Die Autoren haben ein vorläufiges Patent angemeldet, das sich auf die in diesem Manuskript besprochene Technologie bezieht.
Danksagungen
Die Autoren danken für den R21-Zuschuss der National Institutes of Health (1R21HG009576) an S. Z. und W. L. und den Institutional Support for Research and Creativity des New York Institute of Technology (NYIT) für S. Z., die diese Arbeit unterstützt haben. Die Autoren danken dem Doktoranden Xuanting Wang (Columbia University) für die Unterstützung bei der Figurenerstellung und danken Prof. Michael Hadjiargyrou (NYIT), Prof. Jingyue Ju (Columbia University), Drs. James Russo, Shiv Kumar, Xiaoxu Li, Steffen Jockusch und anderen Mitgliedern des Ju-Labors (Columbia University), Dr. Yongdong Wang (Cerno Bioscience), Meina Aziz (NYIT) und Wenhao Ni (NYIT) für hilfreiche Diskussionen und Vorschläge für unser Manuskript.
Materialien
| Name | Company | Catalog Number | Comments |
| 5' DNA Adenylation kit | New England Biolabs | E2610S | 50uM concentration |
| 6550 Q-TOF mass spectrometer | Agilent Technologies | 5991-2116EN | Coupled to a 1290 Infinity LC system |
| A(5´)pp(5´)Cp-TEG-biotin-3´ | ChemGenes | 91718 | HPLC purified |
| ATPγS | Sigma-Aldrich | 11162306001 | Lithium salt |
| Bicine | Sigma-Aldrich | B8660 | BioXtra, ≥99% (titration) |
| Biotin maleimide | Vector Laboratories | SP-1501 | Long arm |
| C18 column | Waters | 186003532 | 50 mm × 2.1 mm Xbridge C18 column with a particle size of 1.7 μm |
| Centrifugal Vacuum Concentrator | Labconco | Refrig 115v/60hz 7310022 | Labconco CentriVap |
| ChemBioDraw | PerkinElmer | ChemDraw Prime | Generate a chemical structure and property data of structures & fragments |
| CMC (N-cyclohexyl-N?-(2-morpholinoethyl)-carbodiimide metho-p-toluenesulfonate) | Sigma-Aldrich | 2491-17-0 | 95% Purifiy |
| Cyanine3 maleimide (Cy3) | Lumiprobe | 11080 | Water insoluble |
| DEPC-treated water | Thermo Fisher Scientific | AM9906 | Autoclaved, certified nuclease-free |
| Diisopropylamine (DIPA) | Thermo Fisher Scientific | 108-18-9 | 99% Alfa Aesar |
| DMSO | Sigma-Aldrich | 276855 | Anhydrous dimethyl sulfoxide, 99.9% |
| EDTA | Sigma-Aldrich | E6758 | Anhydrous, crystalline, BioReagent, suitable for cell culture |
| Formic acid | Merck | 64-18-6 | 98-100%, ACS reag, Ph Eur |
| Hexafluoro-2-propanol (HFIP) | Thermo Fisher Scientific | 920-66-1 | 99% Acros Organics |
| LC-MS sample vials | Thermo Fisher Scientific | C4000-11 | Plastic screw thread vials |
| LC-MS vial caps | Thermo Fisher Scientific | C5000-54A | Autosampler vial screw thread caps |
| Na2CO3 buffer | Sigma-Aldrich | 88975 | BioUltra, >0.1 M Na2CO3, >0.2 M NaHCO3 |
| Oligo Clean & Concentrator | Zymo Research | D4060 | Spin column |
| OriginLab | OriginLab | OriginPro | Data analysis and graphing software |
| pCp-biotin | TriLink BioTechnologies | NU-1706-BIO | 20 ul (1 mM) |
| RNA #1--#6 | Integrated DNA Technologies | Custom RNA oligos | 19nt-21nt single-stranded RNAs, used without further purification |
| Rocking platform shaker | VWR | Orbital Shaker Standard 1000 | Speed Range 40 to 300 rpm |
| Streptavidin magnetic beads | Thermo Fisher Scientific | 88816 | Binding approx. 55ug biotinylated rabbit lgG per mg of beads |
| Sulfonated Cyanine3 maleimide | Lumiprobe | 11380 | Water soluble |
| T4 DNA ligase 1 | New England Biolabs | M0202S | 400 units/uL |
| T4 polynucleotide kinase | Sigma-Aldrich | T4PNK-RO | From phage T4 am N81 pse T1 infected Escherichia coli BB |
| Tris-HCl buffer | Sigma-Aldrich | T6455 | Tris-HCl Buffer, pH 10, 10×, Antigen Retriever |
| Urea | Sigma-Aldrich | 81871 | Urea for synthesis. CAS No. 57-13-6, EC Number 200-315-5. |
Referenzen
- Addepalli, B., Venus, S., Thakur, P., Limbach, P. A. Novel ribonuclease activity of cusativin from Cucumis sativus for mapping nucleoside modifications in RNA. Analytical and Bioanalytical Chemistry. 409 (24), 5645-5654 (2017).
- Gao, H., Liu, Y., Rumley, M., Yuan, H., Mao, B. Sequence confirmation of chemically modified RNAs using exonuclease digestion and matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 23 (21), 3423-3430 (2009).
- McLuckey, S. A., Van Berkel, G. J., Glish, G. L. Tandem mass spectrometry of small, multiply charged oligonucleotides. Journal of The American Society for Mass Spectrometry. 3 (1), 60-70 (1992).
- Fountain, K. J., Gilar, M., Gebler, J. C. Analysis of native and chemically modified oligonucleotides by tandem ion-pair reversed-phase high-performance liquid chromatography/electrospray ionization mass spectrometry. Rapid Communications in Mass Spectrometry. 17 (7), 646-653 (2003).
- Taucher, M., Breuker, K. Characterization of modified RNA by top-down mass spectrometry. Angewandte Chemie International Edition in English. 51 (45), 11289-11292 (2012).
- Kellner, S., Burhenne, J., Helm, M. Detection of RNA modifications. RNA Biology. 7 (2), 237-247 (2010).
- Thomas, B., Akoulitchev, A. V. Mass spectrometry of RNA. Trends in Biochemical Sciences. 31 (3), 173-181 (2006).
- Bjorkbom, A., et al. Bidirectional direct sequencing of noncanonical RNA by two-dimensional analysis of mass chromatograms. Journal of the American Chemical Society. 137 (45), 14430-14438 (2015).
- Zhang, N., et al. A general LC-MS-based RNA sequencing method for direct analysis of multiple-base modifications in RNA mixtures. Nucleic Acids Research. 47 (20), 125(2019).
- Zhang, N., et al. Direct sequencing of tRNA by 2D-HELS-AA MS Seq reveals its different isoforms and dynamic base modifications. ACS Chemical Biology. 15 (6), 1464-1472 (2020).
- Bakin, A., Ofengand, J. Four newly located pseudouridylate residues in Escherichia coli 23S ribosomal RNA are all at the peptidyltransferase center: analysis by the application of a new sequencing technique. Biochemistry. 32 (37), 9754-9762 (1993).
- Cantara, W. A., et al. The RNA Modification Database, RNAMDB: 2011 update. Nucleic Acids Research. 39 (Database issue), D195-D201 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten