Method Article
2D-HELS MS Seq:一种基于 LC-MS 的通用方法,用于对具有不同核苷酸修饰的 RNA 混合物进行直接和从头测序
摘要
在这里,我们描述了一种基于 LC-MS 的测序方法的详细方案,该方法可用作在没有 cDNA 中间体的情况下对短 RNA(每次运行 <35 nt)进行测序的直接方法,以及作为在单个研究中以单碱基精度对不同核苷酸修饰进行测序的通用方法。
摘要
基于质谱 (MS) 的测序方法已被证明可用于 RNA 直接测序,而无需互补 DNA (cDNA) 中间体。然而,这种方法很少用作 从头 RNA 测序方法,而主要用作有助于确认纯化单链 RNA 样品已知序列的质量保证的工具。最近,我们通过将二维质量保留时间疏水末端标记策略集成到基于 MS 的测序 (2D-HELS MS Seq) 中,开发了一种直接 RNA 测序方法。该方法能够对单个 RNA 序列以及包含多达 12 个不同 RNA 序列的混合物进行准确测序。除了四种经典核糖核苷酸(A、C、G 和 U)外,该方法还能够对含有修饰核苷酸的 RNA 寡核苷酸进行测序。这是可能的,因为修饰的核碱基要么具有本质独特的质量,可以帮助其鉴定及其在 RNA 序列中的位置,要么可以转化为具有独特质量的产物。在这项研究中,我们使用了 RNA,掺入了两个具有代表性的修饰核苷酸(假尿嘧啶 (Ψ) 和 5-甲基胞嘧啶 (m5C)),以说明该方法在单个 RNA 寡核苷酸以及 RNA 寡核苷酸混合物的从 头 测序中的应用,每个核苷酸具有不同的序列和/或修饰的核苷酸。使用标准高分辨率 LC-MS 系统时,本文描述的对这些模型 RNA 进行测序的程序和方案适用于其他短 RNA 样品 (<35 nt),也可用于修饰的治疗性 RNA 寡核苷酸的序列验证。未来,随着更稳健算法的发展和更好的仪器,这种方法可以允许对更复杂的生物样本进行测序。
引言
基于质谱 (MS) 的测序方法,包括自上而下的 MS 和串联 MS 1,2,3,4,已被开发用于 RNA 的直接测序。然而,在质谱仪中有效生成高质量 RNA 分子量标准的原位碎裂技术目前不能应用于从头测序 5,6。此外,分析传统的一维 (1D) MS 数据以对一个纯化的 RNA 序列进行从头测序也不是一件容易的事,对于混合 RNA 样品的 MS 测序来说,这将更具挑战性 7,8。因此,开发了一种基于二维 (2D) 液相色谱 (LC)-MS 的 RNA 测序方法,该方法采用 2D 质量保留时间 (tR) 分子量标准来取代 1D 质量分子量标准,从而更容易识别 RNA 从头测序所需的分子量标准组分8。然而,基于 2D LC-MS 的 RNA 测序方法主要局限于纯化的合成短 RNA,因为它不能仅基于一个分子量标准读取完整的序列,而必须依赖于两个共存的相邻分子量标准(5'-和 3'-分子量标准)8。更具体地说,这种方法需要双向双端读长来读取低质量区域8 中的末端核碱基。双端读数增加的复杂性导致该方法不适用于 RNA 混合物的测序,因为对于未知样品,哪个分子量标准片段属于哪个分子量标准会引起混淆。
为了克服基于 MS 的 RNA 测序方法中的上述障碍并扩大此类在直接 RNA 测序中的应用,必须解决两个问题:1) 如何生成可用于读取完整序列的高质量质量梯,从 RNA 链中的第一个核苷酸到最后一个核苷酸,以及 2) 如何有效识别复杂 MS 数据集中的每个 RNA/质量梯。结合良好控制的酸降解,我们通过在基于 MS 的测序技术中引入疏水末端标记策略 (HELS) 开发了一种新的测序方法,并通过在待测序 RNA 的 5' 和/或 3' 末端添加疏水标签成功解决了这两个问题9。该方法从 RNA 创建一个"理想"序列分子量标准——每个分子量标准片段都来自每个磷酸二酯键处的位点特异性 RNA 切割,两个相邻分子量标准片段之间的质量差是该位置的核苷酸或核苷酸修饰的精确质量 8,9,10.这是可能的,因为我们包括一个高度受控的酸性水解步骤,在将 RNA 注入仪器之前,平均每个分子对 RNA 进行一次碎裂。因此,每个降解片段产物都在质谱仪上检测到,并且所有片段一起形成测序梯形图 8,9,10。这种新策略能够从 RNA 链的一个阶梯中完整读取 RNA 序列,而无需从 RNA 的另一个阶梯中读取双端读数,并且还允许对包含组合核苷酸修饰的多个不同链的 RNA 混合物进行 MS 测序9。通过在 RNA 的 5' 和/或 3' 端添加标签,标记的分子量标准片段显示出 tR 的显著延迟,这有助于区分两个质量分子量标准彼此以及噪声低质量区域。添加疏水标签引起的 mass-tR 偏移有助于质量阶梯鉴定,并简化序列生成的数据分析。此外,添加疏水标签可以帮助识别链中的末端碱基,防止其相应的梯子片段由于标签引起的质量和疏水性增加而处于嘈杂的低质量t R 区域,从而允许从单个梯子中识别 RNA 的完整序列;不需要双端读取。因此,我们之前已经证明了在不使用任何高级测序算法的情况下,可以成功对多达 12 个 RNA 不同链的复杂混合物进行测序9,这为对包含经典核苷酸和修饰核苷酸的 RNA 进行从头 MS 测序打开了大门,使其更适合混合和更复杂的 RNA 样品的测序。事实上,使用 2D-HELS MS Seq,我们甚至成功地对 tRNA 样品的混合群体进行了测序10,并正在积极将其应用扩展到其他复杂 RNA 样品。
为了帮助 2D-HELS MS Seq 直接对更广泛的 RNA 样品进行测序,我们将重点介绍这种测序方法的技术方面,并将涵盖将该技术应用于 RNA 样品直接测序时所需的所有基本步骤。将使用具体示例来说明测序技术,包括合成的单个 RNA 序列、多个不同 RNA 序列的混合物以及包含经典核苷酸和修饰核苷酸的修饰 RNA,例如假尿嘧啶 (ψ) 和 5-甲基胞嘧啶 (m5C)。由于 RNA 都含有磷酸二酯键,因此在最佳条件下,任何类型的 RNA 都可以进行酸水解,从而产生用于 2D-HELS MS Seq 的理想序列分子量标准 8,9。然而,给定 RNA 的所有分子量标准片段的检测取决于仪器。在标准高分辨率 LC-MS (40K) 上,对纯化的短 RNA 样品 (<35 nt) 进行测序的最小上样量为每次运行 100 pmol。然而,当必须进行额外的实验时(例如,为了区分具有相同质量的同分异构碱基修饰),则需要更多的材料(每个 RNA 样品高达 400 pmol)。用于对模型合成修饰的 RNA 进行测序的方案也适用于更广泛的 RNA 样品的测序,包括碱基修饰未知的生物 RNA 样品。然而,需要更大的样品量,例如使用标准 LC-MS 仪器对 tRNA (~76 nt) 进行测序需要 1000 pmol 才能对所有修饰进行测序,并且必须为其从头测序10 开发一种先进的算法。
研究方案
1. 设计 RNA 寡核苷酸
- 设计不同长度(19 nt、20 nt 和 21 nt)的合成 RNA 寡核苷酸,包括一个同时具有经典核苷酸和修饰核苷酸的 RNA 寡核苷酸 (RNA #6)。ψ 被用作非质量改变修饰的模型,这对 MS 测序来说具有挑战性,因为它的质量与 Um 相同。选择5C 作为质量改变修饰的模型,以证明该方法的稳健性。
RNA #1:5'-HO-CGCAUCUGACUGACCAAAA-OH-3'
核糖核酸 #2:5'-HO-AUAGCCCAGUCAGUCUACGC-OH-3'
RNA #3:5'-HO-AAACCGUUACCAUUACUGAG-OH-3'
RNA #4:5'-HO-GCGUACAUCUUCCCCUUUAU-OH-3'
核糖核酸 #5: 5'-HO-GCGGAUUUAGCUCAGUUGGGA-OH-3'
RNA #6:5'-HO-AAACCGUψACCAUUAm5CUGAG-OH-3'
- 将每个合成的 RNA 溶解在无核酸酶的焦碳酸二乙酯 (DEPC) 处理的水(除非另有说明,否则表示为 DEPC 处理的 H2O)中,以获得 100 mM RNA 储备液。储备溶液在 -20 °C 下长期储存。
- 为避免可能的 RNA 样品降解,请使用不含 RNase 的实验备件,包括经 DEPC 处理的水、微量离心管和移液器吸头。经常使用 RNase 消除湿巾擦拭实验室用品的表面。
2. 用生物素标记 RNA 的 3'-端
- 两步反应方案(腺苷酸化和连接)
- 将 1 μL 含有 50 mM 乙酸钠(pH 6.0)、10 mM MgCl2、5 mM 二氯二苯基三氯乙烷 (DTT)、0.1 mM 乙二胺四乙酸 (EDTA)、1 μL 1 mM ATP、1 μL 100 μM 生物素化胞苷二磷酸 (pCp-生物素)、1 μL 50 μM Mth RNA 连接酶和 6 μL DEPC 处理的 H2O(总体积为 10 μL)加入无 RNase 的薄壁 0.2 mL PCR 管中。
注:在两步反应之前,将试剂储存在 -20 °C 下。在室温下解冻试剂,并在加入反应之前通过涡旋和离心充分混合。 - 将反应物在 PCR 机中于 65 °C 孵育 1 小时,并在 85 °C 下灭活反应 5 分钟。
- 加入 3 μL 含有 50 mM 三(羟甲基)氨基甲烷 (Tris)-HCl,pH 7.8、10 mM MgCl2、1 mM DTT、1.5 μL 待测序 RNA 样品储备液的 10x T4 RNA 连接酶反应缓冲液,在不含 RNase 的薄壁 0.2 mL PCR 管中进行连接步骤,该管含有 10 μL 上一步的反应溶液, 3 μL 无水二甲基亚砜 (DMSO) 达到 10% (v/v),1 μL T4 RNA 连接酶(10 单位/μL)和 11.5 μL DEPC 处理的 H2O(总体积为 30 mL)。将反应物在 16 °C 下在 PCR 机器中孵育过夜。
注:由于 DMSO 的凝固点高 (18.45 °C),因此在室温下混合反应组分。 - 将反应物在 16 °C 下孵育过夜。
- 通过柱纯化淬灭和纯化反应,以去除酶和游离的pCp-生物素,使用Oligo Clean & Concentrator (Zymo Research, Irvine, CA, USA)。试剂盒中提供寡核苷酸结合缓冲液、DNA 洗涤缓冲液、离心柱和收集管。在添加结合缓冲液之前,向反应溶液中加入 20 mL 经 DEPC 处理的 H2O,以达到 50 mL 样品体积。
- 向每种反应溶液中加入 100 mL 结合缓冲液。加入 400 μL 乙醇,通过移液混合,然后将混合物转移到色谱柱中。以 10,000 x g 离心 30 秒。丢弃流出物。
- 向色谱柱中加入 750 μL DNA 洗涤缓冲液。分别以 10,000 x g 和最大速度离心 30 秒和 1 分钟。
- 将色谱柱转移至 1.5 mL 微量离心管中。向色谱柱中加入 15 μL 经 DEPC 处理的 H2O,并以 10,000 x g 离心 30 秒以洗脱 RNA 产物。
注:样品在此阶段可储存在 -20 °C 下,直到执行下一步。
- 将 1 μL 含有 50 mM 乙酸钠(pH 6.0)、10 mM MgCl2、5 mM 二氯二苯基三氯乙烷 (DTT)、0.1 mM 乙二胺四乙酸 (EDTA)、1 μL 1 mM ATP、1 μL 100 μM 生物素化胞苷二磷酸 (pCp-生物素)、1 μL 50 μM Mth RNA 连接酶和 6 μL DEPC 处理的 H2O(总体积为 10 μL)加入无 RNase 的薄壁 0.2 mL PCR 管中。
- 一步反应方案
- 混合 2 μL 150 μM 腺苷-5'-5'-二磷酸-{5'-(胞嘧啶-2'-O-甲基-3'-磷酸-TEG}C-生物素(AppCp-生物素)、3 μL 10x 连接酶反应缓冲液、1.5 μL 100 mM 待测序 RNA 样品储备液、3 μL 无水 DMSO 以达到 10% (v/v)、1 μL T4 RNA 连接酶(10 单位/μL)、 和 19.5 μL 经 DEPC 处理的 H2O(总体积为 30 mL),装在 1.5 mL 不含 RNase 的微量离心管中。
- 将反应物在 16 °C 下在 PCR 机器中孵育过夜。
- 如上所述在步骤 2.1.5-2.1.8 中进行色谱柱纯化。
注:为每个 RNA 样品准备一个单独的/专用反应管(150 pmol 规模的 RNA)。可能需要用磺基花青3 (Cy3) 或 Cy3 标记 RNA 的 5'-末端(例如,用于双向测序验证)。该方法与 3'-生物素化的方法不同,在之前的出版物9 中进行了描述。
3. 在链霉亲和素珠上捕获生物素化 RNA 样品
- 通过在 1.5 mL 不含 RNase 的微量离心管中加入 200 μL 1x B&W 缓冲液(5 mM Tris-HCl,pH 7.5,0.5 mM EDTA,1 M NaCl)来激活 200 μL 链霉亲和素 C1 磁珠。涡旋该溶液并将其放在磁架上 2 分钟。然后小心地吸出溶液,丢弃上清液。
- 用 200 μL 溶液 A(DEPC 处理的 0.1 M NaOH 和 DEPC 处理的 0.05 M NaCl)洗涤珠子两次,并在 200 μL 溶液 B(DEPC 处理的 0.1 M NaCl)中洗涤一次。对于每个洗涤步骤,涡旋溶液并将其放在磁架上 2 分钟,然后弃去上清液。然后加入 100 μL 的 2x B&W 缓冲液(10 mM Tris-HCl,pH 7.5,1 mM EDTA,2 M NaCl)。
- 向生物素化 RNA 样品中加入 1x B&W 缓冲液,直至体积为 100 μL。然后将此溶液添加到储存在 100 μL 2x B&W 缓冲液中的洗涤珠中。在室温下在摇床上以 100 rpm 的速度孵育 30 分钟。将试管放在磁铁架上 2 分钟,然后弃去上清液。
- 在 1x B&W 缓冲液中洗涤包被的珠子 3 次,并通过 Nanodrop 测量每个洗涤步骤中上清液的最终浓度以进行回收分析,以确认目标 RNA 分子保留在珠子上。
- 将珠子在 10 mM EDTA(pH 8.2)和 95% 甲酰胺中于 65 °C 下在 PCR 机器中孵育 5 分钟。将试管放在磁铁架上 2 分钟,然后用移液管收集上清液(包含从链霉亲和素珠子中释放的生物素化 RNA)。
注:酸降解前的这一物理分离步骤仅用于 图 1c 中 RNA#1 的测序,对于 2D-HELS MS Seq 不是必需的,因为疏水性生物素标记会导致 3' 标记的分子量标准片段在 LC-MS 测量过程中具有显著延迟的 tR ,这可以清楚地区分 2D mass-tR 图中标记的 3'-分子量标准片段和未标记的 5'-分子量标准片段。
4. RNA 酸水解生成用于测序的 MS 分子量标准
- 将每个 RNA 样品分成三个相等的等分试样。例如,将体积为 15 μL RNA 样品的 RNA 样品分成三个 5 μL 的等分试样。
- 加入等体积的甲酸,使反应混合物中含有 50% (v/v) 的甲酸 8,9。
- 在 PCR 机中于 40 °C 孵育反应,一个反应分别运行 2 分钟、一个反应 5 分钟和一个反应 15 分钟。
- 每次反应结束后,立即将样品冷冻在干冰上,以淬灭酸降解。
- 使用离心真空浓缩器干燥样品。样品通常在 30 分钟内完全干燥,并且在干燥过程中将甲酸与 H2O 一起去除,因为甲酸的沸点 (100.8 °C) 与 H2O (100 °C) 相似。
- 将总共三个干燥样品悬浮并混合在 20 μL 经 DEPC 处理的 H2O 中,用于 LC-MS 测量。
注:在此阶段,样品可以储存在 -20 °C 下,同时等待 LC-MS 测量。
5. 将 ψ 转化为 CMC ψ加合物
- 将 80 μL 经 DEPC 处理的 H2O 加入含有 0.0141 g N-环己基-Nʹ-(2-吗啉乙基)-碳二亚胺甲基对-甲苯磺酸盐 (CMC) 和 0.07 g 尿素的 1.5 mL 无 RNase 微量离心管中。加入 10 μL 100 μM 待测序 RNA 样品储备液、8 μL 1 M Bicine缓冲液 (pH 8.3) 和 1.28 μL 0.5 M EDTA。加入经 DEPC 处理的 H2O 以达到 160 μL 的总体积。最终浓度为 0.17 M CMC、7 M 尿素和 4 mM EDTA 在 50 mM 比辛 (pH 8.3) 中11。
注:该方案适用于单个合成 RNA 序列或 RNA 混合物。 - 将 160 μL 反应溶液分成四个等分试样,放入不含 RNase 的薄壁 0.2 mL PCR 管中,并在 37 °C 下在 PCR 机中孵育 20 分钟。
注:每管 50 μL 是 PCR 机中可以使用的最大反应体积。 - 用 10 μL 1.5 M 乙酸钠和 0.5 mM EDTA (pH 5.6) 淬灭每个反应。
- 根据步骤 2.1.5-2.1.8 中描述的程序,使用四个平行离心柱进行色谱柱纯化以去除过量的反应物。将纯化产物溶解在每个 1.5 mL 不含 RNase 的微量离心管中的 15 μL DEPC 处理的 H2O 中。
- 将纯化的产物转移到四个不含 RNase 的薄壁 0.2 mL PCR 管中。向每 15 μL 纯化产物中加入 20 μL 0.1 M Na2CO3 缓冲液 (pH 10.4),并加入经 DEPC 处理的 H2O,使每个反应管(共四管)的最终体积为 40 μL。将反应物在 37 °C 下在 PCR 机器中孵育 2 小时。
- 如步骤 2.1.5 所述,用四个平行离心柱进行柱纯化,淬灭和纯化反应。将 CMC-ψ 转化的产物洗脱到 1.5 mL 无 RNase 的微量离心管中,每个管用 15 μL DEPC 处理的 H2O。
- 将来自四个收集管的纯化 CMC-ψ 转化样品合并到一个管中。根据步骤 4.1-4.6 中描述的程序进行 50% (v/v) 甲酸降解,以生成用于测序的 MS 分子量标准。
6. LC-MS 测量
- 制备用于 LC-MS 测量的流动相。流动相 A 为 25 mM 六氟-2-丙醇和 10 mM 二异丙胺的 LC-MS 级水溶液;流动相 B 为甲醇。
- 将样品转移至 LC-MS 样品瓶中进行分析。每个样品进样体积为 20 μL,含有 100-400 pmol RNA。
- 使用以下液相色谱条件:柱温 35 °C,流速 0.3 mL/min;在 15 分钟内从 2% 至 20% 流动相 B 的线性梯度,然后用 90% 流动相 B 进行 2 min 洗涤步骤。
注:对于第 2 节中提到的疏水性更强的末端标记,例如 Cy3 和 sulfo-Cy3,样品洗脱可能需要更高百分比的有机溶剂(即,可以使用类似的梯度,但流动相 B 的百分比范围更大)。例如,在 30 分钟内从 2% 增加至 38% 的流动相 B,并使用 90% 流动相 B 进行 2 分钟的洗涤步骤。 - 在安捷伦 Q-TOF(四极杆飞行时间)质谱仪上分离和分析样品,该质谱仪与配备自动进样器和 MS HPLC(高效液相色谱)系统的液相色谱系统联用。液相色谱柱是 50 mm x 2.1 mm C18 色谱柱,粒径为 1.7 μm。使用以下 MS 设置:负离子模式;范围,350 m/z 至 3200 m/z;扫描速率,2 光谱/秒;干燥气体流量,17 L/min;干燥气体温度,250°C;雾化器压力,30 psig;毛细管电压,3500 V;和碎片电压,365 V。请注意,这些参数特定于所用质谱仪的类型或型号。
- 使用 Agilent MassHunter 采集软件采集数据。使用安捷伦分子特征提取 (MFE) 工作流程提取化合物信息,包括质量数、保留时间、体积(相应离子种类的 MFE 丰度)和质量评分等。 使用以下 MFE 设置:"质心数据格式,小分子(色谱),峰高度≥ 100,最大 1000,质量评分 ≥ 50"。
注:优化 MFE 设置以提取尽可能多的潜在化合物,最多 1000 种,质量评分为 ≥ 50。
7. 通过计算算法自动生成 RNA 序列
注意:此程序仅显示在 图 1c 中的 RNA #1。
- 按体积(峰强度)和 tR 的顺序对 MFE 提取的化合物进行排序。 通过以下方式 进行数据预选:1) 将 tR 设置为 4 到 10 分钟,以选择由生物素标记的 RNA 片段,因为生物素标记的质量梯组分的 tRs 转移到这个 tR 窗口(4 分钟到 10 分钟),以及 2) 使用比梯子片段数量高一个数量级的输入化合物进行算法计算,以减少基于体积的数据量。例如,对于 20 nt RNA,需要 20 个标记的 mass-tR 分子量标准组分来对 20 nt RNA 进行测序,因此,将根据体积从 MFE 数据文件中选择 200 个化合物。请注意,当使用不同类型或型号的质谱仪时,tR 窗口可能会有所不同。
- 使用已发布算法的修订版本进行 RNA #1 的数据处理和序列生成8。修订后的算法的源代码如前所述 (https://academic-oup-com.remotexs.ntu.edu.sg/nar/article/47/20/e125/5558343#supplementary-data)9。
- 除了使用该算法自动生成序列外,还可以手动计算两个相邻梯形组分之间的质量差异以进行碱基检出。RNA 中的所有碱基都可以手动调用并与 RNA 核苷酸和修饰数据库中的理论碱基匹配8;因此,可以手动准确读出 RNA 链的完整序列,用于确认算法报告的序列读取的准确性。在 RNA 修饰数据库12 中可以找到更多的 RNA 修饰结构,其相应的理论质量可通过 ChemBioDraw 获得。在表 S1–S2 中,将特定梯形组分的观测质量与其理论质量进行比较时,会显示 ppm(百万分之一)质量差异,小于 10 ppm 的值被认为与每个碱基检出都匹配良好。
8. 对 RNA 混合物进行测序
- 使用步骤 2.2 中描述的一步方案,用 A(5')pp(5')Cp-TEG-生物素标记五条 RNA 链(RNA #1 至 #5)在其 3'-末端的混合物。在总体积为 150 μL 的反应溶液中,加入 15 μL 10x T4 RNA 连接酶反应缓冲液、1.5 μL 每条 RNA 链(分别为 100 μM RNA #1 至 #5 储备液,总体积为 7.5 μL)、10 μL 150 μM A(5')pp(5')Cp-TEG-生物素、15 μL 无水 DMSO、 5 μL T4 RNA 连接酶(10 单位/μL)和 97.5 μL DEPC 处理的 H2O。将反应溶液均匀分成 5 个等分试样。每个不含 RNase 的微量离心管含有 30 μL 反应溶液。
- 将反应物在 16 °C 下在 PCR 机器中孵育过夜。
- 根据步骤 2.1.5-2.1.8 中描述的程序使用五个平行的离心柱进行柱纯化。将 3'-生物素化的 5 条 RNA 链(RNA #1 至 #5 的混合物)的混合物样品洗脱到 1.5 mL 不含 RNase 的微量离心管中,每个管含有 15 μL DEPC 处理的 H2O。
- 将来自五个收集管的纯化混合物样品合并为一个管。根据第 4 节中描述的程序进行甲酸降解。
- 如第 6 节所述,通过 LC-MS 测量样品,并使用具有优化 MFE 设置的数据分析软件分析数据,以提取包含质量、tR 和体积的数据,如步骤 6.5 中所述。由于混合导致数据复杂性显著增加,因此不应用典型的处理和碱基调用算法。混合样品的 RNA 中的所有碱基都以类似于第 7.3 节的方法手动调用,并与 RNA 核苷酸和修饰数据库8 中的理论碱基非常匹配,因此混合样品中所有五条 RNA 链的完整序列被准确读出。 表 S7–S11 列出了所有信息,包括观察到的质量数、tR、体积、质量评分和 ppm 质量差。
结果
将生物素标签引入 RNA 的 3' 端,以产生易于识别的 mass-tR 分子量标准。2D-HELS MS Seq 方法的工作流程如图 1a 所示。与未标记的对应物相比,引入 RNA 3' 端的疏水性生物素标记(参见第 2 节)增加了 3' 标记的分子量标准组分的质量和 tRs。因此,在 2D 质量-tR 图中,3' 阶梯曲线会移动到更大的 y 轴值(由于 tRs 的增加),并会移动到更大的 x 轴值(由于质量的增加)。图 1b 显示了样品制备方案,包括将生物素标签引入 RNA 的 3'-末端以进行 2D-HELS MS Seq。图 1c 显示了 3'-分子量标准与 5'-分子量标准和其他不需要的片段在 2D mass-tR 图上的分离,该图基于 RNA #1 的 3'-生物素标记的 mass-tR 分子量标准片段的 tRs 的系统变化。单独的 3'-阶梯曲线给出了 RNA #1 的完整序列,而没有显示 tR 偏移的 5'-阶梯曲线提供了相反的序列,但它需要末端配对才能读取末端碱基8。使用这种 2D-HELS 策略,不需要像以前报道的那样进行末端配对,并且只需一条标记的梯形曲线即可完全读出整个 RNA 序列8。因此,可以对含有多个 RNA 的混合样品进行测序,例如,两条不同长度的 RNA 链(分别为 RNA #1 和 RNA #2,分别为 19 nt 和 20 nt),每个 RNA 处具有 5'-生物素标记(图 1d)。
将ψ转化为其 CMC ψ加合物,用于 2D-HELS MS Seq。对于基于 MS 的测序,ψ 是一种困难的核苷酸修饰,因为它的质量与尿苷 (U) 相同。为了区分这两个碱基,我们用 CMC 处理 RNA,CMC 将 ψ 转化为 CMC ψ加合物(参见第 5 节)。加合物的质量与 U 不同,可以在 2D-HELS MS Seq 中区分。图 2a 显示了将反应ψ转化为 RNA #6 中 CMC 加合物的反应粗产物的 HPLC 曲线。通过对它们的 UV 峰进行积分,我们计算了转化率,在第 5 节所示的过程之后,42% ψ转化为其 CMC ψ加合物。在酸降解和 LC-MS 测量后,我们根据从算法处理数据中鉴定出的非 CMC 转换分子量标准和 CMC 转换分子量标准手动采集序列 8,9。由于 ψ 部分转化为 CMC ψ 加合物,从 RNA #6 中第 8 位的 ψ 开始,红色曲线从灰色曲线分支出来(图 2b)。由于 CMC 的质量和疏水性,与未转化的梯形组分相比,这种转化导致每个含有 CMC ψ加合物的分子量标准组分的质量增加 252.2076 道尔顿,tR 显著增加。因此,在 2D mass-tR 图中可以观察到从 RNA #6 中的位置 8 开始的显著变化,表明位置 8 确实是 RNA #6 中的ψ。
对 RNA 混合物进行测序。通过具有 3' 端标记的 2D-HELS MS Seq 方法对五条不同 RNA 链的混合物进行测序(参见第 8 节)。对混合 RNA 进行测序的担忧是,当 2D mass-tR 图中的多条梯形曲线共享相同的起点时(2D mass-tR 图中的疏水标签),它们可能会相互重叠。然而,碱基检出是逐个进行的,每个碱基检出都基于 MFE 数据中两个相邻梯形片段之间的质量差异。只要每个质量数差异与数据池中经典核苷酸或修饰核苷酸的理论质量数之一匹配(PPM MS 差异< 10),就可以进行正确的碱基检出 8,9。在多重 RNA 样品的分析中,图 1 和图 2 中使用的典型处理和碱基检出算法未使用,主要是因为混合物导致数据复杂性显著增加。通过计算两个相邻质量梯子片段之间的质量差,并将其与数据池中核苷酸的理论质量进行比较,手动对这些序列进行碱基检出9。任何质量 PPM <10 的匹配碱基都被选为该位置的碱基标识。通过这种逐个碱基的手动碱基检出计算,混合物中的所有序列都被准确测序。OriginLab 软件用于重建 2D mass-tR 图,其中每个序列的起始 tR 被系统地标准化,以便更好地可视化五个不同的 RNA 序列(图 3)。如果没有这种归一化,所有五个 RNA 序列的字母代码(即 A、C、G 和 U)将在图中挤在一起(图 S1),与图 3 中报告的情况相比,可视化的便利性较低。测序结果表明,2D-HELS MS Seq 方法不仅限于纯化的单链 RNA 的测序,更重要的是,还包括具有多条 RNA 链的 RNA 混合物的测序。目前正在开发算法,以自动化碱基识别和序列生成过程。
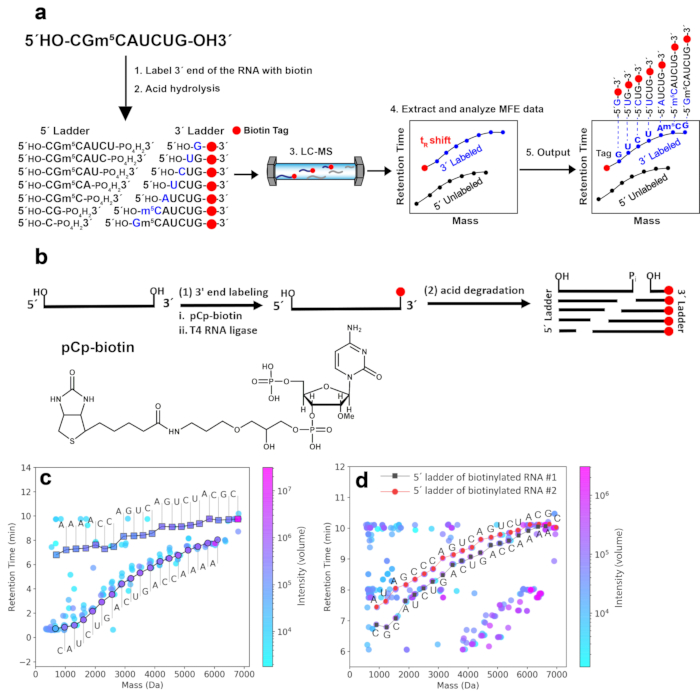
图 1.代表性 RNA 样品的 2D-HELS MS Seq。 (a) 2D-HELS MS Seq 的工作流程。主要步骤包括 1) 待测序 RNA 的疏水标签标记,2) 酸水解,3) LC-MS 测量,4) MFE 数据的提取和分析,以及 5) 通过算法或手动计算生成序列。(b) 样品制备方案,包括将生物素标签引入 RNA 的 3'-末端以进行 2D-HELS MS 测序。(c) 根据 RNA #1 (19 nt) 的 3'-生物素标记的 mass-tR ladder 片段的 tR的系统变化,在 2D 质量保留时间 (tR) 图中分离 3'-分子量标准与 5'-分子量标准和其他不需要的片段。序列是从头的,并通过碱基调用算法9 直接自动读出。(d) 5'-生物素标记的 RNA #1 和 RNA #2 同时测序,分别为 19 nt 和 20 nt。将生物素标签引入 RNA 的 5'-末端的方法与 3'-生物素化的方法不同,可以在之前发布的方案9 中找到。两个 RNA(RNA #1 和 RNA #2)的 5'-末端被生物素化,它们的 5'-生物素化分子量标准可以很容易地鉴定;在 LC-MS 后的 2D mass-tR 图中,两种 5'-生物素化分子量标准都很容易与未标记的 3'-分子量标准分离,因为生物素化分子量标准组分由于生物素的疏水性而具有较大的 tR 偏移,而未标记的分子量标准组分位于较低的 tR 区域。尽管 5'-ladders 和 3'-ladders 共存,但它们不会干扰两条混合 RNA 链的序列解释。这两种 RNA 的每个序列都是根据计算算法处理的数据从 5'-生物素化分子量标准中手动获取的 8,9。该图是从 Zhang 等人 9 修改而来的。请单击此处查看此图的较大版本。

图 2.将假尿嘧啶 (ψ) 转化为其加合物,用于 2D-HELS MS Seq。 (a) 在含有 1 ψ的 20 nt RNA (RNA #6) 中将反应ψ转化为其 CMC 加合物的反应粗产物的 HPLC 曲线。(b) 含 ψ 的 RNA #6 的测序。由于 CMC 的质量和疏水性,ψ转化为 CMC ψ 加合物 (ψ*) 导致质量增加 252.2076 道尔顿,tR 显著增加。因此,在 mass-tR 图中可以观察到从 8 位置开始的剧烈偏移,表明这是 RNA 序列中 8 位置的ψ。序列是根据计算算法处理的数据手动获取的 8,9。该图是从 Zhang 等人 9 修改而来的。请单击此处查看此图的较大版本。

图 3.对含有 5 种不同 RNA 的 RNA 混合物进行测序。生物素用于在 2D-HELS MS Seq 之前标记每个 RNA 的 3' 端。对于每个序列,将起始 tR 值系统地标准化为以 7 分钟的间隔开始,以便于可视化。对于五个 RNA 中的每一个,起始 tR 值和随后的 tRs 之间的绝对差异保持不变,因此更容易在同一图中可视化它们中的每一个。通过手动计算两个相邻分子量标准组分的质量差异并将其与 RNA 核苷酸和修饰数据库中的理论质量差异匹配来鉴定所有碱基8;图 3 的图是使用 OriginLab 根据手动碱基检出和测序数据重建的(参见代表性结果中的 RNA 混合物测序部分)。未经 tR 归一化的 5 种混合 RNA 的 2D mass-tR 图如图 S1 所示。请单击此处查看此图的较大版本。
图 S1. 请点击此处下载此文件。
讨论
与基于串联的 MS 片段化不同,2D-HELS MS Seq 方法使用高度受控的酸性水解,在使用质谱仪分析之前对 RNA 进行片段化 9,10。因此,仪器可以检测到每个酸降解片段,形成相当于测序分子量标准品。在最佳条件下,该方法平均每个分子一个位点特异性 RNA 切割仅在磷酸二酯键处从 RNA 创建一个"理想"序列梯 8,9,10。在质谱仪单次运行中测量每个降解片段后,两个相邻分子量标准片段之间的质量差对应于该位置的 RNA 核苷酸或修饰的精确质量。每种 RNA 修饰要么具有内在的独特质量,可以帮助在 RNA 中识别和定位它,要么可以转换为具有独特质量的 RNA。因此,从理论上讲,这种方法可以报告经典核苷酸和修饰核苷酸的身份和位置,用于任何 RNA 的从头和直接测序。然而,不同的序列分子量标准可能相互重叠,使 MS 数据分析复杂化,并使在实践中难以通过 MS 进行 RNA 测序。
3'-疏水标签的一个优点是它克服了任何片段化方法中的一个主要挑战, 即每个 RNA 分子必须被切割成两个片段(理想情况下不能再切割成两个片段):一个片段包含原始的 5'-末端,另一个片段包含 RNA 的原始 3'-末端。因此,每个切割事件都会产生两个片段,产生两个分子量标准——一个从 5' 端测量,另一个从 3' 端测量。在确定哪个 MS 峰属于哪个阶梯时,总是存在歧义。由于会产生大量重叠的序列分子量标准,这在几种不同 RNA 的混合物中变得更加成问题。然而,由于来自 3' 端的所有梯子片段都用疏水标签标记,因此它们表现出更长的 tRs(图 1a)。因此,我们可以在 2Dmass-t R 数据中获得清晰明确的分子量标准,这些数据仅来自 3' 标记的 RNA。值得注意的是,我们正在优化使用不同的化学偶联方法选择性标记任何 RNA 的 5' 或 3' 端的方法 9。我们还可以进行双向测序,这里不用于确定末端碱基,而是用于在从 5' 和 3'- 方向读取时提供两次相同的序列信息(即双向测序验证),从而进一步提高测序的准确性。
对于未知 RNA 样品的 从头 测序,尤其是复杂的生物样品,需要一种通用且稳定的算法来处理大量 LC-MS 数据以准确高效的方式生成序列,最近已通过其他已发表的工作获得该算法 10。尽管这些算法已用于对更复杂的样本进行测序10,但在这项研究中,除非另有说明,否则我们执行了手动碱基检出以生成序列。我们的目标是涵盖 2D-HELS MS Seq 中的所有关键步骤,并希望说明即使在不使用额外的测序算法的情况下,我们仍然可以手动读出要测序的 RNA 序列的过程。为了便于可视化并更快地识别 2D mass-tR 图中测序所需的梯形片段,除非另有说明,否则每次 LC-MS 运行的 MFE 文件在读取其序列之前,都会通过已发布算法8 的修订版本进行处理。已发布的算法不能直接用于从 LC-MS 数据中读出序列,但其部分功能仍可用于处理数据——通过该算法对质量加合物进行分层聚类将增加每个梯形组分的强度,从而降低数据复杂性,尤其是在产生序列读数的关键区域8,9.
2D-HELS MS Seq 样品制备过程中的关键步骤之一是提高 RNA 疏水标签末端标记效率。高标记效率有助于减少生成序列数据所依赖的 MS 信号所需的 RNA 样品量。为了提高标记效率,我们采用了新的标记策略,包括使用活化的 AppCp-生物素来避免在标记 RNA 的 3'-末端时进行腺苷酸化步骤。使用这种一步法,用生物素标记 19 nt RNA 的 3'-末端的反应产量(参见步骤 2.2)可以从 60% 提高到 ~95%99 。通过高效标记,我们能够对包含多达 12 个不同 RNA 的混合样品进行测序,如前所述9。在这项研究中,我们使用 5 种 RNA 的混合物作为代表性示例来说明测序过程。我们还检测准确测序所需的所有分子量标准片段,并读出混合物中五个 RNA 序列中每个序列的完整序列。更高的标记效率不仅有助于最大限度地减少样品上样量,还有助于显著降低下游数据分析期间的数据复杂性,以便生成序列。目前正在开发新的反应,以实现在 5' 和 3' 末端标记 RNA 的定量产量。
如图 1c 所示对 RNA #1 进行测序时,使用链霉亲和素捕获和释放步骤在酸降解之前物理分离生物素化 RNA #1(参见第 3 节)。这去除了一小部分未标记的 RNA,从而更容易在 2D mass-tR 图中直观地识别标记的质量梯子。然而,物理分离步骤不是强制性的,因为与未标记的对应物相比,由于生物素标签的疏水性,生物素化的 RNA 分子量标准片段具有延迟/更长的 tRs。此外,碱基检出不依赖于物理分离,而是依赖于相邻质量阶梯组分的质量差异,因此,只要两个相邻阶梯组分的质量差异与特定核苷酸的相应质量或 RNA 核苷酸和修饰日期碱基8 的相应质量匹配,就可以实现正确的碱基检出。目前正在开发一种计算算法,用于自动化碱基识别和序列生成。
将原始 LC-MS 数据(文件类型为 .d)导出为电子表格文件时,MFE 设置对于数据处理和后续序列生成至关重要(参见第 6.5 节)。例如,我们在 100 到 1000 的范围内测试了 MFE 设置"峰随高度",并注意到设置为 100 时,设置为 100 可以为我们提供的化合物数量多 2 倍。为了避免丢失任何 ladder 组分,我们可以在测序工作流程中调整 MFE 设置。此设置可能取决于仪器质量数分辨率、质量数 ladder 碎片离子的数量和数据复杂性。此外,对于小分子,使用质心数据集和色谱类型设置也很重要。根据数据质量,质量分数可以在 50% 到 100% 之间变化。
我们在研究中使用的 LC-MS 仪器的质量分辨率上限为 ~40K,限制了该方法只能对长度小于 35 个碱基的 RNA 进行测序。但是,该方法的确切读取长度取决于仪器;具有更高分辨率的更高级仪器可能会导致更长的读长。同样,通量( 即在单次 LC-MS 运行中可以同时测序多少个 RNA 序列)仍有待探索,尽管我们手动测序了多达 12 个不同 RNA 链的 RNA 样品混合物,即使没有使用任何算法9。在当前的工作流程中,每次 LC-MS 运行需要 ~100 pmol 短 RNA (<35 nt)。当需要额外的实验时,上样量会增加:为了区分异构核苷酸修饰,通常需要高达 400 pmol 的 RNA。对于特异性 tRNA(如tRNA Phe)的测序,可能需要 ~1000 pmol 的样品进行测序和修饰分析。但是,我们预计在灵敏度更高的 LC-MS 仪器上所需的样品上样量将减少。随着样品标记效率、测序算法以及仪器灵敏度和分辨率的提高,我们预计我们的方法将适用于更广泛的 RNA 样品,尤其是那些具有各种 RNA 修饰的样品。
披露声明
作者已经申请了与本手稿中讨论的技术相关的临时专利。
致谢
作者感谢美国国立卫生研究院 (1R21HG009576) 向 S. Z. 和 WL 提供的 R21 赠款,以及纽约理工学院 (NYIT) 对研究和创造力的机构支持向 S. Z. 提供的赠款,该赠款支持了这项工作。作者感谢博士生 Xuanting Wang(哥伦比亚大学)对图表制作的帮助,并感谢 Michael Hadjiargyrou 教授(纽约理工学院)、Jingyue Ju 教授(哥伦比亚大学)、James Russo 博士、Shiv Kumar、Xiaoxu Li、Steffen Jockusch 博士以及 Ju 实验室的其他成员(哥伦比亚大学)、Yongdong Wang 博士(Cerno Bioscience)、Meina Aziz (NYIT) 和 Wenhao Ni (NYIT) 为我们的手稿提供了有益的讨论和建议。
材料
| Name | Company | Catalog Number | Comments |
| 5' DNA Adenylation kit | New England Biolabs | E2610S | 50uM concentration |
| 6550 Q-TOF mass spectrometer | Agilent Technologies | 5991-2116EN | Coupled to a 1290 Infinity LC system |
| A(5´)pp(5´)Cp-TEG-biotin-3´ | ChemGenes | 91718 | HPLC purified |
| ATPγS | Sigma-Aldrich | 11162306001 | Lithium salt |
| Bicine | Sigma-Aldrich | B8660 | BioXtra, ≥99% (titration) |
| Biotin maleimide | Vector Laboratories | SP-1501 | Long arm |
| C18 column | Waters | 186003532 | 50 mm × 2.1 mm Xbridge C18 column with a particle size of 1.7 μm |
| Centrifugal Vacuum Concentrator | Labconco | Refrig 115v/60hz 7310022 | Labconco CentriVap |
| ChemBioDraw | PerkinElmer | ChemDraw Prime | Generate a chemical structure and property data of structures & fragments |
| CMC (N-cyclohexyl-N?-(2-morpholinoethyl)-carbodiimide metho-p-toluenesulfonate) | Sigma-Aldrich | 2491-17-0 | 95% Purifiy |
| Cyanine3 maleimide (Cy3) | Lumiprobe | 11080 | Water insoluble |
| DEPC-treated water | Thermo Fisher Scientific | AM9906 | Autoclaved, certified nuclease-free |
| Diisopropylamine (DIPA) | Thermo Fisher Scientific | 108-18-9 | 99% Alfa Aesar |
| DMSO | Sigma-Aldrich | 276855 | Anhydrous dimethyl sulfoxide, 99.9% |
| EDTA | Sigma-Aldrich | E6758 | Anhydrous, crystalline, BioReagent, suitable for cell culture |
| Formic acid | Merck | 64-18-6 | 98-100%, ACS reag, Ph Eur |
| Hexafluoro-2-propanol (HFIP) | Thermo Fisher Scientific | 920-66-1 | 99% Acros Organics |
| LC-MS sample vials | Thermo Fisher Scientific | C4000-11 | Plastic screw thread vials |
| LC-MS vial caps | Thermo Fisher Scientific | C5000-54A | Autosampler vial screw thread caps |
| Na2CO3 buffer | Sigma-Aldrich | 88975 | BioUltra, >0.1 M Na2CO3, >0.2 M NaHCO3 |
| Oligo Clean & Concentrator | Zymo Research | D4060 | Spin column |
| OriginLab | OriginLab | OriginPro | Data analysis and graphing software |
| pCp-biotin | TriLink BioTechnologies | NU-1706-BIO | 20 ul (1 mM) |
| RNA #1--#6 | Integrated DNA Technologies | Custom RNA oligos | 19nt-21nt single-stranded RNAs, used without further purification |
| Rocking platform shaker | VWR | Orbital Shaker Standard 1000 | Speed Range 40 to 300 rpm |
| Streptavidin magnetic beads | Thermo Fisher Scientific | 88816 | Binding approx. 55ug biotinylated rabbit lgG per mg of beads |
| Sulfonated Cyanine3 maleimide | Lumiprobe | 11380 | Water soluble |
| T4 DNA ligase 1 | New England Biolabs | M0202S | 400 units/uL |
| T4 polynucleotide kinase | Sigma-Aldrich | T4PNK-RO | From phage T4 am N81 pse T1 infected Escherichia coli BB |
| Tris-HCl buffer | Sigma-Aldrich | T6455 | Tris-HCl Buffer, pH 10, 10×, Antigen Retriever |
| Urea | Sigma-Aldrich | 81871 | Urea for synthesis. CAS No. 57-13-6, EC Number 200-315-5. |
参考文献
- Addepalli, B., Venus, S., Thakur, P., Limbach, P. A. Novel ribonuclease activity of cusativin from Cucumis sativus for mapping nucleoside modifications in RNA. Analytical and Bioanalytical Chemistry. 409 (24), 5645-5654 (2017).
- Gao, H., Liu, Y., Rumley, M., Yuan, H., Mao, B. Sequence confirmation of chemically modified RNAs using exonuclease digestion and matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 23 (21), 3423-3430 (2009).
- McLuckey, S. A., Van Berkel, G. J., Glish, G. L. Tandem mass spectrometry of small, multiply charged oligonucleotides. Journal of The American Society for Mass Spectrometry. 3 (1), 60-70 (1992).
- Fountain, K. J., Gilar, M., Gebler, J. C. Analysis of native and chemically modified oligonucleotides by tandem ion-pair reversed-phase high-performance liquid chromatography/electrospray ionization mass spectrometry. Rapid Communications in Mass Spectrometry. 17 (7), 646-653 (2003).
- Taucher, M., Breuker, K. Characterization of modified RNA by top-down mass spectrometry. Angewandte Chemie International Edition in English. 51 (45), 11289-11292 (2012).
- Kellner, S., Burhenne, J., Helm, M. Detection of RNA modifications. RNA Biology. 7 (2), 237-247 (2010).
- Thomas, B., Akoulitchev, A. V. Mass spectrometry of RNA. Trends in Biochemical Sciences. 31 (3), 173-181 (2006).
- Bjorkbom, A., et al. Bidirectional direct sequencing of noncanonical RNA by two-dimensional analysis of mass chromatograms. Journal of the American Chemical Society. 137 (45), 14430-14438 (2015).
- Zhang, N., et al. A general LC-MS-based RNA sequencing method for direct analysis of multiple-base modifications in RNA mixtures. Nucleic Acids Research. 47 (20), 125 (2019).
- Zhang, N., et al. Direct sequencing of tRNA by 2D-HELS-AA MS Seq reveals its different isoforms and dynamic base modifications. ACS Chemical Biology. 15 (6), 1464-1472 (2020).
- Bakin, A., Ofengand, J. Four newly located pseudouridylate residues in Escherichia coli 23S ribosomal RNA are all at the peptidyltransferase center: analysis by the application of a new sequencing technique. Biochemistry. 32 (37), 9754-9762 (1993).
- Cantara, W. A., et al. The RNA Modification Database, RNAMDB: 2011 update. Nucleic Acids Research. 39 (Database issue), D195-D201 (2011).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。