Method Article
2D-HELS MS 염기서열분석(2D-HELS MS SEQ): 뉴클레오티드 변형이 다른 RNA 혼합물의 직접 및 새로운 염기서열분석을 위한 일반적인 LC-MS 기반 방법
요약
여기에서는 cDNA 중간체 없이 짧은 RNA(실행당 <35nt)를 염기서열분석하는 직접 방법과 단일 염기 정밀도에서 단일 연구에서 다른 뉴클레오티드 변형을 염기서열분석하는 일반적인 방법으로 사용할 수 있는 LC-MS 기반 염기서열분석 방법에 대한 자세한 프로토콜에 대해 설명합니다.
초록
질량분석법(MS) 기반 염기서열분석법(sequencing approach)은 상보적 DNA(cDNA) 중간체 없이 RNA를 직접 염기서열분석(sequencing)하는 데 유용한 것으로 나타났습니다. 그러나 이러한 접근법은 de novo RNA 염기서열분석 방법으로 적용되는 경우는 거의 없으며, 주로 정제된 단일 가닥 RNA 샘플의 알려진 염기서열을 확인하기 위한 품질 보증을 지원할 수 있는 도구로 사용됩니다. 최근에는 2차원 질량 머무름 시간 소수성 말단 라벨링 전략을 MS 기반 염기서열분석(2D-HELS MS Seq)에 통합하여 직접 RNA 염기서열분석 방법을 개발했습니다. 이 방법은 단일 RNA 염기서열뿐만 아니라 최대 12개의 개별 RNA 염기서열을 포함하는 혼합물을 정확하게 염기서열분석할 수 있습니다. 4개의 표준 리보뉴클레오티드(A, C, G 및 U) 외에도 이 방법은 변형된 뉴클레오티드를 포함하는 RNA 올리고뉴클레오티드를 염기서열분석할 수 있는 능력을 가지고 있습니다. 이는 변형된 핵염기가 RNA 염기서열에서의 식별 및 위치에 도움이 될 수 있는 본질적으로 고유한 질량을 갖거나 고유한 질량을 가진 산물로 전환될 수 있기 때문에 가능합니다. 이 연구에서는 두 가지 대표적인 변형 뉴클레오티드(슈도우리딘(Ψ) 및 5-메틸시토신(m5C))을 통합한 RNA를 사용하여 각각 다른 염기서열 및/또는 변형된 뉴클레오티드를 가진 단일 RNA 올리고뉴클레오티드와 RNA 올리고뉴클레오티드의 혼합물의 새로운 염기 서열분석에 대한 방법의 적용을 설명했습니다. 이러한 모델 RNA의 염기서열분석을 위해 여기에 설명된 절차 및 프로토콜은 표준 고분해능 LC-MS 시스템을 사용할 때 다른 짧은 RNA 샘플(<35 nt)에 적용할 수 있으며, 변형된 치료용 RNA 올리고뉴클레오티드의 염기서열 검증에도 사용할 수 있습니다. 미래에는 더 강력한 알고리즘과 더 나은 기기가 개발됨에 따라 이 방법을 통해 더 복잡한 생물학적 샘플의 염기서열분석을 수행할 수 있을 것입니다.
서문
RNA의 직접 염기서열분석을 위해 하향식 MS 및 탠덤 MS 1,2,3,4를 포함한 질량분석법(MS) 기반 염기서열분석법이 개발되었습니다. 그러나 질량분석기에서 고품질 RNA ladder를 효과적으로 생성하기 위한 현장 단편화 기술은 현재 de novo sequencing 5,6에 적용할 수 없습니다. 또한, 정제된 RNA 염기서열서열(1)을 하나라도 분석하기 위해 기존의 1차원(1D) MS 데이터를 분석하는 것은 그리 간단하지 않으며, 혼합된 RNA 샘플의 MS 염기서열분석은 훨씬 더 어려울 수 있습니다 7,8. 따라서 2차원(2D) 액체 크로마토그래피(LC)-MS 기반 RNA 염기서열분석 방법이 개발되어 1D 질량 래더를 대체하기 위해 2D 질량 보유 시간(tR) 래더 생산을 통합하여 RNA8의 새로운 염기서열분석에 필요한 래더 구성 요소를 훨씬 쉽게 식별할 수 있습니다. 그러나 2D LC-MS 기반 RNA 염기서열분석 방법은 하나의 단일 ladder만으로는 완전한 염기서열을 판독할 수 없고 두 개의 공존하는 인접한 ladder(5'- 및 3'-ladder)8에 의존해야 하기 때문에 주로 정제된 합성 짧은 RNA에 국한됩니다. 보다 구체적으로, 이 접근 방식은 저질량 영역8에서 말단 핵염기를 판독하기 위해 양방향 쌍단 판독을 필요로 합니다. paired-end reading의 복잡성이 추가됨에 따라 이 방법은 알려지지 않은 샘플에 대해 어떤 ladder fragment가 어떤 ladder에 속하는지에 대한 혼동이 발생하기 때문에 RNA 혼합물의 sequencing에 사용할 수 없습니다.
위에서 언급한 MS 기반 RNA 염기서열분석 접근법의 장벽을 극복하고 직접 RNA 염기서열분석에서 이러한 응용 분야를 넓히기 위해서는 1) RNA 가닥의 첫 번째 뉴클레오타이드에서 마지막 뉴클레오타이드까지 완전한 염기서열을 판독하는 데 사용할 수 있는 고품질 질량 래더를 생성하는 방법과 2) 복잡한 MS 데이터 세트에서 각 RNA/질량 래더를 효과적으로 식별하는 방법의 두 가지 문제를 해결해야 합니다. 잘 제어된 산 분해와 함께 MS 기반 염기서열분석 기술에 소수성 말단 라벨링 전략(HELS)을 도입하여 새로운 염기서열분석 방법을 개발했으며, 염기서열분석 9를 수행할 RNA의 5' 및/또는 3' 말단에 소수성 태그를 추가하여 이 두 가지 문제를 성공적으로 해결했습니다. 이 방법은 RNA로부터 "이상적인" 서열 사다리를 생성하며, 각 사다리 단편은 각 포스포다이에스테르 결합에서 독점적으로 부위 특이적 RNA 절단에서 파생되며, 인접한 두 사다리 단편 사이의 질량 차이는 해당 위치에서 뉴클레오티드 또는 뉴클레오티드 변형의 정확한 질량입니다 8,9,10. 이는 기기에 주입하기 전에 분자당 평균적으로 한 번씩 RNA를 단편화하는 고도로 제어된 산성 가수분해 단계를 포함하기 때문에 가능합니다. 결과적으로, 각 분해 단편 생성물은 질량 분석기에서 검출되고 모든 단편은 함께 시퀀싱 래더 8,9,10을 형성합니다. 이 새로운 전략은 RNA의 다른 사다리에서 쌍을 이루는 말단 판독 없이 RNA 가닥의 한 단일 사다리에서 RNA 염기서열을 완전히 판독할 수 있게 하며, 추가로 조합 뉴클레오티드 변형을 포함하는 여러 다른 가닥을 가진 RNA 혼합물의 MS 염기서열분석을 가능하게 합니다9. RNA의 5' 및/또는 3' 말단에 태그를 추가함으로써 표지된 ladder fragments는 tR의 상당한 지연을 나타내며, 이는 두 개의 mass ladder를 서로 구별하고 잡음이 있는 low-mass region과도 구별하는 데 도움이 될 수 있습니다. 소수성 태그 추가로 인한 mass-tR shift는 질량 ladder 식별을 용이하게 하고 염기서열 생성을 위한 데이터 분석을 단순화합니다. 또한, 소수성 태그를 추가하면 태그로 인한 질량 및 소수성 증가로 인해 해당 사다리 단편이 시끄러운 저질량-tR 영역에 있는 것을 방지하여 가닥의 말단 염기를 식별하는 데 도움이 될 수 있으며, 따라서 단일 사다리에서 RNA의 전체 서열을 식별할 수 있습니다. 쌍단 읽기가 필요하지 않습니다. 그 결과, 당사는 고급 염기서열분석 알고리즘9을 사용하지 않고도 최대 12개의 개별 RNA 가닥의 복잡한 혼합물에 대한 성공적인 염기서열분석을 입증했으며, 이는 표준 뉴클레오티드와 변형된 뉴클레오티드를 모두 포함하는 RNA의 새로운 MS 염기서열분석의 문을 열고 혼합된 더 복잡한 RNA 샘플의 염기서열분석을 보다 실현 가능하게 합니다. 실제로, 2D-HELS MS Seq를 사용하여 혼합된 tRNA 샘플10 집단을 성공적으로 시퀀싱했으며 다른 복잡한 RNA 샘플로 적용을 적극적으로 확장하고 있습니다.
2D-HELS MS Seq가 더 광범위한 RNA 샘플의 염기서열분석을 용이하게 하기 위해 여기에서는 이 염기서열분석 접근법의 기술적 측면에 초점을 맞추고 RNA 샘플의 직접 염기서열분석에 기술을 적용할 때 필요한 모든 필수 단계를 다룰 것입니다. 구체적인 예는 합성 단일 RNA 서열, 여러 개의 별개의 RNA 서열의 혼합물 및 슈도우리딘(ψ) 및 5-메틸시토신(m5C)과 같은 표준 및 변형된 뉴클레오티드를 모두 포함하는 변형된 RNA를 포함하는 염기서열분석 기술을 설명하는 데 사용됩니다. RNA는 모두 포스포다이에스테르 결합을 포함하고 있기 때문에 모든 유형의 RNA를 산 가수분해하여 최적의 조건 8,9에서 2D-HELS MS Seq에 대한 이상적인 서열 사다리를 생성할 수 있습니다. 그러나 주어진 RNA의 모든 ladder fragments의 검출은 기기에 따라 다릅니다. 표준 고분해능 LC-MS(40K)에서 정제된 짧은 RNA 샘플(<35nt)의 염기서열분석을 위한 최소 로딩량은 실행당 100pmol입니다. 그러나 추가 실험을 수행해야 하는 경우(예: 동일한 질량을 공유하는 이성질체 염기 변형을 구별하기 위해) 더 많은 재료가 필요합니다(RNA 샘플당 최대 400pmol). 모델 합성 변형 RNA의 염기서열분석에 사용되는 프로토콜은 염기 변형이 알려지지 않은 생물학적 RNA 샘플을 포함하여 더 광범위한 RNA 샘플의 염기서열분석에도 적용할 수 있습니다. 그러나 모든 변형이 포함된 완전한 tRNA의 염기서열분석을 위해서는 표준 LC-MS 기기를 사용하여 tRNA 염기서열분석을 위한 1000 pmol(~76 nt)과 같은 훨씬 더 많은 샘플이 필요하며, de novo 염기서열분석10을 위해 고급 알고리즘을 개발해야 합니다.
프로토콜
1. RNA 올리고뉴클레오티드 설계
- 표준 뉴클레오티드와 변형 뉴클레오티드를 모두 가진 하나(RNA #6)를 포함하여 다양한 길이(19 nt, 20 nt 및 21 nt)의 합성 RNA 올리고뉴클레오티드를 설계합니다. ψ는 비질량 변형 변형에 대한 모델로 사용되며, 이는 U. m5C와 동일한 질량을 갖기 때문에 MS 염기서열 분석에 어려움이 있습니다.
RNA #1: 5'-HO-CGCAUCUGACUGACCAAAA-OH-3'
RNA #2: 5'-HO-AUAGCCCAGUCAGUCUACGC-OH-3'
RNA #3: 5'-호-아아아구아카우아쿠가그-오-3'
RNA #4: 5'-HO-GCGUACAUCUUCCCCUUUAU-OH-3'
RNA #5: 5'-HO-GCGGAUUUAGCUCAGUUGGGA-OH-3'
RNA #6: 5'-HO-AAACCGUψACCAUUAm5CUGAG-OH-3'
- 각 합성 RNA를 뉴클레아제가 없는 디에틸 피로카보네이트(DEPC) 처리된 물(달리 표시되지 않는 한 DEPC 처리된H2O로 표현됨)에 용해시켜 100mM RNA 원액을 얻습니다. 원액은 -20°C에서 장기간 보관됩니다.
- RNA 시료 분해 가능성을 방지하려면 DEPC 처리된 물, 미세 원심분리기 튜브 및 피펫 팁을 포함한 RNase가 없는 실험 용품을 사용하십시오. RNase 제거 물티슈를 사용하여 실험실 소모품의 표면을 자주 닦습니다.
2. RNA의 3'-말단에 비오틴을 표시하십시오.
- 2단계 반응 프로토콜(아데닐화 및 결찰)
- 50mM 아세테이트나트륨, pH 6.0, 10mM MgCl2, 5mM 디클로로디페닐트리클로로에탄(DTT), 0.1mM 에틸렌디아민테트라아세트산(EDTA), 1mM ATP 1μL, 100μM 비오틴화 시티딘 비스포스페이트(pCp-biotin) 1μL, 50μM Mth RNA 리가제 1μL, DEPC 처리된 H2O6μL(총 부피 10μL)를 포함하는 10x 아데닐화 반응 완충액 1μL를 RNase가 없는 얇은 벽 0.2mL PCR 튜브에 추가합니다.
참고: 2단계 반응 전에 시약을 -20°C에서 보관하십시오. 시약을 실온에서 해동하고 반응에 추가하기 전에 볼텍싱과 원심분리로 잘 혼합합니다. - PCR 기계에서 65°C에서 1시간 동안 반응을 배양하고 85°C에서 5분 동안 반응을 비활성화합니다.
- 염기서열분석 대상 RNA의 100mM 샘플 스톡의 50 mM 트리스(하이드록시메틸)아미노메탄(트리스)-HCl을 포함하는 3 μL의 10x T4 RNA 리가제 반응 완충액 3 μL를 첨가하여 이전 단계의 반응 용액 10 μL를 함유하는 RNase-free, 얇은 벽의 0.2 mL PCR 튜브에서 결찰 단계를 수행하고, 10%(v/v)에 도달하기 위한 3μL의 무수 디메틸 설폭사이드(DMSO), 1μL의 T4 RNA 리가제(10단위/μL) 및 11.5μL의 DEPC 처리된 H2O(총 부피 30mL)에 도달합니다. PCR 기계에서 16°C에서 밤새 반응을 배양합니다.
참고: DMSO의 높은 어는점(18.45 °C)으로 인해 실온에서 반응 성분을 결합합니다. - 16 °C에서 밤새 반응을 배양합니다.
- Oligo Clean & Concentrator를 사용하여 효소를 제거하고 pCp-biotin을 유리시키기 위해 컬럼 정제를 통해 반응을 퀀칭 및 정제합니다(Zymo Research, Irvine, CA, USA). Oligo Binding Buffer, DNA Wash Buffer, spin columns 및 collection tube가 키트에 포함되어 있습니다. 결합 완충액을 추가하기 전에 20mL의 DEPC 처리된 H2O를 반응 용액에 추가하여 50mL 샘플 부피에 도달합니다.
- 각 반응 용액에 결합 완충액 100mL를 추가합니다. 400μL의 에탄올을 첨가하고 피펫팅으로 혼합한 후 혼합물을 컬럼으로 옮깁니다. 10,000 x g 에서 30초 동안 원심분리기. 플로우 스루를 폐기합니다.
- 750μL의 DNA 세척 완충액을 컬럼에 추가합니다. 10,000 x g 의 원심분리기, 최대 속도 각각 30초 및 1분.
- 컬럼을 1.5mL 마이크로 원심분리 튜브로 옮깁니다. DEPC 처리된 H2O 15μL를 컬럼에 추가하고 10,000 x g 에서 30초 동안 원심분리하여 RNA 산물을 용리합니다.
참고: 샘플은 다음 단계가 수행될 때까지 이 단계에서 -20°C에서 보관할 수 있습니다.
- 50mM 아세테이트나트륨, pH 6.0, 10mM MgCl2, 5mM 디클로로디페닐트리클로로에탄(DTT), 0.1mM 에틸렌디아민테트라아세트산(EDTA), 1mM ATP 1μL, 100μM 비오틴화 시티딘 비스포스페이트(pCp-biotin) 1μL, 50μM Mth RNA 리가제 1μL, DEPC 처리된 H2O6μL(총 부피 10μL)를 포함하는 10x 아데닐화 반응 완충액 1μL를 RNase가 없는 얇은 벽 0.2mL PCR 튜브에 추가합니다.
- 1단계 반응 프로토콜
- 150μM 아데노신-5'-5'-디포스페이트-{5'-(시티딘-2'-O-메틸-3'-인산염-TEG}C-비오틴(AppCp-비오틴) 2μL, 10x 리가제 반응 완충액 3μL, 염기서열분석 대상 RNA의 100mM 샘플 스톡 1.5μL, 10%(v/v)에 도달하기 위한 무수 DMSO 3μL, T4 RNA 리가제(10단위/μL) 1μL를 결합하여 1단계 라벨링 반응을 수행합니다. 및 1.5mL RNase가 없는 마이크로 원심분리 튜브에서 19.5μL의 DEPC 처리된 H2O(총 부피 30mL).
- PCR 기계에서 16°C에서 밤새 반응을 배양합니다.
- 위의 2.1.5-2.1.8단계에서 설명한 대로 컬럼 정제를 수행합니다.
참고: 각 RNA 샘플(RNA의 150pmol 스케일)에 대해 분리/전용 반응 튜브를 준비합니다. RNA의 5'-말단을 sulfo-Cyanine3 (Cy3) 또는 Cy3로 표지하는 것이 필요할 수 있습니다(예: 양방향 염기서열분석 검증을 위해). 이 방법은 3'-비오틴화(3'-biotinylation)와 다르며 이전 간행물9에 설명되어 있습니다.
3. streptavidin beads에서 biotinylated RNA 시료 캡처
- 1.5 mL RNase-free 미세원심분리 튜브에 200 μL의 1x B&W 완충액(5 mM Tris-HCl, pH 7.5, 0.5 mM EDTA, 1 M NaCl)을 첨가하여 200 μL의 streptavidin C1 자석 비드를 활성화합니다. 이 용액을 소용돌이치고 자석 스탠드에 2분 동안 놓습니다. 그런 다음 용액을 조심스럽게 피펫으로 빼내어 상층액을 버리십시오.
- 200μL의 용액 A(DEPC 처리된 0.1M NaOH 및 DEPC 처리된 0.05M NaCl)로 비드를 두 번 세척하고 200μL의 용액 B(DEPC 처리된 0.1M NaCl)에서 한 번 세척합니다. 각 세척 단계에 대해 용액을 소용돌이치고 자석 스탠드에 2분 동안 놓은 다음 상등액을 버립니다. 그런 다음 100 μL의 2x B&W 버퍼(10 mM Tris-HCl, pH 7.5, 1 mM EDTA, 2 M NaCl)를 추가합니다.
- 부피가 100 μL가 될 때까지 비오틴화된 RNA 샘플에 1x B&W 완충액을 추가합니다. 그런 다음 이 용액을 100 μL의 2x B&W 완충액에 보관된 세척된 비드에 첨가합니다. 실온에서 100rpm의 흔들 플랫폼 셰이커에서 30분 동안 배양합니다. 튜브를 자석 스탠드에 2분 동안 놓고 상층액을 버립니다.
- 코팅된 비드를 1x B&W 버퍼에서 3회 세척하고 회수 분석을 위해 Nanodrop으로 각 세척 단계에서 상층액의 최종 농도를 측정하여 타겟 RNA 분자가 비드에 남아 있는지 확인합니다.
- PCR 기계에서 5분 동안 65°C에서 95% 포름아미드와 함께 10mM EDTA, pH 8.2에서 비드를 배양합니다. 튜브를 자석 스탠드에 2분 동안 유지하고 피펫으로 상층액(스트렙타비딘 비드에서 방출된 비오틴화된 RNA 포함)을 수집합니다.
참고: 산 분해 전 이 물리적 분리 단계는 그림 1c에서 RNA#1의 염기서열분석에만 사용되며, 소수성 비오틴 라벨로 인해 LC-MS측정 중에 3 ' 표지된 ladder 단편이 상당히 지연될 수 있기 때문에 2D-HELS MS 염기서열분석에는 필수가 아니며, 이는 2D mass-tR 플롯에서 표지된 3' ladder 단편과 표지되지 않은 5' ladder 단편을 명확하게 구별할 수 있습니다.
4. 염기서열분석을 위한 MS ladder를 생성하기 위한 RNA의 산 가수분해
- 각 RNA 샘플을 3개의 동일한 부분 표본으로 나눕니다. 예를 들어, 15μL RNA 샘플 부피의 RNA 샘플을 5μL의 부분 표본 3개로 나눕니다.
- 반응 혼합물 8,9에서 50%(v/v) 포름산을 얻기 위해 동일한 부피의 포름산을 첨가합니다.
- PCR 기계에서 40°C에서 반응을 각각 2분, 5분, 15분 동안 각각 한 번 배양합니다.
- 각 반응이 끝난 후 샘플을 드라이아이스에서 즉시 동결하여 산 분해를 억제합니다.
- 원심 진공 농축기를 사용하여 샘플을 건조시킵니다. 샘플은 일반적으로 30 분 이내에 완전히 건조되며, 포름산은 H2O (100 ° C)와 유사한 끓는점 (100.8 ° C)을 갖기 때문에 건조 과정에서H2O와 함께 포름산이 제거됩니다.
- LC-MS 측정을 위해 20μL의 DEPC 처리된 H2O에 총 3개의 건조된 시료를 현탁 및 결합합니다.
참고: LC-MS 측정을 기다리는 동안 이 단계에서 샘플을 -20°C에서 보관할 수 있습니다.
5. ψ CMC-ψ 부가물로 변환
- 0.0141g의 N-cyclohexyl-Nʹ-(2-morpholinoethyl)-carbodiimide metho-p-toluenesulfonate(CMC)와 0.07g의 요소가 들어 있는 1.5mL RNase가 없는 미세 원심분리 튜브에 DEPC 처리된 H2O 80μL를 추가합니다. 염기서열분석할 RNA의 100μM 샘플 스톡 10μL, 1M bicine 완충액(pH 8.3) 8μL 및 0.5M EDTA 1.28μL를 추가합니다. DEPC 처리된 H2O를 추가하여 총 부피 160 μL에 도달합니다. 최종 농도는 50 mM bicine(pH 8.3)에서 0.17 M CMC, 7 M 요소 및 4 mM EDTA입니다11.
참고: 이 프로토콜은 단일 합성 RNA 염기서열 또는 RNA 혼합물에 적용할 수 있습니다. - 160 μL 반응 용액을 RNase-free, 얇은 벽의 0.2 mL PCR 튜브에서 4개의 동일한 부분 표본으로 나누고 PCR 기계에서 37°C에서 20분 동안 배양합니다.
참고: 튜브당 50μL는 PCR 기계에서 사용할 수 있는 최대 반응량입니다. - 10 μL의 1.5 M 소듐 아세테이트와 0.5 mM EDTA(pH 5.6)로 각 반응을 담금질합니다.
- 2.1.5-2.1.8 단계에 설명된 절차에 따라 과도한 반응물을 제거하기 위해 4개의 병렬 스핀 컬럼으로 컬럼 정제를 수행합니다. 정제된 생성물을 각 1.5mL RNase가 없는 마이크로 원심분리기 튜브에 15μL의 DEPC 처리된 H2O에 용해시킵니다.
- 정제된 산물을 RNase가 없고 벽이 얇은 0.2mL PCR 튜브 4개로 옮깁니다. 각 15μL의 정제된 생성물에 20μL의 0.1M Na2CO3 버퍼(pH 10.4)를 추가하고 DEPC 처리된 H2O를 추가하여 각 반응 튜브(총 4개의 튜브)에 대해 40μL의 최종 부피를 만듭니다. PCR 기계에서 37°C에서 2시간 동안 반응을 배양합니다.
- 2.1.5단계에 설명된 대로 4개의 평행한 스핀 컬럼으로 컬럼 정제를 통해 반응을 퀀칭 및 정제합니다. CMC-ψ로 변환된 제품을 각각 15μL의 DEPC 처리된 H2O가 있는 1.5mL RNase가 없는 마이크로 원심분리 튜브로 용리합니다.
- 4개의 채취 튜브에서 정제된 CMC-ψ 변환된 시료를 하나의 튜브로 결합합니다. 4.1-4.6단계에 설명된 절차에 따라 포름산 분해 50%(v/v)를 수행하여 염기서열분석을 위한 MS ladder를 생성합니다.
6. LC-MS 측정
- LC-MS 측정을 위한 이동상을 준비합니다. 이동상 A는 LC-MS 등급의 물에 10mM 디이소프로필아민이 포함된 25mM 헥사플루오로-2-프로판올입니다. 이동상 B는 메탄올입니다.
- 분석을 위해 시료를 LC-MS 시료 바이알로 옮깁니다. 각 시료 주입량은 100-400 pmol의 RNA를 포함하는 20 μL입니다.
- 다음 LC 조건을 사용하십시오: 컬럼 온도 35°C, 유속 0.3mL/분; 15분 동안 2–20% 이동상 B에서 선형 그래디언트를 수행한 후 90% 이동상 B를 사용한 2분 세척 단계.
참고: 섹션 2에서 언급한 바와 같이 Cy3 및 sulfo-Cy3와 같은 소수성 말단 라벨의 경우 샘플 용리에 더 높은 비율의 유기 용매가 필요할 수 있습니다(즉, 유사한 그래디언트를 사용할 수 있지만 이동상 B의 백분율 범위가 증가함). 예를 들어, 30분 동안 2–38% 이동상 B, 90% 이동상 B로 2분 세척 단계. - 오토샘플러와 MS HPLC(High Performance Liquid Chromatography) 시스템이 장착된 LC 시스템과 결합된 Agilent Q-TOF(Quadrupole Time-of-Flight) 질량 분석기에서 시료를 분리하고 분석합니다. LC 컬럼은 입자 크기가 1.7μm인 50mm x 2.1mm C18 컬럼입니다. 다음 MS 설정을 사용하십시오 : 음이온 모드; 범위, 350 m/z - 3200 m/z; 스캔 속도, 2 스펙트럼 / s; 건조 가스 흐름, 17 L / min; 건조 가스 온도, 250 °C; 분무기 압력, 30psig; 모세관 전압, 3500V; 및 단편기 전압, 365V. 이러한 매개변수는 사용 중인 질량 분석기의 유형 또는 모델에 따라 다릅니다.
- Agilent MassHunter 수집 소프트웨어로 데이터를 수집하십시오. 애질런트 분자 특징 추출(MFE) 워크플로우를 사용하여 질량, 머무름 시간, 부피(각 이온 종에 대한 MFE 함량) 및 품질 점수 등을 포함한 화합물 정보를 추출합니다. 다음 MFE 설정을 사용합니다: "중심 데이터 형식, 소분자(크로마토그래피), 높이가 100≥ 피크, 최대 1000, 품질 점수 ≥ 50".
참고: MFE 설정을 최적화하여 ≥ 50의 품질 점수로 최대 1000까지 가능한 한 많은 잠재적 화합물을 추출합니다.
7. 계산 알고리즘을 통한 RNA 염기서열 생성 자동화
참고: 이 절차는 그림 1c의 RNA #1에 대해서만 표시됩니다.
- MFE에서 추출한 화합물을 부피(피크 강도)가 감소하는 순으로 정렬하고tR을 선택합니다. 1) 비오틴 표지된 질량 ladder 구성 요소의 tRs가 이 tR 창(4분에서 10분)으로 이동하기 때문에 비오틴으로 표지된 RNA 단편을 선택하기 위해t R을 4분에서 10분으로 설정하고 2) 알고리즘 계산을 위해 사다리 단편 수보다 더 높은 크기의 입력 화합물을 사용하여 부피에 따라 데이터 양을 줄이는 것을 통해 데이터 사전 선택을 수행합니다. 예를 들어, 20 nt RNA의 경우 20 nt RNA의 염기서열분석을 위해 20개의 표지된 mass-tR ladder 구성 요소가 필요하므로 MFE 데이터 파일에서 200개의 화합물이 부피에 따라 선택됩니다. tR 창은 다른 유형 또는 모델의 질량 분석기를 사용하는 경우 다를 수 있습니다.
- 발표된 알고리즘의 개정된 버전을 사용하여 RNA #1의 데이터 처리 및 서열 생성을 수행합니다8. 개정된 알고리즘의 소스 코드는 앞서 (https://academic-oup-com.remotexs.ntu.edu.sg/nar/article/47/20/e125/5558343#supplementary-data)9에 설명되어 있습니다.
- 알고리즘을 사용하여 염기서열 생성을 자동화하는 것 외에도, 염기 호출(base calling)을 위해 인접한 두 래더 구성 요소 간의 질량 차이를 수동으로 계산할 수 있습니다. RNA의 모든 염기는 수동으로 호출될 수 있으며 RNA 뉴클레오티드 및 변형 데이터베이스8의 이론적인 염기와 일치할 수 있습니다. 따라서 RNA 가닥의 전체 염기서열을 수동으로 정확하게 판독할 수 있으며, 이는 알고리즘에 의해 보고된 염기서열 판독의 정확성을 확인하는 데 사용됩니다. RNA 변형의 더 많은 구조는 RNA 변형 데이터베이스12에서 찾을 수 있으며, 이에 해당하는 이론적 질량은 ChemBioDraw에 의해 얻어집니다. 표 S1–S2에서, 관찰된 질량을 특정 래더 성분에 대한 이론적 질량과 비교할 때 ppm(백만분율) 질량 차이가 표시되며, 10ppm 미만의 값이 각 염기 호출에 대해 양호한 일치로 간주됩니다.
8. RNA 혼합물 염기서열분석
- 2.2단계에서 설명한 1단계 프로토콜을 사용하여 5개의 RNA 가닥(RNA #1 - #5)을 A(5')pp(5')Cp-TEG-비오틴으로 3' 끝에서 혼합물을 라벨링합니다. 총 부피의 150 μL 반응 용액에 15 μL의 10x T4 RNA 리가제 반응 완충액, 각 RNA 가닥 1.5 μL(총 부피 7.5 μL에 대해 각각 RNA #1 내지 #5의 100 μM 스톡), 150 μM A(5')pp(5')Cp-TEG-비오틴 10 μL, 무수 DMSO 15 μL, 5 μL의 T4 RNA 리가제(10 units/μL) 및 97.5 μL의 DEPC 처리된 H2O. 반응 용액을 5개의 부분 표본으로 균등하게 분배합니다. 각 RNase-free 마이크로 원심분리기 튜브에는 30 μL의 반응 용액이 포함되어 있습니다.
- PCR 기계에서 16°C에서 밤새 반응을 배양합니다.
- 2.1.5-2.1.8단계에 설명된 절차에 따라 5개의 병렬 스핀 컬럼을 사용하여 컬럼 정제를 수행합니다. 3'-비오틴화된 5 RNA 가닥(RNA #1 - #5 혼합물)의 혼합물 샘플을 각각 15μL의 DEPC 처리된 H2O가 있는 1.5mL RNase가 없는 미세 원심분리 튜브로 용리합니다.
- 5개의 수집 튜브에서 추출한 정제된 혼합물 샘플을 하나의 튜브로 결합합니다. 섹션 4에 설명된 절차에 따라 포름산 분해를 수행합니다.
- 섹션 6에 설명된 대로 LC-MS로 샘플을 측정하고 최적화된 MFE 설정으로 데이터 분석 소프트웨어를 사용하여 데이터를 분석하여 6.5단계에 설명된 대로 질량, tR 및 부피를 포함하는 데이터를 추출합니다. 일반적인 처리 및 기본 호출 알고리즘은 혼합으로 인해 데이터 복잡성이 크게 증가하기 때문에 적용되지 않습니다. 혼합된 샘플의 RNA에 있는 모든 염기는 섹션 7.3과 유사한 방법으로 수동으로 호출되고 RNA 뉴클레오티드 및 변형 데이터베이스8의 이론적인 염기와 잘 일치하므로 혼합 샘플에서 5개의 RNA 가닥 모두의 완전한 염기가 정확하게 판독됩니다. 표 S7–S11에는 관찰된 질량, tR, 부피, 품질 점수 및 ppm 질량 차이를 포함한 모든 정보가 나열되어 있습니다.
결과
RNA의 3'-말단에 비오틴 태그를 도입하여 쉽게 식별할 수 있는 mass-tR ladder를 생성합니다. 2D-HELS MS Seq 접근 방식의 워크플로우는 그림 1a에 나와 있습니다. RNA의 3'-말단에 도입된 소수성 비오틴 표지(섹션 2 참조)는 표지되지 않은 사다리 성분과 비교할 때 3' 표지된 사다리 성분의 질량과 tR을 증가시킵니다. 따라서 3'-래더 곡선은 2Dmass-t R 플롯에서 더 큰 y축 값(tRs의 증가로 인해)으로 이동하고 더 큰 x축 값(질량 증가로 인해)으로 이동합니다. 그림 1b는 2D-HELS MS 염기서열(Seq)에 대해 RNA의 3'-말단에 비오틴 태그를 도입하는 것을 포함한 시료 전처리 프로토콜을 보여줍니다. 그림 1c는 RNA #1의 3'-비오틴 표지 mass-tR 래더 단편의 tRs의 체계적인 변화를 기반으로 하는 2D mass-tR 플롯에서 5' 사다리 및 기타 원치 않는 단편에서 3' 사다리를 분리하는 것을 보여줍니다. 3'-ladder curve만으로도 RNA #1의 완전한 염기서열을 얻을 수 있고,t R shift를 보여주지 않는 5'-ladder curve는 역서열을 제공하지만, 말단 염기8을 판독하기 위해서는 end-pairing이 필요합니다. 이러한 2D-HELS 전략을 사용하면 이전에 보고된 바와 같이 말단 페어링이 필요하지 않으며 전체 RNA 염기서열은 단 하나의 표지된 ladder curve8에서 완전히 판독할 수 있습니다. 이와 같이, 여러 RNA를 포함하는 혼합 샘플, 예를 들어 길이가 다른 두 개의 RNA 가닥(RNA #1 및 RNA #2, 각각 19 nt 및 20 nt)을 각 RNA에서 5'-비오틴 표지로 염기서열분석하는 것이 가능합니다(그림 1d).
ψ를 2D-HELS MS 시퀀스용 CMC-ψ 부가물로 변환. ψ는 우리딘(U)과 질량이 같기 때문에 MS 기반 염기서열분석을 위한 뉴클레오티드 변형이 어렵습니다. 이 두 염기를 서로 구별하기 위해 RNA를 CMC로 처리하여 ψ를 CMC-ψ 부가물로 변환합니다(섹션 5 참조). 부가물은 U와 질량이 다르며 2D-HELS MS 염기서열에서 구별할 수 있습니다. 그림 2a는 RNA #6에서 ψ을 CMC-부가물로 변환하는 반응의 조질 생성물의 HPLC 프로파일을 보여줍니다. UV 피크를 적분하여 섹션 5에 설명된 프로세스 후에 CMC-ψ 부가물로 변환되는 42%와 ψ 백분율을 계산했습니다. 산 분해 및 LC-MS 측정 후, 알고리즘 처리 데이터 8,9에서 식별된 CMC로 변환되지 않은 래더와 CMC로 변환된 래더를 기반으로 염기서열을 수동으로 획득했습니다. 빨간색 곡선은 CMC-ψ 부가물로의 ψ의 부분 변환으로 인해 RNA #6(그림 2b)의 8번 위치에서 ψ부터 회색 곡선에서 갈라집니다. CMC의 질량 및 소수성으로 인해 이 변환은 변환되지 않은 것과 비교할 때 각 CMC-ψ 부가물 함유 사다리 구성 요소에 대해 질량이 252.2076 Dalton 증가하고 tR이 크게 증가합니다. 따라서 RNA #6의 위치 8에서 시작하는 극적인 이동은 2D mass-tR 플롯에서 관찰될 수 있으며, 이는 위치 8이 실제로 RNA #6의 ψ임을 나타냅니다.
RNA 혼합물 염기서열분석 5개의 서로 다른 RNA 가닥의 혼합물은 3' 말단 표지를 사용한 2D-HELS MS Seq 접근법에 의해 염기서열분석됩니다(섹션 8 참조). 혼합 RNA 염기서열분석에 대한 우려는 2D mass-tR 플롯의 여러 래더 곡선이 모두 동일한 시작점(2D mass-tR 플롯의 소수성 태그)을 공유할 때 서로 겹칠 수 있다는 것입니다. 그러나 기본 호출은 MFE 데이터에서 인접한 두 래더 조각 간의 질량 차이를 기반으로 하나씩 이루어집니다. 각 질량 차이가 데이터 풀에서 표준 또는 변형 뉴클레오티드의 이론적 질량 중 하나와 잘 일치하는 한(PPM MS 차이 < 10)올바른 염기 호출을 수행할 수 있습니다 8,9. 다중화된 RNA 샘플의 분석에서, 그림 1 및 2에 사용된 일반적인 처리 및 염기 호출 알고리즘은 주로 혼합물로 인한 데이터 복잡성이 크게 증가하기 때문에 사용되지 않습니다. 이러한 염기서열은 인접한 두 질량 사다리 단편 사이의 질량 차이를 계산하고 이를 데이터 풀9에 있는 뉴클레오티드의 이론적 질량과 비교함으로써 수동으로 염기 호출됩니다. 질량 PPM이 <10인 일치하는 염기가 이 위치에서 염기 항등식으로 선택됩니다. 염기 추출을 위한 이 염기별 수동 계산을 통해 혼합물의 모든 염기서열이 정확하게 염기서열 분석됩니다. OriginLab 소프트웨어는 5개의 서로 다른 RNA 염기서열을 더 잘 시각화하기 위해 각 염기서열에 대한 시작 tR이 체계적으로 정규화되는 2D 질량-tR 플롯을 재구성하는 데 사용됩니다(그림 3). 이러한 정규화가 없으면 5개 RNA 모두의 염기서열에 대한 문자 코드(즉, A, C, G, U)가 플롯에 함께 밀집되어(그림 S1) 그림 3에 보고된 것보다 시각화가 덜 쉬워집니다. 염기서열분석 결과는 2D-HELS MS 염기서열분석 접근법이 정제된 단일 가닥 RNA의 염기서열분석에만 국한되지 않고 더 중요하게는 여러 RNA 가닥과의 RNA 혼합물임을 보여줍니다. 알고리즘은 현재 base-calling 및 sequence 생성 프로세스를 자동화하기 위해 개발 중입니다.
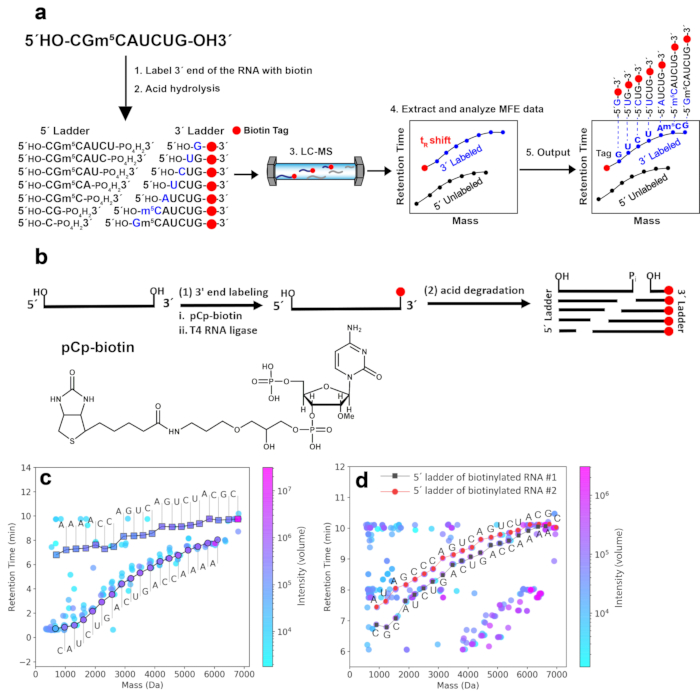
그림 1. 대표 RNA 샘플의 2D-HELS MS 염기서열. (a) 2D-HELS MS 시퀀스에 대한 워크플로우 주요 단계에는 1) 염기서열 분석할 RNA의 소수성 태그 라벨링, 2) 산 가수분해, 3) LC-MS 측정, 4) MFE 데이터 추출 및 분석, 5) 알고리즘 또는 수동 계산을 통한 염기서열 생성이 포함됩니다. (b) 2D-HELS MS 염기서열분석을 위해 RNA의 3'-말단에 비오틴 태그를 도입하는 것을 포함한 샘플 준비 프로토콜.(c) RNA #1의 3'-비오틴 표지 질량-tR 래더 단편의 tRs의 체계적인 변화를 기반으로 하는 2D 질량 보유 시간(tR) 플롯에서 5' 사다리 및 기타 원치 않는 단편에서 3' 사다리 분리 (19 nt). 시퀀스는 de novo이며 기본 호출 알고리즘9에 의해 자동으로 직접 판독됩니다. (d) 5'-비오틴 표지 RNA #1 및 RNA #2, 19 nt 및 20 nt의 동시 염기서열분석. RNA의 5'-말단에 비오틴 태그를 도입하는 방법은 3'-비오틴화(3'-biotinylation)와 다르며, 이전에 발표된 프로토콜9에서 찾을 수 있습니다. 두 RNA(RNA #1 및 RNA #2)의 5'-말단은 비오틴화되어 있으며 5'-비오틴화 사다리를 쉽게 식별할 수 있습니다. 두 5'-비오틴화된 사다리는 LC-MS 이후 2D mass-tR 플롯에서 비오틴화된 사다리 성분이 비오틴의 소수성으로 인해 더 큰 tR 이동을 갖는 반면, 비오틴화된 사다리 성분은 더 낮은 tR 영역에 있기 때문에 두 5'-비오틴화 사다리 모두 비오틴화 사다리와 쉽게 분리됩니다. 5' 사다리와 3' 사다리가 공존하지만, 두 개의 혼합된 RNA 가닥의 염기서열 해석을 방해하지 않습니다. 이 두 RNA의 각 염기서열은 계산 알고리즘으로 처리된 데이터 8,9를 기반으로 5'-biotinylated ladder에서 수동으로 획득됩니다. 이 그림은 Zhang et al.9에서 수정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2. 슈도우리딘(ψ)을 2D-HELS MS 염기서열(2D-HELS MS Seq)에 대한 부가물로 전환.(a) 하나의 ψ 포함하는 20 nt RNA (RNA #6)에서 ψ CMC 부가물로 변환하는 반응의 조질 생성물의 HPLC 프로필. (b) ψ 함유 RNA #6의 염기서열분석. ψ를 CMC-ψ 부가물(ψ*)로 변환하면 CMC의 질량 및 소수성으로 인해 질량이 252.2076 Dalton이 증가하고 tr이 크게 증가합니다. 따라서, 8의 위치에서 시작하는 극적인 이동은 mass-tR 플롯에서 관찰될 수 있으며, 이는 이것이 RNA 염기서열에서 8의 위치에서의 ψ임을 나타냅니다. 시퀀스는 계산 알고리즘으로 처리된 데이터 8,9를 기반으로 수동으로 획득됩니다. 이 그림은 Zhang et al.9에서 수정되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3. 5개의 별개의 RNA를 포함하는 RNA 혼합물 염기서열분석. 비오틴은 2D-HELS MS 염기서열분석 전에 3'-말단에서 각 RNA를 라벨링하는 데 사용됩니다. 각 시퀀스에 대해 시작 tR 값은 시각화를 용이하게 하기 위해 7분 간격으로 시작하도록 체계적으로 정규화됩니다. 시작t R 값과 후속 tRs 사이의 절대 차이는 5개의 RNA 각각에 대해 변경되지 않은 상태로 유지되므로 동일한 플롯에서 각각을 시각화하는 것이 더 쉽습니다. 모든 염기는 인접한 두 사다리 구성 요소의 질량 차이를 수동으로 계산하고 이를 RNA 뉴클레오티드 및 변형 데이터베이스8의 이론적 질량 차이와 일치시킴으로써 식별됩니다. 그림 3의 플롯은 수동 염기 추출 및 염기서열분석 데이터를 기반으로 OriginLab을 사용하여 재구성되었습니다(대표 결과의 RNA 혼합물 염기서열분석 섹션 참조). tR 정규화가 없는 5개의 혼합 RNA의 2D mass-tR 그림은 그림 S1에 나와 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
그림 S1. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
탠덤 기반 MS 단편화와 달리, 고도로 제어된 산성 가수분해는 질량 분석기 9,10로 분석하기 전에 RNA를 단편화하기 위해 2D-HELS MS Seq 접근법에 사용됩니다. 그 결과, 산으로 분해된 각 단편은 기기에 의해 검출될 수 있으며, 이는 염기서열분석 래더와 동일한 것을 형성합니다. 최적의 조건에서 이 방법은 평균적으로 포스포디에스테르 결합 8,9,10에서 독점적으로 분자당 하나의 부위 특이적 RNA 절단을 통해 RNA에서 "이상적인" 서열 래더를 생성합니다. 분해된 각 단편을 질량 분석기로 한 번의 실행으로 측정한 후, 인접한 두 사다리 단편 간의 질량 차이는 해당 위치에서 RNA 뉴클레오티드 또는 변형의 정확한 질량에 해당합니다. 각 RNA 변형은 RNA에서 이를 식별하고 찾는 데 도움이 될 수 있는 고유한 고유 질량을 가지고 있거나 고유한 질량을 가진 질량으로 변환될 수 있습니다. 따라서 이론적으로 이 방법은 모든 RNA의 de novo 및 direct sequencing을 위해 canonical 및 modified nucleotides의 정체와 위치를 보고할 수 있습니다. 그러나 서로 다른 염기서열 래더가 서로 겹칠 수 있어 MS 데이터 분석이 복잡해지고 실제로 MS에 의한 RNA 염기서열분석이 어려울 수 있습니다.
3'-소수성 태그의 이점 중 하나는 모든 단편화 방법, 즉 모든 RNA 분자가 정확히 두 개의 단편으로 절단되어야 한다는 것(이상적으로는 그 이상은 아님)을 극복한다는 것입니다. 따라서 각 분열 이벤트는 두 개의 단편을 생성하여 두 개의 사다리를 생성하는데, 하나는 5' 끝에서 측정되고 다른 하나는 3' 끝에서 측정됩니다. 어떤 MS 피크가 어떤 래더에 속하는지 결정하는 데는 항상 모호함이 있습니다. 이것은 많은 수의 중첩 서열 래더(overlapping sequence ladder)의 생성으로 인해 여러 다른 RNA의 혼합물에서 더 문제가 됩니다. 그러나 3' 말단의 모든 사다리 단편은 소수성 태그로 표시되어 있기 때문에 훨씬 더 긴 tRs를 나타냅니다(그림 1a). 그 결과, 3' 표지된 RNA에서만 독점적으로 파생된 2D mass-tR 데이터에서 명확하고 모호하지 않은 ladder를 얻을 수 있습니다. 특히, 우리는 다양한 화학적 접합 방법을 사용하여 RNA의 5' 또는 3' 말단을 선택적으로 라벨링하는 접근 방식을 최적화하고 있습니다9. 또한 양방향 염기서열분석을 수행할 수 있는데, 이는 여기에서 말단 베이스를 결정하는 데 사용되지 않지만 5'- 및 3' 방향에서 읽을 때 동일한 염기서열 정보를 두 번 제공하는 데 사용되므로(즉, 양방향 염기서열분석 검증) 따라서 염기서열분석의 정확도를 더욱 향상시킬 수 있습니다.
미지의 RNA 샘플, 특히 복잡한 생물학적 샘플의 새로운 염기서열 분석을 위해서는 정확하고 효율적인 방식으로 서열분석을 위한 많은 양의 LC-MS 데이터를 처리하기 위해 일반적이고 강력한 알고리즘이 필요하며, 이는 최근 다른 발표된 연구를 통해 사용할 수 있게 되었습니다10. 이러한 알고리즘은 더 복잡한 샘플(10)의 염기서열분석에 사용되었지만, 본 연구에서는 달리 명시되지 않는 한 염기서열 생성을 위한 수동 염기 호출을 수행하였다. 우리는 2D-HELS MS Seq의 모든 주요 단계를 다루는 것을 목표로 하며, 추가 염기서열분석 알고리즘을 사용하지 않고도 염기서열분석이 필요한 RNA의 염기서열을 수동으로 판독할 수 있는 프로세스를 설명하고자 합니다. 시각화의 용이성을 위해 그리고 2D mass-tR plot에서 염기서열분석에 필요한 ladder fragment를 보다 신속하게 식별하기 위해, 각 LC-MS 실행의 MFE 파일은 달리 명시되지 않는 한 서열을 읽기 전에 게시된 알고리즘8 의 수정된 버전에 의해 처리됩니다. 공개된 알고리즘은 LC-MS 데이터에서 염기서열을 판독하는 데 직접 사용할 수 없지만, 그 기능의 일부는 여전히 데이터를 처리하는 데 사용될 수 있습니다 - 이 알고리즘을 통해 질량 부가물을 계층적으로 클러스터링하면 각 래더 구성 요소의 강도가 증가하여 특히 염기서열 판독이 생성되는 중요한 영역에서 데이터 복잡성을 줄일 수 있습니다8, 9.
2D-HELS MS Seq에 대한 시료 전처리 중 중요한 단계 중 하나는 RNA 소수성 태그 말단 라벨링 효율성을 개선하는 것입니다. 높은 라벨링 효율은 염기서열 데이터가 의존하는 MS 신호를 생성하는 데 필요한 RNA 샘플의 양을 줄이는 데 도움이 될 수 있습니다. 라벨링 효율성을 높이기 위해 당사는 RNA의 3'-말단을 라벨링할 때 아데닐화 단계를 피하기 위해 활성화된 AppCp-biotin을 사용하는 것을 포함한 새로운 라벨링 전략을 채택합니다. 19 nt RNA의 3'-말단을 비오틴으로 표지하기 위한 반응 수율(단계 2.2 참조)은 이 1단계 방법을 사용하여 60%에서 ~95%9 로 개선할 수 있습니다. 효율적인 라벨링을 통해 앞서설명한 바와 같이 최대 12개의 서로 다른 RNA를 포함하는 혼합 샘플의 염기서열을 분석할 수 있습니다 9. 본 연구에서는 염기서열분석 과정을 설명하기 위해 5개의 RNA 혼합물을 대표적인 예로 사용합니다. 또한 정확한 염기서열분석에 필요한 모든 ladder fragment를 검출하고 혼합물에 있는 5개의 RNA 염기서열 각각의 전체 염기서열을 판독합니다. 라벨링 효율성이 높아지면 시료 로딩량을 최소화하는 데 도움이 될 뿐만 아니라 염기서열 생성을 위한 다운스트림 데이터 분석 중에 데이터 복잡성을 크게 줄이는 데도 도움이 됩니다. 현재 5'말단 및 3'말단 모두에서 RNA를 표지할 때 정량적 수율을 달성하기 위한 새로운 반응이 개발 중입니다.
그림 1c와 같이 RNA #1을 염기서열분석할 때, 스트렙타비딘 포획 및 방출 단계는 산 분해 전에 비오틴화된 RNA #1을 물리적으로 분리하는 데 사용됩니다(섹션 3 참조). 이렇게 하면 표지되지 않은 RNA의 작은 부분이 제거되고 결과적으로 2D mass-tR 플롯에서 표지된 질량 ladder를 시각적으로 더 쉽게 식별할 수 있습니다. 그러나 비오틴화된 RNA ladder 단편은 표지되지 않은 단편과 비교할 때 비오틴 태그의 소수성으로 인해 tR이 지연되거나 길어지기 때문에 물리적 분리 단계가 필수는 아닙니다. 또한, 염기 호출은 물리적 분리에 의존하지 않고 인접한 질량 ladder 구성 요소의 질량 차이에 의존하므로 인접한 두 ladder 구성 요소의 질량 차이가 RNA 뉴클레오티드 및 변형 datebase8의 특정 뉴클레오티드 또는 변형의 해당 질량과 잘 일치하는 한 올바른 염기 호출을 달성할 수 있습니다. 현재 기본 호출 및 시퀀스 생성을 자동화하기 위한 계산 알고리즘이 개발 중입니다.
원본 LC-MS 데이터를 스프레드시트 파일로 내보내는 동안(.d 파일 형식) MFE 설정은 데이터 처리 및 후속 시퀀스 생성에 매우 중요합니다(섹션 6.5 참조). 예를 들어, 100에서 1000 사이의 범위에서 MFE 설정 "높이가 있는 피크"를 테스트한 결과 100으로 설정하면 1000으로 설정하는 것보다 2배 더 많은 화합물을 제공할 수 있다는 것을 알게 되었습니다. 래더 구성 요소가 누락되지 않도록 시퀀싱 워크플로우 중에 MFE 설정을 조정할 수 있습니다. 이 설정은 기기 질량 분해능, 질량 ladder 조각의 양 및 데이터 복잡성에 따라 달라질 수 있습니다. 또한 소분자에 대해 centroid 데이터 세트 및 크로마토그래피 유형 설정을 사용하는 것이 중요합니다. 품질 점수는 데이터 품질에 따라 50%에서 100%까지 다양할 수 있습니다.
이 연구에서 사용하는 LC-MS 기기는 ~40K의 상위 질량 분해능을 가지고 있어 길이가 35개 미만인 RNA만 염기서열분석을 수행하는 것으로 제한됩니다. 그러나 이 방법의 정확한 판독 길이는 기기에 따라 다릅니다. 더 높은 분해능을 가진 더 발전된 기기는 판독 길이가 길어질 수 있습니다. 마찬가지로, 처리량 , 즉 단일 LC-MS 실행에서 얼마나 많은 RNA 염기서열을 동시에 염기서열 분석할 수 있는지는 아직 밝혀지지 않았지만, 알고리즘을 사용하지 않고도 최대 12개의 서로 다른 RNA 가닥까지 RNA 샘플 혼합물을 수동으로 염기서열 분석했습니다9. 현재 워크플로우에서는 각 LC-MS 실행에 ~100 pmol short RNA(<35 nt)가 필요합니다. 추가 실험이 필요할 때 로딩량이 증가합니다: 이성질체 뉴클레오티드 변형을 구별하기 위해서는 일반적으로 최대 400pmol의 RNA가 필요합니다. tRNAPhe와 같은 특정 tRNA를 염기서열분석하려면 염기서열분석 및 변형 분석을 위해 ~1000pmol의 샘플이 필요할 수 있습니다. 그러나 감도가 더 높은 LC-MS 기기에서 필요한 시료 로딩량이 감소할 것으로 예상됩니다. 시료 라벨링 효율성, 염기서열분석 알고리즘, 기기 감도 및 분리능이 개선됨에 따라 당사의 방법이 더 넓은 범위의 RNA 샘플, 특히 다양한 RNA 변형이 있는 샘플에 적용할 수 있을 것으로 기대합니다.
공개
저자는 이 원고에서 논의된 기술과 관련된 임시 특허를 출원했습니다.
감사의 말
저자는 미국 국립보건원(National Institutes of Health, 1R21HG009576)이 S. Z. 및 W. L.에 제공한 R21 보조금과 S. Z.에 대한 NYIT(New York Institute of Technology)의 연구 및 창의성 기관 지원(Institutional Support for Research and Creativity) 보조금이 이 작업을 지원한 것을 인정합니다. 저자는 그림 제작에 도움을 준 박사과정 학생인 Xuanting Wang(Columbia University)에게 감사의 뜻을 전하며, Michael Hadjiargyrou 교수(NYIT), Jingyue Ju 교수(Columbia University), James Russo 박사, Shiv Kumar, Xiaoxu Li, Steffen Jockusch 및 기타 Ju 연구실 구성원들, Yongdong Wang 박사(Cerno Bioscience), Meina Aziz(NYIT), Wenhao Ni(NYIT)에게 감사의 뜻을 전한다.
자료
| Name | Company | Catalog Number | Comments |
| 5' DNA Adenylation kit | New England Biolabs | E2610S | 50uM concentration |
| 6550 Q-TOF mass spectrometer | Agilent Technologies | 5991-2116EN | Coupled to a 1290 Infinity LC system |
| A(5´)pp(5´)Cp-TEG-biotin-3´ | ChemGenes | 91718 | HPLC purified |
| ATPγS | Sigma-Aldrich | 11162306001 | Lithium salt |
| Bicine | Sigma-Aldrich | B8660 | BioXtra, ≥99% (titration) |
| Biotin maleimide | Vector Laboratories | SP-1501 | Long arm |
| C18 column | Waters | 186003532 | 50 mm × 2.1 mm Xbridge C18 column with a particle size of 1.7 μm |
| Centrifugal Vacuum Concentrator | Labconco | Refrig 115v/60hz 7310022 | Labconco CentriVap |
| ChemBioDraw | PerkinElmer | ChemDraw Prime | Generate a chemical structure and property data of structures & fragments |
| CMC (N-cyclohexyl-N?-(2-morpholinoethyl)-carbodiimide metho-p-toluenesulfonate) | Sigma-Aldrich | 2491-17-0 | 95% Purifiy |
| Cyanine3 maleimide (Cy3) | Lumiprobe | 11080 | Water insoluble |
| DEPC-treated water | Thermo Fisher Scientific | AM9906 | Autoclaved, certified nuclease-free |
| Diisopropylamine (DIPA) | Thermo Fisher Scientific | 108-18-9 | 99% Alfa Aesar |
| DMSO | Sigma-Aldrich | 276855 | Anhydrous dimethyl sulfoxide, 99.9% |
| EDTA | Sigma-Aldrich | E6758 | Anhydrous, crystalline, BioReagent, suitable for cell culture |
| Formic acid | Merck | 64-18-6 | 98-100%, ACS reag, Ph Eur |
| Hexafluoro-2-propanol (HFIP) | Thermo Fisher Scientific | 920-66-1 | 99% Acros Organics |
| LC-MS sample vials | Thermo Fisher Scientific | C4000-11 | Plastic screw thread vials |
| LC-MS vial caps | Thermo Fisher Scientific | C5000-54A | Autosampler vial screw thread caps |
| Na2CO3 buffer | Sigma-Aldrich | 88975 | BioUltra, >0.1 M Na2CO3, >0.2 M NaHCO3 |
| Oligo Clean & Concentrator | Zymo Research | D4060 | Spin column |
| OriginLab | OriginLab | OriginPro | Data analysis and graphing software |
| pCp-biotin | TriLink BioTechnologies | NU-1706-BIO | 20 ul (1 mM) |
| RNA #1--#6 | Integrated DNA Technologies | Custom RNA oligos | 19nt-21nt single-stranded RNAs, used without further purification |
| Rocking platform shaker | VWR | Orbital Shaker Standard 1000 | Speed Range 40 to 300 rpm |
| Streptavidin magnetic beads | Thermo Fisher Scientific | 88816 | Binding approx. 55ug biotinylated rabbit lgG per mg of beads |
| Sulfonated Cyanine3 maleimide | Lumiprobe | 11380 | Water soluble |
| T4 DNA ligase 1 | New England Biolabs | M0202S | 400 units/uL |
| T4 polynucleotide kinase | Sigma-Aldrich | T4PNK-RO | From phage T4 am N81 pse T1 infected Escherichia coli BB |
| Tris-HCl buffer | Sigma-Aldrich | T6455 | Tris-HCl Buffer, pH 10, 10×, Antigen Retriever |
| Urea | Sigma-Aldrich | 81871 | Urea for synthesis. CAS No. 57-13-6, EC Number 200-315-5. |
참고문헌
- Addepalli, B., Venus, S., Thakur, P., Limbach, P. A. Novel ribonuclease activity of cusativin from Cucumis sativus for mapping nucleoside modifications in RNA. Analytical and Bioanalytical Chemistry. 409 (24), 5645-5654 (2017).
- Gao, H., Liu, Y., Rumley, M., Yuan, H., Mao, B. Sequence confirmation of chemically modified RNAs using exonuclease digestion and matrix-assisted laser desorption/ionization time-of-flight mass spectrometry. Rapid Communications in Mass Spectrometry. 23 (21), 3423-3430 (2009).
- McLuckey, S. A., Van Berkel, G. J., Glish, G. L. Tandem mass spectrometry of small, multiply charged oligonucleotides. Journal of The American Society for Mass Spectrometry. 3 (1), 60-70 (1992).
- Fountain, K. J., Gilar, M., Gebler, J. C. Analysis of native and chemically modified oligonucleotides by tandem ion-pair reversed-phase high-performance liquid chromatography/electrospray ionization mass spectrometry. Rapid Communications in Mass Spectrometry. 17 (7), 646-653 (2003).
- Taucher, M., Breuker, K. Characterization of modified RNA by top-down mass spectrometry. Angewandte Chemie International Edition in English. 51 (45), 11289-11292 (2012).
- Kellner, S., Burhenne, J., Helm, M. Detection of RNA modifications. RNA Biology. 7 (2), 237-247 (2010).
- Thomas, B., Akoulitchev, A. V. Mass spectrometry of RNA. Trends in Biochemical Sciences. 31 (3), 173-181 (2006).
- Bjorkbom, A., et al. Bidirectional direct sequencing of noncanonical RNA by two-dimensional analysis of mass chromatograms. Journal of the American Chemical Society. 137 (45), 14430-14438 (2015).
- Zhang, N., et al. A general LC-MS-based RNA sequencing method for direct analysis of multiple-base modifications in RNA mixtures. Nucleic Acids Research. 47 (20), 125 (2019).
- Zhang, N., et al. Direct sequencing of tRNA by 2D-HELS-AA MS Seq reveals its different isoforms and dynamic base modifications. ACS Chemical Biology. 15 (6), 1464-1472 (2020).
- Bakin, A., Ofengand, J. Four newly located pseudouridylate residues in Escherichia coli 23S ribosomal RNA are all at the peptidyltransferase center: analysis by the application of a new sequencing technique. Biochemistry. 32 (37), 9754-9762 (1993).
- Cantara, W. A., et al. The RNA Modification Database, RNAMDB: 2011 update. Nucleic Acids Research. 39 (Database issue), D195-D201 (2011).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유